Mit der Geniom-Microarray-Technologie HPV
Werbung
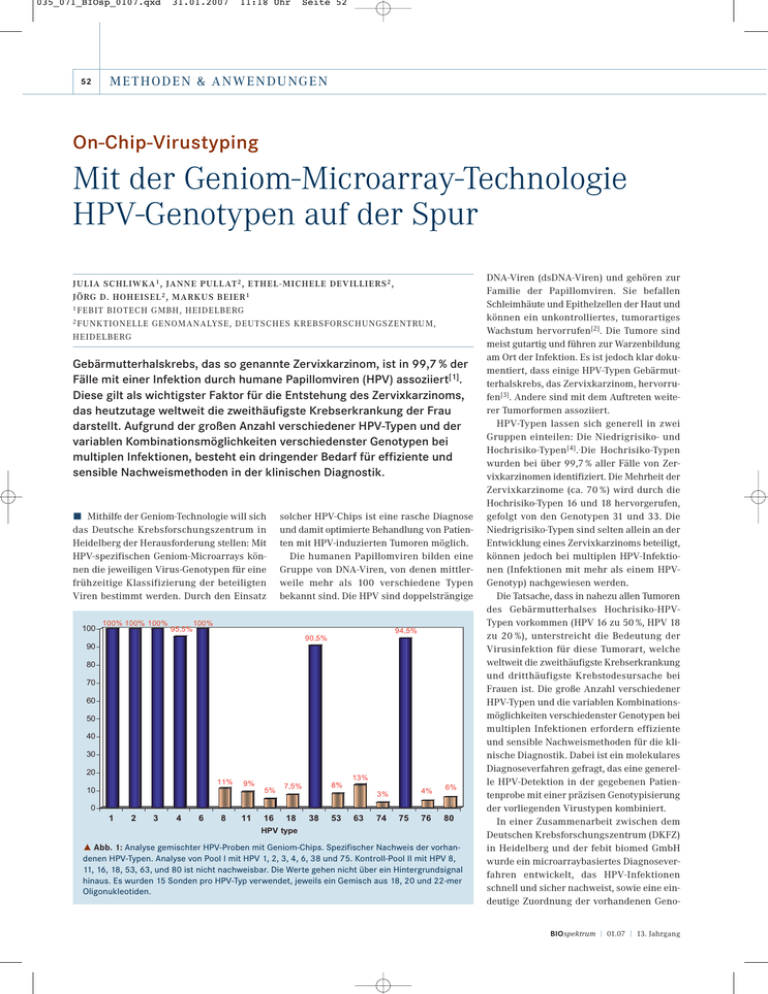
035_071_BIOsp_0107.qxd 52 31.01.2007 11:18 Uhr Seite 52 MET H ODE N & AN WE N DU NGEN On-Chip-Virustyping Mit der Geniom-Microarray-Technologie HPV-Genotypen auf der Spur JULIA SCHLIWKA 1 , JANNE PULLAT 2 , ETHEL -MICHELE DEVILLIERS 2 , JÖRG D. HOHEISEL 2 , MARKUS BEIER 1 1 FEBIT BIOTECH GMBH, HEIDELBERG 2 FUNKTIONELLE GENOMANALYSE, DEUTSCHES KREBSFORSCHUNGSZENTRUM, HEIDELBERG Gebärmutterhalskrebs, das so genannte Zervixkarzinom, ist in 99,7 % der Fälle mit einer Infektion durch humane Papillomviren (HPV) assoziiert[1]. Diese gilt als wichtigster Faktor für die Entstehung des Zervixkarzinoms, das heutzutage weltweit die zweithäufigste Krebserkrankung der Frau darstellt. Aufgrund der großen Anzahl verschiedener HPV-Typen und der variablen Kombinationsmöglichkeiten verschiedenster Genotypen bei multiplen Infektionen, besteht ein dringender Bedarf für effiziente und sensible Nachweismethoden in der klinischen Diagnostik. ó Mithilfe der Geniom-Technologie will sich das Deutsche Krebsforschungszentrum in Heidelberg der Herausforderung stellen: Mit HPV-spezifischen Geniom-Microarrays können die jeweiligen Virus-Genotypen für eine frühzeitige Klassifizierung der beteiligten Viren bestimmt werden. Durch den Einsatz 100 100% 100% 100% 95,5% solcher HPV-Chips ist eine rasche Diagnose und damit optimierte Behandlung von Patienten mit HPV-induzierten Tumoren möglich. Die humanen Papillomviren bilden eine Gruppe von DNA-Viren, von denen mittlerweile mehr als 100 verschiedene Typen bekannt sind. Die HPV sind doppelsträngige 100% 94,5% 90,5% 90 80 70 60 50 40 30 20 11% 10 9% 5% 8% 7,5% 13% 4% 3% 6% 0 1 2 3 4 6 8 11 16 18 38 53 63 74 75 76 80 HPV type ˚ Abb. 1: Analyse gemischter HPV-Proben mit Geniom-Chips. Spezifischer Nachweis der vorhandenen HPV-Typen. Analyse von Pool I mit HPV 1, 2, 3, 4, 6, 38 und 75. Kontroll-Pool II mit HPV 8, 11, 16, 18, 53, 63, und 80 ist nicht nachweisbar. Die Werte gehen nicht über ein Hintergrundsignal hinaus. Es wurden 15 Sonden pro HPV-Typ verwendet, jeweils ein Gemisch aus 18, 20 und 22-mer Oligonukleotiden. DNA-Viren (dsDNA-Viren) und gehören zur Familie der Papillomviren. Sie befallen Schleimhäute und Epithelzellen der Haut und können ein unkontrolliertes, tumorartiges Wachstum hervorrufen[2]. Die Tumore sind meist gutartig und führen zur Warzenbildung am Ort der Infektion. Es ist jedoch klar dokumentiert, dass einige HPV-Typen Gebärmutterhalskrebs, das Zervixkarzinom, hervorrufen[3]. Andere sind mit dem Auftreten weiterer Tumorformen assoziiert. HPV-Typen lassen sich generell in zwei Gruppen einteilen: Die Niedrigrisiko- und Hochrisiko-Typen[4]..Die Hochrisiko-Typen wurden bei über 99,7 % aller Fälle von Zervixkarzinomen identifiziert. Die Mehrheit der Zervixkarzinome (ca. 70 %) wird durch die Hochrisiko-Typen 16 und 18 hervorgerufen, gefolgt von den Genotypen 31 und 33. Die Niedrigrisiko-Typen sind selten allein an der Entwicklung eines Zervixkarzinoms beteiligt, können jedoch bei multiplen HPV-Infektionen (Infektionen mit mehr als einem HPVGenotyp) nachgewiesen werden. Die Tatsache, dass in nahezu allen Tumoren des Gebärmutterhalses Hochrisiko-HPVTypen vorkommen (HPV 16 zu 50 %, HPV 18 zu 20 %), unterstreicht die Bedeutung der Virusinfektion für diese Tumorart, welche weltweit die zweithäufigste Krebserkrankung und dritthäufigste Krebstodesursache bei Frauen ist. Die große Anzahl verschiedener HPV-Typen und die variablen Kombinationsmöglichkeiten verschiedenster Genotypen bei multiplen Infektionen erfordern effiziente und sensible Nachweismethoden für die klinische Diagnostik. Dabei ist ein molekulares Diagnoseverfahren gefragt, das eine generelle HPV-Detektion in der gegebenen Patientenprobe mit einer präzisen Genotypisierung der vorliegenden Virustypen kombiniert. In einer Zusammenarbeit zwischen dem Deutschen Krebsforschungszentrum (DKFZ) in Heidelberg und der febit biomed GmbH wurde ein microarraybasiertes Diagnoseverfahren entwickelt, das HPV-Infektionen schnell und sicher nachweist, sowie eine eindeutige Zuordnung der vorhandenen GenoBIOspektrum | 01.07 | 13. Jahrgang 035_071_BIOsp_0107.qxd 31.01.2007 11:18 Uhr Seite 53 53 HPV6 HP V 80 HP V 76 HP V 75 HP V 74 HP V 53 HP V 38 HP V 18 HPV74 HP V 16 HP V 11 HP V 8 HP V 6 HP V 4 HP V 3 HP V 2 HP V 1 HPV11 HP V 63 16 14 12 10 8 6 4 2 0 ˚ Abb. 2: Virus-Genotyping mit Geniom-Microarrays. Sogar stark homologe HPV-Typen werden eindeutig unterschieden. Es finden keine Kreuzhybridisierungen statt. typen ermöglicht. Ziel war es, einen spezifischen Biochip zu generieren, der die simultane Detektion von Infektionen mit jedem bislang sequenzierten HPV-Typ erlaubt. Dabei sollte eine leichte Handhabung, hohe Reproduzierbarkeit und Sensitivität gewährleistet, sowie eine Automatisierung und hoher Probendurchsatz möglich sein. Die flexible Geniom-Microarray-Plattform der febit biomed GmbH vereint die Optimierung von Design, Produktion und Analyse individueller Microarrays in einem Gerät[5, 6]. Im Falle der HPV-Genotypisierung konnten neueste Sequenzinformationen über HPVTypen herangezogen und unmittelbar für den Entwurf der entsprechenden Oligonukleotide verwendet, sowie die Sonden aufgrund erster Ergebnisse neu kombiniert und optimiert werden. Die Genome von Papillomviren sind etwa 8 kb groß und enthalten sechs frühe (early; E1 – E7) und zwei späte (late; L1 und L2) Gene. Da der L1-ORF (open reading frame, ORF) der am meisten konservierte ORF ist, erfolgen formale Klassifizierungen standardisiert durch Analysen dieser Sequenz-Region des HPV-Genoms. Für jeden HPV-Typ wurden spezifische Oligonukleotide entworfen und diese, mit dem Ziel einer gesteigerten Spezifität, experimentell optimiert. Die Flexibilität der Geniom-Plattform ermöglichte die direkte Umsetzung erzielter Ergebnisse in optimierende Maßnahmen wie BIOspektrum | 01.07 | 13. Jahrgang Änderungen der Sondensequenzen in ihren Längen oder Zusammenstellungen. Somit konnte zeiteffizient ein Biochip entwickelt werden, der für die parallele Analyse gemischter HPV-Proben herangezogen wurde. Mit 15 Sonden unterschiedlicher Länge und Spezifität pro HPV-Typ, konnten Gemische aus verschiedenen Virus-Genotypen analysiert, und die einzelnen Virustypen eindeutig nachgewiesen und bestimmt werden (Abb. 1). Für den Einsatz als molekulares Diagnostik-Werkzeug bedeutet dies eine Sensitivität und Präzision, die es erlaubt, selbst bei multiplen Virusinfektionen jeden einzelnen Virustyp zu identifizieren und klassifizieren, und damit eine sofortige Zuordnung der beteiligten Viren zur Niedrigrisiko- bzw. HochrisikoGruppe vorzunehmen. Der rasche, praktikable und sichere Nachweis in der Routinediagnostik ist besonders wichtig im Hinblick auf die Prävention von HPV-induzierten Zervixkarzinomen. Ein routinemäßiges VirusScreening ermöglicht bei Patienten mit auffälligen Befunden eine frühzeitige und gezieltere Behandlung, sodass eine eventuell drohende Entartung befallenen Gewebes verhindert werden kann. Die Spezifität der Geniom-Microarrays wird bei der Analyse stark homologer Virustypen deutlich (Abb. 2). Der Nachweis eines einzelnen spezifischen Virus in einem Virus-Gemisch erfolgt hochspezifisch, und es finden keine Kreuzhybri- disierungen mit Sonden statt, die spezifisch für andere, homologe Virustypen sind. Das DKFZ nutzt die Vorteile der Geniom-Technologie, um HPV-Chips für den routinemäßigen Einsatz in der molekularen Diagnostik zugänglich zu machen[7]. ó Literatur [1] Walboomers, J. M., Jacobs, M. V., Manos, M. M., Bosch, F. X., Kummer, J. A., Shah, K. V., Snijders, P. J., Peto, J., Meijer, C. J., Munoz, N. (1999): Human papillomavirus is a necessary cause of invasive cervical cancer worldwide. J. Pathol. 189(1): 12–19. [2] Galloway, D. A., McDougall, J. K. (1989): Human papillomaviruses and carcinomas. Adv. Virus Res. 37: 125–171. [3] zur Hausen, H. (2002): Papillomaviruses and cancer: from basic studies to clinical application. Nat. Rev. Cancer. 2(5): 342–350. [4] zur Hausen, H. (1986): Intracellular surveillance of persisting viral infections. Human genital cancer results from deficient cellular control of papillomavirus gene expression. Lancet. 2(8505): 489–491. [6] Mund, C., Beier, V., Bewerunge, P., Dahms, M., Lyko, F., Hoheisel, J. (2005): Array-based analysis of genomic DNA methylation patterns of the tumour suppressor gene p16INK4A promoter in colon carcinoma cell lines. Nucleic Acids Res. 33 (8): e73. [7] Pullat et al., zur Veröffentlichung eingereicht Korrespondenzadresse: Dr. Julia Schliwka febit biotech GmbH Im Neuenheimer Feld 519 D-69120 Heidelberg Tel.: 06221-6510-300 Fax: 06221-6510-329 [email protected] www.febit.de











