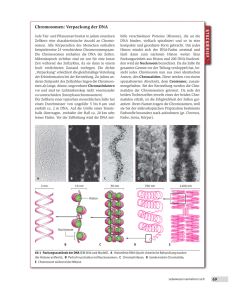
Zusammenfassung Genetik I SS 04
Werbung

zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr Zusammenfassung Genetik I SS 04 für Bioinformatiker (also nicht wirklich für Biologen etc. zur Vorbereitung geeignet – bitte Disclaimer & Copyright lesen) Def.: Genetik (griech.) „Ursprung, Entstehung, Leben“ Ziel: Grundlagen zellulärer Differenzierung als Grundlage der Entstehung morphologisch hochkomplexer Organismen zu verstehen - Abkürzungen: o Å - 1 Angstrom = 1.0 × 10-10 Meter o bp – Basenpaar/Basenpaare o Da – Dalton, Einheit der molekularen Masse o ds – doppelsträngig o kb – Kilobase: 1.000 Basen/Basenpaare o M – Molarität o o o o o o o Mb – Megabasen MG – Molekulargewicht Mol – absolute Menge einer Sobstanz OD – optische Dichte ss – einzelsträngig λ – Wellenlänge λmax – max Wellenlänge am Absorpionsmaximum - 6 Hauptrichtungen: o Klassische Genetik - Erforschung der Grundelemente der Vererbung - materieller + räumlicher Manifestation der Vererbung in der Zelle - Mechanismen der Erbmaterialverteilung bei Zellteilung o Molekulare Genetik - biochemische Grundlagen der Vererbung - molekularer Aufbau - Funktionsausübung in Zelle und Gesamtorganismus o Entwicklungsgenetik - genetische Mechanismen der Zelldifferenzierung - Entwicklung der Organismen o Verhaltensgenetik - genetischen Elemente tierischen und menschlichen Verhaltens o Neurogenetik - genetische Grundlagen der Nervensystementwicklung o Populationsgenetik - für Individuengruppen geltende genetischen Regeln - Auswirkung der Regeln auf deren Zusammenspiel - Auswirkung der Regeln auf Evolution der Organismen - Modellorganismen: o Bäckerhefe o C-Elegans o Drosophila (melanogaster) o Einige Bakterien (Escherichia coli, Pneumococcus u.a.) erstaunliche Ähnlichkeiten zwischen niederen und höheren Lebewesen - 3 essentielle Funktionen des genetischen Material (durch Mendel herausgefunden) o Replikation (genotypische Funktion) - Speicherung der genetischen Information - Übertragung von Generation zu Generation o Genexpression (phänotypische Fkt.) - Kontrolle der Entwicklung des Phänotyps eines Organismus - d.h. Wachstum und Differenzierung von Zygote bis Adulten Seite 1 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr o Mutation (evolutionäre Fkt.) - Veränderungen des gen. Material zur Organismusanpassung an Umweltmodifikationen - DNA-Grundstruktur o Unabhängigkeit von der besonderen Abfolge der einzelnen Nucleotide o Struktur an sich nicht von Bedeutung o Abfolge der Basen wichtig Codierung der Aminosäuren (AS) für Polypeptid o Struktur erlaubt Erhalt und Weitergabe ihrer Sequenz o DNA-Molekül: 2 Stränge – Korrespondenz der jeweiligen Sequenz mit der anderen - Fehlerkorrektur - nicht-kovalente Bindungen „leichte“ Trennung - genetischer Code o Beziehung zw. einer DNA-Sequenz und der Sequenz des entsprechenden Proteins - Transformation (Bsp. – Griffith) o Maus vs. Pneumococcus (eig. „Streptococcus pneumoniae“ - verursacht Pneumonie) - Virulenz durch Kapselpolysaccharid bestimmt - Stämme (I, II, III) haben glatte Oberfläche durch andere Polysaccharide (smooth – „S-Stämme“) können Stämme ohne Polysaccharid hervorbringen (raue Oberfläche – rough – „R-Stämme“) nicht tödlich, da Bakterium so zerstörbar 1) lebender S-Stamm Maus stirbt 2) lebender R-Stamm Maus gesund 3) durch Hitze abgetöteter S-Stamm Maus gesund 4) lebender R-Stamm + abgetöteter S-Stamm Maus stirbt lebende Zellen des S-Stammes aus toter Maus isoliert 1944 Nachweis durch Avery, dass DNA das transformierende Element ist - transformierende Aktivität nicht durch Hitze, Protease und RNAse zerstörbar - aber durch DNAse zerstörbar muss DNA sein o nachfolgende Überprüfung ob DNA auch in einem ganz anderen System das genetische Material darstellt o Bakteriophage T2 befällt E. coli - Markierung der T2-Phagen radioaktiv • entweder DNA mit 32P • oder Proteinhülle mit 35S - Phage und Bakterium mischen - Phage kann Bakterium infizieren - Lösen der Phagenköpfe von Bakterium mit Mixer (Schubkraft) • 32P: Bakterium radioaktiv / Phagenkopf minimal radioaktiv • 35S: Bakterium minimal radioaktiv / Phagenkopf radioaktiv - Zentrifugieren Trennung von Phagen und Bakterium • 32P: Überstand wenig radioaktiv / Zellniederschlag radioaktiv • 35S: Überstand radioaktiv / Zellniederschlag wenig radioaktiv - Bildung von „Verhältnissen“ (Ratios) o zur Erkennung von Störungen durch Verunreinigungen o mit Hilfe der optischen Dichte Absorption des Lichts bei versch. Wellenlängen o reine Proben sollten einen Quotienten (A260/A230) >2,0 haben Seite 2 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - C-Wert-Paradox o C-Wert = Gesamtmenge an DNA in einem Genom in bp; charakteristisches Merkmal einer gegeben Tier- o. Pflanzenart; steigt i a. mit zunehmendem Komplexitätsgrad o Paradoxon: Organismen mit vergleichbar komplexem Körperaufbau können manchmal drastische Unterschiede in DNA-Menge aufweisen - Amphibien-Arten mit weniger als 109 und mehr als 1011 bp - Blütenpflanzen mit etwa 5*108 bis 1011 bp o Unterschiede beruhen auf mehr oder weniger großen Anteil an DNA zw. den Genen o Lilien-Genom ist 10mal so groß, wie Menschen-Genom - chemische Grundlagen o Pyrimidin-Basen (NCNCCC) - Cytosin - Uracil - Thymin o Purin-Basen (NCNCCCNCN) - Adenin - Guanin o Desoxyribose – keine Hydroxylgruppe o Ribose o Basen kommen in der DNA verschiedener Arten in unterschiedlichen Mengen vor gen. Information - Franklin – 1952 – Röntgenstrahlen - Watson/Crick – 1953 – Doppelhelixmodell der DNA (heutige B-Form) o Regelmäßige Helix o 2 Polynucleotidketten o Purin-Pyrimidin-Paarung - DNA-Struktur o kleine Furche o große Furche o Windungen im Uhrzeigersinn - Replikation o Verdopplung des Erbgutes o 2 allgemeine Prinzipien - der Beginn der DNA-Replikation verpflichtet die Zelle zu einer weiteren Teilung - schreitet Replikation fort, so wird Teilung erst erlaubt, wenn Prozess abgeschlossen o Replicon = DNA-Einheit, an der ein einzelner Replikationsvorgang abläuft - jedes Replicon „feuert“ pro Zellzyklus ein Mal - besitzt Ursprung (origin) und Terminus - typ. Anzahl in Säugergenom: 103 bis 104 o unidirektionale Replikation o bidirektionale Replikation o semikonservativer Replikationsmechanismus - d.h. in jedem der beiden neuen DNA-Moleküle ist ein Elternstrang enthalten - (konservativ – ein neues Molekül aus 2 Tochtersträngen; dispersiv – zerstückelt) Seite 3 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - entscheidender Nachweis über E. coli • mehrere Generationen in Gegenwart von 15N wachsen lassen DNA der Bakterien somit durch zentrifugieren unterscheidbar von „normaler“ Bakterien-DNA modifizierten Bakterien in Normalmedium (14N) überführt nach genau einer Zellteilung Hybrid aus einem „leichten“ und einem „schweren“ Strang (durch Dichtemessung) o wichtigste Funktion der Doppelhelix bei Replikation: - Möglichkeit der Strangtrennung ohne kovalente Bindungen zu brechen o Ablauf: o Initiation - Trennung der beiden DNA-Stränge (in Wirklichkeit Schmelzreaktion über kurzen Bereich) • rechts in OriC binden DnaA-Proteine an Strang (vier 9bp lange Bereiche) leitet Bildung des Initationskomplexes ein • 20 – 40 DnaA-Moleküle binden noch zusätzlich OriC im DnaA-Komplex gewunden • Helicase DnaB mit DnaC bindet an DnaA links im OriC (drei 13bp) DnaC löst sich von DnaB • DnaB kann mit Entwindung des Strangs beginnen - Entwindungspunkt beginnt DNA entlang zu wandern (markiert Erzeugung einer Replikationsgabel; wandert während Elongation weiter) - Synthetisierung der ersten Nucleotide der neuen Kette zu einem Primer (nur einmal für Leistrang nötig; wiederholt sich aber zu Beginn jedes OkazakiFragments auf dem Folgestrang) • immer Synthetisierung in 5’-3’-Richtung • d.h. ein Primer auf Leitstrang an den geheftet wird • mehrere Primer auf Folgestrang, da nur Abschnittsweise Synthetisierung von Okazaki-Fragmenten in 5’-3’-Richtung - Elongation • Verlängerung der synthetisierten Kette durch Replisom mit Nucleotiden • Replisom wandert an DNA entlang, Elternstränge entwinden sich, Tochterstränge werden synthetisiert o Ablauf an Replikationsgabel: - DNA-B-Helicase bewegt sich in 5'-3'-Richtung entlang des Stranges, an den sie gebunden ist und entwindet die Doppelhelix unter Verbrauch von ATP - entstehenden Einzelstrang-Bereiche werden durch Einzelstrang-bindendeProteine (SSB-Proteine) abgedeckt - DNA-Polymerase III heftet auf dem Leitstrang (Vorwärtsstrang) neue Desoxynukleotide an das 3'-OH-Ende des wachsenden DNA-Stranges - Auf dem Folgestrang (Rückwärtsstrang) bildet die Primase kurze RNA-Stücke (Primer), welche durch die DNA-Polymerase III zu Fragmenten von 1000-2000 Nukleotiden verlängert werden. Die Primer werden von der 5'-3'-Exonuclease der Polymerase I entfernt und die entstehenden Lücken durch DNA ersetzt - DNA-Ligase verknüpft aufeinanderfolgende Okazaki-Fragmente miteinander o Termination - am Ende Trennung der verdoppelten Chromosomen o beteiligte Komponenten bei DNA-Replikation: - DNA-Polymerasen Seite 4 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - - - - - - 1. Kontrolle der Basenpaarung des eintretenden Nucleotids mit Base auf Matrizenstrang 2. Proofreading – Fehlerkorrektur und Reparatur Helicasen • trennt den von der Topo entwundenen DNA-Strang enzymatisch auf SSB (Einzelstrang-Bindeproteine) • Helix-destabilisierende Proteine, d.h. verhindert dass die Einzelstränge wieder den doppelsträngigen Zustand annehmen • binden an ss-DNA und erleichtern Arbeit der Replikationsenzyme Primosom • Proteinkomplex • beinhaltet Primasen, für (RNA-)Primer auf Folgestrang o Primer: Startstück – kurzes RNA-Stück mit einem freien 3’-OHEnde zur Elongation • initiiert auch die Synthese von Okazaki-Fragmenten DNA-Ligase • schließt Lücken der Okazaki-Fragmente • Bakterien haben 1 • Eukaryoten haben mindestens 2 versch. Topoisomerasen • Verhinderung der Überdrillung der Helix vor der Replikationsgabel • Arbeitsprinzip: o 3 neg. Überspiralisierungen Topo bindet sich Schnitt in einem DNA-Strang geschnittenes 3’-Ende wird unter anderem Strang durchgeführt DNA wird neu verknüpft Topo dissoziiert 2 neg. Überspiralisierungen Replisom • Proteinkomplex (DNA-Polymerase-III, Helicasen, Primosom) in Replikationsgabel • Entwindung und Neusynthese der DNA Telomere • verhindern Verkürzung der DNA („Endreplikationsproblem“) - Transkription o kopieren von DNA-Sequenzen in RNA durch RNA-Polymerase o Unterschiede zur Replikation: - nur einer der beiden DNA-Stränge wird in RNA umgeschrieben - nur ein kleiner Teil der genetischen Gesamtinformation wird genutzt o „antisense make sense“ - d.h. der transkribierte Strang ist der antisense-Strang (codogener) - in vitro: Gegenteil (entstehend: Antisense-RNA) o ablesen in 3’ 5’-Richtung o dabei entstehende mRNA (Uracil statt Thymin) ist sense-Strang (codierender) o Ablauf: - Initiation: RNA-Polymerase bindet an Promotor und knüpft erste NucleotidBindungen • Promotor: DNA-Bereich am Anfang des Gens; schließt das erste Basenpaar mit ein, das transkribiert wird; wird aber selbst nicht transkribiert; kein Primer notwendig! Seite 5 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr • - - Sigma-Faktor: Teil der Polymerase – reduziert Affinität des Kernenzyms zu „Nicht-Promotor“-Regionen, d.h. notwendig um gewünschtes Gen zu finden; löst sich am Ende des Initiationsstadiums ab • In Pro- und Eukaryoten sehr ähnlich, aber Eukaryoten benutzen mehr Proteine zur Promotorerkennung o Prokaryoten: Consensus-Sequenz (-35, -10 – Pribnow-Box) o Eukaryoten: TATA-Box Elongation: RNA-Polymerase läuft entlang der Matrize und synthetisiert RNA • 5’ 3’ • spannerraupen-artig o linkes Ende rutscht gleichmäßig, rechtes sprunghaft • DNA regional entwunden • Nucleotide kovalent an 3’-Ende des wachsenden RNA-Strangs angefügt Termination: Stopp bei Terminatorsequenz • Transkriptionsblase fällt in sich zusammen RNA:DNA-Hybrid wird getrennt DNA wieder Doppelhelix, RNA frei • Termination manchmal von rho-Faktor abhängig o Entstandenes Produkt: Primärtranskript - RNA vom Promotor bis Terminator - Trägt noch das ursprüngliche 5’- bzw. 3’-Ende - fast immer instabil - Prokaryoten: • entweder: rascher Abbau (mRNA) o mRNA ist dann meist polycistronisch – von einer Gruppe benachbarter Gene (Operons) wird eine einzige mRNA transkribiert • oder: Zerlegung in fertige Endprodukte (tRNA, rRNA) • während Transkription wird schon mit Translation (am 5’-Ende) begonnen • 1 Polymerase - Eukaryoten: • entweder: Modifizierung der Enden der Primärtranskripte (mRNA) o mRNA meist monocistronisch – nur ein Strukturgen o Abbau der RNA meist langsamer • oder: zurechtschneiden in fertige Endprodukte (sämtliche RNAs) RNA muss vor Translation noch prozessiert und modifiziert werden (Spleißen, Modifikation der Enden) Transkription und Translation nicht gleichzeitig • 3 Polymerasen (II: mRNA, proteincodierende Gene; III: tRNA, I: rRNA) o einige allgemeine Transkriptionsfaktoren: - Promotoren • TATA-Bindeprotein – Einleitung der Ausbildung des Initiationskomplexes • auch Initiator statt TATA-Box (in Genen für Haushalts-Proteine) - Enhancer – verstärken Transkription - Silencer – schwächen Transkription ab - Terminatoren Seite 6 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr RNA-Polymerase Kein Primer, sondern Promotor Liest begrenzten, selbst definierten Abschnitt der DNA Keine Nukleaseaktivität Endprodukt ist Einzelstrang - - DNA-Polymerase Primer Komplette Kopie des DNAStrangs entsteht Fehlererkennung und – korrektur Kompletter identischer Doppelstrang Gene als tandemartig repetitive Sequenzen o Klassisches Beispiel: rRNA-Repeats - 3 Teilgene (18S, 5,8S, 28S) werden zu Primärtranskript transkribiert zu reifer rRNA prozessiert - Trennung der codierenden Bereiche durch Spacer dann RNA-Polymerase II Transkriptionsaktivatoren o Zink-Finger-Motiv o Helix-turn-Helix-Motiv o Leucin-Motiv - Genstruktur/Expression o Exons/Introns - Exons • Sequenzen, welche die reife mRNA enthält • Sequenzen in verschied. Spezies gleich - konserviert lässt sich für Isolierung verwandter Gene bei versch. Spezies verwenden - Introns • dazwischenliegende Sequenzen, welche bei Prozessierung des Primärtranskripts zur reifen mRNA entfernt werden • keine Codierungsfunktion • Position der Introns in versch. Spezies gleich – konserviert • ABER: Sequenzen variieren o Gene sind aufgeteilt - unterbrochen - d.h. Reihenfolge der Abschnitte eines unterbrochenen Gens stimmt im Genom und gereifter mRNA überein - je höher der Eukaryot entwickelt ist, desto mehr Gene sind unterbrochen Größe eines Genes hauptsächlich durch Introns bestimmt o Regulation der Genexpression - 2 Möglichkeiten der Regulation: • Induktion – Anschalten von Genen / Synthese von Enzymen bei Auftreten eines bestimmten Substrats • Repression – Abschalten von Genen / stoppen der Synthese bei Auftreten eines bestimmten Substrats - jeweils mit Kontrollmechanismus: • positiv (Induktion) – Produkt des Regulatorgens nötig um Expression anzuschalten Seite 7 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr • negativ (Repression) – Produkt des Regulatorgens nötig um Expression abzuschalten o Lac-Operon (als Beispiel für Enzyminduktion; neg. Genregulation) - Lac-Operons enthält drei Strukturgene (Operons) lacZ, lacY und lacA - Gene tragen die Information zur Synthese von Enzymen des LactoseStoffwechsels - in Gegenwart von Lactose (Induktor) als einziger Kohlenstoffquelle nimmt Menge an Lactose-Enzymen um mehr als das 1000-fache zu • Grund: die von der Zelle aufgenommene Lactose bzw. ein Umwandlungsprodukt (Allolactose - Induktor) regt die Transkription des Lac-Operons an alle Gene werden miteinander exprimiert (oder nicht exprimiert) - Repressorprotein kontrolliert Initiierung der Transkription • Repressorprotein wird durch Regulatorgen lacI codiert Lactose induziert Bildung der Lactoseabbauenzyme - Regulation: • ist Induktors abwesend wird Gengruppe nicht transkribiert Repressor bindet an den Operator (Operator liegt zwischen Promotor und lacZ-Leseraster) Bindung des Repressors hindert RNA-Polymerase an der Initiation der Transkription am Promotor verhindert Transkription des Operons • wird Induktor hinzu gegeben, startet die Transkription Induktor (Allolactose) bindet an Repressor Komplex bindet nicht mehr an Operator RNA-Polymerase kann an DNA binden lac-mRNA wird transkribiert • Transkription stoppt, sobald der Induktor entfernt wird in sehr kurzer Zeit wird sämtliche lac-mRNA zerstört (Halbwertszeit ca. 3 Minuten) & Zelle stoppt Enzymproduktion o Katabolitrepression (bei lac-Operon; pos. Genregulation) - Glucose reprimiert la-Operon - wenn Zelle genügend Glucose (eins der Abbauprodukte von Lactose) hat, braucht sie Lactose nicht mehr umzuwandeln - CAP und CAP-Stelle beteiligt • CAP bindet nur an CAP-Stelle, wenn cAMP vorhanden ist • cAMP-Menge wird durch Glucose beinflusst o viel Glucose wenig cAMP o wenig Glucose viel cAMP - Regulation: • wenig Glucose CAP-cAMP-Komplex bildet sich bindet an CAPStelle stimuliert Bindung von RNA-Polymerase an Promotor stimuliert Transkription des lac-Operon • viel Glucose kein CAP-cAMP-Komplex CAP-Stelle unbesetzt lac-Operon nur in geringem Ausmaß transkribiert (auch wenn Lactose vorhanden und lac-Repressor nicht an Operator gebunden) Seite 8 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr o Tryptophan-Operon (als Beispiel für Enzymrepression; neg. Genregulation) - besteht aus Promotorregion, Operator, leader-attenuator-Region und 5 Strukturgenen - Trp-Repressor durch trpR-Gen codiert • Repressor inaktiv codiert (Aporepressor) • Aktivierung erst durch Bindung mit Tryptophan (Corepressor) o Bindung des Repressors an Operator nur, wenn an jedem Monomer ein Tryptophan-Molekül gebunden ist ist kein Tryptophan vorhanden, wird es durch die Zelle selbst erzeugt (alle Enzyme, die dafür nötig sind, bilden Operon und werden somit gleichzeitig synthetisiert) - Regulation - Mechanismus 1: • Tryptophan abwesend inaktiver Repressor wird nicht aktiviert kann nicht an Operator binden RNA-Polymerase kann mRNA-Molekül transkribieren, welche die 5 Gene für den Tryptophanstoffwechsel repräsentiert • Tryptophan anwesend bindet an inaktiven Repressor dieser wird aktiviert lagert sich an Operator Transkription wird unterbrochen - Regulation – Mechanismus 2 - Attenuation: • zwischen Promotor und dem ersten Gen des trp-Operons bilden sich im Transkript zwei Stamm-Schleife-Strukturen aus • die relative Lage dieser Sequenzen verhindert, dass sich beide Strukturen gleichzeitig ausbilden. • größere, stabilere Struktur hat keinen Einfluss auf die Transkription • kleinere Stamm-Schleife-Struktur (Haarnadelstruktur) ist Terminator o wenn sich diese Struktur bildet, beendet sie die Transkription, bevor das Operon erreicht ist keine Expression • ausreichend Tryptophan vorhanden Ribosom bewegt sich schnell durch die ORF- Sequenz (Sequenz ohne Terminations-Codon) verhindert Ausbildung der großen Stamm-Schleife kleine Schleife (Terminator) bildet sich Transkription kommt zum Stillstand. • zu wenig Tryptophan vorhanden Ribosom bleibt in der ORF-Sequenz stecken große Stamm-Schleife bildet sich Haarnadelstruktur kann sich nicht bilden Transkription geht weiter • Bedeutung für Mutanten: bei trp-Repressor-Mutanten ist Expession des Operons noch von Tryptophankonzentration in Zelle abhängig o Deletion des Leader führt zu stark gesteigerter Expression des Operons - Spleißen o meisten eukaryotischen Gene durch nicht-codierende Sequenzen (Introns) unterbrochen o wichtige Bereiche (Exons) müssen von Introns getrennt werden und Exons dann neu zusammengefügt werden o dafür nötig: Spleißosome – snRNA (Ribonucleoproteinkomplex: >50 Proteine und 5 kleine RNA-Moleküle) o Ablauf: o Spleißosome erkennen Consensus-Sequenzen und Intron-Exon-Übergänge o spalten Phosphodiesterbindungen auf • 2’OH-Gruppe eines Adenosins in Verzweigungsstelle aus dem Inneren des Introns greift 5’-Spleißstelle des Introns an (nucleophile Attacke) o verknüpfen Exonenden miteinander Seite 9 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr • Angriff der freien 3’OH-Gruppe des stromaufwärts liegenden Exons auf die 3’-Spleißstelle zwischen Ende des Introns und stromabwärts liegenden Exon - Translation o Übersetzung der mRNA in ein Protein o beteiligte Komponenten - Ribosomen • 2 Grundtypen o 70S – Prokaryoten o 80S – Eukaryoten • große & kleine Untereinheit • lagern sich an mRNA an Polysomen - tRNA • Adapter zwischen Aminosäure und Trinucleotid • ca. mittig auf tRNA sitzt Trinucleotid (Anticodon) ist basenkomplementär zum Codon der mRNA o wobble: erste Base in Anticodon (tRNA) manchmal durch Inosin ersetzt Redundanz des gen. Codes an dritter Position im Codon (mRNA) o (20 AS, aber 64 möglich; < 61 tRNAs) • Beladung der tRNA mit der richtigen Aminosäure o Enzym (Aminoacyl-tRNA-Synthetase) hat 2 Bindungsstellen o Bindung & Aktivierung einer bestimmten AS aktivierte AS wird an passende tRNA gebunden o (AS + ATP + tRNA AA-tRNA + AMP + PP) o einige Details zum genetischen Code - ist degeneriert, d.h. meist wird eine Aminosäure durch mehrere Codonen festgelegt - Startcodonen • Prokaryoten: AUG o. GUG • Eukaryoten: hauptsächlich AUG (selten GUG) • Erkennung des Startcodons bei Pro- und Eukaryoten unterschiedlich o Prokaryoten: Erkennungssequenz – Shine-Delgarno-Sequenz o Eukaryoten: Scanning-Mechanismus um erste AUG-Sequenz zu finden - 3 Stoppcodonen o Ablauf - Initiation • zunächst bindet kleine Untereinheit zusammen mit Initiations-tRNA an mRNA • nach erfolgreicher Erkennung des Startcodons, folgt große Untereinheit - Elongation • 2 Bereiche: o A-Stelle: für AA-tRNA o P-Stelle: für PP-tRNA (Peptidyl-tRNA) • P-Stelle schon mit Initiations-tRNA besetzt, A-Stelle noch frei Seite 10 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr 1. Bindung einer neuen AA-tRNA an A-Stelle Basenpaarbildung von Anticodon und Codon 2. Polypeptidkette von PP-tRNA abgetrennt an AA-tRNA gebunden (neue PP-tRNA) 3. Verschiebung der neuen PP-tRNA an P-Stelle (Ribosom bewegt sich dabei um 3 Nucleotide weiter) & dabei Freilassung der (alten) freien (PP-) tRNA A-Stelle wieder frei - • 1. – 3. wiederholt sich bis zum Stoppcodon • kontinuierlich in 5’ 3’-Richtung • in 3er-Gruppen • nicht überlappend, kommafrei Termination • an einem Stoppcodon • für Stoppcodon gibt es keine tRNA mit passenden Anticodon • stattdessen: Bindung von Proteinen (Release-Faktoren) Release-Faktor löst Polypeptid-Kette von der tRNA an der P-Stelle (durch Wassermolekül) mRNA und tRNA dissoziieren, Ribosom zerfällt in Untereinheiten - Zellzyklus o M-Phase - Mitose (Teilung des Zellkerns in 2 gleichartige Tochterzellen) • Prophase o Chromatin kondensiert zu Chromosomen (2 Chromatiden) o Bildung der Mitosespindeln im Cytoplasma ausgehend von den beiden Centrosomen (aus Mikrotubuli) Wanderung zu Zellpolen • Prometaphase o Auflösung der Kernmembran o Spindelfasern der Centrosomen wachsen in Kernbereich hinein verbinden sich mit Kinetochoren der Chromatiden • Metaphase o Chromosomen lagern sich senkrecht in Äquatorialebene an • Anaphase o Trennung der Chromatiden jetzt eigenständige Chromosomen o Kinetochorfasern verkürzen sich ziehen Chromosomen zu Zellpolen o Zellpole rücken auseinander • Telophase o Chromosomen erreichen Pole o Kinetochorfasern lösen sich auf o Um Chromosomen wird neue Kernhülle gebildet o Chromosomen dekondensieren - Cytokinese • Teilung des Cytoplasmas • Einschnürung der Zelle o Interphase - G1-Phase • Wachstum & Verdopplung der Zellorganellen & Differenzierung - S-Phase Seite 11 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - • Wachstum & Verdopplung der Chromosomen G2-Phase • Vorbereitung für Mitose Meiose o Meiose I (Trennung der homologen Chromosomen) - Prophase I • Chromosomen beginnen sich zu verdichten • homologe Chromosomen paaren sich (4 Chromatiden) • Bildung der Mitosespindeln im Cytoplasma ausgehend von den beiden Centrosomen (aus Mikrotubuli) Wanderung zu Zellpolen • Auflösung der Kernmembran • Spindelfasern der Centrosomen wachsen in Kernbereich hinein verbinden sich mit Kinetochoren der Chromatiden - Metaphase I • Chromosomen paarweise in Äquatorialebene angelagert • Kinetochor eines Zellpolsn ist mit je einem Chromosom eines Paares verbunden - Anaphase I • Kinetochorfasern verkürzen sich ziehen (ganze) Chromosomen zu Zellpolen homologe Chromosomen jeweils zum gegenüberliegenden Pol • Zellpole rücken auseinander - Telophase I & Cytokinese • Chromosomen erreichen Pole (haploider Satz, aber Chromosomen sind noch doppelt) • Kinetochorfasern lösen sich auf • um Chromosomen wird neue Kernhülle gebildet • Trennung der Zelle zu 2 Tochterzellen • KEINE Verdopplung des gen. Materials o Meiose II (Trennung der Schwesterchromatiden) - Prophase II • Neuer Spindelapparat • Chromosomen bewegen sich zur Äquatorialebene - Metaphase II • Anlagerung der Chromosomen an Äquatorialebene wie bei Mitose (senkrecht) - Anaphase II • Schwesterchromatiden trennen sich im Centromer individuelle Chromosomen • Bewegung zu Zellpolen - Telophase II & Cytokinese • Kernbildung an Zellpolen • Zellteilung 4 Tochterzellen mit haploiden Chromosomensatz o Interphase - jedes Chromosom verdoppelt sein genetisches Material Seite 12 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - Vergleich Mitose – Meiose DNA-Replikation Zahl der Teilungen Paarung homologer Chromosomen Zahl der Tochterzellen Rolle Mitose Während jeder Interphase vor Kernteilung 1 (P, M, A, T) Nein Meiose Einmal vor Meiose I 2 (jeweils P, M, A, T) Ja 2 – diploid – gen. identisch mit Elternzelle 4 – haploid – gen. nicht identisch mit Elternzelle oder untereinander Gameten, gen. Varibiabilität Wachstum, Gewebsregeneration - DNA – Organisationsstrukturen - Chromosomen o Verpackungsproblem bei DNA in Phagen, Viren, Bakterienzellen und eukaryotischen Zellkernen – DNA-Molekül ist zu lang starke Komprimierung notwendig o Chromatin - Protein-DNA-RNA-Komplex - Interphaseform des gen. Materials in einem Zellkern - 2 Zustände: • Heterochromatin o kondensiert und dicht gepackt; auffällig färbbar o besteht aus ganzen Chromosomen oder Teilen von Chromosomen o findet sich häufig in der Nähe von Centromeren und Telomere o genetisch inaktiv (inert) o während Interphase kondensiert – Transkription ist unterdrückt wird spät in S-Phase repliziert o relativer Anteil von H. in Zelle schwankt o konstitutives Heterochromatin chromosomale Regionen, die in allen Zellen in beiden homologen Chromosomen an der gleichen Stelle heterochromatisch sind Bsp.-Regionen: Centromere, Telomere sehr hoher Anteil an nicht-codierenden Sequenzen Bsp.: Positionseffektvariegation bei Drosophila m. • - Euchromatin o aufgelockert und wenig dicht gepackt; weniger anfärbbar o euchromatische Bereiche existieren während Interphase und Mitose in versch. Kondensationsphasen o zur Transkription fähig o Packung kann zwischen Interphase und Zellteilung zyklisch wechseln o fakultatives Heterochromatin (! Bez. eig. falsch !) Zustand des Euchromatins Inaktivierung ganzer Chromosomen o. sogar vollständiger Chromosomensätze durch dichtere Verpackung betrifft immer nur einen von 2 homologen Partnern Bsp.: (zufällig ausgewählte) Inaktivierung eines XChromosoms bei weibl. Säugern (sog. Barr-Körper) • aber 2tes X-Chromosom darf nicht fehlen, da sonst Turner-Syndrom (klein & steril) genetisch inaktiv ≠ heterochromatisch !!! Seite 13 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr • genetische Inaktivität kann o durch nicht-codierende Sequenzen bedingt sein, als auch o aus Repression von potentiell transkribierbaren Sequenzen resultieren Anwendung des Begriffs Heterochromatin auf beide Phänomene verdeckt wesentliche Unterschiede - Positionseffektvariegation • Somatisch instabiler Positionseffekt • Mutanten sind durch Mosaike aus normalen und mutantem Phänotyp charakterisiert • betroffenes Gen funktioniert nur in einem Teil der Zellen und Genprodukt wird zwischen Zellen nicht ausgetauscht • je näher ein Gen zum Heterochromatin positioniert ist, um so wahrscheinlicher ist es der Positionseffektvariegation zu unterliegen und umso größer ist der Anteil der Zellen, in denen das Gen inaktiv ist • Bsp.: white-mottled Drosophila m. – gescheckte Augen o Nichthistone - alle Proteine des Chromatins, außer den Histonen - zwischen versch. Geweben und Spezies wahrsch. größere Variabilität als Histone - geringerer Teil der Proteinmenge, als Histone - viel größere Anzahl versch. Proteine, als Histone - Funktionen: • Steuerung der Genexpression • Aufrechterhaltung der übergeordneten Strukturen - HMG-Proteine • Abgegrenzte, genau definierte Unterklasse der Nichthistone - Bsp.: RNA-Polymerase o Centromer - die Stelle des Chromosoms, die Bewegung ermöglicht - ist essentiell für Segregation • wenn Bruch ein Fragment mit Centromer und azentrisches Fragment ohne Centromer erzeugt, wird azentrisches nicht an Spindelfaser befestigt fehlt in einem der Tochterzellkerne o Metaphasechromosomen - extrem dicht verpackt ~ 100mal stärker als Lampenbürsten- / Polytänchromosomen - bereits verdoppelt - 2 Untereinheiten: • Chromatiden = Tochterchromosomen • Centromer = Spindelfaseransatzstelle - in Schleifen organisiert • Schleifen an zentraler Proteingerüst verankert • in Schleifen: codierende DNA-Sequenzen - aufgrund des hohen Kondensationsgrads Gene transkribtiv inaktiv o Lampenbürstenchromosomen - in Amphibien etwa 0,5 mm lang Kondensationsfaktor 100 Seite 14 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - bilden sich in Prophase der Meiose I aus (in Oogenese und Spermatogenese) bereits verdoppelt symmetrische Schleifen • i.d.R. 4 homologe Schleifen o bestehen aus einer DNA-Faser als Achse, die mit Transkriptionsprodukten dicht bepackt ist Christbaum • Schwesterchromatiden bilden Chromosomenachse • an der Basis stark kondensiert o Polytänchromosomen - während des gesamten Zellzyklus lichtmikroskopisch sichtbar - ca. 100mal größer als Metaphasechromosomen - entstehen durch schrittweise Verdopplung der DNA ohne Kernteilung • d.h. resultierende Kerne sind polyploid (Erdbeere, Banane) o Hierarchie der Faltung von Chromatin - Nucleosomen – Perlenschnur aus DNA (ca. 200 bp) und Histon-Molekülen (2x4 + 1) - 30-nm-Chromatin-Faser – Verdichtung der Perlenschnur zur Chromatinfaser - Schleifendomänen – Faltung der 30-nm-Faser zu Schleifen; an Nicht-HistonProteine geheftet - Metaphase-Chromosom – durch weitere Faltung bildet typ. hochkompakte Form des Chromosoms o Bänderungsverfahren - von sehr praktischer Bedeutung - aber Mechanismus noch unklar o Puff - - Bereich, indem die Chromosomenfäden aus ihren normalen Packungszustand freigesetzt werden Bereiche, in denen RNA-Synthese stattfindet (Relaxion der DNA notwendig, damit RNA synthetisiert werden kann) • Ecdyson (genaktivierend) in Larve injiziert Puffs, da mRNA, die translatiert wird, gebunden wird gehen normalerweise von einzelnen Banden aus Muster steht im Zusammenhang mit Genexpression • jedes einzelne aktive Gen kann einen Puff verursachen • in jedem Gewebe gibt es zu jedem beliebigen Zeitpunkt ein charakteristisches Muster von Puffs - Genom-/Chromosomenmutation o Verursacher: ionisierende Strahlung; radiometrisch wirkende Chemikalien; UV-Licht; spontane Ereignisse o Chromosomenmutationen – Variation der Chromosomenstruktur - Chromosomenbrüche, die zu strukturellen Umlagerungen führen entweder Reihenfolge der Gene innerhalb einer Kopplungsgruppe verändert oder Gruppe von Genen mit anderer Kopplungsgruppe verbunden - Art der Mutation hängt von Anzahl der Brüche und Verteilung ab - Deletion Seite 15 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - - - • Fehlen eines Chromosomenabschnittes • bei einem Bruch Inversion • Umkehrung der Genreihenfolge in einem Chromosomenabschnitt • bei 2 Brüchen auf einem Chromosom • perizentrische o Inversionsschleife umfasst Centromer • Parazentrische o Centromer außerhalb der Inversionsschleife Duplikation • Verdopplung eines Chromosomenabschnittes • Tandemduplikation o direkt (gleiche Reihenfolge) o invertiert • umgesetzte Duplikation o direkt o invertiert • ungleiches Crossing-over o bei tandemartig angeordneten Sequenzen o ungleicher Strangaustausch 1. Augenbildung bei Drosophila • kleine Tandemduplikation Bar-Mutation (bandförmige Augen) • auf einem Chromatid geht Bar-locus verloren • auf anderen Mutation Locus verdoppelt • bei mehr als 2 Brüchen Translokation • Übertragung eines Chromosomenabschnittes von einem Chromosom auf ein anderes • einfache Translokation • reziproke Translokation • Verschiebung • bei jeweils einem Bruch auf unter 2 unterschiedlichen Chromosomen o Genommutationen – Variation der Chromosomenzahl - Quantitative Änderungen des Chromosomensatzes - Euploidie • betrifft Anzahl vollständiger Chromosomensätze • • • Haploidie o ein vollständiger Chromosomensatz – n Diploid o normal – 2n Polyploidie – 3n, 4n, … o Autopolyploidie • durch Vervielfältigung des eigenen Chromosomensatzes o Allopolyploidie • durch Kombination unterschiedlicher Genome o Autoallopolyploidie • vereinigt Merkmale von Allo- und Autopolyploidie Seite 16 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr o Beispiele: • Bienen: Königin – diploid; Drohnen – haploid • Bananen, Weintrauben, Birnen – triploid • Baumwolle, Kartoffeln, Kaffee, Erdnüsse – tetraploid • heutiger Weizen – hexaploid - Aneuploidie – gutes Beispiel für Gendosiseffekte • betrifft einzelnen Chromosomen eines Satzes • • • • • • Monosomie - Hypoploidie o 2n-1 – es fehlt ein Chromosom • Gendosis um 50% verringert o doppelte Monosomie – 2n-1-1 – es fehlen 2 nichthomologe Chromosomen o Nullisomie – 2n-2 – es fehlt ein Chromosomenpaar Trisomie – Hyperploidie o 2n+1 – ein Chromosom doppelt • Gendosis um 50% gesteigert o doppelte Trismoie – 2n+1+1 – 2 zusätzliche nichthomologe Chromosomen Tetrasomie – Hyperploidie o 2n+2 – ein Chromosom 4fach bilateraler Gynandromorph bei Drosophila o nach erster mitotischen Teilung verliert eine Zelle das 2te XChromosom (links) o linke Seite: aus männlichen Zellen – X0 – weißes Auge, kleine Flügel o rechte Seite: aus weiblichen Zellen – XX – heterozygot für die rezessiven Allele – schwarzes Auge, normaler Flügel Menschen – heterosomal o 45, X0 – Turner-Syndrom • Zwergwuchs o 47, XXY – Klinefelter • unfruchtbar • geringer IQ • sehr groß Mensch - autosomal o 47, XY, +21 – Down-Syndrom • Herzfehler • Schwachsinn • Vierfingerfurche • kurzer Schädel o 47, XY, +13 – Pätau-Syndrom o 47, XY, +18 – Edward-Syndrom o Genmutationen - Punktmutationen - submikroskopische Veränderungen von DNA-Sequenzen • einzelnen Basen oder Tripletts - Detektionen von Mutationen bei Drosophila Seite 17 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - - - Sex-linked recessiv lethal system Autosomal o. Sex-linked visible mutations Morgan – bewies zuerst die Präsenz eines Genes auf einem Chromosom • bei Augenfarbe von Drosophila • white eyes determinierende Gen und sein Allel auf X-Chromosom • ebenfalls: yellow body color und miniatur wings Sichelzellanämie chemisch induzierte • Basenpaar-Substitution o theoretische Bedeutung o Bedeutung bei Klärung von Schwesterchromatidenaustausch o anstelle der richtigen Base wird Analogon eingebaut o z.B. 5-Bromuracil o dient zur Markierung spontane • Basenpaar-Substitutionen aufgrund Tautomerie der DNA-Basen o Transition • Purin durch Purin • Pyrimidin durch Pyrimidin • menschl. Hämoglobin o Transversion • Purin durch Pyrimidin • Pyrimidin durch Purin • menschl. Hämoglobin o Missense-Mutationen • Austausch einer Aminosäure im Genprodukt • entweder: Funktion des Moleküls wird nicht gestört • oder: vollständiger Funktionsverlust möglich o Neutrale Mutationen • es entsteht trotzdem richtige Aminosäure (3. Base!) o Nonsense-Mutationen • entweder kein Genprodukt • oder verkürztes Genprodukt; i.d.R. nicht funktionsfähig o Rasterschubmutation • völlige Veränderung einer ganzen Aminosäuresequenz • • - Desaminierung (chemisch) o Entfernung der Aminogruppe einer Base Depurinierung/-pyrimidierung o Base geht verloren durch Auflösung der Verbindung zur Desoxyribose temperatursensitive • normale Funktion bei normalen Temperaturen • Störung bei höheren o. niedrigeren Temperaturen (Tod, o.ä.) • Bsp: Mutante von Drosophila ist bei 29°C innerhalb weniger Sekunden gelähmt Seite 18 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr o Geschlechtsbestimmung bei Drosophila - Weibchen normalerweise 2X - Männchen normalerweise XY - aber Y nicht entscheidend, sondern Verhältnis von X-Chromosomen zu Autosomensatz • X:A ≥ 1 Weibchen • X:A ≤ 0.5 Männchen • 2X und 3A steriler Intersex mit mosaikartigen weibl. und männl. Zügen o Gendosiskompensation in Drospohila - Dosiskompensation wichtig, da Anzahl von X sich bei Geschlechtern unterscheidet Regulation der Menge an Genprodukten - Strategie bei Drosophila: Hyperaktivität im X-Chromosom des Männchens • ein Multi-Proteinkomplex verstärkt Transkription im X-Chromosom - Dosiskompensation wird früh in Embyogenese initiiert - Dosiskompensationskomplex aus RNA und Proteinen • Nicht-codierende roX-RNAs und MSL-Proteinen • bindet an X und bewirkt Hypertranskription o MSL-Proteine können ohne roX-RNA nicht effizient an X binden o Transkriptionsstellen der roX-RNAs sind gleichzeitig Entstehungsort des Komplexes und 2 Ansatzstellen für Ausbreitung auf X (noch ca. 35 weitere Ansatzstellen) - Sex-lethal-Gen und daughterless-Gen haben Schlüsselrolle • ist Sxl angeschalten (normalerweise beim Weibchen), existiert aktives Proteinprodukt des Genes keine Dosiskompensation • ist Sxl ausgeschalten (normalerweise beim Männchen), existiert aktives Protein nicht Dosiskompensation • ohne da-Gen kann Sxl nicht aktiviert werden o Geschlechtsbestimmung bei Säugern - bestimmt durch Y - während Embryonalentwicklung noch kein Unterschied zw. männl. u. weibl. - ohne Y wird Gonadenanlage zu Ovarien Östrogene - mit Y wird Gonadenanlage zu Hoden Testosteron & Anti-Müllersche Hormon o Replika-Technik - Samt-Stempel wird auf Ausgangsplatte gedrückt und dann auf mehrere Selektionsplatten Übertragung mit gleicher Orientierung auf unterschiedliche Nährböden Identifikation der einzelnen Stämme immer noch möglich o Spreading technique – Fluktuationstest - belegt das Phagenresistenz auf zufälligen Mutationsereignissen, bereits vor dem Selektionsereignis, beruhen • Ansetzen einer Bakterienkultur Probe wird auf antibiotikumhaltigem Nährboden ausgebreitet alle Bakterien sterben ab, da keine resistenten Zellen vorhanden sind Anfangskultur werden zwei Ansätze A und B entnommen Ansatz A wird in Reagenzglas belassen Seite 19 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - Ansatz B wird auf andere Reagenzgläser aufgeteilt beide Kulturen werden auf jeweils mehrere antibiotikumhaltige Nährböden verteilt bei A auf allen Platten etwa die gleiche Anzahl antibiotikaresistenter Kolonien. Ansatz B zeigt starke Unterschiede im Ergebnis (Fluktuationen genannt) Gesamtzahl antibiotikaresistenten Bakterien ist gleich - Streuung der Anzahl resistenter Kolonien auf Platten des Ansatzes B widerlegt, dass Mutationen durch Kontakt mit Antibiotikum entstanden sind – sonst wäre Anzahl resistenter Bakterien aus Ansatz B auf allen Platten etwa gleich gewesen Fluktuationstest weißt nach, dass die Resistenz ein zufälliges Mutationsergebnis ist, und schon vor dem Kontakt mit dem Antibiotikum entsteht - DNA-Repair 1. Photoreaktivierung UV-geschädigter DNA o UV-Strahlung verursacht Bindung zwischen benachbarten Pyrimidine o. Purinen - Wasserstoffbrücke zu gegenüberliegender Base nicht mehr vorhanden DNA-Struktur wird verzerrt - Thymin-Dimer am häufigsten o Reparatur durch Enzym Photolyase - bindet (auch im Dunkeln) an Pyrimidindimer - trennt diese bei Licht in einer Photoreaktion o im Prinzip Schutz gegen Hautkrebs 2. Nucleotidexzisionsreparatur o vielseitigster Weg, was Vielzahl der erkannten und behobenenSchäden angeht o Ablauf - DNA-Schaden bzw. Verzerrung der DNA wird erkannt - betroffene Strang wird an beiden Seiten des Schadens mit wenigen Basen Abstand eingeschnitten und beschädigter Teil wird entfernt - entstandene Lücke wird durch Reparatursynthese gefüllt und freie Enden verschlossen o Suppressormutation - 2 Mutationen, wobei die 2te Mutation in der Lage ist die phänotypische Ausprägung der ersten Mutation zu hemmen bzw. zu unterdrücken - - intergene Suppression – Behebung durch anderes Gen • Mutation in einem Gen für Aminoacyl-tRNA (AA-tRNA) o tRNA wird mit falschen AS beladen • Mutation in einer tRNA o Stopp-Codon kann übergangen werden Bindung einer Aminosäure, die eigentlich da hin sollte • Mutation in einem Ribosomen o Genauigkeit der Ablesung wird verringert Fehlablesungen können supprimiert werden Intragene Suppression – Rückmutation auf demselben Gen • Transition eines Nuccleotids wird durch erneute Transition behoben Genetik von Bakterien und Viren Seite 20 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr o Konjugation - Sexualvorgang bei Bakterien - 2 Zellen fusionieren Austausch gen. Materials über Cytoplasmabrücke - Mobile genetische Elemente: Transposons o Transposition - „illegitime“ Form der Rekombination zwischen nichthomologen DNASequenzen - wesentlich: DNA-Synthese ist beteiligt o in Prokaryoten 20-40% aller Mutationen aufgrund Transposons - in E. coli einfache IS-Elemente (Insertionssequenzen) • einfache DNA-Segmente; invertierte Repeats - in E. coli auch komplexere Transposons • tragen oft Gene für Antibiotikaresistenz o Eukaryoten - viele unterschiedliche Gruppen von Transposons - Häufigkeit im Genom variiert - durch Umwelteinflüsse, oder gen. induziert - resultieren oft in Mutationen - durch flankierende Duplikationen an Insertionsstelle gekennzeichnet o 2 Möglichkeiten: - Exzision/Insertion • präzises Herausschneiden des Elementes an den Enden Aufschneiden der Wirts-DNA, mit versetztem Schnitt Einbau des herausgeschnittenen Elements Reparatur der Lücken durch Duplikation von Bereichen des gerade eingesetzten Stückes (flankierende Duplikationen) - Verdopplung • gleiches Prinzip, nur Verdopplung des Elements und Beibehaltung der ursprünglichen Position o 4 Gruppen - Insertions-Elemente (-Sequenzen) • bestehen nur aus der DNA, die für Transpositionsvorgang selbst verantwortlich ist • keine weiteren Gene • ein Gen auf dem DNA-Abschnitt codiert Transposas – eingerahmt von inverted repeats als Begrenzung (codieren selbst nicht) - einfache Transposonen – Tn3-Familie - zusammengesetzte Transposonen • beinhalten weitere Gene, z.B. für Antibiotikaresistenz • Mutagene - transposible und mutagene Phagen o Retrotransposonen - große Ähnlichkeit mit Struktur der Retroviren, bilden aber keine infektiösen Partikel Seite 21 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - Transposition erfolgt über RNA-Zwischenstufe festgelegte Struktur mit LTRs (long terminal repeats) proteincodierende Gene – u.a. reverse Transkriptase o Retro-Viren - ähnlich Retrotransposom - Transposition erfolgt über RNA-Zwischenstufe Genom besteht aus RNA und wird bei Replikation in DNA umgeschrieben - codieren zusätzlich infektiöse Partikel - können als Proviren in Genom eingebaut werden und an Nachkommen vererbt werden - Induzierung von Tumoren möglich - können auch zelluläre Gene in ihr Genom aufnehmen o Pseudogen: Gen-ähnlicher DNA-Abschnitt, der nicht funktionsfähig ist - 2 Arten: • durch reverse Transkription von polyadenylierter mRNA und Einbau ins Genom • duplizierte Gene, die Defekte haben und nicht funktionsfähig sind - Mendel o leitete die Entwicklung der Formalgenetik ein o kontrollierte Kreuzungsversuche an Erbsen o Beobachtung von Merkmalsausprägungen - Samenform, Farbe der Samenschalen und Erbsen, Form der Hülsen, Stand der Blüten u.a. 1. Mendelsche Gesetz – Uniformitäts- und Reziprozitätsgesetz Kreuzt man reinerbige Eltern, die sich in einem Merkmal unterscheiden, so sind alle Nachkommen (F1) unter sich gleich, uniform. Dabei ist gleichgültig, welcher Kreuzungspartner als Vater und welcher als Mutter dient; reziproke Kreuzungen sind gleich. 2. Mendelsches Gesetz - Spaltungsgesetz Kreuzt man die Individuen der F1-Generation untereinander o. mit sich selbst, so erhält man in der F2-Generation eine Aufspaltung in festen Zahlenverhältnissen. beim intermediären (unvollständige Dominanz) Erbgang 1:2:1; beim dominanten Erbgang: 3:1 3. Mendelsches Gesetz – Unabhängigkeitsgesetz, Gesetz von der Neukombination Kreuzt man reine Rassen, die sich in mehreren Merkmalen (in mehreren Genen) unterscheiden, so werden die Allele verschiedener Gene frei kombiniert und unabhängig voneinander vererbt. (Gesetz nicht universell gültig, nur eine bestimmte Kategorie jeweils betrachteter Gene) o unvollständige Dominanz - keins der zwei Allele setzt sich voll durch - rosa Löwenmäulchen neuer Phänotyp o Codomianz - beide Allele manifestieren sich unabhängig voneinander ausgeprägt Seite 22 von 27 beide werden zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr - MN-Blutsystem beim Menschen o Multiple Allelie - Merkmale durch verschiedene Allele in einer Organismengruppe gekennzeichnet - AB0-Blutsystem des Menschen o Polygenie - Merkmale durch mehrere Gene beeinflusst - Hautpigmentierung des Menschen o Pleiotropie - Gen hat Einfluss auf mehrere phänotypische Merkmale o Expressivität - Merkmale können in unterschiedlichem Maß zur Ausprägung kommen o Penetranz - Merkmale kommen gar nicht zur Ausprägung o Epistasie - Gene können in mutanter Form die Ausprägung anderer Gene unterdrücken - Albino bei Mäusen o Superdominanz (monogene Heterosis) - Phänotyp der Heterozygoten liegt außerhalb der von den Eltern vorgegeben Grenzen o Kopplungsgruppe - Chromosom, da lineare Anordnung von hunderten von Genen Rekombinationshäufigkeiten ermitteln Lage von Genen relativ zueinander und ihr Abstand im Chromosom Unabhängigkeit der Gene ermittelbar o Komplementationstest - einfach Methode zur groben Lokalisierung von genetischen Defekten (Mutationen) - liegen in einem Gen zwei Mutationen a und b vor, dann muss der Phänotyp der Heterozygote a/b einen mutierten Phänotyp zeigen - falls die Mutationen a und b in verschiedenen Genen vorliegen, so ist der heterozygote Genotyp a+/+b vom Phänotyp des Wildtyps - Drosophila melanogaster o Achsenbildung - bereits im unbefruchteten Ei im Ovar der Mutter - über maternale Genprodukte - Festlegung der Körperachsen durch 4 Gruppen (Polaritätsgene): • die anteriore Region (Kopf, Thorax) o Hauptgen bicoid • die posteriore Region o Hauptgen nanos • die dorsale Achse o Hauptgen dorsal Seite 23 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr • - Terminales System o Hauptgen Y (noch nicht näher identifiziert) Ausfall eines oder mehrerer der Gene bewirkt charakteristische Effekte und Defekte o Segmentbildung - stimuliert durch maternale Gene - 3 Klassen von Segmentierungs-Genen • legen Anzahl und Polarität der Körpersegmente fest • Gap-Gene o legen grobe Segmentierung des Embryos in 5 Bereiche fest (Acron, Kopf, Thorax, Abdomen, Telson) o hunchback o Krüppel o knirps o giant o tailless & huckebein • Paar-Regel-Gene o Feinsegmentierung und periodisches Streifenmuster, welches Anzahl und Polarität der 14 Segmente bzw. 7 Segment-Paare festlegt o even-skipped o hairy • Segmentpolaritätsgene o bestimmen interne Segmentdifferenzierung in Längs- und Dorsoventral-Achse o Segmente werden voneinander abgetrennt und intern untergliedert o engrailed o gooseberry o hedgehog o wingless o fused - homöotische Gene • stimuliert durch Gap- und Paar-Regel-Gene • spezifizieren die Identität eines ganz bestimmten Körpersegmentes (Spezifität, Identität, Reihenfolge der Körpersegmente) • genregulatorische Funktion mit Hilfe der Homöobox o Homöobox: • DNA-Region, oft nahe dem 3’-Ende der jeweiligen Transkriptionseinheit • codiert Polypeptidsequenz von 60 Aminosäuren Homöodomäne • bildet Helix-turn-Helix-Motiv • bindet mit hoher Affinität an spezifische Sequenzen in der Kontrollregion des zu regulierenden Gens • Antennapedia-Komplex o bestimmt die spezifische Differenzierung der Kopf- und ThorakalSegmente • Bithorax-Komplex Seite 24 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr o Gen-Cluster o bestimmt die Identität der hinteren Thorax- und AbdominalSegmente - Gentechnik o direkte Manipulation von Genen zur praktischen Anwendung o Arbeitsschritte zur gezielten gentechnischen Veränderung eines Organismus - Isolierung der DNA o. des DNA-Segments der Spenderzelle - Einbau des isolierten DNA-Segments in einen Vektor dient als DNAÜberträger - Einschleusen der neukombinierten DNA in eine Zielzelle - Etablierung der neukombinierten DNA als extrachromosomales Element o. Integration in das Genom - Analyse der geno- und phänotypischen Veränderung o Werkzeuge der Gentechnik: - Restriktionsenzyme • schneiden DNA sequenzspezifisch • Teil des Schutzmechanismus von Bakterien gegen fremde DNA - Ligase • zusammenfügen von DNA - Vektoren - Wirtsorganismen o Vektoren für die Klonierung - ein Vektor ist ein Nucleinsäuremolekül, das als Träger für fremde DNA dient - Klonierungsvektor kann sich als eigenständige, vermehrungsfähige Einheit in Wirtszelle replizieren • hat dazu ori – origin of replication - Plasmidvektoren • extrachromosomales doppelsträngiges DNA-Molekül • mit wenigen Ausnahmen zirkulär geschlossen - Transformation von Bakterien: • Bakterien in Kälte in CaCl2-Lösung inkubiert Zellwand und –membran wird durchlässig Bakterien und Plasmid-DNA auf Agarplatten mit Nährmedium und Antibiotikum Bakterienkolonien, die das Plasmid mit dem entsprechenden Resistenzgen haben, wachsen Transformation erfolgreich - transgene Pflanzen • 3 Möglichkeiten • 1 davon: biolistische Transformation o z.B. für Getreide und auch für Mitochondrien und Plastide o um Zellwand der Pflanzenzelle zu durchdringen, beschießt man sie mit DNA-beschichteten Gold- o. Wolframpartikeln o Benutzung von Shuttle-Vektoren • besitzen Selektionsmarke für Pflanzen Seite 25 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr • - Klonierungsstelle zw. pflanzenspezifischen Promotor und Terminator transgene Organismen (Drosophila) • einbauen der Fremd-DNA in P-Element spritzen der rekombinanten DNA in posteriore Region von Drosophila-Ei dort Entwicklung der Urgeschlechtszellen durch Transposase wird Fremd-DNA stabil ins Genom aufgenommen o Synthese komplementärer DNA - mRNA wird mit Enzym reverse Transkriptase zusammengefügt - kurzer DNA-Primer wird hinzugefügt, welcher Hybridisierung ermöglicht - reverse Transkriptase bildet cDNA aus DNA-RNA-Hybrid entsteht - mRNA wird entfernt - DNA-Polymerase benutzt cDNA als Template um gegensätzlichen DNA-Strang zu synthetisieren o DNA-Klonierung - Herstellung von DNA-Fragmenten - Vermehrung eines DNA-Fragments in theoretisch unbegrenzter Menge - Verknüpfung von Fremd-DNA mit dem Vektor - Einschleusung der künstl. rekombinanten Moleküle in eine Wirtszelle, in der sie repliziert werden können - Selektion von Klonen, die das rekombinante Molekül aufgenommen haben o PCR - Polymerasekettenreaktion damit können geringste Mengen von DNA vermehrt und für Untersuchung zugänglich gemacht werden o DNA-Sequenzierung - die Bestimmung der DNA-Sequenz, also der Nukleotid-Abfolge, von genomischer DNA Seite 26 von 27 zuletzt aktualisiert: 09.08.2004 - 23:12 Uhr benutze Offline-Quellen - Script Genetik I - Prof. Sass (2002/2004) inkl. Mitschriften von Steffi R. (danke noch mal ☺) - Lehrbuch der Genetik - Seyffert (2. Auflage) - Biologie – Campell (6. Auflage) - Zoologie – Wehner/Gehring (23. Auflage) benutzte Online-Quellen - http://www.aum.iawf.unibe.ch/vlz/BWL/Gen_Kurs/start.htm - http://zope.reaktor.fhfurtwangen.de/portal/natural_sciences/nuclear_sciences/ student%20project%201%20radiation%20biology/Genetik - http://www.schirkonyer.de/genetik.htm - http://www.dev-biologie.de/drosophila/drosophila.htm weitere Danksagungen ☺ gehen an – in alphabetischer Reihenfolge: - dem Bildungsministerium für die Finanzierung meines Studiums - meinen Mitbewohnern, dafür, dass ich 14 Tage aus dem Putzplan genommen wurde - den ZIMO Copyshop und seinen Mitarbeitern für die freundliche Hilfe, ohne die eine Vervollständigung des Scripts nicht möglich gewesen wäre, da das Mädel, was sich für die Folien verantwortlich erklärt hatte, auf halber Strecke nur noch die aktualisierten Folien im Copyshop hinterlegt hat und somit ein sehr unvollständiges Script von 2004 entstanden ist, welches sich nur schlecht mit dem von 2002 verkomplettieren lies Disclaimer & Copyright Ich übernehme keine Garantie für die Korrektheit der von mir hier zusammengetragenen Informationen. Trotz gründlicher Arbeit, kann ich sowohl Tippfehler, als auch Inkonsistenzen, die sich durch unterschiedliche – evtl. auch widersprechende – Quellen ergeben, nicht ausschließen. Ich übernehme keine Verantwortung für Schäden, die aus der Verwendung dieses Dokuments resultieren. Das Risiko bei der Verwendung dieser Zusammenfassung liegt alleine beim Benutzer. Ich habe keinen Einfluss auf die Seiten, deren Links ich angegeben habe. Fehler können mir gerne mitgeteilt werden unter [email protected] Sie dürfen auf beliebigen Medien unveränderte Kopien dieses Dokumentes, wie sie es erhalten haben, anfertigen und verbreiten; Änderungen, die nicht vom Autor genehmigt wurden, sind jedoch nicht erlaubt. Mit der Verwendung dieser Zusammenfassung erklären Sie sich mit den hier angegebenen Bedingungen einverstanden. Seite 27 von 27