Wiederholunng
Werbung

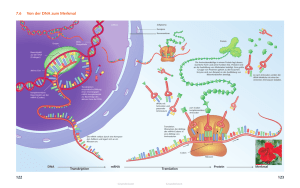
Wiederholunng Klassische Genetik Mendelsche Regeln Uniformitätsregel Spaltungsregel Freie Kombinierbarkeit Koppelung von Genen Polygene: mehre Gene für ein Merkmal Pleiotropie: 1 Gen steuert mehrere Merkmale Epistasie: 1 Gen steuert die Wirkung eines anderen Wiederholung Folgen nicht Mendel: Polygene Gekoppelte Gene Cytoplasmatische Vererbung Chromosomen beim Menschen: 46 Chromosomen (diploid) 1 Chromosomenpaar geschlechtsbestimmend: XX = ♀ XY = ♂ Wiederholung Mutationen: Genommutationen Chromosomenmutationen: Deletion, Translocation, Duplication, Inversion Genmutationen Unterschiedliche Chromosomenzahl beim Menschen: Trisomie (21 = Down-Syndrom) Ullrich-Turner-Syndrom (45,X): unterentwickelte weibliche Geschlechtsmerkmale, eine kleine Statur, einen tiefen Haaransatz, eine ungewöhnliche Augen- und Knochenentwicklung, eine Trichterbrust und sind meist unfruchtbar Triplo-X-Syndrom (47,XXX). Das Triplo-X-Syndrom ist die klinisch unauffälligste Chromosomenaberration Klinefelter-Syndrom (fast immer 47,XXY; selten 48,XXXY oder 49,XXXXY) Molekulare Genetik DNA und molekularer Bau: schon besprochen Desoxyribose, Phosphat, Purin und Pyrimidinbasen Doppelhelix Genetischer Code 4 verschiedene Basen Für 20 Aminosäuren müssen Basentriplets kodieren (42 = 16, 43 = 64) Triplet = Codon (mRNA) Codon teterminierende Teil auf DNA = Codogen komplimentärer Teil bei der t-RNA = Anticodon Aufklärung des genetischen Codes Bis zur Mitte der 60er Jahre alle 64 möglichen Basentripletts funktionell zugeordnet 61 der 64 Codons legen Aminosäuren fest, die drei verbleibenden sind Stoppcodons, die den Translationsvorgang beenden Der genetische Code ist redundant, aber nicht gänzlich zufällig; kein Codon codiert mehr als eine Aminosäure Das Leseraster ist für die Zelle zum Verständnis der molekularen Texte in ihrer Erbsubstanz von großer Bedeutung Die Evolution des genetischen Codes Der genetische Code ist fast universell gültig und findet sich in gleicher Form von den einfachsten Bakterien bis zu den kompliziertesten Tieren und Pflanzen Gene lassen sich oft problemlos transkribieren und translatieren, wenn sie von einer Art in eine andere verpflanzt werden Replikation der DNA DNA-Polymerasen Semikonservativ Schrauben werden entwunden Komplementärer Strang wird ergänzt Fehler (1:104) werden durch Enzym korrigiert: Exonuklease (entfernt nichtgepaarte Nucleotide) Replikation der DNA (Eukaryonten) Wesentlich langsamer als bei Bakterien 5´ 3´ leicht Antiparallel: Okazaki-Fragmente (auch 5´ 3´) An mehreren Stellen gleichzeig (Replicon); 1000 und mehr pro DNA-Doppelhelix DNA-Polymerasen Eigene Startpunkte (Iniationspunkte) Replikationsgabeln (bidirektional) DNA-Replikation im Detail. (a) In einem DNA-Molekül sind die Zucker-PhosphatGerüste der beiden Stränge in entgegengesetzten Richtungen orientiert. (b) Wegen der entgegengesetzten Orientierung synthetisiert die DNA-Polymerase einen Strang kontinuierlich und den anderen stückweise. DNA-Ligase setzt die kurzen Stücke zusammen. Helicase entwindet die Eltern-DNA, so dass die Replikationsgabel weiter voranschreitet. Reparatur Erkennung von Abweichungen der Idealstruktur Stelle wird erkannt durch Endonuclease erkannt und aufgespalten/ausgeschnitten Lücke wird durch DNAPolymerase (eigene für kleine Reparaturen, größere Reparaturen: normale DNAPolymerase) gefüllt Ligase verknüpft Große Schäden: letzte Rettung: Rekombinatonsreparatur (Schwesterchromosom als Matritze) Wie arbeiten Gene? Zentrale Dogma der Molekularbiologie: DNA RNA Protein Transkription Ablesen der DNA Translation mRNA Protein RNA Transkription RNA-Synthese: Sequenz der DNA wird abgelesen RNA-Polymerase Eukaryonten (mindstens 3: tRNA, rRNA und mRNA) mRNA-Vorläufer im Kern (Precursor); werden noch modifiziert Transkription RNA-Polymerase bindet an den Promotor (in der Nähe vor dem transkribierenden Gen) Partielle Endwindung der DNA RNA Polymerase bewegt sich entlang des DNAStranges als „Template“, Anlagerung und Verbindung der RNA-Nukleotide Terminationsstelle: RNA-Polymerase und RNA wird frei Die molekularen Komponenten des Transkriptionsapparates Das Enzym RNA-Polymerase zieht die Stränge der DNA auseinander und verknüpft die RNA-Nucleotide kovalent miteinander Wie die DNA-Polymerasen der Replikation, können auch RNA-Polymerasen Polynucleotide nur in 5’3’-Richtung bilden, sie sind dabei aber nicht auf einen Primer angewiesen Der Teil des Gens, an den die RNAPolymerase anfänglich bindet und der den Initiationsort der Transkription darstellt, heißt Promotor Die Basenfolge, die das Ende der Transkription signalisiert, wird als Terminator bezeichnet Der zu einer RNA transkribierte DNABereich heißt Transkriptionseinheit Processing Jedes Ende eines prä-mRNA-Moleküls wird in bestimmter Weise modifiziert: das zuerst synthetisierte 5’-Ende erhält eine 5’-Cap-Struktur (modifiziertes Guaninnucleotid) am 3’-Ende bildet sich ein Poly-A-Schwanz Diese Modifikationen üben mehrere wichtige Funktionen aus: sie zeigen an, dass die mRNA zum Export ins Cytoplasma bereit ist sie schützen die mRNA vor einem vorzeitigen Abbau durch Ribonucleasen sie unterstützen die Bindung eines Ribosoms an das 5’-Ende Processing Die meisten Gene und ihre primären RNA-Transkripte enthalten große Bereiche nicht-codierender (= nicht translatierter) Nucleotidfolgen Die zwischen den Exons eines offenen Leserahmens liegenden nicht-codierenden Abschnitte heißen Introns Die für Aminosäurefolgen codierenden Abschnitte eines offenen Leserasters heißen Exons Bei der Transkription schreibt die RNA-Polymerase II Exons, Introns und andere untranslatierte Bereiche in ein Primärtranskript um Translation mRNA + Ribosomen tRNA + Aminosäure Information der mRNA wird in Proteine umgesetzt Basentripletts; Codon, Anticodon Iniation Aktivierung der Aminosäuren (ATP, AS durch Aminoacyl-tRNA-Synthetase an tRNA) Ribosomen Elongation Aktivierte AS zu Ribosomen Termination Ribosomen zerfallen in ihre Untereinheiten Prokaryoten Modell einer t-RNA Aminosäurenakkzeptor Anticodon Kontrolle der Translation Synthese und Abbau der m-RNA Polyamine (z. B. Spermidin, Spermin) Ribosomensynthese, Transkrption, Translation Postsynthetische Modifikation Bei ca. 50 % der Proteine: Entfernung des N-terminalen Methioninrests durch Peptidasen freie NH2-Gruppe durch Acetylierung blockiert Proteintopogenese Reversible Modifikationen (Phosphorylierungen, Acetylierungen, Methylierungen…) Proteinfaltung, Chaperone Raumstruktur prinzipiell durch Primärstruktur Chaperone (Proteine als sog. „Chaperon-Maschinen) helfen bei Faltung, verhindern Schäden 2 Klassen von Proteinen