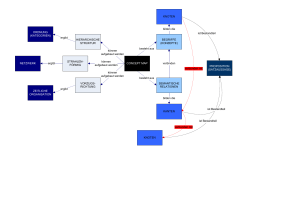
Implementierung eines effizienten Parameter - G-CSC
Werbung

Ruprecht-Karls Universität Heidelberg
Fakultät für Mathematik und Informatik
Lehrstuhl Technische Simulation
Bachelor-Arbeit
Implementierung eines effizienten
Parameter-Vererbungsalgorithmus zur
Konfiguration des Softwarepakets NeuGen
Sergei Wolf
Matrikelnummer: 2241758
Betreut durch
Prof. Dr. Gabriel Wittum
und Dr. Jens P. Eberhard
5. Dezember 2007
Erklärung
Hiermit versichere ich, dass ich diese Bachelor-Arbeit selbstständig verfasst und nur die
angegebenen Quellen und Hilfsmittel verwendet habe.
....................................................
Datum: 5. Dezember 2007
Danksagung
Ich danke Herrn Prof. Dr. Gabriel Wittum und Herrn Dr. Jens P. Eberhard für die Vergabe des interessanten Themas. Besonderen Dank schulde ich Alexander Wanner und
Simone Eberhard für zahllose kritische Kommentare und Verbesserungsvorschläge.
Abstract
The computer-based generation, computation and post-processing of neuronal networks
in 3D is an important research field of the neurosciences today. In order to make progress
in this field, the detailed reconstruction and analysis of neuronal nets is an indispensable
instrument. For this purpose the software NeuGen has been developed and is utilized in
the presented study.
NeuGen is written in C++ and Java, and it provides algorithms for the generation of
detailed neurons as well as neuronal networks in a three-dimensional cortical column.
A configuration editor can read, handle and modify the necessary parameters for the
configuration of the geometry. For an efficient inheritance of these parameters for the
different neuron classes, a faster method is in order. The presented thesis deals with the
development, implementation and run-time analysis of a new algorithm. The algorithm
uses so-called dependence graphs, and the resulting run-time is of linear order.
Zusammenfassung
Die computergestützte Erzeugung, Berechnung und Verarbeitung neuronaler Netze in
drei Raumdimensionen (3D) ist ein wichtiges Teilgebiet der Neurowissenschaften heutzutage. Um in diesem Gebiet die Forschung voranzutreiben, ist die detailgetreue Rekonstruktion und Analyse neuronaler Netze ein unverzichtbares Hilfsmittel. Das zu diesem
Zweck entwickelte Softwarepaket NeuGen ist Gegenstand dieser Bachelor-Arbeit.
NeuGen ist in C++ und Java geschrieben und enthält Algorithmen zur Erzeugung detaillierter Nervenzellen sowie neuronaler Netze in einer kortikalen Kolumne in 3D. Die
zur Konfiguration erforderlichen Geometrieparameter werden über einen Konfigurationseditor eingegeben bzw. eingelesen und modifiziert. Zur effizienten Vererbung dieser Parameter auf die einzelnen Neuronen-Klassen ist ein schnellerer Algorithmus notwendig geworden. Diese Arbeit beschäftigt sich mit der Entwicklung, Implementierung
und Laufzeitanalyse eines solchen Algorithmus. Der neue Algorithmus verwendet sogenannte Abhängigkeitsgraphen und zeigt insgesamt ein lineares Laufzeitverhalten.
Inhaltsverzeichnis
1
2
Einleitung
1
1.1
Motivation . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
2
1.2
Aufbau der Arbeit . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
4
Grundlagen
5
2.1
Neurobiologie . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
5
2.1.1
Das Gehirn . . . . . . . . . . . . . . . . . . . . . . . . . . . .
5
2.1.2
Die Nervenzelle . . . . . . . . . . . . . . . . . . . . . . . . .
8
NeuGen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
10
2.2.1
Neuronenklassen . . . . . . . . . . . . . . . . . . . . . . . . .
11
2.2.2
Parameterdateien . . . . . . . . . . . . . . . . . . . . . . . . .
13
2.2.3
Vererbungsalgorithmus . . . . . . . . . . . . . . . . . . . . . .
15
2.2.4
Konfiguration . . . . . . . . . . . . . . . . . . . . . . . . . . .
15
2.2.5
Visualisierung . . . . . . . . . . . . . . . . . . . . . . . . . .
17
2.2.6
Simulation . . . . . . . . . . . . . . . . . . . . . . . . . . . .
17
Graphentheorie . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
18
2.2
2.3
3
Vererbung der Geometrieparameter
21
3.1
Motivation . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
21
3.2
Grundbegriffe . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
22
3.3
Definition von Vererbung . . . . . . . . . . . . . . . . . . . . . . . . .
24
3.3.1
Beziehung erster Ordnung . . . . . . . . . . . . . . . . . . . .
24
3.3.2
Beziehung n-ter Ordnung . . . . . . . . . . . . . . . . . . . . .
25
3.3.3
Reihenfolge der Vererbung . . . . . . . . . . . . . . . . . . . .
26
Datenstrukturen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
28
3.4
i
Inhaltsverzeichnis
3.5
3.6
3.7
4
Traversierung von Bäumen . . . . . . . . . . . . . . . . . . . .
28
3.4.2
Implementierungen . . . . . . . . . . . . . . . . . . . . . . . .
31
Default-Recursive-Inheritance-Vererbungsalgorithmus . . . . . . . . .
34
3.5.1
Anhängen von Knoten . . . . . . . . . . . . . . . . . . . . . .
34
3.5.2
Vervollständigen von Knoten . . . . . . . . . . . . . . . . . . .
36
3.5.3
Vererben von Knoten . . . . . . . . . . . . . . . . . . . . . . .
39
3.5.4
Umkehrung der Vererbung . . . . . . . . . . . . . . . . . . . .
41
3.5.5
Implementierungen . . . . . . . . . . . . . . . . . . . . . . . .
42
3.5.6
Kosten der Funktionen . . . . . . . . . . . . . . . . . . . . . .
43
Default-Inheritance-Vererbungsalgorithmus . . . . . . . . . . . . . . .
47
3.6.1
Programmablauf . . . . . . . . . . . . . . . . . . . . . . . . .
47
3.6.2
Bestimmung von Siblingketten . . . . . . . . . . . . . . . . . .
48
3.6.3
Bestimmung und Ergänzung von abhängigen Teilbäumen . . . .
50
3.6.4
Wurzel-Siblingketten . . . . . . . . . . . . . . . . . . . . . . .
53
3.6.5
Teilbäume für die Bestimmung der zweiten Ordnung . . . . . .
56
3.6.6
Vererbung von Blättern . . . . . . . . . . . . . . . . . . . . . .
58
3.6.7
Implementierungen . . . . . . . . . . . . . . . . . . . . . . . .
61
3.6.8
Kosten der Funktionen . . . . . . . . . . . . . . . . . . . . . .
61
Simplified-Inheritance-Vererbungsalgorithmus . . . . . . . . . . . . . .
64
3.7.1
Parameterwerte aktualisieren . . . . . . . . . . . . . . . . . . .
64
3.7.2
Implementierungen . . . . . . . . . . . . . . . . . . . . . . . .
66
3.7.3
Kosten der Funktion . . . . . . . . . . . . . . . . . . . . . . .
67
Evaluierung
68
4.1
Vergleich der Vererbungsalgorithmen . . . . . . . . . . . . . . . . . .
68
4.1.1
Default-Recursive-Inheritance-Vererbungsalgorithmus . . . . .
70
4.1.2
Default-Inheritance-Vererbungsalgorithmus . . . . . . . . . . .
72
4.1.3
Simplified-Inheritance-Vererbungsalgorithmus . . . . . . . . .
73
Zusammenfassung . . . . . . . . . . . . . . . . . . . . . . . . . . . .
75
4.2
5
3.4.1
Zusammenfassung
76
Abbildungsverzeichnis
78
ii
Inhaltsverzeichnis
Algorithmenverzeichnis
81
Literaturverzeichnis
82
iii
1 Einleitung
Während Sie diese Zeilen lesen, ist es schon aktiv: Ihr Gehirn. Die Informationsverarbeitung im Gehirn zu verstehen und zu simulieren, ist eines der Forschungsziele der
Neuroinformatik. Dazu werden geometrische Modelle von Nervenzellen und ihrer Vernetzung benötigt. Auf solchen Modellen kann dann die Signalausbreitung in einem
neuronalen Netz berechnet werden. Aufgrund technischer Beschränkungen ist die mikroskopische Aufnahme und Rekonstruktion von biologischen neuronalen Netzwerken
nicht möglich. Daher wurde das Softwarepaket NeuGen entwickelt [4, Eberhard].
NeuGen erzeugt realistische Morphologien verschiedener Klassen von Neuronen und
synaptisch verbundener neuronaler Netzwerke in drei Raumdimensionen. Auf solchen
synthetischen Netzen kann dann die Signalausbreitung simuliert werden. Damit die Nervenzellen den in der Natur gefundenen möglichst gut entsprechen, werden Geometrieparameter für die Generierung benötigt. Beispiele für solche Geometrieparameter sind
der Radius des Zellkörpers, sowie Gesamtlänge und Anzahl der Verzweigungen des
Dendritenbaumes. Diese Parameter werden aus experimentellen Daten extrahiert.
Die Geometrieparameter werden zur Konfiguration von NeuGen verwendet. Sie können mithilfe eines Konfigurationseditors eingestellt werden. Da viele der Parameter für
die unterschiedlichen Nervenzellarten identisch sind, müssen sie nicht für jede Zelle
einzeln festgelegt, sondern können vererbt werden. Vererbung bedeutet, dass ein Wert
eines Parameters eines Neurons für bestimmte andere Neuronen unverändert übernommen werden kann. Dieses Vorgehen spart Speicherplatz und Bearbeitungszeit. Auch in
natürlichen neuronalen Netzen werden bei verschiedenen Neuronen übereinstimmende
Parameter gefunden.
1
1 Einleitung
Der bisher in NeuGen implementierte Algorithmus für die Vererbung neuronaler Morphologieparameter ist rekursiv. Da die Laufzeit exponentiell anwächst, ist dieser Algorithmus für große Parameteranzahlen nicht gut geeignet. Dies ist besonders dann ein
Problem, wenn viele Geometrieparameter gleichzeitig im Konfigurationseditor geändert
werden.
In dieser Bachelor-Arbeit wird der bisherige Vererbungsalgorithmus analysiert. Darauf
aufbauend wird dann ein neuer, schnellerer Algorithmus für die Vererbung von Geometrieparametern entwickelt und in NeuGen implementiert. Die Analyse des neuen Algorithmus und ein Vergleich mit dem bisherigen wird ebenfalls für verschiedene Testfälle
durchgeführt. Es wird gezeigt, dass der neue Algorithmus eine effizientere Generierung
von synthetischen neuronalen Netzen in drei Raumdimensionen ermöglicht. Damit leistet die vorliegende Arbeit einen kleinen Beitrag zum Fortschritt der Neuroinformatik.
1.1 Motivation
Um einen Parameter für ein Standard-Netz im Konfigurationseditor zu editieren, benötigt der bisherige Recursive-Tree-Inheritance-Vererbungsalgorithmus (RTI), der in
C++ geschrieben ist, ca. 3.7 Sekunden. NeuGen wurde in C++ und die zugehörige graphische Benutzeroberfläche in Java implementiert. Aus Kompatibilitätsgründen
wurde ein Programmierwerkzeug SWIG (Simplified Wrapper and Interface Generator)
eingesetzt, um die graphische Benutzeroberfläche und den RTI-Vererbungsalgorithmus
zu verbinden. Die Gründe für die hohe Zeit sind die exponentielle Laufzeit des RTIVererbungsalgorithmus und der Zugriff des Codes auf eine andere Programmiersprache
mithilfe der generierten Funktionen von SWIG. Darauf wird im Detail im Kapitel 4
nochmals eingegangen.
Im ersten Schritt wurde der RTI-Vererbungsalgorithmus und die für die Vererbung benötigten Klassen in Java implementiert. Infolgedessen ist die Zeit von 3.7 auf 0.5 Sekunden gesunken, gemessen auf einem Intel Pentium 4 mit einer Taktfrequenz von 3.00
GHz und 1 GB Arbeitsspeicher. Den Algorithmus auf der Java-Seite bezeichnen wir
2
1.1 Motivation
mit Default-Recursive-Inheritance-Vererbungsalgorithmus (DRI).
Der DRI-Vererbungsalgorithmus verwendet als Datenstruktur für die Geometrieparameter einen Baum. Entlang des Baumes werden Knoten ermittelt, die von anderen Knoten den Inhalt erben sollen. Die Knoten mit den zu vererbenden Geometrieparametern
werden in bestimmte Knoten kopiert und als vererbt markiert. Wird ein Wert im Konfigurationseditor geändert, müssen die Knoten, die von anderen erben, wieder bestimmt
werden. Für Bäume mit wenigen vererbten Geometrieparametern, ist DRI im Allgemeinen schnell, im anderen Fall, bei vielen vererbten Geometrieparametern ist DRI langsam, da es dazu kommen kann, dass der Baum mehrmals durchlaufen werden muss.
DRI hat im schlechtesten Fall exponentielle Laufzeit.
Ein verbesserter Default-Inheritance-Vererbungsalgorithmus (DI) wird entwickelt, der
eine geringere Laufzeit aufweist. Es wird ein Abhängigkeitsgraph aufgebaut, in dem
alle Informationen über abhängige Knoten und Erblasser zu jedem Knoten enthalten
sind. Der Abhängigkeitsgraph wird für die Vererbung von Knoten verwendet.
Für die graphische Benutzeroberfläche entwickeln wir einen vereinfachten SimplifiedInheritance-Algorithmus (SI). Dieser Algorithmus verwendet den oben erwähnten Abhängigkeitsgraphen. Im neuen Algorithmus müssen nur bestimmte Knoten durchlaufen
werden. Im schlimmsten Fall muss der ganze Graph durchlaufen werden, um abhängige
Knoten, die auf vererbt gesetzt sind, mit dem Inhalt von deren Erblasser zu überschreiben.
Danach werden die Algorithmen DI und SI für die Parameterdatei Param.neu mittels
Tests mit DRI verglichen. Dabei werden zufällige Werte in gleiche Parameterbäume
eingetragen und die Vererbung mit verschiedenen Algorithmen ausgeführt. Zum Schluss
vergleichen wir, ob die Algorithmen gleiche Bäume erzeugen.
3
1 Einleitung
1.2 Aufbau der Arbeit
Diese Bachelorarbeit besteht aus fünf Teilen. Kapitel 2 beschäftigt sich mit den Grundlagen. Dabei werden zunächst die biologischen Grundlagen der Nervenzelle und der
Kolumne, danach die Komponenten der Software NeuGen beschrieben. Anschließend
werden die notwendigen Definitionen aus der Graphentheorie gegeben. Kapitel 3 stellt
die Implementierung der neuen Vererbungsalgorithmen vor. Im darauf folgenden Kapitel 4 werden Algorithmen zum Testen der neuen Vererbungsalgorithmen vorgestellt.
Kapitel 5 schließt mit einer Zusammenfassung.
4
2 Grundlagen
Bevor wir uns näher mit den Details des Vererbungsmechanismus beschäftigen, ist es
notwendig, einige grundlegende Begriffe zu klären, die zum Verständnis der Arbeit erforderlich sind.
2.1 Neurobiologie
2.1.1 Das Gehirn
Das menschliche Gehirn besteht im Durchschnitt aus etwa 100 Milliarden Nervenzellen, die synaptisch miteinander verbunden sind, wiegt 1,35 Kilogramm und ist eines
der größten Organe des menschlichen Körpers [1, Neil A. Campbell]. Es lässt sich in
Vorderhirn (Prosencephalon) und Hirnstamm (Truncus cerebri) unterteilen.
• Das Vorderhirn umfasst die Regionen Großhirn (Telencephalon) und Zwischenhirn (Diencephalon). Das Großhirn ist in eine linke und eine rechte Großhirnhemisphäre unterteilt.
• Als Hirnstamm bezeichnet man die Regionen Mittelhirn (Mesencephalon) und
Rautenhirn (Rhombencephalon). Das Rautenhirn besteht aus drei Untereinheiten Brücke (Pons), verlängertes Rückenmarkt (Medulla oblongata) und Kleinhirn
(Cerebellum).
5
2 Grundlagen
Abbildung 2.1: Schemazeichnung der wichtigsten Gehirnstrukturen aus [6, Kandel]
Die Großhirnrinde (Cortex) ist der größte Teil des Gehirns mit einer Oberfläche von etwa 0,5 Quadratmeter. Der Cortex ist aus verschiedenen Schichten aufgebaut. Der Neocortex (Isocortex) wird in sechs übereinanderliegende Schichten (Layer) unterteilt [2,
Anja Schierloh].
Eine kortikale Kolumne (s. Abbildung 2.2) oder Säule (Column) ist eine funktionale
Verarbeitungseinheit, die alle Schichten des Cortex umfasst. Die Nervenzellen einer Kolumne sind untereinander sehr stark verbunden und bilden ein lokales Netzwerk (neuronal network). Eine Kolumne besteht aus mehreren horizontal verbundenen Minikolumnen (minicolumn), die sich durch die Schichten II-IV erstrecken. Die säulenartige Anordnung der Nervenzellen ist ein grundlegendes Strukturprinzip der Großhirnrinde.
Abbildung 2.2 zeigt synaptische Konnektivität innerhalb einer kortikalen Kolumne. Die
Dreiecke stellen Pyramidenzellen, Kreise die Sternzellen dar. Rote Bereiche zeigen erregende (exzitatorische) Synapsen, blaue Bereiche hemmende (inhibitorische) Synapsen,
Pfeile die Weiterleitungsrichtung der Signale.
6
2.1 Neurobiologie
• Schicht I (Molekularschicht) enthält keine Nervenzellen und besteht aus Fortsätzen von Pyramidenzellen.
• In Schicht II/III sind kleine Pyramidenzellen vorhanden. Schicht II bezeichnet die äußere Körnerschicht und Schicht III die äußere Pyramidenschicht.
• Schicht IV (innere Körnerschicht) ist hauptsächlich
aus Nicht-Pyramidenzellen aufgebaut.
• Schicht V (innere Pyramidenschicht) weist große
Pyramidenzellen auf.
• Schicht VI (multiforme Schicht) setzt sich
aus kleineren Pyramidenzellen
Pyramidenzellen zusammen.
und
Nicht-
Abbildung 2.2: Kolumne nach [4, Eberhard]
7
2 Grundlagen
2.1.2 Die Nervenzelle
Die Neuronen (gr. neuron, Nerv) sind die kleinsten Einheiten des Gehirns. Ihre Aufgabe
ist die Aufnahme, Verarbeitung und Weitergabe von Reizen und Informationen. Eine
Nervenzelle (s. Abbildung 2.3) besteht aus einem Zellkörper (Soma) und den davon
ausgehenden Fortsätzen, den Dendriten und dem Axon.
Abbildung 2.3: Aufbau einer Nervenzelle aus [1]
• Das Soma (gr. Körper) enthält den Zellkern (Nucleus) mit der genetischen Information. Im Soma findet der Stoffwechsel der Zelle statt.
• Die Dendriten (gr. dendron, Baum) sind kurze, dünne verzweigte Fortsätze, die
Informationen von anderen Neuronen empfangen und in Richtung des Zellkörpers
leiten.
8
2.1 Neurobiologie
• Das Axon (gr. Achse) ist ein langer Fortsatz, über den elektrische Signale vom Soma zu anderen Nervenzellen weitergeleitet werden. Der Ursprungsort des Axons
heißt Axonhügel. Die elektrischen Signale werden als Aktionspotentiale bezeichnet.
• Zwischen den Nervenzellen gibt es Verbindungsstellen. Diese Kontaktpunkte
nennt man Synapsen. Sie ermöglichen die Informationsübermittlung von einer
Nervenzelle auf die andere.
Es existieren verschiedene Typen von Nervenzellen. Sie werden nach Form, wie der Anzahl der vorhandenen Fortsätze, Größe und Funktion unterschieden. Die Nervenzellen
können in zwei Hauptklassen eingeteilt werden: die Pyramiden- und NichtpyramidenZellen.
Pyramidenzellen haben einen pyramidenförmigen Zellkörper und ein langes Axon. Von
der Spitze des Somas entspringt der apikale Dendrit. An der Grundfläche des Somas
befinden sich die basalen Dendriten.
In dieser Arbeit werden wir uns mit fünf unterschiedlichen Neuronentypen befassen:
den L2/3-Pyramidenzellen, den L4-Sternzellen, den L4-Spiny-Stellate-Zellen, den L5APyramidenzellen und den L5B-Pyramidenzellen.
9
2 Grundlagen
2.2 NeuGen
NeuGen1 ist ein Softwarepaket zur Generierung von realistischen kortikalen Neuronen
und neuronalen Netzen in 3D. Zur Generierung verwendet NeuGen Geometrieparameter, die die Struktur und andere Eigenschaften von verschiedenen Nervenzelltypen beschreiben. Ein Vererbungsmechanismus vererbt alle oder einige Geometrieparameter
von einem Basis-Neuron auf die einzelnen Neuronen-Klassen. Anhand der vererbten
Geometrieparameter können detaillierte Nervenzellen generiert werden.
Eine schematische Darstellung der Neuronen- und Neuronennetz-Erzeugung von NeuGen zeigt Abbildung 2.4. Zunächst werden die Geometrieparameter eingelesen und
durch einen Vererbungsalgorithmus verändert. Mit den vererbten Geometrieparametern
als Eingabe wird die Erzeugung von Neuronen und neuronalen Netzen gestartet.
Daten importieren
Parameterdatei
Parameter
Vererbungs-
Parameter
algorithmus
editieren
Konfigurationseditor
Vererbte Geometrieparameter
NeuronenGenerierung
Daten exportieren
hoc-Datei
dx-Datei
Abbildung 2.4: Schematische Darstellung von NeuGen
1
http://www.neugen.org/
10
3D-Visualisierung
2.2 NeuGen
2.2.1 Neuronenklassen
NeuGen unterstützt fünf verschiedene kortikale
Neuronentypen, L4-Spiny-Stellate-Zellen, L2/3Pyramidenzellen, L5A-Pyramidenzellen, L5BPyramidenzellen und L4-Star-Pyramidenzellen.
Neuron
axon
...
subclasses
L4Stellate
In den Abbildungen 2.6-2.10 sind verschieaxon
dene Nervenzelltypen zu sehen. Blaue Struk...
turen stellen das Axon, rote den Zellkörper
synapse
pyramidal
und gelbe die Dendriten dar. In Abbildung 2.5
axon
ist die Neuronen-Klassen-Hierarchie in Neu...
Gen vereinfacht dargestellt. Die Zellen vom
subclasses
L23pyramidal
Typ L4Stellate, Pyramidal, Starpyramidal eraxon
ben Geometrieparameter direkt von der Klasse
...
Neuron. L23pyramidal und L5pyramidal erben
synapse
L5pyramidal
von pyramidal, die selbst Geometrieparameter
axon
von der Klasse Neuron erbt. L5Apyramidal und
...
L5Bpyramidal erben von der Klasse L5pyramidal.
subclasses
L5Apyramidal
axon
...
synapse
L5Bpyramidal
axon
...
synapse
synapse
starpyramidal
axon
...
synapse
soma
synapse
Abbildung 2.5: NeuronenklassenAbbildung 2.6: L4-Star-Pyramidenzellen
Hierarchie
11
2 Grundlagen
Abbildung 2.7: L4-Spiny-Stellate-Zelle
Abbildung 2.8: L2/3-Pyramidenzellen
Abbildung 2.9: L5A-Pyramidenzellen
Abbildung 2.10: L5B-Pyramidenzellen
12
2.2 NeuGen
2.2.2 Parameterdateien
Die Konfiguration von NeuGen umfasst Geometrieparameter der einzelnen Neuronenklassen, die in XML-Dateiformat gespeichert werden. Eine XML-Datei lässt sich als
Baum darstellen und besteht aus Elementen, markiert durch Tags, die einen Knoten repräsentieren. Die Knoten besitzen einen Tag-Namen und eine Menge von Attributen
mit Wertzuweisungen. Attribute liefern Zusatzinformationen über einen Knoten. Die
Blätter des Baumes bilden den Inhalt (character data). Diese sind als Knoten in den
Parameterdateien string, double, integer, object mit einem Attribut key definiert. Die
object-Knoten haben ein zusätzliches Attribut classdescriptor und können Kinderknoten besitzen. Im folgenden ist eine Beispiel dargestellt: Das äußerste Element <object>
<object key=" dendrite " classdescriptor= " " >
<object key=" gen_0 " classdescriptor=" " >
<object key=" branch_angle " classdescriptor=" " >
<real key=" max " >60</real>
</object>
<object key=" siblings " classdescriptor=" siblings " >
<object key=" siblings " classdescriptor=" " >
</object>
</object>
</object>
</object>
mit dem Attribut-Name key und Attribut-Wert dendrite umfasst den gesamten Inhalt und
hat ein Kindknoten mit Attribut-Wert gen_0. Knoten gen_0 besitzt zwei Knoten mit den
Attribut-Werten branch_angle und siblings. Der Knoten branch_angle hat ein Element
real mit dem Inhalt 60. Der Attribut-Name classdescriptor mit Attribut-Wert sibling
wird vom Vererbungsalgorithmus verwendet.
Diese Geometrieparameter beschreiben detailliert die morphologische Struktur eines
Neurons. In Abbildung 2.11 ist ein vereinfachter Parameterbaum bestehend aus dem Basisneuron und der Neuronenklasse L4Stellate dargestellt. Es ist erkennbar, dass die beiden Klassen eine etwa gleiche Parameterstruktur besitzen. Die Klasse L4Stellate kann
gleichnamige Parameter vom Basisneuron erben. Die vererbbaren Parameter werden
nicht in der Datei gespeichert, um möglichst übersichtliche und leicht erweiterbare Parameterdateien zu erhalten.
13
2 Grundlagen
L4Stellate
axon
len_param
z: 375
nbranch: 26.0
nparts: 90
rad
max: 0.5
min: 0.1
dendrite
gen_0
branch_angle
max: 60
min: 30
len_param
x: 75
y: 0
z: 0
nbranch_param: 4
nparts_density: 0.25
siblings
siblings
branch_angle
max: 60
min: 30
len_param
x: 50
y: 0
z: 0
nbranch_param: 2
nparts_density: 0.25
siblings
...
rad
max: 1.5
min: 0.25
deviaton
x: 1.0
y: 1.0
z: 1.0
napiden: 0
nden: 7
soma
rad: 5
synapse
rad: 1.0
Abbildung 2.11: Parameterdatei
14
max 10 Sibling-Generationen
max 10 Sibling-Generationen
Neuron
axon
len_param
z: 400
nbranch: 10
nparts: 100
rad
max: 0.5
min: 0.1
dendrite
gen_0
branch_angle
max: 60
min: 30
len_param
x: 20
y: 0
z: 0
nbranch_param: 3
nparts_density: 0.25
siblings
siblings
branch_angle
max: 60
min: 30
len_param
x: 10
y: 0
z: 0
nbranch_param: 3
nparts_density: 0.25
siblings
...
rad
max: 1.5
min: 0.25
deviaton
x: 1.0
y: 1.0
z: 1.0
napiden: 0
nden: 11
subclasses
L4Stellate
...
pyramidal
...
starpyramidal
...
soma
rad: 5
synapse
rad: 1
2.2 NeuGen
2.2.3 Vererbungsalgorithmus
Die Aufgabe des Vererbungsalgorithmus besteht darin, die Geometrieparameter entlang
der Neuronen-Klassenhierarchie zu vererben. Der Algorithmus prüft für jede Klasse in
der Hierarchie, ob alle notwendigen Geometrieparameter vorhanden sind. Die Neuronenklassen besitzen die Parameter für das Axon, die Dendriten, das Soma und die Synapse. Beispielsweise kann man deren Radius in µ m im Konfigurationseditor einstellen.
Parameter, die nicht explizit gesetzt sind, erben von ihrer Oberklasse.
In Abbildung 2.11 kann L4Stellate von der Oberklasse Neuron die Werte der gleichnamigen Parameter erben. Parameter können innerhalb derselben Klasse vererbt werden,
z. B. enthält der Dendrit die Parameter für die Fortsätze des dendritischen Baumes.
Wenn ein Parameter des ersten Fortsatzes (gen_0) des dendritischen Baumes explizit
gesetzt ist, dann können die Parameter der nachfolgenden Fortsätze (siblings) die Parameter von ihren vorhergehenden Fortsätzen erben. Wenn Parameter des ersten Fortsatzes von ihrer Oberklasse erben, dann erben auch die nachfolgenden Fortsätzen von ihrer
Oberklasse.
2.2.4 Konfiguration
NeuGen verfügt über einen graphischen Konfigurationseditor (s. Abbildung 2.12). Mithilfe des Konfigurationseditors lassen sich Geometrieparameter einer Parameterdatei
einstellen. Die Funktionen des Konfigurationseditors sind das Laden und das Speichern
der Dateien, sowie das Editieren der Parameter. Für das Speichern und Laden von Parameterdateien in Form von XML-Dateien wird die libxml-Bibliothek2 verwendet.
Nach dem Laden einer Parameterdatei, wird ein Baum aufgebaut, auf den der Vererbungsalgorithmus angewendet wird, um die Parameter entlang der Neuronenklassen zu
vererben. Zusätzlich wird eine Datei mit Kommentaren zu den Parametern geladen.
Auch die Kommentare werden vererbt. Vor dem Speichern einer Parameterdatei werden die vererbten Parameter aus dem Baum entfernt.
Über die Baumansicht auf der linken Seite des Konfigurationseditors können die ein2
http://xmlsoft.org/
15
2 Grundlagen
zelnen Parameter per Mausklick ausgewählt werden. Die Parameter vom Typ object
enthalten andere Parameter. Für Parameter vom Typ string, double und integer können
Werte in einen Eingabedialog eingegeben werden. Eine Funktion bestimmt Parameter,
die von ihrer Oberklasse oder innerhalb der eigenen Klasse erben können. Es ist möglich diese Parameter auf vererbt (set inherited), oder auf nicht vererbt (set uninherited)
zu setzen. Falls falsche Werte wie z. B. eine Fließkommazahl anstelle einer ganzen Zahl
eingegeben werden, wird eine Fehlermeldung ausgegeben. Nach einer fehlerfreien Änderung eines Parameters werden seine abhängigen Parameter, die auf vererbt gesetzt
sind, in eigener Klasse und in den Unterklassen aktualisiert. Das bedeutet, dass durch
eine einzige Änderung viele Änderungen bewirkt werden.
Der rechte Teil zeigt die Erklärungen zu den einzelnen Parametern. Die Zuordnung zwischen den Elementen der Parameterdateien und den dazu gehörigen Erklärungen der
Kommentardateien, findet per Mausklick statt.
Abbildung 2.12: Konfigurationseditor (links ist der Baum der Parameter, rechts ist die
Erklärung)
16
2.2 NeuGen
2.2.5 Visualisierung
Die erzeugten Netze können schließlich mit Hilfe von Java3D 3 in NeuGen visualisiert
werden. NeuGen kann die Geometrie für die Software OpenDX 4 zur Visualisierung von
neuronalen Netzen in 3D exportieren.
2.2.6 Simulation
NeuGen erzeugt die Neuronengeometrie vernetzter dreidimensionaler Zellen in einem
Netzwerk und ein elektrophysiologisches Modell zur Simulation. Für die Simulation der
Signalverarbeitung auf den künstlichen neuronalen Netzen kann NeuGen hoc-Dateien
für Software NEURON 5 generieren. Die hoc-Datei mit der erzeugten Neuronengeometrie und dem detaillierten Neuronenmodell kann in NEURON eingelesen und eine
Simulation durchgeführt werden.
3
4
5
http://java.sun.com/products/java-media/3D/
http://www.opendx.org/
http://www.neuron.yale.edu/neuron/
17
2 Grundlagen
2.3 Graphentheorie
Wir verwenden im folgenden Eigenschaften und Begriffe im Zusammenhang mit Graphen, die nach [5, Ottmann u.Widmayer] wie folgt definiert sind:
• Ein gerichteter Graph G = (V, E) (englisch: directed graph) besteht aus einer
Menge V von Knoten und (englisch: vertices) und einer Menge E = V × V von
Kanten (englisch: edges).
• Ein Paar (v, v0 ) mit v, v0 ∈ V bezeichnet eine Kante e, v heißt Anfangs- und v0
Endknoten.
• Ein Weg (englisch: path) von v nach v0 ist eine endliche Folge von Kanten
0
p = (v0 , v1 ), . . . , (vm−1 , vm ) mit v = v0 , v = vm und (vi , vi+1 ) ∈ E
für 0 < i < m. m ist die Länge des Weges. v0 ist direkter Vorgänger von v1 , v1 ist
direkter Nachfolger von v0 . v1 heißt Vorgänger von vi , vi heißt Nachfolger von v1
für i > 1.
• Ein Weg p heißt Zyklus, wenn er geschlossen ist, also wenn vm = v0 gilt.
• Der Eingangsgrad (englisch: indegree) indeg(v) eines Knotens v ist die Anzahl
seiner direkten Vorgänger v0 , also indeg(v) = |{v0 |(v0 , v) ∈ E}|. Der Ausgangsgrad
(englisch: outdegree) outdeg(v) ist die Anzahl der direkten Nachfolger v0 von v0 ,
also outdeg(v) = |{v0 |(v, v0 ) ∈ E}|.
• Ein Baum t (englisch: tree) ist ein gerichteter Graph, wenn t keine Zyklen enthält
und es existiert ein Knoten v0 mit indeg(v0 ) = 0, für alle anderen Knoten v gilt
indeg(v) = 1.
• Der Nachfolger v von einem Knoten v0 heißt Sohn, oder Kindknoten.
• Der Vorgänger v von einem Knoten v0 heißt Vater, oder Elternknoten.
18
2.3 Graphentheorie
• Ein Knoten ohne Vater heißt die Wurzel des Baumes.
• Ein Knoten mit outdeg(v) = 0 heißt Blatt, andere Knoten, die weder Blätter noch
Wurzel sind nennt man innere Knoten.
• Zwei Knoten v und v0 , die Kinder desselben Vaters sind, heißen Geschwister, oder
Nachbarknoten.
• Ein n-ärer Baum ist ein Baum, bei dem jeder Knoten höchstens n Nachfolger hat.
Für n = 2 heißt der Baum ein Binärbaum.
• Seien t1 , . . . ,tm Bäume, man bekommt einen weiteren Baum, indem die Wurzeln
von t1 , . . .,tm zu Söhnen einer neuen Wurzel r (englisch: root) werden. ti (r) heißt
i-ter Teilbaum neuer Wurzel r (s. Abbildung 2.13).
• Die Höhe h (englisch: height)) eines Baumes ist die maximale Länge des Weges
von der Wurzel zu einem Blatt. Die Höhe eines leeren Baums, der aus keinem
Knoten besteht ist h = 0. In nicht leeren Bäume ist h = 1 + max{h(t1 ), . . ., h(tm)}.
• Die Tiefe d eines Knotens ist die Länge des Weges von der Wurzel zu dem Knoten. Die Knoten mit derselben Tiefe gehören zum selben Niveau l (englisch:
layer).
Wir werden innere Knoten eines Baumes als Kreise, Blätter als Quadrate und Kanten
als Pfeile zwischen zwei Knoten darstellen (s. Abbildung 2.14).
19
2 Grundlagen
Wurzel
t1
t2
...
tm
Abbildung 2.13: Teilbäume
Wurzel
innerer Knoten
Blatt
Abbildung 2.14: Darstellung eines Baumes
20
3 Vererbung der Geometrieparameter
In diesem Kapitel untersuchen wir die Vererbungsalgorithmen. Zuerst besprechen wir,
warum die Vererbung der Geometrieparameter notwendig ist. Danach werden die notwendigen Begriffe erklärt. Im Abschnitt 3.4 beschreiben wir die vorhandenen Datenstrukturen. Als Nächstes betrachten wir den bisher in NeuGen eingesetzten Vererbungsalgorithmus DRI. Danach wird der neue Algorithmus DI vorgestellt, der den Abhängigkeitsgraph erzeugt und die Knoten vererbt. Am Ende dieses Kapitels betrachten wir
den im Konfigurationseditor eingesetzten Vererbungsalgorithmus SI, der den erstellten
Abhängigkeitsgraphen von DI verwendet, um die Parameterwerte zu vererben.
3.1 Motivation
Die zur Konfiguration erforderlichen Geometrieparameter können über einen Konfigurationseditor eingegeben werden. Der Vererbungsalgorithmus sorgt dafür, dass nicht explizit gesetzte Parameterwerte von ihren Oberklassen geerbt werden.
Die gesetzten Parameterwerte sind nach dem Editieren in der Konfigurationsdatei gespeichert und die vererbten Geometrieparameter treten in der Konfigurationsdatei nicht
auf. Mehrfach vorhandene Daten in derselben Konfigurationsdatei würden dazu führen,
dass neuere Informationen an den verschiedenen Stellen eingetragen werden müssten.
Das führt dazu, dass die Konfigurationsdatei sehr groß und unübersichtlich wird. Durch
den Algorithmus bleibt die Konfigurationsdatei kompakt, neue Daten können schnell
hinzugefügt, oder entfernt werden.
Die Geometrieparameter besitzen Kommentare, die in einer Kommentardatei gespei-
21
3 Vererbung der Geometrieparameter
chert werden. Auch die Kommentare zu den Geometrieparametern werden mithilfe des
Vererbungsalgorithmus vererbt.
Im Folgenden führen wir die benötigten Konzepte, die für die Vererbung wichtig sind,
ein.
3.2 Grundbegriffe
• Ein Knoten v speichert einen Schlüssel k und einen Vererbungszustand, der angibt,
ob v den Inhalt von einem anderen Knoten geerbt hat.
• Das i-te Kind eines Knotens v bezeichnen wir mit vi für i > 0.
• Ein innerer Knoten v kann eine Typbeschreibung besitzen. Die Typbeschreibung
kann Alias oder Sibling sein.
• Ein Knoten v mit der Typbeschreibung Sibling heißt Sibling. Ein Sibling hat mindestens einen Vorgänger und einen inneren Knoten als Nachfolger.
• Ein Knoten v mit der Typbeschreibung Alias heißt Alias. Ein Alias besitzt mindestens einen Vorgänger und Nachfolger. Der Vorgänger eines Alias ist kein Kind
eines Sibling.
• Sei ein Weg p(v, v0 ) von v nach v0 durch eine Folge (v0 , . . . , vm ) von Knoten mit
v0 = v, vm = v0 und deren Schlüsselmenge S(v, v0 ) = {k0 , . . ., km } gegeben, dann
bezeichnet S(v, v0) den Schlüsselpfad von v nach v0 .
Seien S(v, v0 ), S1 (v, v0 ) und S2 (v, v0 ) Teilmengen von S(v, v0 ) für die gilt:
S(v, v0 ) enthält keine Schlüssel der Siblings und deren direkten Nachfolger.
S1 (v, v0 ) enthält keine Schlüssel der Alias Knoten.
S2 (v, v0 ) enthält keine Schlüssel der Nachfolger von Alias.
22
3.2 Grundbegriffe
• Ein Knoten w der von einem anderen Knoten v den Inhalt erben kann, heißt abhängiger Knoten. Der Knoten v, von dem der Inhalt geerbt wird, heißt Erblasser.
• Gegeben sei ein Weg p von v nach v0
0
p = (v0 , v1 ), . . ., (vm−1 , vm ) mit v = v0 , v = vm und (vi , vi+1 ) ∈ E
für 0 < i < m. Für i gerade ist vi ein Vorgänger bzw. Nachfolger von Sibling
und für i ungerade ist vi ein Sibling. Dann bezeichnen wir die Folge von Knoten
(v0 , . . .vm ) als Siblingkette.
In Abbildung 3.1 veranschaulichen wir den Sibling graphisch durch eine Raute und den
Alias durch ein Dreieck. Die gepunkteten Verbindungspfeile skizzieren einen Schlüsselpfad vom einen Knoten zum anderen Knoten. Der Schlüssel k von v, w und deren
Schlüsselpfade S(r, v), S(r, w) sind gleich. Im nächsten Unterkapitel wird diese Eigenschaft beschrieben.
r
Sibling
S(r,w)
p(r,w)
b
b
b
S(r,v)
p(r,v)
b
b
w k
b
b
b
b
v
k
Abbildung 3.1: Sibling, Siblingkette und Alias
23
3 Vererbung der Geometrieparameter
3.3 Definition von Vererbung
In diesem Abschnitt definieren wir die Vererbung von Knoten.
3.3.1 Beziehung erster Ordnung
Seien v und w Knoten mit einem minimalen gemeinsamen Teilbaum t(r) und r, v, w
sind keine Siblinge und keine Aliase. w ist ein indirekter Nachfolger von einem Sibling.
Wenn folgendes gilt:
1. S1 (r, v) = S2 (r, w),
2. |p(r, v)| < |p(r, w)|,
3. es existiert kein w0 mit 1, 2 und |p(r, w0 )| < |p(r, w)|.
dann gibt es eine Beziehung erster Ordnung zwischen v und w. Zur Beschreibung von
Beziehungen zwischen Knoten verwenden wir den Pfeil →. Wir schreiben für die Be1
ziehung erster Ordnung zwischen v und w: v −
→ w. Das bedeutet, dass w von v erben
kann.
r
S1 (r,v)
p(r,v)
k
v
S2 (r,w)
p(r,w)
w k
Abbildung 3.2: Beziehung erster Ordnung
24
3.3 Definition von Vererbung
Abbildung 3.2 zeigt ein Beispiel für die Beziehung erster Ordnung zwischen den Knoten
v und w ohne Alias im Teilbaum t(r).
a)
b)
r
r
S1 (r,v)
p(r,v)
S1 (r,v)
p(r,v)
k
v
S2 (r,w)
p(r,w)
k
S2 (r,w)
p(r,w)
k
v
w k
w
Abbildung 3.3: Beziehung erster Ordnung mit Alias im Teilbaum t(r)
In Abbildung 3.3 ist die Beziehung erster Ordnung zwischen den Knoten v und w mit
Alias im t(r) dargestellt. Im Beispiel a) wird die Teilmenge S2 (r, w) bestimmt, indem die
Schlüssel der direkten Nachfolger w von Alias aus S2 (r, w) entfernt werden. Im Beispiel
b) wird S1 (r, v) bestimmt, indem die Schlüssel von Alias aus S1 (r, v) entfernt werden.
3.3.2 Beziehung n-ter Ordnung
Gegeben seinen die Knoten v und w. Falls es zwei minimale Teilbäume t1 (r1 ), t2 (r2 )
gibt, wobei r1 , r2 keine Siblinge oder Aliases sind, sowie v aus t1(r1 ), w aus t2 (r2 ) ist,
und wenn folgende Eigenschaften gelten:
1. S1 (r1 , v) = S2 (r2 , w),
2. |S1 (r1 , v)| = |S2 (r2 , w)|,
25
3 Vererbung der Geometrieparameter
1
3. es existiert eine Beziehung r1 −
→ r2 ,
1
4. es existiert keine Beziehung v −
→ w,
1
5. es existiert ein v0 mit v0 −
→ w.
2
dann existiert eine Beziehung zweiter Ordnung zwischen v und w: v −
→ w. Die Bezien
hung n-ter Ordnung v →
− w wird analog definiert wie die Beziehung zweiter Ordnung.
r
r1
k
S1 (r, v)
r2
k
v
k
v0
S2 (r2 , w)
w k
Abbildung 3.4: Beziehungen zweiter Ordnung
In Abbildung 3.4 ist ein Beispiel für die Beziehung zweiter Ordnung zwischen den Knoten v und w dargestellt. Zwischen v0 und w existiert eine Beziehung erster Ordnung.
3.3.3 Reihenfolge der Vererbung
Wir unterscheiden drei Fälle, wann ein Knoten w von einem anderen Knoten v erbt. Sei
n
eine Beziehung v →
− w gegeben, w ist auf vererbt gesetzt und es gebe kein v(1) , n(1) < n
n(1)
mit v(1) −−→ w, von dem w erbt.
26
3.3 Definition von Vererbung
Fall 1: Sei v nicht auf vererbt gesetzt, dann erbt w von v.
n(2)
Fall 2: Sei v auf vererbt gesetzt und es existiert kein Knoten v(2) , n(2) > n mit v(2) −−→ w,
dann erbt w von v.
Fall 3: Sei v auf vererbt gesetzt und es gilt:
Fall 1
Fall 2
v
Fall 3 i)
v
n
v
n
w
n
w
w
>n
Abbildung 3.5: Reihenfolge bei der Vererbung (Fall 1-3 i)
n(3)
i) es existiert kein v(3) , n(3) mit v(3) −−→ v für n(3) > n, dann erbt w von v.
n(3)
ii) es existiert ein v(3) , n(3) mit v(3) −−→ v für n(3) > n und
a) falls v von einem Knoten u über eine Ordnung > n erbt, dann erbt w von
einem Knoten u0 über eine Ordnung > n.
b) falls v von einem Knoten u über eine Ordnung ≤ n erbt, dann erbt w von v.
a)
b)
≤n
>n
v
>n
n
>n
v
n
w
>n
w
Abbildung 3.6: Reihenfolge bei der Vererbung (Fall 3 ii)
27
3 Vererbung der Geometrieparameter
3.4 Datenstrukturen
Listen und Bäume sind die wichtigsten Datenstrukturen der Informatik. Die Liste ist eine endliche Folge von Daten-Elementen. Der Nachteil von Listen ist, dass der Suchaufwand proportional zur Anzahl der gespeicherten Elemente ist (lineare Suchzeit O(n)),
d. h. jedes Element muss bei einer erfolglosen Suche betrachtet werden. Die Suchwege
lassen sich durch baumartige Datensturkturen verkürzen.
Bevor wir die Implementierungen für die Knoten betrachten, beschäftigen wir uns mit
der Frage, wie man den Baum durchläuft.
3.4.1 Traversierung von Bäumen
Die Traversierung (Durchquerung) eines Baumes bedeutet, alle seine Knoten systematisch aufzusuchen, um an den Knoten spezielle Operationen durchführen zu können.
In Bäumen gibt es verschiedene Reihenfolgen, wie die Knoten besucht werden können
(aus [3, Robert Sedgewick]), (s. Abbildung 3.7):
• Preorder: wir besuchen den Knoten, dann alle Teilbäume die sich links vom jeweiligen Vaterknoten, dann alle Teilbäume die sich rechts davon befinden.
• Inorder (bei Binärbäumen): wir besuchen den linken Teilbaum, dann den Knoten
und dann den rechten Teilbaum.
• Postorder: wir besuchen alle Teilbäume die sich links vom jeweiligen Vaterknoten, dann alle Teilbäume die sich rechts davon befinden und dann den Knoten
selbst.
• Levelorder: wir besuchen den Knoten, dann alle Wurzel der Teilbäume die sich
links von ihm, dann alle Wurzel der Teilbäume die sich rechts von ihm befinden,
dann die nächste Ebene.
Der Algorithmus für Postorder-Traversierung kann wie folgt programmiert werden:
28
3.4 Datenstrukturen
Preorder:
1
1, 2, 3, 4, 5, 6, 7, 8, 9
Inorder:
3, 2, 4, 1, 7, 6, 8, 5, 9
Postorder:
5
2
3, 4, 2, 7, 8, 6, 9, 5, 1
Levelorder:
1, 2, 5, 3, 4, 6, 9, 7, 8
3
4
9
6
7
8
Abbildung 3.7: Traversieren von Bäumen
Algorithm 3.1 postorder_traverse(v)
Input: v
if v == null then
2:
return v
3: if v is leaf then
1:
return null
5: for i = 1 to outdeg(v) do
6:
vi ← v.getChild(i)
7:
postorder_traverse(vi )
4:
8:
doSomething(v)
29
3 Vererbung der Geometrieparameter
Die Funktion 3.1 bekommt als Argument den Knoten eines Baumes und traversiert zunächst die Unterbäume eines Knotens, bevor der Knoten selbst von der Funktion doSomething bearbeitet wird. Wenn wir den Aufruf von doSomething vor die for-Schleife
mit dem rekursiven Aufruf verschieben, bekommen wir die Preorder-Traversierung. Es
fällt auf, dass wir im Baum in die Tiefe gehen und als erstes den Knoten unten links bearbeiten. Deswegen werden alle Verfahren außer levelorder als Tiefensuche (engl. depth
first search) bezeichnet.
Der Zeitaufwand bei der Tiefensuche ist O(n) für einen Baum mit n Knoten, da jeder
Knoten mindestens einmal besucht wird. Im schlimmsten Fall (entarteter Baum) sind
insgesamt n − 1 Aufrufe von postorder_traverse(vi ) für den Durchlauf notwendig.
Durch die Verwendung einer Liste können wir die Rekursion vermeiden (s. Funktion
3.2).
Algorithm 3.2 levelorder_traverse(v)
Input: v
1: LinkedList q
q.addFirst(v)
3: while q.size() > 0 do
4:
v ← q.getLast()
2:
5:
6:
7:
8:
9:
10:
q.removeLast()
doSomething(v)
for k ← 0 to outdeg(v) do
vi ← v.getChild(i)
if v! = null && v != leaf then
q.addFirst(vi )
Der Baum wird in Levelorder-Reihenfolge durchlaufen, indem wir eine FIFO-Datenstruktur benutzen (engl. first in first out (als erster rein, als erster raus)). Die Knoten
werden am Anfang der Liste eingefügt und in der while-Schleife der letzte Knoten geholt. Die Knoten des Baumes werden von oben nach unten und von links nach rechts
besucht. Hier gehen wir nicht zuerst in die Tiefe, sondern in die Breite (engl. breadth
first search). Die Laufzeit ist O(n), da jeder Knoten einmal besucht wird.
30
3.4 Datenstrukturen
3.4.2 Implementierungen
Die Daten der Parameterdatei werden in einer Baumstruktur gespeichert. Jeder Knoten
enthält elementare Daten wie Knotenname, Knotenwert und eventuelle Kinderknoten.
Ein DefaultXMLObject-Objekt ist eine Wurzel oder ein innerer Knoten, der eine Liste
von Instanzen der Klasse DefaultXMLNode enthält. Ein DefaultXMLNode-Objekt kann
ein innerer Knoten oder ein Blatt sein. Für die Liste benutzen wir die Java-Klasse ArrayList.
Eine Übersicht der für die Knoten verwendeten Klassen ist in Abbildung 3.8 zu sehen. In der Abbildung ist ein Klassendiagramm mit den Klassen, ihren Attributen und
Methoden dargestellt. Die Vererbungsbeziehung zwischen einer allgemeineren Klasse
(Oberklasse) und einer spezialisierten Subklasse (Unterklasse) wird durch eine Linie
mit einem Dreieck repräsentiert. Die Spitze des Dreiecks zeigt auf die Oberklasse. Dieselbe Darstellung wird für die Schnittstellenvererbung benutzt, wenn Oberklasse und
Unterklasse eine Schnittstelle (Interface) ist. Wenn eine Schnittstelle von einer normalen Klasse implementiert wird, dann wird die Linie gestrichelt gezeichnet.
bildet die Basisschnittstelle und dient dazu, den Zugriff auf ein
Knotenobjekt festzulegen. Die Schnittstelle enthält folgende Methoden. getParent() liefert den Elternknoten, getName() den Schlüssel, isA() den Typ, isInherited() den VererXMLNodeInterface
bungsstatus; setInherited() trägt den Status als vererbt, setUninherited() als nicht vererbt
ein.
DefaultXMLNode
implementiert die Schnittstelle XMLNodeInterface. Die Klasse
DefaultXMLNode repräsentiert ein Knotenobjekt. Ein Knoten besitzt einen Schlüssel,
einen Elternknoten und einen Vererbungsstatus. Die Klasse enthält zusätzlich die Methode getPath(), die den Pfad von der Wurzel bis zum Knoten liefert.
sind abgeleitete Klassen, sie erben alle Eigenschaften von der Vaterklasse DefaultXMLNode. Die Klassen repräsentieren die Blatt-
DefaultXMLString, -Real, -Bool, -Int
31
3 Vererbung der Geometrieparameter
Abbildung 3.8: Übersicht der verwendeten Klassen
32
3.4 Datenstrukturen
objekte. Sie unterscheiden sich von DefaultXMLNode dadurch, dass sie zusätzlich einen
Wert und einen Typ für ein Blatt speichern. Die Klasse enthält zusätzlich die Methode
convert(), die verwendet wird, um ein DefaultXMLNode in ein -String, -Real, -Bool oder
-Int umzuwandeln. Sie überprüft, zu welcher Klasse ein Objekt gehört.
ist abgeleitet von XMLNodeInterface, die Schnittstelle erbt alle Methodendefinitionen der Basisschnittstelle. Sie definiert folgende Methoden. addChild() fügt einen neuen Kindknoten an das Ende der Liste an, removeChild() entfernt
XMLObjectInterface
einen beliebigen Knoten, getChild(i) ermöglicht indizierten Zugriff auf die Kinder, getChildrenCount() ermittelt die Anzahl der Kinder, hasUninheritedChildren() kann abfragen, ob ein Teilbau vererbte Kinder besitzt, addDependants() fügt für einen Knoten
einen abhängigen Knoten in eine Menge ein, getAllDependants() liefert alle abhängigen
Knoten, getObjectClassName() ermittelt den Klassennamen eines Knotens und getSupreme() liefert den Erblasser.
DefaultXMLObject
implementiert die Schnittstelle XMLObjectInterface. Die Klasse
repräsentiert die inneren Knoten. Ein innerer Knoten speichert seine Kinder in einer
ArrayList children. Die Knoten von denen geerbt wird und die erben werden, werden
als eine sortierte Menge von geordneten Paaren gespeichert. Für die Menge benutzen
wir die Java-Klasse TreeMap, die alle Schlüssel in einem Binärbaum sortiert. Diese Datenstruktur verbindet einen Schlüssel mit einem Wert, vergleichbar beispielsweise mit
einem Telefonbuch. Ein Name ist ein Schlüssel und die Nummer der Wert. In unserem Fall wird in die TreeMap die Ordnung (Schlüssel) und die Menge der abhängigen
Knoten (Wert) eingetragen. Die Menge der abhängigen Knoten wird in der Java-Klasse
HashSet, die keine Duplikate enthält, gespeichert. Die Erblasser werden direkt mit der
Ordnung in die TreeMap eingetragen.
Die Klasse enthält folgende Methoden. printAllDependants(), die Ausgabe aller Knoten, die von einem Knoten erben könnten, unterstützt uns beim Debuggen, convert()
kann abfragen, ob ein Knoten ein innerer Knoten ist, getRoot() liefert die Wurzel.
33
3 Vererbung der Geometrieparameter
3.5 Default-Recursive-InheritanceVererbungsalgorithmus
In diesem Kapitel wird der bisher in NeuGen implementierte Default-RecursiveInheritance-Vererbungsalgorithmus (DRI) beschrieben. Bevor wir die Funktion für das
Vererben von Knoten betrachten, betrachten wir zunächst die Funktionen für das Anhängen und Vervollständigen von Knoten: copyAppend und complete.
3.5.1 Anhängen von Knoten
Mit Hilfe der rekursiven Funktion 3.3 copyAppend wird eine Kopie eines Knotens erzeugt und an einen anderen Knoten angehängt. Die Funktion bekommt zwei Knoten
v und w der Teilbäume t(v) und t(w) als Argumente. Beide Teilbäume werden mittels
der rekursiven Tiefensuche in Preorder-Reihenfolge durchlaufen und es wird nach den
Schlüsseln der Nachfolger von v in w gesucht. Wenn ein Schlüssel nicht gefunden wird,
dann erzeugen wir eine Kopie des gesuchten Knotens und hängen sie an den entsprechenden Vaterknoten an. Dabei werden folgende Fälle unterschieden: v ist ein Alias, v
ist ein Blatt, v ist ein innerer Knoten und kein Alias. (s. Zeilennummern in der Funktion
3.3)
1-9: Fall 1 (s. Abbildung 3.9): v ist ein Alias, dann werden alle seine Kinder vi durchlaufen. Wir suchen einen Knoten w(k) von w mit demselben Schlüssel k wie vi .
Die Funktion wird rekursiv für alle vi und w aufgerufen, um vi an w anzuhängen,
wenn w(k) nicht gefunden wird. Andernfalls wird die rekursive Funktion complete (s. 3.4) aufgerufen. In dieser Funktion prüfen wir, ob alle Knoten von t(vi )
in t(w) enthalten sind, um fehlende Knoten durch den Aufruf von copyAppend
anzuhängen.
10-11: Fall 2 (s. Abbildung 3.10a): v ist ein Blatt, dann erzeugen wir eine Kopie von v
und hängen sie an w.
34
3.5 Default-Recursive-Inheritance-Vererbungsalgorithmus
12-16: Fall 3 (s. Abbildung 3.10b): v ist ein innerer Knoten und kein Alias, dann wird
ein Knoten v0 mit dem Schlüssel und Inhalt von v erzeugt und an w angehängt.
Danach wird die Funktion rekursiv für alle vi und v0 aufgerufen, um beide Knoten
zu bearbeiten.
Algorithm 3.3 copyAppend(v, w)
Input: v and w
1: if v is Alias then // Fall 1
2:
for i = 1 to outdeg(v) do
3:
vi ← v.getChild(i)
4:
5:
6:
k ← vi .getKey()
w(k) ← w.getChild(k)
if w(k) == null then
copyAppend(vi , w)
8:
else
9:
complete(vi , w(k))
10: else if v is leaf then // Fall 2
7:
new Leaf(v.getKey(), w, v.getValue())
12: else if v is inner node then // Fall 3
13:
v0 ← new Node(v.getKey(), w, v.getType())
14:
for i = 1 to outdeg(v) do
11:
15:
16:
vi ← v.getChild(i) // hänge vi an v0
copyAppend(vi , v0 )
v
w
1
2
3
4
w
=⇒
w
1
3
=⇒
1
2
3
4
Abbildung 3.9: Anhängen von Knoten (Fall 1)
35
3 Vererbung der Geometrieparameter
Abbildung 3.9 veranschaulicht die Arbeitsweise von copyAppend. Der Alias v wird nicht
an w angehängt, stattdessen sucht die Funktion nach den Schlüsseln der Nachfolger von
v in w. Wenn kein Schlüssel gefunden wird, dann wird der gesuchte Knoten erzeugt und
an t(w) angehängt.
v 1
w
=⇒ w
v 1
w
=⇒ w
1
(a) Fall 2
1
(b) Fall 3
Abbildung 3.10: Anhängen von Knoten (Fall 2 und Fall 3)
3.5.2 Vervollständigen von Knoten
Die rekursive Funktion 3.4 complete sucht nach den Schlüsseln eines Teilbaumes in
einem anderen Teilbaum. Dabei werden die Teilbäume mithilfe der rekursiven Tiefensuche in Preorder-Reihenfolge durchlaufen. Wird ein Schlüssel nicht gefunden, dann
wird durch Aufruf der Funktion copyAppend eine Kopie des gesuchten Knotens erzeugt
und angehängt. Die Funktion complete bekommt zwei Knoten v und w als Argumente.
Die beiden Knoten werden verglichen und es ergeben sich folgende Fallunterscheidungen:
1-2: Fall 1: w ist ein Blatt, dann terminiert der Aufruf.
3-6: Fall 2 (s. Abbildung 3.11): w ist ein Alias, dann werden alle seine Kinder vervollständigt, d. h. v wird mit wi verglichen.
7-21: Fall 3 (s. Abbildung 3.12): w ist eine innerer Knoten und kein Alias, dann wird
getestet, ob ein Nachfolger vi ein Alias ist. Ist das der Fall, dann wird geprüft,
ob die Schlüssel der Kinder von Alias in w enthalten sind. Ansonsten wird nach
36
3.5 Default-Recursive-Inheritance-Vererbungsalgorithmus
den Knoten w(k) mit den gleichen Schlüsseln wie vi in w gesucht. Die Funktion
copyAppend wird aufgerufen, um eine Kopie von vi zu erzeugen und sie an w
anzuhängen, wenn der Schlüssel nicht gefunden wird. Wenn der gesuchte Knoten
existiert und kein Alias ist, dann wird vi mit w(k) verglichen. Falls w(k) ein Alias
ist, dann wird vi mit allen Nachfolgern des Alias verglichen.
Algorithm 3.4 complete(v, w)
Input: v and w
1: if w is leaf then // Fall 1
2:
return
3: if w is Alias then // Fall 2
for i = 1 to outdeg(w) do
5:
wi ← w.getChild(i)
6:
complete(v, wi )
7: else // Fall 3
4:
8:
9:
10:
11:
12:
13:
14:
15:
16:
17:
18:
19:
20:
21:
for i = 1 to outdeg(v) do
vi ← v.getChild(i)
if vi is Alias then
complete(vi , w); continue
k ← v.getKey()
w(k) ← w.getChild(k)
if w(k) == null then
copyAppend(vi , w)
else if !w(k) is Alias then
complete(vi , w(k))
else
for j = 1 to outdeg(w(k)) do
w j ← w(k).getChild( j)
complete(vi , w j )
Die Abbildung 3.11 zeigt ein Beispiel, wie der Teilbaum t(w), mit w ist ein Alias, vervollständigt wird. Das Verfahren sucht in den Kindern von Alias nach den Knoten mit
den Schlüsseln 2 und 3. Durch den Aufruf von copyAppend werden Kopien der Knoten
mit den Schlüsseln 2, 3 für alle Nachfolger von Alias erzeugt und angehängt.
37
3 Vererbung der Geometrieparameter
w
v 1
2
3
4
5
w
1
w
1
=⇒
=⇒
2
3
2
3
2
3
4
5
4
5
4
5
Abbildung 3.11: Vervollständigen von Knoten (Fall 2)
v 1
w 1
w 1
=⇒
2
2
3
3
Abbildung 3.12: Vervollständigen von Knoten (Fall 3)
38
1
3.5 Default-Recursive-Inheritance-Vererbungsalgorithmus
3.5.3 Vererben von Knoten
Sehen wir uns die rekursive Funktion 3.5 processNode für das Vererben von Knoten
in einem Baum an. Der Baum wird mithilfe der rekursiven Tiefensuche in PostorderReihenfolge durchlaufen. Die Funktion bekommt einen Knoten v als Argument.
1-14: Wenn v kein Blatt ist, dann werden alle seine Kinder vi besucht und geprüft, ob
einer von ihnen ein Sibling ist. Die Funkion processNode wird für vi aufgerufen,
wenn er kein Sibling ist, ansonsten für die Kinder des Siblings. Wenn vi ein Sibling ist, wird s = vi initialisiert, um anschließend testen zu können, ob ein Sibling
gefunden wurde.
15-27: Wird ein Sibling s gefunden, dann werden alle seine Nachbarknoten vi und alle seine Kinder s j durchlaufen. Es wird nach dem Kind s(k) mit dem gleichen
Schlüssel k wie vi in s j gesucht. Wenn der Schlüssel nicht gefunden wird, dann
wird die rekursive Funktion copyAppend aufgerufen und die Knoten vi , s j übergeben. Ansonsten, wenn der Schlüssel gefunden wird, wird die rekursive Funktion
complete aufgerufen und die Knoten vi , s(k) übergeben.
28-30: Zum Schluss wird processNode auf alle Nachfolger des Siblings angewendet. Im
Falle einer Siblingkette können die Knoten von oben nach unten vererbt werden.
Wenn ein Knoten vererbt wird, dann muss dieser auch in die Unterbäume vererbt werden. Deswegen wird die Funktion für die Kinder des Siblings nochmal
aufgerufen.
In Abbildung 3.13 ist die Arbeitsweise von processNode an einem Beispiel veranschaulicht. Die Knoten v1 und v2 werden an die Kinder des Siblings vererbt. Die Funktion
copyAppend erzeugt die zu vererbenden Knoten und hängt sie an die Teilbäume t(s1)
und t(s2) an. Die Knoten von v2 werden an die Kinder des Alias angehängt.
Die nachfolgende rekursive Funktion processNode implementiert das Verfahren.
39
3 Vererbung der Geometrieparameter
Algorithm 3.5 processNode(v)
Input: v
1: if v == null then
return v
3: if v is leaf then
4:
return null
2:
s ← null
6: for i = 1 to outdeg(v) do
7:
vi ← v.getChild(i)
8:
if vi is Sibling then
5:
9:
10:
11:
12:
s ← vi
for j = 1 to outdeg(s) do
s j ← s.getChild( j)
processNode(s j )
else
14:
processNode(vi )
15: if s! = null then // Sibling gefunden
13:
16:
17:
18:
19:
20:
21:
22:
23:
24:
25:
26:
27:
28:
29:
30:
31:
40
for i = 1 to outdeg(v) do
vi ← v.getChild(i)
if v(i) is Sibling then
continue
for j = 1 to outdeg(s) do
s j ← s.getChild( j)
k ← v.getKey()
s(k) ← s j .getChild(k)
if s(k) == null then
copyAppend(vi , s j )
else
complete(v(i), s(k))
for i = 1 to outdeg(s) do // Vererbung auf Teilbäume von Sibling
si ← s.getChild(i)
processNode(si )
return v
3.5 Default-Recursive-Inheritance-Vererbungsalgorithmus
s1
v
v1 1
3
v2 2
4
5 s1
s2
⇒ 1
s
s2
3
2
4
5
⇒ 1
3
2
2
4
5
5
Abbildung 3.13: Vererben von Knoten
3.5.4 Umkehrung der Vererbung
Die Funktion 3.6 für die Umkehrung der Vererbung reverseProcess wird verwendet,
um vererbte Knoten eines Baumes zu entfernen. Dabei wird der Baum mithilfe der
rekursiven Tiefensuche in Postorder-Reihenfolge durchlaufen. Ein innerer Knoten wird
entfernt, wenn er keine Kinder hat, und er kein Nachfolger eines Alias oder Sibling
ist.
1-3: Zuerst prüfen wir, ob der Knoten v ein Blatt oder ein innerer Knoten ist. Wenn der
Knoten ein Blatt und vererbt ist, dann wird er entfernt.
4-13: Falls v ein innerer Knoten ist, dann rufen wir die Funktion rekursiv für alle seine
Kinder auf. Wir löschen v, wenn er keine Kinder hat und kein Nachfolger eines
Alias oder Sibling ist.
41
3 Vererbung der Geometrieparameter
Algorithm 3.6 reverseProcess(v)
Input: v
1: if v is leaf && v.isInherited() then
v.getParent().removeChild(v)
3:
return null
4: if v is inner node then
2:
5:
6:
7:
8:
9:
10:
11:
12:
13:
for i = 0 to outdeg(v) do
vi = v.getChild(i)
reverseProcess(vi )
if outdeg(v) = 0 then
w = v.getParent()
if w! = root && !w is Alias && !w is Sibling then
w.removeChild(v)
return null
return v
3.5.5 Implementierungen
Abbildung 3.14: Klasse DefaultRecursiveInheritance
42
3.5 Default-Recursive-Inheritance-Vererbungsalgorithmus
Die Klasse DefaultRecursiveInheritance (s. Abbildung 3.14) wurde in Java implementiert. Sie dient der Vererbung von Knoten und enthält folgende Methoden. process bekommt einen fertigen Baum und ruft die Methode processNode auf, um die Knoten zu
vererben, isAlias und isSibling fragen ab, ob ein Knoten ein Sibling oder Alias ist, copyAppend erzeugt eine Kopie von einem Knoten und hängt sie an einen anderen Knoten
an. Die Methode complete vervollständigt fehlende Knoten eines Knotens mit dem entsprechenden Knoten eines anderen Knotens. Die Methode reverseProcess entfernt alle
vererbten Knoten des Baumes.
3.5.6 Kosten der Funktionen
Die Funktion complete wird auf zwei Teilbäume t(v),
t(w) eines n-ären Baumes mit N inneren Knoten angewendet. Die Laufzeit ist linear,
Vervollständigen von Knoten
weil jeder Knoten aus t(w) genau einmal besucht wird. Im schlimmsten Fall können für
jeden inneren Knoten aus t(v) mehrere Aliase in t(w) existieren. In Abbildung 3.15 sind
die Teilbäume t(v) und t(w) dargestellt.
w
v 1
2
1
3
2
3
2
3
Abbildung 3.15: Aufwand für complete
Die Anzahl der Knoten des Niveaus l mit l ≥ 1 von t(v) bezeichen wir mit pl und
von t(w) mit ql . Um den Teilbaum t(w) in dem Niveau l zu vervollständigen, wird die
43
3 Vererbung der Geometrieparameter
Funktion pl · ml , wobei ml =
1
pl (q2l−1 + q2l ),
mal aufgerufen.
p 1 · m1 + p 2 · m2 + · · · + p l · ml = q 1 + q 2 + · · · + q l
h
=
∑ ql
l=1
= O(N)
In einem n-ären Baum mit Höhe h gibt es auf dem Niveau l maximal nl−1 Knoten. Die
Funktion benötigt für zwei Teilbäume
h
∑n
l=1
l−1
h
(n2l−2 + n2l−1 )
= ∑ (n2l−2 + n2l−1 )
·
l−1
n
l=1
Aufrufe. Zum Beispiel benötigen wir für den Baum aus Abbildung 3.15
2
∑ (22l−2 + 22l−1) = 3 + 12 = 15
l=1
Aufrufe von complete. Davon sind drei Aufrufe für den Schlüssel 1 und jeweils 6 für
die Schlüssel 2 und 3. Das Ergebnis ist die Anzahl der Knoten von t(w). Die Laufzeit
wird dominiert von der Anzahl N der Knoten des gesamten Baumes. Der Aufwand ist
von der Größenordnung O(N).
Vererben von Knoten
Die Laufzeit zum Ausführen der Funktion processNode, hängt
von der Anzahl n der Siblinge des Baumes ab. Im schlimmsten Fall ist der Baum mit
N Knoten eine Siblingkette mit maximal n = N−1
2 Siblingen. In Abbildung 3.16 ist eine Siblingkette mit der Anzahl der Funktionsaufrufe dargestellt. Die Siblingkette wird
in Postorder-Reihenfolge traversiert. Wenn ein Sibling gefunden wird, dann wird die
Funktion für die Kinder des Siblings (Schlüssel 2,3,4) einmal aufgerufen. Dann können
die notwendigen Anweisungen ausgeführt werden, wie das Anhängen oder das Vervollständigen von Knoten. Im letzten Schritt wird die Funktion wieder für die Kinder des
Siblings aufgerufen, um die Knoten, die zunächst oben vererbt werden, nach unten zu
vererben.
44
3.5 Default-Recursive-Inheritance-Vererbungsalgorithmus
1 1
2 2
4 3
8 4
Abbildung 3.16: Aufwand für processNode
1 + 2 + 4 + . . . = 20 + 21 + · · · + 2n
n
=
∑ 2i
i=0
=
1 − 2n+1
1−2
= 2n+1 − 1 = O(2
N+1
2
− 1)
= O(2N )
Im schlimmsten Fall ist der Gesamtaufwand von der Größenordnung O(2N · N), wenn
die Funkion copyAppend oder complete aufgerufen wird.
Die Laufzeit zum Ausführen der Funktion copyAppend für
einen n-ären Baum mit N Knoten ist im schlechtesten Fall von der Größenordnung
Anhängen von Knoten
O(N), wenn ein Teilbaum t1 mit N −3 Knoten erzeugt und angehängt wird (s. Abbildung
3.17).
45
3 Vererbung der Geometrieparameter
b
=⇒
b
t1
t1
b
0
t1
Abbildung 3.17: Aufwand für copyAppend
Die Laufzeit zum Ausführen der Funktion reverseProcess ist O(N), da der Baum, um vererbte Knoten zu löschen, in Postorder-Reihenfolge
durchlaufen wird.
Umkehrung der Vererbung
Die Gesamtlaufzeit des DRI-Vererbungsalgorithmus ist von der Laufzeit der Funktion
processNode abhängig. Folglich ist die Gesamtlaufzeit des DRI-Vererbungsalgorithmus
O(2N · N).
46
3.6 Default-Inheritance-Vererbungsalgorithmus
3.6 Default-Inheritance-Vererbungsalgorithmus
In diesem Kapitel wird ein neuer Default-Inheritance-Vererbungsalgorithmus (DI) für
die Vererbung von Knoten beschrieben. Der DRI-Vererbungsalgorithmus speichert keine Informationen über die abhängigen Knoten und hat eine exponentielle Laufzeit
O(2N · N). Der DI-Vererbungsalgorithmus erzeugt einen Abhängigkeitsgraphen, indem
alle Beziehungen erster und zweiter Ordnung zwischen den Knoten gespeichert werden,
und hat eine lineare Laufzeit.
3.6.1 Programmablauf
Der DI-Vererbungsalgorithmus wird in folgenden Schritten durchgeführt (s. Tabelle
3.1). Zuerst bestimmen wir alle Siblingketten eines Baumes, um in den Siblingketten die
Siblingketten bestimmen
Teilbäume ergänzen und die Beziehungen erster Ordnung speichern
Wurzel-Siblingketten bestimmen
Teilbäume für die Beziehungen zweiter Ordnung bestimmen
Teilbäume ergänzen und die Beziehungen zweiter Ordnung speichern
Blätter vererben
Tabelle 3.1: Programmablauf
Beziehungen erster Ordnung zwischen den Knoten zu dokumentieren. Dann vererben
wir innere Knoten in den Teilbäumen der Siblingketten und speichern die Beziehungen
erster Ordnung zwischen den Knoten. Als Nächstes bestimmen wir die Siblingketten,
die nicht von anderen Siblingketten abhängen. Danach bestimmen wir Teilbäume für die
Beziehung zweiter Ordnung. Anschließend speichern wir die Beziehungen zweiter Ordnung. Zum Schluss vererben wir die Blätter. Auf Details wird in folgenden Abschnitten
eingegangen.
47
3 Vererbung der Geometrieparameter
3.6.2 Bestimmung von Siblingketten
Die Funktion 3.7 für die Bestimmung von Siblingketten getDependanceTrees durchläuft den Baum in Level-Order-Reihenfolge. Zur Implementierung der Traversierung
verwenden wir eine Warteschlange (Queue) q. Dabei suchen wir nach Siblingketten und
fügen sie in die Warteschlange sibChainQueue ein. Die zweite Warteschlange q2 wird
verwendet, um die Siblingketten zu durchlaufen und die Beziehungen erster Ordnung
zwischen dem Vorgänger und den Nachfolgern des Siblings zu speichern. Für die Warteschlangen q und q2 wird die Java-Klasse LinkedList und für sibChainQueue ArrayList
verwendet.
1-3: Nachdem die Warteschlange initialisiert ist, wird die Wurzel des Baumes am Anfang der Warteschlange eingefügt. Solange noch Knoten in der Warteschlange
liegen, wird der letzte Knoten aus der Warteschlange geholt.
4-7: Um einen Knoten nicht mehrmals zu bearbeiten, wird geprüft, ob er abhängige
Knoten hat. Falls der Knoten keine abhängigen Knoten hat, wird die zweite Warteschlange initialisiert und der Knoten hinzugefügt. Solange noch Knoten in der
zweiten Warteschlange sind, wird die nachfolgende Anweisung durchgeführt.
9-17: Der letzte Knoten wird aus der zweiten Warteschlange geholt und entfernt. Wenn
ein Sibling s gefunden wird, dann wird die Beziehung erster Ordnung zwischen
dem Vorgänger und den Nachfolgern von s gespeichert. Die Kinder des Siblings
werden in die zweite Wartschlange eingefügt, um die Siblingkette zu durchlaufen. Falls ein Knoten der Anfang einer Siblingkette ist, dann wird er in die Warteschlange sibChainQueue hinzugefügt. Um die Teilbäume der Siblingketten zu
besuchen, werden die Knoten in die erste Warteschlange hinzugefügt.
18-21: Um den Baum in Level-Order-Reihenfolge zu traversieren, werden die Nachfolger des Knotens, die keine Siblinge sind, in die Warteschlange q eingefügt.
48
3.6 Default-Inheritance-Vererbungsalgorithmus
Algorithm 3.7 getDependanceTrees(r, sibChainQueue)
1: Queue q; q.addFirst(r) // Liste zum Traversieren des Baumes
2: while q.size() > 0 do
3:
4:
5:
6:
7:
8:
9:
10:
11:
12:
13:
14:
15:
16:
17:
18:
19:
20:
21:
v ← q.getLast(); q.removeLast()
H ← v.getAllDependants() // Menge der abhängigen Knoten
if H.size() = 0 then // verhindert Duplikate in der Queue sibChainQueue
Queue q2; q2.addFirst(v) // Liste zum Traversieren der Siblingketten
while q2.size() > 0 do
w ← q2.getLast(); q2.removeLast()
if w! = v then // füge nicht besuchte Knoten in q ein
q.addFirst(w)
s ← getSibChild(w)
if s! = null then
for i = 1 to outdeg(s) do
si ← s.getChild(i); q2.addFirst(si )
1
w.addDepentants(1, si ) // speichere w −
→ si in H von w
if w is SiblingChain then // w ist eine Siblingkette
sibChainQueue.addFirst(w) // Siblingketten des Baumes
for i = 1 to outdeg(v) do
vi ← v.getChild(i)
if !vi is Sibling then
q.addFirst(vi )
49
3 Vererbung der Geometrieparameter
1
1
1
Abbildung 3.18: Beziehungen erster Ordnung zwischen dem Vorgänger und den Nachfolgern des Siblings
3.6.3 Bestimmung und Ergänzung von abhängigen Teilbäumen
Im vorherigen Verfahren getDependanceTrees werden alle Knoten die, der Anfang einer
Siblingkette sind, in die Warteschlange sibChainQueue eingefügt und die Beziehung
erster Ordnung zwischen dem Vorgänger und den Nachfolgern des Siblings gespeichert.
Für die Vererbung ist es notwendig, die Beziehungen erster und zweiter Ordnung für die
Knoten in den Teilbäumen der Siblingketten zu speichern, um alle Abhängigkeiten zu
dokumentieren. Die Funktion 3.8 getDependancies erhält zwei Parameter, die Ordnung
n und die Warteschlange sibChainQueue mit den Siblingketten.
1-10: Zuerst wird die Warteschlange zum Traversieren der Siblingketten und deren Teilbäume initialisiert, dann werden die Siblingketten, die in sibChainQueue liegen,
durchlaufen. Danach wird die Menge der abhängigen Knoten des aktuellen Knoten ermittelt. Falls die Kinder des Knotens keine Siblinge und keine Blätter sind,
werden sie zur childrenQueue zum Speichern der Beziehung und zur q zum Traversieren der Teilbäume hinzugefügt.
11-15: Hier wird die Menge der abhängigen Knoten durchlaufen. Für jeden abhängigen
Knoten werden die Kinder des Erblassers durchlaufen.
50
3.6 Default-Inheritance-Vererbungsalgorithmus
16-25: Fall 1: Falls das Kind v j des Erblassers v ein Alias ist und der abhängige Knoten w kein Kind w(k) mit dem gleichen Schlüssel wie v j hat, werden die Kinder
des Alias erzeugt und in w angehängt. Dann wird die Funktion getDependanceTrees(w(k), sibChainQueue) aufgerufen. Wenn w(k) eine Siblingkette ist, wird
sie zur sibChainQueue hinzugefügt und die Beziehungen erster Ordnung in ihr
gespeichert. Wenn v j eine Siblingkette ist, wird w(k) zur sibChainQueue hinzugefügt. Danach werden die Beziehungen der Ordnung n zwischen den Knoten
gespeichert.
26-34: Fall 2: Ansonsten, wenn w(k) nicht existiert, wird v j kopiert und an w angehängt.
Fall 3: Falls w(k) existiert und kein Alias ist, wird die Beziehung zwischen den
Knoten gespeichert.
Fall 4: Ist w(k) ein Alias, so wird die Beziehung zwischen dem Kind des Erblassers und den Kindern des Alias gespeichert.
Fall 1
n
v
w
n
v
n
=⇒ v j
vj
w
1
2
n
Fall 2
1
2
1
2
3
4
3
4
n
v
w
n
v
vj 1
=⇒ v j 1
2
2
n
w
1
Abbildung 3.19: Anhängen von Knoten und Speichern von Beziehungen (Fall 1 und
Fall 2)
51
3 Vererbung der Geometrieparameter
Algorithm 3.8 genDependancies(n, sibChainQueue)
1: Queue q, childrenQueue
for i = 1 to sibChainQueue.size() do // Siblingketten
3:
p ← sibChainQueue.get(i); q.addFirst(p)
4:
while q.size() > 0 do // Teilbäume der Siblingkette
2:
5:
6:
7:
8:
9:
10:
11:
12:
13:
14:
15:
16:
17:
18:
19:
v ← q.getLast(); q.removeLast()
H ← v.getAllDependants(n) // Abhängige Knoten n-ter Ordnung
for i = 1 to outdeg(v) do
vi ← v.getChild(i)
if !vi is leaf && !is Sibling then
q.addFirst(vi ); childrenQueue.addFirst(vi )
for all nodes in H do // Abhängige Knoten von v
w ← H.next(); q.addFirst(w)
for j = 0 to childrenQueue.size() do // Kinder von v
v j ← childrenQueue.get( j); k ← v j .getKey();
w(k) ← w.getChild(k)
if v j is Alias && w(k) == null then // 1.Fall
for i = 1 to outdeg(v j ) do
v ji ← v j .getChild(i); k ← v ji .getKey();
w(k) ← w.getChild(k)
if w(k) == null then // hänge v ji an w(k)
copyAppend(v ji , w)
getDependanceTrees(w(k), sibChainQueue)
if v ji is SiblingChain then
20:
21:
22:
23:
sibChainQueue.add(w(k))
n
→ w(k) in H von w
v ji .addDepentants(n, w(k)) // speichere v ji −
24:
25:
26:
27:
28:
29:
30:
31:
32:
33:
34:
52
else
if w(k) == null then // Fall 2
. . . // wie 20-25 mit v ji ← v j
else if !w(k) is Alias then // Fall 3
. . . // wie 23-25 mit v ji ← v j
else // Fall 4
for l = 1 to outdeg(w(k)) do
w(k)l ← w(k).getChild(l);
. . . // wie 23-25 mit v ji ← v j und wk ← w(k)l
3.6 Default-Inheritance-Vererbungsalgorithmus
Fall 3
n
v
vj 1
w
1
2
n
v
w
n
=⇒ v j 1
1
2
Fall 4
n
v
vj 1
2
w
1
n
v
=⇒ v j 1
2
w
n
1
n
Abbildung 3.20: Anhängen von Knoten und Speichern von Beziehungen (Fall 3 und
Fall 4)
3.6.4 Wurzel-Siblingketten
Um die Beziehungen zweiter Ordnung zu dokumentieren, ist es notwendig, die Siblingketten zu bestimmen, die nicht von anderen Knoten abhängen. Ein Knoten v sei die
n
Wurzel einer Siblingkette. Wenn es für v kein v0 −
→ v gibt, heißt v Wurzel-Siblingkette.
Als Erstes erzeugen wir mithilfe der Funktion 3.9 getDependanceChains zwei Maps.
Die Schlüsselmenge der Maps ist die Menge aller Siblingketten. Die Datenmenge der
ersten Map (dependanceTreeMap) ist die Menge der abhängigen Siblingketten und die
der zweiten Map (innerDependancies) die Menge von restlichen abhängigen Knoten,
die kein Anfang einer Siblingketten sind.
1-7: Zuerst kopieren wir alle Siblingketten in die Menge sibChainSet. Dann durchlaufen wir alle Siblingketten v. Für jedes v durchlaufen wir seine abhängigen Knoten
w.
8-11: Falls w in der Menge der Siblingketten enthalten ist, fügen wir v (Erblasser) in
die Schlüsselmenge und w (abhängige Siblingkette) in die Datenmenge von de-
53
3 Vererbung der Geometrieparameter
pendanceTreeMap ein. Ansonsten, wenn w keine Siblingkette ist, fügen wir v
(Schlüssel) und w (Wert) in innerDependancies ein.
Algorithm 3.9 getDependanceChains(sibChainQueue)
1: dependanceTreeMap, innerDependancies
sibChainSet ← sibChainQueue
3: for i = 1 to sibChainQueue.size() do // Siblingketten
4:
v ← sibChainQueue.get(i)
5:
H ← v.getAllDependants(1) // Abhängige Knoten erster Ordnung
2:
6:
7:
8:
9:
10:
11:
for all nodes in H do // Abhängige Knoten von v
w ← H.next()
if sibChainSet.contains(w) then
dependanceTreeMap.put(v, w)
else
innerDependancies.put(v, w)
Zum Auffinden der Wurzel-Siblingketten verwenden wir dependanceTreeMap. Die
Funktion 3.10 getRootCandidate bestimmt eine Teilmenge von Schlüsseln aus der
Schlüsselmenge von dependanceTreeMap, die nicht in ihrer Datenmenge vorkommen.
1-4: Wir kopieren die Schlüsselmenge von dependanceTreeMap in zwei Mengen: rootCandidateSet und keySet. Dann durchlaufen wir alle Knoten v von keySet.
5-8: Für jedes v holen wir seine Datenmenge von dependanceTreeMap. Dann durchlaufen wir alle Knoten w aus der Datenmenge und entfernen w (abhängige Siblingketten) aus rootCandidateSet.
Als Ergebnis erhalten wir die Menge rootCandidateSet, die Wurzel-Siblingketten enthält. In Abbildung 3.21 ist der Abhängigkeitsgraph mit Beziehungen erster und zweiter Ordnung zwischen den Knoten dargestellt. Die Knoten v und w sind zwei WurzelSiblingketten.
54
3.6 Default-Inheritance-Vererbungsalgorithmus
Algorithm 3.10 getRootCandidate(dependanceTreeMap)
1: rootCandidateSet ← dependanceTreeMap.keySet() // Schlüsselmenge der Map
2: keySet ← dependanceTreeMap.keySet()
for all nodes in keySet do
4:
v ← keySet.next()
5:
depSet ← dependanceTreeMap.get(v) // Datenmenge von v
3:
6:
7:
8:
for all nodes in depSet do
w ← depSet.next()
rootCandidateSet.remove(w) // entferne w aus der Menge
1
v
1
1
w
k
1
1
2
1
2
1
2
1
2
k
1
1
k
Abbildung 3.21: Abhängigkeitsgraph mit den Wurzel-Siblingketten v und w
55
3 Vererbung der Geometrieparameter
3.6.5 Teilbäume für die Bestimmung der zweiten Ordnung
Als Nächstes bestimmen wir die Teilbäume für die Beziehungen zweiter Ordnung. Die
Funktion 3.11 createSecondOrderTrees geht von der Menge der Wurzel-Siblingketten
aus. Die Funktion kann wie folgt beschrieben werden:
1-8: Hier durchlaufen wir alle Wurzel-Siblingketten und fügen sie in die Warteschlange q ein. Für jede Siblingkette sibChain aus q durchlaufen wir alle ihre abhängigen Siblingketten depSibChain. Die depSibChain fügen wir in q ein. Das Paar
(sibChain, depSibChain) fügen wir in die zweite Warteschlange q2 ein.
11-15: Solange q2 Paare von Knoten enthält, holen wir das Paar mit den Knoten v und
seinem abhängigen Knoten w. Für v durchlaufen wir alle seine restlichen abhängigen Knoten v0 aus der Menge innerDepSetv.
20-26: Nun durchlaufen wir alle restlichen abhängigen Knoten w0 von w. Wenn w0 den
gleichen Schlüssel hat wie v0 , speichern wir die Beziehung zweiter Ordnung zwi2
schen den beiden Knoten. Wenn für v0 kein u mit der Eigenschaft u −
→ v0 existiert,
fügen wir v0 in die Menge secondOrderTrees ein. Um die Teilbäume zu durchlaufen, fügen wir das Paar(v0 , w0 ) in q2 ein.
27-29: Wenn wir keinen Knoten mit dem Schlüssel von v0 finden, verlängern wir die
abhängige Siblingkette, d. h., wir erzeugen einen Sibling und hängen ihn an w
an. Dann wird ein Knoten w0 mit dem Inhalt von v0 erzeugt und an den Sibling
2
angehängt. Danach speichern wir die Beziehung zweiter Ordnung v0 −
→ w0 . Wenn
2
es keinen Knoten u mit der Eigenschaft u −
→ v0 gibt, fügen wir v0 in die Menge
secondOrderTrees. Anschließend fügen wir das Paar(v0 , w0 ) in q2 ein.
In Abbildung 3.22 ist ein Baum mit den Siblingketten v und w dargestellt. Die Siblingkette mit der Wurzel v ist eine Wurzel-Siblingkette. Die Knoten mit den Schlüsseln 2
und 3 bezeichen wir als Teilbäume zweiter Ordnung.
56
3.6 Default-Inheritance-Vererbungsalgorithmus
Algorithm 3.11 createSecondOrderTrees(dependanceTreeMap, innerDependancies,
rootCandidateSet, order=1)
1: for all nodes in rootCandidateSet do // Wurzel-Siblingketten
2:
p ← rootCandidateSet.next()
3:
Queue q, q2; q.addFirst(p)
4:
5:
6:
7:
8:
9:
10:
11:
12:
13:
14:
15:
16:
17:
18:
19:
20:
21:
22:
23:
24:
25:
26:
27:
28:
29:
while q.size() > 0 do
sibChain ← q.getLast(); q.removeLast()
dependanceSet ← dependanceTreeMap.get(sibChain)
for all nodes in dependanceSet do // Abhängige Siblingketten
depSibChain ← dependanceSet.next()
q.addFirst(depSibChain)
q2.addFirst(Pair(sibChain, depSibChain))
while q2.size() > 0 do
pair ← q2.getLast(); q2.removeLast()
v ← pair.first
w ← pair.second
innerDepSetv ← innerDependancies.get(v)
for all nodes in innerDepSetv do // Abhängige von v
0
v ← innerDepSetv .next()
innerDepSetw ← innerDependancies.get(w)
found = false
for all nodes in innerDepSetw do // Abhängige von w
w0 ← innerDepSetw .next()
0
if w0 .getKey().equals(v .getKey()) then
0
0
v .addDepentants(order + 1, w )
0
secondOrderTrees.add(v )
0
0
q2 .addFirst(Pair(v , w ))
found = true
if !found then
w0 ← completeDepChain(v0 , w) // verlängere abhängige Siblingkette
. . . // wie 23-25
57
3 Vererbung der Geometrieparameter
r
r
=⇒
v
v
1
1
1
1
1
2
1
1
1
w
1
2
1
3
1
2
1
1
w
2
3
1
2
2
1
3
Abbildung 3.22: Teilbäume der zweiten Ordnung
3.6.6 Vererbung von Blättern
Als Nächstes wollen wir die Blätter innerhalb eines Baumes vererben. Dabei wird der
Baum in Level-Order-Reihenfolge traversiert.
Die Funktion 3.12 inherit bekommt die Wurzel des Baumes und die Ordnung als Argumente.
1-13: Zuerst fügen wir die Wurzel r des Baumes in die Warteschlange q ein. Solange
noch Knoten in q liegen, wird der letzte Knoten v geholt. Um alle Knoten einmal
zu besuchen, durchlaufen wir alle Kinder vi des aktuellen Knotens und fügen sie
in q ein. Für alle vi durchlaufen wir alle abhängigen Knoten w von v. Dabei prüfen
wir, ob w ein Kind w(k) mit demselben Schlüssel wie vi hat.
14-20: Falls wir den Knoten w(k) nicht finden, betrachten wir folgende Fälle:
Fall 1 (s. Abbildung 3.23): Falls vi nicht auf vererbt gesetzt ist, oder w keine
Beziehung höherer Ordnung zu einem anderen Knoten hat, erzeugen wir mithilfe
58
3.6 Default-Inheritance-Vererbungsalgorithmus
von complete eine Kopie von vi und hängen sie an w an.
Fall 2 (s. Abbildung 3.24): Wenn vi auf vererbt gesetzt ist und v kein Anfang einer
abhängigen Siblingkette ist, erzeugen wir eine Kopie von vi und hängen sie an w
an.
Algorithm 3.12 inherit(r, order)
1: Queue q; q.addFirst(r)
2: while q.size() > 0 do
3:
4:
5:
6:
7:
8:
9:
10:
11:
12:
13:
14:
15:
16:
17:
18:
v ← q.getLast(); q.removeLast()
for i = 0 to outdeg(v) do
vi ← v.getChild(i)
if !vi is leaf then
q.addFirst(vi )
else
H ← v.getDependants(order)
for all nodes in H do
w ← H.next()
k ← vi .getKey()
w(k) ← w.getChild(k)
if w(k) == null then
if !vi .isInherited() || w.getSupreme(order+1) == null then // Fall 1
w(k) ← complete(vi , w)
else if !isTreeRoot(v) then // Fall 2
w(k) ← complete(vi , w)
In Abbildung 3.25 ist ein Fall dargestellt, wann ein Blatt nicht vererbt wird. Der Knoten
w hat eine Beziehung höherer Ordnung n zu einem anderen Knoten u. Wenn folgendes
1
2
gilt: v −
→ w und u −
→ w, wird v j nicht an w vererbt.
59
3 Vererbung der Geometrieparameter
Fall 1 v
n
w
⇒
vj 1
v
n
vj 1
w
1
Abbildung 3.23: Vererbung von Blättern (Fall 1)
Fall 2
≤n
v
≤n
n
w
⇒
vj 1
v
n
vj 1
w
1
Abbildung 3.24: Vererbung von Blättern (Fall 2)
Sonst
≤n
v
vj 1
≤n
>n
n
w
;
v
>n
n
vj 1
Abbildung 3.25: Keine Vererbung von Blättern
60
w
1
3.6 Default-Inheritance-Vererbungsalgorithmus
3.6.7 Implementierungen
Die Klasse DefaultInheritance (s. Abbildung 3.26) enthält folgende Methoden. getDependanceTrees bestimmt alle Siblingketten eines Baumes und speichert Beziehungen
zwischen dem Vorgänger und den Nachfolgern des Siblings. genDependancies speichert
Beziehungen erster und zweiter Ordnung und vererbt innere Knoten. getDependanceChains bestimmt zwei Maps. Die Datenmenge der ersten Map enthält alle abhängigen Siblingketten, die der zweiten Map die restlichen von einer Siblingkette abhängigen
Knoten. Die Methode getRootCandidate ermittelt die Menge der Wurzel-Siblingketten.
createSecondOrderTrees bestimmt Teilbäume für die Beziehungen zweiter Ordnung.
inherit vererbt die Blätter.
Abbildung 3.26: Klasse DefaultInheritance
3.6.8 Kosten der Funktionen
Die Funktionen werden auf einen n-ären Baum mit N Knoten angewendet.
Die Laufzeit der Funktion getDependanceTrees ist
O(N). Der Baum wird in Level-Order-Reihenfolge durchlaufen. Dabei kommt jeder
Bestimmung von Siblingketten
61
3 Vererbung der Geometrieparameter
Knoten höchstens einmal in den Warteschlangen q und q2 vor.
Bestimmung und Ergänzung von abhängigen Teilbäumen
Die Laufzeit der Funk-
tion getDependanceTrees ist O(N). Jeder Knoten wird höchstens einmal in die Warteschlange q eingefügt, um die Beziehungen zwischen den Knoten zu speichern und
innere Knoten zu vererben.
Der Laufzeit der Funktion getDependanceChains ist von der
Größenordnung O(N). Es werden alle Siblingketten und deren abhängige Knoten durchWurzel-Siblingketten
laufen. Für jeden abhängigen Knoten wird geprüft, ob er in der Menge rootCandidateSet
ist. Der Aufwand ist Nsib · Nab · c < N · c, wobei Nsib die Anzahl der Siblingketten, Nab
die Anzahl der abhängigen Knoten und c der Suchaufwand in der Menge ist. Dabei
wird jeder abhängige Knoten höchstens einmal besucht. Für die Menge verwenden wir
die Java-Klasse HashSet. Die Laufzeit dieser Klasse zum Suchen ist konstant. In der
Anwendung sind Nsib und Nab klein.
Die Funktion getRootCandidate hat eine Laufzeit von O(N). Es werden alle Siblingketten und deren abhängige Knoten aus der Datenmenge von dependanceTreeMap durchlaufen. Die abhängigen Knoten, die in der Menge rootCandidateSet sind, werden gelöscht. Die Laufzeit zum Löschen eines Knotens ist konstant.
Teilbäume für die Bestimmung zweiter Ordnung
Die Laufzeit der Funktion crea-
teSecondOrderTrees ist O(N). Es werden alle Wurzel-Siblingketten und ihre abhängigen Siblingketten durchlaufen, um Teilbäume mit Beziehung zweiter Ordnung zu dokumentieren. Dabei werden die Knoten der abhängigen Siblingkette höchstens einmal
besucht.
Vererbung von Blättern
Die Laufzeit der Funktion inherit ist O(N). Um die Blätter
zu vererben, wird der Baum in der Level-Order-Reihenfolge traversiert. Jeder innere
Knoten wird höchstens einmal in die Warteschlange eingefügt.
62
3.6 Default-Inheritance-Vererbungsalgorithmus
Aufgrund der Laufzeiten der Funktionen von der Größenordnung O(N) ist die Gesamtlaufzeit des Default-Inheritance-Vererbungsalgorithmus O(N).
63
3 Vererbung der Geometrieparameter
3.7 Simplified-Inheritance-Vererbungsalgorithmus
In diesem Kapitel beschreiben wir den Simplified-Inheritance-Vererbungsalgorithmus
(SI) für den Konfigurationseditor von NeuGen. Der SI-Vererbungsalgorithmus verwendet den erzeugten Abhängigkeitsgraphen des DI-Vererbungsalgorithmus, um die Abhängigkeiten gezielt abzulaufen und die Parameterwerte zu vererben.
3.7.1 Parameterwerte aktualisieren
Wenn ein Parameter im Konfigurationseditor verändert wird, ist es notwendig, alle seine
abhängigen Parameter im Abhängigkeitsgraphen zu aktualisieren. Wird ein Parameter
auf vererbt gesetzt, wird der Wert von seinem Erblasser übernommen und die abhängigen Parameter aktualisiert. Die Funktion 3.13 updateAllInheritedValues stellt diese
Funktionalität bereit. Die Funktion, die ein Blatt als Argument erhält, kann wie folgt
beschrieben werden:
1-10: Zuerst initialisieren wir die Warteschlange q und fügen den Vater des Blattes p in
q ein. Solange q Knoten enthält, holen wir den letzten Knoten v und entfernen ihn
aus q. Dann holen wir ein Kind v(k) von v, das den gleichen Schlüssel wie der
Knoten p besitzt. Wenn v(k) auf vererbt gesetzt ist, ermitteln wir den Erblasser
u(k) und kopieren anschließend mithilfe der Funktion setInheritedValue den Wert
von u(k) in v(k).
11-20: Hier durchlaufen wir für die erste und zweite Ordnung alle abhängigen Knoten v0
von v. Dann prüfen wir, ob das Kind v0 (k) mit dem gleichen Schlüssel wie v(k) auf
vererbt gesetzt ist. Wenn v0 (k) auf vererbt gesetzt und nicht in der Menge procSet
enthalten ist, fügen wir v0 in q ein. Zum Schluss wird v0 in procSet eingefügt, um
den Knoten nicht mehrmals in die Warteschlange zu legen.
In Abbildung 3.27 ist ein Beispiel dargestellt. Die Blätter des Baumes haben den gleichen Schlüssel. Das Blatt v(1) von v mit dem Wert 10 (value) wird auf vererbt gesetzt
64
3.7 Simplified-Inheritance-Vererbungsalgorithmus
Algorithm 3.13 updateAllInheritedValues(p)
1: Queue q
2: q.addFirst((p.getParent())
3: while q.size() > 0 do
4:
5:
6:
7:
8:
9:
10:
11:
12:
13:
14:
15:
16:
17:
18:
19:
20:
v ← q.getLast(); q.removeLast()
k ← p.getKey()
v(k) ← v.getChild(k)
if v(k).isInherited() then // wenn der Knoten vererbt ist
u ← v.getSupreme()
u(k) ← u.getChild(v(k).getKey())
setInheritedValue(u(k), v(k)) // kopiere den Wert von u(k) in v(k)
order ← 2
for i = 1 to order do
Hv ← v.getDependants(i)
for all nodes in Hv do // die abhängigen Knoten von v
v0 ← Hv .next()
k ← v(k).getKey()
v0 (k) ← v0 .getChild(k)
if v0 (k).isInherited() && !procSet.contains(v0) then
q.addFirst(v0 )
procSet.add(v0 )
65
3 Vererbung der Geometrieparameter
=⇒
u
value = 5 1
u
value = 5 1
v
v
value = 10 1
value = 5
v0
value = 10
1
v0
1
value = 5
1
Abbildung 3.27: Vererbung von Parameterwerten
und erbt anschließend den Wert 5 von dem Blatt u(1) des Erblassers u. Danach wird das
auf vererbt gesetzte Blatt v0 (1) des abhängigen Knotens v0 aktualisiert.
3.7.2 Implementierungen
Abbildung 3.28: Klasse SimplifiedInheritance
Die Klasse SimplifiedInheritance (s. Abbildung 3.28) enthält folgende Methoden:
getHeritage ermittelt den Erblasser eines Knotens. setInheritedValue kopiert den Wert
des Erblassers in einen abhängigen Knoten. getInheritedValue liefert für einen Knoten
den Wert des Erblassers. updateAllInheritedValues aktualisiert die abhängigen Knoten
eines Knotens.
66
3.7 Simplified-Inheritance-Vererbungsalgorithmus
3.7.3 Kosten der Funktion
Parameterwerte aktualisieren
Die Laufzeit der Funktion updateAllInheritedValues
ist O(N). Im ungünstigsten Fall wird der ganze Baum mit N Knoten einmal durchlaufen.
Für die Menge verwenden wir die Java-Klasse HashSet. Die Laufzeit zum Suchen eines
Knotens in der Menge procSet ist konstant.
Folglich ist die Gesamtlaufzeit des SI-Vererbungsalgorithmus O(N).
67
4 Evaluierung
In diesem Kapitel werden die im vorherigen Kapitel beschriebenen Vererbungsalgorithmen Default-Inheritance und Simplified-Inheritance mittels Tests überprüft.
Zur Evaluierung wurde eine Testumgebung in NeuGen implementiert, in der die Geometrieparameter der Parameterdatei verändert werden können, um danach die Ergebnisse der verschiedenen Vererbungsalgorithmen zu vergleichen. Zusätzlich wurde ein
Algorithmus zum Erzeugen eines beliebigen Baumes programmiert, um die Laufzeit
der Vererbungsalgorithmen für große Bäume zu messen.
4.1 Vergleich der Vererbungsalgorithmen
Wir vergleichen die drei Vererbungsalgorithmen anhand von zwei Kriterien: Gleichheit
der Parameterbäume nach der Vererbung und Laufzeit.
In Tabelle 4.1 sind die Laufzeiten für die Vererbung von Geometrieparameter der Parameterdatei Param.neu mit 3622 Knoten, durchgeführt auf einem Intel Pentium 4 mit
einer Taktfrequenz von 3.00 GHz und 1 GB Arbeitsspeicher, zusammengefasst.
Für den Test wurde jeweils ein Parameter zufällig geändert und dann der Vererbungsalgorithmus angewandt. Insgesamt wurden so 2306 Parameter nacheinander bearbeitet, davon bekamen 847 einen zufälligen Wert, 1147 wurden auf vererbt und
312 auf nicht vererbt gesetzt. Der Eintrag für die Parameteranzahl 1 ist aus dem
Eintrag für 2306 Parameter als Mittelwert berechnet. In Tabelle 4.1 sind zum Vergleich auch die Laufzeiten der bisherigen Vererbungsalgorithmen Recursive-Tree-
68
4.1 Vergleich der Vererbungsalgorithmen
Inheritance und Default-Recursive-Inheritance. Es zeigt sich, dass die Vererbung mit
dem Simplified-Inheritance-Vererbungsalgorithmus weniger Zeit in Anspruch nimmt
als mit dem Default-Inheritance-Vererbungsalgorithmus. Dies war zu erwarten, da
der Simplified-Inheritance-Vererbungsalgorithmus den erzeugten Abhängigkeitsgraphen des Default-Inheritance-Vererbungsalgorithmus verwendet. Wegen der exponentiellen Laufzeit benötigen der bisherige Recursive-Tree-Inheritance- und der DefaultRecursive-Inheritance-Vererbungsalgorithmus die meiste Zeit.
Parameteranzahl
Zeit in Sekunden
neue Verfahren
bisherige Verfahren
Default-
Simplified-
Default-Recursive-
Recursive-Tree-
Inheritance
Inheritance
Inheritance
Inheritance
2306
250
0.1
1128
8532
1
0.1
4.2E-5
0.49
3.7
Tabelle 4.1: Laufzeiten für die Vererbungsalgorithmen
Der Recursive-Tree-Inheritance-Vererbungsalgorithmus wurde in C++ und die graphische Benutzeroberfläche in Java implementiert. Um den C++-Code unter Java nutzen
zu können, wird das Programmierwerkzeug SWIG1 (Simplified Wrapper and Interface
Generator) verwendet. SWIG generiert eine Art Hülle (Wrapper-Code) um den vorhandenen C++-Code, welche dem Java-Code den Zugriff auf den C++-Code ermöglicht.
Durch den vielfachen Wechsel zwischen den verschiedenen Programmiersprachen
können Probleme entstehen. Der Grund für die hohe Laufzeit des Recursive-TreeInheritance-Vererbungsalgorithmus ist das Übersetzen von C++-Klassen und Methoden nach Java mit SWIG. Die Laufzeit des Default-Recursive-Inheritance-Vererbungsalgorithmus auf der Java-Seite ist besser, als die Laufzeit des Recursive-TreeInheritance-Vererbungsalgorithmus mit dem Zugriff auf den C++-Code mithilfe von
SWIG.
1
http://www.swig.org
69
4 Evaluierung
Um die Ergebnisse der Vererbungsalgorithmen zu prüfen, wurde in jedem Schritt der
editierte Baum mit den Geometrieparametern der Parameterdatei Param.neu kopiert.
Auf die Bäume wurden jeweils verschiedene Vererbungsalgorithmen angewendet. Danach wurden die Bäume durchlaufen und die Knoten miteinander verglichen. Der
Default-Inheritance- und der Simplified-Inheritance-Vererbungsalgorithmus liefern den
gleichen Baum, wie der bisher in NeuGen eingesetzte Default-Recursive-InheritanceVerer-bungsalgorithmus.
Im Folgenden wollen wir die Laufzeiten der Vererbungsalgorithmen betrachten. Dazu
wurde ein Baum mit Siblingketten mit Beziehungen erster und zweiter Ordnung erzeugt
und in jedem Schritt die Siblingketten vergrößert. Auf die Bäume wurde die Vererbung
angewendet und die Laufzeit gemessen.
4.1.1 Default-Recursive-Inheritance-Vererbungsalgorithmus
In diesem Abschnitt wollen wir uns die Laufzeit des Default-Recursive-InheritanceVererbungsalgorithmus ansehen. Dazu verlängern wir die Siblingketten und vererben
dann die Knoten im Baum.
In Abbildung 4.1 sehen wir, dass der Anteil der Siblinge erster oder zweiter Ordnung
an der Gesamtanzahl der Siblinge einen Einfluss auf die Laufzeit hat. Wenn eine der
beiden Ordnungen überwiegt, nähert sich die Struktur des Baumes der des in Kapitel
3.5 beschriebenen schlimmsten Falles an. In diesem Fall besitzt der Baum eine große
Tiefe. Wenn der Anteil der Siblinge erster oder zweiter Ordnung etwa gleich ist, ist die
Struktur des Baumes breiter. In diesem Fall sind weniger Durchläufe nötig.
Wie wir in den Abbildungen 4.1 und 4.2 sehen, ist die gemessene Laufzeit des DefaultRecursive-Inheritance-Vererbungsalgorithmus für Bäume mit einer Gesamtanzahl von
unter 100 Siblingen fast gleich. Ab etwa 100-110 Siblingen steigt die Kurve deutlich an.
Bei einer großen Anzahl von Siblingen zeigt sich, dass die Laufzeit exponentiell wächst.
Dies stimmt mit dem im Kapitel 3.5 berechneten Aufwand von der Größenordnung
O(2N · N) überein.
70
4.1 Vergleich der Vererbungsalgorithmen
Abbildung 4.1: Entwicklung der Laufzeit von Default-Recursive-Inheritance für Bäume
mit bis zu 13 × 13 Siblingen
DefaultRecursiveInheritance
14.000
Process performance
12.000
Zeit t [sec]
10.000
8.000
6.000
4.000
2.000
0.000
0
20
40
60
80
100
Siblinge
120
140
160
180
200
Abbildung 4.2: Entwicklung der Laufzeit von Default-Recursive-Inheritance für die
Gesamtanzahl der Siblinge
71
4 Evaluierung
4.1.2 Default-Inheritance-Vererbungsalgorithmus
Aus den Abbildungen 4.3 und 4.4 kann man entnehmen, dass der Default-InheritanceVererbungsalgorithmus eine bessere Laufzeit als der Default-Recursive-InheritanceVererbungsalgorithmus hat. Auch sehen wir in Abbildung 4.3, dass der Anteil der Ordnungen im Gegensatz zum vorher beschriebenen Algorithmus DRI keinen starken Einfluss auf die Laufzeit hat. Weiter erkennt man, dass die gemessene Laufzeit sich der
theoretischen Laufzeit von O(N) für eine große Anzahl von Siblingen annähert. Dass die
Kurve in Abbildung 4.4 nicht linear ist, können wir dadurch erklären, dass der CacheSpeicher für kleinere Bäume effizienter genutzt werden kann, was die Ausführungszeit
verkürzt. Für große Bäume wird der Cache-Speicher nicht effizient verwendet und hat
daher keinen Einfluss auf die Laufzeit. Außerdem verwenden wir im Vererbungsalgorithmus Datenstrukturen, die im Laufe des Algorithmus ihre Größe dynamisch ändern.
Dadurch müssen die Daten umkopiert werden, was zu einer höheren Laufzeit führt. Die
Peaks sind Artefakte des Betriebssystems, die das Ergebnis nicht beeinflussen.
Abbildung 4.3: Entwicklung der Laufzeit von Default-Inheritance für Bäume mit bis zu
50 × 50 Siblingen (Peaks sind Artefakte, die das Ergebnis nicht beeinflussen)
72
4.1 Vergleich der Vererbungsalgorithmen
DefaultInheritance
4.500
Process performance
4.000
3.500
Zeit t [sec]
3.000
2.500
2.000
1.500
1.000
0.500
0.000
0
500
1000
1500
2000
2500
Siblinge
Abbildung 4.4: Entwicklung der Laufzeit von Default-Inheritance für die Gesamtanzahl
der Siblinge
4.1.3 Simplified-Inheritance-Vererbungsalgorithmus
Wir wollen in diesem Abschnitt die Laufzeit des Simplified-Inheritance-Vererbungsalgorithmus ansehen, der den erzeugten Abhängigkeitsgraphen des Default-InheritanceVererbungsalgorithmus verwendet.
Wenn wir uns die Abbildungen 4.5 und 4.6 ansehen, stellen wir fest, dass die Laufzeit
besser ist, als die des Default-Inheritance-Vererbungsalgorithmus und wie bei diesem
nicht von der Struktur des Baumes abhängt. Dies können wir dadurch erklären, dass
der Abhängigkeitsgraph nur einmal durchlaufen werden muss, um die Knoten zu vererben.
Die Peaks sind Artefakte des Betriebssystems, die das Ergebnis nicht beeinflussen. Aufgrund der kurzen Laufzeiten sind mehr Peaks zu sehen, als in Abbildung 4.3.
73
4 Evaluierung
Abbildung 4.5: Entwicklung der Laufzeit von Simplified-Inheritance für Bäume mit bis
zu 100 × 100 Siblingen (Peaks sind Artefakte des Betriebssystems, die
das Ergebnis nicht beeinflussen)
SimplifiedInheritance
0.250
Process performance
0.200
Zeit t [sec]
0.150
0.100
0.050
0.000
0
1000
2000
3000
4000
5000
Siblinge
6000
7000
8000
9000
10000
Abbildung 4.6: Entwicklung der Laufzeit von Simplified-Inheritance für die Gesamtanzahl der Siblinge
74
4.2 Zusammenfassung
4.2 Zusammenfassung
Es zeigt sich, dass der Default-Inheritance-Vererbungsalgorithmus und der für die grafische Benutzeroberfläche entwickelte Simplified-Inheritance-Vererbungsalgorithmus
eine bessere Laufzeit als der bisher in NeuGen implementierte Default-RecursiveInheritance-Vererbungsalgorithmus aufweisen. Bereits ab 100 Siblingen benötigt der
Default-Inherit-ance-Vererbungsalgorithmus deutlich mehr Zeit für die Vererbung. Bei
den Tests liefern alle Vererbungsalgorithmen gleiche Parameterbäume für die gleiche
Parameterdatei Param.neu.
75
5 Zusammenfassung
In dieser Bachelor-Arbeit wurde der bisher in NeuGen implementierte Default-Recursive-Inheritance-Vererbungsalgorithmus betrachtet und ein schnellerer Default-Inheritance-Vererbungsalgorithmus vorgestellt. Für die grafische Benutzeroberfläche wurde
der Simplified-Inheritance-Vererbungsalgorithmus beschrieben, der den Abhängigkeitsgraphen des Default-Inheritance-Vererbungsalgorithmus mit den gespeicherten Beziehungen erster und zweiter Ordnung zwischen den Knoten verwendet.
Der betrachtete rekursive DRI-Vererbungsalgorithmus ist auch für die Vererbung mit
Beziehungen höherer Ordnung verwendbar. Der Nachteil ist seine exponentielle Laufzeit. Außerdem werden keine Beziehungen zwischen den Knoten gespeichert, weswegen es nicht möglich ist, den Wert des Erblassers und der abhängigen Knoten zu bestimmen. Für jeden editierten Knoten im Konfigurationseditor muss der Vererbungsalgorithmus auf dem ganzen Baum ausgeführt werden.
Um die Algorithmen zu testen, wurde eine Testumgebung implementiert, in der die
Ergebnisse des bisher in NeuGen eingesetzten DRI-Vererbungsalgorithmus mit den Ergebnissen der neuen Algorithmen verglichen werden. Dabei liefern die Vererbungsalgorithmen gleiche Resultate, d. h. sie liefern für gleiche Parameterdateien gleiche Parameterbäume. Zusätzlich wurde die Laufzeit der Algorithmen für große Parameterbäume
gemessen.
Die Vorteile der beiden Vererbungsalgorithmen DI und SI liegen in den kurzen Ausführungszeiten. Vor allem aber sind sie für große Parameterdateien geeignet. Mithilfe des
Abhängigkeitsgraphen kann der Wert des Erblassers und der abhängigen Knoten festgestellt werden. Die beiden Vererbungsalgorithmen sind nur für die Vererbung mit den
76
Beziehungen erster und zweiter Ordnung geeignet. Eine Anwendung der Vererbung mit
den Beziehungen höherer Ordnungen wäre für den Benutzer im Konfigurationseditor
unübersichtlich.
Die Vererbung von Geometrieparametern bildet die in der Natur gefundenen Zusammenhänge zwischen den einzelnen Nervenzellklassen in sinnvoller Weise ab. Vor allem
aber muss sie schnell und platzsparend sein. Beide Kriterien werden von der vorliegenden Arbeit erfüllt. Durch die Implementierung des vorgestellten neuen Algorithmus
in das Softwarepaket NeuGen ist die effizientere Generierung von größeren synthetischen neuronalen Netzen ermöglicht worden. Diese Arbeit ist somit ein weiterer kleiner
Schritt auf dem Weg zur Modellierung des Gehirns.
77
Abbildungsverzeichnis
78
2.1
Schemazeichnung der wichtigsten Gehirnstrukturen aus [6, Kandel] . .
6
2.2
Kolumne nach [4, Eberhard] . . . . . . . . . . . . . . . . . . . . . . .
7
2.3
Aufbau einer Nervenzelle aus [1] . . . . . . . . . . . . . . . . . . . . .
8
2.4
Schematische Darstellung von NeuGen . . . . . . . . . . . . . . . . . .
10
2.5
Neuronenklassen-Hierarchie . . . . . . . . . . . . . . . . . . . . . . .
11
2.6
L4-Star-Pyramidenzellen . . . . . . . . . . . . . . . . . . . . . . . . .
11
2.7
L4-Spiny-Stellate-Zelle . . . . . . . . . . . . . . . . . . . . . . . . . .
12
2.8
L2/3-Pyramidenzellen . . . . . . . . . . . . . . . . . . . . . . . . . . .
12
2.9
L5A-Pyramidenzellen . . . . . . . . . . . . . . . . . . . . . . . . . . .
12
2.10 L5B-Pyramidenzellen . . . . . . . . . . . . . . . . . . . . . . . . . . .
12
2.11 Parameterdatei . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
14
2.12 Konfigurationseditor (links ist der Baum der Parameter, rechts ist die
Erklärung) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
16
2.13 Teilbäume . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
20
2.14 Darstellung eines Baumes . . . . . . . . . . . . . . . . . . . . . . . . .
20
3.1
Sibling, Siblingkette und Alias . . . . . . . . . . . . . . . . . . . . . .
23
3.2
Beziehung erster Ordnung . . . . . . . . . . . . . . . . . . . . . . . .
24
3.3
Beziehung erster Ordnung mit Alias im Teilbaum t(r) . . . . . . . . . .
25
3.4
Beziehungen zweiter Ordnung . . . . . . . . . . . . . . . . . . . . . .
26
3.5
Reihenfolge bei der Vererbung (Fall 1-3 i) . . . . . . . . . . . . . . . .
27
3.6
Reihenfolge bei der Vererbung (Fall 3 ii) . . . . . . . . . . . . . . . . .
27
3.7
Traversieren von Bäumen . . . . . . . . . . . . . . . . . . . . . . . . .
29
3.8
Übersicht der verwendeten Klassen . . . . . . . . . . . . . . . . . . . .
32
3.9
Anhängen von Knoten (Fall 1) . . . . . . . . . . . . . . . . . . . . . .
35
Abbildungsverzeichnis
3.10 Anhängen von Knoten (Fall 2 und Fall 3) . . . . . . . . . . . . . . . .
36
3.11 Vervollständigen von Knoten (Fall 2) . . . . . . . . . . . . . . . . . . .
38
3.12 Vervollständigen von Knoten (Fall 3) . . . . . . . . . . . . . . . . . . .
38
3.13 Vererben von Knoten . . . . . . . . . . . . . . . . . . . . . . . . . . .
41
3.14 Klasse DefaultRecursiveInheritance . . . . . . . . . . . . . . . . . . .
42
3.15 Aufwand für complete . . . . . . . . . . . . . . . . . . . . . . . . . .
43
3.16 Aufwand für processNode . . . . . . . . . . . . . . . . . . . . . . . .
45
3.17 Aufwand für copyAppend . . . . . . . . . . . . . . . . . . . . . . . . .
46
3.18 Beziehungen erster Ordnung zwischen dem Vorgänger und den Nachfolgern des Siblings . . . . . . . . . . . . . . . . . . . . . . . . . . . .
50
3.19 Anhängen von Knoten und Speichern von Beziehungen (Fall 1 und Fall 2) 51
3.20 Anhängen von Knoten und Speichern von Beziehungen (Fall 3 und Fall 4) 53
3.21 Abhängigkeitsgraph mit den Wurzel-Siblingketten v und w . . . . . . .
55
3.22 Teilbäume der zweiten Ordnung . . . . . . . . . . . . . . . . . . . . .
58
3.23 Vererbung von Blättern (Fall 1) . . . . . . . . . . . . . . . . . . . . . .
60
3.24 Vererbung von Blättern (Fall 2) . . . . . . . . . . . . . . . . . . . . . .
60
3.25 Keine Vererbung von Blättern . . . . . . . . . . . . . . . . . . . . . .
60
3.26 Klasse DefaultInheritance . . . . . . . . . . . . . . . . . . . . . . . .
61
3.27 Vererbung von Parameterwerten . . . . . . . . . . . . . . . . . . . . .
66
3.28 Klasse SimplifiedInheritance . . . . . . . . . . . . . . . . . . . . . . .
66
4.1
Entwicklung der Laufzeit von Default-Recursive-Inheritance für Bäume mit bis zu 13 × 13 Siblingen . . . . . . . . . . . . . . . . . . . . .
4.2
Entwicklung der Laufzeit von Default-Recursive-Inheritance für die
Gesamtanzahl der Siblinge . . . . . . . . . . . . . . . . . . . . . . . .
4.3
72
Entwicklung der Laufzeit von Default-Inheritance für die Gesamtanzahl der Siblinge . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
4.5
71
Entwicklung der Laufzeit von Default-Inheritance für Bäume mit bis zu
50 × 50 Siblingen (Peaks sind Artefakte, die das Ergebnis nicht beeinflussen) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
4.4
71
73
Entwicklung der Laufzeit von Simplified-Inheritance für Bäume mit bis
zu 100 × 100 Siblingen (Peaks sind Artefakte des Betriebssystems, die
das Ergebnis nicht beeinflussen) . . . . . . . . . . . . . . . . . . . . .
74
79
Abbildungsverzeichnis
4.6
80
Entwicklung der Laufzeit von Simplified-Inheritance für die Gesamtanzahl der Siblinge . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
74
Algorithmenverzeichnis
3.1
3.2
3.3
postorder_traverse(v) . . . . . . . . . . . . . . . . . . . . . . . . . . .
levelorder_traverse(v) . . . . . . . . . . . . . . . . . . . . . . . . . . .
copyAppend(v, w) . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
29
30
35
3.4
3.5
3.6
complete(v, w) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
processNode(v) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
reverseProcess(v) . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
37
40
42
3.7
3.8
3.9
3.10
getDependanceTrees(r, sibChainQueue)
genDependancies(n, sibChainQueue) . .
getDependanceChains(sibChainQueue)
getRootCandidate(dependanceTreeMap)
49
52
54
55
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
3.11 createSecondOrderTrees(dependanceTreeMap, innerDependancies, rootCandidateSet, order=1) . . . . . . . . . . . . . . . . . . . . . . . . . . 57
3.12 inherit(r, order) . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 59
3.13 updateAllInheritedValues(p) . . . . . . . . . . . . . . . . . . . . . . . 65
81
Literaturverzeichnis
[1] Neil A. Campbell. Biologie. Spektrum Akademischer Verlag, 1 edition, 1997.
[2] Anja Schierloh. Neuronale netzwerke und deren plastizität im barrel-kortex der
ratte. http://deposit.ddb.de/cgi-bin/dokserv?idn=97305395x,
2003.
[3] Robert Sedgewick. Algorithmen in C++. Pearson Studium, 3 edition, 2002.
[4] J. P. Eberhard, A. Wanner und G. Wittum. Neugen: A tool for the generation of realistic morphology of cortical neurons and neural networks in 3d.
http://dx.doi.org/10.1016/j.neucom.2006.01.028, 2006.
[5] Thomas Ottmann und Peter Widmayer. Algorithmen und Datenstrukturen. Spektrum Akademischer Verlag, 4 edition, 2002.
[6] Eric R. Kandel, James H. Schwarz und Thomas M. Jessell. Neurowissenschaften Eine Einführung. Spektrum Akademischer Verlag, 1 edition, 1996.
82