V12 Stochastische Effekte
Werbung

V12
Stochastische Effekte
31. Januar 2013
Übersicht
Stochastische Effekte
=> was sind das?
=> warum wichtig?
=> wie behandeln?
Gillespie-Algorithmus <=> direkte Implementierung
Softwarewerkzeuge WS 12/13 – V12
2
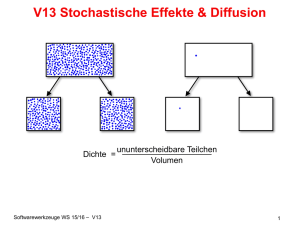
Stochastische Effekte
ununterscheidbare Teilchen
Dichte =
Volumen
Softwarewerkzeuge WS 12/13 – V12
3
Dichtefluktuationen
N = 10
0
N = 100
1.67
6
6
4
4
2
2
0
0
0
N = 1000
3
6
0
9
6
6
4
4
2
2
0
0
0
3
Softwarewerkzeuge WS 12/13 – V12
6
0
N = 10000
55.6
9
0
0
5.56
3
6
0
9
556
3
6
9
4
Poisson-Verteilung
Betrachte Kontinuum w mit im Mittel λ Ereignissen pro Einheitsintervall Δw
Annahmen:
i)
Seltenheit:
Ereignis
ii)
Proportionalität:
iii)
<<1 Ereignisse in [w, w+Δw], höchstens ein
<N> = λ Δw
Geschichtslosigkeit (Markov-Prozess)
Wahrscheinlichkeit, dass k Ereignisse
pro Einheitszeitintervall auftreten:
Mittelwert:
Varianz:
Relative Streuung (Fehler):
Mittlere Teilchenzahl
100
1000
1 Mol
relative Unsicherheit
10%
3%
1e-12
Softwarewerkzeuge WS 12/13 – V12
5
Reaktionen im Teilchenbild
Assoziation:
A + B => AB
Kontinuierliche Ratengleichung:
Anzahl neuer AB in V während Δt:
Reaktionsrate kAB => Reaktionswahrscheinlichkeit PAB
Softwarewerkzeuge WS 12/13 – V12
6
Direkte Implementierung
A + B => AB
Achtung: didaktische Implementierungen!
Softwarewerkzeuge WS 12/13 – V12
7
Beispiel: Reaktions-Kette
A => B => C
Raten:
Zeitentwicklung aus den kontinuierlichen Raten:
NA, NB, NC / NA0
1.00
0.50
0.00
0
10
20
30
time
k1 = k2 = 0.3
Softwarewerkzeuge WS 12/13 – V12
8
Stochastische Simulation
A => B => C
A0 = 1000 Teilchen bei t = 0
t=7
1.00
400
A
C
300
frequency
A, B, C / A0
A
0.50
B
0.00
0
10
20
C
200
100
0
0.00
30
time
k1 = k2 = 0.3
B
0.25
0.50
0.75 1.00
Werte bei t = 7 (1000 Läufe)
=> Fluktuationen
Softwarewerkzeuge WS 12/13 – V12
9
A0
Weniger Teilchen => Mehr
Rauschen
= 100
1.00
A, B, C / A0
A, B, C / A0
1.00
0.50
0.00
0
10
20
0.50
0.00
30
0
10
time
20
30
1.00
A, B, C / A0
A, B, C / A0
30
time
1.00
0.50
0.00
20
0
10
20
time
Softwarewerkzeuge WS 12/13 – V12
30
0.50
0.00
0
10
time
10
Noch weniger Teilchen
A0 = 30
1.00
A, B, C / A0
A, B, C / A0
1.00
0.50
0.00
0
10
20
0.50
0.00
30
0
10
time
20
30
1.00
A, B, C / A0
A, B, C / A0
30
time
1.00
0.50
0.00
20
0
10
20
time
Softwarewerkzeuge WS 12/13 – V12
30
0.50
0.00
0
10
time
11
Varianz vs. Teilchenanzahl
Poisson:
relative Abweichung
300
A
A0 = 1000
B
200
Fit der Verteilung mit Gaussvert.
(Normalverteilung)
<A> = 0.13,
wA = 0.45
<B> = 0.26,
wB = 0.55
<C> = 0.61,
wC = 0.45
Softwarewerkzeuge WS 12/13 – V12
100
rel. Häufigkeiten
1000 Simulationsläufe,
Werte sichern bei t = 7.
0
0.00
A
300
0.50
B
A0 = 1001.00
C
150
0
0.00
400 A
0.50
B
1.00
A0 = 30
C
200
0
0.00
0.50
1.00
12
Gillespie-Algorithmus
naive Integration
=> shortcomings
Gillespie
=> Idee an der Tafel
=> Parallelen zu den
Poisson-Annahmen
D. Gillespie, J. Phys. Chem. 81 (1977) 2340–2361
Softwarewerkzeuge WS 12/13 – V12
13
Das Problem
Zerfallsreaktion: A => Ø
Wahrscheinlichkeit für ein Ereignis in (t, t+Δt) bei A(t) Molekülen = A(t) k Δt
Naiver Algorithmus:
A = A0
For every timestep:
get random number r ε [0, 1)
if r ≤ A*k*dt:
A = A-1
Es geht, aber: A(t)*k*dt << 1 — Genauigkeit
=> meistens passiert nichts
Gillespie überlegte sich: "Wann ist das nächste Ereignis?"
Softwarewerkzeuge WS 12/13 – V12
14
Exponentiell verteilte Wartezeiten
Idee: wann findet die nächste Reaktion statt?
Mit A(t) Molekülen:
g(A(t), s) = Wahrscheinlichkeit, dass keine Reaktion in (t, t+s) stattfindet
+ Poisson-Annahmen
=> DGL für Verteilung der Wartezeiten:
mittlere Wartezeit:
Softwarewerkzeuge WS 12/13 – V12
15
Gillespie – Schritt 1
Idee: Finde heraus, wann die nächste Reaktion stattfinden wird!
(Zwischen den diskreten Reaktionen passiert nichts …
Es seien A(t) Moleküle zum Zeitpunkt t im System
f(A(t), s) = W‘keit. dass für A(t) Moleküle die nächste Reaktion im Intervall
(t+s, t+s+ds) stattfindet, mit ds => 0
g(A(t), s) = W‘keit. dass für A(t) Moleküle in (t, t+s) keine Reaktion passiert.
Dann:
Keine Reaktion während (t, t+s):
W‘keit. für Reaktion in (t+s, t+s+ds)
Softwarewerkzeuge WS 12/13 – V12
16
Wahrscheinlichkeit für (keine
Reaktion)
Wir brauchen nun g(A(t), s)
Verlängere g(A(t), s) ein wenig:
noch etwas A(t+s) = A(t) und sortiere um:
Mit g(A, 0) = 1 ("keine Reaktion während Zeit = 0")
=> Verteilung der Wartezeiten zwischen diskreten
Reaktionsereignissen:
Lebensdauer = mittlere Wartezeit:
Softwarewerkzeuge WS 12/13 – V12
17
Gillespie-Algorithmus: 1 Spezies
r ∈ [0,1]
Exponentielle Verteilung der Wartezeiten:
Mit
1
0
0
life time
t
=> Gillespie-Algorithmus für Reaktion A => Ø:
A = A0
While(A > 0):
get random number r ∈ [0, 1)
t = t + s(r)
A = A-1
Softwarewerkzeuge WS 12/13 – V12
keine Abfrage!!!
einfache Sprung
in der Zeit
18
Gillespie- vs. direkte Integration
Naiv:
Gillespie:
"Was ist die
Wahrscheinlichkeit, dass im
nächsten Δt ein Ereignis
stattfindet?"
=> kleine feste Zeitschritte
"Wie lange dauert es
bis zum nächsten
Ereignis?"
=> variable Zeitschritte
=> Näherung erster
Ordnung
=>
exakt
• Gillespie
30
• Gillespie
30
• naive
- analytic
20
• naive
- analytic
NA
NA
25
10
20
0
0
1
2
Softwarewerkzeuge WS 12/13 – V12
t
3
4
0.00
0.20
t
0.40
19
Gillespie – viele Spezies
Für beliebige Anzahl an möglichen Reaktionen:
(i) Wahrscheinlichkeiten für jede einzelne Reaktion: αi i = 1, …, N
gesamte W‘keit α0 = Σ αi
(ii) Wartezeit bis zum nächsten beliebigen Ereignis:
(iii) Wähle eine der Reaktionen aus gemäß:
1
0
2
3
1 12
123
4
5
1…4
6
1…5
0
(iv) Update der Teilchenzahlen
Softwarewerkzeuge WS 12/13 – V12
20
Ein Beispiel mit zwei Spezies
Reaktionen:
k1
k2
A + A => Ø
A + B => Ø
k3
k4
Ø => A
Ø => B
k3 = 1.2 s–1
k4 = 1 s–1
Ratengleichungen:
Gleichgewichtszustand:
mit
k1 = 10–3 s–1
k2 = 10–2 s–1
=> Ass = 10, Bss = 10
Softwarewerkzeuge WS 12/13 – V12
21
Erban, Chapman, Maini, arXiv:0704.1908v2 [q-bio.SC]
Gillespie-Algorithmus
Softwarewerkzeuge WS 12/13 – V12
22
Stochastisch vs. Deterministisch
k1
A + B => Ø
k3
Ø => A
k4
Ø => B
Erban, Chapman, Maini, arXiv:0704.1908v2 [q-bio.SC]
A + A => Ø
k2
Softwarewerkzeuge WS 12/13 – V12
23
Stationärer Zustand
k1
k2
A + A => Ø
A + B => Ø
k1 = 10–3 s–1
k2 = 10–2 s–1
Erban, Chapman, Maini, arXiv:0704.1908v2
kontinuierliches Modell:
Ass = 10, Bss = 10
Softwarewerkzeuge WS 12/13 – V12
<=>
k3
k4
Ø => A
Ø => B
k3 = 1.2 s–1
k4 = 1 s–1
Aus langen GillespieSimulationen:
Ass = 9.6, Bss = 12.2
24
Photosynthese ist…
…die Umwandlung von Lichtenergie in chemische Energie
(einer der wichtigsten Prozesse weltweit)
biologischer Überblick
Stochastische SImulation aus einzelnen
Reaktionen
=> Unterschiede zu Gillespie?
=> Reihenfolge von Näherung und Simulation
Vesiweb
Softwarewerkzeuge WS 12/13 – V12
25
Photosynthese in Rb. sphaeroides
Lichtenergie
elektronische
Anregungen
Softwarewerkzeuge WS 12/13 – V12
e–-H+Paare
Protonengradient
chemische
Energie
26
Chromatophoren-Vesikel
45 nm
=> einfach:
4 Proteine + 2 Transporter + H+; Kristallstrukturen u.
Reaktionen
=> klein:
<30 Proteine + LHCs; alles Prozesse im Vesikel
=> abgeschlossen: definierte Randbedingungen für die Simulation
=> praktisch:
Anregung mit Licht, spektroskopische Messungen
Softwarewerkzeuge WS 12/13 – V12
Geyer, Helms, BPJ 91 (2006) 921 + 927
27
Modellierung der Proteine
RC: Photon =>
Ladungstrennung
Boxer et al, JCP 93
(1989) 8280
1AIJ.pdb
Softwarewerkzeuge WS 12/13 – V12
28
Stochastische Reaktionen
Wenn BS frei ist => Assoziation möglich: BS + X => BS:X
1) sind alle Bedingungen erfüllt?
2) chemische Reaktionskinetik:
Reaktionsrate:
Bindungs-W.keit pro BS:
for each timestep Δt:
for each reaction:
conditions fulfilled?
determine probability:
perform reaction
Geyer, Lauck, Helms, J Biotech 129 (2007) 212
Softwarewerkzeuge WS 12/13 – V12
29
Bsp: Elektronentransfer im RC
// R1: transfers an electron to the Quinone
//
using the energy from an exciton{
if
(bs_Q && (e_P == 1) && (e_Q == 0) &&
((He_Q == 0) || (He_Q == 1)))
{
if (LHPoolp->take_out(LH_kon))
{
e_P = 0;
e_Q = 1;
writeInternals();
}
}}
Bedingungen?
Wahrscheinlichkeit?
Reaktion!
Protein = {BS; Reaktionen(Zustand)}
Geyer, Lauck, Helms, J Biotech 129 (2007) 212
Softwarewerkzeuge WS 12/13 – V12
30
"Pools-and-Proteins"–Modell
H+ outside
Light
QH2
LHC
E
RC
Q
bc1
ADP + P
ATPase
ATP
titratab le
grou ps
c2red
c2ox
H+ inside
40 aktive Proteine
19 passive Pools
• unabhängig voneinander
• stochastische Reaktionen mit je 1 Molekül • ein Pool pro Metabolit
• hier: Diffusion ist schnell
• Anzahl wie auf dem Vesikel
Verbindungen definieren das biologische System
=> Pfade als "emergent behavior"
Geyer, Lauck, Helms, J Biotech 129 (2007) 212
Softwarewerkzeuge WS 12/13 – V12
31
Web Interface
Simulationen über Konfigurationsdatei oder
web-interface @ service.bioinformatik.uni-saarland.de/vesiweb
• Verstehen der Prozesse
• Modell-Verifikation + Parametrisierung gegen Experimente
[Florian Lauck. T.G., 2006]
Softwarewerkzeuge WS 12/13 – V12
32
Stochastische Effekte
Oxidationszustand des Cytochrom c-Pools bei kontinuierlicher Beleuchtung
4.4 W/m2 const.
Softwarewerkzeuge WS 12/13 – V12
33
Steady State <=> Fluktuationen
60 Sek. bei konstanter Beleuchtung mit 10 RC/LHC-Dimeren und 4 bc1-Dimere
=> Oxidationszustand des Cytochrom c-Pools
20
3 W/m2
8 W/m2
<#c2ox>
15
10
5
0
4.5 W/m2
0
3
6
I [W/m2]
9
12
5.5 W/m2
6.5 W/m2
=> weicher Übergang mit starken Fluktuationen
Softwarewerkzeuge WS 12/13 – V12
34
Deterministisch vs. Stochastisch
20
20
t=1s
t=3s
10
15
<#c2ox>
# c2ox
15
t=0.3s
5
5
0
10
0
0
3
6
9
I [W/m2]
0
3
6
I [W/m2]
9
12
Gleichungen mitteln, dann simulieren<=> Simulieren, dann Ergebnisse mitteln
scharfer Übergang
<=>
weicher Übergang
auch für lange Zeiten
nur numerische Unsicherheiten
<=>
Fluktuationen ≈ Signal
Reproduzierbare Werte
<=>
nur Mittelwert reproduzierbar
Softwarewerkzeuge WS 12/13 – V12
35
Vesimulus: Warum nicht Gillespie?
Gillespie:
Pools-and-Proteins:
hohe Anzahl von Spezies/Zuständen Belegung unabhängiger Bindungsstellen
=> Komplexität!
=> Zustände als Filter
Jeweils eine Reaktion
Mehrere Reaktionen pro Zeitschritt
Numerisch effizient, exakt
Natürliche Modellierung
Viele unabhängige Proteine
Mittelung
Gemittelte Proteine
Simulation
Belegungszahlen pro Protein
?
gemittelte Belegungszahlen = gemittelte Belegungszahlen
Simulation
Softwarewerkzeuge WS 12/13 – V12
Mittelung
37
Zusammenfassung
Stochastische Effekte sind oft wichtig — wann?
Modellierung I: Gillespie
• numerisch effizient
• numerisch exakt
• biochemische Prozesse
=> stochastische Integration kontinuierlicher Ratengleichungen
Modellierung II: Pools-and-Proteins
• direkte Modellierung individueller Proteine
• Netzwerktopologie bleibt erhalten (auf allen Ebenen)
=> mikroskopisches Verhalten jedes einzelnen Proteins
Softwarewerkzeuge WS 12/13 – V12
38