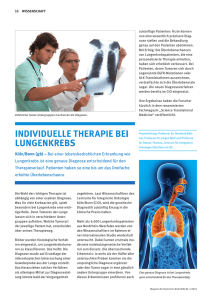
Computational Diagnosis - Computational Diagnostics Group
Werbung

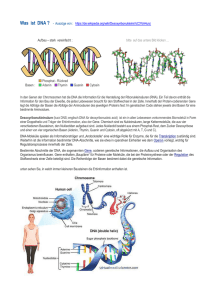
Gutartig oder bösartig? Die Diagnose aus dem Rechner Eine Krankheit Drei Therapien Klinische Studie Im Mittel 75% 55% Erfolg 35% Drei unterschiedliche Formen dieser Krankheit A B C A B C 100% 60% 65% 40% 40% 85% 10% 90% 5% 100% A 90% B 85% C 91,7% Der Behandlungserfolg ist durch die verfeinerte Diagnose angestiegen 91,7% 75% Ohne daß eine neue Therapie entwickelt wurde Eine hochauflösende Unterteilung einer Krankheit in Subtypen führt zu erhöhtem Therapieerfolg Wie erhält man eine höhere Auflösung der Diagnose, die Therapie relevant ist? Der Blick auf das Äußere der Zelle Das Mikroskop Ein Detail des Stoffwechsels: Das Blutbild Diagnose auf Umwegen • Stoffwechselstörungen in den Zellen verursachen die Krankheit • Sie führen manchmal auch zu charakteristischen Veränderungen der Zellform, der Gewebestruktur oder des Blutbildes Diagnose auf der Grundlage von Details • Einige wenige Stoffwechselprodukte werden routinemäßig gemessen • Einige wenige genetische Veränderungen werden routinemäßig festgestellt Wünschenswert • Der Blick in die Zelle statt auf die Zelle • Statt eines Bildes der Zelle ... • ... ein Protokoll über das, was die Zelle gerade macht Außerdem wünschenswert • Der große Überblick • Der Stoffwechsel des Patienten aus der Vogelperspektive Der Zugang: Genomics Die Chip-Technologie Genexpression im großen Maßstab Mehr als 10 000 Messungen auf einmal DNA Chip Gewebe ExpressionsProfil Ein ExpressionsProfil ist eine lange Liste von Zahlen Für jedes Gen eine ExpressionsIntensität Das Profil gewährt einen Blick in die Zellen der Gewebeprobe Es ist ein sehr komplexer diagnostischer Befund Wie erhält man eine höhere Auflösung der Diagnose, die therapierelevant ist ? A B Gruppiere die Patienten nach Therapieerfolg, und versuche in dem Wust von Expressionsinformation ein Muster zu finden, das es erlaubt den Therapieerfolg für unterschiedliche Therapiestrategien vorherzusagen C Welche Behandlung ist am besten für den Patient? Welche Behandlung ist am besten für den Patient? An dieser Lücke arbeiten wir • Wozu braucht man dazu Bioinformatiker? • Sollte man das nicht den Mediziner oder Genetikern überlassen? Also wo liegt das Problem ? Angenommen wir hätten nur die Expressionswerte von zwei Genen A B Ein neuer Patient A B A Der neue Patient A B Es gibt keine Gerade, die die Gruppen trennt A B Gen A ist wichtig A Gen A hoch Gen A niedrig B Gen B ist wichtig A Gen B niedrig Gen B hoch B Neuer Patient ? A B Problem 1: Keine Gerade Problem 2: Zuviele unterschiedliche Geraden In der Praxis untersuchen wir tausende Gene und im allgemeinen mehr Gene als Patienten ... • Problem 1 entsteht nie! • Problem 2 entsteht praktisch immer! Überlegen Sie sich das einmal kurz in drei Dimensionen Also für drei Gene, zwei Patienten mit bekannter Diagnose, einem neuen Patienten und einer Trennebene statt einer Trenngerade OK! Wenn alle Punkte auf einer Geraden liegen, geht es nicht immer. Das ist bei Messungen aber sehr unwahrscheinlich und kommt praktisch nie vor. Aus den Daten alleine kann man weder feststellen, welche Gene wichtig für die Diagnose sind, noch kann man zweifelsfrei eine Diagnose für den nächsten Patienten stellen. Dieses Problem hat mit Medizin wenig zu tun. Es ist ein geometrisches Problem. Das Problem kenne ich. Statt der Trennebene nimmt man besser: - neuronale Netze - Support-Vector-Maschinen - Nearest-Neighbor-Klassifikatoren - etc. Habe ich alles schon ausprobiert. Funktioniert so lala ... Davon verstehe ich nichts. Kann man das denn nicht auch biologisch angehen? Aus der Lage der Trennebene können wir schließen, welche Gene für die Diagnose wichtig sind. Leider gibt es immer viele Trennebenen ... Gen A Gen B Beide ... und bei tausenden Genen erhält man von jeder Trennebene natürlich eine ganze Liste von Genen, die für die Diagnose wichtig sind. ... Von vielen Gene kennt man ihre Funktion in der Zelle Das wollen wir uns zu Nutze machen Trennebene 1 • DNA Repair • DNA Repair • Ligase • DNA Repair • Replikation • Ligase • DNA Repair ... Trennebene 2 • Imunsystem • Zellskelett • O Transport 2 • Ionenkanal • Histon • Kinase • Replikation ... Die erste Liste macht Sinn, die zweite sagt mir nichts! Sehe ich auch so Interessant! Ich habe noch ein Paar tausend solcher Listen. Könnt ihr euch die auch noch anschauen! Nein! Das kann man auch systematischer angehen! Schließlich bestimmen wir die Trennebene oben ja auch nicht durch ausprobieren! Wir sollten das Problem mit gemeinsamen Kräften angehen! Die Computational Diagnostics Gruppe und einige Kooperationspartner Stefan Bentink Prof. Ludwig Claudio Lottaz Florian Markowetz Prof. Hagemeier Dr. Seeger Stefanie Scheid Rainer Spang Prof. Nevins Dr. Kirschner Jochen Jäger Prof. West Dr. Ruiz Prof. Schreiber Dr Ivandic Um nur ein Paar zu nennen Projekte - Leukämie - Brustkrebs - Herz-Chip - Morbus-Krohn Zusammenfassung 1. Hochauflösende Diagnostik verbessert Therapieerfolg. 2. Genexpressionprofile sind sehr umfassende diagnostische Befunde mit häufig direktem Bezug zur Ursache der Krankheit. 91,7% 75% 3. Diagnostik basierend auf hochdimensionalen Befunden führt zu Identifikations-Problemen (zuviele Trennebenen). 4. Wir vermuten, daß in Datenbanken gespeicherte Informationen über die Funktion einzelner Gene uns dabei helfen werden, dieses Problem zu überwinden. Unser Forschungsprogramm Das Umfeld in Berlin Kommen Sie gut nach Hause! Gute Nacht!