PDF - BioProducts
Werbung

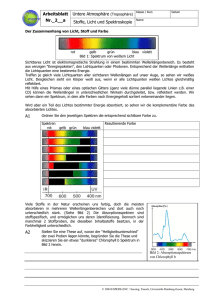
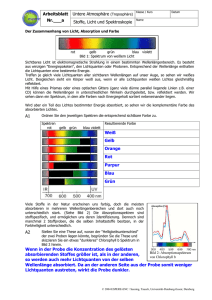
KRAS – ein prädiktiver Marker beim Dickdarmkarzinom Angelika Reiner-Concin Pathologisch-Bakteriologisches Institut Donauspital Wien Mammakarzinom Dickdarmkarzinom KRAS-Mutation • • • • • meist früh in der Karzinogenese > 30% der Colonca 3000 KRAS-Punktmutationen beschrieben single Aminosäurensubstitution unabhängig von EGFR-Mutationen • 82% Codon 12 • 17% Codon 13 • Codon 12 und 13-Mutationen – entscheidend für Progression – prädiktiv für Therapie EGF, TGFα EGF receptor mitogenic responses KRAS BRAF MEK ERK/MAP Patienten mit Wildtyp-KRAS 1.0 p < 0.0001 0.9 Median Mean Events/N (%) In Weeks In Weeks Proportion with PFS 0.8 0.7 0.6 Pmab + BSC 115/124 (93) BSC Alone 114/119 (96) 12.3 7.3 19.0 9.3 HR = 0.45 (95% CI: 0.34–0.59) Stratified log-rank test, p < 0.0001 0.5 0.4 0.3 0.2 0.1 0.0 2224242626282830 3032 3234 3436 3638 3840 4042 4244 4446 4648 4850 5052 52 0 2 4 6 8 10 12 14 16 18 20022 Weeks R.G. Amado, European Journal of Cancer Supplements, Vol 5 No 6, Page 8 Testung von KRAS-Mutationen • Diskriminierung zwischen mutierten und Wildtyp-Allelen • Basis für therapeutischen Entscheidung • Bei jedem primären Colonca Stadium II und III • Unterschiedliche Detektionslimits für einzelne Mutationen • Mutation heterozygot oder homozygot • Heterogenität des Untersuchungsmaterials – Mischung von Tumor und benignem Gewebe • PCR hohe Sensitivität – aus DNA vom Gewebsextrakt JHJM van Krieken et al. Virchows Arch 2008 Paraffinblock und Tumorareal auswählen Im Idealfall > 70% Ca Was entdeckt der Array -Test? Wild Typ Supressor Compound Interpretation Erfahrung erforderlich für Einstellung des Tresholds zur Reduktion des Hintergrundes! Fall 10 des Ringversuchs 10% Tumorgewebe 9ng/µl H2O 0,1 WSC 0,5 WSC Eigene Ergebnisse n = 91 Wildtyp 53 58% Mutationen 38 42% LCD Array 87% Cd 12 Cd 12 – Val 9 Cd 12 – Ala 3 Cd 12 – Asp 11 Cd 12 – Ser 3 Cd 12 – Cys 5 Cd 12 – Arg 2 23% 29% 13% Cd 12 – Ile Cd 12 – Leu 13% Cd 13 Cd 13 – Asp 4 Cd 13 – Arg Cd 13 – Cys 1 Ergebnisse des Pilotversuchs zum KRAS-Ringversuchs der ÖGP % Tumor Teilnehmer Methode 1 2 3 DxS Dxs DHPLC, Sequ, RTPCR 4 5 6 7 8 9 10 11 LCD Array Kapillarseq uenzierung DxS DxS DxS DxS Strip Assay LCD Array Probe 1 WT 60 WT 12ASP WT WT WT WT WT WT WT WT WT Probe 2 12VAL 70 12VAL 12VAL/ 12ASP 12VAL 12VAL 12VAL 12VAL 12VAL 12VAL 12VAL 12VAL 12VAL Probe 3 12ALA 5 12ALA 12ALA 12ALA 12ALA 12ALA 12ALA 12ALA 12ALA 12ALA 12ALA 12ALA Probe 4 WT 70 WT WT WT WT WT WT WT WT WT WT WT Probe 5 12ASP 70 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP Probe 6 12ASP 25 12ASP 12ASP 12ASP WT 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP 12ASP Probe 7 WT WT WT WT WT WT WT WT WT WT WT WT Probe 8 13ASP 10 13ASP 13ASP 13ASP WT 13ASP 13ASP 13ASP 13ASP 13ASP 13ASP 13ASP Probe 9 WT 50 12ASP 12ASP 12ASP WT WT WT WT WT WT WT 12ASP Probe 10 12 CYS 10 12 CYS 12 CYS 12 CYS 12 CYS WT 12 CYS 12 CYS 12 CYS 12 CYS 12 CYS 12 CYS 5 Was ist speziell am Array-Test? • Unterdrückung des Wildtyps • einfaches Equipment – Software zur Auswertung – Einstellung der Treshholds für Auswertung • DNA-Quantifizierung im Test nicht gefordert