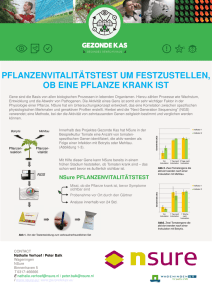
Monika Neugebauer - Universität Hohenheim
Werbung

Universität Hohenheim Institut für Physiologie und Biotechnologie der Pflanzen Prof. Dr. A. Schaller Zur Rolle der Subtilase 3 aus Tomate (Solanum lycopersicum) in der Pathogenabwehr Diplomarbeit Monika Neugebauer Studiengang Biologie, Fakultät Naturwissenschaften September 2008 betreut von Dr. Anna Cedzich In Gedenken an Birgit für Harald und Jana Zusammenfassung Subtilasen stellen eine der größten Gruppen der in Pflanzen vorrangig präsenten Familie der Serinproteasen dar. Bisher wurden 15 Subtilasen in Solanum lycopersicum identifiziert. Für viele wird eine Funktion in der Pathogenabwehr postuliert. Im Rahmen dieser Arbeit sollte die Expression der SlSBT3 nach Pathogenbefall mittels SlSBT3-Promotor::GUS-Pflanzen, semiqRT-PCR, Northern und Western Blot analysiert werden. Darüber hinaus erfolgte eine vergleichende Untersuchung des Wachstumsverhaltens und der Krankheitssymptome verschiedener Tomatenpathogene in SlSBT3 RNAi-, Überexpressions– und Wildtyppflanzen. Über die exogene Zugabe von Salicylsäure konnte eine Abhängigkeit der Expression der SlSBT3 von diesem Signalmolekül der biotrophen Pathogenabwehr festgestellt werden. Eine Induktion der SlSBT3 wurde sowohl nach Infektion durch avirulente, virulente oder hrp-defiziente Stämme eines biotrophen Bakteriums, als auch nach Infektion durch einen hemibiotrophen Oomyceten und nekrotrophen echten Pilz nachgewiesen. Dabei erwies sich die Expression am stärksten induziert durch Infektion mit dem avirulenten Stamm Xanthomonas campestris pv. vesicatoria, nach einer langen Besiedlungsphase von Phytophthora infestans und Sclerotinia sclerotiorum. Die Infiltration von Katalase in Tomatenblätter gefolgt von einer Inokulation mit S. sclerotiorum führte zu einer früheren Induktion von SlSBT3 im Vergleich zu Kontrollen die nur mit S. sclerotiorum infiziert waren. Dies wies daraufhin, dass H2O2 eine reprimierende Wirkung auf die Expression in diesem Pathogen-WirtspflanzeSystem hat. Als übereinstimmendes Charakteristikum der sonst so unterschiedlichen Pathogene wurde die Abhängigkeit der SlSBT3-Expression von der Einleitung des Zelltods genauer untersucht. So konnte auch unabhängig von Pathogenbefall mit dem Zellgift Cantharidin einer Induktion der SlSBT3 festgestellt werden. In der Pflanze der RNAi-Linie war das Wachstum der biotrophen Bakterien tendenziell am schlechtesten. Zusammen mit dem verstärkten Auftreten von Krankheitssymptomen in der RNAi-Linie durch den hrp-defizienten X. campestris und S. sclerotiorum lässt diese Beobachtung eine indirekte Beteiligung an der Pathogenabwehr auf Ebene der Zelltodregulation vermuten. I Inhalt 1 Zusammenfassung I Inhalt II Abkürzungsverzeichnis VII Abbildungsverzeichnis X Einleitung 1 1.1 Die Besiedlung von Pflanzen durch Pathogene 1 1.2 Die Erkennung von Pathogenen durch pflanzliche Organismen 1 1.3 Abwehrmechanismen und Signalkomponenten in der Pathogenabwehr 3 1.3.1 Hypersensitive Reaktion und systemisch erworbene Resistenz 4 1.3.2 Reaktive Sauerstoffspezies in der Pathogenantwort 5 1.3.3 Abwehrstrategien gegen biotrophe oder nekrotrophe Pathogene 6 1.3.4 Pflanzliche Proteasen 7 1.3.5 Pflanzliche Subtilasen 9 1.3.6 Die Subtilasenfamilie in Tomate (Solanum lycopersicum) 11 Zielsetzung 12 1.4 2 2.1 Material und Methoden 15 Material 15 2.1.1 Pflanzen 15 2.1.2 SlSBT3-Promotor::GUS-Linien 15 2.1.3 SlSBT3-RNAi-Linien 15 2.1.4 SlSBT3-Überexpressions-Linien 16 II 2.2 2.1.5 Bakterien 16 2.1.6 Pilze: Sclerotinia sclerotiorum 16 2.1.7 Oomyceten: Phytophthora infestans 17 2.1.8 Plasmide 17 2.1.9 Oligonukleotide 17 2.1.10 Enzyme 18 2.1.11 Antikörper 18 2.1.12 Chemikalien 18 2.1.13 Geräte 19 Methoden 20 2.2.1 Anzucht von Tomatenpflanzen 20 2.2.1.1 2.2.1.2 Behandlung der Tomatensamen zur Dekontamination von Viren 20 Anzuchtbedingungen 20 2.2.2 Kultivierung von Xanthomonas campestris pv. vesicatoria 20 2.2.3 Kultivierung von Sclerotinia sclerotiorum 21 2.2.4 Kultivierung von Phytophthora infestans 22 2.2.5 Infiltration von Xcv in Tomatenblätter 22 2.2.6 Reisolierung von Xcv aus Blattgewebe 23 2.2.7 Statistische Auswertung des Wachstumsverhaltens von Xcv 24 2.2.8 Inokulation von Tomatenblättern mit S. sclerotiorum 25 2.2.9 Inokulation von Tomatenblätter mit P. infestans 25 2.2.10 Behandlung von Tomatenpflanzen mit Salicylsäure 26 2.2.11 Infiltration von Katalase 26 2.2.12 Infiltration von H2O2 27 2.2.13 Infiltration von Cantharidin 27 2.2.14 GUS-Färbung 27 2.2.15 DNA-Extraktion 28 III 2.2.16 Gelektrophorese von DNA 28 2.2.17 RNAse-freies Arbeiten 29 2.2.18 RNA-Extraktion 29 2.2.19 Semiquantitative RT-PCR 31 2.2.19.1 RT-Reaktion 31 2.2.19.2 PCR 32 2.2.20 Northern Blot 34 2.2.20.1 Sondenherstellung für Northern Blot 34 2.2.20.2 Gelelektrophorese von RNA 35 2.2.20.3 RNA-Transfer auf Membranen 36 2.2.20.4 Hybridisierung mit einer radioaktiv-markierten Sonde 37 2.2.20.5 Strippen von Membranen 39 2.2.21 Proteinanalyse 39 2.2.21.1 Herstellung von Gesamtproteinextrakten 39 2.2.21.2 Bestimmung der Proteinkonzentration nach Bradford 39 2.2.21.3 SDS-PAGE 40 2.2.21.4 Coomassie-Färbung 41 2.2.21.5 Immunologischer Nachweis von Proteinen 2.2.21.6 mit Western Blot 41 Ponceau-Färbung 43 IV 3 Ergebnisse 44 3.1 Überprüfung von SlSBT3-RNAi- und -Überexpressions-Linien in der T3Generation 44 3.2 Expression von SlSBT3 nach Behandlung mit Salicylsäure 45 3.3 Expression von SlSBT3 in Tomatenblättern nach Infektion durch Xcv 46 3.4 Wachstumsverhalten von Xcv in SlSBT3 RNAi-, 3.5 Überexpressions- und Wildtyppflanzen 54 3.4.1 Wachstum des virulenten Stamms Xcv 85-10 54 3.4.2 Wachstum des avirulenten Stamms Xcv 85-10 pDsK 200 55 3.4.3 Wachstum des hrp-defizienten Stamms Xcv 85-10 hrpA-G 56 Vergleich der Krankheitssymptome in SlSBT3 RNAi-, Überexpressions- und Wildtyppflanzen nach Infektion durch Xcv 3.6 Expression von SlSBT3 in Tomatenblättern nach Infektion durch S. sclerotiorum 3.7 4 63 Expression von SlSBT3 in Tomatenblättern nach Infektion durch P. infestans 3.9 59 Vergleich der Krankheitssymptome in SlSBT3 RNAi-, Überexpressions- und Wildtyppflanzen nach Infektion durch Xcv 3.8 57 64 Expression von SlSBT3 nach Behandlung mit Katalase und Cantharidin 66 3.9.1 GUS-Repotergenanalyse nach Infiltration von Katalase 67 3.9.2 GUS-Reportergenanalyse nach Infiltration von Cantharidin 68 Diskussion 71 4.1 Beeinflussung des Expressionsstatus der SlSBT3 durch Pathogenbefall 71 4.2 Einfluss von Salicylsäure und H2O2 auf die Expression der SlSBT3 77 V 4.3 4.4 Einfluss der SlSBT3 auf das Wachstum von Pathogenen in Tomatenblättern 79 Die Rolle der SlSBT3 in der Pathogenabwehr 82 5 Ausblick 87 6 Literatur 89 Erklärung XII Danksagung XIII VI Abkürzungsverzeichnis Ø Durchmesser ‚ Minute ’’ Sekunde °C Grad Celsius Cef Cefotaxim Ci Curie Abb. Abbildung BPB Bromphenolblau bp Basenpaare BSA Rinderserumalbumin ca. circa cDNA complementary DNA cfu colony forming units ddH2O doppelt destilliertes Wasser dH2O einfach destilliertes Wasser DEPC Diethylpyrocarbonat DMSO Dimethylsulfoxid dNTP Desoxy-Nukleotidtriphosphat DNA Desoxyribonukleinsäure DNase Desoxyribonuklease ECL enhanced chemiluminescence E. coli Escherichia coli EDTA Ethylendiamintetraessigsäure EtOH Ethanol Fw forward g Erdbeschleunigung g Gramm GUS β-Glucuronidase h Stunde HR hypersensitive Reaktion hrp hypersensitive Reaktion und Pathogenese VII kb Kilobasenpaare kDa KiloDalton Konz. Konzentration µ mikro (10-6) m milli (10-3) M molar (mol/Liter) mA milliAmpere max. maximal min Minute(n) MOPS 4-Morpholinpropansulfonsäure mRNA Messenger-RNA ORF open reading frame PAMP pathogen-associated patterns pBS plasmid Blue Script PCR Polymerase-Kettenreaktion PR Pathogenisis related pv Pathovar OE Überexpression Rif Rifampicin RNA Ribnukleinsäure RNAi RNA-Interferenz RNase Ribonuklease rpm Umdrehungen pro Minute RT Raumtemperatur RT-PCR Reverse Transkriptions-PCR rv Reverse SA Salicylsäure SBT Subtilase sec Sekunden SDS Natriumdodecylsulfat SDS-PAGE SDS-Polyacrylamid-Gelelektrophorese Spec Spectinomycin t Zeit TAE tris-Essigsäure-EDTA VIII TNP Trinatriumphosphat TTSS Typ-III-Sekretionssystem Tween20 Polyoxyethylensorbitanmonolaurat U Enzymeinheiten UBQ Ubiquitin ÜN über Nacht UV Ultraviolett V Volt v/v Volumenprozent verd. verdünnt Vol. Volumen wt Wildtyp w/v Gewichtsprozent XC Xylencyanol Xcv Xanthomonas campestris pv. vesicatoria X-Gluc 5-bromo-4-chloro-3-Indolylglucuronid IX Abbildungsverzeichnis Abb. 3.1: Überprüfung der SlSBT3-Überexpressions-Linie G2f1D und SlSBT3-RNAi-Linie 21-7 Abb. 3.2.1: Expressionsanalyse von SlSBT3 von Tomatenblättern nach Besprühung mit Salicylsäure mit semiqRT-PCR Abb. 3.2.2: Expressionsanalyse von SlSBT3 in Tomatenblättern nach Zufuhr von Salicylsäure durch den Transpirationsstrom mit semiqRT-PCR Abb. 3.3.1: Expressionsanalyse von SlSBT3 nach Infektion durch Xcv 85-10 mit semiqRT Abb. 3.4.1: Wachstum virulenter Stamms von Xcv in SlSBT3–RNAi, Überexpressions- und Wildtyppflanzen. Abb. 3.4.2: Wachstum avirulenter Stamm Xcv 85-10 pDSk200 in SlSBT3– RNAi, -Überexpressions- und Wildtyppflanzen. Abb. 3.4.3: Wachstum hrp-defizienter Stamm Xcv 85-10 A-G in SlSBT3– RNAi, - Überexpressions- und Wildtyppflanzen Abb. 3.5.1: Krankheitsymptome durch Xcv 85-10 in SlSBT3–RNAi, Überexpressions- und Wildtyppflanzen Abb. 3.5.2: Krankheitsymptome durch Xcv hrp A-G in SlSBT3–RNAi, Überexpressions- und Wildtyppflanzen Abb. 3.6.1: Expressionsanalyse von SlBT3 nach Inokulation mit S.sclerotiorum X Abb. 3.6.2: Histochemische Lokalisation der SlSBT3-Promotor::GUS Aktivität nach Inokulation mit S. sclerotiorum Abb. 3.7: Krankheitssymptome durch S. sclerotiorum in SlSBT3–RNAi, Überexpressions- und Wildtyppflanzen Abb. 3.8.: Expressionsanalyse von SlBT3 nach Inokulation mit P. infestans Abb. 3.9.1: Histochemische Analyse der SlSBT3-Promotor::GUS Aktivität nach Infiltration mit Katalase und gleichzeitiger Inokulation mit S. sclerotiorum Abb. 3.9.2: Histochemische Analyse der SlSBT3-Promotor::GUS Aktivität nach Infiltration von Cantharidin XI Einleitung Einleitung 1.1 Die Besiedlung von Pflanzen durch Pathogene Die Besiedlung von Pflanzen durch mikrobielle Krankheitserreger ist in der Natur ein allgegenwärtiges Ereignis. Für das Wachstum und die Entwicklung eines Pathogens auf einer Pflanze ist die Überwindung der so genannten basalen Inkompabilität notwendig. Auf Nichtwirtspflanzen können Pathogene die vorliegenden Barrieren nicht überwinden. Dazu tragen die bestehenden strukturellen oder chemischen Eigenschaften der jeweiligen Pflanzen bei, die bereits ohne Einwirkung von Pathogenen vorhanden sind. Zu strukturellen Barrieren gehören die pflanzlichen Abschlussgewebe wie z. B. die Kutikula auf der Epidermis. Zu chemischen Barrieren gehören eine Reihe von niedermolekularen, antimikrobiell wirkenden Stoffen. Die so genannten Phytoantizipine werden konstitutiv im Sekundärstoffwechsel gebildet; um dabei in bestimmte Strukturen oder in bestimmten Kompartimenten gelagert zu werden und entfalten ihre antimikrobielle Wirkung oft erst nach Gewebezerstörung (Osbourn, 1996). Die Überwindung dieser präfomierten Resistenzfaktoren der Pflanze durch Ausbildung entsprechender Pathogenitätsfaktoren ermöglicht dem Pathogen die Besiedlung des Pflanzengewebes. Entscheidend dabei sind in jedem Fall die Genotypen der Interaktionspartner. Damit ist die Ausprägung einer basalen Kompatibilität spezifisch gegen den jeweiligen Organismus gerichtet. 1.2 Die Erkennung von Pathogenen durch pflanzliche Organismen Neben den präformierten passiven Resistenzfaktoren werden potentielle Pathogene durch Signal-Sensor-Reaktionen erkannt. Die Erkennung typischer Pathogen-assoziierter Strukturen (pathogen- associated Patterns, PAMPS; microbial-associated Patterns, MAMPS) von Rezeptoren der Zelloberfläche von Pflanzen wie z. B. die eines 22 Aminosäuren großen Peptids flg22 des hochkonservierten Aminoterminus 1 Einleitung von Flagellin über die leuzinreiche (LRR)-Rezeptorkinase FLS2 aus Arabidopsis (Felix et al., 1999; Chinchilla et al., 2006) stellt einen ersten Schritt der effektiven Pathogen-Abwehr dar. Die daraus resultierende erfolgreiche Abwehr wird auch PAMP-abhängige Immunität genannt oder vereinfacht mit dem Begriff der basalen Resistenz umschrieben (Chisholm et al., 2006; Jones und Dangl, 2006). Durch Pathogene, die diese erste Abwehr überwinden können, folgt eine Effektoren-gesteuerte Anfälligkeit in der Pflanze, die in einer weiteren Phase durch die spezifische Erkennung des Effektors in einer Effektor-vermittelten Immunität enden kann (Jones und Dangl, 2006). Das Phänomen der Immunität von Pflanzen wurde schon 1956 in der Pathogen-Wirtspflanze-System Flachsrost (Melamspora lini) von Flachs beobachtet (Linum (Flor usitatissimum) 1956). und Genetische Untersuchungen in diesem Pathosystem führten zu der „Gen für Gen“– Hypothese, nach der für jedes Resistenz-Gen (R-Gen) in einer Pflanze ein komplementäres Avirulenz-Gen (Avr-Gen) im Pathogen existiert (Flor, 1971). Zwei unterschiedliche molekulare Mechanismen werden für die Erkennung von Avr-Genen durch die R-Gene postuliert. Bei dem Rezeptor-Liganden Modell wird angenommen, dass das RGenprodukt als Rezeptor des Avr-Genprodukts dient und ein Komplex aus beiden die Resistenz induziert. Viele R- und Avr-Gene wurden identifiziert (Baker et al., 1997) und eine direkte Interaktion von R- und Avr-Genprodukt konnte bisher vereinzelt nachgewiesen werden. So für das Avr-Pto aus Pseudomonas syringae und Pto aus Tomate (Tang et al., 1996) oder PopP2 aus Ralstonia solanacearum und rRRS1-PR aus Arabidopsis (Deslandes et al., 2003). Die identifizierten R-Gene zeichnen sich oft durch hohe Strukturähnlichkeit in rezeptor-ähnlichen Leuzin-reichen (LRR)-Domänen und Nukleotid-bindende-Sequenzen (NBS) aus, was nahe legt, dass die Sensoren der Pathogenerkennung und die eingeleiteten Abwehrsignalwege konserviert sind (Hammond-Kosack und Parker, 2003). Typische LRR-NBS-Strukturen sind bekannt für das RPM1 aus Arabidopsis thaliana, das gegen avrRPm1 aus Pseudomonas syringae gerichtet ist (Grant et al., 1995), oder das RPS2 aus Arabidopsis, welches mit avrRpt2 aus Pseudomonas interagiert (Bent et al., 1994; Mindrinos et al., 1994). 2 Einleitung Die Beobachtung, dass das Resistenzgenprodukt Pto aus Tomate, eine Serin/Threonin-Proteinkinase, mit einem weiteren Protein mit typischer NBSLRR-R-Protein-Struktur, dem Prf, interagiert, stützt das Modell eines weiteren molekularen Mechanismus neben dem postulierten Rezeptor-LigandenModell. Nach dem Guard-Modell kommt es durch Interaktion von Avirulenzgenprodukt und einem ersten Resistenz- oder Virulenzzielprotein zur strukturellen Konformationsänderungen, die von einem zweiten Resistenzprotein, dem Guard oder Wächter, erkannt wird. Erst dadurch wird letztendlich eine Abwehrreaktion ausgelöst (Dangl und Jones, 2001). Können die Genprodukte des Avirulenzgens nicht von denen des RGenprodukts erkannt werden, folgen Wachstum der Pathogene und die Ausprägung ihrer Virulenzfaktoren, was zur Bildung von Krankheitssymptomen führt. Diese kompatible Interaktion resultiert in der vollständigen Etablierung der Pathogens im Blattgewebe der susceptiblen Pflanze. Bei der inkompatiblen Reaktion wird durch Induktion von spezifischen Abwehrmechanismen eine Besiedlung der Pflanze durch das Pathogen unterdrückt. Hier wird das Genprodukt des Avr-Gens von R-Genprodukten erkannt. Bei dieser R-Gen-vermittelten Resistenz oder Wirtsresistenz kommt es vergleichbar der PAMP-abhängigen Immunität zur Einleitung von Abwehrmechanismen. 1.3 Abwehrmechanismen und Signalkomponenten in der Pathogenabwehr Im Gegensatz zu dem Immunsystem der tierischen Organismen verfügen Pflanzen über keine spezialisierten Zellen, die in einem zirkulatorischen System einer Abwehr von Pathogenen dienen könnten. Im pflanzlichen Organismus verfügt jede einzelne Zelle über das Potential Abwehrmechanismen zu entwickeln und systemische Signale, als Folge eines Angriffs mikrobieller Krankheitserreger, auszusenden und zu empfangen (Ausubel et al., 2005; Dangl und Jones, 2001). Hierzu zählt die Induktion von PR (pathogenesis-related)–Proteinen, die Aktivierung oder denovo-Synthese von Resistenzfaktoren. ZU den Resistenzfaktoren gehört die 3 Einleitung Bildung von reaktiven Sauerstoffspezies, Stickoxiden, die Aktivierung der Synthesewege des Sekundärstoffwechsels resultierend in der zusätzlichen Bildung von chemischen Barrieren, wie Phytolalexinen, und mechanischen Barrieren, wie z. B der Verstärkung der Zellwand durch Bildung und Einlagerung von Kallose, Silikaten und Lignin und der Induktion von Zellwand-vernetzender Proteine (Glazebrook et al.,2005). Ungeachtet der Vielfalt der Pathogen-Wirtspflanze-Interaktionen können die Abwehrmechanismen vereinfacht auf wenige molekulare Mechanismen zurückgeführt werden: Die Induktion von PR-Genen, die Auslösung von Zelltod-Mechanismen und die vermehrte Bildung von Signalmolekülen, wie Salicylsäure, Jasmonsäure oder Ethylen, die ihre Wirkung nicht nur auf infizierte Bereiche sondern auch weiter entfernte Bereiche entfalten können. 1.3.1 Hypersensitive Reaktion und systemisch erworbene Resistenz Eine der ersten Reaktionen der R-Gen-vermittelten Resistenz bei Pathogenbefall der Pflanze ist häufig eine lokal begrenzte Resistenzreaktion. Die Bildung reaktiver Sauerstoffspezies (reactive oxygen species, ROS) im Apoplasten, der „oxidative burst“, führt zur hypersensitiven Reaktion und zur Akkumulation von Salicylsäure (Glazebrook et al., 2005; Grant et al., 2000). Die hypersensitive Reaktion (HR) ist durch schnellen Zelltod der infizierten Zellen charakterisiert (Hammond-Kosack et al., 1996). Neben den Abwehrmechanismen, die durch die Erkennung in der betroffenen Zelle ausgelöst werden, findet auch eine Immunisierung des umliegenden Gewebes und der gesamten Pflanze statt. So kommt es in den restlichen Pflanzenorganen nach der Bildung von Nekrosen, die ein Teil der HR oder auch Krankheitssymptom sein können, zur so genannten systemisch erworbenen Resistenz (SAR). Die SAR zeichnet sich durch die Induktion von PR-Proteinen und Akkumulation von Salicylsäure als Regulator der PRProtein-Induktion in den nicht befallenen Geweben und Organen aus. Infolgedessen kann eine lang anhaltende, bisweilen lebenslange Resistenz gegenüber einem breiten Spektrum von Pathogenen beobachtet werden (Durrant und Dong, 2004). 4 Einleitung 1.3.2 Reaktive Sauerstoffspezies in der Pathogenantwort Die Bildung reaktiver Sauerstoffspezies gehört zu den ersten universellen Reaktionen auf biotischen Stress. Die Bildung von ROS ist auf die Aktivität von pH-abhängigen Zellwand-Peroxidasen und der NADPH-Oxidase zurückzuführen (Torres et al., 2006). So zeigten z. B. atrbohD und atrbohF, Mutanten für Homologe der katalytischen Untereinheit der NADPH-Oxidase in Arabidopsis, reduzierte HR nach Befall durch einen avirulenten Stamm von Pseudomonas syringae (Torres et al., 2002). Durch die Aktivität dieser Enzyme wird im Apoplasten aus Sauerstoff das Superoxidanionradikal (O2) generiert, das durch die Superoxiddismutase (SOD) in Wasserstoffperoxid (H2O2) umgesetzt werden kann. Beide können wiederum in Gegenwart von Metall-Ionen in einer so genannten Haber-Weiss-Reaktion in hochreaktive Hydroxylradikale umgesetzt werden (Gechev et al., 2006). ROS sind sehr instabil. Sie erweisen sich als direkt toxisch gegenüber Pathogenen und sind in hohen Konzentrationen auch toxisch für die Pflanzenzelle. Bis auf das Hydroxylradikal ist es der Pflanzenzelle möglich, die ROS zu detoxifizieren und ein Gleichgewicht aufrechtzuerhalten (Mittler et al., 2004). Dazu dienen verschiedene reduzierende Moleküle und Enzyme wie Ascorbat, Gluthathion und Ascorbat-Peroxidasen, Superoxiddismutasen oder Katalasen. Durch diese Kontrolle sind vielfältige Funktionen möglich. ROS tragen durch Vernetzung von Lignin und Verknüpfung von hydroxyprolin-reichen Proteinen mit Polysacchariden der Zellwand zur Bildung zusätzlicher mechanischer Barrieren bei. Daneben ist nachgewiesen, dass ROS als Signale in der Abwehr-abhängigen Genaktivierung wirken und zusammen mit anderen Signalmolekülen, wie z.B. Salicylsäure (SA) und Stickoxiden (NO), regulatorische Funktion übernehmen können. Das stabilste Intermediat, Wasserstoffperoxid (mit einer Halbwertsszeit von etwa 1 ms), das frei über die Zellmembran diffundieren und in das umliegende Pflanzengewebe entweichen (Gechev et al., 2006) kann, gilt dabei als Signalmolekül in der Auslösung des programmierten Zelltods, bei der Induktion bestimmter Wasserstoffperoxid-abhängiger Gene und von PRGenen (Levine et al., 1994, Vandenabeele et al., 2003, Torres et al., 2006). 5 Einleitung Demzufolge beeinflusst die Aktivität der Katalase das Ausmaß des programmierten Zelltods. So zeigt z. B. die cat1 Mutante in Tabak unter abiotschem Stress Induktion von PR-1 und vermindertes Wachstum eines avirulenten Stamms von Pseudomonas syringae (Chamnongpol et.al., 1996). 1.3.3 Abwehrstrategien gegen biotrophe oder nekrotrophe Pathogene Die Aktivierung einer Salicylsäure-abhängigen Resistenz ist als eine Folge des „oxidative burst“ und des R-Gen vermittelten Abwehrmechanismus gegenüber biotrophen Pathogenen zu betrachten (Glazebrook et al.; 2005). Modifizierte Pflanzen, die keine oder nur vermindert Salicylsäure in produzieren, zeigen eine verstärkte Anfälligkeit gegenüber biotrophen Organismen (Delaney et al.; 1994). Die HR als Ergebnis dieser Aktivierung erweist sich als erfolgreiche Abwehrstrategie zur Begrenzung des Pathogenbefalls, da durch das Absterben eines lokalen Bereichs dieser Art von Pathogenen die Nährstoffquelle entzogen wird. Die Akkumulation von Salicylsäure führt zur Induktion von PR-Proteinen in umliegenden Geweben und Organen und kann außerdem in flüchtiger Form als Methylsalicylat auch zwischen Pflanzen zur SAR führen (Durrant und Dong, 2004; Shulaev et al., 1997; Park et al., 2007). Die Auslösung von HR stellt bei Befall durch Nekrotrophe kein effektiver Abwehrmechanismus dar. Abwehrreaktionen gegen solche Pathogene sind im Allgemeinen auf Jasmonsäure (JA)- und Ethylen-induzierte Mechanismen zurückzuführen und überlappen mit den Abwehrmechanismen ausgelöst durch Verwundung und Insektenbefall (Glazebrook, 2005). In Arabidopsis konnte nach Inokulation mit dem nekrotrophen Pilz Alternaria brassicicola gezeigt werden, dass die antimikrobiellen Peptide Thi2.1 und PDF1.2 unabhängig von Salicylsäure durch Jasmonsäure und Ethylen induziert werden. Außerdem wurde festgestellt, dass die Expression von Thi2.1 und PDF1 in der Jasmonsäure-insensitiven Mutante coi1 und Ethylen-insensitiven Mutante ein1 in Arabidopsis ausbleibt (Penninckx et al., 1996). Unabhängig von Salicylsäure wird in Arabidopsis durch Jasmonsäure (JA) und Ethylen die PR-Genexpression induziert und die Abwehr von virulenten Stämmen von Pseudomonas syringae und von Fusarium oxysporum eingeleitet (Dong, 1998). Diese antagonistische Interaktion zwischen SA und JA-abhängigen 6 Einleitung Signalwegen von Pathogene zur erfolgreichen Infektion genutzt. In Arabidopsis ausgelöst konnte durch nach Aktivierung Pseudomonas des syringae SA-ahängigen eine verstärkte Signalwegs Anfälligkeit gegenüber dem nekrotrophen Pilz Alternaria brassicicola beobachtet werden (Koorneef und Pieterse, 2008). Synergistische Interaktionen zwischen SAund JA eingeleiteten Abwehrwegen sind daneben auch bekannt (Van Wees et al., 2000). Diese Abhängigkeiten der jeweiligen Signalwege ermöglicht damit einer Reihe von Pathogenen die Besiedlung der Pflanze (Pieterse und Dicke, 2007). Insgesamt ist die Abwehr von nekrotrophen und biotrophen nicht nur auf SA oder JA-abhängige Signalwege beschränkt, sondern kann auch durch den Einfluss anderer Moleküle mit Signalcharakter, wie Abscisinsäure und Ethylen, zurückgeführt werden (Koorneef und Pieterse, 2008). 1.3.4 Pflanzliche Proteasen Über 500 Proteasen sind im Arabidopsis-Genom codiert (Beers et al., 2004). Die größte Funktion der proteolytisch aktiven Enzyme wurde bisher bei der Regulierung Proteasen des eine Proteinhaushalts komplexe vermutet. regulatorische Mittlerweile Funktion im wird vielen pflanzlichen Organismus aufgrund ihrer hohen Substratspezifität, die durch limitierte Proteolyse erreicht wird, zugeordnet (van der Hoorn und Jones, 2004; Xia, 2004). Die Rolle von pflanzlichen Proteasen in der Pathogenabwehr ist in Modellen in der Abbildung 1 zusammengefasst. Es ist möglich, dass Proteasen entsprechende Elicitoren erkennen und als Folge dieser Interaktion Abwehrreaktionen ausgelöst werden. So können Elicitoren durch Proteasen aktiviert werden, oder aber die Elicitoren beeinflussen die Aktivität der Proteasen, was in Abwehrreaktionen resultiert (Abb. 1. a-c). Desweiteren gibt es Hinweise, dass durch die proteolytische Aktivität Regulatoren prozessiert werden, die daraufhin ihre Wirkung in einem bestimmten Abwehrsignalweg entfalten können (Abb. 1. d und e). Zuletzt können Effektoren durch proteolytischen Abbau direkt unschädlich gemacht, peptidbasierende Toxine freigesetzt oder weitere Abwehrproteine aktiviert werden (Abb. 1. f-h). 7 Einleitung Abb. 1.1 : Modelle für die Rolle von Proteasen in der Pathogenabwehr. Proteasen können in der Rezeption von Elicitoren fungieren (a-c); durch Prozessierung von Effektoren die Abwehr beeinflussen (d, e); Abbau von Effektoren (f) Prozessierung peptidbasiendender Toxine (g) oder inaktiven Abwehrproteinen (h) dienen. Grüne Pfeile und rote t-Balken dienen zur Kennzeichnung positiver oder negativer Signalwege (van der Hoorn und Jones, 2004). Entsprechend dem Virulenzzielproteine cdr1 (constitutive Guard-Modell diskutiert. disease werden Mit resistance) pflanzliche der konnte Proteasen als T-DNA-Insertionsmutante eine Beteiligung einer apoplastische Aspartat-Protease bei Pathogenbefall gezeigt werden. Die Expression von CDR1 erweist sich nach Pathogenbefall induziert und die cdr1-Mutante zeigt neben vermindertem Wachstum auch Resistenz gegen avirulenten Stamm von P. syringae (Xia et al., 2004). Neueste Beweise für die Beteiligung von Proteasen bei Abwehrmechanismen gegen Pathogene lieferte der Nachweis der Interaktion des Avirulenzgenprodukts AVR2 aus dem Erreger der Blattfleckenkrankheit an Tomate Cladosporium fulvum mit RCR3, einer apoplastischen Cysteinprotease aus Tomate (Rooney et al., 2005). 2008 konnte durch heterologe Expression von AVR2 in Arabidopsis und Tomate eine gestiegene Anfälligkeit nach Befall durch verschiedene prokaryontische und eukaryontische Phytopathogene demonstriert werden. Auf diese Interaktion und die Inhibierung des nachgeschalteten Signalwegs 8 Einleitung über das LRR-NBS-R-Protein Cf2 in planta wird das Abschwächen der basalen Resistenz zurückgeführt (van Esse et al., 2008). Weitere Hinweise auf die Beteiligung von Proteasen bei der Pathogenabwehr ergeben sich aus der Ähnlichkeit pflanzlicher Proteasen zu tierischen Cysteinproteasen, den so genannten Caspasen. Unter Caspasen versteht man Cystein-Proteasen, die in tierischen Organismen an der Apoptose beteiligt sind. Bisher konnte nur die Inhibition von bestimmten pflanzlichen Cystein- und Serinproteasen durch typische tierische Caspaseinhibitoren gezeigt werden, die über eine Funktion solcher Proteasen im Prozess des programmierten Zelltods spekulieren lässt (Bonneau et al., 2008). Die HR der Pathogenabwehr stellt dabei eine Sonderform des programmierten Zelltods (PCD) dar (Dangl und Jones, 2001). Die Entdeckung von Pathogen-sekretierten Protease-Inhibitoren legte Proteasen abermals als Virulenzziele von Effektoren der Pathogene nahe. Pathogene erreichen eine Umgehung der Resistenz durch die Unterdrückung der pflanzlichen Abwehrstrategien. Dazu nutzen sie neben Toxinen und genregulatorisch wirkenden Molekülen im Wesentlichen Proteine zur Änderung des pflanzlichen Metabolismus (Abramovitch und Martin, 2004). So konnten aus dem Erreger der Kraut- und Knollenfäule Phytophtora infestans gleich mehrere sekretierte Peptide identifiziert werden, die mit Cystein- oder Serinproteasen interagieren und ihre Funktion inhibieren (Tian et al., 2007; Tian et al., 2005; Tian et al., 2004). 1.3.5 Pflanzliche Subtilasen Subtilasen gehören zu der Familie der Serin-Proteasen. In der MEROPS Datenbank für Proteasen sind sie aufgrund von Sequenzähnlichkeiten in ihrer Primärstruktur im Clan SB mit der Familie S8 basierend auf Vergleichen der Tertiärstruktur zu finden. Serinproteasen zeichnen sich durch den charakteristischen Aufbau des aktiven Zentrums aus Serin (Ser), His (His) und Asparagin (Asp) in einer katalytischen Triade aus. In dieser räumlichen Anordnung kommt es zum Ladungsaustausch zwischen den Aminosäuren und infolgedessen zum nukleophilen Angriff der Serins an die Carbonylgruppe des Substratpeptids. 9 Einleitung Namensgebend für die Subtilisin-ähnlichen Proteasen oder Subtilasen war das Subtilisin Carlsberg aus Bacillus licheniformis. Anhand von Ähnlichkeiten in ihrer Primärsequenz werden die Subtilasen in weitere sechs Subfamilien unterteilt: Subtilisin, Thermitase, Proteinase K, lantibiotische Peptidasen, Kexine und Pyrolysine (Siezen und Leunissen, 1997). Die Proteasen der Kexin-Subfamilie enthalten eine große Gruppe von Proproteinkonvertasen (PC). Sie sind die ersten Subtilasen die in eukaryontischen Organismen in Form der Kex2p-Protease aus Saccharomyces cerevisiae entdeckt wurden (Julius et al., 1984). Es stellte sich heraus, dass die PCs in der Aktivierung regulatorischer Proteine bzw. Peptide eine Rolle spielen wie z. B. bei der Reifung von Peptidhormonen und Wachstumsfaktoren (Barr 1991, Seidah und Chrétien, 1999). Lediglich zwei der neun bekannten tierischen Subtilasen mit Eigenschaften der PCs werden der nicht der Kexin-Familie zugeordnet, die Site-1-Protease/Subtilisin-KexinIsoenzym-1 der Pyrolysin-Familie (Sakai et al. 1998, Seidah et al., 1999) und die NARC oder PCSK9 Protease K-Familie (Seidah et al., 2003). In der Pyrolysin-Subfamilie sind alle bekannten pflanzlichen Subtilasen zu finden. Die Zahl der Subtilasen, die im Genom von Pflanzen codiert sind, übersteigt die derer in tierischen Organismen. Die erste in Pflanzen identifizierte Subtilase war Cucumisin aus Cucumis melo (Yamatagata et al., 1989 und 1994). Von den 56 in Arabidopsis thaliana identifizierten Subtilasen konnten bis jetzt nur wenigen eine Funktionen zugeordnet werden. Den meisten bisher entdeckten wird eine Funktion ähnlich der PC nachgesagt. Die Analyse der sdd1 (stomatal density and distribution 1)-Mutante in Arabidopsis zeigte, dass die SDD1-Subtilase eine wesentliche Funktion in der Steuerung der Dichte und Verteilung der Stomata erfüllt. Expressionsstudien ergaben, dass SDD1 stark in den stomatären Vorläuferzellen exprimiert wird (Berger und Altmann, 2000). Das SDD1 wird vermutlich in den Apoplast der Zellen sekretiert, wo es an der Prozessierung der für die Kontrolle der Stomatadichte verantwortlichen Proteine beteiligt sein könnte (von Groll et al., 2002). 10 Einleitung Die ALE1 (abnormal leaf shape)-Subtilase wird gewebespezifisch in der Embryoepidermis exprimiert und zeigt sich notwendig für die Bildung der Embryoepidermis (Tanaka et al., 2001). Mit einer T-DNA-Insertions-Mutante für AtS1P konnte deren Beteiligung bei der Salzstressantwort nachgewiesen werden. In-vitro-Experimente zeigten, dass AtS1P einen salz-stressabhängigen bZip-Transkriptionsfaktor spaltet (Liu et al., 2007). 2008 konnte Rautengarten et al. mit einer T-DNA-Insertionslinie für die Arabidopsis Subtilase atSBT1.7 eine weitere Rolle von Subtilasen während der Samenentwicklung nachweisen. AtSbt1.7 wird vorwiegend in den sich entwickelnden Samenschalen exprimiert und die Deletion erweist sich verantwortlich für eine veränderte Bildung der Schleimschicht der äußeren Samenhülle. Eine weitere Funktion von Subtilasen legte die Entdeckung der so genannten Saspasen nahe. Als Saspasen werden Serin-Proteasen mit caspaseähnlichen Eigenschaften bezeichnet (Bonneau et al., 2008). In Avena sativa werden Saspasen während einer durch Dunkelheit induzierten Seneszenz exprimiert. Dabei konnte nachgewiesen werden, dass sie in dem Abbau der Ribulose-1,5-bisphosphat-carboxylase/-oxygenase (RuBisCO) involviert sind, was eine eventuelle Rolle bei dem darauf folgenden programmierten Zelltod nicht ausschließt (Coffeen und Wolpert, 2004). 1.3.6 Die Subtilasenfamilie in Tomate (Solanum lycopersicum) Die Familie der Pyrolysin-Subtilasen in Tomate umfasst 15 identifizierte Mitglieder. Sie ist aufgrund von Sequenzhomologien in fünf Gruppen unterteilt: die Einzelgenfamilen tmp, slsbt1 und slsbt2 und die Multigenfamilien p69 und slsbt3, slsbt4. (Meichtry et al., 1999). Bis auf tmp enthält keines dieser Subtilasengene Introns. Die Ähnlichkeit der jeweiligen Aminosäuresequenzen innerhalb einer Unterfamile beläuft sich auf bis zu 98 %, die der Familien untereinander liegt zwischen 34 % und 54 % (Meichtry et al., 1999). 11 Einleitung Abb. 1.2: Phylogenetischer Baum der Subtilasenfamilie in Tomate (Solanum lycopersicum). Dargestellt ist ein Evolutinsbaum basierend auf Aminosäuresequenzen hergeleitet aus cDNA und den Subtilasengenen. Die Nummern geben die PAM-Distanzen zwischen den Sequenzen an (accepted point mutations per 100 residues). (Meichtry et al., 1999) Über die Funktion der Tomatensubtilasen ist bisher wenig bekannt. In ersten Studien wurde den Tomatensubtilasen aufgrund von Strukturähnlichkeiten eine Funktion ähnlich der PCs zu geschrieben, wie sie mit Identifikation von SBP50, einer Subtilase der Kex2p-ähnlichen Familie, nahe gelegt wurde. SBP50 interagiert mit Systemin, dem Peptidhormon für die wundinduzierte Abwehrreaktion (Pearce et al., 1991) und konnte mit einem Antiserum gegen PCs aus Drosophila nachgewiesen werden (Schaller und Ryan, 1994). Eine Rolle der Tomatensubtilasen in der Pathogenabwehr konnte erstmals 1996 in Viroid-befallenen Tomatenpflanzen entdeckt werden (Tornero et al., 1996 a). Die Spaltung des Proteins LRP, eines extrazellulären Proteins mit LRR-Struktur aus Tomate, wird auf die Induktion von P69-Subtilasen zurückgeführt (Tornero et al., 1996 b). Nachdem weiterer Mitglieder der P69Familie identifiziert wurden, ist diese Funktion auf die Familienmitglieder P69B und P69C eingegrenzt worden. Beide Subtilasen werden nach Inokulation mit einem avirulenten Stamm von Pseudomonas und nach 12 Einleitung exogener Zugabe von Salicylsäure zusammen mit PR-Genen exprimiert (Jordá und Vera, 2000). Für die weiteren Vertreter der P69 Subtilasen kann eine Rolle in der Pathogenabwehr aufgrund ihrer gewebe- und entwicklungsabhängigen Expressionsmuster nicht ausgeschlossen werden. (Jordá und Vera, 2000). Die meisten charakterisierten Subtilasen werden in Geweben oder Entwicklungsstadien exprimiert, die in Form von natürlichen Öffnungen wachstumsfördernde, nährstoffreiche Bereiche für eine erfolgreiche Besiedlung durch Pathogene bezeichnet werden können und ideale Eintrittsorte für Pathogene in die Pflanze darstellen. So konnte zum Beispiel über semiquantitaive RT-PCR und GUS-Reportergenanalysen P69E in allen Entwicklungsstadien der Wurzel und P69F in Hydathoden nachgewiesen werden (Jordá et al., 1999). Expressionsstudien legten für die Vertretrer der SlSBT-Subfamilien ähnliche Expressionsmuster offen, somit kann für die Vertreter der SlSBT-Subfamilien eine ähnliche Funktion in der Pathogenabwehr vermutet werden (Ullrich, Diplomarbeit; Huttenlocher, unveröffentlicht). GUS-Reportergenanalysen zeigten für SlSBT1 eine Expression in Abzissionszonen der Blüten, Früchten und Blättern (Cedzich, pers. Mitteilung). Analysen mit Reportergen, semiquantitativer RT-PCR und Northern Blot konnten für SlSBT2 eine Expression in allen grünen Organen und hier interessanterweise ausschließlich in Zellen der Stomata nachweisen. Zudem konnte das Transkript auch im mikropylaren Endosperm und auf der Oberfläche der Stigma nachgewiesen werden (Kathrin Ullrich, Diplomarbeit). SlSBT3 wurde über GUS-Reportergenanalyse, semiquantitativer RT-PCR und Northern Blot-Analysen in allen Organen und Entwicklungsstadien nachgewiesen, wobei sich eine besonders starke Expression im Wurzelgewebe detektieren ließ. Daneben wurde eine abhängige Induktion in Blättern nach Verwundung und Befall von Fraßschädlingen als auch nach Inokulation mit Xanthomonas campestris nachgewiesen (Franziska Huttenlocher, unveröffentlicht). 13 Einleitung 1.4 Zielsetzung Im Rahmen dieser Arbeit sollte eine mögliche Rolle der SlSBT3 in der Pathogenabwehr detaillierter untersucht werden. Hierzu sollte die Expression nach Befall durch biotrophe Bakterien, einen hemibiotrophen Oomyceten und einen nekrotrophen Pilz und nach Behandlung mit ausgewählten Signalmolekülen untersucht werden. Außerdem sollte eine phänotypische Charakterisierung in SlSBT3-RNA-Interferenz (RNAi)- und SlSBT3- überexpremierenden Tomatenpflanzen bezüglich des Wachstumsverhaltens der Bakterien und der Unterschiede der gebildeten Krankheitssymptome im Vergleich zum Wildtyp durchgeführt werden. 14 Material und Methoden Material und Methoden 2.1 Material 2.1.1 Pflanzen Die verwendeten Tomatenpflanzen (Solanum lycoperiscum; Sl) waren vom Kultivar UC82B. Entsprechende Samen des Wildtyps wurden von Royal Sluis (Enkhuizen, NL) bezogen. 2.1.1.1 SlSBT3-Promotor::GUS-Linien Für die SlSBT3Promotor::GUS Pflanzen war durchgeführt von E. Sieferer, ein 1,9 kb großes Promoterfragment der SlSBT3-Gens mit dem uidA-Gen aus E.coli, das für die für die β-Glucuronidase (GUS) kodiert, in den binären Vektor pBI101 kloniert worden. Durch die Transformation der TomatenWildtyppflanzen mit Agrobakterien wurde eine kanamycinresistente T0Generation generiert, von der drei T2-Linien für die Versuche verwendet wurden: GUS-12-1, GUS-14-2, GUS-5-3. 2.1.1.2 SlSBT3-RNAi-Linien In den RNAi-Linien wurde durch RNA-Interferenz-Technik eine Supression der mRNA der SlSBT3 erzielt. Dazu war von E. Sieferer ein ca. 250 bp großes Fragment vom 5’-Ende der SlSBT3-cDNA in sense und anitsense Orientierung in pHannibal kloniert worden und anschließend die gesamte Expressionskassette in den binären Expressionsvektor pART27 umkloniert worden. Durch Agrobakterien vermittelte Transformation wurde das konstrukt in Tomatenpflanzen eingebracht. Von den fünf zur Verfügung stehenden Linien wurden für die folgenden Versuche die Samen der T2-Generation der Linie SlSBT3 HP-21-7 verwendet. 15 Material und Methoden 2.1.1.3 SlSBT3-Überexpressions-LInien Die SlSBT3 überexprimierenden Pflanzen wurden von Prof. Dr. A. Schaller zur Verfügung gestellt. Hier war der ORF der SlSBT3-cDNA in den binären Vektor pRD400 Modifizierung mit der dem CaMV-35S-Promotor Pflanzen wurde mit kloniert worden. Agrobakterien Die vermittelter Transformation erreicht. Für die durchgeführten Versuche wurde die homozygote Linie G2f1D verwendet. 2.1.2 Bakterien Stamm gentechnisch modifiziert Pathogenität Xcv 85-10 - virulent Xcv 85-10 pDSk200 avirulent Xcv 85-10 hrpA-G nicht pathogen Für die vorliegende Arbeit wurden drei isogene Linien vom Stamm Xanthomonas campestris pv. vesicatoria (Xcv) 85-10 verwendet. Alle wurden von dem Institut für Pflanzengenetik der Universität Halle (Prof. Dr. U. Bonas) für die vorliegende Arbeit zur Verfügung gestellt. 2.1.3 Pilze: Sclerotinia sclerotiorum Name Sclerotinia sclerotiorum (SSD) Isolat Dachbrand Herkunft aus Tabak (Nicotiana benthaminiana) Das Isolat wurde für die Versuche der vorliegenden Arbeit vom Institut für Phytomedizin (Dr. A. Walz) der Universität Hohenheim zur Verfügung gestellt. 2.1.4 Oomyceten: Phytophtora infestans Name Phytophtora infestans Isolat Freising 16 Material und Methoden Herkunft aus Kartoffel (Solanum tuberosum) Das Isolat wurde für die Versuche der vorliegenden Arbeit vom Institut für Botanik (AG Prof. Dr. Spring) der Universität Hohenheim zur Verfügung gestellt. 2.1.5 Plasmide pBluescript/SlSBT3 enthält die SlSBT3-cDNA pBluescipt/SlPR1a enthält die SlPR1a-cDNA pBluescript/SlPR3a enthält die SlPR3a-cDNA pBluescript/SlUBQ enthält die SlUBQ-cDNA Die Plasmide wurden von Prof. Dr. A. Schaller zur Verfügung gestellt. 2.1.6 Oligonukleotide Primer Sequenz Solanum lycopersicon Actinfor-RT TGTGGGAGATGAAGCTCAATCG Actinrev-RT TCAAACTATCAGTGAGGTCACG SlSBT3, for ACTCCTCAAGATTACGTAAATCTCC SlSBT3, rev CAATAATAGGAGATGTTACTATCGG SlACO, for ACGCAGGAGGCATCATACTTCTGT SlACO, rev AGGCTAATGACATTCGTGTCCCGT 17 Material und Methoden Sclerotinia sclerotiorum SSgpd* OL742 ATCCCATGGGCTGAGTCTGAG SSgpd* OL743 ATTGGCGGCCTTCTTGATG Phytophtora infestans PiEF2α, for TGACGCTATCGCCAAGGAATC PiEF2α, rev TAACGCTGAGCCGTAATGGGGG Oligonukleotide wurden von der Firma Operon (Köln) generiert. 2.1.7 Enzyme DNAse I DNAse I MBI-Fermentas, St.Leon-Rot Reverse Transkriptase RevertAID™ M-MuLV Reverse Transkriptase MBI-Fermentas, St.Leon-Rot Taq-Polymerase thermostabile DNA-Polymerase aus Thermus aquaticus PEQlab, Erlangen 2.1.8 Antikörper Anti-SlSBT3 Polyklonal, (aus Kaninchen), von Prof. Dr. A. Schaller zur Verfügung gestellt Anti-Kaninchen IgG 2.1.9 Peroxidase Konjugat, Calbiochem, Darmstadt Chemikalien Agarose Life Technologies, Eggenstein Bacto-Trypton Difco Laboratories, Detroit Biomalz Difco Laboratories, Detroit Bradford Reagenz (Protein Assay) BioRad, Hercules CA Brilliant Blau R Roth, Karlsruhe 18 Material und Methoden Cantharidin Sigma-Aldrich, München dNTP-Mix Bioline, London EDTA Fluka AG, St. Louis Fotoentwickler Kodak, Paris Fotofixierer Kodak, Paris Gemüsesaft Amecke, Menden GeneRuler™1kb DNA Ladder MBI-Fermentas, St.Leon-Rot Katalase Sigma-Aldrich, München Magnesiumchlorid Fluka AG, St. Louis Plant Agar Duchefa, Haarlem Ponceau S Sigma-Aldrich, München Potato Dextrose Agar (PDA) Roth, Karlsruhe Protease Inhibitor Mix P Serva, Heidelberg Salicylsäure Sigma-Aldrich, München Sarcosin ICN, Aurorar Weizenstärke Sigma-Aldrich, München X-Gluc Duchefa, Haarlem Yeast Extract Difco Laboratories, Detroit 2.1.10 Geräte Blottingapparatur LKB Bromma/Pharmacia Elektrophoreseapparatur EasyCast Elektrophoresis (für Agarosegele) Systems, Owl Scientific Inc. Elektrophoreseapparatur Bio-Rad (für Polyacrylamidgele) Photometer Biophotometer, Eppendorf Typhoon Trio + Variable Mode Imager, Amersham Biosciences UV-Crosslinker UV-Stratalinker 1800, Stratagene 19 Material und Methoden 2.2 Methoden 2.2.1 2.2.1.1 Anzucht von Tomatenpflanzen Behandlung der Tomatensamen zur Dekontamination von Viren Alle Samen wurden gegen einen möglichen Befall von Viren wie mit z. B. dem Tomaten Mosaikvirus (ToMV) oder dem Tabak Mosaik Virus (TMV) behandelt. Dazu wurden die Samen über Nacht bei 70 °C behandelt und danach 5 min in 70 % Ethanol gewaschen. Anschließend wurde das Ethanol in zwei kurzen Waschschritten durch ddH2O ersetzt und nach Entfernen des Wassers die Samen in ausreichend 10% Trinatriumphosphat (TNP) aufgenommen. Die in der Lösung frei schwimmenden Samen wurden für 3 h unter ständigem Schütteln so dekontaminiert. Vor der Aussaat wurde das TNP durch drei- bis fünfmaliges Waschen mit ddH2O entfernt und die Samen einzeln 5-10 mm tief von Erde bedeckt ausgebracht. 2.2.1.2 Anzuchtbedingungen Die Tomatenpflanzen wurden im Gewächshaus bei Langtagbedingungen (16 h Licht) und einer Mindesttemperatur von 26 °C angezogen. Die Aussaat erfolgte in Töpfen (Ø 4 cm). Nach vollständiger Durchwurzelung, spätestens nach 4 Wochen, wurden die Pflanzen in größere Töpfe (Ø 14 cm) umgetopft. Die Bedingungen bei Versuchen im Klimaschrank waren: 22 °C, 16 h Licht mit einer Lichtintensität von 25 µmol Photonen/m2 s). 2.2.2 Kultivierung von Xanthomonas campestris pv. vesicatoria Nutritient yeast growth 5 g/l Bactopepton (NYG) 3 g/l Hefeextrakt 20 % (w/v) Glycerin 20 Material und Methoden Selektionsmedien Stamm Antibiotikum Xcv 85-10 Rifampicin 100 µg/ml Xcv 85-10 pDSk200 Rifampicin 100 µg/ml Spectinomycin 50 µg/ml Xcv 85-10 ∆hrpA-G Rifampicin 100 µg/ml Xanthomonas campestris pv. vesicatoria (Xcv) wurde auf NYG-Medium kultiviert. Bei diesem Medium handelt es sich um ein Minimalmedium, das zur Induktion der Virulenz der Bakterien verwendet wird. Für die einzelnen Stämme wurde abhängig von der jeweiligen Resistenz das entsperchende Antibiotikum in der angegebenen Menge zugegeben. Für Dauerkultur auf Agarplatten wurden die Bakterien vereinzelt und für 48 h bei 30 °C inkubiert. Diese Dauerkulturen wurden max. 10 Tage bei 4 °C aufbewahrt. Zum Animpfen von Über Nachtkulturen wurden davon 3-4 Kolonien entnommen. Aus einer gut bewachsenen Über Nachtkultur angelegt wurde für alle Stämme Glycerinkulturen angelegt (Endkonzentration Glycerin: 20 %). Die Lagerung von Glycerinkulturen erfolgte und bei -80 °C. 2.2.3 Kultivierung von Sclerotinia sclerotiorum Biomalz-Peptonagar 2% (w/v) Malzin (BMP) 0,2 % (w/v) Pepton (Fleischextrakt) 1,5 % (w/v) Agar ½ Potato Dextrose Agar 1,95 % (w/v) PDA (PDA) 0,2 % (w/v) Hefeextrakt 1 % (w/v) Weizenstärke Zur Erhaltung der Virulenz wurde die Dauerkultur bei 20 °C auf BMP inkubiert. Zur Inokulation wurde das Isolat von BMP auf ½ PDA überführt und für 4 Tage im Dunkeln bei 20 °C inkubiert. Für eine Glycerinkultur wurden von einer 10 Tage bewachsenen Platte (½ PDA) die Dauerstadien (Sclerotien) in Glycerin aufgenommen und bei 21 Material und Methoden -80 °C eingefroren bzw. abgenommen und in einem Reaktionsgefäß bei Raumtemperatur trocken gelagert. 2.2.4 Kultivierung von Phytophtora infestans Gemüseagar Rye B Agar (zur Sporulation) 2 g/l CaCO3 25 % (v/v) Gemüsesaft 12 g/l Agar 15 g/l Bacto-Agar 20 g/l Saccharose in Roggenextrakt Zur Erhaltung der Virulenz wurde das Isolat zur Inokulation max. acht Generationen auf Gemüseagar bei 16 °C inkubiert und danach eine Generation auf Rye Agar B zur Sporulation oder auf Gemüseagar zusammen mit einer oberflächensterilisierten Kartoffelscheibe im Dunkeln bei 16 °C inkubiert. Für Rye Agar B wurde 60 g Roggen über Nacht mit Wasser überschichtet und bei Raumtemperatur gequollen. Der Überstand wurde entnommen und aufbewahrt. Der Roggen wurde ohne zu mischen in 2 l ddH2O 1 h aufgekocht und dabei nach Bedarf auf 2 l aufgefüllt. Der noch warme Roggen wurde über ein Tuch abfiltriert und die aufgenommene Flüssigkeit aus dem Sud gepresst. Beide Extrakte wurden vereinigt, mit Agar und Saccharose versetzt und autoklaviert. 2.2.5 Infiltration von Xcv in Tomatenblätter Zur Inokulation wurden aus einer Über Nachtkultur 100 µl ausplattiert und für 48 h bei 30 °C inkubiert. Die Bakterien wurden steril von Agarplatten entnommen und in 25 ml 1 mM Mg2Cl aufgenommen. Die optische Dichte der Suspension wurde photometrisch bei 600 nm bestimmt. Ausgehend von diesem Wert wurde dann die Bakeriendichte auf 108 cfu/ml (OD600: 0,4) eingestellt; für die Erstellung der Wachstumskurven wurden die Bakterien auf eine cfu/ml von 104 verdünnt. 22 Material und Methoden Zum Nachweis der Induktion der SlSBT3 wurden pro Probenzeitpunkt das vordere Fiederblatt des drittältesten Blattstadiums von drei fünf Wochen alten Pflanzen abgeschnitten und infiltriert. Hierfür wurden die Blätter eines Probenzeitpunkts in 200 ml der Bakteriensuspension mit 0,04 % Silwet L77 im Exikator infiltriert (3 x 10 min; 100 mbar). So war eine Inokulation der gesamten Blattoberfläche sichergestellt. Alternativ wurde die Lösung mit einer 2 ml Spritze mit bis zu 4 Infiltrationspunkten an der Blattunterseite infiltriert ohne die Blätter davor abzuschneiden. Zur Untersuchung der Symptome und des Bakterienwachstums im Blattgewebe wurde die Suspension an der Blattunterseite mit der Spritze injiziert. Die Suspension wurde in den vorderen Teil der zwei vorderen benachbarten Fiederblätter des drittältesten Blattstadiums an der Blattunterseite mit der Spritze bis in die Blattspitze infiltriert. Dabei wurden möglichst wenige Infiltrationspunkte gesetzt, d.h. maximal drei, damit eine homogene Verteilung der Bakterien im Blattgewebe gewährleistet war. 2.2.6 Reisolierung von Xcv aus Blattgewebe Pro Probenzeitpunkt wurden mit einem Korkbohrer (Durchmesser: 1 cm) aus den zwei infiltrierten vorderen Fiederblättern des drittältesten Blattstadiums von je drei fünf Wochen alten Pflanzen eine Blattscheibe aus der Blattspitze ausgestanzt. Nach kurzer Oberflächensterilisation der 6 Blattscheiben in 25 ml 70 % Ethanol und anschließendem kurzen Waschen in 1 mM MgCl2 wurden die Blattscheiben auf Zellstoff getrocknet und jeweils in 100 µl 1 mM MgCl2 in einem 1,5 ml Reaktionsgefäß mit einem sterilen Plastikpistill mazeriert. Der Pistill wurde mit 900 µl 1 mM MgCl2 abgespült. Ausgehend von dieser ersten Verdünnung (1:10) wurden die folgenden Verdünnungen abhängig vom Bakterienstamm bis zu einer Verdünnung von 1:108 hergestellt. Nach Erstellung der Verdünnungstufen wurden 10 µl auf eine Agarplatte getropft, die Agarplatte um 90° gedreht, damit die Suspension bis ca. 1 cm vor dem unteren Rand sich verteilt, und vollkommen trocknen gelassen. Um eine Kontamination mit Pilzen zu vermeiden wurden dem verwendeten Selektionsmedium (s. 2.2.2 zusätzlich Cefotaxim (50 µg/ml) als Antimykotikum zugesetzt. Die so beimpften Platten wurden anschließend mit 23 Material und Methoden Parafilm verschlossen und drei Tage bei 30 °C inkubiert. Anschließend wurden die Kolonien pro Verdünnung ausgezählt. 2.2.7 Statistische Auswertung des Wachstumsverhaltens von Xcv im Blattgewebe Für die statistische Auswertung wurde nur die cfu/ml einer Verdünnungsstufe berücksichtigt und hierbei die niedrigste Verdünnungsstufe gewählt, bei der eine Dokumentation der Kolonienzahl möglich war. Von den sechs ermittelten Werten wurde als Lagemaß der arithmetische Mittelwert mit Stichprobenvolumen (n=4) berechnet, dazu wurden der jeweils höchste und der niedrigste ermittelte Wert gestrichen. Die Standardabweichung wurde ausgehend von einem Stichprobenvolumen n-1 ermittelt. a) Berechnung der cfu/cm2: cfu/Blattscheibe=cfu*Verdünnung cfu/cm2=cfu*Verdünnung/(0,52)ת b) Berechnung des Mittelwerts: c) Berechnung der Standardabweichung einer Stichprobe: Dabei entspricht n dem Stichprobenumfang und x dem Stichprobenmittelwert. 24 Material und Methoden 2.2.8 Inokulation von Tomatenblättern mit S. sclerotiorum Zur Inokulation wurden aus vier Tage inkubierten Agarplatten Blöcke von einem Durchmesser von 0,5 cm mit einem Korkbohrer ausgestanzt. Dabei wurden ausschließlich Bereiche des jüngsten Myzels verwendet. Die Blöcke wurden mit der Seite des Myzels auf die Oberseite eines vorderen Fiederblatts des drittältesten Blattstadiums von fünf Wochen alten Pflanzen inokuliert. Die Blätter wurden hierfür abgeschnitten und drei Blätter pro Probenzeitpunkt in durchsichtige Plastikboxen gelegt. Für die Erhaltung dieser Blätter über den Versuchszeitraum wurden die Boxen mit feuchtem Zellstoff (20 ml ddH2O auf zwei Lagen Zellstoff) bis zum Rand ausgelegt, mit Frischhaltefolie am Boden bedeckt und bis zu vier Tagen im Klimaschrank belassen. 2.2.9 Inokulation von Tomatenblätter mit P. infestans Die Inokulation mit Phytophtora infestans erfolgte über das Stadium der Sporangien. Dafür wurde das Myzel von zehn Tage alten Gemüseagarplatten mit ca. 10 ml 4 °C kalten dH2O überflutet, mit Hilfe eines Glasspatels eine Suspension hergestellt und über eine einfache Lage Miracloth (Calbiochem, Porengröße: 22-25 µm) abfiltriert, um die Sporangien von den Myzelresten zu trennen. Die Sporendichte wurde mit Hilfe der Neubauer-Kammer am Mikroskop bestimmt und durch Zentrifugieren bei 100 xg und Resuspendieren der Sporen in einem geringeren Volumen dH2O auf 1000 Sporen pro ml eingestellt. Die Suspension wurde in 10 µl Tropfen an vier Stellen auf die Blattunterseite getropft. Die Blätter wurden hierfür abgeschnitten und pro Probenzeitpunkt wurde ein vorderes Fiederblatt des drittältesten Blattstadiums fünf Wochen alter Pflanzen in durchsichtige Plastikboxen gelegt. Für die Erhaltung dieser Blätter über den Versuchszeitraum wurden die Boxen mit feuchtem Zellstoff (20 ml ddH2O auf zwei Lagen Zellstoff) bis zum Rand ausgelegt, mit Frischhaltefolie am Boden bedeckt und bis zu vier Tagen mit der Blattunterseite nach oben im Klimaschrank belassen. 25 Material und Methoden 2.2.10 Behandlung von Tomatenpflanzen mit Salicylsäure Lösung 1,5 mM Salicylsäure 10 mM Kaliumphosphatpuffer Zur Behandlung von Tomatenpflanzen mit pH 6.8 Salicylsäure wurde eine entsprechende Menge in 10mM Kaliumphosphat lichtgeschützt über Nacht gelöst. Für eine unterschiedliche exogene Methoden Zugabe verwandt. der Zur Salicylsäure Applikation wurden zwei wurde eine Salicylsäurelösung mit 0,1 % Tween 20 verwendet. Diese wurde mit Hilfe einer Sprühflasche auf die Blattoberfläche 2 Wochen alter Tomatenpflanzen gegeben. Entsprechende Kontrollpflanzen wurden mit 10 mM Kaliumphosphatpuffer mit 0,1 % Tween 20 behandelt. Alternativ wurden Pflanzen gleichen Alters an der Bodengrenze abgeschnitten und in Reaktionsgefäße mit 100 µl Salicylsäurelösung überführt. Die Pflanzen konnten so über das Leitgewebe die Salicylsäurelösung aufnehmen. Erst wenn das gesamte Volumen aufgenommen war (nach max. 2 h), wurden die Pflanzen in Glaskolben mit H2O überführt. Die Kontrollpflanzen wurden jeweils in 100 µl Kaliumphosphatpuffer gestellt und genauso behandelt. Die Probenentnahme erfolgte 0 h, 2 h, 4 h, 6 h, 8 h, 12 h, 24 h und 48 h nach Behandlung statt. Für jede Probe wurden jeweils die zei entwickelten Fiederblätter von drei Pflanzen vereinigt und in flüssigem Stickstoff eingefroren. 2.2.11 Lösung Infiltration von Katalase 2000 U/ml Katalase 10 mM Kaliumphosphatpuffer pH 7.0 Für die Infiltration mit Katalase wurden die Blätter wie für die Inokulation mit pathogenen Pilzen abgeschnitten. Die Katalase wurde unmittelbar vor der Infiltration in Puffer gelöst und mit Hilfe einer Spritze an der Blattunterseite infiltriert. Der infiltrierte Bereich umfasste dabei einen Durchmesser von etwa 2 cm. 26 Material und Methoden 2.2.12 Infiltration von H2O2 Lösung 10 mM H2O2 in ddH2O Für die Infiltration mit Wasserstoffperoxid wurden die Blätter abgeschnitten und mit einer 2 ml Spritze an der Blattunterseite infiltriert. Die Blätter wurden wie für die Inokulationen mit pathogenen Pilzen in Plastikboxen im Klimaschrank belassen. Die Probenentnahme erfolgte 48 h nach Infiltration. 2.2.13 Infiltration von Cantharidin Lösung 5 µM Cantharidin Für die Infiltration wurde zunächst eine 50 mM Stammlösung angesetzt. Dazu wurde die die entsprechende Menge Cantharidin in 1 ml 70 % Ethanol gelöst wurde und dann auf das entsprechende Volumen mit ddH2O eingestellt. Die Blätter wurden abgeschnitten und mit einer 2 ml Spritze an der Blattunterseite infiltriert. Anschließend wurden sie wie für die Inokulationen mit pathogenen Pilzen in Plastikboxen im Klimaschrank belassen. Die Probenentnahme erfolgte bei Bildung von Läsionen nach 72 h. 2.2.14 GUS-Färbung GUS-Puffer 100 mM Natriumphosphatpuffer 10 mM EDTA 3 mM K4[Fe(CN6)] 0,5 M K3[Fe(CN6)] 0,1 % Triton X-100 1 mg/ml X-Gluc pH 7.0 pH 8.0 Die komplette Blattfläche wurde in bis zu 10 ml GUS-Färbelösung 3x 1 min bei 100 mbar vakuuminfiltriert und im Dunkeln über Nacht bei 37 °C inkubiert. Anschließende Entfärbung zur besseren Sichtbarkeit des gebildeten Präzipitats erfolgte mit 70 % bzw. 100 % Ethanol bis zur völligen Entfernung des Chlorophylls aus dem Blattgewebe. 27 Material und Methoden 2.2.15 DNA-Extraktion DEX-Puffer TE-Puffer 0,14 M Sorbitol 0,22 M Tris-HCl pH 8.0 0,22 M EDTA pH 8.0 0,8 M NaCl 0,8 % CTAB 1% Sarcosin 2% β-Mercaptoethanol 10 mM Tris-HCl 1 mM EDTA pH 8.0 Für die Extraktion genomischer DNA wurden ca. 10 mg Blattmaterial mit flüssigem Stickstoff und mit der Hilfe eines gekühlten Pistills zu einem feinen Pulver zermörsert. Anschließend wurden 100 µl DEX-Puffer zugesetzt und kurz resuspendiert. Nach Zugabe von 200 µl Chloroform wurde die Probe 30 min lang bei 65 °C inkubiert und anschließend zentrifugiert (10000 x g, 5 min, RT). Der Überstand wurde in ein neues Eppendorfgefäß überführt. Die in dem Überstand befindliche DNA wurde mit 100 µl Isopropanol für 15 min bei RT gefällt und danach abzentrifugiert (15 min, 4 °C, 13000 x g). Das Präzipität wurde dann mit 70 % Ethanol gewaschen und nach einem weiteren Zentrifugationsschritt (10 min, 4 °C, 13000 x g) in 30 µl TE gelöst. Für die PCR wurde 1 µl DNA als Matrize verwendet. Der Rest wurde bei -20 °C eingefroren. 2.2.16 Gelektrophorese von DNA 50х TAE (1l) Ladepuffer (10x) 242 g Tris-HCl 57,1 ml Essigsäure 100 ml 0,5 M EDTA 50 % Glycerin 1 mM EDTA 0,03 % Bromphenolblau oder Xylencyanol 28 Material und Methoden Die Auftrennung von Nukleinsäuren erfolgte auf TAE-Agarosegelen (1,5 % Agarose, 1 % Ethidiumbromid) in 1x TAE-Puffer bei 80 V bis 120 V. Die aufgetragenen Proben wurden mit 10x Ladepuffer versetzt (Endkonzentration Ladepuffer: 1x). Als Größenstandard wurden 5 µl GeneRuler™ 1 kb DNA ladder aufgetragen. Nach der Auftrennung wurde die DNA unter UV-Licht im Gel sichtbar gemacht und durch Fotos dokumentiert. 2.2.17 RNAse-freies Arbeiten Um ein RNAse-freies Arbeiten zu ermöglichen wurden alle angesetzten Lösungen mit 0,01 % DEPC-behandelt und zweimal 45 min autoklaviert. Glaswaren wurden über Nacht bei 250 °C sterilisiert, Reaktionsgefäße, Spitzen und weiteres Verbauchsmaterial aus Plastik wurden 45 min autoklaviert. Apparaturen, Schlitten und Kämme der Gelektrophoreseapparatur wurden mit herkömmlichen Spülmittel gewaschen, über Nacht in einer Spülmittellösung eingeweicht und danach mit dH2O ausgespült. 2.2.18 RNA-Extraktion Extraktionspuffer 75 mM NaCl 25 mM Tris/HCl 25 mM EDTA pH 8.0 1% SDS 1M β-Mercaptoethanol PClphenol 50:24:1 Phenol:Chloroform:Isoamylalkohol PCl 25:24:1 Phenol:Chloroform:Isoamylalkohol 5x Ladepuffer 2 µl 0,5 M EDTA 25 µl 1 % BPB 50 µl Glycerol 1 µl 10 mg/ml EtBr 22 µl ddH2O pH 8.0 29 Material und Methoden Für die RNA-Isolation wurden 300 mg Blattmaterial in flüssigem Stickstoff zu einem feinen Pulver zerrieben. Anschließend wurde das Pulver in ein Reaktionsgefäß überführt, mit 800 µl Extraktionspuffer versetzt und gemischt. Dann wurden 750 µl PClphenol zugegeben und der Ansatz gut resuspendiert. Für die Aufbereitung mehrerer Proben wurden die so präparierten Proben für max. 2 h auf Eis mit zwischenzeitlichem Resuspendieren gelagert. Das Gemisch wurde 10 min abzentrifugiert (4° C, 13000 x g). Die obere Phase wurde abgenommen, zweimal mit einem Volumen PCl extrahiert und zentrifugiert (4° C, 13000 x g). Anschließend erfolgte ein zusätzlicher Extraktionsschritt mit einfachem Volumen Chloroform. Das Extraktionsgemisch wurde erneut abzentrifugiert (4 °C, 13000 x g) und die wässrige Phase zur Fällung der RNA mit dem 0,25 - fachem Volumen 10 M LiCl versetzt und ÜN bei auf Eis inkubiert. Am nächsten Tag wurde das Präzipitat abzentrifugiert (4 °C, 13000 x g). Das RNA-Sediment wurde mit 70 % Ethanol gewaschen und in 400 µl ddH2O gelöst. Es folgte eine weitere Fällung mit 0,1 - fachem Volumen 3 M NaAc pH 4.8 und dem 2,5 - fachem Volumen an 99 % Ethanol. Danach wurde das Präzipität mit 70 % Ethanol gewaschen, für 5 min bei Raumtemperatur getrocknet und anschließend in 30 µl ddH2O gelöst. Die Konzentration und Reinheit der isolierten RNA wurden photometrisch durch Messung der Extinktion bei 260 nm und 280 nm bestimmt. Die RNA wurde bei -80 °C bzw. -20 °C aufbewahrt. 30 Material und Methoden 2.2.19 Semiquantitative RT-PCR 2.2.19.1 RT-Reaktion Ansatz für DNAse-Verdau 8 µg RNA (40µl) 4 µl 10x DNAse I-Puffer 8U DNAse I 2 µl 25 mM EDTA Ansatz für RT-Reaktion 10 µl RNA (DNAse I verdaut) (20 µl) 1 µl 100 µM Oligo (dT) 4 µl 5x RT-Reaktionspuffer 2 µl 10 mM dNTP 1 µl ddH2O (DEPC) 1 µl(=200 U) Reverse Transkriptase Die isolierte RNA wurde für die Synthese von komplementärer DNA (cDNA) mit DNAse I im Thermocycler für 30 min bei 37 °C verdaut. Durch Zugabe von 2 µl 25 mM EDTA und Erhitzen des Ansatzes auf 70 °C wurde die Reaktion abgestoppt und die DNAse I denaturiert. Zur Überprüfung der Integrität wurden 8 µl der DNAse I verdauten RNA auf ein TAE-Agarosegel aufgetragen. Die RNA wurde anschließend mit der Reversen Transkriptase (RT) in einer Reaktion im Thermocycler in cDNA umgeschrieben. Hierfür wurden in einem ersten Schritt die Oligo(dT)-Nukleotide 5 min bei 70 °C an den poly(A)-Kette der mRNA angelagert dann in einem zweiten Schritt nach Zugabe von 7 µl von einem Mastermix bestehend aus Wasser, Nukleotiden und 5x RTReaktionspuffer 5 min bei 37 °C inkubiert. Nach Zugabe von 1 µl Reverser Transkriptase wurde für 1 h bei 42 °C die cDNA-Synthese eingeleitet. Die Lagerung der cDNA erfolgte bei -20 °C. 31 Material und Methoden 2.2.19.2 PCR 5x PCR-Puffer 15 mM MgCl2 100 mM (NH4)2SO4 0,08 % Triton X-100 20 % DMSO 250 mM KCl 50 mM Tris-HCl pH 8.3 PCR-Programm Funktion Primer SSgpd PiEF2α EPI1 EPI10 95°C 10’ 95°C 5’ 95°C 5’ 95°C 5’ Denaturierung 95°C 30’’ 95°C 30’’ 95°C 30’’ 95°C 30’’ Annealing 56°C 30’’ 66°C 45’’ 60°C 30’’ 68°C 45’’ Elongation 72°C 1’ 72°C 45’’ 72°C 45’’ 72°C 45’’ 72°C 10’ 72°C 10’ 72°C 10’ 10’ 72°C PCR-Programm Funktion Primer Actin, PR1a ACO1 LeSBT3 95°C 5’ 95°C 5’ 95°C 5’ Denaturierung 95°C 30’’ 95°C 30’’ 95°C 30’’ Annealing 58°C 30'' 60°C 30’’ 58°C 30'' Elongation 72°C 45’’ 72°C 45’’ 72°C 45’’ 72°C 10’ 72°C 10’ 72°C 7’ Die RT-PCR wurde in einem Volumen von 50 µl mit 5 µl 5x PCR-Puffer, 1 µl dNTPs (10 mM), 10 U Taq-DNA-Polymerase und je 1 µl von 10 µM „Forward“ und „Reverse“ Primer durchgeführt. Das Endvolumen wurde duch Zugabe von entsprechenden Mengen ddH20 erreicht. Das Volumen der cDNA als 32 Material und Methoden Matrize im jeweiligen PCR-Ansatz variierte abhängig von der jeweiligen Konzentration der cDNA im Ausgangsmaterial. Ein Angleichen der cDNAKonzentration der einzelnen Proben eines Versuchsansatzes erfolgte über Angleichen des PCR-Produkts eines konstitutiv exprimierten Gens. 2.2.20 Northern Blot 2.2.20.1 Sondenherstellung für Northern Blot Mix 1 (25µl) Mix 2 (75µl) 5 µl 5×PCR-PuffeR 4 µl (5U) Taq Polymerase 16µl ddH20 15 µl 5x PCR-Puffer 2 µl 10 mM dNTPs 2 µl 10 µM Primer forward 2 µl 10 µM Primer reverse 2 µl Plasmid-DNA 52 µl ddH20 PCR-Programm Funktion SBT3-Sonde PR3a-Sonde UBQ-Sonde 95°C 2’ 95°C 2’ 95°C 2’ Denaturierung 95°C 20’’ 95°C 20’’ 95°C 20’’ Annealing 55 40’’ 54°C 40’’ 55 40’’ Elongation 72°C 30’’ 72°C 30’’ 72°C 30’’ Für diese PCR wurden zwei Mastermixe angesetzt. Der erste Mastermix (Mix 1) enthielt die Matrize-DNA und Oligonukleotide und der zweite (Mix 2) enthielt die Nukleotide und die Taq-Polymerase. Um eine optimale Zyklenzahl zu bestimmen, wurde zuerst ein Reaktionsansatz aus 75 µl Mix 1 und 25 µl Mix 2 angesetzt und die PCR gestartet. Nach 20, 25, 30 und 35 Zyklen 33 Material und Methoden wurden 10 µl Reaktionsansätze entnommen, auf Eis abgekühlt, mit 1 µl Ladepuffer versetzt und gelelektrophoretisch analysiert. Nachdem die optimale Zyklenzahl bestimmt war, wurden Mix 1 und Mix 2 vereinigt und in 100 µl Reaktionsansätze die Sonde über PCR amplifiziert. Zur Kontrolle wurde 1 µl eines Reaktionsansatzes über Gelelektrophorese analysiert. Die Reinigung der Sonde erfolgte dann durch Phenol/Chloroform-Extraktion mit anschließender Ethanol-Fällung. Hierfür wurde der Ansatz mit 600 µl Phenol/Chloroform (1:1) ausgeschüttelt, 2 min bei 4 °C und 10000 x g abzentrifugiert und der Überstand in 0,1- fachem Volumen Natriumacetat 3 M pH 5.2 und 2,5 - fachem Volumen 99 % EtOH versetzt. Danach erfolgte die Fällung der DNA für 20 min auf Eis. Nach 20 min Zentrifugieren bei 4 °C und 10000 ×g wurde das Sediment mit 70 % EtOH gewaschen, 10 min bei 4 °C und 10000 ×g abzentrifugiert. Abschließend wurde das Sediment getrocknet und in 30 µl ddH2O aufgenommen. Die Konzentration der Sonde wurde anhand eines standartisierten Markers bestimmt. Hierfür wurden 1 µl und 2 µl der Sonden–DNA auf ein Gel aufgetragen und mit der definierten Menge des Markers verglichen. 34 Material und Methoden 2.2.20.2 Gelelektrophorese von RNA Ladepuffer 10x Gelpuffer Agarose-Gel (1,2%) 50 % Glycerol 1 mM EDTA pH 8.0 0,03 % Bromphenolblau 2% Ethidiumbromid 0,4 M MOPS 100 mM Na-Acetat 10 mM EDTA pH 7.0 (eingestellt mit NaOH) 1,5 g Agarose 90 ml ddH2O 12,5 ml 10x Gelpuffer 22,5 ml Formaldehyd Für die RNA-Analyse mittels Northern Blot wurde ein denaturierendes Gel gegossen. Hierfür wurden 1,5 g Agarose in 90 ml Wasser geschmolzen, 12,5 ml 10x Gelpuffer, 22,5 ml Formaldehyd unter Schwenken zugefügt, zügig gegossen und für mindestens 0,5 Stunden erhärten gelassen. 5,5 µg der zu analysierenden RNA-Proben wurden mit 1 µl 10x Gelpuffer, 3,5 µl Formaldehyd, 10 µl Formamid versetzt und 15 min bei 65 °C denaturiert. Danach wurden die Proben kurz auf Eis abgekühlt und in der Tischzentrifuge (13000 ×g, 10 sec) abzentrifugiert. Die denaturierte RNA wurde bei 60 V etwa 5 h elektrophoretisch in 1x Gelpuffer aufgetrennt. Zur Dokumentation wurde das Gel unter UV-Beleuchtung fotografiert. 35 Material und Methoden 2.2.20.3 RNA-Transfer auf Membranen 20×SSC 3M NaCl 0,3 M Na-Citrat 5g Ficoll 400 5g Polyvinylpyrrolidon 5g BSA Fraktion V Transferpuffer 10x SSC Hybridisierungslösung 50 % Formamid 5x SSC 50 mM KPP pH 7,0 2x Denhardts 100 µg/ml sssDNA 0,5 % SDS 1x SSC 0,5 % SDS 0,2x SSC 0,5 % SDS 0,04x SSC 0,04% SDS 50x Denhardts (500ml) Waschlösung 1 Waschlösung 2 Waschlösung 3 Für den Blot wurde das denaturiende Gel 2x 15 min in ausreichend ddH2O und anschließend 15 min in 10x SSC-Puffer geschwenkt. Anschließend erfolgte der Aufbau des Blots: 36 Material und Methoden Abb. 2.1: Aufbau Northern Blot Das Gel wurde auf 2 Lagen entsprechend großen 3MM Whatman Filterpapier sgelegt, die auf einer Glasplatte liegend Kontakt mit dem 10x SSC-Puffer hatten. Dann erfolgte die Abdeckung der Ränder mit Parafilm. Die Nitrocellulosemembran wurde kurz in ddH2O, anschließend in 10x SSC angefeuchtet und luftblasenfrei auf das Gel positioniert. Auf die Membran wurden drei Lagen 3MM Whatman Filterpapier in Größe des Gels gelegt. Von einem Stapel Zellstoff und einer Glasplatte bedeckt, die mit ca. 200 g beschwert wurde, fand der Transfer über Nacht statt. Nach dem Transfer der RNA von dem Agarose-Gel auf die NitrocelluloseMembran wurde der Zellstoff entfernt, die Membran zur Orientierung markiert und kurz in 10x SSC geschwenkt. Anschließend wurde die Membran auf 3MM Whatman Filterpapier mit der geblotteten bzw. RNA-Seite nach oben soweit getrocknet, dass kein überschüssiges SSC als Flüssigkeitsfilm auf der Membran lag. Die transferierte RNA wurde dann durch UV-Crosslink auf der Membran fixiert (1200 µJx100). Die Membran wurde vollständig bei Raumtemperatur getrocknet und aufbewahrt. 2.2.20.4 Hybridisierug mit einer radioaktiv-markierten Sonde Für die Hybridisierung wurde die Membran zuerst in ddH2O angefeuchtet und dann in eine Schale mit 5x SSC überführt, wo sie mit Hilfe zweier steriler Pipetten aufgerollt und in eine Hybridisierungsröhre überführt wurde. Die 37 Material und Methoden Röhre wurde dann mit ca. 10 ml Hybridisierungslösung gefüllt. Anschließend wurde die Membran für mindestens 2 h bei 42°C prähybridisiert. Die radioaktive Markierung der Sonde wurde unter Verwendung des „RadPrime DNA Labeling System“-Kit der Firma Invitrogen durchgeführt. 30 g DNA wurden in 21 µl H2O denaturiert, anschließend auf Eis abgekühlt und kurz anzentrifugiert. Es wurden zugefügt: - 1 µl 500 µM dATP - 1 µl 500 µM dTTP - 1 µl 500 µM dGTP - 20 µl 2,5x Random Primer Lösung - 5 µl (50 µCi) [α-32P]-dCTP - 1 µl Klenow-Fragment Der Ansatz wurde gut gemischt, kurz anzentrifugiert und 10 min bei 37°C inkubiert. Die Reaktion wurde durch Zugabe von 5 µl Stop-Puffer beendet. Danach folgte die Reinigung der Sonde über eine Bio-Spin-Column, um nicht eingebaute Nukleotide zu entfernen. Hierfür wurde die Säule unten geöffnet, in ein 2 ml Eppendorfgefäß gesetzt und der Deckel abgenommen, um die Säulenflüssigkeit auslaufen zu lassen. Zur vollständigen Entfernung der Flüssigkeit wurde das Säulchen 2 min bei ca. 4000 x g zentrifugiert. Die radioaktive Probe wurde in die Mitte der Säule pipettiert und 4 min bei ca. 4000 x g zentrifugiert. Zur Überprüfung der Einbaurate wurde dann die Aktivität der Sonde im Durchlauf bestimmt. Die Probe wurde dann durch Erhitzen auf 100°C für 5 min denaturiert, auf Eis abgeschreckt und kurz anzentrifugiert. Aus der Hybridisierungsröhre wurde die Prähybridisierungslösung entfernt, durch 10 ml frische Hybridisierungslösung ersetzt und die denaturierte Sonde dazupipettiert. Die Hybridisierung erfolgte ÜN bei 42°C. Am nächsten Tag wurde die Membran in mehreren Schritten gewaschen, um unspezifisch gebundene Sonden zu entfernen. Zunächst wurde sie kurz mit Waschlösung 1 gespült und dann 5 min in dieser Lösung gewaschen, während die Temperatur im Hybridisierungsofen auf 60°C erhöht wurde. Es folgten ein 30-minütiger Waschschritt bei 60°C in Waschlösung 1 und danach ein ebenfalls 3038 Material und Methoden minütiger Waschschritt bei 60°C in Waschlösung 2. Die Exposition erfolgte gegen eine Storage Phosphor Screen Platte (Amersham Biosciences) und wurde über Typhoon Trio+ Imaging System (GE Healthcare) radiographisch dokumentiert. 2.2.20.5 Strippen von Membranen Für die Hybridiserung mit weiteren Sonden wurde die Membran gestrippt. Dazu wurden die Blots mit ca. 400 ml kochender Waschlösung 3 übergossen, 1 min in der Mikrowelle erhitzt und unter leichtem Schwenken abgekühlt. Dieser Vorgang wurde bis zu zwei Mal wiederholt. Anschließend wurden die Membranen bei RT getrocknet. 2.2.21 2.2.21.1 Proteinanalyse Herstellung von Gesamtproteinextrakten 5xExtraktionspuffer 500 mM NaCl 250 mM Tris/HCl 2,5 % Triton-X 100 50 mM β-Mercaptoethanol 0,1 % Protease Inhibitor Mix P pH 7.5 Das in flüssigem Stickstoff zerriebene Blattmaterial wurde in 200 µl Extraktionspuffer resuspendiert und so aufgetaut. Zelltrümmer wurden durch eine Zentrifugation für 2 min bei 4 °C und 13000 x g entfernt. Die Aufbewahrung der Proben erfolgte bei -20 °C. 2.2.21.2 Bestimmung der Proteinkonzentration nach Bradford Hierfür wurde ein 1 ml Ansatz bestehend aus 2 µl Proteinextrakt bzw. Wasser als Leerwert, 200 µl Bradford-Reagenz und 798 µl ddH2O vorbereitet. Anschließend wurde die Absorption im UV/Vis Spektrophotometer bei 595 nm gemessen und die Proteinkonzentration anhand einer BSA-Eichgerade errechnet. 39 Material und Methoden 2.2.21.3 SDS-PAGE Trenngelpuffer Sammelgelpuffer 4×Ladepuffer 1,5 M Tris/HCl pH 8.8 0,4 % SDS 0,5 M Tris/HCl 0,4 % SDS 200 mM Tris/HCl 400 mM Dithiothreitol (DTT) 8% SDS 0,4 % Bromphenolblau 40 % Glycerin pH 6.8 pH 6.8 Gelrezeptur (für 2 Gele, 1,5 mm Dicke): 9 %(w/v) Trenngel 4,5 %(w/v)Sammelgel Die 4,2 ml 40 % (w/v) Acrylamidstammlösung 9,6 ml ddH2O 4,6 ml 4x Trenngelpuffer 200 µl 10 % (w/v) APS 20 µl TEMED 1,1 ml 40% (w/v) Acrylamidstammlösung 6,3 ml ddH2O 2,5 ml 4x Sammelgelpuffer 100 µl 10 % (w/v) APS 10 µl TEMED Gesamtproteinextrakte wurden über ein 9% Polyacrylamidgel aufgetrennt. Hierzu wurden die Proteinproben in 1x SDS-Ladepuffer auf eine bestimmte Konzentration verdünnt, bei 100 °C 5 min denaturiert und kurz auf Eis abgekühlt. Als Größenstandard diente ein vorgefärbter Proteinmarker. Die Elektrophorese wurde bei 100 V für 1,5 h in 1x SDS-Laufpuffer durchgeführt. 40 Material und Methoden 2.2.21.4 Coomassie-Färbung Färbelösung Entfärbelösung 2,5 g Coomassie Brilliant Blue R250 450 ml ddH2O 450 ml Methanol 100 ml Essigsäure 600 ml ddH2O 300 ml Methanol 100 ml Essigsäure Nach der Elektrophorese wurde dafür ein SDS-Gel über Nacht in etwa 20 ml in Färbelösung bei Raumtemperatur und ständigem Schütteln inkubiert. Danach erfolgte mit jeweils ca. 20 ml Entfärbelösung unter Schütteln bei Raumtemperatur die Entfärbung des Gels. Das Gel wurde in ddH2O bei 4 °C aufbewahrt. Die Dokumentation erfolgte durch einscannen. 2.2.21.5 Immunologischer Nachweis von Proteinen mit Western Blot Kathodenlösung Anodenlösung 1 Anodenlösung 2 2×TBS-Puffer Blockierungslösung Waschlösung 40 mM 6-Aminohexanlösung 20 % (v/v) Methanol 0,3 M Tris/ HCl 20 % (v/v) Methanol 0,25 M Tris/HCl 20 % (v/v) Methanol 40 mM Tris/HCl 270 mM NaCl pH 10.4 pH 10.4 pH 7.5 0,1 % Tween20 6 % (w/v) Magermilchpulver 1× TBS 0,1% Tween20 41 Material und Methoden ECL-A ECL-B ECL-Lösung 1x TBS 5 ml 1 M Tris/HCl pH 8.5 45 ml ddH2O 110 µl 90 mM ρ-Cumarsäure 250 µl 250 mM Luminol 100 µl 30 % H2O2 900 µl ddH2O 10 ml ECL-A 30 µl ECL-B Das Polyacrylamid-Gel wurde nach dem Semi-Dry-Verfahren geblottet. Pro Gel wurden 15 Blatt Blotting-Papier als auch die Nitrocellulose-Membran auf Gelgröße zugeschnitten und vor dem Blotten ca. 10 min in der jeweiligen Lösung inkubiert. Die Nitrocellulose-Membran wurde vor dem Equilibrieren in Anodenpuffer einmal kurz in autoklaviertem ddH2O geschwenkt. Der Aufbau erfolgte nach Abtropfen der jeweiligen Lösung vom Papier sukzessiv. Abb. 2.2: Aufbau Western Blot. Nach dem Auflegen der letzten Lage von Papier wurden eventuelle Luftblasen durch vorsichtiges Ausstreichen entfernt. Der Transfer fand für 1 Polyacrylamidgel bei 100 mA bei 2 Gelen bei 150 mA für 1 h bis 1,5 h statt. Die Nitrocellulose-Membran wurde unmittelbar nach dem Elektrotransfer in ca. 20 ml Blockierungs-Lösung überführt und über Nacht bei Raumtemperatur 42 Material und Methoden oder 4 °C unter Schütteln inkubiert. Danach wurde die Blockierungslösung, durch den primären anti-SBT3-Antikörper (polyklonales Serum1:2000 in Blockierungslösung) ersetzt und unter Schütteln 2 h oder über Nacht inkubiert. Vor Zugabe des sekundären Antikörpers erfolgten 2-3 Waschschritte mit 50-100 ml Waschlösung a` 10 min bei Raumtemperatur. Als sekundärer Antikörper wurde die anti-Kanninchen IgG-Peroxidase Konjugat in einer Verdünnung von 1:10000 in Blockierungslösung verwendet und abgedunkelt bei Raumtemperatur 1 h unter Schütteln inkubiert. Der nicht gebundene Antikörper wurde durch dreimaliges Waschen in jeweils ca. 50100 ml Waschlösung entfernt und der Blot 2 min in 1x TBS inkubiert. Die Membran wurde ca. 2 min in 10 ml ECL-Lösung inkubiert, anschließend luftblasenfrei in eine durchsichtige Folie gepackt und gegen einen Röntgenfilm exponiert. 2.2.21.6 Ponceau-Färbung Ponceau-Lösung 2g Ponceau S (10x) 30 g Trichloressigsäure 30 g Sulfosalicylsäure mit ddH2O auf 100 ml aufgefüllt Die zu färbende Membran wurde 5 min in Ponceau-Lösung geschwenkt. Danach wurde die Membran mit H2O entfärbt. Abschließend wurde die Membran eingescannt und getrocknet. 43 Ergebnisse Ergebnisse 3.1 Überprüfung von SlSBT3-RNAi- und -ÜberexpressionsLinien in der T2-Generation Zu Beginn der vorliegenden Diplomarbeit standen homozygote Samen der T2- und T3-Generation von drei Kanamycin-resistenten RNAi-Linien zur Verfügung. Für die Durchführung der Versuche wurde die Linie SlSBT3 HP21-7 ausgewählt, da diese nur eine Kopie des Transgens enthält und eine vollständige Unterdrückung der Expression von SlSBT3 in der T2-Generation aufwies. Als Überexpressionslinie wurde die Linie G2f1D ausgewählt, da diese die stärkste Überexpression von SlSBT3 in der T2-Generation zeigte (Franziska Huttenlocher, unveröffentlicht). Vor der Untersuchung der Krankheitssymptome und des Wachstumsverhaltens von X. campestris pv. vesicatoria 85-10 bzw. S. sclerotiorum wurden die verwendeten Linien mittels Western Blot analysiert, um die Expression von SlSBT3 zu überprüfen. Das Ergebnis der Western Blot-Analyse ist in der Abbildung 3.1 gezeigt. Das SlSBT3-Protein konnte in keiner Pflanze der RNAi-Linie SlSBT3 HP-21-7-5 detektiert werden. Somit ist auf Proteinebene die Expression von SlSBT3 vollständig unterdrückt. Die Nachkommen der Überexpressions-Linie (OE) zeigten kein einheitliches Muster. Dennoch ist eine deutlich stärkere Expression der SlSBT3-Subtilase im Vergleich zum Wildtyp (Wt) zu sehen. Damit liegen mit der Linie 21-7 eine erfolgreich unterdrückte SlSBT3-RNAiLinie und mit G2f1D eine echte SlSBT3-Überexpressionslinie vor. 44 Ergebnisse Abb. 3.1: Überprüfung der SlSBT3-Überexpressions-Linie G2f1D (A) und SlSBT3-RNAi-Linie 21-7 (B). Um SlSBT3 (79 kDa) nachzuweisen wurden 20 µg Gesamtproteinextrakt wurde von 11 Pflanzen der Übereexpressions-Linie über SDS-Page aufgetrennt, auf Nitrocellulose übertragen und mit polyklonalem anti-SlSBT3-Antiserum nachgewiesen. Als Vergleich dienten Proteinextrakte aus Wildtyppflanzen. 3.2 Expression von SlSBT3 nach Behandlung mit Salicylsäure Da in ersten Versuchen eine Induktion von SlSBT3 nach Pathogenbefall beobachtet werden konnte (Huttenlocher, unveröffentlicht) sollte der Einfluss von Salicylsäure (SA), als endogenes Signal einer Pathogenabwehr, detaillierter untersucht werden (Durrant und Dong, 2004). Hierzu wurden wie unter Kapitel 2.2.8 beschrieben drei Wochen alte Tomatenpflanzen mit einer 1,5 mM Salicylsäure-Lösung behandelt. Die Kontrollpflanzen wurden mit der entsprechenden Pufferlösung behandelt. Nach bestimmten Zeitpunkten wurden die Blätter geerntet und die Expressionsanalyse von SlSBT3 mit semiqRT-PCR durchgeführt. 45 Ergebnisse Abb. 3.2.1: Expressionsanalyse von SlSBT3 von Tomatenblättern nach Besprühung mit Salicylsäure mit semiqRT-PCR. Blätter von 3 Wochen alte Tomatenpflanzen wurden mit 1,5 mM Salicylsäure in 10 mM Kaliumphosphatpuffer und 0,1 %Tween (A) oder mit 10 mM Kaliumphosphatpuffer und 0,1 %Tween (B) besprüht. Wie in Abbildung 3.2.1 zu sehen, kann das Besprühen von Tomatenpflanzen mit Salicylsäure zu einer Induktion des SlSBT3-Transkripts mit einem Maximum nach acht und zwölf Stunden führen. Als Positivkontrolle für die Versuchsdurchführung wurde die Expression des PR3a-Gens, der sauren Chitinase mitverfolgt. In den entsprechenden Kontrollpflanzen war auch eine Induktion der SlSBT3 nachzuweisen. Allerdings war in diesem Fall die Stärke der Induktion bei acht und zwölf Stunden im Vergleich zu denen der SAbehandelten Pflanzen deutlich schwächer. Auch die Expression des PR3aGens erwies sich in den Kontrollpflanzen ab vier Stunden als induziert. Die Induktion beider Gene könnte auf das Vorhandensein von Detergenz Tween 20 in der Sprühlösung oder auf die angewandte Applikationstechnik zurückzuführen sein. 46 Ergebnisse Abb. 3.2.2: Expressiosanalyse von SlSBT3 in Tomatenblättern nach Zufuhr von Salicylsäure durch den Transpirationsstrom mit semiqRT-PCR. Blätter von 3 Wochen alten Tomatenpflanzen wurden abgeschnitten und über den Transpirationsstrom mit 1,5 mM Salicylsäure in 10 mM Kaliumphosphatpuffer (A) oder 100 µl Kaliumphosphatpuffer (B) zugeführt. Um den Einfluss von Tween 20 oder der Applikationstechnik auszuschließen, wurde in einem zweiten Versuch die Salicylsäure über den Transpirationsstrom zugeführt. Aus dem geernteten Blattmaterial wurde RNA isoliert und die Expression über semiqRT-PCR analysiert. Die Abbildung 3.2.2 sind zeigt, dass die Expression von SlSBT3 nach acht und zwölf Stunden erhöht war. Die Expression des PR3a-Gens zeigte sich bereits nach zwei Stunden induziert. Die Kontrollpflanzen zeigten wie im vorhergehenden Versuch eine schwache Induktion von SlSBT3 mit einem Maximum nach zwölf Stunden. Eine gesteigerte Induktion des PR3a-Gens war auch mit dieser Methode in den Kontrollpflanzen zu beobachten. Die schwache Induktion des PR3a-Gens in den Kontrollen könnte durch Stress induziert werden, der bei dem Abschneiden der Pflanzen oder bei der Applikation von 47 Ergebnisse Salicylsäure ausgelöst wird. Mit diesem Versuch konnte das vorherige Ergebnis bestätigt werden. SlSBT3 ist durch Behandlung mit Salicylsäure induzierbar. 3.3 Expression von SlSBT3 in Tomatenblättern nach Infektion durch Xcv Nachdem die Induzierbarkeit von SlSBT3 durch Applikation von Salicylsäure nachgewiesen worden war, sollte die Abhängigkeit der SlSBT3-Expression nach Infektion durch biotrophe, prokaryontische Pathogene untersucht werden. Die eingesetzten Stämme von Xcv waren mit Ausnahem der Gene, die für die Resistenzreaktion in Wirtspflanzen verantwortlich sinfd, isogen. Es wurden ein virulenter, ein avirulenter (AvrBs3) und ein hrp-defizienter Stamm eingesetzt, der durch Mutation der hrp-Gene A bis G des Typ-IIISekretionsapparats nicht pathogen ist (Schornack, Inst. Pflanzengenetik Universität Halle, pers. Mitteilung) Für den Versuch wurden fünf Wochen alte Pflanzen eingesetzt. Pro Probenzeitpunkt wurden drei Blätter von drei Pflanzen in einem definierten Volumen Bakteriensuspension vakuuminfiltriert (s. Kap. 2.2.5). Für den virulenten Stamm und den hrp-defizienten Stamm wurde eine Bakteriendichte von 108 cfu/ml gewählt. Für den avirulenten Stamm wurde aufgrund der zu erwartenden starken Symptome und dem damit einhergehenden frühen Absterben der Gewebe eine geringere Dichte von 104 cfu/ml gewählt. Die Infiltration der Kontrollpflanzen erfolgte mit 1mM MgCl2. Die Probenentnahme fand in einem Rhythmus von 24 Stunden bis fünf Tage nach Inokulation statt. Aufgrund zu starker Symptombildung und beginnender Nekrotiersierung des Gewebes war eine RNA-Isolierung danach nicht mehr durchführbar. Alle Stämme von X. campestris pv. vesicatoria 85-10 induzieren die Expression von SlSBT3. Nach Infiltration mit dem virulenten Stamm (Abb.3.3.1 A) und dem hrp-defizienten (Stamm Abb. 3.3.1 C) war ein Induktionsmaximum nach dem ersten und zweiten Tag, während durch Infektion mit dem avirulenten Stamm (Abb. 3.3.1 B) eine über den gesamten beobachteten Zeitraum gleichmäßig starke Induktion zu beobachten. Wie unter 3.2.1 konnte auch hier eine relativ starke Induktion von SlSBT3 in den 48 Ergebnisse Kontrollpflanzen beobachtet werden, die durch die mechanische Beanspruchung oder Behandlung der Blätter mit Detergenz erklärt werden könnte. Dadurch kann an dieser Stelle keine eindeutige Aussage über die spezifische Induktion von SlSBT3 nach Infektion durch Xcv getroffen werden. 49 Ergebnisse Abb. 3.3.1: Expressionsanalyse von SlSBT3 nach Infektion durch Xcv 85-10 mit semiqRT. Blätter von 5 Wochen alten Pflanzen wurden jeweils in 200 ml Bakteriensuspension von virulentem X. campestris pv. 8 vesicatoria 85-10 mit 10 cfu/ml(A), avirulentem X. campestris pv. vesicatoria 85-10 pDSk 200 mit 10 8 cfu/ml (B), X. campestris pv. vesicatoria ∆hrpA-G (C) mit 10 cfu/ml und 1 mM MgCl2 4 (D) vakuuminfiltriert. Um eine Beeinflussung durch mechanische Belastung und unspezifische chemische Induktoren wie Detergenzien auszuschließen wurde eine Infiltration der Bakteriensuspension in Blätter direkt an der Pflanze durchgeführt. Hierfür wurden für alle Stämme mit Bakteriensuspensionen gleicher Dichte (108 cfu/ml) gewählt. Eine vollständige Inokulation der gesamten Blattfläche gelang aufgrund der unterschiedlichen Turgeszenz nicht. So konnte eine Infektion des Blatts auch nur partial stattfinden (siehe Kap. 2.2.5). 50 Ergebnisse Die Probenentnahme erfolgte in einem Rhythmus von 24 Stunden über eine Dauer von zehn Tagen. Dafür wurden drei Blätter gleichen Alters von drei unterschiedlichen Pflanzen in einer Probe vereinigt. Aus den geernteten Blättern wurde RNA isoliert und einer Northern Blot-Analyse mit einer SlSBT3-spezifischen Sonde unterzogen. Danach wurde mit der PR3a-Sonde und mit der Ubiquitin-Sonde hybridisiert. Zuletzt wurde nochmals zu Bestätigung der erfolgreichen Infektion mit der PR1a-Sonde hybridisiert. Die Hybridisierung mit der PR1a Sonde führte aber nur zu undeutlichen Signalen, die wahrscheinlich auf die Beeinträchtigung der Membranen durch das mehrmalige Strippen zurückzuführen sind. Aus diesem Grund sind die Ergebnisse dieser Hybridiesierung in der folgenden Abbildung 3.3.2 nicht gezeigt. Die untersuchten Pflanzen zeigten 48 Stunden nach Inokulation mit dem avirulenten Stamm erste Anzeichen einer hypersensitiven Reaktion. In Pflanzen, die mit dem virulenten Stamm inokuliert waren, konnten am fünften Tag nach Inokulation nekrotische Bereiche an den Infiltrationsstellen beobachtet werden. Pflanzen, die mit dem hrp-defizienten und die mit Kontrolllösung behandelt wurden, zeigten keine auffälligen Veränderungen der Blattfläche über den gesamten Versuchzeitraum. In den Kontrollpflanzen konnte keine erhöhte Expression von SlSBT3 und des PR-Proteins PR3a beobachtet werden (Abb. 3.3.2 D). Damit erwies sich die Durchführung des Versuchs erfolgreich. Die mit Xcv infizierten Blätter zeigten ein mit denen aus der semiqRT-Analyse vergleichbares Expressionsmuster. Die Induktion des PR3a-Gens in allen infizierten Proben wies auf eine erfolgreiche Infektion durch die Bakterien hin. Dabei lässt sich in den Blattproben, die mit dem virulenten Stamm infiziert waren ein späteres Induktionsmaximum nach fünf Tagen als durch den avirulenten Stamm nach bereits einem Tag beobachten. Die Signale des „housekeeping“-Genes Ubiquitin waren nach Infiltration des avirulenten und hrp-defizienten Stämmen gleichmäßig stark, was zeigt, dass vergleichbare Menge der pflanzlichen RNA aufgetragen wurden. Die geringe Abnahme der Signale des „housekeeping“-Genes bei den mit dem virulenten Stamm infizierten Blattproben ist am steigenden Anteil bakterieller RNA zu erklären, was im starken das Wachstum des virulenten Stamms liegt. 51 Ergebnisse Die Expression von SlSBT3 zeigte sich nach Infektion durch alle Stämme induziert. Sie war im Falle der Infektion durch den avirulenten Stamm (B) am stärksten, mit einem Maximum am dritten Tag nach Inokulation (Abb. 3.3.2 B). In Pflanzen, die mit dem virulenten Stamm (Abb.3.3.2 A) infiziert wurden, war eine starke Induktion von SlSBT3 fünf Tage nach Inokulation zu beobachten. Durch Infektion mit dem hrp-defizienten Stamm (Abb. 3.3.2 C) konnte eine Induktion der SlSBT3 acht Stunden nach Inokulation beobachtet werden. Die SlSBT3-Signale nach 24 Stunden bis zum fünften Tag nahmen rapide ab. 52 Ergebnisse Abb. 3.3.2: Expression von SlSBT3 in Tomatenblättern nach Infektion durch X. campestris pv. vesicatoria 858 10 mit Northern Blot. Blätter von 5 Wochen alten Pflanzen wurden an der Blattunterseite mit 10 cfu/ml Bakterien infiltriert. Virulenter X. campestris pv. vesicatoria 85-10 (A), avirulenter X. campestris pv. vesicatoria 85-10 pDSk 200 (B), X. campestris pv. vesicatoria ∆hrpA-G (C) und 1 mM MgCl2 (D). Je 5 µg RNA wurden elektrophoretisch aufgetrennt, auf eine Membran transferriert und mit entsprechenen radioaktiv-markierten Sonden hybridisiert. 53 Ergebnisse 3.4 Wachstumsverhalten von Xcv in SlSBT3 RNAi-, Überexpressions- und Wildtyppflanzen Über das Wachstumsverhalten der Bakterien sollte der Einfluss von SlSBT3 auf die Etablierung der Population der verschiedenen Stämme von X. campestris pv. vesicatoria spezifiziert werden. Hierzu wurden fünf Wochen alte SlSBT3-RNAi, SlSBT3-überexpermierende und Wildtyp-Tomatenpflanzen mit jeweils einer geringen Bakteriendichte (104 cfu/ml) infiltriert. Damit konnten die lokale Symptombildungen unterdrückt und die exponentielle Phase des Bakterienwachstums im Blattgewebe nachvollzogen werden. Die Größe der Bakterienpopulation wurde als Zahl der koloniebildenden Einheit proBlattfläche berechnet (vgl. Kap. 2.2.5). Die abgebildeten Wachstumskurven zeigen das Wachstum der drei isogenen Stämme von X. campestris pv. vesicatoria 85-10 über eine Zeitraum von acht Tagen nach Infiltration. Danach konnten aufgrund von beginnenden Symptombildungen in Pflanzen, die mit dem virulenten und avirulenten Stamm inokuliert wurden, keine aussagekräftigen Ergebnisse erzielt werden. Vereinzelt beginnende Nekrotisierung des Gewebes acht Tage nach Infiltration durch den avirulenten Stamm wurde in der Auswertung vernachlässigt. Insgesamt wurde ein tendenziell besseres Wachstum in der Überexpressionslinie und ein tendenziell schlechteres Wachstum in der RNAi-Linie von SlSBT3 beobachtet unabhängig von dem verwendeten Stamm von X. campestris pv. vesicatoria 85-10. 3.4.1 Wachstum des virulenten Stamms Xcv 85-10 Der vorliegende Wildtyp X. campestris pv. vesicatoria 85-10 ist auf Wirtspflanzen virulent (Thieme et al., 2005). Dieser Stamm führt zur Ausbildung von Krankheitssymptomen und löst somit eine kompatible Resistenzreaktion aus. Nach Infiltration des virulenten Stamms von Xcv kam es tendenziell zum gleichmäßigen Wachstum in allen drei Genotypen. Damit kann durch Überexpression der SlSBT3 keine erfolgreiche Abwehr gegenüber dem virulenten Stamm von Xcv erreicht werden. 54 Ergebnisse Die Tendenz des reduzierten Wachstums in der RNAi-Linie entspricht dem allgemeinen Trend der Wachstumskurven mit den anderen Stämmen. 1,E+05 relative log cfu/cm^2 1,E+04 1,E+03 OE Wt RNAi 1,E+02 1,E+01 1,E+00 1,E-01 0 2 4 6 8 dpi Abb. 3.4.1: Wachstum virulenter Stamm von X. campestris pv. vesicatoria in SlSBT3–RNAi, - 4 Überexpressions- und Wildtyppflanzen. Blätter von 5 Wochen alten Pflanzen wurden mit 10 cfu/ml inokuliert. Die Reisolierung erfolgte aus ausgestanzten Blattscheiben der infiltrierten Bereiche. Die 2 danach bestimmte cfu/cm wurde in der Darstellung am Tag 0 auf 1 normiert. 3.4.2 Wachstum des avirulenten Stamms Xcv 85-10 pDsK 200 Bei X. campestris pv. vesicatoria 85-10 pDsK 200 handelt es sich um den avirulenten Stamm. Durch Transformation eines AvrBs3 tragenden Plasmids, früher AvrBs4 (Schornack, Inst. für Pflanzengenetik, Universität Halle, pers. Mitteilung), löst dieser Stamm in Wirtspflanzen eine inkompatible Resistenzreaktion hervor (Ballvora et al., 2001). Bei dem avirulenten Stamm von X. campestris pv. vesicatoria ist ein gleichmäßiges Wachstum in allen drei Genotypen nachweisbar. Damit kann durch Überexpression von SlSBT3 keine erfolgreiche Abwehr gegenüber dem avirulenten Stamm von X. campestris pv. vesicatoria erreicht werden. Die Tendenz des reduzierten Wachstums in der RNAi-Linie entspricht dem allgemeinen Trend der Wachstumskurven mit den anderen Stämmen. Die Populationsgröße inkompatiblen entspricht Reaktion der sollte des der virulenten Stamms. Bei einer avirulente Stamm eine geringere Populationsgröße als der virulente Stamm erreichen, was mit einer 55 Ergebnisse fehlerhaften Expression oder Erkennung des Avirulenzgens in diesem Pathosytem erklärt werden kann. 1,E+05 relative log cfu/ml^2 1,E+04 1,E+03 OE Wt RNAi 1,E+02 1,E+01 1,E+00 1,E-01 0 2 4 6 8 dpi Abb. 3.4.2: Wachstum avirulenter Stamm X. campestris pv. vesicatoria 85-10 pDSk200 in SlSBT3–RNAi, 4 Überexpressions- und Wildtyppflanzen. Blätter von 5 Wochen alten Pflanzen wurden mit 10 cfu/ml inokuliert. Die Reisolierung erfolgte aus ausgestanzten Blattscheiben der infiltrierten Bereiche. Die 2 danach bestimmte cfu/cm wurde in der Darstellung am Tag 0 auf 1 normiert. 3.4.3 Wachstum des hrp-defizienten Stamms Xcv 85-10 hrpA-G Der hrp (hypersensitive Reaktion und Pathogenität)-defizienten Stamm X. campestris pv. vesicatoria 85-10 hrpA-G ist im Genom in den Genen hrp A, B, C, D, E, F und G deletiert und daher nicht in der Lage einen funktionsfähigen Typ-III-Sekretionsapparat auszubilden, der die EffektorProteine über die pflanzliche Zellmembran in das Cytoplasma der Pflanzenzelle sekretiert und dessen Proteine selbst als Effektoren wirken können (Rossier et al., 1999, Büttner und Bonas, 2002). Dieser hrp-defiziente Stamm ist in Wirtspflanzen nicht pathogen und kann keine Modifizierung des pflanzlichen Zellstoffwechsels zur Steigerung seines Wachstums durch TypIII sekretierte Effektoren vollziehen. 56 Ergebnisse Im Wachstum des hrp-defizienten Stamms von Xcv waren die größten Unterschiede der Populationsgrößen in den drei SlSBT3-Genotypen zu beobachten. Das Wachstum ist in der SlSBT3-RNAi-Linie gegenüber dem Wildtyp vermindert und in der Überexpressions-Linie erhöht. 1,E+04 relative log cfu/cm^2 1,E+03 1,E+02 OE Wt 1,E+01 RNAi 1,E+00 1,E-01 0 2 4 6 8 dpi Abb. 3.4.3: Wachstum hrp-defizienter Stamm X. campestris pv. vesicatoria 85-10 hrpA-G in SlSBT3–RNAi, 4 Überexpressions- und Wildtyppflanzen. Blätter von 5 Wochen alten Pflanzen wurden mit 10 cfu/ml inokuliert. Die Reisolierung erfolgte aus ausgestanzten Blattscheiben der infiltrierten Bereiche. Die 2 danach bestimmte cfu/cm wurde in der Darstellung am Tag 0 auf 1 normiert. 57 Ergebnisse 3.5 Vergleich der Krankheitssymptome in SlSBT3 RNAi-, Überexpressions- und Wildtyppflanzen nach Infektion durch Xcv Nachdem eine Induktion von SlSBT3 durch Infektion mit Xcv festgestellt wurde, sollte der Einfluss der SlSBT3-Protease auf die Entwicklung der Krankheitssymptome untersucht werden. Die geänderte Expression von SlSBT3 in den RNAi- und Überexpressionspflanzen sollte bei einer tragenden Rolle von SlSBT3 in der Pathogenabwehr einen qualitativen Unterschied in der Bildung der Krankheitssymptome im Vergleich zum Wildtyp deutlich machen. Um dies zu untersuchen, wurden fünf Wochen alte Pflanzen mit Bakteriensuspensionen von 108 cfu/ml des jeweiligen Stamms von X. campestris pv. vesicatoria infiltriert, um die lokale Symptombildung zu forcieren. Die Kontrolle wurde mit 1 mM MgCl2 behandelt. Die hier dokumentierten Symptome wurden in mindestens zwei unabhängigen Versuchen über einen Zeitraum von 14 Tagen nach Inokulation beobachtet und stellen eine Dokumentation der zeitlichen bzw. qualitativen Änderungen zum Wildtyp dar. Abb. 3.5.1: Krankheitsymptome durch X. campestris pv. vesicatoria 85-10 in SlSBT3–RNAi, Überexpressionsund Wildtyppflanzen. Blätter 5 Wochen alter Wildtyp (WT)-, RNAi- und Überexpressionspflanzen (OE). 8 Dargestellt ist die Blattoberseite 10 Tage nach Inokulation mit 10 cfu/ml. 58 Ergebnisse Bis zum zehnten Tag nach Inokulation mit dem avirulenten und virulenten Stamm von Xcv waren keine Unterschiede in den Krankheitssymptome in der RNAi- und der Überexpressions-Linie im Vergleich zum Wildtyp zu beobachten (Abb. 3.5.1). In den Kontrollpflanzen waren keine Symptome zu erkennen. In allen drei Genotypen ist die Infiltrationsstelle anhand der Kontaktfläche mit der Spritze zu erkennen. Am 10.Tag konnte in den Blättern die dem hrp-defizienten Stamm ausgesetzt waren bei zwölf von 15 Pflanzen der RNAi-Linien Nekrosen beobachtet werden, die dem des virulenten Wildtyps in Konsistenz und Farbe ähnlich waren. In einem zweiten Versuch konnte diese Beobachtung wiederholt werden. Hier wurde der Versuchszeitraum auf 14 Tagen ausgeweitet. Danach zeigten alle 15 SlSBT3-RNAi-Pflanzen die erwähnten Nekrosen. Daneben konnte die Beobachtung der Nekrotisierung auch in 6 von 15 Pflanzen des Wildtyps beobachtet werden, während keine Pflanze der überexprimierenden Linie Symptome aufwies (Abb. 3.5.2). Die Symptome ausgelöst durch die anderen pathogenen Stämme änderten sich nicht. Abb. 3.5.2: Krankheitsymptome hervorgerufen durch den hrp-defizienenten Stamm X. campestris pv. vesicatoria hrp A-G in SlSBT3–RNAi, Überexpressions- und Wildtyppflanzen. Blätter 5 Wochen alter Wildtyp (WT)-, RNAi- und Überexpressionspflanzen (OE). Dargestellt ist die Blattoberseite (links) 8 und die Blattunterseite (rechts) 14 Tage nach Inokulation mit 10 cfu/ml. 59 Ergebnisse 3.6 Expression von SlSBT3 in Tomatenblättern nach Infektion durch S. sclerotiorum In Kapitel 3.2 konnte eine gestiegene Expression von SlSBT3 nach Befall durch biotrophe Bakterien gezeigt werden. Um das Spektrum der phytopathogen Organismen zu erweitern, wurden weitere Organismen mit einem anderen Infektionsmechanismus und Lebensweise gewählt. Hierfür wurde S. sclerotiorum, ein Ascomycet mit breitem Wirtspflanzenkreis, mit der besonderen Eigenschaft unter Kulturbedingungen im Labor nicht zu sporulieren, als eukaryontisches Phytopathogen mit nekrotropher Lebensweise gewählt (Bolton et al., 2006, Walz, Inst. für Phytomedizin, pers. Mitteilung). Die Inokulation der Blattoberfläche erfolgte wie unter 2.2.8 beschrieben mit Myzel bewachsenen Agarstücken auf die Blattoberfläche. Die Probenentnahme erfolgte nach einer ersten erfolgreichen Besiedlung des Blattgewebes durch den Pilz nach 17 Stunden und darauf folgend nach 24, 48 und 72 Stunden nach Inokulation. Eine weitere Probenentnahme wurde nicht durchgeführt, da 72 h nach Inokulation ca. 2/3 der Blattflächen nekrotisch waren und daher für die RNA-Analyse ungeeigenet waren. Pro Zeitpunkt wurden drei gleich alte Blätter vereinigt und für die Expressionsanalyse auf RNA-Ebene mit semiquantitativer RT-PCR und auf Proteinebene mit Western Blot verwendet. Die Expression von SlSBT3 zeigte sich mit zunehmendem Wachstum S. sclerotiorum erhöht. Das Wachstum des Pilzes wurde mit pilzspezifischen Primern für die Glyceraldehyd-Phosphat-Dehydrogenase (GPD bzw. GAPDH) als „house-keeping“-Gene von S. sclerotiorum in der semiqRT-PCR quantifiziert (Abb. 3.6.1 A). Im Western Blot konnte eine Zunahme der Signale für SlSBT3 ebenfalls beobachtet werden (Abb. 3.6.1 B). Die Signale oberhalb des Signals für SlSBT3 sind in der Positivkontrolle, dem gereinigten SlSBT3-Protein, nicht nachweisbar. Hierbei könnte es sich um unspezifische Signale handeln, die durch Kreuzreaktion des SlSBT3-Antiserums mit pilzlichen Proteinen entstehen. Die Zunahme der Signalstärke wäre also durch das Wachstum des Pilzes zu erklären. Auf Proteinebene ist auch in den Kontrollen eine schwache Zunahme der Signale von SlSBT3 nachweisen, die in der semiqRT-PCR-Analyse nicht nachweisbar war. Dies könnte durch einen posttranslationellen Mechanismus, etwa eine erhöhte Stabilität des 60 Ergebnisse Proteins zu erklären sein, oder durch die Akkumulation von anderen Subtilasen, die mit dem polyklonalen SlSBT3-Antikörper nachgewiesen werden können. Abb. 3.6.1: Expressionsanalyse von SlBT3 nach Inokulation mit S. sclerotiorum. Blätter von 5 Wochen alten Tomatenpflanzen wurden abgeschnitten und mit S. Sclerotiorum inokuliert. Als Kontrolle dienten Blätter, die abgeschnitten wurden und nicht inokuliert waren. A) SemiqRT-PCR. Das Wachstum von S. sclerotiorum wurde über Primer für die Glyceraldehyd Phosphat Dehydrogenase (GPD) nachvollzogen. B) Western Blot (oben) und Coomassie gefärbtes SDS-Gel (unten). 25 µg Gesamtproteinextrakt wurden über SDS-PAGE aufgetrennt und im Western Blot mit gereinigtem anti-SlSBT3-Antikörper nachgewiesen. Als Positivkontrolle dienten 10 ng gereinigte SlSBT3. Zudem wurde die Induktion des SlSBT3-Promotors mit Hilfe des GUSReportergens analysiert. Um die Lokalisation der SlSBT3-Expression in dem befallenen Blatt in SlSBT3-Promotor::GUS-Pflanzen zu bestimmen, wurden drei Wochen alte Pflanzen mit einem myzelbewachsenen Agarblock inokuliert und in Plastikboxen im Klimaschrank inkubiert. Die Probenentnahme zur Infiltration der GUS-Lösung erfolgte nach 24, 48 und 72 Stunden nach Inokulation. Die Versuche wurden mit den Linien 5-3, 12-1 und 14-2 61 Ergebnisse durchgeführt. Alle Linien zeigten qualitativ ähnliche Ergebnisse. Die Linie 142 erwies sich für diese Untersuchungen am besten, da sie die geringste Färbung im nicht-induzierten Blatt zeigte. Nur diese Linie wurde für die vorliegenden Abbildungen v (Abb. 3.6.2). 24 Stunden nach Inokulation war der Pilz im Gewebe voll etabliert und es waren wässrige Läsionen mit einem Durchmesser von ca. 0,5 cm bis 0,8 cm zu erkennen (s. Abb. 3.6.2 A und B). Durch anschließende Färbung dieser Blätter mit X-Gluc-Lösung wurde eine Blaufärbung und damit die Aktivität der SlSBT3-Promotors 24 Stunden nach Inokulation vor allem in den Grenzbereichen der Nekrose gezeigt. Vereinzelt waren auch Färbungen an den Blatträndern zu beobachten. Nach etwa 40 Stunden hatte die Nekrose das gesamte inokulierte terminale Fiederblatt erfasst und breitete sich über die Radis in die distalen und proximalen Fiederblättchen aus (Abb.3.6.2 C). 62 Ergebnisse Abb. 3.6.2: Histochemische Lokalisation derSlSBT3-Promotor::GUS Aktivität nach Inokulation mit S. sclerotiorum. (GUS-SlSBT3 14-2). Ein terminales Fiederblatt von 3 Wochen alten Tomatenpflanzen wurde mit einem mycelbewachsenen Agarblock inokuliert. Zu den angegebenen Zeitpunkten wurden die Blätter mit X-Gluc gefärbt. A) Totalaufnahme von Blättern vor der GUS-Färbung. B) Totalaufnahme der XGluc gefärbten und mit 70 % Ethanol entfärbten Blätter. C) Ausschnitte mit einer 6x Vergrößerung der gleichen Blätter Der Pilz wächst vom inokulierten terminalen Fiederblatt (24 hpi) über die Radis zu den benachbarten distalen (48 hpi) und proximalen (72 hpi) Fiederblättern. 63 Ergebnisse 3.7 Vergleich der Krankheitssymptome in SlSBT3 RNAi-, Überexpressions- und Wildtyppflanzen nach Infektion durch S. sclerotiorum Abb. 3.7: Krankheitssymptome durch S. sclerotiorum in SlSBT3–RNAi, -Überexpressions- und Wildtyppflanzen. Blätter von 5 Wochen alten Tomatenpflanzen 36 h nach Inokulation. Wie unter 3.4 sollte die Bildung von Nekrosen in Tomatenpflanzen des Wildtyps, in entsprechenden SlSBT3 unterdrückten- und überexpremierenden Tomatenpflanzen auf zeitliche oder qualitative Änderungen untersucht werden. Hierzu wurden, wie unter 2.2.8 beschrieben, die Blätter mit einem von S. sclerotiorum myzelbewachsenen Agarblock inokuliert. 17 Stunden nach Inokulation konnte ein gleichmäßiges Anwachsen von S. sclerotiorum an der Blattunterseite beobachtet werden. Danach breitete sich die Nekrose in acht von zehn untersuchten Blättern der gesilenceten Linie schneller aus als im Wildtyp und der überexpremierenden Linie. Die Unterschiede in der Ausbreitung der gebildeten Nekrose wurden nach 36 Stunden nach Inokulation am besten deutlich (Abb. 3.6). 64 Ergebnisse 3.8 Expression von SlSBT3 in Tomatenblättern nach Infektion durch P. infestans Unter 3.1 und 3.4 konnte gezeigt werden, dass nach Infektion durch biotrophe prokaryontische und nekrotrophe eukaryontische Pathogen eine Induktion von SlSBT3 stattfindet. Um die Abhängigkeit dieser Induktion von den jeweiligen Pathogenen und deren Eigenheiten bei dem Infektionsmechanismus auszuschließen, wurde ein weiteres Pathogen gewählt. Bei dem Oomyceten Phytophtora infestans handelt es sich um einen hemibiotrophen, eukaryontischen Organismus, der nach einer ersten biotrophen Besiedlungsphase nach etwa 5-6 Tagen seine Lebensweise ändert und fortan nekrotroph leben kann. Daneben ist bekannt, dass die von P. infestans gebildeten Kazal-Inhibitoren die gereinigte SlSBT3 in-vitro inhibieren können, was eine nähere Untersuchung die Pathogen- Wirtspflanze-Beziehung anhand eines veränderten Expressionsstatus von SlSBT3 rechtfertigt (Tian et al., 2005, Knappenberger, unveröffentlicht) Die Inokulation der Tomatenblätter erfolgte wie in Kapitel 2.2.9 beschrieben über das Stadium der Sporangien. Bedingt durch die Tatsache, dass das verwendete Isolat in der Erkennung durch Tomaten als Wirtspflanzen nicht genauer charakterisiert ist, kann hier keine eindeutige Aussage getroffen werden, ob es sich um ein avirulentes oder virulentes Kultivar handelt. Unter Berücksichtigung der beobachteten großflächigen Nekrosen um die Inokulationsstellen, die nach fünf Tagen sichtbar wurden und bis zum Ende des Versuchszeitraums zunahmen, wurde davon ausgegangen, dass es sich um ein virulentes Kultivar von P. infestans handelt. Für die Probenentnahme wurden drei Blätter von drei Pflanzen vereinigt. Die Entnahme erfolgte in einem Rhythmus von 24 Stunden bis zum vierten Tag und nochmals acht Tage nach Inokulation. Somit konnte sichergestellt werden, dass die Entnahme während einer biotrophen Phase und einer nekrotrophen Phase erfolgte, was auch in einer Nekrose am achten Tag zu äußerte (nicht abgebildet). Die anschließende Analyse der Expression von SlSBT3 erfolgte mit semiqRTPCR und mit Western Blot. Das Wachstum von P. infestans im Blattgewebe wurde mit spezifischen Primern für das Gen des Elongationsfaktors von P. infestans nachvollzogen. Neben der Expression vonSlSBT3 wurde auch die 65 Ergebnisse der ACC-Oxidase, eines Enzyms der Ethylensynthese, untersucht. Sowohl mittels RT-PCR als auch mit Western Blot konnte eine gestiegene SlSBT3Expression nach Inokulation mit P. infestans festgestellt werden. Auf der Transkript-Ebene wurde eine erster Anstieg der SlSBT3 Expression ein Tag nach Inokulation beobachtet werden. Die maximale Induktion konnte vier Tage nach Inokulation nachgewiesen werden (Abb. 3.8.1 A). Das Wachstum von P. infestans konnte nicht über eine Zunahme der Expression des Elongationsfaktors nachgewiesen werden. So muss davon ausgegangen werden, dass die Infektion mit P. infestans ungleichmäßig erfolgt war. Zur Kontrolle der Infektion wurde der Anstieg der AminocyclopropancarboxylatOxidase (ACO)-Expression überprüft (Smart et al., 2003). Hier zeigte sich, dass eine erfolgreiche Infektion stattgefunden hatte. Mit der Western Blot-Analyse konnte eine Induktion nach drei Tagen und eine starke Expression acht Tage nach Inokulation gezeigt werden. Wie unter Kapitel 3.6 wurde eine Zunahme der SlSBT3-Expression auch in den Kontrollen auf Proteinebene detektiert, obgleich sie mit der semiqRT-PCR nicht nachweisbar war (Abb. 3.8.1 B). Wahrscheinlich erkennt der polyklonale Antiköper andere Subtilasen, die durch die Behandlung der Blätter induziert werden. Zusammenfassend konnte mit diesem Versuch eine erhöhte Expression von SlSBT3, insbesondere in einer späten Phase der Besiedlung der Pflanzengewebes durch P. infestans gezeigt werden. 66 Ergebnisse Abb. 3.8.: Expressionsanalyse von SlBT3 nach Inokulation mit P. infestans. Blätter von 5 Wochen alten Tomatenpflanzen wurden abgeschnitten und mit 10 µl einer Sporensuspension von P.infestans (1000 Sporen/ml) inokuliert. Als Kontrolle wurden uninokulierte Blätter mitgeführt. A) SemiqRT-PCR. Das Wachstum von P.infestans wurde über Primer für den Elongationsfaktor (PiEF) nachgewiesen. Der Nachweis der Infektion des Blattgwebes erfolgte über mit Primern der ACC-Oxidase (ACO). B) Western Blot (oben) und Coomassie gefärbtes SDS-Gel (unten). 25 µg Gesamtproteinextrakt wurden über SDSPAGE aufgetrennt und im Western Blot mit anti-SlSBT3-Antikörper detektiert. Als Positivkontrolle dienten 10 ng gereinigte SlSBT3. 3.9 Expression von SlSBT3 nach Behandlung mit Katalase und Cantharidin Aufgrund der Beobachtung, dass sich eine starke Expression von SlSBT3 bei Befall durch den avirulenten Stamm von X. campestris, den nekrotrophen Pilz S. sclerotiorum und in der nekrotrophen Phase der P. infestans-Infektion 67 Ergebnisse nachwiesen ließ, wurde die Abhängigkeit der Expression von SlSBT3 von der Einleitung des programmierten Zelltods vermutet, die im folgenden genauer untersucht werden sollte. Hierzu wurde der Einfluss von Wasserstoffperoxid nach Infektion durch S. sclerotiorum und einer exogenen Zugabe des Zellgifts Cantharidin mit GUS-Reportergenpflanzen untersucht. 3.9.1 GUS-Repotergenanalyse nach Infiltration von Katalase Bei Pathogenbefall kommt es zur Bildung reaktiver Sauerstoffspezies (ROS). In diesem Versuch sollte eine eventuelle Beeinflussung der Expression durch das relative stabile ROS H2O2 untersucht werden. Dazu wurden wie unter 3.5 beschrieben drei Wochen alte Pflanzen der GUSLinie 12-1 verwendet. In beide Blatthälften wurde zunächst großflächig Katalase infiltriert. Katalase ist ein pflanzliches Enzym, das die Umwandlung von H2O2 in Sauerstoff und Wasser katalysiert. Durch Infiltration wird dadurch gesamte H2O2 entfernt, das während der Interaktion mit S. sclerotiorum entsteht. Danach wurde eine Blatthälfte S. sclerotiorum inokuliert. Kontrollpflanzen wurden nur mit Pufferlösung infiltriert und anschließende mit S. sclerotiorum inokuliert bzw. nur mit S. sclerotiorum inokuliert. Nach 17 Stunden war ein weitgehend gleichmäßiges Anwachsen des Mycels zu beobachten. Nur in einem von drei Blättern war in dem Katalaseinfiltrierten Bereich war zu diesem Zeitpunkt eine minimal verstärkte Ausbreitung der Nekrose auszumachen (nicht abgebildet). Die Nekrosen vor der X-Gluc Färbung 24 Stunden nach Inokulation waren in ihrer Größe nicht unterschiedlich. Die Reportergen-Analyse in den Abbildungen 3.9.1 zeigte, dass die Aktivität des SlSBT3-Promotors in den mit S. sclerotiorum inokulierten und Katalase-infiltrierten Blättern Blättern stark erhöht wurde. In Gegenwart von Katalase erreichte die Blaufärbung schon nach 24 Stunden eine Intensität, die durch die Pilzinfektion alleine erst nach 48 Stunden zu beobachten war. Wie unter 3.6 war hier eine starke Induktion des SlSBT3Promotors im Grenzbereich der Nekrose und im Gewebe des Infiltrationsbereichs zu sehen. Die Kontrollinfiltration mit Katalase auf der anderen Blattseite zeigte keine großflächige Blaufärbung. Die drei Kontrollblätter die mit S. sclerotiorum, aber nicht mit Katalase infiltriert waren, zeigten im Gewebe um die Nekrose wie unter 3.6 eine vergleichbar 68 Ergebnisse schwächere Blaufärbung. 48 Stunden nach Inokulation war in diesen Blättern eine vergleichbar Kontrollpflanzen starke die mit Färbung zu Pufferlösung erkennen infiltriert (nicht waren abgebildet). zeigten eine vergleichbare Blaufärbung zu der Kontrolle, die nur mit S. sclerotiorum inokuliert waren (nicht abgebildet). Eine Untersuchung in den Katalaseinfiltrierten Blättern nach 48 Stunden erschien als nicht sinnvoll, da nach diesem Zeitraum das Wachstum des Pilzes den Infiltrationsbereich überschritten hatte. Blätter, die nicht infiltriert und nicht inokuliert waren, zeigten keine ausgeprägte Färbung des Blattgewebes. Damit kann angenommen werden, dass es durch H2O2 zu Reprimierung der Expression von SlSBT3 kommt. Abb. 3.9.1: Histochemische Analyse der SlSBT3-Promotor::GUS Aktivität nach Infiltration mit Katalase und gleichzeitiger Inokulation mit S. sclerotiorum. Dargestellt ist die Färbung von Tomatenblätter (GUSLinie 12-1) mit X-Gluc zum Zeitpunkt 0, Infiltration von 2000 U Katalase und gleichzeitiger Infektion mit S. sclerotiorum nach 24 h, Infektion durch S. sclerotiorum nach 24 h. Die Pfeile deuten die infiltrierten Bereiche an. 3.9.2 GUS-Reportergenanalyse nach Infiltration von Cantharidin Der Einfluss des Zellgifts Cantharidin wurde ebenfalls mit GUS-Reportergen analysiert. Die Blätter wurden hierfür an der Blattunterseite mit Cantharidin infiltriert und nach 72 Stunden mit X-Gluc Lösung gefärbt. Die Kontrollpflanzen wurden nicht infiltriert. Nach 72 Stunden war in dem infiltrierten Bereich eine großflächige Nekrose sichtbar (Abb. 3.9.2). Um diese 69 Ergebnisse Nekrose konnte ein Bereich chlorotischen Gewebes anhand der Gelbfärbung beobachte werden. Nach Färbung der Pflanzen zeigte sich in den Cantharidin-behandelten Blättern eine starke unregelmäßige Blaufärbung im gesamten Blattgewebe. Die Kontrollpflanzen zeigten nur eine erhöhte Induktion in den Blattadern, aber keine Färbung im Gewebe zwischen den Blattadern. Somit ist mit diesem Versuch eine Aktivität des SlSBT3-Promotors unabhängig von Pathogenen nach einem lokal begrenzten Zelltod nachgewiesen worden. Abb. 3.9.2: Histochemische Analyse der SlSBT3-Promotor::GUS Aktivität nach Infiltration von Cantharidin. (GUS-SlSBT3 12-1, rechts oben). Infiltration von 5 µg Cantharidin führt nach 72 h zu lokal begrenzten Nekrosen (links oben). Färbung mit X-Gluc und Entfärbung mit 70 %Ethanol (links unten). Kontrollen waren unbehandelt (rechts unten). 70 Diskussion Diskussion Über ein Drittel der pflanzlichen Proteasen sind Serinproteasen. Mit mehr als 50 codierten Genen in Arabidopsis stellt die S8-Familie der Subtilisinähnlichen Proteasen die größte Familie der Proteasen in Pflanzen dar (Van der Loon und Jones, 2004). In Tomate sind bisher 15 Mitglieder identifiziert worden. Sie werden in fünf Subfamilien: tmp, P69, SlSBT1, SlSBT2 und SlSBT3/4 unterteilt (Meichtry et al., 1999). Mit der Charakterisierung von Vertretern der P69-Subfamilie ist einmalig die Induktion pflanzlicher Subtilasen nach Pathogenbefall gelungen (Tornero et al., 2000; van der Hoorn und Jones, 2004). Um die Funktion der Subtilasen in planta genauer zu bestimmen, sollten in der vorliegenden Arbeit die Beziehungen der SlSBT3 zu Abwehrmechanismen und anderen durch Phytopathogene hervorgerufene Reaktionen beschrieben werden. So wurde die Expression der SlSBT3 untersucht, nachdem die Pflanze mit virulenten, avirulenten oder hrpdefizienten biotrophen Bakterien der Gattung Xanthomonas campestris pv. vesicatoria sowie mit einem hemibiotrophen Oomyceten, Phytophthora infestans, und einem nekrotrophen echten Pilz, Sclerotinia sclerotiorum, infiziert wurden. Daneben wurden der Einfluss bekannter Signalmoleküle der Pathogenabwehr und am Zelltod beteiligter Substanzen auf die Expression der SlSBT3 untersucht. Außerdem wurden unter Verwendung von transgenen Pflanzen, die sich in der Expressionsstärke der SlSBT3 unterschieden, das Bakterienwachstum und die auftretenden Krankheitssymptome untersucht. 4.1 Beeinflussung des Expressionsstatus der SlSBT3 durch Pathogenbefall Da bereits in Vorversuchen (Huttenlocher, unveröffentlicht) eine Änderung der Expression der SlSBT3 nach Befall durch Xanthomonas campestris beobachtet werden konnte, wurde m Rahmn dieser Arbeit eine genauere Analyse der Expressionnach Pathogenbefall durchgeführt. 71 Diskussion Die Untersuchungen wurden auf mehrere repräsentative Vertreter von Phytopathogenen ausgeweitet. So wurde die Wirtspflanze Tomate mit virulenten, avirulenten oder hrp-defizienten Bakterien, mit einem Oomyceten bzw. mit einem echten Pilz infiziert. Damit waren aus drei phylogenetisch verschiedenen Klassen repräsentative Vertreter von mikrobiellen Schaderregern ausgewählt, die gleichzeitig verschiedene pathogenetische Strategien, von biotroph über hemibiotroph bis hin zu nekrotroph, verfolgen. Die Ergebnisse der Expressionsanalyse mittels semiqRT (Abb. 3.2.1) nach Infektion durch X. campestris pv. vesicatoria sind nicht aussagekräftig. Durch die Induktion der SlSBT3 in der Kontrollbehandlung müssen in diesem Versuch Störfaktoren, wie eine zu große mechanische Beanspruchung der Blätter durch die Vakuuminfiltration oder ein Einfluss des Detergenz Silwet L77 berücksichtigt werden. Dies wurde durch die in den Kontrollen auftretende Induktion des PR3a-Gens in Folge in diesem Experiment ebenfalls deutlich. Durch Inokulation mit der Spritze und Analyse des Expressionsspiegels mit Northern Blot (Abb. 3.2.2) konnten trotz der geringeren Sensitivität der Nachweismethode aussagekräftigere Ergebnisse erzielt werden. Damit ist dieses Vorgehen besser geeignet, um einen eindeutigen Beweis der Expression der SlSBT3 nach bakterieller Infektion zu erbringen. Durch den avirulenten Stamm von X. campestris pv. vesicatoria wurden über den gesamten beobachteten Zeitraum die stärksten Signale detektiert. Die Induktion des PR-3a Gens zeigte sich in diesem Experiment einen ähnlichen zeitlichen Verlauf. Die unerwartet schwachen Signale des PR-3a Gens nach erfolgter Infektion könnten durch eine schwache Hybridisierung zu erklären sein oder auf eine unvollständige Erkennung des AvrBs3-Genprodukts im Pathosystem mit dem Tomatenkultivar UC82B zurückzuführen sein. Die Einleitung einer für die inkompatible Interaktion typischen hypersensitiven Reaktion (HR) spricht aber aufgrund der frühzeitig beobachteten Symptome und deren Unterschiede zu denen des virulenten Stamms von Xcv (vgl. Kap. 3.5 für eine partielle Erkennung von Xcv pDsK 200 als avirulentes Pathogen. Das Pr-3a Signal 24 Stunden nach Inokulation weicht in der Intensität von 72 Diskussion den anderen beobachteten stark ab. Damit muss angenommen werden, dass die untersuchen Proben zu diesem Zeitpunkt nicht ausreichend infiziert waren, was das Ausbleiben eines Signals nach der Hybridisierung mit der SlSBT3-Sonde zu diesem Zeitpunkt erklären könnte. Die unregelmäßige Verteilung des Signals nach 48 Stunden muss als ein Artefakt des RNATransfers auf die Membran gedeutet werden, da sich nur zu diesem Zeitpunkt ein undeutliches Signal zeigt. Die Induktion der SlSBT3 durch den avirulenten Stamm deutet auf ein R-Gen-vermitteltes Induktionssignal hin. Mit der R-GenErkennung kommt es zur Einleitung einer effektiven Pathogenabwehr verbunden mit der Induktion von PR-Proteinen und der Einleitung der HR. Nach Inokulation mit dem virulenten Stamm von Xcv lässt sich eine Induktion der SlSBT3 erst nach fünf Tagen, vergleichbar mit einer Infektion mit dem avirulenten Stamm gleichzeitig mit Krankheitssymptomen beobachten, wobei den das ersten sichtbaren Expressionssignal nur geringfügig stärker ist als das Signal der konstitutiv exprimierten SlSBT3 zum Zeitpunkt Null. Zum gleichen Zeitpunkt ist auch die stärkste Induktion des PR3a-Gens zu beobachten. Mit dieser Beobachtung könnte die Induktion der SlSBT3 auf eine Effektor-vermittelte Erhöhung der Genexpression oder auf das lokale Absterben des Gewebes infolge der Infektion zurückzuführen sein. Mit dem Nachweis der Induktion der SlSBT3 ausgelöst durch den nicht pathogenen hrp-defizienten Stamm von Xcv kann grundsätzlich eine spezifische TTS-vermittelte Induktion der SlSBT3 in diesem PathogenWirtspflanze-System ausgeschlossen werden. Mit dem hrp-defizienten Stamm ist eine Induktion der SlSBT3 acht Stunden nach Inokulation festzustellen. Die Expression von PR-3a war nach acht Stunden im Vergleich zum Zeitpunkt Null nicht erhöht. Die Abnahme des Signals der PR3a-Hybridisierung scheint im weiteren Verlauf schwächer zu sein als das Signal vor der Inokulation. Somit kann eine geringe Induktion der PR-Gene der Versuchspflanzen vor der Inokulation nicht ausgeschlossen werden. Das untersuchte RNA-Isolat zum Zeitpunkt Null war für alle durchgeführten Northern Blot-Analysen identisch. Daher kann die geringere Expression des PR-3a-Gens der hrp-Stamm infizierten Proben im Vergleich zum Zeitpunkt Null nicht als spezifisches Merkmal dieser Infektion gedeutet 73 Diskussion werden. Vielmehr muss von einer minimalen Beeinträchtigung der Versuchspflanzen ausgegangen werden, die allerdings aufgrund der geingen Unterschiede für die Aussage des gesamten Versuchs vernachlässigt werden kann, da die entsprechenden Kontrollpflanzen keine Abweichungen in der Pr-3a Genexpression zeigen. Demnach besteht in diesem Fall keine Abhängigkeit der SlSBT3-Expression von der Induktion des PR-3a-Gens. Schlussfolgernd kann eine Induktion der SlSBT3 mit einer starken Induktion von PR-Genen, dem Eintreten von sichtbaren Blattveränderungen, wie HR oder Krankheitssymptomen, und der Erkennung von Bakterien im Apoplasten in Verbindung gebracht werden. Die Induktion der SlSBT3 nach acht Stunden und die schnelle Abnahme dieses Signals im weiteren Verlauf der Analyse, würden hierbei für eine Funktion in der frühen basalen Resistenz sprechen. Untersuchungen zur differentiellen Genexpression nach Inokulation mit einem hrp-defizienten Stamm von P. syringae ergaben eine induzierte Expression von etwa 800 regulierten Genen in Arabidopsis nach zwei bis zwölf Stunden (Truman et al., 2005). Im Zusammenhang mit der frühen basalen Resistenz konnte festgestellt werden, dass es sich dabei insbesondere um Gene handelt, die für extrazelluläre Proteine codieren. So wurde für eine in der frühen basalen Resistenz nachgewiesene extrazelluläre Chitinase ein vergleichbarer Zeitverlauf der Expression mit einem Maximum nach acht Stunden gezeigt (Szatmari et al., 2006). In der Literatur ist bereits ein ähnliches Expressionsmuster mit einem Induktionsmaximum acht Stunden nach Inokulation für die apoplastische Pepsin-ähnliche Aspartatprotease CDR1, eine extrazelluläre Protease mit Funktion in der effektiven Abwehr von P. syringae, beschrieben (Xia et al., 2004). Außerdem wurde für die Subtilasen-Gene bereits früher eine schnelle stressinduzierte Expression postuliert, da sie keine Introns beinhalten (Meichtry et al., 1999). Im weiteren Verlauf der Untersuchungen wurde die Expressionsanalyse der SlSBT3 nach erfolgter Infektion durch Eukaryonten ausgeweitet. Die Translokation von Effektoren durch eukaryontische Pathogene ist nicht 74 Diskussion beschrieben (Ellis et al., 2006). Daher finden die Erkennung und die erste Abwehr von Oomyceten und echten Pilzen im extrazellulären Bereich statt, wo die Aktivität der SlSBT3-Protease vermutet wird. Kompatible hemibiotrophe Organismen, wie das hier verwendete Kultivar von P. infestans, durchlaufen eine Phase biotrophen Wachstums, in der sich die Population etabliert, gefolgt von einer Phase des nekrotrophen Wachstums. Pflanzliche extrazelluläre Proteine stellen bei der Etalierung der Population ein bekanntes Ziel von Effektoren der Oomyceten und echten Pilzen dar. Für einen Vertreter der oben genannten P69-Subtilasen aus Tomate, P69B, konnte bereits eine funktionelle Interaktion mit solchen Effektoren nachgewiesen werden (Tian et al., 2004; Tian et al., 2005). Auch für SlSBT3 wurde eine Hemmung der proteolytischen Aktivität durch Virulenzfaktoren von P. infestans gezeigt (Knappenberger, unveröffentlicht). Für weitere extrazelluläre Proteasen konnte eine vergleichbare inhibierende Funktion durch P. infestans sekretierte Peptide gezeigt werden. Darunter die CysteinProtease PIP1 deren Expression bereits zwei Tage nach Infektion bzw. nach Behandlung mit einem SA-Analogon induziert wird (Tian et al., 2007). Unter Vorraussetzung, dass der polyklonale Antikorper eine ausreichend spezifische Identifizierung der SlSBT3 erlaubt, wurde die Induktion der SlSBT3 in Tomatenblättern mittels Western Blot besonders am vierten und achten Tag nach Inokulation beobachtet (Abb. 3.8). Dies sind Zeitpunkte bei denen bereits ein Wechsel von biotropher zu nekrotropher Lebensweise bei P. infestans angenommen werden kann. So muss davon ausgegangen werden, dass die SlSBT3-Genexpression durch diesen Wechsel der Nahrungsgrundlage beeinflusst wird und eine rein biotrophe Lebensweise dieses Pathogens keine stark induzierenden Signale freisetzt. Aus Phytophthora sojae ist ein Nekrose induzierender Peptid-Elicitor, PsojNIP, bekannt, der 48 Stunden nach Inokulation auf Transkriptebene in der Wirtspflanze nachzuweisen ist (Kamoun, 2006, Vleeshouwers et al., 2006). Aufgrund der nahen Verwandschaft der beiden Oomyceten kann ein solcher Elicitor auch für P. infestans vermutet werden. Der Einfluss eines solchen Elicitors würde das Maximum der SlSBT3-Expression auf Transkriptebene vier Tage nach Inokulation erklären und eine Stabilisierung des Proteins 75 Diskussion durch eine postranslationelle Veranderungen beweisen. Mit der Beobachtung , dass am achten Versuchtag grossflächige Nekrosen sihtbar wurden, kann die Vermutung aus der Expressionsanalyse nach Infektion durch Bakterien bestätigen, dass die Induktion der SlSBT3 mit dem Absterben von Geweben einhergeht. Da die erhöhte Expression der SlSBT3 auch mit dem Wachstum eines nekrotrophen Pilzes, S. sclerotiorum, im Blattgewebe beobachtet werden kann (Abb. 3.6.1), ist eine Abhängigkeit der Induktion der SlSBT3 von einer R-Gen-vermittelten Abwehr auszuschliessen. Eine solche Effektor-vermittelte Abwehr führt zu keiner erfolgreichen Eingrenzung des Wachstums, da durch den eingeleiteten programmierten Zelltod in Form von einer HR dem Pilz nicht die Lebensgrundlage entzogen wird (Mur et al., 2008; Govrin und Levine, 2000; Jones und Dangl, 2006). Somit bestätigt sich, dass die induzierte Expression der SlSBT3 nicht auf die spezifische Erkennung des TTSS-Effektors AvrBs3 des biotrophen Prokaryonten X. campestris pv. vesicatoria zurückgeführt werden kann. Demnach kann eine Induktion der SlSBT3 nicht als HR-spezifisch betrachtet werden, sondern ist allgemeiner gefasst mit einer Einleitung von Zelltod und damit verbundenen Prozessen assoziiert. Spezifische Pathogen-assozierte Strukturen und Effektoren von P. infestans können ebenfalls als spezifische induzierende Signale ausgeschlossen werden. Die Besiedlung von Wirtspflanzen durch Nekrotrophe erfolgt in der Abtötung des Gewebes Plasmamembran sowohl durch angreifen, als Elicitoren, auch welche durch die pflanzliche wirtspezifische oder wirtsunspezifische Toxine (Govrin und Levin, 2000; Mayer et al, 2001; Cotton et al., 2002). Ein bekanntes wirtsunspezifisches Toxin aus S. sclerotiorum ist Oxalsäure (Hegedus und Rimmer, 2005). Oxalsäure ist ein Elicitor, der programmierten Zelltod einleitet (Kyoung et al., 2008). Wie in Kap. 3.6 beschrieben, konnte unter Verwendung der SlSBT3Promotor::GUS-Pflanzen eine Reportergen-Induktion in Bereichen um die gebildetete Nekrose und eine Ausbreitung über die gesamte Blattfläche mit zunehmenden Wachstum des Pilzes beobachtet werden. Dies würde 76 Diskussion zusammen mit den vorhergehenden Ergebnissen den Zelltod als induzierendes Signal bestätigen. Die Zunahme der GUS-Färbung scheint dabei mit einem zunehmenden Wachstum von S. sclerotiorum korreliert zu sein. So kann die Induktion des SlSBT3-Promotors durch ein mobiles Signal nach Befall durch S. sclerotiorum bzw. der Auslösung von Zelltod vermutet werden, da durch die die GUS-Aktivität im proximalen Fiederblatt verstärkt wird. Mit der Infiltration von Cantharidin sollte die Induktion der SlSBT3 durch ein Zelltodsignal, unabhängig von Pathogenbefall, bestätigt werden. Cantharidin löst in Pflanzenzellen durch Proteindephosphorylierung über einen Cytochrom c-unabhängigen Weg programmierten Zelltod aus (Robson und Vanlerberghe, 2002). Die über GUS-Reportergen analysierten Expressionsmuster der SlSBT3 nach Infiltration von Cantharidin stimmten mit dem durch S. sclerotiorum nach 48 bzw. 72 Stunden in soweit überein, dass es zu einer Aktivität des SlSBT3Promotors über die gesamte Blattfläche kommt (Abb. 3.6.2 und Abb. 3.9.2). Im Gegensatz dazu führt die durch Cantharidin hervorgerufene Nekrose in den direkt benachbarten Zellen nicht zu einer verstärkten Induktion des Promotors, wie sie für die inokulierten Blätter nach 48 und 72 Stunden auftraten. Das könnte auf Chlorosen in diesem Bereich der Cantharidininfiltrierten Blätter gefolgt von einer Beeinträchtigung der Expression des Reportergens zurückzuführen sein. Mit diesem Ergebnis kann ein mobiles induzierendes Signal ausgehend von der Nekrose vermutet werden. Weiterhin zeigt dieser Versuch, dass eine von Pathogenbefall unabhängige Induktion der SlSBT3 durch ein Zelltod-assoziiertes Signal beobachtet werden kann. 4.2 Einfluss von Salicylsäure und H2O2 auf die Expression der SlSBT3 Nach Auslösung des „oxidative burst“ durch Pathogenbefall kommt es über die Akkumulation von Salicylsäure zur Änderung des Redoxstatus der Zelle und damit verbunden zur Aktivierung des Salicylsäure-abhängigen Transkriptionsfaktors NPR1, der eine Reihe von Salicylsäure-abhängien Reaktionen auf Pathogenbefall kontrolliert (Beckers und Spoel, 2005). 77 Diskussion SA-vermittelte Signale werden daher sowohl mit der R-Gen-vermittelten als auch mit der basalen Abwehr von biotrophen Pathogenen verbunden (Glazebrook, 2005). Eine induzierte Expression durch Salicylsäure wurde bereits für die Subtilasen P69B und P69C über GUS-Reportergenanalysen gezeigt (Jordá et al., 1998, Jordá et al., 2000). Vergleichbare Expressionsmuster mit einem Induktionsmaximum nach acht und zwölf Stunden (Abb. 3.2.2) konnten z. B. für die Carboxypeptidase OsBISCPL1 aus Oryza sativa in einer inkompatiblen Interaktion gezeigt werden (Liu et al., 2008). Daneben wird die Auslösung von programmiertem Zelltod, in Form von HR, auf die Akkumulation von Salicylsäure direkt über die Erkennung positiver HR-Regulatoren, wie dem Transkriptionsfaktor AtMYB30 in Arabidopsis, in Verbindung gebracht (Raffaele et al., 2006). Im Rahmen der hier durchgeführten Versuche wurde auch eine Induktion in den Kontrollpflanzen festgestellt, die allerdings schwächer war als in den SAbehandelten Pflanzen. Möglicherweise haben Stressfaktoren wie mechanische Beanspruchung der Blätter infolge von Besprühen oder Infiltrieren bzw. Detergenzien wie Tween 20 als chemische Induktoren zu der beobachteten Induktion der SlSBT3 und dem PR-Protein PR-3a wie bei der Expressionsanalyse nach Bakterieninfektion mit der Methode der Vakuuminfiltraion und mit Hilfe des Detergenz Silwet L77 geführt. In Arabidopsis wurden bereits vergleichbare Beobachtungen zur induzierten Genexpression durch Detergenzien wie Tween 20 beschrieben (Hunzicker et al., 2004). Mit dem Ziel die Abhängigkeit der SlSBT3-Expression von H2O2 zu analysieren, wurde das bei der Interaktion mit Pathogenen gebildete H2O2 durch Infiltration von Katalase abgefangen. ROS spielen bei der effektiven Pathogenabwehr eine tragende Rolle. Durch sie kommt es z. B. durch Reaktion mit Zellwandstrukturen zur Bildung mechanischer Barrieren, die ein Eindringen von Pilzen unterdrücken. H2O2 wirkt daneben als Signalmolekül bei der Auslösung des Pathogenbefall abhängigen Zelltods (Mur et al., 2008, Gechev und Hille, 2005). Durch das Abfangen von H2O2 an der Kontaktfläche zu S. sclerotiorum scheint die GUS-Aktivität schneller induziert zu werden (Abb. 3.9.1). Im 78 Diskussion Umkehrschluss kann dadurch im Pathosystem mit S. sclerotiorum eine reprimierende Aktivität von H2O2 auf den SlSBT3-Promotor angenommen werden. Auch ohne zusätzliche Infiltration von Katalase kann bei Besiedlung durch S. sclerotiorum der „oxidative burst“ mit dem Herabsenken des pHWerts mittels Oxalsäure unterdrückt wird (Hegedus und Rimmer, 2005). Daher kann die Expression der SlSBT3 in diesem Pathogen-WirtspflanzeSystem als H2O2 unabhängig betrachtet werden. Da die genauen Abläufe der pH-Wert Änderung und der Aktivierung der für die Bildung von ROS verantwortlichen Enzyme in diesem Pathosystem nicht genau bekannt sind, kann ein induzierendes Signal auch vor der Bildung von H2O2 nicht ausgeschlossen werden. So wäre es möglich, dass andere ROS oder die pH-Wert Absenkung direkt zu einer Induktion der SlSBT3 beitragen. Das Pilztoxin Fusicoccin verursacht in Tomatensuspensionskulturzellen durch Aktivierung der H+-ATPase eine Absenkung des pH-Wertes, durch die bereits zur Induktion von PR-Genen nahgewiesen werden konnte (Schaller und Oecking, 1999). Es wäre also denkbar, dass die Ansäuerung des Apoplasten durch die von S. sclerotiorum produzierte Oxalsäure zur Induktion der SlSBT3 führt. Ob diese Induktion physiologische Konsequenzen in einem solchen Milieu hat, ist fraglich, da in-vitro Untersuchungen keine proteolytische Aktivität für SlSBT3 im sauren pH-Bereich vorraussagen (Huttenlocher, unveröffentlicht). Die Induktion durch ein Zelltod-auslösendes Signal wäre demnach auch unabhängig Tabaksuspensionszellkulturen von nach H 2O 2. So wurde Zugabe von einem bereits Elicitor in aus P. infestans eine H2O2-unabhängige Einleitung von programmiertem Zelltod durch Hemmung von Serinproteasen beobachtet (Yano et al., 1999; Sasabe et al., 2000). 4.3 Einfluss der SlSBT3 auf das Wachstum von Pathogenen in Tomatenblättern Die erfolgreiche Besiedlung der Pflanze äußert sich in einer Etablierung der Population des Phytopathogens im Blattgewebe. Im Falle einer Funktion der SlSBT3 in der Pathogenabwehr sollte der Expressionsspiegel der SlSBT3 in Überexpressions-Linien und RNAi-Linien einen Einfluss auf das Wachstum und die Ausbreitung der Pathogene haben. 79 Diskussion Das Wachstum der biotrophen Bakterien zeigte sich in der RNAi-Linie bei allen Untersuchen Stämmen von X. campestris pv. vesicatoria 85-10 erniedrigt. Das Wachstum in SlSBT3-Überexpressionspflanzen und Wildtyppflanzen zeigte durch Infektion mit dem virulenten und avirulenten Stamm keine signifikanten Unterschiede (Abb. 3.4.1 und Abb. 3.4.2). Im Wachstum des hrp-defizienten Stamms wurden die Unterschiede die sich im Wachstum der beiden anderen pathogenen Stämme andeuteten am besten deutlich (Abb. 3.4.3). Auch wenn die beobachteten Populationsgrösse in der RNAi-Linie am zweiten Probentag, um ein vielfaches das der Untersuchungen mit dem virulenten und avirulenten Stammes unterschreitet, kann der allgemeine Trend totzdem an den anderern erzielten Datenpunkten erkannt werden. Zudem konnte ein verbessertes Wachstum in Überexpressionspflanzen beobachtet werden. Das Wachstum des virulenten Stamms zeigt nur in den SlSBT3-RNAi-Linien ein geringeres Wachstum und spiegelt das Ergebnis der Beobachtungen der Wachstumskurven der beiden anderen Stämme. Das Wachstum des avirulenten Stamm von X. campestris pv. vesicatoria 8510 mit einem Maximum nach sechs Tagen lässt einen Effekt hervorgerufen durch eine spezifische Erkennung des AvrBs3-Genprodukts ausschließen. Das stimmt mit der Aussage der Expressionsanalysen in soweit überein, als das auch hier kein Einfluss des AvrBs3-Effektors auf die Expression der SlSBT3 beobachtet werden kann. Die beobachtete Populationsgröße von 104 cfu/ cm2 scheint hierbei die Vermutung der Expressionsanalyse zu bestätigen, wonach die Erkennung des AvrBs3-Genproduktes nur unvollständig in dem verwendeten Tomatenkultivar UC82B stattfindet. Durch die Expression dieses AVR-Genproduktes findet keine effektive Abwehr von Xcv durch Auslösung einer vollständigen inkompatiblen Interaktion statt. Die Einleitung einer hypersensitiven Reaktion scheint davon unbetroffen zu sein. Die Tatsache, dass in diesen Pflanzen mit einer niedrigen koloniebildenden Einheit (cfu/ml) der infiltrierten Bakteriensuspension nach acht Tagen in den untersuchten Pflanzen HR zu beobachten war, die bei der Untersuchungen mit dem virulenten Stamm ausblieben, wie die Inkubation auf 80 Diskussion unterschiedlichen Selektionsmedien, schliessen ein Vertauschen von virulentem und avirulentem Stamm aus. Damit lassen sich die Ergebnisse der Expressionsanalyse bestätigen. TTSSsekretierte Effektoren haben keinen diekten Einfluss auf eine durch SlSBT3 geänderte Pathogenantwort, unter der Berücksichtigung, dass die Erkennung des AvrBs3-Gens nur unvollständig erfolgt. Mit dem besseren Wachstum des hrp-defizienten Stamm in der Überexpressionslinie ließe sich prinzipiell eine Pathogen abwehrende Funktion ausschließen. Liu et al. teilten die Pathogen-induzierten Proteine basierend auf ihrer Funktion in fünf Gruppen. Die erste Gruppe stellen Enzymen dar, die an der Synthese von Phytoalexinen beteiligt sind. Zur zweiten Gruppen gehören Proteine, die zur Lyse der Zellwand von Pathogenen beitragen, wie Chitinasen oder β1-3 Glucanasen. Defensine, die eine Limitierung der Ausbreitung von Pathogenen erreichen, indem sie mit der mikrobiellen Zellmembran interagieren, gehören zur dritten Gruppe. Bei der vierten Gruppe, der durch Pathogenbefall induzierten oder aktivierten Proteine, handelt es sich um Proteasen, die in ihrer Beteiligung am Zelltod als HR-assozierten Proteine klassifiziert werden (Liu et. al., 2001). Die Expressionsanalyse berücksichtigend, könnte SlSBT3 in diese vierte Gruppe der HR involvierten Proteasen zu gezählt werden. Mit der Beobachtung der Unterstützung der Population durch Überexpression der SlSBT3 wäre SlSBT3 in die letzte Gruppe nicht weiter spezifizierter Proteine einzuordnen. Eine mögliche Erklärung wäre, dass einer Alkalisierung des Apoplasten durch Pathogenbefall eine erhöhte proteolytischen Aktivität folgt, die der Prozessierung von wachstumsfördernden Faktoren für X. campestris pv. vesicatoria 85-10 in Tomate dient. Eine direkte Pathogen abwehrende Rolle der SlSBT3 in diesem Pathogen-Wirtspflanze-System hätte dabei eine untergeordnete Funktion. Die beobachtenden Krankheitssymptome von X. campestris pv. vesicatoria 85-10 zeigten in der Bildung der Nekrosen ausgelöst durch den virulenten und avirulenten Stamm keine qualitativen makroskopischen Unterschiede 81 Diskussion (Abb. 3.5.1). Da keine explizite Beobachtung der Krankheitssymptome in einem geringeren Abstand als 24 Stunden durchgeführt wurde, ist eine zeitlich versetzte Symptombildung bedingt durch die biologische Variabilität der beiden Interaktionspartner nicht berücksichtigt worden. Dadurch konnte in allen drei SlSBT3-Genotypen für den virulenten und avirulenten Stamm kein phänotypischer Unterschied offengelegt werden. Die Beobachtung, dass sich in der RNAi-Linie Krankheitssymptome durch den hrp-defizienten Stamm zeigten, die sich im Wildtyp vermindert ausgeprägt erwiesen (Abb. 3.5.1 und 3.5.2), spricht für eine mögliche Beteiligung der SlSBT3 in der basalen Resistenz durch die Erkennung von Bakterien oder als eine Antwort auf den Stressfaktor des Wachstums bzw. Stoffwechsels der hrp-defizienten Bakterien im Blattgewebe, wie sie mit der erhöhten Expression der SlSBT3 angedeutet wurde. Das Auftreten von Krankheitssymptomen durch nicht pathogene Mikroorganismen kann als erfolgreiche Abwehr einer Nichtwirtspflanzen-Resistenz betrachtet werden und ist mehrfach beschrieben (Desaki et al., 2006; Abramovitch und Martin, 2004). Mit der Beobachtung, dass es bei Infektion durch S. sclerotiorum in der RNAiLinie zur besseren Wachstum kommt (Abb. 3.6), lässt sich dagegen die Annahme untermauern, dass die Funktion der SlSBT3 in der Auslösung des Zelltods zu vermuten ist. Der Mangel der SlSBT3 führt zu einer erfolgreicheren Besiedlung des Blattgewebes durch S. sclerotiorum. Demnach könnte SlSBT3 als induziertes Abwehrprotein eine Funktion in der basalen Resistenz gegenüber S. sclerotiorum, bzw. das Ergebnis der Expressionsanalyse von P. Infestans mit einbeziehend, gegenüber nekrotroph lebenden Eukaryonten haben. Hiermit kann eine Zelltod unterdrückende Funktion für SlSBT3 angenommen werden. 4.4 Die Rolle der SlSBT3 in der Pathogenabwehr Basierend auf den hier vorgelegten Beweisen für eine von Pathogenbefall abhängige Induktion der SlSBT3, kann dieses Enzym, wie die Tomatensubtilasen aus der P69-Subfamilie, als PR-Protein, bezeichnet 82 Diskussion werden (Hock und Elstner, 1995). Eine offensichtliche Pathogen abwehrende Funktion muss dabei nicht nachgewiesen sein (van der Loon et al., 2006). Zusammenfassend kann eine direkte Beteiligung der SlSBT3 an der Pathogenabwehr von Xanthomonas campestris pv. vesicatoria 85-10 ausgeschlossen werden. SlSBT3 kann als Ziel einer direkten TTSSvermittelten Effektor-Immunität in diesem Pathogen-Wirtspflanze-System ausgeschlossen werden. Diese Schlussfolgerung wird mit den Wachstumskurven bestätigt. Durch das verbesserte Wachstum des hrpdefizienten Stamms in der SlSBT3-Überexpressions-Linie findet auch keine Einschränkung des Wachstums von nicht pathogenen Bakterien statt. Damit ist die Beteiligung an einer direkten PAMP-vermittelten basalen Abwehr von Prokaryonten ebenfalls anzunehmen. Damit könnte die die erhöhte Expression der SlSBT3 nach Inokulation mit dem hrp-defizienten Stamm nur auf eine indirekte Erkennung der Bakterien zurückgeführt werden. Die starke Induktion der SlSBT3 nach Infektion mit einem Oomyceten und einem echten Pilz könnten auf eine Beteiligung bei der PAMP-vermittelten basalen Abwehr gegenüber Eukaryonten hinweisen. Dies bestätigte das verbesserte Wachstum von S. sclerotiorum in der RNAi-Linie. Da es sich bei den untersuchten Interaktionspartnern der PathogenWirtspflanze-Beziehungen um Vertreter verschiedener Reiche und Klassen von Pathogenen mit enormen Unterschieden in der Besiedlungsstrategie handelt, ist ein gemeinsames Pathogen-assoziertes Signal weitgehend auszuschließen. In der Literatur sind einzelne PAMPs nur für einzelne Vertreter von bestimmter Klassen beschrieben (Nürnberger et al., 2004). Ein über Reiche oder Klassen übergreifende verbreitetes PAMP ist nicht beschrieben und ist durch die vielseitigen strukturellen Unterschiede von Mikroorganismen einleuchtend erklärbar. Demzufolge kann ein induzierendes Signal nur durch ähnliche Infektionsstrategien oder wie im Fall des avirulenten Interaktionspartners durch die Gemeinsamkeit der ausgelösten Reaktion gesucht werden (Schwessinger und Zipfel, 2008). 83 Diskussion Alle Ergebnisse gemeinsam betrachtet, weisen auf eine starke Zelltodassozierte Expression und Funktion der SlSBT3 in der Pflanze hin, die unabhängig von einer Bildung von H2O2 zu sein scheint. Zelltod-Regulierung ist in Pflanzen und Tieren verbunden mit effektiver Pathogenabwehr ein weit verbreiteter Mechanismus (Greenberg et al. 1994; Lam et al., 1999). Demnach könnte die Pathogen abwehrende Rolle in der Beteiligung der SlSBT3 bei der Auslösung von Zelltodprozessen vermutet werden. In der Literatur sind viele Zelltod-assoziierte Proteasen beschrieben, die sich durch identifizierte Elicitoren aus Oomyceten und Pilzen induziert zeigen (Beers et al., 1997; Beers et al.; 2000; Bonneau et al., 2008). Solche Proteasen werden, aufgrund der tierischen Proteasen analoge Funktion in der Auslösung von Zelltod beteiligt zu sein, Caspasen genannt. Eine den tierischen Caspasen ähnliche Cathepsin B-ähnliche Protease zeigt sich in Solanum tuberosum durch avirulenten Stamm von Erwinia amylovora und P. infestans induziert (Avrova et al., 2004, Gilroy et al., 2007). Serinproteasen mit Zelltodfunktion sind aus Avena sativa bekannt (Coffeen und Wolpert, 2004). Eine Reihe von Proteasen werden bei der Regulierung des Zelltods in Pflanzen diskutiert. Interessanterweise zeigten bisher postulierte Vertreter solcher Proteasen eine ähnliche Expression nach Pathogenbefall. Zwei sogenannte Metacaspasen, AtMCO1a und AtMCP1b, zeigen sich in Northern Blot Analysen sowohl durch virulente als auch durch avirulente Stämme bakterieller Pathogene induziert. Eine andere vermutete Metacaspase aus Tomate, LeMCA1, konnte nach Befall durch Botrytis cinerea, einem nekrotrophen Ascomyceten, mit einer Erhöhung des Transkriptspiegels nachgewiesen werden (Watanabe und Lam, 2004). Die Beteiligung solcher Cystein-Proteasen an der Regulierung des Zelltods wird nur aufgrund von Sequenzähnlichkeit zu tierischen Caspasen hergeleitet. Homologe Proteine zu Caspasen oder Regulatoren mit anti- oder proapoptischer Funktion sind nicht bekannt. Eine Identifikation von Funktions- und Strukturhomologen wird zudem aufgrund der unterschiedlichen Entwicklung von tierischen und 84 Diskussion pflanzlichen Systemen bis auf oben genannte Strukturvergleiche ausgeschlossen (Watanabe und Lam, 2004). Die Untersuchungen zum Wachstumsverhalten der Bakterien würde eine solche physiologische Funktion für SlSBT3 mit den beobachteten frühen Symptomen in der SlSBT3 RNAi-Linie unter dieser Annahme erklären. Eine mögliche antiapoptotische Wirkung der SlSBT3 wäre in der Überexpressions-Linie verstärkt und würde eine effektive Abwehr von biotrophen Pathogenen in Form von vermindertem Wachstum verhindern, wohingegen in der RNAi-Linie eine antiapoptotische Wirkung vermindert ist was eine Bildung von Symptomen verstärkt oder einen früheres Auftreten bedingt. Solche Unterschiede wären bei Untersuchungen mit kompatiblen oder inkompatiblen biotrophen Phytopathogenen unter Umständen nur in einem zeitlich versetzten Auftreten oder nur auf mikroskopischer Ebene deutlich. Mit der hier durchgeführten Probennahme in einem 24 Stunden-Rhythmus kann eine solche phänotypische Erscheinung für den virulenten und avirulenten Stamm von Xanthomonas campestris pv. vesicatoria nicht ausgeschlossen werden. Das frühe Auftreten von Nekrosen durch den hrp-defizienten Stamm, beobachtet in der SlSBT3-RNAi-Linie, könnte mit einer solchen antiapoptotischen Funktion der SlSBT3 in Verbindung gebracht werden. Beispiele von vergleichbaren Phänotypen finden sich in Mutanten von Zelltodregulatoren nach Zellstod-Stimuli wie Pathogenbefall. So konnte durch Expression des antiapoptotischen p35 aus Baculoviren eine verstärkte Resistenz in Form von geringeren Symptomen nach Alternaria– Befall in Tomatenfrüchten gezeigt werden (Lincoln et al., 2002). Infiltration von tierischen Caspase-Hemmern führte zu verminderten Krankheitssymptomen von P. syringae in Tabak und X. campestris pv. vesicatoria in Tomate (Richael et al. 2001). In Zelltod-Mutanten wie der ACD2 (accelareted cell death), ein Protein der Plasmamembran aus Arabidopsis, konnte in der Überexpessions-Linie 85 Diskussion ebenfalls keine messbaren Unterschiede im Wachstum von avirulenten Bakterien im Blattgewebe offen legen, zeigten aber eine verminderte Bildung von Krankheitssymptomen in Überexpressionspflanzen (Mach et al., 2001). 86 Ausblick Ausblick In dieser Arbeit konnte gezeigt werden, dass SlSBT3 durch Pathogenbefall induziert wird. Weitere Arbeiten mit Pathogenen wie Pseudomonas syringae oder Alternaria solani könnten die beobachteten Ergebnisse letztendlich bestätigen. Desweiteren wäre interessant zu sehen, ob sich der beobachtete Phänotyp in der SlSBT3-RNAi-Linie nach Infektion durch Sclerotinia sclerotiorum mit Real-time-PCR quantitativ erfassen lässt und ob eine Infiltration mit aktivem SlSBT3 eine komplementierende Wirkung in der SlSBT3 RNAi-Linie hat. Ähnliche Ansätze wären auch für das beobachtete Wachstum und der Ausprägung von Symptomen mit dem hrp-defizienten Stamm von Xanthomonas campestris pv. vesicatoria möglich. Da alle bis jetzt durchgeführten Wachstumsexperimente nur in jeweils einer SlSBT3Linie gemacht wurden, sollten zukünftig die gleichen Versuche in mehreren unabhängigen SlSBT3-RNAi und -Überexpressions-Linien getestet werden. Unterschiede in der Entwicklung von Krankheitssymptomen durch den virulenten und avirulenten Stamm von X. campestris pv. vesicatoria könnten eventuell durch Beobachtung in einem zeitlich engeren Rahmen und in histologischen Schnitten besser beobachtet und herausgestellt werden. Unabhängig von Pathogenen wäre es interessant zu sehen, ob andere Zelltodsignale zu einem ähnlichen Verlauf der Bildung von Symptomen in SlSBT3-RNAi und –Überexpressionspflanzen führen. Mit dem Ziel eine eindeutige Aussage über die Abhängigkeit der SlSBT3Expression von Salicylsäure zu bekommen, sollte die Expressionsanalyse in verschiedenen Mutanten in der endogenen Salicylsäure-Perzeption des ausgelösten Signals endgültig bestimmt werden. In der nahG Mutante kann Salicylsäure nicht akkumulieren. Dadurch bleibt die Salicylsäure-abhängige Induktion von PR-Proteinen aus (Achuo et al., 2004). Mit dieser Mutante wäre bei einer Abhängigkeit von Salicylsäure keine Induktion von SlSBT3 nach Pathogenbefall nachweisbar. 87 Ausblick Die hier im Rahmen dieser Arbeit durchgeführten Versuche der Aktivität des SlSBT3-Promotors nach Behandlung mit Cantharidin und H2O2 sind nur als erster Versuche zu verstehen. Eine endgütige Aufklärung der Abhängigkeit von H2O2 könnten spezifische Nachweise der SlSBT3-Expression auf Transkriptunabhängig oder von Proteinebene einer bestätigt Infektion werden. vergleichbare Daneben müssten Untersuchungen mit Oxalsäure, Superoxiddismutase und H2O2–generierenden Substanzen wie Glucose und Glucose-Oxidase durchgeführt werden. So könnte auch untersucht werden, ob sich eine Induktion auch durch andere Zellgifte nachweisen ließe. Im Hinblick auf die von Phytophthora infestans sekretierten Kazal-Inhibitoren müsste nach den hier vorgelegten Daten ein Phänotyp in den SlSBT3-RNAiund -Überexpressions-Linien zu identifizieren sein. Dabei könnte mit der Bestätigung der Inhibierung des SlSBT3 durch Kazal-Inhibitoren in vivo die Relevanz dieser Protease in der Pathogenabwehr auf ein wirtschaftlich interessantes Pathogen-Wirtspflanzen-System bezogen werden. 88 Literatur Literatur Abramovitch R.B., Martin G.B. (2004): Strategies used by bacterial pathogens to suppress plant defenses. Cur Op Plant Biol 7, 356-364 Achuo E.A., Audenaert K., Meziane H., Höfte M. (2004): The salicylic aciddependent defence pathway is effective against different pathogens in tomato and tobacco. Plant Pathology 53, 65-72 Baker B., Zambryski P., Staskawicz B., S.P., Kumar D. (1997): Signaling in plant-microbe interactions. Science 276, 726-733 Barr P.J. (1991): Mammalian subtilisins: the long-sought dibasic processing endoproteases. Cell 66, 1-3 Beckers G.J.M., Spoel S.H. (2005): Fine-tuning plant defence signalling: Salicylate versus jasmonate. Plant Biol 8, 1-10 Beers E.P. (1997): Programmed cell death during plant growth and development. Cell Death Diff 4, 649-661 Beers E.P., Woffenden B.J., Zhao C. (2000): Plant proteolytic enzymes: possible roles during programmed cell death. Plant Mol Biol 44, 399-415 Bent A. F., Kunkel B. N., Dahlbeck D., Brown K. L., Schmidt R., Giraudat J., Leung J., Staskawicz B.J. (1994): RPS2 of Arabidopsis thaliana: a leucinerich repeat class of plant disease resistance genes. Science 265, 1856-1860 Berger D., Altmann T. (2000): A subtilisin-like serine protease involved in the regulation of stomatal density and distribution in Arabidopsis thaliana. Genes Dev. 14, 1119-1131 89 Literatur Bonneau L., Ge Y., Drury G. E., Gallois P. (2008): What happened to plant caspases? J Exp Bot 59, 491-499 Chamnongpol S., Willekens H., Langebartels C., Van Montagu M., Inzé D., Van Camp W. (1994): Transgenic tobacco with a reduced catalase activity develops necrotic lesions and induces pathogenesis-related expression under high light. Plant J 10, 491-503 Chen Z., Kloek A.P., Cuzick A., Moeder W., Tang D., Innes R.W., Klessig D.F., Kunkel B.N. (2004): The Pseudomonas syringae type III effector AvrRpt2 functions downstream or independently of SA to promote virulence on Arabidopsis thaliana. Plant J 37, 494-504 Ciardi J., Tieman D., Jones J., Klee H.J. (2001): Reduced expression of the tomato ethylene receptor LeETR4 enhances the hypersensitive response to Xanthomonas campestris pv. vesicatoria. Mol Pant Mic Int 14, 487-495. Chinchilla D., Bauer Z., Regenass M., Boller T., Felix G. (2006): The Arabidopsis receptor kinase FLS binds flg22 and determines the specificity of flagellin perception. Plant Cell 18, 465-476 Chisholm S.T., Coaker G., Day B., Staskawicz B.J. (2006): Host-microbe interactions: shaping the evolution of plant immune response. Cell 124, 803814 Coffeen W.C., Wolpert T.J. (2004) Purification and characterization of serine proteases that exhibit caspase-like activity and are associated with programmed cell death in Avena sativa. Plant Cell 16, 857-873 Cotton P., Rascle C., Fevre M. (2002): Characterization of PG2, an early endoPG produced by Sclerotinia sclerotiorum, expressed in yeast. FEMS Microb Lett 213, 239-244 90 Literatur Dangl J.L., Jones J.D.L. (2001): Plant pathogen and integrated defense responses to infection. Nature 411, 826-833 Desaki Y., Miya A., Venkatesh B., Tsuyumu S., Yamane H., Kaku H., Minami E., Shibuya N. (2006): Bacterial lipopolysaccharides induce defense responses associated with programmed cell death in rice cells. Plant Cell Physiol 47, 1530-1540 Deslandes L., Olivier J., Peeters N., Feng D.X., Khounlotham M., Boucher C., Somssich I., Marco Y. (2003): Physical interaction between RRS-R, a protein conferring resistance to bacterial wilt, and PopP2, a type-III effector targeted to the plant nucleus. Proc Natl Acad Sci USA 100, 8024-8029 Dong, X. (1998): SA, JA, ethylene, and disease resistance in plants. Curr Op Plant Biol 1, 316-323 Durrant W.E, Dong X. (2004): Systemic aquired resistance. Annu Rev Phytopath 42, 185-209 Ellis J., Catanzariti A. M., Dodds, P. (2006): The problem of how fungal and oomycete avirulence proteins enter plant cells. Trends Plant Sci 11, 61-63 Felix F., Duran J.D., Volko S., Boller T. (1999): Plants have a sensitive perception system for the most conserved domain of bacterial flagellin. Plant J 18, 265-276 Flor H.H. (1955): Host-parasite interaction in flax rust - its genetics and other implications. Phytopath 45, 680-685. Flor H.H. (1971): Current status of the gene-for-gene concept. Annu Rev Phytopathol 9, 275-296. Gechev T., Hille J. (2005): Hydrogen peroxide as a signal controlling plant programmed cell death. J Cell Biol 168, 17-20 91 Literatur Gilroy E.M., Hein I., Van Der Hoorn R., Boevink P.C., Venter E., McLellan H., Kaffarnik F., Birch P.R.J. (2007) Involvement of cathepsin B in the plant disease resistance hypersensitive response. Plant J 52, 1-13 Glazebrook J. (2005): Contrasting mechanisms of defense against biotrophic and necrotrophic pathogens. Annu Rev Phytopathol 43, 205-227 Golldack D., Vera P., Dietz K. J. (2003): Expression of subtilisin-like serine proteases in Arabidopsis thaliana is cell-specific and responds to jasmonic acid and heavy metals with developmental differences. Physiol. Plant 118, 64-73 Govrin E.M., Levine A. (2000): The hypersensitive response facilitates plant infection by the necrotrophic pathogen Botrytis cinerea. Curr Biol 10, 751-757 Grant J.J., Loake G.J. (2000): Role of oxygen intermediates and cognate redox signalling in disease resistence. Plant Physiol 124, 21-29 Grant M.R., Godiard L., Straube E., Ashfield T., Lewald J., Sattler A., Innes R.W., Dangl J.L. (1995): Structure of the Arabidopsis RPM1 gene enabling dual specificity disease resistance. Science 269, 843-846 Greenberg J.T., Guo A., Klessig D.F., Ausubet F.M. (1994): Programmed cell death in plants: A pathogen-triggered response activated coordinately with multiple defense functions. Cell 77, 551-563 Hammond-Kosack K.E., Jones, J.D.G. (1996): Inducible plant defence mechanisms and resistance gene function. Plant Cell 8, 1773-1791 Hauck P., Thilmony R., He S.Y. (2003): A Pseudomonas syringae type-IIIeffector suppresses cell wall-based extracellular defense in susceptible Arabidopsis plants. Proc Natl Acad Sci USA 100, 8577-8582 92 Literatur Hegedus D.D., Rimmer S.R. (2005) Sclerotinia sclerotiorum: When "to be or not to be" a pathogen? FEMS Microb Lett 251, 177-184 Hock B., Elstner E. (1994): Schadwirkungen auf Pflanzen. Spektrum, Heidelberg, Berlin, 324 f. Jones J.D., Dangl J.L. (2006): The plant immune system. Nature 444, 323329 Jordá L., Coego A., Conejero V., Vera, P. (2000): A genomic cluster containing four differentially regulated subtilisin-like processing protease genes is in tomato plants. J Biol Chem 274, 2360-2365 Jorda L., Conejero V., Vera, P. (1999): Characterization of P69E and P69F, two differentially regulated genes encoding new members of the subtilisin-like proteinase family from tomato plants. Plant Physiol 122, 67-73 Jorda L., Vera P. (2000): Local and systemic induction of two defense-related subtilisin-like protease promotors in transgenic Arabidopsis plants-Luciferin induction of PR gene expression. Plant Physiol 124, 1049-1057 Julius D., Brake A., Blair L., Kunisawa R., Thorner J. (1984): Isolation of the putative structural gene for the lysine-arginine-cleaving endopeptidase required for processing of yeast prepro-alpha-factor. Cell 37, 1075-1089 Kamoun S. (2006): A catalogue of the effector secretome of plant pathogenic oomycetes. Annu Rev Phytopath, 41-60 Kinal H., Park Chung-Mo, Berry J.O., Koltin Y., Bruenn J.A. (1995): Processing and secretion of a virally encoded antifungal toxin in transgenic tobacco plants: Evidence for a Kex2p pathway in plants. Plant Cell 7, 677-688 Koornneef A., Pieterse C.M. (2008): Cross talk in defense signaling. Plant Physiol 146, 839-844 93 Literatur Kyoung S.K., Min J.-Y., Dickman M.B. (2008): Oxalic acid is an elicitor of plant programmed cell death during Sclerotinia sclerotiorum disease development. Mol Plant Mic Int 21, 605-612 Lam E., Pontier D., Del Pozo O. (1999): Die and let live-Programmed cell death in plants. Curr Opin Plant Biol 2, 502-507 Lincoln J.E., Richael C., Overduin B., Smith K., Bostock R., Gilchrist D.G (2002): Expression of the antiapoptotic baculovirus p35-gene in tomato blocks programmed cell-death and provides broad-spectrum resistence to disease. Proc Natl Acad Sci USA 99, 2002, 15217-15221 Liu H., Wang X., Zhang H., Yang Y., Ge X., Song F. (2008): A rice serine carboxypeptidase-like gene OsBISCPL1 is involved in regulation of defense responses against biotic and oxidative stress. Gene 420, 57-65 Liu J.X., Srivastava R., Che P., Howell S.H. (2007): Salt stress responses in Arabidopsis utilize a signal transduction pathway related to endoplasmic reticulum stress signaling. Plant J 51: 897-909. Liu Y., Dammann C., Bhattacharyya M.K. (2000): The matrix metalloproteinase gene GmMMp2 is activated in response to pathogenic infections in soybean. Plant Physiol 127, 1788-1797 Lu H., Rate D.N., Song J.T., Greenberg J.T. (2003): ACD6, a novel ankyrin protein, is a regulator and an effector of Salicylic Acid signaling in the Arabidopsis defense response. Plant Cell 15, 2408-2420 Mach, J.M., Castillo, A.R., Hoogstraten R., Greenberg J.T. (2001): The Arabidopsis-accelerated cell death gene ACD2 encodes red chlorophyll catabolite reductase and suppresses the spread of disease symptoms.Proc of Nat Ac Sci USA 98, 771-776 94 Literatur Mayer A.M., Staples R.C., Gil-ad N.L. (2001): Mechanisms of survival of necrotrophic fungal plant pathogens in hosts expressing the hypersensitive response. Phytochemistry 58, 33-41 Meichtry J., Amrhein N., Schaller A. (1999): Characterization of the subtilase gene family in tomato. Plant Mol Biol 39, 749-760 Mindrinos M., Katagiri F., Yu G.-L., Ausubel F.M. (1994): The A. thaliana disease resistance gene RPS2 encodes a protein containing a nucleotidebinding site and leucine-rich repeats. Cell 78, 1089-1099 Mittler R., Vanderauwera S., Gollery M., Van Breusegem F. (2004): Reactive oxygen gene network of plants. Trends Plant Sci 9, 490-498 Mur L.A.J., Kenton P., Lloyd A.J., Ougham H., Prats E. (2008): The hypersensitive respnse; the centenary is upon us but how much do we know? J Exp Bot 59, 501-520 Osbourn A.E. (1996): Preformed antimicrobial compounds and plant defense against fungal attack. Plant Cell 8, 1821-1831 Park S.-W., Kaimoyo E., Kumar D., Mosher S., Klessig D.F. (2007): Methyl salicylate is a critical mobile signal for plant systemic acquired resistance. Science 318, 113-116 Pearce G., Stydom D., Johnson S., Ryan C.A. (1991): A polypeptide from tomato leaves induces wound-inducible proteinase inhibitor proteins. Science 253, 895-898 Penninckx, I.A.M.A., Eggermont, K., Terras, F.R.G., Thomma, B.P.H.J., De Samblanx, G.W., Buchala, A., Métraux, J.-P., Broekaert, W.F. (1996): Pathogen-induced systemic activation of a plant defensin gene in Arabidopsis follows a salicylic acid-independent pathway. Plant Cell 8, 2309-2323 95 Literatur Pieterse C.M.J., Dicke M. (2007): Plant interactions with microbes and insects: from molecular mechanisms to ecology. Trends Plant Sci 12, 564569 Prell H.H., Day P.R. (2001): Plant-fungal pathogen interaction – a classical and molecular view. Springer-Verlag, Berlin Heidelberg, 1-7 35ff Qutob P.J., Kamoun S., Gijzen M. (2002): Expression of a Phytophthora sojae necrosis-inducing protein occurs during transition form biotrophy to necrotrophy. Plant J 32, 361-373 Raffaele S., Rivas S., Roby D. (2006): An essential role for salicylic acid in AtMYB30-mediated control of the hypersensitive cell death program in Arabidopsis. FEBS Lett 580, 3498-3504 Rate D.N., Cuenca J.V., Bowman G.R., Guttman D.S., Greenberg J.T. (1999): The gain-of-function Arabidopsis acd6 mutant reveals novel regulation and function of the salicylic acid signaling pathway in controlling cell death, defenses, and cell growth. Plant Cell 11, 1695-1708 Rautengarten C., Usadel B., Neumetzler L., Hartmann J., Büssis D., Altmann T. (2008): A subtilisin-like serine protease essential for mucilage release from Arabidopsis seed coats. Plant J 54, 466-480 Richael C., Lincoln J.E., Bostock R.M., Gilchrist D.G. (2001): Caspase inhibitors reduce symptom development and limit bacterial proliferation in susceptible plant tissues. Physiol Mol Plant Pathology, 213-221 Robson C.A., Vanlerberghe G.C. (2002): Transgenic plant cells lacking mitochondrial alternative oxidase have increased susceptibility to mitochondria-dependent and -independent pathways of programmed cell death. Plant Physiol 129, 1908-1920 96 Literatur Kay S., Hahn S., Marois E., Hause G., Bonas U. (2007):A bacterial effector acts as a plant transcription factor and induces a cell size regulator. Science 318, 648-651 Rooney H.C.E., Van't Klooster J.W., Van der Hoorn R.A.L., Joosten M.H.A.J., Jones J.D.G., De Wit P.J.G.M. (2005): Cladosporium Avr2 inhibits tomato Rcr3 protease required for Cf-2-dependent disease resistance. Science 308, 1783-1786 Rossier O., Wengelnik K., Hahn K., Bonas U. (1999): The Xanthomonas hrp type-III system secretes proteins from plant and mammalian bacterial pathogens. Proc Natl Acad Sci USA 96, 9368-9373 Sakai J., Rawson R.B., Espenshade P.J., Cheng D., Seegmiller A.C., Goldstein J.L., Brown M.S. (1998): Molecular identification of the sterolregulated luminal protease that cleaves SREBPs and controls lipid composition of animal cells. Mol Cell 2, 505-514 Sasabe M., Takeuchi K., Kamoun S., Ichinose Y., Govers F., Toyoda K., Shiraishi T., Yamada T. (2000): Independent pathways leading to apoptotic cell death, oxidative burst and defense gene expression in response to elicitin in tobacco cell suspension culture. Eur J Biochem 267, 5005-5013 Schaller A., Oecking C (1999): modulation of plasma membrane H+-ATPase activity differentially activates wound and pathogen defense responses in tomatop. Plant Cell 11, 263-272 Schaller A., Roy P., Amrhein N. (2000): Salicylic acid-independet induction of pathogenesis-relatd gene expression by fusicoccin. Planta 210, 599-606 Schaller A., Ryan C.A. (1994): Identification of a 50-kDa systemin-binding protein in tomato plasma membranes having Kex2p-like properties. Proc Natl Acad Sci USA 91, 11802-11806 97 Literatur Schwessinger B., Zipfel C. (2008): News from the frontline: recent insights into PAMP-triggered immunity in plants. Curr Opin Plant Biol 11, 389-395 Seidah NG, Chrétien M. (1997): Eukaryotic protein processing: endoproteolysis of precursor proteins. Curr Opin Biotechnol 8, 602-607 Seidah N.G., Benjannet S., Wickham L., Marcinkiewicz, J., Bélanger J, S., Stifani S., Basak A., Chrétien M. (2003): The secretory proprotein convertase neural apoptosis-regulated convertase 1 (NARC-1): Liver regeneration and neuronal differentiation. Proc Natl Acad Sci USA 100, 928-933 Shulaev, V., Silverman, P., Raskin, I. (1997): Airborne signalling by methyl salicylate in plant pathogen resistance. Nature 385, 718-721 Siezen R.J., Leunissen J. (1997) Subtilases: the superfamily of subtilisin-like serine proteases. Protein Science 6, 501-523 Smart C.D., Myers K.L., Restrepo S., Martin G.B., Fry W.E. (2003): Partial resistance of tomato to Phytophthora infestans is not dependent upon ethylene, jasmonic acid, or salicylic acid signaling pathways. Mol Plant Mic Int, 141-148 Szatmari A., Ott P.G., Varga G.J., Besenyei, E., Czelleng A., Klement Z., Bozsó Z. (2006): Characterisation of basal resistance (BR) by expression patterns of newly isolated representative genes in tobacco. Plant Cell Reports 25, 728-740 Tanaka H., Onouchi H., Kondo M., Hara-Nishimura I., Nishimura M., Machida C., Machida Y. (2001): A subtilisin-like serine protease is required for epidermal surface formation in Arabidopsis embryos and juvenile plants. Development 128, 4681-4689 98 Literatur Taylor A.A., Horsch A., Rzepczyk A., Hasenkampf C.A., Riggs C.D. (1997): Maturation and secretion of a serine proteinase is associated with events of late microsporogenesis. Plant J 12, 1261-1271 Thaler J.S., Owen B., Higgins V.J. (2004): The role of the jasmonate response in plant susceptibility to diverse pathogens with a range of lifestyles. Plant Physiol 135, 530-538 Tian M., Benedetti B., Kamoun S. (2005): A second Kazal-like protease inhibitor from Phytophthora infestans inhibits and interacts with the apoplastic pathogenesis-related protease P69B of tomato. Plant Physiol 138, 1785-1793 Tian M., Huitema E., Da Cunha L., Torto-Alalibo T., Kamoun S. (2004): A Kazal-like extracellular serine protease inhibitor from Phytophtora infestans targets the tomato pathogenesis-related protease P69B. J Biol Chem 279, 26370-26377 Tian M., Huitema E., Da Cunha L., Torto-Alalibo T., Kamoun S. (2004): A Kazal-like extracellular serine protease inhibitor from Phytophthora infestans targets the tomato pathogenesis-related protease P69B. J Biol Chem 279, 26370-26377 Tian M., Kamoun S. (2005): A disulfide bridge Kazal domain from Phytophthora exhibits stable inhibitory activity against serine proteases of the subtilisin family. BMC Biochem 6 Tian M., Win J., Song J., Van Der Hoorn R., Van Der Knaap E., Kamoun S. (2007): A Phytophthora infestans cystatin-like protein targets a novel tomato papain-like apoplastic protease. Plant Physiol 143, 364-377 Tornero P., Conejero V., Vera P. (1996): Primary structure and expression of a pathogen-induced protease (PR-P69) in tomato plants: Similarity of functional domains to subtilisin-like endoproteases. Proc Natl Acad Sci USA 93, 6332-6337 99 Literatur Tornero P., Conejero V., Vera P. (1997): Identification of a new pathogeninduced member of the subtilisin-like processing protease family from plants. J Biol Chem 272, 14412-14419 Tornero P., Gadea J., Conejero V., Vera P. (1997): Two PR-1 genes from tomato are differentially regulated and reveal a novel mode of expression for a pathogenesis-related gene during the hypersensitive response and development. Mol Plant Mic Int 10, 624-634 Tornero P., Mayda E., Gómez M.D., Cañas L., Conejero V., Vera P. (1996): Characterization of LRP, a leucine-rich repeat (LRR) protein from tomato plants that is processed during pathogenesis. Plant J 10,315-330 Torres M.A., Dangl J.L., Jones J.D. (2002): Arabidopsis gp91phox homologues AtrbohD and AtrbohF are required for accumulation of reactive oxygen intermediates in the plant defense response. Proc Natl Acad Sci USA 99, 517-522 Truman W., De Zabala M.T., Grant M. (2006): Type III effectors orchestrate a complex interplay between transcriptional networks to modify basal defence responses during pathogenesis and resistance. Plant J 46, 14-33 Van der Hoorn R.A.L., Jones J.D. (2004): The plant proteolytic machinery and its role in defence. Curr Op Plant Biol 7, 400-407 Van Esse H.P., van't Klooster J.W., Bolton M.D., Yadeta K.A., Van Baarlen P., Boeren S., Vervoort J., de Wit P.J.G.M., Thomma B.P.H.J. (2008): The Cladosporium fulvum virulence protein Avr2 inhibits host proteases require for basal defense. Plant Cell Van Loon L.C., Rep M., Pieterse C.M.J. (2006): Significance of inducible defense-related proteins in infected plants. Ann Rev Phytopath 44, 135-162 100 Literatur Von Groll U., Berger D., Altmann T. (2002): The subtilisin-like serine protease SDD1 mediates cell-to-cell signaling during Arabidopsis stomatal development. Plant Cell 14, 1527-1539 Van Wees S.C.M., De Swart E.A.M., Van Pelt J.A., Van Loon L.C., Pieterse C.M.J. (2000): Enhancement of induced disease resistance by simultaneous activation of salicylate- and jasmonate-dependent defense pathways in Arabidopsis thaliana. Proc Natl Acad Sci USA 97, 8711-8716 Xia, Y. (2004): Proteases in pathogenesis and plant defence. Cell Microb 6, 905-913 Yamagata H., Masuzawa T., Nagaoka Y., Ohnishi T., Iwasaki T. (1994): Cucumisin, a serine protease from melon fruits, shares structural homology with subtilisin and is generated from a large precursor. J of Biol Chem 269, 32725-32731 Yang H., Yang S., Li Y., Hua J. (2007): The arabidopsis BAP1 and BAP2 genes are general inhibitors of programmed cell death. Plant Phys 145. 135146 Yano A., Suzuki K., Shinshi H. (1999): A signaling pathway, independent of the oxidative burst, that leads to hypersensitive cell death in cultured tobacco cells includes a serine protease. Plant J 18, 105-109 101 Eidesstattliche Erklärung Ich versichere hiermit, dass ich die vorliegende Diplomarbeit selbständig angefertigt, keine anderen als die angegebenen Hilfsmittel benutzt und sowohl wörtliche, als auch sinngemäß entlehnte Stellen als solche kenntlich gemacht habe. Diese Arbeit wurde in gleicher oder ähnlicher Form noch keiner anderen Prüfungsbehörde vorgelegt und auch nicht veröffentlicht. Hohenheim, ____ September 2008 Monika Neugebauer XII Danksagung Danke an Herrn Prof. Dr. Schaller sowohl für die Überlassung dieses interessanten Themas als auch für die vorbehaltslose Unterstützung und die Freiheit, die er mir bei der Bearbeitung der Fragestellung gelassen hat. Frau Dr. Anja Cedzich danke ich neben der hervorragenden wissenschaftlichen Hilfe und Anleitung für die gute Zusammenarbeit, ihrem Glauben an mein Fortkommen und Ihrem Vertrauen in mich und meine Arbeit. Herrn Dr. Andreas Walz vom Fachbereich für Phytopathologie des Instituts für Phytomedizin danke ich für die Zweitkorrektur. Sein ernstes, aufrichtiges Interesse an dem Thema, sein Engagement und sein Vertrauen in meine Fähigkeiten haben mein Vorankommen wesentlich beeinflusst. Ohne seine wissenschaftliche Hilfe wären viele Fragen der PathogenWirtspflanze-Beziehungen offen geblieben. Mathias und Benni vom Reitscheuerflügel danke ich für die Tipps und kleinen ungezählten Hilfen im Laboralltag, dem Team vom Gewächshaus für die Hilfe bei der Anzucht der Tomaten und dem ganzen Institut dafür mich bei Laune zu gehalten zu haben. Danke an Sebastian. Danke an Sabrina. Zum Schluss danke ich meinen Eltern, die mir das Studium in dieser Form möglich gemacht haben. XIII