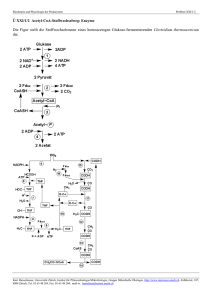
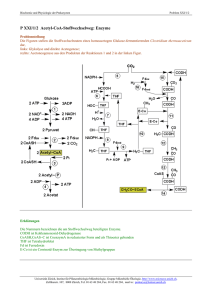
The malonate decarboxylase enzyme system of - ETH E
Werbung

Diss. ETH No. 10766 The malonate decarboxylase enzyme system of Malonomonas rubrai identification, purification and biochemical characterization of components A dissertation submitted to the SWISS FEDERAL INSTITUTE OF TECHNOLOGY ZÜRICH for the degreeof DOCTOROF NATURAL SCffiNCES presentedby HUBERTFRANZPlUS HILBI Dip!. Natw. ETH bom May 30, 1965 citizenofZug (ZG) and Floms (SO) accepted on the recommendation of Prof. Dr. P. Dimroth, examiner Prof. Dr. T. Leisinger, coexaminer Zürich 1994 1 ZUSAMMENFASSUNG Malonomonas rubra, ein mikroaerotolerantes, strikt Na"-abhängiges Faulschlamm- Bakterium, wächst anaerob auf Malonat als einziger Kohlenstoff- und Energiequelle. Malonat Decarboxylase, das Schlüsselenzym dieser Fermentation, setzt dabei das Substrat quantitativ zu Acetat und CO2 um. Die Energieausbeute dieser Reaktion beträgt nur -17.4 kJ pro Mol, und daher muss die ATP-Synthese über chemiosmotische Prozesse erfolgen. Die Decarboxylierung von Malonat stellt ein chemisches Problem dar, weil die C-C-Bindung für die Spaltung aktiviert werden muss. Ziel der vorliegenden Arbeit war die Aufklärung der Malonat-Aktivierung und die biochemische Charakterisierung der Malonat-Decarboxylase. Zellfreier Extrakt von M. rubra decarboxyliert Malonat mit einer beträchtlichen Aktivität von 2.7 U/mg Protein. Zugabe von ATP und Acetyl-CoA zeigt keine Wirkung in frisch präpariertem Extrakt Da Malonyl-CoA zehnmallangsamer als freies Malonat umgesetzt wird, ist letzteres offensichtlich das Substrat Indizien für einen Radikal-Mechanismus sind keine gefunden worden. Stattdessen wird Malonat Decarboxylase durch eine Acetylierung aktiviert, ein Mechanismus, der für Citrat Lyase bekannt war. Aktives Enzym wird vollständig gehemmt durch desacetylierende Reagentien wie Hydroxylamin, Mercaptoethanol oder Thiocyanat Diese Inhibition ist sowohl enzymatisch (mittels einer spezifischen Ligase und ATP/Acetat) als auch chemisch (mit Acetanhydrid) reversibel Dithioerythritol erhöht die reaktivierbare Aktivität, was auf die Beteiligung eines Thiols schliessenlässt Im Verlauf der Katalyse wird der Enzym-gebundene Acetyl-Rest durch einen Malonyl-Rest ersetzt Das aktivierte Substrat der Malonat Decarboxylase ist daher ein Protein-gebundener Thioester der Malonsäure, nämlich Malonyl-Thio-Acyl-Carrier-Protein (Malonyl-SACP). Acetyl-S-Acyl Carrier Protein: Malonat Acyl Carrier Protein-SH Transferase katalysiert die eigentliche Aktivierung von Malonat. Diese Transferase setzt als nichtphysiologische Substrate auch die entsprechenden Coenzym A (CoA)-Derivate um. Mit Malonyl-CoA und Acetat als Alternativ-Substraten ist eine lösliche CoA Transferase gereinigt worden, deren Menge 4 % des Proteins im Extrakt beträgt und die an der Malonat Decarboxylierung beteiligt ist Das monomere Enzym besitzt ein apparentes Molekulargewicht von 67'000 und ein pH-Optimum von 5.5. Die KmWerte für die CoA Substrate sind 1.9 mM (Malonyl-CoA) und 6.9 mM (Acetyl-CoA) und damit etwa zwei Grössenordnungen grösser als diejenigen von physiologischen CoA Transferasen. Der katalytische Mechanismus läuft nicht über ein kovalentes Transferase-CoA Intermediat, das für physiologische CoA Transferasen nachgewiesen 2 ist. Der Umsatz von CoA Derivativen ohne Beteiligung eines kovalenten Enzym-CoA Intermediates ist ebenfalls von der Citrate Lyase bekannt Das Malonat Decarboxylase System enthält neben cytoplasmatischen auch Membran-gebundene Komponenten. Ausserdem ist Biotin als Cofaktor beteiligt, und die Umsetzung von Malonat wird im Extrakt spezifisch durch Na" (Km = 0.8 mM) oder U+ (Km = 3.3 mM) stimuliert Diese Eigenschaften sind typisch für NarTransport Decarboxylasen, die die Decarboxylierungs-Energie zum Aufbau eines elektrochemischen Na"-Gradienten nutzen. Beim Wachstum von M. rubra auf Malonat wird ein einziges Biotin Protein von ungewöhnlicher Grösse (120 kD) exprimiert Aufgrund biochemischer Analysen (Inhibition durch Avidin, Western Blots) als auch mittels Elektronen-Mikroskopie ist dieses Protein im Cytoplasma lokalisiert worden. Das Malonat Decarboxylase Enzym System von Malonomonas rubra ist bezüglich der Substrat-Aktivierung (Acetylierung, ACP-Thiol Transfer) mit der Citrat Lyase verwandt, und es gleicht den Na+-Transport Decarboxylasen bezüglich der SubstratDecarboxylierung (Carboxyltransfer auf Biotin, Decarboxylierung des CÜ2-Biotin) und der Energiekonservierung. 3 SUMMARY Malonomonas rubra, a microaerotolerant, strictly Na"-dependent bacterium isolated from anoxic sediments grows anaerobically on malonate as sole source of carbon and energy. Malonate decarboxylase, the key enzyme of this fermentation pathway, decarboxylates the substrate quantitatively to acetate and C02. This reaction yields only -17.4 kJ per mol and consequently, the ATP synthesis mandatorily involves chemiosmotic processes. The decarboxylation of malonate imposes a chemical problem, since the C-C-bond has to be activated for cleavage. Aim of the work presented here was the elucidation of malonate activation and the biochemical characterization of malonate decarboxylase. Cell free extracts of M. rubra decarboxylate malonate with the considerable activity of 2.7 U/mg protein, Addition of ATP and acetyl-CoA has no effect on freshly prepared extracts. Since malonyl-CoA is decarboxylated ten times slower than free malonate, the latter is apparently the substrate. No evidences for a radical mechanism have been found. Instead. malonate decarboxylase is activated by an acetylation reaction, a mechanism which is also known for citrate lyase. Active enzyme is completely inhibited by deacetylating reagents such as hydroxylamine, mercaptoethanol or thiocyanate. This inhibition is enzymatically (involving a specific ligase and ATP/acetate) and chemically (with acetic anhydride) reversible. Dithioerythritol increases the reactivatable acitvity, which implicates the participation of a thiol residue. During catalysis the enzyme-bound acetyl-residue is exchanged for a malonyl-residue. The activated substrate of malonate decarboxylase is, therefore, a protein-bound thioester of malonate, i.e. malonyl-thio-acyl carrier prorein (malonyl-S-ACP). Acetyl-S-acyl carrier protein: malonate acylcarrier protein-SH transferase catalyzes the activation of malonate. This transferase also accepts the respective coenzyme A (CoA) derivatives as nonphysiological substrates. With malonyl-CoA and acetäte as alternative substrates a soluble transferase has been purified, which amounts to 4 % of the total prorein in cell free extracts and which participates in the decarboxylation of malonate. The monomeric enzyme has an apparent molecular weight of 67'000 and a pH optimum of 5.5. The Km-values for the CoA substrates are 1.9 mM (malonyl-CoA) and 6.9 mM (acetyl-CoA), respectively, and thus about two orders of magnitude higher than those of physiological CoA transferases. The catalytic mechanism does not involve the formation of a covalent transferase-CoA intermediate. which has been demonstrated for the physiological transferases. CoA derivatives as alternative substrates and a mechanism of CoA transfer which does not involve the participation of a covalent enzyme-CoA intermediate are also knownfor citrate lyase. 4 The malonate decarboxylase enzyme system contains cytoplasmic and membranebound components. Biotin is involved as cofactor, and in cell free extracts the decarboxylation ofmalonate is specifically stimulated by Na"- (Km =0.8 mM) or li+ions (Km = 3.3 mM). These features are typical for Naf-transport decarboxylases which use the decarboxylation energy to build up an electrochemical Nat-gradient, Upon growth of M. rubra on malonate only one biotin protein is expressed, with the unusually high molecular mass of 120 kD. This protein has been shown to be located in the cytoplasm by means of biochemical techniques (inhibition with avidin, Western blot) and by means of electron microscopy. The malonate decarboxylase enzyme system of Malonomonas rubra is related to citrate lyase with respect to substrate activation (acetylation, ACP-thiol transfer) and similar to the Nar-transport decarboxylases with regard to the decarboxylation of the substrate (carboxyltransfer to biotin, decarboxylation of a COrbiotin) and the energy conservation strategy.