Biologische neuronale Netze: Computer von morgen?
Werbung

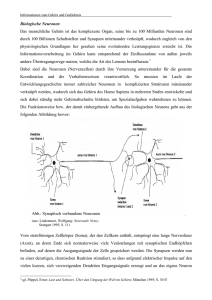
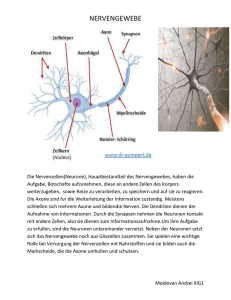
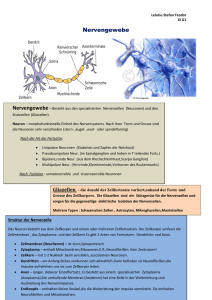
Biologische neuronale Netze: Computer von morgen? Axel Blau The Italian Institute of Technology, Department of Neuroscience and Brain Technologies, via Morego 30, 16163 Genua, Italien, [email protected], www.iit.it Zusammenfassung Das Gehirn versteht es, eine Vielzahl komplexer Aufgaben zu bewältigen. Es wertet sensorische Informationen aus, verbindet kausale Zusammenhänge zwischen einem Stimulus und dessen Relevanz und passt das Verhalten einer gegebenen Situation entsprechend an. In diesem Beitrag wird ein auf in-vitro Mikroelektrodentechnologien basierender ‚bottom-up‘ Ansatz zur Entwicklung eines hybriden Computersystems aus Neuronen und konventioneller Computertechnik skizziert, um das Rechenpotenzial biologischer neuronaler Netzwerke (BNNs) technologisch zu erschließen. Mithilfe eines Kokultursystems aus untereinander vernetzten lichtsensitiven retinalen und kortikalen Neuronen lassen sich grundlegende neuronale Informationsverarbeitungsprozesse und stimulusinduzierte funktionelle wie auch morphologische Änderungen sowohl elektrophysiologisch als auch mikroskopisch untersuchen. In Plastizitätsstudien werden Signalausbreitung in neuronalen Netzwerken und dem Lernen zugrunde liegende Phänomene wie Langzeitpotenzierung und –depression zugänglich. Das vorgestellte System zur lichtinduzierten Programmierung neuronaler Netzwerke zielt langfristig darauf ab, sowohl Beiträge zur neurobiologischen Grundlagenforschung zu leisten, als auch praktisch in der Objekterkennung sowie Roboternavigation Anwendung zu finden. Einleitung: Vergleich zwischen künstlichen computerbasierten und biologischen neuronalen Netzen und Erörterung deren Unterschiede Viele Leistungen des Gehirns lassen sich mit heutigen Computersystemen noch nicht oder unvollständig nachbilden. Dies gilt insbesondere für die synchronisierte parallele Datenverarbeitung, die assoziative Abstraktion in der Objekterkennung und –Kategorisierung und letztlich für Phänomene wie Bewusstsein, Intelligenz und Kreativität. Seit Jahren wird daher versucht, mittels verschiedener Rechnerarchitekturen und künstlicher neuronaler Netze (KNNs) die Fähigkeiten des Gehirns zu simulieren. Eine Übersetzung neuronaler Prozesse und deren Abbildung in einem Computermodell ist allerdings aufgrund grundsätzlicher Unterschiede zwischen der Funktionsweise biologischer Komponenten und Computertechnologien mit Schwierigkeiten behaftet, wie im Folgenden dargelegt wird. Ein derzeit durchschnittlicher Computerprozessor kann 6·109 Fließkommaoperationen pro Sekunde (FLOPS) seriell verarbeiten. Sein Gesamtvolumen beträgt etwa 100 mL, seine Leistungsaufnahme etwas über 100 W. Zu den heute leistungsfähigsten Computern gehört der im Jahr 2005 von IBM am Lawrence Livermore National Laboratory in Betrieb genommene Parallelcomputer ‚Blue Gene/L‘ mit 131.072 Prozessoren, die zusammen 281·1012 FLOPS durchführen können. Seine Leistungsaufnahme beträgt 1,5 Megawatt, sein Stellvolumen über 500 m3. Da Neuronen keine einfachen Ein-Ausschalter oder Transistoren sind, wie bei den ersten Ansätzen künstlicher neuronaler Netze häufig angenommen wurde, sondern an sich schon komplexe Recheneinheiten mit variablen Ausgangssignalsequenzen, lässt sich in Analogie zur Computertechnologie sagen, dass das Gehirn über 100 Milliarden Prozessoren verfügt, die über schätzungsweise 1015 Verknüpfungen hochparallel untereinander verschaltet sind. Das Gehirn kann unterschiedlichen Betrachtungen zufolge zwischen 100·1012 und 10.000·1012 FLOPS durchführen. Diese Rechenleistung übertrifft also das derzeit weltweit leistungsstärkste Computersystem um bis zu zwei Größenordnungen. Die Leistungsaufnahme beträgt dabei ca. 20 W, das Gesamtrechnervolumen ca. 1,4 Liter. Reichte es angesichts der nicht mehr allzu großen Diskrepanz der Rechenleistung zwischen Parallelcomputern und dem Gehirn nicht, einfach auf die nächste Generation bewährter Computertechnologie oder gar auf Quantencomputer zu warten, die sich mit den Leistungen des Gehirns messen könnten? Nach Ray Kurzweil wird doch im Jahre 2020 die Rechenleistung des Gehirns für etwa 1000 Dollar zu haben sein [8]. Möglicherweise führt jedoch die alleinige Betrachtung der Rechenleistung zu einer falschen Schlussfolgerung, bezieht sie sich doch lediglich auf die Zahl der Rechenoperationen pro Sekunde, lässt allerdings offen, welche Rechnerarchitektur zum Einsatz kommt. Bei genauer Betrachtung lassen sich nämlich grundsätzliche Unterschiede zwischen konventionellen Computersystemen mit einer ‚Von-Neumann-Architektur‘ und einem Gehirn identifizieren. Zunächst verarbeiten die meisten Computer Informationen seriell, das Gehirn dagegen hochgradig parallel. Doch selbst Rechnersysteme mit parallel verschalteten Prozessoren unterscheiden sich noch erheblich von biologischen neuronalen Netzwerken. Zum einen kodieren Neuronen Informationen nicht allein mittels eines Orts-Zeit-gekoppelten (Frequenz-) Codes, Abfolgen von ‚digitalen‘ Aktionspotenzialen, sondern nutzen darüberhinaus auch analoge Signalverarbeitungsmechanismen. Letztere werden nicht allein über ein weites Typen- und Konzentrationsspektrum an Neurotransmittern am synaptischen Spalt ermöglicht, sondern schließen allgemein jegliche Art neuromodulatorischer Einflüsse sowie Fluktuationen im Zellmetabolismus ein. Zudem können im Gehirn Prozessor und Speicher als eine Einheit betrachtet werden. Neuronale Aktivität vermag also nicht allein Informationsflüsse zu steuern, sondern kann gleichzeitig mit einer Modifikation von Speicherinhalten einhergehen. Diese Form der ‚Plastizität‘ natürlicher neuronaler Netze gestattet es, sowohl die ‚Hardware‘ als auch die ‚Software‘, die in diesem Fall miteinander verschränkt sind, neu zu organisieren, sei es durch stimulusinduzierte Neuknüpfung oder Umorganisation synaptischer Verbindungen, die Neugewichtung bestehender Verknüpfungen nach dem Hebb’schen Postulat [5] oder die Änderung zellulärer Biochemie hinunter bis zur Modulation der Genexpression. Demnach wird sich die Informationsverarbeitungsleistung eines Gehirns nicht allein über die Summe seiner Einzelelemente und die Zahl der theoretisch gleichzeitig durchführbaren Rechenoperationen zum Ausdruck bringen lassen, wie es der eingangs zitierte Vergleich zunächst plausibel erscheinen ließ. Zudem ist die Funktionalität und der Speicher des Gehirns außerordentlich fehlertolerant und robust gegenüber Störeinflüssen, seien sie chemischer oder elektromagnetischer Natur. Selbst bei gravierenden Eingriffen in die Gehirnorganisation, z.B. bei Unfällen, ist das Gehirn häufig in der Lage, sich derart umzustrukturieren, dass nach einiger Zeit die durch den Eingriff abhanden gekommene Funktionalität wieder verfügbar ist. Experimenteller Ansatz zum Verständnis grundlegender neuronaler Prozesse und deren Verwertung in der Mustererkennung und Roboternavigation In den vergangenen hundert Jahren ist es der Neurowissenschaft gelungen, eine Vielzahl grundlegender Funktionsabläufe im Gehirn zu verstehen. Sie bedient sich vornehmlich zweier Herangehensweisen, dem deduktiven ‚top-down‘-Ansatz, ausgehend vom Gehirn als Ganzem, und dem induktiven ‚bottom-up‘-Ansatz mithilfe von Gehirnschnittpräparaten oder einzelnen in Zellkultur gehaltenen Neuronen. In beiden Fällen wird meistens eine ‚wenn-dann‘-Analyse durchgeführt. Ein Stimulus-Eingangssignal A wird zu einer Antwort A‘ verarbeitet, die direkt messbar oder ersichtlich ist. Im ‚top-down‘-Ansatz lässt sich dank der Molekularbiologie und Genetik anhand von Mutanten klären, welche funktionellen Änderungen einer Systemmodifikation folgen. Dennoch bedarf es, bedingt durch die außerordentliche Verschaltungskomplexität des Gehirns, eines langen Atems, den Details funktionaler Zusammenhänge auf die Spur zu kommen. Dies soll anhand eines Gedankenexperiments veranschaulicht werden, das von Oscar DeFeo, derzeit am University College Cork, vorgeschlagen wurde: Nehmen Sie an, sie hätten noch nie einen Computer gesehen und wüssten demnach nicht, welche Technologie es ermöglicht, den Zeiger auf dem Bildschirm zu bewegen, wenn Sie die Computermaus verschieben. Um es herauszufinden, beginnen Sie also mittels eines deduktiven Ansatzes, den Computer aufzuschrauben und nach dem zu Grunde liegenden Ursache-Wirkungs-Prinzip zu suchen. Sie begreifen irgendwann, dass die kleinste funktionelle Einheit und Kerntechnologie der Computertechnik der Transistor ist, dessen EingangsAusgangs-Verhalten sich genau beschreiben lässt. Mit Ihrem Oszilloskop testen sie, an welchen Leiterbahnen welche Spannungsmuster auftreten, wenn Sie die Maus bewegen. Diese Herangehensweise ist nicht ohne Tücken. Sie könnten z.B. geneigt sein, ein spezielles Transistorelement als den ‚Maustransistor‘ zu identifizieren, da er immer aktiviert wird, sobald Sie die Maus bewegen. In der Neurobiologie entspräche diese Interpretation dem veralteten Modell des ‚Großmutterneurons‘, das immer dann besonders aktiv wird, wenn die Großmutter gesehen wird. Aufgrund der Überlagerung von Prozessen ist es jedoch nicht trivial, das in der Software abgebildete Modell ‚Verschiebe Mauszeiger auf Bildschirm bei Mausbewegung‘, das auf das Vorhandensein einer ganz speziellen Hardwarearchitektur angewiesen ist, zu verstehen. Die Erkenntnis, dass Transistoren dabei eine Schlüsselrolle spielen, ist dabei nur eine notwendige, nicht jedoch hinreichende Voraussetzung zur Erlangung eines vollständigen Verständnisses der Komplexität computerinterner Verarbeitung einer einfachen Mausbewegung. Im ‚bottom-up‘-Ansatz wird lediglich eine ‚überschaubare‘ Teilmenge untereinander vernetzter Neuronen bezüglich ihres Stimulus-Antwort-Verhaltens in Abhängigkeit ihrer Vernetzungsarchitektur untersucht. Er stützt sich auf das seit knapp einem Jahrhundert bekannte Phänomen, dass sich Neuronen in Zellkultur zu ‚funktionalen‘ Netzwerken selbstorganisieren [6]. In einer Reihe von Studien wurde bereits gezeigt, dass neuronale Netzwerke in Zellkultur ähnliche bis identische Eigenschaften zu den Netzwerken im Gehirn zeigen. Sie reagieren mit demselben Antwortverhalten auf externe Stimuli, wobei Mechanismen wie Langzeitpotenzierung und –depression (LTP, LTD) wiederzufinden sind, die im Zusammenhang mit Lernprozessen diskutiert werden [11,12,15,17]. Darüberhinaus konnte bereits eine dem Netzwerk eingeprägte Stimulusdiskriminierung demonstriert werden, die sich als Nachweis eines rudimentären Einprägungs- bzw. Lernvorgangs interpretieren lässt [10]. Wäre es daher denkbar, in einem biohybriden Ansatz unter Zuhilfenahme sich selbst organisierender biologischer neuronaler Netze bestimmte Leistungen des Gehirns viel effektiver zu imitieren, als es digitale Computer vermögen? Die Idee, biologische neuronale Netzwerke als Objekterkennungs- und Navigationsprozessoren einzusetzen, stützt sich auf die Beobachtung, dass selbst kleine Tiere wie Insekten, Vögel und Nagetiere mit relativ kleinen Gehirnen Navigationsaufgaben exzellent lösen. So kann sich der in der Forschung beliebte Wurm C. elegans mit seinen genau 302 Neuronen als Antwort auf endogene wie exogene Reize bereits räumlich orientieren. Fruchtfliegen mit einem Gehirn der Größe eines Sandkorns (ca. 100.000 Neuronen) sind in der Lage, im dreidimensionalen Raum zu navigieren [16]. Bienen (ca. 850.000 Neuronen) sowie Wirbeltiere wie Vögel (ca. 50 Millionen Neuronen) und Nagetiere wie Mäuse und Ratten (ca. 100 Millionen Neuronen) zeigen darüberhinaus ein komplexes Sozialverhalten [4]. Obwohl nicht zu erwarten ist, dass neuronale Netze in absehbarer Zeit zu Jetpiloten werden [9], ist davon auszugehen, dass sie sich zur Lösung von Mustererkennungs- (Abb. 1a) und Navigationsaufgaben einsetzen lassen. b) a) Abb. 1 a) Nutzung biologischer neuronaler Netze in der Objekterkennung. Die Arbeitshypothese, dass einfache Netzwerke in der Lage sind, Objekte zu erkennen, in einen Kontext einzuordnen und dies mit charakteristischen Ausgangssignalen zu ‚bestätigen‘, stützt sich auf die in der Natur beobachtbaren Fähigkeiten selbst einfacher Gehirne. Die Ausgangsignale ließen sich in einem nächsten Schritt z.B. zur Steuerung von Robotern nutzen. b) Schematischer Aufbau eines rückgekoppelten Systems zur fokussierten Lichtstimulation photorezeptiver Neuronen in Kokultur mit einem informationsverarbeitenden Netzwerk aus nicht-lichtrezeptiven kortikalen Neuronen. Sowohl einfache Lichtreize unterschiedlicher Farbe und Intensität als auch detailreichere Bilder oder Videosequenzen lassen sich auf die Zellschicht projizieren. Die durch eine Lichtstimulation erzeugte Aktivität im Netzwerk dient nach Voranalyse in Rückkopplungsexperimenten dazu, die folgende Lichtstimulation zu variieren. Wird als Stimulus ein Video der Umgebung angeboten, ist ein Training des Netzwerkes für Navigationsaufgaben eines Roboters denkbar. Wir verfolgen daher nach Braitenberg-Manier [3] einen ‚bottom-up‘-Ansatz, bei dem zerebrale und retinale Neuronen in verschiedenen Anordnungen zueinander auf Mikroelektrodenarrays gemeinsam in Zellkultur genommen werden. Über einen computergesteuerten LCD-Projektor werden die photorezeptiven Neuronen gezielt stimuliert. Ihre Erregung wird synaptisch an die kortikalen Neuronen weitergeleitet. Damit können die photorezeptiven Neuronen als sensorische Eingangskanäle zur kontrollierbaren Stimulation des übrigen Netzwerkes genutzt werden. In einer Rückkopplungsschaltung wird die spontane sowie licht-evozierte Netzwerkaktivität elektrophysiologisch über die Mikroelektroden ausgelesen und nach ihrer Analyse zur Steuerung der folgenden Lichtstimuli oder für motorische Befehle genutzt. Das Prinzip ist schematisch in Abbildung 1b gezeigt. Ein Schwerpunkt der Arbeiten liegt dabei auf der Entwicklung von Enabling Technologies, die die Voraussetzungen für Langzeitstudien und eine spätere industrielle Umsetzungen schaffen. Dazu gehören u.a. eine Mikrofluidik für den automatisierten Nährstoffaustausch und die Applikation von Neuromodulatoren, Technologien zur Etablierung physiologischer Umgebungen (‚künstlicher Körper‘) außerhalb einer LaborInfrastruktur, kostengünstige Transducer (Mikroelektrodenarrays), reproduzierbare Zellkulturen unter Verwendung neuronaler Vorläufer-Zelllinien, Vororganisation neuronaler Vernetzung mithilfe strukturiert deponierter Zelladhäsionsvermittler, sowie Trainings- und Datenanalysealgorithmen zur Übersetzung neuronaler Codes in abstrakte mathematische Modelle und Steuerungsbefehle. Teilergebnisse zu einigen dieser Arbeiten werden in den nachstehend genannten Veröffentlichungen beschrieben [1,2,7,13,14]. Literatur [1] [2] [3] [4] [5] [6] [7] [8] [9] [10] [11] [12] [13] [14] [15] [16] [17] A. Blau, T. Ugniwenko, Induction and analysis of cell adhesion and differentiation on inkjet micropatterned substrates, physica status solidi (c) 4 (2007) 1873–1876. A. Blau, T. Ugniwenko, C. Ziegler, Replica-molded poly(dimethylsloxane) cell culturing lids contribute to cell culture longevity, Proceedings of the 5th International Meeting on Substrate-Integrated Micro Electrode Arrays, July 4-7, 2006, Reutlingen, Germany (2006) 218-210. V. Braitenberg, Vehicles: Experiments in Synthetic Psychology. The MIT Press, 1986. A.G. Dyer, C. Neumeyer, L. Chittka, Honeybee (Apis mellifera) vision can discriminate between and recognise images of human faces, J Exp Biol 208 (2005) 4709-14. B. Godde, R. Leonhardt, S.M. Cords, H.R. Dinse, Plasticity of orientation preference maps in the visual cortex of adult cats, Proc Natl Acad Sci 99 (2002) 6352-7. R.G. Harrison, The outgrowth of the nerve fiber as a mode of protoplasmic movement, J Exp Zool 9 (1910) 787-846. F. Heer, S. Hafizovic, T. Ugniwenko, U. Frey, W. Franks, E. Perriard, J.C. Perriard, A. Blau, C. Ziegler, A. Hierlemann, Single-chip microelectronic system to interface with living cells, Biosensors & bioelectronics 22 (2007) 2546-53. R. Kurzweil, Homo Sapiens. Leben im 21.Jahrhundert. Was bleibt vom Menschen? Kiepenheuer und Witsch, 1999. S. Lovgren, "Brain" in Dish Flies Simulated Fighter Jet, National Geographic News, November 19, 2004. M.E. Ruaro, P. Bonifazi, V. Torre, Toward the neurocomputer: image processing and pattern recognition with neuronal cultures, IEEE transactions on bio-medical engineering 52 (2005) 371-83. G. Shahaf, S. Marom, Learning in networks of cortical neurons, J Neurosci 21 (2001) 8782-8788. T. Tateno, Y. Jimbo, Activity-dependent enhancement in the reliability of correlated spike timings in cultured cortical neurons, Biological Cybernetics 80 (1999) 45-55. T. Ugniwenko, A. Blau, Developmental diversity of intrinsically photosensitive retinal ganglion cells in the chick embryo, (article in preparation) (2007). T. Ugniwenko, C. Ziegler, A. Blau, Multielectrode monitoring of developmental features in the chick retina, 5th Forum of European Neuroscience, July 8-12, Vienna, 2006. D.A. Wagenaar, Z. Nadasdy, S.M. Potter, Persistent dynamic attractors in activity patterns of cultured neuronal networks, Phys Rev E Stat Nonlin Soft Matter Phys 73 (2006) 051907. J. Weiner, Time, Love, Memory - A Great Biologist and His Quest for the Origins of Behavior Vintage 2000. L.I. Zhang, M.M. Poo, Electrical activity and development of neural circuits, Nat Neurosci 4 (2001) 12071214.