Transkription Teil 2
Werbung

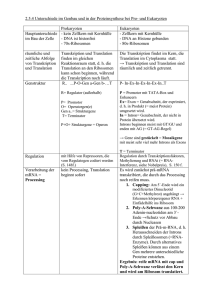
Transkription Teil 2 - Transkription bei Eukaryoten - Inhalte: • Unterschiede in der Transkription von Pro- und Eukaryoten • Die RNA-Polymerasen der Eukaryoten • Cis- und trans-aktive Elemente • Promotoren • Transkriptionsfaktoren • Initiierung der Transkription Das heißt? Wie beginnt im Grunde die Proteinbiosynthese? Sie beginnt an der DNA. Aber welche Enzyme sind notwenig und woher wissen sie, was sie wo zu tun haben? Wer „sagt“ ihnen das? Unterschiede zwischen Pro - und Eukaryoten I: Bei Eukaryoten ist im Gegensatz zu den Prokaryoten die Translation zeitlich und räumlich von der Transkription getrennt. Bei Eukaryoten findet die Transkription im Zellkern und die Translation (später) im Cytoplasma statt. Das führt bei Eukaryoten zu einer besseren Regulation der Genexpression, was wiederum zu einer Vielfalt eukaryotische Formen und Funktionen führt Bei Prokaryoten sind diese beiden Prozesse eng miteinander gekoppelt, so dass die mRNA sogar schon translatiert wird, während sie am anderen Ende erst noch gebildet wird. Unterschiede zwischen Pro - und Eukaryoten II: Die Weiterverarbeitung der mRNA: Ihre tRNA und rRNA verändern beide, doch nur Eukaryoten verändern auch noch ihre mRNA. A. Das Einfügen von Cap- und PolyA-Strukturen dient: • der Stabilisierung und dem Schutz der mRNA vor enzymatischen Abbau. • ist für den Transport durch die Kernmembran und die Anheftung an die Ribosomen notwendig. B. Das alternative Spleißen der Eukaryoten vergrößert das Repertoire an Proteinen und bietet so eine Erklärung, weshalb das Proteom weit aus größer und komplexer ist wie das Genom. 40.000 proteincodierende Gene vs. 100.000 verschiedene Proteine Die RNA-Polymerasen: Ein weiterer Unterschied zwischen Pro- und Eukaryoten ist, dass Eukaryoten drei und Prokaryoten nur eine RNA-Polymerase besitzen. Die drei eukaryotischen RNA-Polymerasen unterscheiden sich in ihrer : • Matrizenspezifität (also in dem was sie produzieren) • Lokalisation im Zellkern • Empfindlichkeit gegenüber Inhibitoren (am Bsp. von α-Amanitin) Die eukaryotischen RNA-Polymerasen: Typ Lokalisation Zelluläre Transskripte Wirkung von α-Amanitin I Nucleolus 18S-, 5,8S-, 28S-rRNA unempfindlich II Nucleoplasma mRNA-Vorläufer + snRNA starke Hemmung III Nucleoplasma tRNA + 5S-rRNA Hemmung erst bei hohen Konz. Das α–Amanitin: Ein zyklisches Oktapeptid, das sich sehr eng an die RNAPolymerasen bindet und dort die Elongation der RNA-Synthese stört. Grüner Knollenblätterpilz (Amanita phalloides) Cis- und trans-aktive Elemente: Eukaryotengene benötigen eben so wie prokaryotische Gene Promotoren, um ihre Transkription starten zu können: • Die RNA-Polymerase I transkribiert nur von Promotoren für rRNA-Genen aus. • Die RNA-Polymerase II hat einfache oder auch komplexere Promotoren, die -wie bei den Prokaryoten- auf dem selben DNAMolekül liegen, auf dem auch das da zugehörige Gen liegt. • Die RNA-Polymerase III reagiert sowohl auf Promotoren, die stromaufwärts vom Gen liegen, als auch auf Promotoren, die stromabwärts von der Initiationsstelle im Gen selbst liegen. Cis-aktive Elemente: Cis-aktive Elemente regulieren Gene und befinden sich auf dem selben DNA-Molekül, wie das zu regulierende Gen! Es gehören dazu: • Promotoren wie z.B. bei der RNA-Polymerase II • Enhancer, die allerdings nur irgendwo auf dem selben DNAMolekül liegen müssen und somit nicht in der Nähe ihres Genes Trans-aktive Elemente: Trans-aktive Elemente regulieren ebenfalls Gene, befinden sich aber nicht auf dem selben DNA-Molekül wie das zu regulierende Gen! Sie werden auch als Transkriptionsfaktoren bezeichnet. Die cis-aktiven Elemente der RNA-Polymerase II: Die Promotoren der RNA-Polymerase II liegen alle auf der ´-Seite (stromaufwärts) des entsprechenden Gens. Essentiell ist die s.g. TATA-Box. Neben ihr gibt es aber auch noch die CAAT- und die GC-Box. 5 Die TATA-Box: • Wird so wegen ihrer typischen Consensussequenz genannt. • Liegt i.d.R. –30 bis –100 Basenpaare vor ihrem Gen. • Schon die Mutation einer einzigen Base der TATA-Box beeinträchtigt ihre Promotoraktivität beträchtlich. Entscheidend ist nicht der hohe AT-Gehalt, sondern die genaue Sequenz. Die GC- und CAAT-Box: Die TATA-Box ist für die Transkription zwar notwendig, reicht meistens aber nicht aus. Zusätzlich werden eine oder mehrere CAAT- und/oder GC-Boxen benötigt. Diese liegen gewöhnlich –40 bis –150 Basenpaare vor dem Gen. Innerhalb dieses Intervalls ist ihre Lage aber variierbar im Gegensatz zu der –35-Region der Prokaryoten. Ebenso können sie auf dem Matrizenstrang (antisense)liegen, wohin gegen die prokaryotische –35-Region auf dem codierenden Strang (sense) liegen muss. Allerdings werden diese cis-aktiven Element (Promotoren) nicht von der RNA-Polymerase II selbst erkannt, sondern nur von Proteinen, den trans-aktiven Elementen (Transkriptionsfaktoren). Beginn der Transkription: Neben den Promotoren sind die Transkriptionsfaktoren für die Transkription in Eukaryoten notwendig. Man kürzt sie TFII ab. TF wegen Transkriptionsfaktor und II für die RNA-Polymerase II. Die einzelnen Faktoren werden durch nachgestellte Buchstaben unterschieden (TFIIA, TFIIB usw.). Der wichtigste Faktor ist TFIID. Er beinhaltet das TATA-BoxBindeprotein (TBP) und dockt deshalb als erster an die TATA-Box an. Die Bindung des TBP ist äußerst spezifisch an der Sequenz TATA und ebenso stark. Sobald das TFIID mit seinem TBP an die TATA-Box angedockt hat, kommt es durch das TBP zu einer Konformationsänderung in der DNA und die Doppelhelix wird teilweise entwunden. Anschließend docken TFIIA, TFIIB und TFIIF an. Wobei TFIIF eine ATP-abhängige Helikase ist und den DNA-Doppelstrang für die RNAPolymerase II trennt. Es folgen TFIIE und die RNAPolymerase II, um abschließend den basalen Transkriptionskomplex zu bilden. Die durch den basalen Transkriptionskomplex initiierte Transkription findet nur mit relativ geringer Häufigkeit statt. Um eine höhere mRNA-Syntheserate zu erzielen, sind zusätzliche Transkriptionsfaktoren notwendig, die an anderen Stellen auf der DNA binden. Solche stromaufwärts gelegenen, stimulierenden Abschnitte eukaryotischer Gene sind in ihrer Sequenz sehr vielfältig und in ihrer Position variabel. Diese Vielfältigkeit lässt auf viele verschiedene, spezifische Gene schließen. Enhancer: Enhancer sind cis-aktive Elemente, welche selbst keine Promotoraktivität besitzen, aber einen Promotor in seiner Aktivität unterstützen. Sie können über mehrere tausend Basenpaare hinweg wirken und können sowohl stromaufwärts, stromabwärts, als auch mitten in dem zu transkribierenden Gen liegen. Ebenso ist es egal in welcher Orientierung oder auf welchem DNA-Strang sie liegen. Das war‘s. Danke.