02.
Werbung
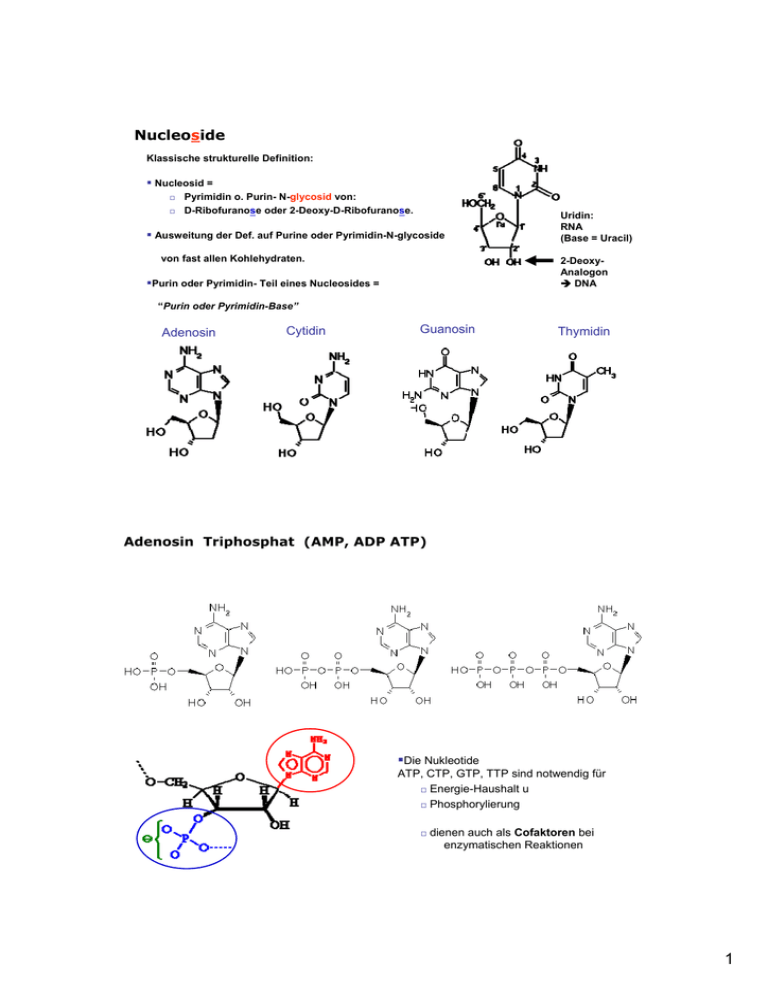
Nucleoside Klassische strukturelle Definition: Nucleosid = Pyrimidin o. Purin- N-glycosid von: D-Ribofuranose oder 2-Deoxy-D-Ribofuranose. Ausweitung der Def. auf Purine oder Pyrimidin-N-glycoside von fast allen Kohlehydraten. Uridin: RNA (Base = Uracil) 2-DeoxyAnalogon Î DNA Purin oder Pyrimidin- Teil eines Nucleosides = “Purin oder Pyrimidin-Base” Adenosin Cytidin Guanosin Thymidin Adenosin Triphosphat (AMP, ADP ATP) Die Nukleotide ATP, CTP, GTP, TTP sind notwendig für Energie-Haushalt u Phosphorylierung dienen auch als Cofaktoren bei enzymatischen Reaktionen 1 Bioenergetik = die Thermodynamik biologischer Stoffwechselprozesse. ΔG beschreibt: ob eine Reaktion Energie freisetzt (Æ exergonisch) oder Energiezufuhr benötigt (Æ endergonisch) Wichtig ist der “Freie Energie Wechsel” (ΔG) wenn ΔG negativ ist -> Spontane Reaktion wenn delta G = 0 ist -> Reaktionsgleichgewicht wenn ΔG positiv ist -> keine spontane Reaktion Hydrolyse des ATP ATP + H2O ADP + HPO42– ΔG°' für die Hydrolyse des ATP zu ADP = –31 kJ/mol ATP: eine“hoch-energetische" Verbindung: im Vergl. zu ADP + HPO42–. Umwandlung von ATP in ADP: Kopplung mit anderen Prozessen Î Lieferung v. Energie Î Umwandlung des endergonischen Prozess Æ exergonischer Prozess 2 Glutaminsre. -> Glutamin O O –OCCH 2CH2CHCO +NH + NH4+ + ATP 3 Reaktion in Verbindung mit der Hydrolyse von ATP: exergonisch ΔG°' = -31 +14 kJ = -17 kJ O – O H2NCCH2CH2CHCO– +NH + ADP + HPO42– 3 Basen-Paarung Watson and Crick: komplementäre Wasserstoffbrückenbindungen H N N A N O H N H CH3 T N 2-Deoxyribose N N 2-Deoxyribose O O N G N 2-Deoxyribose N H H N H C N N N N H H O 2-Deoxyribose 3 Bedeutung und Aufbau der Proteine Æ Aufbau von zell-eigenem Eiweiss – Strukturproteine Æ Aufbau von Enzymen und Hormonen Æ Für die Reproduktion der Zellsubstanz Æ Für Stütz und Schutzfunktion Æ Wasserbindung und Wassertransport Æ Nährstofftransport - Alle Carrier (Ionen-Transport durch die Zellmembran) Æ Energiegewinnung Peptid – Protein - Struktur Primär- Struktur Sekundär- Struktur TeritiärStruktur QuarternärStruktur 4 Struktur der Proteine Primärstruktur: Reihenfolge der Aminosäuren (AS) in einem Protein (= AS-Sequenz) z.B.: ...-Gly-Leu-Tyr-Ala-Pro-His-... Æ Jedes Protein hat eine durch die DNA eindeutig festgelegte AS-Sequenz Bereits geringfügige Abweichungen können zum Verlust d. biolog. Aktivität führen Kombinationen der 20 AS Î Nahezu unbegrenzt hohe Anzahl möglicher Proteine! Physikal-chem. Kräfte zwischen den Kettengliedern bedingen räumliche Anordnung der AS-Kette (Konformation): H+ -Brückenbindung Kovalente Bindung (Disulfid-Brücken) Ionen-Bindung hydrophobe Wechselwirkungen Sekundärstruktur: Raumanordnung eines Proteins: α-Helix oder ß-Faltblattstruktur durch Ausbildung von H-Brückenbindungen zw. Peptidbindungen (R-C=O...H-N-R´) 8 essentielle Aminosäuren Herkömmlich: „Biol. Wertigk.: BW“ Kartoffel: 75 > Soja: 72 > Getreide: 50 > Hülsenfrüchte: 45 Diese 8 AS sind für den Menschen essenziell, d.h. er muss sie über die Nahrung aufnehmen: Æ Valin Æ Isoleucin Æ Methionin Æ Phenylalanin Æ Leucin Æ Threonin Æ Lysin Æ Tryptophan Alternative Methode: “Protein digestibility-corrected amino acid score (PDCAAS)” Protein Quality Evaluation, Report of the Joint FAO/WHO Expert Consultation. Rome: FAO Food and Nutrition Paper No. 51, 1991. 5 Funktionelle Eigenschaften der Proteine Faserproteine Proteine tragen also bei zu: V.a. Strukturelle Aufgaben: wiederholte Strukturelemente Z. B.: Kollagen (α-Helix), Hydratation Löslichkeit Viskosität Seidenfibrin (β-Faltblatt) Gelbildung Texturierung Globuläre Proteine Æ Teig-Stabilität (v.a. Glutene) Æ Schäumung Æ Aroma-Bindung (z.B. Butylamin) Æ Emulgation (z.B. Öl – H2O) Æ Wechselwirkung mit anderen LebensmittelBestandteilen Biol. wirksame Proteine Komplexe Tertiärstrukturen mehrere Arten v Sekundärstrukturen innerhalb derselben Polypeptidkette Aminosäuren Hydratation Löslichkeit Viskosität Gelbildung Texturierung Glycin Alanin Valin Leucin Isoleucin unpolare Seitengruppe Ernährungsphysiologie Methionin Phenylalanin Tryptophan Prolin Serin Threonin Cystein Tyrosin Lysin Arginin Polare, ungeladene Seitengruppe: Seitengruppen: polar unpolar geladen ungeladen basisch sauer Asparagin polare, ungeladene Seitengruppe basische Seitenkette Histidin Glutamin Asparagin Glutamin säure säure saure Seitenkette 6 Zentrales ´Dogma´ der Mol Bio 93 Der genetische Code - Wobble U Erste Position U C A G UUU UUC UUA UUG CUU CUC CUA CUG AUU AUC AUA AUG GUU GUC GUA GUG C Phe Phe Leu Leu Leu Leu Leu Leu Ile Ile Ile Met Val Val Val Val UCU UCC UCA UCG CCU CCC CCA CCG ACU ACC ACA ACG GCU GCC GCA GCG A Ser Ser Ser Ser Pro Pro Pro Pro Thr Thr Thr Thr Ala Ala Ala Ala UAU UAC UAA UAG CAU CAC CAA CAG AAU AAC AAA AAG GAU GAC GAA GAG G Tyr Tyr Stop Stop His His Gln Gln Asn Asn Lys Lys Asp Asp Glu Glu UGU UGC UGA UGG CGU CGC CGA CGG AGU AGC AGA AGG GGU GGC GGA GGG Cys Cys Stop Trp Arg Arg Arg Arg Ser Ser Arg Arg Gly Gly Gly Gly Zweite Position 3. (Wobble) Position 94 7 ProteinBiosynthese: einfacher Überblick Lys 95 Aufnahme- und Enzym-Kinetik beschäftigt sich mit den Geschwindigkeitsgesetzen enzymkatalysierter Reaktionen Enzyme (Î incl. Carrier) Enzyme sind Biokatalysatoren, die die Gleichgewichtslage einer Reaktion beschleunigen, ohne das Gleichgewicht zu verändern. Dies erfolgt durch Erniedrigung der für die Reaktion nötigen Aktivierungsenergie Ea: Ohne Enzym: Mit Enzym: Energie ÜbergangsStadium Reaktanten AktivierungsEnergie AktivierungsEnergie . Produkte 111 8 Biokatalysatoren Reaktionsspezifische Katalysatoren Reaktionsspezifität: die meisten Enzyme katalysieren in der Zelle nur einen einzigen Reaktionstyp Gruppenspezifität: selektiv gegenüber best. chem. Gruppen in ihren Substraten Substratspezifität: Enzym setzt nur ein bestimmtes Zwischenprodukt des Stoffwechsels um, z. B.: Urease spaltet nur Harnstoff = absolute Spezifität Dehydrogenasen, Esterasen: Breite Substrat-Spezifität Î können auch substratanaloge Substanzen metabolisieren Stereospezifität: von optischen Isomeren (D- bzw L-Form) eines Substratmoleküls wird nur 1 Form umgesetzt 113 Cofaktoren . für die Aktivität von Enzymen ist oft Bindung an ein Nicht-ProteinMolekül (Cofaktor) nötig Apoenzym ( inaktiv) + Cofaktor Æ Coenzyme nicht kovalent an Enzym gebunden: Metallionen z.B. NAD+, CoA ATP PALP Mg 2+ Ca 2+ Fe 2+/3+ Zn 2+ Holoenzym (aktiv) Prosthetische Gruppen: kovalent an Enzym gebunden z.B. Häm gruppe Hämoglobin Myoglobin .. MIKRO-NÄHRSTOFFE PALP = Pyridoxalphosphat 115 9 Temperatur RGT Regel ReaktionsgeschwindigkeitsTemperatur-Regel Aktivität Temperatur-Optimum Erhöhung der T um 10 °C pH Optimum Î Verdoppelg. d Reaktionsgeschwindigkeit! T > 50 °C Î Denaturierung, Î Enzymaktivität fällt ab. pH: Enzyme sind nur in einem schmalen pH Korridor aktiv 116 NO3- und NH4+ Transport Reduktion Translokation Akkumulation Schema der Transport-Mechanismen für NO2-, NO3- und NH4+ am Plasmalemma. PM = Plasmalemma P = Protonenpumpe (H+ ATPase, Æ Ladungsausgleich) C = Carrierproteine ___ = Kanäle für pH- und Ladungsausgleich NR = NO3- Reduktase NiR = NO2- Reduktase 124 10