Musterlösung zum 1. Teil der Nachklausur
Werbung
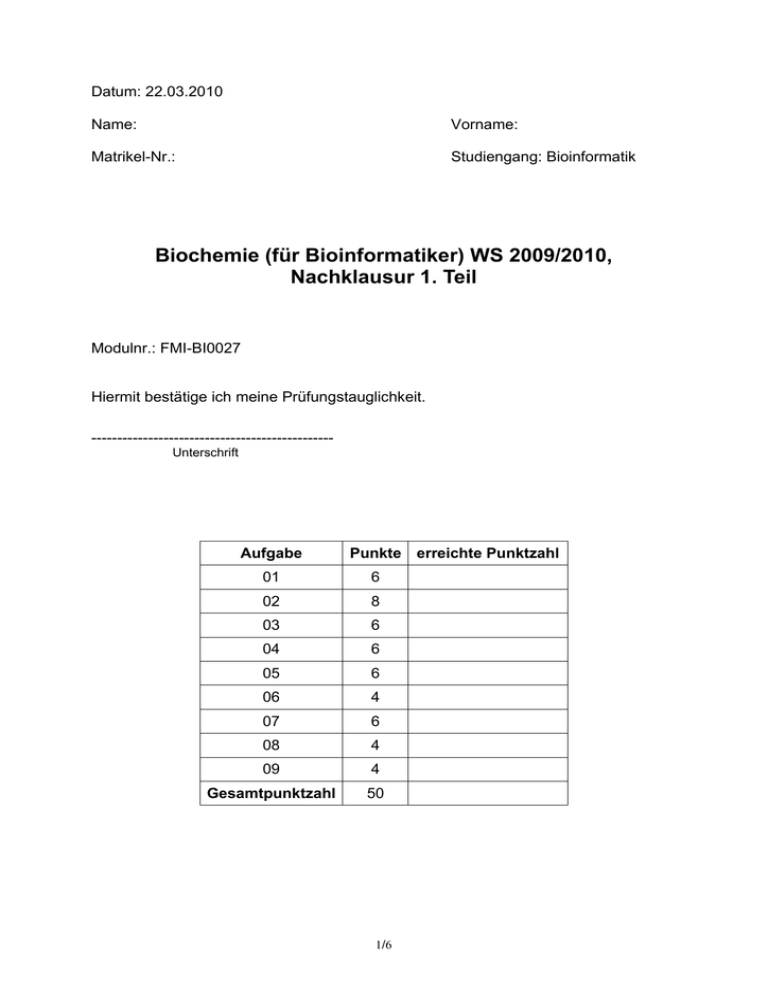
Datum: 22.03.2010 Name: Vorname: Matrikel-Nr.: Studiengang: Bioinformatik Biochemie (für Bioinformatiker) WS 2009/2010, Nachklausur 1. Teil Modulnr.: FMI-BI0027 Hiermit bestätige ich meine Prüfungstauglichkeit. ----------------------------------------------Unterschrift Aufgabe Punkte erreichte Punktzahl 01 6 02 8 03 6 04 6 05 6 06 4 07 6 08 4 09 4 Gesamtpunktzahl 50 1/6 01. (6 Punkte) Nennen und zeichnen Sie jeweils eine Aldohexose und eine Ketohexose. Geben Sie an, welches die Aldohexose und welches die Ketohexose ist. Beispiel Aldohexose: Beispiel Ketohexose: OH O H2C HC C O HO C H OH H C OH OH H C OH H C OH HO C H H C H C H2C H2C OH D-Glucose OH D-Fructose 02. (8 Punkte) Zeichnen Sie ein Dipeptid aus einer Aminosäure mit Ringstruktur und einer sauren Aminosäure. Benennen Sie die Aminosäuren, ordnen Sie die Eigenschaft zu und markieren Sie N- und C-Terminus. Beispiel: CH HC CH HC CH O C + N-Terminus - C CH2 NH3 O H CH2 C C N C H O H Phenylalanin Ringstruktur COO Asparaginsäure saure AS 2/6 - C-Terminus 03. (6 Punkte) Wodurch wird die Richtung (5'-3') eines DNA-Einzelstranges bestimmt? Zeichnen und benennen Sie ein Nukleotid, und geben Sie die Richtung in der Zeichnung an. Benennen Sie die paarende Base. Die Richtung eines DNA-Einzelstranges wird durch das 5’- und das 3’Kohlenstoffatom in der Desoxyribose bestimmt. Beispiel: NH2 N N O O - P O O - N 5' O N 1' 4' 3' 2' OH Desoxyadenosinmonophosphat (dAMP) Die zu Adenin paarende Base ist Thymin. 04. (6 Punkte) Gegeben sei folgende hypothetische genomische Sequenz: 5'-ATGCCAGTGACGTTAAGGCATGTGACAG-3' Übersetzen Sie mit Hilfe der beigefügten Tabelle diese Sequenz in allen möglichen Leserastern. Die Sequenzen müssen dabei nicht mit einem StartCodon beginnen oder mit einem Stop-Codon enden. Aminosäuresequenzen der 6 möglichen Leseraster: 3 entstehen durch die 3 Leseraster des gegebenen Einzelstranges: Met/Start Pro Val Thr Leu Arg His Val Thr Cys Gln Stop Arg Stop Gly Met/Start Stop Gln Ala Ser Asp Val Lys Ala Cys Asp 3 entstehen durch die 3 Leseraster des paarenden Stranges (5'-CTGTCACATGCCTTAACGTCACTGGCAT-3'): Leu Ser His Ala Leu Thr Ser Leu Ala Cys His Met/Start Pro Stop Arg His Trp His Val Thr Cys Leu Asn Val Thr Gly 3/6 05. (6 Punkte) Beschreiben Sie den Vorgang der Transkription. 3 Phasen: • Initiation • Elongation • Termination Prokaryoten: Initiation: • RNA-Polymerase sondiert über σ-Untereinheit die Promotorsequenz und bindet daran • Initiationskomplex windet kurzes Segment des Doppelstrangs von ca. 15 Nucleotiden auf ö offener Promotorkomplex • Abgreifen der Information auf dem Matrizenstrang in 3’ö5’ Richtung und synthetisieren des RNA-Strangs in 5’ö3’-Richtung • RNA-Strang entspricht Gegenstrang (codierender Strang) bis auf, dass T gegen U ausgetauscht ist • σ-Untereinheit wird freigesetzt, wenn der naszierende RNA-Strang eine Länge von zwölf Nucleotiden überschreitet • Hinter einem Stück eines „gemischten Stranges“ mit der Matrizen-DNA, wird der synthetisierte RNA-Strang vom nichttranskribierten Strang verdrängt. Dort bildet sich wieder ein DNA-Doppelstrang aus und der RNA-Strang verlässt den Komplex. Elongation: • Stetige Synthese entlang der DNA. Das 3’-OH-Ende des vorhergehenden Nucleotids greift die Phosphorsäureanhydrid-Bindung zwischem dem α- und dem β-Phosphat des nächsten anzuheftenden Nucleotids an, welches über Basenpaarung am Matrizenstrang fixiert ist. Das dabei anfallende Pyrophosphat wird weiter zu 2 Pi hydrolysiert. Somit werden pro eingebautem Nucleotid zwei energiereiche Bindungen gespalten. • Kein Primer nötig. Termination: • RNA-Polymerase trifft auf Stoppsignal: G-C-reiche Struktur gefolgt von kurzem poly-U-Segment. • Gebildetes G-C-reiches RNA-Segment lagert sich selbst zu Haarnadelschleife zusammen und katapultiert Enzym vom Matrizenstrang 4/6 06. (4 Punkte) Was versteht man unter alternativem Spleißen? Alternatives Spleißen bedeutet, dass neben den Introns auch ausgewählte Exons ganz oder teilweise entfernt werden. Durch alternativen Gebrauch von Donor- bzw. Akzeptorstellen in der primären RNA entstehen verschiedene mRNAs und somit mehr Genprodukte. 07. (6 Punkte) Beschreiben Sie das Prinzip der PCR. • • • • • • • • • • Voraussetzung: Teile der interessierenden DNA-Sequenz sind bekannt Mit dieser Information Herstellung von zwei DNA-Oligonucleotiden von je 1520 Resten, die jeweils an einen der beiden komplementären Stränge hybridisieren und die zu vervielfältigende Region begrenzen Denaturierung durch Erhitzen auf 95°C Annealing: beim Abkühlen auf ca. 55°C binden die Primer an die komplementären Einzelstränge Die DNA-Polymerase-Reaktion setzt an den Primern ein und verläuft in 5’-> 3’-Richtung entlang der einzelnen Matrizenstränge ab Unterbrechung der Initialreaktion durch kurzzeitiges Erhitzen auf 95°C Folge: Denaturierung der neu gebildeten Doppelstränge Wiederum Absenken der Temperatur, Bindung der Primer, weitere Polymerisation Ergebnis: 4 Filialstränge Wiederholung dieser Prozessierungsschritte, bis nach 20-30 Zyklen das betreffende DNA-Segment millionen- bis milliardenfach amplifiziert ist 08. (4 Punkte) Nennen Sie zwei Typen der Enzyminhibition Ihrer Wahl. Beschreiben Sie die Auswirkungen auf KM und vmax. Beispiele: Kompetitive Inhibition: KM erhöht vmax unverändert Unkompetitive Inhibition: KM verringert vmax verringert 5/6 09. (4 Punkte) Erklären Sie das folgende Ramachandran-Diagramm des Modellpeptids Polyalanin. Da die Peptidbindung Doppelbindungscharakter hat, ist sie planar und starr. Die Bindungen N-Cα und Cα-CO sind als Einfachbindungen jedoch drehbar. Die zugehörigen Dreh- oder Torsionswinkel werden als φ und ψ bezeichnet. Jede Peptidbindung in der Hauptkette eines Polypeptids nimmt eine bestimmte Kombination dieser beiden Winkel an. Die Gesamtheit der Winkelkombinationen definiert die Konformation eines Proteinrückgrats. Die möglichen Kombinationen von φ und ψ sind jedoch aufgrund eventueller sterischer Hinderungen zwischen Hauptund Seitenketten eingeschränkt. Der Raum möglicher Konformationen wird mittels des Ramachandran-Diagramms visualisiert. Die mit α und β bezeichneten Bereiche geben an, welche Winkelkombinationen zu den Sekundärstrukturelementen α-Helix und β-Faltblatt führen. Genetischer Code: 1. Pos. 2. Pos. U U C A G Phe Phe Leu Leu Leu Leu Leu Leu Ile Ile Ile Met / Start Val Val Val Val C Ser Ser Ser Ser Pro Pro Pro Pro Thr Thr Thr Thr Ala Ala Ala Ala 3. Pos. A Tyr Tyr Stop Stop His His Gln Gln Asn Asn Lys Lys Asp Asp Glu Glu 6/6 G Cys Cys Stop Trp Arg Arg Arg Arg Ser Ser Arg Arg Gly Gly Gly Gly U C A G U C A G U C A G U C A G