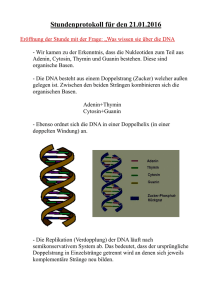
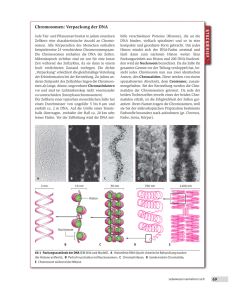
Struktur von Nukleinsäuren Woraus besteht DNA?
Werbung

Struktur von Nukleinsäuren Woraus besteht DNA? James D. Watson & Francis H. C. Crick entwarfen 1953 mithilfe der Daten von Chargaff und der Röntgenkristallographie-Daten von Rosalind Franklin und Maurice Wilkins das Modell der DNA-Doppelhelix. Hinweis: Im Atelier finden Sie beim Posten "Geschichte" interessante Berichte über die Aufklärung der Struktur der DNA, darunter auch Originalliteratur ( Dokument G1 „Die DNADoppelhelix“). DNA, die Desoxyribonukleinsäure, und die heterogene Gruppe der RNAs, Ribonukleinsäuren, gehören in die Familie der Nukleinsäuren. Nukleinsäuren bestehen ganz allgemein aus drei Bausteinen: aus einem Zucker, einer Phosphorsäure und aus Basen. Betrachten wir vorerst nur die DNA: Die DNA enthält als Zucker - wie schon der Name vermuten lässt - 2-Desoxyribose, ein C5Kohlenhydrat. Die Hydroxylgruppe am zweiten C-Atom fehlt, so dass zur Verknüpfung der anderen Bausteine nur noch drei Möglichkeiten zur Verfügung stehen, nämlich die Hydroxylgruppen an C1, C3 und C5: Die Phosphorsäure ist mit zwei Zuckern verestert. Sie verbindet das C3 der einen Desoxyribose mit dem C5 einer andern. Indem auf jede Phosphorsäure ein Zucker folgt und auf diesen wiederum eine Phosphorsäure, entsteht ein langes, lineares Molekül. Nur an einem der beiden Enden, am sogenannten 5'-Ende (sprich: 5 Strich), befindet sich eine Phosphorsäure. Das andere 1 Ende, entsprechend 3'-Ende genannt, ist eine Desoxyribose, welche als einzige am C3 nicht verestert ist. Unsere Zucker-Phosphat-Kette erhält so eine Richtung: Die DNA enthält (neben Zucker und Phosphat) vier Basen, die man aufgrund ihrer Struktur in Purine und Pyrimidine unterteilt. Purine bestehen aus zwei Ringen (einem 6- und einem 5-Ring), Pyrimidine haben nur einen Ring, den 6-Ring: Die in der DNA regelmässig vorkommenden Basen sind die Purine Adenin und Guanin, sowie die Pyrimidine Thymin und Cytosin. Die ebenfalls aufgeführte Pyrimidin-Base Uracil kommt nur in der RNA vor. 2 Wie sind die Basen mit den andern Bausteinen verknüpft? Wie auf dem Bild (Struktur der Purine und Pyrimidine) gezeigt, sind zwei der drei Hydroxylgruppen der Desoxyribose bereits mit der Phosphorsäure verestert, so dass die Verbindung Zucker-Base nur noch über C1 erfolgen kann. Da die Koppelung über N9 bei den Purinen bzw. über N1 bei den Pyrimidinen erfolgt, nennt man die Bindung N-glykosidisch. Die Primärstruktur An jede Desoxyribose ist eine der vier Basen (Adenin, Guanin, Cytosin oder Thymin) Nglykosidisch gebunden. Die Abfolge der Basen kann beliebig variieren und stellt die eigentliche Erbinformation dar. So ist es möglich, dass die DNA (trotz ihres einfachen Baus) Träger von Information ist! Die Sequenz der Basen einer Nukleinsäure nennt man Primärstruktur. Sie liest sich vereinbarungsgemäss immer von 5' nach 3'. Als Beispiel sei ein DNA-Einzelstrang mit der Basenfolge 5'-Adenin-Thymin-Guanin-3' gezeigt: Die Sekundärstruktur In Zellen existiert ein DNA-Einzelstrang jeweils nur kurzfristig und nur über kurze Abschnitte, z.B. während der Zellteilung oder während der Transkription. Normalerweise paart sich ein DNA-Einzelstrang nämlich immer mit einem zweiten Einzelstrang. Diese Paarung ergibt sich dadurch, dass immer eine Base des einen Einzelstrangs mit einer bestimmten Base des anderen Strangs Wasserstoffbrücken ausbildet. Die beiden Basen, welche sich gegenüberstehen, nennt man komplementär. Adenin paart sich mit Thymin, Guanin mit Cytosin: 3 Sehen Sie sich die Basenpaare etwas genauer an: einer Purin-Base steht immer eine PyrimidinBase gegenüber und umgekehrt. Das bedeutet, dass in der DNA immer gleichviele Purine wie Pyrimidine eingebaut sind. Diese als Chargaff-Regel bekannte Tatsache besagt auch, dass die Summe der Adenine gleich der Summe der Thymine und die Summe der Guanine gleich der Summe der Cytosine ist: A+G=T+C A/T=G/C=1 Die Sekundärstruktur einer Nukleinsäure wird durch die Abfolge komplementärer Basen bestimmt. Im Falle der DNA bleiben die Doppelstränge nicht einfach in einer Ebene, vielmehr verdrillen sie sich schraubenartig zu einer Doppelhelix, wobei die hydrophoben Basen innen liegen, die Zuckerringe und die Phosphodiesterbrücken hingegen das Rückgrat bilden und aussen liegen: Die DNA-Doppelhelix hat folgende Eigenschaften: Sie dreht nach rechts und hat einen Durchmesser von etwa 2 nm. Der Abstand zwischen zwei benachbarten Basen beträgt 0,34 nm und der helicale Drehungswinkel der benachbarten Basenpaare beträgt 36°. Das ergibt pro Umdrehung 10 Basenpaare und eine Hubhöhe von 3,4 nm. Die planaren Basen liegen horizontal übereinander und können durch diese Stapelung ("base stacking") die Konformation der DNA stabilisieren. Weil sich die Zucker-Base-Bindungen der komplementären Basenpaare nicht direkt gegenüber liegen, also nicht diametral zueinander stehen (ihr Abstand beträgt weniger als der Durchmesser der Helix, vergl. obige Skizze), sind die vertikalen Abstände zwischen den beiden 4 Einzelsträngen der DNA nicht immer gleich gross, sie bilden entweder eine kleine oder eine grosse Grube. Die beiden Einzelstränge der DNA sind zueinander antiparallel angeordnet, d. h. das 5'-Ende des einen Strangs liegt dem 3'-Ende des andern gegenüber und umgekehrt. Durch die Basenpaarung ergibt sich, dass aus der Basenfolge des einen Strangs zwingend die Basenfolge im andern (komplementären) Strang bestimmt werden kann. Uebung: Sie können dies an einem einfachen Beispiel üben: Versuchen Sie aus folgender DNASequenz den komplementären Strang zu finden: 5'-AATCAGCGATCGCTG-3' Lösung: S. 13 Hinweise: Vergewissern Sie sich, dass Sie alle Eigenschaften der gezeigten DNA verstehen. Sie ist die am häufigsten vorkommende Konformation der DNA und wird als B-Form bezeichnet: siehe Stereobild auf dem Plakat. Man kann das Stereobild auch ohne eine Stereobrille als ein einziges, räumliches Bild sehen, wenn man es durch Üben schafft, zwei normalerweise miteinander verknüpfte Sehreflexe zu entkoppeln. Man muss die Augen streng parallel stellen, als wären sie auf ein fernes Objekt gerichtet, die Augenlinsen aber zugleich für nahes Sehen fokussieren:Achtung! Beim Betrachten des Stereobildes ohne Stereobrille dreht die Helix nach links statt nach rechts! Sie können sich die Struktur der DNA wahrscheinlich am besten vorstellen, wenn Sie das dreidimensionale Modell zuhilfe nehmen. Im Atelier finden Sie ein DNA-Modell. Es zeigt eine vollständige Helixwindung einer B-DNA im Masstab 1:108; der Durchmesser des Modells beträgt also 20 cm (1 nm entspricht ungefähr 10 cm). Versuchen Sie, die folgenden Fragen zu beantworten: • Welches ist die Phosphorsäure, welches der Zucker? • Sehen Sie die grosse und die kleine Grube? • Wo liegt das 3'- und wo das 5' - Ende eines Stranges? • Sehen Sie, wie die Basen übereinandergestapelt und gegeneinander versetzt sind? • Betrachten Sie ein Basenpaar: welches ist die Purinbase und welches die Pyrimidinbase? Vielleicht erkennen Sie die Basen sogar und damit die Basensequenz! Die Basensequenz eines DNA-Abschnitts bestimmt, welche Konformation die Doppelhelix annimmt. Alternative Konformationen sind wichtig für die Erkennung bestimmter Abschnitte durch Eiweisse (z.B. bei der Replikation der Transkription), weil die Basen dadurch für Eiweisse besser zugänglich gemacht werden. A-DNA: Sie kommt z.B. als Intermediäres bei der Transkription vor (DNA/RNA-Helix). Die zusätzliche Hydroxylgruppe am C2 der Ribose verhindert, dass das DNA-RNA-Hybride die BKonformation annehmen kann. Aus demselben Grund kommt die A-Form auch bei doppelsträngiger RNA vor (z.B. in Viren). Die A-Form ist zwar ebenfalls rechtsdrehend, sie ist aber "dicker" als die B-Form und beide Gruben sind etwa gleich gross: siehe Stereobild auf dem Plakat. Z-DNA: Alternierende Purin-/Pyrimidinsequenzen wie z.B. 3´-CGCGCGCGCG-5´ 5´-GCGCGCGCGC-3´ 5 bilden eine linksdrehende Doppelhelix, welche etwa 12 Basenpaare pro Umdrehung hat. Die ZDNA verdankt ihren Namen ihrem "zick-zack"-artigen Zucker-Phosphat-Rückgrat: siehe Stereobild auf dem Plakat. Woraus besteht RNA? RNA ist eine heterogene Gruppe von Nukleinsäuren, deren Aufgabe es ist, Information in der DNA in Proteine umzusetzen. Die verschiedenen Klassen (tRNA, rRNA, hnRNA und mRNA) werden deshalb erst bei den Kapiteln „Transkription“ und „Translation“ genauer erläutert. Hier sei vorerst nur der allgemeine Bau aufgezeigt. Die Ribonukleinsäure, kurz RNA, ist grundsätzlich gleich gebaut wie DNA, zeigt aber einige wichtige und interessante Unterschiede, welche zum Teil schon erwähnt worden sind: • • • als Zucker dient Ribose, nicht 2-Desoxyribose die Pyrimidin-Base Uracil ersetzt Thymin. Alle andern Basen bleiben dieselben RNA besteht (in der Regel) nur aus einem Einzelstrang; es treten aber intramolekulare Basenpaarungen auf, wobei sich Adenin mit Uracil, Guanin mit Cytosin paaren Nukleoside und Nukleotide Purine und Pyrimidine sind nicht nur Bestandteile von Nukleinsäuren, sondern auch von Energieträgern im Cytosol, den sogenannten Nukleotiden, welche z.B. bei diversen Biosynthesen, bei Membrantransporten oder bei Muskelarbeit essentiell sind. 6 Sind nur Zucker und Base im Molekül vorhanden, sprechen wir von einem Nukleosid; ist am C5 noch ein Phosphorsäure-Rest gebunden, sprechen wir von einem Nukleotid: Nukleosid: Base + Zucker Nukleotid: Base + Zucker + Phosphat Für die diversen Verbindungen ergeben sich dadurch folgende Bezeichnungen: freie Base Nukleoside1 Nukleotide2 Adenin (A) Adenosin Adenosinmonophosphat (AMP) Guanin (G) Guanosin Guanosinmonophosphat (GMP) Cytosin (C) Cytidin Cytidinmonophosphat (CMP) Thymin (T) Thymidin Thymidinmonophosphat (TMP) Uracil (U) Uridin Uridinmonophosphat (UMP) 1 Nukleoside enden bei den Purin-Derivaten auf -osin, bei den Pyrimidin-Derivaten auf -idin. Enthalten Nukleoside Desoxyribose als Zucker, nennt man sie Desoxynukleoside; entsprechendes gilt auch für die Nukleotide (Abkürzung: dAMP, dGMP, ... ) 2 Nukleotide können mit einem, zwei oder gar drei Phosphorsäure-Resten verknüpft werden. Entsprechend nennt man zum Beispiel die Adenin-Derivate Adenosinmonophosphat (AMP), Adenosindiphosphat (ADP) und Adenosintriphosphat (ATP). Als Beispiel für ein Nukleotid sei im folgenden Adenosintriphosphat (ATP) gezeigt, der wichtigste Energiespeicher und -überträger des Intermediärstoffwechsels: 7 Nukleinsäuren sind also unverzweigte, lineare Biopolymere aus Nukleotid-Monophosphaten, wobei dAMP, dGMP, dCMP und dTMP die Bausteine für die DNA, und AMP, GMP, CMP und UMP diejenigen für die RNA darstellen! Folgende Darstellung veranschaulicht diesen Sachverhalt: B = Base, Z = (Desoxy)-Ribose, P = Phosphat Hinweis: Sie haben die Möglichkeit, in der Videoecke den Videofilm „Struktur von Nukleinsäuren“ (Dauer: 4.5 Min.) anzuschauen. Eer fasst das bisher Dargestellte in leicht verständlicher Form zusammen. Superstruktur der DNA Die menschliche DNA einer diploiden Zelle besteht aus ungefähr 6 x 109 Basenpaaren, verteilt auf 46 Chromosomen. Jeder Zellkern enthält somit 46 DNA-Moleküle unterschiedlicher Grösse (vgl. DNA in Zellorganellen wie Mitochondrien und Chloroplasten). Würde man die DNA in den 46 Chromosomen zu einer einzigen Doppelhelix zusammenfügen, käme man auf eine Länge von 2 Metern DNA pro Zelle! Es ist einleuchtend, dass die DNA Strukturen annehmen muss, die es erlauben, sie in den Zellkern zu packen und sie vor Scherkräften zu schützen, die die nur 2 nm Dicke Doppelhelix in Stücke reissen könnten. Aus diesem Grund binden bestimmte Proteine an die DNA und verformen diese zu stabilen Superstrukturen. Solche DNA-Proteinkomplexe nennt man Chromatin. Bei Zellteilungen – Mitosen und Meiosen -, müssen sich die Chromosomen zu Mitosechromosomen maximal verdichten (kondensieren), damit sie überhaupt auf die Tochterzellen verteilt werden können. Betrachten wir also vorerst die sogenannten Interphasenchromosomen. Die an DNA gebundenen Proteine machen einen beträchtlichen Teil des Chromatins aus. Tatsächlich besteht Chromatin etwa zur Hälfte aus Proteinen, welche noch in Histone und NichtHiston-Proteine unterteilt werden können. Histone sind kleine, basische Eiweisse, die nur in Eukaryonten vorkommen. Mit ihrem hohen Anteil an positiv geladenen Aminosäuren wie Arginin und Lysin können sie relativ unspezifisch an die DNA binden und die negativen Ladungen der Phosphatgruppen des "DNA-Rückgrats" kompensieren. Aufgabe: Überlegen Sie sich, welche Auswirkungen es auf die Stabilität der DNA hätte, wenn die negativen Ladungen der Helix nicht aufgehoben würden. Lösung: S. 13 8 Es gibt insgesamt fünf verschiedene Klassen von Histonen: H1, H2A, H2B, H3 und H4. Gewisse eukaryontische Zellen - wie z.B. die Erythrocyten des Huhns - haben noch einen weiteren Typ, das Histon H5, welches als Variante von H1 angesehen wird. Je zwei Moleküle H2A, H2B, H3 und H4 verbinden sich zu einem Histon-Octamer. Die DNA-Doppelhelix wickelt sich nun in Linkswindungen um dieses Octamer wie ein Faden um eine Spule. Die dadurch entstehende Struktur ist das Nucleosom. Weitere Angaben: Core-Partikel Chromatosom Nukleosom Core-Partikel: Die Doppelhelix wickelt sich 1 3/4-mal um das Histon-Oktamer. Dies entspricht 146 Basenpaaren (bp) DNA. Chromatosom: Es enthält zwei volle Windungen DNA (166 bp DNA).Daran bindet zusätzlich noch das Histon H1 (vertikaler Zylinder, rechts). Nukleosom: Es besteht ebenfalls aus zwei Windungen DNA, dem Histon-Octamer und H1, hat aber noch sog. Linker-DNA von etwa 30 bp, welche benachbarte Nukleosomen miteinander verbindet. Ein Nukleosom enthält also etwa 200 bp DNA. Behandelt man Chromatin mit einer stark hypotonen Lösung, so lassen sich die Chromosomen als Nukleosomenfäden im Elektronenmikroskop darstellen. Sie gleichen einer Perlenkette: Die nächsthöhere strukturelle Einheit des Chromatins ist das Solenoid. Für die Ausbildung des Solenoids ist das Histon H1 essentiell: es verbindet benachbarte Nukleosomen zu einer 30 nm dicken Chromatinspirale. Wie sich dieser Prozess genau vollzieht, ist aber noch weitgehend unklar. Folgendes EM-Bild veranschaulicht die dichte Packung der Nukleosomen: 9 Die Solenoid-Faser wird gelegentlich von nicht-Histon-Proteinen in Segmente unterteilt, wie folgendes Modell zeigt: Diese Proteine binden meist sequenzspezifisch an die DNA und stellen zum Teil GenRegulationsproteine (z.B. Transkriptionsfaktoren) dar, andere helfen bei der Initiation der DNAReplikation. Eine weitere Gruppe von Proteinen organisiert das Solenoid in Schleifen, welche von der Hauptachse des Chromosoms abstehen. Ohne die Ausbildung von Schleifen, als lineares Solenoid also, würde ein durchschnittlich grosses Chromosom den Zellkern als 30-nm-Faser von 1 mm Länge etwa hundertmal durchmessen. Auch hier sind die Details der Faltung noch unbekannt und Gegenstand intensiver Forschung. Unten ein Schema, das das Prinzip der Faltungen wiedergibt: Hinweis: D. Hewish und L. Burgoyne waren unter den ersten, welche sich mit der Strukturaufklärung des Chromatins beschäftigten. Lesen Sie dazu das Dokument "Hewish & Burgoyne, 1973" am Geschichtsposten: Dokument G 24. 10 Sowohl in Eukaryonten wie in Prokaryonten gibt es Enzyme, die Topoisomerasen, die unter ATP-Verbrauch einen oder beide DNA-Stränge auftrennen, durch Rotation um die Helixachse die Windungszahl pro Längeneinheit ändern und dann die freien Enden wieder verknüpfen (ligieren). Die Topoisomerase I spaltet dabei nur einen, die Topoisomerase II beide DNA-Stränge. Nimmt die Windungszahl pro Längeneinheit zu, spricht man von positivem Supercoiling, nimmt sie ab, von negativem Supercoiling. Diese Konformationsänderung setzt die DNA unter Torsionsspannung. Hat die DNA ein freies Ende, so kann sie sich wieder entspannen, indem sie um ihre Achse rotiert. Sind dagegen beide Enden fixiert, wie bei der Ring-DNA der Prokaryonten oder bei Chromatin-Schleifen, kann die DNA die Überspannung nur durch Ausbildung einer superhelicalen Schleife kompensieren (zwei fixe Enden können nicht frei rotieren). Bei dieser DNA-Superhelix ist die DNA zusätzlich zu den Doppelhelixwindungen umeinander verdrillt: Sie können sich die Entstehung dieser Konformation am besten vorstellen, wenn sie an ein spiraliges Telefonkabel denken, welches Sie aus einem gedehnten in einen entspannten Zustand führen. Hinweis: Gute Dienste leistet ein Schnurmodell aus zwei umeinander verdrillten Fäden, an dem Sie die Auswirkungen von positivem und negativem Supercoiling studieren können. Die Schnur und eine Anleitung finden Sie beim Posten. Topoisomerasen benötigen für die Rückführung (Relaxation) in den Normalzustand kein ATP; die Energie steckt nämlich in der Supercoil-Konformation selbst: 11 Was ist denn der Zweck dieser Superstrukturen? Negatives Supercoiling erleichtert wahrscheinlich Prozesse wie Replikation oder Transkription, weil die "unterwundene" DNA den Zugang bestimmter Proteine zu Steuersequenzen erleichtert. Quellennachweis Die Texte und Abbildungen des vorliegenden Scripts entsprechen den Seitenangaben aus folgenden Werken: • Bruce Alberts et al.: „Molekularbiologie der Zelle", VCH, 2. Auflage, p. 57, 62-63, p. 108113, p. 571, 577, 583-594, p. 690-692 • Walther Traut: „Chromosomen - klassische und molekulare Cytokinetik", Springer-Lehrbuch, p. 57-84 Hinweis: In der „Lese-Ecke“ stehen Ihnen Lehrbücher zum vertieften Studium zur Verfügung 12 Lösungen ♦ Versuchen Sie aus folgender DNA-Sequenz den komplementären Strang zu finden: 5'-AATCAGCGATCGCTG-3' 3‘-TTAGTCGCTAGCGAC-5‘ Der komplementäre Strang hat also die Basensequenz 5'-CAGCGATCGCTGATT-3' Sequenzen werden in 5'-> 3'-Richtung hingeschrieben! ♦ Überlegen Sie sich, welche Auswirkungen es auf die Stabilität der DNA hätte, wenn die negativen Ladungen der Helix nicht aufgehoben würden. Die negativen Ladungen würden sich gegenseitig abstossen und die Doppelhelix destabilisieren. Uebungsaufgaben mit Lösungen finden Sie in der Internetversion des Ateliers! Hinweis: Das „Repetitorium Molekularbiologie“ definiert den Stoff, welcher in den Prüfungen verlangt wird. Wegen seiner Kürze eignet es sich allerdings nicht als primäre Informationsquelle! 13 Chemie der Nukleinsäuren Hinweis: Im Atelier finden Sie die CD "The Nature of Genes". Mittels Tutorials und Aufgaben werden die wichtigsten Themen der Molekularbiologie leicht verständlich vermittelt. Synthese und Abbau der Pyrimidinbasen Hinweis: Synthesen und Abbau der Pyrimidin- und Purinbasen sind Stoff des 2. Studienjahres. Sie werden deshalb hier nur summarisch dargestellt. 1 Die Biosynthese der Pyrimidine erfolgt in 3 Phasen: • Durch die cytoplasmatische Carbamoylphosphat-Synthetase II wird Carbamoylphosphat gebildet: 2 ATP + Glutamin + CO2 + H2O -> 2 ADP + Glutamat + Carbamoyl-P Carbamoylphosphat, das ein grosses Gruppenübertragungspotential hat, reagiert mit Aspartat zu Carbamoyl-Aspartat, das in einer Gleichgewichtsreaktion zu Dihydroorotat zyklisiert (Enzym: Aspartat-Transcarbamoylase). Dieses wird zu Orotat dehydriert (Enzym: Dihydroorotase). • Orotat reagiert mit 5-Phosphoribosyl-1-Diphosphat (PRPP) zum Orotidin-5'-Phosphat welches zum Uridin-5'-Phosphat (UMP) decarboxyliert wird. • Phosphorylierung zum UTP durch Kinasen Die anderen Nukleotide (CTP und TTP) bzw. deren Desoxyribose-Derivate dCTP und dTTP für die DNA gehen aus Uridin-Derivaten hervor! Abbau: der Ring wird partiell hydriert und zwischen N-3 und C-4 hydrolytisch geöffnet. Da jedoch bei der Synthese zwischen Orotsäure und Uridin CO2 abgespalten wurde, erscheint als Abbauprodukt nicht Asparaginsäure sondern Alanin. Frei werdendes NH3 wird im Harnstoffzyklus zu Harnstoff weiter metabolisiert und so ausgeschieden. Synthese und Abbau der Purinbasen Die Biosynthese der Purine verläuft wesentlich umständlicher als diejenige der Pyrimidine. Die Synthese beginnt am Ribose-5-Phosphat-Teil des Nucleotids: Ribose-5-Phosphat + ATP -> 5-Phosphoribosyl-1-Pyrophosphat (PRPP) Die Synthese des Purinrinsystems beginnt mit einem NH3-Transfer: PRPP + Glutamin + H2O -> 5-Phosphoribosyl-1-amin + Glutamat + PP Diese Reaktion ist limitierend für die gesamte Purinsynthese und wird durch AMP und GMP gehemmt (Endprodukthemmung). Phosphoribosylamin wird darauf in mehreren Schritten in Inosinmonophosphat und dieses in Anenosin- und Guanosinmonophosphat überführt. Abbau: Der Abbau wird in zwei Schritte unterteilt: • • Von den Nukleotiden wird zuerst Phosphat (durch eine Phosphatase) und darauf der Riboserest durch eine Nucleosidphosphorylase abgespalten. Auf die freien Purine und Nukleoside wirken nun zwei Enzyme: Desaminasen, die AminoPurine in Oxy-Purine verwandeln und Xanthinoxidasen, die Hypoxanthin über Xanthin in Harnsäure ueberfuehren, welche ausgeschieden werden kann. Bei den meisten Säugern wird Harnsäure durch Uricase noch weiter zu Allantoin abgebaut. Dieser Schritt ist aber beim Menschen nicht möglich, da das Enzym Uricase fehlt. 2 Chemie der Basen Vier Basen sind am Aufbau der DNA beteiligt, nämlich Adenin (A), Guanin (G)[PurinDerivate], Cytosin (C) und Thymin ( T) [Pyrimidin-Derivate]. RNA enthält Uracil (U) an Stelle von Thymin. Die Purin- und Pyrimidinbasen sind planar und hydrophob. Sie bestehen aus heterocyclischen Ring-Verbindungen, welche Stickstoff und Kohlenstoff enthalten und periphere apolare Kohlenwasserstoff- und Stickstoffwasserstoff- Gruppen tragen. Sie werden als Basen bezeichnet, weil sie bei physiologischem pH H+ anlagern. C, T und U sind einfache Derivate eines Pyrimidin-Sechsringes, A und G sind Purin-Verbindungen (ein Fünfring mit dem Sechsring kondensiert). Das Absorptionsmaximum dieser Basen liegt bei 260 nm, also im UV-Bereich. Tautomere Formen der Basen Einer von vielen Gründen für eine Mutation ist das Auftreten von tautomeren Formen der Basen. Man versteht darunter eine Veränderung der Basen, die spontan, aber mit kleiner Häufigkeit auftritt. Diese tautomere Form paart nun mit einer anderen Base, was zu einer Mutation führt, z.B.: 3 Normale Form A (Amino) paart mit T C (Amino) paart mit G G (Keto) paart mit C T (Keto) paart mit A Seltene tautomere Form A (Imino) paart mit C C (Imino) paart mit A G (Enol) paart mit T T (Enol) paart mit G Tautomere sind Strukturisomere, die sich im Gleichgewicht befinden. Der Unterschied in den Strukturen kommt durch die Verschiebung eines Protons zustande. Das Gleichgewicht liegt stark auf der Seite der Amine und Ketone, ein kleiner Anteil Imine und Enole liegt jedoch auch vor. Die H-Brücken zwischen Basen brauchen einen H+-Donor und einen H+-Akzeptor. Liegt nun eine andere tautomere Form vor, weil sich ein Proton innerhalb einer Verbindung verschoben hat, dann verändern sich je nach Base Akzeptor bzw. Donor, und es können nicht mehr dieselben H+Brücken aufgebaut werden. 4 Andere Möglichkeiten der Veränderung bieten zwei spontane chemische Reaktionen, die ernste DNA-Schäden in den Zellen bewirken können. Es sind die Desaminierung und die Depurinierung der Basen, bei denen entweder durch Abspaltung von Ammoniak eine andere Base entsteht (Desaminierung) oder aber die Base ganz vom Zucker abgespalten wird (Depurinierung). Stoffwechsel der Zucker Die Zucker für die Nucleosid-Synthese können auf zwei Wegen bereitgestellt werden, nämlich auf einem oxidativen und einem nicht-oxidativen Weg: • oxidativer Weg (Hexosemonophosphat-Weg): 5 • zweiter Weg (Zuckerumwandlungen): Hinweis: Der Stoffwechsel der Zucker ist Stoff der Biochemie behandelt und wird deshalb hier nur summarisch dargestellt. Chemie der Zucker Man unterscheidet zwischen Desoxyribonukleinsäure (DNA) mit 2-Desoxyribose und Ribonukleinsäure (RNA) mit Ribose als Zucker. Die Zucker bilden das "Rückgrat" der Doppelhelix, indem sie aussen liegen und gegen "oben" und "unten" miteinander über Phosphat verbunden sind. Die 2'-OH-Gruppe der Ribose ist für die chemische Reaktivität der RNA verantwortlich. Wenn diese OH-Gruppe durch eine Base (z.B. OH- ) angegriffen wird, entsteht ein Alkoholat (O- ). Dieses greift das benachbarte Phosphoratom an und spaltet damit die RNAKette (nukleophiler Angriff). Gegen das Zentrum der Helix hin sind die Zucker N-glycosidisch an eine Purin- resp. Pyrimidinbase gebunden. Wenn man sich die Basen und die Zucker als planare Scheiben vorstellt, dann bilden diese einen rechten Winkel: 6 Chemie der Nukleinsäuren Das Zentrum der DNA wird von den Basen gebildet und ist demzufolge hydrophob. Die basischen Ringe, die nach innen ragen, sind den Stufen einer Wendeltreppe vergleichbar und liegen senkrecht zur Helixachse. Es liegen sich immer zwei Basen gegenüber, welche durch Wasserstoffbrücken miteinander verbunden sind. Ein Purin-Ring bildet immer mit einem Pyrimidin-Ring ein Basenpaar und umgekehrt (d.h. sie sind komplementär), da zwei sich gegenüberliegende Purin-Ringe in einer Doppelhelix zuviel Platz beanspruchen würden, während zwei Pyrimidin-Ringe den Raum nicht ausfüllen könnten. Die Anzahl der Wasserstoffbrücken bestimmt unter anderem die Stabilität der Doppelhelix. H-Brücken bilden sich zwischen einem H einer OH- oder NH-Gruppe und einem einsamen Elektronenpaar eines O-Atomes bzw. eines NAtomes aus, wenn die Gruppen sich auf eine Distanz von ca. 0,28nm annähern. Die Bindung ist nicht sehr stark, mehr als 10mal schwächer als eine Hauptvalenzbindung. Unterschiede in der Stabilität entstehen dadurch, dass die Basenpaare unterschiedliche Anzahlen von H-Brücken ausbilden: während Guanin und Cytosin drei H-Brücken bilden, findet man zwischen Adenin und Thymin nur zwei. Dies hat zur Folge, dass die Stabilität der Doppelhelix mit höherem Anteil an G - C -Paarungen zunimmt. 7 Den grössten Einfluss auf die Stabilität der Doppelhelix haben die hydrophoben Kräfte, die zwischen den relativ nah übereinander liegenden ("stacked") Basen wirken (der Abstand beträgt etwa 0.26-0.34nm). Jedes Basenpaar ist im Vergleich zum Nachbar-Basenpaar um ca. 36° um die Achse der Helix gedreht, so dass etwa 10 Basenpaare eine vollständige Umdrehung von 360° ergeben. Die oben beschriebene DNA ist die häufigste Form und wird als B-Form bezeichnet. Je nach Wasser- und Salzgehalt des Mediums kann die DNA aber eine unterschiedliche Geometrie einnehmen. Andere charakteristische Strukturen sind die A- und die B-DNA, über die Sie im Atelier am Posten "Struktur von Nukleinsäuren" näheres erfahren können. Das "Rückgrat" der Doppelhelix wird durch die Verknüpfung der Zucker mit den Phosphaten gebildet, womit die Peripherie der Helix zu einer hydrophilen Hülle wird. Die negativen Ladungen der Phosphatreste bewirken eine Abstossung der beiden Stränge. Dies wird durch die Bindung von Kationen an die Phosphate vermindert oder verhindert (Neutralisation von Ladungen). Zucker und Phosphat sind esterartig verbunden, Zucker und Base sind N-glycosidisch verbunden. Aufgabe: Warum bilden Adenin und Guanin in der DNA keine Basenpaare? Lösung: S. 11 Wie isoliert man DNA? Wenn man DNA untersuchen will, muss man sie zuerst aus Zellen isolieren. Dazu sind folgende Schritte notwendig: 1. Beschaffen von Zellmaterial 2. Aufschliessen der Zellen 3. Unlösliche Teile abzentrifugieren 4. Nukleinsäuren von Proteinen trennen und reinigen 5. Wässrige Phase sammeln 6. Nukleinsäuren mit Alkoholen fällen 7. Nukleinsäuren in Puffer lösen 8. DNA und RNA trennen Hinweis: Sehen Sie sich den Film „DNA-Isolation“ in der Video-Ecke an (Dauer: ca. 7 Min.) Details zur DNA-Isolation finden Sie in der aufgelegten Broschüre. Denaturierung von DNA Was geschieht, wenn man DNA erwärmt? Die Doppelhelix bleibt nicht stabil, sondern trennt sich in die beiden Einzelstränge. Diesen Vorgang nennt man Denaturierung oder Schmelzen. 8 Die Stränge der DNA trennen sich, weil die Wasserstoffbrücken zwischen den komplementären Basenpaaren bei hohen Temperaturen (z.B. 90°C) gelöst werden (die benötigte Temperatur ist unter anderem von der Länge der DNA und vom G-C-Gehalt abhängig). Dabei ändern sich die physikalischen Eigenschaft der DNA, z.B. Viskosität, Lichtabsorption. Man spricht bei diesem Aufbrechen der Helixstruktur von einem Schmelzvorgang und definiert den Schmelzpunkt Tm der DNA als die Temperatur, bei der die Hälfte der DNA als Einzelstränge vorliegt. Wie wir vorher gesehen haben, bilden die beiden möglichen Basenpaare A-T resp. G-C eine unterschiedliche Anzahl an Wasserstoffbrücken aus (G-C drei, A-T zwei). Somit ist der Schmelzpunkt abhängig von der Anzahl G-C Basenpaare, weil diese der Helix eine grössere Stabilität verleihen und der Schmelzpunkt Tm einer G-C reichen DNA höher als derjenige einer A-T reichen DNA. Zur Illustration der Veränderung der Lichtabsorption der DNA bei der Denaturierung dient folgendes Beispiel: Die Absorption der DNA-Moleküle der Bakterienstämme Pneumococcus und Serratia wird bei 260 nm mit zunehmender Temperatur gemessen. Einzelsträngige DNA absorbiert bei 260 nm etwa 1.4 mal mehr (also 40% mehr) als doppelsträngige DNA. Die Zunahme der Absorption man nennt diesen Vorgang den hyperchromen Effekt - ist demzufolge ein Mass für den Gehalt an einzelsträngiger DNA der untersuchten Probe: 9 Die Lage der Schmelzkurve kann beeinflusst werden, da sie vom Lösungsmittel abhängt: Erniedrigung der Salzkonzentration, Erhöhung des pH und die Anwesenheit gewisser organischer Lösungsmittel verschieben die Kurve nach links. Auf der rechten Seite der Grafik ist die Abhängigkeit des Schmelzpunktes vom G-C Gehalt der jeweiligen Bakterien-DNA gut ersichtlich. Aufgaben: Weshalb ist der Schmelzpunkt Tm von Pneumococcus-DNA tiefer als der von Serratia-DNA? Welche Bedeutung hat eine Linksverschiebung der Schmelzkurve? Lösungen: S. 11 Renaturierung Unter geeigneten Bedingungen lassen sich denaturierte ("geschmolzene") DNA-Abschnitte wieder in die doppelsträngige Form überführen. Diesen Vorgang nennt man Renaturierung. Die Geschwindigkeit dieses Vorganges ist von verschiedenen Faktoren abhängig: - von der Konzentration der DNA und der Anzahl gleichartiger Sequenzen - von der Konzentration an Kationen, welche negative Ladungen von Phosphaten neutralisieren - von der Temperatur (die günstigste Temperatur liegt ca. 25°C unter dem Tm) Die Geschwindigkeit der Reassoziation wird als Produkt der DNA-Konzentration Co und der Renaturierungszeit t ausgedrückt, sogenannter cot-Wert. Praktisches Vorgehen bei der Renaturierung • • • die zu untersuchende DNA wird durch Scherkräfte in Fragmente von ca. 500 Nukleotiden zerlegt, so dass ein Vergleich verschieden grosser Genome unabhängig von der DNA-Länge möglich ist: erhitzen auf 100°C damit eine vollständige Trennung erreicht wird Beobachtung der Rückbildung von Doppelstrand-DNA bei 65°C und bei 0.15-0.2 molarer Natrium-Ionenkonzentration, indem man sich den hyperchromen Effekt zu Nutze macht. Die Geschwindigkeit dieser Reaktion hängt nur noch von der Ausgangskonzentration der komplementären Sequenzen C ab. Hybridisierung Der Vorgang der Renaturierung kann erweitert werden, indem man zwei beliebige komplementäre Nukleinsäuresequenzen miteinander paaren und eine doppelsträngige Struktur bilden lässt. Wenn Nukleinsäuren verschiedenen Ursprungs beteiligt sind, dann spricht man von Hybridisierung z.B. RNA mit DNA oder DNA mit strukturell verwandter DNA. Das Prinzip der Hybridisierung besteht darin, zwei Präparationen mit einzelsträngiger Nukleinsäure zu mischen 10 und dann den doppelsträngigen Anteil, der gebildet wurde zu bestimmen. Dieser Vorgang ist die Basis moderner molekularbiologischer Techniken. Hinweis: Kenntnisse des Prinzips der Hybridisierung brauchen Sie später beim Posten „Genklonierung“ Quellennachweis Die Texte und Abbildungen des vorliegenden Scripts entsprechen den Seitenangaben aus folgenden Werken: • Lewin B.: "Gene - Lehrbuch der molekularen Genetik", 2. Auflage; S.65-71; 81-84 • Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D.: "Molekularbiologie der Zelle" 2. Auflage; Seiten 57-63, 108-113, 260-261, 281 • Karlson P., Doenecke D., Koolman J.: "Kurzes Lehrbuch der Biochemie für Mediziner und Naturwissenschafter",14. Auflage, Seite 5, 9, 106-111, 231-235 • Knippers R.: "Molekulare Genetik", 4. Auflage; Seiten 5, 35-45, 49-53 Hinweis: In der „Lese-Ecke“ stehen Ihnen Lehrbücher zum vertieften Studium zur Verfügung Lösungen • Warum bilden Adenin und Guanin in der DNA keine Basenpaare? Adenin und Guanin sind beides Purine. Sie würden in der Helix als Basenpaar zuviel Platz beanspruchen. • Weshalb ist der Schmelzpunkt Tm von Pneumococcus-DNA tiefer als der von Serratia-DNA? Serratia-DNA hat einen höheren Gehalt an den Basen G und C (ca. 60% gegenüber 38% bei Pneumococcus) und ist somit stabiler. Die Folge: mehr Energie in Form von Wärme wird zur Denaturierung benötigt • Welche Bedeutung hat eine Linksverschiebung der Schmelzkurve? Eine Linksverschiebung bedeutet, dass die Stabilität der DNA herabgesetzt ist. Die Folge davon: Denaturierung bei tieferer Temperatur, der Schmelzpunkt Tm ist somit erniedrigt Uebungsaufgaben mit Lösungen finden Sie in der Internetversion des Ateliers! Hinweis: Das „Repetitorium Molekularbiologie“ definiert den Stoff, welcher in den Prüfungen verlangt wird. Wegen seiner Kürze eignet es sich allerdings nicht als primäre Informationsquelle! 11 12 Zellen, Organellen und ihre Nukleinsäuren Prokaryonten Hinweis: Im Atelier koennen Sie sich das Video "Die Prokaryonten" ansehen. Zellstruktur Zu den Prokaryonten gehören Bakterien, Blaualgen und Mykoplasmen. Es sind Organismen, in denen das genetische Material der Zellen (DNA) in Form eines Pronukleus organisiert ist, wobei dieser nicht durch eine Kernmembran vom Cytoplasma getrennt ist. Sie sind einzellige Organismen, die keine Organellen enthalten. a: Kapsel; b: Zellwand; c: Zellmembran; d: Cytosol; e: Nukleoid; f: Flagellen; g: Pili Kapsel Sie ist eine relativ feste Schleimschicht, die vorwiegend aus Polysacchariden aufgebaut ist. Bakterien, die in einen Wirtsorganismus gelangen, werden durch die Kapsel vor Phagozytose geschützt. 1 Zellwand Die Zellwand ist starr und gibt der Zelle mechanische Stabilität. Man unterscheidet zwischen gram-positiven und gram-negativen Zellwänden. Die gram-positiven sind etwa 500 A° dick und bestehen aus ca. 20 Schichten von Peptidoglycanen, die ein Zuckergerüst enthalten, dessen Synthese durch Penicillin gehemmt wird. Penicillin verhindert damit die Vermehrung gram-positiver Bakterien. Die gram-negativen Zellwände haben eine dünnere Peptidoglycanschicht, dafür aber eine weitere Schicht, die aus Lipiden, Proteinen und Lipopolysacchariden besteht. Gram-positive Zellwand: Die Zellwand ist (sehr komplex) aus Makromolekülen, die man Mureine nennt, aufgebaut. Der Grundbaustein ist ein Disaccharid aus N-Acetylglucosamin-NAcetylmuraminsäure. Die Muraminsäure ist ein Aether des N-Acetylglucosamins und enthält Lactat (Nr. 1). Jede zweite Einheit des Polysaccharid-Grundgerüstes bildet mit Lactat an der OHGruppe bei C-3 einen Aether (Nr. 2). Die freie Carboxylgruppe des Lactats bildet ihrerseits Peptidbindungen mit Aminosäuren, die eine Quervernetzung zwischen verschiedenen Polysaccharidketten herstellen. Dies verleiht der Zellwand eine hohe Stabilität. Zellmembran Die Zellmembran umhüllt das Cytoplasma und bildet eine Diffusionsbarriere, die für Ionen, hydrophile Partikel und solche, die ein grösseres Molekulargewicht als 103 Dalton haben, nicht mehr frei durchgängig ist. Da der Stoffwechsel aber von einem ständigen Austausch von Nährstoffen und Stoffwechselendprodukten zwischen Cytoplasma und dem Medium, in dem die Zelle sich aufhält, abhängig ist, verfügt die Zellmembran über selektive Transportsysteme. Membranen sind auch der Ort (einiger) enzymatischer Reaktionen, z.B.die Biosynthese von Phospholipiden oder die oxidative Phosphorylierung. Membranen bestehen etwa zu gleichen Teilen aus Lipiden und Proteinen. Bakterien, die photosynthetisch aktiv sind, haben Thylakoide, d.h. spezialisierte Fortsätze, welche photosynthetisch aktive Pigmente, sowie Elektronentransport- und Photophosphorylierungssysteme enthalten. 2 Transportsysteme der Zellmembran: Membranen sind für hydrophile Moleküle schlecht durchlässig, weil sie einen apolaren Innenbereich haben. Nur sehr kleine, ungeladene Moleküle wie O2, N2, CO2, NH3 und andere Gase, sowie Wasser (obwohl stark polar!), können die Membranen durchdringen. Die Wasserdurchlässigkeit ist enorm wichtig, da Wasser für die Aufrechterhaltung des osmotischen Druckes verantwortlich ist. Dieser Wasserfluss wird durch die Verschiebung von Ionen bewirkt, resp. ist an diese gekoppelt und erfolgt passiv. Für kleinen polare Moleküle besitzen die Zellen spezifische Transportsysteme, mit denen sie Nahrungsstoffe aufnehmen, wasserlösliche Stoffwechselprodukte ausscheiden, Ionenungleichgewichte erzeugen und diese zur Energiegewinnung und Signalübertragung nutzen kann. Mit Hilfe dieser Transportsysteme werden die Membranen selektiv durchlässig. Man spricht von passivem Transport, wenn ein Stoff einem Konzentrationsgradienten folgt, wie z.B. die Aufnahme von Glucose aus dem Blut in die Muskelzelle. Ein aktiver Transport führt gegen einen Konzentrationsgradienten, sozusagen "bergauf", wozu Energie notwendig ist. Wenn die Energie an die Hydrolyse von ATP gekoppelt ist, spricht man von primärem aktivem Transport. Wenn der Transport an den Co-Transport eines anderen Möleküls gekoppelt und von diesem angetrieben ist, spricht man von einem sekundär aktiven Transport. Oxydative Phosphorylierung: Als oxydative Phosphorylierung bezeichnet man eine komplizierte Folge von chemischen Reaktionen, welche die Funktion haben, die chemische Energie, die bei der Oxidation eines Substrates mit O2 entsteht, in chemischer Form von ATP zu speichern. ATP ist also nicht nur Baustein der Nukleinsäuren (in Form von dAMP in DNA, resp. AMP in RNA), sondern auch der wichtigste Energieträger in der Zelle. Cytosol Das Cytosol oder Cytoplasma ist eine "wässrige Salzlösung", die die Zelle ausfüllt, und in der Proteine und Ribosomen zu finden sind. Das grösste Molekül im Innern eines Prokaryonten ist die DNA. Da es in der prokaryontischen Zelle keine Organellen hat, also auch keinen Kern, liegt die DNA frei im Cytoplasma vor. Neben der DNA finden wir auch Plasmid-DNA. Plasmide sind kleine, ringförmige DNA-Moleküle. Ausser DNA finden wir auch verschiedene Arten von RNA, die an Synthesevorgängen beteiligt sind. Nukleoid Da Bakterienzellen, wie vorher erwähnt,keinen Zellkern haben, liegen das Chromosom (ein ringförmiges DNA-Molekül) und die Plasmid-DNA frei im Cytoplasma vor. Die Menge variiert je nach Zellart. Flagellen Wie man aus der schematischen Zeichnung erraten kann, haben die Flagellen etwas mit der Fortbewegung der Prokaryonten zu tun. Es sind Proteinfäden, die schraubenartig verdreht sind und wie eine Schiffsschraube die Bewegung der Zelle steuern. In Verbindung mit spezialisierten Rezeptoren für Aussenreize ermöglichen sie das Ansteuern oder das Vermeiden von Reizquellen (Chemotaxis). Pili Pili sind viel kürzer als Flagellen und haben eine ganz andere Funktion. Sie dienen zur Übertragung von DNA von einer Zelle auf eine andere bei der Konjugation. Sie ermöglichen zudem die Adhäsion einer Zelle an ein Wirtsgewebe. 3 Aufgabe: Wie gross ist das Verhältnis von Nukleinsäuren zu Proteinen (in Bezug auf ihre Masse) in einer prokaryontischen Zelle? Lösung: S. 9 Als Beispiel einer Bakterienzelle: Escherichia coli E. coli ist ein Bakterium, das zu den Colibakterien gehört. Es hat eine Stäbchenform. Seine Grösse ist 1.7x0.65 µm. Es enthält 1 doppelsträngige DNA mit einer Länge von etwa 1.3 mm bestehend aus 4.6x106bp Basenpaaren. E. coli-Zellen lassen sich im Labor gut züchten, weil sie einen einfachen Nährstoffbedarf haben. Sie vermehren sich in einer wässrigen Salzlösung, welche einen Zucker, z.B. Glucose, als Energiequelle enthält. E. coli baut das Kohlenstoffgerüst des Zuckers ab und synthetisiert unter Verwendung von Phosphat, organischem Stickstoff und Sulfat im Medium, Aminosäuren und die Bausteine für Nukleinsäuren. Wenn die E. coli-Zellen genug Nährstoffe haben, verdopplen sie ihre DNA und vergrössern Zellmembran und Zellwand. Nach dem DNA-Verdoppelungszyklus (=Replikation) entsteht eine querverlaufende Scheidewand zwischen den neu entstandenen DNA-Molekülen. Anschliessend teilt sich die Zelle. Die Zeit, die von einer Zelle gebraucht wird, um sich zu verdoppeln, wird Generationszeit genannt. Bei E. coli beträgt diese Zeit unter optimalen Bedingungen (37°C, Vollmedium) 20-25 Min. In Minimalmedium und 37°C beträgt die Generationszeit 60-90 Min. Die kürzere Zeit im Vollmedium rührt daher, dass man neben Glucose auch vorgefertigte Bausteine für Synthesen, beispielsweise für die DNA-Synthese, verabreicht. Ein solches Medium enthält u.a. flüssigen Fleischextrakt. 20-25 Min. ist die Minimalzeit zur Verdoppelung der Zellen, weil Makromoleküle durch die notwendigen Enzyme nicht schneller zusammengebaut werden können. Die Generationszeit einer 500 mal grösseren Säugerzelle beträgt im Vergleich dazu etwa 24 h. 4 Klinik: E. coli ist der häufigste Erreger von Harnweginfektionen, Gallenweg- und Gallenblasenentzündung, Bauchfellentzündung, Blinddarmentzündung, Wundinfektionen und Meningitis (Hirnhautentzündung v.a. bei Säuglingen). Aufgabe: Welche Vorteile hat Fleischextrakt im Nährmedium für E. coli? Lösung: S. 9 Eukaryonten Hinweis: Im Atelier koennen Sie sich das Video "Die Eukaryonten und die Unterschiede zwischen Prokaryonten und Eukaryonten" ansehen. Zu den Eukaryonten gehören die Mitglieder des Tierreiches und des Pflanzenreiches, sowie die Protisten und die Pilze. Die meisten tierischen Zellen haben einen Durchmesser von ungefähr 30-40 µm. Es gibt spezielle Zellen, die viel grösser sind, so z.B. die Muskel- und Nervenzellen (1-40 mm lang, bei einem Durchmesser von 10-40 µm). Im allgemeinen sind eukaryontische Zellen etwa 1000 mal grösser als prokaryontische und viel komplexer aufgebaut. Im Unterschied zu den prokaryontischen Zellen haben sie einen Kern, welcher von einer Doppelmembran umgeben ist. Der Kern enthält die DNA. Es gibt noch weitere Kompartimente im Cytosol, welche von einer Membran umgeben sind. Sie werden als Organellen bezeichnet. Zellkern Der Zellkern ist von einer Doppelmembran umgeben. Jede der beiden Membranen besteht aus einer Lipiddoppelschicht, die wie die Einheitsmembran aufgebaut ist. Es gibt Stellen, an welchen die äussere und die innere Membran mit Proteinen zusammengehalten werden, sogenannte Poren. Diese "Löcher" machen bei einem Durchmesser von 600-700 A° bis zu 10% der Kernoberfläche aus. Die Poren dienen dem Transport von verschiedenen Materialien aus dem Kern ins Cytoplasma und umgekehrt. Der Zellkern ist der Aufbewahrungsort der genetischen Informationen, die in dicht gepackten Knäueln von DNA-Fäden, den Chromosomen gespeichert wird. Eine eukaryontische Zelle enthält etwa 10-1000 mal mehr DNA als eine prokaryontische Zelle. Die morphologisch auffälligsten Strukturen des Zellkernes sind die Nukleolen, in denen die ribosomale RNA-Synthese und der 5 Zusammenbau von Ribosomen stattfindet. Der Kern ist vor allem der Ort, in welchem die DNAReplikation, die DNA-Transcription und das RNA-Splicing stattfinden. Zellmembran Membranen sind charakteristische Strukturen , die aus Lipiden aufgebaut sind, und in denen Proteine eingelagert sind. Eine Plasmamembran enthält etwa die gleiche Gewichtsmenge an Lipiden und Proteinen, während innere Membranen (z.B. Mitochondrien) mehr Proteine enthalten. Das typische Membranmilieu entsteht durch die amphipathischen Eigenschaften der meisten Lipide: das eine Ende des Moleküls besteht aus einem hydrophilen Kopf, das andere Ende aus einem hydrophoben Schwanz. Diese Lipide sind bestrebt, im wässrigen Milieu den Kopf zum Wasser zu strecken und den Schwanz gegen das Wasser abzuschirmen. Das erreichen sie, indem sie flache Lipiddoppelschichten bilden, in welchen die Köpfe nach aussen und die Schwänze nach innen ragen und dort ein hydrophobes Milieu schaffen. Eine der wichtigsten Eigenschaften der Membranlipide ist ihre Fähigkeit, sich in der Doppelschicht bewegen zu können (Fluidität). Die wichtigsten Faktoren, die die Fluidität der Membran beeinflussen, sind Art und Länge der Fettsäuren. Je gesättigter die Fettsäuren, desto starrer die Membran. Die Plasmamembran, die die Zelle umschliesst, stellt die Grenze zwischen zellulärem Milieu im Innern und der Umgebung dar, welche bei einem Vielzeller auch aus weiteren Zellen bestehen kann. Mit den eingelagerten spezifischen Transportproteinen reguliert sie den Fluss grosser und kleiner Moleküle. Eine weitere wichtige Gruppe von Proteinen sind die Rezeptoren. Sie dienen der Kommunikation zwischen Zellen und haben auf der Membranaussenseite eine Struktur, welche einen Liganden erkennt. Die Bindung des Liganden verändert die räumliche Struktur des Proteins, welches aufgrund dieser Aenderung weitere biochemische Veränderungungen in der Zelle bewirkt. Man bezeichnet diesen Vorgang als Signalübertragung. Im Gegensatz zu den Prokaryonten sind die Enzyme der oxidativen Phosphorylierung nicht in der Zell- Membran lokalisiert, sondern in der inneren Membran der Mitochondrien. Cytoplasma Das Cytoplasma der eukaryontischen Zelle ist im Prinzip ähnlich zusammengesetzt wie das Cytoplasma der Prokaryonten: es ist eine sehr konzentrierte Proteinlösung, welche Salze enthält. Viele Reaktionen der Eukaryonten laufen jedoch im Gegensatz zu den Prokaryonten in Organellen ab, also in Kompartimenten, welche durch eine Membran vom Cytoplasma getrennt sind. Organellen Im Cytoplasma von eukaryontischen Zellen gibt es Strukturen, die von einer Membran umgeben sind und so Kompartimente bilden, welche unterschiedliche Stoffwechselaktivitäten aufweisen können. Zu diesen Kompartimenten gehören die folgenden Organellen: Mitochondrien, Chloroplasten, rauhes und glattes endoplasmatisches Retikulum, Golgi-Apparat, Peroxysomen und Lysosomen. Sie werden im Folgenden noch etwas ausführlicher besprochen: 6 Mitochondrien In einer Zelle finden wir gewöhnlich 100-1000 Mitochondrien, die einen Durchmesser von 0,2 - 1 µm haben. Es sind Organellen der Zellatmung, deren Funktion in der Umwandlung von Nahrungsstoffen, wie Kohlenhydrate, Fette etc in brauchbare Energie liegt. Sie sind also gewissermassen die "Kraftwerke der Zelle". Über die Enzyme der Atmungskette und damit der oxidativen Phosphorylierung wird ATP synthetisiert. Es handelt sich dabei um eine Folge von Elektronen-Transfer-Reaktionen, die einen H+-Gradienten zwischen der Aussen- und der Innenseite der inneren mitochondrialen Membran schaffen. Dieser H+-Gradient dient dazu, die ATP-Synthese anzutreiben. ATP ist der Hauptenergieträger in einer lebenden Zelle. Mitochondrien haben eine äussere und eine innere Membran. Die äussere Membran, welche das Mitochondrium umgibt, hat Membrankanäle, welche relativ frei permeabel sind. Die innere Membran, welche eingestülpt ist und Cristae bildet, ist ein perfekter Isolator. Über sie wird der oben beschriebene elektrochemische Gradient aufgebaut. Die innere Membran besteht zu 20% aus Lipiden und zu 80% aus Proteinen. An diesen Proteinen findet die oxidative Phosphorylierung statt Ein Teil der Proteine der Mitochondrien wird nach einem genetischen "Rezept" hergestellt, das sich im Matrixraum der Mitochondrien selbst befindet. Die Mitochondrien verfügen also über eigene DNA-Moleküle, welche die Informationen für ca. 20 mitochondriale Proteine gespeichert haben. Dies entspricht in etwa 5% der gesamten Syntheseleistung, die für die Aufrechterhaltung des Stoffwechsels der Mitochondrien nötig ist. Diese für die Mitochondrien-Proteine kodierenden DNA-Moleküle werden auch in den Mitochondrien hergestellt, während der grösste Teil der sich in den Mitochondrien befindlichen Proteine im Zellkern kodiert ist, im Cytoplasma transliert wird und dann über Membranproteine in die Mitochondrien aufgenommen wird. Chloroplasten Chloroplasten sind linsenförmige Organellen, die mit einer Länge von 5 µm und einem Durchmesser von 2-3 µm relativ gross sind. Sie sind von einer doppelten Membran umgeben, welche sie gegen das Cytoplasma der Pflanzen- bzw. der Algenzellen abgrenzt. Der Innenraum wird Stroma genannt, und in ihm finden wir die Enzyme der CO2-Fixierung und des CalvinZyklus (Aufbau von Kohlenhydraten). Das Stroma ist durchzogen von Thylakoidmembranen, die in der gestapelten Form als Grana bezeichnet werden. 7 Wie die Mitochondrien sind auch die Chloroplasten energieproduzierende Organellen. Sie kommen in Pflanzen und Grünalgen vor, in denen sie mittels Photosynthese ATP produzieren. Die Multi-Enzym-Komplexe der Lichtreaktion sind wie der ATP-Synthase-Komplex an den Thylakoidmembranen lokalisiert. Wie die Mitochondrien enthalten auch die Chloroplasten ihre eigene DNA, die für ca. 30 Chloroplastenproteine, die ribosomale RNA und etwa 30 tRNAs kodiert. Der grosse Teil der Proteine wird jedoch wie bei den Mitochondrien im Kern kodiert. Endoplasmatisches Retikulum Das endoplasmatische Retikulum (ER) stellt die Hauptmasse an Membranen in einer eukaryontischen Zelle dar. Es ist ein stark verwinkeltes "Membranlabyrinth". Das Labyrinth kommt durch die Faltung einer einzigen Lipiddoppelschicht zustande, welche das ER-Lumen umschliesst, und die an der äusseren Kernmembran entspringt. Man kann zwei Typen von ER unterscheiden, das rauhe und das glatte ER. Das rauhe ER ist dadurch charakterisiert, dass an seine Aussenfläche Ribosomen geheftet sind. Die Ribosomen sind an der Proteinsynthese beteiligt. Golgi-Apparat Der Golgi-Apparat liegt zwischen ER und Plasmamembran. Er besteht aus plattenartigen Zisternen. Der Golgi-Apparat ist stark polarisiert, die cis-Seite steht mit dem ER in Verbindung, während die trans-Seite zur Plasmamembran hin gerichtet ist. Im ER modifizierte Proteine treten auf der cis-Seite in den Golgi-Apparat ein und werden auf ihrem Weg zur trans-Seite weiter modifiziert und dann freigesetzt. ER und Golgi-Apparat haben also die Funktion, Proteine zu sortieren, zu modifizieren und dann ihrem "Verbraucher" abzugeben. Da die Membran fluid ist, können die Proteine in Bläschen (Vesikel) verpackt werden und aus der Zelle ausgestossen werden (Exocytose). Lysosomen Lysosomen sind kleine, kugelförmige, membranumhüllte Körperchen, welche hydrolytische Enzyme enthalten, die dem Abbau von Makromolekülen dienen. Sie werden am Golgi-Apparat gebildet, der die Enzyme in Membranen hüllt und diese abschnürt. Die einzelnen Vesikel können dabei unterschiedliche Enzyme enthalten. Peroxysomen Sie sind Organellen von ähnlicher Grösse wie die Lysosomen und ebenfalls membranumhüllt. Das "Hauptenzym", das in ihnen gefunden wird, die Katalase, ist mit anderen Enzymen an einer Reihe von Oxidationen beteiligt. Cytoskelett Je komplizierter eine Zelle aufgebaut ist, desto wichtiger sind Strukturen, die die verschiedenen Kompartimente an der richtigen Stelle halten. Alle Eukaryonten haben ein inneres Skelett, das Cytoskelett, das der Zelle ihre Form aber auch die Fähigkeit zur Bewegung gibt. Es ist ebenfalls dazu befähigt, Organellen zu verschieben und zu positionieren. Es besteht aus einem Netzwerk von Filamenten. Die wichtigsten sind Aktin-Filamente, intermediäre Filamente und Mikrotubuli. 7 Zellwand Zellwände werden nur bei den niederen Eukaryonten wie Protozoen, Hefe- und Pflanzenzellen gefunden. Die Säugetierzellen dagegen weisen eine kompakte Glycoproteinschicht auf, welche Funktionen bei der Zel-Zell-Erkennung hat. Quellennachweis • Lewin B.: "Gene - Lehrbuch der molekularen Genetik", 2. Auflage; Seiten 20-35 • Alberts B., Bray D., Lewis J., Raff M., Roberts K., Watson J.D.: "Molekularbiologie der Zelle", 2. Auflage, Seiten 9-24, 470-473 • Karlson P., Doenecke D., Koolman J.: "Kurzes Lehrbuch der Biochemie für Mediziner und Naturwissenschafter", 14. Auflage, Seiten 380-384 • Knippers R.: "Molekulare Genetik", 4. Auflage; Seiten 15-18, 23-31, 323-324 Hinweis: In der „Lese-Ecke“ stehen Ihnen Lehrbücher zum vertieften Studium zur Verfügung Lösungen • Wie gross ist das Verhältnis von Nukleinsäuren zu Proteinen (in Bezug auf ihre Masse) in einer prokaryontischen Zelle? 7% Nukleinsäuren stehen etwa 15% Proteine gegenüber (nach Massenprozenten). Zahlenmässig sind es etwa 1 000 000 Proteine und 460 000 Nukleinsäuren. • Welche Vorteile hat Fleischextrakt im Nährmedium für E. coli? Fleischextrakt enthält vorgefertigte Bausteine für Nukleinsäure- und Proteinsynthese. Da die E. coli-Zellen in einem solchen Nährmedium diese Bausteine nicht synthetisieren müssen, sparen sie Zeit, und so verkürzt sich die Generationszeit. Uebungsaufgaben mit Lösungen finden Sie in der Internetversion des Ateliers! Hinweis: Das „Repetitorium Molekularbiologie“ definiert den Stoff, welcher in den Prüfungen verlangt wird. Wegen seiner Kürze eignet es sich allerdings nicht als primäre Informationsquelle! 7 7 Zellteilung Chromosomen Hinweis: Im Atelier finden Sie die CD "The Nature of Genes". Mittels Tutorials und Aufgaben werden die wichtigsten Themen der Molekularbiologie leicht verständlich vermittelt. Chromosomen sind die Träger der genetischen Information. Jede menschliche Körperzelle enthält 46 Chromosomen, wovon eine Hälfte mütterlicher, die andere väterlicher Herkunft ist. Bei der Vereinigung einer männlichen mit einer weiblichen Keimzelle verschmelzen nämlich zwei einfache (haploide) Chromosomensätze mit je 23 einzelnen Chromosomen. Es resultiert eine befruchtete Eizelle mit einem doppelten (diploiden) Chromosomensatz aus 46 Chromosomen, welche bei den folgenden Zellteilungen an neue Generationen weitergegeben werden. Jedes Chromosom hat also ein homologes (d.h. ein ihm entsprechendes) Chromosom vom andern Elter; man spricht deshalb vereinfachend von 23 Chromosomenpaaren, obwohl dies streng genommen nur für 44 der 46 Chromosomen, die sogenannten Autosomen, zutrifft. Die zwei Geschlechtschromosomen (auch: Gonosomen oder Heterosomen) beim männlichen Geschlecht tragen nämlich die Bezeichnung X und Y, womit angedeutet ist, dass die beiden Chromosomen morphologisch nicht identisch sind. Tatsächlich ist das der Samenzelle entstammende YChromosom viel kleiner als das X-Chromosom aus der Oocyte. Frauen tragen zwei gleichgrosse X-Chromosomen und sind homogametisch, Männer wegen der XY-Konstellation folglich heterogametisch. Chromosomensätze anderer Spezies Folgende Aufstellung zeigt, dass auch zwischen einander verwandten Arten die Variabilität im Chromosomensatz sehr gross sein kann: Spezies Hefe Mais Reis Zahl der Chromosomen (diploid) 32 20 24 1 Weizen Tabak Farn-Arten Drosophila Hausfliege Ameise Frosch Karpfen Hund Katze Maus Ratte Rind Rhesusaffe Mensch 42 48 >600 8 12 48 26 104 38 64 40 42 60 42 46 Um bei einer beliebigen Zelle die Art des Chromosomensatzes (haploid oder diploid) sowie ihren DNA-Gehalt angeben zu können, hat man folgende Schreibweise eingeführt: Einen haploiden Chromosomensatz bezeichnet man mit n, einen diploiden mit 2n, wobei n die Anzahl Chromosomen darstellt (beim Menschen ist n=23). Unter 1c versteht man den DNA-Gehalt einer haploiden Zelle. Eine normale Körperzelle erhält also das Kürzel 2n, 2c. Für eine Keimzelle gilt entsprechend: 1n, 1c. Nicht immer ist n=c: Vor jeder Teilung verdoppelt nämlich eine Zelle ihren DNA-Gehalt durch Replikation. Eine Zelle, die diese Synthese bereits durchlaufen hat, sich aber noch nicht geteilt hat, beschriftet man mit 2n, 4c. Achtung! Einige Lehrbücher bezeichnen den DNA-Gehalt mit n statt mit c, was immer wieder grosse Verwirrung stiftet. Wenn Sie sich aber an obige Abmachungen halten, werden Sie keine Probleme beim Studieren dieser Unterlagen haben. Wie sind Chromosomen aufgebaut? Die 46 Chromosomen eines Menschen sind verschieden lange DNA-Stücke, die mit bestimmten Proteinen assoziiert zu Strukturen höherer Ordnung organisiert sind. Die DNA ist um HistonOktamere gewickelt, und diese Nucleosomen sind weiter zu einer 30-nm-Faser, dem Solenoid, verdichtet. Die Ausbildung von Schleifen ermöglicht schliesslich die Integration des Chromatins im Zellkern. Die beschriebenen Superstrukturen reichen aber nicht aus, um den Kräften, die sich bei Zellteilungen ergeben, genügend Widerstand zu leisten. Deshalb müssen die Chromosomen während der Pro- und Prometaphase zu stabilen Metaphasen-Chromosomen kondensiert werden. Die genauen Vorgänge, die zu dieser Spiralisation führen, sind noch weitgehend unbekannt. Folgende Abbildung gibt aber wenigstens Auskunft über Prinzip und Ausmass der Kondensationen: 2 Mitose-Chromosomen (Abb. unten), d.h. maximal kondensierte Metaphase-Chromosom, bestehen wegen der bereits erfolgten DNA-Verdoppelung aus zwei Chromatiden, welche bis zu ihrer Trennung in der Mitose am Centromer zusammenhängen. Die kürzeren Abschnitte werden mit p, die längeren mit q bezeichnet. Am Centromer binden Proteine, welche das sogenannte Kinetochor (Abb. unten) bilden, die Ansatzstelle für die Spindelfasern. Centromer und Kinetochor wurden früher als Synonyme gehandelt, jetzt weiss man aber, dass es sich um verschiedene Strukturen handelt. Die Enden eines Chromosoms nennt man Telomere. 3 Anhand der Lage des Centromers unterscheidet man metazentrische, submetazentrische und acrozentrische Chromosomen: Metazentrische Chromosomen haben ihr Centromer in der Mitte, submetazentrische Chromosomen etwas weiter oben. Bei acrozentrischen Chromosomen liegt das Centromer so nahe beim Telomer, dass nebst einem langen höchstens noch ein sehr kurzer Arm erkennbar ist, den man Satellit nennt. Die Satelliten-DNA der fünf acrozentrischen Chromosomen 13, 14, 15, 21 und 22 ist an der Organisation des Nucleolus beteiligt. Sie trägt Gene, welche für die ribosomale RNA kodieren. Bei einigen Organismen kommen noch telozentrische Chromosomen vor. DNA-Gehalt in Chromosomen Die 3x109 Basenpaare der DNA einer haploiden menschlichen Zelle sind auf 23 unterschiedlich grosse Chromosomen verteilt, wie folgende Tabelle zeigt: Chromosom-Nr. 1 2 3 4 5 6 7 8 9 10 11 12 Basenpaare (in Mio.) 263 255 214 203 194 183 171 155 145 144 144 143 Chromosom-Nr. 13 14 15 16 17 18 19 20 21 22 X 4 Basenpaare (in Mio.) 114 109 106 98 92 85 63 72 50 60 164 Das Karyogramm und der Karyotyp Chromosomen lassen sich durch ihre Grösse, die Lage des Centromers und durch ihr spezifisches Bandenmuster eindeutig voneinander unterscheiden. Die Chromosomen des Menschen werden bestimmten Gruppen zuordnet: Gruppe A (Nr. 1-3), Gruppe B (Nr. 4-5), Gruppe C (Nr. 6-12), Gruppe D (Nr. 13-15), Gruppe E (Nr. 16-18), Gruppe F (Nr. 19-20), und Gruppe G (Nr. 21-22). Die Geschlechtschromosomen werden gesondert behandelt, obwohl sie nach obigen Kriterien sehr wohl klassifiziert werden könnten: das X-Chromosom zu Gruppe C und das Y-Chromosom zu Gruppe G. Für genetische Fragestellungen werden die Metaphasen-Chromosomen geordnet dargestellt. Eine solche Darstellung bezeichnet man als Karyogramm und das charakteristische Bild des Chromosomensatztes als Karyotyp. Ein normaler weiblicher Karyotyp des Menschen wird mit 46, XX bezeichnet, ein normaler männlicher Karyotyp mit 46, XY. Wie erstellt man ein Karyogramm? Zu allererst muss eine genügend grosse Zahl sich teilende Zellen gewonnen werden. Zellen können nötigenfalls mit mitogenen Stoffen (meist Lectine) zur Teilung angeregt werden. Da ein Karyogramm aus Chromosomen im Metaphasenstadium besteht, müssen noch Metaphasenblocker zugegeben werden. Das Alkaloid Colchicin eignet sich dafür ausgezeichnet: es depolymerisiert die Mikrotubuli und bewirkt so eine Ansammlung von mitotischen Metaphasestadien in der Zellkultur. Die Zellen werden daraufhin durch hypotonen Schock aufgeschlossen und die Chromosomen auf einem Objektträger fixiert. Standardfärbungen (Orcein, Feulgen oder Giemsa), wie sie früher verwendet wurden, geben nur Auskunft über Genommutationen (Polyploidien, Aneuploidien, siehe Posten "Mutation, Mutagenese und Reparaturmechanismen"). Will man auch bestimmte Strukturaberrationen von Chromosomen 5 diagnostizieren, müssen moderne Bandfärbungstechniken angewendet werden, mit welchen auch die Zuordnung der Chromosomen zu einzelnen Gruppen eindeutig gelingt. Im mikroskopischen Bild liegen die Chromosomen in ungeordneten Gruppierungen. Zur Kartierung in einem Karyogramm stellt man Fotographien der Chromosomengruppen her, schneidet die Bilder der Chromosomen aus und ordnet sie in Reihen an. Geschlechtsbestimmung Eines der beiden X-Chromosomen ist in Zellen von weiblichen Individuen hochkondensiert und genetisch inaktiv. Es liegt als sog. "Barr-Körperchen" der Kernmembran innen an und kann mit einiger Übung im Lichtmikroskop (Pfeil) erkannt werden: Quinacrin ist ein Fluoreszenzfarbstoff mit grosser Affinität zum Y-Chromosom. Somazellen eines Mannes sind somit unter Zugabe von Quinacrin leicht an einem kleinen Punkt - eben dem Y-Chromosom - erkennbar: 6 Mitose Bei der Mitose (Kernteilung) wird das gesamte Genmaterial einer Zelle erbgleich an zwei Tochterzellen weitergegeben. Solche Zellteilungen sind nötig, wenn ein Organismus durch Zellvermehrung wachsen will oder wenn er zugrunde gegangene Zellen ersetzen muss. Hinweis: Im Atelier koennen Sie sich das Video "Mitose" ansehen. Der Zellzyklus Zellen, die sich teilen, durchlaufen eine regelmässige Abfolge von Zuständen, welche sich von Generation zu Generation wiederholen. Man spricht deshalb von einem Zellzyklus. Die eigentliche Zellteilung, welche als M-Phase bezeichnet wird, macht nur einen kleinen Teil des Zyklus aus. Die Interphase, die Zeit zwischen zwei Zellteilungen, beansprucht dafür etwa 95 % des ganzen Zellzyklus. Sie setzt sich aus G1-Phase, G2-Phase (G für Gap, Lücke, da keine DNASynthese nachzuweisen ist) und dazwischenliegender S-Phase (S für DNA-Synthese) zusammen. Die Dauer der einzelnen Phasen ist stark vom Zelltyp und den äusseren Bedingungen abhängig. Schematische Darstellung des Zellzyklus: Die angegebenen Zeiten gelten für eine MausHepatom-Zelllinie. 7 Wie bestimmt man die Dauer der einzelnen Zyklusphasen? Die Dauer einer Zyklusphase entspricht ungefähr dem Bruchteil an Zellen, die sich zu einem beliebigen Zeitpunkt in dieser Phase befinden, multipliziert mit der Gesamtdauer des Zyklus. Aber wie weiss man, in welcher Phase sich eine Zelle befindet? Radioaktiv markiertes [3H]Thymidin kann in DNA, nicht aber in RNA eingebaut werden. Gibt man nun einer Zellkultur für unterschiedlich lange Zeiträume [3H] -Thymidin-Lösung zu, so lassen sich die Zellen, die während der S-Phase [3H] -Thymidin in die DNA eingebaut haben, mittels Autoradiographie bestimmen. Mikroskopisch muss noch ermittelt werden, wieviele Mitosezellen sich unter den radioaktiv markierten Zellen befinden. Das Prinzip der Zellteilung Eine Zelle, die sich teilen will, muss in der S-Phase ihren DNA-Gehalt verdoppeln. Im Verlaufe der M-Phase trennen sich die beiden Chromatiden eines jeden Chromosoms und wandern zu entgegengesetzten Zellpolen. Damit ist sichergestellt, dass die bei der darauffolgenden Teilung entstehenden Tochterzellen dasselbe Erbgut wie ihre Mutterzelle erhalten. In dieser Unterlage wird bei einigen Abbildungen eine spezielle Schreibweise für Chromosomen verwendet, die sich zum Verständnis von Mitose und Meiose gut eignet. Sie werden wahrscheinlich später immer wieder auf folgende Darstellung der Syntax zurückgreifen müssen: 8 Beispiele: 11m und 11w bilden ein Chromosomenpaar. 11m ist also das homologe Partnerchromosom von 11w. zeigt Chromosom 6 männlicher Herkunft, das seine DNA repliziert hat. Es besteht aus zwei Chromatiden (6mα bzw. 6mβ), welche am Zentromer miteinander verbunden sind. zeigt Chromosom 9w, das mit seinem homologen Partnerchromosom 9m in der Prophase I der Meiose einen Komplex bildet. In diesem Komplex findet homologe Rekombination statt. zeigt Chromosom 17 weiblicher Herkunft nach einem Crossing-overEreignis: Chromatid 17w hat eine homologe Rekombination mit seinem Partnerchromosom 17m (entweder mit Chromatid α oder β ) durchgeführt. Chromatid 17wβ hingegen hat kein Crossing-over erfahren. Wie wird die Mitose gesteuert ? Damit eine Zellteilung reibungslos abläuft, sind Steuersignale nötig, die ungewollte Aktionen verhindern und stattdessen ein koordiniertes Nebeneinander vieler Reaktionen gewährleisten sollen. So soll etwa der Eintritt in die M-Phase verwehrt werden, wenn die Zelle ihre DNA-Replikation noch nicht abgeschlossen hat. Unter anderen sind folgende Steuersignale bekannt: - der S-Phase-Aktivator: er setzt die DNA-Replikation in Gang. - der M-Phase-Verzögerungsfaktor: er ist möglicherweise mit dem S-Phase-Aktivator identisch und verhindert den Übertritt von der G2- in die M-Phase, indem er die Synthese des M-PhaseFörderfaktors unterdrückt. - der M-Phase-Förderfaktor MPF (engl. „Mitose-promoting-factor"): er ist eine Kinase und leitet durch Phosphorylierung verschiedener Proteine (darunter das Histon H1) die Prophase der Mitose ein. 9 Eine Zelle kann nur dann die Mitose beginnen, wenn in der G2-Phase genügend MPF produziert worden ist. MPF kann erst synthetisiert werden, wenn die Wirkung des M-Phase-Verzögerungsfaktors abgeklungen ist, und dieser kann zusammen mit dem S-Phase-Aktivator erst verschwinden, wenn die DNA-Synthese vollendet ist. Wie wird die Mitose gesteuert? Experimentelle Befunde bestätigen die Regelmechanismen:: • • Eine Zelle A im G2-Stadium kann durch Fusion mit einer Zelle B im S-Stadium daran gehindert werden, in die M-Phase überzutreten. Interpretation: offenbar wird während der S-Phase ein M-PhaseVerzögerungsfaktor produziert, der verhindert, dass sich eine Zelle mit nicht replizierter DNA (fatal!) teilt. Fusioniert man eine Zelle A, welche sich in der M-Phase befindet, mit einer Zelle B in der Interphase (G1-, S- oder G2-Phase), so beginnt sich Zelle B zu teilen, obwohl sie unter Umständen ihre DNA noch gar nicht repliziert hat! Interpretation: Zelle A enthält den sehr potenten M-Phase-Förderfaktor (MPF), der in jeder Phase des Zellzyklus wirkt und in Zelle B eine Mitose auslöst. Obwohl der MPhase-Verzögerungsfaktor die Synthese von MPF normalerweise verhindert, bis die gesamte DNA repliziert ist, kann er gegen einen künstlich zugegebenen MPF nichts ausrichten. Prophase Die durch die MPF katalysierte Phosphorylierung von Laminin führt zur Auflockerung und schliesslich zur Auflösung der Faserschicht um den Kern. Es wird angenommen, dass analog dazu die Phosphorylierung des Histons H1 die Chromosomenkondensation herbeiführen könnte. Mit der langsam fortschreitenden Verdichtung der Chromosomen löst sich auch der Nucleolus auf. Die beiden Zentriolenpaare trennen sich zunehmends voneinander und wandern der Aussenseite der Kernmembran entlang auf entgegengesetzte Seiten. Die Mikrotubuli, die in der Interphase noch Bestandteil des Cytoskeletts waren, zerfallen und organisieren sich als Polstrahlen, welche sternförmig von den Zentriolenpaaren ausgehen. Am Centromer eines jeden Chromosoms haften verschiedenste Proteine und bilden gegen Ende der Prophase an jedem Chromatid ein Kinetochor aus. Prometaphase Die beginnende Auflösung der Kernmembran in kleine Vesikel markiert den Übergang von der Prophase in die Prometaphase. Dadurch, dass die das Kerngebiet schützende Hülle langsam verschwindet, können die Polstrahlen auch zu den Chromosomen vordringen. Einige haften an die Kinetochoren und werden deshalb als Kinetochormikrotubuli bezeichnet. Mikrotubuli, die zwar an der Ausbildung der Spindel teilhaben, aber nicht an die Chromosomen gebunden sind, nennt man Polmikrotubuli. Mikrotubuliassoziierte Proteine binden an die Enden von solchen Polmikrotubuli, welche von verschiedenen Zentriolen ausgehen, und schützen diese vor spontanem Zerfall. Astraltubuli sind ganz kurze 10 Mikrotubuli, welche gar nicht zu den Chromosomen gelangen, sondern auf die nahgelegene Zellmembran weisen. Die beiden Kinetochoren eines Chromosoms müssen über Kinetochormikrotubuli mit verschiedenen Spindelpolen verbunden werden. Dadurch ist nämlich gewährleistet, dass später die Schwesterchromatiden auch wirklich in verschiedene Tochterzellen gelangen. Die Prometaphase ist ein Zeitraum heftiger Aktivität: Die Chromosomen, die sich weiter verdichten, werden - an den Mikrotubuli aufgehängt - wie Marionetten auf eine Ebene gelenkt. Die Zugkräfte, die auf die Chromosomen wirken, sind nämlich umso grösser, je grösser der Abstand zwischen Kinetochor und Zentriole ist. Dadurch wird nach einigem Hin- und Herpendeln ein stabiler Gleichgewichtszustand in der Metaphasenplatte gefunden. Metaphase Auch jetzt, wo sich die maximal kondensierten (Metaphase-) Chromosomen auf der Metaphasenplatte oder Äquatorialebene befinden, wirken noch starke Zugkräfte. Ein Experiment zeigt es: Trennt man die Verbindung zwischen Kinetochor und Zentriole auf nur einer Seite mit einem Laser durch, so bewegen sich die Chromosomen zu dem Spindelpol, mit dem sie noch verbunden sind. Die Metaphase dauert verhältnismässig lange und ist deshalb auch im Mikroskop oft sichtbar. Man nimmt an, dass Chromosomen, deren Kinetochoren noch nicht an Mikrotubuli gekoppelt sind, ein Signal aussenden, welches die Trennung der Chromatiden und damit die Anaphase hinauszögert. In der Praxis werden Metaphase-Chromosomen zur Erstellung eines Karyogramms analysiert. Anaphase In der Anaphase trennen sich die Chromatiden abrupt voneinander und werden von den Kinetochormikrotubuli zu den beiden Zellpolen gezogen. Die Segregation der Schwesterchromatiden wird durch Anstieg der Ca2+-Konzentration im Cytosol initiiert. 11 Experimente zur Anaphase • Kappt man die Verbindung zwischen Kinetochor und Centrosom auf beiden Seiten der Metaphasenplatte, so trennen sich die Chromatiden immer noch voneinander. • Injiziert man einer teilungsfähigen Zelle das Protein Aequorin, so beobachtet man während der Anaphase einen Calciumanstieg um das Zehnfache des Normalwerts.Aequorin kann zur Messung von Calcium verwendet werden, weil es in Anwesenheit von Calcium Lichtblitze emmitiert. • Eine Anaphase kann frühzeitig ausgelöst werden, indem man einer Zelle im Metaphasenstadium Calcium von Aussen zugibt. • In der Nähe der Spindelpole konnten Membranvesikel gefunden werden, welche reich an Calcium waren. Es wäre möglich, dass diese Vesikel - analog dem sarkoplasmatischen Retikulum in Muskelzellen - die nötigen Calciumionen bereitstellen. Zwei Mechanismen bewirken nun die Verschiebung der getrennten Chromatiden zu den Zellpolen: In der Anaphase A verkürzen sich die Kinetochormikrotubuli unter Dissoziation der Mikrotubuliuntereinheiten an dem dem Kinetochor zugewandten Ende, so dass sich die Chromosomen zwangsläufig den Zentriolen nähern müssen. Die Anaphase B ist gekennzeichnet durch das Längenwachstum der Polmikrotubuli. Da entgegengesetzt polarisierte Polmikrotubuli in der Metaphasenplatte durch Proteine miteinander verbunden sind, kann man sich leicht vorstellen, dass sich durch ständigen Zuwachs an Mikrotubuliuntereinheiten die beiden Zentriolen voneinander „abstossen". Hinzu kommt, dass die Spindelpole offenbar unter Einwirkung der Astralmikrotubuli an die Zellmembran gelenkt werden. Die Kinetochormikrotubuli sind in der späten Anaphase fast völlig zerfallen und die Chromosomen somit an ihrem Bestimmungsort, den Polen, angelangt. Telophase In der Telophase bildet sich um jede der beiden Chromosomengruppen durch Fusion von Vesikeln eine neue Kernmembran und die Chromosomen werden zum Interphasen-Chromatin entspiralisiert. Die RNASynthese wird wieder in Gang gesetzt und neue Proteine werden synthetisiert. Der Nucleolus als Ort der rRNA-Synthese ist nun mikroskopisch wieder zu sehen. Die RNA-Synthese wird wieder in Gang gesetzt und neue Proteine werden synthetisiert. Der Nucleolus als Ort der rRNA-Synthese ist nun mikroskopisch wieder zu sehen. 12 Cytokinese Obwohl die Cytokinese, die Teilung des Zytoplasmas, schon in der Anaphase beginnt, wird sie aus didaktischen Gründen erst jetzt behandelt. Die Teilung der Zelle in zwei Tochterzellen erfolgt durch einen kontraktilen Ring, der Actin und Myosin enthält. Ein schwaches Verbindungsstück, der Mittelkörper, bleibt noch eine Weile als dünne Brücke zwischen den beiden Zellen bestehen. Er enthält die überlappenden Mikrotubuli. Weil sich die Teilungsfurche immer auf Höhe der Äquatorialebene ausbildet, ist gewährleistet, dass jede künftige Tochterzelle nur einen Kern erhalten wird. Meist liegt die Metaphasenplatte gerade in der Mitte der Mutterzelle, und es entstehen so zwei gleich grosse Tochterzellen; in bestimmten Fällen können sich durch asymmetrische Lage der Spindel zwei unterschiedlich grosse Zellen entwickeln. Lage der Teilungsfurche Folgende Experimente zeigen, dass die Mitosespindel die Lage der Teilungsfurche bestimmt: • Eine beginnende Furchung erkennt man an einer leichten „Runzelung“ der Zellmembran. Verändert man in einer solchen Zelle die Lage der Spindel, so verschwindet die Furchung wieder und es bildet sich eine neue dort, wo sich nun die Spindel befindet. • Wenn man die Spindel einer Mitosezelle zur Seite schiebt, bildet sich eine einseitige Furchung aus und es entsteht eine zweikernige Zelle: 13 Die genauen Mechanismen, die zur Trennung der Mutterzelle in zwei Tochterzellen führen, sind noch nicht in allen Details bekannt. Fest steht aber, dass dabei grosse Kräfte wirken: So wird z. B. eine in die Zelle eingeführte feine Glasnadel durch den Druck des immer enger werdenden Rings entzwei gebrochen! Hinweis: Sehen Sie sich im Atelier einige Mitosen an. Können Sie die verschiedenen Phasen zuordnen? Denken Sie daran, dass die Metaphase am längsten dauert und deshalb viele Zellen in diesem Stadium zu finden sind. Entsprechend werden Sie weniger Zellen in der Anaphase entdecken. Zellorganellen und Zellteilungen In vielen Lehrbüchern wird zwar ausführlich über das Verhalten der Chromosomen bei Zellteilungen berichtet, wie es sich aber mit andern Zellorganellen - die ja volumenmässig den grösseren Teil ausmachen und für eine Zelle von ebensogrosser Wichtigkeit sind - auf sich hat, wird oft verschwiegen. In diesem Abschnitt wird kurz geschildert, wie einige wichtige Zellorganellen auf die Tochterzellen verteilt werden: Kernhülle: Wie schon oben gezeigt, löst sie sich am Ende der Prophase in kleine Vesikel auf. In der Telophase fusionieren diese um jede Chromosomengruppe wieder zu zwei vollständigen Kernmembranen. Zentriolen: In der G1-Phase des Zellzyklus rücken die Zentriolen ein kleinwenig auseinander. Rechtwinklig zu jeder alten Zentriole wächst nun zwischen der S- und der G2-Phase je eine Tochterzentriole heran. Die beiden Zentriolenpaare sind anfangs noch gemeinsam in ein Centrosomenmaterial eingebettet, trennen sich dann zu Beginn der M-Phase zunehmends voneinander und erhalten so je ein eigenes Mikrotubuliorganisierendes Zentrum. Cytoskelett: Die Actinfilamente und die Mikrotubuli zerfallen in ihre Untereinheiten (G-Actin und Tubulin) und werden so mehr oder weniger gleichmässig auf die Tochterzellen weitergegeben. Das Netz 14 von Intermediärfilamenten, das in der Interphase den Zellkern umgibt, dehnt sich während der M-Phase aus, umgibt später beide Tochterzellkerne und wird schliesslich durch den kontraktilen Ring in zwei Teile gespalten. Mitochondrien und Chloroplasten: Diese Organellen können nicht aus Untereinheiten zusammengesetzt werden und müssen so als kompakte Strukturen weitergegeben werden. Sie sind in der Mutterzelle meist so grosser Zahl vertreten, dass jede Tochterzelle einen Teil davon erhält. Dort können sie sich durch Teilung vermehren und somit ebenso zahlreich werden wie in der Mutterzelle. Eine Tochterzelle muss aber bei einer Zellteilung wenigstens ein Mitochondrium erhalten, damit sie weiterleben kann. Golgi-Apparat und Endoplasmatisches Retikulum: Sie zerfallen ähnlich wie die Kernhülle in kleine Vesikel und können so gleichmässig aufgeteilt werden. Zellteilung bei Prokaryonten Da Prokaryonten oft nur ein Chromosom besitzen (das Nukleoid), geht die Zellteilung viel einfacher vonstatten als bei Eukaryonten. So ist beispielsweise kein Spindelapparat nötig, um die Schwesterchromosomen voneinander zu trennen: Die DNA der Prokaryonten heftet sich nach der Replikation an der Zellmembran an, und die dazwischenliegende Membran wächst dann gegen innen, so dass zwei Tochterzellen mit je einem neuen Nukleoid entstehen: Meiose Bei der Verschmelzung einer männlichen Keimzelle (Spermium) mit einer weiblichen Keimzelle (Oozyte) entsteht eine befruchtete Eizelle (Zygote). Damit sich die Anzahl der Chromosomen nicht von Generation zu Generation verdoppelt, muss sie bei der Keimzellreifung irgendwann um die Hälfte reduziert werden, und zwar in geordneter Form, so dass sich jeweils die homologen Chromosomen voneinander trennen und Zellen mit haploiden Chromosomensätzen entstehen. Dies ist die Aufgabe der Meiose (Reifeteilung). Die Meiose besteht aus zwei Zellteilungen: 15 • • In der Meiose I (1. Reifeteilung) werden homologe Chromosomen separiert, so dass zwei haploide Tochterzellen entstehen. Die Meiose I heisst wegen der Reduktion von diploid nach haploid auch Reduktionsteilung. In der Meiose II (2. Reifeteilung) werden genau wie bei der Mitose die Chromatiden voneinander getrennt. Die Meiose II ist also eine Mitose ausgehend von einem haploiden Chromosomensatz. Hinweis: Im Atelier koennen Sie sich das Video "Meiose" ansehen. Die erste Reifeteilung (Meiose I, Reduktionsteilung) Vor der eigentlichen Teilung wird in der S-Phase des Zellzyklus die DNA repliziert. Die Chromosomen, welche nun aus zwei Chromatiden bestehen, verdichten sich in der besonders lange dauernden und aus verschiedenen Stadien bestehenden Prophase I. Im Unterschied zur Mitose paaren sich aber die homologen Chromosomen, d. h. sie ordnen sich parallel zueinander an, so dass entsprechende Genabschnitte exakt nebeneinander liegen. Man nennt diese Vierergruppe aus zwei mal zwei Chromatiden eine Tetrade. Die Chromatiden winden sich dann umeinander und scheinen sich an einigen Stellen zu überkreuzen. Man spricht deshalb von einem Chiasma. Ein Chiasma ist der morphologische Ausdruck für einen wichtigen Vorgang, den man Crossing-over oder homologe Rekombination nennt. Bei diesen Überkreuzungen werden nämlich Chromosomenstücke zwischen Nicht-Schwesterchromatiden 16 ausgetauscht und damit das Genmaterial neu verteilt. Jedes Chromosom einer reifen Keimzelle wird also eine Mischung aus „mütterlichen" und „väterlichen" Genabschnitten sein. Verschiedene Stadien der Prophase I Im Gegensatz zur mitotischen Prophase dauert erste meiotische besonders lange: Bei der Spermiogenese 20-24 Tage, bei der Oogenese gar mehrere Jahre! Man unterscheidet mehrere Phasen: Leptotän: die Chromosomen kondensieren zu dünnen Fäden. Ihre Enden sind an der Kernhülle befestigt. Zygotän: die homologen Chromosomen treffen sich und bilden eine Tetrade. Pachytän: dies ist das Stadium der homologen Rekombination, dem Stückaustausch zwischen NichtSchwesterchromatiden. Diplotän: die homologen Chromosomen lösen sich voneinander, bleiben aber vorläufig an den Überkreuzungen (Chiasmata) noch verbunden. Diakinese: die Chromosomen verdichten sich weiter, und die Chromatiden werden sichtbar. Sie haften nun nicht mehr an der Kernhülle. Die Mikrotubuli jeder Seite verbinden sich nun mit den Kinetochoren der Chromosomen, allerdings so, dass ein Chromosom mit dem einen Pol, sein Partnerchromosom mit dem andern in Verbindung steht. Nur so ist gewährleistet, dass sich in der Anaphase I die homologen Chromosomen voneinander trennen: 17 Aufgabe: Wieviele Kombinationsmöglichkeiten gibt es bei der Verteilung der väterlichen und mütterlichen Chromosomen bei einem Organismus mit 4 Chromosomenpaaren? Die zweite Reifeteilung (Meiose II) Die bei der ersten Reifeteilung entstandenen Zellen haben einen haploiden Chromosomensatz. Jedes der 23 Chromosomen besteht aber noch aus zwei Chromatiden. Diese werden in der zweiten Reifeteilung voneinander getrennt. Die verschiedenen Phasen der Meiose II gleichen der Mitose, mit dem wichtigen Unterschied, dass die in der Anaphase II zu trennenden Chromatiden genetisch nicht identisch sind, dies als Folge des Crossing-over der Prophase I: 18 Aufgabe: Wieso ist die Meiose II soviel kürzer als die Meiose I ? Non-Disjunction Fällt die Trennung zwischen den homologen Chromosomen (1. Reifeteilung) bzw. die Trennung der Chromatiden (2. Reifeteilung) aus, gelangen beide Chromosomen bzw. beide Chromatiden in dieselbe Tochterzelle. Durch dieses Ereignis, welches man Non-Disjunction nennt, erhält eine Keimzelle 24, die andere nur 22 Chromosomen. So entstehen bei der Befruchtung Organismen mit 47 (Trisomie) bzw. mit 45 (Monosomie) Chromosomen. Aufgaben • Unten sind die beiden Reifeteilungen am Beispiel von Chromosom 12 schematisch gezeigt. Vergleichen Sie die Meiose mit der Mitose. • Versuchen Sie, die gezeigte Darstellung auf eine Meiose mit zwei Chromosomenpaaren anzuwenden. Nehmen Sie als Beispiel die Chromosomen 2m/2w (grosses Chr.) und 19m/19w (kleines Chr.) und zeichnen Sie alle möglichen Kombinationen auf, die sich bei einem einmaligen Crossing-over zwischen 2m und 2w ergeben. 19 Spermatogenese und Oogenese beim Menschen Spermatogenese: Mit der Pubertät differenzieren sich Spermatogonien zu Spermatocyten 1. Ordnung. Diese teilen sich in der ersten Reifeteilung und werden zu Spermatocyten 2. Ordnung. In der zweiten Reifeteilung gehen daraus Spermatiden hervor. In der Spermiohistogenese reifen die Spermatiden zu Spermien. Aus einer einzigen Spermatogonie entstehen somit vier Spermien, zwei davon haben ein X als Geschlechtschromosom, zwei ein Y. Die mitotische Teilung einiger Spermatogonien stellt zeitlebens den Bestand an Zellausgangspopulationen für die Spermatogenese sicher. 20 Oogenese: Schon vor der Geburt differenzieren sich Oogonien zu Oocyten 1. Ordnung, welche von einer einschichtigplatten Epithelschicht bedeckt sind. Bei der Geburt gibt es etwa 700 000 bis 2 Millionen dieser Primordialfollikel. Die Oocyten 1. Ordnung gehen in die Prophase der ersten meiotischen Teilung über und verbleiben bis zur Pubertät in einem Ruhezustand, dem Diktiyotän-Stadium. Von der Menarche (erste Menstruation) bis zur Menopause reifen jeden Monat einige Primordialfollikel über Primär- und Sekundärfollikel zu Tertiärfollikeln. Ein einziger Tertiärfollikel wird besonders gross. Er wächst zum sprungreifen Graaf-Follikel. Die Oocyte 1. Ordnung beendet kurz vor der Ovulation (Eisprung) die erste Reifeteilung, wird damit zur Oocyte 2. Ordnung und geht in die zweite Reifeteilung über, welche im Metaphasenstadium unterbrochen wird und erst bei einer allfälligen Befruchtung beendet wird. Aus einer weiblichen Keimzelle entstehen vier Tochterzellen mit einem X als Geschlechtschromosom. Nur eine Zelle wird zur Eizelle, die drei sogenannten Polkörperchen degenerieren in der weiteren Entwicklung. Quellennachweis Die Texte und Abbildungen des vorliegenden Scripts entsprechen den Seitenangaben aus folgenden Werken: • Bruce Alberts et al.: "Molekularbiologie der Zelle", VCH, 2. Auflage, ISBN 3-527- 27983-0, p. 867880, p. 910-937, p. 1006-1017 • Walther Traut: "Chromosomen - klassische und molekulare Cytokinetik", Springer-Lehrbuch, ISBN 3-540-53319-2, p. 5-28, p. 135-174 • O. Bucher / H. Wartenberg: "Cytologie, Histologie und mikroskopische Anatomie des Menschen", Verlag Hans Huber, 11. Auflage, ISBN 3-456--81803-3, p. 83-93 21 • Rolf Knippers: "Molekulare Genetik", Georg Thieme Verlag, 6. Auflage, ISBN 3-13-477006-7, p. 142-158, p. 195-198 • Jan Langman: "Medizinische Embryologie", Georg Thieme Verlag, 8. Auflage, ISBN 3-13-446608-2, p. 2-18 • M. Hirsch-Kauffmann, M. Schweiger: "Biologie für Mediziner und Naturwissenschaftler", 2. Auflage, Georg Thieme Verlag, ISBN 3-13-706502-X Hinweis: In der „Lese-Ecke“ stehen Ihnen Lehrbücher zum vertieften Studium zur Verfügung Lösungen Wieviele Kombinationsmöglichkeiten gibt es bei der Verteilung der väterlichen und mütterlichen Chromosomen bei einem Organismus mit 4 Chromosomenpaaren ? Es gibt 2 hoch 4 = 16 Möglichkeiten (allgemein: 2 hoch n Möglichkeiten) Beim Menschen wären dies also 2 hoch 23 = 8 288 608 Möglichkeiten! Auch wenn es kein Crossingovergäbe, wäre die Wahrscheinlichkeit, dass ein Mensch Gameten aus nur mütterlichen oder nur väterlichen Chromosomen produziert 1:2 hoch 23, also nur 0,0000119 %. • • Wieso ist die Meiose II soviel kürzer als die Meiose I ? Dies hat verschiedene Gründe: Einmal ist für die zweite Reifeteilung keine DNA-Replikation nötig, da die Chromosomen schon aus zwei Chromatiden bestehen. Zweitens findet die zeitaufwendige homologe Rekombination nur in der Prophase I statt. Und schliesslich ist die Kondensation der Chromosomen beim Eintritt in die Prophase II schon weit fortgeschritten. Versuchen Sie, obige Darstellung auf eine Meiose mit zwei Chromosomenpaaren anzuwenden. Nehmen Sie als Beispiel die Chromosomen 2m/2w (grosses Chr.) und 19m/19w (kleines Chr.) und zeichnen Sie alle möglichen Kombinationen auf, die sich bei einem einmaligen Crossing-over zwischen 2m und 2w ergeben. Resultat: 8 mögliche Fälle (hätte Chromosom 19 ebenfalls ein Crossing-over erfahren, wären 16 verschiedene Gameten möglich gewesen): • 22 Uebungsaufgaben mit Lösungen finden Sie in der Internetversion des Ateliers! Hinweis: Das „Repetitorium Molekularbiologie“ definiert den Stoff, welcher in den Prüfungen verlangt wird. Wegen seiner Kürze eignet es sich allerdings nicht als primäre Informationsquelle! 23 Mechanismus der DNA-Replikation Hinweis: Im Atelier finden Sie das Video „Die Replikation“ sowie die CD "The Nature of Genes". Das Video bietet einen guten Einstieg in das Thema; die CD macht mittels Tutorials und Aufgaben die wichtigsten Themen der Molekularbiologie leichter verständlich. Allgemeines DNA als Matrize Am Beispiel der Replikation wurde zu ersten Mal gezeigt, dass Makromoleküle mit Hilfe einer molekularen Matrize synthetisiert werden. Mit der Aufklärung der Struktur der DNADoppelhelix durch Watson & Crick wurde ersichtlich, wie die DNA als Matrize, d.h. als Vorlage für die Vervielfältigung und Weitergabe der genetischen Information dienen könnte. Jeder Strang der DNA ist komplementär zum anderen. Somit kann ein Strang als Matrize dienen und der zweite Strang kann aufgrund der Regeln für die Basenpaarung synthetisiert werden Dies soll am folgenden Beispiel klar werden: Wenn der folgende Strang als Matrize dient: 5'-AGCGTTAGCGATCAT-3' muss der neu synthetisierte Strang die Basenfolge 3'-TCGCAATCGCTAGTA-5' aufweisen, da A mit T und G mit C basenpaaren. Das Replicon Ein DNA-Abschnitt, der von einem Replikationsstartpunkt aus repliziert wird, heisst Replicon. Ein eukaryotisches Chromosom wird von mehreren Stellen aus repliziert, hat also mehrere Replicons. Die Replikation beginnt am am „Origin“ (Ursprung, Startpunkt) und läuft, wenn sie einmal begonnen hat, solange weiter, bis das gesamte Replicon verdoppelt ist. 1 In E. coli (Bakterium) stellt das Genom ein einziges Replicon dar, d.h. die Initiation an einem einzigen Startpunkt führt zur Replikation des gesamten Genoms. Bakterien können zusätzlich noch Plasmide enthalten: ein Plasmid ist ein ringförmiges autonomes DNA-Molekül, das ein eigenes Replicon darstellt. Ebenso ist jede Phagen- oder Virus - DNA ein Replicon, das während des Infektionszyklus viele Male aktiviert werden kann. Replikationstypen Man kann sich verschiedenen Arten der Replikation vorstellen. Watson und Crick schlugen schon 1953 bei der Beschreibung der Doppelhelix folgenden Typ vor: Eine Doppelhelix (Mutterstrang) wird in ihre zwei Einzelstränge getrennt. Dabei entsteht eine Y-Figur mit einem doppelsträngigen Stamm und zwei zunächst einzelsträngigen Zweigen, deren Nukleotidfolgen als Matrizen für die Synthese neuer komplementärer Stränge dienen. An jedem von diesen „mütterlichen“ Einzelsträngen wird nun durch spezifische Enzyme ein „Tochterstrang“ synthetisiert. Diese Art der Replikation nennt man semikonservativ. Es gäbe theoretisch noch andere Arten der Replikation, beispielsweise die konservative Replikation. Bei dieser Form der DNA-Verdoppelung bliebe der ursprüngliche Doppelstrang erhalten und neben ihm würde ein neuer Doppelstrang synthetisiert (Abb.1). Bei der dispersen Replikation verteilen sich die Elternstränge gleichmässig auf die Nachkommen-Doppelstränge (Abb.2): Das Meselson-Stahl Experiment Matthew Meselson und Franklin Stahl zeigten1957, dass die DNA-Replikation semikonservativ verläuft. Sie züchteten E. coli Zellen über mehrere Generationen auf einem künstlichen Nährboden, welcher als Stickstoffquelle das "schwere" Stickstoff-Isotop 15N an Stelle des "leichten" Isotopes 14N enthielt. 2 Die DNA, welche 15N-Isotope in ihren Bausteinen enthält, ist um etwa 1% schwerer als 14N-DNA. Sie lässt sich mittels Zentrifugation in einem CsClDichtegradienten von der 14N-DNA trennen. Die E. coli Zellen, die 15N-DNA enthielten wurden nun auf ein frisches Medium übertragen, welches nur das Isotop 14N enthielt. Die 15N-DNA diente nun als „Elternmolekül“ für weitere Generationen. Meselson und Stahl liessen die Zellen solange wachsen, bis sich die Zellpopulation gerade verdoppelt hatte. Im CsCl-Gradienten ergab die DNA der ersten Tochtergeneration nur eine einzige Bande, woraus sich folgern liess, dass alle DNA-Doppelstränge gleich schwer sein müssen und jeder einen "alten Strang" (15N) und einen "neuen" (14N) haben muss. Damit sprach das Ergebnis gegen eine konservative Replikation. Darauf wurden die Zellen für eine erneute Verdoppelung in 14N-Medium gesetzt und die DNA wiederum im CsCl-Gradienten analysiert. Das Resultat waren zwei Banden, eine mit der leichten 14N-DNA und eine mit der gemischten 14N/15N-DNA. Somit war semikonservative Replikation für die DNA von E. coli bewiesen. Aufgabe: Nehmen Sie die Stränge der zweiten Generation des Experimentes oben und zeichnen Sie die DNA-Stränge der dritten Generation, die in einem 15N-Medium entstehen würden, farbig auf. Lösung: S. 15 Hinweis: Weitere Informationen zum Meselson-Stahl Experiment finden Sie beim Posten „Geschichte“ im Dokument-Nr. G4. 3 Replikations-Elemente Die Replikationsgabel Die Replikation beginnt an einem Startpunkt. Sie kann uni- oder bidirektional erfolgen, je nachdem ob sich eine oder zwei Replikationsgabeln vom Startpunkt (Origin) wegbewegen. Bei der bidirektionalen Replikation bewegen sich die beiden Replikationsgabeln in entgegengesetzter Richtung: Mittels radioaktivem Thymin im Nährmedium (Experiment) fand John Cairns heraus, dass an einem oder beiden Enden der gebildeten Replikationsgabel die Elternstränge entspiralisiert und die aufgetrennten Stränge rasch repliziert werden. Experiment von John Cairns John Cairns gelang es, mittels der Technik der Autoradiographie die Replikation als hochgradig koordinierten Vorgang zu beschreiben, bei dem die Elternstränge aufgewunden und gleichzeitig repliziert werden. Er erzeugte radioaktiv markierte DNA in E. coli Zellen, indem er sie in einem Nährmedium kultivierte, welches an Stelle des normalen Thymins das mit Tritium (3H) markierte Thymin enthielt. Er isolierte diese DNA, liess sie sich auf einem Elektronenmikroskop-Objektträger ausbreiten und überschichtete sie mit einer photographischen Emulsion. Binnen mehrerer Wochen erzeugten die radioaktiv markierten Thyminreste Bahnen aus Silberkörnchen, welche ein Abbild der DNA darstellten. Diese Bahnen zeigten, dass die DNA von E. coli aus einem einzelnen ringförmigen Molekül mit einer Länge von 1.7 mm besteht. Die radioaktiv markierte DNA, die während der Replikation aus der Zelle isoliert worden war, wies eine zusätzliche Schleife auf. Auf Grund des Vergleiches der DNA-Mengen in den Schleifen führte Cairns die zweite Schleife darauf zurück, dass zwei zu den Elternsträngen komplementäre Tochterstränge gebildet worden waren. DNA - Polymerasen In der Replikationsgabel synthetisiert ein Multienzymkomplex die beiden Tochterstränge. Dieser Enzymkomplex enthält neben anderen Proteinen die DNA - Polymerasen, welche Desoxynukleotide zu langen Polynukleotidketten verknüpfen. Das Bakterium E. coli enthält drei verschiedene DNA - Polymerasen, die meisten eukaryontischen Zellen dagegen deren fünf oder mehr, die sich in ihrer Struktur und Funktion unterscheiden. 4 DNA-Polymerasen bei E. coli Bezeichnung DNA-Polymerase I DNA-Polymerase II DNA-Polymerase III Aufbau 1 Untereinheit 928 Aminosäuren 103 Kilodalton (kDa) 88 kDa 10 Untereinh. (ca. 900kDa) Funktionen 3' - 5'–Exonuclease, 5'-3'-Exonuclease, Entfernung von RNA-Primer, Reparatur von DNA-Schäden Reparatur DNA-Polymersae, Replikation DNA-Polymerasen in eukaryotischen Zellen DNA- Polymerase katalytische Untereinheit (kDa) 180 Andere Untereinheiten (kDa) 86 58 48 3'-5'- Exonuclease Hauptfunktion nein 40 125 50 nein ja 125 200 53 mehrere ja ja Komplex mit Primase, Synthese kurzer DNAStücke nach Start durch Primase Reparatur von DNA mitochondriale DNAReplikation Replikation, Reparatur Replikation, Reparatur Die grundlegende Reaktion der DNA-Polymerasen ist immer dieselbe: Das Enzym katalysiert den nukleophilen Angriff der 3'-Hydroxylgruppe des Nucleotides am 3'-Ende des wachsenden DNA-Stranges auf das 5' α-Phosphoratom des anzuhängenden 2'-Desoxynukleotid-5'triphosphates: 5 Die allgemeine Reaktionsgleichung lautet: Für die Polymerisation (Verlängerung) sind zwei zentrale Voraussetzungen notwendig: • Es braucht eine Matrize • Es ist ein Primer (Startermolekül) erforderlich Ein Primer ist ein kurzer Nukleinsäurestrang mit einer freien 3'-Hydroxylgruppe am Ende. DNAPolymerasen können nur Nukleotide an einen bereits vorhandenen Strang anfügen, nicht aber die Synthese eines neuen Stranges starten. Prozessivität Wenn die DNA-Polymerase an einen Primer gebunden und das erste Nukleotid angehängt hat, gibt es für sie zwei Möglichkeiten: entweder sie dissoziiert ab und lagert sich später wieder an oder sie bewegt sich auf der Matrize vorwärts und hängt weitere Nukleotide an. Diese Eigenschaften wird als Prozessivität einer Polymerase definiert: Der Grad der Prozessivität ist durch die Anzahl Nukleotide gegeben, die die Polymerase im Durchschnitt an den neuen Strang anhängt, bevor sie abdissoziiert. Die Prozessivität wird durch Anlagerung eines Proteinkomplexes an die DNA-Polymerase gesteuert. Aufgabe: Überlegen Sie sich, was die Prozessivität einer Polymerase steigern könnte. In der Lösung finden Sie die Angaben bezüglich der Prozessivität der drei DNA-Polymerasen von E. coli. Lösung: S. 15 Energieverhältnisse der Polymerisation Bei der Polymerisationsreaktion finden grundsätzlich zwei Reaktionen statt; einerseits wird ein Phosphorsäureanhydrid hydrolysiert (im Desoxynukleotidtriphosphat, dNTP, von welchem Pyrophosphat abgespalten wird) und andererseits wird eine Phosphodiesterbindung gebildet und zwar an der 3'-OH Gruppe der neusynthetisiertenen DNA. Die Zucker-Phosphat-Bindung, welche neu geknüpft wird, ist energiearm, also in der Grössenordnung von 10-15kJ/mol: soviel ist aufzuwenden (+), wenn sie neu gemacht wird, soviel wird frei (-), wenn sie hydrolysiert wird. Die Polymerase hydrolysiert eine energiereiche Anhydridbindung und stellt damit gegen -30kJ/mol zur Verfügung. Es sollten also ca. -15kJ/mol freie Energie "übrigbleiben", wenn ein neues Nukleotid in die wachsende Kette eingefügt wird. Es ist aber sogar mehr! Basenpaarung und Basenstapelung tragen zur Sabilisierung der eingebauten Base bei: sie tragen um die -10kJ/mol freie Energie bei. Die Kopplung dieser Reaktionen bewirkt eine starke thermodynamische Triebkraft in Richtung der Polymerisation mit einem G°von insgesamt etwa -25kJ mol-1. 6 Genauigkeit Die DNA-Polymerasen arbeiten sehr exakt: Bei E. coli tritt nur bei jedem 109 bis 1010 Einbau eines Nukleotids ein Fehler auf. Aufgabe: Das Genom von E. coli hat ca. 4.6x106 Basenpaare. Wieviele Replikationen des Genoms können stattfinden, bis ein Fehler auftritt? Lösung: S. 15 Während der Polymerisation beruht die Unterscheidung zwischen "richtigen" und "falschen" Nukleotiden auf den Wasserstoffbrücken, die eine korrekte Paarung zwischen den komplementären Basen festlegen. Falsche Basen können nicht die richtigen Wasserstoffbrücken bilden und werden damit zurückgewiesen. Dies allein reicht aber noch nicht aus, um die hohe Präzision der Polymerisation zu erklären. Die Fehlerrate wird in vivo durch zusätzliche enzymatische Mechanismen weiter reduziert, z.B. durch 3'-5'-Exonuclease-Aktivität. Sie ist eine eigenständige Aktivität der Polymerase. Diese Nukleaseaktivität ermöglicht es dem Enzym, ein gerade eingebautes Nukleotid wieder zu entfernen. Wenn ein falsches Nukleotid eingebaut wurde, ist die Weiterbewegung der Polymerase zur Bindungsstelle des nächsten Nukleotids blockiert. Durch die 3'-5'-Exonucleasewirkung wird das falsche Nukleotid entfernt und das korrekte Nukleotid eingebaut. Diese Funktion wird als Korrekturlesen bezeichnet. Die Richtung der Replikation Wir haben gesehen, dass ein Nukleotid immer an die 3'-Hydroxylgruppe des neuen Stranges angehängt wird und die neue Kette somit in 5'-3'-Richtung wächst. Diese Tatsache führt aber zu einem Problem: Wenn die Synthese immer in 5'-3'-Richtung vorwärts schreitet, wie können dann beide DNAStränge gleichzeitig synthetisiert werden? Stellen wir uns eine Replikationsgabel vor. Der Eltern-Doppelstrang wird aufgetrennt und dient als Matrize. Die beiden Stränge weisen aber nicht dieselbe Orientierung auf: da sie antiparallel angeordnet sind, weist der eine Strang eine 3'-5'-Orientierung auf und der andere eine 5'-3'Orientierung. Sehen wir uns zuerst den 3'-5'-Elternstrang an, dieser kann in 5'-3'-Richtung direkt repliziert werden (gestrichelt): Der 5'-3'-Strang aber hat die "falsche" Richtung. Reiji Okazaki fand in den 60er Jahren heraus, dass an diesem Strang viele kurze Stücke in 5'-3'-Richtung synthetisiert werden, welche man nach ihm als Okazaki-Fragmente bezeichnet. Die Länge dieser Fragmente hängt vom Zelltyp ab und beträgt einige hundert bis zweitausend Nucleotide: 7 Somit wird der eine Strang kontinuierlich (Leitstrang) und der andere diskontinuierlich (Folgestrang) synthetisiert. Der kontinuierliche Strang ist derjenige, bei welchem die Synthese des neuen Stranges in der gleichen Richtung läuft wie die Replikationsgabel. Beim diskontinuierlichen Strang läuft die Synthese in die Gegenrichtung der Replikationsgabel. Vorgänge in der Replikationsgabel Bei der Replikation wirken in der Replikationsgabel neben der Polymerasen noch viele weitere Enzyme und Proteine mit, welche in ihrer Gesamtheit als DNA-Replikase-System resp. Replisom bezeichnet werden. RNA-Primase / DNA-Ligase Zu Beginn der Replikation wird ein Primer benötigt. Am Leitstrang kann von diesem Primer aus der neue Strang kontinuierlich gebildet werden, da immer ein basengepaartes Kettenende für die weitere Synthese zur Verfügung steht. Am Folgestrang jedoch kann die Polymerase nur eine kurze Zeit lang wirken, dann muss der Polymerisationsprozess weiter vorne von neuem beginnen. Dafür ist jedes Mal von neuem ein Primer nötig. Dieser basengepaarte Primer-Strang wird durch ein Enzym, die RNA-Primase, synthetisiert. Bei Eukaryonten haben die Primer eine Länge von ungefähr 10 Nukleotiden. Diese RNAStücke werden in Abständen am Folgestrang synthetisiert und anschliessend durch die DNA-Polymerase zu einem OkazakiFragment verlängert. Diese Synthese endet am 5'-Ende des vorhergehenden Fragments. Damit nun ein kontinuierlicher Strang entsteht, der keine RNA-Stücke enthält, tritt noch während der Replikation ein Reparaturmechanismus in Aktion: Eine RNase H (in E. coli Teil der DNAPolymerase I) entfernt die RNA-Primer und eine DNA-Polymerase füllt die entstandene Lücke mit DNA. Die DNA-Ligase schliesst dann die Bindung vom 3'-Ende des neuen zum 5'-Ende des alten DNAStückes und beendet damit den Vorgang. 8 DNA-Ligasen bei Bakterien und Eukaryoten E. coli besitzt eine, Eukaryoten mindestens zwei verschiedene DNA-Ligasen. Ihre Funktion ist dieselbe, sie unterscheiden sich aber im Cofaktor. Während die Bakterien-Ligase NAD als Cofaktor benötigt, ist der Cofaktor von Eukaryoten-Ligasen ATP. Mechanismus: • AMP (aus ATP oder NAD) bindet an die Lysin-Seitenkette des Enzyms • Übertragung des AMP-Restes auf das 5'-Phosphatende der DNA • Bildung der Phosphodiester-Bindung über den Angriff der 3'-OH-Gruppe auf das aktivierte 5'Phosphatende DNA-Helicasen Damit die Replikation überhaupt ablaufen kann, muss der DNA-Doppelstrang (Elternstrang) getrennt werden. Dies bedeutet Schmelzen der DNA über einen kurzen Bereich. Das Entwinden wird von den DNA-Helicasen katalysiert, Enzymen, welche sich entlang der DNA bewegen und die Stränge mittels chemischer Energie aus ATP voneinander trennen. Bei dieser Hydrolyse kann die Form des Proteinmoleküls zyclisch verändert werden, so dass es mechanische Arbeit verrichten kann. Die freiliegenden DNA-Stränge werden durch DNA-bindende-Proteine stabilisiert. In Eukaryoten sowie Prokaryoten wurden bisher eine ganze Reihe von verschiedenen DNAHelicasen gefunden. Die Einteilung der Helicasen erfolgt biochemisch nach der Polarität ihrer Bewegung (3'-5' oder 5'-3') oder nach Art des Substrats. Viele Helicasen benötigen beispielsweise überstehende Einzelstrangenden als Eintrittsstellen, während andere von Doppelstrangenden aus wirken können. DNA-Topoisomerasen Das Auftrennen der Stränge erzeugt topologisch bedingte Spannungen in der helicalen DNAStruktur: Diese werden durch die Wirkung von Topoisomerasen beseitigt. Die Mithilfe bei der Replikation ist aber nur eine von vielen Aufgaben der Topoisomerasen. Bei allen Reaktionen, bei 9 denen die Topologie der DNA verändert wird, spielen die Topoisomerasen eine wichtige Rolle, also zum Beispiel bei der Transkription von Genen, bei der Rekombination u.a. Topoisomerasen öffnen vorübergehend den Phosphodiester-Rückgrat der Doppelhelix, leiten den einen DNA-Strang durch die entstandene Lücke und stellen dann die Phosphodiesterbindung wieder her. Das Ergebnis dieser Reaktion ist die Änderung der Windungszahl pro Längeneinheit und damit der Topologie der DNA. Im Wesentlichen arbeiten die Topoisomerasen der Eukaryoten und der Prokaryoten gleich. Hinweis: Sie finden weitere Informationen in der aufgelegten Brochure „Topoisomerasen von Eukaryoten und Prokaryoten“ Aufgabe: Worin liegt der Unterschied in der Funktion einer DNA-Topoisomerase und einer DNA-Helicase? Lösung: S. 15 Mechanismus Die wesentlichen Vorgänge in der Replikationsgabel in Bakterien: • • • • • Die DNA-Helicase bewegt sich in 5'-3'-Richtung entlang des Stranges, an den sie gebunden ist und entwindet die Doppelhelix unter Verbrauch von ATP. Die entstehenden Einzelstrang-Bereiche werden durch Einzelstrang-bindende-Proteine (SSBProteine) abgedeckt. Die DNA-Polymerase III heftet auf dem Leitstrang (Vorwärtsstrang) neue Desoxynukleotide an das 3'-OH-Ende des wachsenden DNA-Stranges. Auf dem Folgestrang (Rückwärtsstrang) bildet die Primase kurze RNA-Stücke (Primer), welche durch die DNA-Polymerase III zu Fragmenten von 1000-2000 Nukleotiden verlängert werden. Die Primer werden von der 5'-3'-Exonuclease der Polymerase I entfernt und die entstehenden Lücken durch DNA ersetzt Die DNA-Ligase verknüpft aufeinanderfolgende Okazaki-Fragmente miteinander. 10 Initiaton Initiator-Protein-Komplexe binden an spezifische Stellen am Replikationsstartpunkt. Sie bilden einen grossen Proteinkomplex, welcher die DNA-Helicase bindet und sie zu einem freiliegenden DNA-Einzelstrang dirigiert. Zusätzlich bindet die RNA-Primase und bildet mit der Helicase das Primosom, welches sich dann vom Startpunkt wegbewegt und RNA-Primer für die erste DNAKette bildet. Daraufhin lagern sich rasch die übrigen Proteine zu einem ReplikationsProteinkomplex zusammen, der sich vom Startpunkt wegbewegt und zwei neue DNA-Stränge (kontinuierlich/diskontinuierlich) synthetisiert. DNA-Replikationsgabeln werden bei Bakterien an speziellen DNA-Sequenzen initiiert. Von diesen Punkten aus bewegen sich zwei Replikationsgabeln in entgegengesetzter Richtung mit einer Geschwindigkeit von etwa 500 Nukleotiden pro Sekunde. Das Genom der Bakterien ist so klein, dass diese zwei Replikationsgabeln das gesamte Chromosom innerhalb von weniger als 40 Minuten replizieren können. Bei den Eukaryoten ist die DNA im Kern als Chromatin organisiert ist. Deshalb wird die Replikation der DNA von Veränderungen in der Chromatinstruktur begleitet: • Entfaltung des Chromatins in der Umgebung der Replikationsgabeln • Nukleosomen unmittelbar vor der Replikationsgabel verlieren ihre Histon H2A/H2B-Dimere, während die verbleibenden Histon H3/H4-Tetramere auf die neu replizierte DNA übertragen 11 • werden. Hinter der Replikationsgabel werden die Tetramere durch Aufnahme von Histon H2A/H2B wieder zu intakten Nucleosomen zusammengefügt. Auf der replizierten DNA werden neu gebildete Histone H3/H4 mit den H2A/H2B Histonen zusammengebaut und neue Nukleosomen gebildet. Somit sind die Nukleosomen auf der replizierten DNA zur Hälfte aus den alten Histonen (des parentalen Stranges) und aus neu gebildeten Histonen zusammengesetzt. Die Geschwindigkeit mit der sich die Replikationsgabel von Eukaryonten bewegt, liegt bei ca. 50 Nukleotiden pro Sekunde, und das Genom ist etwa 1000 mal grösser. Daraus ergibt sich die Notwendigkeit, die Replikation von mehreren Replikationsstartpunkten aus zu beginnen, da die Replikation sonst mehrere Wochen dauern würde: • • • Es werden immer mehrere Initiationspunkte aktiviert. Die Initiationspunkte haben einen Abstand von 30'000 - 300'000 Basenpaaren. Die Replikationsgabeln bewegen sich von einem gemeinsamen Startpunkt weg und stoppen, wenn sie mit einer anderen Replikationsgabel zusammenprallen. Auf diese Weise können viele Replikationsgabeln gleichzeitig und unabhängig auf jedem Chromosom operieren und dabei zwei komplette Tochter-DNA-Helices synthetisieren. Kontrolle der Replikation Damit das gesamte Genom einer eukaryontischen Zelle beim Zellzyklus verdoppelt wird, muss die Replikation an vielen Origins gleichzeitig oder kurz nacheinander initiiert werden. Da alle Startpunkte "gezündet" werden, aber nicht alle Replicons gleichzeitig repliziert werden können, ergeben sich Probleme der Kontrolle. So müssen gewisse Merkmale replizierte von nicht replizierten Replicons unterscheidbar machen, damit bereits replizierte Replicons kein zweites Mal repliziert werden. Dies wird (kurz zusammengefasst) folgendermassen erreicht: Origins binden nach erfolgter Replikation einen PräReplikationskomplex (Eiweiss-Komplex) und bereiten so eine neue Replikation vor. Wenn die Zelle in einen neuen Replikationszyklus eintritt, werden in der späten G1-Phase spezifische Proteinkinasen synthetisiert und aktiviert, welche ihrerseits in der S-Phase die Prä-Replikationskomplexe aktivieren. Dies führt zur Replikation der DNA. Komponenten der Replikationskomplexe werden darauf inaktiviert und Proteinkinasen zerstört (Proteolyse). 12 Termination Prokaryoten: Bei der Termination treffen am Terminator zwei Replikationsgabeln aufeinander. Dadurch kann es bei ringförmiger DNA (E. coli) zur Ausbildung von sog. Catenan-Strukturen kommen. Der Terminator liegt gegenüber dem Origin. An die Terminatorsequenz bindet das Protein Tus (terminator utilization substance), das die DNA-Helicase blockiert und damit die Wanderung der Replikationsgabeln zum Stillstand bringt. Die beiden Nachkommen-DNA hängen noch über einen unreplizierten Abschnitt zusammen. Dieser kann direkt durch die Einwirkung einer Topoisomerase gelöst werden. Die entstehenden Lücken werden sofort aufgefüllt Oft beobachtet man aber eine weitergehende DNA-Synthese mit der Ausbildung von Catenanen (ineinander verhängte Ringe). Zur Auflösung der Catenane und zur Freisetzung der replizierten DNA ist die Typ-II-Topoisomerase notwendig. Hinweis: Im Atelier können Sie sich das Video "Replikation" ansehen. Eukaryoten: Telomere und Telomerase: Da eukaryontische Chromosomen linear sind, stellt die Replikation der Enden ein besonderes Problem dar. Eine einfache Überlegung zeigt, dass die von Primern vermittelte diskontinuierliche DNA-Synthese quasi automatisch zu einem Verlust endständiger DNA führen muss, weil zumindest der DNA-Abschnitt, an dem die Synthese des letzten RNA-Primers stattfindet, unrepliziert bleiben muss. Die Lösung dieses Problems ist durch die besondere Struktur der Chromosomen-Enden (Telomere) möglich: Telomere bestehen aus Folgen kurzer repetitiver DNA-Sequenzen. An den Enden der Chromosomen des Menschen und anderer Vertebraten findet man viele hundert bis über tausend 6-Nukleotid-Blöcke vor: z.B. die Folge 5'-TTAGGG-3' in monotonen Wiederholungen auf dem einen DNA-Strang und die entsprechende Komplementär-Sequenz auf dem anderen DNA-Strang. Die Telomere werden im Verlauf der vielen Replikationsrunden von normalen Säugetierzellen in Zellkultur und im Tier kürzer. Es handelt sich dabei um einen normalen und unvermeidlichen Alterungsprozess. Reifende Geschlechtszellen, Embryonalzellen, Zellen aus Tumoren und alle schnell proliferierenden Zellen dagegen enthalten ein Enzym, das den Abbau der Telomerenden ausgleicht. Das Enzym heisst Telomerase. Telomerase besteht aus zwei funktionellen Teilen, einem Proteinanteil und einer RNA von etwa 160 Basen. Die RNA enthält einen Sequenzabschnitt, der mit den Telomer-Wiederholungen Basenpaare ausbilden kann. Die RNA dient gleichsam als wandernde Matrize für die Synthese von Telomeren, wie aus dem folgenden Reaktionsablauf hervorgeht: 13 • Es bilden sich Basenpaare zwischen dem überstehenden DNA-Ende und der Telomerase-RNA. • Desoxynukleotide werden an das 3'-OHEnde der DNA nach Massgabe der Matrizensequenz in der RNA angeheftet. • Es findet eine Translokation der Telomerase mit der Bereitstellung neuer Matrizensequenzen statt. Quellennachweis Die Texte und Abbildungen des vorliegenden Scripts entsprechen den Seitenangaben aus folgenden Werken: • Lehninger: "Prinzipien der Biochemie"; 2. Auflage; Spektrum Akademischer Verlag; ISBN 3-86025-106-6: Seiten 927-945 • Knippers R.: "Molekulare Genetik"; 6. neubearbeitete Auflage; Georg Thieme Verlag Stuttgart. New York; ISBN 3-13-477006-7, 1996: Seiten 162-193 • Albert et al: "Molekularbiologie der Zelle"; 2. Auflage; VCH Verlagsgesellschaft mbH, Weinheim (D); ISBN 3-527-27983-0, 1990: Seiten 266-279, 609-620 • Lewin B.: "Gene";2. Auflage, ; VCH Verlagsgesellschaft mbH, Weinheim (D) 1991; ISBN 3-527-28052-9: Seiten 374-399 Hinweis: In der „Lese-Ecke“ stehen Ihnen Lehrbücher zum vertieften Studium zur Verfügung 14 Lösungen • Zeichnen Sie die DNA-Stränge der dritten Generation, die in einem 15N-Medium entstehen würden farbig auf. Als Ausgangs-DNA dient diejenige der zweiten Generation. Das Resultat sieht folgendermassen aus: • Überlegen Sie sich, was die Prozessivität einer Polymerase steigern könnte. Die Prozessivität einer Polymerase hängt von der Dauer der Bindung an die Matrize ab. Je länger sie bindet, desto grösser ist die Zahl der angehängten Nukleotide und damit die Prozessivität. Als Beispiel sollen die DNA-Polymerasen von E. coli dienen: Polymerase I Polymerase II Polymerase III Prozessivität (angehängte Nukleotide vor dem Abdissoziieren) Polymerisationsgeschwindigkeit (angehängte Nukleotide / Sekunde) 3-200 > 10'000 > 500'000 16 - 20 ca. 7 250 - 1'000 • Das Genom von E. coli hat ca. 4.6x106 Basenpaare. Wieviele Replikationen des Genoms können stattfinden, bis ein Fehler auftritt? Bei E. coli kommt alle 1'000 bis 10'000 Replikationen ein Fehler vor • Worin liegt der Unterschied in der Funktion einer DNA-Topoisomerase und einer DNAHelicase? Die Topoisomerase überwindet die Superwindung der DNA, indem sie einen Strang spaltet, abwickelt und wieder verschliesst. Damit macht sie die DNA der Helicase zugänglich, die nun die Stränge voneinander trennen kann. Uebungsaufgaben mit Lösungen finden Sie in der Internetversion des Ateliers! Hinweis: Das „Repetitorium Molekularbiologie“ definiert den Stoff, welcher in den Prüfungen verlangt wird. Wegen seiner Kürze eignet es sich allerdings nicht als primäre Informationsquelle! 15 Mutation, Mutagenese Und Reparatur Hinweis: Im Atelier finden Sie das Video „Die Replikation“ sowie die CD "The Nature of Genes". Das Video bietet einen guten Einstieg in das Thema; die CD macht mittels Tutorials und Aufgaben die wichtigsten Themen der Molekularbiologie leichter verständlich. Was ist eine Mutation? Mutationen sind Änderungen im Erbmaterial Auf molekularem Niveau handelt es sich bei einer Mutation um eine veränderte (mutierte) DNABasensequenz. Die Folgen der genetischen Veränderung betreffen nicht nur die Zelle, in welcher die Veränderung stattfindet, sondern auch deren Nachkommen: die Mutation wird in allen künftigen Generationen kopiert werden, weil die "falschen" Basensequenzen ebenso repliziert werden wie die "richtigen". Eine Mutation in einem lebenswichtigen Gen kann die Inaktivierung eines Enzyms zur Folge haben und damit zum Tod der Zelle führen. Eine solche Mutation wird sich nicht durchsetzen können, weil das Individuum gestorben ist, bevor es sein Erbmaterial an Nachkommen weitergeben konnte. In diesem Fall geht die veränderte DNA-Sequenz wieder verloren, die Mutation hat sich selbst vernichtet. In sehr seltenen Fällen bewirkt eine Mutation aber auch die Synthese eines "besseren", d.h. für die jeweiligen Umweltbedingungen geeigneteren Enzyms. Dieser Organismus hat einen Vorteil, und das modifizierte Gen wird schliesslich das ursprüngliche Gen im grössten Teil der Population durch natürliche Auslese ersetzen. Das Erbmaterial muss also einerseits sehr genau (erbgleich) an die Tochterzellen weitergegeben werden, damit das Überleben der Art gewährleistet ist, andererseits soll es sich auch auf veränderte Umweltbedingungen einstellen können. Evolution, die Entwicklung der Formenvielfalt von Lebewesen, wäre also ohne Mutationen gar nicht möglich! 1 Aber längst nicht jede Änderung der Basensequenz hat wirklich Auswirkungen auf die Struktur oder die biochemischen Prozesse einer Zelle. Solche Mutationen nennt man deshalb "stille Mutationen". Die Organisation der Gene und des genetischen Codes, welche Ihnen an anderer Stelle vorgestellt werden, sind dafür verantwortlich: • Nur etwa 5% der DNA des Menschen enthält Gene. • Eukaryontische Gene enthalten nicht-kodierende Sequenzen (Introns). • Mehrere Basentripletts kodieren für dieselbe Aminosäure. • Gewisse Aminosäuren eines Proteins können durch andere Aminosäuren ersetzt werden, ohne dass das Protein funktionell verändert wird. Arten von Mutationen Mutationen können sich auf einzelne Gene beschränken (Genmutationen), Teile von Chromosomen (Chromosomenmutationen) oder noch grössere Anteile des Erbmaterials (Genommutationen) betreffen. 1. Genommutationen Normalerweise ist jedes der 23 Chromosomen des Menschen doppelt vorhanden, sie bilden ein Chromosomenpaar. Das eine stammt vom Vater, das andere von der Mutter. Liegen in einer Zelle nun drei oder mehr Ausführungen desselben Chromosoms vor, spricht man von Polyploidie, der Chromosomensatz ist dann nicht diploid (2n), sondern eben triploid (3n), tetraploid (4n), pentaploid (5n), usw. Die Knochenmarksriesenzellen (Megakaryozyten) sind gute Beispiele für menschliche polyploide Zellen: ihr Chromosomensatz erreicht durch Endoreplikation (DNASynthese ohne anschliessende Kernteilung) eine Grösse von 32n oder gar 64n! Es handelt sich dabei aber nicht um eine Mutation, sondern um eine gezielte Genomvergrösserung. Diploide Keimzellen entstehen durch Ausbleiben der Reduktionsteilung bei der Meiose. Bei einer eventuellen Befruchtung entstehen triploide Embryonen. Diese sind aber nicht lebensfähig und sterben meist noch im Mutterleib. Folgendes Karyogramm zeigt den triploiden Chromosomensatz eines spontan abortierten menschlichen Fetus: 2 Ebenfalls zu den Genommutationen gehören die Aneuploidien, bei welchen einzelne Chromosomen in Überzahl oder Unterzahl vorhanden sind. Ein Chromosom im Überschuss (also 47 statt 46) bezeichnet man als Trisomie, fehlt ein Chromosom, spricht man von Monosomie. Aneuploidien entstehen durch Fehlverteilung der Chromosomen bei der Gametogese. Down-Syndrom Prominentestes Beispiel für eine Trisomie ist das Down-Syndrom (Trisomie 21), früher "Mongolismus" genannt, bei welchem das Chromosom 21 in dreifacher Ausführung vorhanden ist. Patienten mit nur einem X-Geschlechtschromosom ohne Y-Chromosom leiden unter dem Turner-Syndrom (45, X0). Weitere Informationen über beide Krankheitsbilder finden Sie auf dem Plakat dieses Postens im Atelier. 2. Chromosomenmutationen Diese Mutationen betreffen Strukturveränderungen von Chromosomen, bei welchen Brüche auftreten und sich die entstehenden grösseren Bruchstücke gegebenenfalls neu arrangieren. Chromosomenmutationen sind mit modernen Färbemethoden (Bänderungstechnik) im Gegensatz zu Genmutationen mikroskopisch sichtbar. Chromosomenbrüche können sich in jeder Phase des Zellzyklus erreignen. Tritt die Mutation beispielsweise in der G1-Phase ein, betrifft dies später beide Chromatiden. Eine Strukturaberration in der G2-Phase beschränkt sich meist auf ein Chromatid. Deletion: Das Chromosom verliert durch das Bruchereignis einen Teil seiner Information. Man unterscheidet einen Zwischenstückverlust von einem Endstückverlust. Inversion: Das Bruchstück wird um 180 gedreht und wieder eingesetzt. Man spricht von einer parazentrischen Inversion, wenn beide Arme des Chromosoms betroffen sind, von einer perizentrischen Inversion, wenn nur einer umgebaut wird. Ringchromosomen: Deletion an beiden Enden des Chromosoms und anschliessende Fusion beider Bruchstücke zu einem Ring. 3 Duplikation: Das Bruchstück wird im homologen Partnerchromosom eingebaut. Liegen die entsprechenden Chromosomenabschnitte direkt hintereinander, spricht man von einer Tandem-Duplikation. Ursache ist wahrscheinlich ungleiches Crossing-Over homologer Chromosomen. Translokationen: Hier werden Bruchstücke zwischen nicht homologen Chromosomen ausgetauscht. Es entsteht deshalb kein Materialverlust Zentrische Fusion: (Robertson'sche Translokation): Sie ist eine Sonderform der oben besprochenen Translokation und betrifft die akrozentrischen Chromosomen 13, 14, 15, 21 und 22. Zwei Chromosomen verlieren dabei ihre Satelliten-DNA und fusionieren zu einem grösseren Chromosom. Erfolgt der Umbau zwischen homologen Chromosomen, entsteht ein metazentrisches, sonst ein submetazentrisches erTranslokationschromosom. Aufgabe: Zeichnen Sie eine Zentrische Fusion der Chromosomen 14 und 21 auf. Wie sieht die Chromosomenverteilung bei der Gametogenese und wie bei einer eventuellen Befruchtung der Eizelle aus (theoretisch sechs verschiedene Fälle)? Welchen Einfluss hat dies auf den Embryo? Lösung: S. 12 4 Hinweis: Die Folgen einer Deletion im kurzen Arm von Chromosom 5, einer Mutation, welche zum sog. Cri-du-chat-Syndrom (Katzenschrei-Syndrom) führt, können Sie dem Plakat im Atelier entnehmen. 3. Genmutationen Genmutationen betreffen einzelne Abschnitte eines Gens und sind deshalb mikroskopisch nicht sichtbar. Oft betreffen sie sogar nur ein einziges Basenpaar auf der DNA: es sind dies sogenannte Punktmutationen. Substitution Beim Kopieren von DNA (Replikation) kann es vorkommen, dass falsche, nicht komplementäre Basen eingebaut werden: wird eine Purin-Base durch eine andere Purin-Base (A zu G) oder eine Pyrimidin-Base durch eine andere Pyrimidin-Base (C zu T) ersetzt, spricht man von einer Transition; bei einer Transversion wird eine Purin- durch eine Pyrimidinbase (A,G zu T,C) oder umgekehrt (T,C zu A,G) ersetzt. Hinweis: Ein gutes Beispiel für eine Basensubstitution ist die Sichelzellanämie. Sie finden im Atelier einen Posten zum Thema "Sichelzellanämie". Deletion und Insertion Der Verlust oder der Einbau einzelner Basen kommen seltener vor. Da das Basentriplett (drei aufeinanderfolgende Basen) die Basis des genetischen Codes darstellt, kommt es bei Deletion oder Insertion einzelner Basen unter Umständen zu einer Verschiebung des Leserasters eines Gens (sog. frame-shift-Mutation). Wie Sie noch sehen werden, führt dies schlussendlich zu einer völlig veränderten Proteinstruktur. Dazu ein einfaches Beispiel: DNA (nur kodierender Strang gezeigt) und entsprechende Basentripletts Insertion von zwei Basen (A, T) und resultierende Basentripletts 5 Deletion einer Base (Cytosin) und resultierende Basentripletts Aufgabe: Versuchen Sie herauszufinden, in welchen Fällen Deletion oder Insertion von Basen zu keiner frame-shift-Mutation führt. Lösung: S. 13 Mutagenese: wie entsteht eine Mutation? Mutationen unterteilt man nach Art ihrer Entstehung in spontane und induzierte Mutationen. Spontane Mutationen geschehen auch unter "normalen" Verhältnissen mit bestimmter Wahrscheinlichkeit. Die Spontanrate ist allerdings individuell verschieden. Der Grund für diese Variation liegt in der unterschiedlichen Aktivität der DNA-Reparatursysteme (siehe weiter unten). Die Mutationsrate wird durch induzierte Mutationen weiter erhöht. Physikalische, chemische oder auch virale Mutagene induzieren Mutationen. Die Grenze zwischen spontanen und induzierten Mutationen ist aber nicht immer einfach zu ziehen: viele Agentien müssten nämlich eigentlich in beiden Klassen aufgeführt werden, weil eine gewisse Dosis eines Mutagens auch unter normalen Bedingungen existiert. Ultraviolette Strahlung, zum Beispiel, ist auch im Spektrum des Sonnenlichts enthalten und ist so auch an der Entstehung der "spontanen" Mutationen beteiligt. Induzierte Mutationen 1. Physikalische Mutagene Ionisierende Strahlen: Ionisierende Strahlen sind energiereiche Strahlen (Röntgenstrahlen, AlphaBeta- oder Gamma- Strahlen radioaktiver Substanzen), die einen Teil der von ihnen getroffenen Moleküle verändern. Röntgenstrahlen z.B. rufen bevorzugt Chromosomenbrüche hervor. UV-Licht: UV-Bestrahlung führt zur Bildung von Dimeren aus zwei benachbarten PyrimidinResten, wie weiter unten genauer beschrieben wird. 2. Chemische Mutagene ENU und EMS: Ethylnitroisoharnstoff (ENU) und Ethylmethansulfonat (EMS) werden experimentell zur Mutagenese eingesetzt. ENU und EMS lösen Punktmutationen aus. Colchizin: Polyploidie kann durch Zugabe von Colchizin induziert werden. Colchizin hemmt nämlich den Spindelapparat und damit die Trennung der Chromatiden bei der Zellteilung, indem es an Tubulin bindet und die Neubildung von Mikrotubuli verhindert. 6 5-Brom-Uracil: 5-Bromuracil ist eine dem Thymin ähnliche Base - sie trägt ein Bromatom anstelle der Methylgruppe. Bei der Replikation wird es aber oft mit Cytosin verwechselt, so dass Guanin anstelle von Adenin in die DNA eingebaut wird. Dies ergibt schliesslich eine Basensubstitution. Salpetrige Säure: Salpetrige Säure (HNO2) ist Katalysator bei diversen Umwandlungsreaktionen: So entstehen durch Abspaltung von NH3 (Desaminierung) und anschliessender Isomerisierung aus Cytosin Uracil, aus Adenin Hypoxanthin und aus Guanin Xanthin. Aufgabe: Eine der beiden Cytosin-Basen im DNA-Abschnitt wird unter Einfluss von HNO2 desaminiert. Welche Folgen hat dies auf diesen DNA-Abschnitt und welche Basensequenz haben die bei der nachfolgenden Replikation entstehenden DNA-Helices? (Tip: Uracil paart normalerweise mit Adenin) Lösung: S. 13 7 3. Virale Mutagene Auch Viren sind mutagen und können zu Chromosomenmutationen führen. Retroviren z.B. schreiben ihr Erbmaterial erst von RNA in DNA um und fügen diese dann in das Wirtsgenom ein. Spontane Mutationen Desaminierungen und Depurinierungen Dass salpetrige Säure die Struktur von Basen ändern kann, wurde schon oben gezeigt. Diese Desaminierung kann aber auch spontan ablaufen, genauso wie die hydrolytische Spaltung der Zucker-Base-Bindung von Purinen (Depurinierung). Pyrimidin-Dimere Benachbarte Pyrimidin-Basen (T-T, T-C, C-C) werden durch ultraviolette Strahlung zu kovalent gebundenen Pyrimidin-Dimeren verbunden. Beispiel: 8 Endoreplikation, Zellkernfusion und Endomitose Polyploide Zellen entstehen entweder durch doppelte DNA-Synthese in der S-Phase des Zellzyklus (Endoreplikation), durch Fusion von Zellkernen (Zellkernfusion) oder durch DNASynthese unter Ausbleiben der Zell- und Kernteilung (Endomitose). Non-disjunction Wenn sich zwei homologe Chromosomen bei der Meiose nicht trennen, erhält die eine Keimzelle beide Chromosomen, die andere keines. Diese Erscheinungsform nennt man meiotische Nondisjunction. Nichttrennung von homologen Chromatiden in der Mitose (mitotische Nondisjunction) hat zur Folge, dass ein Chromosom zuviel oder zuwenig in eine somatische Zelle gelangt. Beide Kategorien von Non-disjunction führen schliesslich zur Aneuplodie: Die Zahl der Chromosomen einer Zelle weicht von 23 Chromosomenpaaren ab. Tautomerien Die in der DNA vorkommenden Basen tragen unter anderem Amino- und Ketogruppen. Sie können aber in seltenen Fällen intramolekulare Umlagerungsreaktionen erfahren und in der Imino- oder Enolform vorliegen. In diesem Fall ändern sich die Paarungseigenschaften: So paart z.B. Cytosin nicht mehr mit Guanin, sondern mit Adenin! Entsprechendes gilt für alle anderen Basen, wie folgende Aufstellung zeigt: Normale Form A (Amino) paart mit T C (Amino) paart mit G G (Keto) paart mit C T (Keto) paart mit A Tautomere Form A (Imino) paart mit C C (Imino) paart mit A G (Enol) paart mit T T (Enol) paart mit G 9 Reparaturmechanismen Zellen verlieren durch spontane Depurinierung pro Tag bis zu 5000 Purinbasen aus der DNA. Die spontanen Desaminierungen von Cytosin zu Uracil werden auf etwa 100 geschätzt. Das Verhältnis zwischen den bekannten DNA-Basen und ihren tautomeren Formen beträgt etwa 10'000 zu 1. Trotzdem wird bei der Replikation nur etwa eine Base auf 109 Basen falsch eingebaut. Wie ist dieser Widerspruch zu erklären? Warum macht ein durchschnittlich grosses Gen nur alle 106 Zellteilungen eine Mutation durch? Obige Zahlen fordern die Existenz von Reparatursystemen, welche die DNA nach Fehlern absuchen und diese korrigieren. Solche Reparatursysteme gibt es tatsächlich. Als Mutationen erscheinen also nur die Genomänderungen, welche von den Reparaturkomplexen nicht erkannt wurden. Zellen verfügen über eine Reihe von Enzymkomplexen, welche verschiedene Typen von DNASchäden erkennen und reparieren können. Vereinfacht bestehen DNA-Reparaturvorgänge aus drei Schritten: • Ein Fehler auf einem DNA-Abschnitt wird von einem Enzymkomplex erkannt. Endonukleasen hydrolysieren darauf die Phosphodiesterbindungen im fehlerhaften Abschnitt und entfernen so die veränderte Nukleotidsequenz. • Der resultierende DNA-Einzelstrang wird durch eine DNA-Polymerase wieder mit komplementären Basen zum Doppelstrang ergänzt. • Eine DNA-Ligase verbindet das zuletzt eingebaute Nukleotid mit seinem Nachbarn und schliesst damit die Lücke. Depurinierte Stellen • Eine Endonuklease spaltet die DNA-Kette gerade vor der defekten Stelle und entfernt den Zucker und das Phosphat. • Eine DNA-Polymerase setzt das passende Nukleotid ein. • Eine DNA-Ligase schliesst die Lücke Enzyme erkennen Veränderungen in der DNA-Doppelhelix Fehler in der DNA-Doppelhelix, welche die Basenpaarung verändern, werden von Glycosylasen erkannt und die entsprechenden Basen entfernt. Glycosylasen sind eine vielfältige Gruppe von Enzymen: es gibt Glycosylasen, die spezifisch desaminierte Adenine, desaminierte Cytosine, verschiedene alkylierte Basen und Basen mit geöffneten Ringen entfernen können. Nach dem Entfernen der defekten Base wird der Schaden wie oben beschieben repariert. Desaminierungen können erkannt werden, weil jede durch eine Desaminierung entstandene Base nicht mehr korrekte Basenpaarung eingehen kann: so wird aus Cytosin Uracil, aus Adenin Hypoxanthin und aus Guanin Xanthin. Hier liegt ein Evolutionsvorteil, den die Verwendung von Thymin anstelle von Uracil im Einbau in die DNA hat: in einer uracil-haltigen DNA könnten normale Uracile nicht von solchen unterschieden werden, die durch Desaminierung von Cytosin entstehen. Es gibt auch Multienzymkomplexe, die grössere Veränderungen in der DNA erkennen und reparieren können. Solche Schäden entstehen unter anderem bei der Anlagerung von Kohlenwasserstoffen (wie z.B. dem Karzinogen Benzpyren) an Basen oder Bestrahlung von DNA mit ultravioletter Strahlung, welche zu kovalenter Verknüpfung von benachbarten Pyrimidinen führt. Solche DNA-Stränge werden vor und nach der schadhaften Stelle von Endonukleasen gespalten, ein Stück DNA von 20-30 Nukleotiden mit der schadhaften Stelle entfernt und die entstandene Lücke wie oben beschrieben repariert. 10 Und wenn beide DNA-Stränge defekt sind ? Sind gleichzeitig beide Kopien am gleichen Basenpaar beschädigt, kann keiner der beiden Stränge mehr als Matrize für die Reparatur gebraucht werden. Aber auch für diesen seltenen Fall gibt es mittels genetischer Rekombinationsvorgänge manchmal eine Rettung, nämlich dann, wenn die defekte Basensequenz noch in einem andern DNA-Abschnitt unversehrt vorhanden ist. Der "Korrekturlese"-Mechanismus der DNA-Polymerasen Die DNA-Polymerase baut bei der Replikation Nukleotide in 5'-> 3' - Richtung in den wachsenden DNA-Strang ein. Dazu braucht sie ein korrektes Basenpaar und ein freies 3' -OHEnde, an welches das nächste Nukleotid angehängt werden kann. Liegen nun zwei nichtkomplementäre Basen vor (wie es beim Einbau von tautomeren Formen der Basen geschehen kann), stoppt die Polymerase und entfernt solange Nukleotide in 3'-> 5' - Richtung, bis wieder ein basengepaartes Ende zur Verfügung steht. Die DNA-Polymerase ist also ein selbst-korrigierendes Enzym, indem es die von ihr falsch eingebauten Basen erkennt und wieder entfernt. 11 Hinweis: Wie wichtig diese Reparaturmechanismen für eine Zelle sind, können Sie dem Plakat im Atelier entnehmen. Patienten mit der Erbkrankheit Xeroderma pigmentosum haben Defekte in gewissen Reparaturenzymen und leiden unter schweren Hautschäden, darunter auch Hautkrebs. Quellennachweis • Walther Traut: "Chromosomen - klassische und molekulare Cytokinetik", p. 29-56, SpringerLehrbuch • Bruce Alberts et al.: "Molekularbiologie der Zelle", 2. Aufl., p. 257-269, p. 113-114 Hinweis: In der „Lese-Ecke“ stehen Ihnen Lehrbücher zum vertieften Studium zur Verfügung Lösungen • Zeichnen Sie eine Zentrische Fusion der Chromosomen 14 und 21 auf. Wie sieht die Chromosomenverteilung bei der Gametogenese und wie bei einer eventuellen Befruchtung der Eizelle aus (theoretisch sechs verschiedene Fälle)? Welchen Einfluss hat dies auf den Embryo? 12 • Versuchen Sie herauszufinden, in welchen Fällen Deletion oder Insertion von Basen zu keiner frame-shift-Mutation führt. Die genetischen Code-Wörter sind Basentripletts. Wenn nun eine Anzahl Basen, die dem Vielfachen von 3 entspricht, in die DNA eingebaut oder entfernt wird, kommt es zu keiner Verschiebung des Leserasters, wie unteres Schema zeigt. Allerdings entsteht dann bei der Proteinsynthese ein zu kurzes Eiweiss oder eines mit zuviel Aminosäuren. • Eine der beiden Cytosin-Basen im DNA-Abschnitt wird unter Einfluss von HNO2 desaminiert. Welche Folgen hat dies auf diesen DNA-Abschnitt und welche Basensequenz haben die bei der nachfolgenden Replikation entstehenden DNA-Helices? Uebungsaufgaben mit Lösungen finden Sie in der Internetversion des Ateliers! Hinweis: Das „Repetitorium Molekularbiologie“ definiert den Stoff, welcher in den Prüfungen verlangt wird. Wegen seiner Kürze eignet es sich allerdings nicht als primäre Informationsquelle! 13