DNA - Computing - Institut für Wissenschaftliches Rechnen
Werbung

DNA - Computing
Seminar 2001
Institut Wissenschaftliches Rechnen
TU Braunschweig
Überblick
Historische Entwicklung
Biologische Grundlagen
Hamilton Path Problem (HPP)
Kombinatorische DNA – Lösung des HPP
Modelle von DNA-Computing
Nanostrukturen der DNA
Ausblick
Einführung
1993
1994
1994
1995
Heute
Adleman lernt die Grundlagen der
Molekularbiologie
Adleman löst das HPP mit Hilfe von DNA
Publizierung seiner Ergebnisse
Formalisierung des ersten Modells durch
Lipton
Breite Forschung mit weltweiten
Konferenzen
Biologische Grundlagen
• DNA als Informationsspeicher des DNA-Computers
• Operationen eines DNA-Computer
• Informationen kodieren
• schreiben
• lesen
• manipulieren
DNA
• 1944 von Avery entdeckt
• Gundbaustein der Natur
• besteht aus einzelnen Nukleotiden (Oligonukleotide)
• Zucker (desoxyribose)
• Phosphatgruppe
• Base
Detaillierter Aufbau eines
Nukleotids
DNA
5‘
3‘
3‘
5‘
Modell der DNA
• -Doppelhelix
• strickleiterartiger Doppelstrang aus
gleichlangen Polynukliotidketten
DNA als Informationsträger
• herkömmliche Computer haben Alphabet X = {0, 1}
• DNA besitzt Alphabet X‘ = {A, T, G, C}
Die DNA eines Menschen umfasst 6 Milliarden Basenpaare
was einer Information von ca. 500 Büchern mit 1500 Seiten entspricht.
Amplifying (Replikation)
Annealing / Melting
Melting
(erhitzen)
Annealing
(abkühlen)
Synthesizing
Cutting
Separating
Extracting
Substituting
Detecting / Reading
• Verwendung von PCR / Gelelektophorese
• Einsatz von Blockmitteln bei der PCR
• daraus resultieren kleinere Einzelstränge,
die mit dem Hauptstrang zusammenhängen
• durch veränderte Gelelektrophorese werden
Positionen bestimmt
Neuere Methoden verwenden Leuchtstofffärbungen statt der PCR,
die bei der Gelelektrophorese erkannt werden.
...weitere Operationen
• Mixing (Crossover)
• Ligating (Konkatenation)
• Marking / Unmarking
• Destroying (Exonuclease Enzyme)
„Ein DNA-Programm“
• Gefäß mit in DNA kodierter Eingabe
• ...Synthesizing...
• ...Marking...
• ...Ligating...
• ...Reading...
• ...
• Ausgabe durch Detection als true / false
oder wieder durch DNA kodiert
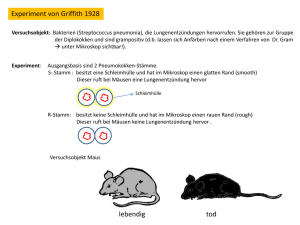
Hamilton Path Problem
Gegeben:
Ein gerichteter Graph, ein Start- und ein Endknoten
Gesucht:
Ein Pfad vom Start- bis zum Endknoten, wobei jeder
Knoten genau einmal besucht werden muß.
5
6
3
1
Richtiger Weg:1326547
7
2
4
Lösen des HPP
Erzeuge eine Menge von zufällig bestimmten
Pfaden durch den Graphen
2. Für alle Pfade in dieser Menge:
1.
a.
Beginnt der Pfad mit dem Startknoten und endet mit
dem Endknoten ?
b. Enthält der Pfad genau |V| Knoten ?
c. Sind alle Knoten im Pfad vorhanden?
3.
Alle Pfade in der verbleibenden Menge sind
Lösungswege, ist die Menge leer gibt es keine
Lösung.
Umsetzung des Algorithmus
mit DNA - Sequenzen
Kodierung der Knoten und Kanten als
DNA-Sequenzen durch Synthetisierung
Länge der Sequenzen jeweils 20 Basen
Jeder Knoten und jede Kante eindeutig
Knoten und Kanten
Knoten B
Knoten A
TCAGGTCGAG TCAGGTCGAG GCCTACGTAG GCCTACGTAG
Komplement
AGTCCAGCTC CGGATGCATC
Kante A B
Erzeugung aller Pfade
1014
Moleküle
100 Mikroliter Kochsalzlösung
TGAACTGGGATCTAGCTAGC
TGAATCGACTTCCGATAGCT
0,9 % NaCL
= TT-100
Erzeugung aller Pfade II
• Hybridisierung von Knoten und Kanten
• Ligation, zur Verstärkung des Rückrats der DNA
Ausfilterung der Pfade ohne
richtigen Start- und Endknoten
• Melting Trennung der Doppelhelix
• Hinzugabe von Sequenzen der Start- und Endknoten als Primer
• DNA-Polymerase vermehrt die DNA-Sequenzen unterschiedlich:
• Mit Start- und Endknoten exponentiell
• Mit Start- oder Endknoten verdoppelt
• Mit keiner Entsprechung gar nicht
• Nach mehren Zyklen des Erwärmens, Abkühlens und Vermehrens
wird eine Probe entnommen, die jetzt fast nur noch Pfade
einen richtigen Start- und Zielknoten enthält.
Kontrolle der Länge
• Ein richtiger Pfad muß genau 140 bp (=Basenpaare) lang
sein 7 Knoten a‘ 20 Basenpaaren
• DNA-Sequenzen laufen elektrophoretisch über ein
Agarose-Gel
Überprüfung der Existenz
jeden Knotens
•Wiederholung für alle Knoten mit entsprechenden Sonden
•Melting, damit DNA-Einzelstränge vorliegen
• Einbringung von Eisensonden mit Komplementstrang eines Knoten
• Durch Anbringung eines Magneten bleiben alle Moleküle haften,
die diesen Knoten enthalten
• Abgießen der Lösung und neue Lösung ansetzen
• Melting trennt Stränge von Sonden
• Abgießen der Lösung in ein neues Reagenzglas
Falls noch DNA vorhanden ist, muß das die Lösung des Problems sein.
Eisensonden mit DNASequenzen
Eisenkugel
Sonden-DNA
Enthaltener
Knoten
Nicht passende DNA
Rückblick
Alle Pfade
Alle Pfade mit vS und vE
Alle Pfade mit der Länge |V|
Knoten 1 enthalten
Knoten 2 enthalten
Knoten n enthalten =
Lösungsmenge
Fehler beim DNA-Computing
Pfad wird nicht erzeugt
Operationen arbeiten nicht fehlerfrei
Falsche Lösungen bleiben erhalten
Fehlerursache Größe, Menge und Masse der
DNA
Nur statistisch wahrscheinliche Lösung !
Beschränktes Modell
DNA-Menge nimmt in der
Berechnungsphase nicht mehr zu
DNA-Menge ist in der Initialisierungsphase
beschränkt
Anwendung von PCR wird ausgeschlossen
Beschränktes Modell
Bitvektoren kodierende Sequenzen werden erzeugt.
X0=1
X1=1
A0
A1
A2
X0=0 X1=0
Xn-1=1
....
An-1
Xn-1=0
An
Vorteil: Wiederverwendung der Bitstrings für andere Probleme
Beschränktes Modell
Operationen
Extraction
Sequenzen werden getrennt, abhängig ob
Subsequenz vorhanden
Merge
Vereinigung zweier Mengen von DNA
Detection
Test, ob DNA überhaupt noch vorhanden
Unbeschränktes Modell
Erweiterung des beschränkten Modelles
durch Adleman
Menge der DNA darf zunehmen
Erweiterte Operation:
Amplify (PCR)
Generator-Modell
Initialisierungsmenge effizienter nutzen
Suchraum einschränken
Generator-Modell
Operationen
• Split
Zufällige Aufteilung der DNA-Menge in zwei Mengen
• Append
Verlängerung der Sequenzen um Sequenzteile
• Merge
Vereinigung von zwei DNA-Mengen
Iteratives Anwenden von Split - Append - Merge erzeugt zufällige
Bitvektoren. Wenn Split - Merge an bestimmten Positionen weggelassen wird, haben alle Sequenzen an der Stelle die gleiche Bitbelegung.
Der Suchraum hat eine Dimension weniger.
Surface-Modell
DNA-Sequenzen fixiert auf einer Oberfläche
Bessere Kontrolle
Einengung des Suchraums
Modell wird verwendet, um Erkenntnisse über
Operationen und deren Fehler zu gewinnen
Nanostrukturen von DNA
DNA-Cube
Nanostrukturen von DNA
• Watson-Crick komplementär bildet Doppelhelix
• eine Zelle bildet verschiedene geometrische
Gebilde
• Verhalten der Kreuzung ist vorhersagbar
• Oligonukleotide bis hin zu Superstrukturen
können zur Realisierung verschiedener
Rechenmodelle benutzt werden
Nanostrukturen von DNA
• „self-assembly“ DNA
• kleine Anzahl von Oligonukleotiden bildet
self-assembled DNA-Stränge
• self-assembly entspricht gewissen Regeln
„self-assembly“ - Einsatz
Durch die Definition verschiedene Operationen auf den
verschiedenen Strukturen kann folgender Einsatz erreicht werden
• linear doppelte DNA-Sequenz entspricht regulärer Sprache
• self-assembled verzweigte DNA kann kontextfreie
Sprachen erzeugen
• doppelte Crossover-Moleküle sind geeignet für
universelle Berechnungen
„small Bricks“
TAE
Triple Doppelhelix mit anti-parallelen Überkreuzungen und einer
geraden Anzahl von Halbdrehungen
TAO
Geeignet für Xor - Funktion / Integer - Addition
DAE
Geeignet für Integer - Addition / Lösen des HPP
Im folgenden Gebrauch schematisch dargestellt als:
W
X
Y
Z
Ein-/Ausgabe mit
Nanostrukturen
„Adleman Graph“
3
2
4
1
7
5
6
Ablauf des Algorithmus
• Erzeugen aller Pfade von Knotenpunkt 1 zu Knotenpunkt N.
• Sortieren der Knoten in jedem Pfad in ansteigender Ordnung.
•Kontrolle für jeden Pfad, dass das Resultat genau “1,2, 3,...N” ist.
• Ausgabe jedes Weges, der den Test erfüllt, wenn es einen gibt.
Erzeugen aller Pfade
Sortieren
Sortieren der einzelnen „Reihen“ durch „Odd-Even-Transposition Sort“
Komplettieren der Struktur
• Einfügen spezieller End - Einheiten
• „DONE“ kann die Struktur in den Finalzustand überführen
Beispielergebnisse
Gültiger Hamilton-Pfad (1,4,5,2,3,6,7)
Beispielergebnisse
Ungültiger Pfad (1,2,3,4,5,2,3,6,7)
Vergleich der Algorithmen
Adleman:
• N (Nodes) + E (Edges) Oligonukleotide
• N Laborschritte (durch Separating)
„self-assembled“:
• E² / N + N² + N DAE Einheiten
• konstante Anzahl von Laborschritten
(Synthesizing, Annealing, Sequenzing)
Ausblick und Bewertung
Vorteile
Hohe Parallelität der Berechnung
– Maximum 270 DNA-Stränge in einem
Reagenzglas
Hohe Energieeffizienz
– 1 Joule schafft 2*1019 Operationen
Geringer Platzbedarf
– 1 Bit braucht 1nm³
Ausblick und Bewertung
Nachteile
Viele Operationen sind zeitaufwendig
– insbesondere Ein- und Ausgabe
NP-Probleme bleiben immer noch NPProbleme
Operationen können fehlerhaft sein
Statistisches Verfahren
Kostenaufwendig
Ausblick und Bewertung
Interessant für Forschung und Technik (Medizin).
Evolutionäre Algorithmen...
DNA-Computer werden in den nächsten 40 Jahren
von-Neumann-Rechner für den Home-User nicht ablösen.
The End
Viel Spaß auf dem Sportfest....
...PROST.