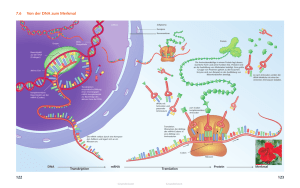
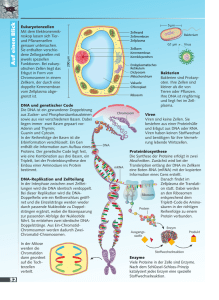
Kapitel 7 - Die Herrschaft der Molekularbiologie Die
Werbung

Befreiungsbiologie 2 Kapitel 7 - Die Herrschaft der Molekularbiologie Die Einsicht hinter Morgans Unverbindlichkeit bezüglich der materiellen Natur der Gene und Johannsens Insistieren auf einer konzeptuellen Trennung von Genotyp und Phänotyp, zwischen denen kein einfaches Kausalverhältnis angenommen werden durfte, ging im Verlauf der Geschichte verloren. Bereits zu Morgans Zeiten sprachen sich manche Genetiker entschieden für ein partikulär-präformationistisches Konzept der Gene aus. R. A. Brink interpretierte Morgans Befunde als Beleg für "diskrete, selbst-replizierende, stabile Körper - die Gene - die als Erbmaterial auf den Chromosomen liegen", was für ihn bedeutete, dass diese "offensichtlich die primären internen Mittel zur Kontrolle der Entwicklung [sind]". (Brink, 1927, in EFK refiguring, p.7???). Morgans Mitarbeiter Hermann J. Müller zeigte sich besonders beeindruckt von der Fähigkeit der Gene, sich selbst zu replizieren und zu mutieren, ohne dabei ihre spezfischen Kausalkräfte zu verlieren. Selbst-Replikation stellte den Vererbungsmechanismus der Gene dar, während stabile Mutationen Evolution durch das Hervorbringen von Merkmalsvariation ermöglichten. Diese beiden Eigenschaften machten das Gen in Müllers Augen so wichtig, dass er in dem ambitioniert betitelten Artikel "The gene as the basis for life" behauptete: "The great bulk […] of the protoplasma [is], after all, only a by-product of the action of the gene material; its 'function’ (its survival-value) lies only in its fostering the genes, and the primary secrets common to all life lie further back, in the gene material itself" (1926, p. 200-201, in EFK refig, p. 8???) Verschiedenes an diesem Zitat fällt auf: die pauschale und unbegründete Behauptung, das meiste Zellmaterial sei nur ein Nebenprodukt des Genoms; die Zuschreibung aktiver Handlungskraft ("action") an das genetsiche Material, sowie die mystifizierende Sprachmetapher der Gene als "Geheimnis" oder "Grundlage des Lebens". Es finden sich hier schon einige Elemente der später im Rahmen des Humangenomprojekts (HGP) populären religiösen Rhetorik, die das Genom als "Buch des Lebens" oder gar als den "heiligen Gral" bezeichnete. Müller hatte aber ausser Rhetorik durchaus handfeste Belege für seine Sicht der Gene. Gegen Ende der 1920er gelang es ihm, Mutationen durch physisches Einwirken auf das Genmaterial zu erzeugen. Er benutzte dazu Röntgenstrahlung und fand, dass die Anzahl induzierter Mutationen linear mit der Strahlendosis zunahm. In manchen Fällen gingen strahleninduzierte Veränderungen der Chromosomen mit veränderten Genkopplungsmustern einher. Da Strahlung in Form von Photonen gequantelt war, vermutete Müller, dass auch das Genom gequantelt sein müsse, wobei die Gene die vermuteten "Elementarpartikel" waren. Mitte der Dreissiger Jahre war ein weiterer von Morgans ehemaligen Studenten, Calvin Bridges, in der Lage, Gene nicht mehr nur relativ zueinander auf einem Chromosom anzuordnen, sondern ihre absoluten Positionen auf dem Chromosom zu bestimmen. Als Positionsmarkierung diente die charakteristische schwarz-weisse Bänderung bestimmter, sehr grosser Chromosomen, die man in der Speicheldrüse von Drosophila entdeckt hatte. Das Resultat bezeichnete man als "physikalische" Genkarte, da die Gene physikalischen Orten auf dem Chromosomen zugewiesen werden konnten, im Gegensatz zu den früheren Genkarten Sturtevants, welche Gene nicht physisch verankerten, sondern nur im relativen Bezug zueinander. Das Versprechen einer zunehmenden physischen Konturierung des Gens rief in den 1930er-Jahren zahlreiche Physiker und Biochemiker auf den Plan. Während die klassische 1 Befreiungsbiologie 2 Transmissionsgenetik weiterhin betrieben wurde, erweiterte sich deren methodisches Arsenal in Richtung der biochemischen Analyse von Genprodukten und der Nutzung biologisch immer einfacherer Systeme, in denen sich die "Wirkung" der Gene möglichst in Reinform studieren liess, sprich: in denen kein nennenswerter Entwicklungsprozess den Genotyp vom Phänotyp trennte. Neben bewährten Versuchsorganismen wie Drosophila, Mäusen und Maispflanzen kamen einfache Pilze und "bakterienfressende" Viren (sog. Phagen) zum Einsatz. In den 1940er Jahren wurden grosse Schritte in Richtung einer molekularen Charakterisierung der Struktur und Funktion von Genen getan. Der Biochemie waren damals sowohl Proteine als auch DNA schon bekannt. Von Proteinen wusste man, dass sie aus 20 verschiedenen Bausteinen (den Aminosäuren) bestehen und als Enzyme eine lebenwichtige Rolle spielen, indem sie Stoffwechselreaktionen beschleunigen. DNA war bereits im 19. Jahrhundert im Nukleus von Zellen gefunden und seine langkettige Form sowie seine biochemischen Bestandeile - ein Zucker, Phosphatreste und vier Basen - waren identifiziert worden. Hätte man damals auf eines der beiden Moleküle als mögliches materielles Substrat der Gene tippen müssen, hätte man die Proteine gewählt. Aufgrund der beinahe endlosen Kombinationsmöglichkeiten ihrer Bausteine konnten sie in zahllosen Varianten auftreten und schienen daher geeignet, die phänotypische Komplexität eines Organismus zu repräsentieren, während DNA dafür zu simpel und repetitiv erschien. 1 Es war daher keine geringe Überraschung, als der Amerikanische Mikrobiologe Oswald T. Avery 1944 einen ersten starken Hinweis darauf lieferte, dass DNA der Stoff der Gene war. Avery und Mitarbeiter studierten in New York Pneumokokken, jene Bakterien also, die beim Menschen Lungenentzündungen hervorrufen können, und fanden einen virulenten (ansteckenden) sowie einen nicht-virulenten Stamm. Faszinierenderweise liessen sich nichtvirulente Bakterien durch Injektion von DNA virulenter Bakterien selbst in virulente Bakterien verwandeln. Es schien, als enthalte DNA die Information, um einen Bakterientyp in einen andern zu transformieren. Der endgültigen Beweis, dass Gene aus DNA bestehen, kam aus der Phagen-Genetik. Phagen sind Viren, die Bakterien befallen und sie zur Selbstvermehrung benutzen. Man hatte bereits gezeigt, dass sog. T-Phagen zwar an der Oberfläche von Bakterienzellen andocken, aber nicht als Ganzes in sie eindringen, und dennoch lösen sich die Zellen nach einer Weile auf und spucken Hunderte von neuen Phagen aus. Offenbar war es bloss ein Teil des Phagen, der die Bakterienzelle enterte, und da T-Phagen nur aus einer Hülle aus Proteinen und einem Kern aus DNA bestehen, kam nur eines dieser beiden Moleküle als Übeltäter in Frage. Im Jahre 1952 kamen Hershey und Chase auf die Idee, Phagen-Protein und -DNA auf verschiedene Weise radioaktiv zu markieren, so dass man sie biochemisch unterscheiden konnte. Damit konnten sie zweifelsfrei nachweisen, dass es nur die DNA der Phagen war, die in die Bakterienzelle eindrang und sie in eine "Fabrik" zur Vermehrung ihrer selbst umfunktionierte. Was Averys Experiment schon angedeutet hatte, bestätigte sich nun: DNA war der Stoff, aus dem Gene gemacht sind. An anderer Front arbeiteten die Amerikaner George Beadle und Edward Tatum an der unmittelbaren Wirkung von Genen auf Zellvorgänge. Sie benutzten radioaktive Strahlung, um Mutationen in dem Schimmelpilz Neurospora crassa zu induzieren, die zu einem Produktionsausfall bestimmter normaler Stoffwechselprodukte führten. Als gezeigt werden konnte, dass einzelne Mutationen für den Ausfall einzelner Substanzen verantwortlich waren, formulierten Beadle und Tatum 1941 die berühmte "Ein Gen - Ein DNA steht für “Desoxyribonucleic acid”, zu deutsch "Desoxyribonukleinsäure" (DNS). Ich benutze in diesem Buch die allgemein gebräuchliche englische Abkürzung. 1 2 Befreiungsbiologie 2 Enzym"- Hypothese, wonach jedes "normale" Gen für die Bildung eines bestimmten Proteins zuständig ist. Durch die Identifikation der Gene mit DNA und dem Wissen, dass ein Gen für ein Protein verantwortlich ist, wurde eine neue Vermutung, die "Kollinearitäts-Hypothese" möglich. Sie besagte, dass die Sequenz von DNA-Bausteinen derjenigen der Aminosäuren eines Proteins entspreche (Proteine bestehen aus Ketten aneinandergereihter Aminosäuren, von denen es 20 verschiedene gibt). Wäre dies der Fall, könnte die Aminosäurensequenz eines Proteins vielleicht von der DNA-Sequenz eines Gens "abgelesen" werden, d.h. die DNA wäre ein Code für die Struktur von Proteinen. Im Laufe weniger Jahre sollte die Kollinearitäts-Hypothese bestätigt werden. Struktur und Replikation der DNA Im Jahre 1953 gelang es dem Briten Francis Crick und dem Amerikaner James Watson in ihrem Labor in Cambridge, England, die molekulare Feinstruktur der DNA aufzuklären. Sie publizierten ihre Resultate in der renommierten Fachzeitschrift Nature (???). Die DNA erwies sich als "Doppelhelix", d.h. als zwei separate, sich spiralförmig umeinander windende Molekülstränge, die sich durch Querverbindungen aneinander "festhalten". . ,-"-. ,-"-. ,-"-. ,-"-. ,-"-. , X | | \ / | | X | | \ / | | X | | \ / / \| | |X| | |/ \| | |X| | |/ \| | |X| `-!-' `-!-" `-!-' `-!-' `-!-' `- (__T A__) (__A T__) \_G C_/ \_/ X /_\ /_T A_\ (__A T__) \_G C_/ \_/ X /_\ /_G C_\ (__T A__) (__A T__) \_C G_/ \_/ X 3 Befreiungsbiologie 2 Die Stränge bestehen aus Zucker-Phosphatmolekülen, und die Querverbindungen werden von Basenmolekülen gebildet, von denen es insgesamt vier verschiedene gibt: Cytosin (C), Thymin (T), Adenin (A) und Guanin (G). Jede Querverbindung wird von jeweils einem Paar von Basen gebildet, die sich über die Lücke zwischen den zwei Strängen hinweg quasi die Hand reichen. Die Paarungen sind nicht beliebig: C paart sich nur mit G, und A nur mit T. Dies ist einfach deshalb der Fall, weil diese Paare chemisch zueinander passen und die anderen möglichen Paare nicht. Fährt man an einem der Stränge entlang und notiert sich fortlaufend die Basen, auf die man trifft, erhält man die DNA-Sequenz oder Basensequenz, die den "genetischen Code" ausmacht. Z.B. TAGTAGGTAC. Fährt man in derselben Richtung am zweiten Strang entlang, erhält man eine dazu komplementäre Sequenz, in diesem Falle ATCATCCATG. Aufgrund der Paarungsregeln lässt sich aus der Sequenz des einen Stranges immer die Sequenz des anderen Stranges ableiten. Diesen Umstand macht sich die Zelle zunutze, wenn aufgrund einer bevorstehenden Zellteilung die Verdoppelung - auch genannt Replikation oder Duplikation - der DNA vonnöten ist. Die beiden DNA-Stränge, sagen wir X und Y, werden zuerst irgendwo gekappt und dann aufgedröselt, so dass sie wie lose Enden in der “Luft” hängen. Nun kann die DNA-Synthesemaschinerie der Zelle an jedem der beiden Enden andocken und den dazu komplementären Strang (X und Y ) synthetisieren, was eben genau durch die Spezifität der Paarungsregeln möglich wird. Jedes Adenin, das sie findet, paart sie mit ein Thymin, jedes Ctyosin mit einem Guanin usw. Der neu synthetisierte Komplementärstrang (z.B. X ) eines Stranges (X) ist somit eine Kopie des ursprünglichen Komplementärstranges (Y). Der DNA-Syntheseapparat arbeitet sich die beiden Stränge entlang, welche dazu kontinuierlich auseinandergerissen und freigelegt werden müssen. Am Ende der Prozedur sind beide Stränge repliziert, was nichts anderes heisst, als dass die gesamte Doppelhelix eines Gens oder Chromosoms repliziert wurde. Die eine Doppelhelix besteht nun aus den Strängen X und X , und die andere aus Y und Y , wobei X = Y und Y = X. 1 1 1 1 1 1 1 Der genetische Code Watson und Crick hatten diesen Mechanismus der DNA-Replikation, dessen genaue Ausarbeitung Jahre dauerte, schon in ihrem Artikel vorgeahnt, merkten sie doch im letzten Satz non-chalant an: "It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material." In einem weiteren Artikel, ebenfalls aus dem Jahre 1953 und ebenfalls in Nature publiziert (???), nahmen sie eine zweite grosse Entdeckung, die des "genetischen Codes", vorweg. Sie postulierten "that the precise sequence of the bases is the code that carries the genetical information" - dass also die genaue Reihenfolge der DNA-Basen den Code ausmache, der die genetische Information trage. Auch diese Vermutung sollte sich später bestätigen. Für ihre Leistungen erhielten Watson und Crick 1962 den Nobelpreis. 2 Diese Schilderung enthält zwar die baren Fakten, ist jedoch zu verklärerisch in dem, was sie auslässt. So war die Entdeckung der DNA-Struktur keinesfalls nur auf Watson und Cricks Mist gewachsen. Tatsächlich hatte ihnen die röntgenmikroskopische Arbeit einer anderen Forscherin in Cambridge, Rosalind Franklin, in die sie ohne deren Erlaubnis Einsicht hatten, entscheidende Hinweise auf die Struktur der DNA geliefert (http://www.exploratorium.edu/origins/coldspring/ideas/printit.html ???). In ihrer Publikation gaben sie aber an, nichts von diesen Untersuchungen gewusst zu haben. Depew und Weber 2 4 Befreiungsbiologie 2 Der "genetische Code" und die Mechanismen, durch welche eine DNA-Sequenz "gelesen" und in ein entsprechendes Protein verwandelt wird, konnten bis etwa Mitte der 1960er-Jahre enträtselt werden. Mit dem genetischen Code ist das Schema gemeint, nach dem die Basensequenz eines Gens in die Aminosäurensequenz eines Proteins "übersetzt" wird. Es gibt wie gesagt vier verschiedene Basen (C, G, A, T) und 20 verschiedene Aminosäuren. Wie könnte ein Code aussehen, der das eine dem anderen eindeutig zuordnet? Ein Code also, bei dem eine gegebene Basenfolge genau eine, und nur eine, Aminosäurenfolge bestimmt? Als Lösung stellte sich heraus, dass jeweils eine Dreiergruppe (ein “Triplett”) von Basen eine Code-Einheit (ein ”Codon”) bilden, die einer bestimmten Aminosäure entspricht. So entspricht etwa dem Basentriplett "GCA" die Aminosäure Alanin, dem Triplett "AGA" die Aminsoäure Arginin und dem Triplett "TCC" die Aminosäure Serin. Aus vier verschiedenen Basen, angeordnet in Dreiergruppen, kann man ingesamt 64 verschiedene Codons bilden. Diese werden im genetischen Code auf die 20 verschiedenen Aminosäuren abgebildet. Allerdings gibt es insgesamt vier Codons, die nicht für Aminosäuren codieren, sondern den Anfang und Schluss eines Gens markieren (sog. Start- und Stopp-Codons). Somit bleiben 61 Codons, die 20 Aminosäuren zugeordnet werden müssen. Dieser Überschuss an DNA-Codons bedeutet, dass in der Regel mehrere verschiedene Codons für ein und dieselbe Aminosäure codieren. Der Code ist somit in der Richtung von Aminosäure zu DNA mehrdeutig, oder redundant. Mit andern Worten, aus dem Wissen, welche Aminosäure synthetisiert wurde, lässt sich nicht eindeutig schliessen, welches Codon dafür codierte. Es kommen stets mehrere in Frage. In der Richtung von DNA zu Aminosäure schien der Code jedoch eindeutig, indem jedes Codon nur auf genau eine Aminosäure verwies. Im Kapitel ??? werde ich das Konzept des genetischen Codes stark relativieren, aber an dieser Stelle benutze ich weiterhin das offizielle Vokabular der Genetik. Genexpression: Transkription und Translation Zur Zeit der Entschlüsselung des genetischen Codes wurden auch die Grundzüge der molekularen Mechanismen, die von einem Gen zu einem bestimmten Protein führen, aufgeklärt. Dieser Prozess verläuft in zwei Schritten, genannt Transkription und Translation. Die beiden Schritte zusammen werden als Gen-Expression bezeichnet. In der Transkription wird die DNA eines Gens abgelesen und eine einsträngige Kopie davon gemacht. Diese sog. messenger RNA (mRNA) ist beinahe identisch zur DNA, ausser dass die Base Thymin durch eine andere Base, Uracil, ersetzt wird. Diese Ersetzung ändert nichts an den Eigenschaften des Codes, und warum sie überhaupt stattfindet, braucht uns hier nicht weiter zu kümmern. Für unsere Zwecke genügt es, darauf zu achten, im genetischen Code den Buchstaben 'T' durch 'U' zu ersetzen (Abbildung ???). DNA-Sequenz: ---CTG-ACT-CCT-GAG-GAG-AAG-TCT--- mRNA-Sequenz: ---CUG-ACU-CCU-GAG-GAG-AAG-UCU--- Abbildung ???: Transkription von DNA zu mRNA. Die RNA-Kopie heisst messenger RNA, weil sie - wie ein Bote - den genetischen Code aus dem Zellkern hinaus ins Zytoplasma trägt, wo dieser "abgelesen" und in das entsprechende Protein umgewandelt wird, was dem Prozess der Translation entspricht (Abbildung???). Dabei wird der mRNA-Strang wie ein Band durch eine Lesemaschine gezogen, die fortlaufend die Basen-Codons - z.B. - AUG - CGT - UAU - … - "abliest", und die dazu gehörigen Aminosäuren aneinanderkettet - Methionin - Arginin - Thyrosin - … Das alles geschieht in einem Tempo von ca. 4 Aminosäuren pro Minute. Die mRNA wandert durch die Lesemaschine (die in Wahrheit ein komplexes Molekül namens "Ribosom" ist) und gleichzeitig wächst an anderer Stelle die neu synthetisierte Aminosäurekette an. Wenn das gesamte mRNA-Band "gelesen" und "übersetzt" ist, wird die fertige Aminosäurekette vom Ribosom abgekoppelt und ins Zytoplasma entlassen, wo sie sich später zu einem dreidimensionalen Protein auffaltet. mRNA-Sequenz: ---CUG-ACU-CCU-GAG-GAG-AAG-UCU--- bezeichnen diese Vorgehensweise als "manipulative und chauvinistic". (p. 346, darwinism evolving ???) 5 Befreiungsbiologie 2 Aminosäuresequenz: ---Leu-Thr-Pro-Glu-Glu-Lys-Ser--Abbildung ???: Translation von mRNA zu Aminosäurenkette Vielleicht fragen Sie sich, wie denn das Ribosom zu jedem mRNA-Codon die "richtige" Aminosäure findet. Die Verbindung zwischen Codon und Aminosäure wird durch ein weiteres Molekül namens Transfer-RNA (tRNA) hergestellt. In aller Einfachheit gesagt bindet die tRNA an einem Ende an das aktuelle mRNA-Codon und am anderen Ende an die passende Aminosäure, so dass letztere an die wachsende Aminosäurekette angehängt werden kann. Natürlich ist das noch keine Erklärung, denn jetzt lautet die Frage, wie denn die tRNA zu jedem Codon die passende Aminosäure findet. Die Antwort darauf lautet letztlich: durch das räumlich selektive Zusammenpassen jeder Aminosäure mit genau derjenigen tRNA, die an das (dieser Aminosäure) entsprechende Codon bindet. Die Bindung einer bestimmten tRNA an ein bestimmtes Codon ist dabei ebenfalls eines Sache der Spezifität des räumlichen Zusammenpassens zwischen dem Codon auf der mRNA und einer dazu komplementären Region namens "Anti-Codon" auf der tRNA. Jedes tRNA Molekül hat also zwei Bindungsstellen: eine für Aminosäuren, und eine, genannt "AntiCodon", für das aktuell "gelesene" mRNA-Codon (Abbildung???). Insgesamt muss es mindestens 20 verschiedene Typen von tRNA-Molekülen für die 20 verschiedenen Aminosäuren geben. Die tatsächliche Anzahl liegt aber eher bei 50, was bedeutet, dass es für manche Aminosäuren mehr als eine Art tRNA-Molekül gibt. Auch dieses System ist also redundant. A C -- Aminosäure C A G--C C--G C--G C--G G--C G--C G--C G A U C U C C C G C G A G | | | | | G U U U C G G A G G G C G | | | C U U G U A G C U A C A G A G A C--G C--G G--C C--G A--U C A U A C A U Anti-Codon - - - - - -G U A - - - - - - - - - - - - - - - - - mRNA mRNA-Codon 6 Befreiungsbiologie 2 Abbildung ???: Transfer-RNA (tRNA). AAC ist die Bindestelle für Aminosäuren, CAU ist das Anti-Codon, GUA das entsprechende Codon auf dem mRNA-Strang. Eine Explosion von Komplexität Die obigen Beschreibungen von Gen-Replikation und Gen-Expression sind Bilderbuchskizzen. Bereits damals, als diese Prozesse aufgeklärt wurden, präsentierten sie sich als komplizierter als hier geschildert. Was jedoch in den kommenden Jahrzehnten folgte, kam einer förmlichen Explosion weiterer Details, Modifikationen, Ausnahmen von der Regel, Verästelungen und Verkomplizierungen gleich. Die bisherigen Ausführungen zeichneten ein klares und einfaches Bild von Struktur und Funktion der DNA, welches einem minimalen Basiswissen entspricht, mit dem Generationen von Schülerinnen und Schüler versorgt wurden. Gemäss diesem Bild besteht das Genom aus einer Reihe von Genen, die auf Chromosomen wie Perlen auf einer Schnur aufgereiht sind. Die Gene bestehen aus DNA. Jedes Gen codiert für ein Protein. Der Code liegt in der linearen Abfolge von DNA-Basen, von denen es vier verschiedene gibt. Er besteht aus Einheiten von jeweils drei Basen ("Basen-Tripplets"), auch "Codons" genannt, wobei jedes Codon genau eine von zwanzig möglichen Aminosäuren spezifiziert. Wenn ein Gen exprimiert wird, wird zunächst eine Kopie der DNA in einem mRNA-Molekül hergestellt. Das mRNA-Molekül enthält denselben Code wie die DNA. Diesen Vorgang nennt man Tranksription. Der mRNA-Code wiederum wird von einem komplizierten molekularen Apparat gescannt, der daraus die entsprechende Aminosäurekette, aus der das Protein besteht, synthetisiert. Diesen Vorgang nennt man Translation. So weit, so gut. An keiner Stelle ist dieses Schema ganz falsch. Aber es ist auch an keiner Stelle ganz richtig. Ich beginne mit dem Aufbau des Genoms. Die erste Überraschung besteht darin, dass keinesfalls jedes Gen ein strukturelles, also protein-codierendes Gen ist. Nicht einmal 2% des Genoms besteht aus protein-codierenden Genen! Der Rest besteht aus verschiedenen anderen DNA-Sequenzen, deren Natur noch immer Gegenstand intensiver Forschungen ist. Von ganzen 90% des Genoms vermutete man zeitweilig, dass es aus "DNA-Müll" (“Junk-DNA”) bestehe, aus Basensequenzen ohne jede Funktion für den Organismus. Heute weiss man, dass etwa die Hälfte des Genoms aus hoch-repetitiven Sequenzen besteht, wovon viele sog. "springende Gene" oder "Transposons" sind. Es handelt sich dabei um mobile DNA-Elemente, die ihre Position innerhalb des Genoms verändern können. Sie springen quasi von einem Ort zu einem anderen. Es wird vermutet, dass diese DNA zu einem grossen Teil von anderen Spezies, insbesondere durch Bakterien, in unser Genom gelangt ist. Es gibt Hinweise, dass manche dieser Transposons, wie auch der Rest der Junk-DNA gen-regulatorische Funktionen haben könnte, aber in welchem Ausmass ist noch weitgehend unbekannt. Jedenfalls wird klar, dass das Genom kaum ein elegant design-tes evolutionäres Programm ist, in dem jedes Element eine spezifische Funktion ausübt, sondern zum grossen Teil etwas Zusammengeschustertes, das viele funktionslose und redundante Elemente enthält. Ein bestechendes Beispiel für solche funktionslosen Elemente betrifft die Transkription. Die Herstellung der mRNA verläuft nämlich über einen seltsam anmutenden Zwischenschritt, den man "Verspleissen" (engl. “Splicing”) nennt. Zunächst wird eine RNA-Kopie einer 7 Befreiungsbiologie 2 DNA-Sequenz erstellt, die "prä-RNA". Aus der prä-RNA werden dann einige Stücke herausgeschnitten, und die losen Enden wieder miteinander "verschweisst". Diese zusammengestückelte RNA ist die mRNA. Damit ist die prä-RNA die eigentliche Kopie der ursprünglichen DNA-Sequenz, während die mRNA bereits ein "editiertes" Produkt ist, das nicht mehr genau der Original-DNA-Sequenz entspricht (Abbildung???). Die herausgeschnittenen Stücke heissen "Introns" und werden herausgeschnitten, weil sie nicht protein-codierend sind und somit nichts zur Herstellung des Protein-Produkts beitragen. Warum diese nutzlosen DNA-Stücke aber überhaupt zwischen die codierenden Bereiche (die "Exons") eingestreut sind, ist noch weitgehend unklar. DNA/prä-RNA: …ATCCTTGGCAGGTAAGCACGAA . . . TTCTCTATTGGAGTAGGTACTACGAG… |_________________________________| Intron mRNA: …ATC-CTT-GGC-AGG-TAC-TAC-GAG… ^ Abbildung ???: Verspleissen von Genen. Fettgedruckt sind Teile der Exons, normal gedruckt das nichtcodierende Intron, das bei der Bildung der mRNA herausgeschnitten wird. Die "Schweissnaht" ist mit ^ gekennzeichnet. Der genetische Code ist generell mit 'T' anstatt 'U' geschrieben. Das wirklich Verrückte ist allerdings, das ein Gen oft nicht nur auf eine, sondern auf mehrere verschiedene Weisen verspleisst werden kann. Das heisst, aus ein- und derselben DNASequenz entstehen dann verschiedene mRNA-Sequenzen und somit verschiedene Proteine. Dies geschieht, grob gesagt, indem nach dem Herausschneiden der Introns nicht immer alle verbleibenden Exons benutzt werden, um das endgültige mRNA-Produkt zusammenzusetzen (Abbildung ???). DNA/prä-RNA: XXXXXXX---XXXXX---XXXXXXXX---XXXXX-----XXXXXXXXXXX [ Exon 1 ] mRNA mRNA mRNA mRNA [Exon 2] 1: [ Exon 1 2: [ Exon 1 3: [ Exon 1 4: [Exon 2][ [ Exon 3 ] [Exon 4] [ ][Exon 2][ Exon 3 ][Exon 4][ Exon 5 ][Exon 2][ Exon 3 ][ Exon 5 ] ][ Exon 3 ][Exon 4] Exon 3 ][Exon 4][ Exon 5 ] Exon 5 ] ] Abbildung ???: Alternatives Verspleissen. Aus einer bestimmten DNA-Sequenz können mehrere verschiedene mRNA-Sequenzen entstehen, die in verschiedene Protein umgesetzt werden. Introns sind durch gestrichelte (---) Linien angedeutet. 8 Befreiungsbiologie 2 Typischerweise werden die alternativen Proteine gewebeabhängig exprimiert. So könnte zum Beispiel in Muskelzellen mRNA 1, in Hirnzellen mRNA 2, in Leberzellen mRNA 3 und in Bindegewebszellen mRNA 4 gebildet werden. Nachdem alternatives Verspleissen zunächst als exotisches Phänomen angesehen wurde, schätzt man heute, dass beim Menschen bis zu 95% der Gene alternativ verspleisst werden können (Pan, Q; Shai O, Lee LJ, Frey BJ, Blencowe BJ (Dec 2008). "Deep surveying of alternative splicing complexity in the human transcriptome by highthroughput sequencing". Nature Genetics 40 (12): 1413–1415. doi:10.1038/ng.259. PMID 18978789???). Es gibt zahlreiche weitere RNA-Modifikationen bei der Transkription. Regelmässig werden nach dem Verspleissen der mRNA Basen hinzugefügt, entfernt oder ersetzt. Ebenfalls werden an den beiden Enden der mRNA bestimmte Sequenzen angehängt. Der genetische Code wird also teilweise umgeschrieben, bevor die Translation in ein Protein stattfindet. Damit ist der Abweichungen von der Bilderbuchskizze der Gen-Expression aber noch nicht genug. Nach der Translation finden eine ganze Reihe sog. postranslationeller Modifikationen statt. Diese dienen dazu, dem Protein den "letzten Schliff" zu verleihen und es für seine spezifische Aufgabe anzupassen. Häufig sind Proteine nach der Synthese inaktiv und müssen durch gezielte Modifikationen aktiviert werden. Dazu gehören das Abspalten eines Teils ihrer Aminosäurenkette oder die chemische Veränderung von Aminosäuren, indem bestimmte Atomgruppen angehängt werden. Eine wichtige Gruppe von Proteinen, die vor allem in der Zellmembran vorkommen, sind die Glykoproteine. Sie sind Teil des chemischen Sensoriums und des Andockappartes an der Aussenhülle der Zelle, wo sie Moleküle oder ganze Zellen an sich binden. Sie entstehen durch das posttranslationelle Anhängen von Zuckerketten an ein Protein. Zu guter Letzt werden etliche wichtige Zellfunktionen wie das Empfangen und Weiterleiten chemischer Signale von grossen Protein-Komplexen ausgeführt, die aus verschiedenen Protein-Untereinheiten bestehen. Das Zusamensetzen dieser Proteingiganten kann ebenfalls erst nach der Translation der einzelnen Bestandteile erfolgen. Genregulation Ein ganz neues Kapitel wird mit der Regulation der Gen-Expression aufgeschlagen. Im Jahre 1959 entdeckten François Jacob und Jacques Monod, dass im Darmbakterium E. coli gewisse DNA-Sequenzen, die an bestimmte strukturelle Gene angrenzen, deren Expression beeinflussen. Damit eröffnete sich die aufregende Möglichkeit, dass Gene nicht nur als Vorlagen für die Bildung von Proteinen dienen, sondern sogar die Aktivität anderer Gene regulieren konnten. Es lohnt sich deshalb, einen genaueren Blick auf die Entdeckung der beiden Franzosen zu werfen. E. coli ernährt sich von Glukose. Wenn diese rar ist, hat es die Fähigkeit, Glukose aus Laktose (Milchzucker) herzustellen. Jacques und Monod fanden heraus, wie die Steuerung der Glukosesynthese aus Laktose in E. coli funktioniert. Sie nannten dieses Steuersystem das lac-Operon. Ein Teil des lac-Operon besteht aus einer Reihe aufeinanderfolgender struktureller Gene (lacZ, lacY, lacA). Diese Gene codieren für verschiedene Proteine, die es dem Bakterium 9 Befreiungsbiologie 2 ermöglichen, Laktose aus dem Umgebungsmedium aufzunehmen und in Glukose umzuwandeln. Daran angrenzend ist eine DNA-Region, die ich hier als "Kontrollregion" bezeichnen werde. Sie bildet den zweiten Teil des lac-Operons und besteht aus zwei DNAStücken, einem "Promotor" und einem "Operator", die beide als Andockstellen für bestimmte Moleküle dienen. Die Regulation der Glukosesynthese aus Laktose basiert darauf, dass manche dieser Moleküle Aktivatoren, andere Inhibitoren, der Expression der lac-Gene sind, je nach dem, welche Umstände vorherrschen. Wenn keine Lactose vorhanden ist, bindet ein Repressor-Molekül an die Operon-Region und behindert die Transkription der lac-Gene, indem es die Anlagerung der Transkriptionsmaschinerie an den Promotor räumlich erschwert. Ist Laktose vorhanden, bindet sie an den Repressor, was in diesem eine räumliche Strukturveränderung herbeiführt, wodurch er nicht mehr an die DNA binden kann. Er löst sich vom Operon. Herrscht ein Mangel an Glukose, bindet ein Protein namens CAP an eine Bindungsstelle in der Promotor-Region und aktiviert die Transkription der lac-Gene, indem es das Andocken der Transkriptionsmaschinerie an den Promotor fördert. Dies ist jedoch nur der Fall, wenn genug Laktose vorhanden ist, da ansonsten der Repressor nach wie vor die Transkription unterdrückt. Wie ein Mangel an Glukose zur erhöhten Bindung von CAP an den Promotor führt, ist noch nicht ganz geklärt. Insgesamt stellt sich die Regulation der Glukoseproduktion aus Laktose als Prozess dar, bei dem, abhängig von den herrschenden Bedingungen, gewisse Moleküle an Kontrollabschnitte der DNA binden und dadurch entweder zu Aktivatoren oder Inhibitoren der Expression angrenzender struktureller Gene werden, die für Proteine codieren, welche die Umwandlung von Laktose zu Glukose ermöglichen. Jacob und Monod kannten noch nicht all diese Details. Entscheidend aber war, dass sie die Kontrollregionen der DNA ebenfalls als Gene bezeichneten und den nicht-genetischen Komponenten des Operons überordneten. Damit war gesagt, dass sie die Kontrolle des Genoms im Genom selbst ansiedelten und es zu einem selbst-regulierenden System erhoben. Nicht nur was Gene taten - nämlich Proteine machen - sondern auch, wann, wo und in welcher Menge sie es taten, wurde nun von Genen selbst bestimmt. Die beiden Franzosen schlossen denn auch den Aufsatz von 1961, in dem sie das lac-Operon beschrieben (???), mit der Bemerkung, Gene enthielten nicht nur Blaupausen für Proteine, sondern ein ganzes Programm zu deren Synthese und die Kontrolle über dessen Ausführung. Jacob und Monods "Kontrollgene" schürten die Hoffnung, die Prinzipien der Genregulation in primitiven Lebewesen wie Bakterien liessen sich auf höhere Organismen anwenden. Deren Studium in den folgenden Jahrzehnten enthüllte dann aber eine ungeahnte Komplexität. Schon angesprochen wurden die zahlreichen Prozesse der Modifikation von Transkription und Translation. Auf Ebene des Genoms zeigte sich, dass es für viele strukturellen Gene multiple Aktivierungs- und Inhibierungsregionen gibt, die auf verschiedene Arten funktionieren und Namen wie Promoter, Enhancer oder Silencer tragen (review: Serfling et al., 1985; Schöler et al., 1988 in Portin 2002???). Verblüffenderweise müssen diese Regionen nicht wie beim lac-Operon an die strukturellen Gene angrenzen, sondern können in beträchtlicher Entfernung davon liegen. Eine DNA-Region kann also die Aktivität eines protein-codierenden Gens, das weit davon entfernt liegt, beeinflussen. Eine weitere Überraschung war die Entdeckung, dass mehrere solcher Kontrollregionen alternativ und auf veschiedene Weise die Transkription eines bestimmten Gens initiieren 10 Befreiungsbiologie 2 können. Solche "alternativen Promotoren" können zum Beispiel die Transkription an unterschiedlichen Stellen des Gens beginnen lassen, woraus unerschiedliche mRNAMoleküle und schliesslich unterschiedliche Proteine resultieren (Schibler & Sierra, 1987 in Portin 2002???). Von einfachen auf höhere Organismen generalisierbar war das Prinzip, dass Kontrollgene bzw. DNA-Kontrollregionen Moleküle an sich binden müssen, um ihre Wirkung zu entfalten (solche Moleküle nennt man allgemein "Transkriptionsfaktoren"). Schon beim lacOperon handelt es sich dabei typischerweise um Mechanismen der räumlichen Interaktion: ein Molekül blockiert den Zugang eines anderen zur DNA, oder es führt zu einer Strukturveränderung, die die Bindungseigenschaften des anderen Moleküls verändern. Auch die DNA ist Teil solcher Mechanismen. Oft bilden DNA-Stücke Schleifen, so dass weit entfernte Regionen miteinander in physischen Kontakt kommen. Auf diese Weise kann ein Transkriptionsfaktor auf einem entfernten DNA-Abschnitt mit einer an ein strukturelles Gen angrezenden Kontrollregion interagieren und die Transkriptionsrate verändern. Ein paar weitere, auf den ersten Blick exotische Phänomene sind überlappende Leseraster, überlappende Gene, Polyprotein-Gene und verschachtelte Gene. Im Falle überlappender Leseraster kann eine protein-codierende DNA-Sequenz auf bis zu 3 Arten abgelesen werden, und jede dieser Arten führt zu einem eigenen Protein. Zum Beispiel könnte die Basenfolge ATTGCGCTAGTG erstens als …ATT-GCG-CTA-GTG…, zweitens als …ATTG-CGC-TAG-TG…, und drittens als …AT-TGC-GCT-AGT-G… gelesen werden. Im Falle überlappender Gene enthält die protein-codierenden Region des einen Gens eine Kontrollregion (z.B. einen Promotor) für die protein-codierende Region eines weiteren, angrezenden Gens. Bei verschachtelten Genen steckt sogar ein ganzes Gen innerhalb eines anderen, nämlich in einem von dessen Introns. Diese Situation widerspricht der klassischen Ansicht, wonach Gene linear auf Chromosomen aufgereiht sind. Polyprotein-Gene schliesslich sind Gruppen von Genen, die zusammen für ein einzelnes Protein codieren. Dies widerspricht der klassischen "Ein Gen- ein Protein" Hypothese. In den 70er-Jahren setzte in der Molekularbiologie ein technologischer Boom ein, der neue Dimensionen der genetischen Manipulation eröffnete. Auf diesem Fortschritt basiert der Grossteil des genetischen Wissens, das nach der Blütezeit der molekularen Revolution angehäuft wurde. Zunächst gelang es, mithilfe bestimmter Enzyme aus einem Stück RNA den dazu komplementären DNA-Strang zu syntethisieren. Andere Enzyme ermöglichten das Isolieren bestimmter DNA-Abschnitte, indem sie sie aus dem DNA-Strang herausschnitten. Dieselben Enzyme konnten auch zum Aufschneiden eines DNA-Strangs und dem Einsetzen eines einzelnen DNA-Fragments benutzt werden. Auf diese Weise wurde das Zusammensetzen von DNA-Stücken beliebiger Herkunft zu einem einzigen DNA-Molekül möglich. Man nannte das Resultat "rekombinante DNA" und die Technik zu ihrer Herstellung "Klonierung" (nicht zu Verwechseln mit dem Klonen ganzer Organismen). Um bei diesem Prozess genügend Ausbeute zu erzielen, musste man die rekombinante DNA in einen lebenden Organismus einschleusen, wo sie durch dessen DNA-Replikationsapparat vermehrt wurde, um schliesslich "geerntet" werden zu können. Mit der Erfindung der polymerase chain reaction (PCR) - Technik in den frühen 80er-Jahren konnte dieser Schritt umgangen werden. Die PCR ermöglichte die rapide Vermehrung beliebiger DNA-Stücke auf rein künstlichem Wege (d.h. in vitro, "im Reagezglas"). Die erste kommerzielle Anwendung der Rekombinationstechnologie war die Herstellung künstlichen Insulins zur Behandlung von Diabetes (Johnson, I. S. (1983). "Human insulin from recombinant DNA technology". Science 219 (4585): 632–637. doi:10.1126/science.6337396???). Dabei wurde das menschliche 11 Befreiungsbiologie 2 Insulin-Gen in das Bakterium E. coli eingefügt, welches dann, in Fermentationstanks gezüchtet, zum Massenproduzent des Hormons wurde. Das Verfahren wurde vom Pharmakonzern Eli Lilly in Zusammenarbeit mit einer der ersten Biochtech-Firmen entwickelt, die vom Molekularbiologen Boyer gegründete "Genentech". Rekombinantes Insulin ist heute die Hauptform dieses Hormons für den therapeutischen Gebrauch. Mitte der 70-er Jahre wurde ein weiterer entscheidender Schritt in Richtung Kontrolle des genetischen Materials getan. Verschiedene Labors entwickelten Methoden zur Sequenzierung von Genen, d.h. zur Bestimmung der Reihenfolge ihrer DNA-Basen. Die ersten Genome, die komplett sequenziert wurden, gehörten Bakteriophagen und Viren. Die Arbeit war langwierig. Alles musste von Hand verrichtet werden. 1986 wurde der erste halb-automatische DNA-Sequenzierer hergestellt, und schon im Jahr darauf kam das erste vollautomatische Gerät auf den Markt. Damit war der Traum, auch das menschliche Genoms vollständig und in nützlicher Frist zu sequenzieren, in Reichweite gerückt. Das Humangenomprojekt In den 80er-Jahren wurden bereits die ersten Fäden für das Humangenomprojekt (HGP) gezogen, das in den 90er-Jahren durchgeführt und anfangs des neuen Jahrtausends offiziell abgeschlossen wurde. Konsortien wurden gegründet und Konferenzen abgehalten, zunächst, um die Machbarkeit des Projekts zu evaluieren, später dann, um dessen Durchführung institutionell zu strukturieren. Auf internationaler Ebene organisierten sich die beteiligten Länder in der Human Genome Organization (HUGO). In den USA schaffte das Nationale Gesundheitsinstitut (NIH = “National Institut of Health”) das nationale Zentrum für Genomforschung, dessen erster Direktor niemand Geringerer als der Mitentdecker der DNA-Struktur James Watson war. Doch die Entschlüsselung des menschlichen Genoms blieb kein rein öffentliches Unterfangen. 1998 verliess der geschäftstüchtige Craig Venter das HGP und trieb die Genomsequenzierung in seiner Privatfirma "Celera Genomics" weiter. Von da an lieferten sich Venter und das internationale HGP ein Wettrennen. Am Ende stand ein arrangiertes Unentschieden. Am 26. Juni 2000 verkündeten USPräsident Bill Clinton und der britische Premierminister Tony Blair gemeinsam, dass ein Grobentwurf des Genoms fertiggestellt sei. Venters Celera und das staatliche HGP kündigten ebenfalls zeitgleich, im Februar 2001, die Publikation ihrer Rohentwürfe an. Venter publizierte in Science, das HGP in Nature. Vollständigere Fassungen wurden 2003 und 2005 verfügbar, doch selbst heute noch bleiben einige Prozente des Humangenoms unsequenziert. Das HGP ist das bisher teuerste und aufwändigste Projekt der Wissenschaftsgeschichte. Es verschlang drei Milliarden Dollar, also ca. 1 Dollar pro Basenpaar des menschlichen Genoms. Ende der 80er Jahre begannen seine Proponenten, aggressiv öffentliche Gelder einzuwerben. Sie bedienten sich dazu einer charakteristischen Rhetorik, die das Genom als Schlüssel zum Leben darstellte, dessen Entschlüsselung revolutionäre Fortschritte im Verständnis und der Behandlung von Krankheiten versprach (???). Das Genom wurde zu etwas Sakralem hochstilisiert und seine Sequenzierung zu einem Vordringen in die tiefsten Geheiminsse des Lebens, oder gar zur Suche nach dem heiligen Gral (???). Politik und 12 Befreiungsbiologie 2 Medien, euphorisiert von dieser Vision, schenkten den Verheissungen der Genetik Glauben. Gelder flossen. Aber es war mehr als die Hoffnung, dem Geheimnis des Lebens auf die Spur zu kommen, die den enormen Reiz des Projekts ausmachten. Seit den Anfängen der Gentechnik war klar, dass sie privaten Unternehmern immense Profitmöglichkeiten eröffnete. Der Weg zum grossen Geld waren Patente, mit denen kommerzielle Firmen gentechnische Anwendungen zu vermarktbaren geistigen Eigentum machen konnten. Im HGP winkte die Möglichkeit, Gensequenzen patentieren zu lassen, die für die krankmachende Unter- oder Überproduktion wichtiger Proteine verantwortlich waren. Als Patentinhaber könnte man im Falle eines defekten Gens das fehlende Protein als Medikament vermarkten, wie es bei der Herstellung künstlichen Insulins getan wird. Im Falle eines überaktiven Gens könnte man ein Produkt entwickeln, welches das überschüssige Protein abbaut. Man konnte dazu das gesamte Genom abgrasen, und drei Milliarden Basenpaare mussten jedem unternehmerisch angehauchten Genetiker wie eine Fundgrube für potentielle Patente erscheinen. Zahlreiche am HGP beteiligten Wissenschaftler hatten ihr Geld in der Biotechnologie stecken, sei es als Gründer, Direktoren oder auch nur Aktionäre von Biotech-Firmen. Der berühmte Genetiker und Wissenschaftskritiker Richard C. Lewontin schrieb in den 90erJahren, dass er keinen prominenten Molekularbiologen kenne, der nicht irgendeinen finanziellen Einsatz im Biotech-Geschäft hätte. Als Beispiel nannte er einen früheren Harvard-Professor, dessen Gentechnik-Firma über 500 Gen-Patente beantragt hatte (ain’t nec so p. 181???). Nobelpreisträger Watson musste im Rahmen einer Untersuchung zu seinen Anteilen an verschiedenen Biotech-Unternehmen sogar von seinem Posten als Direktor des nationalen Zentrums für Genomforschung der USA zurücktreten (ain’t nec so p. 163/4???). Die beispiellose Verflechtung von Wissenschaft und Wirtschaft im HGP war einer seiner problematischsten Aspekte. Das Potential für Interessenskonflikte war gross. Dass Wissenschaft allgemein - und ein Projekt dieses Ausmasses im Speziellen - wesentlich durch das persönliche Profitstreben von zu Unternehmern gewordenen Biologen gesteuert werden könnte, wirft grundsätzliche Fragen über Ethik und Verantwortung in den Wissenschaften auf. In unserem Zusammenhang interessiert vor allem, wie sich die Proponenten des Projekts gezielt einer Rhetorik des semantischen, deterministischen Gens bedienten, um einen machtvollen ideologischen Kontext zu kreieren, der ihren Ambitionen den Weg ebnete. Darauf werde ich im Kapitel zum Aufstieg der Informationsmetapher zurückkommen. Gentherapie Schon in den 90er-Jahren wurde eines der erhofften Wunder aus der Zauberkiste der Genetik intensiv verfolgt: die Heilung von Krankheiten durch Korrektur der zugrundeliegenden defekten Gene. Das Prinzip der Gentherapie schien einfach: da Gene die Spezifikation des Aufbaus und Funktionierens eines Organismus sind, müssen sie auch seine Anfälligkeit für verschiedenste Krankheiten repräsentieren. Wird die jeweilige genetische Grundlage einer Krankheit entdeckt, braucht man nur die fehlerhaften Gene durch funktionierende zu ersetzen. Die Schwierigkeit dabei war, die "gesunde DNA" so in den lebenden Organismus einzuschleusen, dass sie in dessen Genom integriert wurde und 13 Befreiungsbiologie 2 das fehlerhafte Gen ersetzte. Eine technische Herausforderung, und ohne Zweifel eine anspruchsvolle, aber eine, der sich die vom in Aussicht stehenden Erfolg beflügelten Forschergruppen voller Elan stellten. 1990 fand am NIH der erste klinische Gentherapie-Versuch bei Krebspatienten statt. Durchgeführt wurde er von W. French Anderson, der behauptete, Gene könnten sicher in Zellen eingeschleust werden und sich über die regulatorischen Hürden, die der Gentherapie entgegen gestellt wurden, beklagte. Zwei Jahre später wurde am medizinischen Institut der University of Pennsylvania das Institut für Humangentherapie gegründet. Der Leiter des medizinischen Instituts, William Kelley, verkündete: „There is increasing evidence that many of the most important breakthroughs in medicine will occur in the area of gene therapy, revolutionizing medicine as we know it today.“ (Zitat aus Moments of Truth???) Der Rest der Welt schien dem zuzustimmen, wurden doch ab 1990 weltweit über 400 Gentherapieversuche durchgeführt. Die Gentherapie funktionierte aber nicht. Im Gegenteil, sie war ein niederschmetternder Misserfolg. Nichts stellte sich als einfach heraus. Die eingeschleuste DNA wurde vom Körper verschluckt und verschwand vom Radar. Die Versuchspersonen reagierten nicht mit der erhofften Verbesserung ihrer Symptome, und so war klar, dass mit der zugeführten DNA alles Mögliche geschehen sein konnte, nur nicht der erfolgreiche Einbau ins Wirtsgenom. Bereits 1998 sagte ein desillusionierter W. French Anderson an einem Konferenzvortrag: „Organisms have spent thousands of years learning how to protect themselves from having exogenous DNA get into their genomes. So we were all a little naive to think that if we made a viral vector and put it into the human body it would work.“(Zitat aus Moments of Truth???) Was ein Jahr später geschah, sollte dem von Anderson konstatierten Misserfolg der Gentherapie eine tragische Dimension verleihen. Am Institut für Humangentherapie begann James Wilson, ein Kollege Kelleys an der University of Pennsylvania, mit Therapieversuchen bei einer erblichen Stoffwechselkrankheit namens OrnithinTranscarbamylase-Mangel (OTC). Er benutzte ein modifiziertes Schnupfenvirus, um die "gesunden" Gene in die Körper der Patienten zu schleusen. Eine seiner Versuchspersonen war der 18-jährige Jessie Gelsinger, der seine Krankheit bislang mit Medikamenten und einer passender Diät in Schach gehalten hatte. Gelsinger reagierte auf die Injektion des Virus mit einem kompletten systemischen Organversagen und nachfolgendem Hirntod. Dieser Fall zeigte in drastischer Weise, dass man von einem Verständnis der genetischen Vorgänge im menschlichen Körper noch weit entfernt war; und wie gefährlich es war, mit einem unverstandenen biologischen System herumzuspielen. Im Nachhall der Ereignisse stoppte die amerikanische Drogenaufsichtsbehörde FDA sämtliche klinischen Versuche an der University of Pennsylvania. Das Institut für Humangentherapie wurde zum Tierversuchslabor umfunktioniert und das NIH revidierte die Sicherheitsrichtlinien für Gentherapieversuche. Die Familie Gelsingers prozessierte gegen die Universität und gegen Wilson, der später von seinem Amt als direktor des Instituts zurücktrat. Im Zuge weiterer Untersuchungen wurde bekannt, dass auch andere Gentherapie-Forscher Nebenwirkungen und medizinische Komplikationen ihrer Versuche 14 Befreiungsbiologie 2 unterschlagen hatten. Es wurde vermutet, dass es mindestens sechs weitere Todesfällge gegeben hatte. (Zitat aus Moments of Truth???) Bei den Forschern machte sich Ernüchterung breit, während bei den Patienten die Bereitschaft, an weiteren Versuchen teilzunehmen, drastisch sank. Um die Jahrtausendwende verschwand die Gentherapie von der Bildfläche, jedenfalls von derjenigen der medialen Öffentlichkeit. Im Stillen wird Gentherapieforschung auf kleiner Flamme und mit heruntergeschraubten Erwartungen weiter betrieben. Zweifellos könnte sie in manchen Fällen funktionieren, und es ist zu hoffen, dass sie es eines Tages wird. Heute, fünfzehn Jahre später, haben wir statt der Gentherapie die "personalisierte Medizin" als neueste Inkarnation der Hoffnungen einer genzentrierten Biomedizin. Die personalisierte Medizin hat inhaltlich keinen besonderen Bezug zur Gentherapie, aber die Hoffnungen und Versprechungen, die sie macht, sind aus einem Guss mit denjenigen, die im Umfeld des HGP gemacht wurden (z.B. http://www.uzh.ch/news/articles/2014/neue-generation-von-biomedizinern.html???). Es wird mit der Aussicht auf umwälzende Fortschritte in der Behandlung von Krankheiten geworben, die uns in ein oder zwei Jahrzehnten bevorstehen sollen. Die Idee der personalisierten Medizin ist eine gute: jedem Patienten eine massgeschneiderte Behandlung zukommen zu lassen, die auf dessen individuelle Konstitution und Situation abgestimmt ist. Es sollen sämtliche verfügbaren gesundheits- und krankheitsrelevanten Daten in einem individuellen Profil gesammelt und in die Ausarbeitung einer spezifischen Behanldung einbezogen werden. Manche der Daten sind herkömmliche und betreffen zum Beispiel die Krankheitsgeschichte und die familiäre Belastung. Daneben sieht die Vision der personalisierten Medizin aber auch vor, neue Hochleistungsapparate zur Überwachung und Datensammlung in Echtzeit einzusetzen, die auf Abweichungen vom Normalzustand sofort reagieren können. Da individualisierte Behandlung unter Berücksichtigung aller verfügbarer Informationen im Prinzip etwas ist, was Ärzte und Therapeutinnen schon immer getan haben, könnte man die personalisierte Medizin lediglich als alten Wein in neuen Schläuchen abtun. Doch selbst wenn dem so wäre, so ist doch die Grundidee nach wie vor eine exzellente, und ihr neues Leben einzuhauchen ein begrüssenswertes Unterfangen. Zudem sind die Hochleistungs-Patientenüberwachsungssysteme ein bisher nicht da gewesener und potentiell interessanter Aspekt dieser Idee. Was hier jedoch von grösserem Interesse ist, ist die Tatsache, dass sich für manche ihrer Proponenten die personalisierte Medizin als hauptsächlich genzentriertes Behandlungsprinzip darstellt. Das heisst, die Hauptquelle behandlungsrelevanten Wissens soll in einem individuellen genetischen Profil des Patienten bestehen. Dieser Ansatz entstand aus der sog. Pharmakogenetik, die mit dem Versuch, Medikamentendosen auf individuelle genetische Parameter des Patienten abzustimmen, einige Erfolge erzielte. Ein Beispiel ist das Medikament Herzeptin gegen Brustkrebs, dessen Wirksamkeit auf eine Gruppe von Patientinnen beschränkt ist, deren Tumoren zuviel von einem Protein namens HER2 produzieren, da das Gen für HER2 überaktiv ist. Ende der 90er-Jahre kam Herzeptin auf den Markt, zusammen mit einem Test für das Vorliegen einer HER2-Überproduktion, und wird seither erfolgreich angewendet. Hier wurde also Information über die Aktivität eines Gens als Basis für die Behandlung einer Krankheit eingesetzt. Das Beispiel ist simpel, aber vielen erschien es als ein Möglichkeitsbeweis für eine Zukunft der genbasierten personalisierten Medizin. Gegenwärtig kämpft die personalisierte Medizin um ein klares Profil, eine solide konzeptuelle Basis und eine realistische Einschätzung ihrer künftigen Möglichkeiten (Gamma 2013???). Wie die Gentherapie und das HGP ist auch sie zu einem Hype geworden, den manche Beteiligten, teilweise zur Beibringung von Forschungsgeldern, durch spektakuläre Prognosen selber nähren, während andere sich mit 15 Befreiungsbiologie 2 dessen schädlichen Auswirkungen auf die Glaubwürdigkeit ihrer Wissenschaft herumschlagen müssen. 16