Proteinabbau in der Zelle
Werbung

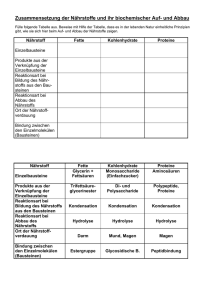
Proteinabbau in der Zelle Intrazelluläre Proteasen Membran-Proteasen Lysosomale Proteasen (Cathepsine) Proteasom-System (Zytosol / Nukleus) Mitochondriale Proteasen Calpaine Caspasen Prozessierende Proteasen Die Halbwertzeit (t1/2) von Proteinen ist sehr variabel Kurzlebige Proteine: Ornithindecarboxylase: <0,2 h RNA-Polymerase I: 1,3 h Tyrosin-Aminotransferase: 2,0 h Serin-Dehydratase: 4,0 h Langlebige Proteine: Aldolase: 118 h Cytochrom b: 130 h Laktat-Dehydrogenase: 130 h Immunglobulin G: 21 Tage Aktin (Skelettmuskel): ca. 3 Monate Linsen-Crystallin: >80 Jahre (Dice and Goldberg, 1975) Wann werden Proteine abgebaut? • Geschädigte, denaturierte Proteine • Aufrechterhaltung des Aminosäurepools • Prozessierung von Vorläuferproteinen • Signaltransduktion (Regulator-Proteine) • Immunantwort • Zellproliferation und Zelltod Lysosom- und Proteasom-vermittelte Proteolyse 1. Endogene Proteine 2. Exogene Proteine (Marker für Abbau) Proteasom-Weg Peptidasen Peptide, Aminosäuren Endosom/ Lysosom-Weg Lysosomen enthalten saure Hydrolasen 0,1-0,5 µM pH 5,2 Proteasen Nukleasen Glycosidasen Lipasen Phospholipasen Phosphatasen Sulphatasen H+ ATP saure Hydrolasen Cathepsine Cytosol pH 7,2 ADP + Pi Area densa elektronendichtes, agglutiniertes Material Proteindegradation im Lysosom 1-3: Heterophagie 4-5: Autophagie Rezeptor-Liganden Komplex Plasmamembran 1.) Rezeptor-vermittelte Endozytose Exogenes Protein 2.) Pinozytose 3.) Phagozytose Frühes Endosom Lysosom Multiv esikuläre s Körperchen 4.) Makroautophagie Mitochondrium 5.) Mikroautophagie Autophagozytose bei Hunger Invaginationsmembran LC3 Marker Autophagische Vakuole Autophagosom LC3 Marker Normal Hunger Aktuelle Forschung: Gestörte Autophagie und Tumorentwicklung Das Ubiquitin-Proteasom-System Ubiquitin • Abbau von zelleigenen Proteinen. • Selektiver und fein regulierter Prozess. Su 2-Stufen Prozess: bs tra t 1.) Substrat-Proteine werden für den Abbau mit Ubiquitin-Molekülen markiert. 2.) Abbau erfolgt über das Proteasom. Viele regulatorische Proteine werden in ihrer Konzentration moduliert. Proteasom Nobel-Preis für Chemie 2004 ”für die Entdeckung Ubiquitin-vermittelter Proteindegradation" Aaron Ciechanover Technion, Haifa Avram Hershko Technion, Haifa Irwin Rose, Irvine, CA, USA „Der Verlierer“ Alexander Varshavsky Auftrennung eines Retikulozyten-Lysats: • Fraktion 1: hitzestabil, ca. 9 kD, (=Ubiquitin). • Fraktion 2: hitzelabil (= Enzyme). 1.) ATP ist notwendig. 2.) Mischen der Fraktionen rekonstituiert proteolyt. Aktivität. 2 Komponenten beteiligt Wie werden Substrate ausgewählt? Ubiquitin: the kiss of death • • • • • • “Stigma” für den Abbau. 8,5 kD (76 AS) Polypeptid. vorhanden in allen eukaryontischen Zellen. hoch konserviert. Lys48 Ub C-terminales Glycin bindet kovalent an Lysin des Substrates (Isopeptidbindung). Lys-48 für Verknüpfung der Polyubiquitinkette. Lysin C-Terminus Gly76 wird mit e-NH2-Gruppe bestimmter Lysine des Substratproteins verknüpft. Die Ubiquitinylierung ist eine 3-Stufen-Reaktion 1.) E1: Ub-aktivierendes Enzym 2.) E2: Ub-konjugierendes Enzym 3.) E3: Ub-Protein Ligase E3 erkennt das spezifische Targetprotein und transferiert Ub auf Lys e-NH2-Gruppe des Substrates. Das 26 S Proteasom, der “Schredder” 20S-Partikel 19S-Partikel katalytisch 19S (Deckel) a b b a 20S (Kern) 19S (Deckel) S = Svedberg-Konstante (Sedimentationskoeffizient) regulatorisch + 26S Der Kern-Partikel (20S) • 2 Kopien von 14 Proteinen. • 7 Proteine bilden eine Ringstruktur. • 4 Ringe (“donuts”) liegen übereinander. • bestimmte ß-Proteine sind proteolytisch aktiv. Die regulatorischen Partikel (19S) • 2 regulatorische Partikel an jedem Ende. • ebenfalls aus 14 Proteinen zusammengesetzt. • dienen der Substrat-Erkennung u. Entfaltung. Abbau im Proteasom - Bindet Ub-konjugiertes Substrat. - Isopeptidase spaltet Ub vom Protein. - Unter ATP-Verbrauch wird Substrat entfaltet (6 ATPasen im 19S-Partikel). a b b 3 Threonin-Proteasen a Proteasom ist multikatalytisch: • Trypsin-Aktivität (Arg, Lys) • Chymotrypsin-Aktivität (Tyr, Phe) • Postglutamyl-Aktivität (Glu, Asp) Kontrolle des Proteinabbaus über die AS-Sequenz 1.) Die N-End-Regel (Degrons) • destabilisierende N-terminale AS (z.B. Arg, Lys, His) • stabilisierende N-terminale AS (z.B. Ala, Gly, Met) 2.) „Destruction Box“ in mitotischen Cyclinen 3.) PEST-Sequenz Proteine mit vielen Pro (P), Glu (E), Ser (S), Thr (T) werden schneller abgebaut. Aktivierung von Degradationssignalen Beispiele: Phosphoylierung des Substratproteins Dissoziation eines Inhibitors E1, E2 & E3 Hierarchie erhöht die Spezifität: • E1: nur 1-3 Enzyme. • E2: ca. 30 homologe Proteine, z.T. unterschiedliche Lokalisation. • E3: >300, oft selektiv für einzelne Substrate, mehrere Familien, in unterschiedlicher E2/E3-Kombination, hochmolekulare Komplexe (garantiert biologische Spezifität). RING Finger E3-Ligasen HECT E3-Ligasen F-Box E3-Ligasen E3 Ligasen werden auch aktiviert Phosphorylierung Liganden-Bindung Allosterische Aktivierung Exkurs: Proteinmodifikation durch Ub-verwandte Prozesse • Mono- und Diubiquitinylierung. • Sumoylierung (Sumo: small ubiquitin-related modifier). • Neddylierung (Nedd). • Autophagosomen-Bildung (Apg-12) • u.a. Ub-verwandte Modifikationen werden: • über E1, E2 und E3-Kaskaden reguliert. • führen nicht zur Proteindegradation, • oft antagonistisch zur Polyubiquitinylierung • regulieren z.B. Transkription, Proteintargeting, DNA-Reparatur, Endozytose u.a. Übersicht: Proteasom–vermittelte Proteolyse in zellulären Prozessen Elimination falsch gefalteter Proteine (ERAD) Signaltransduktion, Transkriptionsfaktoren Entzündungsregulation Zellzyklusprogression DNA-Reparatur Programmierter Zelltod (Apoptose) Circadiane Rhythmik (biologische Uhr) Antigenprozessierung u. -präsentation (MHC-I) Der Transkriptionsfaktor NF-kB wird durch phosphorylierungsinduzierte Proteolyse aktiviert Prinzip: Aktivierung von NF-kB durch Dissoziation seines Inhibitors NF-kB-Aktivierung durch Stressfaktoren: • Zytokine • Bakterien,Viren verstärkte Aktivierung in: • Entzündung: rheumatoide Arthritis, Morbus Crohn. • Tumoren, z.B. Hodgkin-Lymphom. Phosphorylierung des Inhibitors IkB stimuliert Ubiquitinylierung. IkB Degradation NF-kB KernTranslokation Targetgene: viele proinflammatorische und anti-apoptotische Gene Zellzyklus: Zyklischer Abbau von Cyclinen Mitose-Phase (Chromosomentrennung) Gap-2 Cyclin-Abbau durch das Proteasom aktiv Gap-1 Cyclin B Synthese-Phase (DNA-Replikation) Cdk1 (Cyclindependent kinase) inaktive Cdk1 Proteasom und DNA-Reparatur Tumorsuppressor p53: Wächter des Genoms Normale Zelle DNA-Schaden STOP p53: Transkriptionsfaktor Stabilisierung STOP p53 Abbau von p53 durch Mdm2 p53 E3 Ligase Mdm2 Aktivierung von p53 Targetgenen p53 p53 Abbau G1 Checkpoint Sind DNA Schäden vorhanden? Ist die Umgebung günstig? Ist die Zelle groß genug? Arrest, Apoptose DNA-Reparatur Tumorsuppression p53 ist in >50% von Tumoren mutiert u. inaktiv Tumorzelle STOP p53 Mutation Papillomviren Kontrollpunkte defekt E6 Mdm2 Amplifikation E3 Ligase E3 Ligase E6-AP Mdm2 p53 p53 inaktiv STOP p53 p53 p53 Abbau p53 Abbau G1 Checkpoint Sind DNA Schäden vorhanden? Ist die Umgebung günstig? Ist die Zelle groß genug? Arrest Apoptose Tumorgenese Ein Proteasom-Inhibitor in klinischer Anwendung Velcade® (Bortezomib) • Zulassung: USA 1/2003, Europa 4/2004 für multiples Myelom. • Zahlreiche klinische Studien für andere Anwendungen Knochenmarksausstrich Multiples Myelom (Plasmacytom) Targets von Velcade®/Bortezomib Hemmung des Zellzyklus (Cyclinabbau). Aktivierung der Apoptose (p53-Stabilisierung). Hemmung von NF-kB (Apoptose-Schutz blockiert). In Zukunft: Entwicklung selektiverer Wirkstoffe, z.B. spezifische E3-Ligase-Hemmer Zusammenfassung Lysosomen verdauen über Endosomen oder Autophagosomen eingeschleuste Proteine. Abbau endogener Proteine erfolgt i.d. R. über das fein regulierte und selektive Ubiquitin/Proteasom-System. Proteine werden unter ATP-Verbrauch durch Isopeptid-Bindung mit mehreren Ub-Molekülen markiert (E1, E2, E3 Enzyme). Unterschiedliche E2/E3-Enzyme garantieren biologische Spezifität. Proteasom-vermittelter Abbau spielt eine regulatorische Rolle in nahezu allen biologischen Prozessen. Ub-verwandte Modifizierungen (z.B. Sumo) führen nicht zum Proteinabbau, sondern besitzen andere Aufgaben. Proteasom-regulierte Signalwege sind in verschiedenen Erkrankungen gestört und bilden neue Drug-Targets.