Monoklonale Antikörper
Werbung

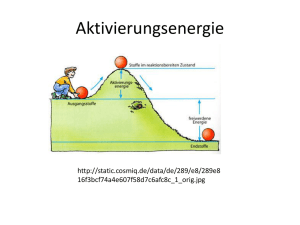
Tumor – Geschwulst – Neoplasma (Neubildung) Abnorme Gewebsmasse, entstanden durch Vermehrung körpereigener Zellen Regulationsstörung Zellteilung (Proliferation) Zellverlust (Apoptose) Tumorwachstum: autonom irreversibel, progressiv, nicht mit normalem Gewebe koordiniert Invasion, Destruktion Metastasen Stoffwechselprodukte (Hormone) Aufbau Tumorparenchym Tumorzellen Tumorstroma Bindegewebe Gefäße Entzündungszellen: Lymphozyten, Makrophagen Dignitätsbeurteilung Merkmal Benigner TU Maligner TU Wachstumsrate Langsam, wenige Mitosen Langsam bis schnell, Mitosen ev. zahlreich, atypische Mitosen Lokale Ausbreitung Schlecht begrenzt Zusammenhängend Invasiv, destruierend Gut begrenzt Expansiv. Verdrängend Kapsel Histologie Hoher Differenzierungsgrad Meist monomorph Differenzierungsverlust Zellatypien Umgebung Kompression Druckatrophie Hormonsekretion Invasion, Destruktion Rezidive Metastasen Zelluläre Atypien (I) Zellpolymorphie: Variable Zellgröße, -form Anisonukleose (-karyose): Variable Kerngröße Kernpolymorphie Unterschiedliche Kernform Kernhyperchromasie: Vergröbertes, stärker anfärbbares Chromatin Erhöhter DNA-Gehalt Zelluläre Atypien (II) Mitosefiguren Vermehrtes Auftreten Atypische Formen: tri-, tetrapolar Nucleolenvergrößerung Verschiebung der Kern/Plasma-Relation Zugunsten des Kerns Vermehrte zytoplasmatische Basophilie Vermehrte RNA – Proliferation Polyploidie: [4N], [8N] Aneuplodie: Hyperdiploid [2,5N], triploid [3N] Intraepitheliale Neoplasie (in-situ Karzinom) Präinvasives Karzinom Proliferation atypischer, neoplastischer Zellen innerhalb des ursprünglichen Epithelverbandes Entwicklung in (mikro-) invasives Karzinom Entfernung verhindert Enstehung eines metastasierungsfähigen Tumors Krebsrisikofaktoren: Umwelt Chemische Verbindungen Zigarettenrauch: Lungenkarzinom Asbest: Pleuramesotheliom Aflatoxin (Schimmelpilz): Leberkarzinom Ernährung: Colonkarzinom Chronische Infektionen Helicobacter pylori: Magenkarzinom Hepatitis B, C: Leberkarzinom HPV: Cervixkarzinom Strahlen: UV: Basaliom, Plattenepithelkarzinom Ionisierende Strahlen: Schilddrüsenkarzinom Helicobacter pylori Krebsrisiko: Genetische Faktoren Angeboren: Keimbahnmutationen Erworben: Somatische Mutationen Onkogene Tumorsuppressorgene Apoptosegene Telomerasegene DNA-Reparaturgene Onkogene (Proto-Onkogene) Normale zelluläre Gene Genprodukte regeln Proliferation Differenzierung Mobilität Mutationen führen zu konstitutiver Aktivierung, „gain of function“, „dominante“ Mutation z.B.: Aktivierung eines Wachstumsfaktor-Rezeptors Onkogengruppen (I) Autokrine Wachstumsfaktoren Wenn Rezeptoren vorhanden – autokrine stimulation Für maligne Transformation allein nicht ausreichend Wachstumsfaktorrezeptoren Extrazelluläre Ligandenbindungsdomäne Transmembranöse Region Intrazelluläre katalytische Domäne, meist mit Protein (Tyrosin-) Kinaseaktivität erbB2(Her-2/neu): Amplifikation Mammakarzinom, Ovar, Magen Mammakarzinom: HER2 +++ FISH IH Onkogengruppen (II) Signaltransduktionskette Molekulare Schalter, Signalverstärkung Rezeptor-Tyrosinkinasen RAS: zentrale Position in zahlreichen Neoplasien durch Mutation permanent aktiviert Zytoplasmatische/nucleäre Tyrosinkinasen Abl-Kinase Translokation BCR-ABL Onkogengruppen III Transkriptionsfaktoren Binden an regulatorische Sequenzen von Zielgenen Regulieren Transkription MYC-, FOS-, JUN-Genfamilien, E2FTranskriptionsfaktor MYC Neuroblastom Prognostisch ungünstig Neuroblastom – Risikofaktoren N-myc Southern-blot 1,2: pos. Ko. (IMR) 3,4: normale DNA 5: Patient 1 6: Patient 2, 10μg 7: Patient 2, 5μg 8: Patient 2, 2μg DNA Aktivierungsmechanismen von Onkogenen Amplifikation: DNA-Vermehrung N-MYC, Her2/NEU Chromosomale Translokationen (Rearrangement) Philadelphia-chromosom t[9;22]: CML BCL-2/IgH t[14;18]: follikuläres Lymphom SYT/SSX1,2: Synovialsarkom Punktmutation H-, Ki-, N-RAS, MEN1, RET Integration einer nicht onkogenen Sequenz HBV: Leberkarzinom Deregulierung der Onkogenexpression Promotor-(De)Methylierung ? Tumorsuppressorgene Normale zelluläre Gene Negative Regulation von wachstumsassoziierten Funktionen „rezessives“ Verhalten bei Mutation Retinoblastomgen (RB-1): Nukleäres Phosphoprotein (p105RB) Regulation d. Zellzyklus P53 – „Guardian of the Genome“ Nukleärer Transkriptionsfaktor Bei Auftreten eines genetischen Schadens hochreguliert, Aktivierung von Zielgenen: Arretierung in G1 Aktivierung von DNA-Reparaturmechanismen: GADD45 Behebung des DNA-Schadens MDM2 inaktiviert p53 Wenn „irreparabel“ ÄApoptose Mutationen von p53 Dickdarm-, Lungen-, Magen-, Mammakarzinom Verlängerte Halbwertszeit, IH nachweisbar Funktionsverlust Akkumulation genetischer Schäden Wachstumsvorteil Blockade der Apoptose (u.a. Resistenz gegen Chemotherapie) Mechanismen der Apoptose Programmierter Zelltod Durch Zelle selbst initiiert, gesteuert Caspasen: ausführende Enzyme Zerstörung von Proteinen Fragmentierung der DNA Aktivierung durch Liganden von Transmembranrezeptoren Proteine der äußeren Mitochondrienmembran Hemmung durch Bcl-2 (Gegenspieler: BAX) Telomere, Telomerasen Fibroblastenkultur: Wachstumsstillstand nach 50 Zellteilungen „mortality stage 1“, M1 Telomerenverkürzung, p53 Aktivierung Wenn p53 inaktiv: 20 weitere Zellteilungen mit weiterer Telomerenverkürzung „mortality stage 2“, M2, „crisis“ Aktivierung von Telomerasen in einigen Zellen Stablilisierung der Telomerenlänge, Immortalisierung DNA Reparaturgene Reparatur von Basenfehlpaarungen, entstanden bei DNA-Replikation Mutagene Einflüsse Störung: genetische Instabilität „Mutatorphänotyp“ MutHLS-System (E.coli): Max. 4 bp repariert Exonuklease, Helicase II, DNA-Polymerase III, DNA-SSB(single strand binding protein), DNA-Ligase Humane Gene: hMSH2, hMLH1, hPMS1, hPMS2 Angeborener Defekt: Xeroderma pigmentosum, HNPCC HNPCC – Hereditary Non-Polyposis Colon Carcinoma Junge Patienten Colon ascendens Muzinöses Adenokarzinom Mutation in DNA-Reparaturgen (hMSH2, hMLH1, hPMS1, hPMS2) Hohe Mutationsrate Mikrosatelliteninstabilität Loss of MMR – protein expression hMLH1 HNPCC MSIAnalyse BAT26 D5 S346 BAT 25 (APC) D17 S250 (Mfd 15CA) D2 S123 Normalgewebe 6-FAM BAT 25: 110-130bp D2 S123: 200230bp TET BAT 26: 100-120bp D17 S250: 140170bp HEX D5 S346: 100130bp TAMRA Interner Standard Tumor: MSS HNPCC MSI-Analyse BAT 25 BAT2 D5 S346 (APC) 6 D5 S346 (APC) Interner Standard NORMALGEWEBE NORMALGEWEBE TUMORGEWEBE: MSI-H TUMORGEWEBE: MSI-H