Phytopathologie
Werbung
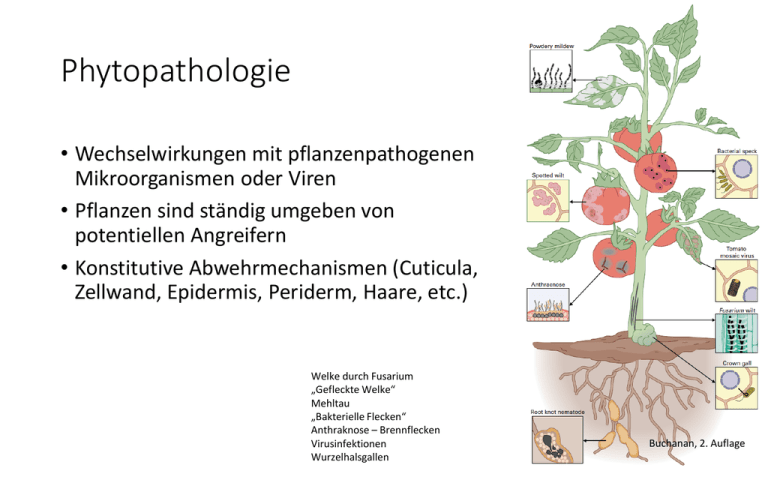
Phytopathologie • Wechselwirkungen mit pflanzenpathogenen Mikroorganismen oder Viren • Pflanzen sind ständig umgeben von potentiellen Angreifern • Konstitutive Abwehrmechanismen (Cuticula, Zellwand, Epidermis, Periderm, Haare, etc.) Welke durch Fusarium „Gefleckte Welke“ Mehltau „Bakterielle Flecken“ Anthraknose – Brennflecken Virusinfektionen Wurzelhalsgallen Buchanan, 2. Auflage Phytopathogene • Sind auf Wirte spezialisiert • Monokulturen fördern Evolution von neuen Pathogenrassen • Pathogene können im Boden verbleiben Formen der Lebensweisen • Biotrophe Mikroorganismen und Viren: • Hohe Wirtsspezifität, brauchen den lebenden Wirt • Z.B. Mehltau • Nekrotrophe Mikroorganismen: • Geringere Spezifität, töten Wirt durch Toxine ab und ernähren sich saprophytisch • Z.B. Botrytis • Hemibiotrophe Mikroorganismen • Zuerst biotrophe Lebensweise, dann töten sie Wirt ab • Z.B. Phytophthora Fusicoccin – ein Welketoxin • Löst unkontrollierte Stomataöffnung aus • Pilz befällt Mandel- und Pfirsichbäume Strasburger, 37. Auflage „Präformierte Abwehrmechanismen“ • Cuticula • (Verholzte) Zellwände • Periderm • Toxische Verbindungen in Zellwänden und Vakuolen Erkennung von Pathogenen • Inhärente Immunität (Duden: „einer Sache innewohnend“, innate immunity) • Erkennungsmoleküle: MAMP (microbe associated molecular pattern) • Erkennung an membranständigen LRRRezeptorkinasen • Einfachste Form: Nicht-Wirts-Resistenz LRR LRR-Rezeptorkinase: LRR= Leucinreiche Region, Erkennung Rezeptorkinasedomäne Nicht-Wirts-Resistenz • Mikroorganismen verraten sich durch eigene Faktoren (Zellwandbestandteile, Chitin, Proteine (Flagellin)) oder freigesetzte pflanzliche Bestandteile (der Cuticula oder Zellwandbestandteile) – MAMPs • Pflanze in gesundem Zustand kann Pathogen abwehren • Biologischer Eustress • Stärkung der Abwehrmechanismen Infektion der Pflanze • Entweder durch Veränderung der MAMPs oder • Signaltransduktion blockieren • Effektoren werden durch Proteinkanäle in die Zellen gebracht • Pathogen befällt Pflanze Resistenz • Effektor wird erkannt • NB-LRR (NB= Nukleotidbindestelle) Gen-für-Gen-Modell • Effektoren sind Avirulenz-Genprodukte, avr • Rezeptoren sind Resistenzgenprodukte, R • Ziel der Züchter ist es die passenden R-Gene in die Nutzpflanze zu bekommen Strasburger, 37. Auflage Resistenz • Effektor wird erkannt • NB-LRR (NB= Nukleotidbindestelle) • NB-LRR-Proteine haben Überwachungsfunktion in der Zelle • Signaltransduktion zur Hypersensitiven Reaktion (HR) Hypersensitive Reaktion • Programmierter Zelltod • Ausbreitung des Pathogenes wird erschwert • Bildung von Abwehrproteinen • Akkumulation von toxischen Verbindungen, insbesondere Phenole • Ausschüttung von Signalstoffen ① Pathogenantwort • Erkennung: MAMP • Signaltransduktion • Signalmoleküle: PR-Proteine und Phytoalexine • Antwort • PR-Proteine (pathogenisis related), Abwehrproteine • Effektoren (E) blockieren Antwort • Phytoalexine, niedermolekulare Substanzen • SA, Salicylsäure ③ ④ ② ⑤ ⑥ Phytoalexine • Niedermolekulare Verbindungen • Meist antimikrobielle Wirkung • Bildung erst nach Infektion • Resistente Pathogene müssen Phytoalexine abbauen Strasburger, 37. Auflage PR-Proteine (pathogenisis related) • Abwehrproteine, z.B. • • • • • Chitinasen Proteinasen RNasen Peroxidasen, Superoxiddismutasen Defensine: kleine Proteine (<50 Aminosäuren) mit einem basischem und einem hydrophoben Teil →Schädigen die Membranen der Pathogene https://de.wikipedia.org/wiki/Defensin ① Pathogenantwort ③ • SA, Salicylsäure löst die SAR aus ④ ② ⑤ ⑥ Systemisch erworbene Resistenz • Lokale Infektion sorgt für erhöhten Salicylsäure in der ganzen Pflanze und erhöhte Abwehrbereitschaft • Systemische Resistenz gegen breites Spektrum von Pathogenen Buchanan, 2. Auflage Phytopathogene Pilze • Biotrophe und nekrotrophe Pathogene • Sporen keimen auf Oberfläche und dringen durch Öffnungen (Stomata, o.ä.), Wunden oder durch die Epidermis ein • Haustorienbildung apsnet.org http://imbi.bio.indiana.edu/ http://journal.frontiersin.org/article/10.3389/fpls.2014.00664/ Haustorien Blumeria (Erysiphe) graminis in cells of grass hosthttps://www.uoguelph.ca/~gbarron/MISC2003/haustori.htm Hyaloperonospora parasitica: Hyphen und Haustorien https://de.wikipedia.org/wiki/Haustorium Phytopathogene Bakterien • Meist fakultative Parasiten • Dringen durch Öffnungen oder Wunden ein • Leben im Apoplasten • Abtöten des Wirtsgewebes nach Erreichen einer kritischen Population (quorum sensing) Apfelbaum mir Erwina Agrobacterium tumefaciens • Wurzelhalstumore • Wirtschaftlich nicht bedeutend – aber wissenschaftlich • Agrobacterium ist ein Bodenbakterium, lebt von organischem Material • Genetische Voraussetzung für Infektion: Ti-Plasmid in 10% der Population Zeitlicher Ablauf der Infektion • Verletzung durch mechanische Einflüsse • Bakterien können Pflanze durch Pflanzenstoffe (z.B. Acetosyringon) lokalisieren • Bakterien gelangen in Pflanze • Binden an Arabinogalactane (Glykoproteine) • Acetosyringon löst Genexpression aus → Vir-Operon Agrobacterium Wirt Zeitlicher Ablauf der Infektion • Expression des Vir-Operons (5) • Einzelstrangkopie der T-DNA (6) →T-DNA zwischen LB und RB (left, right border) • Verpacken (7) und Transport (8) der DNA in Pflanzenzelle • Kernimport (10) • Einbau in Kerngenom (11) Die T-DNA • Promotoren und Terminatoren für pflanzliche Transkriptionsmaschinerie • Gene der T-DNA codieren für • AUX- und CK-Synthese • Opinsynthese AUX CK Opine Tumorentstehung • AUX und CK fördern Zellteilung • Keine Differenzierung • Tumoröses Wachstum Opine • Opine sind Derivate von Aminosäuren • Können von der Pflanze nicht abgebaut werden • Gelangen in den Wurzelraum • Abbau durch Occ-Genprodukte • Dienen Agrobacterium als C- und NQuelle • Tra-Gene ermöglichen Weitergabe des Ti-Plasmids an andere Agrobakterien Synthese Abbau Agrobakterien-vermittelter Gentransfer The Plant Journal: Volume 47, Issue 6, pages 969-976 • T-DNA kann ersetzt werden durch beliebige Gene • Transformation der Blüten oder von Kallusgewebe Viren • Gelangen nur durch Verletzung in Pflanzen • Insekten (oder auch Menschen) • Nebenfarbveränderungen auch eingerollte Blätter, gestauchtes Wachstum, etc. Mosaikviren auf Abutilon (ABMV) Tabak, Kontrolle, TMV-infiziert Tabakmosaikvirus, (TMV) Das Tabakmosaikvirus TMV • ssRNA-Virus • ▼ sind Promotoren für 3 mRNAs (RdRP, CP, MP) • (+)-RNA dient als mRNA für RNAabhängige RNA-Polymerase (RdRP) • RdPR bildet (-)-RNA in VirusReplikationskomplex (VRC) • Gene für Hüllproteine (CP) und Movement-Proteine (MP) werden exprimiert • Neue Viren entstehen oder • Nukleinsäure wird systemisch transportiert Ausbreitung • ssRNA ist an MP (movement protein) gebunden • Ein Helferprotein vermittelt Durchtritt durch Plasmodesmos • MP erweitert Plasmodesmos • Proteinkinase phosphoryliert MP • MP löst sich in Nachbarzelle von RNA • Translation der RNA beginnt sofort nach Durchtritt Blumenkohlmosaikvirus • Cauliflower mosaic virus (CaMV) • Hülle aus einer Proteinisoform • dsDNA-Virus mit 8.000 bp • 8 Gene, davon 6 mit bekannter Funktion Weiler, Nover Blumenkohlmosaikvirus • Virus gelangt in Zelle • Transkription beginnt • 2 mRNAs entstehen (19S und 35S, S ist Maß für das Zentrifugationsverhalten) • 19S-RNA unterstützt 35S Expression • 35S ist polycistronisch • TAV (Gen VI) gewährleistet Reinitiation an internen Start-Codons • 35S-RNA wird dann als Matrize für Reverse Transkriptase verwendet RNA→DNA Blumenkohlmosaikvirus • 35S-mRNA-Promoter ist ein kurzer, starker Promoter in vielen Pflanzen und den meisten Geweben Strasburger, 37. Auflage











