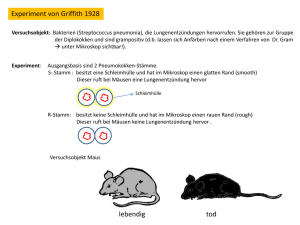
Praktikum Zelluläre Biochemie WS 2013/2014
Werbung

BCII am PEI, 04.11.2013-28.11.2013 Praktikum Zelluläre Biochemie WS 2013/2014 4-wöchiges Praktikum im Bachelor-Studiengang der Biochemie Paul-Ehrlich-Institut Paul-Ehrlich-Str. 51-59 D-63225 Langen Tel.: 06103-77-0 Wichtig: • Alle Teilnehmer bitte am Dienstag, den 29.10.2013 um 10:00 Uhr an der Pforte des Paul-Ehrlich-Institutes melden • Einführungsveranstaltung für alle Teilnehmer am Di. 29.10.2013 , 10:1514:30 Uhr, Praktikumsstart ist am Montag, 4.11.2013 9:00 Uhr BCII am PEI, 04.11.2013-28.11.2013 Praktikumsleitung: Prof. Dr. Christian J. Buchholz Tel.: 06103-774011 Email: [email protected] Prof. Dr. Ralf R. Tönjes Tel.: 06103-774010 Email: [email protected] Beteiligte Arbeitsgruppen Abt. Medizinische Biotechnologie Fachgebiet 6/0 (Dr. Ivics) Fachgebiet 6/3 (Prof. Dr. Schumann) Fachgebiet 6/4 (Prof. Dr. Tönjes) Forschungsgruppen des Präsidenten Pr1 (Prof. Dr. Buchholz) Pr2 (Dr. Mühlebach) Abt. Veterinärmedizin Fachgebiet 4/0 (Dr. von Messling) Fachgebiet 4/3 (Dr. Bastian) Abt. Immunologie Fachgebiet 3/0 (PD Dr. van Zandbergen) Fachgebiet 3/3 (Dr. Kirberg) Nachwuchsgruppen NG1 (Dr. Toda) NG3 (Dr. König) Abt. Virologie Fachgebiet 2/0 (Prof. Dr. Hildt) LOEWE-Professur Prof. Dr. Modlich Inhaltliche Struktur: Jede Gruppe absolviert je ein Experiment Gentransfer (GT), Immunologie (I), Genmodifikation (GM) und Virologie (V). Die Experimente werden parallel in verschiedenen Gruppen angeboten. GT1 Prof. Dr. Buchholz GT2 Dr. Mühlebach GT3 Prof. Dr. Modlich I1 I2 I3 PD Dr. van Zandbergen und Dr. Kirberg Dr. Toda und Dr. Mühlebach Dr. Bastian GM1 Prof. Dr. Schumann GM2 Dr. Ivics GM3 Dr. von Messling V1 V2 V3 Prof. Dr. Hildt Dr. König Prof. Dr. Tönjes 2 BCII am PEI, 04.11.2013-28.11.2013 Gruppeneinteilung BC-II Praktikum WS 2013/14 Gruppe 1 2 3 4 5 6 7 8 9 10 Teilnehmer Bengs, Christian Hirschhäuser, Alexander Forster, Marcel Christian Stach, Oliver Langer, Danny Weber, Miriam Menche, Constantin Weber, Christiane Brunnberg, Jamina Bergs, Amelie Edelmann, Martin Krug, Dennis Steinmann, Annebirth Steinmetz, Tobit Grecht, Michael Frombach, Ann-Sophie Schneider, Martin Munzinger, Josephine Sieger, Raika Languille, Bojana Rossin, Marius Gruppe 11 12 13 14 15 16 Göös-Dombrowski, Anne-Marie 17 Aydin, Nagihan Reimer, Teresa El Abdi, Mohamed Pannek, Andreas Höß, Carsten Außem, Jan Bergmann, Michaela Kück, Anja Ade, Jens Von Dungen, Annika Adrion, Dennis Nikorowski, Dennis Wernig, Florian 18 3 Teilnehmer Engel, Marcial Beyer, Daniel Dörr, Mareike Baumgärtner, Anne Wendrich, Kim Wirth, Franziska Raupach, Jan Röschert, Isabelle Schmanke, Paul Laßhof, Lena Reinig, Inga Linker, Stephanie Stasiuk, Iryna Kuchyriska, Oleysa Kalliontzi, Mersini Knuth, Anne-Kathrin Fresenborg, Leonard Henß, Thilo Bauer, Rebekka Huhn, Meik Henritzi, Sandra Grensing, Jana Ulshöfer, Corinna Maier, Andreas Schader, Tim Marnet, Mario BCII am PEI, 04.11.2013-28.11.2013 Übersicht zum Zeitplan GT1 1. Woche 4.11.-7.11. Gr. 1/2 2. Woche 11.11.-14.11. Gr. 7/8 3. Woche 18.11.-21.11. Gr. 13/14 GT2 Gr. 3/4 Gr. 9/10 Gr. 15/16 GT3 V1 Gr. 5/6 Gr. 1/2 Gr. 17/18 Gr. 7/8 Gr. 11/12 Gr. 13/14 V2 Gr. 3/4 Gr. 9/10 Gr. 15/16 V3 I1 Gr. 11/12 Gr. 7/8 Gr. 5/6 Gr. 13/14 Gr. 17/18 Gr. 1/2 I2 Gr. 9/10 Gr. 15/16 Gr. 3/4 I3 GM1 Gr. 11/12 Gr. 13/14 Gr. 17/18 Gr. 1/2 Gr. 5/6 Gr. 7/8 GM2 Gr. 15/16 Gr. 3/4 Gr. 9/10 GM3 Gr. 17/18 Gr. 11/12 4. Woche 25.11.-28.11. Gr. 5/6 Protokolle: Jede Gruppe fertigt zu den Experimenten GT, I, GM und V je ein Protokoll an. Das Protokoll wird bis spätestens 10.12.13 an den jeweiligen Versuchsbetreuer per Email geschickt. Das Protokoll wird einmal korrigiert (üblicherweise auf Papier). Das Protokoll wird entsprechend den Korrekturen überarbeitet. Nach der einmaligen Korrektur muss eine akzeptable Endversion vorliegen, die dann abgezeichnet wird. 4 Experiment GT1/GT2 Experiment Gentransfer GT1/GT2: Gentransfer mit retro- und lentiviralen Vektoren Prof. Dr. Christian Buchholz (GT1) Dr. Michael Mühlebach (GT2) 5 Experiment GT1/GT2 Verlaufsplan: Tag 1: - Transduktion der Zielzellen - Isolierung viraler RNA - reverse Transkription viraler RNA Tag 2: - Isolierung zellulärer DNA - PCR an RNA, cDNA und genomischer DNA Tag 3: - Mikroskopische Auswertung der Transduktionen Versuchshintergrund und -ablauf: Sowohl γ-Retroviren wie auch Lentiviren gehören zur Familie der Retroviren und integrieren ihre genetische Information nach Infektion ins Genom der Wirtszelle. Retrovirale Vektoren, die zur genetischen Veränderung von bestimmten Zielzellen verwendet werden, sind im Gegensatz zu den Viren, von denen sie abgeleitet sind, replikationsinkompetent. Sie können durch verschiedene Methoden hergestellt werden: zum einen gibt es stabil transfizierte Verpackungszellen, welche alle Komponenten des Vektors (Transfervektor, Gag/Pol, Env) konstitutiv bilden. Andererseits können die für diese Vektorbestandteile kodierenden Plasmide transient (nicht stabil, „vorübergehend“) in geeignete Zelllinien ko-transfiziert (d.h. mit einer speziellen Technik in die einzelnen Zellen eingebracht) und so „transient transfizierte Verpackungszellen“ generiert werden (Abb. 1). Hierfür sind 293TZellen (humane embryonale Nierenmarkszellen) aufgrund der hohen erreichbaren Transfektionseffizienzen besonders geeignet. Für diesen Versuch wurden im Vorfeld verschiedene transient transfizierte 293TVepackungszellen zur Produktion von Murinen Leukämie Virus (MLV)-abgeleiteten γretroviralen Vektoren sowie von Humanen Immundefizienz Virus (HIV)-abgeleiteten lentivirale Vektoren hergestellt und die entsprechenden Vektoren präpariert. Die dabei erzeugten Vektormengen und die Eigenschaften der Vektorpartikel werden innerhalb des Praktikums untersucht. 6 Experiment GT1/GT2 Abb. 1: Herstellung und Analyse von retroviralen Vektoren durch Triple-Transfektion. Die erzeugten γ-retroviralen MLV-abgeleiteten Vektoren bestehen alle aus MLVKapsidpartikeln (Gag/Pol), und besitzen den gleichen Transfervektor, der für das grün fluoreszierende Protein (GFP) kodiert. Sie unterscheiden sich aber durch die verwendeten Hüllproteine (Abb. 2), welche das Wirtszellspektrum (Tropismus) der Vektorpartikel durch die Bindung an spezifische zelluläre Rezeptoren bestimmen. Folgende Hüllproteine (Env) werden hier verwendet: 1. Env des ecotropen MLV: Ecotropes MLV (vollständiges Virus, nicht der Vektor!) repliziert nur in Nagerzellen und nutzt den zellulären Rezeptor MCAT-1 zum Anbinden und den Eintritt in die Wirtszelle. 7 Experiment GT1/GT2 2. Env des amphotropen MLV: Amphotropes MLV repliziert in Nagerzellen sowie auch in humanen Zellen und nutzt den Rezeptor RAM-1 (receptor for amphotropic MLV 1). 3. Env des GaLV (Gibbon Ape Leukemia Virus): Das Hüllprotein des GaLV bindet an den Rezeptor Glvr-1 (GaLV receptor 1), der (wie RAM-1) in fast allen humanen Zelltypen, jedoch nicht in murinen Zellen gebildet wird. Allerdings scheint Glvr-1 in einigen für die Gentherapie besonders interessanten Zellpopulationen (z. B. hämatopoetischen Stammzellen) stärker exprimiert zu werden als RAM-1. Daher wird GaLV Env zur Pseudotypisierung (Verwendung eines heterologen Env, d.h. Verwendung eines Hüllproteins eines anderen Virus auf der Vektoroberfläche) von retroviralen MLV-abgeleiteten Vektorpartikeln verwendet. Somit verändert man den Vektortropismus und kann in bestimmten Zielzellen die Gentransfereffizienz erhöhen. Für gentherapeutische Anwendungen ist es wünschenswert, wenn die verwendeten Vektorpartikel ihre genetische Information spezifisch nur in die Zielzellpopulation übertragen würden und nicht in angrenzende Nicht-Zielzellen. Dies würde sowohl die Sicherheit, wie auch die Effizienz der Vektoren erhöhen. Der Tropismus der oben beschriebenen Hüllproteine erlaubt zwar eine Speziesunterscheidung, aber keine Unterscheidung zwischen verschiedenen humanen Zelltypen. Hier am PEI, in der AG Buchholz, wurde ein System entwickelt, das spezifischen Gentransfer in Zielzellen erlaubt. Dazu werden HIV-abgeleitete lentivirale Vektoren mit den Hüllproteinen des Masernvirus (MV) pseudotypisiert, dem Hämagglutinin (H)-Protein, welches für die Rezeptorerkennung zuständig ist, und dem Fusions (F)-Protein, welches anschließend die Membranfusion zwischen Virus und Wirtszelle vermittelt. Zur Inkorporation in die lentivirale Hülle, mussten die zytoplasmatischen Domänen beider Proteine spezifisch um 18 bzw. 30 Aminosäuren verkürzt werden (HΔ18 und FΔ30). Des Weiteren ist das H Protein durch Punktmutationen für die natürlichen Masernvirusrezeptoren verblindet. Als Rezeptorerkennungsdomäne wird ein Einzelkettenantikörperfragment (scFv) an die Ektodomäne des H Proteins fusioniert. Dieses vermittelt spezifische Bindung an sein Antigen (s. Abb. 3). 8 Experiment GT1/GT2 Abb. 2: Konstrukte, die für die Generierung der MLV- und HIV-abgeleiteten Vektoren verwendeten werden. Die Transfervektoren (pMg-EGFP und pSEW) besitzen die von MLV bzw. HIV abgeleiteten flankierenden LTRs (long terminal repeats), das Verpackungssingnal "Ψ", welches die Verpackung der Transkripte in MLV- bzw. HIV-Kapsidpartikel gewährleistet sowie einen internen Promotor, der die Expression des Transgens (EGFP) antreibt. Die 3`-LTR von pSEW enthält eine Deletion in der U3 Region, was diesen Vektor zu einem sog. SIN (Self inactivating) Vektor macht (erhöhte Sicherheit). Das Verpackungskonstrukt pHIT60 beinhaltet die Strukturgene gag und pol des MLV, deren Genprodukte in der Lage sind, leere, nicht umhüllte Kapsidpartikel des MLV zu bilden. Das Verpackungskonstrukt pCMVΔR8.9 enthält die Strukturgene gag und pol des HIV. Im Gegensatz zu MLV besitz HIV noch sechs akzessorische Gene, von denen zwei auf dem Verpackungskonstrukt pCMVΔR8.9 zu finden sind. Die Genprodukte Tat und Rev sind für eine effiziente Bildung von infektiösen Vektorpartikeln notwendig. Die Hüllproteinkonstrukte, die für die Pseudotypisierung der MLV-Vektoren verwendet werden, kodieren für die Hüllproteine des eco- bzw. amphotropen MLV bzw. des GaLV. Die Hüllproteinkonstrukte, die für die Pseudotypisierung der HIV-Vektoren verwendet werden, kodieren für die modifizierten Hüllproteine des Masernvirus (MV) oder des Vesikulären Stomatitis Virus (VSV). Alle Gene werden von internen Promotoren getrieben. 9 Experiment GT1/GT2 Abb. 3: Notwendige Modifikationen in den MV H und F Proteinen zur Erzeugung von HIVabgeleiteten Vektoren mit zielgerichtetem Zelleintritt. Der dargestellte HIV-Vektor, der die beschriebenen modifizierten MV Hüllproteine in seine Membran eingebaut hat, kann sein Transfergen (GFP) ausschließlich in CD105-positive Zellen übertragen. Auf diese Weise wurden unter anderem Vektoren, die spezifisch für das BZelloberflächenprotein CD20, den Endothelzellmarker CD105, den hematopoetischen Stammzellmarker CD133 oder den murinen Glutamatrezeptor, der auf Neuronen exprimiert ist, hergestellt (Funke et al. 2008, Anliker et al. 2010). Dies zeigt auch gleichzeitig die Flexibilität des Systems: durch Präsentation von scFvs mit verschiedener Spezifität, können die unterschiedlichsten Zelltypen gezielt angesteuert werden. In diesem Praktikumsversuch soll der Zelltropismus des CD105-spezifischen Vektors im Vergleich zum Standardvektor, der mit dem Hüllprotein des Vesikulären Stomatitis Virus (VSV-G) pseudotypisiert ist, verglichen werden. VSV-G Pseudotypen weisen eine hohe Stabilität auf und es können Vektoren mit einem hohen Titer erzeugt werden. Diese Vektoren sind allerdings unspezifisch und können nahezu alle humanen Zellen transduzieren (Transduktion: Gentransfer mittels viraler Vektoren; im Gengensatz zu Infektion: Gentransfer mittels 10 Experiment GT1/GT2 replizierender Viren). Die Konstrukte zur Generierung der Vektoren sind in Abb. 2 dargestellt. Ein Vorteil in der Verwendung von HIV-abgeleiteten Vektoren in der Gentherapie liegt darin, dass HIV-Vektoren, im Gegensatz zu MLV-Vektoren, auch sich nicht-teilende Zellen transduzieren können. Die Vektorpartikel werden für das Praktikum zur Verfügung gestellt. Dafür wurden im Vorfeld zunächst 293T Zellen mit den für die Vektorkomponenten kodierenden Plasmiden (Transfervektor, Verpackungskonstrukt, Hüllproteinkonstrukt/e) transfiziert (Abb. 1, 2). Diese Zellen geben dann die Vektorpartikel in den Zellkulturüberstand ab. Zwei Tage Zellkulturüberstände „geerntet“ und nach Transfektion dieser Verpackungszellen direkt bei -80°C wurden (viral gelagert die vektorpartikelhaltigen packaging (MLV-Vektoren) cells; VPC) oder durch Ultrazentrifugation über ein 20%iges Sucrosekissen ankonzentriert und dann bei -80°C gelagert (HIV-Vektoren). Der eigentliche Praktikumsversuch beginnt dann mit der Transduktion verschiedener Zielzellen, d.h. die Zellen werden mit vektorpartikelhaltigem Überstand inkubiert, um sie durch Einschleusen des Transfervektors ins Genom genetisch zu verändern. Drei Tage nach erfolgter Transduktion werden die Zellen auf die Anwesenheit des transferierten Reportergens hin untersucht. Die Menge der generierten infektiösen Vektorpartikel und deren Tropismus lassen sich so nachweisen. Im Rahmen dieses Versuches sollen auch die RNA Genome aus den Vektorpartikeln isoliert, in cDNA umgeschrieben und dann mit PCR detektiert werden. Ebenso soll mittels PCR in der genomischen DNA der Zielzellen, die Integration des Transfergens nachgewiesen werden. Vor dem Praktikum vorbereitet und durchgeführt: Herstellung von MLV- bzw. HIV-abgeleiteten Vektoren Transfektion mittels Polyethylenemin (PEI): • 24 h vor Versuchsbeginn werden in T175 Flaschen 2 x 107 Zellen ausgesät • die DNA (Transfervektor, Verpackungskonstrukt, Hüllproteinkonstrukt/e) wird in einem Falkon vorgelegt (siehe Pipettierschema Tab. 1) • dazu werden 2,3 ml DMEMohne Zusätzte gegeben und gevortext 11 Experiment GT1/GT2 • in einem zweiten Falkon werden140 µl PEI und 2,2 ml DMEMohne Zusätzte durch vortexten vermischt • dann werden die beiden Ansätze (DNA und PEI) vereinigt, gevortext und für 20 Min. bei RT inkubiert • in der Zwischenzeit wird das Medium von den am Tag zuvor ausgesähten 293T Zellen gegen 10 ml DMEM15% FCS gewechselt • dann wird der Transfektionsansatz zu den Zellen gegeben und durch leichtes Schwänken der Flasche verteilt • am nächsten Tag wird ein Mediumwechsel gegen 16 ml DMEM10% FCS durchgeführt • 24 h später wird der Überstand abgenommen und durch einen 0,45 µm Filter filtriert (geerntet) und entweder direkt aliquotiert und bei -80°C gelagert oder durch Ultrazentrifugation über ein 20%iges Sucrosekissen ankonzentriert (28.000 rpm, 4°C, 3h). Nach der Zentrifugation wird der Überstand verworfen und das Pellet in 120 µl PBS resuspendiert, aliquotiert und dann bei -80°C gelagert. Tab. 1: Kombinationen der Vektorpartikelkomponenten kodierenden Plasmid-DNAs zur Transfektion von 293T Zellen. Ansatz Verpackungskonstrukt Transfervektor Hüllproteinkonstrukt Name MLV HIV MLV HIV 1 pHIT60 x pLEGFP-N1 x pALF-GaLVwt GaLV-MLV 2 pHIT60 x pLEGFP-N1 x pHIT123 3 pHIT60 x pLEGFP-N1 x pHIT456 4 x pCMVΔR8.9 x pSEW pCG-HΔ18-αCD105/pCG-Fd30 Eco-MLV AmphoMLV αCD105HIV 5 x pCMVΔR8.9 x pSEW pMDG2 VSV-G-HIV 12 Experiment GT1/GT2 Versuchsdurchführung: Alle Zellkulturarbeiten werden steril und unter der Werkbank durchgeführt Experiment Transduktion von Zielzellen mit MLV-abgeleiteten Vektoren: Zielzellen: NIH 3T3, HT1080, und D17 (canine [„Hunde“] Osteosarkomzellen) 3,5x105 NIH 3T3 und 3x105 HT1080 bzw. D17 Zellen wurden in Sechs-Loch-Platten am Tag zuvor ausgesät und werden zur Verfügung gestellt: • Es werden jeweils 2 Zellkulturplatten benötigt: a) für GFP-Detektion b) für die Isolierung von zellulärer DNA (s. Schema Abb. 4). • Vektorpartikelhaltige Überstände wurden vor Beginn des Praktikums generiert und stehen nun für Transduktionsversuche zur Verfügung. Es sollen 550 µl einer 1:10 Verdünnung hergestellt werden (nach Absprache mit Versuchsbetreuern!). • Zielzellen mit PBS waschen. • je 500 µl der unverdünnten und 1:10 verdünnten vektorhaltigen filtrierten Überstande zu den Zielzellen geben (siehe Abb. 4; beschriften!). • 2 h nach Transduktion Zellen mit frischen Medien versorgen. • Für die Transduktionen, aus deren Zellen später die genomische DNA isoliert werden soll, stehen DNAse verdaute Vektoren zur Verfügung. Warum sind diese Vektoren mit DNAse verdaut worden? Experiment Transduktion von Zielzellen mit HIV-abgeleiteten Vektoren: Zielzellen: 293T, HT1080Rot, und 70/30 Mischung aus 293T/HT1080Rot (HT1080Rot Zellen sind HT1080 Zellen, die stabil das rot fluoreszierende Protein dTomato exprimieren) 6x104 293T, 6x104 HT1080Rot bzw. 4,2x104 293T/1,8x104 HT1080Rot Zellen wurden in Vierundzwanzig-Loch-Platten am Tag zuvor ausgesät und werden zur Verfügung gestellt: • Ankonzentrierte vektorpartikelhaltige Überstände wurden vor Beginn des Praktikums generiert und stehen nun für Transduktionsversuche zur Verfügung. Es sollen für jeden Vektortyp (VSV-G-HIV und αCD105-HIV) entsprechende Verdünnungen hergestellt werden (nach Absprache mit Versuchsbetreuern!). • Zielzellen mit PBS waschen. • je 200 µl der Verdünnungen werden zu den Zielzellen geben (siehe Abb. 5; beschriften!). • 2h nach Transduktion Zellen mit frischen Medien versorgen. 13 Experiment GT1/GT2 Experiment Isolierung von Nukleinsäuren und PCR Detektion: Isolierung viraler RNAs Um die viralen RNAs aus den MLV-abgeleiteten Vektorpartikeln zu isolieren, wird der vektorhaltige Überstand direkt eingesetzt und mittels des QIAamp Viral RNA Kit aufgereinigt. QIAamp viral RNA Mini Kit The sample is first lysed under highly denaturing conditions to inactivate RNases and to ensure isolation of intact viral RNA. Buffering conditions are then adjusted to provide optimum binding of the RNA to the QIAamp membrane, and the sample is loaded onto the the QIAamp spin column. After contaminants are washed away, high-quality RNA is eluted in a special RNase-free buffer, ready for direct use or safe storage. Preparation of virus RNA (according to Qiagen) Use RNase-free material only after the lysis with buffer AVL • Pipet 560µl of prepared Buffer AVL (containing carrier RNA) into a 1.5ml tube • Add 140µl virus-containing supernatant, mix by votexing for 15 sec. • Incubate at RT for 10 min. • Briefly centrifuge tube to remove drops from inside the lid • Add 560µl EtOH (96-100%), mix by vortexing, briefly spin down • apply 630µl of sample to provided column (in a 2-ml collection tube) • centrifuge 1 min (8000rpm) and discard filtrate, place the QIAamp column in a clean collection tube • apply residual sample to column • centrifuge 1 min (8000rpm) and discard filtrate, place the QIAamp column in a clean collection tube • add 500µl buffer AW1, centrifuge 1 min (8000rpm) and discard filtrate, place the QIAamp column in a clean collection tube • add 500µl buffer AW2, centrifuge 3 min (13000rpm) and discard filtrate, place the QIAamp column in a clean collection tube. • repeat centrifugation to get rid of residual filtrate, place the QIAamp column in a clean 1.5-ml microcentrifuge tube. • add 60µl buffer AVE, incubate 1 min at RT • centrifuge 1 min (13000rpm) • Take off 20 µl for DNase digest, store on ice • store rest of eluate at –80°C 14 Experiment GT1/GT2 DNAse digestion of RNA 20 µl + 67.5 µl H2O 15 Experiment GT1/GT2 16 Experiment GT1/GT2 Herstellung von cDNA der viralen RNA • heat one incubator to 65°C and one to 42°C • prepare RT-reaction-Mastermix for all samples: for one sample (Σ=13µl): 5x Buffer 4µl RNase OUT 1µl DTT 2µl Reverse Transkriptase 1µl • preparation of the RNA samples: mix 4µl RNA + 1µl (10pmol) Primer + 1µl dNTPs + 6µl H2O (control: one sample without template) • incubate these 12µl samples 5min at 65°C, then put them directly on ice • add 8µl reaction-mix to each sample, incubate 1h at 42°C • store samples at -80°C Experiment Transduktion von Zielzellen: • 72h nach Transduktionsbeginn können die Zielzellen mittels Fluoreszenzmikroskopie (EGFP) auf den erfolgten Gentransfer hin untersucht werden. Auswertung des Experiments Transduktion von Zielzellen (EGFP): Die transduzierten Zellen werden unter dem Axiovert (Mikroskop) untersucht und die Zahl der transduzierten Zellkolonien pro betrachteter Fläche bestimmt. Hochgerechnet auf die gesamte Fläche der Kulturschale kann die Zahl der infektiösen Vektorpartikel pro Volumeneinheit Überstand bestimmt werden (t.u./ml; t.u. = transducing units). Diese Titer können graphisch dargestellt werden. Vergleiche die Titer der verschiedenen Vektorpartikel in den unterschiedlichen Zielzellen. - Bei den NIH 3T3 und HT1080-Zellen handelt es sich um eine humane und eine murine Zelllinie. Welche ist was? (Hinweis: Der Tropismus der verwendeten Hüllproteine für die MLV-abgeleiteten Vektoren sollte eine Aussage erlauben) - Welche Rezeptoren sind auf der caninen Zelllinie D17 vorhanden? 17 Experiment GT1/GT2 - Welche Zellen, 293T oder HT1080, exprimieren CD105 auf ihrer Oberfläche? - Demonstriere anhand von Fotos der transduzierten Mischungen aus 293T und HT1080Rot Zellen die unterschiedliche Spezifität der verwendeten HIVPseudotypen. - Wie können Unterschiede im Titer zwischen den einzelnen Vektortypen erklärt werden? Experiment Isolierung von Nukleinsäuren und PCR Detektion: Isolierung zellulärer DNAs Die Gesamt-DNA der MLV-Vektor-transduzierten Zellen soll isoliert werden und die Anwesenheit der Vektorsequenzen mit PCR nachgewiesen werden. Wir nutzen zur Isolierung der DNA das QIAGEN DNeasy Tissue Kit. DNeasy Tissue procedure: The kit uses advanced silica-gel-membrane technology for rapid and efficient purification of total cellular DNA. The buffer system is optimized to allow direct lysis followed by selective binding of DNA to the DNeasy membrane. DNeasy purified DNA typically has an A260/A280 ratio between 1.7 and 1.9, and is up to 50 kb in size. The DNeasy procedure also efficiently recovers DNA fragments as small as 100 bp. Before lysis of cells, these have to be detached from monolayer culture and washed: • Remove medium • Wash cells 1x with 1 ml PBS/well • Detach cells by adding 500 µl Trypsin-EDTA + 500 µl PBS and incubate for 5 min at RT • Transfer cells into Eppendorff Tube • Spin down (5 min 2000 rpm) • Wash cells with 1 ml PBS • Spin down (5 min 2000 rpm) 18 Experiment GT1/GT2 Purification of total DNA from cultured animal cells: • • • • • • • • • • • • • • • Resupend pellet in 200µl PBS Add 20µl proteinase K and 200µl buffer AL (do not add proteinase K directly to buffer AL), mix by votexing Incubate 56°C 10 min Add 200 µl EtOH, mix by vortexing Transfer mixture to DNeasy mini spin column (placed in a 2 ml collection tube) spin 1 min at 8000 rpm Replace the collection tube, add 500µl buffer AW1 spin 1 min at 8000 rpm Replace the collection tube, add 500µl buffer AW2 spin 3 min at 14000 rpm place the column in clean 1.5 ml centrifuge tube add 200 µl buffer AE on the membrane incubate 1 min spin 1 min at 8000 rpm to elute DNA determine the DNA concentration Experiment Isolierung von Nukleinsäuren und PCR Detektion: → Detektion von GFP Sequenzen mittels PCR an RNA, cDNA und DNA • prepare PCR-reaction-mix for all samples (additional: one plasmid control to check the PCR and to check for DNA content in the RNA: Instead of cDNA use 0,5µl RNA (DNase I digested and undigested) as Template in the PCR): for one sample (Σ=50µl): sample (cDNA or DNA or RNA or H20) 10x Buffer dNTP (10mM) Primer 1 (10pmol/µl) Primer 2 (10pmol/µl) Dream-Taq-Polymerase H20 in 10µl 5µl 1µl 1µl 1µl 0,25µl 31,75µl • program: 1. 3min 95°C 2. 30sec 95°C 3. 30sec 58°C (Temp depends on the Primer) 4. 30sec 72°C (Time depends on expected product length: 1 min / kb) 5. 5min 72°C 6. hold at 4°C 30 repeats from step 2 to 4 • Analyse samples on agarose gels. 19 Experiment GT1/GT2 Abb. 4: Pipettierschema für die MLV-abgeleiteten Vektoren. Abb. 5: Pipettierschema für die HIV-abgeleiteten Vektoren. 20 Experiment GT3 Experiment Gentransfer GT3: Gentransfer mit retroviralen Vektoren Prof. Dr. Ute Modlich (GT3) 21 Experiment GT3 Verlaufsplan: Tag 1: - Transduktion der Zielzellen - Isolierung viraler RNA - reverse Transkription viraler RNA Tag 2: - Isolierung zellulärer DNA - PCR an RNA, cDNA und genomischer DNA Tag 3: - Mikroskopische Auswertung der Transduktionen - Durchflusszytometrischen Analyse der Transduktionen - Durchflusszytometrischen Analyse zur regulierten Genexpression Versuchshintergrund und -ablauf: Sowohl γ-Retroviren wie auch Lentiviren gehören zur Familie der Retroviren und integrieren ihre genetische Information nach Infektion ins Genom der Wirtszelle. Retrovirale Vektoren, die zur genetischen Veränderung von bestimmten Zielzellen verwendet werden, sind im Gegensatz zu den Viren, von denen sie abgeleitet sind, replikationsinkompetent. Sie können durch verschiedene Methoden hergestellt werden: zum einen gibt es stabil transfizierte Verpackungszellen, welche alle Komponenten des Vektors (Transfervektor, Gag/Pol, Env) konstitutiv bilden. Andererseits können die für diese Vektorbestandteile kodierenden Plasmide transient (nicht stabil, „vorübergehend“) in geeignete Zelllinien ko-transfiziert (d.h. mit einer speziellen Technik in die einzelnen Zellen eingebracht) und so „transient transfizierte Verpackungszellen“ generiert werden (Abb. 1). Hierfür sind 293TZellen (humane embryonale Nierenzellen) aufgrund der hohen erreichbaren Transfektionseffizienzen besonders geeignet. 22 Experiment GT3 Abb. 1: Herstellung und Analyse von retroviralen Vektoren durch Triple-Transfektion. 23 Experiment GT3 Für diesen Versuch wurden im Vorfeld verschiedene transient transfizierte 293TVepackungszellen zur Produktion von murinen Leukämie Virus (MLV)-abgeleiteten γretroviralen Vektoren hergestellt und die entsprechenden Vektoren präpariert. Die dabei erzeugten Vektormengen und die Eigenschaften der Vektorpartikel werden innerhalb des Praktikums untersucht. Die erzeugten γ-retroviralen MLV-abgeleiteten Vektoren bestehen alle aus MLVKapsidpartikeln (Gag/Pol), und besitzen den gleichen Transfervektor, der für das grün fluoreszierende Protein (GFP) kodiert. Sie unterscheiden sich aber durch die verwendeten Hüllproteine (Env, Abb. 2), welche das Wirtszellspektrum (Tropismus) der Vektorpartikel durch die Bindung an spezifische zelluläre Rezeptoren bestimmen. Die Verwendung von heterologen Env, d.h. die Verwendung eines Hüllproteins eines anderen Virus auf der Vektoroberfläche, wird als Pseudotypisierung bezeichnet. Folgende Hüllproteine werden hier verwendet: 4. Env des ecotropen MLV: Ecotropes MLV (vollständiges Virus, nicht der Vektor!) repliziert nur in Nagerzellen und nutzt den zellulären Rezeptor MCAT-1 zum Anbinden und den Eintritt in die Wirtszelle. 5. Env des pantropen Vesicular Stomatitis Virus (VSV): Pantropes VSV hat ein sehr breit gefächertes Wirtsspektrum wie Insekten aber auch Säugetiere wie z.B. Rinder, Pferde, Schweine und Menschen. Der Eintritt erfolgt über Endocytose wobei LDL (Low Density Lipoprotein) Rezeptoren eine zentrale Rolle spielen. 6. Env des xenotropen RD114 Virus (Felines Endogene Virus): Das Hüllprotein des RD114 Virus bindet u.a. an humane Natrium-abhängige Aminosäuretransporter (ASCT) und erlaubt die Infektion humaner, aber nicht muriner Zelllinien. 24 Experiment GT3 Abb. 2: Konstrukte, die für die Generierung der MLV- abgeleiteten Vektoren verwendeten werden. Der Transfervektor RSF91-eGFP (Schambach et al. 2006) besitzt die von MLV abgeleiteten flankierenden LTRs (long terminal repeats), das Verpackungssingnal "Ψ", welches die Verpackung der Transkripte in MLV- Kapsidpartikel ermöglicht. Die Expression des Transgens (EGFP) erfolgt hierbei über die Promotor/Enhancer-Region der 5´-LTR. Die 3`-LTR von pES.1-T6-lmg* enthält eine Deletion in der U3 Region, was diesen Vektor zu einem sog. SIN (Self inactivating) Vektor macht (erhöhte Sicherheit). Der interne Minimalpromotor T6 erlaubt regulierte Genexpression nach Bindung des Tetracyclin-gesteuerten Transaktivors. Das Verpackungskonstrukt p-gag/pol beinhaltet die Strukturgene gag und pol des MLV, deren Genprodukte in der Lage sind, leere, nicht umhüllte Kapsidpartikel des MLV zu bilden. Die Hüllproteinkonstrukte, die für die Pseudotypisierung der MLVVektoren verwendet werden, kodieren für die Hüllproteine des ecotropen MLV, des Pantropen VSV bzw. des xenotropen RD114. Alle Gene werden von internen Promotoren getrieben. Für gentherapeutische und biotechnologische Anwendungen ist es wünschenswert, das gewünschte (Reporter-)Gen nicht konstitutiv, also permanent, sondern reguliert zu exprimieren. Eines der am meisten genutzten Systeme zur transkriptionellen Genregulation ist das sog. Tetracyclin-regulierte System (Tet-System, Gossen und Bujard, 1992). Dieses basiert auf dem bakteriellen Tetracyclin Resistenzoperon, welches auf dem Transposon 10 (TN10) aus Escherichia coli kodiert ist. Als Aktivator der Transkription (Transaktivator) dient ein Fusionsprotein, bestehend aus dem tet25 Experiment GT3 Repressor (tetR) und der aktivierenden Domäne des Virionproteins 16 (VP16) aus dem Herpes Simplex Virus. In dimerisierter Form bindet nun der Transaktivator (tTA) mit der tetR-DNA bindenden Domäne an die Sequenz des tet-Operators. Zur Optimierung wird in der Regel eine heptamerisierte Operatorkassette (tetO7) benutzt. Die VP16-Domäne ist nun in der Lage, einen an tetO7 fusionierten Minimalpromotor zu stimulieren und damit die Genexpression zu induzieren (Abb. 3). Der Minimalpromotor wurde durch das Entfernen der Enhancerregion aus dem humanen Cytomegalovirus immediate early Promotor (CMVie) erzeugt. Der daraus entstandene Minimalpromotor (-53/+75) steuert die Transkription nun in Abhängigkeit des an tetO7 gebundenen tTA. In Anwesenheit des Antibiotikums Tetracyclin ist der Transaktivator durch Veränderung seiner Tertiärstruktur nicht mehr in der Lage, an die tet-Operatoren zu binden. Daher kommt es zu keiner Induktion des Minimalpromotors durch tTA (TetOff-Transaktivator). VP16 tTA (Dimer) Tetracyclin tetR Ohne Tetracyclin Expression des Transgens +1 tetO7 P Transgen Mit Tetracyclin Keine Expression des Transgens Abbildung 3: Schematische Darstellung der Funktionsweise des Tet- Systems Die tet- Repressordomäne (tetR) des Transaktivators tTA bindet als Dimer an die tet- Operatoren (tetO7) nur in Abwesenheit von Tetracyclin. Dabei stimuliert die VP16- Domäne den Minimalpromotor (P) aus dem humanen CMVie-Promotor, und das Transgen wird transkribiert. In Anwesenheit von Tetracyclin ist die tetR-Domäne nicht mehr in der Lage, an tetO7 zu binden. Die VP16 Domäne ist dementsprechend nicht fähig, stimulierend auf den Minimalpromotor zu wirken. Dieses ursprüngliche System wurde in den vergangen Jahren immer wieder optimiert. Dadurch erfuhr das Tet-System eine Reihe von Modifikationen der Operatorsequenz und des Transaktivators zur verbesserten Regulation, d.h. eine niedrige Aktivität im ausgeschalteten und eine hohe Genexpression im angeschalteten Zustand. Die tet-Operatoren im ursprünglichen System besitzen 26 Experiment GT3 einen Abstand der Zentren von 42 nt. In einer optimierten Variante beträgt der Abstand der Zentren 36 nt. Eine weiterhin deutlich verbesserte Regulation wurde durch Modifikationen des Minimalpromotors erreicht. Hierbei wurde ein synthetischer Minimalpromotor basierend auf einer pflanzlichen viralen Sequenz generiert (Loew et al. 2010, Heinz et al. 2011). Der Transaktivator besitzt nicht mehr die ursprüngliche VP16 Transaktivierungsdomäne, sondern drei daraus abgeleitete sogenannte Minimaldomänen. Weiterhin wurde dieser durch Mutagenese-Screens derart verändert, dass die daraus resultierende Variante einen reversen Phänotyp generiert, d.h. der Transaktivator bindet nun ausschließlich in Gegenwart von Doxycyclin, einem Tetracylin Derivat (TetOn-Transaktivator). Weiterhin besitzt diese Variante (rtTA2s-M2) eine niedrigere Bindeaktivität zu den Operatoren im ausgeschalteten Zustand und eine deutlich höhere Sensitivität zu Doxycyclin. Damit das Tet-System in der Zielzelle funktional ist, sind zwei Komponenten in derselben Zielzelle erforderlich: • der Transaktivator, und • die Tet-Operatoren mit Minimalpromotor und Transgen Dies kann auf zwei prinzipiellen Wegen erreicht werden: - Die beiden Komponenten befinden sich auf zwei unabhängigen Vektoren Zwei-Vektor-System - Die beiden Komponenten befinden sich auf demselben Vektor Ein-Vektor-System In diesem Praktikum soll die Genregulation anhand des Zwei-Vektor-Systems untersucht werden. Dabei wird eine Zelllinie verwendet, welche bereits den Transaktivator kontinuierlich im zellulären Hintergrund exprimiert. Diese wird mit einem γ-retroviralen Vektor transduziert und mit bzw. ohne Zugabe von Doxycyclin auf GFP-expression untersucht. Im Vergleich dazu wird eine zweite Zelllinie getestet, welche keinen Transaktivator beinhaltet. Die Vektorpartikel werden für das Praktikum zur Verfügung gestellt. Dafür wurden im Vorfeld zunächst 293T Zellen mit den für die Vektorkomponenten kodierenden Plasmiden (Transfervektor, Verpackungskonstrukt, Hüllproteinkonstrukt/e) transfiziert 27 Experiment GT3 (Abb. 1, 2). Diese Zellen geben dann die Vektorpartikel in den Zellkulturüberstand ab. Zwei Tage nach Transfektion wurden die vektorpartikelhaltigen Zellkulturüberstände dieser Verpackungszellen „geerntet“ und direkt bei -80°C gelagert (MLV-Vektoren). Der eigentliche Praktikumsversuch beginnt dann mit der Transduktion verschiedener Zielzellen, d.h. die Zellen werden mit vektorpartikelhaltigem Überstand inkubiert, um sie durch Einschleusen des Transfervektors ins Genom genetisch zu verändern. Dabei wird auch durch Zugabe von Doxycyclin die regulatorische Eigenschaft des Tet-Systems untersucht. Zwei Tage nach erfolgter Transduktion werden die Zellen auf die Anwesenheit des transferierten Reportergens hin untersucht. Die Menge der generierten infektiösen Vektorpartikel und deren Tropismus lassen sich so nachweisen. Im Rahmen dieses Versuches sollen auch die RNA Genome aus den Vektorpartikeln isoliert, in cDNA umgeschrieben und dann mit PCR detektiert werden. Ebenso soll mittels PCR in der genomischen DNA der Zielzellen, die Integration des Transfergens nachgewiesen werden. Vor dem Praktikum vorbereitet und durchgeführt: Herstellung von MLV- abgeleiteten Vektoren Transfektion mittels Kalzium-Phosphat: • 6 24 h vor Versuchsbeginn werden in 10cm-Zellkulturschalen 5 x 10 Zellen (293T) ausgesät • HEPES-gepufferte Phosphat-Lösung wird in einem Falcon-Röhrchen vorgelegt • DNA (Transfervektor, Verpackungskonstrukt, Hüllproteinkonstrukt/e) und CaCl2 werden gemischt (siehe Pipettierschema Tab. 1) • Das DNA/CaCl2-Gemsich wird unter ständiger Erzeugung von Luftblasen (mittels Pipetierhilfe) in die Phosphat-Lösung getropft • Der Transfektionsansatz wird dann für 20 Min. bei RT inkubiert • in der Zwischenzeit wird das Medium von den am Tag zuvor ausgesäten 293T Zellen gegen 10 ml DMEM / 10%FCS / 20mM HEPES / 25µM Chloroquine gewechselt 28 Experiment GT3 • dann wird der Transfektionsansatz zu den Zellen gegeben und die Zellen über Nacht inkubiert • am nächsten Tag wird ein Mediumwechsel gegen 10 ml DMEM / 10%FCS / 20mM HEPES durchgeführt • 24 h später wird der Überstand abgenommen und durch einen 0,22 µm Filter filtriert (geerntet), direkt aliquotiert und bei -80°C gelagert Tab. 1: Kombinationen der Vektorpartikelkomponenten kodierenden Plasmid-DNAs zur Transfektion von 293T Zellen. Ansatz Verpackungskonstrukt Transfervektor Hüllproteinkonstrukt Name MLV MLV 1 p-gap/pol pRSF91-eGFP p-eco 2 p-gap/pol pRSF91-eGFP p-RD114 RD114-MLV 3 p-gap/pol pRSF91-eGFP p-VSV-G VSVG-MLV 4 p-gap/pol pES.1-T6-lmg* p-VSV-G Tet-MLV eco-MLV Versuchsdurchführung: Alle Zellkulturarbeiten werden steril und unter der Werkbank durchgeführt Experiment Transduktion von Zielzellen mit MLV-abgeleiteten Vektoren: Zielzellen: SC-1, HT1080, D17 3*105 SC-1, HT1080, D17 und eine 50/50 Mischung der beiden Zelllinien SC-1 und HT1080 wurden in Sechs-Loch-Platten am Tag zuvor ausgesät und werden zur Verfügung gestellt: • Es werden jeweils 2 Zellkulturplatten benötigt: a) für GFP-Detektion b) für die Isolierung von zellulärer DNA (s. Schema Abb. 4). • Vektorpartikelhaltige Überstände wurden vor Beginn des Praktikums generiert und stehen nun für Transduktionsversuche zur Verfügung. Es sollen 500 µl einer 1:10 Verdünnung hergestellt werden (nach Absprache mit Versuchsbetreuern!). 29 Experiment GT3 • Zielzellen mit PBS waschen und 1ml frisches Medium mit 4µg/ml Protaminsulfat hinzugeben • je 200µl der unverdünnten und 1:10 verdünnten vektorhaltigen und filtrierten Überstande zu den Zielzellen geben (siehe Abb. 4; beschriften!) und über Nacht inkubieren. • Für die Transduktionen, aus deren Zellen später die genomische DNA isoliert werden soll, stehen DNAse verdaute Vektoren zur Verfügung. Warum sind diese Vektoren mit DNAse verdaut worden? Experiment Genregulation: Zielzellen: HT1080 und A1 3*105 A1 und HT1080 Zellen wurden in je 1 Sechs-Loch-Platte am Tag zuvor ausgesät und werden zur Verfügung gestellt: • Vektorpartikelhaltige Überstände wurden vor Beginn des Praktikums generiert und stehen nun für Transduktionsversuche zur Verfügung. • Zielzellen mit PBS waschen und 1ml frisches Medium mit 4µg/ml Protaminsulfat hinzugeben. Eine Hälfte der Zellen erhält zusätzlich 1µg/ml Doxycyclin. • je 200µl des unverdünnten vektorhaltigen und filtrierten Überstandes zu den Zielzellen geben (siehe Abb. 5; beschriften!) und über Nacht inkubieren. Experiment Isolierung von Nukleinsäuren und PCR Detektion: Isolierung viraler RNAs Um die viralen RNAs aus den MLV-abgeleiteten Vektorpartikeln zu isolieren, wird der vektorhaltige Überstand direkt eingesetzt und mittels des QIAamp Viral RNA Kit aufgereinigt. QIAamp viral RNA Mini Kit The sample is first lysed under highly denaturing conditions to inactivate RNases and to ensure isolation of intact viral RNA. Buffering conditions are then adjusted to provide optimum binding of the RNA to the QIAamp membrane, and the sample is 30 Experiment GT3 loaded onto the the QIAamp spin column. After contaminants are washed away, high-quality RNA is eluted in a special RNase-free buffer, ready for direct use or safe storage. Preparation of virus RNA (according to Qiagen) Use RNase-free material only after the lysis with buffer AVL • Pipet 560µl of prepared Buffer AVL (containing carrier RNA) into a 1.5ml tube • Add 140µl virus-containing supernatant, mix by votexing for 15 sec. • Incubate at RT for 10 min. • Briefly centrifuge tube to remove drops from inside the lid • Add 560µl EtOH (96-100%), mix by vortexing, briefly spin down • apply 630µl of sample to provided column (in a 2-ml collection tube) • centrifuge 1 min (8000rpm) and discard filtrate, place the QIAamp column in a clean collection tube • apply residual sample to column • centrifuge 1 min (8000rpm) and discard filtrate, place the QIAamp column in a clean collection tube • add 500µl buffer AW1, centrifuge 1 min (8000rpm) and discard filtrate, place the QIAamp column in a clean collection tube • add 500µl buffer AW2, centrifuge 3 min (13000rpm) and discard filtrate, place the QIAamp column in a clean collection tube. • repeat centrifugation to get rid of residual filtrate, place the QIAamp column in a clean 1.5-ml microcentrifuge tube. • add 60µl buffer AVE, incubate 1 min at RT • centrifuge 1 min (13000rpm) • Take off 20 µl for DNase digest, store on ice • store rest of eluate at –80°C 31 Experiment GT3 DNAse digestion of RNA + 67,5 µl H2O 20 µl 32 Experiment GT3 33 Experiment GT3 Herstellung von cDNA der viralen RNA • heat one incubator to 65°C and one to 42°C • prepare RT-reaction-Mastermix for all samples: for one sample (Σ=13µl): 5x Buffer 4µl dNTP (10mM) 1µl RNase OUT 0,5µl Reverse Transkriptase 0,5µl DTT 2µl 5µl H20 • preparation of the RNA samples: mix 4µl RNA + 1µl (10pmol) Primer + 2µl H2O (control: one sample without template) • incubate these 7µl samples 1min at 65°C, then put them directly on ice • add 13µl reaction-mix to each sample, incubate 1h at 42°C • store samples at -80°C Experiment Transduktion von Zielzellen und Genregulation: • 72h nach Transduktionsbeginn können die Zielzellen mittels Fluoreszenzmikroskopie (EGFP) und Durchflusszytometrie auf den erfolgten Gentransfer und die Induktion der Genexpression hin untersucht werden. Auswertung des Experiments Transduktion von Zielzellen und Genregulation (EGFP): Die transduzierten Zellen werden unter dem Axiovert (Mikroskop) untersucht. Um eine präzise quantitative Aussage über den Virustiter zu erhalten, werden die Zellen abgelöst und mittels des BD Accuri Durchflusszytometer auf die Gentransferrate analysiert. Dadurch kann die Zahl der infektiösen Vektorpartikel pro Volumeneinheit Überstand bestimmt werden (t.u./ml; t.u. = transducing units). Diese Titer können graphisch dargestellt werden. Zur Unterscheidung von humanen und murinen Zellen, werden die Zellen zur Analyse der regulierten Genexpression zusätzlich mit einem Antikörper gefärbt, welcher gegen HLA(A,B,C)-Oberflächenmoleküle gerichtet ist. Anschließend wird auch hier die Genexpression und Oberflächenfärbung mittels Durchflusszytometrie bestimmt. 34 Experiment GT3 Vergleiche die Titer der verschiedenen Vektorpartikel in den unterschiedlichen Zielzellen. - Bei den SC-1 und HT1080-Zellen handelt es sich um eine humane und eine murine Zelllinie. Welche ist was? - Welche Aussage über die vorhandenen Rezeptoren können getroffen werden? - Welche Wirtsspezifität (human oder murin) hat der verwendete Antikörper? - Welche Zelllinie exprimiert den Transaktivator und welcher Typ (TetOn/TetOff) wird exprimiert? - Wie können Unterschiede im Titer zwischen den einzelnen Vektortypen erklärt werden? Experiment Isolierung von Nukleinsäuren und PCR Detektion: Isolierung zellulärer DNAs Die Gesamt-DNA der MLV-Vektor-transduzierten Zellen soll isoliert werden und die Anwesenheit der Vektorsequenzen mit PCR nachgewiesen werden. Wir nutzen zur Isolierung der DNA das QIAGEN DNeasy Tissue Kit. DNeasy Tissue procedure: The kit uses advanced silica-gel-membrane technology for rapid and efficient purification of total cellular DNA. The buffer system is optimized to allow direct lysis followed by selective binding of DNA to the DNeasy membrane. DNeasy purified DNA typically has an A260/A280 ratio between 1.7 and 1.9, and is up to 50 kb in size. The DNeasy procedure also efficiently recovers DNA fragments as small as 100 bp. Before lysis of cells, these have to be detached from monolayer culture and washed: • • • • • • Remove medium Wash cells 1x with 1 ml PBS/well Detach cells by adding 500 µl Trypsin-EDTA + 500 µl PBS and incubate for 5 min at RT Transfer cells into Eppendorff Tube Spin down (5 min 2000 rpm) Wash cells with 1 ml PBS • Spin down (5 min 2000 rpm) 35 Experiment GT3 Purification of total DNA from cultured animal cells: • • • • • • • • • • • • • • • Resupend pellet in 200µl PBS Add 20µl proteinase K and 200µl buffer AL (do not add proteinase K directly to buffer AL), mix by votexing Incubate 56°C 10 min Add 200 µl EtOH, mix by vortexing Transfer mixture to DNeasy mini spin column (placed in a 2 ml collection tube) spin 1 min at 8000 rpm Replace the collection tube, add 500µl buffer AW1 spin 1 min at 8000 rpm Replace the collection tube, add 500µl buffer AW2 spin 3 min at 14000 rpm place the column in clean 1.5 ml centrifuge tube add 200 µl buffer AE on the membrane incubate 1 min spin 1 min at 8000 rpm to elute DNA determine the DNA concentration Experiment Isolierung von Nukleinsäuren und PCR Detektion: → Detektion von GFP Sequenzen mittels PCR an RNA, cDNA und DNA • prepare PCR-reaction-mix for all samples (additional: one plasmid control to check the PCR and to check for DNA content in the RNA: Instead of cDNA use 0,5µl RNA (DNase I digested and undigested) as Template in the PCR): for one sample (Σ=50µl): sample (cDNA or DNA or RNA or H20) 10x Buffer dNTP (10mM) Primer 1 (10pmol/µl) Primer 2 (10pmol/µl) Taq-Polymerase H20 in 10µl 5µl 1µl 1µl 1µl 0,5µl 31,5µl • program: 7. 4min 95°C 8. 40sec 95°C 9. 40sec 58°C (Temp depends on the Primer) 10. 1min 72°C (Time depends on expected product length: 1 min / kb) 11. 10min 72°C 12. hold at 4°C 30 repeats from step 2 to 4 • Analyse samples on agarose gels. 36 Experiment GT3 Abb. 4: Pipettierschema für die MLV-abgeleiteten Vektoren. 37 Experiment GT3 Abb. 5: Pipettierschema für den MLV-abgeleiteten Vektor zur regulierten Genexpression. Referenzen: Gossen M, Bujard H. Tight control of gene expression in mammalian cells by tetracycline-responsive promoters. Proc Natl Acad Sci U S A. 1992 Jun 15;89(12):5547-51. Heinz N, Schambach A, Galla M, Maetzig T, Baum C, Loew R, Schiedlmeier B. Retroviral and transposon-based tet-regulated all-in-one vectors with reduced background expression and improved dynamic range. Hum Gene Ther. 2011 Feb;22(2):166-76. doi: 10.1089/hum.2010.099. Epub 2010 Dec 19. Loew R, Heinz N, Hampf M, Bujard H, Gossen M. Improved Tet-responsive promoters with minimized background expression. BMC Biotechnol. 2010 Nov 24;10:81. doi: 10.1186/1472-6750-10-81. Schambach A, Mueller D, Galla M, Verstegen MM, Wagemaker G, Loew R, Baum C, Bohne J. Overcoming promoter competition in packaging cells improves production of self-inactivating retroviral vectors. Gene Ther. 2006 Nov;13(21):1524-33. Epub 2006 Jun 8. 38 Experiment I1 Experiment Immunologie I1: Isolation of Peripheral Blood Mononuclear Cells (PBMC’s) and Macrophage infection experiment with Leishmania parasites PD. Dr. Ger van Zandbergen Immunologische Nachweismethoden Dr. Jörg Kirberg Versuchstag 1 (AG Kirberg) Teil I Präparation von Zellen aus den verschiedenen Organen der verschiedenen Mausstämme, FACS-Färbung, FACS-Messung Teil II FACS-Auswertung und Analyse der Ergebnisse Versuchstag 2 (AG van Zandbergen) Isolation of Peripheral Blood Mononuclear Cells (PBMC’s) Versuchstag 3 (AG van Zandbergen) Infection of PBMC with Leishmania parasites 39 Experiment I1 Versuchstag 1: Montag Entwicklung der Lymphozyten: Analyse von primären und sekundären lymphatischen Organen (Thymus, Knochenmark, bzw. Milz und Lymphknoten) mittels Durchflußzytometrie (FACS, Fluorescence Activated Cell Sorting/Analysis) Lymphozyten reifen in den primären lymphatischen Organen aus Vorläuferzellen, welche letztendlich auf hämatopoietische Stammzellen (HSCs) zurückzuführen sind. HSCs sitzen im Knochenmark sowie temporär, während der foetalen Entwicklung, in der foetalen Leber. Der Reifungsprozess der Lymphozyten beinhaltet die erstmalige Expression der Antigen-spezifischen Rezeptoren. Hierbei entwickeln sich die B Zellen im Knochenmark. B Zellen exprimieren als Antigenspezifische Rezeptoren den B Zell Rezeptor (BCR, als Serumprotein auch Immunoglobulin, Ig, oder Antikörper, Ak, (antibody, Ab) genannt). BCRs sind membranständige Heterodimere, bestehend aus leichter und schwerer Kette, die von den B Zellen als Effektormolekül auch sezerniert werden. Die Gene, welche für den BCR kodieren, liegen als Gensegmente vor, welche während der Entwicklung der B Zellen durch Gen-Umlagerungen, die sog. V(D)J-Rekombination, zusammengefügt werden um einen kodierenden Leserahmen zu erhalten. T Zellen entwickeln sich im Thymus. Dazu wandern kontinuierlich Vorläuferzellen, die aus dem Knochenmark stammen, über den Blutstrom in den Thymus ein. T Zellen tragen als membranständigen Rezeptor den T Zell Rezeptor (TCR); Dies sind Heterodimere deren Gene, wie bei den B Zellen, aus Gensegmenten per V(D)J-Rekombination während ihrer Entwicklung, welche im Thymus erfolgt, erstellt werden. Nach ihrer Reifung besiedeln die Lymphozyten die verschiedenen sekundären lymphatischen Organe, wie Milz, Lymphknoten, Peyer’s Patches, Omentum, Peritoneum, etc.. Dort verleiben sie nicht statisch sondern sind i.d.R. ausgesprochen mobil. Als reife Lymphozyten re-zirkulieren sie kontinuierlich zwischen dem Blutstrom und den sekundären lymphatischen Organen. Dazu treten sie z.B. aus dem Blut durch das HEV (high endothelial venules) in die Lymphknoten ein. Diese verlassen sie über die efferenten Lymphgefässe, die schlussendlich zum Thoracic duct führen ("Milchbrustgang“, Lymphsammelstamm) der in der Nähe des Herzens in den linken Venenwinkel und damit ins Blut mündet. Neben BCR und TCR bzw. den Rezeptoren von NK-Zellen lassen sich verschiedene SubPopulationen von Lymphozyten durch verschiedene weitere, auf ihrer Zelloberfläche differenziell exprimierte Moleküle, unterscheiden. Den verschiedenen Markern wird meist eine CD-Nummer zugewiesen (Clusters of Differentiation, CD-Marker, z.B. CD4, welches ursprünglich dem Marker "L3T4“ bzw. "Ly-4“ (murine T-Helfer Zellen) bzw. "T4“ (humane T-Helfer Zellen) entsprach). Durch spezifische Färbung der verschiedenen Oberflächenmarker und Analyse per Durchflußzytometrie (FACS, Fluorescence Activated Cell Sorting/Analysis) können quantitative 40 Experiment I1 und qualitative Veränderungen der einzelnen Sub-Populationen festgestellt werden. Dies kann z.B. zur Diagnostik von Krankheiten bzw. der Überwachung einer Therapie dienen. In diesem Praktikum werden Zellen aus primären sowie sekundären lymphatischen Organen isoliert, die verschieden, genetisch manipulierten Mäusen, entnommen werden. Die Mäuse sind transgen bzw. knock-out für Gene die in der Entwicklung der Lymphozyten bedeutsam sind. Die Teilnehmer sollen die zu beobachtenden Veränderungen gegenüber dem Normalzustand beschreiben und versuchen diese Beobachtungen den möglichen genetischen Veränderungen zuzuordnen. Folgende Mausstämme/Allele können vorkommen: Wildtyp (nicht manipuliert), z.B. Stamm C57BL/6J (B6) oder BALB/c (C), B6.Rag-1-KO oder C.Rag-2-KO (Recombination Activating Gene 1 bzw. 2), C.Ova23-3-TCR-tg (transgen für einen Ovalbumin-Peptid 323-339, I-Ad-restringierten TCR), B6.JHT-KO (deletion der J Genelemente im IgH (schwere Kette Ak)-Locus). Materialien • Eppendorf • Zentrifuge (für Röhrchen bzw. für 96-well Tubes Verdünnungen Platten; 5°C) • Pipetten (P20, P200, P1.000) (1.5 bei ml) Zellzahlbestimmung, Antikörper • Vortex • 14 ml Falcon Röhrchen mit Deckel • Eisbox • 6 ml Falcon Röhrchen mit Deckel • Gestelle für Röhrchen • 2 ml Spritzen, Plastik, • CO2 zum Töten der Spendertiere (bzw. per • Petrischalen (6 cm Ø) • 96-well Platten, U-Boden Genickbruch durch Betreuer) • 95 % Ethanol (zum Anfeuchten) • Micron Röhrchen für FACS Analyse • FACS-Puffer (PBS mit 2 % FCS (fetal calf • Nylon Netze (100 μm, 'grob') • Spritzen mit aufgesetzten 40 μm ('fein') serum)), steril • ACT für Puffer Nylon Netzen (Ammoniumchlorid-Tris • Pasteur Pipetten aus Glas, kurz, Puffer), steril • graue ‘Nuggis’ • Trypan Blau Lösung, 0.4 % (w/v) • Fluoreszensmarkierte Antikörper (FITC, PE, APC, PerCP) für • Instrumente (Schere, Pinzette) verschiedene • Bechergläser Oberflächen-Antigene (z.B. CD4, CD8, mit 95 % Ethanol für Instrumente TCRβ, etc.) • Zählkammer (Neubauer/Bürki) • [ PI (Propidium-Iodite) Stock-solution I • Accuboy oder Pipettierball (0.5 mg/ml in PBS) ] • Plastik Pipetten (10 ml, 5 ml, 2 ml, 1 ml) • Gestell für Micron Röhrchen • Pipetten Spitzen, Gelb und Blau • Schema für 96-well Platten 41 Experiment I1 Teil I Präparation von Zellen aus den verschiedenen Mausstämme, FACS-Färbung, FACS-Messung Organen der verschiedenen Verschiedene Mäuse stehen zur Verfügung. Sie stammen aus der Versuchstierzucht des Instituts. Die Tiere werden durch cervicale Dislokation oder mit CO2 getötet (erfolgt ausschließlich durch die/den BetreuerIn). Entnahme der Organe Tiere werden auf einer Korkplatte oder Styropor-Brett platziert und mit Ethanol befeuchtet. Die Organe werden entnommen (Milz, Thymus, evtl. Lymphknoten, Knochenmark) und in ca. 5 ml FACS-Puffer (PBS mit 2 % FCS) in einer Petri-Schale auf Eis platziert. Herstellung einer Einzelzell-Suspension und Lyse der Erythrozyten (Milz, Knochenmark) Die weichen lymphoiden Organe werden zwischen zwei Lagen von Nylon-Netzen platziert und mit einem Pistill oder einer gebogenen Pinzette ausgeklopft/-gequetscht. Das Knochenmark wird mit Kanülen ausgespült. Die Zellen werden unter mehrmaligem Nachspülen in einem 14 ml Falcon-Röhrchen gesammelt. Die Zellen werden pelletiert (10’, 300 g (ca. 1200 rpm), 5°C). Bei Milz und Knockenmark sollen die Erythrozyten lysiert werden. Dazu werden die Zellen in ca. 2 ml ACT (RBC-Lysis-Puffer) resuspendiert und kurz (max. 2’ !!!) bei 37°C in einem Wasserbad inkubiert. Sofort danach wird die Lyse durch Auffüllen mit FACS-Puffer gestoppt (Deckel aufsetzen und zum Mischen 2x invertieren). Die Zellen werden anschließend pelletiert (10’, 300 g (ca. 1200 rpm), 5°C). Die Zellen werden in 2 ml FACS-Puffer resuspendiert. Die Zellen werden durch 40 µm Filter gegeben um Aggregate/Klumpen zu entfernen. Die Zellen werden auf Eis gelagert. 42 Experiment I1 Zellzahlbestimmung Die Zellen werden 1:10 in einem geeigneten Volumen vorverdünnt (z.B. 20 µl Zellen + 180 µl FACS-Puffer). Etwas verdünnte Zellsuspension wird mit Trypan-Blau-Lösung zu gleichen Teilen gemischt (z.B. 20 µl Zellen + 20 µl Trypan-Blau-Lösung). Die Mischung wird in eine Zählkammer (Neubauer, Bürki) überführt und die lebenden (ungefärbten) Zellen werden gezählt. Die Zellkonzentration ergibt sich aus der Multiplikation von Zellzahl (Mittelwert der Quadranten), den Verdünnungsfaktoren und dem Kammerfaktor (104). Schematische Darstellung einer Neubauer Zählkammer mit vier Quadranten à 16 Kleinquadraten Resultate der Zählung notieren; Verteilung der Zellen für die anschließende Färbung berechnen (ca. 1,5 x 106 Zellen pro Färbung pro Well): Organ MausStamm x 106 Zellen/ml (2 ml) 43 x 106 Zellen/Organ für FACS: 1,5 x 106 Zellen Experiment I1 FACS-Färbung und Analyse der Zellen Die Zellen werden in die Wells einer 96-well-Platte anhand des Färbeschemas verteilt (evtl. Zellen mit anderen Teilnehmern austauschen). Sollte das Volumen kleiner als 100 µl sein, bitte 100 µl FACS-Puffer zugeben. Die Färbelösungen ansetzen: Unter Beachtung welche Verdünnungen erforderlich sind, wie viele Proben Sie mit der jeweiligen Färbelösung anfärben möchten, und dem Färbevolumen (100 µl pro Well). Die Antikörper werden in FACS-Puffer verdünnt. Wenn die Färbelösungen bereit sind werden die Zellen pelletiert (2’, 5°C, 450 g (ca. 1’500 rpm)), dann ‚ausgeflickt’ + abgetupft, und resuspendiert (Vortex). Evtl. werden 10 µl 2.4G2 Hybridom-Überstand zu jedem Well zugegeben und ge-vortext (2.4G2 blockiert FcRezeptoren (CD16/32)). Dann wird die jeweilige Färbelösung (100 µl) bzw. FACS-Puffer zugegeben; 20’ auf Eis inkubieren. Nach der Färbung werden überschüssige Antikörper durch waschen entfernt: Mit 100 µl FACS-Puffer verdünnen, zentrifugieren, ‚ausgeflickt’ + abgetupft, Zellen in 200 µl FACSPuffer aufnehmen. Nochmals waschen. Zuletzt die Zellen in 100 µl FACS-Puffer aufnehmen. Die Zellen sind nun bereit für die FACS-Analyse. Sie dazu in die Micron-Röhrchen überführen. Die FACS-Messungen erfolgen am Durchflusszytometer (FACSCalibur, LSR II, o.ä.). Ein mögliches Färbeschema: 44 Experiment I1 Teil II FACS-Auswertung und Analyse der Ergebnisse Die FACS-Daten können mit verschiedenen Programmen analysiert werden (FACSDiva, CellQuest, FlowJo, WinMDI, etc.). Zur Besprechung der Ergebnisse beantworten Sie bitte folgende Fragen: 1. Welche Zelltypen lassen sich mit Hilfe der verwendeten Reagenzien detektieren? 2. Welche (prozentuale) Verteilung an T und B Zellen ist in den verschiedenen Organen zu erwarten und was finden Sie? 3. Welche Subpopulationen von Zellen erwarten Sie in den verschiedenen Organen (z.B. bezüglich CD4, CD8 und TCR Expression in Thymus und Lymphknoten)? 4. Welchen Einfluss hat die Deletion des Rag1, Rag2-Gens bzw. der JH-Elemente auf die Entwicklung der verschiedenen Lymphozyten? 5. Was ist das Resultat der Komplementierung mit (fertig rearrangierten) αβTCR-Genen in Rag-defizienten Tieren in Bezug auf die Entwicklung von T und B Zellen? 6. Welche absoluten Anzahlen von T und B Zellen finden sich in der Milz von Mäusen? 45 Experiment I1 Versuchstag 2 und 3: Dienstag und Donnerstag Isolation of Peripheral Blood Mononuclear Cells (PBMC’s) and Macrophage infection experiment with Leishmania parasites Introduction 1.1 Leishmaniasis Leishmaniasis is a parasitic infection with the protozoan genus Leishmania which is endemic in 88 countries, mainly prevalent in the tropical and subtropical regions of the world. Currently approximately 12 million people are suffering from Leishmaniasis, however about 350 million people are worldwide threatened and the estimated incidence of 2 million new cases arises each year. Up to date here are about 21 Leishmania species known to be pathogenic for humans. According to the Leishmania species initiating infection and the immunologic status, humans can develop a large spectrum of symptoms ranging from selfhealing lesions to a severe organ-infiltrating manifestation of the disease. Four major forms of human Leishmaniasis have been described: cutaneous, diffuse cutaneous, mucocutaneous and visceral Leishmaniasis. The localized cutaneous Leishmaniasis (LCL), which is primarily caused by Leishmania major (L. major) and Leishmania tropica (L. tropica) produces selfhealing skin ulcers on exposed parts of the body, which is also known as Aleppo boil. On the other hand, chronic diffuse cutaneous Leishmaniasis (DCL) caused by Leishmania aethiopica (L. aethiopica) and Leishmania mexicana amazonensis (L. mexicana amazonensis) produces widespread skin lesions all over the body which resemble leprosy. Another more severe form is the mucocutaneous Leishmaniasis (MCL) characterized by the infiltration of the mucousal membranes, especially those of the nose and mouth leading to extensive tissue damage and disfiguration (Espundia). The causative agents of MCL are Leishmania braziliensis (L. braziliensis) and Leishmania mexicana pifanoi (L. mexicana pifanoi). The most severe and life threatening form is the visceral Leishmaniasis (VL) also named Kala azar caused by the Leishmania donovani complex, including Leishmania donovani, Leishmania infantum and Leishmania chagasi (L. donovani, L. infantum and L. chagasi). This form affects internal organs such as the lymph nodes, the liver, the spleen and the bone marrow and is lethal if untreated. 1.2 Life cycle of L. major parasites The Leishmania parasite is a dimorphic unicellular parasite belonging to the class of Kinetoplastida because of the prominent DNA-containing mitochondrion, the kinetoplast. The life cycle of Leishmania is characterized by the alteration between two hosts, a sand fly and numerous mammals. The flagellated promastigote life stage of Leishmania spp. lives and replicates extracellular in the digestive tract of the female sand fly of the subgenera 46 Experiment I1 Phlebotomus and Lutzomyia. In the midgut of the insect vector, the promastigotes mature through a differentiation process called metacyclogenesis from a non-virulent procyclic form into the virulent metacyclic form. In contrast to gut epithelial attached procyclic parasites, metacyclic promastigotes detach and accumulate in the anterior parts of the digestive tract, like the glands from where they can be inoculated into the skin of a mammalia during a blood meal of the sand fly. Inside the mammalian host, Leishmania parasites are only able to survive intracellular. Therefore the parasite attracts polymorphonuclear granulocytes (PMN) to the site of infection via a chemotactic factor termed Leishmania chemotactic factor (LCF). The virulent inoculum of Leishmania consists of viable and apoptotic promastigotes leading to a silent uptake of the promastigotes and to a higher intracellular survival rate inside the PMN by evading their antimicrobial killing mechanisms. After engulfment by the recruited PMN, the parasites take advantage of the fact that aging neutrophils die by apoptosis and simultaneously recruit macrophages (MF) via MIP-1 beta (CCL4) release for their clearance. Hiding inside apoptotic PMN, the Leishmania promastigotes are transferred into their final host cells the MF, by using PMN as Trojan horses. Inside MF the non-multiplying promastigotes are located within specialized compartments, called phagolysosomes, where they differentiate into the non-motile amastigote life stage, which is adapted to the acidic and hydrolase-rich environment within the phagolysosomes. Amastigotes are able to multiply inside MF and are responsible for the maintenance and propagation of the disease by infecting surrounding phagocytes causing Leishmaniasis. The life cycle is completed when a sand fly take a blood meal from an infected mammal. The free or in MF resident amastigotes are then able to rapidly differentiate into the promastigote life stage in the gut of the insect. The adaptation to both an arthropod vector and a mammalian host is a typical feature of the obligatory intracellular Leishmania parasite and is illustrated in Fig. 1. Figure 1: Life cycle of Leishmania spp. [Source: http://en.wikipedia.org/wiki/File:Leishmaniasis_life_cycle_diagram_en. svg, accessed on 04/05/2012] 47 Experiment I1 1.3 The interaction with human MF In contrast to PMN where Leishmania promastigotes do not differentiate into the amastigote life stage, MF are known to be the final host cells. Inside MF promastigotes transform into amastigotes and start to multiply. However, MF are a heterogeneous population of cells with various immune and homeostatic functions in the human body. They consist of mature MF and circulating immature monocytes which can migrate into tissue and differentiate after stimulation by different signals into tissue resident MF, such as microglia in the central nervous system or alveolar MF in the alveoli of the lung. For cutaneous Leishmaniasis it is not known which distinct subtype of MF is infected. Blood derived human monocytes were shown to be able to differentiate into two different phenotypes of MF in vitro after stimulation with different growth factors. These phenotypes were termed type I and type II MF. Type I MF (MF I): The incubation with GM-CSF polarizes human blood monocytes into type I MF or classical activated MF. The morphology of this phenotype is fried egg-shaped and the cells are CD14 positive, but CD163 negative. MF I produce pro-inflammatory cytokines when stimulated such as TNF alpha, IL-1, IL-23, and IL-12(p40). In addition they are efficient producers of antimicrobial effector molecules like reactive oxygen and nitrogen intermediates. Type I MF play an important role in the clearance of apoptotic cells and support the Th-1 response by the secretion of IL-12 and IL-6. Therefore, type I MF are termed pro-inflammatory phagocytes. Type II MF (MF II): M-CSF incubation leads to the differentiation of monocytes into type II MF or alternative activated MF. MF II are wide stretched cells and show a spindle-like shape. In contrast to MF I, MF II have a higher phagocytosis capacity. Furthermore, they play an important role in the clearance of necrotic cells. A particular feature of MF II cells is the expression of CD14 and the scavenger receptor CD163, which is supposed to be involved in anti-inflammatory processes. The MF II phenotype is hallmarked by a lack of microbicidal activity as well as IL12(p40) secretion and release anti-inflammatory IL-10 as the signature cytokine upon activation. Moreover, MF II down-regulate the IL-12 production, have poor antigen presentation and were shown to secrete TGF beta upon uptake of apoptotic cells. Therefore, type II MF are termed anti-inflammatory phagocytes. Despite the recognition and characterization of these distinct subtypes of human MF, the consequences of such differently polarized MF interacting with L. major like uptake and parasite propagation remain unclear. 48 Experiment I1 Tag1: Isolation of Peripheral Blood Mononuclear Cells (PBMC’s) 1. Dilute a buffy coat (∼ 30 ml) 2-3 times with prewarmed PBS (30mL blood + 70mL PBS) to a final volume of ∼ 100ml 2. Layer 15 ml pre-warmed (37°C) Leukocyte separation medium 1077 at the bottom 3. of 4 50 ml tubes (put the LSM back in the fridge as soon as possible as it is light sensitive) Layer carefully 25 ml buffy coat-dilution on top 4. Centrifuge at 1600 rpm for 30 min at room temperature without brake! 5. 6. Harvest the cells from the interphase and the medium above and put them in 6 new 50 ml tubes Add wash-medium (PBS + 5% medium) till a final volume of 50 ml 7. Centrifuge 8 min at 2200 rpm RT and discard supernatant to remove residual LSM 8. Centrifuge 8 min at 1600 rpm RT and discard supernatant 9. Centrifuge 8 min at 800 rpm RT to remove thrombocytes and discard supernatant 10. Add 10 ml cold Ammoniunchloride 0,15M (in H2O) to each tube for ery-lysis for 10 – 15 min. [4 g in 500 ml H2O] 11. Add wash-medium (PBS + 5% medium) till a final volume of 50 ml 12. Centrifuge 8 min at 800 rpm RT and discard supernatant 13. 14. 15. 16. Wash some more when the supernatant is not clear enough or still red Pool the cells in one tube and repeat step 12 6 Count them, if there are less than 240 · 10 use MACS for isolation of monocytes Plastic adherence: seed max. 40 - 50 · 106 PBMC’s in a flask in 5 ml medium + 1% plasma 17. Incubate the cells for 1,5 h in an incubator 37°C, 5% CO2 18. Remove the non-adherent cells by washing the flasks gently 2 times with 4ml prewarmed wash-medium (37°C) 19. Add culture medium supplemented with 10 ng/ml GM-CSF (5 ml) themed 30 ng/ml M-CSF (4 ml) 20. Incubate the cells for 5-7 days at 37°C, 5% CO2 Type I (pro-inflammatory): 10 ng/ml GM-CSF (store working aliquot at 4°C) 100 µg/ml stock (at -80°C, box 16) Type II (anti-inflammatory): 30 ng/ml M-CSF (store working aliquot at -20°C) 50 µg/ml stock (at -80°C, box 14) Material: Single buffy coat LSM 1077 Lymphocyte Separation Medium, 500ml (PAA: J15-004) 1x PBS without Calcium and Magnesium 49 Komplett Medium M-CSF, 50µg (R&D) GM-CSF, 100µg (Leukine) Experiment I1 Tag 2: Macrophage infection experiment with Leishmania parasites 1. Counting the macrophages Material: Complete medium: 500 ml RPMI (or other cell culture medium) + 5 ml L- Glutamin + 5 ml Penicillin/ Streptomycin + 5 ml Hepes-Buffer + 1 ml 25mM ß- mercaptoethanol + 10 % FCS (50ml) Counting chamber (Neubauer Hemocytometer Improved): 2 Depth: 0,100 mm x 0,0025 mm Macrophages (MF): Differentiate monocytes isolated from Buffy-Coat with either M-CSF (MF type I) or GM-CSF (MF type II) for 6-9 days Procedure: • Put macrophages on ice for 30 min • Detach macrophages from culture flask with a cell scraper (or other tool to detach cells) • Wash flasks with 2 ml cold PBS (4°C) and check in the microscope if the macrophages are detached from the bottom of the culture flask • Centrifuge the cells: 1800 rpm, 8 min, RT and resuspend the pellet in 1 to 5 ml medium depending on the size of the pellet • Fill the counting chamber with 10µl cell suspension • Count 16 squares 1 square • Calculate the cell number: Counted cells x 10 000 (chamber factor) = cells/ ml 2. Adherence of the macrophages (MF) • Resuspend MF in complete medium to a final concentration of 1 x 106 MF/ml Centrifugation of macrophages: 1800 rpm, 8 min, room temperature (RT) • Pipette 100µl per well of the cell suspension in a 96-well plate • Let the MF attach to the bottom of the well at RT (or 37°C) for 30 min 50 Experiment I1 3. Counting the Leishmania parasites Counting chamber (Neubauer Hemocytometer Improved): Depth: 0,02 mm x 0,0025 mm2 Procedure: • Dilute stationary phase (stat-phase) parasites in complete medium (1 : 10) e. g. 100µl parasite suspension + 900µl complete medium • Fill the counting chamber with 5µl parasite suspension • Count 16 squares (count both viable and dead parasites) • Calculate the parasite number: Counted parasites x 50 000 (chamber factor) x 10 (dilution factor) = parasites/ ml 4. Co-incubation of the macrophages with the parasites (Best point to start practical work with the students) Procedure: • Resuspend Leishmania parasites in complete medium to a final concentration of 20 x 106 MF/ml Centrifugation of parasites: 2400 g, 8 min, room temperature (RT) • Remove the medium from the adherent MF with a pipette and add 100µl of the Leishmania suspension (multiplication of infection (MOI) 1:20) Pro-inflammatory MF Leishmania parasites stat-phase + Anti-inflammatory MF • Centrifuge the parasites on to the plate for 4 min at 1200 rpm, RT • Incubate for 2 h at 37°C • Wash the 96-well plate with pre-warmed washing buffer (PBS with 5% complete medium) by washing from the four sides of the well to remove the extracellular parasites 51 Experiment I1 5. Staining of the infected macrophages using Diff-Quik Material: Fixation solution (blue) Staining solution I (orange) Staining solution II (violet) Procedure: • Remove the supernatant carefully to not lose so much cells • Dry the MF containing plate at RT for 5 min • Fix the cells with 100 µl Fixation solution (methanol) for 5 min (carefully pipet it to the edge of the well!) • Remove the Fixation solution and add 100 µl of Staining solution I (Eosin) for 2 min • Remove Staining solution I and add 100 µl of Staining solution II (Thiazine) for 2 min • Remove Staining solution II and wash the MF containing plate with tap water and airdry the plate • Count the infection rate under a microscope (400x magnification) by counting at least 200 MF at four different spots in the well (see Figure). Choose field of vision that have similar distribution • Count parasite burden by counting the number of parasites per MF of at least 20 MF Examples of infected MF with Leishmania major 52 Experiment I2 Experiment Immunologie I2: Messung der Antigen-spezifischen CD4+ T-Zellaktivierung Dr. Masako Toda Achtung: Der Versuchsteil I2 besteht aus 2 unabhängigen Blöcken. Der erste Teil findet am Montag und Donnerstag im Labor Toda statt, der zweite Teil findet am Dienstag im Labor Mühlebach statt. 53 Experiment I2 Versuchsteil I, Labor Toda Hintergrund T-Zellen stellen eine Untergruppe der Lymphozyten dar. Sie spielen eine kritische Rolle in der adaptiven Immunantwort. T-Zellen werden in zwei große Untergruppen unterteilt, basierend auf der Expression der Co-Rezeptoren CD4 oder CD8 auf der Zelloberfläche. CD4+ T-Zellen, auch T-Helfer-Zellen genannt, assistieren anderen Leukozyten bei der Ausführung ihrer immunologischen Aufgaben: Sie unterstützen die Reifung von B-Zellen in Plasma-Zellen (Antikörper-produzierende Zellen) und die Aktivierung und Expansion von CD8+ T-Zellen und Makrophagen. Dagegen differenzieren CD8+ T-Zellen überwiegend in zytotoxische Zellen welche Virus-infizierte Zellen und Tumor-Zellen eliminieren können. Für die Aktivierung von naiven CD4+ T-Zellen werden zwei Signale benötigt. Das erste Signal besteht aus der Interaktion des T-Zell-Rezeptors (TCR) mit Antigen-Peptid/MHCKlasse II-Molekül Komplexen, welche auf der Oberfläche von Antigen-präsentierenden Zellen (APCs), z.B. dendritischen Zellen und Makrophagen, präsentiert werden. Der TCR bindet dabei neben dem MHC Molekül nur an ein Peptid-Fragment des Antigens, welches im Inneren der präsentierenden Zelle vorher aus dem Antigen prozessiert wurde. Diese Prozessierung findet in lysosomalen bzw. endosomalen Kompartimenten der APCs statt. Die Peptide werden also durch die MHC-Klasse II Moleküle auf der Oberfläche der APCs den CD4+ T-Zellen präsentiert. Aufgrund der großen Variabilität der möglichen Antigene ist für eine effektive Immunantwort eine mindestens ebenso große Variabilität der TCRs erforderlich. Diese wird durch die VDJ-Rekombination der Gen-Elemente der TCR Ketten während der T-Zell-Reifung im Thymus gewährleistet. Das zweite Signal zur T-Zell-Aktivierung ist Antigen-unspezifisch und wird als 'Co-Stimulus’ bezeichnet. Die bedeutendsten co-stimulatorischen Signale für die Aktivierung von naiven TZellen werden durch Mitglieder der B7-Familie (B7-1: CD80; B7-2: CD86) vermittelt, welche ebenfalls auf der Oberfläche von APCs präsentiert werden. Der dazugehörige Interaktionspartner auf den T-Zellen ist CD28. Naive T-Zellen exprimieren den IL-2 Rezeptor (IL-2R), welcher aus einer Kette gebildet wird. Dabei hat die - und einer -Kette eine niedrige Bindungsaffinität für IL-2. Bei der Co-Stimulierung von T-Zellen über CD28 mit B7-1 oder B7-2, in Kombination mit einer TCRAktivierung, wird die Synthese von IL-2 induziert. IL-2 ist ein essentieller Wachstumsfaktor für T-Zellen, der die T-Zellproliferation fördert. Die autokrine und/oder parakrine Verfügbarkeit von IL-2 ist daher ein entscheidender Schritt für die adaptive Immunantwort. Es ermöglicht den wenigen für ein bestimmtes Antigen spezifischen T-Zellen sich effizient zu 54 Experiment I2 vermehren. Ohne diese Expansion kann keine effektive Immunantwort ausgeführt werden (z.B. gegen Pathogene). Um die T-Zell vermittelte Immunität zu untersuchen werden vielfach transgene Mäuse verwendet, die einen vorgefertigten TCR von bekannter Spezifität in den T Zellen exprimieren. Dies hat gegenüber normalen [Wildtyp (WT)]-Mäusen den Vorteil, dass in diesen Tieren T-Zellen einer bestimmten Antigen-spezifität in großer Zahl produziert werden. Dies vereinfacht die experimentelle Detektion einer Antigen-spezifischen Immunantwort erheblich. WT-Mäuse haben dagegen eine große Variabilität in ihrer T-Zell-AntigenSpezifität (polyklonale TCRs), und haben deswegen nur eine sehr geringe Anzahl von TZellen, welche spezifisch für ein gegebenes Antigen sind. DO11.10 Mäuse sind ein Beispiel für eine Mauslinie, die einen transgenen TCR trägt. Die meissten CD4+ T-Zellen aus diesen transgenen Mäusen tragen auf ihrer Oberfläche einen TCR, der an den Komplex aus den Aminosäuren 323-339 des Ovalbumins (ein Hauptallergen des Eiweißes) und präsentiert auf I-Ad Klasse II MHC Molekülen bindet. In diesem Praktikum detektieren wir die Ovalbumin-induzierte T-Zellaktivierung von Lymphozyten aus DO11.10 Mäusen. Dazu wird den Mäusen die Milz entnommen. Als sekundäres lymphatisches Organ enthält die Milz sowohl T-Zellen wie APCs. Es wird eine Einzel-Zellsusupensionen hergestellt und die Zellen werden in vitro mit OVA stimuliert. Falls OVA-spezifische T-Zellen aktiviert wurden sollte IL-2 sekretiert werden und im Überstand der Zellen zu detektieren sein. Das produzierte IL-2 wird mittels Enzyme Linked-ImmunoSorbent Assay (ELISA) detektiert und quantifiziert. 1. Arbeits-Tag (Montag): Ansetzen der T-Zell Stimulation Mäuse Der verwendete Mausstamm DO11.10 wurde unter spezifiziert pathogen-freien Bedingungen (SPF) in den S1-Räumen der zentralen Tierhaltung im PEI gezüchtet. Als Kontrollmäuse werden wildtyp Mäuse (Stamm BALB/c, von Charles River) verwendet. Die Haltung/Zucht/Tötung der Mäuse erfolgt im Einklang zur relevanten Gesetzgebung. Das PEI wird durch das Regierungspräsidium Darmstadt dementsprechend überwacht. Medium Als Basalmedium wird RPMI 1640, dem 100 U/ml Penicillin, 100 mg/ml Streptomycin und 5x10-5 M 2-Mercaptoethanol zugegeben werden, genutzt. Sollte das Medium kein Glutamin enthalten, muss diese Aminosäure noch zugegeben werden. Dies ist das "Komplettmedium“: 55 Experiment I2 Materialien für "Komplettmedium“: RPMI 1640 (Invitrogen) : 500 ml Penicillin, Streptomycin-Lösung : 5.0 ml (100x konzentriert Invitrogen) Glutamine (100x konzentriert PEI) : 5.0 ml 2-Mercaptoethanol (14 M, Sigma Aldrich) : 3.5 µl Es sind ZWEI verschiedene Medienansätze [(A) mit/ohne Antigen, (B) "Zellkulturmedium“] daraus zu erstellen: (A): Dem Komplettmedium wird kein/wird Antigen/Mitogen zugegeben (s.u.) (B): Dem Komplettmedium wird FCS (fötales Rinderserum) zugegeben: 500 ml Komplettmedium + 50 ml FCS = "Zellkulturmedium“ Herstellung der Antigen-Lösung (A) - als Antigen wird OVA (Sigma Aldrich; Grade V) verwendet - Es werden Lösungen mit einer Konzentration von 20, 200 und 2000 µg/ml OVA in Komplettmedium (also ohne FCS) angesetzt - als T-Zell Mitogen (positive Kontrolle) wird, Concanavalin A (Con A, Sigma Aldrich) verwendet - Es werden Lösungen mit einer Konzentration von 2,0 und 10 µg/ml Con A in Komplettmedium (also ohne FCS) angesetzt - 100 µl der gewünschten Antigen-Lösung werden in die vorgesehen Wells der 96er Multiwellplatte einpipettiert - Während der Präparation der Zellen sollte die Platte bei Raumtemperatur gelagert werden (sollte die Präperation der Zellen längere Zeit in Anspruch nehmen, ist es besser sie in den Kühlschrank zu stellen; die Platte sollte aber Raumtemperatur haben, wenn die Zellen ausgesät werden) Herstellung der Milz-Zellsuspension - die Milz wird unter sterilen Bedingungen aus der Maus entnommen, die Arbeit erfolgt unter der sterilen Bank (Laminar Air Flow) - das Organ wird in eine Petrischale (Ø 6 cm) mit 5.0 ml Zellkulturmedium gelegt - um die Zellen der Milz zu erhalten, wird diese mit dem Rücken eines Stempels einer Spritze zerquetscht. Die Zellen schwimmen nun im Medium. - Zellsuspension über ein Zellsieb in ein 50 ml Falcon geben - die Petrischale mit 5.0 ml Zellkulturmedium spülen um restliche Zellen aufzunehmen - nun wird die Zellsuspension in ein 15 ml Falcon überführt 56 Experiment I2 - mit Zellkulturmedium auf 15 ml auffüllen - 10 min bei 1300 rpm bei Raumtemperatur (RT) zentrifugieren - den Überstand verwerfen; etwas Überstand im Falcon belassen, damit das Pellet bedeckt ist, nun das Falcon schütteln, bis sich das Pellet gelöst hat - Lysierpuffer zugeben (150 mM NH4Cl; 1-2 ml je Milz) um die Roten Blutzellen zu lysieren, 1 min bei RT inkubieren - 10 min bei 1300 rpm bei RT zentrifugieren - der Überstand sollte rot sein, das Pellet weiß - der Überstand wird verworfen und das Pellet wird in 12 ml RPMI mit 10 % FCS gelöst - 10 min bei 1300 rpm bei RT zentrifugieren - der Überstand wird verworfen und der Waschschritt 2 x wiederholt - die Zellen werden in einer geeigneten Menge Zellkulturmedium gelöst (5 ml je Milz) - die Zellen werden gezählt und die Zellzahl auf 8 x 106 Zellen eingestellt - 100 µl der Zellsuspension werden je Well in eine 96er Multiwellplatte einpipettiert - zusätzlich sollte 100 µl Antigen-Lösungen in den Wells sein, somit ist das Gesamtvolumen je Well 200 µl Platte jeweils als Dreifachbestimmung/pro gleichen Probenbedingung (z.B.: A1,2,3; A4,5,6; ...) Milzzell-Kultur - die Zellen werden in einem CO2-Inkubator bei 37°C , 5% CO2 und 95% relative Luftfeuchte kultiviert - Der Zellüberstand wird nach 24 h kultivieren abgenommen - der Überstand sollte, bis zur ELISA-Messung, bei -20°C gelagert werden 57 Experiment I2 2. Arbeits-Tag (Donnerstag): Detektion von IL-2 mittels ELISA Beobachtung der Zellen - an Tag 3 der Zellkultur werden die Zellen unter dem Mikroskop angesehen - wenn die T-Zellen durch die Antigenstimulation aktiviert wurden, bilden sie „Blasten“, welche größer sind als nichtaktivierte Zellen - die Zellen werden unter dem Mikroskop fotografiert Zytokin ELISA Die IL-2 Konzentration im Zellkulturüberstand wird mit Hilfe eines ELISA ermittelt. (Medien/Puffer/Reagenzien untenstehend) - der anti-Maus-IL-2-Antikörper wird in Coating-Puffer auf eine Konzentration von 0.5 µg/ml verdünnt - eine ELISA-Platte wird mit 50 µl/well Erstantikörper (=anti-MausIL-2) gecoatet - die Platte wird über Nacht im Kühlschrank inkubiert - die Platte wird 3x mit je 150 µl/well Waschpuffer gewaschen - die freien Bindungsstellen werden durch Zugabe von 100 µl/well Blocking-Puffer und Inkubation der Platte für 1 h bei RT und 80 rpm auf dem Schüttler abgeblockt - die Platte wird 3x mit je 150 µl/well Waschpuffer gewaschen - die Proben und der Zytokin-Standard (von 4000 pg/ml, 2000 pg/ml, 1000 pg/ml …, serielle Verdünnungsreihe; mindestens 4 verschiedene Konzentrationen werden für die Standardkurve benötigt, mehr wären besser) werden in RPMI (mit 5 % FCS) verdünnt. Von den Verdünnungen werden 50 µl/well in die Platte gegeben - die Platte wird für 2 h bei RT und 80 rpm auf dem Schüttler inkubiert - die Platte wird 3x mit je 150 µl/well Waschpuffer gewaschen - der Detektionsantikörper wird in Assay-Puffer auf eine Konzentration von 0.5 µg/ml verdünnt - es werden 50 µl/well Detektionsantikörper in die Platte gegeben - die Platte wird für 1 h bei RT und 80 rpm auf dem Schüttler inkubiert - die Platte wird 3x mit je 150 µl/well Waschpuffer gewaschen - Streptavidin konjugiert mit Meerrettichperoxidase (= HRP-Strep) wird 1:4000 in Assay-Puffer verdünnt - es werden 50 µl/well verdünnte HRP-Strep Lösung in die Platte gegeben - die Platte wird für 30 min bei RT und 80 rpm auf dem Schüttler inkubiert - die Platte wird mindestens 3x mit je 150 µl/well Waschpuffer gewaschen - die TMB-Substratlösungen A und B werden im Verhältnis 1:1 gemischt 58 Experiment I2 - es werden 100 µl/well TMB-Substrat in die Platte gegeben - die Platte wird maximal 30 min bei RT, 80 rpm und im Dunkeln auf dem Schüttler inkubiert (klar - blau) zum Abstoppen der Reaktion werden 50 µl/well 1 N H2SO4 in die Platte gegeben (blau → gelb) - Messung der Absorption bei 450 nm und 630 nm Referenzfilter im ELISA-Reader Platten-Layout: Jede Probe von Tag 1with doppelt im ELISA bestimmt Puffer und Lösungen für den ELISA Coating-Puffer: 50 mmol/l Natriumcarbonatpuffer pH 9,6 (1.696 g Na2CO3 + 2.856 g NaHCO3 ad 1 l mit Reinstwasser) Waschpuffer: PBS + 0.05 % Tween 20 (250 µl Tween in 500 ml PBS) Blocking-Puffer: PBS + 2 % BSA Medium: RPMI 1640 + 5 % FCS Medium wird NUR für die Verdünnung der Proben und des Standards verwendet!!! Assay-Puffer: PBS + 0.05 % Tween 20 + 1 % BSA TMB-Substrat: Tetramethylbenzidine (TMB) substrate reagent set (BD Bioscience) Antikörper Anti-Maus-IL-2 monklonaler Antikörper (Klon; JES6-1A12, Biolegend ) Biotin markierter anti-murine IL-2 monklonaler Antikörper (Klon; JES6-5H4, Biolegend) Standard rekombinantes murines IL-2 (PeproTech) 59 Experiment I2 Versuchsteil II (Dienstag), Labor Mühlebach Diagnostischer Western Blot zum Nachweis von Masernviren Die humorale Immunantwort ist auf die Produktion von Pathogen-spezifischen Antikörpern ausgerichtet, die von B-Lymphozyten produziert werden. Die Aktivierung von naiven BLymphozyten erfolgt durch B-Zellrezeptor-spezifischen Antigenkontakt und benötigt in der Regel zusätzliche Stimulation durch T-Helferzellen (CD4+) mit gleicher Spezifität. Aktivierte B-Lymphozyten differenzieren sich anschließend zu antikörperproduzierenden Plasmazellen und Gedächtnis-B-Lymphozyten aus. Antikörper können auf drei verschiedenen Wegen gegen Pathogene wirken. Einer dieser Wirkungsmechanismen ist die Neutralisation. Hierbei werden Pathogene (z.B. Viren oder intrazelluläre Bakterien) von den sogenannten neutralisierenden Antikörpern an denjenigen Strukturen gebunden, die für den Eintritt in die Zelle benötigt werden; die Bindung zwischen Pathogen und Zelle kann hierdurch nicht mehr stattfinden. Der zweite Wirkungsmechanismus von Antikörpern ist die Induktion von Phagozytose durch Opsonisierung. Hierbei werden an Pathogene gebundene Antikörper von phagozytierenden Zellen vermittelt durch Fc-Rezeptoren, die an die an die konstante Region („Fc“, siehe unten) der Antikörper binden, erkannt. Diese Bindung wirkt als auslösendes Signal für die Phagozyten, das Pathogen aufzunehmen und zu inaktivieren. Als dritter Wirkungsmechanismus können an auf z.B. Virus-infizierten Zellen präsentierten Pathogen-Strukturen gebundene Antikörper die Komplementkaskade (ein Teil des angeborenen Immunsystems) aktivieren. Die Aktivierung der Komplementkaskade führt schließlich zur Abtötung der infizierten Körperzellen. Die Effektormechanismen von Antikörpern werden durch ihre Klassen (IgA, IgD, IgE, IgG, IgM) definiert, die sich aus dem Aufbau ihres konstanten Fc-Teils ergeben. IgAs werden hauptsächlich in Schleimhäuten sezerniert und sind Bestandteil der mukosalen Immunität. Die immunologische Aufgabe von IgEs ist relativ unklar. Es wird vermutet, dass sie eine Rolle bei der Abwehr von Parasiten spielen; unter den in unserem Lebensbereich vorherrschenden Lebensbedingungen sind IgEs jedoch hauptsächlich aus den fehlgeleiteten Immunantworten der Allergien bekannt. Für die humorale Immunabwehr einzelliger Krankheitserreger wie Bakterien und Viren sind hauptsächlich IgMs und IgGs verantwortlich. IgMs werden ind er frühen Phase von Infektionen ausgeschüttet, in denen die Antigenspezifität der B-Zellen und der ausgeschütteten Antikörper noch sub-optimal ist. Nach wenigen Tagen, in denen die B-Zellen reifen und immer spezifischer Rezeptoren und Antikörper produzieren, switched die Produktion von Pentamer organisierten IgMs auf Dimer organisiert IgGs: Abbildung 1: Aufbau eines IgG Antikörpers (Quelle: http://www.biokurs.de/skripten/12/bs12-54.htm) 60 Experiment I2 Ein IgG-Molekül besteht aus vier Untereinheiten, wobei jeweils zwei identisch zueinander sind: zwei schwere (H) Ketten und zwei leichte (L), die über kovalente Disulfidbrücken miteinander verbunden sind (siehe Abbildung 1). Die variablen Regionen der leichten und schweren Ketten vermitteln die spezifische Antigenbindung. Antikörper werden aufgrund ihrer sehr hohen Antigenspezifität in vielen biochemischen und molekularbiologischen Verfahren eingesetzt, wie zum Beispiel Western Blot. Hier werden zunächst Proteine gelelektrophoretisch aufgetrennt und anschließend auf eine Membran transferiert, auf der sie anschließend über spezifische Antikörper detektiert werden können. Die Antikörperdetektion findet in zwei Schritten statt, daher spricht man hier auch von einer indirekten Markierung. Zunächst wird die Membran mit einem Primärantikörper inkubiert, der spezifisch für das zu detektierende Protein ist. Anschließend wird mit einem zweiten Antikörper inkubiert, der spezifisch an den Fc-Teil des Erstantikörpers bindet und an eine Peroxidase gekoppelt ist, die nach Zugabe eines Substrats dieses in ein lumineszierendes Produkt katalysiert. Diese Lumineszenz wird mit einem Röntgenfilm dokumentiert und markiert also die Stelle auf der Membran, auf die das gesuchte Protein transferiert worden ist. Umgekehrt ist das Vorhandensein Pathogen-spezifischer Antikörper im Serum von Patienten aber auch ein Zeichen für einen vorherigen oder aktuellen Kontakt (= Infektion oder Immunisierung) mit dem entsprechenden Pathogen. Daher kann hier der Nachweis spezifischer Antikörper zur Diagnose einer vergangenen oder aktuellen Infektion mit einem bestimmten Krankheitserreger genutzt werden. Diagnostische Western Blot Analysen waren z.B. bis zur Einführung PCR-basierender Nachweismethoden das Mittel der Wahl zur Diagnose einer Infektion mit dem humanen Immundefizienzvirus (HIV). Bei einem solchen sogenannten diagnostischen Western Blot dient Serum oder Plasma eines Patienten als Primärantikörper. Das Serum oder Plasma enthält aber eine heterogene Mischung aus verschiedenen Antikörpern, die spezifisch für diverse Antigene sind. Inkubiert man diese Antikörpermischung auf einer Membran, auf der verschiedene Proteine des entsprechenden Krankheitseregers immobilisiert sind, lassen sich so nach Wegwaschen der nicht an die auf dieser Membran immobilisierten Proteine bindenden unspezifischen Antikörper und folgender Inkubation des Blots mit einem Zweitantikörper spezifisch für humane Immunglobuline diejenigen Antikörper des Patienten nachweisen, die spezifisch für den gesuchten Erreger sind. Im Rahmen dieses Praktikums soll mittels diagnostischer Western-Blot Analysen nachgewiesen werden, welche „Patienten“ Kontakt mit Masernviren, dem hochkontagiösen Erreger der Masern, hatten. Dazu sollen Masern-spezifische Antikörper in „Patienten“-Seren nachgewiesen werden. Masernviren bestehen aus pleomorphen, umhüllten Partikeln, die ein bis mehrere Ribonukleoproteinkomplexe (RNP) enthalten können (siehe Abbildung 2). Abbildung 2: Aufbau eines Masernvirions (nach Flint et al., 2001) Ein solcher Komplex besteht aus dem viralen Genom und drei weiteren Proteinen: Nukleokapsid (N, 60kDa), Large-Protein (L (Polymerase), 200kDa) und Phosphoprotein (P, 61 Experiment I2 72kDa). Das Matrix-Protein (M-Protein, 37kDa) ist an die Membran der Virushülle angelagert, die aus der Wirtszellmembran enstammt und integral das Fusionsprotein (F, 60kDa) und Hämagglutinin (H, 79kDa), zwei Glykoroteine, enthält. Die Antikörper-vermittelte anti-Masernvirus Immunantwort richtet sich hauptsächlich gegen das Hämagglutinin sowie das Nukleokapsidprotein. Western-Blot (der Versuch beginnt für die Studenten ab Punkt 4. Immunodetektion!) 1. Proben für den Western-Blot 6 Für den Western-Blot werden 2 x 10 Vero-αHis-Zellen in 10 cm Schalen mit dem Masernvirus MVNSe-GFP(N) (MOI = 0,1) infiziert und nach 48 h mit 10 ml PBS für 5 min auf Eis gewaschen und durch Zugabe von 2 ml RIPA-Lysispuffer für 10 Minuten auf Eis inkubiert. Danach werden die Zellen ein 1,5 ml Eppendorf-Reaktionsgefäß (Eppendorf AG) überführt und mindestens 10 Minuten bei 13.000 rpm in einer Tischzentrifuge bei 4°C zentrifugiert und bei -80°C eingefroren. Vor dem Auftragen werden die Proben mit 5 x SDSProbenpuffer versetzt und für 5 Min. bei 95°C denaturiert. Als Negativkontrolle dienen uninfizierte Vero-αHis-Zellen. RIPA-Lysispuffer: 10 mM Tris pH 7,4 1% Natriumdeoxycholat 150 mM NaCl 1% Triton X-100 2. SDS-Polyacrylamid-Gelelektrophorese Die Auftrennung von Proteinen erfolgt mit SDS-Polyacrylamid-Gelen. Die Gele bestehen aus einem unteren 10%igem Polyacrylamid-Trenngel und einem oberen 6%igen Sammelgel. Die Lösung für ein 10%iges Trenngel setzt sich aus 19,8 ml A. bidest., 16,7 ml einer 30%igen Bisacrylamid-Mischung (Protogel®, national diagnostic), 12,5 ml einer 1,5 M TrisHCl pH 8,8 und 0,5 ml einer 10%igen SDS-Lösung zusammen. Danach werden 500 µl einer 10%igen Ammoniumpersulfat-Lösung und 20 µl TEMED (Bio-Rad Laboratories GmbH) zugesetzt. Das Trenngel wird bis auf eine Höhe von zirka einem Zentimeter unterhalb des Taschenformers gegossen. Zur Ausbildung einer planen Oberfläche wird die Gellösung mit 0,2 bis 0,5 ml A. bidest. überschichtet. Das 6%ige Sammelgel setzt sich aus 3,6 ml A. bidest., 3,4 ml einer 30%igen Bisacrylamid-Mischung (Protogel®, national diagnostic), 2,5 ml einer 1,0 M Tris-HCl Lösung mit pH 6,8, 0,2 ml einer 10%igen SDS-Lösung, 0,2 ml einer 10%igen Ammoniumpersulfat-Lösung und 20 µl TEMED zusammen. Nach der Polymerisation des Trenngels wurde das A. bidest. abgegossen, mit der Sammelgel-Lösung überschichtet und der Taschenformer (Bio-Rad Laboratories GmbH) eingesetzt. Das polymerisierte Gel wird in eine Doppelelektrophoresekammer (Bio-Rad Laboratories GmbH) 62 Experiment I2 eingespannt und die Kammer mit 1 x WB-Laufpuffer aufgefüllt. 130µl µl der vorbereiteten Proben (siehe Fehler! Verweisquelle konnte nicht gefunden werden.) werden mit speziellen Gelloader-Spitzen (ART GEL, Molecular Bioproducts Inc.) in die Geltaschen gefüllt. Die Elektrophorese erfolgt bei 80 -120 Volt für zirka 1,5 Stunden. 10 x WB-Laufpuffer: 1 x WB-Laufpuffer: 25 mM Tris-Base 1 Teil 10 x WB-Laufpuffer 192 mM Glycin 9 Teile A. bidest. 1% SDS 3. Elektrotransfer von Proteinen Nach der Gelelektrophorese (siehe Fehler! Verweisquelle konnte nicht gefunden werden. , Fehler! Verweisquelle konnte nicht gefunden werden.) der Proteinproben findet ein Transfer der Proteine aus dem SDS-Polyacrylamid-Gel auf eine PVDF-Membran nach der Semi-Dry-Transfer-Methode von KYHSE-ANDERSEN (KYHSE-ANDERSEN 1984) statt. Für den Transfer wird eine Graphitblotapparatur (Fastblot B33/B34 Whatman Biometra, Biometra GmbH i.L.) verwendet. Nach der elektrophoretischen Trennung der Proteine wird das Gel abgemessen und 10 Whatman-Filterpapiere (3MM-Papier, Whatman Biometra, Biometra GmbH i.L.) und eine PVDF-Membran (Bio-Rad Laboratories GmbH) auf die Größe des Gels zurechtgeschnitten. Die PVDF-Membran wird zur Aktivierung in 100%iger Methanol-Lösung getränkt. Die Whatman-Filterpapiere und die PVDF-Membran werden dann anschließend in Transferpuffer getränkt. Der Aufbau der Blotapparatur erfolgt (von der Kathode zur Anode) nach folgendem Schema: Anode 5 x Whatman-Filterpapier PVDF-Membran SDS-Polyacrylamid-Gel 5 x Whatman-Filterpapier Kathode Der elektrophoretische Transfer erfolgt bei 15 V für 60 Min und wird durch Anfärbung der PVDF-Membran mit Ponceau S-Lösung überprüft. Das Ponceau S kann durch Waschen der Membran mit PBS wieder entfernt werden. Das Gel wird anschließend in zirka 0,5 cm breite Streifen geschnitten. 5 x Salze für Transferpuffer: 25 mM Tris-Base 1 x Transferpuffer: 20% 5 x Salze 20% Methanol 192 mM Glycin 63 60% A. bidest. Experiment I2 2,63 mM Ponceau S, Na- Ponceau S-Lösung: 1% Essigsäure Salz 4. Immunodetektion von Proteinen auf PVDF-Membranen Blockieren (1h): Zur Blockierung unspezifischer Bindungsstellen erfolgt eine Inkubation der PVDFMembranen (2x Masern-Lysat, 2x Negativkontrolle) für eine Stunde bei RT in TBS + 0,5% Tween (TBS-T) mit 5% Milchpulver (MP). Waschen (30min): Es folgen sechs Waschschritte mit TBS-T (jeweils 5 Minuten bei RT) Seruminkubation (2h): Anschließend werden zwei Rhesus-Affen Seren je in der Verdünnung 1:500, in TBS-T mit 5% MP aufgenommen und in einer Schale auf die PVDF-Membranen gegeben: Streifen 1 Streifen 2 Masern-Lysat Streifen 3 Streifen 4 Negativ-Lysat Serum Masernpositiv Serum Masernpositiv Serum Masernnegativ Serum Masernnegativ Der Ansatz wird auf einer Schwenkapparatur (WT 16, Whatman Biometra, Biometra GmbH i.L.) für 2h bei RT inkubiert. Waschen (30min): Es folgen sechs Waschschritte mit TBS-T (jeweils 5 Minuten bei RT) Zweitantikörperinkubation (1h): Anschließend folgt die Inkubation mit dem zweiten Antikörper, ein HRPO-gekoppelter AntiRhesus Antikörper aus der Ziege (Rockland), für 60 Minuten bei RT in einer 1: 10.000 Verdünnung in PBS +1% BSA. Waschen (30min): Es folgen sechs Waschschritte mit TBS-T (jeweils 5 Minuten bei RT) Detektion: Die Detektion der Proteine erfolgt mittels Chemolumineszenz (Lumilight™, Roche Diagnostics GmbH) nach Angaben des Herstellers. Hierfür wird die mit ChemolumineszenzLösung bedeckte Membran in eine Plastikfolie gegeben und ein Röntgenfilm (BioMax MR-1, Kodak GmbH) für 5 bis 60 Sekunden aufgelegt. Die Entwicklung des Films erfolgt mit Hilfe eines Röntgenentwicklergerätes (Curix 60, Type 9462/106, Agfa Deutschland Vertriebsgesellschaft mbH & Cie.KG). Da der Proteinstandard („prestained protein marker“, NEB BioLabs) nicht mit Peroxidase markiert ist, werden dessen Proteine nicht auf dem Film dargestellt. Durch erneutes Auflegen des Films auf die PVDF-Membran, auf der der Proteinstandard erkennbar ist, ist die Bestimmung der Größe der Proteine möglich. 64 Experiment I3 -1. Einleitung Experiment Immunologie I3: Charakterisierung der adaptiven Immunantwort gegen Mycobacterium tuberculosis Dr. Max Bastian (4/3) Inhalt: 1. Einleitung 2. Experimente 3. Protokolle 4. Pipettierschemata 5. weiterführende Literatur 65 Experiment I3 -1. Einleitung 1. Einleitung Mycobacterium tuberculosis ist der Erreger der Tuberkulose des Menschen. Die Tuberkulose ist weltweit verbreitet. Nach WHO Schätzungen ist ca. ein Drittel der Weltbevölkerung, d.h. ca. 2 Milliarden Menschen mit dem Erreger infiziert. Insbesondere nach Koinfektionen mit HIV kommt es zu schweren, oft letalen Krankheitsverläufen. Daher ist von der Seuche insbesondere das südliche Afrika, aber auch Asien und Russland betroffen. Während in westlichen Industrieländern die Inzidenz in den vergangenen fünfzig Jahren nicht zuletzt aufgrund der Entwicklung antimycobakterieller Antibiotika stetig gesunken ist, steigen die Fallzahlen in den genannten Regionen. Besondere Sorge bereitet dabei, das massive Auftreten von multiresistenten Bakterienstämmen, die teilweise durch keines der gebräuchlichen Antibiotika mehr zu therapieren sind. Angesichts dieser Entwicklungen wäre ein sicherer, effizienter Impfstoff mehr denn je erforderlich. Ein solcher Impfstoff steht allerdings nicht zur Verfügung. Bacille-Calmette-Guerin (BCG), der 1921 entwickelte, attenuierte Lebendimpfstoff, schützt zwar vor besonders schweren, systemischen Tuberkuloseformen im Kindesalter. Er schützt aber nicht gegen die epidemiologisch bedeutsamste Lungenmanifestation. Die Tatsache, dass 130 Jahre nach der Erstbeschreibung des Erregers durch Robert Koch nach wie vor kein Impfstoff zur Verfügung steht, hängt letztlich damit zusammen, dass sich der Erreger im Laufe einer langen Coevolution an seinen Wirt, den Menschen, hervorragend angepasst hat und meisterhaft gelernt hat, die verschiedenen Effektormechanismen des Immunsystems zu umgehen. Abb 1: Die Immunantwort gegen Mtb Mykobakterien gelangen über Aerosole in die Lunge, werden von Alveolarmakrophagen aufgenommen und verursachen eine lokale Entzündung. Antigenspezifische T Zellen koordinieren die lokale Reaktion. Es kommt zur Bildung von Granulomen. Im inneren, verkäsenden Bereich sind die Bakterien den Effektorfunktionen des Immunsystems entzogen. Sie können dort über lange Zeiträume persistieren. Bei einer Schwächung des Immunsystems bricht die Struktur auf, infektiöse Aerosole werden abgehustet. Der Infektionszyklus ist geschlossen. Mykobakterien sind ca. 2-3 µm große, säurefeste Stäbchen, die sich durch eine besonders wachs- und lipidreiche Zellwand auszeichnen. In der Regel werden sie aerogen übertragen. Sie werden inhaliert und infizieren zunächst Alveolarmakrophagen. Obwohl diese über ein reiches Armamentarium verfügen, um Bakterien und Pilzsporen zu inaktivieren, gelingt es den Mykobakterien, die Makrophagen umzuprogrammieren. Sie gelangen ins Phagosom und persistieren dort, wobei sie die Ansäuerung und Reifung des Phagosoms aktiv 66 Experiment I3 -1. Einleitung inhibieren. Gleichzeitig provozieren sie eine lokale Entzündungsreaktion, im Verlauf derer Granulozyten und Monozyten in das infizierte Gewebe einwandern. Dendritische Zellen verbringen antigenes Material, bzw. lebende Bakterien in den lokalen Lymphknoten, wo es zur Aktivierung antigenspezifischer T Zellen kommt. Diese T Zellen wandern zurück ins infizierte Gewebe und koordinieren dort die lokale Entzündungsreaktion. Das Schlüsselzytokin ist hierbei Interferon-γ. Unter anderem durch die Wirkung dieses prototypischen T Helfer 1 Zytokines kommt es zur Bildung von Granulomen, hochdynamischen, multizellulären Strukturen, die einen sehr typischen Aufbau haben: In der Mitte befindet sich ein stark hypoxischer Bereich mit zahlreichen, nekrotischen Zellen. Dieser zentrale Bereich entwickelt im Lauf der Zeit eine amorphe, käseartige Konsistenz. Der Bereich enthält in diesem Stadium keine lebende Zellen mehr, wohl aber lebende Mykobakterien, die in dieser sauerstoffarmen Umgebung in eine latente Phase übertreten. Um den inneren Bereich findet sich ein dichter Ring an mehrkernigen, sogenannten Langerhans’schen Riesenzellen, eine spezialisierte Form von Makrophagen. Dieser innere Zellring wird von einem Wall an Lymphozyten und Gewebsmakrophagen umgeben. Obwohl derartige Granulome über lange Zeiträume im Gewebe liegen und sich in Ort und Größe kaum zu verändern scheinen, unterliegt insbesondere der äußere Zellwall einem ständigen Wandel. Lymphozyten und Monozyten wandern ein, migrieren innerhalb des Gewebes, werden apoptisch, müssen ersetzt werden etc. Ständig gelangen dabei aktive Mykobakterien aus dem inneren, verkäsenden Bereich in die peripheren, zellulären Bereiche und müssen dort durch zytotoxische T Zellen oder aktivierte Makrophagen abgetötet werden. Insofern ist die Eindämmung der Bakterien im Inneren des Granuloms ein äußerst aktiver Prozess. Ist die Dynamik des Granuloms gestört –sei es durch eine virus- oder altersbedingte Immunsuppression (z.B. HIV) oder durch andere Faktoren (z.B. Alkoholismus)– kommt es zu einem Aufbrechen der Struktur. Insbesondere in der Lunge gelangen dann lebende Mykobakterien in die luftführenden Atemwege, sie werden abgehustet und werden in Form von bakterientragenden Aerosolen von einem nächsten Individuum eingeatmet. Aus dem engen Zusammenhang zwischen einer HIV-infektion und dem Auftreten einer klinischen Tuberkulose lässt sich ermessen, welche wichtige Rolle insbesondere CD4positive T Helferzellen bei der Immunabwehr gegen M.tuberculosis und eben vor allem bei der Aufrechterhaltung der Granulomstruktur innehaben. Allgemein wird die Bildung von Granulomen in erster Linie als Schutzmechanismus des Immunsystems verstanden, und in der Tat ist die schwerste, klinische Form der Tuberkulose, die Miliartuberkulose –vulgo „die gallopierende Schwindsucht“– durch das Fehlen granulomatöser Strukturen gekennzeichnet. Trotzdem nutzt der Erreger diese Struktur zur Aufrechterhaltung der Infektion. Er ist im inneren, verkäsenden Bereich den Effektormechanismen des Immunsystems weitgehend entzogen. Daher nimmt das Granulom und die zur Bildung des Granuloms erforderlichen T Zellen eine Schlüsselstellung in der Infektionsstrategie des Erregers ein. In diesem Sinne wurde kürzlich gezeigt, dass immundominante T Zellepitope von M.tuberculosis eine geringere Sequenzvariabilität aufweisen als beispielsweise essentielle Strukturproteine oder Enzyme. Das deutet daraufhin, dass Mykobakterien nicht nur effiziente Evasionsmechanismen entwickelt haben, um der Immunantwort zu entgehen, sondern dass 67 Experiment I3 -1. Einleitung sie die T zellvermittelte Immunantwort nachgerade für die Schaffung ihrer Persistenznische benötigen. Bislang waren die Strategien zur Entwicklung eines neuen Tuberkuloseimpfstoffes in erster Linie durch die Vorstellung geprägt, man müsse möglichst starke, Interferon-γ sezernierende, antigenspezifische T Zellen induzieren. Mittlerweile zeichnet sich ab, dass diese Herangehensweise möglicherweise zu kurz greift, denn auch die natürliche Infektion induziert starke Interferon-γ dominierte TH1-Antworten. Trotzdem führt genau dies eben nicht zur Elimination des Erregers und mithin zu einem Unterbruch der Infektionskette, sodern lediglich zur jahrzehntelangen Persistenz des Erregers. Ein Impfstoff, der das Potential hätte, die Tuberkulosepandemie zu kupieren, darf daher nicht einfach nur die natürliche Infektion nachahmen, er müsste „better than nature“ sein. Im Moment gibt es keine klare Vorstellung, wie das zu erreichen wäre. Obwohl seit der Erstbeschreibung des Erregers die biomedizinische Forschung eine ganze Reihe von Quantensprüngen vollzogen hat, sind die hochkomplexen Zusammenhänge zwischen Mykobakterien und dem menschlichen Immunsystem nur in Ansätzen verstanden. Es wird noch viel Detailarbeit in relevanten Tiermodellen erfordern, insbesondere die Zusammenhänge, die zur Ausprägung des verkäsenden Granulomes führen, besser zu verstehen. Umgekehrt ist dies die Voraussetzung für die Entwicklung von Impfstrategien, die die Granulombildung verhindern und damit dem Erreger seine Persistenznische entziehen. In diesem Zusammenhang untersuchen wir in unserer Arbeitsgruppe mykobakterienspezifische T Zellantworten in sensibilisierten Meerschweinchen. Ziel ist zum einen, mykobakterielle Antigene zu identifizieren, die von den Mykobakterien verwendet werden, um TH1 Antworten zu induzieren, die –wie oben ausgeführt– zur Bildung von Granulomen beitragen. Zudem soll das Meerschweinchenmodell verwendet werden, um anschließend die funktionelle Rolle dieser T Zellen untersuchen zu können. Ziel unseres Praktikumsteils ist es, einige der unserer Arbeit zugrundeliegenden Konzepte vorzustellen und einige Experimente durchzuführen, die wir in diesem Zusammenhang verwenden. 68 Experiment I3 -2. Experimente 2. Experimente Experiment 1 – T Zellassay: Es soll die T Zellantwort auf mykobakterielle Antigene untersucht werden. Im Gegensatz zu B Lymphozyten, die ihre Antigene in löslicher Form binden können, erkennen T Zellen ihr Antigen immer im Zusammenhang mit einem antigenpräsentierenden Molekül auf der Zelloberfläche von anderen Zellen. Im Fall von CD8-positiven T Zellen sind dies MHC-I Moleküle, die in erster Linie antigenes Material aus dem Zytosol binden und präsentieren. Im Fall von CD4-positiven T Helferzellen sind dies MHC-II Moleküle, die ihrerseits in erster Linie phagozytiertes, exogenes Material aus dem endosomalen Kompartment binden und präsentieren. Erkennen T Zellen „ihr spezifisches Antigen“ reagieren sie je nach Funktionszustand mit Proliferation oder Freisetzung von Effektormolekülen, wie z.B. Zytokinen oder zytotoxischen Molekülen. In unserem Experiment untersuchen wir „zentrale T Gedächniszellen“. Diese Zellen beginnen nach einer Antigenstimulation heftig zu proliferieren. Als Meßparameter verwenden wir im vorliegenden Experiment daher die Zellproliferation. Dazu gewinnen wir Blut von BCG-immunisierten Meerschweinchen. Aus dem Blut isolieren wir mittels Ficoll-Dichtegradientenzentrifugation PBMCs. Das Akronym steht für Peripheral Blood Mononuclear Cells. Diese Zellfraktion enthält damit –in Abgrenzung zu polymorphkernigen Granulozyten und kernlosen Erythrozyten– alle mononukleären Zellen aus dem peripheren Blut, das sind in erster Linie B- und T-lymphozyten, Monozyten und in geringem Maße auch dendritsche Zellen. Damit sind in der Zellfraktion alle Zellen enthalten, die wir für das Experiment benötigen: Die T Zellen stellen die Population dar, die wir untersuchen wollen, während Monozyten und B Zellen sehr effizient Antigene aufnehmen und auf ihren MHC-Molekülen präsentieren können. Um die Proliferation der T Zellen messen zu können, färben wir alle Zellen mit CFSE (Carboxy-Fluorescin-Succimidyl-Ester), einem grünen Fluoreszensfarbstoff, der die Zellmembran permeiert und über eine Reaktion mit freien Aminogruppen kovalent an membranständige sowie zytosolische Moleküle gebunden wird. Während der Mitose verteilt sich der Fluoreszenzfarbstoff zu gleichen Teilen auf die Tochterzellen. Die daraus resultierende Abnahme der Fluoreszenz kann im Durchflußzytometer quantifiziert werden und ist ein Maß für die Proliferationsaktivität der Zellen. Zur Untersuchung der antigenspezifischen Immunantwort nehmen wir die aufgereinigten und gefärbten T Lymphozyten in einer sterilen Mikrotiterplatte in Kultur und geben mykobakterielle Antigene hinzu. In unserem Fall verwenden wir a) ein Gesamtextrakt von BCG – dieses Material wird hergestellt, indem die Bakterien mit Ultraschall behandelt werden und anschließend nichtlösliches Material abzentrifugiert wird; b) Tuberkulin – hierbei handelt es sich um wasserlösliches, hitzinaktiviertes Kulturmaterial, das durch eine Proteinfällung aufkonzentriert wurde; sowie c) einen Lipidextrakt von BCG – dieses Material 69 Experiment I3 -2. Experimente wird gewonnen, indem Kulturmaterial eingetrocknet wird und anschließend Lipide und Lipopeptide mit einem Chloroform:Methanol-gemisch herausgelöst werden. Als Positivkontrolle verwenden wir PHA, Phytohämagglutinin, ein unspezifisches Mitogen. Als Negativkontrolle verwenden wir einen Ansatz, der nur Medium und kein Antigen enthält, und zum anderen ein Lysat von Salmonellen, einem Erreger, dem die Tiere bislang nicht ausgesetzt sein sollten. Wir geben die Antigene zu den Zellen hinzu und inkubieren die Zellen für vier Tage bei 37°C und 5% CO2. Nach dieser Inkubationsphase untersuchen wir die Proliferation im Durchflußzytometer. Zur Vorbereitung des Experimentes sind die folgenden Protokolle: I – Ficoll-aufreinigung, II – CFSE-Färbung sowie III – T Zellassay, sowie das Pipettierschema I – Proliferationsassay zu beachten. Experiment 2 – Antigenanalyse: Um zu verstehen, womit wir die T-Zellen stimuliert haben, wollen wir in einem zweiten Schritt die verwendeten Antigenpräparationen mittels SDS-PAGE und anschließendem Westernblot, bzw. mittels Dünnschichtchromatographie untersuchen. Dazu gießen wir entsprechend der Anweisung 12%-ige SDS-Gele und beladen je zwei Gele mit a) Gesamtextrakt von BCG, b) Tuberkulin, c) Lipidextrakt von BCG und d) dem Gesamtlysat von Salmonella typhimurium. Anschließend lassen wir die Antigene im elektrischen Feld wandern. Sobald die Migrationsfront den unteren Gelrand erreicht hat, unterbrechen wir den Lauf. Jeweils ein Gel wird mit Coomassie Brillantblue angefärbt. Das andere wird auf eine Nitrocellulosemembran geblotted. Die unspezifische Proteinbindung der Nitrocellulose wird abgeblockt. Immunogene Antigene werden anschließend mittels Westernblot detektiert. Als Quelle von Primärantikörpern verwenden wir Serum von BCG-immunisierten Meerschweinchen. Gebundene Antikörper werden anschließend mit einem Peroxidase-gekoppelten Sekundärantikörper und einem entsprechenden Farbsubstrat sichtbar gemacht. Parallel dazu untersuchen wir alle Antigene mittels Dünnschichtchromatographie. Hierfür verwenden wir Silikatplatten, die mit einem Fluoreszenzfarbstoff versetzt sind. Wir tragen je 5 µl Antigenlösung auf und geben die Platte in eine Migrationskammer, deren Boden bis zu einem Zentimeter mit einem Chloroform:Methanol gemisch bedeckt ist. Sobald die Migrationsfront ca. 1 cm vom Plattenende entfernt ist, unterbrechen wir den Lauf. Wir trocknen die Platte unter der chemischen Abzugshaube und untersuchen das Punktmuster unter der UV-lampe. Zur Vorbereitung dieser Experimente sind die Protokolle IV – SDS-PAGE und Westernblot und V – Dünnschichtchromatographie, sowie die Pipettierschemata II – Antigenanalyse und III – Dünnschichtchromatographie zu beachten. 70 Experiment I3 -2. Experimente Experiment 3 – Infektionsversuch: Im dritten Experiment wollen wir untersuchen, wie Mykobakterien in Makrophagen eindringen. Dazu färben wir BCG mit einem lipophilen Fluoreszenzfarbstoff. Wir präinkubieren die Bakterien für eine Stunde mit Serum von BCG immunisierten Meerschweinchen, das mykobakterienspezifische Antikörper enthält. Anschließend geben wir präinkubierte, bzw. nichtpräinkubierte Bakterien zu Meerschweinchenmakrophagen. Nach Übernachtinkubation waschen wir nicht eingedrungene Bakterien ab, fixieren die Zellen mit 4% Formaldehyd, permeabilisieren die Zellen und inkubieren sie mit einem Antikörper das endosomale Kompartment anfärbt. Anschließend untersuchen wir die Makrophagen mittels Fluoreszenzmikroskopie. Zur Vorbereitung dieses Experimentes sind die Protokolle VI – Bakterienfärbung und VII – intrazelluläre Antikörperfärbung, sowie das Pipettierschema IV –Fluoreszenzmikroskopie zu beachten. Zeitplan: Montag – Tag 1: – – – Ficoll Dichtegradientenzentrifugation CFSE Färbung Ansetzen des Proliferationsassays – Bakterienfärbung und Infektion von Makrophagen Dienstag – Tag 2: – SDS-PAGE – Dünnschichtchromatographie – intrazelluläre Färbung von Makrophagen – Fluoreszenzmikroskopie Donnerstag – Tag 3: – Durchflußzytometrische Analyse der T Zellproliferation – Entwicklung des Westernblots – Abschlußbesprechung 71 Experiment I3 -3. Protokollee 3. Protokolle Protokoll I – Ficoll gradient PBMC isolation 1. Material: 1. Anti-coagulant: e.g. PBS including 10 mM Sodium Citrate 2. Ficoll Solution: e.g. Ficoll-PaqueTM Plus GE Healthcare (density of 1.077 g/ ml; GE Healthcare 17-1440-03) 3. Lysing solution Ammonium chloride (NH4Cl) Potassium bicarbonate (KHCO3) EDTA H2O dist 0.15M 10mM 0.1mM 8.29 g 1g 37 mg q.s. 1 l filter with 0.45µm filter, store at RT. + 4. Wash buffer: PBS/ 2% FCS 2. Method: 1. Equilibrate 15 ml of Ficoll in a 50 ml Falcon tube to room temperature (RT). 2. Carefully overlay 15 ml Ficoll with up to 30 ml of uncoagulated whole blood mixed 1:1 with PBS 3. Centrifuge 30 min at RT at 500xg, switch off break 4. Carefully aspirate buffy coat from the interphase 5. Centrifuge for 10 min at 1000xg 6. Discard supernatant, resuspend cell pellett in 15 ml wash buffer 7. Centrifuge for 10 min at 200xg (The slow centrifugation step is required to remove platelets) 8. OPTIONAL: Discard supernatant; if pellett contains erythrocytes proceed with ammonium chloride lysis, otherwise go to step 11: 9. Resuspend pellett in 2 ml Lysing solution 10. Allow the red cells to lyse for ~ 10 min at RT, do not extend this step too much because PBMCs might suffer from prolonged incubation 11. Add 13 ml wash buffer, centrifuge for 10 min at 400xg 12. Resuspend pellett in appropriate amount of medium and count. 72 Experiment I3 -3. Protokollee Protokoll II – CFSE staining Principle: 5(6)-carboxyfluorescin diacetate N succinimidyl ester (CFSE) diffuses into cells, intracellular esterases remove the acetate residues. This renders the dye fluorecent and inable to leave the cell. It associates with aminogroups and distributes evenly during mitosis. 1. Material: 1. 37°C waterbath 2. PBS 3. PBS/ 2% wash-serum 4. CFSE stock (Alexis, HLX-610-030-M025, box 48 –70°C) is aliquoted in 10 µl at 5mM in DMSO, working aliquots contain 10 µl of 0.5 mM CFSE in DMSO working aliquots are finally diluted 1:250 into PBS (without serum) 2. Method: 7 1. wash PBMC in PBS, resuspend thoroughly to 1x10 ¢/ ml 2. add CFSE to a final concentration of 2.0 µM 3. mix well by vortexing 4. incubate ~ 10’ at 37°C in a preheated waterbath 5. fill tube completely with PBS/ 2% wash-serum 6. wash cells twice with PBS/ 2% wash-serum, resuspend in complete medium Note: Staining at 0.5-2.0 µM CFSE allows for additional staining with PE-labeled antibodies. Still, additional Fl2-%Fl1 compensation is necessary! 73 Experiment I3 -3. Protokollee Protokoll III – T cell assay 1. Material: 1 1. T cell medium (RPMI 1640; 10%FCS ; Pen/Strep; L-Glu) 2. 96 rd bottom cell culture plates 3. flow cytometer 4. PHA, e.g. Sigma-AldrichL1668-5MG 5. antigens 2. Method: 5 1. Wash and adjust Ficoll-purified, CFSE stained PBMCs to 2 x 10 cells/ml 2. Put the appropriate antigen into the corresponding well (e.g. dilute the respective antigen to a standard concentration of 20 µg/ml and add 50 µl to the appropriate well) 5 3. Add 1x10 PBMCs in 50 µl to the corresponding well (final volume 100 µl) 4. Incubate cells for 5 days at 37°C, 5% CO2, moist saturated 5. Harvest cells and analyze for CFSE dilution by flowcytometry Notes: if only few T cells are available, reduce set up from triplicates to duplicates, reduce cell numbers while maintaining proportions ; additional staining for surface markers is possible 1 Alternatively, use autologous guinea pig serum. 74 Experiment I3 -3. Protokollee Protokoll IV – SDS PAGE and Westernblot I. SDS-PAGE 1. Material: 4 x lower buffer: 1.5 M Tris/ HCl (90.85 g/ 500 ml) 0.4% SDS (2 g/ 500 ml) adjust to pH 8.8 Upper buffer: 30 % Acrylamide/ Bisacrylamide (37.5/ 1); Boehringer Mannheim 10 % APS (Ammonium Persulfate), 1 g APS ad 10 ml H2O TEMED reagent, BioRad H2O saturated Isobutanol 5 x Electrode buffer: 15.1 g Tris base 94.0 g Glycine 50 ml 10% SDS add 1000 ml H2O, adjust to pH 8.3 2x loading buffer: 1.7 ml Upper buffer 2.0 ml Glycerol 4.5 ml 10% SDS 1.0 ml β-Mercaptoethanol 0.8 ml Bromphenol blue Coomassie stain: 0.5 g Coomassie Brillant Blue R250 10 ml Acetic Acid 45 ml Methanol 45 ml H2O Destain: 10 ml Acetic Acid 90 ml H2O Gel chamber, glass plates, spacers power supply 0.5 M Tris/ HCl (30.25 g/ 500 ml) 0.4% SDS (2g/ 500 ml) adjust to pH 6.8 2. Method: 1. Clean and assemble the glass plates into the running device, it’s essential to wear gloves throughout the whole casting process 2. Mix the Acrylamide Polymer according to the following scheme: 10% SDS-gel H2O 4x lower buffer 30 % AC/BAC 10 % APS TEMED 2 gels 6.20 ml 3.75 ml 5.00 ml 150 µl 20 µl 3. Mix the components in the order shown. Polimerization will start as soon as the TEMED has been added. Carefully pour the solution in the gap between the glass plates. 4. Overlay the polymer with H2O-saturated isobutanol 5. Let the gel polymerize for 45‘ at RT 6. After polymerization pour off the overlay and rinse the gel with deionized H2O , remove any remaining water with a paper towel 7. Mix the stacking gel according to the following scheme: 75 Experiment I3 -3. Protokollee stacking gel H2O Upper buffer 30% AC/ BAC 10% APS TEMED 2 gels 3.00 ml 1.25 ml 0.625 ml 150 ml 20 ml 8. Mix the components in the order shown. Polimerization will start very rapidly as soon as the TEMED has been added. Pour the solution and immediately insert the comb into the gap between the glass plates. 9. Let the stacking gel polymerize for 30‘ at RT 10. Remove the comb and add the 1x running buffer into the upper and lower buffer reservoires, rinse the slots with a syringe and a needle Preparation and loading of the protein samples: 11. Dilute protein samples 1:2 in the 2x loading buffer and denature sample for 5‘ at 95°C 12. Centrifuge samples once at high speed to collect the solution at the bottom of the tube 13. Load samples unto the bottom of the slots (10-slot comb: one slot will hold ~ 20 ml), don’t forget the molecular weight marker (~ 5 ml) 14. Connect the running device to the power supply and run the gel with 70–100 V Coomassie stain: 15. Prepare Coomassie staining bath: Methanol 450 ml; Acetic Acid 75 ml; Coomassie Brillant Blue R250 1.25 g add H2O to 1 l 16. Stain the gel for 1-2 h in Comassie stain 17. Destain the gel in destaining solution o/n: Methanol 450 ml; Acetic Acid 75 ml add H2O to 1l II. Transfer of proteins to the nitrocellulose membrane 1. Material/ Reagents: Anode Solution 1: 0.3 M Tris/ HCl (3.63 g/ l) 20% Methanol (200 ml/ l) adjust to pH 10.4 Anode Solution 2: 0.025 M Tris/ HCl (0.3 g/ l) 20% Methanol (200 ml/ l) adjust to pH 10.4 Cathode Solution: 0.04 M e-Amino-n-capronic Acid (5.2 g/ l) 20% Methanol (200 ml/ l) adjust to pH 7.6 Nitrocellulose: Filter paper: Protran Nitrocellulose 0.45 mm, BA 85, Fa. Schleicher & Schuell Whatman No1, Standard; Fa. Merk 76 Experiment I3 -3. Protokollee 2. Method: 1. Prepare filter paper and nitrocellulose membrane in the size of the SDS gel 2. Assemble the blotting sandwich according to the following scheme: → → → → → 2 filter sheets soacked with Anode Solution 1 1 filter sheets soacked with Anode Solution 2 Nitrocellulose soacked with Anode Solution 2 SDS gel 3 filter sheets soacked with Cathode Solution 3. Squeeze out any air bubbles and place the sandwich into the blotting chamber 2 4. Apply a current of 0.8 mA/ cm 1 minigel ~ 47 mA; let it blot for ~ 2 hrs III. Developing the blotted nitrocellulose membrane 1. Material/ Reagents: Ponceau S Solution: 0.1% Ponceau S red in 5% Acetic Acid Blocking Buffer: 500 ml PBS 2g Skim Milk Powder adjust to pH 7.4 1st Antibody: 1:10 prediluted serum of BCG sensitized guinea pigs 2nd Antibody: goat a guinea pig IgG, horse-raddish peroxidase conjugated, 3-Amino-9-Ethylcarbazol (AEC) tablets; Sigma-Aldrich (A-6926) Substrate buffer: 0.05 M Sodium acetate adjust to pH 5.0 3% H2O2 solution (Sigma Product No. H-1009) 2. Method: 1. Transfer the blotted nitrocellulose membrane to a tray containing Ponceau S Solution, incubate it for ~ 5 min 2. when the bands are clearly visible you may take a picture; mark the bands of the molecular weight marker with a pencil 3. Pour of the Pon S and add ~ 15 ml blocking buffer 4. block the blotted membrane over night at RT 5. Pour of the blocking buffer and add 5 ml blocking buffer containing the 1st Ab ( 1:10 diluted guinea pig serum) 6. Incubate membrane for 1 hr at RT 7. Pour of the 1st Ab and wash the membrane with blocking buffer 3x for 10 min at RT 8. Pour of any remaining blocking buffer and add 5 ml blocking buffer containing the 2nd Ab HRP 1:5000 diluted in blocking buffer) (goat a gpIgG 9. Incubate it for 2 hrs at RT 10. Pour of the 2nd Ab and wash the membrane with blocking buffer 3x for 10 min at RT 11. Wash the membrane 1x with PBS for 10 min at RT 12. Wash the membrane 1x with substrate buffer for 5 min at RT 13. Prepare the substrate: Dissolve 1 Tablett of 3- Amino-9- Ethylcarbazol (AEC) in 3 ml DMF. Add 300 µl of resolved AEC stock solution to 5 ml of substrate buffer. Add 25 μl of fresh 3% hydrogen peroxide immediately prior to use. 14. ncubate the membrane until the specific bands appear in a dark violett colour 77 Experiment I3 -3. Protokollee Protokoll V – Thin Layer Chromatography 1. Material/ Reagents: Solvent: 180 ml Chloroform 75 ml Methanol 12 ml H2O ® Thin Layer Plates: TLC LuxPlate Silica gel 60 F254 (Merck Darmstadt; # 1.05802.0001) UV-lamp 2. Method: 1. Insert filter paper into a beaker glass and fill in chloroform:methanol:water (65:25:4) until the bottom is covered, let the chamber equilibrate for about 1 hour. 2. Prepare a thin layer plate and mark the start line about 1 cm above the lower rim of the plate with a soft pencil 3. Add the samples (max 5 µl per spot) and let the material dry completely; if more material should be added, let the solvent dry and apply additional 5 µl 4. Put the plate into the chamber such that the solvent does not reach the samples; close the lid of the chamber 5. Let the samples migrate by solvent drag for about 30-45 min until the front is about 1 cm below the upper rim of the plate. 6. Mark the migration front with a soft pencil and let the plate dry. 7. Analyze the plate under a UV-lamp. 8. Protokoll VI – PKH26 staining of BCG 1. Material/ Reagents: PKH-26 dye, Sigma-Aldrich (# PKH26GL) PBS/ 0.5% FCS FCS 2. Method: 1. resuspend BCG bacteria in 1 ml PBS. 2. centrifuge at 10.000 x g for 10 min, carefully aspirate supernatant 3. thouroughly resuspend bacterial pellet in 500 µl Diluent C 4. prepare a working dilution of PKH26 (add 1 µl PKH-26 dye to 500 µl Diluent C), mix well 5. add the working dilution to the resuspended cell pellet 6. incubate 5 min at toom temperature 7. add 500 µl FCS to stop the reaction 8. incubate 5 min at room temperature 9. centrifuge the bacteria as in step 2 10. resuspend the bacteria in cell culture medium 78 Experiment I3 -3. Protokollee Protokoll VII – intracellular macrophage staining 1. Material/ Reagents: 8-chamber slides PBS/ 0.5% FCS 4% formaldehyde permeabilization buffer: PBS/ 1% saponin/ 0.5 % FCS anti-LAMP FITC conjugate 2. Method: 1. Remove medium supernatant. 2. Wash preincubated, adherent macrophages twice with 250 µl PBS/ 0.5% FCS. 3. Carefully remove wash buffer and add 4% formaldehyde, incubate 5 min. 4. Wash with 250 µl PBS/ 0.5% FCS. 5. Add 250 µl permeabilization buffer. Incubate for 5 min at room temperature. FITC 6. Remove buffer, add 50 µl prediluted anti-LAMP buffer) conjugate (1: 500 in permeabilization 7. Incubate 30 min at 4°C 8. Wash twice with 250 µl permeabilization buffer. 9. Remove plastic wall 10. Add 20 µl glycerol and carefully add cover slip 11. Fix cover slip with nail polish ( dark magenta; Rossman # 45.7765) 12. Analyze under fluorescence microscope 79 Experiment I3 -4. Pipettierschemata 4. Pipettierschemata Blatt I – Proliferationsassay Zellen: 6 Zellen (x10 ) PBMCs MS I PBMCs MS II PBMCs MS III PBMCs MS IV Assay: 5 PBMCs: CFSE gefärbt (2.0 µM); 1x10 / well Medium: SF-IMDM; 5% autolog Serum Waschmedium: PBS/ 0.5% FCS Antigene: Arbeitskonzentration: PHA (0.5 mg/ ml) ……………………………………… 1 µg/ ml BCG-lysat (10 mg/ ml) ……………………………………… 10 µg/ ml PPD (300.000 IU/ ml) ………………………………… 30 IU/ ml …………………………… 10 µg/ ml LipidextraktBCG (10 mg/ml) Salmonellenlysat (10 mg/ ml) …………………………… 10 µg/ ml Plattenschema: 1-2 3-4 5-6 7-8 A Medium Medium Medium Medium B PHA PHA PHA PHA C BCG-lysat BCG-lysat BCG-lysat BCG-lysat D PPD PPD PPD PPD E total Lipid total Lipid total Lipid total Lipid F Salmonella-lysat Salmonella-lysat Salmonella-lysat Salmonella-lysat 9-10 11-12 G H MS I MS II MS III MS IV Zellen: ...................................................................................................................................... 5 6 15x (1x10 ¢ in 50 µl) = 1.5 x10 ¢ in 750 µl T¢ medium (10% autolog. Serum) ...................................................................................................................................... ...................................................................................................................................... ...................................................................................................................................... Antigene: ...................................................................................................................................... PHA: 10x (0.1 µg in 50 µl) = 1 µg (d.h. 2 µl der stock-lösung) in 500 µl ...................................................................................................................................... BCG-lysat: 10x (1µg in 50 µl) = 10 µg in 500 µl ...................................................................................................................................... ...................................................................................................................................... PPD: 10x (3 IU in 50 µl) = 10 µl working-stock (1:100 Vorverdünnung) in 500 µl ...................................................................................................................................... Total-lipid: 10x (1 µg in 50 µl) = 10 µg (d.i. 1 µl des working stock) in 500 µl ...................................................................................................................................... Salmonellen-lysat: 10x (1µg in 50 µl) = 10 µg in 500 µl ...................................................................................................................................... ...................................................................................................................................... ...................................................................................................................................... .................................................. 80 Experiment I3 -4. Pipettierschemata Blatt II – Antigenanalyse Probenaufbereitung: 1. nimm 50 µl BCG lysat und gib 50 µl 2x loading dye hinzu 2. nimm 20 µl PPDtub12 in 100 µl 2x loading dye auf 3. Trockne 200 µg total lipidBCG ein und nimm in 100 µl 2x loading dye auf lade jeweils 15 µl pro slot SDS-PAGE: 1. SDS-Gel mit non-reduzierendem SDS-Puffer: Coomassie / Blot o/n bei 30 mA; 2. Block: 2h in PBS/Tw20/2%MP geblockt; 3. 1st Antikörper: 1:10 sensibilisiertes Meerschweinchen-serum; 1h RT 4. 2nd Antikörper: 1:1000 anti-gpIgGHRP; 1 h RT 5. AEC-Entwicklung Gel-belegung: slot Probe 1 – 2 3 marker BCG lysat 4 – 5 PPD 6 – 7 TLBCG 8 marker 9 – 10 – ...................................................................................................................................... ...................................................................................................................................... ...................................................................................................................................... Blatt III – Dünnschichtchromatographie Probenaufbereitung: gib jeweils a. b. c. 5 µl BCG lysat 5 µl PPDtub12 5 µl LipidextraktBCG auf die entsprechend markierten Bereiche der TLC Platte TLC: 1. 2. 3. 4. stelle die Platte in eine mit Chloroform:Methanol:H2O (60:25:4) vorbereitete Migrationskammer lass die Antigene wandern, bis die Lauffront ca 1cm unterhalb des Plattenrandes steht trockne die Platte und untersuche das Bandenmuster unter der UV-lampe bestäube die Platte mit Ninhydrin oder Phosphomolybdän und erhitze die Platte vorsichtig mit dem Bunsenbrenner; scanne das Ergebnis Platten-belegung: slot Probe 1 – 2 BCG lysat 3 PPD 4 TLBCG .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... 81 Experiment I3 -4. Pipettierschemata Blatt IV – Fluoreszenzmikroskopie Zellen: 6 Zellen (x10 ) Mφ MS I Mφ MS II Bakterien: Nimm ein Aliquot BCG und färbe die Zellen entsprechend Protokoll VI inkubiere die Bakterien für 1 h ± Serum von BCG sensibilisierten Meerschweinchen Assay: Medium: SF-IMDM; 5% FCS Waschmedium: PBS/ 0.5% FCS 1. 2. 3. 4. 5 Gib 1x10 Mφ in 200 µl Kulturmedium Gib 0, 5, 10, 50 µl gefärbte Bakteriensuspension hinzu Inkubiere die Zellen o/n bei 37°C 5% CO2 Wasche und färbe die Zellen wie im Protokoll VII beschrieben Platten-belegung: slot – serum + serum 1 – – 2 5 µl 5 µl 3 10 µl 10 µl 4 50 µl 50 µl .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .................................................................................................................................................................... .............................................................................................................................................. 82 Experiment I3 -5. Weiterführende Literatur 5. Weiterführende Literatur 1. The success and failure of BCG - implications for a novel tuberculosis vaccine P. Andersen and T. M. Doherty; Nat Rev Microbiol; 2005 (Vol. 3, pp 656-62) Over the past 50 years, the Mycobacterium bovis bacille Calmette-Guerin (BCG) vaccine against tuberculosis (TB) has maintained its position as the world's most widely used vaccine, despite showing highly variable efficacy (0-80%) in different trials. The efficacy of BCG in adults is particularly poor in tropical and subtropical regions. Studies in animal models of TB, supported by data from clinical BCG trials in humans, indicate that this failure is related to pre-existing immune responses to antigens that are common to environmental mycobacteria and Mycobacterium tuberculosis. Here, we discuss the potential mechanisms behind the variation of BCG efficacy and their implications for an improved TB vaccination strategy. 2. Tuberculosis vaccines - an update P. Andersen; Nat Rev Microbiol; 2007 (Vol. 5, pp 484-7) The current tuberculosis (TB) vaccine Mycobacterium bovis bacillus Calmette-Guérin (BCG) is the most widely used vaccine worldwide, but it does not prevent the establishment of latent TB or reactivation of pulmonary disease in adults. Peter Andersen looks at the progress of the candidates to improve or replace BCG. 3. Human T cell epitopes of Mycobacterium tuberculosis are evolutionarily hyperconserved I. Comas, et al.; Nat Genet; 2010 (Vol. 42, pp 498-503) Mycobacterium tuberculosis is an obligate human pathogen capable of persisting in individual hosts for decades. We sequenced the genomes of 21 strains representative of the global diversity and six major lineages of the M. tuberculosis complex (MTBC) at 40- to 90-fold coverage using Illumina next-generation DNA sequencing. We constructed a genome-wide phylogeny based on these genome sequences. Comparative analyses of the sequences showed, as expected, that essential genes in MTBC were more evolutionarily conserved than nonessential genes. Notably, however, most of the 491 experimentally confirmed human T cell epitopes showed little sequence variation and had a lower ratio of nonsynonymous to synonymous changes than seen in essential and nonessential genes. We confirmed these findings in an additional data set consisting of 16 antigens in 99 MTBC strains. These findings are consistent with strong purifying selection acting on these epitopes, implying that MTBC might benefit from recognition by human T cells. 4. Mycobacterium tuberculosis and the intimate discourse of a chronic infection D. G. Russell; Immunol Rev; 2011 (Vol. 240, pp 252-68) Mycobacterium tuberculosis is an extremely successful pathogen that demonstrates the capacity to modulate its host both at the cellular and tissue levels. At the cellular level, the bacterium enters its host macrophage and arrests phagosome maturation, thus avoiding many of the microbicidal responses associated with this phagocyte. Nonetheless, the intracellular environment places certain demands on the pathogen, which, in response, senses the environmental shifts and upregulates specific metabolic programs to allow access to nutrients, minimize the consequences of stress, and sustain infection. Despite its intracellular niche, Mycobacterium tuberculosis demonstrates a marked capacity to modulate the tissues surrounding infected cells through the release of potent, bioactive cell wall constituents. These cell wall lipids are released from the host cell by an exocytic process and induce physiological changes in neighboring phagocytes, which drives formation of a granuloma. This tissue response leads to the generation and accumulation of caseous debris and the progression of the human tuberculosis granuloma. Completion of the life cycle of tuberculosis requires damaging the host to release infectious bacteria into the airways to spread the infection. This damage reflects the pathogen's ability to subvert the host's innate and acquired immune responses to its own nefarious ends. 83 Experiment GM1 Experiment Genmodifikation GM1: Bestimmung der Retrotranspositionsrate des humanen LINE-1 Elements Prof. Dr. Gerald Schumann 84 Experiment GM1 Einleitung: Ziel des vorliegenden Versuchs ist die Bestimmung der Retrotranspositionsaktivität des humanen L1 Retrotransposons in verschiedenen Zelllinien. Dabei wird die Aktivität des Elements mit Hilfe eines Luziferase-basierenden Assays unter Verwendung unterschiedlicher Plasmide bestimmt. Folgende Ansätze sollen, jeweils in Tetraplikaten, untersucht werden: 1) pWA355 + Rluc (phRGTK) 4) pYX015 2) pWA366 + Rluc (phRGTK) 5) pYX017 3) pYX014 6) pEGFP Die Eigenschaften und Karten der hier verwendeten Plasmide finden sich am Ende dieses Versuchsprotokolls. Zusätzlich soll die Expression von humanen LINE-1-Elementen in vivo in verschiedenen Zellllinien nach Transfektion mit einem Reporterelement (pDK101/L1R-T7) durch Immunfluoreszenzfärbung analysiert werden. Versuchsablauf: Tag 1: Aussäen der Zellen zur Vorbereitung der Transfektionen und Immunfluoreszenzfärbung Die nachfolgenden Versuche werden mit zwei verschiedenen Zelllinien in 12well-Platten durchgeführt. Bei den verwendeten Zelllinien handelt es sich um die humane Teratokarzinomlinie 2102EP und die humane Cervixkarzinomlinie HeLa. Für die nachfolgenden Versuche werden die Zellen in einer Zellzahl von 1x105 pro Einzelwell verwendet. Bestimmung der Zellzahl: Jede Gruppe erhält für die Versuche je eine T75-Flasche beider Zelllinien zur weiteren Verwendung. Zur Bestimmung der Zellzahl wird wie folgt vorgegangen: Medium absaugen Zellen mit 10ml PBS waschen PBS absaugen pro Flasche 1,5ml Trypsin(0,25%)/EDTA/PBS zugeben Inkubation für 5min im Brutschrank Ablösen der Zellen durch vorsichtiges Klopfen an die Flaschen Zugabe von 8,5ml Medium; Zellen resuspendieren Zelllösung in 15ml-Falcon überführen 50µl abnehmen, 1:1 mit Trypanblau mischen und in die Neubauer-Zählkammer überführen. Trypanblau wird aufgrund der Membrandurchlässigkeit nur von toten Zellen aufgenommen, die nachfolgend nicht mit ausgezählt werden. Zellzahlbestimmung während der Zellzahlbestimmung beide Falcons für 5min bei 800rpm und Raumtemperatur abzentrifugieren je 1ml Medium pro Einzelwell in die 12well-Platten vorlegen Überstand nach dem Abzentrifugieren abnehmen und Zellen in einem entsprechenden Volumen Medium resuspendieren (siehe Beispielrechnung unten) pro Einzelwell 1x105 Zellen aussäen 5ml der Zellsuspension in eine neue T75-Flasche mit 15ml Medium überführen, den Rest der Zellen verwerfen. 12well-Platten und T75-Flaschen in den Brutschrank überführen. 85 Experiment GM1 Neubauer-Kammer Die Neubauerkammer hat in der Mitte ein Kreuz aus engen Linien. Die 4 Eckfelder (Gruppenquadrate) sind in je 16 Einzelquadrate unterteilt. Zum Auszählen werden die Zellen in mindestens 2 (bei besonders niedriger Zellzahl 4) Eckfeldern gezählt. Berechnung der Zellzahl: Auszählen von 2x16 Kleinquadraten Berechnung des Mittelwertes aus den 2 Eckfeldern Kammerfaktor: 1x104 Berechnungsformel: Zellzahl/ml = Mittelwert x Verdünnungsfaktoren x Kammerfaktor Beispiel: 300 Zellen wurden in 2 Eckfeldern gezählt. 50μl Zellsuspension wurde mit 50μl Trypanblau verdünnt (Verdünnungsfaktor 2). Es liegen 10ml Zellsuspension insgesamt vor. Der Kammerfaktor (um vom Einschlussvolumen auf 1 ml umzurechnen) beträgt 1x104. → 300 Zellen x 2 x 10ml x 1x104 Zellen/ml = 6x107 Zellen/ml Berechnung des einzusetzenden Medienvolumens: Volumen = tatsächliche Zellzahl/ml / gewünschte Zellzahl/ml Beispiel: Die Zellzahl wurde auf 6x107 Zellen/ml berechnet. Ausgesät werden sollen aber 1x105 Zellen/ml. Daraus ergibt sich folgende Berechnung: Volumen = 6x107 Zellen/ml / 1x105 Zellen/ml = 600ml Medium → Zellen in 600ml Medium aufnehmen und je 1ml in die 12well-Platten geben oder: → Zellen in 1/100 Medium aufnehmen (also 6ml) und je 10µl der Suspension zu den je 1ml vorgelegtem Medium geben Immunfluoreszenzfärbung zum Nachweis der Expression des LINE-1 codierten ORF-1 Proteins Zum Nachweis der Expression des LINE-1-codierten ORF1-Proteins wird in diesem Versuchsteil eine Immunfluoreszenzfärbung durchgeführt. Dabei wird mit einem Fluoreszenz-gekoppelten Antikörper, der den T7-Taq am Carboxyterminus des ORF1-Proteins erkennt, die subzelluläre Lokalisation dieses Proteins analysiert. Die Immunfluoreszenzfärbung wird in HeLa-Zellen und 2102EP-Zellen durchgeführt. Die Zellen wurden bereits donnerstags zuvor von der vorherigen Gruppe auf Deckgläschen ausgesät und vom Betreuer freitags mit einem Plasmid transfiziert, das für das L1.3Element mit einem ORF1-T7-Fusionsprotein kodiert (siehe Plasmidkarte pDK101/L1.3). Dadurch kann 86 Experiment GM1 die Analyse der Expression des ORF1-Proteins einfach über die Detektion des T7-Taqs erfolgen. Gefärbt werden außerdem Aktin und der Zellkern (DAPI-Färbung). Folgende Ansätze wurden vorbereitet: 1) Erstantikörperkontrolle (ein Deckglas) 2) Zweitantikörperkontrolle (ein Deckglas) 3) pDK101-L1.3 (3 Deckgläser) Fixierung der Zellen Medium absaugen Zellen mit PBS waschen Fixierung mit 4% Paraformaldehyd/PBS (pH 7.4) auf dem Schüttler bei Raumtemperatur für 15 min Permeabilisierung der Zellen zur Permeabilisierung Paraformaldehyd-Lösung absaugen die Zellen für 10 min bei Raumtemperatur auf dem Schüttler in 1% Triton-X-100/PBS (pH 7.4) inkubieren Waschen der Zellen und Blockieren Permeabilisierung-Lösung vorsichtig absaugen die Zellen 3x2 min in PBS auf dem Schüttler bei Raumtemperatur waschen anschließend Sättigung von unspezifischen Bindestellen durch Blockieren mit 5% BSA/0,1% Triton-X-100/PBS für 30 min bei Raumtemperatur PBS (ml) 5% BSA/PBS (ml) Triton-X-100 (µl) 1% Triton-X-100/PBS 0,1% Triton-X-100/5% BSA/PBS Inkubation mit dem Erstantiköper Blockingpuffer absaugen Zugabe des Erstantikörpers. Für den Nachweis des LINE-1-Elementes wird ein anti-T7Antikörper aus dem Kaninchen verwendet (Verdünnung von 1:250 in 5% BSA/PBS; 120µl pro Deckglas). Der anti-Aktin-Antikörper ist bereits mit einem Fluoreszenzfarbstoff markiert und wird zusammen mit dem Zweitantikörper gegen den T7-Antikörper zugegeben. 1h bei Raumtemperatur inkubieren 5% BSA/PBS (ml) Antikörper (µl) Erstantikörperkontrolle Zweitantikörperkontrolle anti-T7-Antikörperlösung anti-Aktin-Antikörperlösung Waschen der Zellen und Inkubation mit dem Zweitantikörper Zellen 3x5 min mit PBS waschen, um nicht gebundenen Erstantikörper zu entfernen Inkubation mit dem Zweitantikörperlösung (120µl pro Deckglas). Dabei handelt es sich für den T7-Taq um einen Fluoreszenz-gekoppelten Anti-Kanichen-Antikörper (AlexaFluor® 488 goat-anti-rabbit; Verdünnung 1:1000 in 5% BSA/PBS). Zum Nachweis von Aktin wird ein Fluoreszenz-gekoppelter Phalloidin-Antikörper (AlexaFluor® 633 Phalloidin; Verdünnung 1:500 in 5% BSA/PBS) eingesetzt. 87 Experiment GM1 Inkubation für 30 min bei Raumtemperatur im Dunkeln! 5% BSA/PBS (ml) Erstantikörperkontrolle Zweitantikörperkontrolle anti-T7-Antikörperlösung anti-Aktin-Antikörperlösung Antikörper (µl) Waschen der Zellen und Zellkernfärbung Zellen 3x5 min in PBS waschen Parallel Lösung mit DAPI (Stock: 1mg/ml → Verdünnung 1:1000 in PBS; 500µl pro Deckglas). 2 min bei Raumtemperatur inkubieren 3x10 min Waschen mit PBS bei Raumtemperatur auf dem Schüttler PBS (ml) Dapi-Stocklösung (µl) Erstantikörperkontrolle Zweitantikörperkontrolle anti-T7-Antikörperlösung anti-Aktin-Antikörperlösung Einbetten der Zellen pro Deckgläschen ein Tropfen Aquapolymount auf einen entsprechend beschrifteten Objektträger auftropfen Deckgläschen mit der „Zellseite“ zum Objektträger auf den Tropfen aufgelegen überschüssiges Einbettmedium vorsichtig abtupfen. die Proben werden bis zur weiteren Analyse (Tag2) bei 4°C im Kühlschrank gelagert. Tag 2: Transfektion und Auswertung der Immunfluoreszenz am Konfokalmikroskop Die Transfektionen werden mit dem Transfektionsreagenz FugeneHD nach Herstellerangaben durchgeführt. Es wird dabei ein Verhältnis von 3:1 (v/w) von Transfektionsreagenz zu eingesetzter Plasmid-DNA verwendet. Für die Transfektion eines Einzelwells werden 1,1µg Plasmid-DNA (gelöst in 52µl OptiMEM) und 3,3µl FugeneHD benötigt. Für die ersten beiden Transfektionen in der unten stehenden Tabelle werden 9/10 von pWA355 bzw. pWA366 mit 1/10 Rluc-Plasmid eingesetzt (Kotransfektion). Für die restlichen Transfektionen wird jeweils nur ein Plasmid verwendet. Plasmid Konzentration µg/µl einzusetzendes Volumen in µl pWA355 + Rluc pWA366 + Rluc pYX014 pYX015 pYX017 pEGFP Durchführung: Medium von den Zellen absaugen pro Einzelwell 1ml frisches Medium zugeben 88 Volumen FugeneHD in µl Volumen OptiMEM in µl Experiment GM1 Zellen wieder in den Brutschrank stellen ausreichende Anzahl von Eppendorf-Reagenzgefäßen beschriften pro Tetraplikat werden für die Transfektionen Mastermixe angesetzt; daher zunächst Berechnung der einzusetzenden Plasmidkonzentrationen mit Hilfe der obigen Tabelle in jedes Eppi entsprechendes Volumen an OptiMEM für den jeweiligen Mastermix vorlegen zum OptiMEM die entsprechende Menge an Plasmid-DNA zufügen zu jedem Ansatz das entsprechende Volumen FugeneHD zugeben mischen, kurz abzentrifugieren 10min bei Raumtemperatur inkubieren zur Bildung der Lipid-DNA-Komplexe pro Einzelwell 50µl Transfektionsansatz zupipettieren, 12wells kurz schwenken und zurück in den Brutschrank stellen Transfektion über Nacht, Mediumwechsel durch Betreuer am darauffolgenden Tag Auswertung der Immunfluoreszenz am Konfokalmikroskop Die Auswertung der Immunfluoreszenzfärbungen erfolgt am Konfokalmikroskop zusammen mit dem Betreuer. Es werden verschiedene Aufnahmen der Färbungen gemacht. Tag 3: Auswertung des Assays und Bestimmung der Transfektionseffizienz (3Tage nach Transfektion): Die Auswertung erfolgt über durch ein Luziferaseassay. Die Messung der Luciferaseaktivität ermöglicht die Bestimmung der Retrotranspositionsaktivität des untersuchten L1-Elements. FireflyLuziferase bietet sich in diesem Falle als Reportersystem an, da ihre Aktivität verhältnismäßig leicht mit Hilfe eines Luminometers bestimmt werden kann. In Anwesenheit von ATP, Magnesiumionen und Sauerstoff katalysiert die Luziferase die Oxidation von Luziferin über das Intermediat Luciferyl-ATP zu Oxoluciferin. Bei dieser Umsetzung seines Substrats wird ein Fluoreszenzlicht emittiert, das vom Luminometer detektiert wird. Luciferin + ATP → Lucifery-AMP + Diphosphat (PP) Luciferyl-AMP + O2 → Oxoluciferin + Photon Wie bereits oben beschrieben, wurde bei den Transfektionsansätzen, bei denen die Plasmide pWA355 und pWA366 verwendet wurden, zusätzlich noch ein weiteres Plasmid, die Rluc, mittransfiziert. Die darauf codierte Renilla-Luziferase wird konstitutiv exprimiert und dient in diesen Versuchen als interne Kontrolle. Die nachfolgend gemessenen Firefly-Luziferaseaktivitäten werden auf die Aktivitäten der Renilla-Luziferase normalisiert, so dass dadurch Pipettierfehler und Unterschiede in der Transfektionseffizienz in verschiedenen Ansätzen ausgeglichen werden können. Da die Renilla-Luziferase ein anderes Substrat als die Firefly-Luziferase verwendet, kann durch Verwendung unterschiedlicher Substrate einfach zwischen beiden Aktivitäten unterschieden werden. Verständisfrage: Warum ist bei den übrigen Transfektionsansätzen mit pYX014, pYX015 und pYX017 keine Kotransfektion mit einem Renilla-Plasmid (Rluc) erforderlich? 89 Experiment GM1 Durchführung des Luziferase-Assays: 1fach-Lysispuffer (Passive Lysis Buffer, Promega) ansetzen Medium von den Zellen absaugen Zellen mit PBS waschen und absaugen pro Einzelwell 150µl Lysispuffer zugeben 15min bei Raumtemperatur auf dem Schüttler inkubieren während der 15min Inkubation Ansetzen der beiden Substrate für die Firefly-Luziferase (Luciferase Assay Reagent II) und die Renilla-Luziferase (Stop&Glow) nach Angaben des Betreuers; pro well werden 50µl des jeweiligen Substrats zuzüglich je 2ml (Totvolumen Luminometer) benötigt. Zellen mit einem Zellschaber ablösen und mit einer Pipette in ein 1,5ml Eppi überführen durch Auf- und Abpipettieren gut homogenisieren 10min bei 4°C und 13.000rpm abzentrifugieren vom Überstand 20µl in eine 96well-Platte überführen Luziferase-Aktivitätsmessung am Luminometer Bestimmung der Transfektionseffizienz: Diese erfolgt mit Hilfe eines Fluoreszenzmikroskops durch Auszählen der grün fluoreszierenden Zellen in den mit pEGFP transfizierten wells. Dazu werden von jedem Einzelwell je ein Durchlichtund ein EGFP-Bild aufgenommen und dann durch Auszählen die Anzahl der transfizierten (grün fluoreszierenden) und der nicht leuchtenden Zellen ermittelt. Anhand dieser Zahlen kann dann die Transfektionseffizienz als % grün leuchtender Zellen an der Gesamtzellzahl berechnet werden. Zelllinie Anzahl Zellen pro Gesichtsfeld Anzahl grüner Zellen pro Gesichtsfeld Transfektionseffizien z in % 2101EP HeLa Vorbereitung der Immunfloureszenzfärbung für die nachfolgende Gruppe (siehe auch Tag 1) Bestimmung der Zellzahl: Jede Gruppe erhält für den Versuch wiederum je eine T75-Flasche beider Zelllinien zur weiteren Verwendung. Zur Bestimmung der Zellzahl wird wieder wie folgt vorgegangen: Medium absaugen Zellen mit 10ml PBS waschen PBS absaugen pro Flasche 1,5ml Trypsin(0,25%)/EDTA/PBS zugeben 90 Experiment GM1 Inkubation für 5min im Brutschrank Ablösen der Zellen durch vorsichtiges Klopfen an die Flaschen Zugabe von 8,5ml Medium; Zellen resuspendieren Zelllösung in 15ml-Falcon überführen 50µl davon nach erneutem Resuspendieren für die Zellzahlbestimmung abnehmen, 1:1 mit Trypanblau mischen und in die Neubauer-Zählkammer überführen. Trypanblau wird aufgrund der Membrandurchlässigkeit nur von toten Zellen aufgenommen, die nachfolgend nicht mit ausgezählt werden. Zellzahlbestimmung während der Zellzahlbestimmung beide Falcons für 5min bei 800rpm und Raumtemperatur abzentrifugieren je 1ml Medium pro Einzelwell in die mit Deckgläschen ausgelegten 12well-Platten vorlegen Überstand nach dem Abzentrifugieren abnehmen und Zellen in einem entsprechenden Volumen Medium resuspendieren (siehe Beispielrechnung unten) pro Einzelwell 1x105 Zellen aussäen 5ml der Zellsuspension in eine neue T75-Flasche mit 15ml Medium überführen, den Rest der Zellen verwerfen. 12well-Platten und T75-Flaschen in den Brutschrank überführen. Auswertung: 1) Bestimmung der Transfektionseffizienz in beiden untersuchten Zelllinien. Zeigen sich Unterschiede? Worauf könnten sie zurückzuführen sein? 2) Berechnung Retrotranspositionsrate in den verschiedenen Ansätzen und Zelllinien 3) Welchen Einfluss hat die verwendete Zelllinie auf die Retrotranspositionsaktivität? 4) Welche Rolle spielen die Transfektionsbedingungen (Einfach- zu Kotransfektion)? 5) Gibt es Unterschiede in der Retrotranspositionsrate bei unterschiedlichen Ansätzen? Wenn ja, worauf sind diese zurückzuführen? 6) Was kann man aus den Immunfluoreszenzbildern bezüglich der Lokalisation der LINE-1Expression ableiten? Gibt es Unterschiede zwischen den Zelllinien? Wenn ja, welche und worauf sind sie zurückzuführen? 7) Beantwortung der Verständisfrage Plasmidkarten: 91 Experiment GM1 pWA355 (kompetent)/pWA366 (inkompetent) pYX014 (kompetent)/pYX015 (inkompetent)/ pYX017 (kompetent) pYX014 (kompetent)/pYX015 (inkompetent) /pYX017 (kompetent) pDK101/L1.3 (kompetent) Prinzip des Retrotranspositionsassays: 92 Experiment GM2 Experiment Genmodifikation GM2: Transposition und transponierbare genetische Elemente Dr. Zoltan Ivics (6/0) Betreuer: Dr. Csaba Miskey, Dr. Oliver Walisko, Dr. Esther Grueso, Marta Swierczek, Lisa Wiedemann 93 Experiment GM2 Transposition und transponierbare genetische Elemente Bei dem Vorgang der Transposition handelt es sich um einen Spezialfall der Rekombination zwischen nicht-homologen DNA Sequenzen, welche zu einer Positionsänderung der transponierbaren (mobilen) DNA im betrachteten Genom führt. Je nach begrifflicher Definition werden solche mobilen (transponierbaren) DNAs als Insertionssequenzen, Transposons oder transponierbare genetische Elemente (TEs) bezeichnet. Transponierbare genetische Elemente sind in der Natur weit verbreitet und sind Bestandteil vieler untersuchter Genome, einschließlich derer von Prokaryonten, niederen und höheren Eukaryonten (einschließlich Hefen, Pflanzen und Wirbeltieren). So sind ca. 45% des menschlichen Erbguts TEs, oder von TEs abgeleitete DNA-Sequenzen. Aufgrund ihrer Mobilität sind TEs in der Lage genetische Informationen zu verändern, in dem sie z.B. in Gene integrieren und somit zu Genmutationen (Insertionen) führen. TE-Integrationen in der Nähe von genregulatorischen Einheiten (Enhancern/Promotoren) können zu Veränderungen der Expression der betroffenen Gene führen. Ein Beispiel für den Einfluss transponierbarer, genetischer Elemente auf die Genfunktion und somit auf den Phänotyp ist der Ziermais (Abbildung 1). Im Ziermais sitzt das Transposon in einem Gen, welches für den dunklen Farbstoff der Maiskörner kodiert und somit gelbe Maiskörner zur Folge hat. Durch erneute Mobilisierung des Transposons kommt es zur Reversion des Pänotyps (dunkle Körner), aber auch zu Farbvariationen. Somit tragen TEs aufgrund ihrer Mobilität wesentlich zur Genomplastizität und -evolution bei. Abbildung 1 Entsprechend ihres Transpositionsmechanismus, Hauptkategorien eingeteilt werden: 1. DNA Transposons (Klasse2-TEs) werden als DNAZwischenstufe aus dem DNA-Strang "ausgeschnitten" und an einer anderen Stelle der Wirts-DNA integriert (Abbildung 2). können TEs in zwei 2. Retrotransposons werden nach Transkription als RNA-Zwischenstufe in eine cDNA umgeschrieben, welche ebenfalls an einer beliebigen Stelle des Genoms re-Integriert wird. Abbildung 2 Struktur & Transpositionsmechanismus von DNA Transposons (Klasse2-TEs) Die Struktur von DNA Transposons ist vergleichbar einfach: Das zentrale Transposase-Gen (die Transposase ist das Enzym, das den Transpositionsvorgang katalysiert) ist zu beiden Seiten von terminalen, invertierten Sequenzwiederholungen (terminal inverted repeats=TIRs) flankiert, die die Bindungsstellen für die Transposase enthalten. 94 Experiment GM2 Während der Transposition (Vergleich Abbildung 2 & 3) bindet die Transposase an die Transposase-Bindungsstellen in den TIRs (transposase binding) und es kommt zu einer Annäherung der beiden Transposonenden (synatic complex). Im synatipschen Komplex schneidet die Transposase die beiden Transposonenden und das ausgeschnittene Transposon integriert in einem neuen Ziellokus (strand transfer). Dieser Mechanismus wird auch "cut-and-paste transposition" genannt. Transposons als Vektorsysteme in der molekularbiologischen und biomedizinischen Forschung Die Fähigkeit von DNA Transposons sich zwischen verschiedenen DNA-Loci durch cut-and-paste zu bewegen, kann für die Entwicklung von Gentransfervektoren verwendet werden (Abbildung 3). Dafür wird das Transposase-Gen entfernt und ein Transgen, welches in die Zielzelle/-organismus eingeschleust werden soll, zwischen die TIRs eingefügt. Das Plasmid mit dem TIR-flankierten Transgen wird zusammen mit der Transposase in die Zielzelle transfiziert, wobei es dann im Verlauf der Transpositionsreaktion zur Integration des TIR-flankierten Transgens in das Genom kommt (Abbildung 3). Solche Transposon-basierten (nicht-viralen) Gentransfersysteme finden in der somatischen Gentherapie und molekularbiologischen Forschung Anwendung. Abbildung 3 Der Praktikumsversuch Ziel des in diesem 3-tägigen Praktikums durchgeführten Versuches wird es sein Ihnen exemplarisch die Anwendung von Transposon-basierten Vektorsystem zu veranschaulichen. Sie werden ein Transgen (in diesem Fall ein NeomycinResistenzgen) mit Hilfe des sog. Sleeping Beauty Transposons von transfizierter Plasmid DNA mobilisieren und in das Genom menschlicher Zellen (Zellkultur) 95 Experiment GM2 integrieren. Sie werden lernen, die Transpositionsreaktion anhand eines GFP (green fluorescent protein)-Markergens zu verfolgen. In das Genom integrierte Transposon DNA (Transgen) werden Sie mit einer speziellen PCR-Technik (Splinkerette-PCR) nachweisen. In der Praxis würde ein solcher Versuch ca. zwei Wochen in Anspruch nehmen, die uns für dieses Praktikum leider nicht zur Verfügung stehen. Deshalb haben wir den Versuchsablauf so angepasst, dass Sie die wesentlichen Techniken innerhalb von 3 Tagen kennnenlernen werden. Der Praktikumsablauf Das Praktikum ist in drei Blöcke aufgeteilt, wobei sich Teil I mit dem Nachweis der Transpositionsreaktion anhand eines GFP-Fluoreszenz-Markers, Teil II mit dem Nachweis genomisch integrierter Transposon DNA mittels PCR beschäftigt und Teil III beschäftigt sich mit einem anderen Ansatz der Genommodifikation, der auf dem Einsatz von Nukleasen beruht. Versuch I: Messung von Sleeping Beauty Transposition in Zellkultur Hintergrund: Ein "Reporter"-Plasmid (pCMV(CAT)-GFP//T2neo) (siehe Anhang), das ein Neomycin-markiertes Sleeping Beauty Transposon (SB) innerhalb eines GFPLeserahmens trägt, dient in diesem Experiment als Marker, um die Transpositionsreaktion nachzuweisen. Dazu wird das "Reporter"-Plasmid zusammen mit einem Expressions-Plasmid, das die SB-Transposase (SB-Tpase) kodiert in humane HeLa-Zellen transfiziert. Die SB-Tpase "schneidet" das SB-Transposon aus dem GFP-Leserahmen aus, was zur Wiederherstellung eines intakten GFP-ORFs und somit zu GFP-Expression führt. Der Anteil GFP-exprimierender Zellen in der Gesamtzellpopulation ist ein Maß für die Effizienz der Transpositionsreaktion, welche sich mittels FACS-Zytometry nachweisen lässt. Materialien: - HeLa Zellen (T75 Flasche, 100% konfluent) - 6-Well Zellkulturplatte - DMEM (+ 1% Pen/Strep, 1mM Glutamin, 10% FCS) - OptiMEM - 1x PBS - 1x Trysin - Trypanblau-Lösung - Transposon: pCMV(CAT)-GFP//T2neo (500 ng/ul) - Transposase: pCMV-SB100X (100 ng/ul) - inactive Transposase: pCMV-SB_inactive (100 ng/ul) - transIT-LTI Transfektionsreagenz - Fixierungsreagenz (1% Formaldehyd in PBS) - BD LSRII Flowzytometer Durchführung: Tag1: Aussaat der Zellen (HeLa) für Transfektion - Legen Sie in 6-Well Zellkulturplatte pro Well 2 ml DMEM vor 96 Experiment GM2 - Ernten Sie von einer T75 Zellkulturflasche die HeLa-Zellen, indem Sie die Zellen zunächst mit 10 ml PBS waschen (PBS zugeben und gleich wieder entfernen) und dann 1 ml 1x Trypsin zugeben - Verteilen Sie das Trypsin auf den Zellen durch vorsichtiges Schwenken und lassen Sie es einige Minuten bei 37°C einwirken - durch vorsichtiges "klopfen" lösen Sie die Zellen vom Boden der Zellkulturschale - Geben Sie 9 ml DMEM zu den abgelösten Zellen und stellen Sie eine homogene Zellsuspension durch starkes "auf & ab" Pipettieren her. - pipettieren Sie anschließend 10 µl der Zellsuspension in ein Eppendorfgefäß und fügen Sie 40 µl Trypanblau-Lösung hinzu (1:5-Verdünnung) - Geben Sie 10µl der Lösung in eine Neubauer-Zählkammer und zählen Sie die Zellen - pipettieren Sie jeweils 200 000 Zellen der Zellsuspension pro Well in die vorbereitete 6-Well-Platte - verteilen Sie die Zellen gleichmäßig durch vorsichtiges Schwenken - kultivieren Sie die Zellen über Nacht bei 37°C, 5% CO2 Tag 2: Transfektion - die am Vortag ausgesäten Zellen sollten zwischen 50-70% konfluent sein - stellen Sie folgende Ansätze her Kontrolle[µl] 100ng Tpase [µl] 200ng Tpase [µl] untransfiziert ul OptiMEM 0 4 ul pCMV-SB100X 0 µl pCMV-SB_inactive 0 ul transIT-LTI 0 250 ul pCMV/CAT-GFP//T2neo 0 0 250 250 4 4 1 2 2 0 0 6 6 6 - Inkubieren Sie die Ansätze20 min bei Raumtemperatur - geben Sie 250 ul der Ansätze zu der 6-Well Platte - vorsichtig schwenken - inkubieren Sie die Ansätze 48 Stunden bei 37°C, 5% CO2 Tag 3: Analyse der Transpositionsaktivität mittels FACS - Saugen Sie das Zellkulturmedium ab und waschen Sie die Zellen vorsichtig mit 10 ml PBS - trypsinieren Sie die Zellen mit 1 ml Trypsin für wenige Minuten bei 37°C - Geben Sie 5 ml DMEM zu den abgelösten Zellen und stellen Sie eine homogene Zellsuspension durch starkes "auf & ab" Pipettieren her - überführen Sie die Zellsuspension in ein 15 ml-Röhrchen und zentrifugieren Sie bei 1000rpm für 5 Minuten - saugen Sie den Überstand vorsichtig ab - resuspendieren Sie das Pellet in 1 ml Fixierungsreagenz und pipettieren Sie die Suspension in ein FACS-Röhrchen - Analysieren Sie die Transpositionsaktivität mittels FACS 97 Experiment GM2 Versuch II: Ligation Mediated PCR to Recover Integration Sites of the Frog Prince Transposon in the Human Genome (Dr. Csaba Miskey) Genomic insertion of exogenous DNA by transposable element-derived vector systems provides the basis of versatile genetic applications including genetic screens for functional annotation of gene function and somatic gene transfer to treat genetic diseases in humans with gene therapy. In these studies it is usually necessary to determine the exact chromosomal position of the integrated transposon vectors. Several PCR-based methods have been devised to amplify insertion sites one of the prevalent ones is ligation mediated PCR (LM-PCR) (Figure 5). When aiming at amplifying vector integration sites by PCR a major technical difficulty is to provide specific binding-sites for the primers used in the PCR reactions. More specifically, the vector- (transposon) specific primers need to form amplicons with primers annealing in the neighborhood of the insertion sites to make it possible to amplify the integration locus. To achieve this goal known sequences need to be placed to the neighborhood of the insertion site on the chromosomes. This aim is achieved by fragmenting the chromosomal DNA containing the insertion(s) with frequently cutting restriction endonucleases and ligating linkers to the chromosomal ends. The enzymes used have recognition sequences of only four nucleotides. Since such short sequences occur statistically frequently on the chromosomes of any genome, digesting DNA with these enzymes results in chromosomal fragments with mean length distribution of a couple of hundred base pairs. This is important because the PCR reactions used later in the protocol have size limitations, but the short DNA fragments generated can efficiently be amplified. Thus, the restriction digest of the chromosomal DNA serves two purposes: (i) fragmenting the long chromosomes into small (thus PCR-amplifiable) pieces, and (ii) provide compatible DNA ends for ligating linker sequences to the chromosomal fragments for the PCR. After the ligation step the specific DNA fragments containing the specific transposon ends are PCR amplified from the large pool of ligation products. Since the linkers are ligated to virtually all pieces of the genomic DNA, the transposon specific primers provide the specificity of the PCR reactions. The DNA to be amplified is very much underrepresented in the ligation reaction, therefore two rounds of PCR is needed to amplify the junction fragments with linker-, and transposon-specific primers.. The second round of PCR reaction is performed on the products of the first round reaction. The primer binding sites of the second (“nested”) round are situated “inside” of the specific first-round PCR products. The final PCR products containing the transposon-genomic DNA junctions can be cloned or sequenced directly. 98 Experiment GM2 Figure 5 Schematic of Ligation Mediated PCR LM-PCR is based on restriction endonuclease digestion of the isolated genomic DNA containing the transposon insertion(s), ligation of double-stranded DNA linkers of a known sequence to the genomic fragments and subsequent PCR amplification of the insertion sites utilizing vector-end and linker-specific primers. 99 Experiment GM2 Practical Day 1 1. Digest 1µg of human genomic DNA isolated from HeLa cells containing integrated Frog Prince transposons with FspBI in 50µl reaction volume, for 4 hours, at 37oC. Concentration of the gDNA:…………………. µg/µl Material Volume water µl gDNA (1 µg) µl 10x Tango Buffer (Fermentas) µl FspBI 2 µl Total: 50 µl 2. Isolate the digested chromosomal DNA with QIAquick Spin Kit: Add 250 µl of Buffer PB to the restriction digest. Place a QIAquick spin column in a provided 2 ml collection tube. To bind DNA, apply the sample to the QIAquick column and centrifuge for 30s. Discard flow-through. Place the QIAquick column back into the same tube. To wash, add 0.75 ml Buffer PE to the QIAquick column and centrifuge for 30s. Discard flow-through and place the QIAquick column back in the same tube. Centrifuge the column for an additional 1 min Place QIAquick column in a clean 1.5 ml microcentrifuge tube To elute DNA, add 25 μl elution buffer to the center of the QIAquickb membrane, let the column stand for 2 min, and then centrifuge for 1min. 3. Ligate the isolated fragmented gDNA to double stranded linkers with T4 ligase at 16oC overnight. Material Volume µl water digested gDNA 5 µl linker (25pmol/ul) 1 µl µl 10x T4 ligase buffer T4 DNA ligase 400U/µl 0.4 µl Total: 20 µl 100 Experiment GM2 Practical Day 2 1. Perform 1st PCR on the ligation reaction. Material Volume water µl 10x Standard Taq buffer µl dNTP (10mM ) 1 µl Linker Primer (10 pmol/μl) 1 µl TXR-BR1 Primer (10 pmol/μl) 2 µl ligated DNA 5 µl 0.25 µl Taq polymerase Total: 50 µl st 1 PCR program: 94°C 3min 10 cycles of: 94°C 30s Ramp 0.5°C/sec to 65°C 65°C 30s 72°C 1min 20 cycles of: 94°C 30s Ramp 0.5°C/sec to 58°C 58°C 30s 72°C 1min 72°C 4°C 5min 101 Experiment GM2 2. Perform 2nd PCR on 1st PCR products (“nested” PCR). Material Volume water µl 10x Standard Taq buffer µl dNTP (10mM ) 1 µl Nested Primer (10 pmol/μl) 1 µl TXR-PF2 Primer (10 pmol/μl) 2 µl 1st PCR pruduct 5 µl Taq polymerase 0.25 µl Total: 50 µl 2nd PCR program: 94°C 3min 10 cycles of: 94°C 30s Ramp 0.5°C/sec to 65°C 65°C 30s 72°C 1min 25 cycles of 94°C 30s Ramp 1°C/sec to 55°C 55°C 20s 72°C 1min 72°C 4°C 5min Practical Day 3 Analyze 10 µl of the “nested” PCR product on a 1% agarose gel with GelRed dye using a DNA ladder as size marker. Make conclusions about the number of integration sites present in your sample. 102 Experiment GM2 Part III: Genome editing with RNA-guided nucleases: the CRISPR-Cas system Bacteria and archaea have evolved adaptive immune defences termed Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR)/CRISPR‐ASociated (Cas) systems that use short RNA to direct degradation of foreign nucleic acids. CRISPR defence involves acquisition and integration of new targeting “spacers” from invading virus or plasmid DNA into the CRISPR locus, expression and processing of short guiding CRISPR RNAs (crRNAs) consisting of spacer‐repeat units, and cleavage of nucleic acids (most commonly DNA) complementary to the spacer (figure 1). Figure 1 Features of the CRISPR-Cas adaptive immune system. Stage 1: CRISPR spacer acquisition. Specific fragments or protospacers (with an adjacent protospacer-associated motif; shown as red bar) of double-stranded DNA from a virus or plasmid are acquired at the leader end of a CRISPR array on host DNA. A CRISPR array consists of unique spacers (colored boxes; spacers are numbered sequentially with the most recently acquired spacer having the highest number) interspaced between repeats (black diamonds). Acquisition occurs by a process that minimally requires Cas1 and Cas2, encoded in the cas locus, usually located in the vicinity of the CRISPR array. Stage 2: CRISPR expression. Pre-CRISPR RNA (Pre-crRNA) is transcribed from the leader region by RNA polymerase and further cleaved into smaller crRNAs that contain a single spacer and a partial repeat (hairpin structures with colored spacers) by Cas proteins. Stage 3: CRISPR interference. crRNA containing a spacer that has a strong match to incoming foreign nucleic acid (plasmid or virus) initiates a cleavage event (shown by scissors); Cas proteins are required for this process. DNA cleavage interferes with virus replication or plasmid activity and imparts immunity to the host. Interference can be mechanistically and temporally separated from CRISPR acquisition and expression (depicted by wavy white bar across the cell). This figure is based on the CRISPR-Cas system in Streptococcus thermophilus, which represents a well-studied and relatively simple CRISPR-Cas system. From Bhaya, D. et al. Annu. Rev. Genet. 2011.45:273-297. 103 Experiment GM2 The Type II CRISPR system, utilizes a single effector enzyme, Cas9, to cleave dsDNA. Originally, the system consists of 3 components: 1) a long pre‐crRNA transcribed from the spacer‐containing CRISPR locus, 2) the multifunctional Cas9 protein, and 3) a tracrRNA important for gRNA (guide RNA) processing. Jinek et al. demonstrated that a tracrRNA‐crRNA fusion, termed a guide RNA (gRNA) (Fig. 2), is functional in vitro, obviating the need for RNase III and the crRNA processing in general (Jinek M. et al. Science 2012;337:816-821). Figure 2 (A) Cas9 can be programmed using a single engineered RNA molecule combining tracrRNA and crRNA features. (Top) In type II CRISPR/Cas systems, Cas9 is guided by a two-RNA structure formed by activating tracrRNA and targeting crRNA to cleave site-specifically–targeted dsDNA. (Bottom) A chimeric RNA generated by fusing the 3′ end of crRNA to the 5′ end of tracrRNA. From M Jinek et al. Science 2012;337:816-821. (B) RNA-guided gene targeting in human cells involves coexpression of the Cas9 protein bearing a C terminus SV40 nuclear localization signal with one or more guide RNAs (gRNAs) expressed from the human U6 polymerase III promoter. Cas9 unwinds the DNA duplex and cleaves both strands upon recognition of a target sequence by the gRNA, but only if the correct protospacer-adjacent motif (PAM) is present at the 3′ end. Any genomic sequence of the form GN20GG can in principle be targeted. From P Mali et al. Science 2013;339:823-826. Type II CRISPR interference is a result of Cas9 unwinding the DNA duplex and searching for sequences matching the crRNA to cleave. Target recognition occurs upon detection of complementarity between a “protospacer” sequence in the target DNA and the remaining spacer sequence in the crRNA. Importantly, Cas9 cuts the DNA only if a correct protospacer adjacent motif (PAM) is also present at the 3’ end. The S. pyogenes system utilized in this work requires an NGG sequence, where N can be any nucleotide. While the S. pyogenes system has not been characterized to the same level of precision than S. thermophilus, DSB formation also occurs towards the 3’ end of the protospacer. If one of the two nuclease domains is inactivated, Cas9 will function as a nickase in vitro and in human cells. 104 Experiment GM2 Genetic engineering Genetic engineering alters the genetic configuration of an organism using techniques that remove heritable material or that introduce DNA prepared outside the organism. This involves using recombinant nucleic acid (DNA or RNA) techniques to form new combinations of heritable genetic material followed by the incorporation of that material either indirectly through a vector system or directly through micro-injection, macro-injection and micro-encapsulation techniques. If genetic material from another species is added to the host, the resulting organism is called transgenic. Genetic engineering can also be used to remove genetic material from the target organism, creating a gene knockout organism (Capecchi, M.R. Nat. Med. 2001;7:1086–1090). Genetically modified animals have been used for research, to model human diseases and the production of agricultural or pharmaceutical products. Targeted genome editing with engineered nucleases Chosen by Nature Methods as the 2011 Method of the Year (Nature Methods 2012.9:1), genome editing is a type of genetic engineering in which DNA is inserted, replaced, or removed from a genome using artificially engineered nucleases, or "molecular scissors." The nucleases create specific double-stranded break (DSB) at desired locations in the genome, and harness the cell’s endogenous mechanisms to repair the induced break by natural processes of homologous recombination (HR) and non-homologous end-joining (NHEJ) (figure 4). There are currently four families of engineered nucleases: meganucleases, zinc finger nucleases (ZFNs), transcription activator-like effector nucleases (TALENs), and CRISPRs (figure 3)(Esvelt K.M. & Wang H.H. Mol. Syst. Biol. 2013;9:641-658; Tan W.S. et al. Adv Genet. 2012;80:37-97). Figure 3 Various categories of engineered nucleases currently used for genome editing. ZFN (zinc finger nuclease) and TALEN (Transcription activator-like effector nuclease) are linked to the active catalytic domains of FokI endonuclease. FokI subunits can be engineered to function as heterodimers only to increase specificity. Naturally occurring meganucleases can be engineered into hybrids to cover greater sequence diversity. RGENs now complement the toolbox of genome engineers. The respective DNA cleavage domains are depicted by the double pentamers in dark blue, and elements that determine DNA target-site specificity are highlighted in red. A zinc finger module (left) contacts three nucleotides of the target sequence on average, a TAL effector module (middle) binds one nucleotide, and one nucleotide of the sgRNA (right) interacts with one nucleotide of the DNA target site. 105 Experiment GM2 Perhaps the most reliable way to learn about the function of a gene or protein is to specifically perturb it and monitor what happens. This reverse genetic approach is routinely applied in many species, but, with a few exceptions, it is challenging or even impossible to make targeted changes at endogenous genomic loci, arguably the most elegant method of genetic perturbation. Instead, the experimenter must settle for more indirect approaches: overexpressing the modified gene from a heterologous location or knocking it down, often only partially, with an approach such as RNA interference. Engineered nucleases—which can be designed, in principle, to cut at any location in the genome of any species and thus to introduce tailored modifications into the endogenous sequence—are set to change this. Figure 4 The gRNA is a hybrid of two components: CRISPR RNA (crRNA; blue) and trans-activating crRNA (tracrRNA; red). (A) Cleavage of the target DNA by the Cas9 nuclease results in mutations that can knock out the target gene. (B) If an appropriate donor DNA molecule is available, genetic information (shown here in pale blue) can be added to the target DNA in a precise manner. (C) By targeting two Cas9 nucleases to different regions of the target DNA, it is possible to delete the genetic information between the two regions. CRISPR: clustered regularly interspaced short palindromic repeats; Cas: CRISPR-associated. From Segal D.J. eLife 2013;2:e00563. In our practical approach, we will mimic the different steps we would have to follow in case of trying to knockout a particular gene/protein. We will use specifically designed CRISPRs cutting into two different exons in the same gene. The DSBs created by the CRISPRs will be repaired by, either HR or NHEJ. Like NHEJ is more prone to mutations, either insertions or deletions (INDELs), we will analyse the presence of this INDELs by a T7 assay (similar to the Surveyor assay in figure 5). 106 Experiment GM2 Figure 5 Overview of procedures. (A) A typical gene modification experiment initiates with CRISPR-Cas9 expression in an appropriate cell type. Cas9 cleave its endogenous target guided by the corresponding RNA, and subsequent repair yields minor mutations in a subset of chromosomes (in this case 20%).(B) A CRISPR-Cas (gRNA) is pictured schematically at the top of the panel bound to genomic DNA (straight double lines) and the resultant cleavage event is depicted by a gap. NHEJinduced mutations in the genomic DNA are denoted by jagged double lines. Bottom panel: The Surveyor assay initiates with cell lysis and PCR amplification of the targeted locus. Cross-annealing of mutated and wild-type sequence then converts mutations into mismatched duplexes. The reannealed amplicon is digested with the Surveyor nuclease (T7 nuclease in our case), which selectively cleaves distorted duplex DNA (Qiu P. et al. Biotechniques 2004;36:702–707). Digestion products are then resolved via PAGE or agarose gels. Horizontal half arrows indicate PCR primers; straight double lines and jagged lines denote, respectively, wild-type and mutated amplicon sequences. The rectangle at bottom schematically depicts a polyacrylamide/agarose gel with banding patterns yielded by CRISPR-treated and control cells. From Guschin D.Y. et al. Methods Mol Biol. 2010;649:247-56. Practical Day 1 In order to get Cas9 and our gRNAs into the cells by transfection, we will trypsinize a 10cm dish with STE cells to get them in suspension. We will count the cells and we will use 106 cells/condition for electroporation. Trypsinization Remove media from the cultured cells and wash cells once with PBS- (no Ca2+ or Mg2+); use at least same volume of PBS- as culture media. 107 Experiment GM2 For harvesting, incubate the cells ~3 minutes at 37 °C with recommended volume of indicated trypsinization reagent (1ml/P10 dish of trypsin-EDTA). Neutralize trypsinization reaction with DMEM+10%FBS when all the cells have been detached. Count an aliquot of the cells using a hemocytometer (=Neubauer chamber) and determine cell density. Nucleofection™ Make sure that the entire supplement is added to the Nucleofector™ Solution. Start 4D-Nucleofector™ System and create experimental settings (Buffer P3 and program EN-138). Prepare 4 M6 cell culture wells and fill them with 2,5ml of DMEM+20%FBS (without antibiotic) and pre-incubate/equilibrate plates in a humidified 37 °C/5 % CO2 incubator. Prepare 4 tubes with the corresponding DNA: TUBE 0 (control) TUBE 1 (gRNA1) TUBE 2 (gRNA2) TUBE 3 (gRNA1+gRNA2) 2ug Cas9 (1ug/ul) 2ug Cas9 (1ug/ul) 2ug Cas9 (1ug/ul) 2ug Cas9 (1ug/ul) 2ug pmaxGFP (1ug/ul) 2ug gRNA1 (1ug/ul) 2ug gRNA2 (1ug/ul) 1ug gRNA1 (1ug/ul) + 1ug gRNA2 (1ug/ul) Centrifuge 4x106 cells at 90xg for 10 minutes at room temperature. Remove supernatant completely. Resuspend the cell pellet carefully in 400ul of room temperature 4DNucleofector™ Solution and add 100ul of the cell mix to every sample tube. Warning: As leaving cells in Nucleofector™ Solution for extended periods of time may lead to reduced transfection efficiency and viability it is important to work as quickly as possible. Transfer the resuspended cells with DNA into the Nucleocuvette™ Vessels (Warning: avoid air bubbles while pipetting!!) and transfect them with the nucleofector (Warning: Gently tap the Nucleocuvette™ Vessels to make sure the sample covers the bottom of the cuvette). After the pulse, resuspend cells with pre-warmed medium in the plates wells using the supplied pipettes and avoid repeated aspiration of the sample. Mix cells by gently pipetting up and down two to three times. Distribute the cells evenly by moving the plates following a “cross” motion. 108 Experiment GM2 Practical Day 2 Today, we will check the transfection procedure under the microscope by GFP expression, and we will isolate the DNA from the cells in order to analyse the genome modification of the CRISPRs-Cas transfected. And we calculate approximately the efficiency of transfection by checking the percentage of GFP+ cells under the fluorescence microscope in comparison with the total number of cells. Homogenisation and Lysis Cell culture cells growing in monolayers will be directly lysed in the dish. Remove media from the cultured cells and wash cells once with PBS- (no Ca2+ or Mg2+); use at least same volume of PBS- as culture media. Add 400 μl DNA Lysis Buffer T, 20 μl Proteinase K solution (20mg/ml) and 30 μl RNase A (10mg/ml). Spread the buffer evenly over the whole surface to ensure complete detaching of the cells. Mix the lysate by pipetting up and down several times. Fill the lysate in a 1,5 ml centrifuge tube. Vortex for 10 sec and incubate at 50 °C for 15 – 30 min in a shaking water bath. Warning: If no shaking water bath is available, mix the sample 3 to 4 times during incubation time by vortexing for 10 sec. Loading and Binding Add 200 μl DNA Binding Buffer per 400 μl DNA Lysis Buffer T and mix thoroughly by pipetting. After addition of the DNA Binding Buffer a precipitate can form, which however has no influence on the DNA isolation. Place a PerfectBind DNA Column in a 2 ml Collection Tube and load up to 750 μl of the preparation (inclusive all precipitates) on the column. Centrifuge the PerfectBind DNA Column with the Collection Tube for 1 min at 10.000 x g*. Discard the flow-through. Repeat this step until the entire preparation is loaded. Discard the flow-through and the Collection Tube. Washing I Add 650 μl of the diluted DNA Wash Buffer (Buffer concentrate plus 1.5 volume absolute ethanol) on the column. Centrifuge the PerfectBind DNA Column with the Collection Tube for 1 min at 10.000 x g*. Discard the flow-through, keep the Collection Tube. Washing II Repeat last step. Dry (Warning: Important! Do not skip this step!) Using the same 2.0 ml Collection Tube, centrifuge PerfectBind DNA Column at 10.000 x g* for 2 min to completely dry the PerfectBind DNA Column. Elution Place the PerfectBind DNA Column into a sterile 1.5 ml microfuge tube and add 100 μl of Elution Buffer (10mM tris HCl PH>7) or water onto the center of the membrane. Allow tubes to sit for 3 min at room temperature. To elute DNA from the column, centrifuge at 6.000 x g* for 1 min. 109 Experiment GM2 DNA Quantification Quantify the amount of DNA by A260/280 using the nanodrop spectrophotometer. PCR amplification Amplify specifically the genome locations framing the CRISPRs targeted exons. PCR1 Volume (per reaction) SAMPLE 0 Material SAMPLE 1 DNA template SAMPLE 2 10 µl dNTP (10mM ) 1 µl Primer 1 fd (10 μM) 2.5 µl Primer 1 rv (10 μM) 2.5 µl Taq (+ control) 0.5 µl water Y µl (=33.5-X) Total: 50 µl PCR2 Volume (per reaction) SAMPLE 0 Material SAMPLE 1 DNA template SAMPLE 2 X (=500ng) 5x Phusion HF buffer 10 µl dNTP (10mM ) 1 µl Primer 2 fd (10 μM) 2.5 µl Primer 2 rv (10 μM) 2.5 µl Phusion polymerase SAMPLE 4 X (=500ng) 5x Phusion HF buffer Phusion polymerase SAMPLE 3 Taq 0.5 µl water Y µl (=33.5-X) Total: 50 µl 110 SAMPLE 3 SAMPLE 4 (+ control) Experiment GM2 PCR program: 98°C 30’’ Purification of the target 35 cycles of: 98°C 10’’ 65°C 20’’ 72°C 15’’ 72°C 10min 4°C ∞ Run 2 µl out of the 50 µl PCR reaction on a 1% agarose test gel. Note: Alternatively, we can measure the amount of DNA by nanodrop. If the amplicon shows the expected size AND it is only one band, purify the remaining PCR reaction with a PCR purification Kit (f.e. Qiagen). Follow the manufacturer’s instructions. Elute the DNA in 30 µl 1xNEB2 buffer and determine the DNA concentration with the nanodrop again. Practical Day 3 After a PCR amplification of the targeted loci, we will melt and randomly reanneal the amplicons, which will convert any mutations into mismatched duplex DNA. The reannealed amplicon will be digested with the T7 nuclease, which selectively cleaves distorted duplex DNA, and digestion products will be then resolved by a 2.5% agarose gel. Amplicon melting and reannealing Perform the program “heteroduplexes” (95°C, 10 min; 95°C to 85°C, -2°C/s; 85°C, 1 min; 85°C to 75°C, -0.3°C/s; 75°C, 1 min; 75°C to 65°C, -0.3°C/s; 65°C, 1 min; 65°C to 55°C, -0.3°C/s; 55°C, 1 min; 55°C to 45°C, -0.3°C/s; 45°C, 1 min; 45°C to 35°C, -0.3°C/s; 35°C, 1 min; 35°C to 25°C, -0.3°C/s; 25°C, 1 min) with the whole purified PCR sample. T7 Endonuclease I assay and gel electrophoresis Use the following set up for the T7 assay, always taking along a positive control (that will be provided by us): 100-200 ng amplicon (in 11 µl of 1xNEB2 buffer) 1 µl T7 endonuclease (in a 1:1 mixture with 1xNEB2) -----------------------------------------------------------------------------------12 µl total volume Incubate the reaction at 37°C for not longer than 15-20 minutes in a waterbath (don’t use heat block!). Stop the reaction with adding 2.4 µl 10x Orange G dye loading buffer and put sample on ice. Load the whole reaction on a 2.5% agarose gel, let run until gaining a good resolution and check the T7 endonuclease cuts by gel documentation. Quantify the degree of genome modification by ImageJ software. 111 Experiment GM3 Experiment Genmodifikation GM3: Entwicklung eines bivalenten “single round replication” Influenzabasierten Impfstoff-Kandidaten Dr. Veronika von Messling (4/0) 112 Experiment GM3 Verlaufsplan: Tag 1: - PCR, Restriktionsverdau - Aussaat von Zielzellen (MDCK) Tag 2: Tag 3: Transfektion von Vektorproduktionszellen (293T) - Transfer des Überstands auf Zielzellen, Titration - Gelreinigung des PCR Fragments, Dephosphorylierung des Vektors - Ligation und Transformation - Miniprep und Kontrollverdau - FACS Analyse der Zielzellen, Titration Versuchshintergrund und -ablauf: Influenzaviren gehören zur Familie der Orthomyxoviren. Sie sind von einer Plasmamembran umhüllt, welche von der Wirtszelle abstammt. Auf der Oberfläche des Viruspartikels befinden sich eingebettet in die Plasmamembran die viralen Proteine Hämagglutinin (HA) und Neuraminidase (NA). Ein weiteres Hüllprotein ist das M2 Protein, welches einen Ionenkanal durch die Plasmamembran formt. Das Matrixprotein M1 kleidet die virale Hülle von innen aus. Das Genom von Influenzaviren besteht aus 8 einzelsträngigen RNA-Segmenten negativer Polarität (Abb. 1). Abb. 1: Schematische Abbildung des Influenza A Virus (Medina et al., Influenza A Viruses: New research developments, Nature Reviews Microbiology 2011) 113 Experiment GM3 Nach der Infektion der Zielzelle transkribiert der virale Polymerasekomplex (Abb. 1) bestehend aus den Nukleokapsid (NP), PA, PB1, und PB2 Proteinen zunächst die mRNAs und anschließend die positivsträngige antigenomische RNA dieser Segmente. Diese antigenomische RNA dient als Vorlage zur Replikation negativsträngiger genomischer RNAs. Die de novo synthetisierten NP Proteine enkapsidieren sowohl die antigenomische als auch die genomische RNA, um eine Erkennung durch das angeborene Immunsystem zu verhindern. Die anderen Proteine des Polymerasekomplexes assoziieren mit der enkapsidierten genomischen RNA und bilden den Ribonukleoprotein-Komplex (RNP). Die RNPs werden dann an die Plasmamembran transportiert und assoziieren dort mit den viralen Hüllproteinen Hämagglutinin (HA), Neuraminidase (NA) und M2, sowie dem Matrixprotein (M) (Abb. 2). Abb. 2: Schematische Abbildung des Influenza A Virus-Lebenszyklus (Medina et al., Influenza A Viruses: New research developments, Nature Reviews Microbiology 2011) Ziel dieses Versuchs ist es, influenzavirale Vektoren herzustellen, die zusätzlich zu den beiden Hauptantigenen HA und NA ein weiteres Antigen exprimieren und die in ihrem Wirt nur einen einzigen Replikationszyklus durchlaufen können. Wir erwarten, dass diese Vektoren zusätzlich zu der von den momentan verwendeten inaktivierten Impfstoffen hervorgerufenen humoralen Immunantwort eine zelluläre Immunantwort gegen die Influenzaeigenen und -fremden Proteine induzieren. 114 Experiment GM3 Der experimentelle Ansatz basiert auf dem Ersatz des offenen Leserasters (ORF) des NP Gens im NP Segment eines bidirektionalen Vektors durch den ORF des zusätzlichen Antigens. Zur Herstellung rekombinanter Viren wird demnach zusätzlich zu den 8 bidirektionalen Vektoren, welche die Influenza-Gene kodieren, ein NP Expressionsplasmid kotransfiziert, so dass es zur Bildung von Viruspartikeln kommt, die das modifizierte NP Segment inkorporiert haben. In den Zielzellen kommt es zur Transkription der mRNAs und nachfolgenden Proteinsynthese von allen Segmenten, aber nicht zu einer Replikation der viralen genomischen RNA, da kein neues NP gebildet wird (Abb. 3). Abb. 3: Schematische Abbildung der Herstellung influenzaviraler Vektoren Im Rahmen des Praktikums wird dieser experimentelle Ansatz mit Hilfe eines NP Segments, das ein eGFP Reportergen trägt, validiert und mit einem replikationsfähigen eGFPexprimierenden Virus verglichen. Darüber hinaus wird das Hauptantigen des respiratorischen Synzytialvirus (RSV) in das genomische NP Plasmid kloniert. Dieses Virus führt häufig zu Koinfektionen mit Influenza, sodass ein Impfstoff, der gegen beide Viren schützt, von großem Vorteil wäre. 115 Experiment GM3 Vor dem Praktikum vorbereitet und durchgeführt: • Herstellung der Plasmid-DNA für die Transfektionen und Klonierung • Aussaat der als Vektorproduktionszellen genutzten 293T Zellen in 6-well Platten mit DMEM + 5% FCS • Vorbereitung von als Zielzellen genutzten MDCK Zellen in T75 Flaschen • Primersynthese • Herstellung kompetenter Bakterien für die Transformation ERINNERUNGEN! Im Labor müssen Kittel getragen werden. Für alle Experimente müssen Handschuhe getragen werden. Ein Kontakt mit Schleimhäuten muss vermieden werden. Alle Zellkulturarbeiten werden steril und unter der Werkbank durchgeführt. Versuchsdurchführung 1. Tag – Vektorvalidierung: Experiment 1: Transfektion der Vektorproduktionszellen mit Influenza-Plasmiden • 5x105 293T Zellen wurden in 6-well Platten am Tag zuvor ausgesät und werden zur Verfügung gestellt. Es werden jeweils 4 wells benötigt: - 2 für die Transfektion aller Plasmide - 2 für die Transfektion der Influenza-Plasmide ohne das NP Expressionsplasmid Generell wird die Transfektion nach dem folgenden Protokoll durchgeführt: in 2 sterile FACS-Röhrchen werden jeweils die folgenden Ansätze pipettiert: Ansatz A) Plasmid-DNA DMEM(ohne Zusätze) 2,0 µg 130 µl Ansatz B) PEI (Polyethylenimin, 18 mM) DMEM(ohne Zusätze) 8,0 µl 130 µl 116 Experiment GM3 zuerst wird der DNA-Mix vorbereitet: Medium vorlegen und anschließend die Plasmide hinzufügen danach wird Ansatz B vorbereitet und beide Lösungen werden kurz gut durchgemischt (Vortexer) nun gibt man Ansatz A zügig zu Ansatz B, wieder wird gut gemischt (Vortexer) und bei Raumtemperatur für 20 Minuten inkubiert nach der Inkubation wird der komplette Mix auf die Zellen gegeben Da Doppelansätze mit jeweils 9 bzw. 8 Plasmiden durchgeführt werden, wird ein Mastermix hergestellt und die Menge der entsprechenden Komponenten berechnet. Der Mastermix soll für drei Ansätze hergestellt werden (alle verwendeten Plasmide haben eine DNAKonzentration von 0,5 µg/µl): Vorbereitung des Mastermix für die Transfektion ALLER Plasmide: Lösung A: Plasmide PB1 PB2 PA HA NP Plasmid-Menge [µg] Entsprechendes Volumen [µl] DMEM (ohne Zusätze) : µl Lösung B: PEI: DMEM µl (ohne Zusätze) : µl Volumen Lösung A + B: Volumen/well: µl µl 117 GFP NA M NS Gesamt Experiment GM3 Vorbereitung des Mastermix für die Transfektion OHNE NP-Espressionsplasmid: Lösung A: Plasmide PB1 PB2 PA HA NP NA M Plasmid-Menge [µg] Entsprechendes Volumen [µl] DMEM (ohne Zusätze) : µl DMEM (ohne Zusätze): µl Lösung B: PEI: µl Volumen Lösung A + B: Volumen/well: µl µl Der Transfektionsmix wird wie folgt auf die Zellen (6 well Format) gegeben: Transfektion ALLER Plasmide 1 Transfektion OHNE NP-Plasmid 1 Kontrolle, nichttransfizierte Zellen 1 Transfektion ALLER Plasmide 2 Transfektion OHNE NP-Plasmid 2 Kontrolle, nichttransfizierte Zellen 2 Abb. 4 118 NS Gesamt Experiment GM3 Experiment 2: Aussaat von Zielzellen • Eine T75 Flasche mit MDCK Zellen wird für jedes Team zur Verfügung gestellt. MDCK Zellen sind Hundenierenzellen, die für die Infektion mit Influenza empfänglich sind. Für jede Transfektionsplatte wird eine 6-well und eine 96-well Platte benötigt. Aussaat der Zellen erfolgt nach dem folgenden Protokoll: Abnehmen des Mediums Anschließend Waschen der Zellen mit 5 ml PBS Hinzugabe von 3 ml Trypsin-EDTA auf die Zellen, Inkubation für 10-15 min im Brutschrank Zugabe von 7 ml DMEM (mit Zusätzen) (Endvolumen = 10 ml) Zellen in der T75-Flasche gut resuspendieren von der Zellsuspension werden 9 ml in ein 15 ml-Falcon-Röhrchen überführt und bei 2000 rpm für 5 min zentrifugiert (RT) (1ml bleiben in der Flasche und werden mit 9 ml Medium aufgefüllt) nach dem zentrifugieren wird der Überstand abgenommen und verworfen das Zellpellet wird in 3 ml DMEM (mit Zusätzen) resuspendiert zur Bestimmung der Zellzahl werden 10 µl der resuspendierten Zellen mit 90 µl Trypanblau verdünnt (1:10 Verdünnnung) von dieser Verdünnung werden 10 µl in eine Neubauer-Zählkammer gegeben und die Anzahl der lebenden Zellen mikroskopisch bestimmt dafür werden alle Zellen in allen vier Großquadraten gezählt (siehe Abb 5.) Großquadrat Abb. 5 anschließend wird die Anzahl der Zellen pro ml nach der folgenden Formel berechnet: 119 Experiment GM3 in die 6 well Platte werden 2 ml DMEM (mit Zusätzen) vorgelegt, anschließend werden 5 3x10 Zellen hinzugegeben: Zellzahl/ml =___________ 3x105 Zellen = ml 6 Zum Aussäen der 96 well Platte werden 1x10 Zellen in ein Reagenzreservoir pipettiert und auf 5 ml mit DMEM (mit Zusätzen) aufgefüllt: 1x106 Zellen = ml ad 5 ml DMEM = ml mit einer Multichannel Pipette wird diese Zellsuspension nun auf die 96 well Platte verteilt (50 µl/well) beide Platten werden anschließend im Brutschrank bei 37°C inkubiert 120 Experiment GM3 1. Tag – Klonierung von RSV G in genomisches NP Plasmid: Experiment 1: PCR Amplifikation des RSV G ORFs • Ein RSV G-haltiges Plasmid wird zur Verfügung gestellt. Zur Durchführung der PCR werden die folgenden Komponenten in den entsprechenden Konzentrationen in einem 0,2 ml PCR-Gefäß zusammen pipettiert: 10x Polymerase Puffer EndKonzentration 1x 10 mM Deoxynucleotid Mix (dNTP) 0.2 mM Forward primer (10 pmol/µl) 10 pmol Reverse primer (10 pmol/µl) 10 pmol DNA template ~ 100 ng Pfu-Polymerase 1.5 U Doppel-destilliertes Wasser ad 50 µl Komponenten DNA-Konzentration des templates: Menge [µl] µg/µl Als Negativkontrolle wird zusätzlich wird ein Ansatz angefertigt, welcher kein Template enthält. Entsprechend wird die Menge an Wasser (ad 50 µl) angepasst. Die PCR-Reaktion wird mit folgendem Programm durchgeführt: Schritt Temperatur Zeit Denaturation 94°C 2 min Denaturation 94°C 10 sec Primer annealing 55°C 30 sec Extension 72°C 2 min Extension 72°C 7 min Hold 4°C 121 25 x Experiment GM3 Experiment 2: Analytisches Agarosegel zur Überprüfung der PCR Zur Überprüfung ob die PCR funktioniert hat, werden 2 µl des PCR-Ansatzes auf ein Agarosegel aufgetragen. Für ein 1%-igen Agarose-Gels werden 1 g Agarose in 100 ml 1x TAE-Puffer durch kurzes Aufkochen in der Mikrowelle gelöst. Von dieser Agaroselösung werden 15 ml zur Polymerisierung des Gels in eine Kammer gegossen. Die DNA-Proben werden mit 6x DNA Ladepuffer gemischt, welcher GelRed zur Färbung der DNA enthält (2 µl einer 1:10-Verdünnung auf 500 µl 6x Lade-Puffer). Anschließend werden sie auf das Gel aufgetragen (12 µl Gesamt-Volumen) (Abb. 6). Als Standard wird DNA-Marker 1kb DNA Ladder von NEB verwendet. Das Gel läuft bei einer Spannung von 100 V für 20 min. Danach wird es unter UV-Licht analysiert. PCR-Probe : 2 µl Marker : 1 µl 6x DNA Ladepuffer: 2 µl 6x DNA Ladepuffer: 2 µl H2O H2O : 8 µl : 9 µl 1 kb DNA ladder, NEB TAE-Puffer (20x) Zusammensetzung: 1 L Menge: Reinstwasser Tris(hydroxymethyl)aminomethan Eisessig oder 50% Essigsäure EDTA 0,5M pH 8,0 ca.700ml Reinstwasser ad 1 L 96,8 g 22,84 ml 45,7 ml 40 ml Abb. 6 122 Experiment GM3 Experiment 3: Reinigung des PCR Fragments Reinigung der DNA erfolgt nach dem folgenden Protokoll (QIAquick PCR Purification Kit von Qiagen): 123 Experiment GM3 Experiment 4: Restriktionsverdau des PCR Fragments und des NP Vektors Restriktionsenzyme schneiden DNA an spezifischen Sequenzen, wobei je nach Enzym bei einem geraden Schnitt glatte Enden (blunt ends) oder bei einem versetzten Schnitt Überhänge (sticky ends) entstehen. Durch einen Restriktionsverdau des PCR-Fragments und des NP-Vektors mit den gleichen Enzymen entstehen kompatible Enden, die später miteinander ligiert werden können. Die beiden Restriktionsansätze werden wie folgt angesetzt: Verdau des PCR-Fragments Verdau des Vektors Komponenten Endkonzentration Menge [µl] Endkonzentration 10x BSA 1x 1x 10x Puffer 1x 1x Enzym 1 1,5 U 1,5 U Enzym 2 1,5 U 1,5 U DNA-Menge 30 µl PCR-Ansatz 2,5 µg H2O ad 50 µl ad 50 µl Menge [µl] Die Ansätze werden für 3 h oder über Nacht bei 37° C inkubiert und anschließend gelelektrophoretisch aufgetrennt und aufgereinigt oder bis zur weiteren Verwendung bei 20°C gelagert. 124 Experiment GM3 2. Tag – Vektorvalidierung: Experiment 1: Transfer der Influenza Partikel-haltigen Überstande auf Zielzellen für spätere FACS Analyse Infektion der Zielzellen in 6-well Platten erfolgt nach dem folgenden Protokoll: die Überstände der transfizierten 293T-Zellen werden in ein 15 ml Falcon überführt (gleiche Ansätze werden vereinigt) und für 3 min bei 1200 rpm und 4°C abzentrifugiert (Zentrifuge vorkühlen!) in dieser Zeit werden entsprechend viele Eppendorf-Tubes vorbereitet und beschriftet Überstand abnehmen und in Eppendorf-Tubes überführen, Tubes ständig auf Eis halten die 6 well Platte mit MDCK-Zellen wird 1x mit serumfreien Medium gewaschen: Überstände abnehmen und verwerfen, anschließend je 2ml serumfreies Medium/well auf die Zellen geben, Platte kurz schwenken, Medium wieder abnehmen und verwerfen in jedes well werden 1,5 ml TPCK-Medium vorgelegt (ohne FCS, mit 1 µg/ml Trypsin; wird von den Betreuern ausgegeben) anschließend werden jeweils 500 µl und 50 µl der virushaltigen Überstände bzw. Kontrolle auf die Zellen gegeben (siehe Abb. 7) und 24 – 48 h im Brutschrank inkubiert Überstand Überstand Überstand nichtTransfektion ALLER Transfektion transfizierter Zellen Plasmide OHNE NP-Plasmid (Kontrolle) 500 µl 500 µl 500 µl 50 µl 50 µl 50 µl Abb. 7 Experiment 2: Titration der produzierten Influenza Partikel 125 Experiment GM3 Die Virus-Titration ist eine Methode zur Quantifizierung der infektiösen Dosis (Virus-Titer). Dazu wird eine Verdünnungsreihe des Virus angefertigt, welche anschließend auf ausgesäte MDCK-Zellen transferiert wird (96-Well Platten). Wichtig ist, dass nach jedem Verdünnungsschritt die Pipettenspitze gewechselt wird. Diese “Limited dilution” Titration erfolgt nach dem folgenden Protokoll (Abb. 8 A): für eine Verdünnungsreihe von 10-1 bis 10-8 in Zehnerschritten werden 450 µl DMEM(ohne Zusätze) in einer 24-well Platte vorgelegt in das erste well werden 50 µl Influenza Partikel-haltiger Überstand hinzugefügt und gemischt aus dieser Verdünnung werden anschließend 50 µl in das nächste well transferiert dieser Vorgang wird fortgesetzt, bis man 8 Verdünnungen hergestellt hat (1:10 bis 1:108) B A Abb. 8 Anschließend werden die Verdünnungen auf die 96-Well Platte mit ausgesäten MDCKZellen überführt, wobei 4 Replikate von jeder Verdünnungsstufe mit je 100 µl/Well angefertigt werden (Abb. 8 B). Damit immer die gleiche Spitze verwendet werden kann, fängt man mit der höchsten Verdünnungsstufe an. Die fertige 96-well-Platte wird 24 – 48 h im Brutschrank bei 37°C inkubiert. 126 Experiment GM3 2. Tag – Klonierung von RSV G in genomisches NP Plasmid: Experiment 1: Agarose Gelreinigung des verdauten PCR Fragments Sowohl das verdaute PCR-Produkt als auch der verdaute Vektor (Tag 1, Exp. 4) werden jeweils auf ein 1 % Agarose-Gel geladen (Anfertigung des Gels und Marker siehe Tag 1, Exp. 2). Dabei werden die kompletten Ansätze (50 µl) mit je 10 µl 6x DNA Loading Puffer gemischt und wie folgt auf das Gel aufgetragen: Abb. 9 Das Gel läuft bei einer Spannung von 100 V für 20 min. Als Kontrolle lässt man den unverdauten Vektor mitlaufen (0,5 µl Vektor, 2 µl 6x Loading Puffer, 9,5 µl H2O). Bevor man das Gel unter UV-Licht analysiert, wird es mit einem Skalpell an den gekennzeichneten Stellen geschnitten (Abb. 9). Anschließend wird lediglich der Teil des Gels unter UV-Licht gelegt, welcher den Marker und den 2 µl-Ansatz, bzw. den unverdauten Vektor enthält und der Abstand zwischen DNA-Bande und Tasche mithilfe eines Lineals bestimmt. Dieser Abstand wird dann ohne UV-Licht auf das Gelstück übertragen, welches den 2 x 29 µlAnsatz enthält, da man an dieser Stelle die entsprechende Bande vermutet. Die Bande wird mithilfe eines Skalpells ausgeschnitten, in ein 2 ml-Eppendorf-Tube überführt und die DNA mithilfe des folgenden Protokolls isoliert (QIAquick® Gel Extraction Kit von Qiagen): 127 Experiment GM3 128 Experiment GM3 129 Experiment GM3 Experiment 2: Reinigung und Dephosphorylierung des verdauten Vektors Um eine Selbstligation des Vektors nach einem Restriktionsverdau zu verhindern, werden die 5‘-Enden enzymatisch dephosphoryliert. Der Dephosphorylierungs-Ansatz wird wie folgt angesetzt: Komponenten Endkonzentration 10x Puffer 1x Alkaline Phosphatase (1 U/µl) 1U Vektor-Menge 8 µl End-Volumen 10 µl Menge [µl] 10 µl Der Reaktionsansatz wird 1 Stunde bei 37° C inkubiert und anschließend für 10 min bei 65° C inaktiviert. Experiment 3: Ligation des PCR-Produkts in den Vektor Für die Ligation werden das Insert (verdautes PCR-Produkt) und der verdaute, dephosphorylierte Vektor in einem Verhältnis von 7 : 1 eingesetzt (End-Volumen 20 µl). Es wird das Quick Ligation Kit von NEB verwendet. Ligations-Ansatz: Komponenten Endkonzentration 2x Ligase-Puffer 1x Ligase 1 µl Insert 7 µl Vektor 1 µl H2O ad 20 µl Menge [µl] Wichtig ist, dass der Ligations-Puffer auf Eis aufgetaut wird. Der Ansatz wird für 5 Minuten (!) bei Raumtemperatur inkubiert, anschließend kurz auf Eis gestellt und entweder direkt zur Transformation in Bakterien genutzt oder bei -20°C gelagert. 130 Experiment GM3 Experiment 4: Transformation der kompetenten Bakterien Der Ligations-Ansatz wird in chemisch kompetente E.Coli-Bakterien transformiert. Vor der Transformation wird folgendes vorbereitet: • Wasserbad auf 42°C vorwärmen • SOC-Medium auf Raumtemperatur vorwärmen • Thermoblock auf 37°C vorwärmen • Agar-Platte auf 37°C vorwärmen Anschließend erfolgt die Transformation der Ligation nach dem folgenden Protokoll: Bakterien auf Eis (!) auftauen lassen den kompletten Ligations-Ansatz (20 µl) hinzugeben 20 Min auf Eis inkubieren den Ansatz für 35 sec bei 42°C im Wasserbad inkubieren Hitzeschock danach sofort für 5 min auf Eis stellen 250 µl vorgewärmtes SOC-Medium (Raum-Temperatur) hinzu geben Inkubation des Ansatzes für 1 Stunde bei 37°C und 200 rpm im Thermoblock danach wird die Bakteriensuspension auf einer Agarplatte (LB Amp-Platte mit 50 µg/ml Ampicillin ) ausplattiert Inkubation über Nacht bei 37° C 3. Tag – Vektorvalidierung: Experiment 1: Vorbereitung der Zellen zur FACS Analyse Ablösung und Fixierung der Zellen erfolgt nach dem folgenden Protokoll: das Medium wird abgenommen und verworfen die Zellen werden mit 2 ml/well PBS gewaschen Zugabe von 0,5 ml Trypsin-EDTA/well Inkubation im Brutschrank, bis Zellen sich abgelöst haben (~ 5 – 10 min) Zugabe von 1,5 ml DMEM (+5% FCS, +1% Glu) 131 Experiment GM3 Transfer der Zellsuspension in ein 15 ml Falcon Zentrifugation bei 1200 rpm, 3 min der Überstand wird abgenommen und verworfen das Pellet wird in 2 ml FACS Wash buffer (PBS, 1% FCS, 0.1% NaN3) resuspendiert und erneut abzentrifugiert (1200 rpm, 3 min) Wiederholung dieses Waschschrittes der Überstand wird abgenommen und verworfen das Zellpellet wird in 200 µl FACS Fix (PBS, 1% Paraformaldehyd) resuspendiert und in ein FACS-Röhrchen transferiert die Lagerung dieser fixierten Probe erfolgt bei 4°C im Dunkeln Experiment 2: FACS Analyse der Proben • Fixierte Kontrollproben von MDCK Zellen, die mit einem eGFP-exprimierenden replikationskompetenten Influenzavirus infiziert wurden, werden zur Verfügung gestellt. Die FACS Analyse erfolgt mit dem FACS-Gerät Accuri von BD. Es werden 10 000 Zellen eingelesen und der entsprechende Prozentsatz GFP-positiver Zellen ermittelt: Transfektion Transfektion Transfektion Transfektion ALLER ALLER OHNE NP- OHNE NP- Plasmide, Plasmide, Plasmid, Plasmid, 500 µl 50 µl 500 µl 500 µl GFP [%] 132 Untransfiziert Untransfiziert e Zellen, e Zellen, 500 µl 500 µl Experiment GM3 Experiment 3: Auswertung der Titration Der Virus-Titer [TCID50/ml] wird nach der Methode von Spearman und Kärber berechnet. Dabei erfolgt die Berechnung des dekadischen Logarithmus, bei der 50% der Ansätze einen zytopathischen Effekt (CPE) bzw. grüne Zellen aufweisen, nach folgender Formel: xk = d= p= ∑p = dekadischer Logarithmus der höchsten Verdünnungsstufe, bei der noch in allen Ansätzen ein CPE bzw. grüne Zellen sichtbar sind dekadischer Logarithmus des Verdünnungsfaktors Anteil an CPE positiven Wells bzw. Wells mit grünen Zellen pro Verdünnungsstufe Summe aller p ab der Verdünnungsstufe xk und den p-Werten aller höheren Verdünnungen mit beobachtetem CPE bzw. grünen Zellen Im Praktischen heißt das, es werden auf der 96 well-Titrationsplatte alle wells markiert, in denen grüne Zellen sichtbar sind, ein Beispiel ist in Abb. 10 gezeigt: Abb. 10 Anschließend wird ermittelt, welches die letzten Verdünnungsstufen sind, in denen man noch grüne Zellen erkennen kann. Dabei wird die Anzahl der Replikate (hier: 4) mit bedacht. In diesem Beispiel sind noch in zwei Replikaten der Verdünnung 1:105 und in allen vier Replikaten der 1:104 Verdünnung grüne Zellen detektierbar. In der folgenden Tabelle kann man nun anhand dieser Informationen den Titer ablesen, welcher in diesem Beispiel 1x106 TCID50/ml ist: 133 Experiment GM3 Letzte Markierungen in den Titer Verdünnungsstufen Verdünnungsstufe [TCID50/ml] Replikat 1 2 3 10-2 x x x 10-2 x x x x 10-2 10-3 x x x x 10-3 10-3 x x x x x 10-3 x x x x 10-3 x x x x -4 x 10-3 x x x x -4 x x 10-4 x x x 10-4 x x x x 10-4 x x x x 10 -5 x 10 -4 x x x x 10-5 x x 10-5 x x x 10-5 x x x x 10-5 x x x x -6 x 10-5 x x x x -6 x x 10 10 10 10 4 1,5 x 103 x 134 3,0 x 103 5,0 x 103 1,0 x 104 1,5 x 104 3,0 x 104 5,0 x 104 1,0 x 105 1,5 x 105 3,0 x 105 5,0 x 105 1,0 x 106 1,5 x 106 3,0 x 106 5,0 x 106 1,0 x 107 Experiment GM3 Ergebnisse der Titration der produzierten Influenza-Partikel: Transfektion ALLER Plasmide Transfektion OHNE NPPlasmid Untransfizierte Zellen Titer [TCID50/ml]: 3. Tag – Klonierung von RSV G in genomisches NP Plasmid: Experiment 1: Miniprep der erhaltenen Kolonien Anzahl der erhaltenen Kolonien: ________ Bereits vorbereitet: • Für jede Ligation wurden 6 Kolonien für Minipreps gepickt und in 5 ml LB Medium mit 0,1 mg/ml Ampicillin über Nacht bei 200 rpm und 37ºC inkubiert. Die DNA Isolierung erfolgt nach dem folgenden Protokoll (QIAprep® Spin Miniprep Kit von Qiagen): 135 Experiment GM3 136 Experiment V1 Experiment Virologie V1: Einfluss des Hepatitis-C-Virus auf die Nrf2/ARE-abhängige Genexpression Prof. Dr. Eberhard Hildt 137 Experiment V1 Einleitung Hepatitis-C-Virus (HCV) Das Hepatitis-C-Virus ist ein einzelsträngiges (+)-Strang-RNA-Virus, das zur Familie der Flaviviridae gehört und dort das Genus der Hepaciviren bildet. Die Analyse der RNA-Sequenzen führte zur Gruppierung der Isolate in 6 verschiedene Genotypen und 30 Subtypen. Die Genotypen und Subtypen zeigen eine unterschiedliche geographische Verteilung. Die Infektion mit HCV kann neben einer akuten Leberentzündung (Hepatitis) zu einer chronischen Infektion führen, aus der heraus eine Leberzirrhose und schließlich ein hepatozelluläres Karzinom (HCC) entstehen können (Tsai et al., 2010; Castello et al., 2010). Gegenwärtig sind ca. 170 Mio. Menschen weltweit mit Hepatitis-C-Virus (HCV) infiziert, eine effiziente Therapie steht jedoch nicht zur Verfügung. Umso wünschenswerter wäre das Vorhandensein eines Impfstoffs gegen HCV, der jedoch nicht verfügbar ist. Das ca. 9.6 kB große (+)-Strang-RNA-Genom kodiert für ein ca. 3.000 Aminosäuren großes Polyprotein. Die Prozessierung des Polyproteins erfolgt durch virale und wirtseigene Proteasen, sodass ausgehend von dem Vorläuferpolyprotein 10 verschiedene HCV-Proteine gebildet werden. Diese können in zwei Gruppen unterteilt werden, zum einen in die Strukturproteine (E1, E2 und Core) und zum anderen in die sog. Nichtstrukturproteine (NS) (Bartenschlager et al., 2004; Giannini et Brechot 2003; Penin et al., 2004). Bei den Proteinen E1 und E2 handelt es sich um Glykoproteine, die in die Membranhülle des Virus eingelagert sind. Das ca. 20 kDa große Core-Protein erfüllt neben seiner strukturellen Funktion auch regulatorische Aufgaben, indem es Signalkaskaden moduliert. Durch einen alternativen open reading frame kann von dem für Core codierenden Bereich das sog. F-Protein gebildet werden, dessen molekulare Funktion jedoch unklar ist (Branch et al., 2005). Die Nichtstrukturproteine von HCV nehmen sehr unterschiedliche Funktionen wahr. Das kleine Protein p7 kann in der Wirtszelle Ionenkanäle bilden (Griffin et al., 2003, Pavlovic et al., 2003; Luik et al., 2009), deren Funktion für die Virusreplikation noch 138 Experiment V1 weitgehend ungeklärt ist. Bei NS2 handelt es sich um eine Metalloprotease, die sich autokatalytisch vom Polyprotein abspaltet (Santolini et al., 1995; Yamaga et al., 2002). Das ca. 70 kDa große NS3 verfügt neben der in der N-terminalen Region lokalisierten Serin-Proteaseaktivität (Kim et al., 1996) auch über eine Helikasefunktion (Tai et al., 1996). Das NS5B-Protein ist eine RNA-abhängige RNAPolymerase (Lesburg et al., 1999, Bressanelli et al., 2002). Dem NS5A-Protein werden regulatorische Funktionen zugeschrieben (Gale et al., 1998; MacDonald and Harris, 2004; Burckstummer et al., 2006, Himmelsbach et al., 2009). Bis vor kurzem war es nicht möglich, signifikante Mengen an infektiösen HCVPartikeln rekombinant zu erzeugen. Ein wesentliches Modellsystem zur Charakterisierung der Virus-Zell-Interaktion stellen sogenannte virus like particles (VLPs; Baumert et al., 1999) bzw. HCV pseudo particles dar (Bartosch et al., 2003). Bei letzteren handelt es sich um chimäre Retroviren, die an ihrer Oberfläche die HCV-Hüllproteine E1/E2 tragen. Aufbauend auf diesen Systemen konnten CD81, der Scavenger-Rezeptor SR-B1, der LDL-Rezeptor, hoch sulfatiertes Heparansulfat sowie L-SIGN/DC-SIGN, Claudin-1 und Occludin als HCV-bindende Proteine identifiziert werden, die als Rezeptoren bzw. Teile des Rezeptorkomplexes fungieren könnten (Bartosch et Cosset, 2006; Barth et al. 2006, Ploss et Rice, 2009, Harris et al., 2010). Ein wesentlicher Durchbruch für die Untersuchung des Lebenszyklus von HCV war die Etablierung eines Systems, das die Analyse des kompletten Lebenszyklus in Zellkultur ermöglicht. Ausgehend von einem HCV-Genotyp 2a-Isolat eines Patienten mit einem fulminanten Verlauf konnten drei Arbeitsgruppen ein replikationsfähiges HCV-Genom (JFH-1) etablieren (Wakita/Pietschmann et al., 2005; Zhong et al., 2005; Lindenbach et al., 2005). Der JFH-1-Klon repliziert in Zellkultur, ohne dass Zellkultur-adaptive Mutationen erforderlich wären. Interessanterweise erlaubt dieses System nach Transfektion des JFH-1-RNA-Genoms oder einer Chimäre, welche die Core- bis NS2-Region des Genotyps 1b (Wakita/Pietschmann et al. 2005) oder 2a (Zhong et al., 2005) enthält, die Bildung infektiöser HCV-Partikel, die dann wiederum in Zellkultur Huh-7.5-Zellen (humane Schimpansen infizieren können. 139 Hepatozyten-Zelllinie) oder in vivo Experiment V1 Die Infektion mit HCV kann akute und chronische Lebererkrankungen verursachen. Bei etwa 25 % der HCV-Infektionen kommt es zum Auftreten einer akuten Hepatitis, die allerdings zumeist recht mild verläuft. In 50 bis 80 % der Erkrankungsfälle nimmt die Hepatitis C einen chronischen Verlauf (Seef, 2002), gekennzeichnet durch eine Viruspersistenz von mehr als 6 Monaten. Bei ca. 20 % der Patienten mit chronischer Hepatitis C entwickelt sich eine Leberzirrhose (Tsai et Chung, 2010, Castello et al., 2010). Vom Zeitpunkt der Infektion bis zum Vollbild der Zirrhose vergehen zumeist 20 bis 30 Jahre. Die WHO geht von etwa 170 Millionen HCV-Infektionen weltweit aus, es sind also etwa 3 % der Weltbevölkerung betroffen (Poynard et al., 2003). Im Unterschied zu vielen anderen Infektionskrankheiten ist HCV auch ein Problem in westlichen Industrienationen, so z. B. den USA und Deutschland (Prävalenz von 1,8 % in den USA und 0,4 % in Deutschland (Higuchi et al., 2002)). Gegenwärtig basiert die Therapie der HCV-Infektion auf einer Kombination von pegyliertem Interferon-α und Ribavirin (DiBisceglie et Hoofnagle, 2002, Ferenci, 2006, Lagging et al., 2009, Rong et al., 2010). Allerdings ist die Effizienz dieses Ansatzes bei gleichzeitig hohen Kosten sehr begrenzt. Die Entwicklung von Protease-Inhibitoren zur Hemmung der viralen Nichtstrukturproteine NS3/4 verspricht eine effizientere Heilung der Infektion in naher Zukunft (Kronenberger and Zeuzem, 2012). Die Genomreplikation von HCV erfolgt unter Beteiligung der Nichtstrukturproteine (NS) auf der zytoplasmatischen Seite des endoplasmatischen Retikulums (ER) in sog. Replikonkomplexen. Wichtig hierbei ist, dass die Genom-Replikation auch ausgehend von subgenomischen Konstrukten, denen die Strukturproteine Core, E1, E2 komplett oder teilweise fehlen, untersucht werden kann. Durch die „Beladung“ des ER mit diesen Replikonkomplexen kommt es zu starken strukturellen Veränderungen, das sogenannte membranous web bildet sich. Für den blauf der Virusreplikation ist es wichtig, dass die de novo gebildeten viralen Genome (gebildet durch NS5B, eine RNA-abhängige RNA-Polymerase) effizient verpackt werden. Dazu werden die neu gebildeten Genome von NS5A gebunden und vom Ort der Genomreplikation zu den in unmittelbarer Nähe befindlichen lipid droplets (LDs) 140 Experiment V1 transportiert. Die LDs sind die Schlüsselorganellen für die Morphogenese. Es handelt sich dabei um kleine intrazelluläre Lipidtröpfchen, die an ihrer Oberfläche mit dem HCV-Core-Protein bedeckt sind. Core und NS5A induzieren die Bildung dieser LDs. Über seine RNA-Bindungsdomäne transportiert NS5A nun die RNA zu den LDs, wo sie mit dem Core-Protein interagieren und wo der Zusammenbau des Capsids beginnt. Anschließend erfolgt die Interaktion mit den Hüllproteinen E1 und E2. Der Export von HCV weist einige Homologien zum Export von Lipoproteinen, insbesondere VLDL (very low density lipoprotein), auf. Dies führt dazu, dass das reife Viruspartikel als Lipoproteinpartikel zu beschreiben ist, was eine strukturelle Untersuchung sehr erschwert. Der Transkriptionsfaktor Nrf2 Der Cap'n'Collar basic leucine zipper Transkriptionsfaktor Nrf2 (NF-E2 related factor) ist ein essentieller Faktor bei der Modulation einer Vielzahl entzündlicher Prozesse (Beyer et Werner, 2008, Kensler et al, 2007). Dabei liegt Nrf2 in seinem inaktiven Zustand an Keap1 gebunden im Zytosol vor und wird sehr rasch degradiert. Im Zuge der Aktivierung von Nrf2 dissoziiert der Nrf2/Keap1-Komplex, Nrf2 wird stabilisiert und wandert in den Zellkern (Tong et al., 2007). Dort interagiert es mit anderen Proteinen, z. B. sMaf-Proteinen, und bindet an die Promotorelemente von Genen, die ein sog. antioxidant response element (ARE) enthalten. Für die Funktionalität von Nrf2 ist die Interaktion mit sMafs im Nukleus essentiell, erst dann kann die Genexpression Nrf2/ARE-abhängig regulierter Gene induziert werden. AREs finden sich in den Promotorelementen von Genen, die antioxidative Enzyme kodieren, wie die katalytische Untereinheit der Glutamat-Cystein-Ligase (GCLC/GCS), GlutathionPeroxidase (GPx), Heme-Oxygenase-1 (HO-1), Thioredoxin-Reductase (TrxR) (Osburn et al., 2007, Kensler et al., 2007) oder Phase II Enzyme des Detoxifizierungsweges wie NAD(P)H: quinone oxidoreductase 1 (NQO1), Isoenzyme der GlutathionS-transferase (GST) oder UDP-Glucuronosyltransferase (UGT) (Lau et al., 2008). Durch die Induktion der antioxidativen Enzyme wird die Sensitivität gegenüber Oxidantien oder ROI (reactive oxygen intermediates) vermindert. Darüber hinaus weisen auch die Promotoren verschiedener für Proteasomuntereinheiten 141 Experiment V1 kodierender Gene ARE-Sequenzen auf und können Nrf2-abhängig induziert werden (Kwak et al., 2003, 2006). Für die Induktion von HO-1 konnte ein inhibierender Effekt auf die HBV-Replikation beobachtet werden (Protzer et al., 2008). Aufgrund der Rolle von Nrf2 bei der Induktion einer Reihe zytoprotektiver Gene wurde Nrf2 in erster Linie eine Schutzfunktion gegenüber genotoxischen Substanzen, die als Karzinogen wirken können, zugeschrieben (Yates et al., 2006; Kwak et al., 2002). Neuere Arbeiten zeigten aber, dass Nrf2 auch eine wesentliche Rolle beim Schutz von Tumorzellen spielt und somit die Entstehung von Tumoren fördern kann. Ein Beispiel ist die starke Induktion von Nrf2 und ARE – abhängig von GSTP1 – bei der HCC-Entstehung (Lau et al., 2008, Ikeda et al., 2004). Nrf2 ist essentiell für die Leberregeneration (Beyer et al., 2008). Interessanterweise bedingt HCV im Unterschied zum Hepatitis-B-Virus (HBV) (Schaedler et al., 2010) eine Hemmung der Induktion von Nrf2, indem sMaf aus dem Zellkern an den Replikonkomplex transloziert wird (Carvajal-Yepes et al. 2011). Im Rahmen des Praktikums soll nun der Einfluss von HCV auf die Nrf2/AREabhängige Genexpression analysiert werden. Um ein gefahrloses Arbeiten zu ermöglichen (gegen HCV gibt es bisher keine Impfung), werden wir im Rahmen des Praktikums mit einem subgenomischen Konstrukt arbeiten, das die Replikation, aber nicht die Synthese von Viren ermöglicht. Dem Konstrukt fehlen die Sequenzen, die für die Hüllproteine kodieren (∆E1E2). Auf der Basis dieses subgenomischen HCV-Replikons (∆E1E2) sollen folgende Aspekte untersucht werden: • Interferenz von HCV mit der Expression Nrf2/ARE-abhängig regulierter Gene mittels Reportergen-Assay • Einfluss von HCV auf die subzelluläre Lokalisation von sMaf mittels konfokaler Immunfluoreszenzmikroskopie • Einfluss von HCV auf den intrazellulären ROI-Spiegel und auf zytoprotektive Mechanismen, die vor ROI-induzierten Schäden schützen (Oxyblot-Analyse) 142 Experiment V1 Lerninhalte Herstellung der Replikon-RNAs mittels in vitro-Transkription Zellkulturarbeiten Elektroporation zur Generierung der Replikonzellen • Fixierung und Färbung für Immunfluoreszenz (IF) • konfokale Laser-Scan-Mikroskopie (CLSM) • Transfektion mit Reportergenkonstrukten; Durchführung von ReportergenAssays • Immunblot-Analysen (Western Blot-Analyse zur Detektion HCV- spezifischer Proteine, Oxyblot-Analyse) Zeitplan Tag 1 (Montag) Linearisieren der Plasmide für die in vitro run-off T7-Transkription, Elektroporation. Tag 2 (Dienstag) Transfektion der Zellen mit dem Luciferase-Reporter-Konstrukt (pNQO1) sowie tBHQ-Stimulation (Positivkontrolle für den Luciferase-Assay), Lyse der Zellen für Oxyblot- und Western Blot-Analysen (Lysate werden vom Betreuer gestellt) und Proteinbestimmung mittels Bradford-Assay, Beladen der Gele für Oxyblot- und Western Blot-Analysen, blotten und anschließendes Färben der Membranen. Tag 3 (Donnerstag) Fixierung der Zellen für die Immunfluorseszenz (12-Loch-Platte) und anschließende Immunfärbung sowie Auswertung am CLSM (konfokale Laser-Scan-Mikroskopie), H2O2-Stimulation der Zellen für die Oxyblot-Analyse (für die nächste Gruppe), Lyse der Zellen für Oxyblot- und Western Blot-Analysen, Luciferase-Assay. 143 Experiment V1 144 Experiment V1 Praktischer Teil Tag 1 In vitro run-off T7-Transkription Mittels in vitro run-off T7-Transkription sollen das subgenomische HCVReplikon JFH1/ΔE1E2 und das replikationsdefiziente Replikon JFH1/GND hergestellt werden. Durch Elektroporation sollen Huh-7.5-Zellen mit diesen Konstrukten transfiziert werden, sodass in den Zellen im Falle von JFH1/ΔE1E2 die Replikation erfolgen kann. DNA-abhängige RNA-Polymerasen können doppelsträngige DNA-Sequenzen in ihre komplementäre RNA transkribieren. Hierfür werden vorzugsweise Bakteriophagensysteme genutzt (Milligan et al.1987). Bei der T7-Transkription erkennt die aus den gleichnamigen Bakteriophagen isolierte DNA-abhängige RNA-Polymerase spezifisch 17 Basenpaare, den T7-Promotor. Als Initiationsnukleotid des RNA-Transkripts ist Guanosin (G) essentiell. Die Effizienz der Polymerase wird gesteigert, indem zwei weitere Guanosine folgen. In vitro handelt es sich um eine run-off Transkription, d. h. die RNA-Polymerase dissoziiert am 3´-Ende der DNA. Die virale RNA kann nun durch T7-Transkription aus linearisierten HCV-ReplikonPlasmiden (mit T7-Promotor) gewonnen werden. Diese RNA wird nach Reinigung durch Phenol/Chloroform-Extraktion für die Elektroporationsexperimente genutzt. Im Rahmen des Praktikums sollen das subgenomische HCV-Replikon JFH1/ΔE1E2 und das replikationsdefiziente Replikon JFH1/GND hergestellt werden. Da RNasen ubiquitär vorkommen, ist es besonders wichtig, bei der Arbeit mit RNA Handschuhe zu tragen und sie auch regelmäßig zu wechseln. Darüber hinaus sollten RNA-haltige Lösungen stets auf Eis gelagert werden, und es sollte nur mit RNase-freien Materialien bzw. DEPC-behandelten Lösungen gearbeitet werden. 145 Experiment V1 Durchführung Linearisieren der Plasmide für die in vitro T7-Transkription Hierfür werden 40 µg Plasmid-DNA (JFH1/GND und JFH1/∆E1E2; werden vom Betreuer gestellt) mit 40 U XbaI (NEBuffer4, 1 x BSA) für 2 h bei 37°C inkubiert. Die linearisierten Plasmide werden im Anschluss mittels Phenol/Chloroform-Extraktion extrahiert. Phenol/Chloroform-Extraktion Zur Reinigung und Extraktion von Nukleinsäuren aus proteinhaltiger Lösung wird die Phenol/Chloroform-Extraktion verwendet. Das Prinzip beruht auf den verschiedenen Löslichkeiten von Proteinen und Nukleinsäuren in wässriger und organischer Phase. Das Chloroform wird zur Reinigung von Phenolresten verwendet. Die DNA/RNA in der wässrigen Phase kann letztendlich mit Ethanol/Isopropanol gefällt werden. Die Arbeiten mit Phenol und Chloroform werden nur mit Nitril-Handschuhen und Schutzbrille unter dem Abzug durchgeführt. • Der Ansatz wird mit 40 µl 3 M Natriumacetat (pH 5,2) versetzt und mit ddH2O auf ein Volumen von 400 µl gebracht. • Zugabe von 400 µl Phenol, vortexen und Zentrifugation für 5 min bei 13.300 rpm und 4°C. • Die wässrige obere Phase wird vorsichtig abgenommen und mit 400 µl Chloroform in einem neuen 1,5 ml Reaktionsgefäß kurz gevortext und wieder 5 min bei 13.300 rpm und 4°C zentrifugiert. • Die wässrige (obere) Phase vorsichtig abnehmen und in ein neues Reaktionsgefäß (gut beschriften) überführen. • Zugabe des 2,5-fachen Volumens Ethanol (100 %) (vorsichtig schwenken). • Pelletieren der DNA durch Zentrifugation bei 13.300 rpm und 4°C für 30 min. • Den Überstand dekantieren (verwerfen). 146 Experiment V1 • Das Pellet mit 75 % eiskaltem Ethanol waschen (Zentrifugation bei 13.300 rpm und 4°C für 10 min). • Den Überstand dekantieren und das Pellet lufttrocknen. • Das Pellet in ca. 30 µl DEPC ddH2O aufnehmen und die Konzentration (1:100 Verdünnung) photometrisch bestimmen (siehe unten). In vitro run-off T7-Transkription Pro Ansatz 4-5 µg linearisierte Plasmid-DNA einsetzen. In vitro Transkriptionsansatz 5 µg DNA 8 µl Transkriptionspuffer (10x) 3,2 µl NTP-Mix (10mM) 2 µl RNase Out 4 µl T7-Polymerase ad 80 µl DEPC-ddH2O Der Transkriptionsansatz wird 2 h bei 37°C inkubiert. Durch Zugabe von 3 μl DNaseI und Inkubation für 1 h bei 37°C wird die template-DNA degradiert und somit die Transkription gestoppt. Die RNA wird mittels Phenol/Chloroform-Extraktion extrahiert. Phenol/Chloroform-Extraktion • Der Ansatz wird mit 40 µl 3 M Natriumacetat (pH 5,2) versetzt und mit DEPC ddH2O auf ein Volumen von 400 µl gebracht. • Zugabe von 400 µl Phenol, vortexen und Zentrifugation für 5 min bei 13.300 rpm und 4°C. • Die wässrige obere Phase wird vorsichtig abgenommen und mit 400 µl Chloroform in einem neuen 1,5 ml Reaktionsgefäß kurz gevortext und wieder 5 min bei 13.300 rpm und 4°C zentrifugiert. 147 Experiment V1 • Die wässrige (obere) Phase vorsichtig abnehmen und in ein neues Reaktionsgefäß (gut beschriften) überführen. • Zugabe des 0,7-fachen Volumens Isopropanol (100 %) (vorsichtig schwenken). • Pelletieren der RNA durch Zentrifugation bei 13.300 rpm und 4°C für 30 min. • Den Überstand dekantieren (verwerfen). • Das Pellet mit 75 % eiskaltem Ethanol waschen (Zentrifugation bei 13.300 rpm und 4°C für 10 min). • Den Überstand dekantieren und das Pellet lufttrocknen. • Das Pellet in ca. 30 µl DEPC ddH2O aufnehmen und die Konzentration (1:100 Verdünnung) photometrisch bestimmen. Jeweils 1 µl der RNA-Lösung wird für die Qualitätsbestimmung im Agarosegel verwendet (siehe unten). Konzentrationsbestimmung am Photometer Die Konzentration von Nukleinsäuren lässt sich photometrisch bei einer Wellenlänge von 260 nm bestimmen. Eine Extinktion von 1 entspricht einer Konzentration von 50 μg/ml doppelsträngiger DNA bzw. 35 μg/ml einzelsträngiger DNA oder RNA und folgt dem Lambert-Beer´schen Gesetz. Lambert-Beer’sches Gesetz A=εxcxd A = Absorption ε = Extinktionskoeffizient c = Konzentration d = Schichtdicke der Küvette Das Verhältnis der OD260 zur OD280 zeigt an, wie stark eine nukleinsäurehaltige Lösung durch Reste von Proteinen verunreinigt ist und sollte zwischen 1,8 und 2,0 liegen. Geringere Werte spiegeln eine Kontamination durch Proteine wider. 148 Experiment V1 Lassen Sie sich vor Beginn der Messung die Handhabung des Photometers vom Praktikumsleiter erklären. • Photometer ca. 15 min vor Benutzung anschalten. • Von der zu messenden Probe (linearisierte Plasmid-DNA bzw. RNA) wird eine 1:50 Verdünnung hergestellt, für den Nullwert werden 100 µl DEPC ddH2O vorbereitet. • Im Photometer wird das DNA- bzw. RNA-Programm gewählt und die Verdünnung eingestellt. • Die Messung erfolgt in Quarzküvetten. • Zunächst wird mit 100 µl DEPC ddH2O eine Nullwerteistellung vorgenommen. Im Anschluss wird die DNA gemessen. Qualitätsbestimmung von RNA mittels Agarose-Gelelektrophorese Der Degradationsgrad der RNA kann anhand eines Agarosegels verifiziert werden. • Hierzu wird eine 1 %-ige Agarose-Lösung (in 1 x TAE) hergestellt und in der Mikrowelle aufgekocht (bis die Agarose vollständig gelöst ist). • Nach dem Abkühlen der Lösung (handwarm) wird diese in eine horizontale Gelkammer gegossen, und es werden ca. 2 µl Ethidiumbromid hinzugegeben und verteilt. (Ethidiumbromid steht unter Verdacht, karzinogen zu sein, sodass hier nur mit Nitrilhandschuhen gearbeitet werden darf!!!) • Je 1 µl RNA wird mit 5 µl DEPC ddH2O und 2 µl 6 x Ladepuffer gemischt und auf das Gel aufgetragen (Vorsicht, die Gelkammer sollte gut gewaschen sein, um eine Hydrolyse der RNA zu vermeiden). • Die Separation der RNA im elektrischen Feld findet bei 100 V statt. • Die RNA kann unter dem UV-Transilluminator bei 245/365 nm visualisiert werden. 149 Experiment V1 Elektroporation von Huh-7.5-Zellen mit den HCV-Replikons Huh-7.5-Zellen sind eine Sublinie der Huh-7-Hepatoma-Zelllinie und hoch permissiv für die HCV-Infektion. Die Huh-7.5-Zellen wurden zwei Tage vor der Elektroporation passagiert und zu einer Dichte von 80 bis 90 % kultiviert. Ernten der Zellen • Die Zellen werden mit 10 ml D-PBS gewaschen und durch Zugabe von 2 ml Trypsin bei 37°C für ca. 5 min vom Flaschenboden gelöst. • Nach Zugabe von 8 ml DMEM complete Medium wird die Zellsuspension in einem 50 ml Röhrchen gepoolt und 10 min bei 1.200 rpm und 4°C zentrifugiert. • Die Zellen werden zweimal gewaschen. Hierfür wird der Überstand jeweils verworfen, das Pellet mit 50 ml eiskaltem D-PBS gewaschen und erneut 10 min bei 1.200 rpm und 4°C zentrifugiert. • Vor der zweiten Zentrifugation wird ein Aliquot für die Zellzahlbestimmung mittels Neubauer Zählkammer abgenommen. • Die Zellen werden in eiskaltem D-PBS resuspendiert, sodass sich eine Konzentration von 5 x 106 Zellen/ml ergibt (Zellen auf Eis lagern). Zellzahlbestimmung mittels Neubauer-Kammer Zur Bestimmung der Zellzahl werden die Zellen in eine Neubauer-Zählkammer überführt und gezählt. Die Zellkonzentration (in Zellen/ml) ergibt sich aus der Multiplikation von Zellzahl (Mittelwert aus 4 Gruppenquadraten), Verdünnungsfaktor und Kammerfaktor (104). 150 Experiment V1 Elektroporation Die Elektroporation ist eine Transfektionsmethode, um Nukleinsäuren in die Zelle zu bringen, indem die Zellmembran sich durch einen kurzen elektrischen Impuls so verändert, dass die Zelle die Nukleinsäuren aus der Umgebung aufnehmen kann. Hierzu werden 10 µg RNA (JFH1/GND, JFH1/∆E1E2) mit 800 µl der zuvor 6 hergestellten Zellsuspension (5 x 10 Zellen/ml) vermischt, in eine Elektroporations- küvette gegeben und mit Hilfe eines elektrischen Pulses des Bio Rad Gene Pulsers transfiziert (bitte zügig arbeiten, weil die RNA hydrolysiert wird). Elektroschock Spannung (V) Kapazitiver Widerstand (C) Elektr. Widerstand (R) Küvettenspalt 300 V 950 µF ∞ 2 mm Nach 10-minütiger Inkubation bei Raumtemperatur wird die Zellsuspension in 12 ml DMEM complete Medium überführt und in eine 6- bzw. 12-Loch-Platte ausgesät (6-Loch-Platte je 2 ml pro Vertiefung, 12-Loch-Platte je 1 ml pro Vertiefung). Nach ca. 4 h wird das Medium gewechselt, um entstandene Stressfaktoren sowie tote Zellen aus dem Überstand zu entfernen. Die Zellen werden 72 h nach Elektroporation (p.e.) geerntet. Es werden Zellen elektroporiert für die nachfolgende Western Blot-Analyse, die Oxyblot-Analyse, das Reportergenexperiment sowie die Immunfluoreszenzfärbung. 151 Experiment V1 Tag 2 Proteinextraktion, Bestimmung Analyse, Oxyblot-Analyse der Proteinkonzentration, Western Blot- Im Rahmen des Praktikums soll untersucht werden, inwieweit sich der Einfluss von HCV auf die Nrf2/ARE-abhängige Genexpression in oxidativem Stress niederschlägt. Dabei soll insbesondere untersucht werden, wie sich HCV auf die Inaktivierung von Peroxiden auswirkt. Dies soll mittels Oxyblot-Analyse geschehen, die eine indirekte Quantifizierung von ROI ermöglicht. Protein-Extraktion (für Western Blot- und Oxyblot-Analyse) Die Zellen vorsichtig mit PBS waschen, PBS absaugen und anschließend 300 µl RIPA-Puffer pro Vertiefung zugeben. Mit Hilfe der Zellschaber die Zellen von der Zellkulturplatte kratzen, in ein 1,5 ml Reaktionsgefäß überführen und für 10 min auf Eis inkubieren. Achtung: Zelllysate immer auf Eis stellen um Proteindegradation zu vermeiden! Um eine vollständige Lyse der Zellen zu erreichen, wird eine Ultraschallbehandlung durchgeführt, da Hochfrequenzschall die Zellen schert und dadurch aufschließt. Hierzu wird das Reaktionsgefäß in ein Becherglas voll mit Eis gestellt, und die Zellen werden 5 sek bei 20 % Intensität mit dem Ultraschallstab aufgeschlossen. Anschließend werden Zelltrümmer 5 min bei 4°C und 13.000 rpm abzentrifugiert. Bestimmung der Proteinkonzentration mittels Bradford-Assay Die Bestimmung der Proteinkonzentration erfolgt mit Hilfe des Bradford-Tests. Das Prinzip beruht darauf, dass der Farbstoff Coomassie-Brillant-Blau G-250 in saurer Lösung mit Proteinen Komplexe bildet. Die ungebundene Form des Farbstoffs besitzt ein Absorptionsmaximum bei 470 nm. Durch Komplexbildung verschiebt sich dieses auf 595 nm und kann photometrisch erfasst werden. 152 Experiment V1 Zur Bestimmung der Proteinkonzentration wird eine Standardreihe pipettiert. Hierfür wird eine BSA-Stammlösung (10 µg/µl; NEB) auf 0,5 µg/µl verdünnt. Für eine Doppelbestimmung alles zweimal pipettieren: • Jeweils 1 ml Bradford Reagenz in Plastikküvetten vorlegen und 0/ 0,5/ 1/ 1,5/ 2 und 3 µg BSA dazu pipettieren. • 1 µl Zelllysat zu 1 ml Bradford-Reagenz pipettieren. • Die Standards und die Lysate werden im Photometer bei 595 nm gemessen. • Die Auswertung erfolgt mit Hilfe von Excel. Herstellung von SDS-Gelen (werden vom Betreuer vorbereitet) Hergestellt werden sogenannte diskontinuierliche SDS-Gele, die aus einem Sammelund einem Trenngel bestehen. Die Proteine werden im Sammelgel konzentriert, was dazu führt, dass schärfere Banden im Trenngel entstehen. • Gelgießstand mit einer Alumina-Platte beschicken, gefolgt von zwei 1,5 mm Spacern und einer Glasplatte (sicherstellen, dass alle Gummidichtungen am Gelgießstand angebracht wurden!!!). • Diese Reihenfolge wird wiederholt bis 8-10 Gele gegossen werden können. • Gelgießstand mit Deckplatte und Klammern verschließen. • Das Trenngel wie folgt herstellen (Handschuhe verwenden). Achtung, Acrylamid ist gifig! Aqua dest. 4 x Trenngelpuffer Rotiphorese 40 TEMED Ammoniumpersulfat-Lösung 10 % (APS) Trenngel 12 % 36 ml 20 ml 24 ml 80 µl 800 µl • Trenngel direkt in den Gelgießstand gießen (Platz für Sammelgel lassen) und jedes Trenngel vorsichtig mit 200 µl Butanol überschichten. • Anhand der übrig gebliebenen Trenngel-Lösung kann die eingesetzte Polymerisation festgestellt werden. 153 Experiment V1 • Butanol vollständig von den Trenngelen abgießen und das Sammelgel wie folgt herstellen (Handschuhe verwenden): Aqua dest. 4 x Sammlgelpuffer Rotiphorese 40 TEMED Ammoniumpersulfat-Lösung 10 % (APS) Sammelgel 4 % 39 ml 15 ml 6 ml 60 µl 600 µl • Die Trenngele überschichten und die Kämme luftblasenfrei einfügen. • Nach dem Polymerisieren die Gele aus dem Gelgießstand nehmen und die Kämme entfernen. Oxyblot-Analyse (Review: Thomas Nyström 2005) Oxidativer Stress wird meist durch reaktive Sauerstoffintermediate (ROI, reactive oxygen intermediates) wie z. B. O2 oder H2O2 hervorgerufen, die während des normalen aeroben Stoffwechsels entstehen. Diese hochreaktiven freien Radikale können mit Hilfe von Metallkatalysatoren (Fe2+ oder Cu2+) die Amine der Aminosäurereste von z. B. Lysin, Arginin, Prolin oder Histidin in Carbonylreste umwandeln. Carbonylisierte Proteine werden normalerweise ubiquitinyliert und dem proteasomalen Abbau zugeführt. Ist jedoch die Regulierung der Proteindegradation in der Zelle gestört, kommt es zur Akkumulation von fehlerhaften Proteinen und zu Fehlfunktionen in der Zelle. Der meistgenutzte Marker zur Messung von Proteinoxidation ist der Carbonylgehalt von Proteinen. Hier macht man sich zu Nutze, dass die Carbonylgruppe mit 2,4-Dinitrophenylhydrazin (DNP) unter Bildung einer Schiff‘schen Base zum entsprechenden Hydrazon Dinitrophenylhydrazon kann reagiert. dann 2,4- entweder photometrisch oder mit Antikörpern im Western Blot nachgewiesen werden. 154 Experiment V1 Während des Praktikums wird das OxyBlot™ protein oxidation detection Kit von Millipore verwendet. Um die Oxidation der Proteine noch zu steigern, werden die Zellen für 30 min mit 2 mM H2O2 (oder für 4 h mit Glucoseoxidase) behandelt. Die DNP-derivatisierten Proteine werden im 12 %-igen SDS-Gel (40 µg Protein pro Spur) aufgetrennt. Dann erfolgt die Western Blot-Analyse wie beschrieben mit einem antiDNPH-Antikörper, gefolgt von der Inkubation mit einem Meerrettichperoxidasegekoppelten Antikörper. Anschließend folgt die Detektion der Banden mit ECLReagenz wie beschrieben. SDS-Gellauf Oxyblot-Analyse • Mische 20 µg Protein (GND und ΔE1E2) mit demselben Volumen 12 % SDS (je Probe zwei Reaktionsansätze). • Mische die erste Probe 1:1 mit 1 x DNPH und die zweite Probe (Negativkontrolle) 1:1 mit der 1 x Derivatisierungskontrolle. • 15 Minuten Inkubation bei Raumtemperatur (nicht länger als 30 Minuten!). • Zugabe des 1,5-fachen Volumens des Proteinlysats an Neutralisationslösung je Reaktionsgefäß. • Laden der vollständigen Proben aufs Gel (man braucht keinen Ladepuffer und kein Erhitzen). • Kombiniere 2,5 µl Proteinstandard mit beigefügten DNP-Resten und 5 µl 4 x Gel-Ladepuffer. Blotten der Proben • Whatman-Papier und Immobilon PVDF-Membran mit einer Größe von 7 x 9 cm zurechtschneiden (Membran nur mit Handschuhen anfassen). • Membran in Methanol legen (aktivieren). • Whatman-Papier wie folgt in die Blotpuffer legen: - 4 Blatt in Anodenpuffer I - 2 Blatt in Anodenpuffer II - 6 Blatt in Kathodenpuffer 155 Experiment V1 • Blot wie folgt zusammenbauen: Kathode 6x Kathode Gel Membran 2x Anode II 4x Anode I Anode • Für 1 h bei 80 mA/Membran blotten. Antikörperinkubation und Detektion • Die Membran in 1 x TBST legen (darf nicht trocknen). • Anschließend die Membran in 1 % BSA in 1 x TBST für 1 h blockieren. • 3 ml Primärantikörperlösung (1 % BSA in 1 x TBST) entsprechend den Anweisungen der Betreuer vorbereiten und die Membran 1h bei Raumtemperatur (auf dem Roller) inkubieren: 1. AK Antibody stock (90451) 1:150 • Membran mit der Proteinseite nach innen gerichtet in das Röhrchen schieben und für 1 h auf dem Roller inkubieren. • Waschen der Membran: 3 x 10 min in 1 x TBST. • 3 ml Sekundärantikörperlösung (1 % BSA in 1 x TBST) entsprechend den Anweisungen der Betreuer vorbereiten und die Membran Raumtemperatur (auf dem Roller) inkubieren: 2. AK Antibody stock (90452) 1:300 • Waschen der Membran: 3 x 10 min in 1 x TBST (im Dunkeln!!!). Anschließend wird der Blot mit ECL-Substrat entwickelt. • Zuschneiden von Folien und Festkleben in einer Entwicklerkassette. 156 1h bei Experiment V1 • Anmischen des ECL-Substrates: pro Blot 200 µl Lösung 1 mit 200 µl Lösung 2 im Eppi mischen. • Oxyblot mit der Pinzette (oder der behandschuhten Hand) aus TBST nehmen, kurz auf ein Tuch abtropfen lassen und auf die Folie in die Entwicklerkassette legen. • ECL-Substrat tropfenweise auf den Blot geben. • Benetzten Blot luftblasenfrei mit einer 2. Folie bedecken. • Exposition mit einem Röntgenfilm in der Dunkelkammer (vorher vom Betreuer erklären lassen!!!). Western Blot-Analyse Mit Hilfe der Western Blot-Analyse soll der Erfolg der Elektroporation überprüft werden. Hierfür werden die Protein zunächst mittels SDS-PAGE aufgetrennt, auf eine PVDF-Membran geblottet und anschließend unter Verwendung HCVspezifischer Antikörper anlysiert. Die Detektion der HCV-Proteine erfolgt mit Antikörpern, die gegen das Core- sowie das NS3-Protein gerichtet sind. SDS-Gellauf Western Blot-Analyse • 75 µg Protein werden mit 4 x SDS-Probenpuffer im Verhältnis 1:4 gemischt und für 5 min bei 95°C gekocht. • SDS-Gel in die Gelkammer einspannen und Reservoir mit 1 x SDS-Laufpuffer auffüllen. • Proben und 10 µl Proteinmarker in die Taschen laden. • Das Gel für ca. 1 h bei 120 V laufen lassen, bis die blau gefärbte Lauffront am Ende des Gels angekommen ist. 157 Experiment V1 Blotten der Proben (siehe Oxyblot-Analyse) Antikörperinkubation und Detektion • Die Membran in 1 x TBST legen (darf nicht trocknen). • Anschließend die Membran in 10 % Milchpulver in 1 x TBST für 1 h blockieren. • 3 ml Primärantikörperlösung (in 10 % Milchpulver in 1 x TBST) entsprechend den Anweisungen der Betreuer vorbereiten und die Membran 1 h bei Raumtemperatur (auf dem Roller) inkubieren: 1. AK NS3 (abcam; mouse) HCV-Core (Pierce; mouse) 1:1.000 1:1.000 • Membran mit der Proteinseite nach innen gerichtet in das Röhrchen schieben und für 1 h auf dem Roller inkubieren. • Waschen der Membran: 3 x 10 min in 1 x TBST. • 3 ml Sekundärantikörperlösung (in 10 % Milchpulver in 1x TBST) entsprechend den Anweisungen der Betreuer vorbereiten und die Membran 1 h bei Raumtemperatur (auf dem Roller) inkubieren (Röhrchen mit Alufolie umwickeln, da Sekundärantikörper mit Fluorophor gelabelt ist!!!): 2. AK IRDye®800 donkey anti-mouse 1:3.000 • Waschen der Membran: 3 x 10 min in 1 x TBST (im Dunkeln!!!) Anschließend wird der Blot mit dem „Odyssey® Imaging System“ entwickelt (wird vom Betreuer erklärt). 158 Experiment V1 PEI-basierte DNA-Transfektion der elektroporierten Huh-7.5-Zellen (vom Vortag) Im Rahmen des Praktikums soll mittels eines sog. Reportergenexperiments der Einfluss von HCV auf die Expression Nrf2/ARE-abhängig regulierter Gene analysiert werden. Dazu werden HCV-replizierende Zellen bzw. die entsprechenden Kontrollzellen mit dem Nrf2/ARE-Reporterkonstrukt pNQO1luc transient transfiziert. Ein Teil der Zellen wird vor der Ernte für 16 h mit 50 µM tert-Butylhydrochinone (tBHQ) stimuliert. tBHQ wirkt als Elektrophil und bewirkt so die Induktion von Nrf2 und die damit verbundene Expression von u. a. NQO1. DNA lässt sich durch eine Reihe unterschiedlicher Verfahren in eukaryotische Zellen einbringen, die alle unter dem Begriff der Transfektion zusammengefasst werden. Grundsätzlich unterscheidet man zwischen transienter (zeitweiliger) und stabiler (dauerhafter) Transfektion. Die unterschiedlichen Transfektionsmethoden lassen sich grob in chemische (Calcium-Phosphat-Präzipitation, Lipofektion, Transfektion durch kationische Polymere oder Nanopartikel) und physikalische (Mikroinjektion, Elektroporation, „particle gun“, Magnet-assistierte Transfektion) Methoden einteilen. Die in diesem Praktikum angewandte Transfektion der Zellen erfolgt mit dem kationischen Polymer Polyethylenimin (PEI). Die Anfang der 90er Jahre entwickelte Methode (Ehrhardt et. al, 2005) nutzt die Eigenschaft dieser Polymere, auf der DNA zu kondensieren und Polykomplexe zu bilden, welche dann von der Zelle aufgenommen werden. Um eine Transfektion so effizient wie möglich zu gestalten, muss der Transfektionsansatz sehr genau pipettiert werden. Dies gilt insbesondere für die Reihenfolge, in der die einzelnen Reagenzien zugegeben werden. Das Volumen des Transfektionsansatzes entspricht 1/10 des Mediumvolumens in dem sich die zu transfizierenden Zellen befinden. Die eingesetzte Menge PEI (1 mg/ml) richtet sich nach der Menge der zu transfizierenden DNA. Hierbei werden pro 1 µg DNA 6 µl PEI eingesetzt. 159 Experiment V1 Der Transfektionsansatz wird nach folgendem Schema pro Vertiefung einer 6-LochPlatte angesetzt: Reagenz Volumen/Ansatz x Ansätze NQO1luc PBS 193 µl DNA (1 µg/µl) 1 µl PEI (1 mg/ml) 6 µl Der Ansatz wird gevortext, für 10 min bei Raumtemperatur inkubiert und anschließend tropfenweise auf die Zellen pipettiert. Nach 8-12 h erfolgt ein Mediumwechsel, da das PEI toxisch für die Zellen ist. Hierzu wird das Medium abgesaugt und die Zellen werden vorsichtig mit PBS gewaschen. Nach Absaugen des PBS werden 1,5 ml frisches Medium auf die Zellen gegeben (wird am nächsten Tag vom Betreuer durchgeführt). Die Analyse der Zellen erfolgt nach 48 h durch den Luciferase-Reportergen-Assay. 160 Experiment V1 Tag 3 Immunfluoreszenzmikroskopie Im Rahmen des Praktikums soll mit Hilfe der konfokalen Immunfluoreszenzmikroskopie einerseits der Einfluss von HCV auf die subzelluläre Lokalisation der sMafs untersucht, andererseits sollen die daran beteiligten viralen Faktoren identifiziert werden. Dies wird unter Verwendung von Huh-7.5-Zellen, die ein subgenomisches replikationskompetentes (∆E1E2) bzw. -defizientes (GND) HCVReplicon tragen, analysiert. Die Immunfluoreszenzmikroskopie ist eine Methode zur Detektion von Proteinen im subzellulären Bereich mit Hilfe Fluorophor-gekoppelter Antikörper. Hierbei kann zwischen der direkten und indirekten Immunfluoreszenz unterschieden werden. Bei der direkten Immunfluoreszenz wird die Probe mit einem spezifischen gegen das Zielprotein gerichteten Antikörper markiert, der direkt mit einem Fluorophor (z. B. FITC) gekoppelt ist. Bei der indirekten Technik werden zwei Antikörper verwendet: ein Primärantikörper, der spezifisch an das Zielprotein bindet, und ein Sekundärantikörper, der mit einem Fluorophor gekoppelt und gegen den Primärantikörper gerichtet ist. Zu Beginn der Färbeprozedur muss die Probe zunächst fixiert werden, um den Zustand zum jeweiligen Zeitpunkt zu erhalten. Die Wahl des Fixativs richtet sich nach dem jeweiligen Protein, das detektiert werden soll bzw. nach dem eingesetzten Antikörper. Aldehyde quervernetzen Proteine und erhalten die natürliche Zellmorphologie. Durch Alkohol- oder Aceton-Fixierung kommt es zur Denaturierung der Proteine. Die Denaturierung führt zum Verlust der biologischen Aktivität und dem Auftreten neuer Gruppen, was die Bindung der Antikörper beeinflussen kann. Nach dem Fixieren wird das Fixativ zunächst ausgewaschen. Anschließend werden mögliche freie Bindungsstellen der Probe durch Inkubation mit Serumproteinen (hier: 10 % BSA in 1 x TBST) blockiert, um unspezifische Bindungen zu vermeiden. Nach dem Blockieren der Probe erfolgt die Inkubation mit dem Primärantikörper, der gegen das Zielprotein gerichtet ist. Nach erneutem Waschen wird mit dem gegen den 161 Experiment V1 Primärantikörper gerichteten Sekundärantikörper inkubiert, der mit dem Fluorophor gekoppelt ist und mit Hilfe von Fluoreszenz detektiert werden kann. Zusätzlich erfolgt eine Färbung der Kerne mit DAPI (4´,6-Diamidino-2-phenylindol), welches sich an die A/T-reiche Region der kleinen Furche der DNA-Doppelhelix anlagert. Mit dieser Methode lassen sich auch bis zu drei verschiedene Zielproteine gleichzeitig markieren, indem mit den entsprechenden Primär- und Sekundärantikörpern inkubiert wird. Dabei ist darauf zu achten, dass die eingesetzten Antikörper nicht miteinander kreuzreagieren. Die Analyse der Immunfluoreszenz erfolgt mit Hilfe eines konfokalen Laser-ScanMikroskops. Konfokale Laser-Scan-Mikroskopie (CLSM) Die konfokale konventionellen Laser-Scan-Mikroskopie bietet Weitfeld-Lichtmikroskopie. einige Während Vorteile bei einem gegenüber der konventionellen Lichtmikroskop das gesamte Präparat ausgeleuchtet wird und das Bild aus einer Überlagerung aller Ebenen entsteht, wird das Präparat beim LSM Punkt für Punkt abgerastert. Durch Messung der Lichtintensitäten des reflektierten oder emittierten Lichtes an allen Punkten des Präparates wird aus den entstehenden Einzelbildern ein digitales Gesamtbild zusammengesetzt. Eine entscheidende Besonderheit der konfokalen Laser-Scan-Mikroskopie besteht darin, dass sich im Strahlengang des detektierten Lichts eine Lochblende (englisch: pinhole) befindet. Dadurch wird eine Steuerung der Schärfentiefe sowie die Ausschaltung von unerwünschten Hintergrundinformationen ermöglicht. Vereinfacht gesagt, es werden nur die Signale aus einer Schicht des Präparats detektiert. Durch die Variation der Öffnung der Blende kann somit die Dicke der analysierten Schicht verändert werden. Eine weitere Möglichkeit besteht in der Anfertigung von seriellen optischen Schnitten der Präparate (z-stack). Bei der CLSM werden meist digitale Bilder von einfach-, doppel- oder dreifach-markierten fluoreszierenden Proben erstellt. Durchführung Für die Immunfluoreszenzfärbung werden 3 x 104 Zellen/ml in 12-Loch-Platten auf Deckgläschen ausgesät und nach 72 h fixiert. Hierfür wird wie folgt vorgegangen: 162 Experiment V1 Fixieren der Zellen • Medium abnehmen und die Zellen einmal mit 1 ml PBS waschen. • Zugabe von 0,8 ml Formaldehyd-Lösung (4 % in PBS) pro Vertiefung • Inkubation für 10 min bei Raumtemperatur. • Nach der Inkubation das Formaldehyd abnehmen und einmal mit 1 ml PBS waschen. Blockieren unspezifischer Bindungen • Zugabe von 0,5 ml BSA (10 % in 1 x TBST) je Vertiefung. • Inkubation 60 min bei Raumtemperatur. • Überstand abnehmen. Inkubation mit dem Primärantikörper (sMafs, NS3) • Antikörperverdünnungen vorbereiten in 10 % BSA-Lösung (jeweils 50 µl pro Deckgläschen) • • sMafs (F/G/K) (H-100) (Santa Cruz; rabbit) → 1:80 • NS3 (Virostat; mouse) → 1:50 50 µl Antikörperverdünnung auf ein Stück (trockenen) Parafilm in einer feuchten Kammer geben. • Deckgläschen mit einer Pinzette aus dem Loch nehmen (wird vom Betreuer vorgeführt) und auf die Antikörperverdünnung legen (darauf achten, dass die Seite mit den Zellen nach unten zeigt!!!). • Inkubation für 60 min bei Raumtemperatur (in einer feuchten Kammer). • Während der Inkubation jeweils 1 ml 1 x TBST in die Vertiefungen der 12Loch-Platte geben (zum Waschen). • Nach der Inkubation die Deckgläschen in die vorbereiteten Vertiefungen geben (Zellen zeigen nach oben!!!). • Waschen: 3 x 5 min in 1 x TBST. 163 Experiment V1 Inkubation mit dem Sekundärantikörper (sMafs, NS3) und DAPI (Ab hier lichtgeschützt arbeiten!!!) • Antikörperverdünnungen vorbereiten in 10 % BSA-Lösung (jeweils 50 µl pro Deckgläschen) • • für sMafs (F/G/K) → Cy3 donkey anti-rabbit; 1:400 • für NS3 → Alexa 488 donkey anti-mouse; 1:1000 • DAPI →1:400 50 µl Antikörperverdünnung auf ein Stück (trockenen) Parafilm in einer feuchten Kammer geben. • Deckgläschen mit einer Pinzette aus der Vertiefung nehmen und auf die Antikörperverdünnung legen (darauf achten, dass die Seite mit den Zellen nach unten zeigt!!!). • Inkubation für 60 min bei Raumtemperatur (in einer feuchten Kammer). • Während der Inkubation jeweils 1 ml 1 x TBST in die Vertiefungen der 12Loch-Platte geben (zum Waschen). • Nach der Inkubation die Deckgläschen in die vorbereiteten Vertiefungen geben (Zellen zeigen nach oben!!!). • Waschen 3 x 10 min in 1 x TBST (lichtgeschützt !!!!). Einbetten der Proben mit Mowiol • Objektträger ordentlich beschriften (Primärantikörper, Sekundärantikörper, Gruppenkürzel)!!! • Ca. 10 µL Mowiol pro Deckgläschen auf den Objektträger tropfen (Vorsicht: sehr viskos; lässt sich nur schwer pipettieren). • Deckgläschen kurz auf einem Tuch abtropfen lassen und möglichst luftblasenfrei mit den Zellen nach unten auf den Objektträger geben (Deckgläschen so wenig wie möglich bewegen, da sonst die Zellen beschädigt werden). • Im Luftstrom der Laminar-Flow trocknen (lichtgeschützt), 164 Experiment V1 Luciferase Assay Das Firefly-Luciferase-Gen eignet sich hervorragend als Reportergen, sowohl bei eukaryotischen als auch bei prokaryotischen Expressionssystemen. Hierbei steht das Luciferase-Gen unter der Kontrolle des jeweiligen Promotors des zu untersuchenden Gens (z. B. NQO1). Durch Transfektion der Zellen mit dem jeweiligen Reporter-Konstrukt und anschließender Stimulation kann somit die Aktivität des gewünschten Promotors bestimmt werden. Die Firefly-Luciferase katalysiert hierbei die Umwandlung von D-Luciferin, Sauerstoff und ATP in Oxyluciferin, AMP, CO2 und Licht (Biolumineszenz), das mit einem Luminometer detektiert werden kann. Die Menge produzierter Biolumineszenz spiegelt hierbei die Aktivität des jeweiligen Promotors wider. ATP + D-Luciferin + O2 Luciferase AMP + PP + D-Luciferin + CO2 + Licht Zelllyse Die mit den Konstrukt pNQO1luc transfizierten Zellen (JFH1/GND und JFH1/∆E1E2) werden 48 h nach der Transfektion geerntet. Hierzu wird das Medium abgesaugt, und die Zellen werden einmal mit PBS gewaschen. Anschließend werden pro Vertiefung 200 µl Luciferase-Lysepuffer zugegeben, und die Platten werden 5 min auf Eis inkubiert. Für eine Doppelbestimmung werden jeweils 50 µl Lysat (das zuvor 5 min bei 13000rpm und 4 Grad zentrifugiert worden war) aus einer Vertiefung in jeweils 2 Vertiefungen einer „weißen“ 96-Loch-Platte (werden vom Betreuer ausgeteilt) pipettiert. Dies wird für alle Proben wiederholt. Messung der Luciferase-Aktivität Die Messung der Luciferase-Aktivität erfolgt im Orion plate reader II. Um die Waschflüssigkeit aus dem System zu entfernen, wird das Gerät mit LuciferinSubstrat gespült (= “Prime“). Die 96-Loch-Platte wird in das Gerät gestellt, das entsprechende Programm wird gewählt (Messzeit 10 sek/Luciferin 30 µl) und die Messung wird gestartet. Die ausgegebenen Werte stellen RLUs (relative light units) 165 Experiment V1 dar. Um die Werte miteinander vergleichen zu können, wird der (relative) Proteingehalt der Lysate mit Hilfe eines Bradford-Assays bestimmt. Die Werte beider Messungen können dann zueinander ins Verhältnis gesetzt werden (RLU/relative Proteinmenge). Proteinbestimmung mittels Bradford Hier wird keine exakte Konzentrationsbestimmung durchgeführt, da hier relative Werte ausreichend sind. Pro Vertiefung einer 96-Loch-Platte werden 5 µl Lysat vorgelegt und jeweils 100 µl Bradford-Reagenz hinzugefügt. Nach 5-minütiger Inkubation bei Raumtemperatur wird die Absorption bei 595 nm im TECAN-Reader gemessen. Die Auswertung erfolgt mit Hilfe von Mircosoft Excel. Literatur: Bartenschlager R, Frese M, Pietschmann T.Novel insights into hepatitis C virus replication and persistence.Adv VirusRes. 2004;63:71-180. Review. Barth H, Liang TJ, Baumert, TF. Hepatitis C virus entry: molecular biology and clinical implications. Hepatology 2006; 43 in press. Bartosch B, Cosset FL. Cell entry of hepatitis C virus. Virology. 2006 Apr 25;348(1):1-12. Bartosch B, Dubuisson J, Cosset FL. Infectious hepatitis C virus pseudo-particles containing functional E1-E2 envelope protein complexes. J Exp Med. 2003 Mar 3;197(5):633-42. Baumert TF, Vergalla J, Satoi J, Thomson M, Lechmann M, Herion D, Greenberg HB, Ito S, Liang TJ.Hepatitis C virus-like particles synthesized in insect cells as a potential vaccine candidate. Gastroenterology. 1999 Dec;117(6):1397-407. Beyer TA, Werner S.The cytoprotective Nrf2 Transkription factor controls insulin receptor signaling in the regenerating liver.Cell Cycle. 2008 Apr;7(7):874-8. Branch AD, Stump DD, Gutierrez JA, Eng F, Walewski JL. The hepatitis C virus alternate reading frame (ARF) and its family of novel products: the alternate reading frame protein/F-protein, the doubleframeshift protein, and others. Semin Liver Dis. 2005 Feb;25(1):105-17. Review. Bressanelli S, Tomei L, Rey FA, De Francesco R. Structural analysis of the hepatitis C virus RNA polymerase in complex with ribonucleotides. J Virol. 2002 Apr;76(7):3482-92. Burckstummer T, Kriegs M, Lupberger J, Pauli EK, Schmittel S, Hildt E. Raf-1 kinase associates with Hepatitis C virus NS5A and regulates viral replication. FEBS Lett. 2006 Jan 23;580(2):575-80. Carvajal-Yepes M, Himmelsbach K, Schaedler S, Ploen D, Krause J, Ludwig L, Weiss T, Klingel K, Hildt E Hepatitis C virus impairs the induction of cytoprotective Nrf2 target genes by delocalization of small Maf proteins.J Biol Chem. 2011 Mar 18;286(11):8941-51. 166 Experiment V1 Castello G, Scala S, Palmieri G, Curley SA, Izzo F.HCV-related hepatocellular carcinoma: From chronic inflammation to cancer.Clin Immunol. 2010 Mar;134(3):237-50. Epub 2009 Nov 11 Di Bisceglie AM, Hoofnagle JH. Optimal therapy of hepatitis C. Hepatology. 2002 Nov;36(5 Suppl 1):S121-7. Ferenci P. Pegylated interferon plus ribavirin for chronic hepatitis C: the role of combination therapy today, tomorrow and in the future. Minerva Gastroenterol Dietol. 2006 Jun;52(2):157-74. Gale M Jr, Blakely CM, Kwieciszewski B, Tan SL, Dossett M, Tang NM, Korth MJ, Polyak SJ, Gretch DR, Katze MG. Control of PKR protein kinase by hepatitis C virus nonstructural 5A protein: molecular mechanisms of kinase regulation. Mol Cell Biol. 1998 Sep;18(9):5208-18. Griffin SD, Beales LP, Clarke DS, Worsfold O, Evans SD, Jaeger J, Harris MP, Rowlands DJ.The p7 protein of hepatitis C virus forms an ion channel that is blocked by the antiviral drug, Amantadine. FEBS Lett. 2003 Jan 30;535(1-3):34-8. Harris HJ, Davis C, Mullins JG, Hu K, Goodall M, Farquhar MJ, Mee CJ, McCaffrey K, Young S, Drummer H, Balfe P, McKeating JA.Claudin association with CD81 defines hepatitis C virus entry.J Biol Chem. 2010 Apr 7. [Epub ahead of print] Higuchi M, Tanaka E, Kiyosawa K. Epidemiology and clinical aspects on hepatitis C. Jpn J Infect Dis. 2002 Jun;55(3):69-77. Himmelsbach K, Sauter D, Baumert T, Ludwig L, Blum H, and Hildt E. (2009)New aspects of an antitumor drug: sorafenib efficiently inhibits HCV replication. GUT, 2009 Dec;58(12):1644-53. Ikeda H, Nishi S, Sakai M.Transcription factor Nrf2/MafK regulates rat placental glutathione Stransferase gene during hepatocarcinogenesis. Biochem J. 2004 Jun 1;380(Pt 2):515-21. Kensler TW, Wakabayashi N, Biswal S. Cell survival responses to environmental stresses via the Keap1-Nrf2-ARE pathway. Annu Rev Pharmacol Toxicol. 2007;47:89-116. Kim JL, Morgenstern KA, Lin C, Fox T, Dwyer MD, Landro JA, Chambers SP, Markland W, Lepre CA, O'Malley ET, Harbeson SL, Rice CM, Murcko MA, Caron PR, Thomson JA. Crystal structure of the hepatitis C virus NS3 protease domain complexed with a synthetic NS4A cofactor peptide. Cell. 1996 Oct 18;87(2):343-55. Kronenberger, B. and Zeuzem, S. 2012 Jan. New developments in hcv therapy. J Viral Hepat, 19, Suppl 1:48–51. Kwak MK, Itoh K, Yamamoto M, Kensler TW. Enhanced expression of the transcription factor Nrf2 by cancer chemopreventive agents: role of antioxidant response element-like sequences in the nrf2 promoter. Mol Cell Biol. 2002 May;22(9):2883-92. Lagging M, Wejstål R, Uhnoo I, Gerdén B, Fischler B, Friman S, Josephson F, Karlström O, Sangfelt P, Schvarz R, Weiland O; Swedish Consensus Group.Treatment of hepatitis C virus infection: updated Swedish Consensus recommendations.Scand J Infect Dis. 2009;41(6-7):389-402. Lau A, Villeneuve NF, Sun Z, Wong PK, Zhang DD Dual roles of Nrf2 in cancer. Pharmacol Res. 2008 Nov-Dec;58(5-6):262-70. Epub 2008 Sep 13. Lesburg CA, Cable MB, Ferrari E, Hong Z, Mannarino AF, Weber PC. Crystal structure of the RNAdependent RNA polymerase from hepatitis C virus reveals a fully encircled active site. Nat Struct Biol. 1999:6(10):937-43. Lindenbach BD, Evans MJ, Syder AJ, Wolk B, Tellinghuisen TL, Liu CC, Maruyama T, Hynes RO, Burton DR, McKeating JA, Rice CM. Complete replication of hepatitis C virus in cell culture. Science. 2005 Jul 22;309(5734):623-6. 167 Experiment V1 Luik P, Chew C, Aittoniemi J, Chang J, Wentworth P Jr, Dwek RA, Biggin PC, Vénien-Bryan C, Zitzmann N.The 3-dimensional structure of a hepatitis C virus p7 ion channel by electron microscopy.Proc Natl Acad Sci U S A. 2009 Aug 4;106(31):12712-6. Epub 2009 Jul 9. Macdonald A, Harris M. Hepatitis C virus J Gen Virol. 2004 Sep;85(Pt 9):2485-502. Review. NS5A: tales of a promiscuous protein. Osburn WO, Karim B, Dolan PM, Liu G, Yamamoto M, Huso DL, Kensler TW. Increased colonic inflammatory injury and formation of aberrant crypt foci in Nrf2-deficient mice upon dextran sulfate treatment. Int J Cancer. 2007 Nov 1;121(9):1883-91. Pavlovic D, Neville DC, Argaud O, Blumberg B, Dwek RA, Fischer WB, Zitzmann N. The hepatitis C virus p7 protein forms an ion channel that is inhibited by long-alkyl-chain iminosugar derivatives. Proc Natl Acad Sci U S A. 2003 May 13;100(10):6104-8. Epub 2003 Apr 28. Penin F, Dubuisson J, Rey FA, Moradpour D, Pawlotsky JM. Structural biology of hepatitis C virus. Hepatology. 2004 Jan;39(1):5-19. Review Ploss A, Rice CM. Towards a small animal model for hepatitis C.EMBO Rep. 2009 Nov;10(11):1220-7. Epub 2009 Oct 16. Protzer U, Seyfried S, Quasdorff M, Sass G, Svorcova M, Webb D, Bohne F, Hösel M, Schirmacher P, Tiegs G.Antiviral activity and hepatoprotection by heme oxygenase-1 in hepatitis B virus infection.Gastroenterology. 2007 Oct;133(4):1156-65. Epub 2007 Jul 25. Rong L, Perelson AS.Treatment of hepatitis C virus infection with interferon and small molecule direct antivirals: viral kinetics and modeling.Crit Rev Immunol. 2010;30(2):131-48. Santolini E, Pacini L, Fipaldini C, Migliaccio G, Monica N. The NS2 protein of hepatitis C virus is a transmembrane polypeptide. J Virol. 1995 Dec;69(12):7461-71. Schädler, S, Krause, J, Klingel, K, Weiss, T. Nassal, M, Werner, S, Hildt, E. (2010) Hepatitis B virus induces expression of antioxidant response element (ARE)- regulated genes by activation of Nrf2 (J. Biol Chem. 2010 Dec 24;285(52):41074-86. Epub 2010 Oct 18.) Seeff LB. Natural history of chronic hepatitis C. Hepatology. 2002 Nov;36(5 Suppl 1):S35-46. Tai CL, Chi WK, Chen DS, Hwang LH. The helicase activity associated with hepatitis C virus nonstructural protein 3 (NS3). J Virol. 1996 Dec;70(12):8477-84. Tsai WL, Chung RT.Viral hepatocarcinogenesis.Oncogene. 2010 PMID: 20228847 HCV-related hepatocellular carcinoma: From chronic inflammation to cancer. Wakita T, Pietschmann T, Kato T, Date T, Miyamoto M, Zhao Z, Murthy K, Habermann A, Krausslich HG, Mizokami M, Bartenschlager R, Liang TJ. Production of infectious hepatitis C virus in tissue culture from a cloned viral genome. Nat Med. 2005 Jul;11(7):791-6. Yamaga AK, Ou JH. Membrane topology of the hepatitis C virus NS2 protein. J Biol Chem. 2002 Sep 6;277(36):33228-34. Yates MS, Kwak MK, Egner PA, Groopman JD, Bodreddigari S, Sutter TR, Baumgartner KJ, Roebuck BD, Liby KT, Yore MM, Honda T, Gribble GW, Sporn MB, Kensler TW. Potent protection against aflatoxin-induced tumorigenesis through induction of Nrf2-regulated pathways by the triterpenoid 1-[2cyano-3-,12-dioxooleana-1,9(11)-dien-28-oyl]imidazole.Cancer Res. 2006 Feb 15;66(4):2488-94. Zhong J, Gastaminza P, Cheng G, Kapadia S, Kato T, Burton DR, Wieland SF, Uprichard SL, Wakita T, Chisari FV. Robust hepatitis C virus infection in vitro. Proc Natl Acad Sci U S A. 2005 Jun 28;102(26):9294-9. 168 Experiment V2 Experiment Virologie V2: RNAi technologies to identify cellular factors affecting viral replication Dr. Renate König 169 Experiment V2 Goal: Using RNAi screening technology to identify factors involved in HIV replication Aim 1 (Experiment 1): The application of synthetic siRNAs and high-throughput screening technology A high-throughput 384-well plate assay will be optimized with the goal to identify siRNAs that influence cell viability. This assay can then be used as a counterscreen for factors that previously had been identified in a genome-wide siRNA screen for cellular factors that influence HIV-1 infection of human 293T cells. To this end, in this course, we will transfect positive and negative control siRNAs into cells (in a 384-well plate using HTS equipment) and test their effects on cell viability measured by ATP readout of the cells. Aim 2 (Experiment 2): The application of vector-based shRNAs and validation of cellular factors in physiological relevant cell types Stable shRNA expressing cells targeting a specific restriction factor (SAMHD1) were generated. In this course, the knockdown efficiency of the shRNAs will be confirmed by Western Blot analysis. In addition, the influence of the absence or presence of SAMHD1 on viral replication will be evaluated after infection of the cells with a lentiviral vector. This will determine the restriction capacity of the cellular factor. Introduction: RNA interference – an overview. Until recently, genome-scale genetic experimentation has been limited to studies in tractable model organisms, such as yeast or C. elegans. However, a number of advances in the manipulation of mammalian cells have made genome-wide gain-of-function and loss-of function analysis possible. The development of high throughput transfection technologies allowed for the introduction of large-scale arrayed libraries of nucleic acids into mammalian cells in a rapid and economical fashion. Furthermore, the emergence of RNA interference (RNAi) as a novel pathway which allows the modulation of gene expression in mammalian cells has facilitated loss of function studies. Consequently, effects of gene dosage on particular cellular phenotypes can be monitored for hundreds to thousands of genes in a single experiment. Large collections of cDNAs, shRNAs, siRNAs, small molecules, and other bioactive agents are currently being compiled and distributed, and the repertoire of possible genetic screens is expansive, only limited by the ability to detect a particular molecular or cellular phenotype. Recently, large-scale genetic analysis and high throughput screening (HTS) in mammalian cells has become accessible to academic institutes, largely because costs associated with these efforts, including libraries and automation, have been reduced significantly. RNAi is a naturally occurring process in eukaryotes by which double-stranded RNAs (dsRNA) trigger the sequence-specific degradation of cognate mRNAs thus preventing expression of their protein products (Fig. 1). First described in C. elegans (REF), RNAi has become a widespread tool for reverse-genetics in invertebrate model systems and has proven useful in large-scale functional genomic screens. In mammalian systems, RNAi can be triggered using small interfering RNAs (siRNAs), small dsRNA fragments of 21-23bp that mimic Dicer processed intermediates while 170 Experiment V2 overcoming undesired antiviral mechanisms (Fig. 2). There are two general strategies to induce RNAi in mammalian cells: a) transient transfection of synthetic double-stranded RNA oligonucleotides; b) stable expression of shRNAs, DNA-based reagents such as plasmids or viral vectors that encode short RNA hairpin sequences. When transferred into cells by transfection or transduction, RNA hairpins are expressed and processed to release endogenously produced siRNAs. Because RNAi reagents can be designed directly from sequence information without need of physical clones, it is possible to generate large collections of them to simultaneously interrogate thousands of genes in live cells. Today, large-scale libraries can be obtained from commercial vendors, which, combined with high throughput techniques, are making it possible to perform large-scale, high speed, reproducible functional screens. Still, limitations to the technology exist, specifically: Variable inhibitory efficiency: siRNA silencing potency varies significantly, depending both on the selected sequence within the mRNA Figure 1. Schematic representation of the RNAi pathway. RNAi involves cleavage of long target and on the target itself. It is clear that dsRNA stretches into small dsRNAs by the there are inherently “difficult genes” for which nuclease Dicer. Coupled to this process is activation of the RNA Interference Signaling the likelihood of selecting efficient siRNAs is Complex (RISC), which in turn uses small lower than usual. Multiple studies have RNA fragments to induce sequence-specific shown that the efficacy of siRNA can be degradation of cognate mRNAs improved, in part, at the level of sequence design [reviewed in Tilesi F et al., Design and validation of siRNAs and shRNAs. Curr Opin Mol Ther. 2009 Apr;11(2):156-64]. Off-target effects: siRNAs are known to produce a “signature” of inhibited transcripts in addition to the intended target (129-131), which can yield potentially misleading results. Several sources of off-target effects can be considered. In fact, they have been associated with significant base-pairing between the 5’ region of the siRNA chains and unintended targets. In addition, siRNAs may work through miRNA-like mechanisms, which affect targets without the need of perfect base-pair matching. As a result, results obtained through RNAi screens need to be reproducible with independent siRNAs To overcome these caveats in large-scale siRNA screens, several strategies can be employed, including: a) Increasing library redundancy: Current commercial libraries contain 3 to 4 siRNAs per target, which allows for prioritization of target hits represented by multiple siRNAs as highly significant hits. b) Empirically-derived screen analysis tools, specifically designed to analyze siRNA screens (Birmingham A, et al, Statistical methods for analysis of highthroughput RNA interference screens. Nat Methods. 2009 Aug;6(8):569-75.). 171 Experiment V2 Figure 2. Schematic representation of the various ways to “feed” siRNAs s into the RNAi pathway. Delivery of synthetic siRNAs into the cell or expression of vectorbased shRNA hairpins that after cleavage through Dicer can engage into the RISC complex or dsRNA (e.g. from viruses) can trigger the RNAi pathway. Cellular innate restriction factors inhibiting HIV-1 – an overview Innate immunity is the first line of defense against foreign pathogen invasion. In general, the innate response is composed of the interferon (IFN) system, as well as cell-based anti-pathogen countermeasures that serve to restrict the replication of pathogens early in infection. In addition, a successful innate response promotes the secretion of cytokines and growth factors that shape the later pathogen-specific, or acquired immune response. The ability to mount a vigorous innate response and to transition to a highly-specific acquired response is thought to be essential for combating and controlling infection. The direct antiviral effect of type I IFNs is exerted by a variety of effectors that are expressed by genes whose transcription is directly stimulated by IFNs, the IFN-stimulated genes (ISGs). A number of innate immune effectors, the so-called restriction factors, have been identified that suppress retroviral replication in vitro and therefore may constitute new avenues for therapeutic intervention. Three of these innate retroviral restriction factors— apolipoprotein B mRNA editing enzyme, catalytic polypeptide 3 (APOBEC3), bone marrow stromal cell antigen 2 (BST-2/tetherin/CD317) and very recently, the dNTP hydrolase SAMHD1, have garnered substantial attention, since they specifically inhibit HIV-1 replication in vitro in human cells (reviewed in Harris et al., JBC, Oct 5th, 2012). In an attempt to identify novel candidate restriction factors, our lab has performed a siRNA screen (Fig. 3) and in addition performed tandem affinity purification followed by mass spectrometry to uncover novel interaction partners of accessory proteins of HIV and identified SAMHD1 as a novel restriction factor. These results corroborated and extended the findings of two other groups demonstrating that SAMHD1 is critical for restriction of HIV-1 infection in monocytes (Berger et al., PLoS Pathog. 2011 Dec;7(12):e1002425, Hrecka et al., Nature. 2011 Jun 29;474(7353):658-61, Laguette et al., Nature. 2011 May 25;474(7353):654-7.). SAMHD1 (Sterile Alpha Motif (SAM) Domain and HD domain-containing protein 1) was initially recognized as IFN-regulated gene and if mutated the cause for a severe neurodegenerative autoimmune disease called Aicardi-Goutières Syndrom (AGS). Patients carrying certain responsible mutations are suffering from neurological 172 Experiment V2 dysfunction, inappropriate immune activation and aberrant IFN-α secretion in the brain. The role as a novel HIV-1 restriction factor has pushed SAMHD1 recently into focus. It had been shown that SAMHD1 hydrolyses intracellular dNTPs in non-cycling cells, thus decreasing the amount of these key substrates, and thereby limiting viral replication (Fig. 4). Figure 4. Taken out of Review “Harris RS, Hultquist JF, Evans DT. THE RESTRICTION FACTORS OF HUMAN IMMUNODEFICIENCY VIRUS. J Biol Chem. 2012 Oct 5. [Epub ahead of print]” SAMHD1 acts to block HIV-1 reverse transcription by depletion of cellular dNTPs in non-cycling cells. HIV-2/SIV Vpx and some Vpr alleles can overcome the SAMHD1 restriction block by acting as an adaptor to an E3 ubiquitin ligase complex that polyubiquitinates SAMHD1 and targets it for degradation by the 26S proteasome. Figure 5. Genome-wide screen to identify host cellular proteins required for viral infection. An collection of siRNAs targeting approximately 24,000 human genes (6 siRNAs/gene) is arrayed in a multi-well plate such that each well contains 2 siRNAs targeting a single gene. These are transfected into mammalian cells, which are subsequently infected by viral particles genetically modified to encode luciferase. Putative host cell factors are identified through analysis of luciferase activity. Experimental outline Experiment 1 (Aim 1) To identify novel cellular factors affecting infection of cells by a VSV-G pseudotyped HIV-1 reporter virus vector, the method of choice is systematic high throughput cellular assays utilizing a genome-wide siRNA library. A counter-screen is necessary to identify those siRNAs that reduce or enhance cell growth/viability. 173 Experiment V2 Here, in this practical course, a 384-well plate-based assay will be optimized to identify siRNAs that influence cell viability. This could serve as a counterscreen for the screen depicted in Fig. 5. To optimize the conditions for the cell growth/viability assay: the optimal ratio of a positive control siRNA (targeting RPS27a, a highly essential gene for cell viability) and a negative control (siGL2, targeting luciferase, not targeting any cellular gene) will be determined. An example of a sequence of a siRNA is depicted in Fig. 6. Figure 6. siRNA against Luciferase The detection reagent ATP-lite will be added to the cell viability assay. ATPLite™ is an Adenosine TriPhosphate (ATP) monitoring system based on firefly (Photinus pyralis) luciferase. This luminescence assay is the alternative to colorimetric, fluorometric and radioisotopic assays for the quantitative evaluation of proliferation and cytotoxicity of cultured mammalian cells. ATP monitoring can be used to assess the cytocidal, cytostatic and proliferative effects of a wide range of drugs, biological response modifiers and biological compounds. Luciferase readouts are extremely attractive detection platforms for screening applications: wide dynamic range, more sensitive than fluorescence technologies, less interference, easy to automate for high-throughput applications and homogenous assay set up (Fig. 7). Figure 7. ATPlite readout Internal validation of the screens provides the assessment of positive control siRNAs to negative control siRNAs. In addition, we will determine the Z`-factor of the screen, a dimensionless calculation used to assess the quality of a high-throughput assay. It compares the mean value of the maximum signal control to the mean value of the minimum control and will have a higher value when (a) there is a wide separation 174 Experiment V2 band between maximum and minimum control and (b) the standard deviations are low. For a good assay, Z-prime values for each plate should be greater than or equal to 0.5. A perfect assay would have a Z-prime value approaching 1.0. In short, it is a measure for the actual assay window relative to the observed noise (Fig. 8). Z’ = 1 – (3*(STDEVNegControl + STDEVPositiveControl) / (MEANNegativeControl – MEANPositive Control) %CV= 100 x (Standard Deviation / Mean) Fig 8 Calculation of Z-prime and CV value Experiment Preparations: Experiment 1 (Aim 1) Have 1 white solid 384-well plate and 1 u-clear black 384-well plate ready with metal lids Calculate the amount of siRNA, transfection reagent (RNAiMax) and cells needed in respective mastermix volumes. Please refer to the respective sections below to determine the amount and volume you need for each step: Day 1 (Monday) Reverse Transfection protocol for 384-well plates: 1. Trypsinize a flask of adherent 293T cells Remove media, wash carefully the monolayer with PBS, remove PBS, add enough 0.25% w/v trypsin solution to cover the monolayer (1.5 ml for T75 flask), Let sit in incubator for ~3 minutes, then check flask under inverted microscope to make sure the monolayer of cells is lifting off of the flask and cells are single cells, add 8,5 ml DMEM-20 (=DMEM including 20% FCS), transfer to a 50 ml Falcon, count cells by transferring an aliquot of cells in a Eppendorf tube (20 ul), mixing it with 80ul trypan blue that stains non-viable cells, fill both sides of the haemocytometer chamber ‘Neubauer’ (see Fig. 9), count viable cells in each of the four corner squares, calculate cell density, prepare 1 cell solution with DMEM-20 (30 ml each) in 50 ml Falcons each. 2. Dispense 0.5 pmole siRNA (20 uM stock concentration) in 10 ul optimem 3. Prepare a Mastermix, for each well: 50nl RNAimax (Invitrogen) in 10 ul opti-mem/well and incubate at RT for 5 min 4. dispense 10 ul transfection reagent/well 5. Allow the transfection reagent and siRNA incubate for 20 min at RT 6. Dispense 20ul DMEM-20 with 3000 cells/well. 175 Experiment V2 Before you start, please review the plate layout and calculate the following (for each group): • Prepare a medium that contains 20% FCS, you need 40 ml in total, how much FCS should be in that 40 ml? • cell number and volume: in total you want to seed 1500 wells, each well with 3000 cells/well, 20 ul/well • amount of siRNAs in Mastermix: you have to calculate for 50 wells for each siRNA (siGL2 and siRPS27a), each well should contain 0.5 pmol siRNA (20 uM stock) in 10 ul • amount of transfection reagent (RNAimax): you have to calculate for 100 wells, each well should contain 50 nl RNAimax Day 4 (Thursday) Read-out: 1. add 30 ul of ATP-lite only to the white solid plate 2. measure luciferase RLU in Plate reader (Pherastar). 3. Visual check of the black u-clear plate (dead cells versus live cells) Plate layout: 176 Experiment V2 Figure 9. “improved Neubauer” haemocytometer Calculate the viable cell concentration per ml using the following formula: 4 C1 = t x tb x 1/4 x 10 t = total viable cell count of four corner squares tb = correction for the trypan blue dilution (counting dilution was 1/tb) 1/4 = correction to give mean cells per corner square 4 10 = conversion factor for counting chamber C1 = initial cell concentration per ml 4 Example: t = 480; tb = 2; C1 = 480 x 2 x 1/4 x 10 = 2.4 x 6 10 cells per ml Analysis Experiment 1 (Aim 1): 1) Graph each experiments (bar graphs) and standard deviations 2) Calculate ratio positive to negative controls for each condition 3) Calculate %CV 4) Calculate Z-Prime 5) Is the assay window and Z`Factor good enough for screening? 6) Which parameter could be optimized to improve the conditions for screening 177 Experiment V2 Experimental outline Experiment 2 (Aim 2) SAMHD1 has been identified as a novel cellular factor restricting HIV-1. In this practical course, you will test the efficiency of HIV-1 restriction by endogenous SAMHD1 and you will correlate this with the expression levels of SAMHD1. We will use THP-1 cells, a cell culture model for monocytic cells. The cells can be differentiated into macrophage-like cells using phorbol 12-myristate 13-acetate (commonly known as PMA or TPA). It had been shown before that after differentiation with TPA the cells became to a large extent refractory to HIV-1 infection. Just recently, it became clear, that the restriction is due to endogenous expression of SAMHD1. We generated stable THP1 cell lines carrying a vectorencoded shRNA targeting SAMHD1 and a control scrambled shRNA with a sequence not complementary to any known gene. In Figure 10 the vector map of the shRNA vector (pLKO) is depicted including the hairpin and the Puromycin selection marker that was used for selection of the stable THP1 cell lines. In this experiment, you will A) test the two cell lines (THP-1 shSAMHD1 and THP-1 shScrambled) on their endogenous expression of SAMHD1 by Western Blot analysis and B) infect the two PMA-differentiated cell lines with a lentiviral vector encoding luciferase and test the restriction capacity of SAMHD1. Figure 9. pLKO shRNA vector Name Description cppt Central polypurine tract hPGK Human phosphoglycerate kinase eukaryotic promoter puroR Puromycin resistance gene for mammalian selection SIN/LTR 3' self inactivating long terminal repeat f1 ori f1 origin of replication 178 Experiment V2 ampR Ampicillin resistance gene for bacterial selection pUC ori pUC origin of replication 5' LTR 5' long terminal repeat Psi RNA packaging signal RRE Rev response element Experiment Preparations: Experiment 2 (Aim 2) Have the two THP1 cell lines ready for the two parts of this experiment. In the first part (A) you will lyse part of the cells with a strong denaturing lysis buffer (RIPA) and you will separate the denatured proteins by electrophoresis. In a next step you will transfer the proteins to a membrane and stain SAMHD1 with a specific antibody. In the second part (B) you will seed live cells, differentiate the cells over night with PMA and then infect the cells with a luciferase expressing lentiviral vector. The infection can be measured by luciferase read-out. Day 1 (Monday) (A) Determine the cell number of the two cell lines THP1-shSAMHD1 and THP1-shScrambled by using a Neubauer counting chamber. To determine the cell number use an appropriate dilution of 1:5 with Trypan Blue (80µl Trypan Blue+ 20µl cell suspension).Transfer 1x106 of the cells in an Eppendorf Tube each and spin down the cells at 300g for 5min. (B) : Take the rest of the cells for seeding. (A) Lysis of the cells: Prepare 5ml RIPA-lysisbuffer add 1/2 tablet of Protease inhibitors to 5ml of RIPA-lysisbuffer Wash the cells with 500µl ice-cold PBS by spinning at 300g for 5min at 4°C Remove the supernatant and resuspend the cells in 100µl RIPA-lysisbuffer Incubate for 30min on ice!! Centrifuge at top speed at 4°C and afterwards transfer the supernatant to a new tube, freeze at -20°C (B) Seed 7500 cells/well in solid-white and u-clear 96-well plates in RPMI medium+10%FCS in 100 ul/well RPMI-10 medium. 179 Experiment V2 Add 5ng/ml PMA for differentiate the cells over night at 37°C Before you start, please review the plate layout (Fig 10) and calculate the following (for each group): you will have to seed each cell line in 200 wells, each well contains 7500 cells, 100 ul medium/well. How many cells do you need in total in how much medium in total for each cell line? You will need to add PMA to your medium before you seed the cells. How much PMA do you need to add for a final concentration of 5 ng/ml? The stock solution is 10 ng/ul. Day 2 (Tuesday) (A) Determine the protein concentration by Bradford-Assay: - Thaw the lysates on ice, - dilution of the samples for measurement 1:5; dilution of the reference (= RIPA) =1:5 - per sample: 200 µL 5x-Bradford reagent (Bio-Rad) is mixed with 800 µL H2O - 995 µL of the 1x-Bradford reagent are transferred to a plastic cuvette - 5 µL of the diluted samples are added; the solutions are mixed and incubated for 5 min at RT - detection at 595 nm with a UV/Vis spectrophotometer (GeneQuant pro, GE) protein concentrations are determined with the help of a previously prepared standard curve (A) Gel electrophoresis and Western Blotting of the samples 1) Protein samples (total protein amount: 20 µg) are prepared by adding NuPAGE LDS Sample Buffer (4X) and NuPAGE Sample Reducing Agent (10X) to a final 1X concentration each. 2) Denature the proteins at 70 °C for 10 min. 3) Prepare NuPAGE Bis-Tris gradient gels (4-12 %): Remove the gel from the pouch. Wash the gel with ddH2O. Peel of the tape from the bottom. 4) Place the gel (and a “dummy gel”) in the Mini-Cell such that the “well” side faces inwards, towards the inner chamber. Seat the gels on the bottom and lock into place with the wedge. 5) Fill the inner chamber with 1X MOPS running buffer (200 mL + 500 µL NuPAGE Antioxidant). 6) Pull the comb out of the gel. Rinse all wells with 1X MOPS running buffer. 7) Load the samples and protein marker. 8) Fill the outer chamber with 1X MOPS running buffer (600 mL). 9) Run the gel for 55 min at 200 V. 10) For Western blotting, prepare 1X NuPAGE Transfer Buffer as follows: 180 Experiment V2 Component Volume NuPAGE Transfer Buffer (20X) 25 mL NuPAGE Antioxidant 0.5 mL Methanol 50 mL 424.5 ddH2O mL Total volume 500 mL 11) In order to activate the PVDF membrane prior to blotting, place the membrane in methanol for 10 s, rinse with ddH2O and place it in 1X NuPAGE Transfer Buffer. 12) Soak the cathode (-) core of the blotting module, six blotting pads and two filter papers in 1X NuPAGE Transfer Buffer. 13) Place three blotting pads in the cathode core, followed by filter paper. 14) Remove the gel from the electrophoresis chamber, open the gel cassette and place the gel on top of the filter paper (without air bubbles!). 15) Place the activated PVDF membrane on the gel, followed by filter paper and three blotting pads. 16) Place the anode (+) core on top of the blotting pads and position the blot module in the buffer chamber. 17) The blot module and outer chamber were filled with 1X NuPAGE Transfer Buffer. 18) Conduct blotting 35 V for 1 h. 19) After blotting, rinse the PVDF membrane with 1X TBST. 20) Place the PVDF membrane in blocking solution (1X TBST + 5 % powdered milk) for 2 days at 4 °C. (B) Infection of PMA differentiated THP1 cell lines 1) Check the u-clear 96-well plate if the cells are differentiated 181 Experiment V2 2) Infect the cells with 50µl of CSII-Luc by spin occulation at 1200rpm for 1,5hours 3) After spin occulation the virus infected cells have to be incubated for two days at 37°C Fig. 10: Generation of lentiviral vector encoding luciferase (CSII-luc) Plate layout for infection: 182 Experiment V2 Day 4 (Thursday) (A) Antibody probing of the blot and detection To visualize proteins in the samples separated by SDS-PAGE and blotted onto a membrane, one chooses an antibody specific for the protein of interest (primary antibody) and applies subsequently a species-specific antibody (secondary antibody), which is directed against the Fc-part of the primary antibody and which is coupled to an appropriate enzyme e.g. horse radish peroxidase (HRP). • • • • We want to detect SAMHD1 protein on the blot membrane with a primary antiSAMHD1 antibody from rabbit (Proteintech) 1:1000 diluted in TBST + 5% milk powder (5 ml total volume needed). As a secondary antibody we will use anti-rabbit IgG-HRP (GE) 1:10000 diluted in TBST+5% milk powder (20 ml total volume needed). Typically GAPDH, tubulin or actin serve as loading controls, they are mostly expressed at a stable level and one of them is usually detected to proof, that variation in the levels of the protein of interest are not due to different amounts of loaded total lysate. We will detect GAPDH with an anti-GAPDH antibody from rabbit (Cell Signaling) 1:5000 diluted in TBST + 5% milk powder (5 ml total volume needed). The secondary antibody will be anti-rabbit IgG-HRP (GE) 1:20000 diluted in TBST+5% milk powder (20 ml total volume needed). Since SAMHD1 and GAPDH have different molecular masses, we will cut the membrane into two pieces (see Fig 11): One upper part where SAMHD1 should run (72kDa) and the other part, where we expect GAPDH (36 kDa) (compare picture). Each part gets incubated with the respective primary antibody for 1 h at RT under constant agitation. The blots get quickly rinsed with TBST three times and then washed three times with TBST for 10 min each. Thereafter the blots are probed with the secondary antibody (use correct dilution with the resp. primary antibodies) for 1 h at RT under constant agitation. The blots get quickly rinsed with TBST three times and then washed three times with TBST for 10 min each. The detection reagent will be ECL (Enhanced chemilumescent) prime from GE. It’s a substrate for detection of horseradish peroxidase enzyme activity: HRP catalyses the oxidation of luminol to 3-aminophtalate via several intermediates. The reaction is accompanied by emission of low-intensity light at 428nm. However, in the presence of certain chemicals, the light emitted is enhanced up to 1000-fold, increasing the sensitivity of the reaction. The ECL reagents need to be mixed in a 1:1 ratio prior to 183 Experiment V2 use, let them equilibrate to room temperature and incubate the mixed reagent for 5 min on the blot in the dark. Proceed with detection in the dark room: For detection of the luminescence reaction we will use photo films, which are placed on top of the membranes with varying detection times and are then developed. (B) Detection of infection Luciferase activity of the virus will be detected using BriteLite reagent (PerkinElmer) (Fig. 12) 1) Add 30µl of BriteLite/well to the white solid plate only 2) Measure luciferase RLU in plate reader (PheraStar) Read-out Analysis Experiment 2 (Aim 2): 1) Graph the infection experiment (bar graphs) and standard deviations 2) Scan the exposed film and mark the sizes of the marker and each lane 3) Is there a difference in expression of SAMHD1 between the two cell lines and what are the consequences on virus infection? 4) What is the cellular activity of SAMHD1 and how would you measure it? 5) What could be the reason that a virus can infect cells (to a certain amount) even in the presence of (high level) SAMDH1? 6) We used a shRNA that is complementary to the sequence of SAMHD1 to perform the experiment. Is there proof that SAMHD1 expression was silenced? Is this evidence enough that the shRNA is specifically targeting SAMHD1 and if not, which additional experiment would you perform? 7) We used stable THP1 cell lines generated with the pLKO vector construct. Look at the vector map and think about 2 different methods to introduce the vector to the cells to make the cells stable for shRNA expression. 8) Which different experiments would you undertake to identify novel restricting factors for virus infections? 184 Experiment V3 Experiment Virologie V3: Reduktion der Virusbelastung in PERV-infizierten humanen Zellen durch siRNAs und Nachweis von PERV-C mittels PCR Prof. Dr. Ralf Tönjes 185 Experiment V3 Ziele des Versuchs: 1. Ermittlung der absoluten Viruslast in infizierten Zell-Linien. 2. Nachweis der spezifischen Reduktion der Virusbelastung durch porzine endogene Retroviren (PERV) in Zellkultur-Überständen mittels PERVspezifischer siRNAs. 3. Nachweis von ekotropen PERV-C mittels PCR in genomischer DNA. Theorie: Hemmung der Genexpression durch siRNA 1990 wurde das Posttranscriptional Gene Silencing (PTGS) in Petunien (Solanaceae) gefunden, nachdem Forschergruppen um Mol und Jorgensen versuchten, die Blütenfärbung von Petunien durch das Einbringen eines zusätzlichen Pigmentgens zu verstärken und dabei das genaue Gegenteil erreichten. Es konnte gezeigt werden, dass die Gene nicht nur auf der Ebene der Transkription abgeschaltet wurden, sondern ebenso die zugehörige mRNA schnell abgebaut wurde. Etwas später wurden ähnliche Erkenntnisse bei Pilzen (Neurospora; „Quelling“), Würmern, Fliegen und Mäusen gewonnen (1, 2). Die Technik der RNA Interferenz (RNAi) wurde 1998 durch A. Fire und C. Mello in Caenorhabditis elegans nachgewiesen (1, 2, 3). Doppelsträngige RNA (dsRNA) führte zu einem effizienten und spezifischen Gene-Knockdown. Wird eine doppelsträngige (ds) RNA mit homologer Sequenz zu einer zelleigenen mRNA in eine Zielzelle eingebracht, startet der Vorgang des PTGS, indem Dicer, eine ATPabhängige Ribonuklease, die dsRNA in viele kleine Stücke schneidet. Es entstehen sogenannte small interfering RNAs (siRNAs). Diese siRNAs wandern zu einem Enzymkomplex, dem sog. RNA-induced-silencing-complex (RISC). Anhand der siRNA wird das komplementäre Stück der zelleigenen mRNA gefunden, zerschnitten und somit die Proteinsynthese des entsprechenden Gens unterbunden (Abb. 1). In höheren Eukaryoten führt lange, dsRNA (mehr als 30 Basenpaare) zu unspezifischen Auswirkungen bzw. zur Apoptose, da diese die enzymatische Zerstörung aller mRNAs und den Stop der Proteinsynthese bewirkt. Erst die Arbeit von S. Elbashir unter der Leitung von T. Tuschl konnte 2001 dieses Problem umgehen, indem kurze dsRNAs von 21 nt Länge verwendet wurden, die am 3’-Ende jeweils 2-3 Nukleotide überstehen. Solche kurzen siRNA-Moleküle können synthetisch hergestellt und in die Zielzellen eingebracht werden. Man steigt dadurch in den Prozess des PGTS zwar erst später ein, erzielt jedoch den gleichen Effekt (1, 2, 4; Abb. 2). Weiterführende Informationen zu siRNA und MicroRNA siehe http://opengenomics.org/CSB2005_RNAiTutorial/3-CSB2005-B&WSlides.pdf Die Lage der synthetischen siRNAs innerhalb der Genomstruktur von PERV, die im Versuch verwendet werden, ist in Abb. 3 dargestellt. Die Nukleotidangaben beziehen sich auf eine Referenzsequenz (PERV-B(33)ATG; Acc.No. AJ133816) und bezeichnen die Positionen, die von den siRNAs erkannt werden. 186 Experiment V3 Abb. 1: Modell der molekularen Schritte des Posttranskriptional Gene Silencing (PTGS), http://www.laborjournal.de/rubric/archiv/stichw ort/w_02_07.html (2) Abb. 2: RNA Interferenz (RNAi) mit siRNA, http://www.invitrogen.com/content.cfm?pageid= 10075 (5) Zum Versuch: PERV-infizierte humane Zellen (293/PERV-B(33)) werden 72 Std. vor Beginn des Praktikums mit PERV-spezifischen siRNAs (einzeln und in verschiedenen Kombinationen) und einer nicht PERV-spezifischen siRNA, sowie MOCK (nur Transfektionsreagenz, keine siRNA im Ansatz) transfiziert. Als Negativkontrolle dienen nicht-transfizierte Zellen. Aus den Überständen der transfizierten Zellen wird virale RNA von PERV mittels des High Pure Viral RNA Kit (Roche) isoliert. Daraufhin wird mittels quantitativer OneStep RT-PCR (LightCycler, Roche) die Anzahl der Virionen pro µl ZellkulturÜberstand bestimmt. Aliquots der Überstände werden bei –80°C eingefroren und dienen als Template für einen reversen Transkriptase (RT)-Test (C-type-RT Activity Assay, Cavidi Tech). Es soll gezeigt werden, dass es durch den Einsatz spezifischer siRNA's möglich ist, die Anzahl der Virionen im Zellkultur-Überstand zu verringern und, dass unspezifische siRNAs keinen Einfluss auf die Virusexpression haben. 187 Experiment V3 Genomstruktur von PERV Position der synthetischen siRNA‘s SA 5’-LTR gag 3’-LTR env pol SD poly A site PBS (Gly4) cap site 1154 2728 gag gag I 1245-1266 si 7 1414-1435 gag III 1949-1970 gag IV 2086-2107 Nukleotidangaben bezogen auf PERV-B(33)ATG (Czauderna et al. 2000) Abb. 3: Genomstruktur von PERV und Position der synthetischen siRNAs Nachweis von PERV-C mittels PCR Anhand der Ergebnisse einer Host Range und Interferenzstudie (14) wurden drei replikationskompetente PERV-Klassen erstmalig beschrieben: Polytrope PERV-A und PERV-B sind in der Lage nicht nur wirtseigene, sondern auch speziesübergreifende Zellen, einschließlich humaner Zellen, in vitro zu infizieren. Ekotropes PERV-C gehört zur bisher jüngsten entdeckten PERV-Klasse (15) und weist ein Wirtstropismus beschränkt auf porcine Zellen auf. Obwohl PERV-C an sich nicht xenotrop ist, stellt es trotzdem ein Risiko für die Xenotransplantation dar (11). PERV-C dient nämlich als Vorlage zur Rekombination mit PERV-A, wobei die resultierende Rekombinante, PERVA/C, die stärkste für PERV bislang detektierte Infektiosität aufweist, mit höherem Virustiter in Zellkultur im Vergleich zu den parentalen Viren (13, 16, 17). Ferner besteht die Gefahr, dass PERV-C in eine humantrope Variante evoluiert, da sich seine die Rezeptorbindedomäne in nur neun Aminosäuren von der von PERV-A unterscheidet (18). Ändern sich bloß vier dieser Aminosäuren durch Mutationen (19), wird der Eintritt von PERV-C in humane Zellen durch den PERV-A-Rezeptor (20) ermöglicht. Aus diesen Gründen ist es notwendig, alle replikationskompetenten PERV aus dem porcinen Genom zu eliminieren, bevor diese für die Xenotransplantation in Betracht gezogen werden. Deshalb wird im Rahmen dieses Praktikums porcine genomische DNA auf Vorkommen von PERV-C über sensitive nested PCR diagnostisch untersucht. Die Polymerase-Kettenreaktion (PCR) wurde zur selektiven Amplifikation von DNAAbschnitten mithilfe von spezifischen Oligonukleotiden entwickelt (8). Die PCRProgramme beginnen mit einer initialen Denaturierung, die dazu dient, die Doppelstränge der genomischen DNA aufzutrennen. Dann folgt ein Wiederholungszyklus (30x) aus einer Dreier-Schleife von Denaturierung, Annealing und Elongation. Durch die Hitze-Denaturierung der DNA werden Einzelstränge für die Oligonukleotide und die Polymerase zugänglich. Während des Annealing-Schrittes lagern sich spezifische Oligonukleotide an den zu amplifizierenden Genabschnitt. Beim Elongationsschritt entsteht das Amplifikat durch Polymerisation. Zum Schluss erfolgt ein finaler Elongationsschritt. 188 Experiment V3 Da PERV-C nur in sehr geringer Kopienzahl im Genom porciner Zellinien bzw. porciner DNA vorliegt (10, 12), reicht eine PCR meist nicht aus, um ein spezifisches Amplifikat zu erhalten. Daher schließt sich an die erste PCR eine nested PCR an mit spezifischen Oligenukleotiden, die innerhalb des Amplifikates der ersten PCR-Runde liegen. Durchführung: ACHTUNG: Die Transfektion wird nicht von den Praktikanten durchgeführt, die folgende Beschreibung dient nur zum besseren Verständnis des Versuches! Pro Reaktionskammer (well) werden am Vortag der Transfektion 2x105 293/PERV-B(33)-Zellen ausgesät. Ansätze (je 1 µg siRNA/well): siRNA 7 (Eurogentec), siRNA I, III u. IV (Dharmacon), je 20 µM siRNA einzeln: je 5 µl siRNA/Ansatz bei 2-er-Kombi: je 2,5 µl siRNA/Ansatz bei 3-er-Kombi: je 1,7 µl siRNA/Ansatz si7 + I si I + + IV III + IV si7 + I si7 1 2 3 7 I III IV untr. Zellen 4 5 6 10 Neg. control siRNA 8 MOCK 9 X X Abb. 4: Transfektionsschema Transfektion: • • • • • • • • • • die zu transfizierenden siRNAs zu je 0,1 ml Opti-MEM I (Invitrogen) in Polystyrolröhrchen geben (A) Mastermix aus 0,9 ml Opti-MEM I und 90 µl Transfektionsreagenz Lipofectamine ansetzen und vortexen (B) je Probe 0,1 ml B zu A geben, vortexen 15–30 min bei RT inkubieren, damit sich die siRNA-Lipidkomplexe bilden können in der Zwischenzeit Zellen 1x mit 2 ml Opti-MEM I pro well waschen je 0,8 ml Opti-MEM I zu A/B’s geben (= Transfektionsmix) je 1 ml Transfektionsmix pro well bei 37°C im Brutschrank 4-5 Std. inkubieren Transfektionsmix absaugen je well 2 ml Medium (DMEM + 10% FKS + Streptomycin/Penicillin + L-Glutamin) zugeben 189 Experiment V3 nach 72 Std.: Überstände (Supernatant, SN) zellfrei filtrieren, Isolierung der viralen RNA aus 200 µl zellfreiem SN, SN für RT-Test aliquotieren (je 1x 100 µl) I) Filtration der Überstände und Aliquotierung: • • • • • 2x 10 Eppi’s analog den 6-well-plates beschriften (also: si7+I+IV, siI+III+IV, … ODER von 1-10) [1x 10 Eppi’s für die Isolierung viraler RNA, 1x 10 Eppi’s für RTTest] 10 5-ml-Röhrchen beschriften und in Ständer einsetzen (= Auffanggefäße für filtrierte SN’s) je ca. 2 ml SN pro well mittels 2-ml-Spritze aufnehmen durch 0.45 µm Sartorius-Filter in vorbereitete 5-ml-Röhrchen filtrieren je 1x 100 µl zellfrei filtrierten SN in vorbereitete Eppi’s geben (für RT-Test), bei -80°C (Gruppe A) bzw. auf Eis (Gruppe B) lagern II) Isolierung viraler RNA (vRNA) (High Pure Viral RNA Kit, Roche): 1) 2) 3) 4) 5) 6) 7) 8) 9) 10) 11) 12) 13) 14) Arbeitslösung herstellen: 5 ml Bindepuffer (grüner Deckel) ergänzt mit 50 µl poly(A) carrier-RNA pro vorbereitetem Eppi (s.o.) je 400 µl Arbeitslösung vorlegen, je 200 µl zellfrei filtrierten SN zugeben und gut mischen (vortexen) Inkubation bei Raumtemperatur (RT) für 10 min High Pure Filter-Tubes in Auffanggefäße setzen und Proben in das obere Reservoir pipettieren 15 sec bei 10.000 rpm in Eppendorf-Tischzentrifuge zentrifugieren, währenddessen neue Auffanggefäße in Ständer stellen Filter-Tubes in neue Auffanggefäße setzen, alte Auffanggefäße samt Durchfluss verwerfen je 500 µl Inhibitor Removal Puffer (Gefäß 3a, schwarzer Deckel) in die oberen Reservoirs pipettieren (geht am besten mit einer Eppendorf Multipette und 12,5 ml-Combitip) 1 min bei 8.000 rpm zentrifugieren, währenddessen neue Auffanggefäße in Ständer stellen Filter-Tubes in neue Auffanggefäße setzen, alte Auffanggefäße samt Durchfluss verwerfen. Es folgen zwei Waschschritte. Für den ersten Waschschritt 500 µl Waschpuffer (blauer Deckel) in die oberen Reservoirs pipettieren (Eppendorf Multipette, 5 ml-Combitip) 1 min bei 8.000 rpm zentrifugieren, währenddessen neue Auffanggefäße in Ständer stellen Filter-Tubes in neue Auffanggefäße setzen, alte Auffanggefäße samt Durchfluss verwerfen. Für den zweiten Waschschritt je 400 µl Waschpuffer zugeben (Eppendorf Multipette, 5 ml-Combitip) 1 min bei 8.000 rpm zentrifugieren, anschliessend 10 sec bei 13.200 rpm zentrifugieren (um Reste des Waschpuffers zu entfernen), währenddessen sterile, Nuklease-freie 1,5 ml-Eppis beschriften Auffanggefäße verwerfen und Filter-Tubes in sterile, Nuclease-freie 1,5 mlEppis einsetzen 190 Experiment V3 15) Je 50 µl Elutionspuffer (Gefäß 4, farbloser Deckel) in die oberen Reservoirs pipettieren (Eppendorf Multipette, 2,5 ml-Combitip) 16) 1 min bei 10.000 rpm zentrifugieren 17) Filter-Tubes verwerfen, Eppis mit vRNA verschließen und auf Eis stellen (Gruppe A) bzw. bei –80°C lagern (Gruppe B). Die Proben dienen als Template für die One-Step RT-PCR mittels LightCycler. III) Quantitative One-Step RT-PCR (LightCycler, Roche): Die PCRs werden mittels des QuantiTect SYBR Green RT-PCR Kits (Qiagen, Cat. No.: 204243) durchgeführt. Die PERV RT-PCR dient zur Bestimmung der Virionenzahl im Überstand PERV-infizierter Zellen. 2x QuantiTect SYBR Green RT-PCR Master Mix, RNase-freies Wasser und Primer (F und R, je 10 µM) auftauen (im PCR-Raum), Template-RNAs (vRNA) und externen homologen Standard auf Eis auftauen (im Labor). Der PCR-MasterMix wird im PCR-Raum pipettiert, die Verdünnung des Standards und die Templatezugabe findet im Labor statt. Externen homologen Standard verdünnen (auf Eis; Wasser in Eppis vorlegen): Externer homologer Standard (= 326 bp T7-in-vitro transkribierte RNA): F/R (NdeI) [115 ng/µl] Berechnung der Kopienzahl: 6x1023 [Kopien/µl] x Konz. [g/µl] / MW [g/mol] MW (RNA) = Basepairs x 340 daltons/bp MW = 326 bp x 340 daltons/bp MW = 1,108x105 daltons Für die Konzentration von 115 ng/µl gilt also: 6x1023 x 1,15x10-7 / 1,108x105 = 6,23x1011 copies/µl Verdünnungsreihe (auf Eis!): 1:6,2 (d.h. 1 µl RNA + 5,2 µl H2O) = 1x1011 copies/µl seriell 1:10 verdünnen von 1010 bis 103 8 Eppis vorbereiten (beschriften von 1010-103) je 18 µl DEPC-H2O vorlegen + 2 µl aus der Lösung mit 1x1011 copies/µl in Eppi 1010, mix + 2 µl aus der 1010-Verdünnung in Eppi 109, mix + 2 µl aus der 109-Verdünnung in Eppi 108, mix usw. bis Eppi 103 Als Standard einsetzen:103 - 109 copies/µl Proben als Doppelbestimmung einsetzen! 191 Experiment V3 MasterMix ansetzen (PCR-Raum!): pro Probe: 5,8 µl RNase-freies Wasser 10 µl 2x QuantiTect SYBR Green RT-PCR Master Mix 1 µl Primer F (0,5 µM Endkonz.) 1 µl Primer R (0,5 µM Endkonz.) 0,2 µl QuantiTect RT Mix 18 µl Gesamtvolumen gesamt (n+2 = 30): 174 µl RNase-freies Wasser 300 µl 2x QuantiTect SYBR Green PCR Master Mix 30 µl Primer F 30 µl Primer R 6 µl QuantiTect RT Mix 540 µl vortexen 18 µl pro Kapillare + je 2 µl Standard bzw. Template-RNA (erst im Labor zugeben!) Probenreihenfolge: 1: 2: 3: 4: 5: 6: 7: 8: 9: 10: 11: 12: 13: 14: Wasser Standard 2 x 103 16: Standard 2 x 104 17: Standard 2 x 105 18: Standard 2 x 106 19: Standard 2 x 107 20: Standard 2 x 108 21: Standard 2 x 109 22: Probe 1 Repl. von Probe 1 Probe 2 Repl. von Probe 2 Probe 3 Repl. von Probe 3 15: Probe 4 Repl. von Probe 4 Probe 5 Repl. von Probe 5 Probe 6 Repl. von Probe 6 Probe 7 Repl. von Probe 7 23: Probe 8 24: Repl. von Probe 8 25: Probe 9 26: Repl. von Probe 9 27: Probe 10 28: Repl. von Probe 10 Kapillaren mit Deckel verschließen und mit Proben zum LightCycler gehen. Probenkarussell mit Kapillaren beladen und in Zentrifuge kurz zentrifugieren. Probenkarussell in LightCycler platzieren. PCR: RT-Reaktion Denaturierung Amplifikation (40 cycles) Schmelzkurve Cooling 50°C 95°C 94°C 58°C 72°C 20 min 15 min 15 sec 15 sec 16 sec 95°C 65°C 97°C 10 sec 10 sec 0 40°C 30 sec slope 2°C/s, single measurement slope 0,1°C/s, cont. measurement (wenn nicht anders angegeben: slope 20°C/s) Nach Beendigung des PCR-Laufs werden die Proben entsorgt und es erfolgt entweder direkt die Auswertung der Daten oder aber am zweiten Tag (je nach Gruppeneinteilung). 192 Experiment V3 IV) RT-Test (C-Type-RT Activity Assay, Protokoll B, Cavidi-Tech) Bestimmung der Enzymaktivität der reversen Transkriptase im Überstand PERV-infizierter Zellen (d.h. in den Viruspartikeln, die sich im Überstand befinden). C-Type Sample Dilution Buffer C-Type Reconstitution Buffer bei 37°C im Wasserbad auftauen Benötigte Anzahl poly(A)-beschichteter Streifen (siehe Tab.1), C-Type Sample Dilution Buffer, 2x C-Type Sample Dilution Components (Flaschen mit weißem Deckel), C-Type RT Reaction Components (Flasche mit grünem Deckel) und MMuLV-rRT Standard (Flasche mit gelbem Deckel, Lot-Nr. 11071) aus –20°C nehmen. Für Gruppe A: Proben aus -80°C nehmen und auf Eis auftauen. Reaktionsmix herstellen: • • • • • 50 ml Falcon beschriften (Reaktionsmix, Datum) 12 ml C-Type Reconstitution Buffer zu einer Flasche C-Type RT Reaction Components geben, gut mischen und in 50 ml Falcon geben 6 ml Wasser zugeben C-Type Sample Dilution Buffer herstellen (je 5 ml C-Type Sample Dilution Buffer pro Flasche C-Type Sample Dilution Components, gut mischen und Inhalt beider ’Flaschen wieder in die C-Type Sample Dilution-Flasche geben, mischen) 6 ml Sample Dilution Buffer in 50 ml Falcon geben, mischen (=Reaktionsmix) Poly(A)-Platten vorbereiten: • • • • entsprechende Anzahl 8-er-Streifen in 96-well-plate-Rahmen spannen (Tab. 1) pro well 200 µl Reaktionsmix mit elektronischer Mehrkanalpipette zugeben Platten mit selbstklebender Folie schließen Präinkubation der Platten für 20-60 min bei 33°C 193 Experiment V3 1 A B C D E F G H 2 3 4 1 407,1 mU/ml 2 180,9 mU/ml 3 80,4 mU/ml 4 35,7 mU/ml 5 15,9 mU/ml 6 7,06 mU/ml 7 3,14 mU/ml 8 1,39 mU/ml Tab. 1: Probenauftragung RT-Test MMuLV-Standard verdünnen (gilt für Lot-Nr. MUL 11071 !): • • • • • • 420 µl Sample Dilution Buffer zu MMuLV-Lyophilisat geben, gut mischen 8 Eppi’s vorbereiten, je 250 µl Sample Dilution Buffer vorlegen 200 µl resuspendierten MMuLV Standard ins erste Eppi geben, vortexen Pipettenspitze wechseln, 200 µl aus dem ersten ins zweite Eppi geben, vortexen Pipettenspitze wechseln, 200 µl aus dem zweiten Eppi ins dritte geben, vortexen, usw. bis Eppi Nr. 8 Standardverdünnungen auf Eis stellen RT-Reaktion starten: • • • • • Poly(A)-Platten aus 33°C-Inkubator nehmen Standard (je 10 µl/well) in die erste Reihe pipettieren (von oben nach unten abnehmend, entsprechend Tab. 1) Proben zugeben (als Duplikate, Pipettierschema ist selbst zu entwerfen und in Tab. 1 einzutragen), je 10 µl/well Platten mit selbstklebender Folie verschließen Platten für 2-3 Std. bei 33°C inkubieren 194 Experiment V3 Platten waschen: • • • • Waschpuffer (2.5 ml Waschpufferkonzentrat, 75 ml 10% Triton X-100 ad 1 l H2O) Platte im ELISA-Washer 3x mit 200 µl Waschpuffer pro well waschen (Programm RT, last strip = 4) Platte umgedreht auf Celltorks aufklopfen Platte kann jetzt entweder eingefroren (-20°C, Gruppe B) oder der Versuch fortgesetzt werden (Gruppe A) Inkubation mit Product Tracer: • • • • 12 ml 1% Triton X-100 zu einer Flasche C-Type RT Product Tracer geben, vortexen benötigt ca. 10-20 min, bis sich das Lyophilisat gelöst hat, zwischendurch vortexen je 100 µl Product Tracer pro well zugeben Inkubation bei 33°C für 90 min Inkubation mit AP-Substrat: • • • • • • Flasche mit Substrat Puffer bei 37°C im Wasserbad auftauen Substrat Tablette zu Substrat Puffer (Flasche mit Alufolie umwickeln) geben, vortexen (braucht ca. 10 min zum Lösen) Platte im ELISA-Washer 3x mit 200 µl Waschpuffer pro well waschen (Programm s.o.) Platte umgedreht auf Celltorks aufklopfen je 125 µl AP-Substrat pro well, Platten mit Kunststoffdeckel schließen, im Dunkeln 10-20 min inkubieren Platten im ELISA-Reader messen (A405) V) PCR PERV envC Templates: • je zwei genomische DNAs von PERV-C-positiven (#1 und #3) und PERV-C-negativen (#112 und #114) Schweinen • genomische DNA der Zelllinien ST-IOWA, ST-IOWA/PERV-C(1312) und HEK-293/5° Name Nukleotide Position PERV-C (1312) (Preuss et al., 2006) PERV-C-PCR Env-C for CTGACCTGGATTAGAACTGG Env-C rev ATGTTAGAGGATGGTCCTGG Env-C for2 GTGCTCTCCTTCAGACCTAGATTAC Env-C rev2 CGTCAAGACCGTATTTGGTCC Tab. 2: Primerliste EnvC-PCR, Diplomarbeit M. Rodrigues Costa 195 6606 – 6625 6867 – 6886 6635 – 6659 6822 – 6842 Experiment V3 Die PCR wird mit Hilfe der Taq DNA Polymerase von NEB (Cat.No. M0267) durchgeführt. Die PERV-envC-PCR dient als diagnostisches Werkzeug zur Bestimmung, ob PERV-C in potentiellen Spendertieren für Xenotransplantation vorhanden ist oder nicht. Alle Proben werden in Doppelbestimmungen durchgeführt. Die für die PCR benötigten Reagenzien werden im PCR-Raum aufgetaut. Dort werden auch die MasterMixe für die zwei PCR-Runden hergestellt und in PCR-Röhrchen aliquotiert, die Zugabe der Templates findet im Labor statt. MasterMixe ansetzen (PCR-Raum!): pro Probe: Gesamt: Master Mix1: (n+1=17) Gesamt: Master Mix 2: (n+1=17) Gesamt: 17,375 µl 2,5 µl 0,5 µl 0,5 µl 0,5 µl 2,5 µl 0,125 µl 24 µl 1 µl Wasser 10x ThermoPol Reaction buffer, NEB 10 mM dNTPs 10 µM envC for Primer 10 µM envC rev Primer 10x Betaine Taq DNA Polymerase, NEB 295,375 µl 42,5 µl 8,5 µl 8,5 µl 8,5 µl 42,5 µl 2,125 µl 408 µl je 1 µl Wasser 10x ThermoPol Reaction buffer, NEB 10 mM dNTPs 10 µM envC for Primer 10 µM envC rev Primer 10x Betaine Taq DNA Polymerase, NEB 24 µl pro tube Template DNA (50 ng) 295,375 µl 42,5 µl 8,5 µl 8,5 µl 8,5 µl 42,5 µl 2,125 µl 408 µl je 1 µl Template DNA (50 ng) Wasser 10x ThermoPol Reaction buffer, NEB 10 mM dNTPs 10 µM envC for 2 Primer 10 µM envC rev 2 Primer 10x Betaine Taq DNA Polymerase, NEB 24 µl pro tube Template DNA (50 ng) Die Amplifikation der DNA erfolgt in Biometra Thermocyclern TPersonal bzw. TGradient unter folgenden Bedingungen: 196 Experiment V3 PCR 1: Denaturierung Amplifikation (30 cycles) Finale Elongation Cooling 95°C 95°C 55°C 68°C 68°C 4°C 30 sec 30 sec 30 sec 15 sec 5 min ∞ Nachdem die erste PCR beendet ist, wird jeweils 1 µl des erhaltenen Amplifikats als Template für die nested PCR eingesetzt. Nested PCR: Denaturierung Amplifikation (30 cycles) Finale Elongation Cooling 95°C 95°C 63°C 68°C 68°C 4°C 30 sec 30 sec 30 sec 15 sec 5 min ∞ Die Analyse der erhaltenen Amplifikate erfolgt über ein 1%-iges Agarosegel (mit 2 dicken 20-er-Kämmen) in TBE-Puffer. Die Amplifikate werden mit je 5 µl 6x Blaupuffer versetzt und die Proben auf das Gel geladen. Elektrophorese: 110 V, 60 min. Auswertung: a) Berechnung der Anzahl der PERV Virionen in den Proben und Darstellung im Balkendiagramm (Mittelwerte bilden und Standardabweichung [Fehlerbalken] eintragen) Anhand der PERV-Standardreihe der RT-PCR ermittelt die LightCycler-Software die RNA-Kopien in 2 µl Probe. Viruspartikel enthalten einen doppelten Satz virale Nukleinsäure (2x vRNA), daher muss dieser Wert durch 4 geteilt werden, um die RNA-Kopienzahl pro µl Probe zu erhalten. RNA-Kopienzahl (in 2 µl) / 4 = Anzahl der Viruspartikel pro µl Probe b) Berechnung bzw. grafische Auswertung (Balkendiagramm aus Mittelwerten der Proben, Standardabweichung eintragen) des RT-Tests c) Vergleich der erhaltenen Daten von RT-PCR und RT-Test d) Ergebnis der diagnostischen PCR Frage: Kann durch spezifische siRNA eine Reduktion der Virusbelastung in PERVinfizierten humanen Zellen qualitativ und quantitativ erreicht werden? 197 Experiment V3 Literatur: 1) 2) 3) 4) 5) 6) 7) 8) 9) 10) 11) 12) 13) 14) 15) 16) 17) 18) 19) http://de.wikipedia.org/w/index.php?title=RNA-Interferenz&printable=yes Hien K. RNA-Interferenz (RNAi). Laborjournal-Ausgabe 07/2002 aus http://www.laborjournal.de/rubric/archiv/stichwort/w_02_07.html Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC: Potent and specific genetic interference by double stranded RNA in Caenorhabditis elegans. Nature 391, 806-811, (1998). Elbashir SM, Harborth J, Lendeckel W, Yalcin A, Weber K, Tuschl T: Duplexes of 21nucleotide RNAs mediate RNA interference in mammalian cell culture. Nature 411, 494498 (2001). http://www.invitrogen.com/content.cfm?pageid=10075 http://opengenomics.org/CSB2005_RNAiTutorial/3-CSB2005-B&WSlides.pdf Laborjournal 11/2008, S. 60-63 Saiki RK, Scharf S, Faloona F, Mullis KB, Horn GT, Erlich HA, Arnheim N.: Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia. Science. 1985 230(4732):1350-1354. http://de.wikipedia.org/wiki/Polymerase-Kettenreaktion Preuss T, Fischer N, Boller K, Tönjes RR.: Isolation and characterization of an infectious replication-competent molecular clone of ecotropic porcine endogenous retrovirus class C. J Virol. 200680(20):10258-61. Denner J, Tönjes RR (2012): Infection barriers to successful xenotransplantation focusing on porcine endogenous retroviruses. Clin Microbiol Rev 25: 318-343. Kaulitz D, Mihica D, Dorna J, Costa MR, Petersen B, Niemann H, Tönjes RR, Denner J (2011): Development of sensitive methods for detection of porcine endogenous retrovirus-C (PERV-C) in the genome of pigs. J Virol Methods 175: 60-65. Kaulitz D, Mihica D, Plesker R, Geissler A, Tönjes RR, Denner J (2011): Absence of infection in pigs inoculated with high-titre recombinant PERV-A/C. Arch Virol 156: 707-710. Takeuchi Y, Patience C, Magre S, Weiss R, Banerjee P, Le Tissier P, Stoye P (1998): Host range and interference studies of three classes of pig endogenous retrovirus. J Virol 72: 9986-9991. Niebert M and Tönjes RR (2005): Evolutionary spread and recombination of porcine endogenous retrovirus in the Suiformes. J Virol 79: 649-654. Wilson C, Wong S, van Brocklin M, Federspiel M (2000): Extended analysis of the in vitro tropism of porcine endogenous retrovirus. J Virol 74: 49-56. Oldmixon B, Wood J, Ericsson T et al. (2002): Porcine endogenous retrovirus transmission characteristics of an inbred herd of miniature swine. J Virol 76: 3045-3048. Gemeniano M, Mpanju O, Salomon M, Eiden V, Wilson A (2006): The infectivity and host range of the ecotropic porcine endogenous retrovirus, PERV-C, is modulated by residues in the C-terminal region of its surface envelope protein. Virol 346: 108-117. Argaw T, Figuerosa M, Salomon D, Wilson A (2008): Identification of residues outside of the receptor binding domain that influence the infectivity and tropism of porcine endogenous retrovirus. J Virol 82: 7483-7491.18) 20) Ericsson T, Takeuchi Y, Templin C et al. (2003): Identification of receptors for pig endogenous retrovirus. PNAS 100: 6759-6764. 198