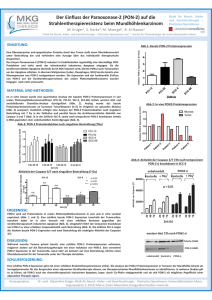
Etablierung eines MYCN-vermittelten murinen
Werbung

EtablierungeinesMYCN‐vermittelten murinenNeuroblastommodellsundAnalyse tumorsuppressivermikroRNAs Inaugural‐Dissertation zur ErlangungdesDoktorgrades Dr.rer.nat. derFakultätfür Biologie ander UniversitätDuisburg‐Essen vorgelegtvon KristinaAlthoff ausHerne August2013 Die der vorliegenden Arbeit zugrunde liegenden Experimente wurden im hämatologisch‐onkologischenLaborderKinderklinikIIIamUniversitätsklinikumEssen durchgeführt. 1.Gutachter: Prof.Dr.JohannesSchulte 2.Gutachter: Prof.Dr.BertramOpalka VorsitzenderdesPrüfungsausschusses: Prof.Dr.PeterBayer TagdermündlichenPrüfung: 12.Dezember2013 Inhaltsverzzeichnis Inha altsverzzeichnis 1 EIINLEITUNG G.................................................................................................................................1 1.1 DasNeurroblastom............................................................................................................................1 1.2 Molekula areMarkerrundRisiko ostratifikattion.......................................................................2 1.3 DieMaussalsNeuroblastom‐M Modellorgan nismus..................................................................4 1.3 3.1 Modelleemitneuralle eisten‐speziffischer,stabilerÜberexpre essioneinesO Onkogens.......................4 1.3 3.2 KonditiionaleModelle....................................................................................................................................................5 1.4 MikroRN NAs..........................................................................................................................................7 1.4 4.1 DieBiogeneseundF Funktionvon nmiRNAs..........................................................................................................7 1.4 4.2 DieRollevonmiRNA AsbeiderPaathogenesedeesNeuroblasttomsundandderen Tumoreentitäten................................................................................................................................................................8 2 ZIIELDERAR RBEIT....................................................................................................................12 3 M MATERIALUNDMETH HODEN...............................................................................................13 3.1 Tierexpe erimentelle eMethoden n................................................................................................13 3.1.1 GenerieerungderLSL L‐MYCNMau s.......................................................................................................................13 vonLSL‐MYCN N;Dbh‐iCreM MäusenundD DetektionvonTumoren..............................................16 3.1.2 Zuchtv handlung.....................................................................................................18 3.1.3 XenogrraftsundNanopartikel‐Beh 3.2 Molekula arbiologisccheMethod den...........................................................................................18 3.2 2.1 Polymeerase‐Kettenrreaktion...................................................................................................................................18 3.2 2.2 Agarose‐Gelelektrop phorese....................................................................................................................................20 3.2 30‐Survivin.............................................21 2.3 KlonierrungderKonstruktepDESST30‐KDM1AundpDEST3 3.2 2.4 Bestimm mungderKonzentrationvvonDNAund dRNA...................................................................................21 3.2 2.5 Isolieru unggenomisccherDNA.................................................................................................................................22 3.2 2.6 mRNA‐‐undmiRNA‐‐Isolierung..............................................................................................................................22 3.2 2.7 ReverseeTranskriptiionvonkomp plementärerDNAausisoliiertermRNA undmiRNA...............22 3.2 2.8 QuantittativePCR...........................................................................................................................................................22 3.2 2.9 Array‐b basiertecomp parativegenoomischeHybrridisierung........................................................................24 3.2 2.10 mRN NAqPCRproffiling..........................................................................................................................................24 3.2 2.11 miRN NAqPCRprofiling.........................................................................................................................................25 3.3 Biochem mischeMeth hoden............................................................................................................26 3.3 3.1 HerstelllungvonPro oteinlysatenu undBestimm mungihrerKonzentration...........................................26 3.3 3.2 Natrium mdodecylsulffat‐Polyacrylaamidgelelekttrophorese.........................................................................26 3.3 3.3 WesterrnBlotundim mmunologisch herNachweisvonProtein nen.............................................................27 3.3 3.4 Proteom manalyse............................................................................................................................................................29 3.3 3.5 HerstelllungvonCalccium‐Phosph hat‐Nanoparttikeln....................................................................................29 3.4 HistologischeMethoden.............................................................................................................30 3.4 4.1 Histologischeundim mmunhistoch hemischeFärrbungenvonG Gewebeschniitten..............................30 3.4 4.2 Elektro onenmikrosko opischeUnteersuchungenv vonGewebesschnitten.................................................30 3.4 4.3 Immun nfluoreszenzfä ärbungenvon nProteineniinhumanenZ Zelllinien.................................................31 I 3.5 Inhaltsverzzeichnis Zellbiolo ogischeMetthoden..........................................................................................................31 3.5.1 KultivieerungvonZellen............................................................................................................................................31 ktionenvonm miRNAundsiiRNA....................................................................................32 3.5.2 TransieenteTransfek abilenZellkloonenundIndu uktionvonko onditionalen Konstrukten n.............33 3.5.3 Generieerungvonsta kopischeAuffnahmenvonZellen...........................................................................................................34 3.5.4 Mikrosk bilitäts‐Assay y...................................................................................................................................................34 3.5.5 Zellviab ose‐Assay.............................................................................................................................................................35 3.5.6 Apopto y...................................................................................................................................................35 3.5.7 Proliferrations‐Assay klusanalyse.........................................................................................................................................................36 3.5.8 Zellzyk 3.5.9 LuziferaseReporterrAssay......................................................................................................................................36 3.6 GensetE EnrichmentAnalyse,sttatistischeM Methodenu undBildbeaarbeitung...........37 3.6 6.1 GensetEnrichmentA Analyse....................................................................................................................................37 3.6 en.................................................................................................................................................38 6.2 StatistischeAnalyse 3.6 6.3 Bildbeaarbeitung............................................................................................................................................................38 4 ER RGEBNISSE..............................................................................................................................39 4.1 DasLSL‐MYCN;Dbh‐‐iCreMausm modell....................................................................................39 4.1.1 Generieerungdertra ansgenenMau uslinieLSL‐M MYCN.....................................................................................39 YCNBeobachttungskohortee.......................................................................................................................39 4.1.2 LSL‐MY ypisierungen.......................................................................................................................................................40 4.1.3 Genoty otoxizitätdessGenotypsLSSL‐MYCN;Dbh h‐iCre...................................................................................40 4.1.4 Embryo n........................................................................................................................41 4.1.5 Tumoriinzidenzund‐lokalisation histologieundExpression nspezifischerrNeuroblasto ommarker..............................................47 4.1.6 Tumorh Dbh‐iCreMau usmodellsmiitderdesetablierten 4.1.7 VergleichderTumorinzidenzdessLSL‐MYCN;D CNMausmod dells............................................................................................................................................48 TH‐MYC h‐iCreTumorren.............................................................49 4.1.8 GenomischeAberrationenvonLSSL‐MYCN;Dbh 4.1.9 mRNAProfiling..............................................................................................................................................................51 4.1.10 miRN NAProfiling......................................................................................................................................................54 4.1.11 miR‐‐542alspote entielltumorm modulierendeemiRNAimN Neuroblastom m.....................................57 4.2 Dietumo orsuppresssiveFunktio onvonmiR R‐542inhum manenNeu uroblastom men undZellllinien..................................................................................................................................58 4.2 2.1 ExpresssionsanalysevonmiR‐542 2undSurviviininhumanenPrimärtum moren.............................58 4.2 2.2 Funktio onvonmiR‐5 542‐3pExpreessioninZelllinien....................................................................................62 4.2 2.3 RegulattionderExprressionvonS urvivindurch hmiR‐542‐3p p.................................................................66 4.2 2.4 DirekteeBindungvon nmiR‐542‐3p panSurvivin n3‘UTR................................................................................67 4.2 2.5 Funktio onvonSurviv vinHerunterrregulationinZelllinien..........................................................................68 4.2 2.6 PartiellleWiederherstellungdesP 3pExpressionnmittels PhänotypsdeermiR‐542‐3 HochreegulationvonSurvivinohn nemiRNA‐Bin ndestelle............................................................................71 4.2 2.7 MiR‐54 42‐3pbeladen neNanoparti kelzurmiRN NA‐Behandlun ngvonNeurooblastomzelle eninvitro undinvvivo........................................................................................................................................................................72 II 4.3 Inhaltsverzzeichnis Dietumo orsuppresssiveFunktio onvonmiR R‐137inhum manenNeu uroblastom men undZellllinien..................................................................................................................................77 4.3 3.1 KDM1A Aalspotentie ellesZieleine rNeuroblasttomtherapie......................................................................77 4.3 3.2 ExpresssionsanalysevonmiR‐137 7inhumanen nPrimärtumo oren..........................................................79 4.3 3.3 Funktio onvonmiR‐1 137inZellliniien...................................................................................................................80 4.3 3.4 ValidierrungvonKDM M1AalsdirekktesZielvonmiR‐137inZ Zelllinien.................................................83 4.3 3.5 Funktio onvonKDM1 1AHerunterr egulationinZ Zelllinien...........................................................................85 4.3 3.6 PartiellleWiederherstellungdesP PhänotypsdeermiR‐542‐3 3pExpressionnmittels Ho ochregulation nvonKDM1A AohnemiRNA A‐Bindestellee.............................................................................................87 4.3 3.7 IdentifiizierungundValidierungvvonAKAP2aalsweiteresZielvonmiR‐1137.................................88 5 DISKUSSION N..............................................................................................................................91 5.1 DasLSL‐MYCN;Dbh‐‐iCreMausm modell....................................................................................91 5.1.1 TumoreedesLSL‐MY YCN;Dbh‐iCreMausmodelllslassensichalsmurineN Neuroblastom ma beschreeiben.....................................................................................................................................................................91 überstellungd derbeidenNeeuroblastom‐MausmodellleLSL‐MYCN N;Dbh‐iCreun nd 5.1.2 Gegenü TH‐MYC CN..........................................................................................................................................................................93 erZieleeiner Neuroblastomtherapiean nhanddesLSSL‐MYCN;Dbh h‐iCre 5.1.3 Identifiikationenure Mausm modells...................................................................................................................................................................96 heWeiterentw wicklungend desLSL‐MYCN N;Dbh‐iCreM Modells,dieneeueErkenntn nissefür 5.1.4 Möglich dieForshcungamhu umanenNeu roblastomgeebenkönnten n...............................................................103 usmodellsfürrdieGenesea andererTum orentitäten.............105 5.1.5 DieRelevanzdesLSL‐MYCNMau 5.2 Tumorsu uppressivemiRNAsim mNeuroblastom................................................................106 5.2 2.1 Dietum morsuppressiveFunktionvvonmiR‐542 2imNeurobla astom....................................................107 5.2 2.2 Dietum morsuppressiveFunktionvvonmiR‐137 7imhumanen nNeuroblastoom…...........................111 6 ZU USAMMEN NFASSUNGUNDAUSB BLICK...........................................................................114 7 ABSTRACT.................... . ........................................................................................................1146 8 LIITERATUR R.............................................................................................................................117 9 ABKÜRZUN NGEN.....................................................................................................................131 10 ABBILDUNG GS‐UNDTA ABELLENV VERZEICHN NIS.............................................................133 10.1 Abbildun ngsverzeich hnis.............................................................................................................133 10.2 Tabellen nverzeichniis...................................................................................................................136 11 ANHANG....................................................................................................................................137 11.1 Danksag gung....................................................................................................................................137 11.2 PublikattionenundKongressb eiträge................................................................................138 11 1.2.1 Publlikationen........................................................................................................................................................138 11 1.2.2 Konggressbeiträge e................................................................................................................................................139 11.3 Lebensla auf......................................................................................................................................141 11.4 Erklärun ngen...................................................................................................................................142 III Ein nleitung 1 Eiinleitun ng 1.1 D DasNeurroblasto om Das Neuroblastom m ist der häufigste h soolide Tumo or des Kind desalters uund verantw wortlich für ca. 15% aller pädiatriscch‐onkologiischen Tod desfälle. Die Prävalennz beträgt 1:7.000, die Inzzidenz 1,3 Neuerkran nkungen aauf 100.000 0 Kinder unter u 15 JJahren, wo obei das mittlereeAlterbeiDiagnoseb bei17,4Moonatenliegt1,2.Neurob blastomeenntwickelnsichaus primitivvenNeurallleistenzelllen,dieenttlangdes sympathisc s henGrenzsstrangesso owieim Nebenn nierenmark klokalisierrtsind3.Hisstologisch wirddasN Neuroblastoomzusamm menmit dem Rh habdomyossarkom, de em Non‐Hoodgkin Lym mphom und d dem Ewinng Sarkom m zu den klein‐, blau‐, rund dzelligen Tumoren T ggezählt1. Ess handelt sich um eiinen heterrogenen, bösartigenTumorr,dersichb beigünstiggerBiologiee(Stadien1 1,2und4s))häufigauchohne Therap pie komplettt zurückbildet oder differenzieert, während eine unngünstige Biologie B (Stadien 3 und 4) 4 trotz Anwendung m modernsteer, multimo odaler Theerapie nich ht selten zum To od des Paatienten fü ührt4. Die Einteilung der Stadiien erfolgtt nach dem m 1988 entwick kelten5 und 1993 modifizierte m en6 Interna ational Neu uroblastom ma Staging System (INSS).Diemittlerre5‐Jahress‐Überleben nsratebetrrägtca.59% %,wobeidiiesestarkm mitdem m assoziiert ist7,8 (T Tab.1). Naach einem Rückfall wird jedo ch eine 5‐Jahres‐ Stadium Überleb bensrate von v nur 8‐31% 8 posstuliert9‐11 und aktu uelle Beha ndlungspro otokolle können n nur 25‐3 35% der Patienten mit hohem m Stadium m heilen4. Demnach ist die Notwen ndigkeitfürrneuezielg gerichteteN Neuroblasttomtherapiiensehrgrooß. 1 Ein nleitung Tab.1:IInternation nalNeurobllastomaSta agingSystem(INSS)3,5,,6 Stadium Definiition 1 2a 2b 3 4 4s lokalissierterTum mor,keinBeefallderLy ymphknoten, vollstäändigeRese ektion lokalissierterTum mor,keinBeefallderLy ymphknoten, unvollständigeResektion lokalissierterTum mor,Befalld deripsilateeralenLymphknoten, vollstäändigeoderrunvollstän ndigeReseektion Tumorrinfiltration n über d die Mittellinie ode er Befall der kontraalateralenL Lymphknotten, inoperrabel Tumorr mit Disssemination nen zu entfernten Lymphknooten, Knoch hen, Knoch henmark, Leber, Haut H und/oder andderen Organeen lokalissierter Prim märtumor mit Disseminationen n begrenztt auf Haut,L Leberund/ /oderKnocchenmark, beschrränktaufK Kinder<1Jaahr 5‐Ja ahres‐ Übe erleben >90 0% >90 0% >90 0% ~75 5% <20 0% ~75 5% 1.2 M Molekula areMarrkerund dRisiko ostratifik kation Neben demPatien ntenalterb beiDiagnosse,demTu umorstadiumsowieddenSerum‐‐Ferritin Spiegeln12,1 3 gibt es genetische g und biologgische Marrker, die und Laktatdehydrrogenase‐S prädikttivfürdenV VerlaufderrErkranku ungsind. EineAm mplifikationdesOnko ogensMYC CN(v‐myc myelocytom m matosis viraal related oncogen, o neurobllastoma derived), d lokalisiert l auf Chro omosom 2p24, ist bei 20% % aller Neurob blastome detektierb bar und mit fortgeschritte enem Staddium, sch hnellem Tumorw wachstum und schlecchter Progn nose assozziiert14,15. DNA‐Sequen D nzanalysen n zeigen nurinsseltenenFäällenMutationeninM MYCNkodierrendenReg gionen16,w wasvermutenlässt, dass daas erhöhte Maß an trranskriptioonell aktiveem Wildtyp p Gen, dass mit hohen n MYCN mRNA Leveleinhergeht,zurrTumorforrmationfüh hrt.MYCN agiertzusaammenmittseinen beiden Co‐Faktorren MAD und MAX X als Tran nskriptionssfaktor un d beeinflu usst die ngundMig grationalsaauchbeiApoptose ExpresssionvonGeenen,diesowohlbei Entwicklun undPro oliferationeineRollespielen17. Die Exxpression des Neu urotrophin nrezeptors NTRK1 korreliert t mit nie edrigem Tumorsstadium und günstig ger Prognoose von Neuroblasto N ompatienteen18, währe end die ExpresssiondesNeeurotrophinrezeptorssNTRK2m mitschlechte erPrognosseassoziierrtist19. Diemeistenfamilliärenund 10%derssporadischeenNeurobllastomeweeisenaktiv vierende Mutatio onen der Anaplastisc A hen Lymph hom Kinasse (ALK) au uf. Die ALK KF1174L Muta ation ist 2 Einleitung nichtnureinederhäufigervorkommendenMutationen,sondernauchdieMutationmit dergrößtenKorrelationzueinemaggressivenKrankheitsverlauf20‐22. Die prognostische Aussagekraft eines teilweisen oder kompletten Zugewinns von Chromosom 17q, der bei 63‐83% aller Neuroblastome auftritt, ist umstritten23. Innerhalb dieser Region liegt das Gen BIRC5 (Baculoviral IAP repeat‐containing 5), welches für das Protein Survivin kodiert23,24. Im Vergleich zu Normalgewebe, MalignomenbeiErwachsenenundnicht‐malignenfetalenNeuroblastenderNebenniere ist die Expression von Survivin im Neuroblastom erhöht. Die Überexpression von Survivin im Tumor korreliert außerdem mit einer ungünstigen Prognose der Patienten25,26.SurvivininteragiertmitAuroraBKinase(AURKB),INCENPundBorealin, die zusammen den chromosomalen Passenger Komplex (CPC) bilden. Dieser Komplex reguliertSchlüsselereignissewährendderMitosewiedieKorrekturvonFehlernbeider AnheftungderChromosomenandieMikrotubuli,dieAktivierungvonKontrollpunkten beim Aufbau des Spindelapparates und die Konstruktion und Regulation des kontraktilenApparates,derdieZytokineseantreibt27.AußerdemstabilisiertSurvivindie MikrotubulidurchdirekteBindungandiese28. Die Lysin‐spezifische Demethylase 1 (KDM1A), die die Entfernung der einfachen oder doppeltenMethylierungsowohlvonLysin4alsauchvonLysin9anHiston3katalysiert, spielteineSchlüsselrolleinderRegulationvonGenenwährendderEntwicklung29.Eine Überexpression von KDM1A, die mit erhöhter Zellproliferation einhergeht, wird in verschiedenen Tumorentitäten postuliert30‐34. In primären Neuroblastomen ist die Expression von KDM1A invers mit Differenzierung und günstigem Krankheitsverlauf korreliert. Die Herunterregulation von KDM1A in humanen Neuroblastomzelllinien führtaußerdemzurReduktionderZellviabilität35. 3 Ein nleitung 1.3 D DieMaussalsNeu uroblasstom‐Mo odellorg ganismu us Tiermo odelle hum maner Erkrrankungen können für f das Ve erständnis der mole ekularen Mechan nismen, diee der Path hogenese d der Erkran nkung unte erliegen, seehr hilfreich sein. Aufgrun nd der kurzen Generation G szeit, derr geringe en Größe,, der einfachen Haltunggsbedingun ngen,derÄ ÄhnlichkeittzumMenschenund deskomp lettsequen nzierten Genoms stellt diie Labormaus (Mus musculus) eines derr geeignettsten Mode elle zur Untersu uchungvon nKrebserkrankungen ndar36.FürrdasKrank kheitsbildddesNeurob blastoms werden n verschied dene gentechnisch‐veeränderte Mausmodel M lle beschrieeben, die allesamt a aufdie Überexpreessioneinesbestimmtten,fürdieeseTumore entitätrelev evanten,Transgens bei wird zwischen konventio onellen unnd kondittionalen zurückzzuführen sind. Dab Mausm modellenun nterschiede en. 1.3.1 M Modelle mit m neuralleisten‐sspezifische er, stabiler Übereexpression n eines O Onkogenss 1.3.1.1 DasTH‐M MYCNMode ell Basiereend auf der Erkenntn nis, dass e ine MYCN‐‐Amplifikattion beim Neuroblasttom ein häufigeesEreignis darstelltgenerierten nWeisseta al.1997da asTH‐MYCN N‐Mausmod dell.Ein Plasmid dkonstruktt wurde en ntwickelt, in das cD DNA für hu umanes M MYCN hinte er einen Tyrosin n Hydroxyllase (TH) Promotor P aaus der Raatte ligiert wurde. M Mittels Pron nukleus‐ Injektio onwurded dasPlasmid dinmurin neBlastozy ysteneingeb brachtunddtransgene eMäuse generieert37. Der Promotorr ist, wie schon 19 992 besch hrieben w wurde, sow wohl im Zentrallnervensysttem als au uch in den n sympath hischen Ganglien undd der Neb benniere aktiv38. Mittels dees TH‐MYC CN Mausmoodells konn nte erstmals gezeigt w werden, da ass eine ÜberexxpressionvonMYCNd dasPotentiialbesitztd dieEntsteh hungmurinnerNeuroblastome zu treiben, da trransgene Mäuse M diesser Linie klein‐, blau‐ und ruundzellige Tumore entwick keln, die unifokal u au us dem G Ganglion co oeliacum entstehen. e Die Inzide enz der Neurob blastomentw wicklungh heterozygotterTiereliiegtim129 9x1/SvJStaammbei70%und imC57Bl/6NStam mmbei5% %.Kommtd dasTransgeenhomozy ygotvor,lieegtdieInzidenzim 129x1//SvJStamm mbeiüber9 90%37. 4 Einleitung 1.3.1.2 DasTH‐ALKF1174LModell Berry et al. generierten das TH‐ALKF1174L Mausmodell, indem sie humanes ALKF1174L hinter den TH Promotor aus der Ratte klonierten und dieses Konstrukt in murine Blastozysten injizierten. Keine der transgenen Tiere entwickelt einen Tumor, während die Verkreuzung mit der TH‐MYCN Maus jedoch zu einer signifikanten Steigerung der Tumorpenetranz dieses Modells führt, vergleichbar mit der homozygoter TH‐MYCN Tiere39. 1.3.2 KonditionaleModelle Das Cre‐lox‐Rekombinationssystem ermöglicht eine genetische Modifikation spezifischer Zell‐ und Gewebearten. Die Cre‐Rekombinase wird dabei hinter einen spezifischenPromotorkloniertundnurindenZellenexprimiert,indenenderPromotor aktiv ist. Ein Gen, das von loxP Sequenzen flankiert ist, wird in Cre‐exprimierenden Zellen aufgrund der Rekombinase‐Aktivität ausgeschnitten. In konditionalen Mausmodellen wird diese Technik zum einen zum Stilllegen von Genen und zum anderen zur Entfernung von Sequenzen, die die Transkription von Transgenen verhindern,verwendet.ImletzterenFallwirddasTransgennachdemAusschneidender loxPSequenzenexprimiert.Eskommthäufigvor,dassnichtalleZellendesZielgewebes dieCre‐Rekombinaseexprimieren,waszueinemmosaikartigenAusschneidenderloxP Stellenführt.DiesesPhänomenspiegeltdieSituationeinesspontanenTumorswider,bei dem ebenfalls nicht alle Zellen des Gewebes, aus dem dieser entsteht, die Mutation aufweisen. Die Aktivität des Promotors, der die Expression der Cre‐Rekombinase antreibt, ist häufig nicht nur auf die Ziel‐Zelltypen beschränkt, was eine genaue UntersuchungdesExpressionsmustersdeszuverwendendenCre‐Modellsunabdingbar macht. Etablierte konditionale Neuroblastommodelle nutzen das Dbh‐iCre Mausmodell zur spezifischenExpressiondesjeweiligenOnkogens.DieCre‐Rekombinasestehthierunter Kontrolle des murinen Dopamin beta‐Hydroxylase (Dbh) Promotors. Stanke et al. zeigten anhand der Verkreuzung mit einer ROSA26 Reportermaus, dass die Cre‐ Rekombinase im Dbh‐iCre Modell ausschließlich in noradrenergen Neuronen des peripherenunddeszentralenNervensystemsexprimiertwird40(Abb.1). 5 Ein nleitung Abb.1: Expression der Cre e‐Rekombin nase im Dbh‐iCre Mausmodell Verk kreuzung mit m einer ROSA26 Reporter R Mauslin nie; β‐Galaktosidase Färbung iin sympatthischen Ganglien n von Emb bryonen (E1 16,5) zur D Detektion der d Cre‐ vermitteelten lacZ Z Expression: (A, B) positiiv im sympath hischen Greenzstrang, (C) negativ in Schweißdrüsen‐ gewebe..ModifizierttnachStank keetal.40. 1.3.2.1 DasLSL‐L Lin28b;Dbh h‐iCreModeell Anhand dderErken nntnis,dasseinehoh eExpressionvonLIN N28Homol ogB(LIN2 28B)mit schlech htem Gesam mtüberlebe en von Neeuroblastom mpatienten n assoziierrt ist, gene erierten Molenaaar et al. ein konditio onales Mau usmodell zur z Überexpression ddes murine en Gens. Verkreu uzt mit dem Dbh h‐iCre Moodell entw wickeln LSL‐Lin28b L Mäuse murine Neurob blastome, die ein ne hohe Expressiion von MYCN aufweisen n. Die Neurob blastomgen nese diesess Modells w wird über die Hochrregulation von MYCN N durch Lin28bvermitteltt41. 1.3.2.2 DasLSL‐A ALKF1174L;Dbh‐iCreMoodell mp et al.. entwickelten ein kondition nales Neu uroblastom mmodell, das d bei Heukam F Verkreu uzung mit einer Cre‐‐Maus hum manes ALKF1174L übere exprimiert.. In vier vo on zehn Tieren konnte die Entwiicklung eiines Neurroblastomss beobachhtet werde en. Die uzung mit dem TH‐M MYCN Mod dell führte zu einer signifikante s en Steigeru ung der Verkreu Tumoriinzidenz42. 6 Ein nleitung 1.4 M MikroRN NAs MikroR RNAs (miRN NAs) sind kurze, hocch‐konserv vierte, nichtt‐kodieren de RNA Moleküle, die die Expression von Zielgenen mit komplemeentären Se equenzen rregulieren können. Über 8 800 verscchiedene humane m miRNAs konnten k bisher b ide ntifiziert werden (www.m mirbase.org) und es wird verm mutet, dasss bis zu 30 0% aller huumanen Ge ene Ziel einerR Regulationd durchmiRN NAssind43. 1.4.1 D DieBiogen neseundF FunktionvvonmiRNA As Die Bio ogenese vo on miRNAss (Abb.2) beginnt mit m der Tra anskriptionn des miRN NA‐Gens durch d die RNA Polymerase P e II, wobei die primääre miRNA A (pri‐miRN NA) gebild det wird (500–3000 Basen n)44,45. Die e erhalten ne Haarnad del‐Struktu ur wird voon Drosha a, einer Endonu ukleasederrRNaseIIIFamilie,aabgespalten nunddiecca.70Base nlangeVo orläufer‐ miRNA (pre‐miRN NA) freigessetzt46. Hieerzu bildett Drosha mit m seinem Co‐Faktor DGCR8 (DiGeorrgesyndrom mecriticalregiongen e8)einenKomplex,d derauchallsMikropro ozessor‐ Kompleexbekannttist47.Mitte elsdesRan n‐abhängigeenKerntransport‐RezzeptorsExp portin‐5 wird d die pre‐m miRNA auss dem N Nukleus in n das Zyttoplasma exportiertt48. Die zytoplaasmatische RNaseIII Dicerprozzessiertdieepre‐miRN NAinca.222Nukleotid delange miRNA‐Doppelstrränge49.Die eausgereift ftemiRNAw wirdmittellseinerRN NA‐Helicase einzwei Einzelsträngekon nvertiert50 undlagert sichmitdemRNA‐in nduzierten silencingK Komplex mplexzusam mmen.Wäh hrendder33‘Einzelstrrangder (RISC) zummiRNA‐RISCEffektor‐Kom miRNA(3p‐Isoforrm)häufigdegradiert twird,interragiertder5‘Einzelsttrang(5p‐Isoform) mitdem mRISC‐Kom mplex51,52. DiemiRNA Aerkenntk komplemen ntäreSequeenzenspezzifischer Ziel‐mR RNAs in deer 3‘untranslatierten n Region (U UTR), meisstens an d er 2‐8 Nuk kleotide langen Bindestellle am 5‘‘Ende, un nd der miRNA‐RISC m C‐Komplex unterdrückt die Genexp pression durch d Inhibierung der Transslation od der Indukktion von mRNA Degrad dation53,54.JJedemiRNA Akannanm mehrerem mRNAsbind denundjeddemRNAkannvon verschiiedenen miiRNAs regu uliert werd den. Mittelss in silico Analysen A kkönnen pottentielle Ziele vvon miRNA As vorherg gesagt weerden (ww ww.targetsccan.org). Jeedoch beruht die biologissche Funkttionalität der d miRNA A nicht nurr auf der Bindung aan komplem mentäre Basen,sondernw wirdauchdu urchanderreFaktoren n,wiederA Anzahlder Zielsequen nzender umliegeendenSequ uenzenderrmRNA55u undderOk kkupationv vonBindesstellendurcchRNA‐ bindend deProteine56,beeinfllusst.Demzzufolgeist esnotwen ndigvorherrgesagteZielevon 7 Ein nleitung miRNAs in biolo ogischen Experiment E ten, beispiielsweise mittels m Luuziferase Reporter R dieren. Analyseen,zuvalid Abb.2: DieBiogen nesevonmiiRNAs(a)T Transkriptio ondesmiRN NAGensinpprimäremiR RNA(pri‐ miRNA))durchRNA APolymeraseII(PolII);Abspaltung gderhaarna adel‐förmigeenVorläuferr‐miRNA hDrosha.(b b)ExportinssZytoplasm madurchExportin‐5unnd(c)Proze essierung (pre‐miRNA)durch Dicer; Inkorporation in den RNA‐iinduzierten silencing Komplex K (RIISC). (d) spezifische durch D mRNA D Degradation n oder Inhib bierung derr Translation von Prote einen. Modiifiziert nach h Garzon etal.57. 1.4.2 D DieRollev vonmiRNA Asbeider rPathogen nesedesNe euroblasto omsunda anderen T Tumorenttitäten MiRNAs spielen eine wichttige Rolle in verschiiedenen ze ellulären M Mechanism men, wie Proliferration, Apo optose, Zelllwachstum m und Zellzyklus, weshalb es niccht verwun nderlich ist, dasss sie auch h mit Krebserkrankun ngen in Veerbindung gebracht w werden. Abhängig davon, ob sie mRNAs m reg gulieren, d die tumorssuppressiv ve oder onnkogene Proteine P ppressivem miRNAs58. kodiereen,wirkensiealsonkogeneoderrtumorsup 8 Einleitung 1.4.2.1 DasonkogenemiR‐17‐92‐Cluster Das bekannteste Beispiel für die Beteiligung von miRNAs an der Pathogenese des Neuroblastoms ist das miR‐17‐92‐Cluster, das sechs miRNAs umfasst (miR‐17,‐18a,‐19a,‐19b‐20aund‐92),dieinNeuroblastomzelllinienmithoherMYCN Expression stärker exprimiert werden als in Zelllinien mit niedrigem MYCN Level. Mittels Luziferase Reporter Analyse konnte gezeigt werden, dass MYCN in verschiedenen Genregionen des miR‐17‐92‐Clusters direkt bindet und die miRNAs hochreguliert. Eine ektopische Expression des miR‐17‐92‐Clusters in der NeuroblastomzelllinieSK‐N‐AS,dienureineKopiedesMYCNGensträgt,führtzueiner gesteigerten Proliferation der Zellen59. Auch in primären Neuroblastomen konnte eine HochregulationinHoch‐Risiko‐Patientengezeigtwerden60,61. 1.4.2.2 TumorsuppressivemiRNAs Die erste miRNA, deren tumorsuppressive Funktion im Neuroblastom beschrieben wurde, ist miR‐34a, die auf der chromosomalen Region 1p36.23 lokalisiert ist. Besonders in humanen Neuroblastomen, die eine MYCN Amplifikation aufweisen, liegt diese Region häufig homozygot deletiert vor. Tumore mit 1p Deletion weisen eine niedrige Expression von miR‐34a auf und die exogene Expression der miRNA in Neuroblastomzelllinien führt zu einer Reduktion der Proliferation und induziert Apoptose62,63. Interessanterweise weist die 3’UTR von MYCN zwei funktionelle BindestellendermiR‐34aaufunddieBindungdermiRNAandieseBindestellenführtzu einer Herunterregulation von MYCN auf Proteinebene64. Es wird vermutet, dass genau diese Regulation den Phänotyp der miR‐34a Überexpression erklärt65. Chen et al. beschreibeneinenweiterenSignalwegindieserAchse.Siekonntenzeigen,dassMYCNin Neuroblastomzelllinien direkt die Expression von Survivin durch Bindung an den SurvivinPromoterreguliert66. Schulte et al. analysierten die Expression von 430 miRNAs in 69 Tumoren von NeuroblastompatientenmittelsmultiplexHochdurchsatz‐qPCR67.AnhandeinerKaplan‐ Meier Analyse können diese Neuroblastompatienten anhand der miRNA Expressionsprofile in Langzeit und‐ Kurzzeit‐Überlebende separiert werden. Auf Basis der 20 miRNAs mit der höchsten positiven und der 20 miRNAs mit der höchsten negativen Beteiligung, die zu einer effizienten Separation der Patienten führt, wurde eine nicht‐überwachte Clusteranalyse durchgeführt (Abb.3). Beide Isoformen der miR‐542sindinTumorenmithohemStadiumniedrigerexprimiertalsinTumorenmit 9 Ein nleitung günstigger Biologie. Die miR R‐542 Exprression ist signifikant niedrigerr in Tumoren mit MYCNA AmplifikatiionimVergleichzuT Tumorenm mitnureine erKopievoonMYCNssowiein Tumoreen von Paatienten mit m Rückfal l im Verg gleich zu Patienten P m mit Tumorr‐freiem Überleb ben67. ung von Neuroblasto ompatienten mit gute er und sch hlechter Prognose Abb.3: Auftrennu anhand dvonmiRN NAExpressiionsprofilen nZwei‐dim mensionaleC Clusteranalyysebasieren ndauf40 miRNAss. Schwarze Balken=Pa atienten miit Rückfall. Grüne G Pfeile e=Expressiion der miR R‐542‐3p 67 undmiR R‐542‐5p.M Modifiziertna achSchulte etal. . 10 Einleitung Bray et al. beschreiben ebenfalls eine Korrelation zwischen fehlender Expression von miR‐542‐5p und sowohl schlechterem Ereignis‐freiem als auch schlechterem Gesamt‐ überleben in 144 Neuroblastompatienten68. Nach exogener Expression dieser miRNA kannzwarkeinUnterschiedinderProliferationsratederNeuroblastomzelllinienKelly, SK‐N‐AS und NB1691 detektiert werden, aber sowohl Kelly als auch SK‐N‐AS Zellen reagieren mit signifikant verringerter Invasivität durch eine Matrigel‐Matrix. Die Expression von miR‐542‐3p induziert eine Reduktion der Proliferationsrate der humanen Lungenkarzinomzelllinie A549, der humanen Zervixkarzinomzelllinie HeLa und der humanen Brustkrebszelllinie MCF769. Des Weiteren reduziert miR‐542‐3p die FähigkeitvonA549ZellenzurKoloniebildung.SowohlinA549alsauchinHeLaZellen konntegezeigtwerden,dassSurvivineindirektesTargetvonmiR‐542‐3pist. EinweiteresBeispielfüreinemiRNAmittumorsuppressiverFunktionistmiR‐137,die aufdemhumanenChromosom1p22lokalisiertist.MiR‐137istinverschiedenenhuma‐ nen Tumorentitäten, wie dem Glioblastom70, dem Kolorektalkarzinom71, dem Mund‐ höhlenkarzinomunddemPlattenepithelkarzinomdesKopfesundNackensherunterre‐ guliert72. Die exogene Expression von miR‐137 in den Mundhöhlenkarzinomzelllinien HSC‐6undHSC‐7reduziertderenWachstumundKonfluenzinvitro72. 11 ZielderArbeit 2 ZielderArbeit ZieldieserDissertationwares,einneueskonditionalesMYCNvermitteltesMausmodell zugenerierenundzuetablieren.FolgendeFragestellungensolltenbearbeitetwerden: 1. Entwickeln LSL‐MYCN;Dbh‐iCre Mäuse Tumore, die als murine Neuroblastomebeschriebenwerdenkönnen? Anhand der Lokalisation sowie der histologischen und molekularen Eigenschaften der Tumore,diesichintransgenenLSL‐MYCN;Dbh‐iCreMäusenentwickeln,solluntersucht werden, ob sich das Modell als murines Neuroblastommodell eignet. Weiterhin sollen verschiedene Bildgebungsmethoden angewendet werden, um das Modell genauer zu charakterisieren. 2. Wie unterscheiden sich die Tumore des LSL‐MYCN;Dbh‐iCre Modells von denendesetabliertenTH‐MYCNModells? IndiesemTeilderArbeitsollendieEigenschaftendesneuenLSL‐MYCN;Dbh‐iCreMaus‐ modells mit denen des etablierten Neuroblastommodells TH‐MYCN verglichen werden, umGemeinsamkeitenundUnterschiedeherauszuarbeiten. 3. Kann das LSL‐MYCN;Dbh‐iCre Mausmodell genutzt werden, um neue Ziele einerNeuroblastomtherapiezuidentifizierenundsinddiesevalidierbar? Anhand des LSL‐MYCN;Dbh‐iCre Mausmodells sollen potentiell tumorsuppressive miRNAs detektiert werden, die sowohl bei der Genese als auch für die Therapie des Neuroblastoms eine Rolle spielen könnten. Nach erfolgreicher Identifikation soll die funktionelle Bedeutung ausgewählter miRNAs in vitro in etablierten humanen Neuro‐ blastomzelllinienanalysiertwerden.DazusollendiemiRNAsektopischexprimiertund die phänotypischen Auswirkungen auf die Zellen untersucht werden. Des Weiteren sollenausanderenTumorentitätenbekannteZielgenediesermiRNAsimNeuroblastom validiertwerden. 12 MaterrialundMe ethoden 3 MateriallundM Methode en Chemik kalienund Verbrauchsmaterialieenwurden,fallsnichttanderserrwähnt,von ndenin Deutsch hland verttretenen Firmen Carrl Roth Gm mbH & Co..KG (Karlssruhe), App pliChem GmbH (Darmstad dt)oderSig gma‐Aldrich hChemieG GmbH(Tau ufkirchen) bezogen.D DieSyn‐ ukleotidprim mernerfolggtebeiEurrofinsMWG GOperon(E Ebersberg)). thesevonOligonu Alle Pu uffer und Lösungen wurden m mit deionissiertem, Millipore‐ger M reinigtem Wasser angesettzt. Puffer und Lösungen, die unter sterrilen Bedin ngungen Veerwendung g finden sollten,,wurdenvo orihrerBe enutzungau utoklaviertt.pH‐Werte ewurdenm mitHClbzw w.NaOH eingesttellt. 3.1 T Tierexpe erimenttelleMethoden Alle im m Rahmen dieser d Arbe eit verwen ndeten Mäu use wurden n im zentrralen Tierla aborato‐ rium dees Universitätskliniku ums Essen gehalten. Die Tiere lebten in eeinem hell‐‐dunkel‐ Rhythm musvonje1 12Std.und dbeieiner konstanten nRaumtem mperaturvoon20±2°C. Wasser undpellletiertesFutter(SsniffSpezialdiiätenGmbH H,Soest)errhieltendieeTiereadllibitum. 3.1.1 G Generieru ungderLSL L‐MYCNMaaus Die LSL L‐MYCN Maus M wurde e im Auftrrag des häämatologiscch‐onkologgischen Labor der Kinderk klinikIIIbeiderFirm maTaconicA ArtemisGm mbH(Köln n)unterreggelmäßigerrgegen‐ seitigerrAbsprachegeneriertt. 3.1.1.1 Konstruk ktiondesLS SL‐MYCNV ektors or mit Rekkombinasee‐vermitteltter Kassetttenaustausschstelle In eineen geschüttzten Vekto (RCME))wurdehu umaneMYC CNcDNA(G Gen‐ID:ENS SG0000013 34323)hinntereinenH Hühner‐ Aktin‐P Promotor (CAG) ( sowiie eine loxxP‐flankiertte Polyaden nyl‐Stoppseequenz (LS SL) und vor ein ne interne ribosomale r e Eintrittssstelle (IRES S) sowie eiin Luziferaase Gen (Fluc) klo‐ niert(A Abb.4(II)).. 13 MaterrialundMe ethoden Abb.4: Schemade erVektorko onstruktion nzurGenerierungtra ansgenerLSSL‐MYCNM Mäuse(I) Schema desROSA26Genlokus derF1RMC CE103emb bryonalenM MausstammzzellenmitR Rekombi‐ Kassettenaustauschstelllen(RCME),,benutztum mden(II)CA AG‐LSL‐MYC CN‐IRES‐ nase‐verrmitteltenK Fluc‐RM MCE‐Austaucchvektorein nzubauen;d derROSA26 Genlokusnachder(III))Insertion deskon‐ ditionalen Allels un nd (IV) dem m Cre‐Rekoombinase‐veermittelten Ausschneidden der Polyadenyl‐ G = Hühner‐‐Aktin‐Prom motor; hGHp pA = Polyad denyl‐Stoppssequenz, he ergestellt Stoppseequenz. CAG aushum manemWacchstumshorm mon; NMYC =offenesL Leserasters vonhumannemMYCN; A1/A2 = PrimerzzurAmplifik kationdesM MYCN‐Transsgens;B1/B B2=PrimerzurValidierrungdesAu usschnei‐ densderrPolyadeny yl‐Stoppsequ uenz.ModifiiziertnachM MilestoneRe eportTaconiicArtemisG GmbH. ungvonLSL‐MYCNtraagendenem mbryonalen nStammzeellen 3.1.1.2 Generieru 03embryon naleMaussstammzelle en(ES‐Zelleen)mitdem mgene‐ TaconiccArtemisF1RMCE10 tischen n Hintergru und 129S6 6;C57Bl6/N N, die im ROSA26 R Genlokus übber RMCE Stellen erten,muriinenFibrob blasten‐ verfügtten(Abb.4(I)), wurdenaufeineermitotiscchinaktivie schicht kultiviert.DieKotransfektiond derES‐Zelllenmitdem mCAG‐LSLL‐MYCN‐IRE ES‐Fluc‐ RMCE‐A Austauchveektor und einem Flp pe‐Rekomb binase exp primierendden Vektorr (pCAG Flpe pA A) erfolgte mittels Lip pofektion u unter Verw wendung eiines Standaardprotoko olls. Der AustausschderKaassettenerffolgtandeenPositionenFRTundF3.PosittiveKlone wurden siebenTagelangm mitG418selektiertun ologischverschiedeneeKolonienwurden ndmorpho isoliert.MittelsSo outhernBlo otAnalysew wurdediekorrekteR Rekombinattionüberprrüft.Die dedientezu umNachwe eisderkor rektenRek kombinationin5‘‐RichhtungdesV Vektors, 5‘‐Sond die3‘‐SSondefürd diekorrekte eRekombi nationin3 3‘‐Richtung g.AnhanddderHybridiisierung mitderrneo‐SondeewurdediieEinzelinttegrationü überprüft.D DieverwenndetenSond denund die zzu erwartenden Fragmenttgrößen bei Verrdau mitt verschiedenen 14 MaterialundMethoden Restriktionsenzymen sind in einer Tabelle zusammengefasst (Tab.2). Validierte Klone wurdenvermehrtundinflüssigenStickstoffgelagert. Tab.2: Sonden für die Southern Blot Analyse Modifiziert nach Milestone Report TaconicAr‐ temisGmbH. Sonde Enzyme Wildtyp‐Allel F1RMCEAllel LSL‐MYCNAllel HindIII EcoRI PciI AvrII PciI StuI HindIII EcoRI AseI 4,4 15,6 9,5 5,3 9,5 4,9 ‐ ‐ ‐ 3,9 4,3 16,4 12,1 16,4 8,3 ‐ ‐ ‐ 7,0 7,8 13,1 6,6 6,7 15,3 7,0 7,8 4,5 5‘ 3‘ neo Fragmentgröße[kb] 3.1.1.3 GenerierungvonchimärenMäusen Tetraploide Maus‐Blastozysten wurden aus superovulierten Balb/c Weibchen (Tag 3 post coitum) isoliert. In 60 Blastozysten wurden je 7‐10 rekombinante ES‐Zellen (C57Bl/6N)injiziert.Je20BlastozystenwurdendanndreischeinträchtigenWeibchenin den Uterus implantiert, woraufhin 10 chimäre Nachkommen geboren wurden. Die chimärenNachkommen,diesichsowohlausdentransgenenES‐Zellenalsauchausden Zellen der Blastozyste entwickelten, wurden weitergezüchtet und keimbahnmutierte männlicheTiere(schwarze/weißeFellfarbe)durchVerpaarungmitC57Bl/6NWeibchen vermehrt. Der Nachweis der Keimbahntransmission erfolgte anhand der schwarzen Fellfarbe. Das Transgen wurde mittels LSL‐MYCN Primern in einer PCR amplifiziert (s.3.2.1–Tab.3),umdenEinbauzuvalidieren.HeterozygoteLSL‐MYCNMäusewurden an unsere Arbeitsgruppe transferiert (Abb4(III)) und durch Verpaarung mit Wildtyp‐ Tierenweitergezüchtet. 15 MaterialundMethoden 3.1.1.4 Genotypisierungen ZurGenotypisierungallerverwendetenMausstämmewurdedenNachkommenanTag5 bis 8 nach der Geburt eine Schwanzbiopsie entnommen. Die Vervielfältigung des LSL‐MYCN‐Amplikons aus genomischer Schwanz‐DNA erfolgte anhand einer PCR mit LSL‐MYCN‐Primern(Abb.4:A1/A2undTab.3)unddieDetektionmitHilfederAgarose‐ Gelelektrophorese (s. 3.2.1 und 3.2.2). Homozygote LSL‐MYCN Mäuse wurden mit ROSA26‐Wildtyp‐Primernidentifiziert(Tab.3).DieGenotypisierungvonDbh‐iCreMäu‐ senerfolgteunterVerwendungvonDbh‐iCre‐Primern(Tab.3). 3.1.2 ZuchtvonLSL‐MYCN;Dbh‐iCreMäusenundDetektionvonTumoren Dbh‐iCreMäusewurdenfreundlicherweisevonProf.Dr.HermannRohrer(Max‐Planck‐ Institut für Hirnforschung, Frankfurt) zur Verfügung gestellt. Heterozygote Dbh‐iCre Tiere wurden mit heterozygoten LSL‐MYCN Tiere verpaart. Doppelt‐transgene Nach‐ kommen wurden mittels LSL‐MYCN‐ und Dbh‐iCre‐Genotypisierungs‐PCRs (s.3.1.1.4) identifiziertundalsLSL‐MYCN;Dbh‐iCrebezeichnet. Durch wöchentliches Abtasten des Abdomens und des Halsbereiches wurden Tumore detektiert. Sowohl mittels Lumineszenz‐Bildgebung (s.3.1.2.1) als auch Ultraschall‐ Bildgebung (s.3.1.2.2) wurden ertastete Tumore validiert. Alle tumor‐tragenden LSL‐MYCN;Dbh‐iCre Mäuse wurden bei Erreichen eines Tumorvolumens von ca. 500mm3 oder bei schlechtem Habitus durch zervikale Dislokation getötet und seziert. Abmagerung,BewegungseinschränkungundAtembeschwerdenaufgrundeinesTumors inderNähederLuftröhrewurdealsschlechterHabitusdefiniert.Mittelseinerhandels‐ üblichen Kamera (Panasonic Lumix DMC‐FZ248) wurden makroskopische Fotos der Tumoreerzeugt.Herz,Hirn,Milz,MuskulaturausdemFemur,Nebennieren(wennnicht mitTumorverwachsen),Nieren,SchwanzundTeilederLebersowiederTumorewur‐ den sofort in flüssigem Stickstoff kryokonserviert und bei ‐80°C gelagert. Außerdem wurden Femur sowie Teile der Leber und der Tumore in 4% Paraformaldehyd in PBS pH7,2(Morphisto,FrankfurtamMain)eingelagert. IndenTumorenwurdedaskonstitutiveAllelanhandderausgeschnittenenPolyadenyl‐ Stoppsequenz im Vergleich zu Kontrollgeweben mittels flox out Primern (Tab.3) vali‐ diert. 16 MaterialundMethoden 3.1.2.1 Biolumineszenz‐BildgebungvonTumor‐tragendenLSL‐MYCN;Dbh‐iCreMäusen ZellenderLSL‐MYCN;Dbh‐iCreMaus,indenendieCre‐Rekombinaseaktivistunddamit dieTranskriptionsabbruchstelle(hGHpA)herausgeschnittenwurde,solltennebendem Onkogen MYCN auch Luziferase exprimieren. Zur Detektion dieser Zellen wurde Mäu‐ sen, die einen tastbaren Tumor trugen, in die Schwanzvene D‐Luciferin in PBS (PerkinElmer, Rodgau) injiziert (50mg/kg Körpergewicht). In Gegenwart von Luzi‐ ferase wird Luciferin oxidiert und bei der daraus resultierenden Abspaltung von TeilgruppenwirdEnergieinFormvonLichtabgegeben.Tierewurdenmit3%Isofluran (Forene®; Abbott, Wiesbaden) in Sauerstoff narkotisiert und in die Kammer des IVIS Lumina II (PerkinElmer), in der eine kontinuierliche Behandlung mit 2% Isofluran in Sauerstoff aufrechterhalten wurde, gelegt. Mittels dieses Luminszenz‐ Bildgebungsgerätes wurde die Intensität der Lumineszenz‐Emission gemessen und die BilderimTIF‐Formatexportiert. 3.1.2.2 Ultraschall‐BildgebungverschiedenerGewebevonLSL‐MYCN;Dbh‐iCreMäusen HochfrequenterSchallaußerhalbdeshörbarenBereichsdesMenschen(>16kHz)wird als Ultraschall bezeichnet. In der Humanmedizin dient die Ultraschall‐Untersuchung (Sonografie)zurBildgebungbeiverschiedenenErkrankungen.ManmachtsichzuNutze, dass die Ultraschallwellen, die von einem Schallkopf ausgesendet werden, je nach Ge‐ webeart entweder absorbiert oder reflektiert werden. Der Schallkopf empfängt die reflektiertenWellenwiederunddieReflektionwirdaufdemBildschirmalsGraustufen dargestellt. Wässrige Strukturen werden schwarz dargestellt, während Luft, die kein Ultraschallleitet,weißerscheint. ImRahmendieserArbeitwurdedasUltraschall‐BildgebungssystemVevo®2100(Visual Sonics, Amsterdam, Niederlande) verwendet, um Gewebe und Tumore der LSL‐MYCN;Dbh‐iCre Maus zu untersuchen. Enthaarte Tiere wurden mit 3% Isofluran (Forene®) in Sauerstoff narkotisiert und auf dem beheizbaren Untersuchungstisch so fixiert, dass eine Ableitung der Herz‐ und Atemfrequenz ermöglicht wurde. Die Ultra‐ schall‐Untersuchung wurde unter kontinuierlicher Narkose mit 2% Isofluran in Sauer‐ stoff mittels eines MicroScan‐Schallkopfs (MS550D‐Visual Sonics) durchgeführt. An‐ hand der 3D‐Funktion wurden in 100µm Abstand Schnitte durch Gewebe gemacht, dessenOberflächeanhanddesGerätsberechnetwurde.DurchAneinanderreihendieser Schnittewurdedie3D‐StrukturdesjeweiligenGewebesrekonstruiertunddasVolumen berechnet. 17 MaterrialundMe ethoden 3.1.3 X XenografttsundNanopartikel‐‐Behandlu ung Sechs‐W WochenalttenthymusslosenNCR R(nu/nu)M Mäusenwu urdensubkkutanindie eFlanke 1·107 W WACII‐Zelleen, suspend diert in 20 00 µl Matriigel™ (BD, Heidelberg H g), injiziert.. Mittels einer elektronisch hen Schieb blehre (Bau umarkt Obii, Essen) wurden w diee Tumore drei d Mal proWo ochevermeessenundd dasVolumeenmittelsd derfolgendenFormel berechnet: Tumorv volumen[m mm3]=½· Länge[mm m]·Breite[mm]·Höhhe[mm] Sobald die Tumorre ein Volu umen von cca. 500mm m3 erreicht hatten,wuurden jewe eils drei Tiere in ntravenös mit miR‐5 542‐3p‐ bzw w. Negativ vkontroll‐m miRNA‐belaadenen Nan noparti‐ keln (H Herstellungg s. 3.3.5) behandelt. Eine zweite Injektio on erfolgtee 24Std. nach der ersten. Nach weitteren 24Sttd. wurden n die Mäusse durch zervikale D islokation getötet. Sowohll Teile von n Lebern alls auch von n Tumoren n wurden für f qPCR‐ und Weste ern Blot Analyseen in Kryo oröhrchen überführt, ü in flüssigeem Sticksto off schockggefroren un nd bei ‐ 80°C geelagert. Fürr histologissche Unterrsuchungen n wurden Teile T von LLebern und d Tumo‐ renin4 4%ParaforrmaldehydinPBS,pH 7,2(Morphisto)fixiert. 3.2 M Molekula arbiolog gischeM Methode en 3.2.1 P Polymerasse‐Kettenrreaktion Saiki ettal. entwicckelte 1985 5 das Prinzzip der Pollymerase‐K Kettenreakttion (PCR),, mittels derDN NA‐Sequenzzenvervielffältigtwerd denkönnen73.Ineine emPufferssystemwerrdendie Ausgan ngs‐DNA (ttemplate), zwei z speziifische Olig gonukleotid dprimer, eeine thermostabile Taq‐Polymerase und Dessoxyribonu ukleosidtrip phosphate (dNTPs) vermisch ht. Die eigentliichePCRerrfolgtindreiReaktion nsschritten n.ImDenatturierungssschritt(94°°C)wird das tem mplate in seine beide en Einzelstrränge aufg getrennt, an n die die sspezifischen n Oligo‐ nukleottidprimeriimAnnealingschritth hybridisieren.DerElo ongationsscchritt,beid demdie Polymeerase aus dem d gebun ndenen Priimer das teemplate ne eu synthetiisiert, findet beim Temperraturoptim mum der Po olymerase statt (72°C C). Jede PCR besteht aus 25‐40 Zyklen, wobeid dieMenged deseingese etztentempplatesbeio optimalenB Bedingungeeninjedem mZyklus verdoppeltwird. 18 MaterialundMethoden IndieserArbeitwurdedieMethodederPCRzurGenotypisierungtransgenerMäuseund zur Validierung der ausgeschnittenen Polyadenyl‐Stoppsequenz benutzt, wobei die GoTaq®HotStartPolymerase(Promega,Mannheim)Verwendungfand.DieStocklösung derPrimerhatteeineKonzentrationvon20mM.FolgendesPipettierschemawurdefür jedenPCR‐Ansatzverwendet: 5xGoTaq®FlexiPuffer MgCl2(25mM) dNTP‐Mix(Bio‐Budget,Krefeld) Primerforward Primerreverse ® GoTaq Polymerase(5U/µl) templateDNA DNase‐freiesH2O 4µl 1,2µl 0,6µl 1µl 1µl 0,1µl 1µl 11,1µl DieAmplifikationdertemplateserfolgteimC1000™ThermalCycler(Bio‐Rad,München), wobei die Annealingtemperatur vom GC‐Gehalt der verwendeten Primer und die Elongationszeit von der Länge des zu synthetisierenden DNA‐templates (60Sek. pro 1kb)abhängigwar.DieverwendetenPrimer,sowiediespezifischenAnnealingtempera‐ turen und Elongationszeiten wurden in einer Tabelle zusammengefasst (Tab.3). Fol‐ gendesallgemeinesProgrammwurdebenutzt: 180Sek. 30Sek. 30Sek. 30‐180Sek. 300Sek. 94°C 94°C 55‐65°C 72°C 72°C 39x 19 MaterialundMethoden Tab.3:OligonukleotidprimerfürPCRfwd=forward;rev=reverse Produkt‐ Annealing‐ Elongations‐ Bezeichnung Sequenz(5‘‐3‘) länge temperatur zeit Dbh‐iCre fwd:ctgccagggacatggccagg rev:gcacagtcgaggctgatcagc 300bp 59°C 45Sek. flox‐out fwd:gcccgcggtgatgcctttgagg rev:cggggactgggcggtggaac 2241bp 703bp 67,7°C 240Sek. LSL‐MYCN fwd:accacaaggccctcagtacc rev:tgggacgcacagtgatgg 168bp 60°C 30Sek. ROSA26‐ Wildtyp fwd:ctcttccctcgtgatctgcaactcc rev:catgtctttaatctacctcgatgg 299bp 62°C 45Sek. Die Auswertung der PCRs erfolgte mittels der Agarose‐Gelelektrophorese (s.3.2.2), wobeijeweils14µldesPCR‐Ansatzesaufgetragenwurden. 3.2.2 Agarose‐Gelelektrophorese Aufgrund ihrer negativen Ladung bewegen sich sowohl DNA‐ als auch RNA‐Stränge in einem elektrischen Feld in Richtung der Anode. Die Laufgeschwindigkeit von Nuklein‐ säure‐SträngeninAgarosegelenkorreliertmitderFragmentgröße,daAgarosegeleeine siebartigeStrukturaufweisenundkleinerenFragmenteneinengeringerenWiderstand bieten. Im Rahmen dieser Arbeit wurden 1‐2%‐ige Agarosegele benutzt, die in Tris‐Acetat‐ EDTA‐Puffer (40mM Tris, 20mM Essigsäure, 1mM EDTA, pH8,0) angesetzt wurden. Die Zugabe von Ethidiumbromid (Endkonzentration 1,25µM), ein Farbstoff, dessen Absorptionsspektrum sich bei Interkalation mit Nukleinsäuren verändert, diente zur Visualisierung der DNA‐Fragmente unter UV‐Licht. Zur Bestimmung der Fragmentgrö‐ ßen wurde parallel zu den zu analysierenden Proben ein DNA‐Standard (LifeTechnologies, Darmstadt) aufgetragen. Die Elektrophorese erfolgte in Tris‐Acetat‐ EDTA‐Puffer bei 120V für 45 bis 60Min. und die Visualisierung am DokumentationsgerätBiodocAnalyzer(Biometra,Göttingen). 20 MaterialundMethoden 3.2.3 KlonierungderKonstruktepDEST30‐KDM1AundpDEST30‐Survivin Humane KDM1A cDNA ohne 3’UTR wurde im Vektor pDONR™ erworben (E1871‐ BC048134.1; THP Medical Products, Wien, Österreich). Humane Survivin cDNA ohne 3’UTR im Gateway® pENTR™ Vektor (LifeTechnologies) wurde freundlicherweise von Fieke Lamers aus der Arbeitsgruppe Humangenetik der Universität Amsterdam zur Verfügunggestellt74.Beidegenesofinterest(GOIs)konntenausdiesenDonorplasmiden anhandderGateway‐KlonierungsehreffizientinZielvektorenkloniertwerden.Mittels Gateway® LR‐Clonase®II Enzym‐Mix (LifeTechnologies) wurden die GOIs nach Herstel‐ leranweisungen in den Zielvektor Gateway® pT‐Rex™‐DEST30 (LifeTechnologies) klo‐ niert.EinKonstruktmitGFPalsGOIwarschoninunsererArbeitsgruppevorhanden.Die Hitzeschocktransformation in chemisch kompetente E.coli TOP10 (LifeTechnologies) erfolgtenachHerstellerangaben.Je50und200µldertransformiertenE.coliwurdenauf LBAMP‐Platten(10%Trypton,0,5%Hefeextrakt,10%NaCl,1%Agar‐Agar,pH7,0,0,01% Ampicillin) über Nacht inkubiert und Einzelzellklone gepickt. Um größere Mengen des PlasmidszugewinnenwurdenEinzelzellklonein100mlLBAMP‐Medium(10%Trypton, 0,5%Hefeextrakt,10%NaCl,pH7,0,0,01%Ampicillin)vermehrt.Plasmid‐DNAwurde mit Hilfe des Plasmid Maxi Kits (Qiagen, Hilden) nach Herstellerangaben isoliert. Um sicherzustellen, dass das GOI fehlerfrei im Zielvektor vorlag, wurden bei der Firma Seqlab(Göttingen)SequenzierungeninAuftraggegeben,wobeidieSequenzierungspri‐ mer benutzt wurden, die vom Hersteller für Gateway® pT‐Rex™‐DEST30 empfohlen wurden. Auf der Homepage des National Center for Biotechnology (NCBI, http://www.ncbi.nlm.nih.gov) wurden die aus der Sequenzierung erhaltenen Ergeb‐ nissemitBLASTNanalysiert. 3.2.4 BestimmungderKonzentrationvonDNAundRNA Die Messung der Nukleinsäurekonzentration erfolgte photometrisch mittels des Nano‐ Drop 2000c (Thermo Scientific). Dabei wurde die optische Dichte (OD) bei einer Wel‐ lenlänge von 260nm, dem Absorptionsmaximum von Nukleinsäuren, gemessen. Eine OD260 von 1 entspricht 50µg/ml doppelsträngiger DNA und 40µg/ml RNA. Eventuelle Verunreinigungen durch Proteine wurden bei 280nm detektiert. Der Quotient aus der OD260 und der OD280 liegt bei reinen DNA‐Präparationen bei 1,8 und bei reinen RNA‐ Präparationenbei2,0.ImRahmendieserArbeitwurdennurreineNukleinsäure‐Präpa‐ rationenweiterverwendet. 21 MaterialundMethoden 3.2.5 IsolierunggenomischerDNA Genomische DNA aus Geweben von LSL‐MYCN;Dbh‐iCre Mäusen wurde mittels des peqGOLD Tissue DNA Mini Kits (peqlab, Erlangen) nach Anweisungen des Herstellers isoliertundin70µlDNase‐freiemWasseraufgenommen. 3.2.6 mRNA‐undmiRNA‐Isolierung Die Extraktion von mRNA aus pelletierten humanen Zelllinien und murinen Geweben erfolgteanhanddesRNeasy®MicrooderMidiKits(Qiagen).MiRNAswurdenausGewe‐ ben mittels des miRNeasy Mini Kits (Qiagen) extrahiert. Sämtliche Kits wurden nach HerstelleranweisungeninklusivedesoptionalenDNase‐VerdausmittelsdesRNase‐free DNase Sets (Qiagen) durchgeführt. Die Lagerung isolierter mRNA und miRNA erfolgte bei‐80°C. 3.2.7 ReverseTranskriptionvonkomplementärerDNAausisoliertermRNAund miRNA DiereverseTranskriptionzurHerstellungvonkomplementärerDNA(cDNA)ausmRNA erfolgtemittelsderSuperScript® IIReverseTranscriptase(Life Technologies).DiecDNA ausmiRNAwurdemittelsdesmiScriptReverseTranscriptionKits(Qiagen)transkribiert. Beide Kits wurden nach Herstellerangaben durchgeführt und die synthetisierte cDNA bei‐20°Cgelagert. 3.2.8 QuantitativePCR DieMethodederquantitativenPCR(qPCR),dieaufdemPrinzipderherkömmlichenPCR beruht,dientzurAnalysederTranskriptionspezifischerGene.DieausisoliertermRNA mittels reverser Transkription generierte cDNA wird während der Amplifikation mit einemFluoreszenzfarbstoffversetzt,derindieDNAinterkaliert.DiemessbareFluores‐ zenz der Probe nimmt proportional zur vorhandenen DNA‐Menge zu. Der Schwellen‐ wert‐Zyklus(Ct‐Wert)beschreibtdenPCR‐Zyklus,andemdieFluoreszenzerstmaligdie Hintergrund‐Fluoreszenzübersteigt. Im Rahmen dieser Arbeit wurde für die Analyse von cDNA, die aus isolierten mRNAs synthetisiertwurden,abhängigvomOligonukleotidprimerderFastSYBR®GreenMaster Mix(LifeTechnologies)oderderTaqMan®FastAdvancedMasterMix(LifeTechnologies) 22 MaterialundMethoden verwendet. Die Detektion von cDNA, die aus miRNAs transkribiert wurde, erfolgte an‐ hand des miScript SYBR Green PCR Kits (Qiagen). Mittels des StepOnePlus™ Real‐Time PCR Systems (Life Technologies) wurde die qPCR durchgeführt. Die dabei benutzten ProgrammevariierteninAbhängigkeitdesbenutztenKits(Tab.4). Tab.4:ProgrammederqPCR Fast SYBR® Green TaqMan® Fast Advanced Master Mix Master Mix miScript SYBR Green PCR Kit 95°C 20Sek. 95°C 20Sek. 95°C 20Sek. 95°C 3Sek. 95°C 1Sek. 95°C 3Sek. 60°C 30Sek. 60°C 20Sek. 60°C 30Sek. 40x SowohldieverwendetenPrimeralsauchdas benutzteKitwurden tabellarischzusam‐ mengefasst(Tab.5). Tab.5:OligonukleotidprimerfürqPCRhs=human;mm=murin;fwd=forward;rev=reverse Sequenz(5‘‐3‘) oder Bezeichnung qPCRKit Bestellnummer fwd:tcatgcacatacaacacaga Fast SYBR® Green Master Mix Mm_Dbh rev:tgcacatagttgacacacat Hs_GAPDH fwd:catcaagaaggtggtgaagc rev:gagcttgacaaagtggtcgt Fast SYBR® Green Master Mix Mm_GAPDH QT01658692(Qiagen) Fast SYBR® Green Master Mix Hs_KDM1A QT00099442(Qiagen) Fast SYBR® Green Master Mix Mm_Phox2b fwd:ggcagaggaattgaggtaag rev:cgtctccacatccatcttt Fast SYBR® Green Master Mix Hs_miR‐542‐3p MS00010073(Qiagen) miScript SYBR Green PCR Kit Hs_MYCN QT00201404(Qiagen) Fast SYBR® Green Master Mix Hs_NEFL QT00096369(Quagen) Fast SYBR® Green Master Mix Hs_RNU6B MS00007497(Qiagen) miScript SYBR Green PCR Kit Hs_Survivin Hs03063352(LifeTechnologies) TaqMan® Fast Advanced Master Mix Hs_TFPI2 fwd:gtcgattctgctgcttttcc rev:ggagacagatctccgcgtta Fast SYBR® Green Master Mix Mm_Th fwd:aggtggtgacacttatccaa rev:agtgccagagaggacaag Fast SYBR® Green Master Mix 23 MaterialundMethoden DerCt‐WertvonzuanalysierendenmRNAs wurdegegendendesubiquitär‐exprimier‐ tenEnzymsGlycerinaldehyd‐3‐Phosphat‐Dehydrogenase(GAPDH)dergleichenSpezies normalisiert.DerCt‐Wertderubiquitär‐exprimiertenmiRNARNU6BdientezurNorma‐ lisierung des Ct‐Wertes der zu analysierenden miRNAs. Anhand der ∆∆‐Ct Methode75 wurdediemRNAbzw.miRNAExpressionrelativzurjeweiligenKontrollekalkuliert. 3.2.9 Array‐basiertekomparativegenomischeHybridisierung Die Technik der Array‐basierten komparativen genomischen Hybridisierung (aCGH) findetindermedizinischenForschungAnwendung,umVerlusteundZugewinnegeno‐ mischerDNAineinemGeweberelativzurKontrollezudetektieren.DabeiwirddieDNA der Referenzprobe und der zu analysierenden Probe mit verschiedenen Fluoreszenz‐ farbstoffen gelabelt und gegen ein Raster von DNA‐Fragmenten hybridisiert. Das Ver‐ hältnisderFluoreszenzsignalegibtAufschlussaufchromosomaleAberrationen. FürdieDetektionchromosomalerAberrationen,dieinTumorendesLSL‐MYCN;Dbh‐iCre Modells auftraten, wurden jeweils 13 Proben und die entsprechenden Kontrollen (Ge‐ webeausdemSchwanzdergleichenMaus)analysiert.GenomischeDNAwurdemitHilfe desQIAampDNAMiniKits(Qiagen)nachHerstellerangabenisoliert.DieProbenwurden auf einem 180K Array (AMADID 027411‐Agilent Technologies, Böblingen), der das komplette Mausgenom enthielt, analysiert. Je 400ng DNA aus Tumorgeweben wurde mittels Random priming (Life Technologies) Cy3 (Perkin Elmer, Rodgau) gelabelt, wäh‐ rend die gleiche Menge DNA aus Schwanzgewebe mit Cy5 (Perkin Elmer) markiert wurde. Die Hybridisierung und Waschung der Proben erfolgte nach Herstellerangaben desArrays.DieMessungderFluoreszenz‐IntensitätenwurdeanhanddesAgilentScan‐ ners G2505C (Agilent Technologies) durchgeführt. Das Software Programm Feature Extractionv10.1.1.1(AgilentTechnologies)fandbeiderExtrahierungderDatenAnwen‐ dung. Diese wurden mit Hilfe von aCGHbase (http://medgen.ugent.be/arraycghbase) weiter bearbeitet. Verluste und Zugewinne von Genen wurden anhand des circular binarysegmentation(CBS)Algorithmusberechnet76,77. 3.2.10 mRNAqPCRprofiling Anhand von Exon‐Mikroarrays kann die mRNA‐Menge bestimmter Gene einer Probe bestimmtwerden.DazuwirddieisoliertemRNAzunächstincDNAodercRNAtranskri‐ biertunddannmitFluoreszenzfarbstoffengelabelt.DerspezifischeMikroarray‐Genchip, 24 MaterialundMethoden dereineAuswahlvonDNA‐Spotsenthält,hybridisiertdiecDNAodercRNAderzuanaly‐ sierenden Probe. Nach Abwaschen der nicht‐gebundenen Nukleinsäuren, gibt die zu normalisierendeFluoreszenzintensitätderSpotsdiemRNAExpressiondesspezifischen GensinderProbewieder. Die Bestimmung der Konzentration und Qualität isolierter Gesamt‐RNA aus Tumoren des LSL‐MYCN;Dbh‐iCre Modells und aus nicht‐malignen Nebennieren von Wildtyp‐ Tieren erfolgte anhand des RNA6000 Nano Assays auf dem Agilent 2100 Bioanalyzer (Agilent Technologies). Die Transkription in cDNA und die Fragmentierung dieser er‐ folgte mit Hilfe des GeneChip® 3‘IVT Express Kit (Affymetrix, Freiburg). Die Hybridisie‐ rungdercDNAaufMausgenom‐Genchips4302.0(Affymetrix),dieSondenzurDetektion aller Gene der Maus enthielten, und das Auslesen erfolgte nach Anweisungendes Her‐ stellers.CELDateienwurdennormalisiertunddieExpressionmittelsderRMAMethode mitPowerToolsv1.15.0(Affymetrix)berechnet. DieKalkulationderMYCNmRNASignaturbasierteaufdemvonFredlundetal.publizier‐ tenAlgorithmus78. 3.2.11 miRNAqPCRprofiling Isolierte miRNAs aus Tumoren des LSL‐MYCN;Dbh‐iCre Modells und nicht‐malignen Nebennieren aus Wildtyp‐Mäusen wurden mittels des TaqMan miRNA Reverse TranscriptionKits(LifeTechnologies)unterVerwendungdesrodentstem‐loopMegaplex RTPrimerPoolsAundB(v2.0‐LifeTechnologies)nachHerstelleranweisungenincDNA transkribiert.Ein12ZyklenlangerAmplifikationsschrittzurHerstellungeinergrößeren Menge an cDNA wurde durchgeführt. Amplifizierte cDNA wurde 1zu4 verdünnt und mittels des TaqMan® MicroRNA Assay 7900 HT (Life Technologies) in folgender qPCR Reaktionquantifiziert: TaqMan®Assay 1,5µl ® TaqMan geneexpressionmastermix 1,75µl cDNA 0,02µl DNase‐freiesH2O 0,23µl Rohdaten der miRNA Expressionswerte wurden anhand des Cq‐cutoff gefiltert und normalisiertanhanddesglobalmean79. DieKalkulationderMYCNmiRNASignaturbasierteaufdemvonMestdaghetal.publi‐ ziertenAlgorithmus80. 25 MaterrialundMe ethoden 3.3 B Biochem mischeM Methode en 3.3.1 H Herstellun ngvonPro oteinlysateenundBesstimmungihrerKon nzentration n Zellpellletsbzw.T Tumorgewe ebewurde für30Min n.aufEisin nRIPA‐Pufffer(50mM MHepes, 150mM M NaCl, 1% % Triton X‐100, 1% N NP‐40, 0,1% SDS, cO Omplete Prootease Inhibitoren und PhosSTOP Ph hosphatase Inhibitoreen (Roche) nach Hersstellerangaaben) lysierrt. Nach Zentrifugatiion bei 14.000g für 3 30Min. wu urde der Überstand inn ein neue es Reak‐ einer Z tionsgeefäßüberfü ührtundbei‐20°Cgelaagert. BeiderrBestimmu ungvonPro oteinkonzeentrationen ninLösung genfindethhäufigdieM Methode nach B Bradford Anwendung g. Diese maacht sich zu Nutze, dass sich das Absorptions‐ maximu um des Trriphenylme ethanfarbsttoff Coomaassie‐Brilla ant‐Blau G‐‐250 bei der d Bin‐ dungan nProteinevon470nm mauf595n nmverschiebt81. ImRah hmendieserrArbeitwu urdenje2 µldeszuaanalysieren ndenProteiinlysatsmiit998µl 1xRoti®‐Quant (Roth) verm mischt un d 5Min. inkubiert. i Die Absorrption wurde am NanoDrrop2000c (ThermoS Scientific,B raunschweeig)gemessenundgeegendenLeerwert normallisiert. Anh hand einerr BSA‐Stan ndardreihe wurde diie Proteinkkonzentrattion der Probeb bestimmt. 3.3.2 N odecylsulffat‐Polyacrrylamidge elelektroph horese Natriumdo Mittels der Methode der d Natriu umdodecyllsulfat‐Poly yacrylamiddgelelektrop phorese (SDS‐PA AGE) könn nen Protein ne nach ih hrer Ketten nlänge, die e proportioonal zur Molekül‐ M masse ist, aufgettrennt werrden. Durcch Zugabee von SDS wird die Eigenladu ung der Protein ne von ein ner konstan nten negattiven Ladu ung überde eckt. Die LLinearisieru ung der Proben n erfolgt du urch Erhitzen auf 95°°C, wobei Sekundär‐ S und u Tertiäärstrukturen durch dieUntterbrechun ngvonWassserstoffbrü ückenbindu ungenaufg gebrochen werden.Aufgetra‐ genauffeinSDS‐G Gelwerden größereP roteineim elektrischenFeldstäärkerzurücckgehal‐ ten als kleine, waas zur Aufftrennung d dieser führrt82. Anhan nd eines M Molekularge ewichts‐ standarrdkanndieeProteingrößenachF FärbungmiiteinemFarbstoffabggeschätztw werden. ImRah hmendieserArbeitwurdenje5 0µgGesam mtproteinm mitLAEMM MLI‐Puffer (50mM Tris‐HC ClpH6,8,2%SDS,0,1%Bromph henolblau,1 10%Glycerrol,10%β‐‐Mercaptoe ethanol) versetzzt, 5Min. bei 95°C im m Heizblockk denaturieert undzussammen m mit dem Molekular‐ gewichttsstandard d Page Ruller (Fermeentas) auf NuPAGE® Novex 4‐112% Bis‐Trris Gele 26 MaterialundMethoden (Invitrogen)aufgetragen.DieelektrophoretischeAuftrennungderProteineerfolgtebei einerkonstantenelektrischenSpannungvon120VjenachLaufweitederFarbfrontfür 90‐120Min. 3.3.3 WesternBlotundimmunologischerNachweisvonProteinen DerWesternBlotisteineproteinbiochemischeMethode,mitderProteinevomSDS‐Gel auf eine Membran aus Nitrozellulose transferiert werden können83. Die Proteine wan‐ dern dabei durch ein senkrecht zum SDS‐Gel angelegtes elektrisches Feld in Richtung der Anode auf die Membran, auf der sie aufgrund hydrophober Wechselwirkungen haftenbleiben.DasandenProteinenangelagerteSDSwirdausgewaschen,wodurchdie Proteine ihre Sekundär‐ und Tertiärstruktur wieder einnehmen. Freie unspezifische Bindungsstellen der Proteine werden mit einer Lösung aus entfettetem Milchpulver blockiert.DieDetektionbestimmterProteinekannmitspezifischenPrimärantikörpern, dieanihrAntigenbinden,erfolgen.MittelseinesSekundärantikörpers,derspezifischan denprimärenbindetundeinReporterenzymenthält,dasdasSubstratLuminolumsetzt, wirddieProteinbandevisualisiert. ImRahmendieserArbeitwurdenaufgetrennteProteinemittelsdesTrans‐Blot®Turbo™ (Biorad) vom SDS‐Gel auf die Hybond‐™C Extra Nitrozellulose‐Membran (Amersham Biosciences, Freiburg) transferiert. Dazu wurden zuerst zwei Filterpapiere (Whatman) mitAnodenpuffer1(300MTris,20%Methanol)getränktundinderBlotkammerüber‐ einanderpositioniert.EinweiteresFilterpapier,mitAnodenpuffer2(25mMTris,20% Methanol) getränkt, wurde auf die beiden Filterpapiere gelegt.Als nächstes wurde die Membran (in Anodenpuffer 2) und das SDS‐Gel, welches mit Kathodenpuffer (25mM Tris, 40mM Glycine, 10% Methanol) getränkt war, aufgestapelt. Drei weitere Filterpa‐ piere (in Kathodenpuffer) bildeten den Abschluss. Bei einer konstanten elektrischen Stromstärkevon90mAwurdendieProteinefür1Std.übertragen.DieMembranwurde mitTBS‐T0,1(0,1%Tween20inTBS)gewaschenunddieProteinemitPonceauS‐Lösung (0,1%PonceauS,5%Essigsäure)zurÜberprüfungdesTransfersundzurEinzeichnung derMarkerbandenangefärbt.NacheinemweiterenWaschschrittwurdendieunspezifi‐ schen Proteinbindestellen für 1 Std. mit Bovine Lacto Transfer Techniques Optimizer (BLOTTO‐5%Milchpulver,50mMTrisHClpH8,80mMNaCl,2mMCaCl2,0,2%NP‐40) blockiert.DieInkubationdesPrimärantikörpersinBLOTTOerfolgteüberNachtbei4°C, gefolgt von einem weiteren Waschschritt. Der Sekundärantikörper, an den Meerettich‐ 27 MaterialundMethoden peroxidase gekoppelt war, inkubierte in TBS‐T0,1 für eine Std. bei Raumtemperatur, ebenfalls gefolgt von einem Waschschritt. Verwendete Verdünnungen der Antikörper wurdentabellarischaufgeführt(Tab.6).ProteinewurdenmittelsdesDetektionsreagenz ECLPlus™(AmershamBiosciences)visualisiert,imFusionFX(peqlab)detektiertunddie Bilderalsjpegexportiert. Tab.6:AntikörperfürdenimmunologischenNachweisvonProteinen Antigen Bestellnummer Verdünnung AuroraBkinase 3094(CellSignaling) 1:1.000 AKAP2 Ab64904(Abcam) 1:1.000 Aktin A3853(Sigma‐Aldrich) 1:2.000 Caspase9 IM3434(Immunotech) 1:1.000 GAPDH MAB374(Millipore) 1:2.000 Kaninchen‐IgG NA9340V(GEHealthcare) 1:5.000 KDM1A 2139(CellSignaling) 1:1.000 Maus‐IgG NA9310V(GEHealthcare) 1:5.000 N‐Myc sc‐53993(SantaCruz) 1:1.000 Survivin 2808(CellSignaling) 1:1.000 TetR 631131(Clontech) 1:2.000 28 MaterrialundMe ethoden 3.3.4 P Proteomanalyse Proteom manalysen wurdenim mRahmen desBMBF‐Projekts„ENGINE“innKooperattionmit der Arb beitsgruppe von Proff. Dr. Kai SStühler (M Molecular Prroteomics LLaboratoryy (MPL), Heinricch‐Heine‐Universität,Düsseldorff)durchgefführt. ImRahmendieserrArbeitwu urdendasP Proteomvo onSHEPZe ellen96Stdd.nachtran nsienter Transfeektion m mit Negatiivkontroll‐ miRNA oder o miR R‐542‐3p verglichen. Die Transfeektionenerrfolgtenin12‐wellPlaatten. 3.3.5 H Herstellun ngvonCalcium‐Phossphat‐Nan nopartikeln n ImRah hmeneiner Kooperationmitder rArbeitgrup ppevonPrrof.Dr.MattthiasEpple(Insti‐ tut für Anorganissche Chem mie, Univerrsität Duisburg‐Essen n) wurdenn miRNA‐b beladene Calcium m‐Phosphatt‐Nanoparttikel synth hetisiert, wie w in Soko olova etall. beschrieben84,85. Nanopaartikel wurrden aus einer e wässsrigen Lösu ung aus Calciumnitraat (6,25m mM) und Diamm monium‐hyd drogenphossphat (3,74 4mM) prääzipitiert und unter ssterilen Be edingun‐ genmittPre‐miR™ ™miRNA‐542‐3pPreccursorMoleekülenbzw w.derPre‐m miR™Nega ativkon‐ trolle ffunktionalisiert (19µ µM). Diesee Konstruk kte wurden n mittels eeiner zusätzlichen Hülle aaus Calcium m‐Phosphat geschützzt und weiiter stabilissiert mit vverzweigtem m Poly‐ Ethylen nimin(PEI–2mg/ml)(Abb.5). In beiden Dispersion nenbetruggendieKon nzentra‐ tionend dermiRNA As1,08µMunddievon µg/ml. nPEI357µ d verwen ndeten miR RNA‐ Abb5: Struktur der beladen nen Calciu um‐Phosph hat‐Nano‐paarti‐ kel Diaagrammatisccher Aufbau u von miR RNA‐ beladen nem Nanopaartikel der Arbeitsgru uppe von Pro of. Dr. Mattthias Epple (Institut für Anorgan nische Chem mie, Universität Duisbu urg‐ Essen). CaP= Calciium‐Phosph hat; PEI= P Poly‐ ert von Diiana Ethyleniimin. Graphik generie Kozlovaa. 29 MaterrialundMe ethoden 3.4 H HistologischeM Methoden n 3.4.1 H Histologisscheundim mmunhisttochemisch heFärbungenvonGeewebeschnitten Die Hämatoxilin‐E Eosin‐Färb bung wird in der Hisstologie angewendet, um verschiedene Struktu urenvonGeewebeschn nittenanzuffärben.Häm malaunfärbtsaureSttrukturenw wieDNA und Riibosomen blau, währrend Eosin n basischee Strukture en wie Plaasmaproteiine und ondrien ro ot erscheine en lässt. In n der Immu unhistoche emie werdeen markierrte Anti‐ Mitocho körperbenutzt,um mProteine einGewebeeschnittensichtbarzu umachen. Formalin‐fixierte Gewebe wurden w in P Paraffin eingebettet und in 5µµm dicke Scheiben geschniitten. Die Schnitte S wurden w mit t Xylol entp paraffiniertt und in eeiner absteigenden Alkoholreihe rehy ydriert. Die Demaskiierung derr Antigene erfolgte m mittels derr Target Retrieva alSolution (Dako,Hamburg)beeipH6für 20Min.beii100°C.Di eSchnitte wurden 20Min..gekühltu undfür5M Min.in3%H H2O2inkub biert.ImDa akoAutosttainer(Dak ko)wur‐ den diee verschied denen Antikörper (Taab.7) nach h Herstellerrangaben iinkubiert. Die D Ent‐ wicklun ngerfolgtemittelsdessEnVision™ Kits(Dako o). mmunhisto ochemische enNachweiisvonProte teinenaufG Gewebe‐ Tab.7: Antikörperrfürdenim schnitte en Antigen Bestelllnummer Verdünn nung Caspase3 9661((CellSignalin ng) 1:200 CD56 ab6123 3(abcam) 1:500 Mib‐1 RM‐91 106(Thermo oScientific) 1:25 TH Ab764 442(abcam) 1:200 3.4.2 E Elektronenmikrosk kopischeUn ntersuchu ungenvonGewebescchnitten Elektro onenmikrosskopische Untersuchu ungen von n Geweben n wurden im Rahme en einer Kooperration mit der Arbeittsgruppe vvon Dr. Lukas Heuka amp (Instittut für Patthologie, Universsitätsklinik kKöln)durchgeführt. Calcium m‐Phosphatt‐Nanoparttikel wurdeen von derr Arbeitsgrruppe von Prof. Dr. Matthias M Epple ((Institut fü ür Anorgan nische Chem mie, Univeersität Duissburg‐Esse n) mittels Raster‐ elektronenmikrosskopieanha andihrerd dynamischeenLichtstre euungcharrakterisiertt. 30 MaterrialundMe ethoden 3.4.3 IImmunflu uoreszenzffärbungen vonProte eineninhu umanenZeelllinien DieImm munfluoresszenzisteinespeziellleImmunffärbung,be eiderdievverwendete enAnti‐ körpermitFluoreeszenzfarbsstoffenmarrkiertwerd den. Für Imm munfluoreszenz‐Färb bungen derr Proteine Survivin und Aurora a B Kinase wurden Zellen aauf 35mm m Glasboden n Kultursch halen (Matttek) transfiziert undd 96Std. in nkubiert. NachdeerFixierun ngderZelle enin4%Paaraformaldehydfür20 0Min.,wurrdendieZe ellenmit 0,1% T Triton‐X‐10 00 in PBS fü ür 10Min. permeabillisiert. Die Inkubationn mit Prim mär‐ und Sekund därantikörp pernerfolgtefürjeweeils1Std.in nderfolgendenReiheenfolge,wo obeialle Antikörrpermit10 0%BSAinP PBSverdün nntwurden n: anti‐AuroraB(BecctonDickin nson,1:500 0) anti‐Ma aus‐Alexa‐F Flour‐568((Invitrogen n,1:1.000) anti‐Surrvivin(Novvus,1:250) anti‐Kaninchen‐Allexa‐Fluor‐‐488(Invitrrogen,1:1.0000). AnschliießendwurrdendieZe ellenmit0,,1%Hoechst33343in nPBSfür110Min.gefä ärbt.Die mikrosk kopische Analyse A erfolgte bei Raumtemp peratur in PBS mitteels eines inversen Fluoresszenz‐Mikroskops (Ax xioObserveer, Zeiss, Darmstadt), D , ausgestatttet mit ein ner digi‐ talen m monochrom matischen Kamera K (A AxioCam MRm M Rev.2CCD, Zeisss) und ein nem Öl‐ Objektiiv (Plan‐Ap pochromat 63x/1.40, Zeiss). Diee Bilder wurden bei gleichen Expositi‐ E onseinsstellungenakquiriertundalsTIF FDateieneexportiert. 3.5 Z Zellbiolo ogischeMethod den 3.5.1 K Kultivieru ungvonZe ellen Die hum manen Neuroblastom mzelllinien IMR‐32, SHEP S und SK‐N‐BE ssowie die humane h embryo onaleNiereenzelllinie HEK‐293w wurdenbeiderDeutsschenSam mmlungvon nMikro‐ organissmen und Zellkulturen GmbH (DMSZ, Braunschwe B eig) erworrben. Die humane h Neurob blastomzellllinie WACIII wurde frreundlicherweise von n Frank Weestermann n (DKFZ, Heidelb berg)zurV Verfügungg gestellt.Ein neAuthentiifizierungd derZelllinieenerfolgte ebeider DSMZ.A AlleKulturrenwurden nanhandd desMycoAlert™Mycop plasmaDete tectionKits(Lonza, Basel, SSchweiz) in n regelmäß ßigen Abstäänden auf Kontamina ationen duurch Mykop plasmen untersu ucht. 31 MaterialundMethoden SämtlicheverwendeteZelllinienwurdeninRPMI1640Mediumsupplementiertmit10% FCS,AmphotericinB(3µg/ml),Penicillin(100U/ml)undStreptomycin(100µg/ml)bei 37°C, 80% Luftfeuchtigkeit und 5% CO2 kultiviert. Bei Erreichen etwa 80%‐iger Kon‐ fluenz wurden die Zellen mit 0,2% Trypsin/EDTA für 2‐3Min. abgelöst. Durch Zugabe vonKulturmediumwurdedieReaktionabgestoppt.DieZellenwurdenmittelsdesTC10™ Automated Cell Counter (BioRad) gezählt und in für die Versuche erforderlichen Ver‐ dünnungenausgesätoderweiterkultiviert. DieKryokonservierungvonZellenerfolgteinFCSmit10%DMSOinflüssigemStickstoff. ZumAuftauenwurdendieKryoröhrchenbei37°Cfürca.1Min.inkubiertunddieZellen inKulturmediumaufgenommen. 3.5.2 TransienteTransfektionenvonmiRNAundsiRNA FürtransienteTransfektionenvonmiRNAundsiRNAinhumaneZelllinieninvitrowur‐ denZelleninverschiedenenZahlenausgesät,abhängigvonihrerWachstumsgeschwin‐ digkeit und Größe. Die im Rahmen dieser Arbeit ausgesäten Zellzahlen wurden tabel‐ larischzusammengefasst(Tab.8). Tab.8:AusgesäteZellzahlenderverwendetenZelllinien Zellzahl Zelllinie 96‐well 12‐well 35 mmGlasboden Kulturschale(Mattek) HEK‐293 2·104 ‐ ‐ IMR‐32 1,5·104 12·104 18·104 SHEP 0,5·104 4·104 6·104 SK‐N‐BE 1·104 8·104 12·104 WACII 0,5·104 4 · 104 6·104 Transiente Transfektionen der Pre‐miRs™ (AM17100‐Life Technologies) miR‐137 (PM10513),miR‐542‐3p(PM11340)undmiR‐542‐5p(PM13010)Precursor Molekülen undderPre‐miR™Negativkontrolle(AM17110‐LifeTechnologies)erfolgtenmittelsdes Transfektionsreagenzes siPort™ NeoFX™ (Life Technologies) in einer Endkonzentration von30nMnachAnweisungendesHerstellers. 32 MaterrialundMe ethoden Die FleexiTube siiRNAs (10 027417‐Qiiagen) Hs__AKAP2_8 (SI042332201), Hs_B BIRC5_5 (SI0029 99453)und dHs_KDM1 1A_5(SI027 780932)so owiedieAlllStarsNegaativkontrolll‐siRNA (10272 280‐Qiagen n)wurdenineinerE Endkonzenttrationvon n33nMmiitHilfedessTrans‐ fektionsreagenzL Lipofectamiine™RNAiM Max(LifeT Technologiees)nachHeerstellerang gabenin dieZellleneingebrracht. 3.5.3 G Generieru ung von stabilen s Z Zellklonen und Indu uktion vo on konditiionalen K Konstruktten Der Vek ktor pcDNA A™6/TR (V V1025‐20‐‐Life Techn nologies) ist ein Expreessionsvek ktor, mit demho oheLeveld desTetracy yclin‐RepreessorProteeinsexprim miertwerdeenkönnen..Außer‐ demveerfügtdasP Plasmidübe erdenSäuggetier‐Selek ktionsmark kerBlasticiidin(Abb.6 6). Vektorkarte e von pcDN NA™6/TR B Besonderheiiten: Ex‐ Abb.6: V pression des Tetracy yclin‐Repressor Proteiins (TetR); Blastici‐ äugetier‐ dinresisteenz zur Seleektion von stabil‐transsfizierten Sä zellen.Moodifiziertnaachwww.life etechnologiees.com. ™6/TRwurrdemittelssElektropooration(Neeon™transfeectionsysteem,Invitrog gen)bei pcDNA™ 1.000V Vund50msinSHEPZ Zelleneinggebracht,w wobeinachH Herstelleraangabenvo orgegan‐ gen wu urde. Durch h Zugabe von Blasti cidin (7,5µg/ml) zum m Medium m wurden positive Klone sselektiert. Einzelzellk klone wurd den mittelss Limited Dilution D ge neriert. Diie Über‐ prüfunggderKloneeerfolgtem mittelsWessternBlot(s.3.3.3). Der Vektor Gatew way® pT‐R Rex™‐DEST3 30 (12301‐016‐Life Technologgies) dient zur Ex‐ pressio onhoherLeeveleinesG GOIsinSäu ugetierzelleen.DerVek ktorverfüggtüberTettracyclin Operato or2Stellen n,dieinTettracyclinR Repressor‐eexprimieren ndenZellenneineTerttacyclin‐ regulierrte Expression des GOI G erlaubeen. Der Säu ugetier‐Selektionsmarrker Neom mycin ist ebenfalllsvorhand den(Abb.7). 33 MaterrialundMe ethoden Abb.7: Vektorkarrte von pT T‐Rex‐DEST T30 Beson nderheiten: TetracycclinOperato or2Stellen (TetO2)zurrTetracyclin n‐regulier‐ tenExprressiondes GOIsinTettracyclinReepressor‐exp primieren‐ den Zelllen; Neomy ycinresistenzz zur Selekt ktion von sttabil‐trans‐ fizierten n Säu ugetierzellen n. M odifiziert nach www.liffetechnologies.com. Die Konstrukte pT‐Rex™‐‐DEST30‐K KDM1A, pT‐Rex™‐DE EST30‐Survvivin (Klonierung s.3.2.3)) und pT‐R Rex™‐DEST T30‐GFP alls Kontrollle wurden mittels Ellektroporattion bei 1.000V Vfür50mssindenvallidiertenEiinzelzellklo onSHEP‐p pcDNA™6/T TReingebrracht.Es wurde nach Hersstelleranwe eisungen geearbeitet. Transfizier T rte Zellen w wurden du urch Zu‐ gabevo onGeneticiin(0,5mg/ /ml)zumK KulturmediiumselektiertundEiinzelzellklo onemit‐ tels Lim mited Diluttion generie ert. Durch Zugabe vo on Tetrazy yklin (1µg//ml) zum Medium M wurde die Expression von GFP, KDM M1A oder Survivin S in nduziert. Eiinzelzellklo one, die induzieerbar GFP exprimierrten, wurd den am Axxiovert 40CFL Mikrooskop (Carl Zeiss MicrosccopyGmbH H,Jena)dettektiert.DieeÜberprüffungderin nduzierbareenExpresssionvon KDM1A AundSurviivinerfolgttemittelsW WesternBlo ot(s.3.3.3).AusgewähhlteKlonewurden mitSHE EP‐GFP,SH HEP‐KDM1A AundSHEP P‐Survivinbezeichnett. 3.5.4 M Mikroskop pischeAuffnahmenvvonZellen Mikrosk kopischeA AufnahmenvonZellen nauf12‐w wellPlatten,,diemitm miRNAstran nsfiziert wurden n, erfolgten n am Axiov vert 40CFL L Mikrosko op (Carl Zeiss Microsccopy GmbH H) unter Verwen ndung der Software ZEN (Verrsion 2011 1 (blue ediition) Carl Zeiss Miccroscopy GmbH).FotoswurrdenalsTIFexportierrt. 3.5.5 Z Zellviabiliitäts‐Assay y Der gellbe, wasserrlösliche Fa arbstoff 3‐ (4,5‐Dimetthylthiazol‐‐2‐yl)‐2,5‐D Diphenyl‐te etrazoli‐ umbrom mid (MTT) wird be ei seiner R Reduktion zu blau‐v violettem, w wasserunlö öslichen Formazzan. Beim MTT‐Test M wird w die m metabolisch he Aktivitätt von Zelleen anhand der Re‐ duktion ndesFarbsstoffsbestimmt86.DieeReduktion nsratehängtvonderGlykolyserrateder Zellenaabundwird ddefiniertalsihreZeellviabilität.. 34 MaterialundMethoden DieAnalysederViabilitätvonZellenerfolgteim96‐wellFormatzuverschiedenenZeit‐ punkten (24, 48, 72, 96 und 120Std.) nach der transienten Transfektion von miRNAs undsiRNAs(s.3.5.2).DerFarbstoffMTTwurdeinPBSgelöst(10mM)und20µlprowell zu den Zellen gegeben (Endkonzentration 1,7mM). Nach einer Inkubationszeit von 2Std.bei37°CwurdederÜberstandabgesaugtunddasentstandeneFormazanin100µl Solubilisierungslösung (10% SDS, 0,6% Essigsäure in DMSO) gelöst. Die Messung er‐ folgte bei einer Wellenlänge von 570nm (Referenzwellenlänge 630nm) im Epoch Spektralphotometer‐System (BioTech, Bad Friedrichshall). Alle Experimente wurden mindestensdreiMal,jeweilsindreifach‐Bestimmungdurchgeführt. 3.5.6 Apoptose‐Assay Zellen, die aufgrund von Apoptose sterben, geben Mono‐ und Oligonukleosome ins Zytoplasmaab.DasCellDeathDetectionELISAPLUSKit(11774425001‐Roche)verwen‐ det Antikörper, die spezifisch gegen DNA und Histone gerichtet sind, um diese Mono‐ und Oligonukleosome zu detektieren. Die Umsetzung eines spezifischen Substrates korreliertmitderFraktionapoptotischerZelleninderProbe. Die Analyse der apoptotischen Zellen erfolgte nach Herstellerangaben im 96‐well For‐ mat96Std.nachTransfektionvonmiRNAsodersiRNAs(s.3.5.2).DieMessungerfolgte anhand des Epoch Spektralphotometer‐Systems (BioTech). Alle Experimente wurden dreiMal,jeweilsindreifach‐Bestimmungdurchgeführt. 3.5.7 Proliferations‐Assay DieSynthesevonDNAisteinindirekterParameterderZellproliferation.DasPrinzipdes CellProliferationELISAKits(1647229001‐Roche)beruhtaufdemEinbauvon5‐bromo‐ 2‘‐Deoxyuridin(BrdU)anstellevonThymidinindiegenomischeDNAvonproliferieren‐ denZellen.MittelseinesAntikörperswirddaseingebauteBrdUgebunden.DerUmsatz desSubstratsgibtAufschlussaufdieProliferationsrateeinerZellprobe. Die Analyse der proliferierenden Zellen erfolgte im 96‐well Format 96Std. nach der Transfektion von miRNAs oder siRNAs (s.3.5.2). Es wurde nach Herstellerangaben vorgegangen. Die Messung erfolgte anhand des Epoch Spektralphotometer‐Systems (BioTech). Alle Experimente wurden mindestens drei Mal und jeweils in dreifach‐Be‐ stimmungdurchgeführt. 35 MaterialundMethoden 3.5.8 Zellzyklusanalyse Der Farbstoff Propidiumiodid interkaliert mit Nukleinsäuren von perforierten Zellen. Bei der Zellzyklusanalyse wird anhand von Durchflusszytometrie der DNA‐Gehalt von Zellen bestimmt. Dieser gibt Aufschluss auf den Zellzyklusstatus einzelner Zellen. Die subG1‐PhaseistcharakterisiertdurchdegradierteDNAunddamiteinCharakteristikum apoptotischerZellen.InderG1‐PhaseliegtinderZelleeinvollständigerChromosomen‐ satzvor.BefindetsicheineZelleinderS‐Phase,inderdieDNArepliziertwird,liegtein 1‐2‐facherChromosomensatzvor.DieG2/M‐PhaseistdurchdasVorhandenseinzweier Chromosomensätzecharakterisiert. Zu analysierende Zellen wurden in 12‐well Platten mit miRNAs oder siRNAs transient transfiziert(s.5.3.2)undnach96Std.analysiert.ZellpelletswurdenmitPBSgewaschen (5Min.bei1.000g),in70%EthanolaufEisfür1Std.fixiertundzentrifugiert(5Min.bei 1.000g).DasPelletwurdein500µlPBSresuspendiertunddieRNAdurchZugabevon RNase A (Endkonzentration 0,25mg/ml) für 1Std. bei 37°C verdaut. Propidiumiodid wurde in einer Endkonzentration von 10µg/ml zugegeben. Der zelluläre DNA‐Gehalt von jeweils 5.000 Zellen wurde mittels des Flow Cytometers FC500 (Beckman Coulter, Krefeld) gemessen und anhand der Software CXP analysiert. Alle Experimentewurden mindestensdreiMaldurchgeführt 3.5.9 LuziferaseReporterAssay DiedirekteBindungeinermiRNAaneinevorhergesagteZiel‐mRNAkannmittelseiner LuziferaseReporterAnalysevalidiertwerden.DasLuziferase‐Reporter‐Plasmidenthält diepotentielleBindestelledermiRNA.DieBindungdermiRNAandieBindestelleführt zurHemmungderLuziferaseExpressionundsomitzurgeringerenAktivität. Die Bindung von miR‐137 an die 3’UTR von KDM1A wurde wie folgt untersucht: Das Glühwürmchen‐LuziferaseReporterPlasmidpMir‐Target(OriGene),welchesdie3’UTR von KDM1A beinhaltete (SC0204616‐NM_015013), das Plasmid pRL‐TK (E224A– Promega, Mannheim), das als Transfektionskontrolle Renilla‐Luziferase exprimierte, und Negativkontroll‐miRNA oder miR‐137 wurden im 12‐well Format sowohl in HEK‐293alsauchinSHEPZellenkotransfiziert. DieValidierungdermiR‐542‐3pBindungandie3’UTRvonSurvivinerfolgtemitfreund‐ licherweisevonDr.KwangheeBaek(GraduiertenschulefürBiotechnologie,KyungHee Universität, Yongin, Korea) zur Verfügung gestellten Psi‐Check2‐Vektoren (Promega), 36 MaterrialundMe ethoden diedie 3’UTRvon nSurvivine enthielten. Aufeinem derbeiden nVektorennwardie3‘UTRso 3p zerstörtt vorlag. HEK‐293 H mutiertt, dass die vorhergessagte Bindeestelle der miR‐542‐3 undSH HEPZellenw wurdenmiitjeweilseeinemderb beidenVek ktorenundentweder mitder Negativvkontroll‐m miRNAoderrmiR‐542‐‐3pkotranssfiziert. DieMessungder Luziferase‐Aktivitäteenerfolgte 48Std.nacchderTrannsfektionm mitdem Dual‐Lu uciferase® Reporter Assay A Systtem (Prom mega) nach Herstellerranweisung gen. Die Expresssion der Glühwürmc G chen‐Luzifeerase wurd de gegen die der Seeefeder‐Lu uziferase normallisiert. 3.6 G Genset Enrichm E ment An nalyse, statistis s sche Meethoden n und B Bildbearrbeitung g 3.6.1 G GensetEnrichmentA Analyse Anhand d einer Genset G Enrrichment A Analyse (G GSEA) mitttels einerr Online‐S Software (www.b broadinstittute.org) wurden w Sign nalwege od der Gruppe en von funnktionell ve erwand‐ ten Gen nen identiffiziert, die in Tumoreen des LSL L‐MYCN;Dbh‐iCre Mauusmodells im Ver‐ gleich zzu nicht‐m malignen Nebenniere N n aus Wildtyp‐Mäusen dereguuliert wurd den. Die Softwarreverwend detdieMollecularSignnaturesDattenbank(M MSigDBv3.11),dieeine eKollek‐ tion etaablierter Gensets G entthält87. Dass Ziel eineer GSEA istt es Grupppen von Ge enen zu identifizieren,diegemeinsam me,biologi scheFunkttionenhabenundamAnfangod derEnde der Raangliste deer verschie eden regu ulierten Geene stehen n. Bei derr GSEA wird w der Enrichm ment Score (ES) als Maß M definieert, wie staark ein Gen nset innerhhalb der Rangliste R derGen neüberrepräsentiertist.DieSign nifikanzdeesESwirdaufBasiseeinesPermu utations p‐Wert tberechnettundanhandderfalssch‐positiv Rate(FDR R)angegebeen.EinFDR R<0,25 wirdallssignifikan ntdefinierrt.BeiderB Berechnun ngdesnorm malisiertennES(NES) werden dieUntterschiedeinderGröß ßederGen nsetsundin nderKorre elationzwisschenGenssetsund Expresssionsdaten n normalisiert. Dadurrch können n Ergebnissse von Anaalysen versschiede‐ nerGen nsetsmiteinanderverrglichenweerden.EinNES>2zeig gteinesehhrstarkeAn nreiche‐ rungdeesGensets anundein nNES=1‐2 einemodeeratebisstarkeAnreiicherung,w während NES<1fürkeineA Anreicherun ngsteht.In ndieserArb beitwurde dieGSEAaaufeineKo ollektion von3.2 272Gensetss(C2)ange ewendet.BeeiderAnallysewurdenGensets, diewenige erals15 Geneod dermehraals500Geneenthielteenausgesch hlossen. 37 MaterialundMethoden 3.6.2 StatistischeAnalysen SämtlicheGraphenwurdenmitGradhPadPrism® 5.0erstellt.StatistischeAnalysenwie derStudent‘st‐Test,derLog‐Rank‐TestundderWilcoxonTesterfolgtenebenfallsmit‐ telsdieserSoftware.StatistischeUntersuchungenzumVerlaufzweierGraphenwurden von Johannes Köster (Genome Informatics, Institut für Humane Genetik, Universität Duisburg‐Essen) anhand eines Permutationstests durchgeführt. Expressionsdaten aus qPCR‐Analysen einzelner mRNAs wurden mit Biogazelle qbasePLUS 2.0 berechnet. Die Signifikanz der Unterschiede zwischen zwei experimentell verschieden behandelten Gruppenwurdemit*(p<0,05),**(p<0,01)und***(p<0,001)gekennzeichnet. 3.6.3 Bildbearbeitung EingescanntehistologischeSchnittewurdenmitPanoramicViewer1.15.2analysiertund als TIF‐Dateien exportiert. Die Qualität von Fotos und anderen Dateien wurde mittels derSoftwarePhotoshop®CS4(Adobe)sinnerhaltendaufgewertet. 38 Erg gebnisse 4 Errgebnissse 4.1 D DasLSL‐MYCN;D Dbh‐iCre eMausm modell 4.1.1 G Generieru ungdertra ansgenenM Mauslinie LSL‐MYCN N DiekorrrekteReko ombination ndesLSL‐M MYCNKonsstruktsinE ES‐Zellenw wurdeanha andvon SoutherrnBlotAnaalysenunte ersucht(Ab bb.8). Abb.8:RepräsenttativeSouth hernBlotA Analyseein nesES‐Zellk klons(a)5‘‐‐Sonde(b) 3‘‐Sonde (c)neo‐‐Sonde.Mod difiziertnach hMilestone Reportvon TaconicArtemisGmbH H. Einer d der validierrten Klone e wurde veermehrt, in n Blastozyssten injizieert und in schein‐ trächtigge Mäuse implantiert i t. Die chim mären Nach hkommen wurden w miit Wildtyp‐‐Mäusen (C57Bl//6N) weiteer verpaartt, nach sch hwarzer Fellfarbe sele ektiert, mitttels PCR validiert v undanunsereArb beitsgruppetransferieert. 4.1.2 L LSL‐MYCN NBeobachttungskoho orte Eine Beobachtun ngskohorte aus 21 h heterozygotten Tieren n der trannsgenen Mauslinie LSL‐MY YCNwurdeaufAuffälligkeitenim mVerhalteenundauf Unterschieedeinder Lebens‐ zeitübeereinenZeeitraumvon n17‐23MoonatenimVergleichzzuWildtyp ‐Mäusenbeobach‐ tet.Aufffälligkeiten nkonntenn nichtfestgeestelltwerd den. 39 Erg gebnisse 4.1.3 G Genotypissierungen AusSch hwanzbiopsienvonpo otentielltraansgenenL LSL‐MYCNMäusenisooliertegeno omische DNA w wurde mitteels PCR auff Vorhandeensein des humanen MYCN‐Trannsgens unttersucht (Primerr LSL‐MYC CN – Tab.3 3). Währen nd bei Wild dtyp‐Mäuse en kein Pr odukt amp plifiziert werden nkonnte,w wiesensow wohlheteroozygoteals auchhomozygoteLSSL‐MYCNT Tiereein 168 Baasenpaar (b bp) langes Amplikon auf (Abb. 9a). Die Zy ygotie des Transgenss wurde anhand d der ROSA A26‐Wildtyp p‐PCR festggestellt (Prrimer ROSA A26‐Wildtyyp – Tab.3 3). Wild‐ typ‐un ndheterozy ygoteMäusezeigteneeine299bp pBande,wä ährendbeiihomozygo ottrans‐ genenT Tierenkein nProdukta amplifizierttwerdenk konnte(Abb b.9b).Diesslagdarinbegrün‐ det,dasssdieElon ngationszeitvon45s imFalle desVorhan d denseinsddesTransge ens,das aus~13 3.000bpbeesteht,zuk kurzwar,u umdasProd duktzubild den.Dbh‐iC Cre‐Mäusewurden ausschlließlich heeterozygot gezüchtet.. Die Geno otypisierun ngs‐PCR (P Primer Dbh h‐iCre – Tab.3) amplifizierteein300 0bplangessProdukt,w welchesinWildtyp‐M Mäusennich htgene‐ riertweerdenkonn nte(Abb.9cc). Abb.9: Repräsenttative Geno otypisierun ngs‐PCRs der d verwen ndeten Mau uslinien LS SL‐MYCN undDbh h‐iCre(a)M MYCNTranssgen(PrimeerLSL‐MYCN N),amplifiziertausgenoomischerDN NAeiner heterozyygotenund einerhomo ozygotenLSSL‐MYCNMaaus.(b)ROS SA26‐Wildtyyp‐Genlokus(Primer ROSA26 6‐Wildtyp), amplifiziert a mischer DNA A einer Wild dtyp‐ und eeiner hetero ozygoten aus genom YCN‐Maus. (cc) Dbh‐iCre Transgen ((Primer Dbh h‐iCre), amp plifiziert auss genomisch her DNA LSL‐MYC einerheeterozygoten n Dbh‐iCre Maus.wt= Wildtyp;+/ /‐=heterozy ygot;+/+= homozygot..Primer: Tab.3. 4.1.4 E EmbryotoxizitätdessGenotypssLSL‐MYCN N;Dbh‐iCre e Die Nacchkommen n aus Verpa aarungen vvon hetero ozygoten LS SL‐MYCN uund Dbh‐iCre Mäu‐ senwu urdenhinsichtlichihresGenotyp psausgezähltundderprozentuualeAnteil berech‐ net (Ab bb.10). Miittels des Chi‐Quadra C at‐Tests wu urde analy ysiert, ob ddie Verteilu ung der GenotypendenMendelschen nRegelnen ntsprach.E Eswurdeve ermutet,daass25%de erNach‐ kommeenkeinesd derTransge eneerben, 50%einesderTransgeneerhalltenund25 5%über 40 Erg gebnisse beide T Transgene verfügen. Es wurde die Nullhy ypothese aufgestellt, a dass kein n Unter‐ schiedzzwischend denzuerwa artendenu unddenbeo obachtetenVerteilunggenderGen notypen vorlag. DerChi‐Qu uadrat‐TesstbestätigttedieseHy ypothese(p p=0,68),w woraussich hschlie‐ ßenließ ß,dassderGenotypL LSL‐MYCN;D Dbh‐iCreniichtunmitttelbarletal war. V der auftrettenden Gen notypen Abb.10: Verteilung aus Verpa aarungen von v heterozzygoten LS SL‐MYCN undDbh‐iiCreMäusen nAnteilderrmöglichen nGenoty‐ pen als Prrozentbalken (n=239)). Chi‐Quadrat‐Test: χ2=0,17; Freiheitsgrrad=1; p==0,68. Beobachtete dererwarteeten. Verteilung entsprichtd 4.1.5 T Tumorinzidenzund d‐lokalisattion Heterozzygote LSL L‐MYCN Mä äuse wurd den mit heeterozygote en Dbh‐iCree Tieren verpaart v und do oppelt‐transgene Nachkommen wöchentlich auf Tum more abgettastet. Mitttels Ka‐ plan‐Meier‐Analysse wurde das Tumoor‐freie Überleben de er doppeltt‐transgene en Tiere (n=38) im Vergleeich zu derr Beobachttungskohorrte (n=21 –s.4.1.2), die aus he eterozy‐ goten L LSL‐MYCN Mäusen bestand, b au ufgezeichneet (Abb.11 1). Währennd keine der d LSL‐ MYCN Mäuse inn nerhalb derr Beobachttungszeit von v 394 bis b 688 Taagen einen Tumor aufwiess, wurde bei b LSL‐MY YCN;Dbh‐iCrre Mäusen n eine Tum morinzidenzz von 76,3 3% beo‐ bachtett, wobei sicch die Tum more im Allter von 26 6‐337 Tage en entwicke kelten. Die meisten Tumoree entwickeelten sich spätestens in einem Alter A von 123 Tagen (96,5%),w während nurein nTumordeerKohorte nachdieseerZeitentstand.Tum mor‐tragenddeMäuseb bildeten in88,8% %derFälleeinenoderzweiTu umoreand denNebenn nierenaus,,während Tumore anden oberenZeervikalgang glien(33,3% %)unddem mGanglion ncoeliacum m(11,1%)sseltener men.HäufiggwarenmehrereTum moreanden nverschiedenenGeweebenineinerMaus vorkam zu find den. Tumorre am Gan nglion coelliacum kam men aussch hließlich inn Verbindu ung mit Nebenn nierentumo oren vor, während w Tu umore am oberen Ze ervikalgangglion auch einzeln auftrateen (11,1%)). Ein Zusa ammenhan g zwischen n diesen Beobachtunggen konnte e statis‐ tischniichtuntersu uchtwerde en,dadieA Anzahldero observierte enFällezu geringwarr. 41 Erg gebnisse 1: Entwickllung von Tumoren T Abb.11 im LSL L‐MYCN;Dbh h‐iCre Mau usmodell Kaplan‐‐Meier‐Anallyse des Tumor‐ freien Überlebenns von LS SL‐MYCN n (Be obachtungsskohorte; Mäusen n=21) Vergleich h zu im YCN;Dbh‐iCrre Tieren (n=38). LSL‐MY Tumore entwicklungg an den Nebennie‐ ren,dem mGanglion coeliacumu unddem oberen Zervikalgganglion. Log‐Rank L Test. Zellen d der LSL‐MYCN;Dbh‐iC Cre Maus, in denen die d Cre‐Rek kombinase e aktiv warr, da die Polyadeenyl‐Stoppsequenzhe erausgesch hnittenwurrde,sollten nnebendeemOnkogenMYCN auch Lu uziferase exprimieren e n. Luziferin n wird in Gegenwart G von Luzifeerase oxidiiert und kann m mittels Lum mineszenz‐‐Bildgebungg visualisiiert werden (Abb.122). Repräse entative Tumoree konnten sowohl an n den Nebeennieren (I,, II und III), als auch am oberen Zervi‐ kalgangglion(I)un ndamGang glioncoeliaccum(III)naachgewiese enwerden.. 2: Luziferasse‐Expressiion dreier repräsenttativer Tum more aus LLSL‐MYCN;D Dbh‐iCre Abb.12 Mäusen n In vivo o Biolumiineszenz B Bildgebung repräsen ntativer Tuumore vo on drei LSL‐MYC YCN;Dbh‐iCree Mäusen an n den Neben nnieren (I, II und III), am a oberen Zervikalgan nglion (I) undamGanglionco oeliacum(IIII).Luziferasee‐Aktivität:blau=niedrrig;rot=hooch. 42 Ergebnisse Die Tumore des LSL‐MYCN;Dbh‐iCre Mausmodells wurden mittels Ultraschall‐Bildge‐ bungweiteruntersucht.WährenddieoberenZervikalganglienaufgrundihrergeringen Größe in heterozygoten LSL‐MYCN Tieren nicht genau dargestellt werden konnten (Abb.13a), waren repräsentative Tumore die aus diesen Geweben entstanden waren, deutlichalsechoärmere,raumforderndeStrukturinderHalsregionrechtsundlinksder Luftröhrenachweisbar(Abb.13b).ZurAuffindungeinerNebennierewurdezunächstdie Lokalisation der Niere anhand ihrer auffälligen Struktur aus Nierenrinde, ‐mark und ‐becken, die im Ultraschall deutlich erkennbar waren, definiert (nicht gezeigt). In RichtungkranialkonntebeiheterozygotenLSL‐MYCNTierendieNebenniereamvorde‐ ren Nierenpol dargestellt werden (Abb.13c). Die Nebennierenrinde war als echoarme Strukturdeutlicherkennbar,währenddasinnenliegendeNebennierenmarkeinstärke‐ res Echo aufwies. Tumore der Nebenniere, die im LSL‐MYCN;Dbh‐iCre Mausmodell auftraten, konnten als echoarme Strukturen am oberen Nierenpol dargestellt werden (Abb.13d).Mittelsder3D‐FunktiondesVevo2100wurdendie3‐dimensionalenStruk‐ turen verschiedener Gewebe rekonstruiert (Abb.13e) und ihre Volumina bestimmt. Nicht‐maligne Nebennieren ausgewachsener Mäuse wiesen ein mittleres Volumen von 2,18mm3(±0,61mm3)auf(Datennichtgezeigt).DieVoluminaderNebennierenvonje sechs LSL‐MYCN und LSL‐MYCN;Dbh‐iCre Tieren wurden über 100Tage wöchentlich gemessenundgegendasAlterderTiereaufgetragen(Abb.13f).ZwischendenVolumina nicht‐maligner Nebennieren von LSL‐MYCN;Dbh‐iCre und LSL‐MYCN Mäusen konnte kein Unterschied detektiert werden. Zwei der doppelt‐transgenen Mäuse entwickelten innerhalb der Beobachtungszeit Tumore an einer der beiden Nebennieren, anhand deren Volumina sich ein exponentielles Wachstum nachweisen ließ. Ein weiteres LSL‐ MYCN;Dbh‐iCre Tier dieser Kohorte bildete einen Tumor des oberen Zervikalganglions mitschnellerVolumenvergrößerungausundmusstenach50Beobachtungstagengetö‐ tetwerden(Datennichtgezeigt).DieTumorinzidenzderhierbeobachtetenKohortelag innerhalbderStreuungderGesamtkohorte. 43 Erg gebnisse Abb.13 3: Ultrasch hall‐Bildge ebung von n Tumore en aus LSL‐MYCN;D L Dbh‐iCre Mäusen (a)DurcchschnittdeerHalsregioneinerheteerozygotenLSL‐MYCNM MausinHöhhederobere enZervi‐ kalganglien (aufgru und der gerringen Größ ße nicht geenau definie erbar). (b) Durchschniitt durch nglien entsstandene Tumore T einner repräse entativen beidseittig aus den oberen Zervikalgan LSL‐MYC YCN;Dbh‐iCreeMaus.(c)D Durchschnitttderamvo orderenNierrenpollokallisiertenNeb benniere (NN)ein nerheterozy ygotenLSL‐MYCNMauss.Nebennierrenrindesch hwarz;Nebeennierenma arkweiß‐ meliert. (d) Durch hschnitt durrch einen aaus der Neb benniere he ervorgeganggenen Tumor einer YCN;Dbh‐iCree Maus. (e)) 3D‐Rekon nstruktion eines e reprässentativen N Nebennierentumors LSL‐MYC einer L LSL‐MYCN;D Dbh‐iCre Maus. M (f) Nebenniereenvolumina von je 6 LSL‐MYC CN und LSL‐MYC YCN;Dbh‐iCreeMäusenmittels3D‐Reekonstruktio onaufgetrag gengegenddasAlterdessjeweili‐ gen Tieres. Zwei der d doppelt‐‐transgenen n Tiere entw wickelten in nnerhalb deer Beobachttungszeit umoranein nerderNebe ennieren. einenTu 44 Erg gebnisse Sobald ein Tumo or ein Volu umen von 500mm3 überschrittt, die Atm mung einess Tieres nd eines die Luftröhrre zudrückkenden Tum mors einge eschränkt w war oder ein e Tier aufgrun einenaallgemeinen nschlechte esHabitusaaufwies,wu urdediesessdurchzerrvikaleDisllokation getötet. Während d der Sektiion wurden n makrosk kopische Aufnahmen A gemacht, anhand derersowohlTum morederN Nebennieren nunddes Ganglioncoeliacum(A Abb.14a)a alsauch desobeerenZervik kalganglion ns(Abb.14b b)dokumeentiertwurden. Abb.14 4: Makroko opische Au ufnahmen repräsenta ativer Tum more des LLSL‐MYCN;D Dbh‐iCre Modellss (a) Darstellung beidseitig entsttandener Neebennierenttumore (recchts und lin nks) und eines Tu umors des Ganglion co oeliacum (M Mitte). (b) Darstellung D eines e Tumoors, hervorg gegangen ausdem moberenZerrvikalganglionnahederrLuftröhre. Stoppseque enzingenoomischerD DNAvon DasVorrhandenseinderspezzifischenPoolyadenyl‐S LSL‐MY YCN;Dbh‐iCre Mäusen n deutete d darauf hin, dass in de em jeweiliggen Geweb be keine Rekomb bination stattgefunden hatte, also keinee Cre‐Reko ombinase eexprimiert wurde. Mittels PCR(Prim merflox‐outt‐Tab.3)kkonnteing genomische erDNAaussHerzgewebevon LSL‐MY YCN;Dbh‐iCre Tieren, ein 224 41bp lang ges Amplik kon nachggewiesen werden (Abb.15). In geno omischer DNA D aus Tu umoren dees LSL‐MYC CN;Dbh‐iCrre Modells, in wel‐ cher die Polyadeenyl‐Stoppsequenz aaufgrund der d Expresssion der Cre‐Rekom mbinase ausgescchnitten wurde, w war das Ampliikon 703bp lang. Die e Aktivieruung des Transgens durcheeinRekomb binationserreigniskon nntesoindenTumore ennachgew wiesenwerd den. 45 Erg gebnisse ngderausg geschnit‐ Abb15:PCRzurValidierun tenen Polyadeny yl‐Stoppseq quenz in vier v re‐ präsen ntativen Tumoren au us LSL‐MY YCN;Dbh‐ iCre Mäusen M Kon nditionales LSL‐MYCN Allel im Herz2 2241bplang,konstituttivesAllelim mTumor der jew weils gleich hen Maus 7703 bp lang g. Primer flox out o (Tab.3 3). wt=W Wildtyp; He e=Herz; Tu=Tumor. Die Exp pression dees Transge ens MYCN w wurde sow wohl auf mRNA‐Ebenee mittels qPCR q als auch au uf Protein‐‐Ebene mitttels Westeern Blot in n Kontrollg geweben voon Wildtyp p‐Tieren undTu umorenvon nLSL‐MYCN N;Dbh‐iCreeMäusenaanalysiert.D DieMYCN mRNAExp pression vier rep präsentativ ver Tumore sowie deer Kontrollgewebe Niiere, Leberr, Herz und d Gehirn wurde relativ zurr Expressio on der niccht‐malingeen Nebenniere aufgettragen (Ab bb.16a). Tumoree zeigten im Vergleicch zu Nebeennieren eiine signifik kant, im M ittel 14,5‐ffach, er‐ höhteE ExpressionvonMYCN NmRNA.Im mVergleich hderTumorezudenaanderenan nalysier‐ ten Kon ntrollgeweeben war die d Hochreegulation von v MYCN mRNA nocch deutlich her. Die erhöhtee Expressio on des Pro oteins MYC CN im Tum mor im Vergleich zum m Herz derr jeweils gleichen Maus wurde in vier repräseentativen LSL‐MYCN;D L Dbh‐iCre M Mäusen darrgestellt (Abb.16b). Abb.16 6: Überexp pression vo on MYCN in Tumoren des LS SL‐MYCN;D Dbh‐iCre Modells M (a)Balk kendiagramm mderMYCN NmRNAExp pressionvieerrepräsenttativerTum moreundKontrollge‐ weben aus einer Wildtyp‐Ma aus relativ zur Expresssion der Nebenniere N dent‘s t‐ (NN). Stud b)MYCNProteinExpreessionimHeerz(He)und dTumor(Tuu)vierrepräsentati‐ Test:p=0,0002.(b use.GAPDHalsLadungsskontrolle. verMäu 46 Erg gebnisse 4.1.6 T Tumorhistologieun ndExpresssionspeziffischerNeu uroblastom mmarker Die Exp pression deer Neurobllastommarkker Dbh, Th T und Phoxx2b wurdee auf mRNA A‐Ebene mittels qPCRinveerschieden nenGewebeendesLSL‐‐MYCN;Dbh h‐iCreModdellsanalysiert.Die Markerr‐Expressio on vier repräsentativeer Tumore und der Kontrollgew K webe Niere e, Leber, Herzun ndGehirnw wurderela ativzurExp pressiondeernicht‐ma alignenNebbenniereaufgetra‐ gen (Ab bb.17). Inn nerhalb de er Kontrolllgewebe war w die Exp pression alller Markerr in der Nebenn niereamhö öchsten.DieTumore wiesenveerglichenm mitnicht‐m malignenNe ebennie‐ ren im Mittel einee 2,4‐fach erhöhte Exxpression von v Dbh, eine 4,2‐facch erhöhte Expres‐ sion vo on Th und eine 22‐fach erhöhtee Expressio on von Pho ox2b auf. D Die Hochreg gulation deranaalysiertenN Neuroblasto ommarkerindenTum morenwarsignifikantt. Abb.17: mRN NA Expresssion von Neuro‐ bla astommark kern in vieer repräsen ntativen Tumoren aus s LSL‐MYCN N;Dbh‐iCre Mäusen Ballkendiagram mm der N euroblastom mmarker mR RNA Expre ession vieer repräse entativer Tum more und Kontrollgeeweben relativ zur Exp pression de er nicht‐maalignen Neb benniere (NN N). Signiffikante H Hochregulatiion in Tum moren im Vergleichh zur Neb benniere (Sttudent‘st‐Te est:Dbh:p==0,005;Th:p=0,02; Pho ox2b: p=0,0006). D Dbh=Dopam min‐beta Hydroxylase;T Th=TyrosinnHydroxylase. Die Strruktur reprräsentative er Tumore aus LSL‐M MYCN;Dbh‐iCre Mäus en wurde anhand histologgischer Sch hnitte, die mit Hämattoxylin und d Eosin an ngefärbt wuurden, untersucht. DieTum morebestaandenausk kleinen,blaauen,rundeenZellen,ty ypischfürddasNeurob blastom, das aauch im humanen n Organissmus diese Strukttur aufw weist1 (Ab bb.18a). Elektro onenmikrosskopische Aufnahmeen zeigten neurosek kretorische Vesikel, die für neuron nales Geweebe spezifissch sind1 ((Abb.18b).. Weiterhin n wurde CCD56, ein neurales n Zelladh häsionsmolekül,dasin nneuronallenundneu uroendokrinenGeweebenundTumoren 8 , immunh exprimiert wird88 histochemi sch dargestellt (Abb b.18c). Diee Expression des Neurob blastommarrkers Th, die d schon aauf Transk kriptionseb bene gezeiggt werden konnte, wurdeaanhandimmunhistochemischerrFärbungen naufProteinebenevaalidiert(Ab bb.18d). 47 Erg gebnisse Abb.18 8: Histolo ogische und u moleekulare Charakteris C tika von n Tumore en des LSL‐MY YCN;Dbh‐iCrreModells (a)HämatooxylinundE EosinFärbung.Überwieegendkleine,runde, blaue Zellen. Maßsstabsbalken: links=10 0µm; rechtts=50µm. (b) Elektroonenmikrosk kopische memitneurrosekretorisschenVesikeeln(Pfeile).Maßstabsb balken=5000nm.(c)Im mmunhis‐ Aufnahm tochemiische Färbu ung von CD D56 (links) und Negativkontrolle e nur mit SSekundäran ntikörper (rechts)). Maßstabssbalken=20 00 µm. (d) Immunhisttochemische e Färbung vvon Th (lin nks) und Negativk kontrollenu urmitSekun ndärantikörrper(rechts)).Maßstabs‐balken=2000µm. 4.1.7 V Vergleich derTumo orinzidenzzdesLSL‐M MYCN;Dbh‐iCreMaussmodellsm mitder d desetablie ertenTH‐M MYCNMau usmodells Das Tumor‐freie Überleben Ü von LSL‐M MYCN;Dbh‐iCre Mäuse en wurde m mit dem des etab‐ lierten TH‐MYCN Neuroblasstommodellls verglich hen. Die LS SL‐MYCN;D Dbh‐iCre Tiiere be‐ fanden sich im gemischte en genetisschen Hinttergrund aus C57Bll6/N (75% %) und 129x1//SvJ (25%)), während d der geneetische Hintergrund der analyysierten TH H‐MYCN Mäuse 100%129xx1/SvJwarr.TH‐MYCN NTiereim gemischten nHintergruundausC5 57Bl6/N %) entwickkelten kein ne Tumore e (Daten nnicht gezeigt), wie (50%) und 129x1/SvJ (50% schon iin vorheriggen Publik kationen beeschrieben wurde37. LSL‐MYCN N;Dbh‐iCre Mäusen zeigten n im Vergleich zu TH H‐MYCN T Tieren ein signifikantt schlechteeres Tumo or‐freies Überleb ben,ermittteltanhand deinerKap plan‐Meier‐Analyse(A Abb.19).D DieTumoriinzidenz des T TH‐MYCN‐M Modells be etrug 48% % und war w damit geringeer als die d des LSL‐MY YCN;Dbh‐iCre Modellls (76,3% %). Das mediane m Tumor‐freiee Überleben von TH‐MYC CN Mäusen n betrug 154 Tage, w während das d von LSL‐MYCN;Dbbh‐iCre Tie eren bei 82,5Taagenlag.DiejenigenTiere,dieim mLaufederrBeobachtu ungmindesstenseinen nTumor 48 Erg gebnisse ausbild deten, entw wickelten diesen im T TH‐MYCN Modell M im Mittel M nachh 85,3 Tage en ihres Lebens,imLSL‐MYCN;Dbh‐iC CreModell nach79,6Tagen. gleich derr Tumoriinzidenz Abb.19: Verg zwischen dem LSL‐MYCN N;Dbh‐iCre Modell und dem TH‐MYCN Mod dell Kaplan‐Meier‐ Analy yse des Tumor‐freie T en Überlebens von heterrozygot transgenen TH‐MYCN Mäusen (n=8 84) und LSL‐MYCN N;Dbh‐iCre Tieren (n=3 38).Tumoriinzidenzin LSL‐MYCN;D Dbh‐iCre Mäussensignifika anthöher(LLog‐RankTe est). heAberra ationenvon nLSL‐MYC CN;Dbh‐iCr reTumoreen 4.1.8 G Genomisch GenomischeAberrrationenvon13LSL‐‐MYCN;Dbh h‐iCreTumo oren(elfTuumorederrNeben‐ nieren undzweiT Tumorede esoberenZ Zervikalgan nglions)wu urdenrelattivzuGew webeaus dem Scchwanz derr jeweils gleichen Maaus mittelss einer aCG GH Analysee detektiertt und in einerÜ Übersichtsggraphikden njeweiligen nChromosomenzuge eteilt(Abb..20a).Diemittlere Zeit biss zur Entw wicklung eiines Tumoors in dieser Kohorte e betrug 880,4 Tage. In zwei Tumoreen wurden n keine chrromosomallen Aberrationen dettektiert. Diee mittlere Zeit bis zur Enttwicklung dieser beid den Tumorre betrug 46 4 Tage. Ein Zusamm menhang zw wischen der Zeiit bis zur Tumorentw T wicklung u und dem Auftreten A ch hromosom maler Aberrrationen konnte aufgrund der gering gen Anzahll an analysierten Tum moren nichht untersuccht wer‐ den. Einer d der analysiierten Tum more einer Nebennieere zeigte eine e teilweeise Zunah hme des Chromo osoms 1. In n acht Tum moren der Nebennierre und eine em Tumor des oberen Zervi‐ kalgangglions wurrde eine Zunahme Z d des kompleetten Chro omosoms 33 detektiert. Eine Zunahm me des kom mpletten Chromosom ms 6 zeigten n fünf der analysierteen Tumore e, wobei sowohll Tumore der d Nebenn nieren als aauch des oberen o Zerv vikalgangliions betrofffen wa‐ ren. Au uf diesem Chromoso om liegt deer ROSA26 6 Genlokuss, in den ddas Transg gen des LSL‐MY YCNMausm modellskloniertwurd de.EinTum morderNe ebenniereuundeinerd desobe‐ ren Zervikalgangglions amplifizierten diesen Lo okus sogarr (Abb.20bb). Eine niedriger frequen ntierte Zun nahme der distalen H Hälfte des Chromosom C ms 11 wurdde in vier der Tu‐ more ggefunden (A Abb.20c), entsprecheend einer Region R auff dem hum manen Chro omosom 17, diee in 50% aller a huma aner Neurooblastome eine Zunahme aufweeist42. In drei d der Tumoreewurdeein nekomplettteZunahm medesChro omosoms1 12festgesteellt.BeideTumore 49 Erg gebnisse desobeerenZervik kalganglion nssowieein nTumorderNebennierewiesenndieseAbe erration auf. Interessanterweisse wies ein ner der Tu umore des oberen Ze ervikalgangglions die gleichen g Aberrattionenauf wiederTu umorausd derNebenn nierederg gleichenMaaus(Abb.2 20a(Tu‐ mor12 2und13)), während derzweiteeanalysiertteTumord desoberen Zervikalga anglions andere Aberratio onen als der dazugeh hörige Tum mor der Nebenniere N e zeigte (A Abb.20a (Tumorr8und9). Teilweiise oder komplette k Verluste gganzer Chrromosomen n wurden in Tumorren des LSL‐MY YCN;Dbh‐iCreModellsnichtdeteektiert. 0: Genomissche Aberrrationen vvon Tumorren des LS SL‐MYCN;D Dbh‐iCre Modells M Abb.20 Analyse von 13 LSL L‐MYCN;Dbh h‐iCre Tumooren relativ v zu Gewebe e aus dem SSchwanz der jeweils n Maus. (a) Überblick über ü genom mische Aberrrationen alle er analysierrten Tumore. blau = gleichen Zunahm me;rot=Abn nahme.(b)R RepräsentattiveDarstelllungdesinzzweiderTuumoreampliifizierten ROSA26 6GenlokusaaufChromossom6.(c)Reepräsentativ veAnsichtd derZunahmeederdistale enHälfte 1 in vier der Tumore.. Graphik geeneriert in Zusammenaarbeit mit Anneleen A des Chrromosoms 11 Beckerss. 50 Ergebnisse 4.1.9 mRNAProfiling Mittels Affymetrix mRNA Arrays wurden mRNA Expressions Profile von drei nicht‐ malignenNebennierenundachtLSL‐MYCN;Dbh‐iCreTumorenerstellt. 4.1.9.1 Gene Set Enrichment Analyse (GSEA) von Tumoren des LSL‐MYCN;Dbh‐iCre Mausmodells Eines der am stärksten angereicherten Gensets innerhalb der in Tumoren von LSL‐MYCN;Dbh‐iCre Mäusen hochregulierten Gene, das anhand der GSEA detektiert wurde, beinhaltete Marker pädiatrischer Krebserkrankungen89 (Abb.21a (oben)). Die‐ ses Genset besteht aus in Xenografts, die acht verbreitete pädiatrische Tumore reprä‐ sentieren,verschiedenexprimiertenGenenimVergleichzuNormalgewebe.Einweiteres Genset, das in Tumoren des LSL‐MYCN;Dbh‐iCre Modells stark angereichert war, bein‐ halteteGene,dieinBZellLymphomen,dieeineaktivierteFormvonMYCexprimieren, hochreguliertsind90(Abb.21a(unten)).WeiterevonMYChochregulierteGensetswaren inTumorenausLSL‐MYCN;Dbh‐iCreMäusenangereichert91‐93,währendvonMYCherun‐ terregulierteGensets90,93,94inNebennierenangereichertwurden(Abb.21b).Zusätzlich waren verschiedene Gene, die die DNA Replikation und den Zellzyklus betreffen95‐98, statistisch signifikant hochreguliert in Tumoren des LSL‐MYCN;Dbh‐iCre Modells. Ein weiteresGenset,dasGenerepräsentierte,diewährendneuralerDifferenzierungherun‐ terreguliert sind99, wurde in Tumoren aus LSL‐MYCN;Dbh‐iCre Mäusen hochreguliert, wasdieHypotheseeinesundifferenziertenPhänotypunterstützt. 51 Erg gebnisse 1:GensetEn nrichmentA AnalyseAch htLSL‐MYCN N;Dbh‐iCreTumoreundddreinicht‐maligne Abb.21 NebennierenvonW Wildtyp‐Mäu usen.(a)Hoochregulatio oneinesGen nsets,dasM Markerpädia atrischer nrepräsentiiert(oben) undeinesM MYCN‐abhän ngigenGenssets(unten).Gezeigt Krebserrkrankungen istderP PlotderRan ngsummend desMSigDB (v3.1)Genssets.(b)Tabelleausgew wählterGen nsetsder MSigDBC2 Kollektiion,dieinL LSL‐MYCN;D Dbh‐iCre bzw w.inNeben nnierensignnifikantange ereichert ng des Genssets innerh halb aller Gensets, G geo ordnet nachh abfallende em NES, waren. Rang=Ran =AnzahlderrGeneinjed demGenset,,NES=norm malisierterE Enrichment Score,FDR=falsch‐ Größe= positivR Rate.GraphiikgeneriertinZusamm menarbeitmiitAnneleenBeckers. 4.1.9.2 Expressio on einer Siignatur MY YC(N) regu ulierter mR RNAs in LSSL‐MYCN;D Dbh‐iCre Tumoren An den n gleichen mRNA Arrray Daten, die für diee GSEA verrwendet w wurden, wu urde auf Basis d der von Freedlund eta al. publizierrten MYC(N N) Signatur humanerr Neurobla astome78 einezw wei‐dimensionaleClussteranalyseedurchgefü ührt(Abb.22).DesW Weiterenwu urdedie Regulattion dieserr mRNAs in humaneen MYCN‐amplifizierrten im Veergleich zu u nicht‐ amplifizzierten Neeuroblastom men78 und d die der Internet‐Datenbank www.myc‐‐cancer‐ gene.orrganhandvonPfeilen ndargestel lt. Die niccht überwaachte Clustteranalyse anhand deer Gene de er MYCN SSignatur fü ührte zu einereiindeutigenSeparation nvonNebeennierenun ndLSL‐MYC CN;Dbh‐iCrreTumoren n. 52 Erg gebnisse Abb,22 2:RegulierttemRNAsin nTumoren ndesLSL‐M MYCN;Dbh‐iC CreMausm modellsZwe ei‐dimen‐ sionale hierarchisch heClusteran nalysebasieerendaufdeerExpressio ondermRNA AsderMYC CNSigna‐ n Fredlund et al.78 in n acht LSL ‐MYCN;Dbh‐iCre Tumo oren und ddrei nicht‐m malignen tur von 7 78 Nebennieren.RegullationinMY YCN‐amplifizziertenhum manenNeuro oblastomen undRegu ulationin myc‐cyncer‐‐gene.org) aals Pfeile. Exxpression: blau=hoch; b ; rot = nied drig. Gra‐ Datenbaank (www.m phikgen neriertinZu usammenarb beitmitAnn neleenBeckers. on von Nra as, die aucch für das humane Neuroblasto N om bekann nt ist100, Die Hochregulatio modellvalid diertwerdeen.DemGenUBE2Cw wirdeinepoositiveRollleinder konnteimMausm uroblastomzelllinien zugeschrieeben101. Auch A in LSSL‐MYCN;D Dbh‐iCre Mitose von Neu hregulation n detektierrt werden. In humanen Neurobblastomen ist eine konnte eine Hoch hohe N NME1 Exprression miit einem u ungünstigeen Stadium m assoziierrt102. Tumo ore des LSL‐MY YCN;Dbh‐iCre Modellss überexprrimierten diese d mRN NA ebenfallls. Der Lak ktat‐De‐ hydrogenase Leveel im Blut von Neurooblastompaatienten dient in der Klinik als Tumor‐ es Gens in n LSL‐MYCN N;Dbh‐iCre Tumoren zeigte, da ass auch markerr13. Die Reggulation de murineeNeuroblasstomediesesEnzymü überexprim mierten.SRM,einbekaanntesnachgeord‐ netes T Target von n MYCN103,, war im LSL‐MYCN N;Dbh‐iCre Modell eb enfalls sig gnifikant regulierrt. 53 Erg gebnisse 4.1.9.3 Regulatio onbekannte erMYCNm mRNATargeetsinTumorendesLSSL‐MYCN;D Dbh‐iCre Mausmod dells Die Exxpression bekannter b MYCN reegulierter Targets wurde w gesoondert unttersucht (Abb.23). Die Carrbamoyl‐Ph hosphat Syynthetase 2 2 (Cad)wirrdin verscchiedenen Publika‐ P tion alss von MYC und MYCN N hochregu uliertes En nzym besch hrieben92,1004,105. Ein ebenfalls vonMY YChochreguliertesZie elistdieCyyclin‐abhän ngigeKinasse4(Cdk4)),dieeinew wichtige Rolle in n der Regu ulation des Zellzykluss spielt106,107. Eine sig gnifikant errhöhte Exp pression konnte auch in Tu umoren de es LSL‐MYC CN;Dbh‐iCre Modells im Verglei ch zu nicht‐malig‐ nen Neebennieren n festgestellt werden n. Die MYC CN‐induzierte Herunnterregulatiion von Dickkop pf‐3(Dkk3))wirdinh humanenN Neuroblasto omzellenm mitderdireektenBindu ungvon miR‐19 9bundmiR R‐92aandie e3’UTRerkklärt108,109.Tumored . erLSL‐MY YCN;Dbh‐iCreMaus zeigten n im Vergleeich zu nich ht‐maligneen Nebenniieren eine signifikantte Herunterregula‐ tionvon nDkk3. 3: Reguliertte MYC Tarrgets in Tu moren dess LSL‐MYCN N;Dbh‐iCre M Modells Ex xpression Abb.23 (log2)M MYCNhochrregulierter((CadundCddk4)undMY YCNherunte erregulierterr(Dkk3)Zie elgenein LSL‐MYC YCN;Dbh‐iCreeTumorenu undnicht‐m malignenNeb bennieren.S Student‘st‐te test. 4.1.10 miRNAPrrofiling urden miRN NA Profilee von fünff nicht‐mallignen Nebbennieren und elf Mittels qPCR wu LSL‐MY YCN;Dbh‐iCreTumore enerstellt. 4.1.10.1 1 Expressio on einer Signatur MYCN reegulierter miRNAs iin Tumorren des LSL‐MYCN N;Dbh‐iCreModells Eine zw wei‐dimenssionale Clusteranalys e wurde au uf Basis de er von Messtdagh eta al. publi‐ zierten MYCSignaaturhuman nerNeurob blastomedurchgeführrt80(Abb.224).DieReg gulation der miiRNAs in humanen h MYCN‐amp plifizierten im Vergleich zu niicht‐ampliffizierten 54 Erg gebnisse Neurob blastomen, auf der die MYCN Siignatur berruht, wurde anhand vvon Pfeilen n darge‐ stellt. Dienich htüberwacchteCluste eranalyseaanhandderrmiRNAsd derMYCNSSignaturfü ührtezu einer eeindeutigen n Separatio on von Neb bennieren und Tumo oren aus LSSL‐MYCN;D Dbh‐iCre Mäusen n. 4: Reguliertte miRNAs in Tumoreen von LSL L‐MYCN;Dbh h‐iCre Mäussen Zwei‐dimensio‐ Abb.24 nalehieerarchischeC Clusteranaly ysebasieren ndaufderE Expressiond dermiRNAssderMYCNSignatur vonMesstdaghetal..80inelfLSL L‐MYCN;Dbhh‐iCreTumo orenundfün nfnicht‐mallignenNebe ennieren. Regulatiion in MYCN‐amplifizie erten humaanen Neurob blastomen als a Pfeile80. Expression n: blau = hoch;ro ot=niedrig.Graphikgen neriertinZu usammenarb beitmitAnn neleenBeckkers. Verschiiedene miR RNAs des bekannten b n onkogeneen miR‐17‐‐92 Clusterrs110 (miR‐‐18a‐3p, miR‐18 8a‐5p, miiR‐19a‐3p, miR‐20aa‐5p, miR R‐20b‐5p, miR‐92a ‐3p) warren in LSL‐MY YCN;Dbh‐iCreTumore ensignifikaanthöhereexprimiertalsinnichtt‐malignenNeben‐ nieren. Dieonkoggene,direktvonMYC Nreguliertte80,miR‐1 181azeigteeeineÜberrexpres‐ N;Dbh‐iCreT Tumoren.V VerschiedeenebeschriebenemiR RNAsmitpo otentiell sionin LSL‐MYCN tumorsuppressiveer Funktion im Neurroblastom wie miR‐1 103‐3p111, miR‐184‐3 3p112,113, 1 undmiR miR‐21 14‐3p114,m miR‐340‐3p115 R‐542‐5p677,116zeigten nauchinLSSL‐MYCN;D Dbh‐iCre Tumoreen eine Heerunterreg gulation. W Weitere miR RNAs, die im humannen Neurob blastom 55 Erg gebnisse gleich rreguliert werden w wie e in Tumorren des LS SL‐MYCN;Db bh‐iCre Moodells ware en miR‐ 15b, m miR‐30c, miR‐103, miiR‐140, miiR‐148a, miR‐326, m miR‐491, m m miR‐500 un nd miR‐ 61580.V VerschiedeenemiRNAss,wiezum BeispieldiiebekannteonkogeneemiR‐9‐5p p,dieim 1 ,zeigteinLSL‐MYC humaneenNeurobllastomvon nMYCindu uziertwird117 CN;Dbh‐iCre eTumo‐ renkeineeindeuttigeRegula ation.Ande remiRNAsswarenentgegengeseetztzumhu umanen Neurob blastom reguliert. Zu um Beispieel ist die miR‐137 in humaneen Primärttumoren signifik kantheruntterreguliert(s.4.3),w währendTu umoredes LSL‐MYCN N;Dbh‐iCre Modells eineein ndeutigeHo ochregulattionzeigten n. 4.1.10.2 2 Regulatio on von miRNAs des miR‐‐17‐92 Cllusters inn Tumore en des LSL‐MYCN N;Dbh‐iCreMausmod ells Die Exp pression bekannter b MYCN reggulierter Taargets des miR‐17‐992 Clusterss wurde ebenfallls untersu ucht (Abb.25). Einee signifikaante Hochregulation von miR R‐17‐5p, miR‐19 9a‐3pundm miR‐92a‐3p pinTumorrenvonLS SL‐MYCN;Db bh‐iCreMääusenimVergleich zu nich ht‐malignen n Nebennieren konn nte validierrt werden. Weiterhinn waren auch a die anderen nMitgliedeerdesClussters(miR‐ 17‐3p,miR R‐18a‐5p,m miR‐18a‐3pp,miR‐20a‐5pund miR‐19 9b‐3p)signifikanthochreguliert (Datennichtgezeigt). Abb.25 5: Reguliertte MYCN miRNA m Targgets in Tum moren des s LSL‐MYCN N;Dbh‐iCre Modells Expression (log2) ausgewähltter MYCN h hochregulieerter miRNA As des miR R‐17‐92 Clu usters in YCN;Dbh‐iCreeTumorenu undnicht‐m malignenNeb bennieren.S Student‘st‐te test. LSL‐MYC 56 Erg gebnisse 4.1.11 m miR‐542a alspotentiielltumorm moduliere endemiRN NAimNeurroblastom m Eine tu umorsupprressive Fun nktion der 5p‐Isoform von miR‐542 ist bereits in Neuro‐ blastom mzellen gezzeigt word den68. Auch h für miR‐5 542‐3p konnte eine tumorsupp pressive Funktio onimKolon nkarzinom mnachgewieesenwerdeen69.DieAnalyseder Expression ndieser miRNA imLSL‐MY YCN;Dbh‐iC CreModell sollteAufsschlussdarrübergebenn,obdiese emiRNA auchim mmurinenNeuroblasttommodelllreguliertw wurde. Es steellte sich heraus, dass d beidee Isoform men der miR‐542 m iin Tumorren aus LSL‐MY YCN;Dbh‐iCre Mäusen n im Verglleich zu niicht‐malign nen Nebennnieren sig gnifikant herunteerreguliertt waren. In n 91% derr analysierrten Tumo ore war diie Expressiion von miR‐54 42‐5psonieedrig,dassdiesenich htdetektierrtwerdenk konnte.Dieesgaltauch hfürdie Expresssion von miR‐542‐3p m p in 55% d der Tumoree. Weiterhin wurde ddie Expresssion von miR‐54 42‐5pinzeh hnunddie emiR‐542‐ 3pExpresssionin21T TH‐MYCNT Tumorenre‐analy‐ siert.AuchindiessenTumore enwarenb beideIsoformensigniifikantheruunterreguliiertund in90%(miR‐542‐‐5p)bzw.7 76%(miR‐5 542‐3p)sogarnichtdetektierbarr(Abb.26)). Abb.26 6: Regulatio on von miR R‐542 in Tu umoren der Mausmod delle LSL‐M MYCN;Dbh‐iCre und TH‐MYC CN Dotplotss der Expression von (a) miR‐5 542‐5p und d der von (b) miR‐54 42‐3p in LSL‐MYC YCN;Dbh‐iCreeundTH‐MYCNTumorrenrelativzurExpressionnicht‐maalignerNebe ennieren (NN).Sttudent‘st‐Teest:***=p< <0,001. Aufgrun nd der bek kannten Ro ollen der Issoformen von v miR‐54 42 in verscchiedenen Tumor‐ zelllinieen und ihrer starke en Regulattion im neeuen LSL‐M MYCN;Dbh‐‐iCre Maussmodell, wurde deren poteentiell tum morsuppresssive Funkttion in hum manen Neuuroblastom men und Zelllinieenweiteraanalysiert((s.4.2). 57 Erg gebnisse 4.2 D Dietumo orsupprressiveF Funktio onvonm miR‐542 2inhum manen N Neurobla astomen nundZe elllinien n Beide IIsoformen der miR‐542 wurden n bei der Analyse A de er miRNA E Expression nsprofile von Tu umoren auss LSL‐MYCN N;Dbh‐iCree Mäusen im Vergleicch zu nichtt‐malignen Neben‐ nieren herunterreguliert ge efunden, w was im TH H‐MYCN Mausmodell validiert werden konnte..Indiesem mTeilderA Arbeitsollteedietumormoduliere endeFunkttiondieserrmiRNA inhumaanenprimäärenNeuro oblastomen nundZelllinienunterssuchtwerdden. 4.2.1 E Expressionsanalyse evonmiR‐5 542undSurvivininhumanen nPrimärtu umoren IneinerrvorherigeenStudiew wurdensow wohldiem miRNAalsa auchdiem mRNAExpre essions‐ profile von 69 Primärtumo P oren humaaner Neurroblastompatienten aanhand von qPCR Datenggeneriert67.ImRahmendieserA ArbeitwurrdedervorrhandeneD Datensatzin nBezug aufdie Regulation ndermiR‐542Isoforrmenre‐analysiert.Ge enaueInforrmationen sowohl überdiePatienten nalsauchd dasStadium munddenVerlaufderrErkrankuungwarenb bekannt (Tab.9)). 58 Ergebnisse Tab.9:Zusammensetzungder69NeuroblastompatientendermiRNAundmRNAExpres‐ sions‐KohorteModifiziertnachAlthoffetal.67. AnzahlPatienten Geschlecht AlterbeiDiagnose Stadium MYCN Amplifikation 11q 1p36 17q Verlauf Ereignis‐freies Überleben[Tage] Gesamt‐Überleben [Tage] männlich weiblich <1Jahr ≥1Jahr 1+2 3+4 4s nein ja Wildtyp Deletion Ungleichgewicht Wildtyp Deletion Ungleichgewicht Zugewinn Rückfall Ereignis‐freies Überleben Tod Überleben Median Bereich Median Bereich 69 37 32 25 44 13 38 18 53 16 36 16 2 44 15 6 13 28 41 20 49 2283 456‐4844 2260 456‐4844 Die Expression beider miR‐542 Isoformen wurde gegen verschiedene bekannte prog‐ nostischeMarkerderKohorteaufgetragen(Abb.27a).SowohlmiR‐542‐5palsauch‐3p waren in Tumoren von Patienten, die später einen Rückfall erlitten, herunterreguliert. WeiterhinkonnteeineKorrelationzwischenniedrigermiR‐542‐5pund‐3pExpression und hohem Stadium sowie MYCN Amplifikation gezeigt werden. Neuroblastome mit hoher NTRK1 Expression wiesen erhöhte Level beider miR‐542 Isoformen auf. Mittels Kaplan‐Meier Analyse konnte gezeigt werden, dass Patienten mit Tumoren mit hoher miR‐542‐5p bzw. ‐3p Expression eine signifikant höhere Wahrscheinlichkeit eines Ereignis‐freien Überlebens aufwiesen, als diejenigen mit niedriger Expression der miRNAs(Abb.27b). 59 Erg gebnisse Abb.27 7Korrelatio onvonmiR‐542‐5pun nd‐3pExprressionmitprognostisschenMark kernund Überleb beninhum manenNeuroblastomen nExpressio onvonmiR‐5 542‐5pund‐3pinPrim märtumo‐ ren von n 69 Neurob blastompatienten anhan nd vorhand dener qPCR Daten67 (Taab.9). (a)V Vergleich der Exp pression derr Tumore vo on Patiente n mit oder ohne Rückffall, verschiiedener Stad dien, mit oderoh hneMYCNAmplifikation n(MNA)un dmithoherroderniedrrigerNTRK11Expression n.Mann‐ =p<0,05;***=p<0,01 ;***=p<0 0,001.(b)Ka aplan‐Meierr‐Analysede esEreig‐ WhitneyyU‐Test:*= nis‐freieen Überlebeens (EFS) vo on Patienten n mit Tumo oren mit hoh her oder nieedriger miR R‐542‐5p bzw.‐3p pExpression n.Log‐Rank Test. derabsoluttenmiR‐54 42‐5pund‐3pExpresssionwurd enmiRNAExpres‐ ZurBesstimmungd sionsprrofile,diem mittelsNextt‐Generatioon‐Sequenzzierungine einemweitterenvorha andenen Datensaatzerstellttwurden1100,re‐analyssiert.DieserDatensatzbestandaausProfilenvonje fünfNeeuroblastom menmitgü ünstigemun ndungünstigemVerlauf.GenauuereInform mationen über deen Krankh heitsverlauff, sowie Sttadium und d MYCN Amplifikatioon waren bekannt b (Tab.10 0). Tab.10 0: Zusamme ensetzung der Patien nten der Ne ext‐Generattion‐Sequen nzierungs‐K Kohorte ModifiziiertnachSch hulteetal.1110. günstigerrVerlauf ungünstiggerVerlauf AnzahlPatiienten 5 5 Stadium 1 4 Median 456 1045 AlterbeiDiagnose [Tage] Bereich 103‐961 496‐48277 MYCNAmplifikation nein ja Todaufgrun ndvonErkrrankung nein ja Median 3109 261 Ereignis‐freeies Überleben[[Tage] Bereich 2856‐374 45 184‐946 Median 3109 Gesamtüberleben 539 [Tage] Bereich 2856‐374 45 207‐13755 60 Erg gebnisse Sowohll miR‐542‐‐5p als aucch miR‐542 2‐3p waren n in Neuro oblastomenn mit ungünstigem VerlauffimVergleiichzuTum morenmitggünstigemV Verlaufherrunterregulliert.Ihrea absolute ExpresssionfielnieedrigeraussalsdieExxpressiond dermiRNAsdesonkoogenenmiR R‐17‐92‐ Clusters,sogarin Neuroblasstomenmittgünstigem mVerlauf((Abb.28a)..EinVergle eichder absoluttenExpresssionsleveld derbeidenmiR‐542‐IIsoformenu untereinannderführte ezudem Ergebnis,dassdieeseetwagle eichhocheexprimiertw wurden(Abb.28b). Abb.28 8:KorrelationvonmiR R‐542‐5pun nd‐3pExpressionmittgünstigem mVerlaufh humaner Neurob blastome Re‐Analyse R von v Next‐Geeneration‐Sequenzierun ngs‐Daten vvon je fünff Neuro‐ blastom men mit günstigem und ungünstigeem Verlauf1110 (Tab.10). (a) Darsteellung der absoluten Expression des miR‐17‐92‐Clu usters und beider miR R‐542‐Isoforrmen. (b)Exxpressionsle evel von 2‐5pund‐3p päquivalent. miR‐542 A Su urvivin alss potentielles Ziel fü ür miR‐5422‐3p vorhe ergesagt Da in iin silico Analysen wurde669,sollteein neKorrelattionzwisch henmiR‐54 42‐3pundS Survivininndenvorha andenen qPCR D Daten der 69 6 Neurob blastompatiienten67 un ntersucht werden. w Diie Korrelattion von niedrigger Survivin n Expressio on und hoh her miR‐54 42‐3p Exprression konnnte anhan nd eines Scatterp plots gezeigt werden n (Abb.29 a). Außerd dem war die d Surviviin Expression von Tumoreenmit hoh hermiR‐54 42‐3p Expreession sign nifikant nie edrigerals von Tumo oren mit niedriggerExpresssiondermiiRNA(Abb..29b).Weiiterhinbesttandeinessignifikante eKorre‐ lation zzwischenh hoherSurviivinExpresssionundsschlechtere emÜberlebbenderPa atienten, analysiertanhand deinerKaplan‐Meier‐A Analyse(A Abb.29c). 61 Erg gebnisse 9: Korrelatiion von miR R‐542‐3p u und Surviviin in humanen Neuro oblastomen Analyse Abb.29 derExp pressionvon nmiR‐542‐3 3pundSurvvivinmRNA inhumanen nNeuroblasstomenanhandvor‐ b.9). (a/b) Korrelation n von hohe er miR‐542‐‐3p Expression mit handeneer qPCR Daaten67 (Tab niedrigeer Survivin Expression anhand vo n Scatter‐ und u Boxplot. Student‘s s t‐Test. (c) Kaplan‐ Meier‐A Analysedes Ereignis‐fre eiesÜberleb bensvonNeeuroblastom mpatientenm mithoheru undnied‐ rigerSurvivinExpreession.Log‐RankTest. 4.2.2 F Funktionv vonmiR‐5 542‐3pExp pressionin nZelllinien n Dieim VergleichzzumKontrrollgewebe niedrigeE Expression vonmiR‐5542‐5pund d‐3pim LSL‐MY YCN;Dbh‐iCre Modell und in hum manen Prim märtumore en ließ verrmuten, dass diese miRNA einetumo orsuppressiveFunktioonimNeuroblastom haben könnnte.Zurw weiteren Überprrüfung diesser Hypoth hese wurdeen verschieedene in vitro v Versuuche in etablierten humaneenNeurobllastomzelllliniendurch hgeführt. Eine fü ür keine miiRNA kodie erende Neggativkontro oll‐miRNA oder eine dder beiden n Isofor‐ mendeermiR‐542wurdenin ndenhumaanenNeuro oblastomze elllinienIM MR‐32,SHEP P,SK‐N‐ BEund dWACIIexprimiert.D DierelativeeZellviabiliitätwurde zuverschiiedenenZe eitpunk‐ tenmitttelsdesMTTAssayg gemessen, aufden24 4Std.WerttderNegattivkontroll‐miRNA normallisiert und gegen die Zeit aufgeetragen (Ab bb.30). Alle analysierrten Zelllin nien, die mitderrNegativko ontroll‐miR RNAtransfi ziertwurden,wiesen SteigerunggenderZelllviabili‐ tät übeer die Zeit auf. Die Ex xpression vvon miR‐542‐5p führrte zu Zellllinien‐spezzifischen Reaktio onen. Währrend IMR‐32, SK‐N‐B BE und WA ACII keine Reduktionn der Zellv viabilität zeigten n,führtedieeExpressio oninSHEP Pzueiner signifikantenVerringgerungder Zellvia‐ bilitätü überdieZeeit.DieExp pressiondeermiR‐542 2‐3phingeg genverringgertedieV Viabilität allerun ntersuchten nZelllinien nimVergleiichzurNeg gativkontro oll‐miRNAssignifikant. 62 Erg gebnisse Abb.30 0: Reduktiion der Viabilität V von hum manen Neu uroblastom mzelllinien mittels miR‐54 42‐3pExpre essionTransienteTranssfektionderrhumanenN Neuroblastoomzelllinien nIMR‐32, SHEP,SK‐N‐BEund dWACIImittNegativkon ntroll‐miRN NA(NK),miR R‐542‐5poddermiR‐542 2‐3pund TTAssay.An ngegebenep p‐WertefürrdieSignifik kanzvon MessunggderZellviabilitätanhanddesMT miR‐542 2‐5poder‐3 3pgegenNK Kberechnet anhandeineesPermutattionstests. Zur gen naueren Untersuchun U ng der Meechanismen n, die der Reduktionn der Zellv viabilität durch m miR‐542‐3p p Expression zugrun de liegen, wurden weitere Anallysen 96Sttd. nach Transfeektion durrchgeführt, da zu dieesem Zeitp punkt die Viabilität aller analy ysierten Zellen aam stärkstten verring gert werden n konnte. Anhand A vo on makroskkopischen Aufnah‐ menmiiR‐542‐3pexprimiere enderZelle nkonnteim mVergleich hzurNegattivkontroll‐miRNA exprimierendenZ ZelleneinedeutlichveerringerteK Konfluenzfestgestellttwerden.A Auchbei Expresssion von miR‐542‐5p m p zeigten N Neuroblasto omzellen eine e geringgere Konflu uenz als nachTrransfektion nmitNegattivkontroll ‐miRNA,jeedochward derEffekthhiernichtssodeut‐ lichwieemitmiR‐5 542‐3p(Ab bb.31). 63 Erg gebnisse Abb.31 1: Reduzierrte Konflue enz von N Neuroblasto omzellen nach n miR‐5 542‐3p Exp pression Mikrosk kopischeAufnahmenvo onNeuroblaastomzellen 96Std.nach hTransfekttionmitNeg gativkon‐ troll‐miR miR‐542‐5podermiR‐5 542‐3p.Maß ßstabsbalken n=200µm. RNA(NK),m Weiterh hinwurde mittelsDu urchflusszyttometried derZellzyklus96Std. nachTransfektion untersu ucht. Währrend die Ex xpression vvon miR‐54 42‐3p im Vergleich V zzur Expresssion der Negativvkontroll‐m miRNA zu einer sign nifikant gessteigerten Fraktion vvon Zellen n in der subG1‐Phase,dem mnachmutm maßlichap poptotischeenZellen,fführte,bliebbdieserEfffektbei m p aus (Datten nicht gezeigt). g Die prozenttualen Anteile der Expresssion von miR‐542‐5p Zellen in den Zellzyklus‐Phasen Postm mitose (G1 1), Synthese (S) und Prämitose/Mitose (G2/M))wurdenaalsBalkend dargestellt (Abb.32). DieExpresssionvonm miR‐542‐5p pführte in keiner der anaalysierten Zelllinien Z zzu einer sig gnifikanten n Veränderuung der einzelnen Zellzyk klus‐Fraktio onen. Dahin ngegen wu urdein alleen mitmiR‐542‐3p trransfizierte en Zellli‐ nienein nesignifikaanteSteigerrungderFrraktionvon nZelleninderS‐Phassedetektierrt,diein IMR‐32 2,SHEPund dWACIIZe ellenmitei nersignifik kantenRed duktionderrG1‐Phaseeinher‐ ging. Eine tenden nziell verringerte G1‐‐Phase kon nnte auch in SK‐N‐BE E Zellen vermutet werden n. 64 Erg gebnisse Abb.32 2: Verschieb bung des Zellzyklus Z d durch gestteigerte S‐P Phase in Neeuroblastomzellen nach m miR‐542‐3p Expression n Analyse d des Zellzyklu us 96Std. nach n Transfe fektion von Negativ‐ kontrolll‐miRNA (N NK), miR‐54 42‐5p oder ‐3p. Anteil der Zellen n in den Zeellzyklus‐Ph hasen G1 (Postmitose), S (Sy ynthese) un nd G2/M (P Prämitose und u Mitose) als Prozennt‐Balken. Student‘s S t‐Test:**=p<0,05;**=p<0,01. DierelaativeAnzah hlapoptotischerZelleenwurde9 96Std.nach hExpressioonbeiderm miR‐542 Isoform menimVerrgleichzurNegativkon ntroll‐miRN NAanhand ddesELISA ACelldeath hAssays untersu ucht.DieBehandlungmitmiR‐5 542‐3pinduziertesignifikantdeenAnteilap poptoti‐ scher Z Zellen, wäh hrend mit miR‐542‐5 5p kein Efffekt detek ktiert wurdde (Abb.33 3a). Die Zellproliferation, dieanhand dderELISA A‐basierten nBrdUInkorporationngemessen nwurde, 96Std.nach hmiR‐542‐‐3pExpres sioninalleenanalysiertenZelllinniensignifikantab, nahm9 währen nd ein deraartiger Effe ekt bei Exp pression von miR‐54 42‐5p nur iin SK‐N‐BE E Zellen erreichtwurde(A Abb.33b). Abb.33 3:Induktion nvonApop ptoseundR Reduktionv vonProlifer rationinNeeuroblasto omzellen nach m miR‐542‐3p Expression n Analyse 9 96Std. nach h Transfektion von Neggativkontrolll‐miRNA (NK), m miR‐542‐5p oder ‐3p. (a a) Balkendi agramm deer Zahl apop ptotischer ZZellen relativ v zu NK, gemesseenmittelsC CelldeathEL LISA.(c)Ballkendiagram mmderZahlproliferierrenderZelle enrelativ zuNK,ggemessenan nhandvonE ELISA‐basierrterBrdUIn nkorporation n.Student‘st‐Test:*=p<0,05; **=p<0,01;***=p p<0,001. 65 Ergebnisse 4.2.3 RegulationderExpressionvonSurvivindurchmiR‐542‐3p Die 3’UTR von Survivin weist eine Bindestelle für miR‐542‐3p auf, die schon in der Adenokarzinomzelllinie A549 und der Brustkrebszelllinie MCF7 validiert wurde69. In diesemTeilderArbeitsolltedieRegulationvonSurvivindurchmiR‐542‐3pinhumanen Neuroblastomzellen validiert werden. Dazu wurden Negativkontroll‐miRNA, miR‐542‐5p oder miR‐542‐3p transient in IMR‐32, SHEP, SK‐N‐BE und WACII Zellen exprimiertunddieExpressionvonSurvivin96Std.nachTransfektionuntersucht. DieSurvivinmRNAExpressionvonmiR‐542‐5pbzw.‐3pexprimierendenZellenwurde relativ zu Negativkontroll‐miRNA exprimierenden aufgetragen. Die Expression von miR‐542‐3p führte zu einer signifikanten Reduktion von Survivin mRNA in IMR‐32, SHEPundWACIIZellenundinSK‐N‐BEZellenkonnteeineTendenzzurHerunterregula‐ tion von Survivin mRNA gezeigt werden. Im Gegensatz dazu zeigte die Expression von miR‐542‐5pkeinenEffektaufdieSurvivinExpression(Abb.34a).InWesternBlotAnaly‐ sen wurden zum einen das Protein Survivin und zum anderen der Komplexpartner AURKB detektiert. Anhand der Ladungskontrolle Aktin konnte eine ungleichmäßige Beladung der Gele ausgeschlossen werden. Sowohl dieProteinexpressionvonSurvivin als auch die von AURKB wurden nach Expression von miR‐542‐3p im Vergleich zur Negativkontroll‐miRNA deutlich herunterreguliert, während dieser Effekt mit miR‐542‐5p nicht erreicht wurde (Abb.34b). In Immunfluoreszenzfärbungen wurde DAPIeingesetzt,umdieZellkernenachBehandlungmitderNegativkontrolleodermiR‐ 542‐3p anzufärben. Die Konfluenz der Zellen nach Transfektion mit miR‐542‐3p war geringer als nach Behandlung mit der Negativkontroll‐miRNA, ein Effekt, der schon in vorherigen Versuchen (Abb.31) gezeigt werden konnte. Sowohl Survivin als auch AURKBwurdeinallenmitNegativkontroll‐miRNAbehandeltenZellendetektiert.Nach Expression von miR‐542‐3p dagegen war die Expression beider Proteine deutlich ver‐ ringert(Abb.34c). 66 Erg gebnisse Abb.34 4: Herunterrregulation n von Surviivin in Neu uroblastom mzellen nacch miR‐542 2‐3p Ex‐ pressio on (a) Survivin mRNA nach n miR‐54 42‐5p und ‐3p Expresssion relativ zu Negativk kontroll‐ miRNA (NK), detek ktiert mitte els qPCR un nd normalisiert gegen GAPDH Exxpression. Student‘s S 01. (b) Wesstern Blot Analyse A der Proteine Suurvivin und d AURKB t‐Test: **=p<0,05; **=p<0,0 nach Exxpression vo on NK, miR‐‐542‐5p odeer ‐3p. Aktin n als Ladungskontrolle . (c) Immun nfluores‐ zenz‐Färbung von Survivin (Alexa‐Fluor‐4 488 ‐ grün) und AURK KB (Alexa‐Fluuor‐568 ‐ rot) r nach 3pExpressio on.Zellkern neimselben nFeldmitte elsDAPI(blaau)angefärrbt.Maß‐ NKoderrmiR‐542‐3 stabsballken=50µm m. 4.2.4 D DirekteBiindungvon nmiR‐542 2‐3panSurvivin3‘UT TR Anhand dvonLuzifferaseRepo orterAnalyysensollte untersuchttwerden,oobdieHeru unterre‐ gulation nvonSurvivinnachd derExpresssionvonmiiR‐542‐3paufeinedirrekteBindungvon miR‐54 42‐3pandie3’UTRvo onSurvivin zurückgefführtwerde enkonnte. BeidePsi‐Check2‐ Vektoreen enthieltten die 3’U UTR von Su urvivin, jedoch war diese auf einem derr beiden Vektoreensomutieert,dassdievorhergeesagteBind destellederrmiR‐542‐ 3pzerstörttvorlag. HEK‐29 93undSHE EPZellenw wurdenmit jeweilsein nemderbeidenVektoorenunden ntweder mit derr Negativk kontroll‐miRNA oder miR‐542‐3p kotranssfiziert. Naach 48Std. wurde sowohlldieAktivittätderSeefeder‐alsaauchdiedeerGlühwürrmchen‐Luzziferasegemessen. Die Akttivität der Glühwürm mchen‐Luzifferasewurrde aufdie der Seefedder‐Luziferrase, die als Traansfektionskontrolle exprimiert e t wurde, no ormalisiertt. Die norm mierte Glüh hwürm‐ 67 Erg gebnisse chen‐Lu uziferase‐A Aktivität,diiebeiExprressionvon nmiR‐542‐‐3pberechhnetwurde e,wurde relativ zur Aktiviität nach Negativkon ntroll‐miRN NA Transfe ektion aufg fgetragen (relative ( Reporteergen‐Aktiv vität).Sow wohlinHEK K‐293alsaauchinSHE EPZellenw wurdedie relative Reporteergen‐Aktiv vität bei Kotransfekt K tion mit deem Wildtyp p 3’UTR voon Survivin n signifi‐ kantreeduziert.Im mGegensattzdazu,kon nntedieserEffektbeiderKotraansfektion mitder mutierttenBindesttellenichtg gezeigtwerrden(Abb.35).DiediirekteBinddungvonm miR‐542‐ 3pand die3’UTRvonSurvivin nkonntedaamitvalidieertwerden. Abb.35 5:Direkte Bindung B vo onmiR‐542 2‐3pan Surrvivin 3‘UTRKotransfeektionvonH HEK‐293 undSHE EPZellenen ntwedermittNegativkon ntroll‐miRN NA(NK)ode ermiR‐542‐33pundeine emWild‐ typ‐ odeer mutierteen Survivin 3’UTR trageenden Luzifferase Repo orter. Messuung der Glü ühwürm‐ chen‐Lu uziferase‐Ak ktivitätnach h48Std.und dNormalisierunggegen nSeefeder‐LLuziferase‐A Aktivität. Studentt‘st‐Test:**=p<0,01;****=p<0,0 001. 4.2.5 F Funktionv vonSurviv vinHeruntterregulattioninZellllinien EinedirrekteBindungvonmiR‐542‐3p andas3’U UTRvonSurvivinund diedamitverbun‐ dene H Herunterreggulation vo on Survivin n mRNA un nd Protein konnte gezzeigt werden. Nun sollte u untersucht werden, ob o eine Heerunterregulation von Survivinn in den hu umanen Neurob blastomzellllinienIMR R‐32,SHEP,,SK‐N‐BEu undWACIIphänotypiischeVerän nderun‐ gen mit sich brin ngen würde, die deneen der miR R‐542‐3p Expression E n entspräch hen. Die Zellenw wurdenentwedermitteinerNeggativkontro oll‐siRNAodereinersspezifischensiRNA gegen SSurvivin (siSurvivin) transient ttransfiziertt.Zur Überrprüfungdeer Effektiv vität von siSurvivvin wurde die Expre ession des Proteins 48Std. nach der Traansfektion mittels WesterrnBlotanallysiert,wob beiGAPDH HalsLadun ngskontrollediente.SoowohldieP Protein‐ expresssion von Su urvivin, alss auch die des Komplexpartners AURKB kkonnte mitttels der siRNAh herunterreguliertwerrden(Abb. 36). 68 Erg gebnisse Abb.36 6: Spezifiscche Redukttion von Su urvivin Prrotein mitttels siRNA W Western Blo ot Analyse der Expression von Survivin und u nter AURKB 48Std. naach transien ktion von Neuroblasto om‐ Transfek zelllinien mit Neegativ‐kontrroll‐ siRNA (NK) oder spezifisccher vin(siSurviv vin). siRNAggegenSurviv GAPDHalsLadungsskontrolle. NA bzw. siS Survivin Die Viaabilität derr Zellen nach Transfeektion mit Negativkontroll‐siRN wurde mittels MT TT Assay untersuchtt und gegeen die Zeitt aufgetraggen. Währe end mit Negativvkontroll‐siiRNA transsfizierte Zeellen einen n Anstieg ihrer Viabillität über die Zeit aufwiessen,zeigten ndiemitssiSurvivinb behandelteenZelleneiineRedukttion.DerVergleich beider Datensätzeebeschrieb bdenVerlaaufderGraapheninnerhalbderaanalysierte enZellli‐ ntverschied den(Abb.3 37). nienalsssignifikan Abb.37 7: Reduktio on der Via abilität hu umaner Ne euroblastom mzelllinien durch He erunter‐ regulation von Su urvivin MTT T Assay nach h transienteer Transfektion mit Neegativkontro oll‐siRNA (NK)od dersiRNAgeegenSurviviin(siSurviviin).Angegeb benep‐WertefürdieSiignifikanzvo onsiSur‐ vivingegenNKbereechnetanha andeinesPeermutationstestsPermu utationstest.. Die Traansfektion von siSurv vivin führtee im Vergleich zur Trransfektionn von Nega ativkon‐ troll‐siR RNAzuein nemsignifik kantenAnsstiegdersu ubG1Phase,dieaufeeineIndukttionvon Apopto osehinwiess(Datennichtgezeigtt).Weiterh hinkonnte beiVergleiichderZelllzyklus‐ Phasen n G1, S und d G2/M (A Abb.38) soowohl in IM MR‐32 und d SHEP alss auch in SK‐N‐BE S 69 Erg gebnisse Zellen eeine signifiikante Steigerung derr S‐Phase mit m einherg gehender R Reduktion der G1‐ Phase d detektiert werden. WACII W Zelleen zeigten n eine Tendenz zur eerhöhten S‐Phase. S SHEPu undWACIIZ Zellenreag giertenauß ßerdemmitteinemsign nifikantenA AnstiegderG2/M‐ Phase. Abb.38 8: Verschie ebung dess Zellzyklu us durch gesteigertte S‐ bzw w. G2/M‐Ph hase in Neurob blastomzellen nach Herunterreggulation vo on Survivin Fraktionenn von Zellen n in den verschieedenen Phaasen des Zellzyklus. Z A Analyse mittels Durchflusszytom metrie 96Sttd. nach transien nter Transffektion von n Negativkkontroll‐siRNA (NK) oder siRN NA gegen Survivin (siSurvivin).Studen nt’st‐Test:* =p<0,05; ***=p<0,0 001. Der Efffekt auf diie Zellproliiferation n nach siSurv vivin im Vergleich zuu Negativk kontroll‐ siRNA wure anh hand der ELISA‐bassierten Brd dU‐Inkorpo oration errmittelt. Alle vier agierten aauf die Herunterreg H gulation vvon Surviv vin mit analysierten Zellllinien rea kanterRedu uktionderZellproliferration(Abb b.39). signifik bb.39: Re eduktion der Proliferation Ab hu umaner Ne euroblastom mzelllinien n durch He erunterreg gulation vo on Survivin n BrdU‐ In nkorporation n von Zeellen 96Std d. nach Trransfektion mit Neegativkontro oll‐siRNA (N NK)odersiR RNAgegenS urvivin(siSurvivin). Sttudent‘s t‐T Test: *=p<<0,05; **=p<0,01; ****=p<0,001. 70 Erg gebnisse 4.2.6 P Partielle Wiederhe erstellung des Phän notyps de er miR‐54 42‐3p Expression m mittelsHo ochregulattionvonSu urvivinoh hnemiRNA A‐Bindesteelle DieHerrunterregu ulationvon Survivinm mittelssiRN NAführteindenanallysiertenhu umanen Neurob blastomzellllinien zu erhöhter Apoptose und verringerter Zellviabilittät und Proliferration.DiesserPhänoty ypähnelte demderE ExpressionvonmiR‐5 42‐3p,der mitder Herunterregulatio on von Surrvivin einh herging. Es sollte nun n untersuchht werden, ob ein menhang zwischen z dem Phänottyp der miR‐542‐3p Expression E n und der Survivin S Zusamm Herunterregulatio on bestand d. Dazu w wurde Surv vivin cDNA A, die keinne Bindesttelle für miR‐54 42‐3penthiielt,stabilm mittelseineesVektorsy ystemsinS SHEPZelleeneingebra achtund Einzelzzellklone seelektiert (SHEP‐Survvivin). Ein Negativkon ntroll‐Klonn, der das gleiche Konstru ukt,jedoch hmiteiner cDNAfürG GFPstattSSurvivintru ug,wurdeeebenfallsge eneriert (SHEP‐GFP). Die Expression n beider cD DNAs konn nte durch Behandlunng mit Tetrrazyklin induzieert werden n. Mittels Western W Bloot wurde die d Induktion der Suurvivin Exp pression 24Std. nachTetraazyklin‐Beh handlungin nSHEP‐SurvivinimV Vergleichzuumnicht‐in nduzier‐ ten und zum SH HEP‐GFP‐Kllon ermitttelt, wobei GAPDH als a Ladunggskontrolle e diente (Abb.40).Aufgrun ndderhoh henIndukti onvonSurrvivinkonn ntenurmitteinemFün nftelder Gesamttproteinmeenge gearb beitet werrden, die in vorherigen Versuuchen verrwendet wurde. Innicht‐in nduzierten SHEP‐Zell enwardesshalbimV Vergleichzuuvorherige enWes‐ ternBlo ots(Abb.34undAbb.36)nureiinegeringeSurvivinE Expression detektierbar. Abb.40 0: Survivin n Überex xpression in SHEP‐ Survivin n nach Ind duktion mitt Tetrazykllin Western n Blot An nalyse der Survivin Ex xpression 2 24Std. nach h Tetrazyk klin‐ (TET) bzw. Lösu ungsmittel‐ (EtOH) Be‐ handlun ng von stab bil‐transfizie erten SHEP P‐Zellen mit einem Tetrazyklin n‐ induzierb baren Vekttorkonstrukt P (SHEP‐GFP P) oder Survvivin cDNA ohne 3’UTR R mit GFP (SHEP‐SSurvivin). Die gen nerierten Zellklone Z SH HEP‐GFP u und SHEP‐SSurvivin wu urden überr 96Std. en ntweder mit Tettrazyklin oder o dem Lösungsmit L ttel Ethano ol behandellt und gleicchzeitig mit Nega‐ tivkonttroll‐miRNA A oder mitt miR‐542‐ 3p transien nt transfiziiert. Mittells des MTT T Assays wurdedieZellviab bilitätallerrAnsätzeb estimmt.D DieExpressionvonmiiR‐542‐3pg gingmit einer ssignifikant reduzierte en Zellviab bilität des SHEP‐GFP P‐Klons, unnabhängig ob mit Tetrazyyklin behan ndelt oder nicht, und d des unbehandelten SHEP‐Survvivin‐Klons einher. DiegleiichzeitigeE ExpressionderSurvivvincDNAoh hneBindestellefürdieemiRNAundmiR‐ 71 Erg gebnisse 542‐3p p führte zw war im Verg gleich zur Negativkon ntroll‐miRN NA zu eineer tendenziell ver‐ ringerteenZellviab bilität,jedocchwardiesserEffektn nichtsignifiikant(Abb..41). 4.2.7 Abb.4 41: Partiellle Wiederh her‐stel‐ lung des miR‐5 542‐3p Phänotyps mittells Überexprression von Survi‐ vin oh hne miR‐5 542‐3p Bin ndestelle MTT Assay A 96 Sttd. nach Ex xpression von Negativkonntroll‐miRNA A (NK) m p und gleich hzeitiger oder miR‐542‐3p Tetrazzyklin‐ (TET T) bzw. Lösu ungsmit‐ tel‐ (E EtOH) Behaandlung von n SHEP‐ Zellen mit einem induzierbaren Vek‐ HEP‐GFP) tor‐konstrukt miit GFP (SH SurvivincDN NAohnemiR R‐542‐3p oderS Bindesstelle (SH HEP‐Survivin n). Stu‐ dent‘s t‐Tesst: *=p<0,05; p<0,001. ***=p M MiR‐542‐3 3p belad dene Naanopartike el zur miRNA‐Beehandlung g von N Neuroblasstomzellen ninvitrou undinvivo Zusätzlich zum Nachweis de er tumorsu uppressiven Funktion n von miR‐‐542‐3p in n Neuro‐ blastom mzelllinien in vitro, so ollte diese auch in Neuroblasto omzellen unntersucht werden, w diealsX XenograftsinMäusen ninvivowu uchsen.DadieInjektio onvonmiR RNAindieM Mauszu einemssofortigen Abbauderselbenführrenwürde,wurdenC Calcium‐Phoosphat‐Nan noparti‐ kel als Transporter verwendet84,85. D Die versch hieden bela adenen Naanopartikell waren sphärissch und haatten einen mittleren Durchmessser von weniger w als 200nm. Ih hr Zeta‐ Potentiial betrug +37mV. Anhand eelektronenm mikroskopiischer Auffnahmen konnten k keine sstrukturelleen Unterscchiede zwisschen Neg gativkontroll‐miRNA‐ und miR‐542‐3p‐ geladen nenNanopaartikelnfesstgestelltw werden(Abb.42). Abb.42:Str A rukturvonmiRNA‐beladenen Nanopartik N kelnElektroonenmikrosk kopische Aufnahmen A von Negaativkontroll‐miRNA‐ (NK)undm ( iR‐542‐3p‐bbeladenenN Nanopar‐ tikeln.Graph t hikerstelltvvonDianaK Kozlova. 72 Ergebnisse Zur Untersuchung der Funktionalität der Nanopartikel in vitro wurden WACII Zellen ausgewählt,dadieseindenvorherigentransientenTransfektionenamstärkstenaufdie Expression der miR‐542‐3p reagiert hatten. Die Zellen wurden mit Negativkontroll‐ miRNA‐ oder miR‐542‐3p‐beladenen Nanopartikeln behandelt und der Effekt auf die ZelleninverschiedenenAssaysuntersucht.DierelativeZellviabilität,gemessenmittels MTTAssay,wurdeanverschiedenenZeitpunktendetektiert,aufden24Std.Wertunbe‐ handelterZellennormalisiertundgegendieZeitaufgetragen.DieBehandlungmitNega‐ tivkontroll‐miRNA beladenen Nanopartikeln führte im Vergleich zu unbehandelten Zellen tendenziell zu einer Reduktion der Zellviabilität über die Zeit, die jedoch nicht signifikantwar.WährenddessenverringertedieBehandlungmitmiR‐542‐3p‐beladenen NanopartikelndieViabilitätderZellensignifikant,sowohlimVergleichzuunbehandel‐ tenalsauchzumitNegativkontroll‐miRNAbeladenenNanopartikelnbehandeltenZellen (Abb.43a). Der Anteil apoptotischer Zellen wurde 96Std. nach Behandlung mit den verschieden beladenen Nanopartikeln anhand des ELISA Cell death Assay untersucht. WieschonbeiderAnalysederZellviabilitätwurdeauchindiesemAssayeintendenziel‐ ler Effekt der mit Negativkontroll‐miRNA beladenen Nanopartikel beobachtet, der jedoch nicht das Signifikanzniveau erreichte. Dagegen reagierten die Zellen bei Be‐ handlung mit miR‐542‐3p beladenen Nanopartikeln mit einer signifikanten Induktion vonApoptose,sowohlimVergleichzuunbehandeltenZellenalsauchzumitNegativkon‐ troll‐miRNA behandelten Zellen (Abb.43b). Die Abnahme der BrdU Inkorporation bei mitmiR‐542‐3p‐beladenenNanopartikelnwarimVergleichzuallenKontrollensignifi‐ kantvermindert.AllerdingszeigtendiemitNegativkontroll‐miRNAbeladenenNanopar‐ tikelbereitseinesignifikantverminderteBrdUInkorporationimVergleichzuunbehan‐ delten Zellen (Abb.43c). Die Funktionalität der miR‐542‐3p Expression nach Behandlung mit miR‐542‐3p‐beladenen Nanopartikeln in WACII Zellen wurde anhand der Expression des Zielproteins Survivin analysiert. Eine Western Blot Analyse zeigte, dass die Behandlung mit miR‐542‐3p‐beladenen Nanopartikeln im Vergleich zu unbe‐ handelten und mit Negativkontroll‐miRNA behandelten Zellen zu einer deutlichen HerunterregulationderSurvivinProteinexpressionführte(Abb.43d).DieSpezifitätder miR‐542‐3p Expression durch Behandlung mit miR‐542‐3p‐beladenen Nanopartikeln invitrokonntesomitbewiesenwerden. 73 Erg gebnisse 3:Reduktio onderZellv viabilitätun ndder Pro oliferation, Induktion vonApopttoseund Abb.43 Herunte erregulatio on von Surrvivin mitteels miR‐542‐3p‐belad denen Nano opartikel in n WACII Zellen in vitro Beehandlung mit m Negativvkontroll‐miiRNA‐ (NK) oder miR‐‐542‐3p‐ be eladenen Nanopartikeln.(a) Zellviabilitä ätrelativzu u24Std.Weertderunbe ehandeltenK Kontrolle,gemessen A Signiffikanz berecchnet anhan nd eines Pe ermutations test. (b) Ba alkendia‐ anhand des MTT Assay. nbehandelten n Kontrollee relativen Anteils A apop ptotischer ZZellen nach 96 Std., gramm des zur un desELISAC Celldeath.Sttudent‘st‐Teest.(c)Balk kendiagramm mdeszuru unbehan‐ gemesseenanhandd delten K Kontrolle reelativen Antteil proliferrierender Zeellen nach 96 9 Std., gem messen anh hand von ELISA‐b basierter BrdU Inkorpo oration. Stud dent‘s t‐Tesst. (c) Westtern Blot Annalyse des Proteins Survivin nnach48Sttd.*=p<0,0 05;**=p<0 0,01;***=p p<0,001. ZurUnttersuchunggdesEffekttsdersyntthetisierten nNanoparttikelaufWA ACIIZellen n,dieals Xenografts in nu//nu Mäusen wuchsen n, wurden Xenograft‐‐tragende M Mäuse sow wohl mit Negativvkontroll‐m miRNA‐ als auch mit miR‐542‐3 3p‐beladen nen Nanopaartikeln be ehandelt und diee Leber so owie die Tumore T anaalysiert. Die Expresssion von m miR‐542‐3p p wurde mittels qPCR gem messen un nd gegen d die Expression von RNU6B R noormalisiert.. In mit miR‐54 42‐3p‐behaandelter Le eber konntte im Verg gleich zu mit Negatiivkontroll‐miRNA‐ behand deltereine 200‐fache erhöhteExp pressiondeermiRNAd detektiertw werden(Sttudent‘s t‐Test: p=0,006; Daten nich ht gezeigt) . Die miR‐542‐3p Expression dder Tumore e wurde relativ zu der Exp pression eines der m mit Negativ vkontroll‐m miRNA behaandelten aufgetra‐ m Mittel waar die miR‐‐542‐3p Exxpression der d mit die eser miRNA A behandellten Tu‐ gen. Im more iim Vergleich zu mitt Negativkoontroll‐miR RNA behan ndelten Tuumoren 25 5,8‐fach, signifik kanterhöhtt(Abb.44a).DieFunkktionalität derindenTumoreneexprimierttenmiR‐ 542‐3p pwurdeanh handdesE EinflussesaaufdieExpressiondessTargetsSSurvivingemessen. In Western Blot Analysen A konnte k einee reduziertte Expression des Suurvivin Pro oteins in 74 Erg gebnisse mit miR‐542‐3p‐b beladenen Nanopartiikeln behandelten Tu umoren geezeigt werd den. Als PDH(Abb. 44b). LadunggskontrolledienteGAP 4: Expressiion von miR‐542‐3p m p und Herrunterregullation von n Survivin mittels Abb.44 miRNA‐‐beladenen n Nanoparrtikeln in WACII‐Xe enografts invivo (a) a) Expressiion von miR‐542 2‐3p in mitt Negativkontroll‐miRN NA‐ (NK) od der miR‐542‐3p‐beladeenen Nanop partikeln behandeeltenNeuro oblastom‐Xenograftsan nhandvonqPCR‐Daten.Student‘st‐‐Test:p=0,,008.(b) Western n Blot Analy yse von Surrvivin Proteiin in den glleichen Tum moren. GAPD DH als Ladu ungskon‐ trolle. Die tum morsuppressive Wirk kung der m miR‐542‐3p p Expression wurde anhand hiistologi‐ scher SSchnitte deer mit Neg gativkontrooll‐miRNA und der mit m miR‐5442‐3p‐beha andelten Tumoreen analysiert. Eine Hämatoxyl H lin‐Eosin‐Färbung offfenbarte ddie Neurob blastom‐ 1, die spezifisschen klein‐, blau‐ und rund dzelligen Tumore T d den X Xenograft bildeten b (Abb.45a).Weiterhinwurde eMib‐1,ein nMarkerffüraktiv‐prroliferierenndeZellen, aufden 3p‐beladen nenNanopa artikel‐behaandeltenTumoren Schnittenangefärbt.DiemitmiR‐542‐3 hweniger aktiv‐proliiferierendeeZellenauffalsdiemiitNegativk kontroll‐ wiesen wesentlich ung von geespaltener Caspase 3 wurde de er Anteil miRNA behandeltten. Mittelss der Färbu optotischen nZellengeffärbt.InmiiR‐542‐3pbehandelte enTumore nwurdeeiinhöhe‐ derapo rer Antteil apopto otischer Ze ellen detekktiert, als in Tumore en, die mitt Negativk kontroll‐ miRNA behandeltt wurden. Der Anteil der Mib‐1 1 bzw. gesp paltene Caaspase 3 ge efärbten Zellen w wurde relaativ zur Ge esamtzellzaahl von drrei repräse entativen A Ausschnitte en jedes Tumorssausgezäh hlt.DieMitttelwertedeermitmiR‐‐542‐3p‐be ehandelten nTumorenwurden relativ zudenenv vonTumore en,diemit tNegativko ontroll‐miR RNAbehanddeltwurden n,aufge‐ tragen. Die Zellprroliferation n der miR‐‐542‐3p‐beehandelten Tumore w war relativ zu den Negativvkontroll‐m miRNA‐behandelten ssignifikant um 85% % reduziertt (Abb.45b). Der Anteilaapoptotisch herZellenw warindiessenTumoreensignifika antumdennFaktor2,9 9erhöht (Abb.45c). 75 Erg gebnisse 5: Hemmun ng der Proliferation u und Induktiion von Ap poptose in m miR‐542‐3p‐expri‐ Abb.45 mierendenNeuroblastom‐Xe enografts(aa)JezweirrepräsentatiiveBildervo vonHämatox xilinund Eosingeefärbtenund dMib‐1(ak ktiv‐proliferiierendeZelllen)sowieg gespalteneCCaspase3(a apoptoti‐ scheZelllen)gefärbttenSchnitte endermitN Negativkontroll‐miRNA‐‐(NK)undm miR‐542‐3p p‐belade‐ nen Nan nopartikeln‐‐behandelte en Neuroblaastom‐Xenog grafts. (b/c)) Mittlere P Proliferation n anhand vonMib b‐1undApo optoseanhan ndvongesp paltenerCasspase3inm miR‐542‐3p‐‐behandeltenTumo‐ renrelaativzumitN Negativkontrroll‐miRNA (NK)behan ndelten.KalkulationduurchAuszählungvon dreirep präsentativeenAusschnitttenrelativ zurGesamttzellzahljed desXenograffts.Student‘st‐Test: *=p<0 0,05. 76 gebnisse Erg 4.3 D Dietumo orsupprressiveF Funktio onvonm miR‐137 7inhum manen N Neurobla astomen nundZe elllinien n 4.3.1 K KDM1Aalspotentie ellesZieleiinerNeuro oblastomtherapie KDM1A A ist in un ndifferenzie erten Neurroblastomeen hochreguliert. In w weiteren Untersu‐ U chungen konnte außerdem a eine Hoch regulation in aggresssiven Meduulloblastom men und Retinob blastomen gezeigt we erden (Proof. Dr. Johaannes Schu ulte, persönnliche Mittteilung). DieReggulationdieesespotenttielltherap peutischrellevantenTa argetswurddedeshalb bauchin TumoreendesLSL‐‐MYCN;Dbh h‐iCreMaussmodellsu untersucht. Tumoree zeigten im Vergleicch zu nichtt‐malignen n Nebennieren eine s ignifikant erhöhte ExpresssionvonK Kdm1a.Zur Validierun ngdiesesR Resultatsw wurdedieE Expression auchin sieben TH‐MYCN Tumoren untersuch ht, die ebenfalls eine e signifikannt erhöhte Kdm1a Expresssionaufwieesen(Abb.46). R vvon Kdm1a a mRNA Abb.46: Regulation in LSL‐MY YCN;Dbh‐iCrre und TH‐MYCN T Tumoren Boxplot dder Kdm1a a mRNA Expression in LSL‐M MYCN;Dbh‐iC Cre und TumorenrellativzurExpression TH‐MYCNT nicht‐malig gner Nebennnieren (NN). Stu‐ dent’st‐Tesst:***=p< 0,001. Aufgrun nd der pottentiell onk kogenen Akktivität von n KDM1A wurden w in in silico Analysen A nach m miRNAs gessucht, für die 3’UTR von humaanem KDM M1A eine ddirekte Bin ndestelle aufweissen. Für miR‐137 m wurde w ein p passende Bindestelle e (7mer‐m m8) an 3’U UTR von KDM1A A vorhergessagt (www w.Targetscaan.org). Inn nerhalb de er 327bp llangen 3’U UTR von KDM1A AlagdieseB Bindestelle ezwischen denPositio onen65un nd71(Abb.47). 77 Erg gebnisse 7: 3’UTR vo on KDM1A mit Bindeestelle von n miR‐137 Schematiscches Diagra amm der Abb.47 vorherggesagten Bindestelle von v miR‐13 37 innerhallb der 3’UT TR von KD DM1A (Targ getscan). Pos.=Position.Mod difiziertnachAlthoffet al.118. In verrschiedenen n humane en Tumorrentitäten wie dem m Kolorekktalkarzinom119,120, Lungen ntumoren1221, Melanomzellen122 und Brustkrebs123 ist eine tumorsupp pressive Funktio ondieserm miRNAbere eitsbekann nt. Die Exp pression vo on miR‐137 wurde a uch in Tum moren aus LSL‐MYCN N;Dbh‐iCre Mäusen analysiert,wobei sichherau usstellte,daasssieindeenTumore enimVerglleichzuNe ebennie‐ renvon nWildtyp‐M Mäusenhocchregulierttwar(Abb..48).Immu urinenNeuuroblastom mkonnte dieHyp potheseein nertumorsuppressiveenFunktion nvonmiR‐‐137demnaachnichtb bestätigt werden n. Dies kön nnte darin n begründeet liegen, dass d dem im humannen Neuroblastom vorherggesagten Target T KDM M1A im mu urinen Orgaanismus ke eine Bindesstelle für miR‐137 m vorherggesagtwird d(www.tarrgetscan.orrg). 8: Regulatio on von miR‐137 in Abb.48 LSL‐MY YCN;Dbh‐iCrre Tumoren Boxplot der Expression von miR R‐137 in YCN;Dbh‐iCree Tumoren n im Ver‐ LSL‐MYC gleich zzur Expression in nichtt‐malignen Nebennieren (NN N). Studentt’s t‐Test: <0,001. ***=p< ImfolgendenTeilldieserArb beitsolltessowohldieepotentiellltumorsupppressiveF Funktion von miR‐137 als auch a das onkogene o T Target KDM M1A im humanen Neuuroblastom m unter‐ suchtw werden. 78 Erg gebnisse 4.3.2 E Expressionsanalyse evonmiR‐1 137inhum manenPrimärtumorren Wie sch hon für miR‐542 m du urchgeführtt, wurde in n den vorh handen qP PCR Daten von 69 Neurob blastompatienten67(T Tab.9)auch hdieExpreessionvonm miR‐137unntersucht. In Bezu ug auf verrschiedene prognostiische Mark ker des Ne euroblastom ms, gegen die die miR‐13 37 Expressiion aufgetrragen wurd de, zeigte sich, s dass eine hohe Expression n dieser miRNA signifikant mit günstigem Stad dium einheerging. Auß ßerdem steellte sich eiine Ten‐ ürdieHeru unterregula ationvonm miR‐137in MYCN‐amplifiziertennTumoren heraus. denzfü Die Exp pression von v NTRK1 1 korrelierrte signifik kant mit de er Expresssion von miR‐137 m (Abb.49a). Mittels Kaplan‐‐Meier‐Anaalysen wurrde gezeigt, dass einne hohe miR‐137 m ExpresssionfürdieePatienten nsowohlm miteinerhöherenWah hrscheinlichhkeitdesE Ereignis‐ freien‐alsauchdeesGesamtü überlebens einherging g(Abb.49b b). Abb.49 9: Korrelattion der Ex xpression von miR‐1 137 mit prognostisch hen Marke ern und Überleb ben in hum manen Prim märtumoren n Expressio on von miR‐‐137 in Prim märtumoren n von 69 67 Neurobllastompatienten anhan nd vorhandeener qPCR Daten D (Tab b.9). (a) Veergleich derr Expres‐ sion von n Patienten verschiedener Stadium m, mit oder ohne MYCN N Amplifikattion (MNA) und mit hoher o oder niedrigger NTRK1 Expression als Balkendiagramm. Student‘s t‐ t‐Test.: **=p<0,01; ***=p< <0,001;n.s. =nichtsign nifikant.(b))Kaplan‐Meeier‐Analyse edesEreignnis‐freien‐(E EFS)und des Gessamtüberleb bens (OS) von Patienteen mit Tum moren mit hoher gegenn niedriger miR‐137 Expression.Wilcoxo on‐Test.ModifiziertnacchAlthoffettal.118. 79 Erg gebnisse Die Ree‐Analyse des vorhandenen Next‐Generation‐Seq quenzierunngs‐Datenssatzes110 (Tab.10 utenExpresssionenvo onmiRNAs infünfNeeuroblastom menmit 0),indem dieabsolu günstiggemundun ngünstigem mVerlaufu untersucht wurden,dieschonfüürmiR‐542 2durch‐ geführttwurde,sollteauchfü ürmiR‐137 7ausgeführrtwerden. Die Exp pression vo on miR‐137 korrelierrte signifik kant mit gü ünstigem V Verlauf (p= =0,046) undfiellsignifikan ntniedrigerrausalsdiiederonko ogenenmiR RNAsmiR‐117(p<0,001)und miR‐92 2a(p<0,00 01;Abb.50)). Abb b.50: K Korrelation von miR R‐137 Exprression mitt günsti‐ gem m Verlauf humaner Neuro‐ bla astome Ree‐Analyse vorhan‐ den ner Next‐‐Generation‐‐Sequen‐ zierrungs‐Datennvonjefün nfNeuro‐ blastomen m it günstige em und günstigem V Verlauf110 (Tab.10). ung Darrstellungdeerabsoluten nExpres‐ sion n von miR‐‐137 und de er onko‐ gen nen miRNAss miR‐17 und u miR‐ 92a a. Student‘ss t‐Test. Mo odifiziert nacchAlthoffettal.118. 4.3.3 F Funktionv vonmiR‐1 137inZellllinien Diehoh heExpressiionvonmiR‐137inh humanenNeuroblasto omenmitgüünstigerTu umorbi‐ ologie u unterstütztte die Thesse einer tu umorsupprressiven Fu unktion dieeser miRNA A. Diese These ssollte mitteels in vitro Studien in n humanen n Neuroblastomzelllinnien validie ert wer‐ den. Wiesch honfürmiR R‐542wurrdendiehu umanenZellllinienIMR R‐32,SHEP PundSK‐N N‐BEmit einerfü ürkeinemiiRNAkodie erendenNeegativkontrroll‐miRNA Aodermitm miR‐137trransient transfizziertundd dieZellviabilitätanhan nddermettabolischen nAktivität derZellen nmittels MTTAsssayunterssucht.Dierrelativzum mjeweiligen nWertnacch24Std.ggemesseneZellvia‐ bilitätw wurdegegeendieZeit aufgetrageen.AlledreeianalysierrtenZelllinnienreagiertenauf die Exp pression vo on miR‐137 im Verggleich zu Negativkont N troll‐miRNA A exprimie erenden Zellenm miteinersiignifikantenVerringe rungderV Viabilitätüb berdieZeit t(Abb.51). 80 Erg gebnisse Abb.51 1: Reduktio on der Via abilität hu umaner Neuroblastom mzelllinien mittels ex xogener miR‐137 Expressiion Transie ente Transfeektion der humanen Neuroblastom N mzelllinien IMR‐32, SHEP un nd SK‐N‐BE E mit Negattivkontroll‐m miRNA (NK K) oder miR R‐137. Verlaauf der Zellv viabilität überdieeZeitsigniffikantversch hieden.StattistischeSignifikanzberrechnetanhhandeinesP Permuta‐ tionstesst.ModifizierrtnachAlthoffetal.118. Makrosskopische Aufnahmen A n der Zelle n zeigten 96Std. 9 nacch Transfekktion mit miR‐137 m einedeeutlichverrringerteKo onfluenzim mVergleich hzurTranssfektionvonnNegativk kontroll‐ miRNA(Abb.52). Ab bb.52: Red duktion de er Kon‐ flu uenz hu umaner Neuro‐ blastomzellliinien nacch exo‐ ge ener miR R‐137 Exp pression Miikroskopischhe Aufnahm men von Ze ellen96Std..nachderT Transfek‐ tio on mit Negaativ‐kontrolll‐miRNA (N NK) oder m miR‐137. Maßstabs‐ ba alken = 2000 µm. Mo odifiziert na achAlthoffeetal.118. d Zellzyk klus konntte ein Ansstieg des Anteils A apooptotischerr Zellen Mittels Analyse des en Zelllinieen nach miR‐137 m Ex xpression ddetektiert werden. w (subG1) in allen analysierte Dieser Effektdom miniertein SHEPZelleen,warabeerauchfürrIMR‐32unndSK‐N‐BE EZellen kant (Abb.5 53a). Die relative r An nzahl apopttotischer Zellen nachh Transfekttion von signifik miR‐13 37 gegen Negativkon N ntroll‐miRN NA wurde anhand des ELISA Cell death Assays bestimm mt und zeiigte eine signifikantee Induktion n von Apop ptose in alllen drei Zelllinien (Abb.53b).Inein nerWestern nBlotAnallysewurdeederAnteiildergespaaltenenCaspase9, ein Efffekt der in n pro‐apop ptotischen Zellen au uftritt, unttersucht. D Die Induktion der Caspasee9 Aktivitäät konnte deutlich geezeigt werrden (Abb. 53c). Anhaand der EL LISA‐ba‐ 81 Erg gebnisse sierten BrdU Inkorporation n gemessen n, wurde die d Proliferrationsratee von miR‐‐137‐ex‐ v zu Negattivkontroll‐‐miRNA ex xprimierennden analyssiert. In primierrenden Zelllen relativ allendrreiZelllinieenreduzierrtemiR‐137 7dieProlifferationsig gnifikant(A Abb.53d). Abb.53 3: Induktion n von Apop ptose und R Reduktion der Prolife eration in m miR‐137 ex xprimie‐ rendenZellenAnaalysendesPhänotypsvoonmiR‐137‐exprimiere endenZelllinnienimVerg gleichzu Negativk kontroll‐miR RNA‐exprim mierenden ((NK) 96Std d. nach Tra ansfektion. ((a) Fraktionen von Zellen in n verschied denen Phase en des Zellzyyklus als Prrozent‐Balke en. SubG1‐FFraktion in miR‐137 exprimierenden Zeellen signifik kant erhöhtt. Student‘s t‐Test: p< 0,05. (b) B alkendiagra amm der optotischer ZellenrelattivzuNK,geemessenmiittelsELISACelldeath. Student‘st‐‐Test.(c) Zahlapo Western n Blot Analy yse von KDM M1A und geespaltener Caspase C 9. Aktin A als Laadungskontrrolle. (d) Balkend diagrammdeerZahlprolliferierenderrZellenrelaativzuNK,g gemessenm mittelsELISA A‐basier‐ ter BrdU U Inkorporaation. Stude ent‘s t‐Test. *=p<0,05 5; **=p<0,,01; ***=p <0,001. Mo odifiziert nachAltthoffetal.1188. neuronaleD DifferenzierungvonN Neuroblastoomzellene einleiten Umzu testen,ob miR‐137n kann, w wurde anhaand einer qPCR die E Expression n von Neurrofilament, einem bek kannten Markerrfürneuron naleDiffere enzierung, gemessenundgegen nGAPDHnoormalisiert.Relativ zu Neggativkontroll‐miRNA‐e exprimiereenden Zelleen wurde deutlich, d daass Neuroffilament mierendenZellensign nifikanthocchregulierttwurde(Abbb.54). inmiR‐‐137‐exprim Abb.54: Einleitung neuronaleer Differen nzierung in miR‐137‐e exprimieren nden Zellen Neurofilam ment (NE EFL) mRNA A nach miR‐137 Expressio on relativ zu Neggativkontrolll‐miRNA Expressio on (NK), detektiert d m mittels qPCR und normaliseert gegen GAPDH. Student‘s t‐Test: *=p<0,0 001.ModifiziertnachAllthoffetal.1118. 82 Erg gebnisse 4.3.4 V Validierun ngvonKDM M1AalsdiirektesZie elvonmiR R‐137inZeelllinien Die vorrhergesagte, direkte Bindung vvon miR‐13 37 an die 3’UTR vonn KDM1A sollte s in diesemTeilderUntersuchun ngvalidierttwerden.D DazuwurdenmitmiR R‐137‐transsfizierte Zellen im Vergleiich zu mit Negativkoontroll‐miR RNA‐transfiizierten auuf ihre Exp pression von KD DM1A unterrsucht. Mitttels qPCR wurde diee KDM1A mRNA m Exppression ge emessen undauffdieExpreessionvon GAPDHnoormalisiert..Relativzu urExpressiioninNega ativkon‐ troll‐miiRNA‐expriimierenden n Zellen, w wurde KDM M1A mRNA A in miR‐1 37‐exprim miernden Zellen signifikantt herunterrreguliert (A Abb.55a). In Westerrn Blot Anaalysen kon nnte die Herunterregulatio onvonKDM M1AauchaaufProteineebenevalid diertwerdeen(Abb.55 5b). Abb.55 5: Herunterrregulation n von KDM1 1A in miR‐137‐exprim mierenden Zellen (a)) KDM1A mRNA n nach miR‐137 Expression relativ zu Negativk kontroll‐miRNA (NK) iin humanen n Neuro‐ blastom mzellen detektiert mitte els qPCR un nd normalisiiert gegen GAPDH G Exprression. Stu udent‘s t‐ Test: *= =p<0,05; **=p<0,01 * 1; ***=p<0 0,001. (b) Western W Blo ot Analyse dder KDM1A A Protein Expression Negativ vkontroll‐miRNA‐ (NK)) und miR‐137‐exprim mierender Zeellen. Aktin n als La‐ ontrolle.Mo odifiziertnacchAlthoffettal.118. dungsko Zur Vallidierung der d direkte en Bindungg von miR‐‐137 an die e Bindesteelle der 3’U UTR von KDM1A A (Abb.46)) wurde eiin Reporteer Assay durchgeführ d rt. Ein Glüühwürmche en‐Luzi‐ ferase R ReporterP Plasmid, we elches die 3’UTRvon n KDM1A be einhaltete, das Plasm mid pRL‐ TK,dassalsTransfektionskontrolleRen nilla‐Luzifeeraseexprimierte,unddNegativk kontroll‐ miRNA oder miR R‐137 wurd den sowoh hl in HEK‐2 293 als auch in SHEP P Zellen ko‐trans‐ fiziert. Relativ zu u Negativk kontroll‐m iRNA‐exprimierenden n Zellen, w wurde die e gegen Seefedeer normaalisierte Glühwürmc G chen‐Luzifeerase‐Aktiv vität signnifikant re eduziert (Abb.56). Die vorrhergesagtte direkte B Bindung von miR‐137 an die 33’UTR von KDM1A konntedamitvalidiertwerden. 83 Erg gebnisse 6:DirekteB Bindungvon nmiR‐137 andie Abb.56 3’UTR von KDM M1A Ko‐T Transfektion n von HEK‐293 und SHEP P Zellen en ntweder mitt Nega‐ und ei‐ tivkontrroll‐miRNA (NK) oder miR‐137 u nem KD DM1A 3’UTR R tragenden n Glühwürm mchen‐ Luziferaase Reportter Plasmid. Messun ng der Glühwürmchen‐Luzziferase‐Akttivitätnach 48Std. ng gegen Se eefeder‐Luziiferase‐ und Normalisierun kontrolle p pRL‐TK. Aktivitäät der Traansfektionsk Studentt‘s t‐Test: **=p<0,01.. Modifizierrt nach Althoffeetal.118. Im Weiteren sollte untersu ucht werdeen, ob die Herunterrregulation von KDM1 1A nach miR‐13 37 Expresssion für Sig gnalwege des Proteiins von Be edeutung iist. Ein bekanntes Target vonKDM1AistTFPI2 2(tissuefacctorpathw wayinhibitor2),dasbeeiHerunterrregula‐ tion vo on KDM1A in Neurob blastomzellllinien hocchreguliert wird35. Diie Expression von TFPI2 iin Negativk kontroll‐miRNA‐ und d miR‐137‐‐exprimiere enden Zelllen wurde anhand einer q qPCR gemessen und gegen g GAPD DH normallisiert. Rela ativ zur Koontrolle wu urde die ExpresssionvonTF FPI2inalle enanalysieertenZelllin nienmitmiR‐137signnifikantho ochregu‐ liert,waasdieFunk ktionalitätderKDM1A AHerunterregulationvalidierte (Abb.57). Abb.57:Rea A aktionvon KDM1AZielenauf miR‐137‐ind m duzierte H Herunterreg gulation vo on KDM1A A TFPI2 (tisssue factor pathway in nhibitor 2) mRNA iin Zelllinie en nach miR‐137 m Exp pression rel ativ zu Neg gativkon‐ trroll‐miRNA (NK), detekktiert mitte els qPCR un nd normalissiert gegen GAPDH Exp pression. Sttudent‘s t‐T Test: *=p<<0,05; **=p<0,01; ****=p<0,00 01. Modifizziert nach Althoff ettal.118. 84 Erg gebnisse 4.3.5 F Funktionv vonKDM1 1AHerunteerregulationinZelllinien KDM1A AwurdealssdirektesT Targetvon miR‐137,d dieandie3 3’UTRbinddet,validierrt.Diese Bindun ng führte so owohl zu einer e Heru unterregulaation auf mRNA‐, m als auch auf Protein‐ P ebene. TPFI2,ein bekanntessZielvonK KDM1A,ko onnteebenfallsvalidieertwerden n.Damit wargezzeigt,dass dieBindun ngvonmiR R‐137and die3’UTRv vonKDM1A Afunktione elleAus‐ wirkun ngenaufdieeNeuroblasstomzellen nhervorrufft,dieauchbeidirekteerHerunterrregula‐ tionvon nKDM1Aeeintreten.N Nunsollteu untersuchttwerden,o obderPhännotypderd direkten KDM1A AHerunterrregulationmittelssiR RNAindenhumanenNeuroblasttomzellenIMR‐32, SHEPu undSK‐N‐B BEdenPhänotypder miR‐137E Expressionähnelnwüürde.DieZelllinien wurden n transientt mit einerr Negativkoontroll‐siRNA oder mit m einer s pezifischen n siRNA gegen KDM1A (ssiKDM1A) transfizierrt. Zunächsst wurde die Speziffität dieserr siRNA nsfektion anhand a ein ner Western n Blot Ana alyse unterrsucht, wob bei eine 48Std. nach Tran DM1AProte einsvalidieertwerdenkonnte(Ab bb.58). RedukttiondesKD Abb.58 8: Spezifiscche Reduk ktion von KDM1A A Protein mittels m siRN NA Western Blot An nalyse der KDM1A Expression 48Std.n nachtransieenterTransffektionvon Neurobllastomzelllin nien mit Negativ‐ kontrolll‐siRNA (NK) oder spezifischer s siRNA ggegen KDM1 1A. Aktin als Ladungs‐ kontrollle. Modifiziert nach h Althoff etal.118. yermitteltteZellviabiilitätwurdeegegendie eZeitaufggetragen(A Abb.59). Dieim MTTAssay gulation vo n KDM1A im Ver‐ Alle analysierten Zelllinien reagierten auf die Heerunterreg vkontrollem miteinersiignifikantrreduzierten nViabilität überdieZeit. gleichzzurNegativ Abb.59 9: Reduktio on der Zellviabilität d durch Heru unterregula ation von K KDM1A MT TT Assay relativ zzum jeweiligen 24Std. Wert nach h transienter Transfekttion von Neegativkontro oll‐siRNA (NK) od der siRNA gegen KDM M1A (siKDM M1A). Angeegebene p‐W Werte für ddie Signifik kanz von siKDM1AgegenNK Kberechneta anhandeineesPermutattionstestsModifiziertnaachAlthoffeetal.118. 85 Erg gebnisse Die dettaillierte Untersuchun ng der Me chanismen n der Redu uktion der Zellviabilittät nach Herunterregulatio on von KDM1A mitteels siRNA wurde w anhand einer Zellzyklusanalyse, einesC CelldeathELISAundd derELISA‐b basiertenB BrdUInkorrporation996Std.nach hTrans‐ fektion untersuch ht.DerAnte eilapoptottischerZellenstiegbe eiTransfekktionvonsiiKDM1A im Verggleich zur Negativkon ntroll‐siRN NA an (Abb b.60a). Mittels Cell deeath ELISA A konnte dieInduktionvon nApoptose validiertw werden(Ab bb.60b).Au ußerdemw wurdeeine eReduk‐ elativzurN Negativkontroll‐miRNAaufgetraagenwurde e,detek‐ tionderProliferattion,diere tiert(A Abb.60c). Abb.60 0: Induktion von Apo optose und d Reduktion der Viab bilität und Proliferattion von Zellend durchHeru unterregula ationvonK KDM1AAnaalysen96Std d.nachTrannsfektionvo onNega‐ tivkontrroll‐miRNA (NK) oder siRNA gegeen KDM1A (siKDM1A). (a) Fraktiionen von Zellen Z in verschieedenenPhassendesZellzyklusalsP Prozentbalkeen.(b)Balkendiagramm mderapopttotischen Zellen, relativ zu NK, gemessen mittells ELISA Cell C death. Student‘s t‐Test: *=p<0,05; B amm der Z ahl proliferrierender Zellen relativv zu NK, gemessen **=p<0,01. (c) Balkendiagra B Inkorp poration. Stu udent‘s t‐Te est: *=p<00,05; ***=p p<0,001. anhand der ELISA‐basierten BrdU ModifiziiertnachAltthoffetal.118. 86 Ergebnisse 4.3.6 Partielle Wiederherstellung des Phänotyps der miR‐542‐3p Expression mittelsHochregulationvonKDM1AohnemiRNA‐Bindestelle Der Phänotyp von Neuroblastomzelllinien nach der Herunterregulation von KDM1A mittelssiRNAähneltedemderExpressionvonmiR‐137.Nunwurdeuntersucht,obder miR‐137‐Phänotyp aufgrund der durch miR‐137 Expression herunterregulierten KDM1A Expression auftrat. KDM1A ohne miR‐137‐Bindestelle (3‘UTR) wurde mittels eines Tetrazyklin‐induzierbaren Vektors stabil in SHEP Zellen eingebracht (SHEP‐KDM1A).AlsKontrollewurdenSHEPZellenmitdemgleichemVektorabereiner induzierbarenExpressionvonGFPgeneriert(SHEP‐GFP).UnabhängigvonderBehand‐ lungmitTetrazyklinwurdedieExpressionvonKDM1AinSHEP‐GFPZellenbeiExpres‐ sionvonmiR‐137reduziert.AuchinSHEP‐KDM1AZellenreduziertedieExpressionvon miR‐137 die Expression von KDM1A in Abwesenheit von Tetrazyklin. Bei Behandlung mit Tetrazyklin wurde die Expression von KDM1A ohne 3‘UTR in SHEP‐KDM1A Zellen induziert,dieresistentgegendieExpressionvonmiR‐137war(Abb.61a).MittelsMTT Assay wurde die Zellviabilität unter den gleichen Bedingungen wie im Western Blot analysiert.DieViabilitätderZellenbeiExpressionvonmiR‐137wurdedazurelativzur jeweiligen Negativkontroll‐miRNA exprimierenden Viabilität aufgetragen. Die Expres‐ sion von miR‐137 reduzierte die Viabilität sowohl von SHEP‐GFP als auch von SHEP‐ KDM1AZelleninAn‐undAbwesenheitvonTetrazyklinsignifikant.InnerhalbderSHEP‐ GFP Zellen konnte kein Unterschied zwischen der Zellviabilität von nicht‐induzierten undinduziertenZellen,diemitmiR‐137behandeltwurden,detektiertwerden.Zwischen miR‐137exprimierendenSHEP‐KDM1AwardieReduktionderZellviabilitätininduzier‐ tenZellensignifikantwenigerstarkalsinnicht‐induziertenZellen(Abb.61b). 87 Erg gebnisse Abb.61 1: Induzierb bare Expre ession von n KDM1A in SHEP‐KD DM1A Zelleen resisten nt gegen miR‐137 Expresssion (a) Western W Bl ot Analysee der KDM M1A Expresssion 96Sttd. nach Tetrazyk klin‐ (TET) oder Lösungsmittel‐B Behandlung von stabil‐transfizierteen SHEP‐Ze ellen mit einemT Tetrazyklin‐iinduzierbarrenVektorkoonstruktmiitGFP(SHEP P‐GFP)oderrKDM1AcDNAohne 3’UTR.G GleichzeitigeeExpressionvonmiR‐1 137.AktinaalsLadungskontrolle.(b b)Balkendiiagramm der Zelllviabilität vo on miR‐137 7 exprimier enden SHEP P‐GFP und SHEP‐KDM11A Zellen relativ zu Negativk kontroll‐miR RNA exprim mierenden iin Ab‐ und Anwesenheit von Tettrazyklin, gemessen mittels MTT Assay y. Student‘ss t‐Test: ***=p<0,01; ***=p<0,001. Modiffiziert nach h Althoff etal.118. erungundValidierun ngvonAK KAP2alsweiteresZieelvonmiR R‐137 4.3.7 IIdentifizie Das du urchgeführtte Rettung gsexperimeent des miR‐137 Phä änotyps deemonstrierte, dass zwar eein Teil dees Effekts der miR‐1 137 Expresssion auf die Herunnterregulatiion von KDM1A Azurückzufführenwarr,aberdasssdergrößeereAnteiln nichtallein aufdieReg gulation von KD DM1A, sond dern allein n oder zusäätzlich auff weitere miR‐137 m Ziiele zurück kgeführt werden nmusste. ZurDettektionweeitererpote entiellerZieelewurde 96Std.nacchTransfe ktioneine Proteo‐ manalyyse von miR‐137 exprimierendeen SHEP Zellen durch hgeführt, w wobei Nega ativkon‐ troll‐miiRNA exprrimierende Zellen alss Vergleich h dienten. Dabei wuurden 38 Proteine P gefundeen, die durrch miR‐13 37 Expresssion signifiikant regulliert warenn (Daten nicht n ge‐ zeigt). Mittels in silico Anallyse wurdee untersucht, welche dieser reggulierten Proteine P sowohlleineBindeestellefürmiR‐137au ufwies,als auchalspotentiellessOnkogeniinFrage kam. 88 Erg gebnisse Das Pro otein AKAP P2 (A kinasse anchor pprotein 2) war in miR R‐137 exprrimierende en SHEP Zellenb beiderAnaalysedesP Proteomsaalssignifikaantherunte erreguliert gefunden wurden (Abb.62a),wasim mWesternBlotvalidieerbarwar((Abb.62b). 2: Herunte erregulation n von AK KAP2 Abb.62 mittels Expressio on von miR R‐137 (a) Ver‐ gleich d der Expressiion von AKA AP2 in Negaativ‐ kontrolll‐miRNA (N NK) exprim mierenden und miR‐137 7exprimierrendenZelle enanhandeeiner Proteom manalyse.(b b)WesternB BlotAnalyseeder gleichen n Proben wie w (a). Aktiin als Ladu ungs‐ kontrollle. Dem Protein AKA AP2, über dessen tu umormodulierende Funktion F b isher noch h nichts bekann ntist,wurdenvierdirekteBindeestellenvon nmiR‐137am3’UTRddermRNAvorher‐ gesagt (www.tarrgetscan.orrg). Innerh halb der 4014bp langen 3’U UTR lagen n diese BindesttellenandenPosition nen912‐91 18(7mer‐1 1A),1634‐1 1640(7meer‐m8),177 75‐1782 (8mer)und3851‐‐3857(7me er‐m8)(Abb b.63). 3: 3’UTR vo on AKAP2 mit Bindesstellen von n miR‐137 Schematiscches Diagra amm der Abb.63 vorherggesagten Bindestellen B n von m miR‐137 innerhalb der 3’U UTR von AKAP2 (www.taargetscan.orrg).Pos.=Position. Anhand d einer Analyse der Exprression vo on AKAP2 2 in hum manen prrimären Neurob blastomen sollte festg gestellt werrden, ob dieses Prote ein eine tuumormodullierende Funktio on im Neurroblastom spielen köönnte. Mitteels einer Kaplan‐Meie K er Analyse e konnte gezeigt werden, dass Patienten mitt Tumoren n mit hoh her AKAP2 2 Expressio on eine 89 Erg gebnisse signifik kantniedriggeWahrsch heinlichkeiitfüreinE Ereignis‐freiesÜberlebbenaufwie esen,als PatienttenmitTum morenmit niedrigerA AKAP2Exp pression.Diesgaltauuchfürdie Analyse desGessamtüberleebens(Abb b.64).DesW Weiterenk korrelierteeineerhöhhteExpresssionvon AKAP2imTumorsignifikanttmitMYCN NAmplifikattion(Daten nnichtgezeeigt). on der Abb.64: Korrelatio Expression n von AKA AP2 mit Überleben n von Neu‐ roblastomp patienten Kaplan‐ Meier‐Analyyse des Ereignis‐ E freien (EFSS) und des Gesamt‐ überlebens (OS) vo on Neu‐ roblastomppatientenmiitTumo‐ ren mit hohher gegen niedriger n AKAP2 Exppression. Wilcoxon‐ W Test. gulation m Im Folggenden sollte die Fun nktion eineer AKAP2 Herunterre H mittels RNA A Intefe‐ renzin SHEPZelleennäheruntersuchtw werden.DiieViabilitättderZellenn,diemitssiAKAP2 deltwurden n,warimV VergleichzzumitNeg gativkontroll‐siRNAtrransfizierte enredu‐ behand ziert(A Abb.65a).W WeiterhininduziertessiAKAP2in nSHEPZellensowohl Apoptosea alsauch eine Reeduktion der d Zellproliferation, gemessen anhand de es Cell deaath ELISA und u der ELISA‐b basiertenB BrdUInkorp poration(A Abb.65b/c). Abb.65 5:ReduktionderZellviabilitätun ndProlifera ationundIn nduktionvo onApoptosseinmit siRNAg gegenAKAP P2transfiziiertenSHEP PZellenTransienteTransfektionvvonSHEPZe ellenmit Negativk kontroll‐siR RNA(NK)od dereinersiR RNAgegen AKAP2(siA AKAP2).(a) VerlaufderrZellvia‐ bilitätü überdieZeittrelativzum mWertnach h24Std.An ngegebenep p‐Wertefür rdieSignifik kanzvon siAKAP2 2gegenNKberechneta anhandeineesPermutatiionstests.(b b)Balkendiaagrammderrapopto‐ tischen Zellen96Sttd.nachTransfektionreelativzuNK K.Student‘stt‐Test:*=p <0,001.(c))Balken‐ mmderZahllproliferiere enderZellen nrelativzu NK96Std. nachTranssfektion.Stu udent‘st‐ diagram Test:*= =p<0,05. 90 Disskussion 5 Diiskussiion Die vorrliegende Arbeit A umffasst die Ch harakterisiierung eines neuen C Cre‐konditionalem MYCN‐vvermittelteen Neurob blastom‐Maausmodells. Anhand dieses M odells sow wie aus weitereenvorhand denen Date en wurden zweiKand didaten‐miR RNAs mitttumormodulieren‐ derFun nktioniden ntifiziert,de erenFunkttioneninhu umanenNe euroblastom menundZelllinien weiteraanalysiertw wurden. 5.1 D DasLSL‐MYCN;D Dbh‐iCre eMausm modell 5.1.1 T TumoredesLSL‐MY YCN;Dbh‐iC CreMausm modellslassensichaalsmurineNeuro‐ b blastomeb beschreiben Eine Überexpresssion oder Amplifikattion von Wildtyp W MYCN geht mit einem m hohen Stadium m und eineer schlechtten Prognoose human ner Neurob blastome eiinher124. Während W die Heiilungschancen eines Neuroblast N toms mit niedrigerem n m Stadium bei jüngerren Kin‐ dernseehrgutsind d,istdieStterberatevvonPatienttenmitSta adium4Neeuroblastom menmit MYCN‐A Amplifikatiionnochim mmerhoch h1.Besondeersfürdie ErforschunngderSign nalwege in diesen bösartiigen Tumoren sind M Mausmodelle, die den genetiscchen Statuss dieser Erkrank kungen wiiderspiegeln, von grooßer Bedeu utung. Weitterhin sindd vorklinische Un‐ tersuch hungen von n potentielll wirksam men spezifisschen Med dikamentenn möglich. Im hier analysiertenLSL‐M MYCN;Dbh‐iCreMaussmodellwu urdeunterssucht,inwieeweitesCh harakte‐ ristika deshuman nenNeurob blastomsin nsichvereiinigt,umA Aufschlüsseeüberneue eThera‐ piendieeserbeimK Kindfatalv verlaufendeenErkrank kungsformzzugeben. Die vom m Dbh‐Promotor getrriebene Ex pression der d Cre‐Rek kombinase findet hau uptsäch‐ lich im m sympathischen Gren nzstrang s tatt40. In LSL‐MYCN; L Dbh‐iCre M Mäusen füh hrte die Expresssion der Cre‐Rekomb C binase zur gewebssp pezifischen Expressio n von MYC CN, was anhand d der Unterrsuchungen n der Tum ore auf mR RNA‐ und Proteinebe P ene gezeigtt wurde. Die anaatomischen n Lokalisattionen diesser MYCN‐vermittelte en Tumoree an Neben nnieren, oberen Zervikalgganglien un nd dem G Ganglion co oeliacum ähnelten ä deer Lage humaner h Neurob blastome, die d sich au us abdomin nalen Gang glien, dem sympathisschen Gren nzstrang undden nNebennieerenentwicckeln4. 91 Diskussion ImhistologischenBildbestandenTumoreausLSL‐MYCN;Dbh‐iCreMäusenauskleinen, blauen,rundenZellen.InderpathologischenUntersuchungvonhumanenpädiatrischen TumorenwirdebenfallszuersteineHämatoxylinundEosinFärbungdurchgeführt.Die Unterscheidung zwischen den Entitäten der klein‐, blau‐ und rundzelligen Tumore erfolgthauptsächlichanhanddesNachweisesspezifischerMarkerinimmunozytochemi‐ schen und genetischen Analysen. Sind diese Untersuchungen nicht eindeutig, werden elektronenmikroskopischeAufnahmenzuRategezogen.Alsspezifischeultrastrukturelle Kennzeichen des Neuroblastoms innerhalb der klein‐, blau‐ und rundzelligen Tumore gelten Neuriten, neurosekretorische Vesikel, Synapsen‐ähnliche Strukturen und zwi‐ schenzelluläre Anschlüsse1. Neurosekretorische Vesikel, die neurale Differenzierung anzeigen,warenauchinTumorenausLSL‐MYCN;Dbh‐iCreMäusendetektierbar. Tumore des LSL‐MYCN;Dbh‐iCre Modells exprimierten sowohl das Oberflächenantigen CD56,alsauchdieNeuroblastommarkerDbh,ThundPhox2b.DieExpressionvonCD56 gilt als Marker für neuroendokrine Tumore. Th katalysiert die Biosynthese der Kate‐ cholamine und wird vor allem in noradrenergen Zellen exprimiert125. Bei der Unterscheidung von Neuroblastomen zu anderen klein‐, blau‐ und rundzelligen Tumo‐ rengiltdieExpressionvonTh,diespezifischfürdasNeuroblastomist,alsMerkmal126. Phox2b wird bei der Entwicklung von sympathischen, parasymphatischen und enteri‐ schen Ganglien exprimiert, die sich von der Neuralleiste ableiten127. Hartomo etal. beschreiben elf Marker, anhand derer sich eine minimale Resterkrankung des Neuro‐ blastomsdetektierenlässt,unterdenensichsowohlDbhundThalsauchPhox2bbefin‐ den128. Anhand der Lokalisation, der histologischen Eigenschaften und der Expression von spezifischen Neuroblastommarkern wurde die Hypothese bestätigt, dass es sich bei in dieser Arbeit analysierten Tumoren des LSL‐MYCN;Dbh‐iCre Mausmodells um murine Neuroblastomehandelte. 92 5.1.2 Gegenüberstellung der beiden Diskussion Neuroblastom‐Mausmodelle LSL‐MYCN;Dbh‐iCreundTH‐MYCN Das TH‐MYCN Mausmodell37 stellt ein exzellentes, weltweit anerkanntes Neuro‐ blastommodelldar,dasinverschiedenenArbeitsgruppen,diesichmitderErforschung desNeuroblastomsbeschäftigen,zuüber200Publikationengeführthat.Esgibtjedoch EinschränkungendesModells,diedieForschungandiesemerschweren.IndiesemTeil derArbeitsollenverschiedeneEigenschaftendesTH‐MYCNunddesLSL‐MYCN;Dbh‐iCre Mausmodells einander gegenübergestellt werden, um abzuschätzen, ob das neue MausmodellVorteilegegenüberdemetabliertenaufweist. DasTH‐MYCNModellwurde1997durchPronukleusinjektiondercDNAvonhumanem MYCNinmurineBlastozystengeneriert37.DieDefinitiondergenauenIntegrationsstelle inderdistalenRegiondesChromosoms18imGenomderMauserfolgteerst2003129.Es ist nicht bekannt, ob der entsprechende Lokus ubiquitär exprimiert oder epigenetisch inaktiviert wird, was zu einer instabilen Expression von MYCN führen könnte. Das LSL‐MYCN;Dbh‐iCre Modell wurde mittels eines Rekombinase‐vermittelten Kassetten‐ austauschineinengenaudefiniertenGenlokusgeneriert,waszurFolgehatte,dassder Genlokus,indendasMYCNTransgenintegriertwurde,genaubekanntwar.DerROSA26 Genlokus stellt bei der Generierung transgener Mausmodelle den am häufigsten ver‐ wendetengenetischenLokusdar,daeineUnterbrechungdiesesubiquitärexprimierten GensbekanntermaßenkeinenPhänotyphervorruft130‐133. VorteiledesLSL‐MYCN;Dbh‐iCreModellsgegenüberdemTH‐MYCNModell,dieIntegra‐ tionsstelledesTransgensbetreffend,sinddemnachzumeinendieGewissheit,dassjede phänotypischeVeränderungdoppelt‐transgenerTiereaufdasVorhandenseindesinte‐ grierten Transgens, nicht aber auf die Unterbrechung eines endogenen Lokus zurück‐ zuführen ist und zum anderen die Kenntnis, dass der Lokus ubiquitär exprimiert und nichtinirgendeinerWeisereprimiertwird. Die Vermehrung und Erhaltung der transgenen Mausstämme LSL‐MYCN und Dbh‐iCre erfolgt durch die Verkreuzung mit Wildtyp‐Tieren oder mit Tieren des gleichen Stam‐ mes.DadiegetrennteZuchtbeiderStämmezukeinemPhänotypführtistdieseunkom‐ plizierter als die des TH‐MYCN‐Modells, das im 129x1/SvJ Hintergrund potentiell Tu‐ more entwickelt. Die Ausbildung eines tödlichen Tumors in einem transgenen Muttertier führt dabei häufig zum Verlust des ganzen Wurfes oder zur Notwendigkeit 93 Diskussion einerAmme.InfolgedessenwerdenhäufigheterozygoteTH‐MYCN‐MännchenundWild‐ typ‐Weibchen für die Erhaltungszucht benutzt, die stets verfügbar sein sollten. Es ist unabdingbar jedes transgene TH‐MYCN Tier nach Tumoren abzutasten, was einen enormenZeitaufwandmitsichbringt.WeiterhinbestehtinnerhalbeinerPopulationaus TH‐MYCN Tieren ein Selektionsvorteil für die Tiere, die keinen Tumor entwickeln, da sich diese mit größerer Wahrscheinlichkeit fortpflanzen. Dass dies auf Dauer zu einer AbnahmederTumorinzidenzführenkann,wurdeauchinunsererArbeitsgruppefestge‐ stellt. TumoredesTH‐MYCNModellsentstammenunifokalausdemGanglioncoeliacum37.Die LokalisationvonTumorendesLSL‐MYCN;Dbh‐iCreModells,dievorallemandenNeben‐ nieren, aber auch an den oberen Zervikalganglien auftraten, spiegelte die humaner Neuroblastome4demnachbesserwideralsdiedesTH‐MYCNModells. MöglicherweiseistdergenetischeHintergrund129x1/SvJsensiblerfürdieEntwicklung bösartigerNeuroblastomealsandereMausstämme,wiez.B.C57Bl/6N,weilsiesichin der Expression nicht‐identifizierter Stamm‐spezifischer Modifizierer unterscheiden134. Trotz des unsensibleren Hintergrundes zeigte die Kaplan‐Meier‐Analyse der beiden Mausmodelle eine signifikant gesteigerte Tumor‐Inzidenz des LSL‐MYCN;Dbh‐iCre Modells. AufgrundderExpressionvonLuziferaseinCre‐RekombinaseexprimierendenZellendes LSL‐MYCN;Dbh‐iCreModellssindTumoredieserMäusemittelsinvivoBiolumineszenz‐ Bildgebungsverfahren detektierbar. Diese Eigenschaft der Tumore kann eingesetzt werden, um Tumorvolumenveränderungen im Vergleich mit anderen Individuen des gleichen Stammes, z. B. während einer Behandlungssimulation, zu untersuchen (hier nichtdurchgeführt).TumoreausTH‐MYCNMäusenexprimierendahingegenkeinRepor‐ tergen, weshalb diese Methode der schnellen und unkomplizierten Bildgebung nicht anwendbarist. Die Kombination von Tumormodellen mit anderen transgenen Mausmodellen, die ein fürdieEntwicklungderTumorentitätpotentiellfunktionellesGenherunter‐oderüber‐ exprimierenisteineinteressanteStrategie,fürdieErkrankungrelevanteSignalwegezu findenundzuuntersuchen.DasLSL‐MYCN;Dbh‐iCreModellentwickelteimnicht‐sensi‐ 94 Diskussion tivengenetischenHintergrund(75%C57Bl/6Nund25%129x1/SvJ)Tumore,während TH‐MYCN Mäuse nur im sensitiven 129x1/SvJ Hintergrund Tumore ausbilden. Eine KombinationvonTH‐MYCNmitanderenMausmodellenistdemnachnurmöglich,wenn dieseweitestgehendaufden129x1/SvJHintergrundzurückgekreuztwurden.Erfahrun‐ gen unserer Arbeitsgruppe zeigten, dass mindestens fünf Rückkreuzungen (96,9% 129x1/SvJ) erforderlich waren, damit Mäuse im gemischten genetischen Hintergrund eine ähnliche Tumorinzidenz erreichten, wie reine 129x1/SvJ Tiere. Der zeitliche Auf‐ wandderRückkreuzungenvonmehrerenMonaten,derbeieinerKombinationmitdem TH‐MYCN Modell aufgewendet werden muss, ist bei Verwendung des LSL‐MYCN;Dbh‐ iCreModellnichterforderlich.DarüberhinaussindtumorspezifischeRegulationenvon GenenbeiKombinationdesLSL‐MYCN;Dbh‐iCremitanderenCre‐konditionalenModel‐ lenohnegroßenAufwandmöglich. Aus der Gegenüberstellung der beiden murinen Neuroblastommodelle TH‐MYCN und LSL‐MYCN;Dbh‐iCreergabensichdiverseVorteilefürdieLSL‐MYCN;Dbh‐iCreMaus,wie dieKenntnisseüberdenGenlokusderTransgenintegration,dieKosten‐undZeiterspar‐ nisbeiderErhaltungszucht,dieanatomischenLokalisationenderentstehendenTumore sowiederenInzidenz,dieOpportunitätderLumineszenz‐Bildgebungsowiedieunkom‐ plizierteundschnelleMöglichkeitderKombinationmitanderentransgenenTiermodel‐ len. Das LSL‐MYCN;Dbh‐iCre Mausmodell überkommt somit die in dieser Arbeit disku‐ tiertenLimitationendesetabliertenTH‐MYCNModells. 95 Diskussion 5.1.3 Identifikation neuer Ziele einer Neuroblastomtherapie anhand des LSL‐MYCN;Dbh‐iCreMausmodells IndiesemTeilderArbeitsollengenomischeAberrationenundreguliertemRNAssowie miRNAsausTumorendesLSL‐MYCN;Dbh‐iCreModellsmitdenenhumanerErkrankun‐ genundmitdenenanderermurinerNeuroblastomeverglichenwerden,umfestzustel‐ len,obdasLSL‐MYCN;Dbh‐iCreModellgenutztwerdenkönnte,umneuepotentiellfunk‐ tionelleZieleeinerNeuroblastomtherapiezudetektieren. 5.1.3.1 Vergleich genomischer Aberrationen von Tumoren aus LSL‐MYCN;Dbh‐iCre MäusenmitdenenhumanerundanderermurinerNeuroblastome Genomische Aberrationen von 13 LSL‐MYCN;Dbh‐iCre Tumoren wurden relativ zu GewebeausdemSchwanzderjeweilsgleichenMausdetektiert. Eine teilweise Zunahme des Chromosoms 1 wurde in einem der Tumore aus LSL‐MYCN;Dbh‐iCreMäusengefunden.DieZunahmedeshumanenChromosoms2q,das ortholog zum murinen Chromosom 1 ist, wird für 12% der humanen Neuroblastome beschrieben135.TeilederhumanenChromosomen1q,2q,5q,6p,8q,13qund18qsind ebenfalls ortholog zu Teilen des murinen Chromosom 1, jedoch ist deren Funktion bisher nicht bekannt. Im TH‐MYCN Modell ist eine Zunahme desChromosoms 1 in 5% bis30%derTumorengefundenworden37,129,136. DieTumoredesLSL‐MYCN;Dbh‐iCreModellsweisenin69%derFälleeineZunahmedes komplettenChromosoms3auf,welchesorthologzuTeilenderhumanenChromosomen 1,3q,4,8qund13qist137.DashumaneChromosom1q,eineRegion,diehäufiginhuma‐ nenNeuroblastomenmehrfachauftritt,istebenfallsortholog138.Daspubliziertemurine Neuroblastommodell, das von der Überexpression von mutiertem ALKF1174L getrieben wird,weistin100%derFälleeineZunahmedesChromosoms3auf42. Das murine Chromosom 6, dessen Zunahme in fünf Tumoren auftrat, ist teilweise or‐ tholog zu den humanen Chromosomen 2p, 3, 4q, 7, 10q und 12p. In humanen Neuro‐ blastomen kommt eine Zunahme des kompletten Chromosoms 7 in 40% der Tumore verschiedener Stadien vor, wobei eine Zunahme von 7q in 12% auftritt und mit hoch‐ Risiko Tumoren assoziiert ist139. Interessanterweise weisen zwei Tumore eine lokale AmplifikationdesChromosoms6auf,diedenROSA26‐Genlokuseinschließt,indendas 96 Diskussion humane MYCN Transgen kloniert wurde. Infolgedessen wurde untersucht, ob beide Tumore, die über diese Aberration verfügen, im Vergleich zu denen ohne Aberration auch eine höhere Expression des Transgens aufweisen und es konnte gezeigt werden, dass dies der Fall war (Daten nicht gezeigt). Schon im TH‐MYCN Mausmodell wurde gezeigt, dass homozygote Mäuse dieser Linie schneller Tumore entwickelten und dass dieseschnellerzumTodführtenalsbeiheterozygoteTiere.MitderhöherenExpression vonMYCNwurdediesbegründet137.Sowirdvermutet,dassdieZunahmedesROSA26‐ LokusimneuenLSL‐MYCN;Dbh‐iCreModelldasTumorwachstumbeschleunigenkönnte. Die Zunahme der distalen Hälfte des Chromosoms 11 in 30% der LSL‐MYCN;Dbh‐iCre Tumore ist ortholog zum humanen Chromosomenarm 17q, der in 50% aller humaner Neuroblastome mehrfach vorkommt und mit ungünstigem Verlauf der Erkrankung sowiemitMYCNAmplifikationkorreliert42.Einbekanntes,potentiellfunktionellesOnko‐ genaufdiesemLokusistSurvivin,dasinderRegion17q25liegt140,141.TH‐MYCNTumore weisen ebenfalls häufig (20‐30%) eine Zunahme der Region auf Chromosom 11 auf129,136,137. DieZunahmedesChromosoms12istorthologzuTeilendeshumanenChromosoms2p, das den endogenen Mycn Lokus enthält, sowie 14q und Teilen von 7p und 7q. Die Zu‐ nahme von 7q tritt, wie schon erwähnt, in 12% humaner Neuroblastome auf ist mit hohemRisikokorreliert139. Der Verlust der Heterogenität von Chromosom 1p36 korreliert in humanen Neuro‐ blastomenmitderAmplifikationvonMYCNundeinerungünstigenPrognose142‐144.Auf diesemLokusliegtmiR‐34a,derinverschiedenenTumorentitäteneinetumorsuppres‐ siveFunktionzugeschriebenwird145,146.DieExpressionvonmiR‐34ainduziertinNeuro‐ blastomzellen Apoptose63 und reguliert direkt die Expression von MYCN64. Weitere potentiell funktionelle Kandidaten auf diesem Lokus sind CHD5147 und KIF1Bβ148. Ein VerlustdesmurinenChromosoms4,dasdasOrthologzumhumanenLokus1p36ent‐ hält,istwederimTH‐MYCNModell136,nochinTumorenvonLSL‐MYCN;Dbh‐iCreMäusen vorhanden.Jedochweisen50%derTumoredesALKF1174L ModellseinenVerlustdieses Lokus auf42. Eine Erklärung für diese Beobachtung könnte sein, dass das Transgen in beiden MYCN‐vermittelten Mausmodellen in Form von cDNA vorliegt und damit keine Bindestelle für miR‐34a aufweist. Dementsprechend wäre ein Verlust dieses Lokus für 97 Diskussion dieMYCN‐getriebenenModellekeinVorteilfürdieEntstehungvonTumoren.Weiterhin wird damit die Hypothese unterstützt, dass in humanen Neuroblastomen durch die RepressionvonmiR‐34adieTumorentwicklungangetriebenwird,wodurchdieFunkti‐ onalitätdesVerlustesdesChromosoms1p36erklärtwerdenkönnte. ImVergleichzudemALKF1174L;Dbh‐iCreNeuroblastommodellsindchromosomaleAber‐ rationen in Tumoren aus LSL‐MYCN;Dbh‐iCre und TH‐MYCN Mäusen seltener 37,42,129. EineÜberexpressionvonWildtypMYCNbenötigtdemnach,umeinenTumorzutreiben, weniger bis keine weiteren spontanen Aberrationen als die Überexpression der ALKF1174L Mutation. Heukamp et al. beschreiben in einem von drei Tumoren von drei‐ fach‐transgenen TH‐MYCN;ALKF1174L;Dbh‐iCre eine Zunahme des Chromosoms 3. Des Weiteren werden nur wenige Verluste einzelner Regionen von Chromosomen detek‐ tiert42.DemnachsindbeigleichzeitigerÜberexpressionvonWildtypMYCNundmutier‐ tem ALKF1174L weniger chromosomale Aberrationen nötig, als bei den beiden MYCN‐ getriebenenModellen. Aus dem Vergleich der chromosomalen Aberrationen des LSL‐MYCN;Dbh‐iCre Modells mitpubliziertenhumanenundanderenmurinenNeuroblastomenergibtsich,dasssich dieSpektrenderchromosomalenAberrationensehrähnlichsind. 5.1.3.2 Vergleich der Genregulationen von Tumoren aus LSL‐MYCN;Dbh‐iCre Mäusen mithumanenundanderenmurinenNeuroblastomenaufmRNAEbene Mittels mRNA Profiling drei nicht‐maligner Nebennieren und acht Tumoren aus LSL‐MYCN;Dbh‐iCreMäusenwurdenimModellreguliertemRNAsidentifiziert. Anhand der GSEA wurden diverse Gensets, die von MYC(N) regulierte Ziele enthalten, als signifikant angereichert in Tumoren des LSL‐MYCN;Dbh‐iCre Modells detektiert, wodurchdieHypothesebestätigtwird,dassMYCNimmurinenSystemähnlicheSignal‐ wege reguliert wie im humanen. Weitere Gensets, die in Tumoren von LSL‐MYCN;Dbh‐iCreMäusenalssignifikantangereichertdetektiertwurden,beinhalteten Gene, die sowohl die DNA Replikation als auch den Zellzyklus betrafen. Dementspre‐ chendreguliertendieseTumoreGene,diefürdieKennzeichenvonTumorzellen,wiedie Stimulation ihres eigenen Wachstums, Resistenzen gegen Wachstumsstopp sowie 98 Diskussion ApoptoseundunendlicheVervielfältigung149,vonBedeutungsind.DieAnreicherungvon Gensets,dieneuronaleDifferenzierungherunterregulieren,bestätigte,dasssichTumore ausLSL‐MYCN;Dbh‐iCreMäusenineinemundifferenziertenStatusbefinden,einweite‐ resbekanntesMerkmalvonTumorzellen150. Mittels einer Clusteranalyse der Gene, die in einer publizierten MYCN Signatur aufge‐ decktwurden78,konntenTumoredesLSL‐MYCN;Dbh‐iCreModellseindeutigvonnicht‐ malignenNebennierensepariertwerden,eindeutlicherHinweisaufdieÄhnlichkeitder MYCNreguliertenmRNAsinmurinenundhumanenTumoren. DieRegulationeinzelnermRNAsderMYCNSignatur78undweitererbekannterZielevon MYCNinTumorendesLSL‐MYCN;Dbh‐iCreModellswurdenimFolgendenmithumanen Neuroblastomenverglichen: Cyclin‐abhängige Kinase 4 (Cdk4) wird während der G1‐Phase des Zellzyklus von Cyc‐ linendesTypesDaktiviert.Molenaaretal.zeigen,dassdiehoheExpressionvonCDK4 mitschlechteremÜberlebenvonNeuroblastompatientenkorreliert.AußerdemistCDK4 sowohl in Tumoren als auch Zelllinien im Vergleich zu verschiedenen nicht‐malignen Geweben hoch exprimiert. Die Herunterregulation von CDK4 in humanen Zelllinien führt sowohl zu einem Arrest in der G1‐Phase des Zellzyklus als auch zur Induktion neuralerDifferenzierung151,152.DieerhöhteExpressionvonCdk4inLSL‐MYCN;Dbh‐iCre TumorenkönntemalignenZellendenDurchgangdurchdieG1‐Phaseerleichtern. DasUbiquitin‐konjugierendeEnzymE2C(Ube2c)spieltbeiderDegradationvonmitoti‐ schen Cyclinen und dem Ablauf des Zellzyklus eine Rolle153. In Gliomzellen führt eine Herunterregulation von UBE2C zu einem Zellzyklusarrest in der G2/M Phase und zu Apoptose154.IndenhumanenNeuroblastomzellenSHEPundSK‐N‐BE(2)‐CwirdUBE2C einewichtigeRolleinderMitosezugeschrieben,wieeinsiRNAScreeningaufdeckte101. LSL‐MYCN;Dbh‐iCreTumorezeigteneineHochregulationvonUbe2c. MYCN‐amplifiziertehumaneNeuroblastomeexprimierensignifikanthöhereMengenan Laktat‐Dehydrogenase(LDH)imVergleichzuTumorenmitnureinerMYCNKopie.Die HerunterregulationvonLDHindenbeidenMYCN‐amplifiziertenNeuroblastomzelllinien LAN5 und Kelly reduziert deren Proliferation. Außerdem verlieren Kelly Zellen bei komplettemVerlustvonLDHihreTumorigenitätinvivo155.LSL‐MYCN;Dbh‐iCreTumore exprimiertenLDHimVergleichzunicht‐malignenNebennierenüber. 99 Diskussion EineÜberexpressionvonNme1korreliertmitungünstigemStadiumvonNeuroblastom‐ patienten. Eine Zunahme des Nme1 Gens, das auf Chromosom 17q lokalisiert ist, tritt häufiginNeuroblastomenmithohemRisikoauf156.AußerdemistdasExpressionslevel vonNme1starkmitderAggressivitätMYCN‐amplifizierterNeuroblastomeassoziiert102. DieHochregulationvonNme1mRNAinLSL‐MYCN;Dbh‐iCreTumorenwurdeparallelzur ZunahmedesChromosoms11,dasorthologzuhumanem17qist,festgestellt. DerFamiliederRAS‐GenewerdeninverschiedenenTumorentitätenonkogeneFunktio‐ nen zugeschrieben157. Einer der drei Hauptvertreter dieser Familie ist NRAS, das ur‐ sprünglichauseinemhumanenNeuroblastomisoliertwurde158.InNeuroblastomzellen mithoherMalignitätwirdNRAShöherexprimiertalsindenenmitniedrigererMaligni‐ tät100.InTumorendesLSL‐MYCN;Dbh‐iCreModellswarNrasebenfallserhöht. Schulteetal.beschreiben,dassKDM1AinundifferenziertenNeuroblastomenhochregu‐ liert ist. Außerdem regulieren differenzierende Neuroblastomzelllinien KDM1A herun‐ ter und die Herunterregulation von KDM1A mittels siRNA führt zur Wachstumsinhibi‐ tion in vitro35. Sowohl LSL‐MYCN;Dbh‐iCre als auch TH‐MYCN Tumore weisen im Vergleich zu nicht‐malignen Nebennieren eine signifikante Überexpression von Kdm1a mRNAauf. LSL‐MYCN;Dbh‐iCreTumorenexprimierienSpermidinSynthase(SRM)über.DieExpres‐ sion diesesEnzyms korreliert stark mit der Expression von MYCN in humanen Neuro‐ blastomen103. Weitere Gene, die im LSL‐MYCN;Dbh‐iCre Modell reguliert wurden, wurden mit einer Internet‐Datenbank Myc‐regulierter Gene verglichen (www.myc‐cyncer‐gene.org), wobei validiert werden konnte, dass diverse Ziele von MYC, die im humanen Organis‐ musreguliertwerden,auchimmurineneinerRegulationunterliegen. Einzelne mRNAs, die im murinen System anders reguliert waren, als im humanen Neuroblastom, wurden ebenfalls detektiert, wodurch der Unterschied zwischen den beidenSpeziesdeutlichwird.VerschiedeneepigenetischeRegulationsmechanismenund verschiedeneSignalwegekönntenhiereineBegründungsein. 100 Diskussion 5.1.3.3 Vergleich der Genregulationen von Tumoren aus LSL‐MYCN;Dbh‐iCre Mäusen mithumanenundanderenmurinenNeuroblastomenaufmiRNAEbene Mittels miRNA Profiling von fünf nicht‐malignen Nebennieren und elf Tumoren aus LSL‐MYCN;Dbh‐iCreMäusenwurdenimModellreguliertemiRNAsidentifiziert. AnhandeinerClusteranalysederGene,dieineinerpubliziertenMYCNmiRNASignatur aufgedeckt wurden80, konnten Tumore des LSL‐MYCN;Dbh‐iCre Modells eindeutig von nicht‐malignen Nebennieren separiert werden, ein deutlicher Hinweis auf dieÄhnlich‐ keitderMYCNreguliertenmiRNAsinmurinenundhumanenTumoren. Die Regulation einzelner mRNAs der MYCN miRNA Signatur80 und weiterer bekannter miRNAZielevonMYCNinTumorendesLSL‐MYCN;Dbh‐iCreModellswurdenimFolgen‐ denmithumanenNeuroblastomenverglichen: TumoreausLSL‐MYCN;Dbh‐iCreMäusenwieseneineerhöhteExpressiondesmiR‐17‐92 Clusters auf. Für das humane Neuroblastom ist bekannt, dass dieses von c‐MYC und MYCN induzierte, onkogene Cluster, Targets mit Schlüsselrollen in der Kontrolle des Zellzyklus und der Apoptose reguliert159, wie zum Beispiel Bim59, E2F1159, p2159 und TGFβ61. Für TH‐MYCN Tumore ist die Hochregulation verschiedener miRNAs des miR‐17‐92Clustersebenfallsbeschrieben160. Im Vergleich zu Neuroblastomen mit günstigem Verlauf, überexprimieren sowohl Tu‐ moremitungünstigemVerlaufalsauchTumoremitMYCNAmplifikationdasonkogene miR‐181 Cluster80,110,112. Mestdagh et al. zeigen, dass miR‐181a zwar in MYCN‐ amplifizierten, abernicht in Tumoren miteinzelner MYCN Kopie hochreguliertist und dassMYCNdirektandenPromotorvonmiR‐181abindet80.AuchinLSL‐MYCN;Dbh‐iCre TumorenwarmiR‐181aüberexprimiert. Die miR‐184 ist herunterreguliert in MYCN‐amplifizierten Tumoren im Vergleich zu Tumoren mit nur einer Kopie von MYCN112. In den humanen Neuroblastomzelllinien KellyundSK‐N‐ASkorrelierteineektopischeExpressionvonmiR‐184mitderInhibie‐ rung des Überlebens und der Herunterregulation von AKT2, einem Mitglied des Phophatidylinositol‐3‐Kinase Signalweges, das eine wichtige Rolle für das Überleben vonverschiedenenTumorzellenspielt.EineÜberexpressionvonMYCNführtzurHerun‐ terregulation der tumorsuppressiven miR‐184112,113,161. Auch Tumore des LSL‐MYCN;Dbh‐iCre Modells wiesen eine Herunterregulation von miR‐184 auf, die vermutlichaufdieÜberexpressionvonMYCNzurückzuführenist. 101 Diskussion EineniedrigeExpressionvonmiR‐340istsignifikantmitMYCNAmplifikation,schlech‐ tem Ereignis‐freiem und Gesamtüberleben korreliert. Außerdem führt die Expression von miR‐340 in den humanen Neuroblastomzelllinien SK‐N‐BE, SH‐SY5Y und Kelly zu einer signifikanten Reduktion der Zellviabilität80,115. Tumore aus LSL‐MYCN;Dbh‐iCre MäusenzeigtenebenfallseinegesteigerteExpressionvonmiR‐340. Schulte et al. beschreiben eine signifikante Korrelation von niedriger miR‐542‐5p Ex‐ pression und hohem Stadium in primären Neuroblastomen. Des Weiteren zeigen sie eineAssoziationvonPatienten,dieeinenRückfallerleidenodereineMYCNAmplifika‐ tion haben und niedrigerer Expression dieser miRNA67. Sowohl miR‐542‐5p als auch miR‐542‐3pwareninLSL‐MYCN;Dbh‐iCreundTH‐MYCNTumorensignifikantherunter‐ reguliert. Sowohl miR‐26a und miR‐26b als auch miR‐30a, miR‐30d und miR‐30e und miR‐328 waren in Tumoren aus LSL‐MYCN;Dbh‐iCre Mäusen niedriger exprimiert als in nicht‐ malignenNebennieren.EineFunktiondiesermiRNAsinhumanenoderTH‐MYCNTumo‐ ren ist zwar noch nicht beschrieben worden, aber sowohl in MYCN‐amplifizierten hu‐ manenNeuroblastomenalsauchinmurinenTumorensindsieherunterreguliert80,160. Weitere miRNAs, die sowohl in humanen Neuroblastomen mit MYCN Amplifikation als auch in Tumoren des LSL‐MYCN;Dbh‐iCre Modells gleich reguliert werden, sind miR‐15b, miR‐30c, miR‐103, miR‐140, miR‐148a, miR‐326, miR‐491, miR‐500 und miR‐61580. Funktionen dieser miRNAs im Neuroblastom sind bisher jedoch nicht be‐ kannt. Sowohl miR‐149, miR‐214 und miR‐324‐5p als auch miR‐331 und miR‐488 sind in Tumoren des LSL‐MYCN;Dbh‐iCre Modells und humanen Neuroblastomen verschieden reguliert. Während humane Neuroblastome mit ungünstiger Prognose eine niedrigere miR‐149 und miR‐324‐5p Expression aufweisen als die mit günstiger Prognose110,112, exprimieren Tumore des LSL‐MYCN;Dbh‐iCre Modells diese miRNAs über. Tumore mit MYCN Amplifikation bzw. ungünstiger Prognose exprimieren miR‐214 über, was ver‐ mutlichaufdiedirekteBindungvonMYCNandenPromotordermiRNAberuht80,wäh‐ rend Tumore der LSL‐MYCN;Dbh‐iCre Maus eine Herunterregulation dieser miRNA aufweisen.HumaneNeuroblastomemitungünstigemVerlaufexprimierenimVergleich zu denen mit günstigem Verlauf niedrige Level der miRNAs miR‐331‐3p und miR‐488‐p110,währendTumoreausLSL‐MYCN;Dbh‐iCreMäusenbeidemiRNAsüberex‐ primieren. 102 Diskussion AusderAnalysederinTumorendesLSL‐MYCN;Dbh‐iCreModellsreguliertenmRNAund miRNA lässt sich zusammenfassen, dass ein großer Teil der im Modell regulierten mRNAsundmiRNAsTargetsvonMYC(N)sindunddassdiversemRNAsundmiRNAsim LSL‐MYCN;Dbh‐iCre Modell ähnlich reguliert sind wie im humanen Neuroblastom. Je‐ dochwurdenauchmRNAsundmiRNAsgefunden,dieimmurinenSystemandersregu‐ liert waren, als im humanen Neuroblastom, woran derUnterschied zwischen den Spe‐ zieserkennbarwird.EineweitermöglicheBegründungdafürkönntesein,dasssichdie miRNA‐Bindestellen mancher mRNAs zwischen den Spezies unterscheiden (www.targetscan.org),waszuverschiedenenRegulationenführt. EinevollständigeÜbertragungvonErkenntnissensowohlaufderEbenechromosomaler Aberrationen als auch auf mRNA und miRNA Level, die im LSL‐MYCN;Dbh‐iCre Modell gewonnen werden, auf das humane Neuroblastom ist demnach, wie in jedem anderen Mausmodell auch, nicht möglich. Jedoch kann das LSL‐MYCN;Dbh‐iCre Mausmodell genutztwerden,umneuepotentielltumormodulierendemRNAsundmiRNAszuidenti‐ fizieren. Ob diese auch bei der humanen Erkrankung eine Rolle spielen, muss dann weiteranalysiertwerden. 5.1.4 Mögliche Weiterentwicklungen des LSL‐MYCN;Dbh‐iCre Modells, die neue ErkenntnissefürdieForschungamhumanenNeuroblastomgebenkönnten Eine interessante Fragestellung betreffend der Neuroblastomentwicklung des LSL‐MYCN;Dbh‐iCreModells,istdienachderZeitspanne,inderdasTransgenexprimiert werden muss, damit es die Entstehung eines Tumors vermittelt. Dazu soll in späteren VersuchendieVerpaarungderLSL‐MYCNMausmiteinerinduzierbarenDbh‐iCreMaus durchgeführt werden. Stubbusch et al. generierten 2011 das Mausmodell Dbh‐CT, des‐ senCre‐Rekombinase‐Aktivitätabhängig voneiner Behandlung mitTamoxifenist. Wie beidemindieserArbeitverwendetenModellvonStankeetal.40stehtdieCre‐Rekombi‐ nase‐Expression unter Kontrolle des Dbh‐Promotors, jedoch handelt es sich um eine inaktive Form der Rekombinase, die nur in Anwesenheit von Tamoxifen aktiviert wird162. Mittels einer Tamoxifen‐Behandlung doppelt‐transgener LSL‐MYCN;Dbh‐CT MäusewährendihrerEntwicklungundzuverschiedenenZeitpunktenihresLebenssoll die Zeitspanne detektiert werden, während der eine MYCN Überexpression die Ent‐ wicklung eines Neuroblastoms vermittelt. Eine Übertragung auf den menschlichen 103 Diskussion Organismus könnte weitere Einblicke in die Entstehung des humanen Neuroblastoms gewähren. Anhand des LSL‐MYCN;Dbh‐iCre Mausmodells, dessen Tumore sich sowohl in ihren chromosomalenAberrationenalsauchihrenmiRNAundmRNARegulationenhumanen Neuroblastomen ähneln, können neue potentielle Targets einer medikamentösen The‐ rapiegefundenundgetestetwerden.DieReaktionderTumoreaufdieBehandlungmit nochnichtetabliertenMedikamentenkannwichtigeAufschlüsseaufdiehumaneThera‐ pierelevanzderMittelgeben. EinweitererinteressanterVersuchsansatzistdieVerkreuzungmitanderentransgenen Mausmodellen,diepotentiellfürdieNeuroblastomgeneserelevanteGeneregulieren. UnsereArbeitsgruppepublizierte2012dasshLsd1‐Mausmodell,dasDoxycyclin‐abhän‐ gig eine shRNA gegen Lsd1 (Kdm1a) exprimiert, die Kdm1a spezifisch herunterregu‐ liert163.DieVerpaarungdiesesModellsmitderLSL‐MYCN;Dbh‐iCreMausunddieInduk‐ tion des Kdm1a Knockdowns könnte ein interessanter Ansatz zur Untersuchung der RollevonKdm1abeiderTumorgenesedesNeuroblastomssein.Eswirdvermutet,dass der Knockdown von Kdm1a, aufgrund seiner onkogenen Funktion35,118, das Wachstum der MYCN‐vermittelten murinen Neuroblastome verlangsamt. Weiterhin könnte der VergleichdermolekularenEigenschaftenvonDoxycyclin‐behandeltenundnicht‐behan‐ deltenTumorenneueEinblickeindenSignalwegvonKdm1ageben. Auch die Verkreuzung mit Mausmodellen, die miRNAs regulieren, ist ein interessanter Versuchsansatz. Eine miR‐17‐92‐Maus, die die miRNAs dieses Clusters Cre‐abhängig deletiert164,stehtunsererArbeitsgruppezurVerfügung.EineHerunterregulationdieses ClustersindenZellendesLSL‐MYCN;Dbh‐iCreModells,diehumanesMYCNexprimieren, könntedieTumorgeneseverlangsamenodersogarunterbinden,schließlichwirddiesen onkogenenmiRNAseinewichtigeFunktionimNeuroblastomzugeschrieben59,61.Weiter‐ hin könnten unbekannte Targets der miRNAs dieses Clusters anhand von Exon‐Arrays entdeckt werden, die nicht nur im Mausmodell sondern auch im humanen Neu‐ roblastomeineRollebeiderFunktiondiesermiRNAsspielenkönnten. 104 Diskussion Die Etablierung einerZelllinie aus Tumorendes LSL‐MYCN;Dbh‐iCre Modells kannvon großer Bedeutung für die Arbeit mit diesem Modell sein. Potentiell wirksame Medika‐ mente könnten vor dem in vivo Experiment an den Zellen in vitro getestet werden, wodurch Tierexperimente eingeschränkt würden. Im Verlauf dieser Arbeit ist es nicht gelungeneineZellliniedesLSL‐MYCN;Dbh‐iCreModellszuetablieren. 5.1.5 Die Relevanz des LSL‐MYCN Mausmodells für die Genese anderer Tumorentitäten Neben der Verpaarung mit der Dbh‐iCre Maus, wurden LSL‐MYCN Tiere mit Alb‐iCre Mäusengekreuzt.Doppelt‐transgeneNachkommenentwickeltenimAltervon246‐286 Tagen mit einer Inzidenz von 86% Tumore, die pathologisch als Leberzellkarzinome beschrieben wurden (Daten nicht gezeigt). Somit konnte gezeigt werden, dass die Ex‐ pression von humanem MYCN in spezifischen Geweben die Entwicklung von weiteren Tumormodellen treiben kann. Besonders für Tumorentitäten, für die noch keine pas‐ sendenMausmodelleexistieren,wäredieseininteressanterAspektfürdieForschung. 105 Disskussion 5.2 T Tumorsu uppresssivemiR RNAsim mNeurob blastom m In versschiedenen Krebsform men wurdeen sowohl miRNAs entdeckt, e ddie überexp primiert sind un nd als onko ogene Regulatoren fu ungieren als a auch miiRNAs, die herunterrreguliert sindun ndalsTumo orsuppresssorenagierren53,57,165,166.Auchim mNeuroblasstomsindm miRNAs beschriieben, die onkogene e oder tum morsuppressive Funk ktionen auufweisen65,,167. Die Expresssion einigeer miRNAs ist in versschiedenen n Neuroblastom Subtyypen unterrschied‐ lich regguliert und d auf Basiss der Expreessionsmuster dieserr miRNAs iist es mög glich die verschiiedenenSu ubgruppen zuidentifizzieren67,1122,116.Demna achkönnteenmiRNAssowohl alsproggnostischeMarkereingesetztw werden,als auchisteiinemiRNA Agerichtete eThera‐ pie,diee,imFallvo ontumorsu uppressiveenmiRNAs,,derenExp pressionerrhöhtoder,,imFall vononk kogenenm miRNAs,derrenExpresssionhemm mt,denkbar116. Im Verrgleich zurr klassischen Chemo therapie, kann k eine zielgericht htete Thera apie die Möglich hkeitgeben nspezifisch hgegenSu ubpopulatio onenvonZ Zellenvorzuugehenunddamit die toxxischen Wirkungen gegen g den Gesamtorg ganismus einzuschrän e nken168. Demnach sind W Wirkstoffe mit m hoher Affinität A un nd Spezifitäät von groß ßer Bedeuttung169. Wiirkstoffe mitderrartigspeziifischerWiirkungbrin ngenjedoch hhäufigRü ückfälleunndResisten nzenmit 1 .MiRNA‐basierteT sich,daaalternativ veSignalwe egeaktivie rtwerden170 Therapienrreduzie‐ renmittgroßerW WahrscheinllichkeitimVergleich zukonven ntionalenuundzielgeriichteten Medikaamenten die d Entwiccklung voon Resisteenzen171. Verschiede V ene Publik kationen beschreeiben die Möglichkei M it einer in vivo Behaandlung miit miRNA‐IInhibitoren n oder ‐ Imitatio onen. Die intratumo orale Injekt ktion von miR‐17‐5p m ‐Inhibitoreen führt zu z einer signifik kantenVerzzögerungd desWachsttumseines XenograftsausLAN‐‐5Zellen59.Tivnan . et al. d demonstrieren, dass eine e zielge richtete Eiinbringung g von miR‐‐34a in Xen nografts ausNeu uroblastom mzellenmitttelsGD2‐kkonjugierten nNanoparttikelnApopptoseeinle eitetund Angiogeenese verm mindert172. Aufgrund dieser Erk kenntnisse kann es vvon großer Bedeu‐ tungseein,neuemiRNAszuidentifiziereen,dieonk kogeneodertumorsupppressiveF Funktio‐ nenfürrverschiedeeneTumorrentitätenh haben. 106 Diskussion 5.2.1 DietumorsuppressiveFunktionvonmiR‐542imNeuroblastom Aufgrund der niedrigen Expression von miR‐542‐3p in murinen Neuroblastomen des neuenLSL‐MYCN;Dbh‐iCreMausmodellswurdevermutet,dassmiR‐542‐3peinetumor‐ suppressiveFunktionimNeuroblastomhabenkönnte.EinesignifikanteHerunterregula‐ tionvonmiR‐542‐3pinhumanenNeuroblastomenmitschlechterPrognoseimVergleich zuTumorenmitguterPrognosebestätigtedieseThese. AnhandvoninvitroAnalyseninetabliertenNeuroblastomzelllinienwurdederNachweis erbracht,dasseineexogeneExpressionvonmiR‐542‐3psowohlApoptoseinduziertals auch die Viabilität und Proliferation der Zellen reduziert. Weiterhin konnte gezeigt werden, dass die Expression dieser miRNA einen Arrest der Zellen in der S‐Phase des Zellzyklus mit sich bringt. Dahingegen resultierte die exogene Expression der 5p‐Isof‐ ormdermiR‐542nurbeieinerderanalysiertenZelllinienineinerReduktionderZellvi‐ abilitätund‐proliferation.WederApoptosenocheinArrestimZellzykluswurdeinden Zelllinien ausgelöst. Dieses Resultat unterstützt die Publikation von Bray etal., dass miR‐542‐5p keinen Effekt auf die Proliferation der Neuroblastomzelllinien Kelly, NB1691 und SK‐N‐AS hat, aber sowohl in Kelly als auch in SK‐N‐AS Zellen zu einer verringerten Invasivität durch eine Matrigel‐Matrix führt68. Entgegen der allgemeinen Annahme, dass die 5p‐Isoform von miRNAs diejenige ist, die den biologischen Effekt ausmacht, wurde in dieser Arbeit gezeigt, dass im Fall der miR‐542 die 3p‐Form als tumorsuppressivfungiert,währenddie5p‐FormkeineneindeutigenEffektzeigt51,52. Yoon et al. publizierten, dass miR‐542‐3p das vorhergesagte Target Survivin in A549 und MCF7 Zellen direkt herunterreguliert69. Im Rahmen dieser Arbeit konnte gezeigt werden, dass in primären humanen Neuroblastomen eine niedrige Expression von SurvivinzumeinenmiteinergesteigertenExpressionvonmiR‐542‐3pundzumanderen einer besseren Prognose für den Patienten einhergeht, wobei die Korrelation mit der Prognosebekanntist173,174.WeiterhinwareineexogeneExpressionvonmiR‐542‐3pin NeuroblastomzelllinienmiteinerHerunterregulationvonSurvivinkorreliert.Außerdem führtediesermiR‐542‐3p‐vermittelteKnockdownvonSurvivinzueinerVerminderung derExpressionvonAURKB,demKomplexpartnervonSurvivin.Esistbekannt,dassdie EntfernungvonSurvivinmitFehllokalisationundverminderterAktivitätvonAURKBin Zusammenhangsteht.AußerdemführtderKnockdownvonSurvivinzuFehlernbeider Zytokinese, die auf die verminderte Aktivität von AURKB zurückgeführt werden175‐177. InteressanterweisewirdbeiderdurchdiePolo‐like‐Kinase1‐vermitteltenPhosphoylie‐ rung von Survivin zuerst Survivin und dann AURKB aktiviert178. Wie schon für A549 107 Diskussion Zellenpubliziert69,wurdeanhandvonLuziferaseReporterAssayseinedirekteBindung dermiR‐542‐3pandieWildtyp3’UTRvonSurvivin,sowohlinfürdiesenAssayetablier‐ ten HEK‐293 als auch in SHEP Zellen, nachgewiesen, während dieser Effekt mit einer mutiertenBindestellenichterreichtwurde. In Neuroblastomzelllinien führte ein Knockdown von Survivin zu einer verminderten Zellviabilitätund‐proliferation,einEffekt,derauchdurchExpressionvonmiR‐542‐3p erreichtwurde.BeiderUntersuchungdesZellzyklusreagiertendreivonvierZelllinien beiKnockdownvonSurvivinmiteinemArrestinderPhaseS,währendeineZellliniemit einemArrestinderG2/M‐Phaseantwortete. Die herausragende Bedeutung von Survivin für die tumorsuppressive Funktion der miR‐542‐3p im Neuroblastomzelllinien, konnte anhand eines Rettungsexperimentes bewiesenwerden.DieÜberexpressionvonSurvivincDNAohnemiR‐542‐3p‐Bindestelle rettetedieNeuroblastomzelllinieSHEPgrößtenteilsvordemPhänotypdermiR‐542‐3p Expression. Daraus lässt sich schließen, dass es einen Zusammenhang zwischen dem Phänotyp der miR‐542‐3p Expression und der Survivin Regulation gibt. Jedoch ist es möglich, dass die Regulation von anderen Zielen dieser miRNA ebenfalls eine funktio‐ nelle Auswirkung auf den Phänotyp humaner Neuroblastomzellen haben könnte. In zusätzlichenExperimenten,diebeimVerfassendieserArbeitnochnichtabgeschlossen waren, sollten weitere von miR‐542‐3p regulierte Targets gefunden werden. Dazu werden mRNA‐Expressionsprofile von mit Negativkontroll‐miRNA oder miR‐542‐3p transfiziertenNeuroblastomzelllinienmiteinanderverglichen.Weiterepotentiellfunkti‐ onelle Targets der miRNA, die dabei evtl. gefunden werden, sollen dann anhand von weitereninvitroStudienvalidiertwerden. Transfektionsreagenzien werden benutzt, um miRNAs in vitro in Zellen einzubringen. Für eine Behandlung in vivo müssen miRNAs vor ihrem Abbau durch Nukleasen ge‐ schütztwerden.DieArbeitsgruppeEppleanderUniversitätDuisburg‐Essenentwickelte Calcium‐Phosphat‐Nanopartikel,mitderenHilfesiesiRNAsinvitroinZelleneinbringen konnten,waszueinerHerunterregulationdessiRNATargetsführte84.Dabeiwerdenmit siRNA‐beladene Calcium‐Phosphat‐Nanopartikel durch Endozytose in die Zelle aufge‐ nommen.ImZytoplasmaschütztderNanopartikeldieNukleinsäurevorihremAbbau.Es istbekannt,dassdasEindringenderNukleinsäureindenZellkernüberKernporenkom‐ plexevermitteltwird,aberesistnochunklar,obderNanopartikeldiesenWegebenfalls nimmt oder im Zytoplasma verbleibt. Im Rahmen dieser Arbeit sollten mit miRNA‐ beladeneCalcium‐Phosphat‐NanopartikelfürdieUntersuchungdertumorsuppressiven 108 Diskussion FunktionvonmiR‐542‐3pinvivoverwendetwerden.ZuerstwurdederenFunktionalität invitroanWACIIZellenvalidiert.ImVergleichzurunbehandeltenKontrollereagierten mit Negativkontroll‐miRNA beladenen Nanopartikeln behandelte Zellen mit einer ten‐ denziellen,abernichtsignifikantenReduktionvonZellviabilitätsowie‐proliferationund mit einer signifikanten Induktion von Apoptose. Demnach schienen die verwendeten Calcium‐Phosphat‐Nanopartikel allgemein einen toxischen Effekt auf WACII Zellen zu induzieren.EineToxizitätaufdieZellviabilitätistinHeLaZellennichtbekannt179.Auf‐ grund der apoptotischen Wirkung von PEI auf verschiedene humane Zelllinien180,181, wurde vermutet, dass auch in WACII Zellen das in den Nanopartikeln vorhandene PEI derenToxizitätausmacht.TrotzdeminduzierteeineBehandlungvonWACIIZellenmit miR‐542‐3pbeladenenNanopartikelnimVergleichzuunbehandeltenundmitNegativ‐ kontroll‐miRNAbehandeltenZellensignifikantsowohleineReduktionderZellviabilität und‐proliferationalsaucheineSteigerungderApoptose.AufBasisdieserTatsacheund des Nachweises, dass miR‐542‐3p‐beladene Nanopartikel die Survivin Protein Expres‐ sion in WACII Zellen reduzieren, wurde die Spezifität der mit miR‐542‐3p‐beladenen Nanopartikel bestätigt. Weiterhin wurden Neuroblastom‐Xenografts‐tragende Mäuse mit miR‐542‐3p beladenen Nanopartikeln über zwei Tage behandelt. Sowohl eine er‐ höhteExpressionvonmiR‐542‐3palsaucheineReduktionvonSurvivinProteinkonnte in den Tumoren aus mit miR‐542‐3p behandelten Mäusen im Vergleich zu den mit Negativkontroll‐miRNAbehandeltenvalidiertwerden,wasdieSpezifitätderNanoparti‐ kel auch in vivo bestätigte. Die signifikante Reduktion von Proliferation und Induktion von Apoptose in diesen Tumoren wies darauf hin, dass der miR‐542‐3p‐vermittelte KnockdownvonSurvivinauchinvivofunktionellwar.DieProduktionvonmiRNA‐bela‐ denenNanopartikelnistbiszumheutigenZeitpunktnochineffektiv,weshalbeineLang‐ zeitstudie zum Effekt der Behandlung auf die Volumenveränderung der Tumore im RahmendieserArbeitnichtdurchgeführtwerdenkonnte. AufgrundderhohenÜberexpressionvonSurvivininverschiedenenTumorentitätenund derniedrigenExpressioninnormalenGewebenwirdSurvivinsowohlalsTumormarker alsauchalsTargetfürdieNeuroblastomtherapiediskutiert182,183.VerschiedeneGruppen beschreiben Medikamente, die die Expression von Survivin in Neuroblastomzellen reduzieren. Demnach ist die proapoptotische Antwort der Neuroblastomzelllinien SK‐SY5Y und LA1‐5S auf Noscapine vermittelt durch die Unterdrückung der Survivin Protein Expression184. Interessanterweise potenziert ein Survivin Knockdown in SK‐ N‐BE2 und SH‐SY5Y Zellen den Effekt von Epigallocatechingallat185, einem 109 Diskussion CarbonsäureesterderGallussäure,deringrünemTeevorkommtunddemapoptotische Wirkung in verschiedenen neuroektrodermalen Tumoren nachgewiesen wurde186. Da ein direkter Knockdown von Survivin in vivo nicht ausführbar ist, könnte hier eine Kombination aus miR‐542‐3p‐beladenen Nanopartikeln und Epigallocatechingallat getestetwerden.YM155,einnieder‐molekularesPräparatzurRepressionvonSurvivin, wurde2007vonNakaharaetal.beschrieben.DieSubstanzsupprimiertdieExpression von Survivin, induziert Apoptose von Prostatakarzinomzelllinien PC‐3 und PPC‐1 in vitro und induziert Tumor‐Regression von Xenografts aus PC‐3 Zellen187. Lamers et al. untersuchten die Effizienz von YM155 in 23 Neuroblastomzelllinien, die auf einen shRNA‐vermittelten Knockdown von Survivin mit massiver Apoptose reagierten. Neun Zelllinien wiesen eine Resistenz gegen YM155 auf, die mit hoher Expression des Mul‐ tidrug‐ResistanceProtein1(MDR1)korrelierte74.UnterdenresistentenZelllinienbefin‐ det sich unter anderen SK‐N‐BE, die wie in dieser Arbeit gezeigt wurde, auf die miR‐542‐3p‐vermittelte Repression von Survivin sehr effizient reagiert. In weiteren StudiensolltedieEffektivitätdermiR‐542‐3pExpressionaufweitereNeuroblastomzell‐ liniengetestetwerden,davermutetwird,dassdertumorsuppressiveEffektdermiRNA nichtdurchResistenzentwicklungaufgehobenwerdenkann. Sobald die Effizienz der Nanopartikelherstellung erweitert und damit effektiver ist, könnte eine Nanopartikel‐basierte Therapie mit ausgewählten miRNAs, wie der miR‐542‐3p, in Zukunft ein neuer Ansatz für die Behandlung humaner Neuroblastome undandererTumorentitätensein. 110 Diskussion 5.2.2 DietumorsuppressiveFunktionvonmiR‐137imhumanenNeuroblastom118 SowohlimneuenLSL‐MYCN;Dbh‐iCrealsauchimetabliertenTH‐MYCNMausmodellfür dasNeuroblastomwarKdm1aimVergleichzunicht‐malignenNebennierensignifikant hochreguliert. Auf Basis dieser Erkenntnis wurde KDM1A als potentielles Ziel einer Neuroblastomtherapie gehandelt. In verschiedenen Tumorentitäten wie Brustkrebs33, kleinzelligem Lungenkarzinom, Kolorektal‐, Blasen‐ und Prostatakrebs188 ist KDM1A überexprimiert. Auch im humanen Neuroblastom ist eine erhöhte Expression im Ver‐ gleich von undifferenzierten zu differenzierten Tumoren bekannt35. Weiterhin ist KDM1AinnerhalbderneuroblastischenTumoreimNeuroblastomhöherexprimiertals imgutartigenGlanglioneuroblastom189.DieEffektivitätverschiedenernieder‐molekula‐ rer Inhibitoren gegen Monoaminoxidasen wurden auf ihre Wirkung gegen KDM1A in NeuroblastomzelllinienSHEP,SH‐SY5YundLAN‐1getestet,jedochwareinEffektaufdie ViabilitätderZellennurinvitro,nichtaberiminvivoXenograft‐Modellnachweisbar35. DieEntwicklungvonneuenspezifischenKDM1AInhibitorenwirdalsKDM1A‐gerichtete TherapiegegendasNeuroblastomdiskutiert35,190. EinemöglicheVerminderungderKDM1AExpressionkönnteauchdurchdieExpression einermiRNAerreichtwerden,diedirektandie3’UTRvonKDM1Abindet.Einepotentiell tumorsuppressive Funktion, die eine Bindestelle in der 3’UTR von humanem KDM1A aufweist, war miR‐137. In murinen LSL‐MYCN;Dbh‐iCre Tumoren war diese miRNA hochreguliert, was die These einer tumorsuppressiven miRNA nicht unterstützte. Am murinen Kdm1a befindet sich jedoch keine Bindestelle für diese miRNA (www.targetscan.org),weshalbdieseRegulationsachseimmurinenSystemkeineRolle spielt.ImhumanenNeuroblastomwurdedieFunktionvonmiR‐137imweiterenVerlauf derAnalysenuntersucht. Eine niedrige Expression von miR‐137 ging sowohl mit ungünstigem Verlauf als auch mit niedrigerem Überleben von Neuroblastompatienten einher. In vitro reduzierte die exogene Expression der miRNA sowohl Zellviabilität und ‐proliferation als auch indu‐ zierte sie Apoptose, Caspase 9 Aktivität sowie neuronale Differenzierung der Neuro‐ blastomzelllinienIMR‐32,SHEPundSK‐N‐BE.WeiterhinwurdeKDM1Adirektdurchdie ExpressionmiR‐137herunterreguliert,wodurchdasKDM1AZielTFPI2ebenfallsregu‐ liertwurde.FürmiR‐137sindverschiedenemRNAZielevorhergesagt,weshalbsichdie Frage stellte, welcher Teil des Effektes von miR‐137 auf die Regulation von KDM1A zurückgingundwelcheraufdenKnockdownanderermiR‐137Ziele.EinRettungsexpe‐ rimentzeigte,dassderEffektdermiR‐137ExpressionteilweiseaufdieRegulationvon 111 Diskussion KDM1Azurückzuführenwar,jedochandereZieledermiRNAebenfallseinefunktionelle Rollespielenmussten.WeiterhinerklärtedasModelldiephänotypischenUnterschiede zwischenderExpressionvonmiR‐137unddesKnockdownvonKDM1AmittelssiRNA, wiezumBeispieldieEinleitungvonApoptoseoderderBlockderProliferationinIMR‐32 Zellen. AnhandderdurchgeführtenProteomanalysewurdeAKAP2alsweiterespotentiellfunk‐ tionelles Target der miR‐137 gefunden. Dieses Protein, welches vier Bindestellen für miR‐137 aufweist, separiert die Prognose von Neuroblastompatienten anhand seiner Expression in den Tumoren, was eine onkogene Funktion vermutet lässt. Der Knock‐ down von AKAP2 mittels siRNA kopierte den Phänotyp der miR‐137 Expression. Im Rahmen dieser Arbeit wurde die Bedeutung der miR‐137‐vermittelten Regulation von AKAP2nichtweiteruntersuchtunddiedirekteBindungdermiRNAandievorhergesag‐ ten Bindestellen bisher nicht validiert. In weiteren Experimenten sollte AKAP2 als po‐ tentiellfunktionellesTargetdermiR‐137näheruntersuchtwerden.BiszudiesemZeit‐ punktistnochkeineFunktionvonAKAP2inKrebserkrankungenbeschrieben. InteressanterweiseistKDM1AauchinneuralenVorläuferzellenexprimiertundintera‐ giert hier mit dem Transkriptionsfaktor NR2E1. Dieser nukleare Rezeptor wird aus‐ schließlichimembryonalenNeuroepitheliumexprimiertundregulierthierdieExpres‐ sionvonmiR‐137.KDM1AgiltalstranskriptionalerKo‐RepressorvonNR2E1undeine erhöhteExpressionvonmiR‐137inneuralenStammzellenführtzureduzierterProlife‐ ration und induziert neuronale Differenzierung191. Die Expression von miR‐137 ist im GlioblastomsignifikantniedrigeralsimGehirnundinneuralenStammzellen.Weiterhin unterstützteineÜberexpressiondermiRNAneuronaleDifferenzierunginGlioblastom‐ zellen. In diesen Zellen wird ein weiteres Zielgen der miR‐137, nämlich RTVP‐1, be‐ schrieben, das direkt von miR‐137 gebunden wird und dessen Herunterregulation tumorsuppressiveWirkunghat192.Lietal.demonstrierten,dassdieExpressionvonmiR‐ 137dasZellwachstumsowiedieInvasionderGlioblastomzellenU87undU373inhibiert und die Expression von CSE1L, einem weiteren vorhergesagten Target dieser miRNA, reguliert193.WeiterhinsupprimiertmiR‐137dieInvasionvonKolorektalkarzinomzellen durchdiedirekteBindunganFMNL2120.AndersrumführtdieSuppressionvonmiR‐137 inKolorektalkarzinomzellenzueinerÜberexpressionvonPaxillin(PXN)undzurInhibi‐ tionderProliferation,MigrationundInvasionderZellen119.InBrustkrebszellenistdie 112 Diskussion Expression von miR‐137 invers mit der von YB‐1 korreliert, einem weiteren Target diesermiRNA194.SowohlCDC42alsauchCDK6sindbeschriebeneTargetsvonmiR‐137 in Lungenkrebszellen, deren Proliferation durch die Expression von miR‐137 inhibiert wird121.DieseundweiterePublikationenunterstützendieTheseeinertumorsuppressi‐ ven Funktion von miR‐137 in verschiedenen Tumorentitäten, die in dieser Arbeit an‐ handdesNeuroblastomsgezeigtwurde.DiebeschriebenenTargetsdermiRNAkönnten auch im humanen Neuroblastom die tumorsuppressive Funktion von miR‐137 weiter erklären. 113 Zusammenfassungund Ausblick 6 ZusammenfassungundAusblick Das Neuroblastom ist der häufigste solide Tumor des Kindesalters und verantwortlich fürca.15%allerpädiatrisch‐onkologischenTodesfälle.EineAmplifikationdesOnkogens MYCN,durchwelchedieprognostischungünstigstePatientengruppedefiniertist,liegtin 20%allerNeuroblastomevor.ImRahmendieserArbeitwurdeeinneuesCre‐konditio‐ nales MYCN‐vermitteltes Mausmodell (LSL‐MYCN) entwickelt. Durch die Kreuzung mit Dbh‐iCre Mäusen wurde die Expression des MYCN Transgens auf Dopamin‐‐Hydro‐ xylaseexprimierendeZellenbeschränkt,waszurEntwicklungvonTumorenderNeben‐ nieren, der oberen Zervikalganglien und des Ganglion coeliacum führte. Die Tumore wiesensowohldiefürdasNeuroblastomspezifischenkleinen,blauenundrundenZellen alsauchneurosekretorischeVesikelauf.WeiterhinkonnteeinesignifikanteÜberexpres‐ sion von humanem MYCN und spezifischer Neuroblastommarker detektiert werden. Genomische Aberrationen sowie mRNA und mikroRNA (miRNA) Expressions‐Profile entsprachendenenhumanerNeuroblastome. MiRNAs sind kurze, nicht‐kodierende RNA Moleküle, die durch Bindung an mRNAs derenStabilitätundTranslationregulieren.DieBedeutungvonmiRNAsinderTumorbi‐ ologie ergibt sich aus der Regulation von Onkogenen und Tumorsuppressorgenen. In humanenprimärenNeuroblastomenwardieniedrigeExpressiondermiRNAsmiR‐542‐ 3pundmiR‐137mitungünstigerPrognoseassoziiertundeineHerunterregulationvon miR‐542‐3pkonnteauchinTumorendesLSL‐MYCN;Dbh‐iCreModellsbeobachtetwer‐ den. Die Funktion beider miRNAs wurde in vitro in präklinischen Modellsystemen des humanen Neuroblastoms detailliert untersucht. Die ektope Expression der miRNAs reduziertedieZellviabilitätundProliferation,erhöhtedieApoptoserateundinduzierte einen Zellzyklusarrest. Weiterhin wurde die direkte Bindung der vorhergesagten Ziel‐ mRNAsKDM1A(miR‐137)undSurvivin(miR‐542‐3p)mittelsLuziferaseReporterAna‐ lysen validiert und die Regulation sowohl auf mRNA‐ als auch auf Proteinebene verifi‐ ziert.DieHerunterregulationderZielgeneinNeuroblastomzellenstellteeinePhänoko‐ piederEffekteeinerexogenenExpressionderjeweiligenmiRNAdar.DieSpezifitätder miRNA‐vermittelten Regulation wurde durch ektope Expression des Zielgens mit mu‐ tierter miRNA‐Bindestelle nachgewiesen. Eine Therapiesimulation in Neuroblastom‐ Xenografts mit miR‐542‐3p‐beladenen Nanopartikeln zeigte sowohl eine Herunterre‐ 114 Zusammenfassungund Ausblick gulation des Zielproteins Survivin als auch eine Reduktion derProliferation und Erhö‐ hungderApoptoserateindenTumorzellen. DieEtablierungdesneuenLSL‐MYCN;Dbh‐iCreNeuroblastom‐Mausmodellskanndabei helfensowohldiePathogenesedesNeuroblastomsbesserzuverstehenalsauchpotenti‐ ellNeuroblastom‐relevanteGeneundmiRNAszuidentifizieren.EineWeiterentwicklung dieses Modells durch Kombination mit anderen Mausmodellen, die potentiell Neuro‐ blastom‐relevante Gene regulieren, könnte im Zusammenspiel mit neuen Behand‐ lungsstrategien Einblicke in die Wirksamkeit von künftigen Neuroblastomtherapien geben. Die Behandlung humaner Neuroblastompatienten mit tumorsuppressiven miRNAs,wiemiR‐542‐3pundmiR‐137,könnteeinmöglicherAnsatzseindenaggressi‐ venVerlaufdieserErkrankungzureduzieren. 115 Abstract 7 Abstract Neuroblastomaisacommonsolidtumorofinfancyandaccountsformorethan15%of pediatric cancer deaths. Amplification of MYCN oncogene defines the most aggressive subtypeofneuroblastomawithpoorprognosis.Inthepresentstudy,anewCre‐condi‐ tionalMYCN‐driventransgenicmousemodelwasgeneratedtoanalysetumorigenesisin vivo.WhentargetingMYCNexpressiontotheneuralcrestintransgenicLSL‐MYCN;Dbh‐ iCremice,palpabletumorsdeveloped,whicharosepredominantlyfrombothadrenals, but also from superior cervical ganglia and ganglion coeliacum. LSL‐MYCN;Dbh‐iCre‐ induced tumors consisted of small round blue cells and possessed neurosecretory vesicles.ExpressionofhumanMYCNandspecificneuroblastomamarkergeneswashigh intumors.Furthermore,murineneuroblastomasrecapitulatedgenomicaberrationsand MYCNmRNAaswellasmicroRNA(miRNA)signaturesdescribedforhumanneuroblas‐ toma.MiRNAsaresmallnoncodingRNAmoleculesthatbindtargetmRNAsresultingin translational repression or degradation of the mRNA. In cancer, they can act as tumor suppressors or oncogenes. In human primary neuroblastomas, low expression of miR‐ 542‐pandmiR‐137washighlycorrelatedwithunfavourableprognosisandmiR‐542‐3p wasupregulatedinmurineLSL‐MYCN;Dbh‐iCretumors.Exogenousexpressionofthese miRNAs in established neuroblastoma cell lines reduced viability and proliferation, increased apoptosis and induced cell cycle arrest. Furthermore, direct interaction be‐ tweenthepredictedtargetssurvivin(miR‐542‐3p)andKDM1A(miR‐137)wasvalidated by luciferase reporter assays and regulation was demonstrated on mRNA as well as protein level. Ectopic expression of miR‐542‐3p or miR‐137 phenocopied the knock‐ down of respective targets. In addition, we were able to validate a direct interaction between miRNAs and their targets by performing rescue experiments. Treatment of micebearingneuroblastomaxenograftswithmiR‐542‐3p‐loadednanoparticlesresulted in downregulation of survivin and induced reduced tumor cell proliferation as well as increasedapoptosis. Inconclusion,thenovelLSL‐MYCN;Dbh‐iCreneuroblastomamousemodelcontributeto abetterunderstandingofMYCN‐drivenneuroblastomaandtoidentificationofrelevant genes and miRNAs. Combination with other transgenic models, regulating potential relevantgenes,couldprovidenewinsightsintoneuroblastomatumorigenesis.Restoring functionoftumorsuppressivemiRNAs,suchasmiR‐542‐3pormiR‐137,couldrepresent afeasibleapproachtoreduceneuroblastomaaggressiveness. 116 Literatur 8 Literatur 1 2 3 4 5 6 7 8 9 10 11 12 13 14 Pizzo, P. & Poplack, D. Principles and practice of pediatric oncology. 6th ed. edn, (LippincottWilliams&Wilkins,2011). Schwab,M.,Westermann,F.,Hero,B.&Berthold,F.Neuroblastoma:biologyand molecularandchromosomalpathology.Thelancetoncology4,472‐480(2003). Matthay, K. K., George, R. E. & Yu, A. L. Promising therapeutic targets in neuroblastoma. Clinical cancer research : an official journal of the American AssociationforCancerResearch18,2740‐2753,doi:10.1158/1078‐0432.CCR‐11‐ 1939(2012). Maris, J. M. Recent advances in neuroblastoma. The New England journal of medicine362,2202‐2211,doi:10.1056/NEJMra0804577(2010). Brodeur, G. M., Seeger, R. C., Barrett, A., Berthold, F., Castleberry, R. P. et al. Internationalcriteriafordiagnosis,staging,andresponsetotreatmentinpatients withneuroblastoma.Journalofclinicaloncology:officialjournaloftheAmerican SocietyofClinicalOncology6,1874‐1881(1988). Brodeur,G.M.,Pritchard,J.,Berthold,F.,Carlsen,N.L.,Castel,V.etal.Revisionsof the international criteria for neuroblastoma diagnosis, staging, and response to treatment.Journalofclinicaloncology:officialjournaloftheAmericanSocietyof ClinicalOncology11,1466‐1477(1993). (2004). Spix, C., Pastore, G., Sankila, R., Stiller, C. A. & Steliarova‐Foucher, E. NeuroblastomaincidenceandsurvivalinEuropeanchildren(1978‐1997):report from the Automated Childhood Cancer Information System project. European journalofcancer42,2081‐2091,doi:10.1016/j.ejca.2006.05.008(2006). Ceschel, S., Casotto, V., Valsecchi, M. G., Tamaro, P., Jankovic, M. et al. Survival afterrelapseinchildrenwithsolidtumors:afollow‐upstudyfromtheItalianoff‐ therapy registry. Pediatric blood & cancer 47, 560‐566, doi:10.1002/pbc.20726 (2006). Simon,T.,Berthold,F.,Borkhardt,A.,Kremens,B.,DeCarolis,B.etal.Treatment and outcomes of patients with relapsed, high‐risk neuroblastoma: results of German trials. Pediatric blood & cancer 56, 578‐583, doi:10.1002/pbc.22693 (2011). Cotterill, S. J., Pearson, A. D., Pritchard, J., Foot, A. B., Roald, B. et al. Clinical prognosticfactorsin1277patientswithneuroblastoma:resultsofTheEuropean NeuroblastomaStudyGroup'Survey'1982‐1992.Europeanjournalofcancer36, 901‐908(2000). Hann, H. W., Evans, A. E., Siegel, S. E., Wong, K. Y., Sather, H. et al. Prognostic importanceofserumferritininpatientswithStagesIIIandIVneuroblastoma:the Childrens Cancer Study Group experience. Cancer research 45, 2843‐2848 (1985). Shuster, J. J., McWilliams, N. B., Castleberry, R., Nitschke, R., Smith, E. I. et al. Serumlactatedehydrogenaseinchildhoodneuroblastoma.APediatricOncology Grouprecursivepartitioningstudy.Americanjournalofclinicaloncology15,295‐ 303(1992). Brodeur, G. M., Seeger, R. C., Schwab, M., Varmus, H. E. & Bishop, J. M. Amplification of N‐myc in untreated human neuroblastomas correlates with advanceddiseasestage.Science224,1121‐1124(1984). 117 15 16 17 18 19 20 21 22 23 24 25 26 27 28 Literatur Canete,A.,Gerrard,M.,Rubie,H.,Castel,V.,DiCataldo,A.etal.Poorsurvivalfor infants with MYCN‐amplified metastatic neuroblastoma despite intensified treatment: the International Society of Paediatric Oncology European Neuroblastoma Experience. Journal of clinical oncology : official journal of the American Society of Clinical Oncology 27, 1014‐1019, doi:10.1200/JCO.2007.14.5839(2009). Pugh,T.J.,Morozova,O.,Attiyeh,E.F.,Asgharzadeh,S.,Wei,J.S.etal.Thegenetic landscape of high‐risk neuroblastoma. Nature genetics 45, 279‐284, doi:10.1038/ng.2529(2013). Amati,B.&Land,H.Myc‐Max‐Mad:atranscriptionfactornetworkcontrollingcell cycle progression, differentiation and death. Current opinion in genetics & development4,102‐108(1994). Shimada, H., Nakagawa, A., Peters, J., Wang, H., Wakamatsu, P. K. et al. TrkA expression in peripheral neuroblastic tumors: prognostic significance and biologicalrelevance.Cancer101,1873‐1881,doi:10.1002/cncr.20557(2004). Nakagawara, A., Azar, C. G., Scavarda, N. J. & Brodeur, G. M. Expression and function of TRK‐B and BDNF in human neuroblastomas. Molecular and cellular biology14,759‐767(1994). Chen,Y.,Takita,J.,Choi,Y.L.,Kato,M.,Ohira,M.etal.OncogenicmutationsofALK kinase in neuroblastoma. Nature 455, 971‐974, doi:10.1038/nature07399 (2008). DeBrouwer,S.,DePreter,K.,Kumps,C.,Zabrocki,P.,Porcu,M.etal.Meta‐analysis of neuroblastomas reveals a skewed ALK mutation spectrum in tumors with MYCNamplification. Clinicalcancerresearch:anofficial journaloftheAmerican AssociationforCancerResearch16,4353‐4362,doi:10.1158/1078‐0432.CCR‐09‐ 2660(2010). Mosse,Y.P.,Laudenslager,M.,Longo,L.,Cole,K.A.,Wood,A.etal.Identification ofALKasamajorfamilialneuroblastomapredispositiongene.Nature455,930‐ 935,doi:10.1038/nature07261(2008). Bown, N., Cotterill, S., Lastowska, M., O'Neill, S., Pearson, A. D. et al. Gain of chromosomearm17qandadverseoutcomeinpatientswithneuroblastoma.The New England journal of medicine 340, 1954‐1961, doi:10.1056/NEJM199906243402504(1999). Combaret, V., Brejon, S., Iacono, I., Schleiermacher, G., Pierron, G. et al. Determination of 17q gain in patients with neuroblastoma by analysis of circulatingDNA.Pediatricblood&cancer,doi:10.1002/pbc.22816(2011). Islam, A., Handley, S. L., Thompson, K. S. & Akhtar, S. Studies on uptake, sub‐ cellular trafficking and efflux of antisense oligodeoxynucleotides in glioma cells using self‐assembling cationic lipoplexes as delivery systems. Journal of drug targeting7,373‐382(2000). Lamers,F.,vanderPloeg,I.,Schild,L.,Ebus,M.E.,Koster,J.etal.Knockdownof survivin (BIRC5) causes apoptosis in neuroblastoma via mitotic catastrophe. Endocrine‐relatedcancer18,657‐668,doi:10.1530/ERC‐11‐0207(2011). Carmena, M., Wheelock, M., Funabiki, H. & Earnshaw, W. C. The chromosomal passenger complex (CPC): from easy rider to the godfather of mitosis. Nature reviews.Molecularcellbiology13,789‐803,doi:10.1038/nrm3474(2012). Altieri, D. C. The case for survivin as a regulator of microtubule dynamics and cell‐death decisions. Current opinion in cell biology 18, 609‐615, doi:10.1016/j.ceb.2006.08.015(2006). 118 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 Literatur Xu, J. & Andreassi, M. Reversible histone methylation regulates brain gene expression and behavior. Hormones and behavior 59, 383‐392, doi:10.1016/j.yhbeh.2010.08.019(2011). Hu, X., Ybarra, R., Qiu, Y., Bungert, J. & Huang, S. Transcriptional regulation by TAL1: a link between epigenetic modifications and erythropoiesis. Epigenetics : officialjournaloftheDNAMethylationSociety4,357‐361(2009). Huang,J.,Sengupta,R.,Espejo,A.B.,Lee,M.G.,Dorsey,J.A.etal.p53isregulated bythelysinedemethylaseLSD1.Nature449,105‐108,doi:10.1038/nature06092 (2007). Kontaki,H.&Talianidis,I.LysinemethylationregulatesE2F1‐inducedcelldeath. Molecularcell39,152‐160,doi:10.1016/j.molcel.2010.06.006(2010). Lim, S., Janzer, A., Becker, A., Zimmer, A., Schule, R. et al. Lysine‐specific demethylase 1 (LSD1) is highly expressed in ER‐negative breast cancers and a biomarker predicting aggressive biology. Carcinogenesis 31, 512‐520, doi:10.1093/carcin/bgp324(2010). Magerl, C., Ellinger, J., Braunschweig, T., Kremmer, E., Koch, L. K. et al. H3K4 dimethylation in hepatocellular carcinoma is rare compared with other hepatobiliaryandgastrointestinalcarcinomasandcorrelateswithexpressionof the methylase Ash2 and the demethylase LSD1. Human pathology 41, 181‐189, doi:10.1016/j.humpath.2009.08.007(2010). Schulte, J. H., Lim, S., Schramm, A., Friedrichs, N., Koster, J. et al. Lysine‐specific demethylase 1 is strongly expressed in poorly differentiated neuroblastoma: implications for therapy. Cancer research 69, 2065‐2071, doi:10.1158/0008‐ 5472.CAN‐08‐1735(2009). Frese, K. K. & Tuveson, D. A. Maximizing mouse cancer models. Nature reviews. Cancer7,645‐658,doi:10.1038/nrc2192(2007). Weiss,W.A.,Aldape,K.,Mohapatra,G.,Feuerstein,B.G.&Bishop,J.M.Targeted expressionofMYCNcausesneuroblastomaintransgenicmice.TheEMBOjournal 16,2985‐2995,doi:10.1093/emboj/16.11.2985(1997). Banerjee,S.A.,Hoppe,P.,Brilliant,M.&Chikaraishi,D.M.5'flankingsequencesof therattyrosinehydroxylasegenetargetaccuratetissue‐specific,developmental, andtranssynapticexpressionintransgenicmice.TheJournalofneuroscience:the officialjournaloftheSocietyforNeuroscience12,4460‐4467(1992). Berry, T., Luther, W., Bhatnagar, N., Jamin, Y., Poon, E. et al. The ALK(F1174L) mutation potentiates the oncogenic activity of MYCN in neuroblastoma. Cancer cell22,117‐130,doi:10.1016/j.ccr.2012.06.001(2012). Stanke,M.,Duong,C.V.,Pape,M.,Geissen,M.,Burbach,G.etal.Target‐dependent specification of the neurotransmitter phenotype: cholinergic differentiation of sympatheticneuronsismediatedinvivobygp130signaling.Development133, 141‐150,doi:10.1242/dev.02189(2006). Molenaar, J. J., Domingo‐Fernandez, R., Ebus, M. E., Lindner, S., Koster, J. et al. LIN28BinducesneuroblastomaandenhancesMYCNlevelsvialet‐7suppression. Naturegenetics44,1199‐1206,doi:10.1038/ng.2436(2012). Heukamp, L. C., Thor, T., Schramm, A., De Preter, K., Kumps, C. et al. Targeted expression of mutated ALK induces neuroblastoma in transgenic mice. Science translationalmedicine4,141ra191,doi:10.1126/scitranslmed.3003967(2012). Bartel,D.P.MicroRNAs:genomics,biogenesis,mechanism,andfunction.Cell116, 281‐297(2004). Lee,Y.,Kim,M.,Han,J.,Yeom,K.H.,Lee,S.etal.MicroRNAgenesaretranscribed by RNA polymerase II. The EMBO journal 23, 4051‐4060, doi:10.1038/sj.emboj.7600385(2004). 119 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 Literatur Cai, X., Hagedorn, C. H. & Cullen, B. R. Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs. Rna 10, 1957‐1966,doi:10.1261/rna.7135204(2004). Lee,Y.,Ahn,C.,Han,J.,Choi,H.,Kim,J.etal.ThenuclearRNaseIIIDroshainitiates microRNAprocessing.Nature425,415‐419,doi:10.1038/nature01957(2003). Han,J.,Lee,Y.,Yeom,K.H.,Kim,Y.K.,Jin,H.etal.TheDrosha‐DGCR8complexin primary microRNA processing. Genes & development 18, 3016‐3027, doi:10.1101/gad.1262504(2004). Bohnsack, M. T., Czaplinski, K. & Gorlich, D. Exportin 5 is a RanGTP‐dependent dsRNA‐binding protein that mediates nuclear export of pre‐miRNAs. Rna 10, 185‐191(2004). Hutvagner,G.,McLachlan,J.,Pasquinelli,A.E.,Balint,E.,Tuschl,T.etal.Acellular function for the RNA‐interference enzyme Dicer in the maturation of the let‐7 small temporal RNA. Science 293, 834‐838, doi:10.1126/science.1062961 (2001). Salzman,D.W.,Shubert‐Coleman,J.&Furneaux,H.P68RNAhelicaseunwindsthe human let‐7 microRNA precursor duplex and is required for let‐7‐directed silencing of gene expression. The Journal of biological chemistry 282, 32773‐ 32779,doi:10.1074/jbc.M705054200(2007). Khvorova, A., Reynolds, A. & Jayasena, S. D. Functional siRNAs and miRNAs exhibitstrandbias.Cell115,209‐216(2003). Schwarz, D. S., Hutvagner, G., Du, T., Xu, Z., Aronin, N. et al. Asymmetry in the assemblyoftheRNAienzymecomplex.Cell115,199‐208(2003). Garzon, R., Calin, G. A. & Croce, C. M. MicroRNAs in Cancer. Annual review of medicine60,167‐179,doi:10.1146/annurev.med.59.053006.104707(2009). Lewis,B.P.,Shih,I.H.,Jones‐Rhoades,M.W.,Bartel,D.P.&Burge,C.B.Prediction ofmammalianmicroRNAtargets.Cell115,787‐798(2003). Grimson, A., Farh, K. K., Johnston, W. K., Garrett‐Engele, P., Lim, L. P. et al. MicroRNA targeting specificity in mammals: determinants beyond seed pairing. Molecularcell27,91‐105,doi:10.1016/j.molcel.2007.06.017(2007). Kedde, M., Strasser, M. J., Boldajipour, B., Oude Vrielink, J. A., Slanchev, K. et al. RNA‐binding protein Dnd1 inhibits microRNA access to target mRNA. Cell 131, 1273‐1286,doi:10.1016/j.cell.2007.11.034(2007). Garzon, R. & Croce, C. M. MicroRNAs and cancer: introduction. Seminars in oncology38,721‐723,doi:10.1053/j.seminoncol.2011.08.008(2011). Miska, E. A. How microRNAs control cell division, differentiation and death. Current opinion in genetics & development 15, 563‐568, doi:10.1016/j.gde.2005.08.005(2005). Fontana,L.,Fiori,M.E.,Albini,S.,Cifaldi,L.,Giovinazzi,S.etal.Antagomir‐17‐5p abolishes the growthof therapy‐resistant neuroblastoma through p21 and BIM. PloSone3,e2236,doi:10.1371/journal.pone.0002236(2008). De Preter, K., Mestdagh, P., Vermeulen, J., Zeka, F., Naranjo, A. et al. miRNA expression profiling enables risk stratification in archived and fresh neuroblastomatumorsamples.Clinicalcancerresearch:anofficialjournalofthe American Association for Cancer Research 17, 7684‐7692, doi:10.1158/1078‐ 0432.CCR‐11‐0610(2011). Mestdagh,P.,Bostrom,A.K.,Impens,F.,Fredlund,E.,VanPeer,G.etal.ThemiR‐ 17‐92microRNAclusterregulatesmultiplecomponentsoftheTGF‐betapathway inneuroblastoma.Molecularcell40,762‐773,doi:10.1016/j.molcel.2010.11.038 (2010). 120 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 Literatur Cole,K.A.,Attiyeh,E.F.,Mosse,Y.P.,Laquaglia,M.J.,Diskin,S.J.etal.Afunctional screenidentifiesmiR‐34aasacandidateneuroblastomatumorsuppressorgene. Molecular cancer research : MCR 6, 735‐742, doi:10.1158/1541‐7786.MCR‐07‐ 2102(2008). Welch,C.,Chen,Y.&Stallings,R.L.MicroRNA‐34afunctionsasapotentialtumor suppressor by inducing apoptosis in neuroblastoma cells. Oncogene 26, 5017‐ 5022,doi:10.1038/sj.onc.1210293(2007). Wei, J. S., Song, Y. K., Durinck, S., Chen, Q. R., Cheuk, A. T. et al. The MYCN oncogene is a direct target of miR‐34a. Oncogene 27, 5204‐5213, doi:10.1038/onc.2008.154(2008). Stallings, R. L. MicroRNA involvement in the pathogenesis of neuroblastoma: potentialformicroRNAmediatedtherapeutics.Currentpharmaceuticaldesign15, 456‐462(2009). Chen,Y.,Tsai,Y.H.&Tseng,S.H.Inhibitionofcyclin‐dependentkinase1‐induced cell death in neuroblastoma cells through the microRNA‐34a‐MYCN‐survivin pathway.Surgery153,4‐16,doi:10.1016/j.surg.2012.03.030(2013). Schulte,J. H.,Schowe,B.,Mestdagh,P.,Kaderali,L.,Kalaghatgi,P.et al.Accurate prediction of neuroblastoma outcome based on miRNA expression profiles. International journal of cancer. Journal international du cancer 127, 2374‐2385, doi:10.1002/ijc.25436(2010). Bray,I.,Tivnan,A.,Bryan,K.,Foley,N.H.,Watters,K.M.etal.MicroRNA‐542‐5p as a novel tumor suppressor in neuroblastoma. Cancer letters 303, 56‐64, doi:10.1016/j.canlet.2011.01.016(2011). Yoon,S.,Choi,Y.C.,Lee,S.,Jeong,Y.,Yoon,J.etal.Inductionofgrowtharrestby miR‐542‐3p that targets survivin. FEBS letters 584, 4048‐4052, doi:10.1016/j.febslet.2010.08.025(2010). Silber,J.,Lim,D.A.,Petritsch,C.,Persson,A.I.,Maunakea,A.K.etal.miR‐124and miR‐137 inhibit proliferation of glioblastoma multiforme cells and induce differentiationofbraintumorstemcells.BMCmedicine6,14,doi:10.1186/1741‐ 7015‐6‐14(2008). Balaguer, F., Link, A., Lozano, J. J., Cuatrecasas, M., Nagasaka, T. et al. Epigenetic silencing of miR‐137 is an early event in colorectal carcinogenesis. Cancer research70,6609‐6618,doi:10.1158/0008‐5472.CAN‐10‐0622(2010). Kozaki, K., Imoto, I., Mogi, S., Omura, K. & Inazawa, J. Exploration of tumor‐ suppressivemicroRNAssilencedbyDNAhypermethylationinoralcancer.Cancer research68,2094‐2105,doi:10.1158/0008‐5472.CAN‐07‐5194(2008). Saiki,R.K.,Gelfand,D.H.,Stoffel,S.,Scharf,S.J.,Higuchi,R.etal.Primer‐directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 239,487‐491(1988). Lamers, F., Schild, L., Koster, J., Versteeg, R., Caron, H. N. et al. Targeted BIRC5 silencingusingYM155causescelldeathinneuroblastomacellswithlowABCB1 763‐771, expression. European journal of cancer 48, doi:10.1016/j.ejca.2011.10.012(2012). Livak, K. J. & Schmittgen, T. D. Analysis of relative gene expression data using real‐timequantitativePCRandthe2(‐DeltaDeltaC(T))Method.Methods25,402‐ 408,doi:10.1006/meth.2001.1262(2001). Menten,B.,Pattyn,F.,DePreter,K.,Robbrecht,P.,Michels,E.etal.arrayCGHbase: an analysis platform for comparative genomic hybridization microarrays. BMC bioinformatics6,124,doi:10.1186/1471‐2105‐6‐124(2005). 121 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 Literatur Olshen, A. B., Venkatraman, E. S., Lucito, R. & Wigler, M. Circular binary segmentationfortheanalysisofarray‐basedDNAcopynumberdata.Biostatistics 5,557‐572,doi:10.1093/biostatistics/kxh008(2004). Fredlund, E., Ringner, M., Maris, J. M. & Pahlman, S. High Myc pathway activity and low stage of neuronal differentiation associate with poor outcome in neuroblastoma. Proceedings of the National Academy of Sciences of the United StatesofAmerica105,14094‐14099,doi:10.1073/pnas.0804455105(2008). Mestdagh,P.,VanVlierberghe,P.,DeWeer,A.,Muth,D.,Westermann,F. et al.A novelanduniversalmethodformicroRNART‐qPCRdatanormalization.Genome biology10,R64,doi:10.1186/gb‐2009‐10‐6‐r64(2009). Mestdagh,P.,Fredlund,E.,Pattyn,F.,Schulte,J.H.,Muth,D. etal.MYCN/c‐MYC‐ inducedmicroRNAsrepresscodinggenenetworksassociatedwithpooroutcome in MYCN/c‐MYC‐activated tumors. Oncogene 29, 1394‐1404, doi:10.1038/onc.2009.429(2010). Bradford,M.M.Arapidandsensitivemethodforthequantitationofmicrogram quantities of protein utilizing the principle of protein‐dye binding. Analytical biochemistry72,248‐254(1976). Schagger, H. & von Jagow, G. Tricine‐sodium dodecyl sulfate‐polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Analyticalbiochemistry166,368‐379(1987). Burnette, W. N. "Western blotting": electrophoretic transfer of proteins from sodium dodecyl sulfate‐‐polyacrylamide gels to unmodified nitrocellulose and radiographic detection with antibody and radioiodinated protein A. Analytical biochemistry112,195‐203(1981). Sokolova,V.V.,Radtke,I.,Heumann,R.&Epple,M.Effectivetransfectionofcells with multi‐shell calcium phosphate‐DNA nanoparticles. Biomaterials 27, 3147‐ 3153,doi:10.1016/j.biomaterials.2005.12.030(2006). Sokolova,V.,Knuschke,T.,Kovtun,A.,Buer,J.,Epple,M.etal.Theuseofcalcium phosphatenanoparticlesencapsulatingToll‐likereceptorligandsandtheantigen hemagglutinin to induce dendritic cell maturation and T cell activation. Biomaterials31,5627‐5633,doi:10.1016/j.biomaterials.2010.03.067(2010). Mosmann, T. Rapid colorimetric assay for cellular growth and survival: application to proliferation and cytotoxicity assays. Journal of immunological methods65,55‐63(1983). Liberzon,A.,Subramanian,A.,Pinchback,R.,Thorvaldsdottir,H.,Tamayo,P.etal. Molecular signatures database (MSigDB) 3.0. Bioinformatics 27, 1739‐1740, doi:10.1093/bioinformatics/btr260(2011). Cunningham,B.A.,Hemperly,J.J.,Murray,B.A.,Prediger,E.A.,Brackenbury,R.et al. Neural cell adhesion molecule: structure, immunoglobulin‐like domains, cell surfacemodulation,andalternativeRNAsplicing.Science236,799‐806(1987). Whiteford, C. C., Bilke, S., Greer, B. T., Chen, Q., Braunschweig, T. A. et al. Credentialing preclinical pediatric xenograft models using gene expression and tissue microarray analysis. Cancer research 67, 32‐40, doi:10.1158/0008‐ 5472.CAN‐06‐0610(2007). Yu, D., Cozma, D., Park, A. & Thomas‐Tikhonenko, A. Functional validation of genes implicated in lymphomagenesis: an in vivo selection assay using a Myc‐ inducedB‐celltumor.AnnalsoftheNewYorkAcademyofSciences1059,145‐159, doi:10.1196/annals.1339.047(2005). Cairo, S., De Falco, F., Pizzo, M., Salomoni, P., Pandolfi, P. P. et al. PML interacts with Myc, and Myc target gene expression is altered in PML‐null fibroblasts. Oncogene24,2195‐2203,doi:10.1038/sj.onc.1208338(2005). 122 92 93 94 95 96 97 98 99 100 101 102 103 104 105 Literatur Schuhmacher, M., Kohlhuber, F., Holzel, M., Kaiser, C., Burtscher, H. et al. The transcriptionalprogramofahumanBcelllineinresponsetoMyc.Nucleicacids research29,397‐406(2001). Zeller,K.I.,Jegga,A.G.,Aronow,B.J.,O'Donnell,K.A.&Dang,C.V.Anintegrated database of genes responsive to the Myc oncogenic transcription factor: identificationofdirectgenomictargets.Genomebiology4,R69,doi:10.1186/gb‐ 2003‐4‐10‐r69(2003). Lee, J. S., Chu, I. S., Mikaelyan, A., Calvisi, D. F., Heo, J. et al. Application of comparative functional genomics to identify best‐fit mouse models to study humancancer.Naturegenetics36,1306‐1311,doi:10.1038/ng1481(2004). Kauffmann,A.,Rosselli,F.,Lazar,V.,Winnepenninckx,V.,Mansuet‐Lupo,A.etal. High expression of DNA repair pathways is associated with metastasis in melanomapatients.Oncogene27,565‐573,doi:10.1038/sj.onc.1210700(2008). Whitfield, M. L., Sherlock, G., Saldanha, A. J., Murray, J. I., Ball, C. A. et al. Identification of genes periodically expressed in the human cell cycle and their expression in tumors. Molecular biology of the cell 13, 1977‐2000, doi:10.1091/mbc.02‐02‐0030.(2002). Eguchi,T.,Takaki,T.,Itadani,H.&Kotani,H.RBsilencingcompromisestheDNA damage‐induced G2/M checkpoint and causes deregulated expression of the ECT2oncogene.Oncogene26,509‐520,doi:10.1038/sj.onc.1209810(2007). Georges, S. A., Biery, M. C., Kim, S. Y., Schelter, J. M., Guo, J. et al. Coordinated regulation of cell cycle transcripts by p53‐Inducible microRNAs, miR‐192 and miR‐215. Cancer research 68, 10105‐10112, doi:10.1158/0008‐5472.CAN‐08‐ 1846(2008). Le, M. T., Xie, H., Zhou, B., Chia, P. H., Rizk, P. et al. MicroRNA‐125b promotes neuronaldifferentiationinhumancellsbyrepressingmultipletargets.Molecular andcellularbiology29,5290‐5305,doi:10.1128/MCB.01694‐08(2009). Han, D., Spengler, B. A. & Ross, R. A. Increased wild‐type N‐ras activation by neurofibromin down‐regulation increases human neuroblastoma stem cell malignancy. Genes & cancer 2, 1034‐1043, doi:10.1177/1947601912443127 (2011). Batra,R.,Harder,N.,Gogolin,S.,Diessl,N.,Soons,Z.etal.Time‐lapseimagingof neuroblastoma cells to determine cell fate upon gene knockdown. PloS one 7, e50988,doi:10.1371/journal.pone.0050988(2012). Carotenuto, M., Pedone, E., Diana, D., de Antonellis, P., Dzeroski, S. et al. Neuroblastoma tumorigenesis is regulated through the Nm23‐H1/h‐Prune C‐ terminalinteraction.Scientificreports3,1351,doi:10.1038/srep01351(2013). Hogarty, M. D., Norris, M. D., Davis, K., Liu, X., Evageliou, N. F. et al. ODC1 is a critical determinant of MYCN oncogenesis and a therapeutic target in neuroblastoma. Cancer research 68, 9735‐9745, doi:10.1158/0008‐5472.CAN‐ 07‐6866(2008). Fernandez,P.C.,Frank,S.R.,Wang,L.,Schroeder,M.,Liu,S.etal.Genomictargets of the human c‐Myc protein. Genes & development 17, 1115‐1129, doi:10.1101/gad.1067003(2003). Li, Z., Van Calcar, S., Qu, C., Cavenee, W. K., Zhang, M. Q. et al. A global transcriptionalregulatoryroleforc‐MycinBurkitt'slymphomacells.Proceedings of the National Academy of Sciences of the United States of America 100, 8164‐ 8169,doi:10.1073/pnas.1332764100(2003). 123 106 107 108 109 110 111 112 113 114 115 116 117 118 Literatur Dauphinot,L.,DeOliveira,C.,Melot,T.,Sevenet,N.,Thomas,V. etal.Analysisof theexpressionofcellcycleregulatorsinEwingcelllines:EWS‐FLI‐1modulates p57KIP2and c‐Myc expression. Oncogene 20, 3258‐3265, doi:10.1038/sj.onc.1204437(2001). Gogolin, S., Ehemann, V., Becker, G., Brueckner, L. M., Dreidax, D. et al. CDK4 inhibition restores G(1)‐S arrest in MYCN‐amplified neuroblastoma cells in the context of doxorubicin‐induced DNA damage. Cell cycle 12, 1091‐1104, doi:10.4161/cc.24091(2013). DeBrouwer,S.,Mestdagh,P.,Lambertz,I.,Pattyn,F.,DePaepe,A.etal.Dickkopf‐3 is regulated by the MYCN‐induced miR‐17‐92 cluster in neuroblastoma. International journal of cancer. Journal international du cancer 130, 2591‐2598, doi:10.1002/ijc.26295(2012). Haug, B. H., Henriksen, J. R., Buechner, J., Geerts, D., Tomte, E. et al. MYCN‐ regulated miRNA‐92 inhibits secretion of the tumor suppressor DICKKOPF‐3 (DKK3) in neuroblastoma. Carcinogenesis 32, 1005‐1012, doi:10.1093/carcin/bgr073(2011). Schulte, J. H., Marschall, T., Martin, M., Rosenstiel, P., Mestdagh, P. et al. Deep sequencing reveals differential expression of microRNAs in favorable versus unfavorable neuroblastoma. Nucleic acids research 38, 5919‐5928, doi:10.1093/nar/gkq342(2010). Annibali,D.,Gioia,U.,Savino,M.,Laneve,P.,Caffarelli,E.et al.Anewmodulein neuraldifferentiationcontrol:twomicroRNAsupregulatedbyretinoicacid,miR‐ 9 and ‐103, target the differentiation inhibitor ID2. PloS one 7, e40269, doi:10.1371/journal.pone.0040269(2012). Chen, Y. & Stallings, R. L. Differential patterns of microRNA expression in neuroblastoma are correlated with prognosis, differentiation, and apoptosis. Cancerresearch67,976‐983,doi:10.1158/0008‐5472.CAN‐06‐3667(2007). Foley,N.H.,Bray,I.M.,Tivnan,A.,Bryan,K.,Murphy,D.M.etal.MicroRNA‐184 inhibits neuroblastoma cell survival through targeting the serine/threonine kinaseAKT2.Molecularcancer9,83,doi:10.1186/1476‐4598‐9‐83(2010). Chen, H., Shalom‐Feuerstein, R., Riley, J., Zhang, S. D., Tucci, P. et al. miR‐7 and miR‐214arespecificallyexpressedduringneuroblastomadifferentiation,cortical development and embryonic stem cells differentiation, and control neurite outgrowth in vitro. Biochemical and biophysical research communications 394, 921‐927,doi:10.1016/j.bbrc.2010.03.076(2010). Das, S., Bryan, K., Buckley, P. G., Piskareva, O., Bray, I. M. et al. Modulation of neuroblastoma disease pathogenesis by an extensive network of epigenetically regulatedmicroRNAs.Oncogene,doi:10.1038/onc.2012.311(2012). Bray, I., Bryan, K., Prenter, S., Buckley, P. G., Foley, N. H. et al. Widespread dysregulationofMiRNAsbyMYCNamplificationandchromosomalimbalancesin neuroblastoma: association of miRNA expression with survival. PloS one 4, e7850,doi:10.1371/journal.pone.0007850(2009). Ma, L., Young, J., Prabhala, H., Pan, E., Mestdagh, P. et al. miR‐9, a MYC/MYCN‐ activated microRNA, regulates E‐cadherin and cancer metastasis. Nature cell biology12,247‐256,doi:10.1038/ncb2024(2010). Althoff, K., Beckers, A., Odersky, A., Mestdagh, P., Koster, J. et al. MiR‐137 functions as a tumor suppressor in neuroblastoma by downregulating KDM1A. International journal of cancer. Journal international du cancer, doi:10.1002/ijc.28091(2013). 124 119 120 121 122 123 124 125 126 127 128 129 130 131 132 Literatur Chen, D. L., Wang, D. S., Wu, W. J., Zeng, Z. L., Luo, H. Y. et al. Overexpression of paxillin induced by miR‐137 suppression promotes tumor progression and metastasis in colorectal cancer. Carcinogenesis 34, 803‐811, doi:10.1093/carcin/bgs400(2013). Liang, L., Li, X., Zhang, X., Lv, Z., He, G. et al. MicroRNA‐137, an HMGA1 target, suppresses colorectal cancer cell invasion and metastasis in mice by directly targeting FMNL2. Gastroenterology 144, 624‐635 e624, doi:10.1053/j.gastro.2012.11.033(2013). Zhu,X.,Li,Y.,Shen,H.,Li,H.,Long,L.etal.miR‐137inhibitstheproliferationof lung cancer cells by targeting Cdc42 and Cdk6. FEBS letters 587, 73‐81, doi:10.1016/j.febslet.2012.11.004(2013). Luo,C.,Tetteh,P.W.,Merz,P.R.,Dickes,E.,Abukiwan,A.etal.miR‐137inhibits the invasion of melanoma cells through downregulation of multiple oncogenic target genes. The Journal of investigative dermatology 133, 768‐775, doi:10.1038/jid.2012.357(2013). Zhao, Y., Li, Y., Lou, G., Zhao, L., Xu, Z. et al. MiR‐137 targets estrogen‐related receptor alpha and impairs the proliferative and migratory capacity of breast cancercells.PloSone7,e39102,doi:10.1371/journal.pone.0039102(2012). Seeger,R.C.,Brodeur,G.M.,Sather,H.,Dalton,A.,Siegel,S.E.etal.Associationof multiple copies of the N‐myc oncogene with rapid progression of neuroblastomas. The New England journal of medicine 313, 1111‐1116, doi:10.1056/NEJM198510313131802(1985). Hoyle,G.W.,Mercer,E.H.,Palmiter,R.D.&Brinster,R.L.Cell‐specificexpression from the human dopamine beta‐hydroxylase promoter in transgenic mice is controlled via a combination of positive and negative regulatory elements. The Journal of neuroscience : the official journal of the Society for Neuroscience 14, 2455‐2463(1994). Gilbert,J.,Haber,M.,Bordow,S.B.,Marshall,G.M.&Norris,M.D.Useoftumor‐ specific gene expression for the differential diagnosis of neuroblastoma from otherpediatricsmallround‐cellmalignancies.TheAmericanjournalofpathology 155,17‐21,doi:10.1016/S0002‐9440(10)65093‐6(1999). Tiveron, M. C., Hirsch, M. R. & Brunet, J. F. The expression pattern of the transcription factor Phox2 delineates synaptic pathways of the autonomic nervoussystem.TheJournalofneuroscience:theofficialjournaloftheSocietyfor Neuroscience16,7649‐7660(1996). Hartomo,T.B.,Kozaki,A.,Hasegawa,D.,Pham,T.V.,Yamamoto,N.etal.Minimal residual disease monitoring in neuroblastoma patientsbased on the expression of a set of real‐time RT‐PCR markers in tumor‐initiating cells. Oncology reports 29,1629‐1636,doi:10.3892/or.2013.2286(2013). Hackett,C.S.,Hodgson,J.G.,Law,M.E.,Fridlyand,J.,Osoegawa,K.etal.Genome‐ wide array CGH analysis of murine neuroblastoma reveals distinct genomic aberrations which parallel those in human tumors. Cancer research 63, 5266‐ 5273(2003). Casola, S. Mouse models for miRNA expression: the ROSA26 locus. Methods in molecularbiology667,145‐163,doi:10.1007/978‐1‐60761‐811‐9_10(2010). Tchorz,J.S.,Suply,T.,Ksiazek,I.,Giachino,C.,Cloetta,D.etal.AmodifiedRMCE‐ compatible Rosa26 locus for the expression of transgenes from exogenous promoters.PloSone7,e30011,doi:10.1371/journal.pone.0030011(2012). Soriano, P. Generalized lacZ expression with the ROSA26 Cre reporter strain. Naturegenetics21,70‐71,doi:10.1038/5007(1999). 125 133 134 135 136 137 138 139 140 141 142 143 144 145 Literatur Luche, H., Weber, O., Nageswara Rao, T., Blum, C. & Fehling, H. J. Faithful activation of an extra‐bright red fluorescent protein in "knock‐in" Cre‐reporter mice ideally suited for lineage tracing studies. European journal of immunology 37,43‐53,doi:10.1002/eji.200636745(2007). Rasmuson, A., Kock, A., Fuskevag, O. M., Kruspig, B., Simon‐Santamaria, J. et al. Autocrine prostaglandin E2 signaling promotes tumor cell survival and proliferation in childhood neuroblastoma. PloS one 7, e29331, doi:10.1371/journal.pone.0029331(2012). Vandesompele, J., Speleman, F., Van Roy, N., Laureys, G., Brinskchmidt, C. et al. Multicentre analysis of patterns of DNA gains and losses in 204 neuroblastoma tumors:howmanygeneticsubgroupsarethere?Medicalandpediatriconcology 36, 5‐10, doi:10.1002/1096‐911X(20010101)36:1<5::AID‐MPO1003>3.0.CO;2‐E (2001). Weiss, W. A., Godfrey, T., Francisco, C. & Bishop, J. M. Genome‐wide screen for allelicimbalanceinamousemodelforneuroblastoma.Cancerresearch60,2483‐ 2487(2000). Rasmuson, A., Segerstrom, L., Nethander, M., Finnman, J., Elfman, L. H. et al. Tumordevelopment,growthcharacteristicsandspectrumofgeneticaberrations in the TH‐MYCN mouse model of neuroblastoma. PloS one 7, e51297, doi:10.1371/journal.pone.0051297(2012). Schleiermacher,G.,Janoueix‐Lerosey,I.,Ribeiro,A.,Klijanienko,J.,Couturier,J.et al. Accumulation of segmental alterations determines progression in neuroblastoma. Journal of clinical oncology : official journal of the American Society of Clinical Oncology 28, 3122‐3130, doi:10.1200/JCO.2009.26.7955 (2010). Stallings, R. L., Howard, J., Dunlop, A., Mullarkey, M., McDermott, M. et al. Are gains of chromosomal regions 7q and 11p important abnormalities in neuroblastoma?Cancergeneticsandcytogenetics140,133‐137(2003). Miller,M.A.,Ohashi,K.,Zhu,X.,McGrady,P.,London,W.B.etal.SurvivinmRNA levels are associated with biology of disease and patient survival in neuroblastoma:areportfromthechildren'soncologygroup.Journalofpediatric hematology/oncology 28, 412‐417, doi:10.1097/01.mph.0000212937.00287.e5 (2006). Ito, R., Asami, S., Motohashi, S., Ootsuka, S., Yamaguchi, Y. et al. Significance of survivin mRNA expression in prognosis of neuroblastoma. Biological & pharmaceuticalbulletin28,565‐568(2005). Attiyeh,E.F.,London,W.B.,Mosse,Y.P.,Wang,Q.,Winter,C.etal.Chromosome 1pand11qdeletionsandoutcomeinneuroblastoma.TheNewEnglandjournalof medicine353,2243‐2253,doi:10.1056/NEJMoa052399(2005). Maris, J. M., Weiss, M. J., Guo, C., Gerbing, R. B., Stram, D. O. et al. Loss of heterozygosity at 1p36 independently predicts for disease progression but not decreased overall survival probability in neuroblastoma patients: a Children's CancerGroupstudy.Journalofclinicaloncology:officialjournaloftheAmerican SocietyofClinicalOncology18,1888‐1899(2000). White,P.S.,Thompson,P.M.,Gotoh,T.,Okawa,E.R.,Igarashi,J.etal.Definition andcharacterizationofaregionof1p36.3consistentlydeletedinneuroblastoma. Oncogene24,2684‐2694,doi:10.1038/sj.onc.1208306(2005). Chim,C.S.,Wong,K.Y.,Qi,Y.,Loong,F.,Lam,W.L.etal.Epigeneticinactivationof the miR‐34a in hematological malignancies. Carcinogenesis 31, 745‐750, doi:10.1093/carcin/bgq033(2010). 126 146 147 148 149 150 151 152 153 154 155 156 157 158 159 Literatur Li,N.,Fu,H.,Tie,Y.,Hu,Z.,Kong,W.etal.miR‐34ainhibitsmigrationandinvasion bydown‐regulationofc‐Metexpressioninhumanhepatocellularcarcinomacells. Cancerletters275,44‐53,doi:10.1016/j.canlet.2008.09.035(2009). Fujita, T., Igarashi, J., Okawa, E. R., Gotoh, T., Manne, J. et al. CHD5, a tumor suppressorgenedeletedfrom1p36.31inneuroblastomas.JournaloftheNational CancerInstitute100,940‐949,doi:10.1093/jnci/djn176(2008). Schlisio,S.,Kenchappa,R.S.,Vredeveld,L.C.,George,R.E.,Stewart,R.etal.The kinesin KIF1Bbeta acts downstream from EglN3 to induce apoptosis and is a potential 1p36 tumor suppressor. Genes & development 22, 884‐893, doi:10.1101/gad.1648608(2008). Hanahan,D.&Weinberg,R.A.Thehallmarksofcancer.Cell100,57‐70(2000). He,S.,Nakada,D.&Morrison,S.J.Mechanismsofstemcellself‐renewal.Annual review of cell and developmental biology 25, 377‐406, doi:10.1146/annurev.cellbio.042308.113248(2009). Molenaar,J.J.,Ebus,M.E.,Koster,J.,vanSluis,P.,vanNoesel,C.J.etal.CyclinD1 and CDK4 activity contribute to the undifferentiated phenotype in neuroblastoma. Cancer research 68, 2599‐2609, doi:10.1158/0008‐5472.CAN‐ 07‐5032(2008). Molenaar, J. J., Koster, J., Ebus, M. E., van Sluis, P., Westerhout, E. M. et al. Copy number defects of G1‐cell cycle genes in neuroblastoma are frequent and correlatewithhighexpressionofE2Ftargetgenesandapoorprognosis.Genes, chromosomes&cancer51,10‐19,doi:10.1002/gcc.20926(2012). Yamanaka, A., Hatakeyama, S., Kominami, K., Kitagawa, M., Matsumoto, M. et al. Cellcycle‐dependentexpressionofmammalianE2‐Cregulatedbytheanaphase‐ promoting complex/cyclosome. Molecular biology of the cell 11, 2821‐2831 (2000). Jiang, L., Bao, Y., Luo, C., Hu, G., Huang, C. et al. Knockdown of ubiquitin‐ conjugatingenzymeE2C/UbcH10expressionbyRNAinterferenceinhibitsglioma cellproliferationandenhancescellapoptosisinvitro.Journalofcancerresearch andclinicaloncology136,211‐217,doi:10.1007/s00432‐009‐0651‐z(2010). Qing, G., Skuli, N., Mayes, P. A., Pawel, B., Martinez, D. et al. Combinatorial regulationofneuroblastomatumorprogressionbyN‐Mycandhypoxiainducible factor HIF‐1alpha. Cancer research 70, 10351‐10361, doi:10.1158/0008‐ 5472.CAN‐10‐0740(2010). Garcia, I., Mayol, G., Rios, J., Domenech, G., Cheung, N. K. et al. A three‐gene expressionsignaturemodelforriskstratificationofpatientswithneuroblastoma. Clinicalcancerresearch:anofficialjournaloftheAmericanAssociationforCancer Research18,2012‐2023,doi:10.1158/1078‐0432.CCR‐11‐2483(2012). Schubbert, S., Shannon, K. & Bollag, G. Hyperactive Ras in developmental disorders and cancer. Nature reviews. Cancer 7, 295‐308, doi:10.1038/nrc2109 (2007). Shimizu, K., Goldfarb, M., Suard, Y., Perucho, M., Li, Y. et al. Three human transforming genes are related to the viral ras oncogenes. Proceedings of the National Academy of Sciences of the United States of America 80, 2112‐2116 (1983). O'Donnell, K. A., Wentzel, E. A., Zeller, K. I., Dang, C. V. & Mendell, J. T. c‐Myc‐ regulated microRNAs modulate E2F1 expression. Nature 435, 839‐843, doi:10.1038/nature03677(2005). 127 160 161 162 163 164 165 166 167 168 169 170 171 172 173 174 Literatur Terrile, M., Bryan, K., Vaughan, L., Hallsworth, A., Webber, H. et al. miRNA expression profiling of the murine TH‐MYCN neuroblastoma model reveals similaritieswithhumantumorsandidentifiesnovelcandidatemiRNAs.PloSone 6,e28356,doi:10.1371/journal.pone.0028356(2011). Testa,J.R.&Bellacosa,A.AKTplaysacentralroleintumorigenesis.Proceedings of the National Academy of Sciences of the United States of America 98, 10983‐ 10985,doi:10.1073/pnas.211430998(2001). Stubbusch,J.,Majdazari,A.,Schmidt,M.,Schutz,G.,Deller,T.etal.Generationof thetamoxifen‐inducibleDBH‐CretransgenicmouselineDBH‐CT.Genesis49,935‐ 941,doi:10.1002/dvg.20773(2011). Sprussel,A.,Schulte,J.H.,Weber,S.,Necke,M.,Handschke,K.etal.Lysine‐specific demethylase 1 restricts hematopoietic progenitor proliferation and is essential forterminaldifferentiation.Leukemia26,2039‐2051,doi:10.1038/leu.2012.157 (2012). Ventura,A.,Young,A.G.,Winslow,M.M.,Lintault,L.,Meissner,A.etal.Targeted deletion reveals essential and overlapping functions of the miR‐17 through 92 family of miRNA clusters. Cell 132, 875‐886, doi:10.1016/j.cell.2008.02.019 (2008). Carleton,M.,Cleary,M.A.&Linsley,P.S.MicroRNAsandcellcycleregulation.Cell cycle6,2127‐2132(2007). Wiemer,E.A.TheroleofmicroRNAsincancer:nosmallmatter.Europeanjournal ofcancer43,1529‐1544,doi:10.1016/j.ejca.2007.04.002(2007). Shohet,J.M.,Ghosh,R.,Coarfa,C.,Ludwig,A.,Benham,A.L.etal.Agenome‐wide search for promoters that respond to increased MYCN reveals both new oncogenic and tumor suppressor microRNAs associated with aggressive neuroblastoma. Cancer research 71, 3841‐3851, doi:10.1158/0008‐5472.CAN‐ 10‐4391(2011). Petrelli, A. & Giordano, S. From single‐ to multi‐target drugs in cancer therapy: when aspecificity becomes an advantage. Current medicinal chemistry 15, 422‐ 432(2008). Schrattenholz, A., Groebe, K. & Soskic, V. Systems biology approaches and tools foranalysisofinteractomesandmulti‐targetdrugs.Methodsinmolecularbiology 662,29‐58,doi:10.1007/978‐1‐60761‐800‐3_2(2010). Modak, S. & Cheung, N. K. Neuroblastoma: Therapeutic strategies for a clinical enigma. Cancer treatment reviews 36, 307‐317, doi:10.1016/j.ctrv.2010.02.006 (2010). Iorio, M. V. & Croce, C. M. MicroRNA dysregulation in cancer: diagnostics, monitoringandtherapeutics.Acomprehensivereview.EMBOmolecularmedicine 4,143‐159,doi:10.1002/emmm.201100209(2012). Tivnan, A., Orr, W. S., Gubala, V., Nooney, R., Williams, D. E. et al. Inhibition of neuroblastoma tumor growth by targeted delivery ofmicroRNA‐34a usinganti‐ disialoganglioside GD2 coated nanoparticles. PloS one 7, e38129, doi:10.1371/journal.pone.0038129(2012). Adida, C., Berrebi, D., Peuchmaur, M., Reyes‐Mugica, M. & Altieri, D. C. Anti‐ apoptosisgene,survivin,andprognosisofneuroblastoma.Lancet351,882‐883, doi:10.1016/S0140‐6736(05)70294‐4(1998). Islam, A., Kageyama, H., Takada, N., Kawamoto, T., Takayasu, H. et al. High expression of Survivin, mapped to 17q25, is significantly associated with poor prognosticfactorsandpromotescellsurvivalinhumanneuroblastoma.Oncogene 19,617‐623,doi:10.1038/sj.onc.1203358(2000). 128 175 176 177 178 179 180 181 182 183 184 185 186 187 188 189 Literatur Adams, R. R., Carmena, M. & Earnshaw, W. C. Chromosomal passengers and the (aurora)ABCsofmitosis.Trendsincellbiology11,49‐54(2001). Chen, J., Jin, S., Tahir, S. K., Zhang, H., Liu, X. et al. Survivin enhances Aurora‐B kinase activity and localizes Aurora‐B in human cells. The Journal of biological chemistry278,486‐490,doi:10.1074/jbc.M211119200(2003). Giodini, A., Kallio, M. J., Wall, N. R., Gorbsky, G. J., Tognin, S. et al. Regulation of microtubule stability and mitotic progression by survivin. Cancer research 62, 2462‐2467(2002). Chu,Y.,Yao,P.Y.,Wang,W.,Wang,D.,Wang,Z.etal.AuroraBkinaseactivation requires survivin priming phosphorylation by PLK1. Journal of molecular cell biology3,260‐267,doi:10.1093/jmcb/mjq037(2011). Sokolova,V.,Kozlova,D.,Knuschke,T.,Buer,J.,Westendorf,A.M.etal.Mechanism of the uptake of cationic and anionic calcium phosphate nanoparticles by cells. Actabiomaterialia9,7527‐7535,doi:10.1016/j.actbio.2013.02.034(2013). Florea,B.I.,Meaney,C.,Junginger,H.E.&Borchard,G.Transfectionefficiencyand toxicityofpolyethylenimineindifferentiatedCalu‐3andnondifferentiatedCOS‐1 cellcultures.AAPSpharmSci4,E12,doi:10.1208/ps040312(2002). Neu,M.,Fischer,D.&Kissel,T.Recentadvancesinrationalgenetransfervector design based on poly(ethylene imine) and its derivatives. The journal of gene medicine7,992‐1009,doi:10.1002/jgm.773(2005). Verissimo,C.S.,Molenaar,J.J.,Fitzsimons,C.P.&Vreugdenhil,E.Neuroblastoma therapy: what is in the pipeline? Endocrine‐related cancer 18, R213‐231, doi:10.1530/ERC‐11‐0251(2011). Duffy,M.J.,O'Donovan,N.,Brennan,D.J.,Gallagher,W.M.&Ryan,B.M.Survivin: a promising tumor biomarker. Cancer letters 249, 49‐60, doi:10.1016/j.canlet.2006.12.020(2007). Li, S., He, J., Li, S., Cao, G., Tang, S. et al. Noscapine induced apoptosis via downregulation of survivin in human neuroblastoma cells having wild type or nullp53.PloSone7,e40076,doi:10.1371/journal.pone.0040076(2012). Hossain,M.M.,Banik,N.L.&Ray,S.K.Survivinknockdownincreasedanti‐cancer effectsof(‐)‐epigallocatechin‐3‐gallateinhumanmalignantneuroblastomaSK‐N‐ BE2 and SH‐SY5Y cells. Experimental cell research 318, 1597‐1610, doi:10.1016/j.yexcr.2012.03.033(2012). Martin, S., Lamb, H. K., Brady, C., Lefkove, B., Bonner, M. Y. et al. Inducing apoptosis of cancer cells using small‐molecule plant compounds that bind to GRP78.Britishjournalofcancer,doi:10.1038/bjc.2013.325(2013). Nakahara, T., Kita, A., Yamanaka, K., Mori, M., Amino, N. et al. YM155, a novel small‐molecule survivin suppressant, induces regression of established human hormone‐refractory prostate tumor xenografts. Cancer research 67, 8014‐8021, doi:10.1158/0008‐5472.CAN‐07‐1343(2007). Hayami,S.,Kelly,J.D.,Cho,H.S.,Yoshimatsu,M.,Unoki,M.etal.Overexpression of LSD1 contributes to human carcinogenesis through chromatin regulation in various cancers. International journal of cancer. Journal international du cancer 128,574‐586,doi:10.1002/ijc.25349(2011). Xu,G.,Xiao,Y.,Hu,J.,Xing,L.,Zhao,O.etal.TheCombinedEffectofRetinoicAcid andLSD1siRNAInhibitiononCellDeathintheHumanNeuroblastomaCellLine SH‐SY5Y. Cellular physiology and biochemistry : international journal of experimental cellular physiology, biochemistry, and pharmacology 31, 854‐862, doi:10.1159/000350103(2013). 129 190 191 192 193 194 Literatur Stavropoulos, P. & Hoelz, A. Lysine‐specific demethylase 1 as a potential therapeutic target. Expert opinion on therapeutic targets 11, 809‐820, doi:10.1517/14728222.11.6.809(2007). Sun,G.,Ye,P.,Murai,K.,Lang,M.F.,Li,S.etal.miR‐137formsaregulatoryloop withnuclearreceptorTLXandLSD1inneuralstemcells.Naturecommunications 2,529,doi:10.1038/ncomms1532(2011). Bier, A., Giladi, N., Kronfeld, N., Lee, H. K., Cazacu, S. et al. MicroRNA‐137 is downregulatedinglioblastomaandinhibitsthestemnessofgliomastemcellsby targetingRTVP‐1.Oncotarget4,665‐676(2013). Li,K.K.,Yang,L.,Pang,J.C.,Chan,A.K.,Zhou,L.etal.MIR‐137SuppressesGrowth andInvasion,isDownregulatedinOligodendroglialTumorsandTargetsCSE1L. Brainpathology23,426‐439,doi:10.1111/bpa.12015(2013). Zhu, X., Li, Y., Shen, H., Li, H., Long, L. et al. miR‐137 restoration sensitizes multidrug‐resistant MCF‐7/ADM cells to anticancer agents by targeting YB‐1. Acta biochimica et biophysica Sinica 45, 80‐86, doi:10.1093/abbs/gms099 (2013). 130 Abkürzungen 9 Abkürzungen °C aCGH AURKB BIRC5 bp BrdU CAG cDNA CDK4 CPC Ct‐Wert Dbh DGCR8 DKFZ DMSO DMSZ DNA dNTP EDTA ES ES‐Zellen etal. FCS FDR Fluc g G1‐Phase G2/M‐Phase GAPDH GFP GOI GSEA H&E HCl INSS IRES i.v. kb KDM1A LB‐Medium LDH GradCelsius Array‐comparativegenomichybridization AuroraBKinase BaculoviralIAPrepeat‐containing5 Basenpaar 5‐bromo‐2‘‐Deoxyuridin Hühner‐Aktin‐Promotor komplementäreDNA Cyclin‐abhängigeKinase4 ChromosomalPassengerKomplex Schwellenwert‐Zyklus Dopaminbeta‐Hydroxylase DiGeorgesyndromecriticalregiongene8 DeutschesKrebsforschungszentrum Dimethylsulfoxid DeutscheSammlungvonMikroorganismenundZellkulturenGmbH Desoxyribonukleinsäure Desoxyribonukleosidtriphosphat Ethylendiamintetraessigsäure EnrichmentScore embryonaleStammzellen undandere fetalcalfserum(fetalesKälberserum) falsch‐positivRate LuziferaseGen Erdbeschleunigung Postmitose PrämitoseundMitose Glycerinaldehyd‐3‐Phosphat‐Dehydrogenase GrünfluoreszierendesProtein geneofinterest GeneSetEnrichmentAnalyse Hematoxylin&Eosin Chlorwasserstoff InternationalNeuroblastomaStagingSystem interneribosomaleEintrittsstelle intravenös kiloBasen Lysin‐spezifischeDemethylase1 lysogenybroth Laktat‐Dehydrogenase 131 LIN28B LSL miRNA MPL MSigDB MTT MYCN NEFL NES NTRK1 OD PBS PCR PEI pri‐miRNA pre‐miRNA qPCR RCME RIPA RISC RNA S‐Phase SDS‐PAGE siRNA SRM TBS TFPI2 TH Tris U UBE2C UTR UV wt Abkürzungen LIN28HomologB loxP‐flankiertePolyadenyl‐Stoppsequenz microRNA MolecularProteomicsLaboratory MolecularSignaturesDatenbank 3‐(4,5‐Dimethylthiazol‐2‐yl)‐2,5‐Diphenyl‐tetrazoliumbromid v‐myc myelocytomatosis viral related oncogen ,neuroblastoma derived Neurofilament normalisierterEnrichmentScore neurotropheRezeptor‐Tyrosinkinase1 optischeDichte PhosphatgepufferteSalzlösung Polymerasekettenreaktion Poly‐Ethylenimin primäremicroRNA Vorläufer‐microRNA quantitativePolymerasekettenreaktion Rekombinase‐vermittelteKassettenaustauschstelle Radioimmunoprecipitationassaybuffer RNA‐induziertersilencingKomplex Ribonukleinsäure SynthesePhasedesZellzyklus Natriumdodecylsulfat‐Polyacrylamidgelelektrophorese silencerRNA SpermidinSynthase TrisgepufferteSalzlösung tissuefactorpathwayinhibitor2 TyronsinHydroxylase Tris(hydroxymethyl)‐aminomethan unit Ubiquitin‐konjugierendeEnzymE2C untranslatierteRegion ultraviolett Wildtyp 132 Abbildun ngs‐und Taabellenverzzeichnis 10A Abbildu ungs‐undTab bellenv verzeich hnis 10.1 A Abbildu ungsverzeichniss Abb.1: ExprressionderCre‐Rekomb binaseimDbh‐iCreMau usmodell…… …………………….6 Abb.2: DieB Biogenesev vonmiRNAs ………………… ………………………………….… ………………… ……8 Abb.3: AufttrennungvonNeuroblasstompatienttenmitgute erundschlecchter P Prognosean nhandvonm miRNAExprressionsproffilen…………… ……….………… …………………….10 Abb.4: ScheemaderVek ktorkonstrukktionzurGeenerierungttransgenerLLSL‐MYCN M Mäuse……… ….………………………………… …………………………………… ………………… ………………… ……14 Abb.5: Strukturderverrwendetenm miRNA‐belaadenenCalciium‐Phosphhat‐ N ………………… ………………… …………………………………… …………………….29 Nanopartikeel……………… Abb.6: Vekttorkartevon npcDNA™6//TR…………… …………………… ………………… …………………… …..33 Abb.7: Vekttorkartevon npT‐Rex‐DE EST30………… ………………… …………………… ………………… ……34 Abb.8: ReprräsentativeSouthernBllotAnalyseeinesES‐Zelllklons……… …………………… …..39 Abb.9: ReprräsentativeGenotypisieerungs‐PCRssderverwendetenMauuslinien L LSL‐MYCNu undDbh‐iCre……………… …………………… ………………… …………………… ………………… …….40 Abb.10: Vertteilungdera auftretenden nGenotypen nausVerpaa arungenvonn h heterozygottenLSL‐MYC CNundDbh‐‐iCreMäuseen……………… ……………..…… …………………….41 Abb.11: Entw wicklungvon nTumoren imLSL‐MYC CN;Dbh‐iCreMausmodeell……………… ……42 Abb.12: Luziiferase‐ExprressiondreieerrepräsentativerTum moreaus L LSL‐MYCN;D Dbh‐iCreMä äusen………… …………………………………… ………………… ………………… ……42 Abb.13: Ultraaschall‐Bildgebungvon nTumorenaausLSL‐MYC CN;Dbh‐iCre Mäusen…… ……44 Abb.14: Mak krokopischeAufnahmen nrepräsentaativerTumoredes L LSL‐MYCN;D Dbh‐iCreMo odells.………… ………………… …………………… ………………… …………………… …..45 Abb.15: PCRzurValidierrungderau sgeschnitten nenPolyade enyl‐Stoppseequenzin vvierrepräseentativenTu umorenaus LSL‐MYCN;D Dbh‐iCreMä äusen………… ………………… …….46 Abb.16: Überrexpression nvonMYCN inTumoren ndesLSL‐MYCN;Dbh‐iCrreModells… ……46 Abb.17: mRN NAExpressionvonNeurroblastomm markerninvierrepräsenntativen T Tumorenau usLSL‐MYCN N;Dbh‐iCreM Mäusen……… ………………… …………………… ………………… ……47 Abb.18: Histologischeun ndmolekulaareCharakteeristikavon Tumorenddes L Dbh‐iCreMo odells………… …………………………………… ………………… ….……………… …..48 LSL‐MYCN;D Abb.19: VerggleichderTu umorinziden nzzwischen ndemLSL‐M MYCN;Dbh‐iCCreModell u unddemTH H‐MYCNMod dell…………… …………………………………… ………………… ………………… …...49 133 Abb.20: Abbildungs‐und Tabellenverzeichnis GenomischeAberrationenvonTumorendesLSL‐MYCN;Dbh‐iCre Modells………………………………………………………………………………..……………………………...50 Abb.21: GensetEnrichmentAnalyse……………………………………………………………………….52 Abb.22: ReguliertemRNAsinTumorendesLSL‐MYCN;Dbh‐iCreMausmodells……….53 Abb.23: RegulierteMYCTargetsinTumorendesLSL‐MYCN;Dbh‐iCreModells………...54 Abb.24: ReguliertemiRNAsinTumorenvonLSL‐MYCN;Dbh‐iCreMäusen……………….55 Abb.25: RegulierteMYCNmiRNATargetsinTumorendesLSL‐MYCN;Dbh‐iCre Modells……………………………………………………………………………………………………………….56 Abb.26: RegulationvonmiR‐542inTumorenderMausmodelle LSL‐MYCN;Dbh‐iCreundTH‐MYCN………………………………………………….……………………57 Abb.27: KorrelationvonmiR‐542‐5pund‐3pExpressionmitprognostischen MarkernundÜberlebeninhumanenNeuroblastomen…………………………………………60 Abb.28: KorrelationvonmiR‐542‐5pund‐3pExpressionmitgünstigemVerlauf humanerNeuroblastome……………………………………………………………………………………..61 Abb.29: KorrelationvonmiR‐542‐3pundSurvivininhumanenNeuroblastomen……62 Abb.30: ReduktionderViabilitätvonhumanenNeuroblastomzelllinienmittels miR‐542‐3pExpression……………………………………………………………………………………….63 Abb.31: ReduzierteKonfluenzvonNeuroblastomzellennachmiR‐542‐3p Expression…………………………………………………………………………………………………………..64 Abb.32: VerschiebungdesZellzyklusdurchgesteigerteS‐Phasein NeuroblastomzellennachmiR‐542‐3pExpression………………………………………………..65 Abb.33: InduktionvonApoptoseundReduktionvonProliferationin NeuroblastomzellennachmiR‐542‐3pExpression………………………………………………..65 Abb.34: HerunterregulationvonSurvivininNeuroblastomzellennachmiR‐542‐3p Expression…………………………………………………………………………………………………………..67 Abb.35: DirekteBindungvonmiR‐542‐3panSurvivin3’UTR………………………………….68 Abb.36: SpezifischeReduktionvonSurvivinProteinmittelssiRNA…………………………69 Abb.37: ReduktionderViabilitäthumanerNeuroblastomzellliniendurch HerunterregulationvonSurvivin………………………………………………………………………….37 Abb.38: VerschiebungdesZellzyklusdurchgesteigerteS‐bzw.G2/M‐Phasein NeuroblastomzellennachHerunterregulationvonSurvivin………………………………….70 Abb.39: ReduktionderProliferationhumanerNeuroblastomzellliniendurch HerunterregulationvonSurvivin………………………………………………………………………….70 Abb.40: SurvivinÜberexpressioninSHEP‐SurvivinnachInduktionmit Tetrazyklin………………………………………………………………………………………………………….71 134 Abb.41: Abbildungs‐und Tabellenverzeichnis PartielleWiederherstellungdesmiR‐542‐3pPhänotypsmittels ÜberexpressionvonSurvivinohnemiR‐542‐3pBindestelle….…………………………..… 72 Abb.42: StrukturvonmiRNA‐beladenenNanopartikeln………………………………………….72 Abb.43: ReduktionderZellviabilitätundderProliferation,Induktionvon ApoptoseundHerunterregulationvonSurvivinmittelsmiR‐542‐3p‐beladenen NanopartikelinWACIIZelleninvitro…………………………………………………………………....74 Abb.44: ExpressionvonmiR‐542‐3pundHerunterregulationvonSurvivinmittels miRNA‐beladenenNanopartikelninWACII‐Xenograftsinvivo………………………………75 Abb.45: HemmungderProliferationundInduktionvonApoptoseinmiR‐542‐3p‐ exprimierendenNeuroblastom‐Xenografts…………………………………………………………...76 Abb.46: RegulationvonKDM1AmRNAinLSL‐MYCN;Dbh‐iCreundTH‐MYCN Tumoren………………………………………………………………………………………………..……………77 Abb.47: 3’UTRvonKDM1AmitBindestellevonmiR‐137………………………………………..78 Abb.48: RegulationvonmiR‐137inLSL‐MYCN;Dbh‐iCreTumoren………………………….78 Abb.49: KorrelationderExpressionvonmiR‐137mitprognostischenMarkern undÜberlebeninhumanenPrimärtumoren………………………………………………………….79 Abb.50: KorrelationvonmiR‐137ExpressionmitgünstigemVerlaufhumaner Neuroblastome……………………………………………………………………………………………………80 Abb.51: ReduktionderViabilitäthumanerNeuroblastomzelllinienmittels exogenermiR‐137Expression……………………………………………………………………………...81 Abb.52: ReduktionderKonfluenzhumanerNeuroblastomzellliniennachexogener miR‐137Expression…………………………………………………………………………………………….81 Abb.53: InduktionvonApoptoseundReduktionderProliferationinmiR‐137 exprimierendenZellen…………………………………………………………………………………………82 Abb.54: EinleitungneuronalerDifferenzierunginmiR‐137‐exprimierendenZellen…82 Abb.55: HerunterregulationvonKDM1AinmiR‐137‐exprimierendenZellen…………..83 Abb.56: DirekteBindungvonmiR‐137andie3’UTRvonKDM1A……………………………84 Abb.57: ReaktionvonKDM1AZielenaufmiR‐137‐induzierteHerunter‐regulation vonKDM1A…………………………………………………………………………………………………………84 Abb.58: SpezifischeReduktionvonKDM1AProteinmittelssiRNA…………………………..85 Abb.59: ReduktionderZellviabilitätdurchHerunterregulationvonKDM1A……………85 Abb.60: InduktionvonApoptoseundReduktionderViabilitätundProliferation vonZellendurchHerunterregulationvonKDM1A…………………………………………………86 Abb.61: InduzierbareExpressionvonKDM1AinSHEP‐KDM1AZellenresistent gegenmiR‐137Expression…………………………………………………………………………………..88 Abb.62: 3’UTRvonAKAP2mitBindestellenvonmiR‐137……………………………………….89 Abb.63: HerunterregulationvonAKAP2mittelsExpressionvonmiR‐137……………….89 135 Abb.64: Abbildun ngs‐und Taabellenverzzeichnis KorrrelationderExpression nvonAKAP2 2mitÜberlebenvon N Neuroblasto ompatienten n……………… ………………… …………………………………… …………………….90 Abb.65: Redu uktionderZ ZellviabilitättundProlifeerationundInduktionvvonApoptosse iinmitsiRNA AgegenAKA AP2transfizziertenSHEP PZellen……… ………………… …………………… …..90 10.2 T Tabellenverzeiichnis Tab..1: InterrnationalNeeuroblastom maStagingSy ystem(INSS S)……………… ……………………..2 Tab..2: Sond denfürdieS SouthernBlootAnalyse… …………………… ………………… ……………………..15 Tab..3: Oligo onukleotidp primerfürP PCR…………… …………………… ………………… …………………… …...20 Tab..4: ProggrammederrqPCR……… ………………… …………………………………… …………………….23 Tab..5: Oligo onukleotidp primerfürq PCR…………………………… …………………… ……23 ………………… Tab..6: Antikörperfürd denimmunoologischenN Nachweisvo onProteinenn…………………..28 Tab..7: Antikörperfürd denimmunh histochemischenNachw weisvonProoteinenauf G nitten………… ………………… …………………… ………………… …………………… ………………… ……30 Gewebeschn Tab..8: AusggesäteZellza ahlenderveerwendeten nZelllinien… …………………… ………………… ……32 Tab..9: Zusaammensetzu ungder69N Neuroblasto ompatienten ndermiRNA AundmRNA A E Expressionss‐Kohorte…………………… …………………………………… ………………… ………………… ……59 Tab..10: Zusaammensetzu ungderPatiientenderN Next‐Generattion‐Sequennzierungs‐ K Kohorte……………………… ………………… …………………… …………………………………… ………………… …….60 136 Anhang 11A Anhang g 11.1 D Danksagung Mein b besonderer Dank giltt Herrn Prrof.Dr.JohaannesSchulte für diee Überlassu ung des hochintteressanten n Themas sowie diie exzellen nte und engagierte e wissensch haftliche Betreuu ungwähren ndmeinerPromotion n. Bei Herrn PDDr.AlexanderrSchramm m und Frau u Prof.Dr. AngelikaE Eggert möcchte ich mich ffür intereessante Diiskussionen n währen nd der Bearbeitunggsphase und u die konstru uktiveKritiikbeiderA AnfertigunggdieserDisssertationsschriftbeddanken. Allen M Mitarbeiterrn des hä ämatologis ch‐onkolog gischen La abors der Kinderkliinik III, besond ders Dr.KathyAstrahantseff, SaabineDreessmann, Joh hannesKösster, SvenL Lindner, EllenM Mahlow, AndreaOde A ersky, An njaRieb, SimonSchä S äfers, NattalieSolom mentsew, Dr.Ann nikaSprüsssel, Dr.HaraldStephaan und Dr.Theresa D aThor, daanke ich für die tatkräfttigeUntersstützung,diiekonstrukktivenDisk kussionenu unddastollleArbeitsklima. B zu u den Projeekten gilt unseren u Ebenfallls mein beesonderer Dank für wichtige Beiträge Kooperrationsparttnern: ‐ Prof.Dr.FrrankSpelem manundAn nneleenBeeckers (Cen nterforMed dicalGenettics,Universitätsklinik kumGhent,,Belgien) ‐ PDDr.Luk kasHeukam mpundAlexxandraFlorrin (InsstitutfürPa athologie,U UniversitätssklinikumK Köln) ‐ PDDr.Lud dgerKlein‐H Hitpass (InsstitutfürZe ellbiologie, Universitäätsklinikum mEssen) Prof.Dr.Sh hirleyKnau uerundSarrahKarczew wski (Zen ntrumfürM Medizinisch heBiotechn nologie,Un niversitätD Duisburg‐Esssen) ‐ ‐ ProfDr.MaatthiasEpp pleundDia naKozlovaa (InsstitutfürAn norganisch heChemie,U UniversitättDuisburg‐‐Essen) nkemeinen nElternund dmeinemM MannChrisstianfürihrreUnterstüützungund dihren Ichdan Rückhaalt. 137 Anhang 11.2 P Publika ationenundKon ngressb beiträge 11.2.1 P Publikatio onen AlthoffK,BeckerssA,OderskyA,MestdaaghP,KöstterJ,BrayIIM,BryanK K,VandesompeleJ, Spelem manF,StallingsRL,Sch hrammA,E EggertA,Sp prüsselA,S SchulteJH. MiR‐13 37 function ns as a tu umor supp pressor in n neurobla astoma byy downreg gulating KDM1A A IntJCancer.2013Feb7.doi::10.1002/iijc.28091.[[Epubahea adofprint] MolenaaarJJ,Domiingo‐FernándezR,Ebu usME,Lind dnerS,KossterJ,DrabeekK,Mestd daghP, vanSlu uisP,ValenttijnLJ,vanNesJ,BroeekmansM,HaneveldF F,Volckma nnR,BrayI, uschK,Kleein‐Hitpass L,Fischer M,Vandesompele Heukam mpL,SprüssselA,ThorrT,Kieckbu J,SchraammA,van nNoeselMM M,VaresioL L,SpelemaanF,EggerttA,StallinggsRL,Caron nHN, VersteeegR,SchultteJH. LIN28B Binducesn neuroblasttomaandeenhancesM MYCNlevellsvialet‐7 7 suppressiion NatGen net.2012No ov;44(11):11 199‐206.do i:10.1038/n ng.2436.Epub2012Occt7. AlthoffK,LindnerrS,Odersky yA,MestdaaghP,Beck kersA,KarcczewskiS,M MolenaarJJJ, BohrerA,KnauerS,Spelema anF,Epple M,KozlovaaD,YoonS,BaekK,Vaandesompe eleJ, A,Schramm mA,SchultteJH EggertA miR‐54 42‐3pexerttstumorsuppressiveefunctionssinneurob blastomab bydown‐ regulattingsurvivvin Manusk kripteingereicht AlthoffK,BeckerssA,Sprüsse elA,LindneerS,ThorT T,dePreterrK,Heukam mpL,Klein‐ HitpasssL,KumpsC,Spelema anF,EggerttA,Schram mmA,SchullteJH AnewC Cre‐condittionalMYC CN‐drivenn neuroblasttomamoussemodelo vercomes limitattionsoftheeestablishe edTH‐MYC CNmodel InVorb bereitung 138 Anhang 11.2.2 Kongressbeiträge Auszüge der Arbeit wurden auf folgenden Konferenzen für einen Vortrag oder eine Posterpräsentationausgewählt: 11.2.2.1 Vorträge AlthoffK,LindnerS,OderskyA,MestdaghP,MolenaarJJ,KnauerS,SpelemanF,BaekK, VandesompeleJ,EggertA,SchrammA,SchulteJH.MiR‐542‐3pexertstumorsuppressive functionsinneuroblastomabydown‐regulatingsurvivin. ‐AdvancedNeuroblastomResearchConference,08.‐21.Juni2012, Toronto,Kanada ‐4thInternationalTuebingenSymposiuminPediatricSolidTumors–FromBench toBedsideinNeuroblastoma,16.‐18.Februar2012,Tübingen 11.2.2.2 Posterpräsentationen AlthoffK,BeckersA,SprüsselA,ThorT,HeukampL,dePreterK,KumpsC,SpelemanF, SchrammA,EggertA,SchulteJH.AnewMYCN‐drivenneuroblastomamousemodelusing Cre‐drivenconditionalexpressionofMYCN. ‐PediatricCancerResearchattheINTERFACE,06.‐08.Juni2013,Wien,Österreich ‐5thAnnualMeetingofNGFN‐PlusandNGFN‐TransferintheProgramofMedical GenomeResearch,11.‐13.Dezember2012,Heidelberg,Deutschland ‐11.ForschungstagderMedizinischenFakultätderUniversitätDuisburg‐Essen, 23.November2012 AlthoffK,LindnerS,OderskyA,MestdaghP,MolenaarJJ,KnauerS,SpelemanF,BaekK, VandesompeleJ,EggertA,SchrammA,SchulteJH.MiR‐542‐3pexertstumorsuppressive functionsinneuroblastomabydown‐regulatingsurvivin. ‐PediatricCancerResearchattheINTERFACE,06.‐08.Juni2013,Wien,Österreich ‐5thAnnualMeetingofNGFN‐PlusandNGFN‐TransferintheProgramofMedical GenomeResearch,11.‐13.Dezember2012,Heidelberg,Deutschland 139 Anhang AlthoffK,BeckersA,OderskyA,MestdaghP,SpelemanF,VandesompeleJ,EggertA, SchrammA,SprüsselA,SchulteJH.MiR‐137functionsasatumorsuppressorin neuroblastomabydownregulatingKDM1A. ‐PediatricCancerResearchattheINTERFACE,06.‐08.Juni2013,Wien,Österreich ‐5thAnnualMeetingofNGFN‐PlusandNGFN‐TransferintheProgramofMedical GenomeResearch,11.‐13.Dezember2012,Heidelberg,Deutschland ‐AdvancedNeuroblastomResearchConference,08.‐21.Juni2012,Toronto, Kanada ‐4thInternationalTuebingenSymposiuminPediatricSolidTumors–FromBench toBedsideinNeuroblastoma,16.‐18.Februar2012,Tübingen,Deutschland 140 Anhang 11.3 L Lebensllauf DerLeb benslaufisttinderOnlline‐Versio onausGrün ndendesDa atenschutzzesnichten nthalten. 141 Anhang 11.4 E Erkläru ungen Hiermitterkläreicch,gem.§6 6Abs.2,fd derPromottionsordnungderMatth.‐Nat.Fak kultäten zurErlaangungderrDr.rer.n nat.,dassic hdasArbeeitsgebiet,d demdasThhema„Etab blierung eines M MYCN‐vermittelten mu urinen Neuuroblastomm modells und Analyse tumorsupp pressiver mikroR RNAs“ zuzu uordnen istt, in Forscchung und d Lehre vertrete undd den Antrrag von KristinaaAlthoffbeefürworte. Essen,d den08.Auggust2013 Prof.Dr.med.JohannesSchuulte Hiermitt erkläre ich, gem. § 7 Abs. 2, c und e der d Promottionsordnuung der Ma ath.‐Nat. des Dr. rrer. nat., dass Fakultääten zur Erlangung E d ich die d vorliegeende Dissertation selbstän ndig verfassst und mich keiner anderen alls der ange egebenen H Hilfsmittel bedient habeun ndallewörrtlichoderinhaltlichü übernomm menenStelle enalssolchhegekennzzeichnet habe. den08.Auggust2013 Essen,d KrristinaAlthhoff d Promottionsordnuung der Ma ath.‐Nat. Hiermitt erkläre ich, gem. § 7 Abs. 2, d und f der Fakultääten zur Errlangung des d Dr. rer.. nat., dasss ich keine anderen P Promotionen bzw. Promottionsversucche in derr Vergangeenheit durcchgeführt habe, h dass diese Arb beit von keiner anderen Fakultät F ab bgelehnt w worden ist, und dass ich die D issertation n nur in diesemVerfahren neinreiche. Essen,d den08.Auggust2013 KrristinaAlthhoff 142