THE CAENORHABDITIS ELEGANS MODEL
Werbung
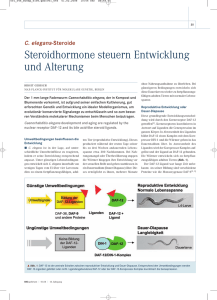
THE CAENORHABDITIS ELEGANS MODEL C. elegans C. elegans Der Name Caenorhabditis elegans stammt aus einer Mischung von griechischen und lateinischen Wörtern. Caeno (griechisch kaeno): neu, ungewöhnlich Rhabdos (griechisch): (griechisch) Rute Stock Rute, Stock Elegans (lateinisch): elegant Der französische Biologe Emile Maupas nannte Caenorhabditis elegans ursprünglich Rhabditis elegans. Erst im Jahre 1952 wurde der Wurm vom deutschen Biologen Günther Osche zu Caenorhabditis elegans umbenannt. Caenorhabditis elegans gehört zu den Nematoden und repräsentiert damit eine weit verbreitete Tierklasse, der ungefähr 20 000 Arten angehören. 1 C. elegans C. elegans Caenorhabditis elegans ist ein im Boden lebender Nematode. Auf Wiesen können bis zu 20 Millionen Nematoden pro m² leben. Nematoden kommen auch als Endoparasiten von Mensch und Tier und als Endo‐ und Ektoparasiten von höheren Pflanzen vor Rund 90 % aller Menschen werden mindestens einmal im höheren Pflanzen vor. Rund 90 % aller Menschen werden mindestens einmal im Leben von parasitischen Nematoden befallen. Der Wurm ist in Bezug auf seine Umweltbedingungen relativ anspruchslos, denn er benötigt für Wachstum und Reproduktion lediglich feuchte Umgebung, gemäßigte Temperatur, atmosphärischen Sauerstoff sowie Bakterien als Nahrungsquelle. C. elegans C. elegans Caenorhabditis elegans ist ein sehr einfach gebauter Nematode, das betrifft sowohl seine Anatomie als auch sein Erbgut, das inzwischen vollständig sequenziert wurde. wurde Die Hermaphroditen (Zwitter) bestehen aus genau 959 Zellen, die Männchen aus 1031 Zellen [Corsi, 2006]. Somit ist Caenorhabditis elegans eine der wenigen Arten, die eine konstante Zellzahl besitzen (Eutelie). Allerdings sterben bei den zwittrigen Individuen von Caenorhabditis elegans durch Apoptose 131 (=12 %) aller im Leben gebildeten somatischen Zellen noch vor Erreichen des Adultstadiums ab. Der adulte Wurm ist ca. 1,2 mm lang und hat einen Durchmesser von maximal 80 µm. Rund 0,2 % der Tiere verlieren während der ersten Phase der Meiose ihr X‐ Chromosom. Deshalb sind die Mehrzahl bei Caenorhabditis elegans g Hermaphroditen (Zwitter). Beide Geschlechter besitzen, mit Ausnahme des Reproduktionstraktes, die gleiche anatomische Grundstruktur, jedoch erscheint der männliche Wurm – unter dem Mikroskop betrachtet – etwas kürzer und schlanker als die Hermaphroditen. Caenorhabditis elegans hat fünf Autosomen und ein X‐Chromosom als Geschlechtschromosom. Die Geschlechtsbestimmung erfolgt durch das Verhältnis von Autosomen zu Heterosomen. Formen mit zwei X‐ Chromosomen sind Hermaphroditen, wo hingegen XO‐Formen Männchen sind. 2 C. elegans C. elegans C. elegans C. elegans Darstellung eines adulten Hermaphroditen von Caenorhabditis elegans A: mikroskopische Aufnahme, zwei ovale Embryos nahe der Vulva sind zu sehen B: schematische Darstellung, linke Seite ist anterior, rechte Seite ist posterior 3 C. elegans C. elegans Zu sehen ist ein erwachsener Hermaphrodit mit Embryos. Sobald die Embryos das 28 – 30 Zellstadium erreichen, werden sie an ihre Umgebung abgegeben. Wenn die Embryogenese abgeschlossen ist, schlüpfen die L1‐Larven und beginnen mit der post‐ embryonalen Entwicklung bei Anwesenheit adäquater Nahrungsquellen. Die Tiere entwickeln sich während des L2‐ entwickeln sich während des L2 L4‐Stadiums bis zum erwachsenen Wurm. Wenn vor der L1/L2‐Häutung ein niedriges Nahrungsangebot, zu viele Tiere oder zu hohe Temperatur vorherrschen, treten die Tiere in einen alternativen Zyklus ein und werden zur Dauerlarve (2d). Als Dauerlarve verweilen sie so lange, bis wieder bessere Bedingungen vorherrschen. Die Größe der Tiere wird in Klammern angezeigt und die Zeit, die die Tiere in jedem Stadium verweilen, ist entlang der Pfeile dargestellt. Unterschiede in Chromosomenzahl und Genomgröße bei verschiedenen biologischen Arten Art Saccharomyces cerevisiae (Hefe) Dictyostelium discoideum (Schleimpilz) Haploide Chromosomenzahl Haploide Genomgröße (Mb) 16 14 7 70 Caenorhabditis elegans (Fadenwurm) 11/12 100 Drosophila melanogaster (Taufliege) 4 170 Gallus domesticus (Huhn) Ga us do est cus ( u ) 39 1.200 00 Mus musculus (Maus) 20 3.000 Xenopus laevis (Krallenfrosch) 18 3.000 Homo sapiens (Mensch) 23 3.000 Zae Mays (Mais) 10 5.000 Allium cepa (Zwiebel) 8 15.000 4 Das Genom von C. elegans RNA INTERFERENCE 5 RNAi RNA interference (RNAi) is the process of mRNA degradation that is induced by double‐stranded RNA in a sequence‐specific manner. RNAi has been observed in all eukaryotes, from yeast to mammals. The power and utility of RNAi for specifically silencing the expression of any gene for which sequence is available has driven its incredibly rapid adoption as a tool for reverse genetics in eukaryotic systems. RNAi RNA interference (RNAi) is the process of mRNA degradation that is induced by double‐stranded RNA in a sequence‐specific manner. RNAi has been observed in all eukaryotes, from yeast to mammals. The power and utility of RNAi for specifically silencing the expression of any gene for which sequence is available has driven its incredibly rapid adoption as a tool for reverse genetics in eukaryotic systems. The RNAi pathway is thought to be an ancient mechanism for protecting the host and its genome against viruses and rogue genetic elements that use double stranded RNA (dsRNA) in their life cycles. double‐stranded RNA (dsRNA) in their life cycles. RNAi is now recognized to be one of a larger set of sequence‐specific cellular responses to RNA, collectively called RNA silencing. These responses have been shown to play a role not only in mRNA and dsRNA stability/degradation, but also in regulation of translation, transcription, chromatin structure, and genome integrity. 6 RNAi Long double‐stranded RNAs (dsRNAs; typically >200 nt) can be used to silence the expression of target genes in a variety of organisms and cell types (e.g., worms, fruit flies, and plants). Upon introduction, the long dsRNAs enter a cellular pathway that is commonly referred to as the RNA interference (RNAi) pathway. First, the dsRNAs get processed into 20‐25 nucleotide (nt) small interfering RNAs (siRNAs) by an RNase III‐like enzyme called Dicer (initiation step). Then, the siRNAs assemble into endoribonuclease‐containing complexes known as RNA‐induced silencing complexes (RISCs), unwinding in the process. The siRNA strands subsequently guide the RISCs to complementary RNA molecules, where they cleave and destroy the cognate RNA (effecter step). Cleavage of cognate RNA takes place near the middle of the region bound by the siRNA strand. RNAi In C. elegans, the demonstration that silencing could be induced by simply feeding nematodes with bacteria that had been engineered to express dsRNA homologous to the target gene, or by soaking worms in such dsRNA drove rapid adoption of RNAi worms in such dsRNA, drove rapid adoption of RNAi as a technology for reverse genetics at the organismal level. 7