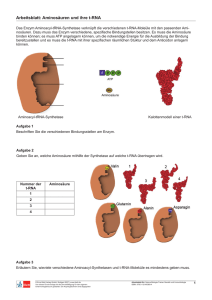
Translation — t-RNA als Vermittler ettelkasten
Werbung
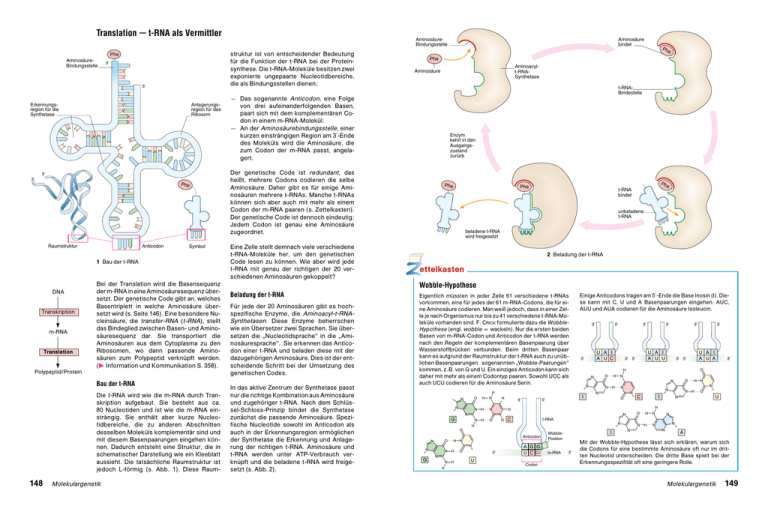
Translation — t-RNA als Vermittler struktur ist von entscheidender Bedeutung für die Funktion der t-RNA bei der Proteinsynthese. Die t-RNA-Moleküle besitzen zwei exponierte ungepaarte Nucleotidbereiche, die als Bindungsstellen dienen: ��� ����������� �������������� �� �� ����������� �������������� ���������� ������������ �������������� ������� �� �� ��� ��������� ������������ ������ 1 Bau der t-RNA DNA Transkription m-RNA Translation Polypeptid/Protein Bei der Translation wird die Basensequenz der m-RNA in eine Aminosäuresequenz übersetzt. Der genetische Code gibt an, welches Basentriplett in welche Aminosäure übersetzt wird (s. Seite 146). Eine besondere Nucleinsäure, die transfer-RNA (t-RNA), stellt das Bindeglied zwischen Basen- und Aminosäuresequenz dar. Sie transportiert die Aminosäuren aus dem Cytoplasma zu den Ribosomen, wo dann passende Aminosäuren zum Polypeptid verknüpft werden. (c Information und Kommunikation S. 358). Bau der t-RNA Die t-RNA wird wie die m-RNA durch Transkription aufgebaut. Sie besteht aus ca. 80 Nucleotiden und ist wie die m-RNA einsträngig. Sie enthält aber kurze Nucleotidbereiche, die zu anderen Abschnitten desselben Moleküls komplementär sind und mit diesem Basenpaarungen eingehen können. Dadurch entsteht eine Struktur, die in schematischer Darstellung wie ein Kleeblatt aussieht. Die tatsächliche Raumstruktur ist jedoch L-förmig (s. Abb. 1). Diese Raum- 148 Molekulargenetik ����������� �������������� ���������� ������ � ���������� ������ ���������� ���������� ������ ����������� — Das sogenannte Anticodon, eine Folge von drei aufeinanderfolgenden Basen, paart sich mit dem komplementären Codon in einem m-RNA-Molekül. — An der Aminosäurebindungsstelle, einer kurzen einsträngigen Region am 3‘-Ende des Moleküls wird die Aminosäure, die zum Codon der m-RNA passt, angelagert. ����� ������������ ��������� ������� ������ Der genetische Code ist redundant, das heißt, mehrere Codons codieren die selbe Aminosäure. Daher gibt es für einige Aminosäuren mehrere t-RNAs. Manche t-RNAs können sich aber auch mit mehr als einem Codon der m-RNA paaren (s. Zettelkasten). Der genetische Code ist dennoch eindeutig: Jedem Codon ist genau eine Aminosäure zugeordnet. Eine Zelle stellt demnach viele verschiedene t-RNA-Moleküle her, um den genetischen Code lesen zu können. Wie aber wird jede t-RNA mit genau der richtigen der 20 verschiedenen Aminosäuren gekoppelt? �� ��� ��� ��� �� � ����� ������ ���������� ����� �������������� ���������������� 2 Beladung der t-RNA - ettelkasten Wobble-Hypothese Beladung der t-RNA Eigentlich müssten in jeder Zelle 61 verschiedene t-RNAs vorkommen, eine für jedes der 61 m-RNA-Codons, die für eine Aminosäure codieren. Man weiß jedoch, dass in einer Zelle je nach Organismus nur bis zu 41 verschiedene t-RNA-Moleküle vorhanden sind. F. Crick formulierte dazu die WobbleHypothese (engl. wobble = wackeln). Nur die ersten beiden Basen von m-RNA-Codon und Anticodon der t-RNA werden nach den Regeln der komplementären Basenpaarung über Wasserstoffbrücken verbunden. Beim dritten Basenpaar kann es aufgrund der Raumstruktur der t-RNA auch zu unüblichen Basenpaarungen – sogenannten „Wobble-Paarungen“ kommen, z. B. von G und U. Ein einziges Anticodon kann sich daher mit mehr als einem Codontyp paaren. Sowohl UCC als auch UCU codieren für die Aminosäure Serin. Für jede der 20 Aminosäuren gibt es hochspezifische Enzyme, die Aminoacyl-t-RNASynthetasen. Diese Enzyme beherrschen wie ein Übersetzer zwei Sprachen. Sie übersetzen die „Nucleotidsprache“ in die „Aminosäuresprache“. Sie erkennen das Anticodon einer t-RNA und beladen diese mit der dazugehörigen Aminosäure. Dies ist der entscheidende Schritt bei der Umsetzung des genetischen Codes. In das aktive Zentrum der Synthetase passt nur die richtige Kombination aus Aminosäure und zugehöriger t-RNA. Nach dem Schlüssel-Schloss-Prinzip bindet die Synthetase zunächst die passende Aminosäure. Spezifische Nucleotide sowohl im Anticodon als auch in der Erkennungsregion ermöglichen der Synthetase die Erkennung und Anlagerung der richtigen t-RNA. Aminosäure und t-RNA werden unter ATP-Verbrauch verknüpft und die beladene t-RNA wird freigesetzt (s. Abb. 2). � � � � � � � � � � �� � �� �� � � � � � � �� �� � � � � � � � �� �� � �� � � � � �� � � � � � � � � � � � � �� � � � � � � � �� ����� ��������� � �� � � � � � � � � � � � � � � �� � � � � � � �� � �� �� �� � � � � � � � � � � � � � Einige Anticodons tragen am 5’-Ende die Base Inosin (I). Diese kann mit C, U und A Basenpaarungen eingehen. AUC, AUU und AUA codieren für die Aminosäure Isoleucin. � � � � � � ��� ����� ����� � ������� �������� �� � � � � � � � � � � Mit der Wobble-Hypothese lässt sich erklären, warum sich die Codons für eine bestimmte Aminosäure oft nur im dritten Nucleotid unterscheiden. Die dritte Base spielt bei der Erkennungsspezifität oft eine geringere Rolle. Molekulargenetik 149