UE Molekulare Biologie und Biochemie
Werbung

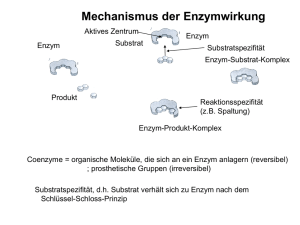
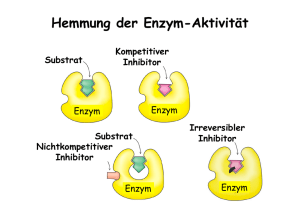
UE Molekulare Biologie und Biochemie Kurs Enzymkinetik: Alkalische Phosphatase Prof. Albert Duschl Kinetik chemischer Reaktionen: Aktivierungs-Energie Enzyme verringern die Aktivierungs-Energie freiwerdende Energie Kinetik chemischer Reaktionen: Geschwindigkeit Energie Verringerung der Aktivierungs-Energie führt zu höherer Reaktionsgeschwindigkeit, weil es mehr Moleküle gibt, deren Energie für eine Überschreitung der Barriere groß genug ist. Y (ohne Enzym) Energie-Verteilung in einem Pool von Molekülen n X (mit Enzym) X Energie Y Alkalische Phosphatase: katalyt. Zentrum Strukturelle Eigenschaften Ser-102 2 Untereinheiten mit je 1 katalytischen Zentrum. Der Rest Ser102 bindet während der enzymatischen Reaktion transient kovalent die Phosphat-Gruppe 2 Zn++ Ionen im katalytischen Zentrum sind für die Aktivität essentiell. 1 Mg++ Ion bindet allosterisch und erhöht die Aktivität. Glycoprotein Typ 4-Membranprotein: Durch kovalent gebundenes Lipid (Phosphatidyl-Inositol-Anker) an der Außenseite der Zell-Membran lokalisiert. A B Alkalische Phosphatase: Funktion Katalysiert die Hydrolyse von Phosphorsäure-Estern: O- O- Ser-102 R—O—P==O R—OH + O- HO—P==O OH2O pH-Optimum im alkalischen Bereich (9 – 11) Beispiele für Substrate: 5‘ Phosphat-Reste an Nucleinsäuren Phosphat-Reste an Proteinen (Ser, Thr, Tyr) ATP Synthetische organische Verbindungen A B Was geschieht im katalytischen Zentrum? Ser-102 O R—O—P==O O- O R—O—P==O O- B Bindung von Zink Zn++ Zn++ O- R—O—P==O OZn++ OR—O—P==O OZn++ Ser-102 Assoziation des Substrats Enzym Ser-102 Ser102 Zn++ O- OH R—O—P==O OZn++ Enzym O R—OH Ser-O-P==O O- Dissoziation des Substrats, Regeneration des Enzyms Enzym Enzym OIOHSer-O-P==O O- O Ser-OH HO-P==O O- Nachweisreaktion p-Nitrophenyl-Phosphat (farblos) Nitrophenolat Anion (gelb) Nitrophenon (gelb) Geschwindigkeit von Enzym-Reaktionen 1) Je mehr Substrat desto höher die Geschwindigkeit, aber ... 2) ... je höher der Besetzungsgrad der vorhandenen Enzym-Moleküle, desto schwieriger ist es für neue Substrat-Moleküle ein freies Enzym zu finden. 3) Wenn alle verfügbaren Enzym-Moleküle mit Substrat besetzt sind, ist die maximal mögliche Geschwindigkeit erreicht. Geschwindigkeit von Enzym-Reaktionen Maximal-Geschwindigkeit 50 Geschwindigkeit Wenn man die ReaktionsVmax Geschwindigkeit gegen die Substrat-Konzentation aufträgt, erhält man eine Vmax / 2 Sättigungskurve, die einem Maximalwert zustrebt 40 30 20 10 0 Die halbmaximale Geschwindigkeit ist erreicht, wenn die Hälfte aller EnzymMoleküle mit Substrat besetzt sind. 0 50 100 150 200 250 Substrat-Konzentration 300 Geschwindigkeit und Affinität Die Michaelis-Konstante 60 Geschwindigkeit Je besser ein Substrat an ein Enzym binden kann, desto geringer ist die SubstratKonzentration, die nötig ist um die Hälfte aller EnzymVmax / 2 Moleküle zu besetzen. 50 40 30 20 10 0 0 200 400 600 800 Substrat-Konzentration 1000 Geschwindigkeit und Affinität Das Michaelis-Menten Diagramm Km ist die Substrat-Konzentration, bei der die halb-maximale Reaktions-Geschwindigkeit erreicht wird. 60 Geschwindigkeit Je höher die Affinität eines Substrates zum Enzym, desto geringer ist die SubstratKonzentration, die nötig ist um die Hälfte aller EnzymVmax / 2 Moleküle zu besetzen. 50 40 30 20 10 0 0 200 400 600 800 Substrat-Konzentration Km ist also eine Konzentration, mit der Dimension mol x l-1 Km 1000 Probleme mit der Hyperbel-Auftragung ? Am Michaelis-Menten Diagramm ist Vmax nur ungenau zu ermitteln. Damit wird auch die Bestimmung von Vmax / 2 ungenau und ... Geschwindigkeit Vmax 50 40 30 20 10 0 0 20 40 60 80 Substrat-Konzentration ... infolgedessen auch die Bestimmung des Km-Werts. Km ungenau 100 Das Lineweaver-Burk Diagramm: ... nur eine andere Darstellung Im Lineweaver-Burk Diagramm werden anstelle von V gegen S die Kehrwerte 1/v gegen 1/S aufgetragen. 0.1 1/v 0.08 0.06 Die Michaelis-Konstante Km finden wir dann exakt am Schnittpunkt der Geraden mit der X-Achse als -1/Km 0.04 0.02 0 -0.01 -0.005 0 0.005 0.01 0.015 0.02 0.025 0.03 1/S - 1 / Km Das Lineweaver-Burk Diagramm: ... hat auch seine Schwächen Im Lineweaver-Burk Diagramm gehen die einzelnen Werte nicht mit gleichem Gewicht ein, einer davon (►) bestimmt die Steigung ganz deutlich. Wenn das Enzym nicht nach Michaelis-Menten geht dann merkt man das nicht immer. 0.1 1/v 0.08 ► 0.06 0.04 0.02 Alternativen: Eadie-Hofstee (v über v/[S]) oder Hanes ([S]/v über [S]). Versuchen sie‘s ruhig mal. 0 -0.01 -0.005 0 0.005 0.01 0.015 0.02 0.025 0.03 1/S - 1 / Km Inhibition von Enzymreaktionen kompetitive Inhibitoren Kompetitive Inhibitoren haben strukturelle Ähnlichkeiten mit dem Substrat und binden reversibel ans katalytische Zentrum. Ein Überschuß an Substrat kann den Inhibitor verdrängen. Km wird dadurch SCHEINBAR erhöht (weil mehr Substrat gebraucht wird, um die Hälfte aller Enzymmoleküle zu besetzen). Geschwindigkeit 60 50 40 30 20 10 0 0 200 400 600 800 Substrat-Konzentration 1000 Inhibition von Enzymreaktionen allosterische Inhibitoren Allosterische Regulatoren binden NICHT an dieselbe Stelle wie das Substrat. Km bleibt daher unverändert, aber Vmax sinkt. Geschwindigkeit 60 50 40 30 20 10 0 0 200 400 600 800 Substrat-Konzentration 1000 Versuch A1 Bestimmung von Km und Vmax der AP 5 Reaktions-Ansätze mit steigenden Substrat-Konzentrationen 1) Pipettieren Sie in der nachstehenden Reihenfolge: Puffer Wasser Substrat 2) Mit jedem Ansatz separat: Starten Sie die Reaktion durch Zugabe des Enzyms, mischen Sie und dann gleich ins Meßgerät 3) Notieren Sie die Absorption genau 20“ und 80“ nach Reaktions-Start. Versuch A1 Bestimmung von Km und Vmax der AP Auswertung: 1. Reaktions-Geschwindigkeit: als Dc/min (aus D OD/min) 1.1. Konzentrations-Änderung: aus DOD nach Lambert-Beer DAbs = e x Dc x d c = DAbs / (e x d) 1.2. V als Dc/min 2. Kehrwerte von V und S 3. Lineweaver-Burk Diagramm zeichnen 4. Km und Vmax grafisch ermitteln Bringen Sie bitte einen Taschenrechner mit! Versuch A2 Kompetitive Inhibition durch Phosphat Als Endprodukt der Reaktion ist Phosphat ein kompetitiver Inhibitor. Durchführung und Auswertung: exakt wie A1, nur mit einem Puffer, der 0.5mM Phosphat enthält. Ki: (beschreibt analog zu Km die Affinität des Inh. zum Enzym die für kompetitive Inhibitionen typische scheinbare Erhöhung von Km läßt sich durch folgende Gleichung ausdrücken: Km‘ = Km x (1 + I/Ki) Km = ohne Inhibitor Km‘ = mit Inhibitor I = Konz. d. Inhibitors [I] kennen Sie aus dem Puffer-Rezept, Km und Km‘ haben Sie gemessen und damit sollten Sie Ki nach obiger Gleichung berechnen können. Versuch B: pH-Optimum der AP Durchführung: wie A1, aber unter Substrat-Sättigung (für Vmax) in allen Ansätzen und in Puffer-Lösungen mit unterschiedlichen pH-Werten Ergebnis: Zeichnen Sie ein Diagramm, in dem die Reaktions-Geschwindigkeit gegen den pH-Wert aufgetragen wird. Versuch C: Allosterische Regulation AP benötigt Zn++ Ionen um aktiv zu sein und wird durch Mg++ zusätzlich aktiviert. EDTA entzieht zweiwertige Kationen und inaktiviert dadurch das Enzym. Dieser EDTA-Effekt läßt sich durch einen Überschuß an Zn++ rückgängig machen. Versuch C: Allosterische Regulation Reaktions-Ansatz 1: 800 µL Puffer 80 µL Substrat 80 µL Wasser 40 µL Enzym Stellen Sie die Küvette ins Photometer und notieren Sie 3 min. lang alle 30“ die Absorption. Reaktions-Ansatz 2: 800 µL Puffer 80 µL Substrat 80 µL Wasser 10 µL EDTA 40 µL Enzym Stellen Sie die Küvette ins Photometer und notieren Sie 3 min. lang alle 30“ die Absorption. Danach geben Sie in dieselbe Küvette 50 µL ZnCl2 und messen nochmals 3 min. wie zuvor. Auswertung: Tragen Sie die Absorptionen aus beiden Experimenten in einem gemeinsamen Diagramm gegen die Zeit auf. Nachtrag: Photometrie Photometrie: Abs = - 10log ( I Io ) Io Beispiele: Abs. I/ Io = 1 - 10log (1) =0 I/ Io = 0.1 - 10log (0.1) =1 I/ Io = 0.01 - 10log (0.01) = 2 I/ Io = 0.001 - 10log (0.001) = 3 I Photometrie: Abs = - 10log ( I Io ) Io Abs = - 10log ( I Io ) =e x I cxd e = ein Proportionalitäts-Faktor (der „molare Extinktions-Koeffizient“) C = die Konzentration D = die Schichtdicke der Küvette Photometrie: Schichtdicke Einfache Schichtdicke 1 Abs: 0.1 1 Abs = - 10log ( I Io ) =e x cxd Doppelte Schichtdicke 1 0.1 0.01 Abs: 1 2 doppelte Schichtdicke ergibt doppelte Absorption Photometrie: Abs = - 10log Konzentration 1 0.1 Abs: 1 1 0.1 0.01 Abs: 1 2 ( I Io ) =e x cxd 1 0.01 Abs: 2 doppelte Konzentration ergibt doppelte Absorption Photometrie: Abs = - 10log Was steckt hinter e, dem molaren Extinktions-Koeffizienten ? ( I Io ) =e x cxd • Die Fähigkeit eines Moleküls, Licht zu absorbieren hängt mit der chemischen Struktur der Substanz zusammen. ( delokalisierte Orbital-Systeme!) • Stellen sie sich vor, Sie lösen einen Löffel Zucker in einer Tasse Wasser. .............. oder einen Löffel Kaffee. ? e ist die Absorption einer 1M Lösung in einer Küvette mit 1cm Schichtdicke [L x mol-1 x cm-1] Protoporphyrin IX (die Grundstruktur von Häm, dem roten Blutfarbstoff)