Reaktionskinetik – Katalyse
Werbung

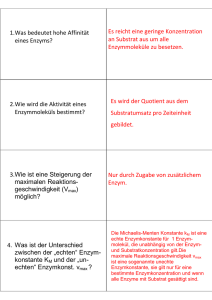
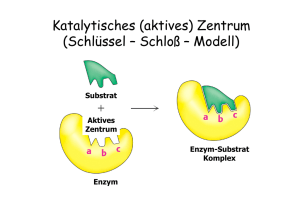
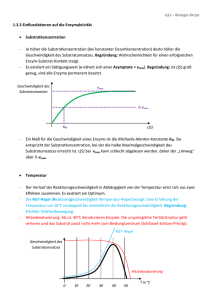
Reaktionskinetik – Katalyse Katalysatoren beschleunigen chemische Reaktionen, ohne das Gleichgewicht zu beeinflussen. Sie beeinflussen nur die Aktivierungsenergie Katalyse Katalysatoren beeinflussen den Reaktionsweg (Mechanismus), nicht aber die Nettoreaktion und Größen selbiger (∆ usw.) Absenken der Aktivierungsenergie Reaktion läuft deutlich schneller ab KEINE Änderung der GG-Konstanten! Die Reaktion muss nicht über den “ganzen Berg” Alter Katalysator (alter Bahntunnel) – geringe Absenkung der Akt-Energie Neuer Katalysator (NEAT-Tunnel): Größere Absenkung der Akt-Energie Erstfeld und Bellinziona bleiben auf gleicher Höhe! Nur Zeit wird verändert! Katalyse Auf eine Reaktion angewendet: Der geschwindigkeitsbestimmende Schritt wird im alternativen Reaktionsweg umgangen Unkatalysierte Reaktion: Ea = 76 kJ/mol Epot Nicht beschleunigt mit I2 als Katalysator: Ea = 57 kJ/mol Um Faktor 2000 beschleunigt 2 H2O2 2 H2O + O2 mit Katalase als Katalysator: Ea = 8 kJ/mol Um Faktor 1015 beschleunigt Rn (Zersetzung von Wasserstoffperoxid) Katalyse Arten der Katalyse: Homogene Katalyse: Katalysator befindet sich in gleichem Phase wie die Reaktanden (z.B. I2 o. Enzyme bei H2O2 Zersetzung) Heterogene Katalyse: Katalysator befindet sich in einer anderen Phase als die Reaktanden (öfters Metalle oder Oxide (Pd, Pt Y2O5) Homogene Katalyse: HBr katalysiert auch die Zersetzung von H2O2 Reaktion verläuft vermutlich über ein vorgelagertes Gleichgewicht. Beobachtetes Geschwindigkeitsgesetz: (Bsp für eine Säurekatalyse) Säure- und Basenkatalyse Säurekatalyse: Zentraler Schritt ist die Übertragung eines Protons auf das Substrat X + HA HX+ + A- HX+ Produkte z.B. Esterhydrolyse, Keto-Enol Tautomerie Basenkatalyse: Zentraler Schritt ist die Übertragung eines Protons vom Substrat XH + B X- X- + BH+ Produkte z.B. Claisenkondensation, Aldolkondensation Enzymkatalyse Enzyme sind meist hochspezifische homogene Biokatalysatoren mit hoher katalytischer Aktivität. Damit gibt es meist schnelle mit keinen Nebenreaktionen Aktives Zentrum zur Bindung von Substraten Polypeptid (Protein Enzym) oder katalytisch aktive RNA ( Ribozym) oder Protein/RNA Komplex (z.B. Ribosom) Spezifisches Substrat – sehr selektive nichtkovalente WW (HBrücken, VdW,…) Enzym (E) + Substrat (S) Enzym-Substrat-Komplex (ES) Enzymkatalyse Enzyme haben regulierbare Aktivität über: a) Feedback: Enzym wird durch Endprodukt einer katalytischen Kette herunter reguliert (reversible Produkthemmung) b) Regulation: Ein Stoff (z.B. Ca2+) bindet an das Enzym und verändert die Konformation, so dass es aktiv wird. Oftmals aktiviert dieser Komplex das nächste Enzym (Regulatorprotein) c) Modifikation: Kovalente Veränderung mit neuen funktionellen Gruppen (z.B. Phosphorylierung) d) Proteolyse: Das aktive Enzym wird durch die Abspaltung eines Peptidstückchens erzeugt (z.B. Trypsin) Katalase Trypsin Amylase Enzymkatalyse Modellvorstellungen: Schlüssel-Schloss-Prinzip (E.Fischer, 1890) 3D-Struktur des Substrats und des aktiven Zentrums passen perfekt zueinander Adsoption von Substrat an die Oberfläche: Geringe cS dann ist Hohe cS dann ist Reaktionsgeschwindigkeit v Besser: Induktiver Fit (Enzym passt sich an Substrat an) Konzentration [S] Michaelis-Menden-Modell Enzym und Substrat stehen mit Enzym-Substrat Komplex im Gleichgewicht Kinetik mit vorgelagertem Gleichgewicht E+S k1 k’-1 ES k2 E+P Wie bei anderen Reaktionen, ist nun ein Ausdruck für die Reaktionsgeschwindigkeit gesucht - in Abhängigkeit von cE und cS Produktbildender Schritt wird als irreversibel angenommen v = k2 cES cES = ? Michaelis-Menten-Modell Wichtige Annahmen zur Herleitung der Formel: Enzym-Substrat-Komplex liegt quasistationär vor Michaelis-MentenKonstante: Damit ergibt sich für cES Falls ≪ dann gilt , Also: Geschwindigkeiten Komplexbildung = Komplexzerfall dcES 0 dt K M cES k 1 k 2 k1 Resultierende Einheit: mol/l c E , 0 cS , 0 cS , 0 K M ≪ , v k 2 cE ,0 vmax Michaelis-Menten-Gleichung: v vmax v k 2 cE , 0 cS , 0 cS , 0 K M Alle Bindungsstellen mit Substrat gesättigt cS , 0 cS , 0 K M Michaelis-Menten-Modell Michaelis-Menten-Gleichung: Bei cS,0 = KM gilt: v vmax 2 v vmax vmax cS , 0 cS , 0 K M Wie werden und KM bestimmt? 1 Lineweaver-Burk-Auftragung KM 1 1 1 v vmax vmax cS 1 1 Alternativ (seltener): Eadie-Hofstee-Auftragung v vmax K M v cS Michaelis-Menten-Modell Bedeutung von KM und vmax Wenn KM = cS Substratkonzentration, bei der die Hälfte der aktiven Zentren besetzt ist Wenn k2 ≪k-1 KM ist die GG-Konstante für die Dissoziation des ES-Komplexes Typische Werte für KM TON = “turnover number” (Katalysevorgänge pro Sekunde) Bsp: Carboanhydrase: CO2 + H20 HCO3- + H+ Katalytische Effizienz mit KM ist meist 10-1 bis 10-7 M , Typische Zahlenwerte: 100 – 105 s-1 Bei 8 ∗ 10 und 6 ∗ 10 perfekte Diffusionskontrolle direkter Umsatz! 108 – 109 ∗