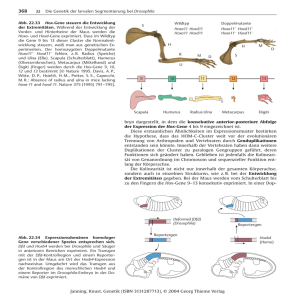
NicoleTroelenbergDissertation
Werbung

Die Funktion von germ cell-less und homeobrain in der Differenzierung terminaler Identitäten im Kurzkeim-Insekt Tribolium castaneum Der Naturwissenschaftlichen Fakultät der Friedrich-Alexander-Universität Erlangen-Nürnberg zur Erlangung des Doktorgrades Dr. rer. nat. vorgelegt von Nicole Troelenberg aus Magdeburg Als Dissertation genehmigt von der Naturwissenschaftlichen Fakultät der Friedrich-Alexander Universität Erlangen-Nürnberg Tag der mündlichen Prüfung: 27.10.2014 Vorsitzende/r des Promotionsorgans: Prof. Dr. Johannes Barth Gutachter/in: Prof. Dr. Martin Klingler Prof. Dr. Manfred Frasch Inhaltsverzeichnis Inhaltsverzeichnis Zusammenfassung.......................................................................................................VI Summary .................................................................................................................. VIII Abbildungsverzeichnis................................................................................................. X Tabellenverzeichnis .................................................................................................. XII Abkürzungsverzeichnis ............................................................................................ XIII 1. Einleitung .............................................................................................................14 1.1 Allgemeine Einleitung .......................................................................................14 1.1.1 Von der Oozyte zum Organismus in 24 Stunden – die embryonale Entwicklung von Drosophila melanogaster .......................................16 1.1.2 Tribolium castaneum als Modellsystem..............................................20 1.2 Ein genomweiter RNAi-Screen als Alternative zum Kandidaten-Gen-Ansatz.24 1.2.1 Ziele des Screens und der einzelnen Analyseschritte..........................26 1.2.2 Etablierung und Aufrechterhaltung der Qualität .................................28 1.3 germ cell-less .....................................................................................................30 1.3.1 germ cell-less in Drosophila ...............................................................30 1.3.2 germ cell-less in Mus musculus...........................................................31 1.3.3 germ cell-less im Menschen ................................................................32 1.4 homeobrain ........................................................................................................34 1.5 Ziele dieser Arbeit .............................................................................................35 2. Material und Methoden ........................................................................................36 2.1 Puffer und Reagenzien ......................................................................................36 2.2 Tribolium castaneum im Labor .........................................................................36 2.3 Parentale RNAi..................................................................................................37 2.3.1 Alter und Geschlecht der parentalen Generation ................................37 2.3.2 dsRNA-Injektion .................................................................................37 2.3.3 Eiablage und Auswertung ...................................................................38 2.4 Fixierung und In-Situ-Hybridisierung ...............................................................39 2.5 Kutikula- und Lebendpräparate .........................................................................41 2.6 Embryonale Injektionen ....................................................................................42 2.7 Amplifikation individueller Genfragmente zur Generierung von dsRNA und DIG-Sonden.......................................................................................................43 2.8 In-Vitro-Transkription der genspezifischen dsRNA .........................................45 2.9 In-Vitro-Transkription genspezifischer Sonden ................................................46 2.10 Fluoreszenzmarkierung spezifischer Proteine ...................................................47 2.10.1 Generierung des gcl-Vektors...............................................................47 2.10.2 Doppelverdau des pCS2-Vektors sowie des gcl-Fragments ...............49 2.10.3 Generierung des gcl-Klons ..................................................................49 2.10.4 Einbau von EGFP................................................................................51 2.11 Antikörperproduktion und Immunohistochemische Färbungen........................52 III Inhaltsverzeichnis 2.11.1 2.11.2 2.11.3 Generierung von polyklonalen Peptid-Antikörpern gegen GCL ........52 Antikörper-Färbung in Embryonen .....................................................52 Antikörper-Färbung im Tribolium Ovar .............................................53 3. Ergebnisse ...............................................................................................................55 3.1.1 Defekte der Ovario- oder Oogenese nach pupaler Injektion ...............57 3.1.2 Defekte der larvalen Körpermuskulatur nach pupaler Injektion .........60 3.1.2 Defekte der Embryogenese nach parentaler RNAi .............................63 3.2 Vom Screen zum Kandidaten-Gen ....................................................................67 3.3 Analyse der Funktionen von germ cell-less und homeobrain ...........................70 3.3.1 Parentale RNAi von germ cell-less und homeobrain induziert posteriore Strukturen am anterioren Pol..............................................70 3.3.2 Tc-gcl wird maternal, Tc-hbn aber zygotisch exprimiert ....................75 3.3.3 Sequenzanalyse von gcl und hbn offenbart konservierte Bindedomänen.....................................................................................78 3.3.4 Veränderungen des Blastoderm-Anlagenplans nach Verlust von gcl und hbn ................................................................................................82 3.3.5 germ cell-less- und torso-Doppel-RNAi führt zum Verlust nahezu aller Segmente .....................................................................................91 3.3.6 hbn ist unabhängig vom Terminalen System ......................................93 3.3.7 Durch gcl-RNAi verursachte Defekte in Keimstreifen .......................94 3.3.8 Veränderung der morphogenetische Bewegungen nach gcl-RNAi ....97 3.3.9 Die Lokalisation von Tc-GCL im frühen Embro und innerhalb der Zellen bleibt ungewiss.......................................................................102 3.3.10 Die Beteiligung bekannter Bindungspartner von Gcl ist in Tribolium fraglich ..............................................................................................104 3.3.11 RNAi von gcl führt zu einem selektiven Verlust maternaler Positionsinformation ........................................................................................108 3.3.12 Embryonale RNAi führt nur bei hbn zum Doppel-Abdomen-Phänotyp110 4. Diskussion ..........................................................................................................112 4.1 Der genomweite RNAi-Screen iBeetle als Methode für die Identifizierung von Entwicklungsgenen..........................................................................................112 4.1.1 Der iBeetle-Screen identifizierte interessante Kandidatengene für alle untersuchten Prozesse .......................................................................112 4.1.2 Grundlagen des Erfolgs von iBeetle..................................................115 4.2 Die Rolle von genetischem Hintergrund und „off-target“-Effekten bei der Verifizierung der Kandidatengene ..................................................................116 4.3 Doppelabdomina im Kurzkeim- Insekt Tribolium ...........................................119 4.3.1 Der Ausbildung von Doppelabdomen-Phänotypen liegen in Tribolium andere Mechanismen zugrunde als in Drosophila ............................119 4.3.2 Tc-germ cell-less und Tc-homeobrain arbeiten mit dem anterioren terminalen System, um anteriore Schicksale einzuleiten ..................122 4.3.3 Wo wirkt Tc-gcl?...............................................................................125 4.3.4 Das Auftreten verschiedener Phänotypen deutet auf multiple Funktionen von hbn...........................................................................128 IV Inhaltsverzeichnis 4.3.5 Differenzierung terminaler Identitäten in Tribolium.........................130 Anhang A – Puffer und Reagenzien .........................................................................134 Anhang B - verwendete Primer.................................................................................135 Anhang C – Vektorkarte pCS2 + gcl-eGFP .............................................................138 Anhang D –Rescreen ................................................................................................139 Anhang E – Ergebnisse zu iB_00736 (staufen) ........................................................141 Anhang F - Ergebnisse zur Positivkontrolle tiotsh ...................................................145 Literaturverzeichnis ..................................................................................................147 Lebenslauf .................................................................................................................165 Publikationen und Vorträge ......................................................................................166 Danksagung...............................................................................................................167 Eidesstattliche Erklärung ..........................................................................................168 V Zusammenfassung Zusammenfassung Im Langkeim-Insekt Drosophila erfolgt die Segmentierung des Embryos entlang der antero-posterioren Achse bereits im Synzytium, initiiert durch drei maternale Gradientensysteme. Der Anlagenplan des phylogenetisch älteren Kurzkeim-Insekts Tribolium castaneum hingegen beinhaltet nur Kopf und thorakale Segmente. Frühere Studien zeigten, dass von den drei maternalen Signalen in Drosophila nur das terminale System in Tribolium eine ähnlich wichtige Rolle in der Segmentierung spielt. Demnach sind essentielle Faktoren für die Etablierung der a-p Achse in Tribolium noch unbekannt. Da die Analyse von Drosophila-Orthologen in Tribolium erschöpft ist, nahm unser Labor am „iBeetle“-Screen teil, einem genomweiten Screen, der auf der Methode der RNA-Interferenz basiert. Mit Hilfe dieses Screens sollten neue Kurzkeim-spezifische Segmentierungsgene in Tribolium identifiziert werden. Der systemische Effekt der RNAi in Tribolium ermöglicht es dabei, wichtige Segmentierungsgene durch die Analyse von L1-Larven dsRNAi-injizierter Mütter zu entdecken. Im ersten Teil dieser Arbeit stelle ich meine Ergebnisse aus dem Screen vor und beschreibe einige interessante Beispiele von Genen / Phänotypen, die ich im Rahmen dieses wissenschaftlichen Großprojekts identifizieren konnte. Anschließend gehe ich auf meine Folgeanalysen möglicher Segmentierungs-Kandidatengene ein. Dabei sollten früh aktive (maternale) Faktoren anhand von hochpenetranten Defekten in den Regionen, die aus den Anlagen nahe der Eipole hervorgehen, identifiziert werden. Im Hauptteil der Arbeit beschreibe ich zwei Gene, deren Knockdown im Screen zu Embryonen mit spiegelsymmetrischen Doppelabdomen führte, ähnlich der bicaudal-Mutante in Drosophila. Die Phänotypen von germ cell-less, einem Gen, das in der Fliege für die Determination der Polzellen notwendig ist, und von homeobrain, das in Drosophila an der Entwicklung des Gehirns beteiligt ist, konnte ich mit Hilfe nicht-überlappender Fragmente verifizieren. Weiterhin bestätigte sich, dass sowohl germ cell-less wie auch homeobrain eine Rolle in der Entwicklung anteriorer Strukturen innehaben. Allerdings basiert diese Funktion im Falle von germ cell-less wahrscheinlich auf der Regulierung maternaler Faktoren in den Nährzellen des Ovars, während homeobrain als Kopf-Gapgen im Blastoderm aktiv wird. In Zusammenarbeit mit dem terminalen System wird so an den Polen das Schicksal anterior vs. posterior festgelegt. VI Zusammenfassung Die Tribolium-Orthologe von germ cell-less und homeobrain unterscheiden sich in ihrer Funktion also signifikant von ihrer Rolle in Drosophila. Diese Arbeit bietet somit neue Einsichten in die Evolution dieser beiden Gene und in die Evolution des terminalen Systems zwischen Kurzkeim- und Langkeim-Insekten. Auch wenn der molekulare Mechanismus der GCL-Funktion in Tribolium letztlich noch nicht geklärt werden konnte, hat diese Arbeit doch gezeigt, dass auch in Kurzkeim-Embryonen die inhärente Symmetrie des terminalen Systems letztlich für Neigung zur Bildung von Doppelabdomina verantwortlich ist, dass aber ein anderer Mechanismus dazu dient, die beiden terminalen Regionen voneinander zu unterscheiden. VII Summary Summary In the embryo of the long germ insect Drosophila melanogaster, three maternal gradient systems pattern the antero-posterior axis during the syncytial blastoderm stage. In contrast, the blastoderm fate map of the short germ beetle Tribolium castaneum only includes head and thoracic segments. Previous studies revealed that of the maternal signals known from Drosophila only the terminal system has a similar function in Tribolium segmentation. Thus, essential maternal factors establishing the a-p axis are still missing. With the candidate gene approach exhausted, our lab participated in the iBeetle screen, a genome wide screen using RNA-Interference, to identify shortgerm specific segmentation genes in Tribolium. RNAi was shown to be systemic in Tribolium, which provides the opportunity to find segmentation genes by analyzing L1-larvae of RNAi-treated mothers. The first part of the thesis is based on that screen. I will describe my results and present examples of genes I identified, which are relevant to the projects involved in this scientific undertaking. Then I explain the follow up analysis of promising candidate genes showing defects/abnormalities with high penetrance in regions derived from primordia near the egg poles - as expected for early maternal patterning functions. In the screen the knockdown of two genes resulted in mirror image embryos with two abdomina, comparable to the bicaudal phenotype in Drosophila: germ cell less, a gene only required for pole cell determination in the fly, and homeobrain which is involved in brain development in Drosophila. These genes were analyzed in the second part of my thesis. I could show that both of them are indeed necessary for proper anterior development. However, my results indicate that germ cell-less probably exerts its function in the nurse cells, possibly by regulating the synthesis or transport of other maternal factors. homeobrain, on the other hand, acts as a transcription factor / gap gene at the blastoderm stage, transforming the anterior terminal region to fates different from those at the posterior pole. Thus, my thesis reveals functions of the Tribolium orthologs of germ cell-less and homeobrain quite different from those in Drosophila. It provides insight into the evolution of these genes as well as the evolution of the terminal system between short germ and long germ insects. While the exact molecular mechanisms of GCL has not VIII Summary yet been unraveled, my work has shown that also in shortgerm embryos the inherent symmetry of the terminal system is responsible for the propensity of these embryos to form mirror-symmetric abdomina, but that a different mechanism is employed to distinguish anterior and posterior terminal regions. IX Abbildungsverzeichnis Abbildungsverzeichnis Abbildung 1: Die Segmentierungskaskade von Drosophila melanogaster ............... 19 Abbildung 2: Anlagenpläne von Lang- und Kurzkeim- Embryo ............................... 21 Abbildung 3: iBeetle-Schema für pupale Screener.................................................... 28 Abbildung 4: Schematische Darstellung parentaler RNAi in Tribolium ................... 38 Abbildung 5: Schematische Darstellung einer embryonalen Injektion ..................... 42 Abbildung 6: iBeetle-Primer-Design nach Eupheria ................................................. 45 Abbildung 7: Übersicht des zur Gcl-Überexpression verwandten Vektors pCS2 ..... 47 Abbildung 8: Schematische Übersicht zur Klonierung von gcl in den Vektor pCS2 50 Abbildung 9: Übersicht des pCS2-gcl-Vektors ......................................................... 50 Abbildung 10: Zusammenfassung meiner Ergebnisse (pupaler RNAi, Stand Dezember 2011) ............................................................................................... 56 Abbildung 11: Ovar-Defekte von pupal injizierten Tieren im Vergleich mit dem Wildtyp . ........................................................................................................... 59 Abbildung 12: Primäre Muskelphänotypen von L1-Larven nach parentaler RNAi .. 62 Abbildung 13: Beispiele für Defekte der Embryonalentwicklung nach pupaler RNAi .......................................................................................................................... 66 Abbildung 14: Vom Screen zum Kandidatengen ...................................................... 69 Abbildung 15: Zusammenfassung der Phänotyp-Verifizierung (Rescreen) .............. 69 Abbildung 16: Phänotypische Serie der Defekte nach pRNAi gegen gcl ................. 71 Abbildung 17: Phänotypen nach gcl-RNAi in Abhängigkeit von der Konzentration 72 Abbildung 18: Phänotypische Serie der Defekte nach pRNAi gegen hbn ................ 73 Abbildung 19: Phänotypen nach hbn-RNAi in Abhängigkeit von der Konzentration .......................................................................................................................... 74 Abbildung 20: Expression von Tribolium-germ cell-less .......................................... 76 Abbildung 21: Expression von Tribolium-homeobrain ............................................. 76 Abbildung 22: Position der für die gcl-RNAi verwendeten dsRNA-Fragmente ....... 79 Abbildung 23: Sequenzvergleich der BTB/POZ-Domäne von GCL in verschiedenen Spezies .............................................................................................................. 79 Abbildung 24: Position des für die hbn-RNAi verwendeten dsRNA-Fragments ...... 80 Abbildung 25: Sequenzvergleich der Aminosäuren von HBN in Tribolium und Drosophila ........................................................................................................ 81 Abbildung 26: Veränderungen des Blastoderm-Anlagenplans nach Verlust von gcl und hbn ............................................................................................................. 85 Abbildung 27: Der Verlust anteriorer Domänen von giant und wingless nach gclRNAi................................................................................................................. 87 Abbildung 28: hbn-RNAi führt zur Expression von caudal im Kopf früher Keimstreifen ..................................................................................................... 88 Abbildung 29: Veränderungen extraembryonaler Anlagen nach gcl- bzw. hbn-RNAi .......................................................................................................................... 90 Abbildung 30: Defekte nach germ cell-less/torso-Doppel-RNAi ............................. 92 Abbildung 31: Verteilung der Phänotypen nach gcl/tor-Doppel-RNAi .................... 92 Abbildung 32: Die Expression von hbn ist nach Torso-RNAi unverändert .............. 93 Abbildung 33: Keimstreifen mit gestörter Segmentierung nach gcl-RNAi............... 95 Abbildung 34a und b: Die Veränderung der morphogenetischer Bewegungen nach gcl-RNAi führt zu einem Riss im extraembryonalen Gewebe ....................... 100 X Abbildungsverzeichnis Abbildung 35: Antikörperfärbung von GCL-Protein in Embryo und Ovar bleibt ergebnislos ...................................................................................................... 103 Abbildung 36: Defekte nach adulter RNAi gegen DP (TC010400) ........................ 106 Abbildung 37: Defekte nach adulter RNAi gegen E2F ........................................... 106 Abbildung 38: Phänotypen nach E2F-Einzel- und Doppel-RNAi .......................... 107 Abbildung 39: Expression maternaler mRNAs nach gcl und hbn-RNAi ................ 109 Abbildung 40: Verifizierung der zygotischen Funktion von hbn durch eRNAi...... 111 Abbildung 41: Modell zur Differenzierung terminaler Identitäten in Tribolium .... 132 Abbildung 42: Vektorkarte des pCS2-gcl-egfp-Plasmids........................................ 138 XI Tabellenverzeichnis Tabellenverzeichnis Tabelle 1: Generationszeiten von Tribolium in Abhängigkeit von der Temperatur .. 37 Tabelle 2: Sequenzidentität unterschiedlicher GCL-Proteine.................................... 79 Tabelle 3: Sequenzidentität von Drosophila- und Tribolium-HOMEOBRAIN im Vergleich ................................................................................................... 80 XII Abkürzungsverzeichnis Abkürzungsverzeichnis A BW bcd C cad d Dmel dsRNA eg en eve F FW gcl hb hbn ISH L NoF OF pBA19 PCR Pg R RNAi RT SB sRNA T Tch Tr Tt U V wg WT abdominales Segment rückwärts (backward) bicoid Coxa caudal destilliert Drosophila doppelsträngige RNA eagle engrailed even-skipped Femur vorwärts (forward) germ cell-less hunchback homeobrain in-Situ-Hybridisierung larvales Stadium mit dem Original iBeetle-Fragment nicht überlappendes Fragment (Non-overlapping fragment) Originalfragment Käferstamm „pearl“ mit eGFP Insertion Polymerase-Ketten-Reaktion (Polymerase chain reaction) Pygopodien Reverse RNA-Interferenz Raumtemperatur Käferstamm San Bernardino einzelsträngige RNA thorakales Segment Trochanter Tropharium Tibiotarsus Urogomphi Vitellarium wingless Wildtyp XIII 1. Einleitung 1. Einleitung 1.1 Allgemeine Einleitung „(...) Neither natural selection nor DNA directly explains how individual forms are made or how they evolved. The key to understanding form is development, the process through which a single-celled egg gives rise to a complex, multi-billion celled animal.“ (Sean B. Carroll, Endless Forms Most Beautiful – The New Science of Evo Devo, 2005) Im Zuge der Evolution erschloss der Mensch logische und abstrakte Vorgänge und begann, die eigene Existenz zu hinterfragen. Neben der Suche nach dem Sinn des Lebens und dem eigenen Platz darin, versucht der Mensch, die Entstehung des Lebens und der vorhandenen Vielfalt, aber auch die unmittelbare Reproduktion der eigenen Art zu verstehen. Die erste dokumentierte ‚methodische’ Abhandlung über Embryogenese geht auf den griechischen Philosophen Aristoteles zurück. Sein Buch De generatione animalium, entstanden im 4 Jh. vor Christus, dokumentiert Geschlechter verschiedener Spezies, klassifiziert erstmals verschiedene Fortpflanzungsformen und diskutiert den Akt der Zeugung sowie die embryonale Entwicklung von Tier und Mensch. (www. http://embryo.asu.edu/ - The Embryo Project Encyclopedia). Im Gegensatz zu anderen Philosophen, wie zum Beispiel Empedokles, welche die Meinung vertraten, dass nicht nur beide Geschlechter einen Samen trügen, sondern, dass diese Samen einen ‚zerlegten’ Organismus beinhalten, der im Leib des Weibchens willkürlich wieder zusammengesetzt wird, folgte Aristoteles der ‚Theorie des bestellten Felds’. Nach dieser birgt der männliche Samen alle wichtigen Informationen, während das Weibchen nur für die Versorgung des Embryonen zuständig ist. Grund für seine Ablehnung der ‚zerlegter Organismus’-Theorie waren Beobachtungen an Hühner- Embryonen, in welchen sich zuerst das Herz, gefolgt von anderen Organen und schließlich äußeren Strukturen forme. Aristoteles folgerte, dass das Weibchen wahrscheinlich mit ihrem menstrualen Blut das Material liefere, das Männchen indes die treibende Energie sei, die den Anstoß zu neuem Leben gäbe und dieses gestalte (Boylan, 1984). Aristoteles’ Theorie der Epigenese, nach der ein Organismus erst nach und nach aus einer undifferenzierten Masse wächst, war bis ins 17. Jahrhundert vorherrschend, 14 1. Einleitung wurde aber im Zuge der Aufklärung von der Präformationslehre abgelöst. Diese basiert auf Anaxagoras, einem Philosophen des 5. Jahrhunderts vor Christus, und besagt, dass ein Organismus in seiner Gesamtheit bereits im Sperma des Männchens vorgebildet ist und die Zeit im Mutterleib einer Phase graduellen Wachstums gleichzustellen ist. Diese Idee hielt sich hartnäckig in den Lehrbüchern, obwohl Caspar F. Wolff im Jahr 1812 am Darmkanal des Hühnchens das Entstehen eines Organs von der Anlage bis zur vollständigen Ausbildung dokumentierte. Ausgehend von Wolffs Arbeiten entwickelte Carl Ernst von Baer das Prinzip der Progression, nach welchem die Differenzierung des Embryos auf eine stufenweise Spezialisierung zurückzuführen ist. Zusammen mit Heinz Christian Pander postulierte er die Keimblatt-Theorie und die Idee, dass ein Embryo zuerst generelle Anlagen der Gruppe ausbildet, zu der er gehört, bevor er sich artspezifisch differenziert (von Baer, 1828). Mitte des 19. Jahrhunderts publizierte Darwin seine Abhandlungen zur natürlichen Selektion und führte darin Variationen zwischen, aber auch innerhalb der Arten, zum Beispiel in Ameisenstaaten, auf äußere Einflüsse zurück (Darwin, 1859). Inspiriert von Darwins Ideen und anderen vorherrschenden Evolutionstheorien ihrer Zeit widmeten sich Forscher wie Weismann und Roux der Untersuchung von Seeigel- und Frosch-Embryonen. Ihre Experimente führten sie letztendlich zu dem Schluss, dass die Strukturen des Embryos in einer komplexen Architektur vordisponiert sein müssen - in den Zellen selbst (Mosaiktheorie nach Roux) oder im Keimplasma (Keimplasmatheorie nach Weismann) (Hertwig, 1906 und 2013). Die Arbeiten von Wolff, von Baer und Darwin sowie die von Mendel aufgestellten Gesetze zur Vererbung von Eigenschaften waren aber auch Inspiration für Entwicklungsbiologen wie Thomas Hunt Morgan. Er mutmaßte, dass die Entwicklung eines Organismus auf dem Zusammenspiel von Umwelt und Kern der befruchteten Eizelle basiere. Seine Arbeit über die Vererbung spezifischer Merkmale in der Taufliege Drosophila melanogaster, in der er die Chromosomen als Träger der Erbinformationen und die Keimzellen wiederum als deren Träger beleuchtete, wurde 1933 mit dem Nobelpreis in Medizin belohnt. Weiterhin etablierte Morgan Drosophila als Modellorganismus (Morgan, 1915) und bereitete damit den Boden für die molekulargenetische Wiedergeburt der Entwicklungsbiologie. 15 1. Einleitung 1.1.1 Von der Oozyte zum Organismus in 24 Stunden – die embryonale Entwicklung von Drosophila melanogaster Gegenwärtig zählt die "Schwarzbäuchige Taufliege" Drosophila melanogaster, neben dem Fadenwurm Caenorhabditis elegans, der Maus Mus musculus und dem Zebrafisch Danio rerio, zu den wichtigsten tierischen Modellorganismen. Durch die Sequenzierung des Genoms im Jahr 2000 sowie der Entwicklung einer Vielzahl molekularbiologischer und genetischer Verfahren wurde die Erforschung zellulärer Prozesse sowie entwicklungsbiologischer Abläufe immens erleichtert (Adams et al., 2000). Die Entwicklung eines neuen Organismus beginnt auch bei Drosophila bereits im Leib der Mutter. Zu Beginn der sogenannten Oogenese durchläuft der Zytoblast vier unvollständige Zellteilungen, an deren Ende ein 16-Zell-Cluster steht, dessen Zellen über Zytoplasmabrücken miteinander verbunden sind (King, 1970). Im Verlauf der Oogenese pumpen die Nährzellen - die 15 polyploiden Schwesterzellen der Oozyte ihr Zytoplasma in das reifende Ei. Dabei werden Nährstoffe, Proteine und Ribonukleinsäuren (RNAs) in das Ei verbracht, die u.a. auch für die Steuerung der Musterbildung im Blastoderm-Embryo, einschließlich der Segmentierung, verantwortlich sind (Sander & Lehmann, 1988; St Johnston & Nüsslein-Volhard, 1992; Forrest & Gavis, 2003; Becalska & Gavis, 2009). Früh in der Oogenese wandert der Kern der Oozyte nach anterior/dorsal und definiert damit durch lokalisierte Translation von gurken die dorso-ventrale Achse des Embryos (van Eeden & St Johnston, 1999). Die Musterbildung im Blastoderm entlang der anterior-posterioren Achse erfolgt über eine Segmentierungskaskade, d.h. eine Gruppe von interagierenden Genen, die als regulatives Netzwerk die Entwicklung des Embryos von der groben Polarisation der Oozyte bis zur Identität der einzelnen Segmente steuern (Nüsslein-Volhard & Wieschaus, 1980; Peel et al., 2005; Abbildung 1). Essentiell für diese Kaskade ist die Lokalisation maternaler mRNAs, deren Proteinprodukte als Morphogene die Blastoderm-Musterbildung regulieren. Der Transport der mRNAs erfolgt entlang des polarisierten Mikrotubuliskeletts der Oozyte, wobei die mRNAs durch Transportkomplexe an Motorproteine wie DYNEIN oder KINESIN binden. An den jeweiligen Polen werden die mRNAs verankert und nach Aktivierung des Eis lokal exprimiert (Pokrywka & Stephenson, 1991; Glotzer et al., 1997; Zimyanin et al., 2008; Macdonald, 2011). 16 1. Einleitung Während die mRNA von bicoid anterior fixiert wird, transportiert KINESIN die oskar-mRNA nach posterior, wo es sofort translatiert wird und die Akkumulation des Polplasmas steuert, welches u. a. nanos-mRNA nach posterior rekrutiert (Driever & Nüsslein-Volhard, 1988a; Ephrussi et al., 1991; Lasko, 2011). Mit Aktivierung des Eis nach der Befruchtung entstehen, ausgehend von den beiden Polen, durch Translation der bicoid- und nanos-mRNAs Proteingradienten. Diese entgegen gerichteten Gradienten gestalten den Embryo entlang der anterio-posterioren Achse, indem sie andere RNAs lokal inhibieren oder nachgeschaltete Gene auf Promotorebene regulieren. In bicoid-Mutanten fehlen zum Beispiel Kopf und Thorax, an deren statt eine Duplikation posteriorer Telson-Strukturen zu beobachten ist. Vice versa führt der Ausfall von nanos zum Verlust des Abdomens. Auch das Entfernen oder die Transplantation von polarem Plasma resultiert in einer Reihe von Phänotypen, die den jeweiligen Mutanten entsprechen und bestätigt somit die Rolle von bicoid und nanos als globale Mustergeber (Nüsslein-Volhard & Wieschaus, 1980; Nüsslein-Volhard et al., 1987, Driever & Nüsslein-Volhard, 1988a, b; Lehmann und Nüsslein-Volhard, 1991). Neben den beiden maternalen Proteingradienten aus BICOID und NANOS, deren Diffusion im syncytialen Blastoderm durch das Fehlen der Zellmembranen ermöglicht wird, benötigt die embryonale Musterbildung zur Komplettierung noch das Terminale System, das für die Musterbildung im Bereich der beiden Pole des Blastodermembryos verantwortlich ist. Hierbei wird der Membranrezeptor TORSO, der über den gesamten Embryo verteilt ist, lokal von TORSO-LIKE aktiviert. Der somit eingeleitete Signalweg reguliert, zusammen mit dem anterioren und dem posterioren System, ein spezifisches Set zygotischer Gene, die die Ausbildung der terminalen Strukturen steuern (Nüsslein-Volhard et al., 1987; Klingler et al., 1988; SavantBhonsale & Montell, 1993; Jiménez et al., 2000). Die Interaktion der maternalen Systeme sowie die Konzentrationen der einzelnen Proteine vermitteln den Zellen räumliche Informationen und induzieren somit je nach Position die Expression unterschiedlicher zygotischer Gapgene. Diese Lückengene interagieren untereinander und unterteilen den Embryo entlang der a-p-Achse in kleinere Abschnitte. Mutationen eines solchen Lückengens resultieren daher im Verlust mehrerer benachbarter Segmente, zum Beispiel vom ersten thorakalen Segment 17 1. Einleitung (T1) bis zum fünften abdominalen (A5) in Mutanten des Gens Krüppel (NüssleinVolhard & Wieschaus, 1980; Wieschaus et al., 1984; Porcher & Dostatni, 2010). Ausgehend vom überlappenden aperiodischen Expressionsmuster der Lückengene und den Informationen der maternalen Faktoren, werden über spezifische EnhancerElemente die Paarregelgene aktiviert. Deren regelmäßiges Streifenmuster kodiert die Entstehung geradzahliger bzw. ungeradzahligerer Segmente indem es die Segmentpolaritätsgene reguliert, welche die Grenzen der Segmente definieren. Hauptverantwortlich dafür sind die Gene wingless und engrailed, die abwechselnd in benachbarten Zellreihen exprimiert sind und sich gegenseitig regulieren (Nüsslein-Volhard & Wieschaus, 1980; Howard & Ingham, 1986; Ingham et al., 1986; DiNardo und O'Farrell, 1987; Martinez-Arias et al., 1988; Dassow et al., 2000; Peel et al., 2005). Die abschließende Identität eines Segments wird über die Aktivität der Hox-Gene vermittelt, einer Gruppe hochkonservierter Transkriptionsfaktoren, die innerhalb der Metazoa konserviert ist und die Individualentwicklung der Segmente entlang der anterio-posterioren Achse steuert. Charakteristisch für Hox-Gene ist die HomöoBox, die eine DNA-Bindedomäne kodiert. Diese Hox-Gene sind bei Bilateria in einem Cluster organisert (der bei Drosophila allerdings in zwei Sub-Cluster zerfallen ist), wobei die Abfolge der genomischen Sequenz den Expressionsdomänen entspricht. Bei Drosophila spezifizieren die Gene des Antennapedia-Komplexes die Strukturen des Kopfes und der ersten beiden thorakalen Segmente, während der Bithorax-Komplex die posterioren Segmente des Körpers, Metathorax bis Abdomen gestaltet. Die Expressionsmuster der Hox-Gene werden dabei sowohl durch die Lückengene als auch Paarregelgene bestimmt (Lewis, 1978; Ingham et al., 1986; Akam, 1989; McGinnis & Krumlauf, 1992; Kenyon, 1994; Krumlauf, 1994; Caroll, 1995; Peel et al., 2005). 18 1. Einleitung Abbildung 1: Die Segmentierungskaskade von Drosophila melanogaster Während der Oogenese werden diverse maternale mRNAs von den Nährzellen in das Ei transportiert, wo sie später die Segmentierung des Embryos steuern. bicoid ist anterior lokalisiert während das Polplasma, eine Mischung aus Proteinen und RNAs, darunter nanos, posterior lokalisiert wird. Die mRNAs von hunchback und caudal sind hingegen ubiquitär verteilt. Mit der Befruchtung des Eis werden die RNAs translatiert. Step 1 zeigt die Expressionsdomänen der maternalen Proteingradienten. Von den Polen aus diffundieren BICOID und NANOS ins Blastoderm, wobei BICOID die Translation von hunchback aktiviert und caudal inhibiert. NANOS hingegen inhibiert hunchback . Step 2 Ausgehend von den Positionsinformationen, die die maternalen Faktoren vermitteln, werden die zygotischen Lückengene aktiviert, deren Expressionsdomänen dann durch gegenseitiger Regulat ion und auch Diffusion präzisiert werden. Step 3 Die teilweise überlappenden Domänen der Lückengene aktivieren, zusammen mit den maternalen Gradienten, die Paarregelgene in einem periodischen Streifenmuster. Wie in den Lückengenen kommt es auch hier zu einer gegenseitigen transkriptionellen Kontrolle, die u.a. die scharfen anterioren Grenzen der Streifen etabliert. Step 4 Wie fast alle Gene der Segmentierungskaskade kodieren auch die Paarregelgene Transkript ionsfaktoren. Diese regulieren die segmentale Expression der Segmentpolaritätsgene und somit die Ausbildung der Segmentgrenzen; die sezernierten Morphogene Hedgehog und Wingless bestimmen dann die Differenzierung segmentaler Organe, z.B. die Position von Dentikeln, Muskelansatzstellen und Sinnesorganen. (Abbildung aus Peel et al., 2005, leicht verändert). 19 1. Einleitung 1.1.2 Tribolium castaneum als Modellsystem Der rotbraune Reismehlkäfer Tribolium castaneum zählt wie Drosophila zu den holometabolen Insekten, vollzieht also eine vollständige Metamorphose von der Larve über die Puppe zum ausgewachsenen Insekt. Er gehört zur Familie der Tenebrionidae, mit geschätzten 15.000 Arten eine der reichsten Familien der Coleoptera. Ihren Ursprung hat die Art vermutlich im Indo-australischen Raum. Internationaler Handel, Vorratshaltung sowie die Erfindung der Zentralheizung begünstigen jedoch seine Verbreitung, so dass Tribolium mittlerweile auch in den gemäßigten Breiten zu den gravierendsten Schädlingen von Getreide und dessen Erzeugnissen gehört (Sokoloff, 1974; Bonneton, 2008). Der anspruchslose Kosmopolit rückte bereits Anfang des 20. Jahrhunderts in den Fokus von Populationsgenetikern, Ökologen und Entomologen (Sokoloff, 1974). Die Verfügbarkeit moderner genetischer und molekularbiologischer Methoden, sowie die generellen Eigenschaften der Art - wie relativ kurze Generationszeiten und geringer Haltungsaufwand in Kombination mit einer hohen Reproduktionsrate - führten dazu, dass auch Entwicklungsbiologen Tribolium als Modell entdeckten - insbesondere, da der Typus der Embryonalentwicklung des Käfers im Vergleich zu Drosophila sowohl evolutionär älter als auch unter den Insekten weiter verbreitetet ist (Tautz et al., 1994; Davis & Patel, 2002; Klingler, 2004; Schröder et al., 2008). Wie fast alle Insekten beginnt auch Tribolium seine Entwicklung in einem Syncytium, d.h. die Nuclei des Embryos liegen ohne Zellmembranen in der Eizelle vor, was die barrierefreie Diffusion von Transkriptionsfaktoren ermöglicht. Während in Drosophila jedoch alle Segmente im Blastodermstadium definiert werden, werden in Tribolium nur die extraembryonalen Membranen (Serosa und Amnion), der Kopf sowie Pro- und Mesothorax in dieser synzytialen Umgebung spezifiziert. Die restlichen Segmente entstehen erst später, nach Bildung des Keimrudiments, wenn sukzessive die posterioren Segmente aus einer „segment addition zone“ auswachsen (SAZ, früher Wachstumszone), welche am posterioren Pol des Embryos entsteht (Seidel et al., 1940; Lohs-Schardin et al., 1979; Nüsslein-Volhard & Wieschaus, 1980; Schoppmeier & Schröder, 2005; Abbildung 2). Die sogenannte Langkeim-Entwicklung von Drosophila findet sich lediglich in höheren Holometabola, wie den Diptera oder Hymenoptera. Kurzkeim-Embryonen oder Embryonen, die einen intermediären Entwicklungstypen aufweisen, kommen 20 1. Einleitung hingegen sowohl in höheren und niederen Insekten, als auch in anderen Klassen der Arthropoda vor (Krause, 1939; Davis & Patel, 2002; Damen, 2007). Die Reduktion der extraembryonalen Membranen Serosa und Amnion zu einer Amnioserosa, sowie die Umformung des larvalen Kopfes (größtenteils eingestülpt) und der larvalen Beine (embryonal als Imaginalscheiben angelegt) waren zusätzliche Gründe, ein basaleres Insekt wie den Käfer Tribolium als Modellorganismus zu etablieren (Schmidt-Ott, 2000; Klingler, 2004). 2004 zeigten Copf et al. die Rolle des maternalen Faktors caudal in der posterioren Achsen-Elongation und Segmentierung in Tribolium (und Artemia franciscana) und lieferten so konkrete Hinweise auf ein konserviertes posteriores Musterbildungssystem. Auch einige andere Studien gaben Hinweise darauf, dass die gesamte Segmentierungskaskade auch in der abdominalen Musterbildung im Wesentlichen konserviert ist, und die unterschiedlichen Entwicklungsmodi von Langkeim- und Kurzkeim-Embryo sich "nur" durch eine andere zeitliche Dynamik der Genexpression in der „segment addition zone“ ergeben (Patel et al., 1994; Liu & Kaufman, 2005; Damen, 2007; McGregor et al., 2009). Jüngere Arbeiten zeigten aber, dass die sequenzielle Generierung der Segmente in der SAZ von Tribolium auf oszillierender Genexpression beruht, also durch Mechanismus welcher Embryo der SoT he Dynamic Nuclear Redistribution of aneinen hnRNP K -homologouszustandekommt, Protein during D rosophila Development and H eat Shock. Flexibility of Tr anscription Sites I n Vivo mitogenese der Vertebraten ähnelt (Sarrazin et al., 2012; El-Sherif et al., 2012). A Amnioserosa Akron Telson Kopf Thorax Abdomen B dorsales Amnion Serosa WZ Thorax Amnion Kopf Abbildung 2: Anlagenpläne und Kurzkeim-Embryo von Lang- In Langkeim-Embryonen (A; Drosophila) werden die Anlangen aller Segmente im Blastoderm festgelegt, in Kurzkeim-Embryonen (B; Tribolium) gilt dies nur für die anterioren Segmente. Weitere Unterschiede werden in Bezug auf die Anlagen der extraembryonalen Membranen deutlich - Amnioserosa in Drosophila (A) und Serosa bzw. Amnion in Tribolium (B) – sowie der terminalen segment addition zone (SAZ), aus der in zellularisierter Umgebung die restlichen Segmente entspringen. (A) Quelle: Buchenau et al., 1997, bearbeitet nach St. Johnston & Nüsslein Volhard; 1992; (B) nach Kotkamp et al., 2010). Buchenau P et al. J Cell Biol 1997;137:291-303 © 1997 Rockefeller University Press 21 1. Einleitung Neben CAUDAL, einem auch bei Vertebraten für die posteriore Entwicklung relevanten Faktor, ist auch NANOS an der embryonalen Musterbildung Triboliums beteiligt, wo es unter anderem (zusammen mit PUMILIO) über die Repression von maternalem hunchback posterior die Expression diverser Lückengene reguliert. Im Vergleich zu Drosophila, wo der Ausfall von nanos zu einem Komplettverlust des Abdomens führt, sind die Knockdown-Phänotypen von nanos/pumilio weniger ausgeprägt - vor allem konnte aber keine Lokalisierung dieser mRNA nachgewiesen werden (Schmitt-Engel et al., 2012). In Drosophila liegt bicoid, das anteriore Morphogen, im Hox-Cluster vor dem Gen zerknüllt (zen), das zygotisch exprimiert ist und für die Bildung der Amnioserosa benötigt wird (Rushlow & Levine; 1990). Obwohl Tribolium wie andere Arthropoden Orthologe der acht homeotischen Gene besitzt, die auch in der gleichen Reihenfolge arrangiert sind wie in der Fliege, findet sich an der Stelle von Hox3 im Komplex kein bicoid sondern zwei zen-Gene (Brown et al., 2001). Wie in Drosophila ist eines dieser Gene (zen-1) für die Ausbildung der Serosa verantwortlich. ZEN-2 hingegen wird zu einem späteren Zeitpunkt aktiv, um die Fusion von Amnion und Serosa bzw. deren Aufreißen zu initiieren, was für die morphogenetischen Bewegungen und den finalen dorsalen Rückenschluss des Embryos notwendig ist (Scuderi & Letsou, 2005; van der Zee et al., 2005). Aber ZEN-2 erfüllt eine weitere Funktion, wie Schoppmeier et al. 2009 aufzeigten. Es übernimmt zusammen mit dem zygotischen Faktor MEX-3 die Funktion von BICOID als anteriorer Repressor von caudal und ist damit maßgeblich an der Musterbildung des Tribolium-Embryos beteiligt. Interessanterweise unterliegt die anteriore Musterbildung in Tribolium dem kanonischen Wnt-Signalweg, der in Vertebraten, aber nicht in Drosophila, an der Achsendetermination beteiligt ist. Die mRNA von axin, einem negativen Regulator dieses Signalwegs, ist maternal am anterioren Pol exprimiert. Der Verlust von axin führt zu einem Verlust anteriorer Strukturen, und folglich zu einer Expansion des Anlagenplans nach anterior (Fu et al., 2012). Es scheint daher so zu sein, dass die räumlich lokalisierten Funktionen von Bicoid und Nanos in Tribolium weitgehend von einem ß-Catenin-Gradienten ausgeübt werden, der von AXIN reguliert wird. Von den drei aus Drosophila bekannten maternalen Gradientensystemen ist in Tribolium das terminale System am besten konserviert (Schoppmeier und Schröder, 2005). Allerdings ergeben sich auch für das terminale System um den TORSO- 22 1. Einleitung Signalweg neue Funktionen. So aktiviert das terminale System in Tribolium zusammen mit ORTHODENTICLE (OTD) anterior die Expression von ZEN-1 und induziert dadurch die Bildung der Serosa, posterior hingegen die Etablierung der „segment addition zone“, aus der das dritte thorakale Segment und das gesamte Abdomen hervorgehen. Das terminale System um TORSO spielt also im Kurzkeimembryo von Tribolium eine zentrale Funktion für die Entwicklung des ganzen Abdomens, während es in der phylogenetisch jüngeren Fliege nur noch für begrenzte Bereiche um die unsegmentierten terminalen Strukturen von Akron und Telson benötigt wird (Schoppmeier und Schröder, 2005; Kotkamp et al., 2010). Um die Kurzkeim-Entwicklung von Tribolium zu verstehen, wurden in den letzten Jahren vorwiegend zwei funktionelle Ansätze benutzt: zum einen kleinere Mutagenese-Screens, die relevante Gene unabhängig von vorgefassten Annahmen identifizieren können, aber den Nachteil haben, dass die Haltung der generierten Linien sehr aufwendig ist. Zum anderen baute man auf den Kandidaten-Gen-Ansatz, d.h. es wurde gezielt nach Homologen von mustergebenden Genen in Drosophila gesucht, und deren Funktion dann in Tribolium mittels RNAi analysiert. Diese Vorgehensweise stößt aber dann an Grenzen, wenn bestimmte Funktion von anderen, nichthomologen Genen übernommen wird (z.B. bicoid/mex-3), oder wenn Gene an einem Prozess beteiligt sind, deren Homologe in Drosophila keine solche Funktion haben (z.B. mille-pattes). Zudem können mit diesem Ansatz nur bereits bekannte Prozesse untersucht werden. Gene, die in Tribolium beispielsweise an der Ausbildung des larvalen Kopfes oder der larvalen Beinmuskulatur beteiligt sind, also Prozessen, die in Drosophila nicht gut verstanden sind, stoßen im Kandidaten-Gen-Ansatz an ihre Grenzen. Mit dem seit 2008 verfügbaren Genom, sowie modernen, preisgünstigen molekulargenetischen Techniken wie der RNA-Interferenz (RNAi) eröffnen sich hier jedoch neue Möglichkeiten (Roth & Hartenstein, 2008; Tribolium Genome Sequencing Consortium, 2008; Schröder et al., 2008; Trauner, 2009). 23 1. Einleitung 1.2 Ein genomweiter Kandidaten-Gen-Ansatz RNAi-Screen als Alternative zum RNA-Interferenz (RNAi) hat die biologische und medizinische Forschung revolutioniert. Sie findet Anwendung bei der Untersuchung von Genfunktionen in Pflanzen, Tieren und menschlichen Zellen, bei der Analyse von Krankheiten und der Entwicklung neuartiger Medikamente (Brown et al., 1999; Lavery & King, 2003; Vaishnaw et al., 2010; Watanabe, Y., 2011). Entdeckt wurde RNAi gewissermaßen im Jahr 1990, als Napoli et al. künstlich generierte Chalconsynthase in Petunien einbrachten, um deren Blütenfarbe zu verändern. Statt der erwarteten violetten Blüten resultierte die Überexpression aber in weißen Blüten, und weitere Untersuchungen ergaben, dass die mRNA des Gens im Vergleich zu wildtypischen Blüten um ein fünfzigfaches reduziert war. Die damals postulierte Co-Suppression weckte reges Interesse, doch verstanden wurde sie erst Jahre später in Caenorhabditis elegans. War man zuvor davon ausgegangen, einzelsträngige (antisense) RNA sei erforderlich für diese Interferenz, so demonstrierten Fire und Mello 1998 eine dramatische Verstärkung des Effekts nach Einbringen von doppelsträngiger RNA – eine Publikation, die ihnen 2004 den Nobelpreis für Physiologie bzw. Medizin einbrachte (Fire et al., 1998; Sen & Blau, 2006). Das Prinzip der RNA-Interferenz macht sich zelleigene Mechanismen zu Nutze. Dabei induziert die eingebrachte doppelsträngige Fremd-RNA den Abbau der komplementären mRNA und führt so zu der posttranskriptionellen Reprimierung des Gens. In vivo schützt sich die Zelle auf diese Weise vor parasitärer RNA, zum Beispiel von dsRNA-Viren. Die eingebrachte dsRNA wird von der Endoribonuklease DICER in Stücke von 21-28 Basenpaaren geschnitten; die einzelnen Fragmente bilden mit anderen Proteinen den RISC-Komplex (RNA-induced silencing complex), der die vorhandenen mRNAs der Zelle nach komplementären Sequenzen absucht und deren Abbau einleitet (Tijsterman et al., 2002; Meister & Tuschl, 2004). Im Gegensatz zu Drosophila konnte für Tribolium ein robuster, systemischer Effekt der RNA-Interferenz nachgewiesen werden. Das zur dsRNA korrespondierende Protein ist also nicht nur in injizierten Zellen oder Syncytien reduziert, sondern nach Injektion in die Hämolymphe - in allen Zellen des Körpers. Im Falle einer Injektion von weiblichen Käfern kann die RNAi daher auch in der F1-Generation beo- 24 1. Einleitung bachtet werden (Brown et al.,1999; Bucher et al., 2002; Tomoyasu, 2008). Dieser Effekt ist so Zielsequenz-spezifisch, dass er bei Tribolium auch zwischen einzelnen Spleißvarianten einer Transkriptionseinheit unterscheiden kann (Arakane et al., 2005). RNAi hat große Vorteile in Bezug auf Effizienz und Logistik für Genanalysen v.a. in Nicht-Modellorganismen: 1. Mutationen mit interessanten Phänotypen müssen nicht aufwendig kartiert bzw. das betroffene Gen molekular identifiziert werden. Genomische Datenbanken sowie Transcriptomics-Analysen ermöglichen die Identifikation der meisten Gene, gegen die dann spezifische dsRNAs generiert werden können (Arziman et al., 2005) 2. Variation des Injektionszeitpunkts ermöglicht es, frühe und späte Funktionen eines Gens zu unterscheiden, bzw. Effekte eines Gens in verschiedenen Entwicklungsstadien bzw. Strukturen getrennt zu untersuchen (Beermann et al., 2004; Tomoyasu & Denell, 2004; Trauner & Büning, 2007; ClarkHachtel et al., 2013). 3. Parentale RNAi bietet die Möglichkeit, durch Injektion weniger Tiere eine Vielzahl von Nachkommen/Embryonen mit induziertem Phänotyp zu generieren, wobei leicht eine phänotypische Serie erzeugt werden kann, die oftmals auch den genetischen Null-Phänotyp einschließt (Brown et al., 1999; Bucher et al., 2002; Cerny et al., 2005). 4. Der Funktionsverlust eines Gens ist, anders als in Drosophila, wo Screens meist entweder zygotische oder maternale Genfunktionen untersuchen, vollständig, d.h. beide Komponenten werden ausgeschaltet (Nüsslein-Volhard & Wieschaus, 1980; Nüsslein-Volhard et al., 1984; Schröder, 2003; Fu et al., 2012). 5. Ein letzter, nicht zu vernachlässigender Aspekt ist die Vereinfachung der Stammhaltung. Statt zahlloser Mutanten muss nur noch der Wildtyp-Stamm gehalten werden, sowie die dsRNAs, die aber mit Hilfe von genspezifischen Primern jederzeit wieder aus cDNA gewonnen werden können. Im Zuge seiner Doktorarbeit untersuchte Christian Schmitt-Engel die Durchführbarkeit eines genomweiten RNAi-Screens im roten Reismehlkäfer Tribolium castaneum und etablierte eine erste Version der Screen-Prozedur (Schmitt-Engel, 2010). Aber 25 1. Einleitung ein genomweiter Screen - das bedeutet die Analyse von schätzungsweise 12.000 Genen (Tribolium Genome Sequencing Consortium, 2008; Zahlen überarbeitet nach M. Stanke, Bioinformatik, Greifswald, AUGUSTUS prediction 3). Um dieses ehrgeizige Vorhaben in die Tat umzusetzen, schlossen sich die Tribolium-Forschergruppen der Georg-August-Universität zu Göttingen (Gregor Bucher und Ernst Wimmer), der Friedrich-Alexander-Universität (Martin Klingler und Michael Schoppmeier) sowie der Universitäten zu Köln (Siegfried Roth) und Rostock (Reinhard Schröder) zu dem Projekt „iBeetle“ zusammen. Jedes Labor bringt dabei nicht nur eigene wissenschaftliche Fragestellungen ein, sondern liefert auch Expertise in den unterschiedlichsten Bereichen. So bestimmten die Schwerpunkte der Forschergruppen und deren Partner den Aufbau des Screens und die die darin berücksichtigten Phänotypklassen. Die Einzelprojekte umfassen sowohl embryonale als auch postembryonale Prozesse, weshalb jede dsRNA in weibliche Larven (larvaler Screen) und Puppen (pupaler Screen) injiziert wurde, um die Funktion jedes Gens in verschiedenen Entwicklungsprozessen untersuchen zu können (Schmitt-Engel et al., in Vorbereitung). Als Mitarbeiter des pupalen Screens möchte ich nachfolgend den Ablauf dieses Screen-Teils darstellen und die einzelnen Analysen kurz erläutern. Der pupale Screen beinhaltet vier Analyseschritte (Abbildung 3), deren Ergebnisse in einer eigens dafür entwickelten Datenbank (Dönitz et al., in Vorbereitung), wenn möglich mit Bildern, dokumentiert wurden. 1.2.1 Ziele des Screens und der einzelnen Analyseschritte Drei Tage nach Injektion der Puppen wurden die Weibchen hinsichtlich möglicher Metamorphose-Defekte untersucht. Hierbei handelte es sich vor allem um die Fälle, wo die Häutung zum Adult-Stadium nicht vollzogen wurde, bzw. aufgrund morphologischer Defekte nicht vollendet werden konnte. An Tag 9 und 11 nach Injektion wurde jeweils eine Eiablage für Kutikula- bzw. Muskelpräparate genommen, wobei gleichzeitig die Menge der gelegten Eier mit dem Wildtyp verglichen wurde, da dies auf eventuelle Defekte in der Oogenese hindeuten könnte. Das telotroph-meroistische Ovar Triboliums, seine Entwicklung und Funktion, sind ein Schwerpunkt von Michael Schoppmeier (Erlangen). Insbesondere die somatischen Stammzellen und ihre Differenzierung stehen dabei im Vordergrund 26 1. Einleitung – Prozesse, die man in Drosophila aufgrund fehlender zeitlicher Trennung schwer untersuchen kann (Trauner und Büning, 2007). Am Tag 13 wurden die Ovare von Weibchen mit reduzierter Einmenge präpariert, und außerdem Kutikula-Präparate aus den an Tag 9 gesammelten Eiern hergestellt, welche später hinsichtlich verschiedenster Defekte ausgewertet wurden. Sowohl Gregor Bucher als auch Ernst Wimmer (beide Göttingen) interessieren sich dabei für Prozesse der embryonalen Kopfentwicklung, die in Drosophila wegen des internalisierten Kopfes nur ungenügend erforscht worden ist (Jürgens et al., 1986). Für diverse Gene, die an der Entwicklung der Neuralplatte von Vertebraten beteiligt sind, konnte in Tribolium bereits eine Rolle in der Musterbildung des Kopfes nachgewiesen werden (Posnien et al., 2011). Klar ausgeprägte Strukturen wie Labrum, Antenne, Mandibel, Maxilla und Labium ermöglichen eine einfache Analyse. Aber der Kopf der L1-Larven weist zudem noch ein charakteristisches Muster von Borsten auf, dessen Veränderung auf subtilere Defekte der Kopfentwicklung schließen lässt (Schinko et al., 2007; Kittelman et al., 2013). Für Gene, die an der Etablierung der dorso-ventralen Achse des Embryos beteiligt sind, interessiert sich die Arbeitsgruppe von Siegfried Roth (Köln), was auch die Entwicklung und Funktion der extraembryonalen Membranen Serosa und Amnion einschließt (van der Zee et al., 2005; van der Zee et al., 2006). Martin Klingler (Erlangen) untersucht die Musterbildung entlang der anterio-posterioren Achse, von der frühen Determination im Embryo bis zu Segmentierung und Musterbildung im wachsenden Keimstreifen (Wolff et al., 1998; Cerny et al., 2008). In vielen Fällen kann bereits vom Charakter eines Kutikulaphänotypen auf den betroffenen Prozess geschlossen werden. Die Inaktivierung früh aktiver Faktoren kann aber auch zum völligen Fehlen einer Kutikula führen, so dass diese so genannten „empty egg“-Phänotypen später noch einmal genauer untersucht werden müssen. Manfred Frasch (Erlangen) beteiligt sich mit einem Projekt zur Analyse der larvalen Muskulatur. Er sucht konservierte Faktoren in der Muskelentwicklung Drosophilas, seien es frühe, an der Fusion der Vorläuferzellen beteiligte, oder solche, die Orientierung und Verankerung der Muskelfasern beeinflussen oder später deren Funktionalität aufrecht erhalten (Schaub & Frasch, 2013). Die Analyse der embryonalen Muskulatur von Tribolium fand am Tag 14 jeder iBeetle Injektions-Runde statt. Hierzu wurde speziell im pupalen Screen der Käferstamm pBA19 verwendet, 27 1. Einleitung der als Enhancertrap-Insertion im Muskelprotein AKTIN somatische Muskeln mit grün fluoreszierendem GFP markiert (Shimomura et al., 1962), sodass Muskelphänotypen ohne histologische Färbung erkannt werden konnten. Neben diesen embryonalen Fragestellungen wurden - vor allem im larvalen Screen, den ich hier nicht näher behandeln möchte - auch nach Phänotypen in Puppen und Adulten gesucht, zum Beispiel in der Morphologie der Beine oder in den Stinkdrüsen, und auch andere Auffälligkeiten, etwa im Verhalten der Tiere, festgehalten (vgl. 1.2.3). Abbildung 3: iBeetle-Schema für pupale Screener Dargestellt ist der schematische Ablauf der pupalen Injektion im Rahmen von iBeetle (blaue Kästen) sowie die and den einzelnen Tagen (- d x -) detektierbaren Phänotypen (grüne Kästen). Metamorphose-Phänotypen beziehen sich meist auf Störungen des Häutungsverhaltens. Bei reduzierter Legeleistung wurden die Ovare von vier Weibchen hinsichtlich Ovarmorphologie und Oogenesedefekten unter dem Mikroskop untersucht. Defekte der Muskulatur wurden mit Hilfe der Muskel-Enhancer-Traps des pBA19-Stammes unter dem Fluoreszenzmikroskop analysiert, embryonale Phänotypen hinsichtlich verschiedener Klassen wie Segmentierung, Kopf- und Beinentwicklung anhand von KutikulaPräparaten nach Tag 13 klassifiziert. Alle Teilanalysen und weitere erdenkliche Defekte / Auffälligkeiten wurden in der iBeetle-Datenbank, wenn möglich mit Fotos, festgehalten. 1.2.2 Etablierung und Aufrechterhaltung der Qualität Ein Screen, so breit angelegt wie iBeetle, bei dem zudem Screener mit diversen wissenschaftlichen Hintergründen zusammenkommen, erfordert nicht nur eine gut abgestimmte Logistik, sondern auch Kontrollen für alle unterschiedlichen Arbeitsschritte, damit eine Vergleichbarkeit der Resultate von Screener zu Screener gegeben ist. Dies wurde im iBeetle-Screen über folgende Maßnahmen erreicht (siehe auch SchmittEngel et al., in Vorbereitung). 28 1. Einleitung Die Screener nahmen zu Beginn des Screens an einem gemeinsamen einwöchigen Symposium teil, in dem die Projektleiter die Hintergründe für die einzelnen Projekte vorstellten, und der Ablauf des Screens sowie die einzelnen Arbeitsschritte besprochen und geübt werden konnten. Zur Etablierung einer einheitlichen Dokumentation injizierten alle Screener dieselben ersten hundert Gene einer Testplatte. Diese Ergebnisse wurden im Anschluss von einem Postdoc ausgewertet und notwendige Anpassungen wurden mit jedem Screener besprochen. Regelmäßige Konferenzschaltungen zwischen den Screening-Zentren Erlangen und Göttingen ermöglichten das Besprechen von Problemen, schwierigen Phänotyp-Annotationen, und hielten zudem das soziale Netzwerk (bzw. die Motivation der Screener) aufrecht. In einem Set von 24 Injektionen (= 1 Repetition) waren immer mindestens eine Pufferkontrolle und eine Positivkontrolle integriert, ersteres zum Erkennen technischer Probleme und als Kontrolle für spontan auftretende Hintergrund-Phänotypen, während letztere die Aufmerksamkeit der Screener prüfen sollte. Für die Positivkontrollen wurde zu Beginn des Screens ein Set von Genen zusammengestellt, deren RNAi-Phänotypen bereits bekannt waren. Dabei war natürlich die Identität des Gens und der zugehörige Phänotyp nur den Betreuern, nicht aber den Screenern bekannt. Innerhalb eines Bundles (fünf Sets von je 24 Injektionen) musste jeweils eine Repetition mit einem fachspezifischen Betreuer vor Ort besprochen werden. In Erlangen betraf dies M. Schoppmeier für Ovarphänotypen, M. Frasch für Muskelpräparate und M. Klingler für Kutikula-Präparate. Um Auskunft über die Reproduzierbarkeit der Ergebnisse zu erhalten wurden am Ende des Screens von jedem Teilnehmer ≥ 10 dsRNAs mit hoch-penetrantem Phänotyp ausgewählt und a) die gleiche dsRNA in zwei unterschiedlichen Konzentrationen in den gleichen Stamm, b) in einen anderen Stamm (San Bernardino) und c) eine nicht überlappende dsRNA des gleichen Gens injiziert. Die Gene, deren Identität auf diese Weise bestätigt werden konnte, wurden für weitere funktionelle Analysen in Erwägung gezogen, darunter die in dieser Arbeit untersuchten Gene germ cell-less und homeobrain. 29 1. Einleitung 1.3 germ cell-less 1.3.1 germ cell-less in Drosophila Eine der ersten Zelltyp-spezifischen Spezialisierungen in Drosophila ist die Etablierung der Keimzellen, die im Verlauf der Zellularisierung aus dem posterioren Polplasma hervorgehen (Illmensee et al., 1976). Neben dem Keimplasma enthält das Polplasma mehrere Determinanten für abdominales Schicksal, weshalb es in Mutanten verschiedener maternaler Effekt-Gene (z.B.: staufen, cappucino) zu Störungen beider Prozesse kommt. Mit Hilfe eines Fertilitätsscreens gelang es 1992 eine Mutante zu generieren, deren Nachwuchs, obwohl morphologisch normal und lebensfähig, sich als steril herausstellte. Weitere Analysen offenbarten einerseits die posteriore Lokalisation der zugehörigen mRNA im Wildtyp, sowie andererseits das Fehlen jeglicher Keimzellen in den Mutanten, weshalb das betroffene Gen germ cell-less (gcl) benannt wurde (Jongens et al., 1992). gcl-mRNA wird von den Nährzellen der Mutter produziert, zusammen mit anderen Determinanten über Ringkanäle in die Oozyte verbracht (vgl. 1.1.1) und akkumuliert im Zuge der Oogenese am posterioren Pol, im Polplasma. GCL Protein hingegen ist früh an der inneren Membran der Nährzellen und des Oozytenkerns lokalisiert. Im Verlauf der Oogenese ist das Protein auch am posterioren Pol der Oozyte in der Kernmembran der Nuclei zu finden, aus denen später die Polzellen (Vorläufer der Keimzellen) hervorgehen (Jongens et al., 1992, 1994). Dies geschieht über transkriptionelles "Silencing", d.h. die Transkription in den für die Polzellen vorgesehenen Nuclei ist für einen gewissen Zeitraum reprimiert (Leatherman et al., 2002) – ein Phänomen, das nicht nur in Drosophila, sondern auch in C. elegans beobachtet wurde (Seydoux et al., 1996). Ektopische Lokalisierung von GCL am anterioren Pol, durch eine Fusion des gcl Transkripts an die 3’ UTR von bicoid, bewirkt die reduzierte Transkription von anterioren somatischen Genen, was auf eine Determinierung in Richtung von Polzellen-Schicksal hindeutet. Mit einsetzender Wanderung der Polzellen in Richtung der embryonalen Gonaden schwindet die GCL-Expression (Jongens et al., 1992, 1994; Leatherman et al., 2002). Eine vertiefende Untersuchung von Robertson et al. (1999) belegt drei distinkte Funktionen von GCL: die Initiierung der Polknospen (spezifische Zellausstülpungen, in welche die Nuclei der Polzellen 30 1. Einleitung einwandern), die Bildung der Polzellen, sowie deren Überleben während sie in die Gonaden einwandern (Robertson et al., 1999) Obwohl gcl-mRNA in geringem Maße in somatischen Zellen der Oozyte zu finden ist, ist GCL nur in den Nuclei der Polzellen detektierbar. Dies beruht auf einer translationellen Kontrolle der mRNA durch das RNA-bindende Protein BRUNO, welches gcl am 3’ UTR über ein spezifisches RNA-Erkennungsmotiv bindet (Moore et al., 2009). 1.3.2 germ cell-less in Mus musculus Leatherman et al. gelang es 2000, gcl in der Maus Mus musculus zu identifizieren. Trotz einer Ähnlichkeit von nur 49% ist Mm-gcl in der Lage, den gcl-NullPhänotypen von Drosophila zu retten. Allerdings ist Mm-gcl nicht nur in den Vorläufer-Keimzellen exprimiert, sondern (wenn auch schwach) in den meisten embryonalen und adulten Geweben. Auffällig hoch ist gcl in männlichen Hoden exprimiert, spezifisch im Pachytän und Diplotän primärer Spermatozyten (Kimura et al., 1999; Leatherman et al., 2000). Mäuse, denen gcl fehlt, zeigen Deformationen der Zellkerne von Leber, Pankreas und Hoden. Neben fehlgebildeten Spermienköpfen sowie ektopischen Flagellen wiesen die Spermien auf molekularer Ebene auch Schädigungen des Chromatins und der Kernstruktur auf. Diese Anomalien fallen zeitlich eng mit dem Auftreten des GCL-Proteins im Pachytän primärer Spermatozyten zusammen und haben eine reduzierte Fertilität der Männchen zur Folge (Kimura et al., 2003; Maekawa et al., 2004). Wie in Drosophila ist Mm-GCL in der Kernmembran lokalisiert, welche die Chromosomen vom Zytoplasma trennt und unter anderem über Kernporen für einen kontrollierten Austausch zwischen den Kompartimenten sorgt, den Zellkern stabilisiert und über assoziierte Proteine die Transkription zu regulieren vermag. Die Kernmembran setzt sich aus einer äußeren und einer inneren Membran, den Poren sowie einer Schicht aus Lamin-Filamenten zusammen. Lamine sind für eine Vielzahl von Funktionen verantwortlich, darunter Chromatinorganisation und Genregulation, welche über ein Set diverser Proteine gesteuert werden. Drei von diesen Proteinen, MAN1, EMERIN und Lamin-assoziiertes-Polypeptid 2β (LAP2β), fungieren als Bindungspartner für GCL (Nili et al., 2001a; Holaska et al., 2003; Mattout-Drubezki 31 1. Einleitung & Y. Gruenbaum, 2003; Gruenbaum et al., 2005; Mansharamani & Wilson, 2005). GCL rekrutiert wiederum den Heterodimer E2F-DP3 an die innere Kernhülle. E2F ist ein Transkriptionsfaktor, der Gene reguliert, die für den Eintritt in die S-Phase verantwortlich und somit am Zellzyklus beteiligt sind (La Thangue, 1994; Lam & La Thangue, 1994). E2F-Proteine werden aber erst funktionell, wenn sie an ein Mitglied der DP-Familie binden. 1999 wurde ein mögliches Ortholog von Drosophila-GCL, das "DP-interacting protein" (DIP) als Bindungspartner von DP3 identifiziert. GCL bindet DP3 über eine BTB/POZ-Domäne (Broad-Complex, Tramtrack und Bric-abrac/Pox-Virus und Zinkfinger), ein konserviertes Protein-Protein-Interaktionsmotiv, und fixiert damit auch E2F an die Kernmembran, was eine Repression der Zielgene von E2F, und somit einen Zellzyklusarrest zur Folge hat (Bardwell & Treisman, 1994; de la Luna et al., 1999; Leatherman et al., 2000; Nili et al., 2001a). Ein weiterer Bindungspartner von Mm-GCL ist das Produkt des Gens „tumor susceptibility gene 101" (tsg101). TSG101 inhibiert den Abbau des Onkoproteins MDM2, welches Teil einer regulatorischen Feedback-Schleife mit dem Tumorsuppressor p53 ist (Masuhara et al., 2003). 2007 konnte ein Ortholog von gcl auch in der Ratte nachgewiesen werden (Rgcl2), wo es im Hoden exprimiert ist. Konfokale Mikroskopie offenbarte, dass RGCL-2 im Zytoplasma wachsender Spermatozoen synthetisiert wird und teilweise ins Flagellum integriert ist (Murayama et al., 2007). 1.3.3 germ cell-less im Menschen Während maternales GCL in Drosophila notwendig ist, um die Keimzellen an sich zu etablieren, scheint gcl in der Maus hauptsächlich in der Spermatogenese zu fungieren. Hs-gcl (Homo sapiens gcl) hingegen wurde 2001 aufgrund seiner Position auf dem 2. Chromosom als Kandidatengen für das Alström-Syndrom charakterisiert. Das Alström-Syndrom ist eine autosomal rezessiv vererbte Krankheit, die mit Taubheit, Augenerkrankungen, Adipositas und Diabetes einhergeht. Die Analyse diverser menschlicher Gewebe ergab, dass Hs-GCL hoch in Pankreas und Hoden, und schwächer in Nieren, Herz und Plazenta exprimiert ist. Diese Daten, im Zusammenhang mit der Protein-Protein-Interaktion der BTB/POZ-Domäne, und den Ergebnissen aus der Maus zur beeinträchtigten Zellkernintegrität, könnten ein Hinweis auf die Funk- 32 1. Einleitung tion von Hs-GCL als möglichen Auslöser des Alström-Syndroms sein, weshalb eine ausführliche Sequenzanalyse des gcl-Gens in Alström-Patienten initiiert wurde (Nili et al., 2001b). Die zahlreichen Deformationen in Mäusespermien und die Expressionsdaten von Nili et al. (2001b) veranlassten Kleiman et al. die Rolle von gcl in sterilen Männern zu untersuchen. Histologische wie zytologische Analysen zeigten einen Zusammenhang zwischen dem Grad der Schädigung und Hs-GCL-Expression. In Abwesenheit von Hs-GCL war zudem eine eingeschränkte Motilität der Spermien zu beobachten. Die Trennung der Chromosomen in der Meiose hingegen verlief normal, was auf eine späte Funktion von Hs-GCL in der Spermatogenese hindeutet (Kleiman et al., 2003). 2012 konnte eine Co-Expression von Hs-GCL und GAGE in Hoden und, wichtiger, verschiedenen Krebstypen wie Lungen-, Leber oder Brustkrebs nachgewiesen werden. Hs-GCL ist kolokalisiert mit Laminen an der Zellmembran und rekrutiert die Proteine der GAGE-Familie aus dem Kernplasma an die Membran. GAGE-Gene sind nur in höheren Primaten zu finden und sind in gesunden Individuen auf die Keimzellen beschränkt. Entartete Zellen exprimieren aber ebenfalls GAGE-Proteine, weshalb sie mit der Tumorgenese in Verbindung gebracht werden. Inwieweit die Rekrutierung von GAGE-Proteinen an die Zellmembran Tumorwachstum begünstigt, ist jedoch ebenso wenig geklärt wie die Art der Bindung und die Funktion von GCL und der GAGE-Proteine an sich (Gjerstoff et al., 2006, 2008 und 2012). 33 1. Einleitung 1.4 homeobrain Die Steuerung der Segmentidentität entlang der Körperlängsachse eines Embryos erfolgt in den Bilateria über die Hox- und Parahox-Gene, eine Gruppe konservierter Transkriptionsfaktoren, die ihre Zielgene über eine spezifische DNA-Binde-Domäne, die Homöobox regulieren (Lewis, 1978; Akam, 1989; McGinis & Krumlauf, 1992; Moreno et al., 2011). 1993 entdeckten Dessain & McGinnis homeobrain (hbn; damals noch 57San) in Drosophila als ein neues Homöobox enthaltendes Gen. hbn liegt in der Nähe der "paired-like homeobox"-Gene orthopedia (otp) und Retinal Homeobox (Rx) auf dem rechten Arm des zweiten Chromosoms. hbn ist bereits im Syncytium in Form eines unten offenen Rings in der Region exprimiert, aus der später der dorsale Kopf hervorgehen wird. Im Verlauf der Zellularisierung verblasst die Expressionsdomäne auf der ventro-lateralen Seite zusehends und eine weitere Domäne wird lateral und posterior der ursprünglichen Domäne sichtbar. Während der frühen Gastrulation unterteilt sich diese mehrfach, und im Keimstreif-Stadium zeigt zusätzlich jedes KörperNeuromer ein Set von Zellen mit hbn-Expression. Später findet sich die mRNA posterior zwischen Mittel- und Dickdarm, im Gehirn und schwach im ventralen Nervenstrang (Walldorf et al., 2000). Ergebnisse von Fröbius und Seaver legen zumindest in Bilateria eine konservierte Funktion von hbn nahe. Ein Ortholog von homeobrain, homeobrain-like (hbnl), wurde im Borstenwurm Capitella sp. nachgewiesen. Wie in Drosophila ist hbnl in der Larvalentwicklung auf wenige Zellen im Gehirn (und den Augen) begrenzt. hbnl findet sich in einigen Zellen der sich ausbildenden Zerebralganglien, im optischen Nerv sowie außerhalb des Gehirns an der Grenze zum Pharynx. Eine Expression entlang des ventralen Nervenstrangs wie in Drosophila tritt jedoch nicht auf (Fröbius & Seaver, 2006). Phylogenetische Studien ergaben zudem, dass der Gen-Cluster um homeobrain (hbn, otp und Rx) bereits partiell in einer der primitivsten Formen der vielzelligen Tiere, dem Placozoa Trichoplax adhaerens, vorhanden ist. Der Cluster ist ebenfalls konserviert in Anthozoa wie der Seeanemone Nematostella vectensis, diversen Insekten, Nessel- und Weichtieren; hbn fehlt jedoch in allen Chordatieren (Mazza et al., 2010). 34 1. Einleitung 1.5 Ziele dieser Arbeit Die vorliegende Arbeit untergliedert sich in zwei aufeinander aufbauende Teile. Zuerst sollten im Rahmen des genomweiten RNAi-Screens „iBeetle" die Funktionen aller Gene des roten Mehlkäfers Tribolium castaneum hinsichtlich verschiedener Entwicklungsprozesse untersucht werden. Zu diesem Zweck wurden dsRNAs von ~ 500 Bp in jeweils zehn weibliche Puppen injiziert und die weitere Entwicklung sowie die F1-Generation der RNAi-Tiere analysiert und Ergebnisse in einer speziell dafür eingerichteten Datenbank dokumentiert (Schmitt-Engel et al., in Vorbereitung; iBeetle-Base, Göttingen). Auf Basis dieser Datenbank sollten am Ende des Screens Gene ausgewählt werden, die gemäß ihres formulierten Phänotyps im Embryo an der Etablierung der anterio-posterioren Achse beteiligt sein könnten. Die Reproduzierbarkeit und Spezifität des Phänotyps wurde durch die Injektion nicht-überlappender Fragmente überprüft, da siRNAs (kurze einzelsträngige oder doppelsträngige Ribo- nukleinsäuren) ungewünschte Gene auf mRNA- oder Proteine-Ebene treffen können (Jackson & Linsley, 2004; Scacheri et al., 2004). Im zweiten Teil der Arbeit sollten die vielversprechendsten Kandidaten (germ cell-less und homeobrain) auf ihre Funktion in der frühen Embryonalentwicklung von Tribolium mit Hilfe von RNAi, in-Situ-Färbungen und anderen molekularbiologischen Methoden untersucht werden. Dies ermöglicht nicht nur ein besseres Verständnis der Entwicklung von Tribolium und Kurzkeim-Insekten per se, sondern im Vergleich mit anderen Arthropoden möglicherweise auch Einblick in die Evolution der Funktion des Gens im Kleinen sowie der Embryonalentwicklung insgesamt (Peel, 2008; Fonseca et al., 2009; Rosenberg et al., 2009). 35 2. Material und Methoden 2. Material und Methoden 2.1 Puffer und Reagenzien Eine Liste der verwendeten Lösungen inklusive zugehöriger Rezepte befindet sich in Anhang A. 2.2 Tribolium castaneum im Labor Mit der Entwicklung transgener Techniken, der Entdeckung parentaler RNAi und der Sequenzierung des Genoms hat sich der rotbraune Reismehlkäfer zusehends als Modellsystem etabliert. Zur Zeit befinden sich in der Abteilung für Entwicklungsbiologie der Friedrich-Alexander-Universität Erlangen eine Vielzahl von Stämmen unterschiedlichen genetischen Hintergrunds. Soweit nicht anders gekennzeichnet, wurden in der vorliegenden Arbeit Individuen des Stammes pBA19 verwendet. Dieser zeichnete sich in Vorversuchen durch hohe Schlupfraten, niedrigen genetischen Hintergrund und eine hohe Überlebensrate nach RNAi-Injektionen aus und besitzt eine eGFP-Expression in der L1-Körper-Muskulatur. Je nach Größe der Population werden die Käfer in verschließbaren Plastikgefäßen der Abmessung 12 cm x 8 cm x 12 cm (L x B x H) bzw. 18 cm x 18 cm x 10 cm mit Lüftungsöffnungen gehalten (5 cm Durchmesser), die zum Schutz vor Milben mit 100 µm Gaze aus Polyamid verschlossen sind. Als Futter wird ein Mehl-HefeGemisch (doppelgriffiges Rosenmehl, Typ 405, Wiener Griessler; Bierhefe Pulver, Cenovis) verwendet, wobei auf 1 kg Mehl 50 g Bierhefe zugesetzt wird. Wasser wird nicht benötigt. Um der Verschleppung von Infektionen entgegenzuwirken, werden die Eier der Stammpopulationen alle drei bis vier Tage durch ein Siebe-System (710 µm und 300 µm Maschenweite, Analysesiebe, Retsch) vom Mehl und den Adulti getrennt und in ein separates Gefäß mit frischem Substrat überführt. In der Regel erfolgt die Aufzucht und Haltung der Tiere bei 25 °C. Im Versuch werden die Tiere zumeist in einen Inkubator auf 32 °C überführt, um die Entwicklung zu beschleunigen. Abhängig von der Temperatur benötigen Tribolium-Eier 48 (25 °C) bzw. 22 Tage (32 °C) für die vollständige Reife zum adulten Tier (Soko- 36 2. Material und Methoden loff, 1974; Tab. 1). Unter beiden Bedingungen halten Luftbefeuchter die Luftfeuchtigkeit in einem Bereich von ca. 35-70 %, da insbesondere kleine Populationen anfällig für Schwankungen sind. Tabelle 1: Generationszeiten von Tribolium in Abhängigkeit von der Temperatur 2.3 Temperatur 25 °C 32 °C Embryonalentwicklung 6,8 d 2,9 d larvale Stadien 31,2 d 14,6 d Puppenstadium 10,2 d 4,6 d Entwicklungsdauer 48 d 22 d Parentale RNAi 2.3.1 Alter und Geschlecht der parentalen Generation Soweit nicht anders gekennzeichnet, fanden die Experimente zur parentalen RNAi an weiblichen Puppen statt, die sich im letzten Drittel ihrer Metamorphose befanden – leicht erkennbar unter dem Stereomikroskop (C-DSD230, Nikon) an sklerotisierten (dunklen) Mandibeln und Flügeldecken. Die Identifizierung der Weibchen erfolgte im gleichen Arbeitsschritt, unter Zuhilfenahme der sexspezifischen Genitalpalpen am posterioren abdominalen Segment (Abbildung 4: Asterisk), die sich im Durchlicht oder durch Belichtung von der Seite einfach erkennen lassen. Die Injektion der adulten Tiere erfolgte in der Regel in den ersten zwei Monaten nach der Metamorphose. 2.3.2 dsRNA-Injektion Zur Vereinfachung der Injektion wurden, je nach Ansatz, 10-25 Puppen auf einem Objektträger aufgereiht und mit Kleber (Fixogum, Marabu) fixiert. Hierbei ist darauf zu achten, dass lediglich das letzte abdominale Segment am Objektträger befestigt wird, da ansonsten das Schlüpfen des Tieres aus der Puppenhülle behindert wird, was zum Tode führt. Die anschließende Injektion mit der genspezifischen Doppelstrang-RNA erfolgte im wesentlichen wie beschrieben (Bucher et al., 2002; Brown et al., 1999; Tomoyasu und Denell, 2004; van der Zee et al., 2006) unter dem Stereomikroskop (SMZ 645, 37 2. Material und Methoden Nikon), wobei der Übergang vom distalen, dunklen zum proximalen, hellen Bereich der Urogomphi der Puppe als Orientierung für eine brauchbare Öffnungsweite der Kapillarenspitze (Glaskapillaren mit Filament, Hilgenberg) diente. Das Optimale Injektionsvolumen erkennt man daran, dass sich die Puppe im Verlauf der Injektion etwas streckt, und die Elytren sich leicht vom Körper abspreizen. Injiziert wurde in der Regel dsRNA mit der Konzentration von 1 µg/µl, soweit nicht anderweitig angegeben. Puppen wurden generell ventro-lateral unterhalb der Flügelspitzen injiziert; adulte Tiere ebenfalls lateral durch die dorsale, dünnhäutige Epidermis (Abbildung 4). Zu Standardisierungszwecken wurde für alle pupalen und adulten Injektionen eine Druckluft-Apparatur (Femtojet express, Eppendorf) verwendet, welche die RNA über Schlauch, Nadelhalter und Kapillare mit einem konstanten Druck von 350 Pa in die Versuchstiere injizierte. Nach Abschluss einer Injektionsserie, oder beim Wechsel der RNA, wurde der Nadelhalter mit gefiltertem Millipore-Wasser und 70 %igem EtOH gespült und im Anschluss mit Druckluft ausgeblasen und bei Zimmertemperatur trocknen lassen. Abbildung 4: Schematische Darstellung parentaler RNAi in Tribolium Gezeigt sind eine weibliche (links) und männliche Puppe (rechts), unterscheidbar an den Genitalpapillen (Asterisk), und ein adulter Käfer. Der Pfeil (grau) markiert die Penetrationsstelle für die dsRNA Injektion. In den Puppen liegt diese seitlich zwischen den letzten abdominalen Segmenten, bei den Adulten muss der weiche Teil der Kutikula durch thorakalen Druck mit einer Federstahlpinzette freigelegt werden. Hier wird ebenfalls seitlich injiziert. 2.3.3 Eiablage und Auswertung Nach der Injektion wurden die Puppen für drei Tage in einer Petrischale mit frischem Mehl auf 32 °C inkubiert, an Tag 3 der Schlupf kontrolliert und darauf Männchen im Verhältnis 1:4 zugesetzt. An Tag 9 erfolgte die erste Eiablage, die für die Herstellung 38 2. Material und Methoden eines Kutikula-Präparates und somit zur Verifizierung der RNAi Verwendung fand. Im Anschluss wurde über einen Zeitraum von zwei Wochen einmal täglich eine Eiablage gewonnen, 0-24 h bzw. 0-16 h, und fixiert (vgl. 2.4), wobei einmal pro Woche, meist über das Wochenende laufend, erneut die Aktivität der RNAi durch ein Kutikula-Präparat überprüft wurde. Im Falle adulter RNAi wurden die Männchen bereits am Tag nach der Injektion zu den Weibchen gesetzt. Die erste Eiablage für Kutikula-Präparate erfolgten für drei Tage. 2.4 Fixierung und In-Situ-Hybridisierung A. Fixierung 1. Embryonen in Siebekörbchen (ø 200 µm) für 2 x 3 min in 50 % DanKlorix (Colgate-Palmolive) dechorionisieren und sorgfältig mit Leitungswasser spülen 2. Transfer in Fixierlösung bestehend aus 6 ml Heptan, 2 ml PEMS und 300 µl 37 %-iges Formol 3. 35-40 min auf Schüttelplattform fixieren, dann unter dem Abzug die untere, aquatische Phase mit Glas-Pipette abnehmen, 10 ml MeOH dazu und 30 s heftig schütteln 4. Zu Boden gesunkene Embryonen in Eppendorf-Gefäß transferieren; Rest im Bedarfsfall mit Spritze (0,7 mm; Primo) mehrmals aufziehen und heftig herauspressen und neuerlich abgesunkene (devitellinisierte) Embryonen auch in das Eppendorf-Gefäß überführen 5. Abschließend 2 x mit MeOH waschen und auf MeOH bei -20 °C lagern B. Hybridisierung und Färbung 1. Fixierte Embryonen mit 50 % MeOH/PBT spülen und Überstand abnehmen 2. 3 x mit PBT spülen 3. 2 x für jeweils 10 min mit PBT auf Drehrad waschen 4. 2 x mit PBT spülen, Überstand abnehmen und dann mit 250 µl PBT/ 250 µl Hybe-B spülen 5. Flüssigkeit abnehmen und 250 µl frisches Hybe-B dazu 39 2. Material und Methoden 6. Flüssigkeit abnehmen und erstmals 250 µl Hybe-A zugeben; im Anschluss für 1 h ins Wasserbad (65 °C) 7. Hybe-A vorsichtig abnehmen, da Embryonen nun leicht durchsichtig, 30 µl Hybe-A dazu sowie 3-5µl der DIG-Sonde und vorsichtig mischen => über Nacht erneut auf 65 °C ins Wasserbad 8. Tag 2: 500 µl Hybe-B dazu und für weitere 20 min im Wasserbad (65 °C) inkubieren 9. Flüssigkeit abnehmen und mit 500 µl Hybe-B 15 min ins Wasserbad (65 °C) 10. Embryonen mit 500 µl Hybe-B/PBT spülen 11. 2 x kurz und 1 x 15 min mit PBT auf Drehrad waschen, dann noch einmal für 20 min mit 1 ml PBT und 200 µl 2,5 % Böhringer Block Reagenz 12. Flüssigkeit abnehmen und 1 h aufs Drehrad mit 1 ml PBT + 1 µl anti-DIG Antikörper (Roche) 13. 3 x kurz, 1 x für 20 min (Rad) und 2 x für 30 min (Rad) mit PBT waschen 14. 2 x für je 5 min mit Färbepuffer waschen, dann zu 1 ml frischen Färbepuffer mit 15 µl NBT/BCIP (Roche) wechseln und Embryonen in Blockschälchen überführen C. 15. Färbung sollte innerhalb von 30-90 min auftreten 16. Reaktionsstopp durch Waschen mit PBT (2 x kurz, 1 x 15 min) 17. Lagerung der Embryonen auf frischem PBT im Kühlschrank DNA-Färbung (optional – erleichtert Altersbestimmung der Blastoderm- Embryonen anhand von Kernzyklus und Kerngrößendifferenz zwischen extraembryonaler Serosa und Embryo) 1. PBT abnehmen 2. 20:1 PBT/Hoechst 33342 (Sigma) dazu (100 µl PBT : 5 µl Hoechst bzw. 200 µl : 10 µl bei vielen Embryonen) und im Dunkeln für 2 h auf dem Drehrad färben oder über Nacht im Kühlschrank lagern 3. 3 x kurz und 2 x für 15 min (Rad) mit PBT waschen 4. Lagerung in frischem PBT im Kühlschrank Die Analyse der in-Situ-Färbungen erfolgte unter dem Stereomikroskop (C-DSD230, Nikon) im Glasschälchen mit PBT. Zur Dokumentation wurden die Embryonen in ein 50 % Glycerin/PBS-Gemisch überführt und unter dem Fluoreszenzmikroskop 40 2. Material und Methoden (Axio Scope A1; Zeiss) mit dem Programm Helicon Focus einzeln fotografiert und mit Methode A zu einem Stapel zusammengefügt. 2.5 Kutikula- und Lebendpräparate Für Kutikula- und Muskelpräparate (iBeetle Screen) wurden die gelegten Eier für drei Tage auf 32 °C belassen, damit alle Embryonen ihre Entwicklung abschließen konnten (Tab.1; Sokoloff, 1974). Während dieser Zeit ruhten die Eier in Sieben mit einer Maschenweite von 300µm. Dies erlaubt es wildtypischen Larven, durchzukrabbeln und in das darunter liegende Speiseöl zu fallen, während Larven mit morphologischen Defekten bzw. embryonal letalen Phänotypen im Sieb verbleiben. Embryonen und nicht geschlüpfte Larven wurden in 200 µm Siebchen überführt und für 2 x 3 min in 50 % DanKlorix (Colgate-Palmolive) dechorionisiert und nach dem Spülen mit H2 O mit einem feinen Marderhaar-Pinsel in 50 % Hoyers/MilchsäureMedium eingebettet und über Nacht bei 60 °C inkubiert, um alle Weichgewebe aufzulösen. Die Auswertung der Präparate erfolgte zeitnah unter dem FluoreszenzMikroskop (Axio Scope A1 bzw. ApoTome; Zeiss). Des Weiteren wurde die Anzahl der ins Öl gefallenen Larven festgehalten. Diese Art der Präparation ermöglicht nicht nur die Analyse kutikulärer Strukturen mittels Fluoreszenz- oder Dunkelfeldmikroskopie, sondern auch die Aufbewahrung der Präparate über einen längeren Zeitraum hinweg und somit eine Re-Analyse. Im Gegensatz zu den Kutikula-Präparaten erfolgte die Analyse der Muskulatur noch am gleichen Tag, da nur lebende Muskeln ein distinktes eGFP-Muster aufweisen. Hierfür wurden die Eier in Sieben mit einer Maschenweite von 180 µm inkubiert, im Anschluss alle Embryonen in Voltalef-Öl (Lehmann & Voss & Co) überführt und unter dem Fluoreszenz-Mikroskop (Axio, Scope A1; Zeiss) hinsichtlich Anzahl, Morphologie und Fluoreszenz-Intensität ausgewertet. Die Präparate im Screen wurden mit dem Programm ProgRes (Mac) von dokumentiert, alle anderen Kutikula-Präparate unter dem ApoTome (Zeiss) mit dem Programm AxioVision unter der Verwendung von Z-Stapeln. 41 2. Material und Methoden 2.6 Embryonale Injektionen Für die embryonale Injektion wurden 1h alte Gelege verwendet (32 °C). Die Embryonen reiften 1h bei 32 °C auf frischem Mehl nach, wurden dann mit H2 O gewaschen und einzeln mit einem feinen Marderhaarpinsel auf einem Objektträger entlang der Längskante so aufgereiht, dass der anteriore abgeflachte Pol zur Kante zeigt (Abb. 5). Die Injektion der RNA erfolgte unter dem Mikroskop (SM-LUX; Leitz-Wetzlar) mit Hilfe eines 3D-Mikromanipulators (Narashige), wobei eine Glaskapillare mit Filament (Hilgenberg) Verwendung fand, die zuvor am Objektträgerrand unter optischer Kontrolle gebrochen wurde. Nach dem Aufreihen wurden die Embryonen mit Voltalef-Öl (Lehmann & Voss & Co) bedeckt, anterior injiziert und in einer Feuchtkammer mit 100 ml 15 % NaCl bei 32°C bis zum Schlüpfen inkubiert (4d). Die DNA wird standardmäßig in einer Konzentration von 1µg/µl injiziert und zur besseren Kontrolle 1:5 mit einer Phenolrotlösung (0,5% in PBS, Sigma) versetzt. Die Embryonen verbleiben für 3 Tage auf 32 °C, dann wird das Öl mit Heptan abgewaschen, die Embryonen kurz trocknen lassen und anschließend wie unter 2.5 beschrieben in Hoyers/Milchsäure eingebettet. Abbildung 5: Schematische Darstellung einer embryonalen Injektion Dargestellt ist ein Objektträger (blau) sowie die darauf aufgereihten Embryonen (schwarz; vergrößerte Darstellung). Der anteriore Pol, erkennbar an abgeflachter Spitze zeigt zur Kante respektive der mit RNA gefüllten Glaskapillare (rote Spitze). Alle Embryonen sind mit Voltalef-Öl bedeckt, (10S, Lehmann & Voss & Co), hier als gelblicher Klecks markiert. 42 2. Material und Methoden 2.7 Amplifikation individueller Genfragmente zur Generierung von dsRNA und DIG-Sonden Die Amplifikation der Tribolium-Genfragmente für den Screen und alle Analysen dieser Arbeit erfolgten in der Regel anhand des Standard-Protokolls von Mirko Theis (Feb.21, 2011) der Firma Eupheria. PCR-1: ∑ 25 µl (Bioline Kit) Wasser 4,8 µl 10x PCR Puffer 2,5 µl 50 mM MgCl2 0,8 µl 100 mM dNTP-Mix (25 mM each) 0,2 µl Template, cDNA 0,3 µl Taq-Red Polymerase (Bioline) 1 µl => 10 µl pro PCR-Tube zusetzten Vorgelegt in PCR-Tube: 2 µM Genspezifischer Primer 2,5 µl (von jedem) Wasser 10 µl PCR-Protokoll: 94 °C 2-3 min 94 °C 60 °C 72 °C 30 sec 30 sec 30 sec x6 94 °C 62 °C 72 °C 30 sec 30 sec 30 sec x6 94 °C 65 °C 72 °C 30 sec 30 sec 30 sec x 22 72 °C 5min 4 °C ∞ 43 2. Material und Methoden Die cDNA wurde aus RNA verschiedener embryonaler Entwicklungsstadien (0-4 h, 0-8 sowie 0-24 h) hergestellt. Hierfür wurde die RNA mit Hilfe des TRIzol®ReagentProtokolls von GibcoBrl gewonnen, anschließend die Konzentration am Photometer (BioPhotometer, Eppendorf) vermessen und mit dem Transcriptor High Fidelity cDNA Synthesis Kit (Roche) unter Verwendung von Random Hexamer Primern zu cDNA umgeschrieben. PCR-2: ∑ 50µl (Bioline Kit) Wasser 39,4 µl 10x PCR Puffer 5 µl 50 mM MgCl2 2 µl 100 mM dNTP-Mix 0,4 µl 100 µM Primer Forward 0,1 µl 100 µM Primer Backward 0,1 µl Taq-Red Polymerase (Bioline) 2 µl => 48 µl in PCR-Tube auf 1 µl PCR-1 Lösung PCR-Protokoll: 94 °C 3 min 94 °C 60 °C 72 °C 30 sec 30 sec 30 sec 72 °C 5 min 4 °C ∞ x 42 Zur Überprüfung der Amplifikation wurden 3 µl des PCR-Produkts direkt auf ein 1 %iges Agarosegel aufgetragen. Anhang B bietet eine Liste der verwendeten Primer (alle Metabion) und Abb. 6 eine Übersicht des iBeetle-Primer-Designs, welches auch für die Generierung eigener Primer herangezogen wurde. So erhielt der genspezifische Forward-Primer (links) eine SP6-Mini-Sequenz und der spezifische Backward-Primer (rechts) eine T7-Mini-Sequenz. Diese Primer wurden in der ersten PCR verwendet, während in der zweiten PCR (siehe unten) die SP6-Mini-Seuqenz durch einen SP6-Primer (für 44 2. Material und Methoden Sondensynthese) bzw. einen T7-SP6-Primer (dsRNA-Synthese) und die T7-MiniSequenz durch einen T7-Primer ersetzt und somit vervollständigt wurden. Abbildung 6: iBeetle-Primer-Design nach Eupheria Das Schema zeigt das Primer-Design für iBeetle, entwickelt von der Firma Eupheria. Im Schritt eins werden Mini-Sequenzen der SP6- und T7-Primer an genspezifische Primer gehängt, welche in einer weiteren PCR durch komplette SP6- und T7-Primer erkannt und ausgebaut werden. Dies ermöglicht die genspezifische Generierung von dsRNA und Sonden. 2.8 In-Vitro-Transkription der genspezifischen dsRNA Protokoll basiert auf T7-Ambion Megaskript Kit. 1 µg Template Je 2 µl von ATP, CTP, GTP und UTP 2 µl 10x Reaktionspuffer => Mischen => zu 18 µl mit H2 O auffüllen 2 µl Enzym => Mischen und kurz zentrifugieren => für 4-16 h auf 37 °C Aufreinigen des RNA-Produkts: 1. 30 µl H2 O (autoklaviert) zur Probe geben 2. 50 µl 10 M NH4 Ac- zu, mischen, 300 µl EtOH (100 %, unvergällt) zugeben, mischen • für 30 min oder über Nacht auf -20 °C fällen 45 2. Material und Methoden 3. für 30 min bei 4 °C in Zentrifuge (15000 rpm) geben, mit 70 %igem EtOH (unvergällt) waschen und erneut für 10 min bei 4 °C in Zentrifuge (15000 rpm) 4. Alkohol sorgfältig abnehmen und ca. 10 min Lufttrocknen bei 37 °C 5. Abschließend Pellet in 15 µl H2 O lösen und Konzentration am Photometer (1 µl RNA in 100 µl H2 O) messen Injiziert wurde, soweit nicht anders angegeben, RNA mit einer Konzentration von 1 µg/µl, wobei auf 10 µl RNA 1 µl Phenolrot (Sigma)beigemischt war. 2.9 In-Vitro-Transkription genspezifischer Sonden x µl DNA (500 ng) x µl H2 O 1 µl 10x Transkriptionspuffer 1 µl rNTP-Mix DIG 0,5 µl RNAse Inhibitor (20 Units) 1 µl RNA Polymerase (T3/T7/SP6) => insgesamt 10 µl Ansatz 1. Ansatz für 2 h auf 37 °C inkubieren 2. Im Anschluss 90 µl H2 O, 1 µl tRNAs (20 µg/µl), 45 µl 10 M NH4 Ac und 435 µl EtOH (100 %, unvergällt) zugeben, mischen 3. 2 h bei -20 °C ausfällen lassen 4. für 20 min bei 4 °C in Zentrifuge (15000 rpm) geben, mit 70 %igem EtOH (unvergällt) waschen und erneut für 10 min bei 4 °C in Zentrifuge (15000 rpm) 5. Alkohol sorgfältig abnehmen und Pellet ca. 10 min lufttrocknen bei 37 °C 6. Abschließend Pellet in 50 µl H2 O lösen und 3 µl in in-Situ testen 46 2. Material und Methoden 2.10 Fluoreszenzmarkierung spezifischer Proteine 2.10.1 Generierung des gcl-Vektors Ausgangsvektor für das angestrebte Fluoreszenzlabelling von mRNA war das Plasmid pCS2+, das von A. Schambony (Universität Erlangen) zur Verfügung gestellt wurde. Obwohl ursprünglich konstruiert für Xenopus-Embryonen, funktioniert pCS2 in einer Vielzahl von Organismen, darunter Säuger, Vögel und Zebrafisch Danio rerio (Turner, Universität von Michigan). Benton et al. nutzten 2013 erfolgreich pCS2 als Template-Plasmid für in vitro Transkription und transiente Expression in Tribolium-Embryos. pCS2 basiert auf dem pBluescript-Vektor und beinhaltet eine Resistenz gegen Ampicillin, mehrere Promotorbindestellen (SP6, T3 und T7) sowie eine SV40 „late“ Polyadenylierungsstelle (Abb. 7). Um die kodierende Sequenz von germ cell-less in den Vektor zu klonieren, wurde mit Hilfe einer „nested“ PCR gcl selbst und ein Teil der nicht-translatierten Regionen der 5’ und 3’ Enden amplifiziert, da diese wichtige regulatorische Elemente enthalten können. Im Gegensatz zu den äußeren Primern, die lediglich Gen-spezifische Sequenzen beinhalten, wurden an den inneren Primern noch Erkennungschnittstellen für Restriktionsenzyme hinzugefügt – für den späteren Einbau in das Plasmid (Abb. 8). Abbildung 7: Übersicht des zur Gcl-Überexpression verwandten Vektors pCS2 Der Vektor pCS2 besteht aus 4095 Basenpaaren. Diese enthalten mehrere Primer-Bindestellen, darunter Promotor für SP6 (hellblau), T7 (blau) und T3 (dunkelblau), die Polyadenylierungsstelle (SV40 late polyA; hellgrau), die Resistenz gegen Ampicillin (AmpR; grau) und ein Cytomegalovirus Promotor (CMV; anthrazit) komplettieren den Vektor. Zwischen SP6 und T3 finden sich Schnittste llen für XBa1 und SnaB1, die für den Verdau Verwendung fanden. 47 2. Material und Methoden Äußere Primer FW-Primer: TATTGTTGGAGAGCGTCGTG (Tm = 59 °C) BW-Primer: AGCAATCACAAAACACATCAACT (Tm = 58 °C) Innere Primer mit Restriktionsschnittstellen FW-Primer: 5’ TTTAATCTAGAGCGGCCGCTGAACTGACTGCAATGGAGGT 3’ Überhang-Xba1-Not1-genspezifischer Primer FW (Tm = 59,6 °C) BW-Primer: 5’ TTTAATACGTAGCGATCGCACCCTCATGCAAGAACACAAAA 3’ Überhang-SnaB1-AsiS1- genspezifischer Primer BW (Tm = 59 °C) „nested“ PCR: ∑ 50µl nach New England Biolabs (NEB) 5x Phusion Puffer HF 10 µl 2 mM dNTP-Mix 5 µl 20 µM Primer Forward 1,25 µl 20 µM Primer Backward 1,25 µl Template x µl (< 250 ng) Phusion Polymerase 0,5 µl => 50 µl mit H2 O PCR-Protokoll 98 °C 30 sec 98 °C 55 °C 72 °C 10 sec 30 sec 20 sec/kb 72 °C 5 min 4 °C ∞ x 30 Im Anschluss an die PCR erfolgte die Aufreinigung via Gelextraktion (MinElute Gel Extraction Kit 50; Qiagen), um Primer, Oligonukleotide usw. zu entfernen. 48 2. Material und Methoden 2.10.2 Doppelverdau des pCS2-Vektors sowie des gcl-Fragments Der Verdau erfolgte nach Protokoll von NEB; 50 µl für 1 µg. x µl DNA (1 µg) 5 µl 10x Puffer 0,5 µl 100x BSA 10 Units der Enzyme zu 50 µl mit H2 O auffüllen Der Ansatz verblieb 2 h bei 37 °C und wurde im Anschluss bei 80 °C für 20 min hitzeinaktiviert. Im Anschluss erfolgte die De-Phosphorylierung des Vektors mit der Antarctic-Phophatase (NEB) für 60 min bei 37 °C, dann die Hitzeinaktivierung bei 65 °C für 5 min. Zum Abschluss wurden Backbone-Vektor (4072 Bp) und Insert (1574 Bp) durch eine Gelextraktion (MinElute Gel Extraction Kit 50; Qiagen) isoliert. 2.10.3 Generierung des gcl-Klons Nach der Aufreinigung erfolgte die Ligation der Konstrukte mit Hilfe der T4-DNALigase (NEB). Für die Berechnung des optimalen Verhältnisses aus Vektor und Insert hat sich im Labor folgende Formel bewährt: Masse Fragment (ng) = 5x Masse Vektor (ng) x Länge Fragment (Bp) / Länge Vektor (Bp) Die Ligation erfolgte über Nacht bei 16 °C. 1 µl der Reaktion wurde in 50 µl E. coliStamm TOP 10 One Shot transformiert (MicroPulser; BIORAD) und die Bakterien in verschiedenen Verdünnungen ausplattiert. Die Bakterien verblieben sodann über Nacht auf 37 °C. Am nächsten Tag erfolgte die Auswahl von 25 isolierten Kolonien, die per Colony-PCR mit den Primern Sp6 und T3 und Testverdau geprüft wurden. Ein viel versprechender Klon wurde mit 4 Primern, die die gesamte Länge des Plasmids abdeckten, zur Sequenzierung zu Seqlab nach Göttingen geschickt (Abb. 9). Primer 1 vorwärts (SP6): 5’ GAT TTA GGT GAC ACT ATA GA 3’ Primer 2 vorwärts (gcl-Vek-in-F): 5’ ACA GCC ACC ATG TTC CAA CT 3’ Primer 3 rückwärts (gcl-Vek-in-BW): 5’AAA ATC CTG CCA CAC CTG AG 3’ Primer 4 rückwärts (T3): 5’AAT TAA CCC TCA CTA AAG GG 3’ 49 2. Material und Methoden Abbildung 8: Schematische Übersicht zur Klonierung von gcl in den Vektor pCS2 Dargestellt ist die Klonierung der kodierenden Sequenz von gcl in den Vektor pCS2. Zuerst wurden mittels PCR Restriktionsschnittstellen an das 5’- und das 3’-Ende des Gens angefügt, sodass eine direktionale Klonierung in den Vektor pCS2 stattfinden kann. Das neue Konstrukt sowie der Vektor wurden im Anschluss verdaut und prozessiert wie unter 2.10.2 und 2.10.3. Abbildung 9: Übersicht des pCS2-gcl-Vektors Dargestellt ist das pCS2-Vektor (oder auch pCS2+) mit dem gcl-Insert. Das fertige Konstrukt enthält mehrere Promotoren, darunter einen für SP6 (hellblau), T3 (blau) sowie die für die Sequenzierung genutzten Primerbindestellen in grün. Weiterhin dargestellt sind die Resistenz gegen Ampicillin (AmpR; grau), die Polyadenylierungss telle (SV40 late polyA; hellgrau) und ein Cytomegalovirus Promotor (CMV; anthrazit). 50 2. Material und Methoden 2.10.4 Einbau von EGFP Der pCS2-gcl-Klon wurde im Anschluss mit SPE1 und AsiS1 verdaut. SPE1 liegt im hinteren Viertel von gcl, ASiS1 hingegen hinter der genspezifischen Sequenz, da es mit dem genspezifischen Primer (vgl 2.10.1) mit in den Vektor kloniert wurde. Die Firma GeneCust synthetisierte für uns eine Sequenz aus ca. 390 bp ces gclTranskripts, fusioniert mit EGFP, das nach dem Verdau mittels der Schnittstellen Spe1 und Sac2 in den pCS2-gcl-Klon ligiert wurde (vgl 2.10.3 für Daten zur Ligation). Das künstliche Konstrukt von GeneCust setzte sich folgendermaßen zusammen: Spe1 - part of Tc-gcl - BspE1 and another TCCGGA (=LeuGly as linker) - EGFP - BspE1 AsiS1 - SV40 - Asc1 - Sac2 ACTAGTCTCAGGTGTGGCAGGATTTTGATGGTGAACGAGCGACATGTGTGGAGGTGGA CCGGATTCCATTTCGGAATGGATTTACTCATGATGACCGATGGCTACACGGTCTCTGTC AAAAGAAACCACAGGTCGGAGTTCGAGCAACTGTTAAGCTTCCAAACCTCGAGACATAT AGCTATAAGGGTAACCGTTGTCAAATTGAACGAGCAAAGACAGATAGTTTACTCCAAGC AGTCAGAGATCCTAAAACTGAGCCTTGCCAAAGGGGAGGAGGTGCAGATAATGGCGCT TGACAAAGACTTGGAGTATCCTATCATCATATCGGCTAATATACTGTACACTTCCCCCGA CTCACAGGAAATAAATCAGGAAGAGGTCGTGCAAACTGTCTCCGGATTGATGGTGAGC AAGGGCGAGGAGCTGTTCACCGGGGTGGTGCCCATCCTGGTCGAGCTGGACGGCGAC GTAAACGGCCACAAGTTCAGCGTGTCCGGCGAGGGCGAGGGCGATGCCACCTACGGC AAGCTGACCCTGAAGTTCATCTGCACCACCGGCAAGCTGCCCGTGCCCTGGCCCACCC TCGTGACCACCCTGACCTACGGCGTGCAGTGCTTCAGCCGCTACCCCGACCACATGAA GCAGCACGACTTCTTCAAGTCCGCCATGCCCGAAGGCTACGTCCAGGAGCGCACCATC TTCTTCAAGGACGACGGCAACTACAAGACCCGCGCCGAGGTGAAGTTCGAGGGCGAC ACCCTGGTGAACCGCATCGAGCTGAAGGGCATCGACTTCAAGGAGGACGGCAACATCC TGGGGCACAAGCTGGAGTACAACTACAACAGCCACAACGTCTATATCATGGCCGACAA GCAGAAGAACGGCATCAAGGTGAACTTCAAGATCCGCCACAACATCGAGGACGGCAGC GTGCAGCTCGCCGACCACTACCAGCAGAACACCCCCATCGGCGACGGCCCCGTGCTG CTGCCCGACAACCACTACCTGAGCACCCAGTCCGCCCTGAGCAAAGACCCCAACGAGA AGCGCGATCACATGGTCCTGCTGGAGTTCGTGACCGCCGCCGGGATCACTCTCGGCAT GGACGAGCTGTACAAGTAATCCGGAGCGATCGCCAGACATGATAAGATACATTGATGA GTTTGGACAAACCACAACTAGAATGCAGTGAAAAAAATGCTTTATTTGTGAAATTTGTGA TGCTATTGCTTTATTTGTAACCATTATAAGCTGCAATAAACAAGTTAACAACAACAATTGC ATTCATTTTATGTTTCAGGTTCAGGGGGAGGTGTGGGAGGTTTTTTGGCGCGCCATCCG CGG Nach erneuter Verifizierung mittels Sequenzierung (Seqlab) erfolgte die Synthese der sense-RNA mit Hilfe des mMessage mMachine® T7 Ultra Kit (Ambion) aus dem mit Spe1-Verdau linearisierten Vektor. Die sense-RNA wurde, wie in 2.6 beschrieben, in einer Konzentration von 2 µg/µl in Embryonen injiziert. Die Embryonen verblieben dann für 3h auf 32 °C und wurden danach unter dem Fluoreszenzmikroskop (Axiophot, Zeiss) untersucht. pT7-LifeAct-EGFP (Benton et al., 2013) diente als interne Kontrolle. Die Vektorkarte des finalen Klons findet sich im Anhang C. 51 2. Material und Methoden 2.11 Antikörperproduktion und Immunohistochemische Färbungen 2.11.1 Generierung von polyklonalen Peptid-Antikörpern gegen GCL Zur Untersuchung der subzellulären Lokalisierung von GCL wurde die Firma Pineda (Antikörper-Service, Berlin) mit dem Design der Immunisierungs-Peptide und der anschließenden Generierung passender Antikörper beauftragt. Folgende Peptidsequenzen wurden zur Gewinnung der Antiseren verwendet: GCL-7 NH2-CKQVLRVNQNDDKGPSEVSN-CONH2 (20 AS) GCL-9 NH2-CTVVKLNEQRQIVYSKQSEI-CONH2 (20 AS) Pro Sequenz wurden jeweils 3 Kaninchen über 2 Monate hinweg immunisiert (Tag 1, Tag 20, Tag 30, Tag 40 + erste Blutentnnahme und Tag 61). Die Auswahl der Tiere traf ich anhand des Verhaltens ihrer Präimmunseren in Testfärbungen. Da auch 160 Tage und 190 Tage nach Abschluss der Immunisierung in zwei Testfärbungen in Ovar und Embryo eine starke Überfärbung auftrat, wurden die Seren im Anschluss von Pineda mittels Affinitätschromatographie aufgereinigt. Nach 300 Tagen wurden die Kaninchen ausgeblutet, die Immunsseren auf -80 °C verbracht bzw. Aliquots für den Gebrauch auf -20 °C 1: 1 mit Glycerin gemischt. Die Seren der letzten Blutung wurden von mir 2x im Ovar und 3x in Embryonen in diversen Verdünnungen getestet. 2.11.2 Antikörper-Färbung in Embryonen Die Antikörper-Färbung erfolgte entweder an fixierten Embryonen (vgl. 2.4) bzw. im Fall der GCL-Antikörper - an Embryonen, die nach der Fixierung sofort in PBT überführt worden sind. Färbeprotokoll: 1. Methanol abnehmen, dann mit Methanol/PBT 2:1 und 1:1 waschen. 2. Embryonen in frisches PBT geben, einmal für 10 min und 2x 20 min in frischem PBT auf Rad waschen. 3. Im Anschluss PBT abnehmen und Embryonen in neuem PBT mit Blocking Reagenz (Roche) für 20 min auf dem Rad blocken (finale Verdünnung 2,5 %) 4. Überstand abnehmen und spezifischen Antikörper in 1 ml PBT zugeben. 52 2. Material und Methoden => bei Raumtemperatur 4 h auf Rad waschen oder über Nacht bei 4 °C 5. Mit PBT spülen und dreimal waschen auf Rad (10 min und 2x 20 min) 6. Überstand abnehmen und 1 ml vorher verdünntem 2. Antikörper (AP- oder Fluoreszenz- markiert) zugeben => für 2 h auf Rad 7. 2x kurz, 1x für 10 min (Rad), 2x für 20 min (Rad) und 1x für 30 min mit 1 ml PBT waschen 8. Bei AP-Antikörper: 2x für 5 min mit Färbepuffer waschen, dann zu 1 ml frischen Färbepuffer mit 15 µl NBT/BCIP (Roche) wechseln; Embryonen in Blockschälchen überführen 9. Färbung mit PBT stoppen: jeweils für 10, 20 und 30 min auf Rad waschen 10. Optional DNA-Färbung Hoechst 33342 (Sigma; vgl. 2.4 Sektion C) 11. Lagerung der Embryonen auf frischem PBT im Kühlschrank 2.11.3 Antikörper-Färbung im Tribolium Ovar 1. Ovare in eisgekühltem PBS präparieren (Salznapf) und rasch in Eppendorf mit Fixierungslösung (140 µl Formol + 860 µl PBS) überführen und für 30 min auf Eis fixieren. 2. 1x waschen mit 1ml PBS; 3. 2x waschen mit 1 ml Ovar-PBT; je 15 min auf Rad bei Raumtemperatur 4. Zugabe des spezifischen 1. Antikörpers, verdünnt in PBT => über Nacht auf 4 °C (vor Licht schützen) 5. Färbelösung abnehmen und 3x mit Ovar-PBT waschen (15 min; Rad; RT) 6. Zugabe des spezifischen 2. Antikörpers, verdünnt in Ovar-PBT (Anhang A) (Wahlweise TRITC-Phalloidin (1:50) und Hoechst (1:20; beide Sigma) zugeben und über Nacht auf 4 °C (vor Licht schützen) 7. 3x mit frischem Ovar-PBT waschen; je 15 min auf Rad bei Raumtemperatur 8. Für DIG-Antikörper: 2x für 5 min mit Färbepuffer waschen, dann zu 1 ml frischen Färbepuffer mit 15 µl NBT/BCIP (Roche) wechseln; Embryonen in Blockschälchen überführen 9. Färbung mit PBT stoppen: jeweils für 10, 20 und 30 min auf Rad waschen. 53 2. Material und Methoden 10. Lagerung der Ovare auf frischem Ovar-PBT im Kühlschrank (nicht länger als eine Woche) Zur Analyse Ovare auf Deckgläschen in einen Tropfen Glycerin/PBS überführen, Ovariolen mit Minutiennadeln vereinzeln und Muskelhülle entfernen; im Anschluss Deckglas mit Wachsfüßchen auflegen. Alle immunohistochemischen Färbungen (Embryonen und Ovar) wurden unter dem ApoTome (Zeiss) mit Hilfe des Programms AxioVision analysiert und dokumentiert. 54 3. Ergebnisse 3. Ergebnisse 3.1 Der genomweite RNAi-Screen identifiziert eine Vielzahl neuer Genfunktionen In diesem Teil der Arbeit werden Ergebnisse meines spezifischen Datasets (n = 987 Gene bzw. dsRNAs, ohne Kontrollen) präsentiert, und mit einem repräsentativen Teil des kompletten Screen verglichen. Dieser repräsentative Teil entspricht den bis Dezember 2011 in die Datenbasis eingegebenen Daten, nämlich circa 3500 dsRNAs im pupalen und 4500 dsRNAs im larvalen Screen (Schmitt-Engel et al., in Vorbereitung). Um die Ergebnisse des Screens besser zu veranschaulichen, werde ich außer meinen eigenen Daten exemplarisch auch einige Beispiele aus den verschiedenen anderen Fragestellungen des Screens darstellen. Insgesamt ergab sich für 558 (57 %) der von mir getesteten 987 dsRNAs ein Phänotyp (Abb. 10), wobei eine Untergrenze von > 30 % Penetranz für Muskel- und Segmentierungsphänotypen, > 50 % für Ovarphänotypen sowie > 80 % in Bezug auf die Letalität angesetzt wurde. Rund 90 der 987 analysierten Gene (9 %) führten innerhalb von 14 Tagen nach Injektion zum Tod (min. 5 von 10), trafen also offenbar essentielle Gene, die zum Beispiel für den zellulären Grundstoffwechsel, oder auf Ebene des Gesamtorganismus für Fressverhalten oder das Vermögen zur Nährstoffaufnahme notwendig sind. Vertiefende Analysen zur Klärung dieser letalen Funktionen waren innerhalb des zeitlichen Rahmens des Screens nicht vorgesehen, da wir uns primär für Entwicklungsgene interessierten. Diese Zahlen decken sich weitestgehend mit den Ergebnissen der anderen Projekte im iBeetle-Screen. So führten insgesamt 56,3 % aller dsRNAs (n = 3.400) zu mindestens einem Phänotyp, wobei 21,1 % nur im pupalen Screen auftraten, 24,6 % hingegen sowohl im pupalen als auch im larvalen Teil (Schmitt-Engel et al., in Vorbereitung). Somit ergibt sich eine Summe von 1555 (45,7 %) Phänotypen nach pupaler Injektion, also etwas niedrigere Quote als bei mir. Dies könnte daran liegen, dass ich die Schwelle für Annotation womöglich etwas stringenter angesetzt habe oder individuelle Unterschiede im ds-RNA-Injektionsvolumen zwischen den einzelnen Screenern vorliegen. 55 3. Ergebnisse Betrachtet man die Gesamtheit der pupalen Injektionen des Screens, so resultierten 202 (6,1 %) der dsRNAs im Tod der injizierten Weibchen. Erneut können hier individuelle Unterschiede der Screener zu Grunde liegen, aber auch die unterschiedlichen Gene, die jeder Screener injiziert hat, könnten hier eine Rolle spielen. Im larvalen Screen waren es 794 (23,5 %). Diese Diskrepanz lässt sich erklären, wenn man bedenkt, dass im larvalen Screen alle für die Metamorphose relevanten Gene ausgeschaltet wurden. Zudem wurden für die Berechnung der Sterblichkeitsrate unterschiedliche Zeitpunkte herangezogen. So zählten im pupalen Screen alle Tiere, die zwischen Tag 0 und Tag 11 verstarben, im larvalen Screen hingegen gab es auch an Tag 22 noch einen Analyseschritt. Knock-downs mit späteren Auswirkungen konnten so im larvalen Teil des Screen noch erfasst werden, wurden im pupalen allerdings ausgelassen. 8% Kein Phänotyp Adult letal (≤ 14 dai) 21% Kachexie 43% Ablageblock Ovarphänotyp 2% Muskelphänotyp 6% Kutikulaphänotyp 3% 8% 9% früh embryonal letal/ unbefruchtet Abbildung 10: Zusammenfassung meiner Ergebnisse (pupaler RNAi, Stand Dezember 2011) Von meinen 987 dsRNA-Injektionen führten 57 % zu einem detektierbaren Phänotyp (558), 9 % (89) resultierten im Tod der Weibchen in den ersten vierzehn Tagen nach Injektion (orange). Eine red uzierte Legeleistung lag in 17 % (163) der Injektionen vor. In 8 % (76) dieser Fälle litten die injizierten Weibchen an Kachexie (rot), einer starken Unterernährung, die sich durch das Fehlen von Fettkörper und Oozyten äußert;. 3 % (29) dieser dsRNAs führten zu "Ablageblock" (magenta), einer Störung der Eiablage, nicht der Eiproduktion selbst, so dass letztendlich nur 6 % (58) einen echten Defekt in der Oogenese- bzw. Ovariogenese aufwiesen (lila). Von den Nachkommen zeigten 2 % (19) der Embryonen einen Muskelphänotyp (grün) und 29 % (287) einen Segmentierungsdefekt (dunkelblau) und 81 (8 %) waren früh embryonal oder unbefruchtet (blau). Für die Wertungen wurden folgen Grenzen angesetzt: Adult letal: mindestens 80 % der Weibchen verstorben innerhalb der ersten 14 Tage; Kachexie, Ablageblock und Ovarphänotyp: mindestens 50 % der Ovare zeigen den Phänotyp; Muskel- und Kutikulaphänotypen > 30 % betroffen; früh embryonal letal / unbefruchtet > 80 %. 56 3. Ergebnisse 3.1.1 Defekte der Ovario- oder Oogenese nach pupaler Injektion In Insekten sind zwei verschiedene Typen der Oogenese bekannt. Ausgehend von der Struktur der Ovariolen unterscheidet man den panoistischen und meroistischen Typus, wobei sich letzterer in zwei Gruppen untergliedert. Tribolium besitzt ein Ovar des telotroph-meroistischen Typus: Das Ovar setzt sich aus zwei Halbovaren zusammen, jedes bestehend aus 5-6 Ovariolen, die jeweils durch ein Filament an der Körperwand fixiert sind. Dem Terminalfilament schließt sich das Tropharium an, der Bereich, in dem die Nährzellen sitzen. Während die Oozyte durch das Vitellarium wandert, bleibt sie über Mikrotubuli-stabilisierte Kanäle (Nährstränge) mit den Nährzellen verbunden und erhält Nährstoffe und maternale Faktoren, die für die Embryogenese notwendig sind. Reife Oozyten / Eier aus beiden Halbovaren werden über einen gemeinsamen Eileiter abgelegt (Bonhag, 1958; Trauner und Büning, 2007). Im Gegensatz zu dem polytroph-meroistischen Typus von Drosophila, bei dem jede Eizelle mit fünfzehn Nährzellen in einem Follikel verpackt ist, ist über die Oogenese Triboliums wenig bekannt. Auffällig ist jedoch, dass die Proliferation der Keimzellen in pupalen Stadien endet, während die somatische Stammzell-Nische über den gesamten Zeitraum der Eiproduktion im adulten Tier aktiv bleibt (Trauner und Büning, 2007). Dies bietet die Möglichkeit, beide Nischen getrennt voneinander zu betrachten und für diese Nische spezifische Faktoren zu entdecken. Defekte am Ovar sollten im Screen durch eine reduzierte Produktion von Eiern an Tag 9 und 11 offenbar werden, woraufhin die Ovare von vier der zehn Weibchen präpariert, fixiert und am gleichen Tag unter dem Lichtmikroskop hinsichtlich ihrer Morphologie untersucht wurden. Insgesamt waren in 8,9 % aller 3400 Injektionen (pupal und larval) spezifische Defekte des Ovars zu beobachten. Von diesen 304 dsRNAs führten 227 zu einem Phänotyp im pupalen Screen, aber nur 100 im larvalen Teil, wobei 23 Gene überlappten (Schmitt-Engel et al., in Vorbereitung). In meinen 987 dsRNA-Injektionen kam es in 213 Fällen (22 %) zu einem reduzierten Legeverhalten. Allerdings konnte ich nur in 58 Fällen (ca. 6 %) einen spezifischen Phänotyp beobachten (Abb.11). In den restlichen Fällen war kein spezifischer Oogenese-Defekt feststellbar bzw. war das Weibchen entweder unterernährt, „kachetisch“ (76 Fällen, 8 %), oder wies einen Ablageblock (29 dsRNAs, 3 %) auf (Ver57 3. Ergebnisse gleiche Abb. 10). Kachexie drückt sich oftmals im noch lebenden Tier durch träge Bewegungen aus. Die Abdomen der Tiere wirken ausgetrocknet. In der Präparation ist auffällig, dass die Oozyten komplett fehlen. Ebenso ist der Fettkörper reduziert bis unauffindbar. Dies deckt sich mit Ergebnissen von Drummond-Barbosa und Spradling (2001), die zeigten, dass die Zellproliferation der somatischen StammzellNische und Keimzellen im Ovar von Drosophila mit der Nahrungsqualität variiert. Beim Ablageblock , auch „held egg“ genannt, hingegen resultiert die Reduktion der Eier aus einer Unfähigkeit der Weibchen, reife Oozyten abzusetzen. Da Phänotypen beider Kategorien regelmäßig auch in nicht injizierten Weibchen zu beobachten sind, gelten sie als unspezifisch. Die spezifischen Phänotypen lassen sich grundsätzlich in drei Klassen einstufen: (a) Die Morphologie des Ovars ist verändert (b) Reduktion der Oozytenzahl mit Defekten des Trophariums (c) Reduktion der Oozytenzahl ohne morphologischer Veränderung des Trophariums Obwohl die Ovariogenese, die Ausbildung des Ovars, mit den frühen Puppenstadien als abgeschlossen gilt (Trauner und Büning, 2007), führten einige dsRNAs zu deutlichen Veränderungen des Ovars. Oftmals war eine deutliche Reduzierung des Vitellariums (Abb. 11 B und G) bzw. das Fehlen ebendieses zu beobachten (Abb. 11 C), wobei die Oozyten, soweit vorhanden, in das Tropharium einwanderten (Abb. 11 C’). In seltenen Fällen kam es zu einer äußerlichen Umgestaltung des Trophariums, von der normalen ovalen Form hin zu einer schmalen, konischen (Abb. 11 E) oder rundlichen (Abb. 11 F). Defekte in der Morphologie, insbesondere im Tropharium, können zu einer Vielzahl von Phänotypen führen, beginnend mit verminderter Produktion von Oozyten, morphologischen Veränderungen der Oozyten oder sichtbaren Störungen in deren Aufreihung (Abb. 11 E-G). Ebenso vielfältig wie die Phänotypen sind die möglichen Ursachen: Apoptose oder eine Störung des Zellzyklus bzw. in der Spezialisierung der Zellen aus den Vorläuferzellen sind ebenso denkbar wie ein Nährstoffmangel. Die Betrachtung im Lichtmikroskop kann dabei nur einen ersten Eindruck vermitteln. Für die Wahl möglicher Kandidatengene zur Fragestellung Keimzell-Nische vs. somatische Stammzell-Nische sollten allerdings nur Phänotypen herangezogen werden, die der Klasse (c) entsprechen, d.h. neben einer Reduktion an Oozyten keinen weiteren morphologischen Defekt aufweisen (z.B. Abb. 11 D). 58 3. Ergebnisse Abbildung 11: Ovar-Defekte von pupal injizierten Tieren im Vergleich mit dem Wildtyp . Gezeigt sind Ovare (A-G) sowie Vergrößerungen einzelner Ovariolen aus meinem Teil des pupalen Screens von iBeetle (E’-G’). Aufgenommen mit dem Programm ProgRes. A Übersicht eines wildtypischen Ovars von Tribolium, bestehend aus zwei Halbovaren mit je 5-6 Ovariolen. A´ zeigt eine einzelne Ovariole mit Tropharium (Tr), das die Nährzellen enthält, und Vitellarium mit Oozyten in unterschiedlichen Entwicklungsstadien (V). B Phänotyp nach pupaler Injektion mit iB_00603 (Dmel\ebi). B’ Die Morphologie des Trophariums ist gestört. Reife Oozyten fehlen, jüngere sind teils fus ioniert (Pfeil). C Phänotyp nach pupaler Injektion mit iB_00647 (Dmel\Egfr). Das Ovar ist in der Größe reduziert. C´ Ovariolen sind deutlich verkürzt, da das Vitellarium fehlt. Oozyten drängen sich im Tropharium (Pfeil). D Phänotyp nach pupaler Injektion mit iB_00977 (kein Dmel-Ortholog). D’ Im Vitellarium akkumulieren junge Oozyten (Pfeil). E Phänotyp nach pupaler Injektion mit iB_01835 (Dmel\orb). E’ Die Form des Trophariums ist anormal, ebenso die Zellstruktur (Pfeil). Das Vitellarium ist verkürzt. Oozyten fehlen. F Phänotyp nach pupaler Injektion mit iB_03577 (Dmel\ l(2)35Be). F’ Das Tropharium ist verkürzt (Pfeil oben). Die Zahl der Oozyten ist reduziert, ihre Form abgerundet. Ebenso ist die Abfolge der Stadien gestört (Pfeil unten). G Phänotyp nach pupaler Injektion mit iB_03595 (Dmel\mirr). G’ Junge Oozyten liegen akkumuliert in einem verkürztem Vitellarium vor. 59 3. Ergebnisse 3.1.2 Defekte der larvalen Körpermuskulatur nach pupaler Injektion Die Muskulatur der Eumetazoa geht aus dem Mesoderm hervor und wird häufig nach Typus der Muskelfasern oder Funktion kategorisiert. In Insekten unterscheidet man drei verschiedene Arten der Muskulatur: zur willkürlich beeinflussbaren Muskulatur zählen die Muskeln des Skelettes, die die Beweglichkeit steuern. Neben dieser somatischen Muskulatur gibt es die unwillkürliche Muskulatur, die das Herz und Teile des Darms umspannt (Seidel et. al.,1940). Trotz struktureller Ähnlichkeiten im grundlegenden Aufbau unterscheiden sich die Muskeln der Skelettmuskulatur in Form, Größe und Reizleitung. So finden sich allein im abdominalen Segment (A1-A7) eines 15h alten Drosophila-Embryos an die 30 verschiedene Muskeln (Bate, 1993). Die Muskulatur in Tribolium gestaltet sich ähnlich komplex (Abb. 12 A und C). Neben einer Längsmuskulatur, die sich dorsal und ventral entlang der anterior-posterioren-Achse erstreckt, finden sich in den thorakalen und abdominalen Segmenten diverse querliegende Muskeln. In 20facher Vergrößerung lassen sich auch in den Beinsegmenten Muskeln erkennen (Abb. 12 C’). Obwohl in den letzten Jahrzehnten zahlreiche Erkenntnisse darüber gewonnen wurden, wie die Muskelzellen entstehen, sich zu Fasern vereinigen und auswachsen, bergen das komplexe Muster und die ihm zugrunde liegenden Prozesse noch viele Fragen, zu deren Klärung ein genomweiter Screen wie iBeetle einen wichtigen Beitrag leisten kann. Die GFP-Insertion, die als Enhancer-Trap im Stamm PBA19 in der somatischen Muskulatur exprimiert ist, liegt im 3'UTR eines der AKTIN Gene (Lorenzen et al., 2007). Bei Bestrahlung mit blauem Licht kommt es so zur Fluoreszenz der larvalen somatischen Muskulatur. Von den 3400 dsRNAs des ersten Teils des iBeetle Screens führten 4,4 % zu einem Defekt der Muskulatur, der als primär, also unabhängig von Segmentierungsdefekten eingeordnet werden kann. 55 der Injektionen (1,6 %) des larvalen Screens resultierten in einem spezifischen Phänotyp der pupalen Flugmuskulatur (detektiert mittels der Enhancertrap-Linie D17). Störungen der larvalen Muskulatur nach parentaler RNAi bewirkten 89 der getesteten dsRNAs (2,6 %) (Schmitt-Engel et al., in Vorbereitung). Diese Zahlen decken sich mit den Ergebnissen meiner pupalen RNAi. Dort konnte in 19 (2 %) der 987 Injektionen ein Muskelphänotyp festgestellt werden (Abb.12), wobei nur Penetranzen über 30 % in die Zählung eingingen. 60 3. Ergebnisse Darunter finden sich Phänotypen, die ein spezifisches Set von Muskeln betreffen, wie im Falle von iB_03580. Hier konnte eine Störung der dorsalen Längsmuskulatur des Prothorax (T1) festgestellt werden. Während hier die Muskeln als kugelförmige Gebilde vorliegen, wahrscheinlich aufgrund einer Störung der Anheftung der Muskeln an die spezifischen Sehnen-Zellen (tendon cells), scheint die restliche Muskulatur intakt (Abb. 12 B und B’). Im Falle des Gens iB_03525 (Dm/baboon) führt die parentale RNAi zu einer sichtbaren Verschmälerung der Skelettmuskulatur – besonders gut erkennbar an der schrägen Muskulatur der abdominalen Segmente und den Beinmuskeln (Abb.12 D und D’). Grund dafür könnte eine Störung der Zellproliferation. So führen Mutationen dieses Gens in Drosophila im Puppenstadium zum Tod, da die Proliferation in den Imaginalscheiben und im Hirngewebe reduziert ist, so dass adulte Strukturen nicht ausgebildet werden können. Parentale RNAi gegen iB_03821 führt zu Larven, deren gesamte Muskulatur zu fehlen scheint – abgesehen von einem Muskel zwischen Kopf und Prothorax (Abb. 12 E). Dies deutet auf eine frühe bzw. essentielle Funktion dieses Gens hin. Erste Vergleiche der Sequenz zeigen Ähnlichkeiten zu dem FGF-Rezeptor 1 (Drosophila). In Drosophila resultieren Mutationen dieses Rezeptors in zahlreichen MuskulaturDefekten, darunter dem Verlust des Herzens, da sich die Herz-Vorläuferzellen, die sich normalerweise in Phase 11 der embryonalen Entwicklung vom Mesoderm abspalten, nicht ausbilden (Beiman et al., 1996). Bei der dsRNA iB_01726 waren einzelne Muskelfasern falsch ausgerichtet bzw. verkürzt (Abb. 12 F). Ebenfalls zu beobachten, ist, dass einzelne Muskelfasern dünner sind, fehlen oder kugelförmig vorliegen, was auf Entwicklungsdefekte bzw. auf eine Störung des Anheftungsprozesses hinweist. Gene, die einzig an der Musterbildung der Beinmuskulatur beteiligt sind, konnten überraschenderweise nicht identifiziert werden. Im Gesamtscreen wurde zwar eine Handvoll Phänotypen annotiert, weitere Analysen konnten jedoch nur einen bestätigen, wobei auch dieses Ergebnis mittlerweile in Frage gestellt wird (Schmitt-Engel et al., in Vorbereitung; interne Kommunikation). 61 3. Ergebnisse Abbildung 12: Primäre Muskelphänotypen von L1-Larven nach parentaler RNAi A-F Dargestellt sind Fluoreszenzaufnahmen der L1- Larven von pBA19 nach parentaler RNAi aus meinem Teil des pupalen Screens von iBeetle im Vergleich zum Wildtyp. Aufgenommen mit ProgRes. Anterior befindet sich stets links. A Dorsale Ansicht einer Wildtyp-Larve. B Dorsale Ansicht des Phänotyps nach pupaler Injektion mit iB_03580 (Dmel\CG42269). B’ zeigt eine Vergrößerung von Kopfes und Prothorax (T1). Die dorsalen Längsmuskeln in T1 sind als kuge lförmige Strukturen zu erkennen. C Laterale Ansicht einer Wildtyp-Larve. C’ zeigt die laterale Ansicht eines Beins. D, D’ Laterale Ansicht des Phänotyps nach pupaler Injektion mit iB_03525 (Dmel\baboon). Die Muskeln in Körper und Bein (D’) sind deutlich dünner ausgeprägt. E Laterale Ansicht des Phänotyps nach pupaler Injektion mit iB_03821 (Dmel\heartless). Nur im ersten thorakalen Segment (T1) ist Muskulatur ausgebildet. Der Rest fehlt. F Laterale und ventrale (F’) Ansicht des Phänotyps nach pupaler Injektion mit iB_01726 (Dmel\11526). Einzelne Muskeln fehlen, sind dünner, fehlorientiert bezüglich ihrer Bindungsstellen oder in sich zusammengefallen. 62 3. Ergebnisse 3.1.2 Defekte der Embryogenese nach parentaler RNAi Neben der Identifizierung von Genen, die an der somatischen Stammzell-Nische des Ovars und der larvalen (Bein-) Muskulatur beteiligt sind, war und ist die embryonale Musterbildung ein zentraler Schwerpunkt des Screens. Von den frühen Prozessen der Achsen-Determination, über die komplizierten Prozesse in der „segment addition zone“ bis hin zur Kopf- und Beinentwicklung – in vielen Bereichen sind nach wie vor Fragen offen bzw. die beteiligten Gene weitgehend ungeklärt (siehe Vorstellung der Einzelprojekte unter 1.2.1). Die Morphologie der Larve mit ihren Segmenten ermöglicht hierbei eine leichte Analyse der Defekte. Mit ~21% (206 der von mir pupal injizierten dsRNAs) stellen die Segmentierungsphänotypen (verändertes larvales Kutikula-Muster) die dominante Gruppe des Screens. Im gesamten Screen hingegen führten ~ 28% (968 dSRNAs) aller pupalen Injektionen zur embryonalen Letalität (Cutoff > 80 %). Davon zeigten ~ 30% lokale oder ubiquitäre Segmentierungsdefekte, während der Rest wildtypische L1-Larven hervorbrachte oder erst gar keine Kutikulas ausbildete (Schmitt-Engel et al., in Vorbereitung). Neben lokal begrenzten und hochspezifischen Defekten resultiert parentale RNAi oftmals in globalen ("pantagmatischen", also alle Körperregionen übergreifenden) Defekten. Superposition mehrerer Phänotypen, die auf Pleiotropie des beteiligten Gens hindeuten, sind ebenfalls keine Seltenheit. Ein Beispiel dafür ist iB_03581. Die parentale RNAi führt zu einem Verlust aller Körperborsten (Abb. 13 B) und einer Reihe weiterer Defekte wie u.a. der Verlängerung der Beinsegmente Coxa (C), Femur (Fe) und Tibiotarsus (Tt) sowie der Reduktion der distalen Kralle (Abb. 13 B’). Weiterhin sind die Proportionen der Kopfanhängsel gestört (Abb. 13 B’’), und die Larven zeigen Defekte in der embryonalen Muskulatur (nicht gezeigt). iB_01702 ist ein perfekter Kandidat für einen spezifischen Faktor in der Entwicklung des embryonalen Kopfes (Abb. 13 C). Die Segmentierung der Larven ist wildtypisch, jedoch findet sich bei näherer Betrachtung der Kopfborsten ein Phänotyp im Gena-Triplet (Abb.13 A’’, blau). Das distinkte Dreieck, das sich lateral am Kopf befindet (Schinko et al., 2008) ist in dsRNA-Injektionen von iB_01702 durch eine Gruppe Borsten ohne ersichtliches Muster ersetzt (Abb. 13 C’’, rot umrandet), was auf eine Störung des interkalaren Segments hindeutet – ein Segment, das neben der Entwicklung des Kopfes auch mit der Gehirnentwicklung in Verbindung gebracht 63 3. Ergebnisse wird (Posnien und Bucher, 2009; Posnien et al., 2011). Dieser Hinweis auf eine Störung der Gehirnentwicklung und eine damit einhergehende frühe Letali- tät/Lebensunfähigkeit der Tiere könnte auch erklären, weshalb keines der Tiere aus seinem Ei geschlüpft ist. Weiterhin fehlte in der Muskelpräparation jegliche Fluoreszenz (nicht gezeigt), was auf eine separate Funktion in der Muskelentwicklung, wahrscheinlicher aber einfach auf einen frühen Tod der Tiere hinweist. Ein Beispiel eines spezifischen Beindefektes ist der Phänotyp von iB_03829. Die parentale RNAi führte hier zu einer Verkürzung des larvalen Beins, wobei nicht auszumachen ist, ob der Femur fehlt oder aber mit dem Tibiotarsus zu einem Segment verschmolzen ist (Abb. 13 D und D’). iB_00992 zeigt mit der Transformation der letzten beiden abdominalen Segmente A9 und A10 ein typisches Beispiel für eine Störung der Segmentidentität. Im Wildtyp fusionieren diese beiden Segmente zu einer terminalen Struktur. Dorsal entstehen dabei die Urogomphi aus A9 und ventral die von A10 abgeleiteten Pygopodien (Sokoloff, 1974). Nach Injektion mit der dsRNA zu iB_00992 bleibt diese Fusion aus. A9 formt ein normales abdominales Segment, A1-A8 ähnlich, während A10 reduzierte Pygopodien anlegt (Abb. 13 E). Ein vielversprechendes Kandidatengen zur Erforschung der dorso-ventralen Achse scheint iB_00641. Eine solch invertierte Larve, deutlich erkennbar an den innen liegenden Beinen und Borsten (Abb. 13 F Asteriske), wurde 2005 von Van der Zee et al. beobachtet und beruht auf einer Störung des dorsalen Rückenschlusses. Im Wildtyp führen morphogenetische Bewegungen der extraembryonalen Membranen dazu, dass die lateralen Seiten des Embryos Richtung dorsal gezogen werden, bis sie entlang der dorsalen Mittellinie fusionieren. Sind die morphogenetischen Bewegungen von Amnion und Serosa gestört, kommt es häufig zu einem "ventralen Rückenschluss" des Embryos und der Internalisierung apikaler Strukturen. Für mein Projekt potentiell interessant sind Phänotypen, bei denen sich nur noch Fragmente larvaler Strukturen finden, seien es Bruchstücke oder identifizierbare Segmente, die aus Strukturen nahe der Ei-Pole erwachsen (Abb. 13 G und H). Solche Phänotypen, ebenso wie eine Anhäufung leerer Eier, können Hinweise sein auf Gene, die an der Etablierung der Achsen oder an der frühen Segmentierung beteiligt sind (Kotkamp et al., 2010; Fu et al., 2013). Allein in meinen 987 dsRNAInjektionen führten 8 % (Abb. 10) zu Eigelegen, die > 80 % leere Eier enthielten; im 64 3. Ergebnisse Gesamtscreen waren es 14,8 % (Schmitt-Engel et al., in Vorbereitung). Die Analyse dieser „empty egg”-Phänotypen sollte in einem gesonderten Projekt getätigt werden. 65 3. Ergebnisse Abbildung 13: Beispiele für Defekte der Embryonalentwicklung nach pupaler RNAi Autofluoreszenz-Aufnahmen der Kutikula-Präparate von L1-Larven nach parentaler RNAi (B-H) im Vergleich zum wildtypischen Phänotyp (A-A’’). Aufgenommen mit ProgRes. Anterior ist stets links und dorsal oben. A Laterale Ansicht einer Wildtyp-L1-Larve. A’ und A’’ Laterale Ansicht des wildtypischen Beins bzw. Kopfes mit Borstenmuster (farbige Dreiecke). T1 markiert das erste thorakale Segment, A1 das erste abdominale. U: Urogomphi (aus A9), P: Pygopodien (aus A10), C: Coxa, Tch: Trochanter, Fe: Femur, Tt: Tibiotarsus. B Phänotyp nach Injektion der dsRNA zu iB_03581 (Dm/shaven). Alle Borsten entlang des Körpers fehlen. Beine (B’) und Kopfanhängsel (B’’) sind verlängert. C Phänotyp nach Injektion der dsRNA zu iB_01702 (Dmel\cortex). Das Borstenmuster des Kopfes ist gestört (C’’ rot umrandeter Bereich). D Phänotyp nach Injektion der dsRNA zu iB_03829 (Dmel\Tret1-1). Die Beine sind verkürzt. E Phänotyp nach Injektion der dsRNA zu iB_00992 (Dmel\Br140). Die Larve hat 9 statt 8 abdominale Segmente, und die Urogomphi fehlen. F Phänotyp nach Injektion der dsRNA zu iB_00641 (Dmel\PDZ-GEF). Die Larve ist nach innen gestülpt. Die Asteriske markieren invertierte Beine. G Phänotyp nach Injektion der dsRNA zu iB_01822 (Dmel\tok). Es sind nur lose Kutikula-Fragmente vorhanden. H Phänotyp nach Injektion der dsRNA zu iB_03546 (kein Dmel-Ortholog). Nur das posteriore Abdomen ist teilweise ausgebildet. Die Pygopodien fehlen. 66 3. Ergebnisse 3.2 Vom Screen zum Kandidaten-Gen Mit Hilfe der in die Datenbank implementierten Suchfunktion wurde nach Abschluss der ersten Screening-Phase (05.12.2011) nach vielversprechenden Kandidaten-Genen für die Bildung der anterior-posterioren-Achse gesucht. Die Suchkriterien wählte ich dabei wenig streng, da die Redundanz in der Annotierungsterminologie dazu führen kann, dass interessante Kandidaten nicht gefunden werden; Larven, denen z. B. der Kopf fehlt, konnten sowohl mit „anteriore Strukturen - nicht vorhanden“ als auch „Kopf - nicht vorhanden“ oder „nur Thorax und Abdomen - vorhanden“ annotiert werden. Daher suchte ich zunächst nach iB-Nummern, bei denen an Tag 13 keine oder nur wenige Larven geschlüpft waren, da dies auf einen Defekt der Embryogenese hindeutet (1260 Ergebnisse). Von diesen schloss ich im Anschluss diejenigen aus, für die an Tag 14 und 15 leere bzw. unbefruchtete Eiern mit einer Penetranz > 80 % annotiert worden war. Zwar können sich in der Klasse der „empty eggs“ starke Defekte von Segmentierungsgenen verbergen (Kotkamp et al., 2010; Fu et al., 2013). Eine spezifische Analyse dieser einzelnen Prozesse war im engen zeitlichen Rahmen von iBeetle allerdings nicht möglich. Zudem war von Anfang an zusätzlich zu iBeetle ein „empty egg“-Screen geplant, bei dem entsprechende iBeetle dsRNAs noch einmal mit Hilfe einer Linie untersucht werden, die im Kern GFP exprimiert (Sarazin et al., 2012). Nach Abzug der „empty egg“-Phänotypen blieben 1006 iB-Nummern. Zu diesen addierte ich dann die Gene (falls noch nicht enthalten), die von den Screenern als besonders interessant für „Segmentierung“ bzw. „anteriore-posteriore Achsenentwicklung“ markiert worden waren. Im Anschluss erfolgte dann die Analyse "per Hand", d.h. ich ging jede dieser Annotierungen einzeln durch und schaute nach Penetranz, Stärke und Spezifität des Phänotyps, wobei ich besonderen Fokus auf Gene legte, die Defekte in Strukturen aufwiesen, die von den beiden Polen der Oozyte entspringen. Im Zuge dieser Suche wählte ich ebenfalls mögliche Kandidaten für das Projekt von Reinhard Schröder (Rostock), der Interesse an Wachstumsprozessen und damit an Phänotypen mit abdominalen und Bein-Defekten hat (siehe 1.2.1). Nach diesen Kriterien konnte die Liste auf 258 Gene reduziert werden (Abb.14). Die 15 aussichtsreichsten Gene von diesen (ein weiteres kam im Verlauf des Screens hinzu) wurden zur Verifizierung des Phänotyps im Zuge eines Rescreens in unter- 67 3. Ergebnisse schiedlicher Konzentration (1 µg/µl und 3 µg/µl) noch einmal in PBA19 sowie den Stamm San Bernadino (SB) injiziert. Letzteres war notwendig, da u. a. in Drosophila, Mus musculus oder auch für die Hefe Saccharomyces cerevisiae nachgewiesen wurde, dass Phänotypen von Mutanten mit dem genetischen Hintergrund des verwendeten Stammes variieren können (Threadgill et al., 1995; Dworkin et al., 2009; Dowell et al., 2010). Die anschließende Injektion der dsRNA eines nicht-überlappenden Fragments (NoF) in den Stamm SB diente hingegen der Verifizierung der Genidentität, also als Kontrolle gegen "off-target"-Effekte (Abb.15). Von den 16 Genen konnte ich 14 mit dem Originalfragment in PBA19 bestätigen (zwei waren also falsch-positive). Zwei weitere ließen sich in SB nicht reproduzieren. Diese Ergebnisse decken sich mit den Ergebnissen der anderen Arbeitsgruppen. Von den 102 Genen, die in PBA19 noch einmal injiziert wurden, zeigten nur 83 % den im Screen aufgetretenen Phänotyp. Von 210 dsRNAs, die in San Bernadino injiziert worden sind, zeigten 11 % einen signifikanten Effekt bezüglich des genetischen Hintergrunds (Schmitt-Engel et al., in Vorbereitung. Kitzmann et al., 2013). Acht weitere Phänotypen wurden nach Injektion der NoFs ausgesondert, da entweder wildtypische Larven auftraten oder eine vermehrte Zahl leerer Eihüllen. Am Ende blieben daher nur vier Gene übrig: Tc-germ cell-less (iB_02693), Tchomeobrain (iB_04564) sowie die Tribolium-Homologe von tout-velu (iB_03835) und RhoGef2 (iB_03492). Für die vertiefende Analyse entschied ich mich für germ cell-less und homeobrain, da die spezifischen Phänotypen – Doppelabdomen mit gespiegelter Symmetrie – auf eine Funktion beider Gene in der terminalen Differenzierung hindeuteten. Komponenten dieses Prozesses waren bisher für Tribolium noch nicht bekannt. Ebenfalls kurz im Fokus der Analyse waren iB_00736 (staufen) und das Gen teashirt. RNAi gegen iB_00736 führte zum Verlust aller Segmente der „segment addition zone“, während RNAi gegen die Positiv-Kontrolle von teashirt ebenfalls zu Doppelabdomen führte. Dies allerdings in geringer Penetranz (< 30 %). Beide Phänotypen konnten auch mit 3 nicht-überlappenden Fragmenten nicht verifiziert werden (nicht gezeigt), weshalb ich die Arbeit an diesen Genen einstellte. Eine ausführliche Liste der untersuchten Gene des Rescreens sowie deren Phänotypen findet sich in Anhang D. Daten zu staufen und teashirt in Anhang E und F. 68 3. Ergebnisse Abbildung 14: Vom Screen zum Kandidatengen Dargestellt sind die Pools, die zur Identifizierung aussichtsreicher Gene herangezogen wurden. 176 von Screenern markierte Gene sowie 1006 aus der Datenbank. Mit Hilfe verschiedener Ausschlusskriterien (Penetranz, Art des Phänotyps etc.) war es möglich die Zahl auf 258 Gene mit möglicher Funktion in der Segmentierung des Tribolium Embryos zu reduzieren. Gene im Rescreen bestätigt mit Originalfragment/ gleicher Stamm bestätigt mit Originalfragment/ anderer Stamm bestätigt mit NoF 0 5 10 Anzahl der Gene 15 20 Abbildung 15: Zusammenfassung der Phänotyp-Verifizierung (Rescreen) Von 16 Kandidatengenen konnten bei erneuter Injektion des Originalfragments in PBA19 nur 14 verifiziert werden. Die Injektion in einen anderen Stamm (San Bernadino) führte zu einem gemischten Ergebnis. Im Falle des Originalfragments konnten 12 Phänotyp en bestätigt werden, im Falle des nichtüberlappenden Fragments (NoF) lediglich 4. 69 3. Ergebnisse 3.3 Analyse der Funktionen von germ cell-less und homeobrain 3.3.1 Parentale RNAi von germ cell-less und homeobrain induziert posteriore Strukturen am anterioren Pol Defekte nach pRNAi gegen germ cell-less (iB_02693) Zur Generierung von gcl-spezifischer RNAi-Phänotypen wurde das gleiche dsRNAFragment von 488 Bp verwendet wie im Screen (vgl. 3.3.3). Die Reduktion von gclmRNA durch parentale RNAi resultiert in Larven, denen sowohl Kopf als auch Thorax fehlen. Stattdessen ist anterior ein zweites Abdomen ausgebildet. Dieses Doppelabdomen liegt entweder gestreckt (Abb. 16 D) oder „kugelförmig“ vor, d.h. beide Abdomen sind dorsal verkürzt und fusioniert (Abb. 16 E). In beiden Fällen ist anhand der Morphologie der abdominalen Segmente (Orientierung der abdominalen Borsten und Position der Tracheenöffnungen) eine Spiegel-Symmetrie erkennbar. Jedes der beiden Abdomen besitzt durchschnittlich 5-7 Segmente und einen Terminus, wobei insbesondere bei den gestreckten Doppelabdomen eines der beiden Abdomen oftmals vollständiger ausgebildet ist als das andere. Bei den kugelförmigen Doppelabdomina sind die Segmente hingegen teilweise fusioniert, die Urogomphi sind aber stets gut zu erkennen (Abb. 16 E). Dorsal ist zudem in vielen Fällen eine Öffnung in der Kutikula vorhanden, welche mittig beide Abdomen überspannt bzw. wie in Abb. 16 F dargestellt - zwischen den beiden Urogomphi lokalisiert ist. Mit Zunahme des Alters der injizierten Weibchen und damit mit Abnahme des RNAi-Effekts steigt die Zahl der vorhandenen Segmente. Mitunter sind zwei vollständige Abdomen (A1-A8 und Terminus) sowie thorakale Segmenten vorhanden, wobei die Duplikation des Thorax immer unvollständig bleibt. So sind lediglich T2 und T3 ausgebildet. Die beiden T2-Segmente liegen oft fusioniert vor (Abb. 16 C). Im den schwächsten gcl-Phänotypen ist die Polarität der Larve zwar wieder hergestellt, allerdings ist der Kopf noch von abdominalen Segmentresten und, seltener, Beinstrukturen durchsetzt (Abb. 16 B). RNAi gegen gcl ist hoch penetrant. Bereits bei einer Injektion von 0,1 µg/µl dsRNA zeigen 18 % der Embryonen Doppelabdomen, bei 1 µg/µl mehr als 90 %. Die Kopfphänotypen sowie wildtypische Larven finden sich lediglich bei 0,4 %, und auch Doppelabdomen mit thorakalen Segmenten treten nur sehr selten auf (Abb. 17). 70 3. Ergebnisse Abbildung 16: Phänotypische Serie der Defekte nach pRNAi gegen gcl Autofluoreszenz-Aufnahmen der Kutikula-Präparate von L1-Larven nach parentaler RNAi gegen gcl (iB_02693) vom schwächsten zum stärksten Phänotyp (B–E, F) im Vergleich zum Wildtyp (A) ApoTome (AxioVision). Anterior ist, soweit erkennbar, stets links und dorsal oben. Die blaue Linie markiert die Symmetrieebene. A Laterale Ansicht einer Wildtyp-L1-Larve mit Kopf, Thorax und Abdomen. T1 markiert das erste thorakale Segment, A“n“ das jeweilige abdominale Segment. U: Urogomphi (aus A9), P: Pygopodien (aus A10) B Schwächster Phänotyp. Die Segmentierung des Kopfes ist gestört. Nur wenige Segmente sind erkennbar (*). C Schwacher Phänotyp. Die Larve besteht aus zwei Abdomen in gespiegelter Symmetrie. T3 und T2 sind ebenfalls dupliziert. Beide Abdomen weisen 8 Segmente und einen Terminus (Pfeilspitze) auf. D Mittlerer Phänotyp. Zwei unvollständigen Abdomen in gespiegelter Symmetrie. Das anteriore A bdomen weist eine unregelmäßige Segmentierung auf. Der Terminus ist an beiden Abdomen vorhand en (Pfeilspitze) E Starker Phänotyp. Kugelförmige Kutikula aus zwei unvollständigen Abdomen. Die Segmentierung ist unregelmäßig. Zwei Paar Urogomphi sind deutlich erkennbar (Pfeilspitze). F Besonderheit des starken Phänotyps. Das Doppelabdomen weist dorsal eine deutliche Öffnung in der Kutikula auf. Die Segmentierung der Abdomen ist gestört. Die Urogomphi sind an beiden Abd omen vorhanden (Pfeilspitze). 71 3. Ergebnisse 100 Doppelabdomen 90 Häufigkeit in % 80 Thorax + Doppelabdomen 70 Kopf/Thorax + ant. Abdomen fusioniert 60 50 leere Eihüllen 40 30 Andere 20 Wildtyp 10 0 0,1 0,2 0,3 0,4 0,5 1 3 Konzentration in µg/µl WT geschlüpfte Larven Abbildung 17: Phänotypen nach gcl-RNAi in Abhängigkeit von der Konzentration Dargestellt ist die prozentuale Verteilung der larvalen Phänotypen nach pRNAi von gcl in Abhängigkeit von der Konzentration der dsRNA. Dunkelrot zeigt die geschlüpften Larven, rot die wildtyp ischen Larven auf dem Objektträger, orange RNAi-unabhängigen Hintergrund bzw. nicht auswertbare Phänotypen, gelb früh letale/unbefruchtete Eier und blau die verschiedenen gcl-Phänotypen. Letztere unterteilen sich in drei Klassen: Kopf/Thorax fusioniert mit dem neuen anterioren Abdomen (hellblau), gespiegelte Doppelabdomen mit (blau) und ohne thorakalen Segmenten (dunkelblau).N pro Konzentration = 83 (0,1), 50 (0,2), 90 (0,3), 78 (0,4), 191 (0,5), 205 (1) und 525 (3) bzw. 317 (WT). Defekte nach pRNAi gegen homeobrain (iB_04564) Auch für den „knock-down“ von hbn wurde das iBeetle-dsRNA-Fragment von 388 bp verwendet (vgl. 3.3.3). Im Gegensatz zu gcl führt die pRNAi von hbn immer (auch bei hohen dsRNA-Konzentrationen) zu einem Spektrum von Phänotypen (Abb. 19). Neben einem hohen Anteil leerer Eier, kommt es bei den meisten Larven zum Verlust von Labrum und Antenne (Abb. 18 B). Mit Zunahme des RNAi-Effekts fehlen komplette anteriore Strukturen, d. h. Kopf, Pro- und seltener Mesothroax. Manchmal sind fusionierte Reste vorhanden (Abb. 18 C). Des Weiteren finden sich Larven mit zusätzlichen thorakalen Segmenten, die symmetrisch zu den „normalen" Beinen angeordnet sind. Die Orientierung der Segmente ist hierbei deutlich anhand der Ausrichtung der Borsten sowie der Öffnung der Tracheen am Mesothorax erkennbar (Abb. 18 D). Die Symmetrieachse kann in T1 liegen, oft ist aber nur T3 in duplizierter Ausführung vorhanden. In 14 % der Fälle schließen sich an den Thorax anterior noch deutlich erkennbare abdominale Segmente an. Die Zahl dieser Segmente schwankt zwischen 2-5, wobei die Segmentierung meist unregelmäßig ist. Terminale Strukturen fehlen am anterioren Abdomen in allen Fällen komplett (Abb. 18 E). 72 3. Ergebnisse Abbildung 18: Phänotypische Serie der Defekte nach pRNAi gegen hbn Autofluoreszenz-Aufnahmen der Kutikula-Präparate von L1-Larven nach parentaler RNAi gegen hbn (iB_04564) vom schwächsten zum stärksten Phänotyp (B–E) im Vergleich zum Wildtyp (A). ApoTome (AxioVision). Anterior befindet sich links und dorsal oben. Die blaue Linie markiert die Symmetrieebene. A Laterale Ansicht einer Wildtyp-L1-Larve mit Kopf, Thorax und Abdomen. T1 markiert das erste thorakale Segment, A“n“ das jeweilige abdominale Segment. U: Urogomphi (aus A9), P: Pygopodien (aus A10). B Schwächster Phänotyp. Abdomen und Thorax sind normal ausgeprägt. Der anteriore Kopf (Labrum und Antennen) fehlt (Asterisk). C Schwacher Phänotyp. Der Kopf ist mit dem Thorax fusioniert. Der Asterisk markiert ein Kopfanhängsel, der Pfeil das distale Ende eines Beines mit partiell fusionierten Podomeren. Nur noch T3 ist komplett ausgebildet. Das Abdomen hingegen ist normal. D Mittlerer Phänotyp. Thorax und Abdomen sind normal ausgeprägt. Anterior schließen sich an T1 zwei weitere thorakale und manchmal ein-zwei abdominale Segmente an. Die zusätzlichen Segmente liegen in gespiegelter Symmetrie vor, erkennbar an den Borsten und der trachealen Öffnung an T2 (Pfeilspitze). E Starker Phänotyp. Zwei fusionierte thorakale Segmente in gespiegelter Symmetrie erkennbar. Daran schließen sich zwei Abdomen an, ein vollständiges mit 8 Segmenten und Terminus sowie anterior ein unvollständiges aus maximal 5-6 Segmenten ohne Terminus, dessen Segmentierung unregelmäßig ist. 73 3. Ergebnisse 100 90 Thorax + unvollst. Doppelabdomen Haäifigkeit in % 80 Doppelthorax 70 ant. Strukturen fehlen 60 50 anteriorer Kopf fehlt 40 leere Eihüllen 30 Andere 20 Wildtyp 10 0 geschlüpfte Larven 1 3 Konzentration in µg/µl WT Abbildung 19: Phänotypen nach hbn-RNAi in Abhängigkeit von der Konzentration Dargestellt ist die prozentuale Verteilung der larvalen Phänotypen nach pRNAi von hbn in Abhängigkeit von der Konzentrationen der verwendeten dsRNA. Dunkelrot zeigt die geschlüpften Larven, rot die wildtypischen Larven auf dem Objektträger, orange RNAi-unabhängigen Hintergrund bzw. aufgrund der Präparation nicht auswertbare Phänotypen, gelb früh letale/unbefruchtete Eier und grün die verschiedenen hbn-Phänotypen. Letztere unterteilen sich in vier Gruppen: im schwachen Phänotyp fehlen die Strukturen des anterioren Kopfes (Labrum und Antennen; mintgrün); der intermediäre Phänotyp, dem anteriore Strukturen (Kopf und teilweise Thorax) fehlen bzw. fusioniert sind (hellgrün); die stärkeren Phänotypen mit zusätzlichen thorakalen Segmenten in gespiegelter Sy mmetrie bzw. bis zu 5-6 weiteren abdominalen Segmenten (dunkelgrün).. N pro Konzentration = 65 (1) und 116 (3) bzw. 317 (WT). 74 3. Ergebnisse 3.3.2 Tc-gcl wird maternal, Tc-hbn aber zygotisch exprimiert Die Expression von Tc-gcl gcl-mRNA ist in Tribolium bereits während der Oogenese im proximalen Tropharium in den Nährzellen und in reifenden Oozyten exprimiert, wobei die mRNA im Verlauf der Oogenese v.a. in der anterioren Hälfte der Oozyte lokalisiert wird (Abb. 20 A). Im frisch abgelegten Embryo, sowie in unbefruchteten Eiern, ist gcl anterior lokalisiert, zudem ist die mRNA um den Polkörper herum konzentriert (Abb. 20 B). Diese Lokalisierung am Polkörper ist unspezifisch und kann auch für andere maternale RNAs nachgewiesen werden, darunter z.B. eagle und pangolin (Bucher et al., 2005; Peel & Averof, 2010). In den folgenden Kernteilungen schwindet die Expression rasch und konnte, trotz intensiver Bemühungen und unter Verwendungen unterschiedlicher Sequenzen (iB-Fragment und NoF) sowie unterschiedlicher PCRs des iB- Fragments für die Sondensynthese und Variationen des Protokolls, in späteren Stadien nicht mehr eindeutig nachgewiesen werden. Späte Färbungen in Keimrudiment und Keimstreif (nicht gezeigt) dürfte auf unspezifische Hintergrundfärbung zurückzuführen sein, wobei eine schwache ubiquitäre Expression allerdings nicht ausgeschlossen werden kann. Tr 75 3. Ergebnisse Abbildung 20: Expression von Tribolium-germ cell-less A-B Nachweis von germ cell-less-mRNA durch in-situ-Hybridisierung aufgenommen und verarbeitet mit dem Programm Helicon Focus . B` Nukleäre Färbung mit Hoechst 33342 des gleichen Embryos. Aufgenommen mit Helicon Focus.In allen Abbildungen ist anterior links. A Dargestellt ist eine einzelne Ovariole. Das gcl-Transkript ist im proximalen Teil des Trophariums (Tr) in den Nährzellen sowie den reifenden Oozyten, zuerst ubiquitär, dann anterior nachweisbar, fehlt jedoch in jungen Stadien. B Im frisch gelegten Ei ist gcl-mRNA ebenfalls anterior, sowie um den Polkörper (*) lokalisiert. Die Expression von Tc-hbn Im Gegensatz zu gcl ist hbn nicht maternal exprimiert (Abb. 21 A), sondern wird erst im frühen undifferenzierten Blastoderm am anterioren Pol exprimiert (Abb. 21 B). Im Zuge weniger Zellteilungen expandiert die Expressionsdomäne zu einer asymetrischen Kappe (Abb. 21 C) – der Expression von zen-1 nicht unähnlich (van der Zee et al., 2005). Mit Differenzierung des Blastoderms, nach Abschluss des 13. Mitosezyklus (Handel et al., 2000), konzentriert sich die Expression von hbn in einem schmalen Streifen entlang der Grenze zwischen Serosa und Embryonalanlage (Abb. 21 D). Im Keimrudiment sowie während der Elongation des Keimstreifens ist hbn-mRNA in verschiedenen Regionen des anterioren Kopfes (Abb. 21 E-H) und der „segment addition zone“ (Abb. 21 E–F) exprimiert. Nach Abschluss der Elongation, im ausgewachsenen Keimstreif, findet sich hbn-Expression als kleine lokal begrenzte Domäne in beiden Kopflappen (Abb. 21 G). Die schwache Färbungen in Thorax und Abdomen (Abb. 21 F und G) dürfte erneut auf unspezifische Hintergrundfärbung zurückzuführen sein. Abbildung 21: Expression von Tribolium-homeobrain A - G Nachweis von homeobrain-mRNA durch in-Situ-Hybridisierung, aufgenommen und verarbeitet mit dem Programm Helicon Focus . A` - E` Nukleare Färbung der Embryos mit Hoechst 33342. Aufgenommen mit Helicon Focus. In allen Abbildungen ist anterior links. A Im frisch abgelegten Ei ist kein hbn-Transkript nachweisbar. A` * markiert den Oozytenkern. B - C zeigt die anteriore Expression und Expansion von hbn in den verschiedenen Blastodermstadien. D Im differenzierten Blastoderm ist hbn als Streifen entlang der Grenze zwischen Serosa und Embryonalanlage. E-F Mit Bildung des Keimrudiments und während der Elongation des Keimstreifens ist hbn-mRNA in spezifischen Regionen (Pfeilköpfe) des anterioren Kopfes sowie in der „segment addition zone“ exprimiert. G Der ausgewachsene Keimstreif weist zwei distinkte Expressionspunkte in den Kopflappen auf. 76 3. Ergebnisse 77 3. Ergebnisse 3.3.3 Sequenzanalyse von gcl und hbn offenbart konservierte Bindedomänen Tribolium germ cell-less Zu Beginn des iBeetle-Projekts wurden neue RNA-Sequenzierungen vorgenommen bzw. Transkriptom-Daten aus der Tribolium-Forschergemeinschaft gesammelt, welche Prof. Stanke (Greifswald) mithilfe verschiedener Varianten seines AUGUSTUSAlgorithmus interpretierte und in einem Genom-Browser zugänglich machte. Neueste bioinformatische Verfahren erlauben dabei die differenzierte Analyse dieser Daten und ein Vergleich verschiedener vorhergesagter Genmodelle. Ausgehend von dieser Vorhersage und den Informationen des National Center for Biotechnology Information (NCBI) liegt das Gen germ cell-less (Tc001571) auf der dritten Komplementationsgruppe und setzt sich aus sieben Exons zusammen. Nach dem Splicing der Introns kodieren 1449 Basenpaare (Bp) für ein 55,64 kD umfassendes Protein, das 482 Aminosäuren enthält (Protein-Molekulargewichts-Rechner von P. Stothart, 2000). Für den Screen und alle weiteren Analysen wurde ein iBeetleFragment von 488 Bp verwendet, welches 22 Bp von Exon 5 sowie die gesamten Exons 6 und 7 beinhaltet (iB_02693). Die Überprüfung der Genspezifität der RNAi erfolgte mit Hilfe zweier nicht-überlappender Fragmente: iB_02693_2 (3’Ende des Exons 5 & Exon 6) mit 196 Bp und iB_02693_3 (Exon 7) mit 217 Bp (Abb. 22). Wie Mm-GCL weist auch Tribolium-GCL (Tc-GCL) eine BTB/POZ-Domäne auf (Leatherman et al., 2000), welche in Tribolium zwischen der 56. Aminosäure (AS) und der 165. AS lokalisiert ist. Der Sequenzvergleich der Amisnosäuren dieser BTB/POZ-Domäne in unterschiedlichen Spezies mit Hilfe des Programms MacVector (Version 12.5.1) offenbart eine Sequenzidentität der Aminosäuren von ≈ 50 % zwischen Tribolium und Drosophila melanogaster bzw. Mus musculus oder Homo sapiens (Abb. 23). Das gesamte Protein ist jedoch mit 32-38% Sequenzidentität speziesübergreifend relativ gering konserviert (Tabelle 2, MacVector). 78 3. Ergebnisse Abbildung 22: Position der für die gcl-RNAi verwendeten dsRNA-Frag mente Dargestellt ist das Gen germ cell-less sowie die im Screen und in den nachfolgenden Versuchen verwendeten iB-Fragmente. Dunkelgrün zeigt das Gen gcl (Tc001571), hellgrün die Exons, gelb das Originalfragment aus dem Screen (iB_02693) und der Folgeanalysen und khakifarben sind die beiden nicht-überlappenden Fragmente (NoFs) (http://bioinf.uni-greifswald.de/gb2/gbrowse/tribolium/; M. Stanke). Abbildung 23: Sequenzvergleich der BTB/POZ-Domäne von GCL in verschiedenen Spezies Der Sequenzvergleich (MacVector) der BTB/POZ-Domänen von GCL-Proteinen unterschiedlicher Spezies zeigt die Konservierung der funktionellen Aminosäuren. Identische Aminosäuren sind fett und grau hinterlegt, andere verblasst dargestellt. T.c.: Tribolium castaneum (Tc001571), D.m.: Drosophila melanogaster (CG8411), M.m.: Mus musculus, H.s.: Homo sapiens. Identität zwischen T.c. und D.m. 52,3 %; T.c. und M.m. 51,4 %; T.c. und H.s. 50,9 %. Tabelle 2: Sequenzidentität der Aminosäuren unterschiedlicher GCL-Proteine Identität in % Dm-GCL Mm-GCL 38,0 33,0 Tc-GCL Tc-GCL 100,0 Hs-GCL 32,7 Dm-GCL 56,2 100,0 27,6 27,7 Mm-GCL 48,7 43,4 100,0 89,2 Hs-GCL 48,8 43,5 93,3 100,0 Anteil identischer Aminosäuren von je zwei GCL-Proteinen im Vergleich zweier NANOS-Proteine im Vergleich 79 3. Ergebnisse Tribolium homeobrain Im Gegensatz zu gcl befindet sich das Gen homeobrain (Tc008926) auf der siebenten Komplementationsgruppe. Es umfasst drei Exons und 6638 Bp, die für ein 29,34 kD umfassendes Protein kodieren, welches 261 Aminosäuren enthält (NCBI; GenomBrowser, Greifswald; Protein-Molekulargewichts-Rechner von P. Stothart, 2000). Im Screen und allen weiteren Analysen wurde ein vorhergesagtes Fragment von 338 bp verwendet, welches 64 bp von Exon 1, das gesamte Exon 2 und 44 bp des dritten Exons enthält (iB_04564). Da zum Zeitpunkt des ReScreens noch kein NoF vorgegeben war, wurde zur Verifizierung der Genspezifität ein Klon von Gregor Bucher (Georg-August-Universität, Göttingen) verwendet. Dieser Klon enthält 735 Bp des dritten Exons 3 von hbn, wobei die Sequenz via spezifischer Primer aus cDNA gewonnen und in den Vektor pCRII kloniert worden ist (lila Balken, Abb. 24). In Drosophila wurde hbn über seine Homeodomäne entdeckt (Dessain & McGinnis, 1993), und auch das Tribolium-Ortholog enthält eine solche Domäne von AS 65 bis AS 123 mit multiplen Bindestellen (nicht gezeigt; NCBI). Der Sequenzvergleich für die Homeodomänen von Tc-HBN und Dm-HBN zeigt, dass dieser spezifische funktionelle Bereich des Proteins zu 96 % konserviert ist, während das gesamte Protein nur eine Überlappung von 31,7 % aufweist (Abb. 25; Tabelle 3, MacVector). Abbildung 24: Position des für die hbn-RNAi verwendeten dsRNA-Fragments Dargestellt ist das Gen homeobrain sowie das im Screen und den nachfolgenden Versuchen verwendeten iB-Fragment. Dunkelgrün zeigt das Gen hbn (Tc08926), hellgrün die Exons, gelb das Originalfragment aus dem Screen (iB_04564) und der Folgeanalysen und in lila die Lage des Klonfragments aus dem ReScreen (http://bioinf.uni-greifswald.de/gb2/gbrowse/tribolium/; M. Stanke). Tabelle 3: Sequenzidentität von Drosophila- und Tribolium-HOMEOBRAIN im Vergleich Tc-HBN Dm-HBN Tc-HBN Dm-HBN 100,0 42,4 31,7 100,0 80 3. Ergebnisse Abbildung 25: Sequenzvergleich der Aminosäuren von HBN in Tribolium und Drosophila Der Sequenzvergleich des HBN-Protein zwischen Tribolium und Drosophila zeigt lediglich in der Homöodomäne (farbig unterstrichen) eine hohe Konservierung (96 %). Identische Aminosäuren s ind fett und grau hinterlegt, andere verblasst dargestellt (Programm MacVector). 81 3. Ergebnisse 3.3.4 Veränderungen des Blastoderm-Anlagenplans nach Verlust von gcl und hbn Wie in 3.3.1 gezeigt, führt die RNAi gegen gcl und hbn zum Auftreten von gespiegelten thorakalen bzw. abdominalen Segmenten an Stelle des Kopfes respektive Thorax. Zur Untersuchung dieses Phänomens wurden 0-24h alte RNAi-Embryonen beider Gene mit Hilfe der Expressionsmuster bekannter Gene untersucht. Der Verlust der gcl-Funktion führt zur anterioren Expression posteriorer Marker Ein Marker für posteriore Anlagen in Arthropoden (und Vertebraten) ist das Gen caudal (cad). cad wird in Tribolium und anderen Kurzkeim-Embryonen für die Ausbildung der meisten Körpersegmente benötigt. Fehlt cad kommt es zum Abbruch der Segmentation, was in Larven resultiert, die nur noch die anterioren Segmente des Kopfes (Antenne und Labrum) aufweisen (Copf et al., 2004). Obwohl im frisch abgelegten Ei zuerst ubiquitär exprimiert, zieht sich die cad Expression im reifen Wildtyp-Blastoderm auf die posteriore Hälfte des Embryos zurück, eine Region, die den Anlagen der gnathalen Kopfsegmente bis zur „segment addition zone“ (Abb. 26 A) entspricht. In gcl-RNAi-Embryonen findet diese Lokalisierung nicht statt. Vielmehr bleibt cad bis zum Eintritt der Gastrulation sowohl posterior als auch anterior exprimiert, was zu einer ubiquitären Färbung des Embryos führt (Abb. 26 B). Eine solche Duplikation posteriorer Expressionsmarker lässt sich auch für vasa (vas) und even-skipped (eve) beobachten. Beide Gene sind im wildtypischen Blastoderm posterior lokalisiert, wobei vas kurz vor Einsetzen der Gastrulation eine Gruppe von Zellen markiert, aus denen später die Keimzellen hervorgehen (Schröder, 2005). eve hingegen ist ein konserviertes Paarregelgen, das in Lang- und Kurzkeimspezies für die Segmentierung als essentiell nachgewiesen ist (Patel et al., 1994; Brown et al., 1994). Während vas im reifen Blastoderm am posterioren Pol lokalisiert ist (Abb. 26 D), bildet eve, ähnlich cad, eine posteriore Domäne, aus der im weiteren Verlauf im differenzierten Blastoderm zwei eve-Streifen und eine kleine posteriore Domäne um das „posterior pit“ (das posteriore Grübchen) herum hervorgehen (Abb. 26 G; Patel et al., 1994; Brown et al., 1994 und 1997). In gcl-RNAi-Embryonen wird vas nun an beiden Polen exprimiert (Abb. 26 E). eve Expression hingegen findet sich erst im späten Blastoderm, nach Initiierung der Mesoderminvagination. Die frühe Blastoderm-Domäne, aus der sich im Wildtyp später die 3 Streifen bilden, fehlt - sie ist weder posterior nachzuweisen noch findet sie 82 3. Ergebnisse sich, wie im Falle von cad, durch die Duplikation als ubiquitäre Expression. Weiterhin ist auffällig, dass die Streifen merklich dünner ausgeprägt sind (Abb. 26 H) als im Wildtyp. Solche Streifen treten im Wildtyp erst im Verlauf der Gastrulation auf, wenn der erste primäre Streifen beginnt sich zu teilen. Während der Formierung des Keimrudiments teilt sich der zweite Streifen auf und in der weiteren Entwicklung gehen weitere Streifen aus der „segment addition zone“ hervor, die sich ebenfalls sukzessive in schmalere segmentale Streifen aufspalten (Patel et al., 1994; Brown et al., 1997). Das Fehlen einer frühen Domäne, die veränderte Form der Streifen (schmal und teilweise unregelmäßig) liefern Hinweise darauf, dass es sich bei den Streifen in Abb. 26 H bereits um sekundäre Streifen späterer Segmente handeln könnte. Dazu passt auch, dass die ersten drei Primärstreifen von eve die Kopfsegmente und thorakale Segmente markieren (eve-1 = Mandibeln und Maxillen; eve-2 = Labium und Prothorax; eve-3 = Meso- und Metathorax; siehe Cerny et al., 2008). Vermutlich sind diese Embryonen bereits älter, wobei aber die fehlende Serosa die Bildung eines Keimrudiments verzögert (s.u.). Betrachtet man die Expression der Gene wingless (wg) und giant (gt) in gcl-RNAEmbryonen, so wird deutlich, dass im Blastoderm von vorneherein keine anterioren Segmente angelegt sind. Im Wildtyp weisen sowohl wg als auch gt neben einer posterioren Domäne eine anteriore Domäne auf. So markieren zwei schmale Streifen von wg die Anlagen der späteren Kopflappen (Abb. 27 A; Nagy & Carroll, 1994), während die dreieckige Domäne von gt (Abb. 27 C) die gnathalen Kopfanlagen umspannt (Bucher & Klingler, 2004). Sowohl die anteriore Expression von wg (Abb.27 B) als auch die gnathale gt-Domäne (Abb. 27 D) fehlen in gcl-RNAi-Embryonen. Stattdessen sind auch hier Duplikationen der beiden posterioren Domänen um die Primitivfurchen erkennbar (die allerdings schwächer exprimiert sind als ihre posterioren Originale). In allen dargestellten gcl-RNAi-Embryonen ist zudem das Auftreten einer anterioren Invagination (ähnlich dem „posterior pit“) zu erkennen (Abb.26 B, D und H; Abb. 27 B und D). 83 3. Ergebnisse RNAi gegen hbn führt zu einer Expansion posteriorer Marker Im Gegensatz zu gcl-RNAi-Embryonen führt der Verlust von hbn durch RNAi nicht zu einer anterioren Duplikation posteriore Expressionsdomänen, sondern zu einer Verschiebung der wildtypischen Expression nach anterior. Während cad im Blastodermstadium des Wildtyps die posteriore Hälfte des Embryos umspannt (Abb. 26 A), ist die Expression nun expandiert auf ca. 20 % Eilänge (Abb. 26 C; dabei entspricht 0% Eilänge dem anterioren und 100 % dem posterioren Pol entspricht). vasa-mRNA ist wie im WT (Abb. 26 D) nur am posterioren Pol lokalisiert. Ähnlich wie cad ist auch die eve Expression nach anterior verschoben (Abb. 26 I), wobei die anteriore Grenze des ersten eve-Streifens auf Höhe der Expressionsgrenze von cad liegt. Der Abstand zwischen den ersten beiden Streifen bleibt wie im Wildtyp, der zweite Streifen ist indes expandiert. In späteren Stadien fällt auf, dass mit Bildung des Keimrudiments im frühen Keimstreifen anterior und posterior distinkte cad-Domänen auftreten. Im Wildtyp ist cad in Keimrudiment und Keimstreifen auf die „segment addition zone“ beschränkt (Copf et al., 2004) und dort durchgehend bis zum vollendeten Keimstreif exprimiert (Abb. 28 A). In hbn-RNAi-Embryonen ist diese Domäne in der posterioren SAZ ebenfalls vorhanden sowie zusätzlich eine kleinere anteriore Domäne. Im frühen Keimstreif ist diese deutlich ausgeprägt (Abb. 28 B) – tatsächlich ist diese cadmRNA in sehr seltenen Fällen anterior schon im Stadium des Serosa-Schlusses exprimiert (nicht gezeigt). Immer auffällig ist jedoch die gestörte Bildung des Kopfes. So lassen sich die Kopflappen nur erahnen und auch der Übergang von Kopflappen zum restlichen Keimstreifen ist weniger prägnant ausgeprägt. 84 3. Ergebnisse Abbildung 26: Veränderungen des Blastoderm-Anlagenpl ans nach Verlust von gcl und hbn Wildtypische Embryonen im Blastodermstadium (linke Spalte), gcl-RNAi-Embryonen (mittlere Spalte) und hbn-RNAi-Embryonen (rechte Spalte). Hellfeldaufnahmen von in-Situ-Hybridisierungen gegen caudal (A-C), vasa (D-F) und even-skipped (G-I). X´ zeigt jeweils die nukleare Färbung des dazugehörigen Embryos X mit Hoechst 33342. Aufgenommen mit Helicon Focus. Anterior ist links und dorsal oben. A In späten wildtypischen Blastodermstadien ist caudal in der posterioren Hälfte des Embryos exprimiert. Posterior ist mit Bildung des „posterior pit“ die Abflachung des Pol erkennbar. B In vergleichbaren Stadien nach gcl-RNAi ist cad-mRNA ubiquitär verteilt. Beide Pole zeigen einen Beginn des „posterior pit“. 85 3. Ergebnisse C In hbn-RNAi-Blastodermstadien kommt es zu einer anterioren Verschiebung von cad. D Im Wildtyp ist vasa am posterioren Pol um die Furchungsgrube lokalisiert. E Vergleichbare Stadien nach gcl-RNAi zeigen eine weitere Domäne am anterioren Pol. An beiden Polen ist die beginnende Einstülpung des Embryos erkennbar. F hbn-RNAi-Embryonen weisen eine wildtypische vasa-Expression auf. G In diesem Wildtyp-Blastoderm sind drei bereits eve-Domänen zu erkennen, welche die posteriore Hälfte des Embryos umfassen. H gcl-RNAi-Embryonen des gleichen Stadiums zeigen eve-Streifen ebenfalls in der anterioren Hälfte. Die Expression ist unregelmäßig, teilweise gestört und zudem verschoben. Die nukleare Färbung mit Hoechst 33342 in H´ zeigt dichtere Kerne und weitere Falten an den beiden Polen, was dafür spricht, dass die eve-Streifen erst in einem späteren Stadium auftauchen. I Nach hbn-RNAi sind die ersten beiden eve-Streifen nach anterior verlagert. Der Abstand zwischen eve-1 und eve-2 scheint unverändert im Vergleich zum Wildtyp. Der zweite Streifen ist expandiert. 86 3. Ergebnisse Abbildung 27: Der Verlust anteriorer Domänen von giant und wingless nach gcl-RNAi Differentialinterferenzkontrast- (DIC)-Aufnahmen wildtypischer Embryonen (linke Spalte) und nach gcl-RNAi (rechte Spalte) im Blastodermstadium. In-Situ-Hybridisierung gegen wingless (A und B) und giant (C und D). X´ zeigt die nukleare Färbung mit Hoechst 33342. Aufgenommen mit Helicon Focus.Anterior ist links und dorsal oben. A Spätes wildtypisches Blastodermstadium. wg ist posterior um die Furchungsgrube exprimiert sowie anterior in zwei ventralen Streifen auf Höhe der Kopfanlangen. B In vergleichbaren Stadien nach gcl-RNAi ist wg-mRNA am posterioren und am anterioren Pol zu finden, wobei die Expression unterschiedlich stark bzw. zeitlich verzögert ist. Die normale Expression im Kopf fehlt dagegen. C Mit Einsetzen der Gastrulation ist gt posterior und in einer dreieckigen Domäne auf Höhe der Anlagen für den gnathalen Kopf exprimiert. D Nach RNAi mit gcl ist an beiden Polen eine gt-Expression zu finden. 87 3. Ergebnisse Abbildung 28: hbn-RNAi führt zur Expression von caudal im Kopf früher Keimstreifen Ventrale Ansicht junger Keimrudimente mit in-Situ-Hybridisierung von caudal im Wildtyp (A) und nach hbn-RNAi (B) und die jeweiligen Kernfärbungen mit Hoechst-33342 (A´ und B´). Aufgenommen mit Helicon Focus.Anterior ist links. A Im wildtypischen frühen Keimstreif ist cad posterior in der „segment addition zone“ exprimiert. B In vergleichbaren Stadien nach hbn-RNAi ist cad-mRNA ebenfalls in der Wachstumszone zu finden. Weiterhin ist cad-mRNA anterior auf Höhe des prägnathalen Kopfes vorhanden, wobei auch die Kopflappen nicht deutlich zu erkennen sind. gcl ist notwendig für die Ausbildung der extraembryonalen Membranen Die gespiegelte Expression posteriorer Gene in der anterioren Hälfte nach gcl-RNAi legt die Frage nahe, inwieweit überhaupt noch extraembryonalen Membranen angelegt werden. Wie in den meisten Insekten sind in Tribolium zwei extraembryonale Membranen ausgeprägt, Serosa und Amnion (van der Zee et al., 2008; Panfilio, 2009; Panfilio & Roth, 2010). Ein verlässlicher Marker für die Serosa ist das Gen zerknüllt-1 (zen-1), welches 1996 von Falciani et al. das erste Mal beschrieben und mit der Entwicklung der Serosa in Verbindung gebracht worden ist. 2005 konnte mittels RNAi die Funktion von zen-1 als Regulator für die Anlage der Serosa nachgewiesen werden (van der Zee et al., 2005). Als brauchbarer Marker für die Anlage des Amnions gilt iroquois (iro), wobei die Ursprünge dieses Gewebes noch nicht im Detail geklärt sind (Nunes da Fonseca et al., 2008 & 2010). Trotz mehrerer Untersuchungen wurde die zeitliche Abfolge der Expressionen von zen-1 und iro erst kürzlich ausführlich beschrieben (Sharma et al., 2013). 88 3. Ergebnisse Im differenzierten Blastoderm ist zen-1 in der Serosaanlage exprimiert, die der Embryonalanlage anterior vorgelagert ist und ungefähr 1/4 des Embryos umspannt (Abb. 29 A). Die Serosaanlage ist dorso-ventral asymmetrisch, was auf regulatorischen Input des antero-posterioren Systems um TORSO, und des dorso-ventralen Systems mit Dpp beruht, die beide an der Etablierung der Serosa und der SerosaEmbryo-Grenze beteiligt sind (van der Zee et al., 2006) - wobei auch das Gen otd eine noch nicht vollständig verstandene Rolle spielt (Kotkamp et al., 2010). Entlang der zen-1 Expressionsgrenze wird auch der Marker für das Amnion iroquois in einem Streifen exprimiert. An diesen schräg im Embryo liegenden Streifen schließt sich eine schmale dorsale Domäne an, die sich bis zum posterioren Pol erstreckt (Abb. 29 D). Nach gcl-RNAi ist hingegen keine Expression von zen-1 mehr vorhanden, was auf einen vollständigen Verlust der Serosaanlage hindeutet (Abb. 29 B). Ferner abwesend ist die anteriore Expression von iro an der Serosa-Embryo-Grenze, während die dorsale Domäne nach anterior expandiert ist (Abb. 29 E). Der Verlust von hbn-Aktivität führt zur Reduzierung der Serosa Nach RNAi konnte unter anderem für eve und cad eine Verschiebung der Expressionsdomänen nach anterior nachgewiesen werden (Abb. 26). Dementsprechend war es nicht überraschend, anterior von bzw. entlang dieser neu etablierten anterioren Grenze die Expression von zen-1 bzw. iro zu finden. Im Vergleich zum Wildtyp kommt es nach hbn-RNAi also zu einer Verkleinerung der zen-1-Domäne. Zudem ist die Abnahme der dorso-ventralen Asymmetrie auffällig (Abb. 29 C). iro hingegen ist wildtypisch exprimiert, was die Stärke der Expression betrifft. Mit Verkleinerung der zen-1-Domäne und der Ausdehnung der Embryonalanlagen kommt es allerdings zu einer Verschiebung der Expression nach anterior, so dass iro nun knapp hinter dem anterioren Pol exprimiert ist (Abb. 29 F). 89 3. Ergebnisse Abbildung 29: Veränderungen extraembryonaler Anlagen nach gcl- bzw. hbn-RNAi DIC-Aufnahmen von in-Situ-Hybridisierungen gegen zerknüllt-1 (A-C) und iroquois (D-F). Wildtypische Embryonen im Blastodermstadium (linke Spalte), gcl-RNAi-Embryonen (mittlere Spalte) und hbn-RNAi-Embryonen (rechte Spalte). X´ zeigt die nukleare Färbung des dazugehörigen Embryos X mit Hoechst 33342. Aufgenommen mit Helicon Focus. Anterior ist links und dorsal oben. A Im differenzierten Blastoderm ist zen-1 schräg von anterior nach dorsal exprimiert und markiert die Anlage der Serosa. B In vergleichbaren Stadien nach gcl-RNAi fehlt jegliche Expression von zen-1. B´ zeigt zudem die Ausbildung einer Furchungsgrube an beiden Polen. C In hbn-RNAi-Blastodermstadien kommt es hingegen zu einer Reduzierung von zen-1. Die Domäne ist nicht nur kleiner, sondern auch weniger schief ausgeprägt. D iro-mRNA ist im wildtypischen Blastoderm entlang der Grenze zwischen Serosa und Keimanlage ausgebildet, sowie dorsal daran anschließend in einer Domäne entlang der dorsalen Seite des Embryos. E Vergleichbare Stadien nach gcl-RNAi zeigen eine Domäne dorsal. Die anteriore Expression fehlt. In E´ sind die beiden polaren Primitivgruben erkennbar. F hbn-RNAi-Embryonen weisen eine nach anterior verschobene iro-Expression auf. 90 3. Ergebnisse 3.3.5 germ cell-less- und torso-Doppel-RNAi führt zum Verlust nahezu aller Segmente Die Resultate der in-Situ-Hybridisierungen (3.3.4) und die larvalen Phänotypen nach gcl-RNAi (3.3.1) implizieren eine Duplikation der „segment addition zone“ am anterioren Pol des Embryos und somit einen frühen Einfluss von gcl (direkt oder indirekt) auf das terminale System um den TORSO-Signalweg. Dieser wird im Wildtyp benötigt für die Etablierung der SAZ am posterioren, und für die Serosa am anterioren Pol (Schoppmeier und Schröder, 2005). Abbildung 30 zeigt die Phänotypen nach gcl-, torso- (tor) und gcl/tor-RNAi im Vergleich zum Wildtyp. A zeigt eine wildtypisch L1-Larve mit der typischen Untergliederung in Kopf, Thorax und Abdomen, B ein typisches Doppelabdomen nach gcl-RNAi und C den tor-RNAi-Phänotyp. torso-RNAi resultiert in einem Verlust der Serosa sowie aller Strukturen, die aus der SAZ erwachsen. Dies führt zu Larven, denen der Metathorax sowie das komplette Abdomen fehlen. Die übrigen Segmente sind hingegen wildtypisch ausgebildet (Abb. 30 C; Schoppmeier und Schröder, 2005). Das gleichzeitige Ausschalten von gcl und tor mittels RNAi resultiert in 60 % der Larven in einem kompletten Verlust anteriorer und posteriorer Segmente. Nur noch ein Kutikularest mit einem oder zwei Beinpaaren ist vorhanden (Abb. 30 D; Abb. 31). Sind zwei Beinpaare vorhanden, scheinen auch diese thorakalen Segmente spiegelbildlich angeordnet (nicht gezeigt). 91 3. Ergebnisse Abbildung 30: Defekte nach germ cell-less/torso-Doppel-RNAi Autofluoreszenz-Aufnahmen der Kutikula-Präparate von L1-Larven nach parentaler RNAi gegen germ cell-less (B), torso (C) und germ cell-less + torso DoppelRNAi (D) im Vergleich zum Wildtyp (A). Anterior ist, soweit erkennbar, stets links und dorsal oben. Die blaue Linie markiert die Symmetrieebene A Laterale Ansicht einer Wildtyp-L1-Larve mit Kopf, Thorax und Abdomen. T1 markiert das erste thorakale Segment, A“n“ das jeweilige abdominale Segment. U: Urogomphi (aus A9), P: Pygopodien (aus A10) B Phänotyp nach gcl-RNAi. Zwei unvollständigen Abdomen in gespiegelter Symmetrie. Das anteriore Abdomen weist eine unregelmäßige Segmentierung auf. Der Terminus ist an beiden Abdomen vorhanden. C Phänotyp nach tor-RNAi. Kopf und zwei thorakale Segmente sind ausgebildet. Die restlichen Segmente fehlen. D Phänotyp nach gcl+tor-RNAi. Lediglich ein Kutikularest sowie einige Beine sind erkennbar. 100% 90% 80% geschlüpfte Larven 70% Wildtyp 60% Andere 50% 40% leere Eihüllen 30% 20% Doppelabdomen 10% Kutikularest mit Beinen 0% gcl/tor-RNAi Wildtyp Abbildung 31: Verteilung der Phänotypen nach gcl/tor-Doppel-RNAi Dargestellt ist die prozentuale Verteilung der larvalen Phänotypen nach pRNAi von gcl/tor mit 1µg/µl dsRNA im Vergleich zum Wildtyp. Dunkelrot zeigt die geschlüpften Larven, rot die wildtypischen Larven auf dem Objektträger, orange RNAi-unabhängigen Hintergrund bzw. aufgrund der Präparation nicht auswertbare Phänotypen, gelb früh letale/unbefruchtete Eier, blau Doppelabdomen (gclPhänotypen) und violett Kutikulareste mit Beinen. N = 85 (gcl/tor), 317 (WT). 92 3. Ergebnisse 3.3.6 hbn ist unabhängig vom Terminalen System Wie unter 3.3.2 gezeigt, ist hbn zygotisch am anterioren Pol exprimiert. Diese Domäne ist der Expression von zen-1 sehr ähnlich, welches durch das Terminale System um den Torso-Signalweg aktiviert wird (Schoppmeier und Schröder, 2005; van der Zee et al.,2005; Vergleiche auch Abb. 21 mit Abb. 29). Die Frage, ob hbn durch TORSO reguliert wird, lag daher nahe. Zu diesem Zweck wurde die wildtypische Expression von hbn mit der Expression in torso-RNAi-Embryonen vergleichen (Abb. 32). Es zeigte sich, dass hbn auch in Abwesenheit von Torso, und daher unabhängig vom Terminalen System, exprimiert wird (Abb. 32 B). Abbildung 32: Die Expression von hbn ist nach Torso-RNAi unverändert Hellfeld-Aufnahmen von in-Situ-Hybridisierungen gegen hbn im Wildtyp (A) und nach Torso-RNAi (B). X´ zeigt die nukleare Färbung des dazugehörigen Embryos X mit Hoechst 33342. Aufgenommen mit Helicon Focus. Anterior ist links und dorsal oben. A Im Blastoderm ist hbn leicht schräg von anterior nach dorsal exprimiert. B Vergleichbares Stadium nach Torso-RNAi. Die hbn-Domäne ist unverändert. 93 3. Ergebnisse 3.3.7 Durch gcl-RNAi verursachte Defekte in Keimstreifen Die kugelförmigen Kutikulas nach gcl-RNAi (3.3.1) deuten darauf, dass es zusätzlich zur Duplikation der „segment addition zone“ weitere Veränderungen des Anlagenplans gibt, was zu einem ernsten Defekt der Segmentbildung führt. Diese lässt sich am besten anhand eines Segmentpolaritätsgens wie wg untersuchen, welches bereits während der Elongation des Keimstreifens in paarigen segmentalen Streifen exprimiert wird (Nagy & Carroll, 1994). Im Zuge der Elongation kommt der Embryo in einem breiten U zu liegen (UStadium), wobei der Kopf am anterioren Pol liegt und der posteriore Keimstreifen mit der „segment addition zone“ am posterioren Pol (Abb. 33 A). Der Kopf ist dabei deutlich anhand der Kopflappen zu erkennen. Die SAZ hingegen lässt sich als Verbreiterung am posterioren Ende des Keimstreifens erkennen. wg ist in den Kopflappen, als posteriore Domäne in der SAZ, und in den Segmenten dazwischen jeweils posterior exprimiert. Die parentale RNAi gegen gcl führt zu einem Spektrum von Phänotypen in den Keimstreifen. In Abb. 33 B zum Beispiel sind die beiden symmetrischen abdominalen Keimstreifen relativ normal entwickelt, wobei man durch die posteriore Expression von wg in jedem Segment die Polarität der Segmente und die Lage der Symmetrieachse erkennen kann. Häufiger zu finden sind Keimstreifen, bei denen eines der beiden Enden aus dem Embryo herauswächst (Abb. 33 C Pfeilkopf). Die Segmentierung ist noch relativ regelmäßig, abgesehen von Verdrehungen eines Abdomens und/oder einer Vergrößerung des Abstand zwischen den beiden segmentalen Streifen, die auf eine Störung oder einen Riss des embryonalen Gewebes hindeuten (Abb. 33 C und C´ offener Pfeilkopf). In stärker betroffenen Embryonen, wie in Abb. 33 D, werden diese Öffnungen entlang der Mittellinie stärker. Die lateralen Seiten des Keimstreifens treffen sich nur noch an der Symmetrielinie und an den beiden „segment addition zone“n, während die restlichen Segmente durch die Löcher entlang der Mittellinie getrennt sind (D´, offener Pfeilkopf). Zudem kann man in den Kernfärbungen (B´-D´) oftmals Störungen oder sogar Risse im extraembryonalen Gewebe identifizieren. Bei der Präparation der gcl-RNAi-Keimstreifen fiel zudem auf, dass sie nicht nur flacher und fragiler sind als die Wildtypen, sondern stärker an der Eihülle zu kleben scheinen, was ein Herauslösen so gut wie unmöglich macht. 94 3. Ergebnisse Abbildung 33: Keimstreifen mit gestörter Segmentierung nach gcl-RNAi Ventrale bzw. laterale Ansicht wachsender Keimstreifen. In-Situ-Hybridisierung von wingless im Wildtyp (A) und nach gcl-RNAi (B-E) und die dazugehörigen Kernfärbungen mit Hoechst-33342 (A´-E´). Aufgenommen mit Helicon Focus. Die blaue Linie markiert die Symmetrieachse in gclRNAi-Embryonen. Anterior ist - soweit erkennbar - links und dorsal oben. A Im wildtypischen frühen Keimstreif ist wg in den Kopflappen, posterior in jedem Segment, sowie in der „segment addition zone“ exprimiert. In A´ lässt sich das dichte Gewebe des Keimstreifens gut vom extraembryonalen Gewebe mit seinen weitverteilten Kernen und dem milchigen Dotter unte rscheiden. B Vergleichbares Stadium nach gcl-RNAi. Statt eines Kopfes ist ein deutlich verlängerter bzw. duplizierter Keimstreif erkennbar. wg mRNA ist an beiden Enden des Keimstreifs lokalisiert, die den beiden „segment addition zone“n entsprechen, sowie segmental, wobei sich die Polarität der Segmente 95 3. Ergebnisse und damit die Lokalisation von wg ändert. Die blaue Line zeigt den Ort der Symmetrieachse, auch erkennbar am gestörten wg-Muster. B‘ enthüllt Unregelmäßigkeiten im extraembryonalen Gewebe. C Auch in diesem Embryo ist der Keimstreif verlängert, und die wg-Expression beidseitig der blauen Linie spiegelbildlich polarisiert.. Eine Seite des Keimstreifens liegt verdreht und ragt am Ende aus dem Embryo (Pfeilkopf). Eines der beiden mutmaßlichen Abdomen weißt mittig ein Loch im Gewebe auf (offener Pfeilkopf), welches in der DAPI-Färbung (C´) deutlicher zu erkennen ist. D und D´ Wie C, nur dass an beiden Seiten des Keimstreifens Öffnungen entlang der Mittellinie au ftreten (offener Pfeilkopf), so dass die rechte und linke Seite des Keimstreifens lateral in den Embryo hineinragen. 96 3. Ergebnisse 3.3.8 Veränderung der morphogenetische Bewegungen nach gcl-RNAi Wie in 3.3.3 gezeigt werden konnte, kommt es nach gcl-RNAi zu einer Expression posteriorer Marker in der anterioren Hälfte des Embryos (z.B. eve und cad). Zudem konnte in diesen RNAi-Embryonen keine Expression von zen-1 bzw. der Verlust der anterioren Expression von iro nachgewiesen werden, was auf einen drastischen Verlust extraembryonalen Gewebes hindeutet. Weiterhin wurden in den mit wggefärbten Keimstreifen (3.3.5) Deformation auffällig, die Hinweise auf gestörte morphogenetische Bewegungen und evl. auch dorso-ventrale Defekte sein könnten. Um dies zu untersuchen und ein besseres Verständnis der Entstehung des gcl-RNAiPhänotyps zu erhalten, konnte Dr. Kristen Panfilio (Universität zu Köln) für eine Kollaboration gewonnen werden. Dr. Panfilio hat sich in den letzten Jahren auf Life Imaging Verfahren spezialisiert und studiert die zellulären Vorgänge der extraembryonalen Membranen im Verlauf der Embryogenese (Lynch et al., 2009; Panfilio, 2009; Panfilio & Roth, 2010; Panfilio et al., 2013). Wie bei allen Insekten finden sich auch in der Embryogenese von Tribolium vier prinzipielle Vorgänge: die Gastrulation, Keimstreifenverlängerung (Elongation) und Verkürzung (Retraktion) und als letzten Schritt der Entwicklung der dorsale Rückenschluss des Embryos (Ausführlich beschrieben in Handel et al., 2000; Benton et al., 2013; Strobl & Stelzer., 2014). Für das Experiment wurde gcl-dsRNA in Weibchen eines transgenen Stammes injiziert, der im Kern lokalisiertes GFP exprimiert (Sarrazin et al., 2012). Die Embryonen wurden im Anschluss über mehrere Stunden hinweg im Zeitraffer gefilmt und im Vergleich zum Wildtyp analysiert. Insgesamt wurden 32 gcl-RNAi und 9 wildtypische Embryonen jüngeren Stadiums, sowie 36 gcl-RNAi und 17 wildtypische ältere Embryonen (zwischen Keimstreifenverkürzung und Dorsalschluss) dokumentiert. Bei Tribolium kommt es gegen Ende des Blastodermstadiums im 13. Zellzyklus zu einer asynchronen Zellteilung, bei der nur die Kerne des späteren Keimrudiments die Mitose durchlaufen. Anschließend differenziert das Blastoderm in Serosa und Embryonalanlage (Handel et al., 2000; Benton et al., 2013; Strobl & Stelzer., 2014). Die Serosa erstreckt sich im vorderen Drittel des Embryos asymmetrisch von anterior nach dorsal und ist deutlich erkennbar an den großen Zellkernen, die weiträumiger verteilt vorliegen als im Keimrudiment (Abb. 34 A). Dann beginnt der Embryo zu gastrulieren, der posteriore Pol flacht zunehmend ab und stülpt sich ein („posterior 97 3. Ergebnisse pit“; Abb.34 B und C). Durch Kompaktion der Zellen der Embryonalanlage bildet sich nun ventral das Keimrudiment, das im weiteren Verlauf posterior in den Dotter sinkt und schließlich zuerst posterior, später auch anterior von den extraembryonalen Membranen überwachsen wird (Abb.34 D-F). In gcl-RNAi-Embryonen hingegen ist mit Eintritt in die Gastrulation kein Unterschied zwischen anterioren und posterioren Nuklei erkennbar. Vielmehr sind alle Zellkerne gleichmäßig verteilt und es sind keine Unterscheide in der Größe auszumachen (Abb. 34 G). Wie im Wildtyp verdichten sich auch in den RNAi-Embryonen die embryonalen Zellen ventral, gleichzeitig jedoch entsteht sowohl am posterioren als auch am anterioren Pol ein „posterior pit“ (Abb. 34 H und I). Während das Keimrudiment zudem im Wildtyp zunächst ventral lokalisiert bleibt, kommt es in gclRNAi-Embryos zu einer Verlagerung zum wahren posterioren Pol (identifiziert anhand der Form des Eis). Im Zuge dieser Verschiebung und der Kompaktion des Gewebes kommt es dorsal zu einem Riss im extraembryonalem Gewebe (Abb. 34 J und L), der das Scheitern des Dorsalschlusses zur Folge haben kann. Im Wildtyp kommt es gegen Ende der Keimstreifverkürzung ebenfalls zu einem Riss der Serosa, eine Vorraussetzung für einen normalen Rückenschluss. Serosa und Amnion ziehen sich zusammen und formen dorsal einen Zellklumpen (Dorsalorgan) (Abb. 34 M-O). Im Verlauf des Dorsalschlusses sinkt dieses dorsale Organ in den Dotter ein und untergeht Apoptose (Panfilio et al., 2013). Ein solches Dorsalorgan ist nur schwächeren gcl-RNAi-Embryonen erkennbar (nicht gezeigt). In den stärkeren, aufgerissenen RNAi-Embryonen aber können die morphogenetischen Bewegungen des Keimstreifs während der Retraktionphase zu einem Austritt von Dotter führen (Abb. 34 P-R). Blockiert dieser das embryonale Gewebe führt dies vermutlich zu einem unvollständigen Dorsalschluss, wie in Abb. 16 F dargestellt. 98 3. Ergebnisse (a) 99 3. Ergebnisse (b) Abbildung 34a und b: Die Veränderung der morphogenetischer Bewegungen nach gcl-RNAi führt zu einem Riss im extraembryonalen Gewebe Ausschnitte aus der Embryogenese zweier gcl-RNAi-Embryonen (früh G-L und spät P-R) im Vergleich zum Wildtyp (A-G bzw. O-Q) unter Verwendung eines Tribolium-Stammes mit nuklearem GFP und Life Imaging (ApoTome, Zeiss). Anterior befindet sich links, dorsal oben. A-B Laterale Ansicht eines differenzierten Blastoderms. Die Serosaanlage umfasst das anteriore Viertel des Blastoderms und ist deutlich erkennbar an der weiträumigeren Verteilung der Zellkerne. Mit Beginn der Gastrulation flacht der posteriore Pol ab zur posterioren Furche. Ventral verdichten sich die Zellen der Keimanlage. C-F Das Keimrudiment ist ventral deutlich erkennbar. Am posterioren Pol beginnt sich das Amnion über das embryonale Gewebe zu stülpen. Die Serosa bedeckt dorsal den Dotter. Das Keimrudiment sinkt vorübergehend posterior in den Dotter, und wird später komplett von extraembryonalem Gewebe bedeckt . G Laterale Ansicht eines späten gcl-RNAi-Blastoderms. Die Serosaanlage ist nicht erkennbar. H-J Es kommt zur Abflachung/posterior pit Bildung an beiden Polen des Embryos. Ventral verdichten sich die Zellen der Keimanlage und verbleiben dort ohne in den Dotter einzusinken. K-L Das Keimrudiment bewegt sich in Richtung des wahren posterioren Pols (erkennbar an der Form des Embryos). Anterior dorsal reißt das Amnion auf (Pfeilkopf). 100 3. Ergebnisse M-O Dorsalschluss eines wildtypischen Embryos. Amnion und Serosa reißen anterior/ventral auf und wandern über den Embryo nach dorsal, wo sie dorsal als flache Scheibe (Dorsalorgan) zu liegen kommen. Dort sinken die Zellen im Zuge des Dorsalschlusses des Embryos in den Dotter ein und sterben. P-R Dorsalschluss eines gcl-RNAi-Embryos. Der Embryo liegt wie der Wildtyp in der Eihülle. Das Doppelabdomen ist deutlich erkennbar. Beide Abdomen liegen dorsal offen. Im Zuge morphogenet ischer Bewegungen sickert Dotter aus dem Embryo. Ein Dorsalorgan ist nicht erkennbar. 101 3. Ergebnisse 3.3.9 Die Lokalisation von Tc-GCL im frühen Embro und innerhalb der Zellen bleibt ungewiss Ausgehend von den bisherigen Ergebnissen scheint GCL mit dem anterioren terminalen System zu interagieren, so dass dort anstelle einer „segment addition zone“ anteriore Strukturen gebildet werden. In Drosophila und Maus ist GCL an der inneren Kernmembran lokalisiert und fungiert als Transkriptionsrepressor - um die Keimzellen von Drosophila transkriptionell zu „silencen“, bzw. um über die Bindung von E2F den Zellzyklus in Mus musculus zu regulieren (Jongens et al., 1992 und 1994; de la Luna et al., 1999; Leathermann et al., 2002). Da gcl-mRNA in späten Oozyten und frisch abgelegten Embryonen in der anterioren Hälfte lokalisiert ist, wäre eine Funktionsweise von GCL als in der Kernmembran verankerter Repressor auch in Tribolium denkbar. Hierbei würde GCL anterior einen bisher noch unbekannten Transkriptionsfaktor binden, der posterior nicht reprimiert und die „segment addition zone“ induzieren würde. Diese Bindung könnte zum Beispiel über die BTB/POZ-Domäne reguliert werden, die auch in Tc-GCL konserviert ist (vgl. 3.3.10). Zur Klärung der intrazellulären Lokalisation von GCL in Tribolium wurden sechs polyklonale Antikörper aus Kaninchen (Pineda) untersucht, die gegen zwei verschiedene Peptide (GCL 7 und GCL 9) induziert worden waren (s. 2.11.1). Allerdings konnte auch 210 bzw. 240 Tagen nach der Immunisierung die erwartete AntigenExpression nicht immunohistologisch nachgewiesen werden. Erst mit Aufreinigung der Seren und sofortige Überführung der fixierten Embryonen in PBT, d.h. Minimalisierung des Kontakts mit Methanol, wurde für GCL 7, aus Tier 1 (GCL 7.1) ein Antigen in der Kernmembran der Nuklei des Embryos lokalisiert (Abb. 35 A). Dies konnte ich mit Hilfe eines Fluoreszenz-markierten 2.Antikörpers und Parallelfärbung mit Hoechst 33342 verifizieren (Abb. 35 A´). Leider zeigte sich bei der Analyse der gcl- RNAi-Embryonen aber, dass dieses Antigen unverändert vorkommt und daher offenbar als unspezifische Bindung des Antikörpers zu betrachten ist (Abb. 35 B). Der Effekt der RNAi wurde dabei parallel durch Kutikula-Präparate überprüft. Auch im Ovar von wildtypischen und von gcl-RNAi-Weibchen ist das vom Antikörper GCL 7.1 detektierte Antigen scheinbar in den Nuklei der Follikelzellen und im Oozytenkern exprimiert (Abb. 36 C und D). Kontrollen mit dem Präimmunserum 102 3. Ergebnisse und dem fluoreszierenden Zweitantikörper Cy2 zeigen lediglich eine schwache Färbung der Follikelzellen, nicht aber im Oozytenkern (Abb. 36 E und F). Auch die Färbung mit einem GCL-Antikörper aus Drosophila, entliehen von der Arbeitsgruppe um Paul Lasko (McGill Universität, Montreal, Kanada), brachte keine Ergebnisse hervor. Die Embryonen wiesen nur fleckige Hintergrundfärbungen auf (nicht gezeigt). Ebenfalls erfolglos blieb ein Ansatz nach der Methode von Benton et al., 2013 (vgl. 2.10). Weder die mRNA von gcl verknüpft mit EGFP aus dem Vektor pCS2, noch die aus dem Vektor pT7, führten nach embryonaler Injektion zu einer Fluoreszenz. Alle Embryonen verstarben zudem wenige Stunden nach der Injektion. Als Positiv-Kontrolle diente mRNA des Plasmids pT7-LifeAct-EGFP, die unserem Labor freundlicherweise von M. Benton (Universität Cambridge, UK) zur Verfügung gestellt wurde (nicht gezeigt), hier wurde das erwartete Fluoreszenzmuster beobachtet. Die Überlebensrate betrug in der Kontrolle 15-20 %. Abbildung 35: Antikörperfärbung von GCL-Protein in Embryo und Ovar bleibt ergebnislos 103 3. Ergebnisse A und B Hellfeldaufnahme (Helicon-Fokus) von Embryonen bzw. C-F Fluoreszenzaufnahmen von Ovaren gefärbt mit dem polyklonalen Antikörper 7, von Tier 1, gegen Tribolium GERM CELL-LESS und Cy2 als 2. Antikörper. A Wildtypisches undifferenziertes Blastoderm. Das Antigen 7 ist entlang der anterior-posterioren Achse des Embryos ubiquitär exprimiert. Subzellulär ist das Antigen im Bereich der Kernmemban lokalisiert. A´ zeigt einen vergrößerten Ausschnitt einer korrespondierenden Fluoreszenzfärbung. Blau dargestellt ist die nukleare Färbung der DNA mit Hoechst 33342, rot der polynukleäre Antikörper 7 gegen GCL. B Undifferenziertes Blastoderm eines gcl-RNAi-Embryos. Die Färbung unterscheidet sich nicht vom Wildtyp. C Wildtypisches Ovar. GCL ist schwach im Gewebe und stärker im Nukleus der Oozyten exprimiert. D gcl-RNAi-Ovar. GCL ist schwach im Gewebe und stärker im Nukleus der Oozyten erkennbar. E und F Kontrollfärbungen: Wildtypisches Ovar mit dem Präimmunserum von Tier 1 bzw. mit dem 2. Antikörper Cy 2. In beiden Fällen ist nur eine schwache Färbung des Gewebes erkennbar. 3.3.10 Die Beteiligung bekannter Bindungspartner von Gcl ist in Tribolium fraglich In Drosophila und Mus musculus erfolgt die Lokalisierung von GCL an die innere Kernmembran über Bindung an die drei Proteine EMERIN, MAN1 und LAP2β (Jonges et al.,1994; Nili et al., 2001; Holaska et al., 2003; Mansharamani & Wilson, 2005). Weiterhin ist bekannt, dass GCL den Transkriptionsfaktor DP bindet und somit DP-E2F-abhängige Gene inhibiert, was den Eintritt der Zellen in die S-Phase verhindert (de la Luna, 1999). Aufgrund der Konservierung der BTB/POZ-Domäne (vgl. 3.3.3) lag eine Beteiligung dieser Bindungspartner in Tribolium im Bereich des Möglichen, weshalb mit Hilfe der Datenbanken von NCBI und OrthoDB nach entsprechenden Orthologen in Tribolium gesucht wurde. Von den drei Proteinen, die in Vertebraten GCL über ihre LEM-Domäne an die Lamine der inneren Kernmembran binden, konnte lediglich MAN1 in Tribolium identifiziert werden (TC030575). Pupale RNAi gegen dieses Gen führte in allen injizierten Weibchen zu Sterilität, aufgrund eines Arrests der Oozyten in der frühen Vitellogenese (nicht gezeigt). Dieser Defekt konnte zwar durch Injektion adulter Weibchen umgangen werden, die adulte RNAi selbst resultierte jedoch zu 100 % in leeren Eiern – womöglich durch einen versteckten Defekt in der Oogenese, denn nach zwei bis drei Wochen konnte auch hier der Ovarphänotyp festgestellt werden. Anders gestalteten sich die Ergebnisse für die Bestandteile des Heterodimers DPE2F. Für DP konnte ein Ortholog gefunden werden (TC010400; Komplementationsgruppe 5), für E2F TC011687 (Komplementationsgruppe 9) und TC005074 (E2F-2; Model; unbekannte Gruppe 59). Beide E2F-Orthologe waren im Zuge des iBeetle- 104 3. Ergebnisse Screens getestet worden und führten zum Tod der injizierten Weibchen innerhalb der ersten 5 Tage nach Injektion (iB_01875 und iB_00800). Auch die von mir durchgeführte pupale RNAi gegen DP (TC010400) resultierte im Tod aller Weibchen. Ich wiederholte die RNAi gegen die beiden E2F Orthologen daraufhin in adulten Weibchen; Im Fall von TC011687) legten die injizierten adulten Weibchen zu 90 % leere Eier, und be E2F-2 (TC005074) wurden gar keine Eier mehr produziert; die Analyse des Ovars offenbarte das Fehlen jeglichen Fettkörpers und einen „starved“-Phänotyp (vgl. 3.1.1). Auch im Falle von DP wurde auf adulte RNAi umgestellt, was zum Überleben der Weibchen, aber der Ablage von 100 % leerer Eier führte. Die Analyse der Eier mit Hilfe der nGFP-Linie resultierte in 43 % unbefruchteten Eiern, sowie Defekten in Keimrudiment und Keimstreif. Bei beiden Genen behielten aber die RNAi-Embryonen (im Gegensatz zu gcl-RNAi) größtenteils die wildtypische Gliederung in Kopf, Thorax und Abdomen bei, lagen aber verdreht und ohne erkennbare Orientierung im Ei (Abb. 36). Die dsRNA-Injektion von E2F (TC011687) in adulte Weibchen resultierte in Larven, denen Teile des Abdomen fehlen, während die vorhandenen Segmente, insbesondere Kopfanhängsel und Beine, aufgequollen wirken. Die Körperborsten fehlen vollständig. Allerdings findet sich dieser Phänotyp in geringer Penetranz von < 10 %. Die adulte RNAi von E2F-2 (TC005074) führte hingegen zu leeren Eiern und wildtypischen Larven. Um eventuelle Genredundanz auszuschalten, wurde Doppel-RNAi gegen beide E2F-Orthologe durchgeführt. Dies führte zu einer Verstärkung des Phänotyps von E2F. Thorax und Metathorax fehlen, die Segmentierung der restlichen Segmente ist gestört und auch im Kopf sind starke Deformationen zu beobachten (Abb. 37 und Abb. 38). Es scheint also, dass gcl seine Funktion in Tribolium nicht über der Komplex E2F-DP entfaltet. Denn sowohl die RNAi gegen E2F als auch DP resultierte in Phänotypen fern der Doppelabdomen und auch die Doppel-RNAi resultierte in Larven, deren grundsätzlicher Bauplan in Kopf, Thorax und Abdomen, trotz Deformationen, noch vorhanden ist. Da sich auch die Phänotypen von gcl in Drosophila und Tribolium unterscheiden ist es daher denkbar, dass der gcl andere Faktoren bindet. 105 3. Ergebnisse Abbildung 36: Defekte nach adulter RNAi gegen DP (TC010400) DP-RNAi-Embryonen (rechts) im Vergleich zum Wildtyp (links) unter Verwendung eines TriboliumStammes mit nuklearem GFP. ApoTome (AxioVision). Anterior, soweit erkennbar, liegt links und dorsal oben. A Wildtypischer Embryo während der Gastrulation. Ventral ist das Keimrudiment deutlich zu sehen. Die extraembryonale Membranen umschließen das Keimrudiment fast vollkommen. B DP-RNAi-Embryo während der Gastrulation. Das Keimrudiment liegt quer auf der ventralen Seite. Anterior ist Dotter vorgelagert. C Wildtypisches U-Stadium. Das Keimband liegt entlang der Eihülle ventral ausgestreckt. Anterior ist der Kopf anhand der Kopflappen erkennbar. D DP-RNAi-Embryo im U-Stadium. Der Keimstreif liegt verdreht im Ei. Abbildung 37: Defekte nach adulter RNAi gegen E2F Autofluoreszenz-Aufnahmen der Kutikula-Präparate von L1-Larven nach parentaler Doppel-RNAi gegen die beiden Drosophila-Orthologe von E2F (TC011687 und TC005074) (C-D) im Vergleich zum Wildtyp (A) und gcl-RNAi. ApoTome (AxioVision). Anterior ist, soweit erkennbar, links und dorsal oben. 106 3. Ergebnisse A Laterale Ansicht einer Wildtyp-L1-Larve mit Kopf, Thorax und Abdomen. T1 markiert das erste thorakale Segment, A“n“ das jeweilige abdominale Segment. U: Urogomphi (aus A9), P: Pygopodie n (aus A10) B Typischer Phänotyp nach parentaler RNAi gegen gcl. Zwei unvollständigen Abdomen in gespiegelter Symmetrie. Das anteriore Abdomen weist eine unregelmäßige Segmentierung auf. Der Terminus ist an beiden Abdomen vorhanden (Pfeilspitze) C Milder Phänotyp nach adulter RNAi gegen E2F und E2F-2. Die Anhängsel des Kopfes sowie die Beine wirken ausgequollen. Die Körperborsten fehlen. Zudem ist die Kutikularisierung im Abdomen gestört. Einzelne Segmente sind kaum auszumachen. D Phänotyp nach adulter RNAi gegen E2F und E2F-2. Der Kopf weist starke Defekte auf. Metathorax und Abdomen fehlen. Verbliebene Anhängsel des Kopfes sowie die Beine sind deformiert. Körpe rborsten fehlen. 100% 90% starker Phänotyp 80% 70% schwacher Phänotyp 60% leere Eier 50% 40% Andere 30% Wildtyp 20% 10% 0% E2f & E2F-2 E2F-2 (TC005074) E2F (TC011687) Abbildung 38: Phänotypen nach E2F-Einzel- und Doppel-RNAi Dargestellt ist die prozentuale Verteilung der Phänotypen nach adulter RNAi gegen E2F, E2F-2 und E2F/E2F-2. Dunkelrot zeigt die geschlüpften Larven, rot die wildtypischen Larven auf dem Objektträger, orange RNAi-unabhängigen Hintergrund bzw. aufgrund der Präparation nicht auswertbare Phänotypen, gelb früh letale/unbefruchtete Eier und blau die verschiedenen Phänotypen. Der milde Phänotyp (hellblau) und starke Phänotypen in blau. 107 3. Ergebnisse 3.3.11 RNAi von gcl führt zu einem selektiven Verlust maternaler Positionsinformation Meine Beobachtungen des gcl-Phänotyps zeigen klar, dass gcl eine Rolle in der frühembryonalen Musterbildung spielt. Obwohl die Lokalisation des Proteins, sowie mögliche Bindungspartner, über die GCL seine Funktion vermittelt, noch offen sind, deuten alle Ergebnisse auf eine Duplikation der „segment addition zone“ und somit eine Beteiligung von GCL auf die Determinierung des terminalen Systems in Bezug auf ein Schicksal anterior vs. posterior hin. Ausgehend von meinem ursprünglichen Modell, welches auf der Lokalisation der gcl-mRNA und der Lokalisation des GCLProteins in Drosophila und Maus beruhte, sollte GCL seine Funktion im abgelegten Embryo ausüben, nach Einsetzen der Translation maternaler RNA (Jongens et al., 1992 und 1994; de la Luna et al., 1999; Leathermann et al., 2002). Eine Überprüfung anterior lokalisierter mRNAs in gcl-RNAi-Embryonen zeigte jedoch, dass dieses Modell Lücken hat. Im wildtypischen Embryo sind pangolin (pan), eagle und axin im frisch abgelegten bzw. unbefruchteten Embryo am anterioren Pol lokalisiert (und noch lokal begrenzt um den Polkörper exprimiert) (Abb. 39 A und D). Eine Funktion konnte allerdings bisher nur für axin nachgewiesen werden (Bucher et al., 2005; Fu et al., 2012). Überraschenderweise zeigte sich aber, dass nach RNAi mit gcl die anterior lokalisierte mRNA von pan komplett fehlt (Abb. 39 B). Die mRNA von axin ist dagegen weiterhin am anterioren Pol lokalisiert (Abb. 39 D). Allenfalls ist die axin-Domäne möglicherweise etwas diffuser verteilt als im WT, und umfasst ungefähr das erste Drittel des Embryos. Die anteriore Expression von eagle scheint hingegen reduziert (nicht gezeigt). All diese Beobachtungen legen nahe, dass Tc-gcl seine Funktion bereits während der Oogenese ausübt (wenn die mRNAs von pan, eagle und axin in der Oozyte lokalisiert werden). Die in-Situ-Hybridisierung von pan und axin in hbn-RNAi-Embryonen zum Vergleich zeigen erwartungsgemäß eine Lokalisierung beider Gene, die dem Wildtyp entspricht (Abb. 39 C und E). eagle wurde nicht getestet. 108 3. Ergebnisse Abbildung 39: Expression maternaler mRNAs nach gcl und hbn-RNAi Frisch abgelegte wildtypische Embryonen (linke Spalte), gcl-RNAi-Embryonen (mittlere Spalte) und hbn-RNAi-Embryonen (rechte Spalte). DIC-Aufnahmen der in-Situ-Hybridisierungen gegen pangolin (A-C) und axin (D-F). X´ zeigt die nukleäre Färbung des dazugehörigen Embryos X mit Hoechst 33342, mit dem Polkörper als weißem Punkt. Anterior ist links. A Im frisch abgelegten Embryo ist pan am anterioren Pol als Kappe, und zusätzlich um den Polkörper lokalisiert. B In vergleichbaren Stadien nach gcl-RNAi fehlt jegliche Färbung von pan. C In hbn-RNAi-Embryonen wird pan wie im Wildtyp als anteriore Kappe und schwach um den Polarkörper exprimiert D axin-mRNA findet sich im Wildtyp als Kappe am anterioren Pol und schwach um den Polarkörper. E Die anteriore Färbung von axin scheint weniger an den anterioren Pol gebunden, und wirkt insgesamt diffuser. Die lokalisierte Färbung um den Polkörper ist gut erkennbar. F Nach hbn-RNAi findet sich axin wie im Wildtyp am anteriore Pol und um den Polarkörper. 109 3. Ergebnisse 3.3.12 Embryonale RNAi führt nur bei hbn zum Doppel-Abdomen-Phänotyp Der selektive Verlust maternaler mRNA-Lokalisierung nach gcl-RNAi wirft die Frage auf, ob GCL seine Funktion wie bisher angenommen im Embryo innehat oder bereits vorher im Ovar wirkt. Der systemische Effekt der RNAi ist hierbei von Nachteil, da mit Injektion der dsRNA alle Gewebe und Genfunktionen getroffen werden (Brown et al.,1999; Bucher et al., 2002; Tomoyasu et al., 2008). Um dies zu umgehen, kann man auf die embryonale RNAi zurückgreifen, die zielgerichtet nur die mRNAs ausschaltet, die sich zum Zeitpunkt der Injektion im Embryo befinden. Dieser direkte Knockdown führt in der Regel zu den stärksten möglichen Phänotypen, wird allerdings selten angewendet wegen der erhöhten Letalität der Embryonen (Injektionsartefakt), sei es durch das Handling, die Injektionswunde oder durch Austrocknung der Embryonen (Brown et al., 1999; Posnien et al., 2014). Außerdem ist es schwieriger, injizierte Embryonen mittels in-Situ-Hybridisierung anzufärben. Die dsRNAs von gcl und hbn wurden sowohl von mir, als auch im Mastermodul 2012/2013 und von Katharina Kalb im Zuge ihrer Bachelor-Arbeit injiziert. Für hbn war es in allen Fällen möglich, mit dsRNA von 1 µg/µl den Kopf-Phänotyp zu reproduzieren (Abb. 40). In einigen Experimenten gelang es auch, DoppelabdomenPhänotypen zu generieren, wegen der gesteigerten Sterblichkeit belief sich die Penetranz hierbei aber lediglich auf 15-20 %. Der Rest der Embryonen entwickelte sich nicht bzw. trocknete aus. Im Falle von gcl hingegen führte die eRNAi in 100 % der Embryonen zum Tod, woraufhin die Konzentration der dsRNA von mir systematisch gesenkt wurde. Erst bei einer Senkung der Konzentration auf 0,1-0,2 µg/µl kam es zum Auftreten von L1-Larven. Hierbei waren neben wildtypischen Larven Embryonen mit starken Defekten zu finden, die allerdings nicht dem gcl-Phänotyp der pRNAi entsprachen. Vielmehr handelte es sich bei den Phänotypen um bekannte globalere Defekte, die immer wieder im Zuge der eRNAi zu beobachten sind, weshalb sie als Artefakte der eRNAi an sich zu betrachten sind (Pufferkontrolle; interne Kommunikation): Einschnürungen im Bereich des Thorax oder des anterioren Abdomens, komplette Trennung der Tagmata und/oder Kopfdefekte (nicht gezeigt). 110 3. Ergebnisse Abbildung 40: Verifizierung der zygotischen Funktion von hbn durch eRNAi Autofluoreszenz-Aufnahme einer exemplarischen L1-Larve nach embryonaler RNAi gegen hbn (B) im Vergleich zum Wildtyp (A). Anterior befindet sich links und dorsal oben. A Laterale Ansicht einer Wildtyp-L1-Larve untergliedert in Kopf, Thorax und Abdomen. T1 markiert das erste thorakale Segment, A“n“ das jeweilige abdominale Segment. U: Urogomphi (aus A9), Pg: Pygopodien (aus A10). B Phänotyp nach eRNAi gegen hbn. T2, T3 und Abdomen sind normal ausgebildet. Anterior von T1 schließen sich zwei weitere thorakale Segment sowie 3 abdominale Segmente an. Die Symmetrie der neuen Segmente ist gespiegelt, wobei die Symmetrieachse (blaue Linie) durch die Fusion von T1 und dem neuen T2-Segment geht, das anterior daran anschließt. T2 ist gut identifizierbar anhand der Öffnung der Tracheen (Pfeilkopf). 111 Diskussion 4. Diskussion 4.1 Der genomweite RNAi-Screen iBeetle als Methode für die Identifizierung von Entwicklungsgenen Der erste Abschnitt dieser Arbeit widmete sich meinen Ergebnissen aus dem pupalen Teil des genomweiten RNAi-Screens iBeetle, wobei neben den allgemeinen Zahlen exemplarisch Beispiele für die unterschiedlichen Klassen an Phänotypen präsentiert wurden, einschließlich der Phänotypen, die potentiell neuartige Segmentierungsgene identifizieren können. Im Abschnitt 3.2 habe ich zudem beschrieben, nach welchen Kriterien ich Kandidatengene für die ausführliche Analyse ausgewählt habe und erörtere nun, wie gut sich dieser methodische Ansatz eines genomweiten Screens bewährt hat. 4.1.1 Der iBeetle-Screen identifizierte untersuchten Prozesse interessante Kandidatengene für alle Ein genomweiter Screen wie iBeetle ist ein extrem nützliches Instrument zur funktionellen Analyse der Gene eines Organismus, insbesondere, wenn zugleich mehrere Prozesse untersucht werden. Gleichzeitig fordert dies aber auch ein enormes Maß an Koordination, Interaktion und Flexibilität. 2010 erstellte daher C. Schmitt-Engel anhand eines Pilotscreens einen Ablaufplan, der die zeitnahe Untersuchung mehrerer Prozesse ermöglicht. Zudem verifizierte er die RNA-Interferenz für einen genomweiten Screen – eine Methode, die in Tribolium zwar bereits genutzt, in großem Maßstab jedoch bis dato nur in C. elegans und in Zellkulturen von Drosophila verwendet worden war (Gönczy et al., 2000; Simmer et al., 2003; Lum et al., 2003; Boutros et al., 2004; Sönnichsen et al., 2005). Tribolium castaneum ist die ideale Plattform für einen genomweiten Screen, da der Effekt der RNAi systemisch ist, die Antwort also in allen Geweben des Körpers, sowie bei parentaler RNAi von Weibchen auch in den Nachkommen, herbeigeführt wird (Brown et al.,1999; Bucher et al., 2002; Tomoyasu & Denell, 2004). In der Tat konnte ich für 57 % der von mir pupal injizierten dsRNAs in den Folgeanalysen mindestens einen Defekt feststellen (Abb.10). Betrachtet man die ~ 3400 dsRNAs 112 Diskussion des gesamten pupalen Screens, so beläuft sich die Zahl der Phänotypen auf 45,7 % (Schmitt-Engel et al., in Vorbereitung). Dies zeugt auf den ersten Blick für einen erstaunlich hohen Output im Vergleich zu Mutagenesescreens in Drosophila (Nüsslein-Volhard & Wieschaus, 1980; Nüsslein-Volhard, Wieschaus, & Kluding, 1984). Vor allem, wenn man bedenkt, dass Gene mit funktioneller Redundanz, d.h. Gene, deren Ausschalten durch ein anderes Gen ausgeglichen wird, Gene mit pleiotropen Funktionen sowie Gene, deren Funktionen auf unterschiedlichen Spleißvarianten beruhen, womöglich übersehen wurden. Im Vergleich mit anderen Screens muss jedoch beachtet werden, dass die meisten dieser Screens sich auf nur einen Aspekt konzentrierten, während iBeetle ein breites Spektrum von Prozessen untersucht, darunter Defekte der Segmentierung, aber auch Muskeldefekte, Störungen der Oogenese oder Stinkdrüsen. Weiterhin handelt es sich bei iBeetle um einen „First pass“-Screen. Jedes dsRNA-Fragment wird also nur ein einziges Mal injiziert. Mögliche falsche Annotierung eines Phänotyps, die auf Krankheiten der injizierten Weibchen, technische und menschliche Fehler oder so genannte „off-target“-Effekte zurückgehen, sind daher bei diesen Zahlen noch nicht einbezogen worden. Solche „off-target“-Effekte wurden bereits bei RNAi-Screens in anderen Modellsystemen beobachtet und basieren auf einer Bindung der kurzen siRNAs, die während der RNA-Interferenz entstehen, mit zielfremden mRNAs, die dann ebenso abgebaut werden (Jackson & Linsley, 2004). Eine vergleichende Studie der Genome des Pilzes Schizosaccharomyces pombe, des Wurms C. elegans und menschlicher Zellkulturen schätzt, dass circa 10 % der vorhergesagten siRNAs „off-target“-Effekte auslösen können (Qiu et al., 2005). Die Mechanismen der RNAi in Tribolium sowie „off-target“-Effekte sind bisher kaum untersucht bzw. verstanden (Tomayasu et al., 2008). Im Rahmen des iBeetle-Screens wurden nun „off-target“-Effekte zum ersten Mal auch für Tribolium beobachtet. Trotz dieser Einschränkungen konnte ich für alle im pupalen Screen untersuchten Prozesse vielversprechende Kandidatengene identifizieren. So führten 6 % (58) der von mir injizierten dsRNAs zu einem Ovarphänotyp und 2 % (19) zu einer erkennbaren Störung der Muskulatur in der F1-Generation. Dieser niedrige Prozentsatz an Muskelphänotypen ist vergleichbar mit einem RNAi-Screen in Drosophila, bei dem ~ 1 % der Gene (17 % der 436 Gene, ausgehend von 10,461 vorhandenen Genen; 113 Diskussion Schnorrer et al., 2010) morphologisch erkennbare Defekte der larvalen Muskulatur aufwiesen. Es wäre dennoch möglich, dass aufgrund pleiotroper Genfunktionen nicht alle relevanten Gene für diese Prozesse entdeckt worden sind. So könnte zum Beispiel der Tod der injizierten Weibchens, wie er in 89 Fällen (9 %) auftrat, jegliche Funktion des gleichen Gens in der Embryogenese (z.B. Muskelentwicklung) verschleiern. In diesem Falle wäre eine vertiefende Untersuchung der dsRNAs notwendig, etwa mittels eRNAi, weil durch die Injektion in Embryonen die Mutter und folglich auch Oogenesefunktionen umgangen werden (Brown et al., 1999). Prinzipiell kann eine Funktion in der frühen Musterbildung die Detektion späterer Phänotypen (z.B. im Nervensystem) immer verhindern oder zumindest erschweren (St Johnston, 2002). Neben der Suche nach Kandidatengenen für die Oogenese und die Entwicklung der larvalen Muskelentwicklung lag das Hauptaugenmerk des pupalen Screens in der Analyse der F1-Generation, um neue für die Embryonalentwicklung relevante Faktoren zu identifizieren. Von meinen 987 Injektionen resultierten 21 % (206) in Kutikulaphänotypen und weitere 8 % (81) der dsRNAs in leeren Eiern, unter denen ebenfalls Faktoren der frühen Achsenentwicklung (antero-posterior und dorso-ventral) sein könnten (Kotkamp et al., 2010; Fu et al., 2013). Ausgehend von der Konservierung wichtiger Signalwege und Prozesse innerhalb der Metazoa, und basierend auf Zahlen bei Drosophila, wird geschätzt, dass circa ein Drittel aller Gene essenziell für die Viabilität einer Art sind (Nüsslein-Volhard, 1994; Miklos & Rubin 1996). Vergleicht man diese Schätzung mit den Zahlen aus dem Screen (Segmentierungsphänotypen, „empty eggs“ und adult letale Gene) so liegen diese mit ca. 40 % über denen von Drosophila. Rechnet man allerdings pleiotrope Effekte sowie mögliche „off-target“-Phänotypen mit ein ergibt sich auch für Tribolium eine ähnliche Größenordnung von ~30 % letaler Gene. Zusammenfassend kann festgehalten werden, dass der iBeetle-Screen seine Ziele erreicht hat. Es konnten nicht nur in allen Prozessen neue Gene gefunden werden (Abb. 10-13), sondern viele dieser Gene wären durch den Homologie-basierenden Kandidaten-Gen-Ansatz nicht in Betracht gezogen worden. Zudem wurden mit der Plattform Tribolium neue Forschungsfelder, wie die Stinkdrüsen-Entwicklung, entwickelt (Schmitt-Engel et al., in Vorbereitung). Dies etabliert Tribolium weiter als Modellsystem und liefert Ansatzpunkte für zahlreiche Forschungsrichtungen, wie 114 Diskussion etwa im Bereich der Schädlingsbekämpfung (Klingler, 2004; Schröder et al., 2008; Brown et al., 2009). 4.1.2 Grundlagen des Erfolgs von iBeetle Das Fundament für ein Projekt wie iBeetle ist zum einen die professionelle Analyse der genomischen Daten und die Auswahl der optimierten dsRNA-Fragmente (Snove & Holen, 2004; Arziman et al., 2005) und zum anderen die Produktion der verwendeten dsRNAs in einem Verfahren, das konstant hohe Qualität gewährleistet. Dies übernahm die Firma Eupheria, die über ein durchdachtes Qualitätsmanagement und ein angepasstes PCR-Verfahren saubere dsRNA von 1 µg/µl für iBeetle lieferte. Auf Ebene der Screener musste auf eine möglich einheitliche Annotation der Phänotypen und gleichbleibende Qualität geachtet werden. Basis hierfür war ein einwöchiges Kick-off-Meeting zu Beginn des Screens, bei dem neben den nötigen praktischen Kenntnissen (Injektionen, Präparationen etc.) die theoretischen Hintergründe der zu untersuchenden Prozesse vermittelt worden sind. Im Verlauf des Screens wurden in regelmäßigen Konferenzschaltungen der beiden Screening-Zentren Erlangen und Göttingen mögliche Probleme und schwierige Phänotypen besprochen sowie die Datenbank des Screens (http://ibeetle-base.uni-goettingen.de) entwickelt. Zur Vereinheitlichung der Phänotypbeschreibungen wurden für die Datenbank „Drop down“-Menüs entwickelt mit einem festen Satz an Begriffen. Die Zahl dieser genormten Begriffe und deren Modifizierungs-Möglichkeiten wurde dabei so gering wie möglich gehalten, um das spätere Durchsuchen der Datenbank so einfach und eindeutig wie möglich zu gestalten. Zudem gab es die Option, getestete dsRNAs/Gene als interessant für ein bestimmtes Thema, z. B. Segmentierung, zu markieren. Eine weitere Instanz der Kontrolle war die stichprobenartige Analyse von Phänotypen durch fachkundige PIs vor Ort. Pro Injektionstag wurden 22 Gene plus zwei Kontrollen injiziert. Fünf dieser Sets bildeten ein „Bundle“, und pro Bundle galt es, fünf willkürlich gewählte Ovare, Kutikula-bzw. Muskelpräparate mit einem erfahrenen PI durchzugehen und zu besprechen. Puffer- und Positivkontrollen mit bereits bekannten Genen boten die Möglichkeit, die Aufmerksamkeit der Screener über den gesamten Zeitraum des Screens zu über- 115 Diskussion prüfen. Insgesamt wurden 85-95 % dieser Positivkontrollen richtig erkannt, wobei die Erkennungsrate im pupalen Screen höher war als im larvalen. Teilweise wurde nur ein Aspekt des Phänotyps übersehen, oder das Nichterkennen des Phänotyps ging auf technische Gründe, wie zum Beispiel den frühen Tod der Weibchen zurück. Pufferkontrollen wurden im pupalen Screen zu weniger als 1% falsch annotiert, und von 37 bereits bekannten Genen, deren dsRNAs im Verlauf des pupalen Screens zufällig injiziert worden waren, wurden alle erkannt (für Details siehe Schmitt-Engel et al., in Vorbereitung). All dies deutet darauf hin, dass zumindest hochpenetrante und einigermaßen gut erkennbare Phänotypen in den allermeisten Fällen auch detektiert wurden. 4.2 Die Rolle von genetischem Hintergrund und „off-target“Effekten bei der Verifizierung der Kandidatengene Wie in Abbildung 10 gezeigt, führten ~ 20 % der dsRNAs im pupalen Screen zu Defekten der Segmentierung, und weitere 8 % der Gene resultierten in Eiern ohne jegliche Kutikula, welche durchaus auf starke Defekte von Segmentierungsgenen hinweisen können (Kotkamp et al., 2010; Fu et al., 2013). Da die Klasse der „empty eggs“ in einem separaten Projekt untersucht werden sollte, beschränkte ich mich auf die iBNummern mit reduzierter Schlüpfrate und die als im Verlauf des Screens als besonders interessant für Segmentierung bzw. antero-posteriore Achsenentwicklung markiert worden sind. Nach der Durchsicht der 1006 erhaltenen Datensätze blieben 258 iB-Nummern über, von welchen ich 16 im Rescreen (Injektion des iB-Fragments in den gleichen Stamm in zwei verschiedenen Konzentrationen sowie in den Stamm San Bernadino) testete. Da eine gewisse Zahl an „false positives“ nicht ausgeschlossen werden kann, war es nicht verwunderlich, dass im „re-screening" dieser 16 dsRNAs (PBA19, OriginalFragment) zwei Phänotypen nicht reproduzierbar waren (ca. 12 %). Dies deckt sich auch mit den Zahlen der anderen Arbeitsgruppen, die insgesamt 83 % der im Screen aufgetretenen Phänotypen reproduzieren konnten (Schmitt-Engel, in Vorbereitung). Interessanter ist die Tatsache, dass der Rescreen Phänotypen und somit Genfunktionen offenlegte, die anscheinend stammesspezifisch sind. So zeigten 11 % von 210 getesteten dsRNAs einen signifikanten Unterschied in verschiedenen genetischen 116 Diskussion Hintergründen (PBA19 vs. SB; Schmitt-Engel, in Vorbereitung). Obwohl ein solcher Effekt des genetischen Hintergrunds bereits für Drosophila, Mus musculus und sogar Saccharomyces cerevisiae nachgewiesen war (Threadgill et al., 1995; Dworkin et al., 2009; Dowell et al., 2010), sind dies die ersten fassbaren Hinweise auf die Relevanz des gewählten Hintergrunds in Tribolium. Die Gruppe von G. Bucher (Göttingen) beschäftigte sich im Zuge dieser Ergebnisse des Screens ausführlicher mit diesem Phänomen und konnte für Tc-importin α1 unterschiedliche Klassen von Phänotypen in Abhängigkeit vom verwendeten Stamm (SB oder BLACK) feststellen, die nicht auf „off-target“-Effekte oder Ähnliches zurückgehen (Kitzmann et al., 2013). Diese Ergebnisse zeigen also, dass auch bei Tribolium der genetische Hintergrund die phänotypische Ausprägung eines Gens beeinflusst - was bei zukünftigen genetischen Untersuchungen in diesem Modellsystem bedacht werden muss (Gibson & Dworkin, 2004). Wie bereits erwähnt, ist RNA-Interferenz generell anfällig für „off-target“Effekte, da die siRNA-Fragmente im RISC-Komplex relativ kurz sind, und zudem auch eine Wirkung auf Ziel-mRNAs haben können, deren Sequenz nicht perfekt komplementär ist (Qiu et al., 2005; Ma et al., 2006). Um „off-target“-Effekte auszuschließen, wurden für die Gene im Rescreen neue, nicht-überlappende dsRNAFragmente konzipiert und in den Originalstamm injiziert. Geht der beobachtete Phänotyp auf einen „off-target“-Effekt zurück, so dürfte mit diesem NoF der Phänotyp nicht auftreten. Im Gegenzug bestätigt das Auftreten des Phänotyps, dass er in der Tat spezifisch ist für die Funktion des Zielgens. Überraschenderweise konnten von den sechzehn Kandidatengenen nur vier verifiziert werden: Tc-germ cell-less (iB_02693), Tc-homeobrain (iB_04564) und die Tribolium-Homologe von tout-velu (iB_03835) und RhoGef2 (iB_03492). Im Gegensatz dazu wurden im gesamten Screen 87 % von 107 getesteten Genen reproduziert. Diese Zahlen liegen im Bereich der 12,5 % „off-target“-Effekte, die in Drosophila mit künstlichen micro-RNAs beobachtet werden konnten (Ni et al., 2011) und repräsentieren wohl die eigentliche Erfolgsrate des Screens, da Phänotypen aller Klassen geprüft worden sind. Die Ursachen für meine geringe Verifikationsrate von 25 % sind ungeklärt, aber meine Screener-Kollegen Jonas Schwirz und Upalparna Majumdar, die ebenfalls embryonale Kutikulaphänotypen analysierten, erhielten recht ähnliche Zahlen (pers. 117 Diskussion Kommunikation). Erste Untersuchungen von U. Majumdar zeigen allerdings, dass unterschiedliche Phänotypen der NoFs eines Gens auf die Stärke des RNAi-Effekts zurückzuführen sein könnten. Als sie die NoFs untersuchte, die in „empty eggs“ oder kaum identifizierbaren Kutikularesten resultierten, zeigte sich nach einiger Zeit mit Abnahme des RNAi-Effekts teilweise ein ähnlicher Phänotyp wie er im Screen annotiert worden war (pers. Kommunikation). Diese Ergebnisse zeigen, dass auch bei der Auswertung der NoFs Phänotypen fälschlicherweise ausgeschlossen werden können, weshalb in Zukunft der zeitliche Aspekt der RNAi-Antwort in die Analyse der Kandidatengene miteinbezogen werden sollte. In den letzten Jahren stieg die Zahl der Studien über die Spezifität der RNAi (Elbashir et al., 2002; Kumar et al., 2003; Beermann et al., 2004; Tomoyasu & Denell, 2004; Trauner & Büning, 2007; Clark-Hachtel et al., 2013). Ein spezielles Augenmerk liegt dabei auf der Konzipierung der RNA-Fragmente auf genomischer Ebene, aber auch dem Zusammenhang zwischen Position im Gen und RNAi-Aktivität (Kumar et al., 2003; Holen, 2005). So kann zum Beispiel die Ratio bestimmter Basen ebenso ausschlaggebend sein für die Aktivität der RNAi wie die Base an erster Position (Jagla et al., 2005). Fest steht, dass der Erfolg von RNAi-Studien wie iBeetle vom Design der RNA-Fragmente abhängig ist, und obwohl modernste InformatikAnalyse-Techniken herangezogen wurden, können „off-target“-Effekte nach wie vor nicht ausgeschlossen werden, weshalb Webtools und Parameter beständig neu angepasst werden müssen (Horn & Boutros, 2010). Im Falle der Gene, bei denen die Injektion der NoFs nur in Wildtyp-Larven oder in ganz anderen Defekten bei der ursprünglichen iBeetle-RNA resultierten, könnte neben „off-target“-Effekten, auch das Vorhandensein von mehreren Isoformen eine Erklärung sein. So wiesen Arakane et al. 2005 über die Analyse der Phänotypen unterschiedliche Funktionen für die beiden Spleißvarianten des Chitin-Synthase Genes TcCHS1 nach. Im Screen lieferte z. B. auch die Injektion der dsRNAs spezifisch für Tc-knirps andere Phänotypen als veröffentlicht, während das nicht-überlappende Fragment den publizierten Phänotypen (Cerny et al., 2008) generierte (Schmitt-Engel et al., in Vorbereitung). In der Tat kam es in zwei Fällen zum Auftreten von „empty eggs“ nach Injektion der NoFs (iB_01719 und iB_3859). Dies könnte ein Hinweis auf einen stärkeren Phänotyp sein, da RNAi gegen Segmentierungsgene zur frühen Letalität führen kann 118 Diskussion (Kotkamp et al., 2010; Fu et al., 2013). Denkbar wäre auch, dass der Phänotyp des NoFs auf einem „off-target“-Effekt beruht, der den genuinen Segmentierungsphänotyp überdeckt. Es wäre daher möglich, dass die Zahl der verifizierten Phänotypen durchaus höher liegt als gedacht. Aufklärung könnte hier nur die detaillierte Analyse mehrerer dsRNA-Fragmente für jedes Gen bzw. eine Studie der Phänotypen in Abhängigkeit der Konzentration liefern, was im zeitlichen Rahmen des Rescreens nicht machbar war. 4.3 Doppelabdomina im Kurzkeim-Insekt Tribolium Im Zuge von iBeetle wurden bislang drei Gene identifiziert, die in Doppelabdomen resultieren, d.h. die Nachkommen der RNAi-behandelten Weibchen entwickeln statt Kopf und Thorax ein zusätzliches Abdomen, das spiegelbildlich zum normalen Abdomen ausgebildet wird. Das erste war die Positivkontrolle teashirt – ein Gen, dessen publizierter Knockdown in Tribolium in relativ milden larvalen Defekten in Kopf und Thorax bestand (Shippy et al., 2008). Als Positivkontrolle für den Screen wurde ein anderes dsRNA-Fragment generiert, das den Doppelabdomen-Phänotyp generierte. Allerdings wies dieser nur eine geringe Penetranz auf und konnte nicht mit dem nicht-überlappenden Fragment verifiziert werden. Auch der Test benachbarter Gene blieb ohne Erfolg, weshalb der Phänotyp als „off-target“-Effekt betrachtet werden muss und nicht weiter untersucht wurde. Im Gegensatz dazu konnte der Doppelabdomen-Phänotyp für Tc-germ cell-less (iB_02693) und Tc-homeobrain (iB_04564) im Rescreen sowohl für die nichtüberlappenden Fragmente als auch in allen getesteten Stämmen (PBA19; SB und Black; nicht gezeigt) reproduziert werden. 4.3.1 Der Ausbildung von Doppelabdomen-Phänotypen liegen in Tribolium andere Mechanismen zugrunde als in Drosophila Im Zuge dieser Arbeit konnte ich zeigen, dass germ cell-less und homeobrain im unterschiedlichen Maß an der Ausbildung der anterio-posterioren Achse in Tribolium beteiligt sind. Im Falle von Tc-gcl kommt es nach parentaler RNAi in der Regel zum Verlust der im Blastoderm angelegten Segmente von Kopf und Thorax. Stattdessen entstehen anterior im Embryo spiegelsymmetrisch angeordnete abdominale Segmen- 119 Diskussion te inklusive Terminus. Komplette Abdomen sowie die Ausprägung eines duplizierten T3-Segments, und somit einer vollständigen „segment addition zone“ sind möglich (Abb. 16). Nach Tc-hbn-RNAi hingegen bleiben sowohl am normalen als auch am neuen zweiten Abdomen i.d.R. thorakale Segmente vorhanden. Zudem bleibt das neue Abdomen stets unvollständig (Abb. 18). Solche spiegelbildlichen Doppelabdomen sind bis dato noch nicht für das Kurzkeim-Insekt Tribolium beschrieben. In Drosophila hingegen wurden die ersten Doppelbadomen 1966 entdeckt. Die Mutation erhielt den Namen bicaudal (Bull, 1966). Aufgrund geringer Penetranz ruhte die Erforschung dieser Mutante jedoch für mehrere Jahrzehnte. Heutzutage ist mit beta NAC, einer Untereinheit des Nascent polypeptide Associated Complex (NAC) das zugehörige Gen dieser Mutation identifiziert und die Ursache des Phänotyps, ektopische Translation von nanos, entschlüsselt (Markesich et al., 2000). Auch die Mutationen Bicaudal C und D, ik2, bullwinkle, alice und Klp38B sowie 4 weiterer Loci resultieren zum Teil in Doppelabdomen. Dabei ist auffällig, dass stets Kopf, Thorax und die ersten 3-5 abdominalen Segmente verloren gehen und durch eine ähnliche Anzahl spiegelsymmetrisch-angelegter posteriorer abdominaler Segmente, inklusive Terminus, ersetzt werden (NüssleinVolhard, 1977; Mohler & Wieschaus, 1986; Wharton & Struhl, 1989; Mahone et al., 1995; Rittenhouse & Berg, 1995; Ruden et al., 1997; Luschnig et al., 2004; Shapiro & Anderson, 2006). Auch in anderen Langkeim-Insekten sind solche unvollständigen Doppelabdomen bekannt, so entwickeln Embryos der Fliegen Megaselia abdita (Stauber et al., 2000) und Episyrphus balteatus (Lemke et al., 2010) nach bicoid-RNAi Doppelabdomen von ~ 4 Segmenten auf jeder Seite - ein Phänotyp, der in Drosophila nur durch das gleichzeitige Ausschalten von bicoid und hunchback erreicht werden kann. Der Verlust von bicoid allein hat in Drosophila indes nur das Auftreten eines zweiten Telsons (posteriorer Terminus) zu Folge (Frohnhöfer & Nüsslein-Volhard, 1968; Gavis & Lehmann, 1992; Simpson-Brose et al., 1994). Trotz gewisser Unterschiede wird in all den erwähnten Fliegenarten die anteroposteriore (auch die dorso-ventrale) Achse über eine präzise zeitlich und räumlich kontrollierte Expression von Proteinen etabliert. Die a-p-Polarität des Embryos wird dabei bereits im Verlauf der Oogenese über die Lokalisierung maternaler mRNAs von bicoid (anterior) und nanos (posterior) festgelegt (Driever & Nüsslein-Volhard, 120 Diskussion 1988a; Ephrussi et al., 1991; Lasko, 2011). Mit der Befruchtung des Eis werden diese RNAs translatiert und die oppositionellen Proteingradienten regulieren die Transkription bzw. Translation weiterer Gene/mRNAs (vgl. Einleitung 1.1.1). In Drosophila reguliert die Konzentration von BICOID die hunchback-Expression und die Transkription weiterer zygotischer Gene, die für die Bildung anteriorer Segmente notwendig sind (Struhl et al., 1989). NANOS fungiert posterior als Repressor für bicoid- und hunchback-mRNA und initiert zusammen mit CAUDAL die Ausbildung posteriorer Strukturen (Schultz & Tautz, 1995). nanos wird dabei von OSKAR an den posterioren Pol rekrutiert (Ephrussi et al., 1991). Kommt es nun zu einer Mislokalisation der oskar-mRNA an den anterioren Pol, wie z. B. im Falle der Mutation von Bicaudal C oder ik2 wird folglich nanos anterior exprimiert (Mahone et al., 1995; Shapiro & Anderson, 2006). Auch in Bicaudal Dbzw. bicaudal-mutierten Fliegen wird nanos, unabhängig von OSKAR, ektopisch im gesamten Embryo exprimiert (Wharton & Struhl, 1989; Markesich et al., 2000). Dies hat die Repression von bicoid und hunchback zur Folge – der beiden Signalgeber für anteriore Schicksale. Mit dem Verlust von BICOID (und HUNCHBACK) wird auch die Translation der caudal-mRNA am anterioren Pol nicht mehr inhibiert. Es werden also über NANOS und CAUDAL in der anterioren Hälfte des Eis die gleichen Gene wie in der posterioren Hälfte aktiviert (Nüsslein-Volhard & Wieschaus, 1980; Nüsslein-Volhard et al., 1987; Driever & Nüsslein-Volhard, 1988a, Simpson-Brose et al., 1994, Lasko 2011). In den untersuchten Langkeim-Insekten beruhen bicaudalPhänotypen also auf einer fehlerhaften Lokalisation des maternalen Faktors nanos bzw. einer Repression von bicoid (und hunchback). Die spiegelbildliche Polarität dieser Eier wird bei all diesen Phänotypen vom terminalen System hergestellt, da BICOID, CAUDAL und NANOS nicht mehr als Gradienten exprimiert sind. Wie bereits erwähnt, konnte im Kurzkeim-Insekt Tribolium castaneum kein Ortholog von bicoid identifiziert werden (Brown et al., 2001). Tc-hunchback ist zwar vorhanden, parentale RNAi resultiert aber nicht in Doppelabdomen, sondern in einer homöotischen Transformation von gnathalen und thorakalen Segmenten zu abdominalem Schicksal sowie posterioren Segmentierungsdefekten. Der anteriore Kopf und die Serosa sind normal ausgebildet (Schröder, 2003; Cerny, 2007). Bezüglich der posterioren Gruppe blieb meine Suche nach einem Oskar-Ortholog in den Transkriptomdatenbanken von Beetlebase, OrthoDB und iBeetle erfolglos. Tc- 121 Diskussion nanos wurde hingegen 2010 beschrieben, scheint im Vergleich zu Drosophila jedoch eine weniger bedeutende Funktion in der Segmentierung einzunehmen. Zum einen resultiert der Knockdown von nanos nicht im kompletten Verlust der aus der „segment addition zone“ hervorgehenden Segmente, da außer dem Thorax immer noch 12 abdominale Segmente entstehen. Zum anderen war es bisher nicht möglich, eine lokalisierte Expression von nanos nachzuweisen (Schmitt-Engel, 2010; SchmittEngel et al., 2012). caudal hingegen wird im Blastoderm posterior exprimiert und markiert im Laufe der Entwicklung die posteriore „segment addition zone“. In caudal-RNAi Larven sind nur noch Labrum, Augen und Antennen vorhanden, was auf eine essentielle Rolle caudals in der Etablierung der antero-posterior-Achse schließen lässt (Copf et al., 2004). In Drosophila wird maternales caudal von BICOID (Mlodzik & Gehring, 1987), zygotisches hingegen von HUNCHBACK inhibiert (Schulz & Tautz, 1995). Im Gegensatz dazu sind in Tribolium augenscheinlich zwei zygotische Gene für die Repression des maternalen caudals verantwortlich: Mex-3 und Zen-2. Bei Abwesenheit dieser beiden kommt es jedoch nicht zur Ausbildung von Doppelabdomen, sondern lediglich zum Verlust anteriorer Segmente (Schoppmeier et al., 2009). Es kann also davon ausgegangen werden, dass die spiegelsymmetrischen Doppelabdomen in Tribolium nicht auf einer bloßen Mislokalisation von nanos oder caudal beruhen – obwohl caudal im Blastoderm nach Tc-gcl-RNAi (Abb.26) bzw. im frühen Keimstreif nach Tc-hbn-RNAi (Abb. 28) auch anterior exprimiert wird. 4.3.2 Tc-germ cell-less und Tc-homeobrain arbeiten mit dem anterioren terminalen System, um anteriore Schicksale einzuleiten In Drosophila ist das terminale System für die unsegmentierten terminalen Strukturen Akron und Telson verantwortlich, sowie für die posteriorsten abdominalen Segmente (vgl. 1.1.1). Das terminale System basiert auf der lokal begrenzten Aktivierung des über die Oozytenmembran verteilten Tyrosin-Kinase-Rezeptors TORSO durch den an den Polen an der Vitellinmembran lokalisierten Liganden TORSOLIKE (Casanova & Struhl, 1989; Sprenger et al., 1989; Stevens et al., 1990; Nüsslein-Volhard, 1991, Stevens et al., 2003). Durch eine Ras/Raf/MAPK-Kaskade werden daraufhin an beiden Polen die zygotischen Gene tailless und huckebein exprimiert (Brönner und Jäckle, 1991; Furriols & Casanova, 2003). Die polaren Domänen 122 Diskussion von tailless sind im synzytialen Blastoderm noch identisch, mit Zellularisierung des Embryos verändert sich die Expression von einer anterioren Kappe zu einem anterior-dorsalen Streifen (Pagnini et al., 1992). In Abwesenheit von BICOID bleibt die anteriore tailless-Domäne jedoch unverändert, und anstelle der anterioren terminalen Struktur Akron entsteht auch anterior ein Telson (Frohnhöfer & Nüsslein-Volhard, 1968; Pagnini et al., 1992). Wie beschrieben (vgl. 1.1.2) weist das Tribolium-Blastoderm einen unvollständigen Anlagenplan auf. Statt aller Segmente (wie in Drosophila) werden nur die extraembryonale Serosa, Kopf, Thorax und „segment addition zone“ angelegt (Seidel et al., 1940; Lohs-Schardin et al., 1979). Aufgrund des veränderten Anlagenplans übernimmt das terminale System in Tribolium eine wichtigere Rolle. Sowohl die Serosa als auch die SAZ unterliegen der Kontrolle des terminalen Systems (Schoppmeier & Schröder, 2005). Über die Regulierung des terminalen Systems ist in Tribolium jedoch nur wenig bekannt. Wie in Drosophila ist torso zwar ubiqitär aktiv und torsolike in den Follikelzellen an beiden Polen exprimiert (Schoppmeier & Schröder 2005), was für einen konservierten Mechanismus spricht. Auch tailless ist in Tribolium vorhanden und wird vom Terminalen System reguliert. Es wird allerdings nur am posterioren Pol exprimiert (Schröder et al., 2000). huckebein scheint nicht am terminales System beteiligt (Kittelmann et al., 2013). Betrachtet man nun die spiegelbildliche Symmetrie der Doppelabdomen, so ist offensichtlich, dass der Phänotyp von gcl und hbn auf das Vorhandensein einer zweiten „segment addition zone“ am anterioren Pol des Embryos zurückzuführen ist. Der anlagenplan ändert sich also von Serosa-Kopf-thorax-SAZ zu SAZ-(Thorax)-SAZ ändert. Beide scheinen also im Wildtyp am anterioren Pol das terminale System so zu modifizieren, dass statt SAZ anteriore Strukturen gebildet werden. In der Tat zeigt die Expressionsanalyse posteriorer Marker nach gcl-RNAi bereits im Blastodermstadium bestehende Spiegelsymmetrien auf. So wird caudal im reifen Blastoderm nicht nur in der posterioren Hälfte des Eis exprimiert (Copf et al., 2004), sondern ubiquitär. vasa findet sich außer am posterioren (Schröder, 2006) nun auch am anterioren Pol, und auch die posteriore Expression von wingless, die die „segment addition zone“ markiert (Nagy & Carroll, 1994; Bolognesi et al., 2008), ist dupliziert. Weiterhin ist schon im Blastoderm die Bildung des „posterior pit“ an beiden Polen auffällig (Abb. 26 und 27). Den Verlust anteriorer Identitäten bestätigt das 123 Diskussion Fehlen der zerknüllt-Expression, als Marker für die Anlage der Serosa (van der Zee et al., 2005), und der anterioren Domänen von iroquois, giant und wingless – Marker für Amnion, gnathale Segmente und Kopflappen (Abb. 27 und Abb. 29; Nagy & Carroll, 1994; Bucher & Klingler, 2004; Sharma et al., 2013). Die frühe Funktion von gcl als Identitätsgeber des terminalen Systems scheint zudem durch die Expressionsdaten von gcl bestätigt. Wie ich in dieser Arbeit zeigen konnte, findet sich gcl-mRNA bereits im frisch abgelegten sowie in unbefruchteten Eiern als Domäne, welche die anteriore Hälfte des Embryos einnimmt. gcl wird also maternal bereitgestellt (Abb. 20). Im Gegensatz zu gcl ist hbn erst später im undifferenzierten Blastoderm als anteriore Kappe exprimiert (Abb. 21). Dies spricht für eine zeitliche Differenzierung der Funktion beider Gene. Tatsächlich wird bereits bei der Analyse der Kutikulas deutlich, dass nicht nur der Phänotyp schwächer ist – es kommt nie zur Ausbildung kompletter Abdomen-, sondern auch die Penetranz von 100 % wie bei gcl-RNAi wird nicht erreicht (was auch die Expressionsanalyse einiger Marker erschwert, Vergleiche Abb.16-19). Ein klarer Unterschied zu gcl RNAi ist jedoch, dass vasa nur posterior - also wildtypisch - exprimiert wird (Abb. 26). caudal, even-skipped und der Amnionmarker iroquois zeigen eine Expansion bzw. Verschiebung der Expressionsdomänen um ein gutes Viertel Eilänge nach anterior (Abb. 26 und Abb. 29). Auch wird zen-1 in hbn-RNAi-Embryonen exprimiert. Die Domäne ist aber, in Übereinstimmung mit der Expansion der posterioren Marker, stark reduziert (Abb. 29). Eine Funktion des Transkriptionsfaktors hbn ist also, dass es offenbar an der Aktivierung von zen-1 beteiligt ist. Mit Bildung des Keimrudiments bzw. im sehr jungen Keimstreifen zeigt sich neben der posterioren cad-Domäne eine zweite kleinere Domäne auf Höhe des Kopfes (Abb. 28). Diese kennzeichnet offenbar eine weitere „segment addition zone“ (Copf et al., 2004), aus der während der Elongation abdominale Segmente auswachsen. Die spätere Etablierung bzw. die unterschiedlichen zugrunde liegenden Mechanismen könnten erklären, warum nach hbn-RNAi weniger Segmente ausgebildet werden als nach gcl-RNAi. Während gcl an der frühen Charakterisierung des anterioren terminalen Systems beteiligt ist, scheint hbn downstream aktiv zu sein, um anterior die WntGene (wg1/wg8) zu reprimieren, die am posterioren Pol für die die Etablierung der „segment addition zone“ und somit caudal-Expression notwendig sind (Bolognesi et 124 Diskussion al., 2008). Ebenso könnte die Aktivierung von zen-1 auf dieser Funktion beruhen, da Fu et al. 2013 zeigten, dass die RNAi von axin, dem Repressor von ß-catenin und somit des Wnt-Signalwegs eine Reduzierung der zen-1-Expression zur Folge hat. 4.3.3 Wo wirkt Tc-gcl? In Drosophila und Mus musculus ist GCL nachweislich an der inneren Kernmembran lokalisiert und fungiert als Transkriptionsrepressor indem es andere Transkriptionsfaktoren an die Membran rekrutiert und so daran hindert, an ihren Zielpromotor zu binden (Jonges et al., 1994; de la Luna et al., 1999; Nili et al., 2001a). Obwohl die intrazelluläre Lokalisation von GCL in Tribolium weder mit sechs induzierten Antiseren noch mithilfe der Injektion einer gcl-egfp mRNA möglich war (vgl. 3.3.9), ist nach wie vor wahrscheinlich, dass Tc-GCL eine ähnliche molekulare Funktion wie seine Homologen in Drosophila und in Vertebraten innehat, zumal die BTB/POZProteinbindedomäne (Bardwell & Treismann, 1994; Abb. 23) konserviert ist. Es wäre daher denkbar, dass GCL am anterioren Pol einen noch unbekannten ubiquitären Transkriptionsfaktor an die Kernmembran rekrutiert, so dass anterior andere zygotische Gene exprimiert werden als posterior. gcl ist im Tribolium-Embryo maternal als anteriore Domäne exprimiert, so dass durchaus Ähnlichkeiten zur Wirkungsweise von Dm-bicoid auf das terminale System bestehen könnten. Aber letztendlich basiert die Transformation des terminalen Systems durch Tc-gcl wohl auf einer anderen Funktion, da mehrere Punkte gegen dieses Modell sprechen: Zum einen zeigt die Analyse der eve-Expression nicht nur eine Duplikation am anterioren Pol, sondern auch eine Kompression und leichte d-v-Verschiebung der Streifen (Abb. 26), was dafür spricht, dass die maternale Positionsinformation im ganzen Embryo verändert ist, nicht nur die Interpretation des terminalen Signals. Ebenfalls auffällig sind die missgestalteten Keimstreifen, die nach gcl-RNAi auftreten können (Abb. 33). Dies wird insbesondere anhand der wg-Färbung deutlich. Statt der segmentalen Streifen kommt es in einigen Fällen nach gcl-RNAi zu einer Aufspaltung entlang der Mittellinie, so dass mittig „Löcher“ im Gewebe entstehen. Weiterhin deuten die starken kugelartigen Phänotypen nach gcl-RNAi auf einen Verlust dorsalen Gewebes hin (Abb. 16). Diese Defekte sind nicht allein durch die Veränderung der antero-posterioren Achse zu erklären, sondern deuten eher auf eine globale Funktion gcls in der Embryogenese hin. 125 Diskussion Sollte gcl allerdings wirklich erst in der Embryogenese an der Etablierung der Achsen beteiligt sein, bleibt zu klären, warum die embryonale RNAi zu 100 % Letalität der Embryonen führt (leere Eier, vergleiche 3.3.12). Zwar wäre denkbar, dass es wie nach RNAi gegen zen-1 durch den Verlust der Serosa zum Austrockenen der Embryonen kommt (Jacobs et al., 2013). Dem widerspricht allerdings, dass auch ein schwacher gcl-Phänotyp mit Senkung der Konzentration nicht herbeigeführt werden konnte (s. 3.3.12) bzw. keine vermehrte Anzahl von „empty eggs“ in der parentalen RNAi gegen gcl beobachtet wird (Abb. 17). Weiterhin ist auffällig, dass nach gclRNAi nur maternal lokalisiertes axin, nicht aber pangolin detektiert werden konnte (Abb. 39). Fehler in der Methode können hierbei weitgehend ausgeschlossen werden, da die in-situ-Hybridisierung in Erlangen eine etablierte Methode ist und die Färbung mehrfach und immer in Verbindung mit dem wildtypischen Embryonen durchgeführt wurde. Die Ursache für die spiegelsymmetrischen Doppelabdomen nach pRNAi gegen gcl muss daher wohl außerhalb des Embryos, in der Oogenese gesucht werden. In Drosophila fußen die a-p-Achse und die d-v-Achse auf maternal lokalisierten mRNAs, z.B. bicoid und gurken, und der asymmetrischen Positionierung des Oozytenkerns (Becalska & Gavis, 2009). Ein solcher Zusammenhang zwischen der Position des Oozytenkerns und der Etablierung der d-v-Achse wurde auch in anderen Insekten wie Tribolium, Gryllus und Nasonia nachgewiesen (Lynch et al., 2010). Die Rolle maternal lokalisierter mRNAs in der Achsenbildung ist mittlerweile in zahlreichen Insekten untersucht (Becalska & Gavis, 2009). Für Tribolium waren abgesehen von dem maternal anterior lokalisierten axin (Fu et al., 2012) aber noch keine diesbezüglichen Funktionen bekannt, obwohl mit eagle und pangolin noch zwei maternal lokalisierte mRNAs identifiziert worden sind, die allerdings keine Funktion zu haben scheinen (Bucher et al., 2005). Wie unter 4.3.1 aufgeführt, resultiert die Misslokalisation von maternalen Faktoren in Drosophila in spiegelsymmetrischen Doppelabdomen. Ursache dieser Misslokalisation können eine Repression der Gene in den Nährzellen selbst sein, ein gestörter Transport von den Nährzellen in die Oozyte oder fehlerhafte Prozesse in der Oozyte selbst (Review St. Johnston, 1995). In der Tat scheint Tc-gcl in den Nährzellen exprimiert zu sein (Abb. 20). In Drosophila konnte sogar GCL-Protein in der Kernmembran der Nährzellen nachgewiesen werden. Welche Funktion es dort möglicherweise erfüllt ist allerdings noch un- 126 Diskussion geklärt (Jongens et al., 1992, 1994). Die Expression des Proteins könnte dennoch ein Hinweis sein auf eine von der Embryogenese unabhängige Funktion von GCL in Tribolium. Inwieweit GCL in der Zelle lokalisiert ist, konnte wie erwähnt nicht identifiziert werden, da der Einsatz beider polyklonaler Antikörper gegen die synthetischen Peptide von GCL und des polyklonalen Antikörpers gegen Drosophila-GCL trotz mehrfacher Abwandlungen des Protokolls erfolglos blieb (vgl. 3.3.9). Nimmt man die Kernmembran-Lokalisation von Fliege und Maus als Grundlage, so wäre es denkbar, dass Tc-GCL bereits in den Nährzellen die Expression maternaler Faktoren über die Rekrutierung eines Transkriptionsfaktors an die Membran (zeitlich) reguliert. Die Analyse bekannter Bindungspartner (Nili et al., 2001; Holaska et al., 2003; Mansharamani & Wilson, 2005) zeigt, dass die Orthologe zu emerin und LAP2β in Tribolium fehlen oder eventuell wegen molekularer Evolution nicht identifiziert werden konnten). Der Knockdown von MAN1 sowie der Bestandteile des Heterodimers E2F-DP führt indes nicht zu Doppelabdomen (vgl. 3.3.10). Sollte GCL im Ovar die Expression von Zielgenen steuern, müsste dies daher wohl über die Bindung anderer Transkriptionspartner oder über die Regulation mehrerer redundanter Faktoren geschehen. Nach der Synthese in den Nährzellen müssen mRNAs über das Zytoskelett in die Oozyten transportiert und dort ggf. lokalisiert werden. Ist das Zytoskelett gestört oder in der Oozyte nicht richtig polarisiert, kann dies Fehlentwicklungen des Embryos zur Folge haben (Review Fyrberg & Goldstein, 1990; Riechmann & Ephrussi, 2001). In Drosophila-Fliegen, die z.B. im Gen mago nashi mutiert sind, wird das Zytoskelett der Oozyte symmetrisch polarisiert. Dies führt u.a. zu einer Lokalisierung von bicoid-mRNA an beide Polen und letztendlich zu einem Zusammenbruch der Achsen (Micklem et al., 1997). In Tribolium wurde die Abhängigkeit der intrazellulären Lokalisierung von pangolin in der Oozyte von der Polarisierung des Mikrotubuli-Skeletts bereits belegt (Peel & Averof, 2010). Der Verlust der maternalen anterioren Expression von pangolin nach gcl-RNAi könnte zwar auf eine fehlende Lokalisierung hindeuten, sollte dies allerdings auf einem Defekt des Zytoskeletts beruhen, so müsste auch axin betroffen sein (Abb. 39). Neben einem polarisierten Zytoskelett hängt der Transport und die Lokalisierung von maternalen mRNAs, wie z.B. bicoid von der Bildung spezieller Partikel ab. In 127 Diskussion diesen Partikeln sind unterschiedliche Proteine vereint, die an dem Transport und der Verankerung der mRNAs beteiligt sind (Macdonald, 2011). Ein Komplex aus u.a. EXUPERANTIA und YPSILON SCHACHTEL ist in Drosophila an der Lokalisierung von oskar in den Nährzellen und der Oozyte beteiligt (Wilhelm, 2000). GCL könnte in den Nährzellen und im Embryo eine ähnliche Funktion einnehmen, da es durch seine BTB/POZ-Domäne nicht nur andere BTB-POZ-Proteine binden kann, sondern auch Proteine ohne eine solche (Bardwell & Treismann, 1994; Bomont et al., 2000). Wahrscheinlicher ist aber wohl, dass GCL die Expression eines solchen Transport-Faktors in den Nährzellen beeinflusst. Zur Klärung, ob GCL früh in der Oogenese bestimmte Gene reprimiert oder aber über die Bindung von Proteinen an Transport und Lokalisierung von mRNAs beteiligt ist, könnte die pangolin-Expression im Ovar von gcl-RNAi-Weibchen untersucht werden. Je nach Funktionsort dürfte pangolin bereits in den Nährzellen fehlen, oder aber in der Oozyte nicht mehr lokalisiert werden. Die intrazelluläre Lokalisation von GCL und somit der genaue Mechanismus kann indes nur über einen neuen Antikörper oder weitere Versuche mit fluoreszenz-markierten mRNAs geklärt werden. Zusammenfassend kann jedoch festgehalten werden, dass gcl in Tribolium an der Differenzierung der Anlagen beteiligt ist, die aus dem terminalen System hervorgehen und somit ein wichtiger Faktor in der Ausbildung der a-p-Achse ist. Obwohl die genaue Funktion nicht geklärt werden konnte, steht der Phänotyp in Kontrast zu der Rolle von gcl in Drosophila, wo es nur für die Differenzierung der Keimzellen, nicht aber die Segmentierung notwendig ist (Jongens et al., 1992). Aufgrund dieser sehr speziellen Funktion ist gcl bis dato nicht in den Fokus der an Achsenentwicklung interessierten Forscher gelangt, weshalb keine Daten aus anderen Insekten herangezogen werden konnten. Neben anderen basalen Insekten wie Oncopeltus, wäre hierbei auch die Honigbiene Apis mellifera als etabliertes Modellsystem von Interesse, die als Hymenoptera stammesgeschichtlich vor Tribolium (Coleoptera) und Drosophila (Diptera) einzuordnen ist. 4.3.4 Das Auftreten verschiedener Phänotypen deutet auf multiple Funktionen von hbn HBN besitzt zwischen der 65 und der 123 Aminosäure eine Homeodomäne (Abb. 25). Eine solche Domäne ist typisch für Transkriptionsfaktoren, da sie eine spezifi- 128 Diskussion sche DNA-Sequenz erkennt und bindet, und dadurch die Aktivität des betreffenden Gens reguliert (Gehring, 1993). Die Expressionsdaten und die Ergebnisse aus eRNAi und pRNAi weisen auf mindestens zwei Funktionen von hbn in Tribolium hin: erstens in der Etablierung bzw. Aufrechterhaltung anterior versus posterior terminaler Strukturen, sowie eine spezifische späte Funktion in der Ausbildung der procephalen Kopfsegmente Labrum und Antenne (Abb. 18). Entsprechend ist hbn in der Embryogenese durchgehend exprimiert: Im frühen Blastoderm als anteriore Kappe und später in Keimrudiment und Keimstreifen lokal in zwei Streifen in den Kopflappen. Es muss angemerkt werden, dass sich die Expressionsanalysen der Markergene in den Blastodermstadien der hbn-RNAi-Embryonen insgesamt schwierig gestaltete, da die spiegelsymmetrischen Phänotypen nicht so penetrant sind (Vergleiche Abb. 17 und 19). Die Auswertung erfolgte jedoch bestens Gewissens, die Färbungen wurden für jeden Hintergrund mindestens zweimal wiederholt und während der Fixierungen beständig das Vorhandensein der Phänotypen über Kutikula-Präparate überprüft. Im Blastoderm ist hbn zunächst ähnlich wie zen-1 exprimiert (Vergleiche van der Zee et al., 2005). Das Fehlen von hbn hat eine Reduzierung der Serosa zu Folge (Abb. 29), was auf eine Funktion hbns in der Aktivierung von zen-1 deutet. Weiterhin ist eine Expansion caudals zu beobachten (Abb. 26). Ähnliche Ergebnisse konnten bereits nach Mex-3-RNAi beobachtet werden, da auch hier die Expression von caudal vergrößert ist. MEX-3 ist an der Repression von caudal beteiligt (Schoppmeier et al., 2009). Nach Mex-3-RNAi fehlen allerdings die ersten beiden primären eve-Streifen im Blastoderm (Schoppmeier et al., 2009). Der Knockdown von hbn hingegen scheint zu nahezu normal ausgeprägten Streifen zu führen, die wie die caudal-Expression lediglich nach anterior expandiert sind (Abb. 26). Es ist dennoch möglich, dass HBN die antero-posteriore Achse zusammen mit bzw. zum Teil über die anteriore Inhibierung von Mex-3 etabliert. Im frühen Keimstreifen ist hbn in den Kopflappen als ein Streifen entlang der a-pAchse exprimiert, sowie in einer Domäne in der Region von Labrum und Stomodeum, und schwach im mandibularen Segment. Das Expressionsmuster im anterioren Kopf ist somit six-3 ähnlich, dessen Knockdown den Verlust des Labrums zu Folge hat (Posnien et al., 2011). HBN scheint damit eine fehlende Komponenten des komplexen Gennetzwerkes um six-3, crocodile u.a. zu sein, das an der Formation des anterioren Kopfes beteiligt ist (Kittelmann et al., 2013). Eine weitere Expression 129 Diskussion findet sich in der „segment addition zone“. Dieser und der Expression im mandibularen Segment konnte durch die Analyse der RNAi-Kutikula-Phänotypen jedoch keine Funktion zugeordnet werden. In Drosophila ist hbn wie in Tribolium im Blastoderm anterior exprimiert. Durch die unterschiedlichen Anlagenpläne sind in beiden Spezies aber verschiedene Bereiche des Embryos betroffen. Die Kurzkeim-Entwicklung mit der antero-dorsalen Position der Serosa und der posterioren „segment addition zone“ wird als ursprüngliche Form der Entwicklung angenommen (Sander, 1976). Es ist daher wahrscheinlich, dass sich der Anlagenplan Drosophilas aus einer Situation ähnlich der Triboliums entwickelt hat, und viele konservierte Gene bzw. ähnliche Genfunktionen, siehe caudal, untermauern diese Annahme (Copf et al., 2004). Während hbn in Tribolium offenbar als Lückengen die a-p-Achse mitgestaltet, scheint hbn in Drosophila nur an der Bildung spezifischer Regionen des Gehirns beteiligt (Walldorf et al., 2000). Mit Verlust der Serosa, der Verschiebung des Anlangenplans und der Evolution von bicoid als neuen Mustergeber wäre es denkbar, dass es zu einer Spezialisierung von hbn bzw. zu einer Reduktion seiner Zielgene kam. Zur Klärung der Evolution von hbn bedarf es vergleichender Studien, denn bislang ist hbn weder in höheren noch basalen Insekten, wie z.B. Gryllus und Oncopeltus untersucht. 4.3.5 Differenzierung terminaler Identitäten in Tribolium In Drosophila hat BICOID drei Funktionen inne: es organisiert über die Aktivierung von Zielgenen wie orthodenticle und hunchback anteriore Schicksale, inhibiert anterior die Translation von caudal und steuert in Zusammenarbeit mit dem terminalen System anterior die Bildung des Akrons (Mlodzik & Gehring, 1987; St Johnston & Nüsslein-Volhard, 1992; Schulz & Tautz, 1995; Gao & Finkelstein, 1998). In Absenz eines bicoid-Orthologs müssen diese Funktionen in Tribolium von anderen Faktoren übernommen werden. Obwohl gcl im Embryo maternal anterior lokalisiert ist, deutet die fehlende Lokalisation des maternalen pangolins (Abb. 39) und erste Resultate von eagle (nicht gezeigt) nach gcl-RNAi darauf hin, dass gcls bereits in der Oogenese an der Expression und/oder Lokalisation maternaler mRNAs beteiligt ist, die für die a-p-Achse essentiell sind. 130 Diskussion Schröder schlug 2003 vor, die beiden Drosophila Orthologe von orthodenticle-1 und hunchback würden in Tribolium bicoid ersetzen, da die kombinierte RNAi den Verlust von Kopf, Thorax sowie dem anterioren Abdomen zu Folge hat. Allerdings sind beide Gene maternal ubiqitär exprimiert, was gegen eine Funktion als anteriores Morphogen spricht (Wolff et al., 1995; Schröder, 2003). Tc-pangolin und Tc-eagle hingegen sind maternal anterior lokalisiert; eine frühe a-p-Funktion konnte allerdings für keines der beiden Gene festgestellt werden (Bucher et al., 2005). Erst 2012 wurde mit axin ein maternal lokalisierter Faktor entdeckt, der für die Ausbildung anteriorer Strukturen notwendig ist (Fu et al., 2012). Das Protein AXIN gehört zu dem Abbaukomplex von ß-CATENIN. Da axin anterior lokalisiert ist, wird dort ß-CATENIN/ ARMADILLO abgebaut und kann nicht mehr in den Nukleus gelangen, um über die Bindung von pangolin wnt-Zielgene zu aktivieren (Hamada et al., 1999, HeegTruesdell & LaBonne, 2006). Wenn man nun davon ausgeht, dass mit AXIN der fehlende Segmentierungsfaktor identifiziert wurde und der daraus folgende Gradient der WNT-Aktivität die a-pPolarität in Tribolium etabliert (Fu et al., 2012), ist anzunehmen, dass auch die anteriore Expression von hbn darüber reguliert wird, während die Differenzierung (Serosa > „segment addition zone“) durch zen-1 über das maternale axin (Fu et al., 2012) und das zygotische, von TORSO unabhängige hbn erfolgen würde (Abb. 29 und 32). Da axin nur anterior exprimiert wird, kann folglich am posterioren Pol ßcatenin/Armadillo zusammen mit TORSO ungestört die Expression von wg aktivieren und caudal und die Gene der „Segmentation Clock“ (Bolognesi et al., 2008; Fu et al., 2012; El-Sherif et al., 2012; Sarrazin et al., 2012;) etablieren (Abb. 41). 131 Diskussion Abbildung 41: Modell zur Differenzierung terminaler Identitäten in Tribolium Interaktion maternaler und zygotischer Gene für die Identitätszuweisun g des terminalen Systems – anterior vs. posterior. Anterior reguliert das terminale System (torso*) zusammen mit axin über die Reprimierung von ß-Catenin die zen-Gene und hbn, die wiederum Serosa und Kopf etablieren. Am posterioren Pol hingegen fehlt axin. ß-catenin und das terminale System (torso*) aktivieren wg und induzieren die Segmentierungskaskade der „segment addition zone“. Punktierte Pfeile stellen mögliche Interaktionen dar. Bei näherer Betrachtung offenbart dieses Modell allerdings Schwächen, die gegen eine Funktion von axin/ ß-catenin in der Differenzierung des terminalen Systems und für einen weiteren, unbekannten Faktor sprechen. So wurde nach axin-RNAi zwar ein Verlust anteriorer Strukturen inklusive Serosa, nicht aber eine anteriore Duplikation von caudal und even-skipped oder Doppelabdomen wie nach gcl-RNAi beobachtet (Fu et al., 2012). Es wäre zwar möglich, dass sich diese Phänotypen in der Klasse der „empty eggs“ oder unter den schwer identifizierbaren Kutikularesten verstecken, die in den ersten zwei Wochen nach axin-RNAi auftraten (Fu et al., 2012). Das ist allerdings fraglich, da nach gcl-RNAi axin nach wie vor anterior lokalisiert ist und somit offenbar als entscheidender Faktor für die Determination des terminalen Systems ausscheidet (Abb. 39). Es wäre aber denkbar, dass gcl neben pangolin auch die mRNA eines weiteren Proteins aus dem ß-catenin-Abbaukomplexes reguliert. Zur Klärung der generellen Rolle von axin wäre es angeraten, die axin-RNAiEmbryonen der ersten Woche („empty eggs“) mit Expressionsmarkern wie z.B. wg 132 Diskussion und caudal, aber auch hbn zu analysieren. Interessant wäre auch die Analyse der Einzel- und Doppel-RNAi gegen die beiden ß-catenin-Kopien (Bao et al., 2012), da diese anteriore Anlagen an beiden Polen ausbilden müssten, die mittels zen-1- oder hbn-Expression nachweisbar wären. Die beiden Gene wurden zwar bereits strukturell untersucht und die RNAi-Phänotypen beschrieben. Da der Knockdown von ßcatenin/armadillo-1 aber den Zusammenbruch der Eiproduktion sowie mittelfristig den Tod der Weibchen zur Folge hat – wahrscheinlich aufgrund gestörter Zelladhäsion – ist eine mögliche Rolle in der Achsenentwicklung von ß-catenin/armadillo-1 ungeklärt (Bao et al., 2012). Um diese Funktion von ß-catenin/armadillo-1 zu umgehen, könnte allerdings auf eRNAi umgestellt werden. Im Zusammenhang mit dem wnt-Signalweg bleibt auch die Rolle von pangolin ungeklärt. Denn wie ich zeigen konnte, reguliert gcl dessen anteriore Lokalisation (Abb. 32). Eine Funktion von pangolin in der Etablierung der a-p-Achse konnte allerdings nicht nachgewiesen werden (Bucher et al., 2005). Auch ich konnte mittels RNAi gegen pangolin keinen Doppelabdomen-Phänotyp nachweisen (nicht gezeigt). Allein kann pangolin die Differenzierung der terminalen Systems daher nicht herbeiführen. Es wäre aber möglich, dass pangolin nur einer von mehreren Faktoren ist, die diese Aufgabe übernehmen und deren Synthese/Transport/Lokalisation von gcl abhängen. Neuste Erkenntnisse aus der Zuckmücke Chironomus riparius (Diptera) könnten diese These untermauern. In Chironomus konnte die Unterscheidung der beiden termianlen Regionen auf ein neues, bis dato unbekanntes Gen zurückgeführt werden: panish. Die strukturelle Analyse des Proteins offenbart Ähnlichkeiten zu pangolin. Zudem ist das Transkript von panish im Chironomus-Embryo wie pangolin in Tribolium anterior lokalisiert, und RNAi gegen panish resultiert in Doppelabdomen (Klomp et al., 2014). Dies deutet auf eine evolutionäre Funktion pangolins in der Etablierung der a-p-Achse, weshalb hbn und zen-1 in pangolin-RNAiHintergrund untersucht werden sollten. Abschließend sei gesagt: obwohl die beteiligten Faktoren und die beteiligten genetischen Interaktionen im Verlauf dieser Arbeit nicht vollständig aufgeklärt werden konnten, liefert sie mit gcl und hbn wichtige erste Einsichten in die Determinierung terminaler Strukturen in Tribolium und möglicherweise der Kurzkeim-Insekten allgemein. 133 Anhang A – Puffer und Reagenzien Anhang A – Puffer und Reagenzien Boehringer Block Reagenz (2,5 %): 1,25 g Reagenz in 50 ml PBT (pH 7-7,5) Färbepuffer: 100 µl 1 M Tris + 50 µl 1 M MgCl2 + 20 µl 5 M NaCl + 5 µl Tween (20 %) + 825 H2 O Färbelösung: 1 ml Färbepuffer + 15 µl NBT/BCIP (Roche) Hoyers: 30 g Gummi arabicum + 50 ml H2 0 (MQ) + 200 mg Chloralhydrat (giftig!) + 20g Glycerin Hybe A: 20 ml Formamid + 10 ml 20x SSC (pH 5,5) + dH2 O zu 40 ml; plus 0,8 ml 10 mg/µl Salmon testis DNA + 200 µl 20 mg/ml tRNA + 40 µl 50 mg/ml Heparin Hybe B: 50 ml Formaid, 25 ml 20x SSC + 25 ml dH2 O Injektionspuffer: 1,4 mM NaCl, 0,07 mM Na2HPO4, 0,03 mM KH2PO4, 4 mM KCl; 2% Phenolrot, Sigma) 10x PBS: 80 g NaCl + 2 g KCl + 2 g KH2 PO4 + 11,5 g Na2 HPO 4 + dH2 O zu 1 l PBT: 50 ml 1x PBS + 0,5 ml 20x Tween + dH2 O zu 1 l PBT (für Ovarpräparationen): 50 ml 1x PBS + 0,5 % Triton + 1 % BSA + dH2 O zu 1 l PEMS: 12,08 g Pipes + 800 µl 1 M MgSO 4 + 800 µl 0,5 M EDTA + dH2 O zu 1 l (pH 6,9) 134 Anhang B - verwendete Primer Anhang B - verwendete Primer NAME SEQUENZ NT001-SP6-DD NT002-T7-SP 6-DD GAATTTAGGTGACA CTA TAGAAGTG AGTAATACGACTCACTA TA GGGA GA TGA CACTA TAGAAGTG AGTAATACGACTCACTA TA GGGA GA TGACACTATAGAAGTGTCAGAAA TCGGGGACAA AAC CTCACTATAGGGAGAA CAA TGTCCAAACCCAGC TC TGACACTATAGAAGTGA TACCGGTTTTGA TGGC ACA CTCACTATAGGGAGA TGAGGAAACA TCGTTTGG TG TGACACTATAGAAGTGGGTGGGGAAGTCAGTG TTGT CTCACTATAGGGAGA TCTTTGAGGCGGA TTTCA TC TGACACTATAGAAGTGGCCA GGAACAA CCAGT CCTA CTCACTATAGGGAGA GTCGAAA TCGCCTCTCAA AA TGACACTATAGAAGTGGTGCGTCGGGACTGA T AAAT CTCACTATAGGGAGA GGTTCTCGTTCTGCTTTG GA TGACACTATAGAAGTGTTGA CACGTCTTTGTTC GTTG CTCACTATAGGGAGA CGTAA TGA TGGCTTTGTT GC TGACACTATAGAAGTGAGGCTCTGCA TCTGGGA AG CTCACTATAGGGAGAA GCGTGGACTAGA GGGA GTG TGACACTATAGAAGTGAGCCCTTTCCGAGTTTG ATT CTCACTATAGGGAGA GCAA GTGCA TA TCGCTTT GA TGACACTATAGAAGTGAGCAA TCCCTACGCTTT GAA CTCACTATAGGGAGAAAA TCTTTGCCGTTTCGT TC TGACACTATAGAAGTGTGTGCGTTCCTAGCGTA CTG CTCACTATAGGGAGA GTCCAGGTTGTTTTCGGT GT TGACACTATAGAAGTGGGCCAACA CACA CGAAA NT003-T7-DD NT004-iB_00736-F NT004-iB_00736-R NT005-IB_01453-F NT005-IB_01453-R NT006-IB_00981-F NT006-IB_00981-R NT007-IB_01968-F NT007-IB_01968-R NT008-IB_00902-F NT008-IB_00902-R NT009-IB_05191-F NT009-IB_05191-R NT0010-t-shirt -F NT0010-t-shirt-R NT0011-IB_03546-F NT0011-IB_03546-R NT0012-iB_03594-F NT0012-iB_03594-R NT0013-iB_03835-F NT0013-iB_03835-R NT0014-iB_03859-F Kommentar Primer für Screen +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini 135 Anhang B - verwendete Primer NT0014-iB_03859-R NT0015-iB_04013-F NT0015-iB_04013-R NT0016-iB_04014-F NT0016-iB_04014-R NT0017-iB_04279-F NT0017-iB_04279-R NT0018-iB_05471-F NT0018-iB_05471-R NT0019-iB_02693-F NT0019-iB_02693-R NT0020-iB_05451-F NT0020-iB_05451-R NT0021-iB_02280-F NT0021-iB_02280-R NT0022-iB_03492-F NT0022-iB_03492-R NT0023-iB_01719-F NT0023-iB_01719-R NT0024-iB_04334-F NT0024-iB_01719-R NT0025-iB_04564-F NT0025-iB_04564-R NT0026-tsh5’-2FRG-F NT0026-tsh5’-2FRG-R NT0027-stau-anf-F NT0027-stau-anf-R ATA CTCACTATAGGGAGA GAACCGTTCCCTCGTTAC AA TGACACTATAGAAGTGCA GTACTTCGCCCCTTT TGA CTCACTATAGGGAGA CAAGTGCGTCTTGAGA TT GG TGACACTATAGAAGTGACTTGGCGCTGTTGGA T AAC CTCACTATAGGGAGA GTCGGTA TGGGTGAGCA GAT TGACACTATAGAAGTGGTGCGAGTA CTGTGGC AAGA CTCACTATAGGGAGA CAAGCTGGTGCTGTTGTT GT TGACACTATAGAAGTGCA GTGCAAAA TTGGCTA CGA CTCACTATAGGGAGA GGTAA CTCCCCGGAA CA TTT TGACACTATAGAAGTGTCAACCAAAACGA CGA T AAGG CTCACTATAGGGAGA TGCA CGA CCTCTTCCTGA TT TGACACTATAGAAGTGCAAA TGCAAA CAGTGCG ATT CTCACTATAGGGAGAAA GGGA TAA CCA GGGAA AGG TGACACTATAGAAGTGGCCCAGTA CGGGTA CG ATAA CTCACTATAGGGAGA CGA GACCGTGAA TCAA C AAA TGACACTATAGAAGTGCA GCTCTGACGACGGTT TTA CTCACTATAGGGAGAA GCCCCGA TCCTTTTTA T TG TGACACTATAGAAGTGGGTACGGCTCCGA TTGT AAA CTCACTATAGGGAGA TTCGCGACCTGCTTCTA C TT TGACACTATAGAAGTGGA TGCAGA CCA TTCA GA GCA CTCACTATAGGGAGA CA CCCTA TCGCTCCTA TC CA TGACACTATAGAAGTGCGTCTACAGCA TCGA CC AGA CTCACTATAGGGAGA CCTTTTCACGCTTTCTCC AC TGACACTATAGAAGTGCGGA CAAACCCTTCAGG TAA CTCACTATAGGGAGA TTA TGA TCTCGGCCTCTC GT TGACACTATAGAAGTG GCAACAAATGGGACCACCAC CTCACTATAGGGAGA +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini 136 NT0028-stau-mid-F NT0028-stau-mid-R NT0029-stau-dwns-F NT0029-stau-dwns-R NT0030-stau2-F NT0030-stau2-R NT0031-cytp450-F NT0031-cytp450 -R NT0032-Stau-Nof-F NT0032-Stau-Nof-R Gcl-Forward Gcl-Backward NT0033Nested_gcl_FW NT0033Nested_gcl_BW NT0034-hbn-Nof-F NT0034-hbn-Nof-R NT0035-iB_00800-F NT0035-iB_00800-R NT0036-iB_01875-F NT0036-iB_01875-R NT0037-Tc-DP-F NT0037-Tc-DP-R NT0038-hbn-NOF2-F NT0038-hbn-NOF2-R TTGCGGTTGCTGGGGTA TAG TGACACTATAGAAGTGGCACCGCCCAGTACTA C TTT CTCACTATAGGGAGA CGATCCCCGACTCTTAACCG TGACACTATAGAAGTG CGTGGTCGTTA GTTGGTGGT CTCACTATAGGGAGA GTTACGGTCTGCTCTGTGCT TGACACTATAGAAGTG ACTCCAACCGTTGCACTCAA CTCACTATAGGGAGA AATTGTGCCGCTTCGTGAA C TGACACTATAGAAGTG TCTACGAAGAGCTTGACGCC CTCACTATAGGGAGA CCAAACGGCATGAAAGCA CA TGACACTATAGAAGTGCGTTCACGAAATTGCTC TCA CTCACTATAGGGAGA GTTGCCGTAA CCCAACTG AT TTTAATCTAGA GCGGCCGCTGAACTGACTGCAA TGGAGGT TTTAATACGTA GCGA TCGCA CCCTCA TGCAAGA ACACAAAA TATTGTTGGAGAGCGTCGTG GCAATCACAAAACACATCAACTG TGACACTATAGAAGTG CGCTTTCCTGTCGCAA TACGTG CTCACTATAGGGAGA ACCAAGACAAGTAAAACACCTGCC TGACACTATAGAAGTGTCGAGGTGTTCCTCTGT CCT CTCACTATAGGGAGAA GAAACGGCTCGTTTGA GAA TGACACTATAGAAGTGCAA GCTGTGGA TTGAGC AAA CTCACTATAGGGAGAAA CAAACCGAA CGCGTAA TC TGACACTATAGAAGTGAAAGAAA TCA GGTGGA T TGGG CTCACTATAGGGAGAAA TGTA TGAA CTCGGGG CTG TGACACTATAGAAGTG ACGCTTTCCTGTCGCAA TAC CTCACTATAGGGAGA GCGAAGAACTCGGCA CTAAA +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini Diverse Bindestellen für Enzyme Primer für gclsenseRNASynthese +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini +SP6mini +T7mini 137 Anhang C – Vektorkarte pCS2 + gcl-eGFP Anhang C – Vektorkarte pCS2 + gcl-eGFP SP6 p r o m o t e r ot e 0 0 l5 ' bu t 300 n 3' 600 ( pa rt ia o 60 0 54 00 0 ro t 90 p R: UT 00 57 gc l r e in 5o10 ri 0 4800 h) et e c 0 3 pR te mo 0 o r p 24 T3 0 2 70 3000 r 00 42 Am 90 21 00 4500 1800 ) 1500 f us io n 0 12 0 pCS2 +gcl-eGFP 6 3 2 4 bp 0 ori 00 3300 m 36 SpeI ( 1 2 4 0 ) e pC GFP S2 ( Cl on ) V CM m p ro SV A o ly P 40 (f ro AsiSI ( 2 3 7 2 ) Abbildung 42: Vektorkarte des pCS2-gcl-egfp-Plasmids Dargestellt ist das pCS2-Vektor (oder auch pCS2+) mit dem gcl-eGFP-Insert. Das fertige Konstrukt enthält mehrere Promotoren, darunter einen für SP6 und T3, die das gcl-eGFP-Insert(blau und grün) einrahmen. Weiterhin dargestellt sind die Resistenz gegen Ampicillin (AmpR; grau), ein Cytomegalovirus-Promotor (CMV; hellblau) und die für die Klonierung von eGFP in den pCS2-gcl verwendeten Schnittstellen der Restriktionsenzyme Spe1 und AsiS1. 138 Anhang D –Rescreen Anhang D –Rescreen iB_Nummer Penetranz des Phänotyps (Screen) Drosophila Ortholog gleiches Fragment in 2 Stämmen Nicht überlappendes Fragment Abdomen fehlt teilweise bzw. komplett, teilweise auch der Metathorax > 80 % CG5753 bestätigt nicht bestätigt Leere Eier, pantagmatische Defekte, die Kopf und Thorax eliminieren > 80 % bestätigt nicht bestätigt Kopf fehlt größtenteils und teilweise der Thorax > 80 % nicht bestätigt nicht bestätigt iB_01453 Großteil von Kopf und Thorax fehlen, teilweise nur Abdomen vorhanden > 80 % -- bestätigt nicht bestätigt iB_01719 Anteriorer Kopf und Abdomen fehlen > 80 % -- teilweise bestätigt nicht bestätigt iB_01968 Nur posteriores Abdomen vorhanden > 80 % -- teilweise bestätigt nicht bestätigt iB_02280 Segmente des Abdomens fehlen, teilweise auch Meso- & Metathorax > 80 % CG13933 nicht bestätigt nicht bestätigt iB_02693 Kopf und Thorax transformiert zu gespiegeltem Abdomen > 80 % CG8411 bestätigt bestätigt Larvale Segmente teilweise fusioniert, Kutikularisierung wirkt gestört > 80 % bestätigt bestätigt Vielzahl leere Eier, wenige Kutikulareste, teilweise nur > 80 % teilweise bestätigt nicht bestätigt iB_00736 iB_00902 iB_00981 iB_03492 iB_03546 Beschreibung des Phänotyps (Screen) Rescreen staufen CG11255 --- CG11448 -- germ cellless CG9635 Rho guanine nucleotide exchange factor 2 CG6863 tolkin 139 Anhang D –Rescreen posteriores Abdomen iB_03594 Vielzahl leere Eier, wenige Kutikulareste, teilweise nur Reste des Kopfes > 80 % nicht bestätigt nicht bestätigt Kopf und Teil des Thorax vorhanden, Rest fehlt oder ist fragmentiert > 80 % bestätigt bestätigt iB_03859 Teile von Kopf, Thorax und Abdomen fehlen bzw. weisen Einschnürungen auf > 80 % CG32579 bestätigt nicht bestätigt iB_04279 Hälfte leere Eier, Rest posteriores Abdomen teilweise mit Kopfanhängeln > 80 % --- bestätigt nicht bestätigt iB_04334 Kopf und Thorax fehlen > 80 % CG13272 nicht bestätigt nicht getestet iB_04564 Kopf und Thorax transformiert zu gespiegeltem Abdomen 30-50 % CG33152 bestätigt bestätigt Teile von Kopf, Thorax und Abdomen fehlen > 80 % bestätigt bestätigt iB_05471 Nur anteriorer Kopf vorhanden > 80 % CG31999 nicht bestätigt nicht bestätigt Positivkontrolle für teashirt Kopf und Thorax transformiert zu gespiegeltem Abdomen 30-50 % CG1374 teilweise bestätigt nicht getestet iB_03835 iB_05451 CG10571 araucan CG10117 tout-velu homeob rain CG9786 hunchb ack 140 Anhang E – Ergebnisse zu iB_00736 (staufen) Anhang E – Ergebnisse zu iB_00736 (staufen) Die RNAi des iBeetle-Fragments iB_00736 resultierte in Tribolium-Larven, denen das Abdomen sowie teilweise der Metathorax, also die Strukturen aus der „segment addition zone“ fehlen (Schoppmeier & Schröder, 2005). iB_00736 umfasst 2 Exons (fett, Gensequenz) des Orthologs zu staufen aus Drosophila (Tc_004615; Gensequenz). Obwohl sich dieser Phänotyp in den Stämmen PBA19 und SB bei erneuter Injektion verifizieren ließ, führte die RNAi des nicht-überlappenden Fragments (rot, Gensequenz) zu wildtypischen Larven. Vorläufige in-Situ-Färbungen zu iB_00736 zeigten eine ubiquitäre Expression (nicht gezeigt) der zugehörigen mRNA. Färbungen von vasa und zen-1 in iB_00736RNAi-Embryonen resultierten in wildtypischen Färbungen (nicht gezeigt), während in eve-Färbungen die Domäne in der SAZ fehlt (nicht gezeigt). Ebenfalls auffällig war, dass in vielen Embryonen keine Formation des „posterior pit“ auftritt. Stattdessen stüplt sich der posteriore Pol nach außen und der Keim wächst außerhalb des Eis (nicht gezeigt). Da die Identität des Gens nach Analyse der Originalfragments und des Nofs ungeklärt war, konzipierte ich zwei weitere Fragmente (stau-Anfang = grün und stauMitte =blau; Gensequenz). Die RNAi beider Fragmente sowie eine erneute Analyse des staufen-Klons von M. Schoppmeier (Erlangen) resultierten in wildtypischen Embryonen. Aus diesem Grund untersuchte ich ebenfalls mit Hilfe der RNAi das Gen, das downstream von staufen liegt (TC004758), da die Readings von RNA-SeqAnalysen überlappen, ein Gen, das Ähnlichkeit zu staufen aufweist (TC008157) sowie das Gen Cytochrom P450 (Tc015293), das im Blast für das Originalfragment ebenfalls als Ergebnis aufgeführt wurde. Auch hier konnten in der Untersuchung der L1-Larven nur wildtypische Larven gefunden werden (nicht gezeigt). Da weder das NoF noch die anderen neu-definierten Fragmente zum Phänotyp des Originalfragments iB_00736 führen, ist davon auszugehen, dass es sich hierbei um einen „off-target“-Effekt handelt. Um die verantwortliche Sequenz und somit das mögliche zweite Zielgen neben staufen zu identifizieren, amplifizierte ich aus dem Originalfragment mit Hilfe neuer Primer zwei Einzelfragmente, iB_00736a und iB_00736b. 141 Anhang E – Ergebnisse zu iB_00736 (staufen) Da iB_00736a den eingangs erwähnten Phänotyp zeigte, teilte ich das Fragment in iB_00736aI und iB_00736II. Erneut war der Phänotyp nur in einem der beiden Fragmente (iB_00736aI) zu beobachten, womit die verantwortliche Gen-Sequenz auf eine Region von ~ 80 bp eingegrenzt werden konnte. Name des Primers NT004-iB_00736-F NT004-iB_00736-R NT0032-stau-Nof-F NT0032-stau-Nof-R NT0027-stau-anf-F NT0027-stau-anf-R NT0028-stau-mid-F NT0028-stau-mid-R NT0029-stau-dwns-F NT0029-stau-dwns-R NT0030-stau2-F NT0030-stau2-R NT0031-cytp450-F NT0031-cytp450 -R NT0041-iB_00736a_F NT0041iB_00736a_BW NT0042-iB_00736b_F NT0042iB_00736b_BW NT0046iB_00736a1_F NT0046iB_00736a1_BW NT0047iB_00736a2_F Sequenz des Primers plus SPAmini (rot) bzw. T7mini (blau) TGACACTATAGAAGTG TCAGAAATCGGGGA CAAAAC CTCACTATAGGGAGA ACAATGTCCAAACCCAGCTC TGACACTATAGAAGTG CGTTCACGAAA TTGCTCTCA CTCACTATAGGGAGA GTTGCCGTAACCCAACTGA T TGACACTATAGAAGTG GCAACAAATGGGACCACCAC CTCACTATAGGGAGA TTGCGGTTGCTGGGGTA TAG TGACACTATAGAAGTG GCACCGCCCAGTACTACTTT CTCACTATAGGGAGA CGATCCCCGACTCTTAACCG TGACACTATAGAAGTG CGTGGTCGTTA GTTGGTGGT CTCACTATAGGGAGA GTTACGGTCTGCTCTGTGCT TGACACTATAGAAGTG ACTCCAACCGTTGCACTCAA CTCACTATAGGGAGA AATTGTGCCGCTTCGTGAA C TGACACTATAGAAGTG TCTACGAAGAGCTTGACGCC CTCACTATAGGGAGA CCAAACGGCATGAAAGCA CA TGACACTATAGAAGTG TCAGAAATCGGGGA CAAAAC CTCACTATAGGGAGA TGGCTTA TTTTGTTGGAAGTTG TGACACTATAGAAGTG CGATTCAAGGTGTTCGTTCA CTCACTATAGGGAGA CCAAACCCAGCTCTGA CAA TGACACTATAGAAGTG TCAGAAATCGGGGA CAAAAC CTCACTATAGGGAGA CTTCCACCAA CAGA CGGA GT TGACACTATAGAAGTG GTGGAAGCGGTGGTAGA CAA Farbe in Gensequenz fett rot grün blau nicht gezeigt nicht gezeigt nicht gezeigt fett und unterstrichen (doppelt und einfach) fett und grau Fett und doppelt unterstrichen 142 Anhang E – Ergebnisse zu iB_00736 (staufen) NT0047iB_00736a2_BW CTCACTATAGGGAGA TGGTCTCCGA CCAGCAATA Gensequenz staufen (Tc004615) – gelb = Exons ATGGGAATGC AACAAATGGG ACCACCACCG AACCATAGGA GCCAGCCAAT GCTCATGAGC ATCCAGCCAC CGGTGACTGC CGCCCATGCA GGTATTTCAC CATTCCACCC ATTTTTTTTT TATTAACGTG GCTGCTTTTA GGAGTTCTGG TTTCTATGCC TCCAGGTCCT CCGCTCATCT CCACCCCGTC TATACCCCAG CAACCGCAAC CCAAAAACAA TGTTCAGCAA GGTAAGGTGT TTGACTGTTT GTACAAAACT TGTCATTCAC TGTTGGCGAT TGTGGCTTTT TTAATTTGGT GGTAGAGTGG CGCTTTATGA CCGCACTCGG TCGTGAATGT TTAGTTCTAC ATAAAGTCAA TCGTAACATG GTTGCCTCCC AAAATTCTTA ATTATTAGGT TGGCGCTGTA TTGAATTTTT TCTTACTAAA ATTTTACTTT AAGCGATCTC AATGTATCTG AGTGAAGATG CGTTTAGGCC GCAGCTCGTT GATCACTGTT GGCCAAAAAA TGAGGATAAT ATGGTAGTAT TTAAACTTTA AAGGCCTGTT TATAGAAAGC TTTGTTAAAA GTTAACACGA ATGGACCAAT CAAATTGGTT TAATTTGGAC ACGTGACCAG TTAATCACGC CCTTAATTGC AGATTGAATT AATGACGCTT CTATAAACCA GTCCTAAAAG TTTAAAATTA TTTAAACACA TTTTCGTGTC TGTAGACGAA ATTTACCAAA ATTCTAACTC TATGAGCCCT AACAACTAAA TGTTTGGACG AATATATTTT TGTAAAGAAA ATGCACGTCA AGCTAGTAAA AAACAAATAT ATTTTGAAGA AATACGACTT GATATTTAAC TTAAAATTAA AACAAAAGTA CAGGAATAAT ATCTGACAAC TATTGTTTGG GCAAAAAAGA AAAACTTCTT AGTAATTCGT GCCATCTCTT TTGGCTTTAA TGACGGCTCG AATGTACGCT GCCATATTTT TAACGAATTT GTCACAGATT TGTGACGTTT TATTGTTCTA AATATGTTCT GTTTCATTTT TTAAATCTTA ATGACCACTG AGGATGTTTC CTAAGATAAC AAAAAGTAAC ATTTTGTTAT TCAATTGTGT CTACCTTATT TTAACCAAGA GTTGTTTTTT ATCCATTTAT ACAATCCTTA AACTGTTTCT ACGGCAATTT TTACAAATGA TCTTCCAGGA CTTTTTCGTT TAATAACTAT CGTACTTCGT ATCCATCTAC TTGACTAATT TGTGCCACTG AAGAATTGTC CGTATTCAGT TTTTTGACAA CTTGTGCCAT AATTACTTTT TATATTTAGT TGCCATTCGT CATTAATATA TTTTTTCTGA TAACTGATTA AAATCAAGCA AAATATTAAC ATTTAGTTGT TAGGACTCGT AATTTAAAGC AAACAACAAA CGGGATTGTT TGTTCTATTT GATGTGTTTT TAATTTTTTG TCACTATTTA TTGCTCGTCG ACATGCTGTA GGTTGTGATC TCGGGAATTT TTATTAACAA TATGTCTGGC TGAGTGAAAC GGTCTATTAT TCGGTAAACA GACCTGCTTT TTTGTACAAT TTGTAGTCAA GTTAGTGTAT TTTCAGTGTT TTTTATATTG AAAAAAATTC TACGAGAAAA AGTGGTTGGT CTCTTCATCG GAGTGTCGTC CCAGATACGT TGAGTGCGCC TAAAGGGAAA TTTACCCGAA ATTTTGGTTT TCGTGGTAGT TGTTCGTATT GTGTAAGACG GTGTTGGTTG TTTGCATGTT GATTTATTAA TACCTGTAGT TTGTGCAATG TTGTATTTTC AGTATATCAC TATGAAAATA CAGCATCGTC AGAAACCGAA CAGGCTCAAC ATAACGACAC TCAACAGCAC CAAACAGGTC CGAACACTAG TACTTCGTCT GCACCGCCCA GTACTACTTT GGCAAACATA AAAGAAAAAA CACCAATGTG CTTGGTCAAT GAATTGGCAA GGTACAATAA AATACAACAC CAGTATCAGC TGACAGAGGA AAGTGGACCA GCACATAAAA AAGTATTTAC AGTCACGTTA AAATTGGGCA ACGAAGAGTA TAAATCAGAG GGTCCGAGTA TTAAAAAAGC CCAACATTCT GCAGCTGCAC AATCACTTGC TAAAACTGAA TTTAAACATC CGCCTTCCAA AACAGCCAGA AATCGGCCCG GAACCAGAGC TACAAACCCC GGGGTGATGA CTCCGACAGT CGAACTGAAT GCTCTAGCCA TGAAACGTGG CGAACGGCCG GTTTACATCG TCGAAAATCC TCCGCCACAT CAGGTATTGC AAAGATAAGC GAGCAAAATA AGAAAATACA ACAGTTTTTG AATAGTTTGA CAAAATTAAC GAAATAATTG CTCCTTCTCT TGTCAGGGTT ATATCAGTCA GGCCGGGTAC TATCCCCGCC AAAACATTTA CGGCACCCAG AACCAGCCAA GATACGGCTA CGACACGCGT CGAAACATGC GCCCCCATTA TCCCTACCAC GAAAATCGCT ACTACGGCCA GTACAGGCCC ACAGGCCCCC ACAATCCCGG TGATCCCTAC ACCGTGCGGT TAAGAGTCGG GGATCGGGAG TATCCCGGTG TTGGGTACAC TGTCCAAGCA GCTCGTCACG ACGCAGCTGC CAAAGCCATC GAAGATATAA AACAACTGGG 143 Anhang E – Ergebnisse zu iB_00736 (staufen) AGACTGCACT GATCAATCAG GGACCGTGGA AGCCCGTAAG TAGGCCGAGA TCAGCGCCAA GTAACCGACA TTTTTATTAA TAGCTGTTGA AAACACAGGA TCGAACGATA TCAATACCGA ACTGAAGTCG CCAATTTCGC TCGTTCACGA AATTGCTCTC AAACGAAATC TTAGTGTCAC GTTTGAAGTG CTCAGCGAGA AAGGGCCGCC GCATATGAAG GTATTTGTAA CGCAGTGTCG GGTTGGCAAC TTTGTTGCAG AAGGCGAAGG CAACGGAAAG AAAATTTCGA AGAAACGTGC AGCTGAAAAA ATGTTGGAAG AGCTGGCCAA ATTGCCGCCG TTGCCCAACA TGCACAACAT GGTACATCTC AAGAGGAAGA GAGTCACTAA TAAGAAGAAA ACCAGGAACT TAATCAAGGT CAATATGGAC AAGTCCAGCG AATTCACCGA AGAAATCAAC CCCATTTCGC GCTTAATCCA AATACAGCAA GCCAACAAGG AGCGAGAGCC GGTCTACACG GTGAGTGTTT CAATCCCCCA ACTGAAACAC GCACAATTTC TTTATTATCA TTATATCAAA ATTAAAGGAA CAGTTTTTAT TTGCTTTATT GATTAGGACA CACGTGTCTT CGGCCTTGCT GTTGATTTAG ATTGTTTCTT AAGGCCTGAG GTTTGCATTT TTAGCTTACA TTAGGAGTAA AATGTATCTA ATTGAATTAG GTGTTGGAAG AGCGTGGCGC CCCCCGGCGC CGCGAGTTCG TGATAGAGGC CTCTGTGAAC GGGCACTCTT GCACCGGAGT GGGCCCTAAT AAAAAAATTG CAAAGCGTAA TGCAGCCGAG GCTCTGTTGA ATCAGTTGGG TTACGGCAAC CCACCTTCGA ATCCGAAACC AGTGTTGAAA ACCGAGAAAG AATCAGAAAT CGGGGACAAA ACCCGGAAAG TAACGTTCGT CGAAGAGAAA ACTGAATCAA CTCCGTCTGT TGGTGGAAGC GGTGGTAGAC AACTAGTCCC TGGAATATTG CTGGTCGGAG ACCAGTCTAC CAACTTCCAA CAAGTAGTAC CCCAGTATTG TTGTATTCAA GTTGGTGATT GATTGTTTCA GAATAAGCCA AAGGAAAGTG TAATGAATAA TAACTCGAAT TCTTGCGTGG ATACTAAGAA GACAGGACAG ACTCAAATGA AAACTCCTCC TCAGCCGATT CAAGGTGTTC GTTCAAAGGA TCAGTTGATG TACTTGGCTC AACTGATGAA CATCCAAGTT CAGTTCTCTG ATTTTCCTAA AGCCAATCAC GAGATGTATT TAACGTTGGT GTCTCTAAGC ACCAATCCGC CGCAGGTTTG TCATGGAGAA GGCCCCACAA CTGAGGCCTC TCATGAGAAG GCTGCTTTAG AAGCACTCAA AGTTTTGTCA GAGCTGGGTT TGGACATTGT TGGCCCCAAC AAGGAAGGTG GTAACGAAAT GTAAGTGGGA GTTGGAGGCG AGTGAATGAA TATTAATGAG GAATTTATTG TTGCAGTACC ACTTCTCAAG GGTCTTCCAA GGTTGGGATC CAAAACGGAG TTAAGAAGTA A 144 Anhang F - Ergebnisse zur Positivkontrolle tiotsh Anhang F - Ergebnisse zur Positivkontrolle tiotsh Ein weiteres Gen, das kurzzeitig im Mittelpunkt des Fokus stand, war das Ortholog der Gene tip-top und teashirt aus Drosophila: Tribolium-tiotsh (Tc_012322;). 2008 beschrieben Shippy et al. den Phänotyp von tiotsh in Tribolium. Das Gen wurde deshalb für den larvalen Screen als Positivkontrolle herangezogen. Das Design des Screens bedingt, das jedes Gen und somit auch die Kontrollen sowohl larval als auch pupal injiziert werden. Obwohl für den larvalen Screen gedacht, resultierte die Positivkontrolle tiotsh auch im pupalen Screen in einem Phänotyp. Statt des Verlustes des ventralen Kopfes, wie beschrieben (Shippy et al., 2008), führte die Positivkontrolle jedoch in 30-50% der Larven zu Doppelabdomen. Der veröffentlichte Phänotyp konnte hingegen nicht gefunden werden. Die Analyse des offiziellen iB-Fragments zu tio-tsh, iB_05191 führte hingegen zu dem von Shippy et al. beschriebenen Phänotyp. Auch konnte der DoppelabdomenPhänotyp nur mit der Positivkontrolle im Stamm PBA19, nicht aber SB beobachtet werden. Die OGS Annotation für tiotsh beschreibt ein Gen, das ca. 80kbp umfasst, wobei die beiden iB-Fragmente von einem ausgedehnten Intron geteilt sind (siehe http://bioinf.uni-greifswald.de/gb2/gbrowse/tcas4; Tc012322). Die unterschiedlichen Ergebnisse für beide Fragmente sowie die AUGUSTUS-Vorhersage (nach M. Stanke, Bioinformatik, Greifswald) legen nahe, dass iB_05191 und die Positivkontrolle zu zwei verschiedenen Genen gehören. Eine entsprechende Analyse ergab, dass sowohl iB_05191 als auch die Positivkontrolle in den späten Keimstreifen in den Kopfanlagen, Beinanlagen und posterior im Stomadeum exprimiert sind. Eine solche Expression ist für Tc-tiotsh beschrieben (Shippy et al., 2008). Diese Ergebnisse sowie die Tatsache, dass im larvalen Screen der veröffentlichte Phänotyp für die Positivkontrolle zu beobachten war, sprechen für die ursprüngliche Vorhersage von tiotsh und einem möglichen „off-target“-Effekt der Positivkontrolle, der die Doppelabdomen-Phänotypen zur Folge hat. Zur Untersuchung des möglichen zweiten Zielgens der Positivkontrolle amplifizierte ich ein Fragment für das Gen, das laut AUGUSTUS Prediction 2 mit tiotsh an der Stelle des Positivkontroll-Fragments überlappt. Die RNAi-Analyse resultierte allerdings in wildtypischen Larven. Der Ursprung des Doppelabdomen-Phänotyps bleibt daher ungeklärt. 145 Anhang F - Ergebnisse zur Positivkontrolle tiotsh Darstellung des mit tc-tiotsh überlappenden Genes X (iBeetle; GBrowse) Positivkontrolle iBeetle tio-tsh Fragm ent A Primer Fragment A: Left Primer + SP6 5’ TGACACTATAGAA GTGCGGACAAACCCTTCAGGTAA 3’ Right Primer + T7 5’ CTCACTATA GGGA GA TTA TGA TCTCGGCCTCTCGT 3’ SEQUENCE SIZE: 349 146 Literaturverzeichnis Literaturverzeichnis Adams, M. D., Celniker, S. E., Holt, R. A. et al. (2000). The Genome Sequence of Drosophila melanogaster. Science, 287: 2185–2195. Akam, M. (1989). Hox and HOM: homologous gene clusters in insects and vertebrates. Cell, 57 (3): 347–479. Arakane, Y., Muthukrishnan, S., Kramer, K. J. et al. (2005). The Tribolium chitin synthase genes TcCHS1 and TcCHS2 are specialized for synthesis of epidermal cuticle and midgut peritrophic matrix. Insect Molecular Biology, 14 (5): 453– 463. Arziman, Z., Horn, T. & Boutros, M. (2005). E-RNAi: a web application to design optimized RNAi constructs. Nucleic Acids Research, 33 (Web Issue): W582588. Bao, R., Fischer, T. Bolognesi, R. et al. (2012). Parallel duplication and partial subfunctionalization of β-catenin/armadillo during insect evolution. Mol Biol Evol, 29: 647–662. Bardwell, V. J. & Treisman, R. (1994). The POZ domain: a conserved proteinprotein interaction motif. Genes & Development, 8: 1664–1677. Bate, M. (1993). The Mesoderm and its Derivatives. The Development of Drosophila melanosgaster. Cold Spring Harbor Laboratory Press: 1013-1090. Becalska, A. N. & Gavis, E. R. (2009). Lighting up mRNA localization in Drosophila oogenesis. Development, 136 (15): 2493–2503. Beermann, A., Aranda, M. & Schröder, R. (2004). The Sp8 zinc-finger transcription factor is involved in allometric growth of the limbs in the beetle Tribolium castaneum. Development, 131 (4): 733–742. Beiman, M., Shilo, B. Z. & Volk, T. (1996). Heartless, a Drosophila FGF receptor homolog, is essential for cell migration and establishment of several mesodermal lineages. Genes Dev., 10 (23):2993-3002. Benton, M. A, Akam, M. & Pavlopoulos, A. (2013). Cell and tissue dynamics during Tribolium embryogenesis revealed by versatile fluorescence labeling approaches. Development, 140: 3210–3220. Bolognesi, R., Farzana, L., Fischer, T. D. & Brown, S. J. (2008). Multiple Wnt genes are required for segmentation in the short-germ embryo of Tribolium castaneum. Current Biology, 18: 1624-1629. 147 Literaturverzeichnis Bomont, P., Cavalier, .L, Blondeau, F. et al. (2000). The gene encoding gigaxonin, a new member of the cytoskeletal BTB/kelch repeat family, is mutated in giant axonal neuropathy. Nat Genet, 26: 370-374. Bonhag, P. F. (1958). Ovarian Structure and Vitellogenesis in Insects. Annual Review of Entomology, 3: 137–160. Bonneton, F. (2008). The beetle by the name of Tribolium typology and etymology of Tribolium castaneum Herbst, 1797. Insect Biochemistry and Molecular Biology, 38: 377–379. Boutros, M., Kiger, A. A., Armknecht, S. et al. (2004). Genome-wide RNAi analysis of growth and viability in Drosophila cells. Science, 303: 832-835. Boylan, M. (1984). The Galenic and Hippocratic Challenges to Aristotle’s Conception. Journal of the History of Biology, 17, (1): 83–112. Brown, S. J., Parrish, J. K., Denell, R. E. & Beeman, R. W. (1994). Genetic control of early embryogenesis in the red flour beetle, Tribolium castaneum. Am. Zool. 34: 343-352 Brown, S. J., Parrish, J. K., Beeman, R. W. und Denell, R. E. (1997). Molecular characterization and embryonic expression of the even-skipped ortholog of Tribolium castaneum. Mechanisms of Development, 61: 165-173. Brown, S. J., Mahaffey, J. P., Lorenzen, M. D. et al. (1999). Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evolution & Development, 1 (1): 11–15. Brown, S. J., Fellers, J., Shippy, T. D., Denell, R. & Stauber, M. (2001). A strategy for mapping bicoid on the phylogenetic tree. Current Biology, 11 (2): 43–44. Brown, S. J., Shippy, T. D., Miller, S., et al. (2009). The Red Flour Beetle, Tribolium castaneum (Coleoptera): A Model for Studies of Development and Pest Biology. Cold Spring Harbor Protocols, 2009 (8): pdb.emo126. Brönner, G & Jäckle, H. (1991). Control and function of terminal gap gene activity in the posterior pole region of the Drosophila embryo. Mechanisms of Development, 35: 205-211 Buchenau, P., Saumweber, H. & Arndt-Jovin, D. J. (1997). The Dynamic Nuclear Redistribution of an hnRNP K-homologous Protein during. Journal of Cell Biology, 137, (2): 291–303. Bucher, G., Scholten, J. & Klingler, M. (2002). Parental RNAi in Tribolium (Coleoptera). Current Biology, 12 (3): 85–86. 148 Literaturverzeichnis Bucher, G. & Klingler, M. (2004). Divergent segmentation mechanism in the short germ insect Tribolium revealed by giant expression and function. Development, 131: 1729-1740. Bucher, G., Farzana, L., Brown, S.J. & Klingler, M. (2005). Anterior localization of maternal mRNAs in a short germ insect lacking bicoid. Evolution & Development, 7: 142-149. Buchon, N. & Vaury, C. (2006). RNAi: a defensive RNA-silencing against viruses and transposable elements. Heredity (Edinb), 96 (2): 195-202. Bull, A. L. (1966). Bicaudal, a Genetic Factor which Affects the Polarity of the Embryo in Drosophila melanogaster. Journal of Experimental Zoology, 161: 221242. Carroll, S. B. (1995). Homeotic genes and the evolution of arthropods and chordates. Nature, 376: 479–485. Carroll, S. B (2005). Endless Forms Most Beautiful – The New Science of Evo Devo, Norton & Company, New York. Casanova J. and Struhl,G. (1989) Localized surface activity of torso, a receptor tyrosine kinase, specifies terminal body pattern in Drosophila. Genes Dev., 3: 2025–2038. Cerny, A. C. (2007). Regulation von Segmentierungs- und Hox-Genen durch GapGene im Kurzkeim-Embryo von Tribolium Castaneum. Dissertation. FriedrichAlexander-Universität. Erlangen. Cerny, A. C., Bucher, G., Schröder, R. & Klingler, M. (2005). Breakdown of abdominal patterning in the Tribolium Krüppel mutant jaws. Development, 132: 5353–5363. Cerny, A. C., Grossmann, D., Bucher, G. & Klingler, M. (2008). The Tribolium ortholog of knirps and knirps-related is crucial for head segmentation but plays a minor role during abdominal patterning. Developmental Biology, 321: 284– 294. Clark-Hachtel, C. M., Linz, D. M. & Tomoyasu, Y. (2013). Insights into insect wing origin provided by functional analysis of vestigial in the red flour beetle, Tribolium castaneum. Proc. N. A. S. USA, 110 (42): 16951–16956. Copf, T., Schröder, R. & Averof, M. (2004). Ancestral role of caudal genes in axis elongation. Proceedings of the National Academy of Sciences, 101 (51): 17711– 17715. Damen, W. G. M. (2007). Evolutionary conservation and divergence of the segmentation process in arthropods. Developmental Dynamics, 236: 1379–1391. 149 Literaturverzeichnis Darwin, C. (1859) On the origin of species by means of natural selection, or the preservation of favoured races in the struggle for life. London: John Murray. Dassow, G. Von, Meir, E., Munro, E. M. & Odell, G. M. (2000). The segment polarity network is a robust developmental module, 406: 188–192. Davis, G. K. & Patel, N. H. (2002). SHORT, LONG, AND BEYOND : Molecular and Embryological Approaches to Insect Segmentation. Annual Review of Entomology, 47: 669–699. de la Luna, S., Allen, K. E., Mason, S. L. & La Thangue, N. B. (1999). Integration of a growth-suppressing BTB/POZ domain protein with the DP component of the E2F transcription factor. The EMBO Journal, 18 (1): 212–228. DiNardo, S. & O’Farrell, P. H. (1987). Establishment and refinement of segmental pattern in the Drosophila embryo: spatial control of engrailed expression by pair-rule genes. Genes & Development, 1: 1212–1225. Dowell, R. D., Ryan, O., Jansen, A. et al. (2010). Genotype to Phenotype: A Complex Problem. Science, 328 (5977): 469. Dönitz, J., Grossmann, D. Schild, I. et al. (2013). TrOn: An Anatomical Ontology for the Beetle Tribolium castaneum. PloS, 8 (7): e70695. Driever, W. & Nüsslein-Volhard, C. (1988a). A gradient of bicoid protein in Drosophila embryos. Cell, 54: 83–93. Driever, W. & Nüsslein-Volhard, C. (1988b). The bicoid protein in Drosophila embryos in a concentration-dependent manner. Cell, 54: 95–104. Driever, W. & Nüsslein-Volhard, C. (1989). The bicoid protein is a positive regulator of hunchback transcription in the early Drosophila embryo. Nature, 337: 138–143. Drummond-Barbosa, D. & Spradling, A. C. (2001). Stem cells and their progeny respond to nutritional changes during Drosophila oogenesis. Developmental Biology, 231 (1): 265–278. Dworkin, I., Kennerly, E., Tack, D. et al. (2009). Genomic Consequences of Background Effects on scalloped Mutant Expressivity in the Wing of Drosophila melanogaster. Genetics, 181: 1065-1076. Elbashir, S. M., Harborth, J. Weber, K. & Tuschl, T. (2002). Analysis of gene function in somatic mammalian cells using small interfering RNAs. Methods, 26: 199–213. El-Sherif, E., Averof, M. & Brown, S. J. (2012). A segmentation clock operating in blastoderm and germband stages of Tribolium development. Development, 139: 4341–4346. 150 Literaturverzeichnis Ephrussi, A., Dickinson, L. K. & Lehmann, R. (1991). Oskar organizes the germ plasm and directs localization of the posterior determinant nanos. Cell, 66: 37– 50. Falciani, F., Hausdorf, B., Schröder, R. et al.(1996). Class 3 Hox genes in insects and the origin of zen. Proceedings of the National Academy of Sciences of the United States of America, 93: 8479–8484. Fire, A.; Yu, S., Montgomery, M. K. et al. (1998). Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 391: 806– 811 Forrest, K. M. & Gavis, E. R. (2003). Live Imaging of Endogenous RNA Reveals a Diffusion and Entrapment Mechanism for nanos mRNA Localization in Drosophila. Current Biology, 13 (14): 1159–1168. Frohnhofer, H. G. & Nüsslein-Volhard, C. (1986). Organization of anterior pattern in the Drosophila embryo by the maternal gene bicoid. Nature, 324 (6093): 120125. Fu, J., Posnien, N., Bolognesi, R., Fischer et al. (2012). Asymmetrically expressed axin required for anterior development in Tribolium. Proceedings of the National Academy of Sciences, Early Edition: 1–5. Furriols, M., & Casanova, J. (2003). In and out of Torso RTKsignalling. EMBO Journal, 22: 1947–1952 Fyrberg, E. A. & Goldstein, L. S. B. (1990). The Drosophila Cytoskeleton. Annual Review of Cell Biology, 6: 559-596. Gao, Q. & Finkelstein, R. (1992). Targeting gene expression to the head: the Drosophila orthodenticle gene is a direct target of the Bicoid morphogen. Development, 125: 4185-4193. | Gehring, W. J. (1993). Exploring the homeobox. Gene, 135 (1-2): 215–21 Gibson, G. & Dworkin, I. (2004). Uncovering cryptic genetic variation. Nat Rev Genet, 5 (9): 681-690. Gjerstorff, M. F., Johansen, L. E., Nielsen, O. et al. (2006). Restriction of GAGE protein expression to subpopulations of cancer cells is independent of genotype and may limit the use of GAGE proteins as targets for cancer immunotherapy. British Journal of Cancer, 94: 1864–1873. Gjerstorff, M. F., Besir, H., Larsen, M. R., Ditzel, H. J. (2010) Expression, purification and characterization of the cancer-germline antigen GAGE12I: a candidate for cancer immunotherapy. Protein Expr Purif, 73: 217–222. 151 Literaturverzeichnis Gjerstorff, M. F., Rösner, H. I., Pedersen, C. B. et al. (2012). GAGE cancer-germline antigens are recruited to the nuclear envelope by germ cell-less (GCL). PloS One, 7 (9): e45819. Glotzer, B. J., Saffich, R., Glotzer, M. & Ephrussi, A. (1997). Cytoplasmic flows localize injected oskar RNA in Drosophila oocytes. Current Biology, 7: 326– 337. Gonzalez-Reyes, A., Elliott, H., &St Johnston, D. (1995). Polarization of both major body axes in Drosophila by gurken-torpedo signalling. Nature, 375: 654–658. Gönczy, P., Echeverri, C., Oegema, K. et al. (2000). Functional genomic analysis of cell division in C. elegans using RNAi of genes on chromosome III. Nature, 408: 331-336. Gruenbaum, Y., Margalit, A., Goldman, R. D. et al. (2005). The nuclear lamina comes of age. Nature Reviews. Molecular Cell Biology, 6: 21–31. Hamada, F., Tomoyasu, Y., Takatsu, Y. et al. (1999). Negative regulation of Wingless signaling by D-axin, a Drosophila homolog of axin. Science, 283 (5408): 1739-1742 Heeg-Truesdell, E. & LaBonne, C. (2006). Wnt signaling: A shaggy dogma tale. Current Biolology, 16 (2): R62-64. Hertwig, O. (1906) Allgemeine Biologie. Fischer Verlag Hertwig, O. (2013) Zeit- und Streitfragen der Biologie: Band 1 (Neuauflage). Dogma Verlag Holaska, J. M., Lee, K. K., Kowalski, A. K. & Wilson, K. L. (2003). Transcriptional repressor germ cell-less (GCL) and barrier to autointegration factor (BAF) compete for binding to emerin in vitro. The Journal of Biological Chemistry, 278: 6969–6975. Holen, T. (2005). Mechanisms of RNAi: mRNA cleavage fragments may indicate stalled RISC. Journal of RNAi and Gene Silencing, 1 (1): 21-25. Horn, T. & M. Boutros (2010). E-RNAi: a web application for the multi-species design of RNAi reagents--2010 update. Nucleic Acids Research, 38 (Web issue): W332-339. Howard, K. & Ingham, P. (1986). Regulatory Interactions between the Segmentation Genes fushi tarazu, hairy, and engrailed in the Drosophila Blastoderm. Cell, 44: 949–957. Huvenne, H. & Smagghe, G. (2013). Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: A review. Journal of Insect Physiology, 56 (3): 227–235 152 Literaturverzeichnis Illmensee, K., Mahowald, A. P. & Loomis, M. R. (1976). The ontogeny of germ plasm during oogenesis in Drosophila. Developmental Biology, 49: 40–65. Ingham, P. W., Ish-Horowicz, D. & Howard, K., R. (1986). Correlative changes in homoeotic and segmentation gene expression in Krüppel mutant embryos of Drosophila. The EMBO journal, 5 (7): 1659–1665. Jackson, A. L. & Linsley, P. S. (2004). Noise amidst the silence : off-target effects of siRNAs ?. Trends in Genetics, 20 (11): 521–524 Jagla, B., Aulner, N. Kelly, P. D. et al. (2005). Sequence characteristics of functional siRNAs. RNA, 11 (6): 864-872. Jiménez, G., Guichet, A., Ephrussi, A., Jime, G. & Casanova, J. (2000). Drosophila terminal and dorsoventral patterning Relief of gene repression by Torso RTK signaling: role of capicua in Drosophila terminal and dorsoventral patterning. Genes & Development, 14: 224–231. Jongens, T. A, Hay, B., Jan, L. Y. & Jan, Y. N. (1992). The germ cell-less gene product: a posteriorly localized component necessary for germ cell development in Drosophila. Cell, 70: 569–84. Jongens, T. A, Ackerman, L. D., Swedlow, J. R. et al. (1994). Germ cell-less encodes a cell type-specific nuclear pore-associated protein and functions early in the germ-cell specification pathway of Drosophila. Genes & Development, 8: 2123–2136. Jürgens, G., Lehmann, R., Schardin, M. & Nüsslein-Volhard, C. (1986). Segmental organisation of the head in the embryo of Drosophila melanogaster - A blastoderm fate map of the cuticle structures of the larval head. Roux’s Archives of Developmental Biology, 195: 359–377. Kenyon, C. (1994). If birds can fly, why can’t we? Homeotic genes and evolution. Cell, 78 (2): 175–180. Kimura, T., Yomogida, K., Iwai, N., Kato, Y. & Nakano, T. (1999). Molecular cloning and genomic organization of mouse homologue of Drosophila germ cell-less and its expression in germ lineage cells. Biochemical and Biophysical Research Communications, 262: 223–230. Kimura, T., Ito, C., Watanabe, S. et al. (2003). Mouse Germ Cell-Less as an Essential Component for Nuclear Integrity. Molecular and Cellular Biology, 23 (4): 1304–1315. King, R. C. (1970). Ovarian Development in Drosophila melanogaster. New York, Academic Press. 153 Literaturverzeichnis Kittelmann, S., Ulrich, J., Posnien, N. & Bucher, G. (2013). Changes in anterior head patterning underlie the evolution of long germ embryogenesis. Developmental Biology, 374 (1): 174–184. Kitzmann, P., Schwirz, J., Schmitt-Engel, C. & Bucher, G. (2013). RNAi phenotypes are influenced by the genetic background of the injected strain. BMC genomics, 14 (5). Kleiman, S. E., Yogev, L., Gal-Yam, E. N. et al. (2003). Reduced Human Germ Cell-Less ( HGCL ) Expression in Azoospermic Men With Severe Germinal Cell Impairment. Journal of Andrology, 24 (5): 670–675. Klingler, M., Erdélyi, M., Szabad, J. & Nüsslein-Volhard, C. (1988). Function of torso in determining the terminal anlagen of the Drosophila embryo. Nature, 335 (6187): 275–277. Klingler, M. (2004). Quick guide Tribolium. Current Biology, 14 (17): 639–640. Klomp, J., Athy, D., Kwan, C. W. et al. (2014). A novel cysteine-clamp gene establishes head-to-tail polarity in the midge Chironomus riparius. 55th Annual Drosophila Research Conference. Abstract. Kotkamp, K., Klingler, M. & Schoppmeier, M. (2010). Apparent role of Tribolium orthodenticle in anteroposterior blastoderm patterning largely reflects novel functions in dorsoventral axis formation and cell survival. Development, 137: 1853–1862. Krause, G. (1939). Die Eitypen der Insekten. Biologisches Zentralblatt, 59: 495–536. Krumlauf, R. (1994). Hox Genes in Vertebrate Development. Cell, 78: 191–201. Kumar, R., Conklin, D. S. & Mittal, V. (2003). High-throughput selection of effective RNAi probes for gene silencing. Genome Research, 13 (10): 2333-2340. la Thangue, N.B. (1994). DRTF1/E2F: an expanding family of heterodimeric transcription factors implicated in cell-cycle control. Trends in Biochemical Science, 19 (3): 108–114. Lam, E.W.-F. & La Thangue, N. B. (1994). DP and E2F proteins: co- ordinating transcription with cell cycle progression. Current Opinion in Cell Biolology. 6 (6): 859–866 Lasko, P. (2011). Posttranscriptional regulation in Drosophila oocytes and early embryos. Wiley Interdisciplinary Reviews, 2: 408–416. Lavery, K. S. & King, T. H. (2003). Antisense and RNAi: powerful tools in drug target discovery and validation. Current opinion in Drug Discovery & Development, 6 (4): 561–569. 154 Literaturverzeichnis Leatherman, J. L., Kaestner, K. H. & Jongens, T. A. (2000). Identification of a mouse germ cell-less homlogue with conserved activity in Drosophila. Mechanisms of Development, 92: 145–153. Leatherman, J. L., Levin, L., Boero, J. & Jongens, T. A. (2002). germ cell-less acts to repress transcription during the establishment of the Drosophila germ cell lineage. Current Biology , 12: 1681–1685. Lehmann, R. & Nüsslein-Volhard, C. (1991). The maternal gene nanos has a central role in posterior pattern formation of the Drosophila embryo. Development, 112 (3): 679–691. Lemke, S., Busch, S. E., Antonopoulos, D. A. et al., (2010). Maternal activation of gap genes in the hover fly Episyrphus. Development, 137: 1709-1719. Lewis, E. B. (1978). A gene complex controlling segmentation in Drosophila. Nature, 276: 565–570. Liu, P. Z. & Kaufman, T. C. (2005). Short and long germ segmentation : unanswered questions in the evolution of a developmental mode. Evolution & Development, 7 (6): 629–646. Lohs-Schardin, M., Cremer, C. & Nüsslein-Volhard, C. (1979). A Fate Mapmap for the larval epidermis of Drosophila melanogaster: localized cuticle defects following irradiation of the blastoderm with an ultraviolet laser microbeam. Developmental Biology, 73: 239–255. Lorenzen, M. D., Kimzey, T., Shippy, T. D. et al. (2007). piggyBac-based insertional mutagenesis in Tribolium castaneum using donor/helper hybrids. Insect Mol Biol. 16: 265-75. Lum, L., Yao, S., Mozer, B. et al. (2003). Identification of Hedgehog Pathway Components by RNAi in Drosophila Cultured Cells. Science, 299: 2039-2045. Luschnig, S., Moussian, B., Krauss, J. et al. (2004). An F1 genetic screen for maternal-effect mutations affecting embryonic pattern formation in Drosophila melanogaster. Genetics, 167 (1): 325-342. Lynch, J. Panfilio, K. A. & Nunes da Fonseca, R. (2009). As Tribolium matures as a model insect, coleopteran community congregates in Cologne. Development Genes and Evolution, 219, (9-10): 531-533. Ma, Y., Creanga, A., Lum, L., & Beachy, P.A. (2006). Prevalence of off-target effects in Drosophila RNA interference screens. Nature, 443: 359-363. Macdonald, P. & Struhl, G. (1985). A molecular gradient in early Drosophila embryos and its role in specifying the body pattern. Nature, 324: 537–545. 155 Literaturverzeichnis Macdonald, P. M. (2011). mRNA localization: assembly of transport complexes and their incorporation into particles. Current Opinion in Genetics & Development, 21: 407–413. Maekawa, M., Ito, C., Toyama, Y., et al. (2004). Stage-specific expression of mouse germ cell-less-1 (mGCL-1), and multiple deformations during mgcl-1 deficient spermatogenesis leading to reduced fertility. Archives of Histology and Cytology, 67 (4): 335–347. Mahone, M., Saffman, E. E. & Lasko, P. F. (1995). Localized Bicaudal-C RNA encodes a protein containing a KH domain, the RNA binding motif of FMR1. The EMBO Journal., 14: 2043-2055. Mansharamani, M. & Wilson, K. L. (2005). Direct binding of nuclear membrane protein MAN1 to emerin in vitro and two modes of binding to barrier-toautointegration factor. The Journal of Biological Chemistry, 280: 13863–870. Martinez-Arias, A., Baker, N. E. & Ingham, P. W. (1988). Role of segment polarity genes in the definition and maintenance of cell states in the Drosophila embryo. Development, 103: 157–170. Masuhara, M., Nagao, K., Nishikawa, M., Kimura, T. & Nakano, T. (2003). Enhanced degradation of MDM2 by a nuclear envelope component, mouse germ cell-less. Biochemical and Biophysical Research Communications, 308: 927– 932. Mattout-Drubezki, A. & Gruenbaum, Y. (2003). Dynamic interactions of nuclear lamina proteins with chromatin and transcriptional machinery. Cellular and Molecular Life Sciences, 60: 2053–2063. McGinnis, W. & Krumlauf, R. (1992). Homeobox Genes and Axial Patterning. Cell, 66: 283–302. McGregor, M., Pechmann, E., Schwager., E. E & Damen, W. G. M. (2009). An ancestral regulatory network for posterior development in arthropods. Communicative & Integrative Biology, 2 (2): 174– 176 Meister, G. & Tuschl, T. (2004). Mechanisms of gene silencing by double-stranded RNA. Nature, 431: 343–9. Micklem, D. R., Dasgupta, R., Elliott, H. et al.(1997). The mago nashi gene is required for the polarisation of the oocyte and the formation of perpendicular axes in Drosophila. Current Biology, 7:468–478. Miklos, G. L. G. & Rubin, G. M. (1996). The Role of the Genome Project in Determining Gene Function: Insights from Model Organisms. Cell, 86: 521-529. 156 Literaturverzeichnis Mlodzik, M. & Gehring, W. (1987). Hierachy of the genetic interactions that specify the anterior-posterior segmentation pattern of the Drosophila embryo monitored by caudal protein expression. Development, 101: 421-435. Mohler, J. & Wieschaus, E. (1986). Dominant maternal-effect mutations of Drosophila melanogaster causing the production of double abdomen embryos. Genetics, 112: 269-284. Moore, J., Han, H. & Lasko, P. (2009). Bruno negatively regulates germ cell-less expression in a BRE-independent manner. Mechanisms of Development, 126: 503–516. Morgan, T. H. (1915). Localization of the Herditary Material in the Germ Cells. Proc Natl Acad Sci USA. 1 (7): 420–429. Murayama, E., Katoh, M., Kanebayashi, A. et al. (2007). Germ cell-less like-2 protein is a new component of outer dense fibers in rat sperm flagella. Reproduction, 134: 749–756. Nagy, L. M. & Carroll, S. (1994). Conservation of wingless patterning functions in the short-germ embryos of Tribolium castaneum. Nature, 367: 460-463. Napoli, C., Lemieux, C. & Jorgensen, R. (1990). Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. The Plant Cell, 2 (4): 279–289. Ni, J. Q., Zhou, R., Czech, B. et al. (2011). A genome-scale shRNA resource for transgenic RNAi in Drosophila. Nat Methods, 8: 405-7. Nili, E., Cojocaru, G. S., Kalma, Y., Ginsberg, D. et al. (2001a). Nuclear membrane protein LAP2beta mediates transcriptional repression alone and together with its binding partner GCL (germ-cell-less). Journal of Cell Science, 114: 3297– 3307. Nili, E., Cojocaru, G. S., Collin, G. B. et al. (2001b). The Human Germ Cell-Less (HGCL): A Candidate Gene for Alström Syndrome. The Journal of Endocrine Genetics, 2: 29–35. Nunes da Fonseca, R., von Levetzow, C. & Kalscheuer, P. et al. (2008). Selfregulatory circuits in dorsoventral axis formation of the short-germ beetle Tribolium castaneum. Developmental Cell, 14: 605–615 Nunes da Fonseca, R., Lynch, J. A. & Roth, S. (2009). Evolution of axis formation: mRNA localization, regulatory circuits and posterior specification in non-model arthropods. Current Opinion in Genetics & Development, 19: 404–411. Nunes da Fonseca, R., van der Zee, M. & Roth, S., (2010). Evolution of extracellular Dpp modulators in insects: the roles of tolloid and twisted-gastrulation in dor- 157 Literaturverzeichnis soventral patterning of the Tribolium embryo. Developmental Biology, 345: 80–93. Nüsslein-Volhard, C. (1977). Genetic analysis of pattern-formation in the embryo of Drosophila melanogaster: characterization of the maternal-effect mutant bicaudal. Roux’ Arch. Dev. Biol, 183: 249-268. Nüsslein-Volhard, C. (1991). Determination of the embryonic axes of Drosophila. Development Supplement, 1: 1-10. Nüsslein-Volhard, C. & Wieschaus, E. (1980). Mutations affecting segment number and polarity in Drosophila. Nature, 287: 795–801. Nüsslein-Volhard, C., Wieschaus, E. & Kluding, H. (1984). Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. Roux’s Archives of Developmental Biology, 193: 267–282. Nüsslein-Volhard, C., Frohnhöfer, H. G. & Lehmann, R. (1987). Determination of Anteroposterior Polarity in Drosophila. Science, 238: 1675–1681. Pignoni, F., Steingrimsson, E. & Lengyel, J. A. (1992). bicoid and the terminal system activate tailless expression in the early Drosophila embryo. Development, 115: 239-251. Panfilio K. A. (2009). Late extraembryonic development and its zen-RNAi-induced failure in the milkweed bug Oncopeltus fasciatus. Developmental Biology, 333: 297–311 Panfilio, K. A. & Roth, S. (2010). Epithelial reorganization events during late extraembryonic development in a hemimetabolous insect. Developmental Biology, 340 (1): 100–115. Panfilio, K. A, Oberhofer, G. & Roth, S. (2013). High plasticity in epithelial morphogenesis during insect dorsal closure. Biology Open, 2 (11): 1108–1118. Patel, N. H., Condron, Barry, G. & Zinn, K. (1994). Pair-rule expression patterns of even-skipped are found in both short- and long-germ beetles. Nature, 367: 429– 434. Peel, A. D., Chipman, A. D. & Akam, M. (2005). Arthropod Segmentation: beyond the Drosophila paradigm. Nature Reviews Genetics, 6: 906–916. Peel, A. D. (2008). The evolution of developmental gene networks: lessons from comparative studies on holometabolous insects. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences, 363: 1539–1547. Peel, A.D. & Averof, M. (2010). Early asymmetries in maternal transcript distribution associated with a cortical microtubule network and a polar body in the bee- 158 Literaturverzeichnis tle Tribolium castaneum. Developmental dynamics. American Association of Anatomists, 239: 2875-2887. Pokrywka, N. J. & Stephenson, E. C. (1991). Microtubules mediatthe localization of bicoid mRNA during Drosophila oogenesis. Development, 113: 55–66. Porcher, A. & Dostatni, N. (2010). The bicoid morphogen system. Current Biology, 20: R249–254. Posnien, N., Schinko, J. Grossmann, D. et al. (2009). RNAi in the Red Flour Beetle (Tribolium). Cold Spring Harbor Protocols. Posnien, N. & Bucher, G. (2010). Formation of the insect head involves lateral contribution of the intercalary segment, which depends on Tc-labial function. Developmental Biology, 338 (1): 107–116. Posnien, N., Koniszewski, N. D. B., Hein, H. J. & Bucher, G. (2011). Candidate gene screen in the red flour beetle Tribolium reveals six3 as ancient regulator of anterior median head and central complex development. PLoS genetics, 7 (12): e1002416. Qiu, S., Adema, C. M., & Lane, T. (2005). A computational study of off-target effects of RNA interference. Nucleic Acids Res, 33 (6): 1834-1847. Rittenhouse, K. R. & Berg, C. A. (1995). Mutations in the Drosophila gene bullwinkle cause the formation of abnormal eggshell structures and bicaudal embryos. Development, 121: 3023-3033. Robertson, S. E., Dockendorff, T. C., Leatherman, J. L.et al. (1999). germ cell-less is required only during the establishment of the germ cell lineage of Drosophila and has activities which are dependent and independent of its localization to the nuclear envelope. Developmental Biology, 215: 288–297. Rosenberg, M. I., Lynch, J. A. & Desplan, C. (2009). Heads and tails: evolution of antero-posterior patterning in insects. Biochimica et Biophysica Acta, 1789: 333–342. Roth, S. & Hartenstein, V. (2008). Development of Tribolium castaneum. Development Genes and Evolution, 218: 115–118. Rual, J.F., Klitgord, N., & Achaz, G. (2007). Novel insights into RNAi off-target effects using C. elegans paralogs. BMC Genomics, 8: 106. Ruden, D. M., Cui, W., Sollars, V. & Alterman M. (1997). A Drosophila kinesin-like protein, Klp38B, functions during meiosis, mitosis and segmentation. Develomental Biology, 191: 284-296. Rushlow, C. & Levine, M. (1990). Role of the zerknüllt gene in dorsal-ventral pattern formation in Drosophila. Advanced Genetics, 27: 277–307. 159 Literaturverzeichnis Sander, K. (1976). Specification of the basic body pattern in insect embryogenesis. Advances in Insect Physiology, 12: 125–238 Sander, K. & Lehmann, R. (1988). Drosophila nurse cells produce a posterior signal required for embryonic segmentation and polarity. Nature, 335: 68–70. Sarrazin, A. F., Peel, A. D. & Averof, M. (2012). A segmentation clock with twosegment periodicity in insects. Science, 336: 338–341. Savant-Bhonsale, S. & Montell, D. J. (1993). torso-like encodes the localized determinant of Drosophila terminal pattern formation. Genes & Development, 7: 2548–2555. Scacheri, P. C., Rozenblatt-Rosen, O., Caplen, N. J. et al. (2004). Short interfering RNAs can induce unexpected and divergent changes in the levels of untargeted proteins in mammalian cells. Proc. N. A. S. USA, 101: 1892-1897. Schaub, C. & Frasch, M. (2013). Org-1 is required for the diversification of circular visceral muscle founder cells and normal midgut morphogenesis. Developmental Biology, 376: 245–259. Schinko, J. B., Kreuzer, N., Offen, N., Posnien, N., Wimmer, E. A. & Bucher, G. (2008). Divergent functions of orthodenticle, empty spiracles and buttonhead in early head patterning of the beetle Tribolium castaneum (Coleoptera). Developmental Biology, 317 (2): 600–613. Schmidt-Ott, U. (2000). The amnioserosa is an apomorphic character of cyclorrhaphan flies. Devolopment Genes and Evolution, 210: 373– 376. Schmitt-Engel, C. (2010). Die Funktion von Genen der posterioren Gruppe und die Etablierung eines genomweiten RNAi-Screens in Tribolium castaneum. Dissertation. Friedrich-Alexander-Universität. Erlangen. Schmitt-Engel, C., Cerny, A. C. & Schoppmeier, M. (2012). A dual role for nanos and pumilio in anterior and posterior blastodermal patterning of the short-germ beetle Tribolium castaneum. Developmental Biology, 364: 224–235. Schnorrer, F., Schonbauer, C., Langer, C. C. H. et al. (2010). Systematic genetic analysis of muscle morphogenesis and function in Drosophila. Nature, 464: 287-291. Schoppmeier, M. & Schröder, R. (2005). Maternal torso signaling controls body axis elongation in a short germ insect. Current Biology, 15: 2131–2136. Schoppmeier, M., Fischer, S., Schmitt-Engel, C., Löhr, U. & Klingler, M. (2009). An ancient anterior patterning system promotes caudal repression and head formation in ecdysozoa. Current Biology, 19: 1811–1815. 160 Literaturverzeichnis Schröder, R., Eckert, C., Wolff, C. & Tautz, D. (2000). Conserved and divergent aspects of terminal patterning in the beetle Tribolium castaneum. PNAS, 97 (12): 6591–6596. Schröder, R. (2003). The genes orthodenticle and hunchback substitute for bicoid in the beetle Tribolium. Nature, 422: 621–625 Schröder, R. (2006). vasa mRNA accumulates at the posterior pole during blastoderm formation in the flour beetle Tribolium castaneum. Development Genes and Evolution, 216: 277-283 Schröder, R., Beermann, A., Wittkopp, N. & Lutz, R. (2008). From development to biodiversity – Tribolium castaneum, an insect model organism for short germband development. Development Genes & Evolution, 217: 119–126. Scuderi, A. & Letsou, A. (2005). Amnioserosa is required for dorsal closure in Drosophila. Developmental Dynamics, 232: 791–800. Seidel, F., Bock, E. & Krause, G. (1940). Die Organisation des Insekteneis. Die Naturwissenschaften. 28: 433-446. Sen, G. L. & Blau, H. M. (2006). A brief history of RNAi: the silence of the genes. The FASEB Journal, 20 (9): 1293-1299. Seydoux, G., Mello, C., Pettitt, J. & Wood, W. (1996). Repression of gene expression in the embryonic germ lineage of C. elegans. Nature, 382: 713–716. Shapiro, R. S. & Anderson, K. V. (2006). Drosophila Ik2, a member of the IκB kinase family, is required for mRNA localization during oogenesis. Development, 133: 1467-1475. Shimomura, O., Johnson, F. H. & Saiga, Y. O. (1962). Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. Journal of Cellular and Comparative Physiology, 59: 223– 239. Shippy, T.D., Tomoyasu, Y., Nie, W. et al. (2008). Do teashirt family genes specify trunk identity? Insights from the single tiptop/teashirt homolog of Tribolium castaneum. Development genes and evolution, 218: 141-152. Simmer, F., Moorman, C., van der Linden, A. M. et al. (2003). Genome-Wide RNAi of C. elegans Using the Hypersensitive rrf-3 Strain Reveals Novel Gene Functions. PLoS Biol, 1 (1): e12. Simpson-Brose, M. Treisman, J. & Desplan, C. (1994). Synergy between the hunchback and bicoid morphogens is required for anterior patterning in Drosophila. Cell, 78 (5): 855–865. 161 Literaturverzeichnis Snove, O. & Holen, T. (2004). Many commonly used siRNAs risk off-target activity. Biochem. Biophys. Res. Commun., 319 (1): 256-263. Sokoloff, A., 1974. The Biology of Tribolium. With special emphasis on genetic aspects. Oxford University Press, Oxford. Sonnichsen, B., Koski, L. B., Walsh, A. et al. (2005). Full-genome RNAi profiling of early embryogenesis in Caenorhabditis elegans. Nature, 434: 462-469. Sprenger, F., Stevens. L. M. & Nüsslein-Volhard, C. (1989). The Drosophila gene torso encodes a putative receptor tyrosine kinase. Nature, 338: 478-483. Stauber, M., Taubert, H., & Schmidt-Ott, U. (2000). Function of bicoid and hunchback homologs in the basal cyclorrhaphan fly Megaselia (Phoridae). Proc. Natl. Acad. Sci. USA, 97: 10844–10849. Stevens, L. M., Frohnhofer., H. G., Klingler, M. & Nüsslein-Volhard, C. (1990). Localized requirement for torsolike expression in follicle cells for the development of terminal anlagen of the Drosophila embryo. Nature, 346: 660-663 Stevens, L. M., Beuchle, D, Jurcsak, Tong, X. und Stein, D. (2003). The Drosophila Embryonic Patterning Determinant Torsolike Is a Component of the Eggshell. Current Biology. 13 (12): 1058-1063. St Johnston, D. & Nüsslein-Volhard, C. (1992). The origin of pattern and polarity in the Drosophila embryo. Cell, 68 (2): 201–19. St Johnston, D. (1995). The intracellular localization of messenger RNAs. Cell, 81 (2): 161–170. St Johnston, D. (2002). The art and design of genetic screens: Drosophila melanogaster. Nat Rev Genet, 3 (3): 176-188. Stothard, P. (2000). The sequence manipulation suite: JavaScript programs for analyzing and formatting protein and DNA sequences. BioTechniques, 28: 11021104. Strobl, F. & Stelzer, E. H. K. (2014). Non-invasive long-term fluorescence live imaging of Tribolium castaneum embryos. Development, (May): 1–8. Struhl, G. (1989). Differing Strategies for Organizing Anterior and Posterior Body Pattern in Drosophila Embryos, Nature, 338: 741 – 744. Tautz, D., Friedrich, M. & Schröder, R. (1994). lnsect embryogenesis - what is ancestral and what is derived?. Development supplement: 193–199. The Embryo Project Encyclopedia (http://embryo.asu.edu/) 162 Literaturverzeichnis Threadgill, D. W., Dulgosz, A. A., Hansen L. A. et al. (1995). Targeted Disruption of Mouse EGF Receptor: Effect of Genetic Background on Mutant Phenotype. Science, 269 (5221): 230-234. Tijsterman, M., Ketting, R. F. & Plasterk, R. H. A. (2002). The Genetics of RNA Silencing. Annual Review of Genetics, 36: 489–519. Tijsterman, M. & Plasterk, R. H. A. (2004). Dicers at RISC. Cell, 117 (1): 1-3. Tomoyasu, Y. & Denell, R. E. (2004). Larval RNAi in Tribolium (Coleoptera) for analyzing adult development. Dev. Genes and Evolution, 214: 575–578. Tomoyasu, Y., Miller, S. C., Tomita, S. et al. (2008). Exploring systemic RNA interference in insects: a genome-wide survey for RNAi genes in Tribolium. Genome Biology, 9 (1): R10. Trauner, J. & Büning, J. (2007). Germ-cell cluster formation in the telotrophic meroistic ovary of Tribolium castaneum (Coleoptera, Polyphaga, Tenebrionidae) and its implication on insect phylogeny. Dev. Genes and Evolution, 217: 13–27 Trauner, J. (2009). Analyse der Oogenese und Insertionsmutagenese in Tribolium castaneum, Dissertation, Friedrich-Alexander-Universität. Tribolium Genome Sequencing Consortium (Richards, S., Gibbs, R. A, Weinstock, G. M. et al. (2008). The genome of the model beetle and pest Tribolium castaneum. Nature, 452: 949–955. Turner, Dave. Department for Neurosciences, University of Michigan, Ann Arbor, MI. http://sitemaker.umich.edu/dlturner.vectors/home Vaishnaw, A. K., Gollob, J., Gamba-Vitalo, C., Hutabarat, R., Sah, D., Meyers, R. & Maraganore, J. (2010). A Status Report on RNAi Therapeutics - Review, Silence - A Journal of RNA Regulation, 1 (14): 1–13. van der Zee, M., Berns, N. & Roth, S. (2005). Distinct functions of the Tribolium zerknüllt genes in serosa specification and dorsal closure. Current Biology, 15 (7): 624–636. van der Zee, M., Stockhammer, O., von Levetzow, C. et al. (2006). Sog/Chordin is required for ventral-to-dorsal Dpp/BMP transport and head formation in a short germ insect. Proceedings of the National Academy of Sciences of the United States of America, 103 (44): 16307–16312. van Eeden, F. & St Johnston, D. (1999). The polarisation of the anterior-posterior and dorsal-ventral axes during Drosophila oogenesis. Current Opinion in Genetics & Development, 9 (4): 396–404. Verdel, A., Jia. S., Gerber. S. et al. (2004). RNAi-Mediated Targeting of Heterochromatin by the RITS Complex. Science, 303 (5658): 672-676. 163 Literaturverzeichnis von Baer, C. E. (1828) Über Entwicklungsgeschichte der Thiere. Beobachtung und Reflexion, Gebrüder Bornträger, Königsberg Watanabe, Y. (2011) Overview of plant RNAi. Methods in Molecular Biology, 744: 1– 11. Wieschaus, E., Nüsslein-Volhard, C. & Kluding, H. (1984). Krüppel, a gene whose activity is required early in the zygotic genome for normal embryonic segmentation. Developmental Biology, 104 (1): 172– 186 Wilhelm, J. E., Mansfield, J., Hom-Booher, N. et al. (2000). Isolation of a ribonucleoprotein complex involved in mRNA localization in Drosophila oocytes. Cell Biol, 148 (3): 427-40. Wolff, C., Sommer, R., Schröder, R. et al. (1995). Conserved and divergent expression aspects of the Drosophila segmentation gene hunchback in the short germ band embryo of the flour beetle Tribolium. Development, 121: 4227-4236. Wolff, C., Schröder, R., Schulz, C., Tautz, D. & Klingler, M. (1998). Regulation of the Tribolium homologues of caudal and hunchback in Drosophila: evidence for maternal gradient systems in a short germ embryo. Development, 125: 3645– 3654. Zimyanin, V. L., Belaya, K., Pecreaux, J. et al. (2008). In Vivo Imaging of oskar mRNA Transport Reveals the Mechanism of Posterior Localization. Cell, 134: 843– 853. 164 Lebenslauf Lebenslauf Persönliche Angaben Name: Nicole Troelenberg Geburtsdatum: 28.01.1984 Geburtsort: Magdeburg (Sachsen Anhalt) Schulbildung 1990-1994: Grundschule Gneisenauring, Magdeburg 1994-2003: Albert-Einstein-Gymnasium, Magdeburg Hochschulausbildung 2003-2009 Studium der Biologie, Martin-LutherUniversität, Halle-Wittenberg Diplomarbeit, Lehrstuhl für Verhaltensbiologie der MLU „Inter- und intraspezifische olfaktorische Diskrimination bei Kurzschwanz-Zwerghamstern (Phodopus spec.)“ Diplom (1,3) 2009-2010 Auslandsaufenthalt 2010-2014 Promotion, Lehrstuhl für Entwicklungsbiologie der Friedrich-AlexanderUniversität (Erlangen) „Die Funktion von germ cell-less und homeobrain in der Differenzierung terminaler Identitäten im Kurzkeim-Insekt Tribolium castaneum“ 165 Publikationen und Vorträge Publikationen und Vorträge Publikationen Troelenberg, N., Weinert, D. & Fritzsche, P. (2009). Inter- und intraspecific olfactory discrimination in dwarf hamsters of the genus Phodopus, Poster, Ethologische Versammlung, Göttingen Troelenberg, N., Dao, V.A., Richter, T., Bucher, G. & Klingler, M. (2013). Tribolium germ cell-less: mirror-image abdomina in a short germ beetle differ from bicaudal phenotypes in Drosophila, Poster, GfE Meeting, Heidelberg Vorträge Troelenberg, N. & Klingler, M. (2012). Mirror Image Embryos and A-P Axis Formation in Tribolium, Tribolium Satellite Meeting, Euro Evo Devo, Lissabon, Portugal Troelenberg, N., Dao V. A., Richter T., Bucher G. & Klingler, M. (2013) Tribolium germ cell-less: mirror-image abdomina in a short germ beetle differ from bicaudal phenotypes in Drosophila, 106. Jahrestagung der DZG, München 166 Danksagung Danksagung Obwohl als Letzte genannt, wäre die vorliegende Dissertation ohne die folgenden Personen so kaum möglich gewesen. Vorneweg möchte ich mich bei Professor Martin Klingler bedanken, für die Möglichkeit, an diesem Projekt zu arbeiten, für die vielen Denkanstöße und Anregungen sowie die Zeit, die er sich für Probleme und fachliche Diskussionen genommen hat. Danke auch an Michael, Ralph und Jochen, die als Dreigestirn immer ein Ohr für Fragen, anregende Diskussionen oder einen Plausch für Zwischendurch hatten. Dank auch Kristen Panfilio, die mit ihren Videos Leben in germ cell-less brachte und viel Enthusiasmus in das Projekt steckte, sowie meinen „Vorgängerinnen“ Jutta und Irene und den anderen Kollegen für die Geduld in der Beantwortung all meiner Fragen, die Anregungen und den täglichen Schwatz, der das Arbeiten hier so angenehm machte; Upal, meiner Labor- und Reisegefährtin, für ihre stets heitere Laune und die gute Zusammenarbeit; Denise und Fabi für ihre Hilfe, wenn mal wieder irgendwas im Argen lag; Doro möchte ich für die lustigen Gespräche und Sequenzanalysen danken und Nadi für die Hilfe sobald es um Ovare und Antikörper ging. Bedanken möchte ich mich auch bei Suparna und den Studenten, die im Zuge eines Praktikums, der Bachelor- oder Masterarbeit bzw. als Hiwi frischen Wind ins Labor gebracht haben: Anna, Christina, Marion, Matthias der Blonde, Matthias der Große und Matthias der Dritte, Angelika, Johannes und Katharina sowie all den anderen aus den Laboren Frasch, Rübsam und Schambony. Mein Dank gebührt weiterhin all den Menschen, die das Labor nicht nur mit ihrer Arbeit, sondern auch durch ihre aufgeschlossene, freundliche Art am Laufen halten: Tina, Claudia, Thomas, Tobi, Maria und Marianne. Zum Abschluss möchte ich mich bei meiner Familie – explizit, aber nicht allein, meinen Eltern – bedanken! Für beständigen Ansporn, Unterstützung und Ratschläge sowie die Ermutigungen, wenn der Druck unüberwindlich schien. Meinen Freunden, insbesondere Anja, Anna und Martina, danke ich fürs Mitleiden und Zuhören, Aufbauen und Bestätigen – ihr seid unbezahlbar! 167 Eidesstattliche Erklärung Eidesstattliche Erklärung Ich erkläre hiermit, dass ich die vorliegende Dissertation mit dem Titel Die Funktion von germ cell-less und homeobrain in der Differenzierung terminaler Identitäten im Kurzkeim-Insekt Tribolium castaneum selbstständig ohne Hilfe Dritter und ohne Benutzung anderer als der angegebenen Quellen und Hilfsmittel verfasst habe. Alle aus Quellen wörtlich oder sinngemäß entnommene Stellen sind als solche kenntlich gemacht. Ich erkläre weiterhin, dass ich nie versucht habe die vorliegende Dissertation in gleicher oder ähnlicher Form in einem Prüfungsverfahren vorzulegen. Ich bin mir bewusst, dass eine falsche Erklärung rechtliche Folgen haben wird. Erlangen, Unterschrift 168