Regulation der Zellteilung in Caulobacter crescentus - Max
Werbung

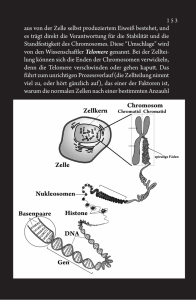
Thanbichler, Martin | Regulation der Zellteilung Tätigkeitsbericht 2008 Mikrobiologie/Ökologie Regulation der Zellteilung in Caulobacter crescentus Thanbichler, Martin; Max-Planck-Institut für terrestrische Mikrobiologie, Marburg Selbständige Nachwuchsgruppe - Prokaryontische Zellbiologie (MPG) Korrespondierender Autor Thanbichler, Martin E-Mail: [email protected] Zusammenfassung In den letzten Jahren wurden im Verlauf der Untersuchung zellulärer Organisation bei Bakterien große Fortschritte gemacht. Es stellte sich heraus, dass Bakterien ähnlich wie Eukaryonten komplexe regulatorische Mechanismen besitzen, um molekulare Prozesse in ihrem Inneren räumlich und zeitlich aufeinander abzustimmen. Die Funktion solcher Systeme soll in diesem Bericht am Beispiel der Koordination von Chromosomensegregation und Zellteilung in Caulobacter crescentus veranschaulicht werden. Abstract Recent advances in the study of bacterial cell biology have demonstrated that bacteria use complex regulatory mechanisms to ensure proper temporal and spatial regulation of cellular processes. In this article, the complexity of such systems is illustrated by the molecular pathway that coordinates chromosome segregation with cell division in Caulobacter crescentus. Einleitung Bakterien gehören, evolutionär gesehen, zu den erfolgreichsten Organismen auf unserem Planeten und sind in fast jeder ökologischen Nische in großer Artenvielfalt vertreten. Auf Grund ihrer überwiegend einzelligen Lebensweise und der Optimierung ihrer Physiologie auf hohe Energieeffizienz und schnelle Wachstumsraten sind ihre Genome im Vergleich zu denen eukaryotischer Zellen sehr klein. Dieser Umstand galt lange Zeit als Indiz für ein weitgehendes Fehlen intrazellulärer Organisation. Die Verbesserung mikroskopischer Verfahren und eine Anpassung zellbiologischer Methoden an prokaryotische Systeme hat in den letzten Jahren jedoch ungeahnte Einblicke in die Struktur und Funktion bakterieller Zellen ermöglicht. Dabei zeigte sich, dass Bakterien über zahlreiche molekulare Systeme verfügen, die zuvor ausschließlich eukaryotischen Zellen zugeschrieben worden waren. Besonders hervorzuheben ist unter anderem ihr reiches Repertoire an Zytoskelett-Elementen und dynamisch lokalisierten Proteinkomplexen, denen bei der räumlichen und zeitlichen Koordination zellulärer Prozesse zentrale Aufgaben zufallen [1]. Die Fortschritte der letzten Jahre führten zur Etablierung einer zellbiologisch ausgerichteten Mikrobiologie, deren Ziel es ist, ein umfassendes Verständnis zellulärer Organisation in Bakterien zu erlangen. Das Problem der Zellteilung Ein grundlegende Fragestellung in der Zellbiologie ist der Mechanismus der Zellteilung, also der Spaltung einer einzelnen Mutterzelle in zwei oder auch mehrere Tochterzellen. Die Nachkommenschaft muss dabei jeweils mit einer vollständigen Kopie der mütterlichen Erbinformationen ausgestattet © 2008 Max-Planck-Gesellschaft www.mpg.de Tätigkeitsbericht 2008 Thanbichler, Martin | Regulation der Zellteilung werden, um lebensfähig zu sein. Voraussetzung dafür sind eine korrekte Replikation des mütterlichen Chromosomensatzes, eine zuverlässige Verteilung der Kopien auf die zukünftigen Tochterzellen und schließlich eine präzise Teilung der Mutterzelle zwischen den separierten Tochterchromosomen. Um ein fehlerfreies Ablaufen der Zellteilung sicherzustellen, ist eine strikte räumliche und zeitliche Koordination der beteiligten Prozesse erforderlich. Die dafür verantwortlichen regulatorischen Mechanismen wurden bisher außer an Escherichia coli [2] vor allem am Modellorganismus Caulobacter crescentus untersucht. Der Zellzyklus von C. crescentus Ein charakteristisches Merkmal von C. crescentus ist dessen asymmetrische Zellteilung, die zur Entstehung von zwei morphologisch und physiologisch unterschiedlichen Tochterzellen führt [3]. Diese Asymmetrie bildet den Ausgangspunkt für einen ungewöhnlichen Entwicklungszyklus, in dessen Verlauf sich die begeißelte Tochterzelle zu einer Stielzelle differenziert (Abb. 1). Dazu wirft sie ihr Flagellum ab und generiert an demselben Zellpol, der vorher das Flagellum trug, einen Stiel. Dieser Entwicklungsschritt ist Voraussetzung für die Initiierung der Zellteilung, die anschließend über die restlichen Stadien des Zellzyklus hinweg abläuft. Nach Bildung eines neuen Flagellums an dem Stiel gegenüberliegenden Zellpol schnüren sich die beiden Tochterzellen voneinander ab. Dadurch entsteht eine neue Schwärmerzelle sowie eine Stielzelle, die unmittelbar mit dem nächsten Teilungszyklus beginnen kann. Dieser morphologische Zyklus ist eng mit der Dynamik der chromosomalen DNA verknüpft [4]. Die Schwärmerzelle enthält exakt eine Kopie eines zirkulären Chromosoms, das die gesamte Erbinformation von C. crescentus trägt. Dessen Replikation wird während der Differenzierung zur Stielzelle eingeleitet und dauert bis kurz vor Abschluss der Zellteilung an. Die beiden Kopien werden dabei im Lauf ihrer Synthese sukzessive auf die beiden Hälften der Stielzelle aufgeteilt, sodass nach deren medialer Teilung zwei Tochterzellen entstehen, die jeweils wieder eine Kopie des Chromosoms enthalten. Abb. 1: Zellzyklus von C. crescentus. Eine Schwärmerzelle, die immer exakt eine Kopie des Chromosoms (blau) enthält, differenziert sich nach Abwerfen des Flagellums zu einer Stielzelle. Diese initiiert die Replikation der chromosomalen DNA, wächst in die Länge und teilt sich medial nach Bildung eines neuen Flagellums. Dadurch werden eine Stielzelle und eine neue Schwärmerzelle freigesetzt, die beide jeweils wieder ein Chromosom tragen. Die Stielzelle kann unmittelbar mit dem nächsten Teilungszyklus beginnen, während die Schwärmerzelle vor der nächsten Zellteilung erst wieder den obligatorischen Differenzierungsschritt zur Stielzelle absolvieren muss. Urheber: Max-Planck-Institut für terrestrische Mikrobiologie/Thanbichler www.mpg.de © 2008 Max-Planck-Gesellschaft Thanbichler, Martin | Regulation der Zellteilung Tätigkeitsbericht 2008 Organisation und Dynamik der chromosomalen DNA Die Organisation bakterieller Chromosomen und die Dynamik ihrer Segregation waren bis vor kurzem noch weitgehend ungeklärt. Vielfach wurde vermutet, dass chromosomale DNA zufällig in der Zelle verteilt ist und die beiden Schwesterchromosomen nach Abschluss der Chromosomenreplikation passiv durch das Längenwachstum der Zelle voneinander separiert werden. Arbeiten an C. crescentus trugen aber wesentlich dazu bei, diese Vorstellung zu revidieren [5]. Die Arbeiten basierten auf einem Verfahren, das es erlaubt, beliebige Bereiche des Chromosoms mit fluoreszierenden Proteinen zu markieren und ihre Lokalisation in der Zelle anschließend mittels Fluoreszenzmikroskopie zu bestimmen. Eine Analyse von 112 verschiedenen Bereichen, die über das gesamte Chromosom verteilt waren, und die Auswertung von mehr als 50000 Zellen führte zu dem Schluss, dass chromosomale DNA eine definierte Architektur aufweist, die jedem Gen eine bestimmte Position innerhalb der Zelle zuweist (Abb. 2). Aufeinander folgende Abschnitte des Chromosoms sind dabei in kondensierter Form wie Perlen auf einer Kette aneinandergereiht und ringförmig in der Zelle angeordnet. Der Replikationsursprung, also der Bereich des Chromosoms, in dem dessen Replikation beginnt, liegt dabei als Fixpunkt am begeißelten Zellpol. Abb. 2: Architektur chromosomaler DNA in C. crescentus. A Schematische Darstellung der Organisation chromosomaler DNA in der Zelle. Der Replikationsursprung (ori) und der Terminus des Chromosoms (ter) sind an gegenüberliegenden Zellpolen positioniert, während alle anderen Bereiche des Chromosoms in kondensierter Form als ringförmige Struktur zwischen diesen beiden Punkten angeordnet sind. B Position verschiedener chromosomaler Abschnitte in der Zelle. Definierte Regionen des Chromosoms wurden mithilfe fluoreszierender Proteine markiert und anschließend mittels Fluoreszenzmikroskopie in der Zelle lokalisiert. Jeder Punkt des Graphen repräsentiert den Durchschnitt von Werten, die aus der Analyse von 500 Zellen mit identischer Markierung resultierten. Urheber: Max-Planck-Institut für terrestrische Mikrobiologie/Thanbichler © 2008 Max-Planck-Gesellschaft www.mpg.de Tätigkeitsbericht 2008 Thanbichler, Martin | Regulation der Zellteilung Das verwendete Markierungsverfahren ermöglichte es auch, die Bewegung von chromosomalen Bereichen während des Replikationsprozesses zu verfolgen. Diese Studien zeigten, dass die beiden Kopien des Replikationsursprungs unmittelbar nach ihrer Entstehung voneinander separiert werden, indem eine von ihnen schnell und in gerichteter Weise zum gegenüberliegenden Zellpol wandert. Ein ähnliches Verhalten wurde auch für andere chromosomale Abschnitte beobachtet. In allen untersuchten Fällen verblieb eine der beiden Kopien an ihrer ursprünglichen konservierten Position, während die andere kurz nach ihrer Synthese an die entsprechende Position in der gegenüberliegenden Zellhälfte bewegt wurde. Diese Ergebnisse führen zu dem Schluss, dass in Bakterien – anders als in Eukaryonten – die Segregation neu synthetisierter DNA schon während der Replikation erfolgt und die definierte Architektur des Chromosoms im Zuge der Segregation duplizierter Chromosomenabschnitte etabliert wird. Der molekulare Mechanismus, der für die Segregation der Chromosomen verantwortlich ist, ist Gegenstand intensiver Forschung, aber bisher noch ungeklärt. Positionierung des Zellteilungsapparats Es zeigte sich, dass die Architektur und Dynamik des Chromosoms in C. crescentus einen entscheidenden Einfluss auf die Positionierung des Zellteilungsapparats und somit auf das korrekte Ablaufen der Zellteilung haben. Die Einschnürung der Zelle wird durch einen Multienzymkomplex vermittelt, der als ringförmige Struktur an der Zellteilungsebene vorliegt [6]. Die Stelle, an der sich dieser Komplex formt, wird im Wesentlichen durch die Position des Proteins FtsZ bestimmt. Es handelt sich hierbei um ein zum eukaryotischen Tubulin homologes Protein, das den zukünftigen Ort der Zellteilung in Form von zellumspannenden, polymeren Filamenten markiert. FtsZ rekrutiert direkt und indirekt alle anderen Komponenten des Zellteilungsapparats und spielt eine entscheidende katalytische Rolle beim anschließenden Konstriktionsprozess. Die regulatorischen Mechanismen, die FtsZ den Weg zur Zellteilungsebene weisen, waren in C. crescentus lange Zeit unbekannt. Vor kurzem konnte jedoch das Protein MipZ als zentraler Regulator der Zellteilung in diesem Bakterium identifiziert werden [7]. Zellbiologische und biochemische Analysen zeigten, dass MipZ über ein Adapterprotein namens ParB mit einem bestimmten Bereich des Chromosoms in der Nähe des Replikationsursprungs interagiert. Genauso wie der Replikationsursprung befindet sich das Protein daher in Schwärmerzellen am begeißelten Zellpol, während es in späteren Stadien des Zellzyklus an beiden Polen der Zelle nachweisbar ist (Abb. 3). Im Gegensatz zu ParB, das einen klar definierten, punktförmigen Komplex bildet, ist der Regulator MipZ jedoch in Form eines Gradienten in der Zelle verteilt, wobei seine Konzentration an den Zellpolen am höchsten ist und zur Zellmitte hin langsam abnimmt. Es zeigte sich, dass MipZ die Polymerisierung des Zellteilungsproteins FtsZ inhibiert. Eine effiziente Assemblierung des Zellteilungsapparats ist daher nur in Bereichen der Zelle möglich, die kein MipZ aufweisen, also in der Regel in der Zellmitte. Dieser Mechanismus stellt sicher, dass die Teilung der Zelle erst nach Beginn der Chromosomenreplikation und -segregation eingeleitet wird und exakt zwischen den beiden neu entstehenden Kopien des Chromosoms stattfindet. www.mpg.de © 2008 Max-Planck-Gesellschaft Thanbichler, Martin | Regulation der Zellteilung Tätigkeitsbericht 2008 Abb. 3: Modell der Koordination von Chromosomensegregation und Zellteilung durch MipZ. Die Schwärmerzelle enthält ein Chromosom, dessen Replikationsursprung am begeißelten Zellpol positioniert ist. MipZ interagiert über das Adapterprotein ParB mit einem bestimmten Bereich des Chromosoms in der Nähe des Replikationsursprungs und wird so ebenfalls an den begeißelten Pol rekrutiert. Die inhibitorische Wirkung von MipZ auf die Polymerisation von FtsZ führt dazu, dass sich FtsZ am gegenüberliegenden Pol sammelt (A). Der Beginn der Chromosomenreplikation führt zur Duplikation des Replikationsursprungs. Beide Kopien werden sofort wieder mit MipZ und ParB besetzt. Im Zuge der Chromosomensegregation bewegt sich einer der beiden Komplexe an das gegenüberliegende Ende der Zelle, während der andere am Ausgangspunkt verbleibt (B). Durch die Positionierung von MipZ an beiden Zellpolen verschiebt sich das Konzentrationsminimum von MipZ (C). Es kommt zur Depolymerisation von FtsZ (C) und zur Bildung neuer FtsZ-Filamente in der Zellmitte, wo nun der inhibitorische Einfluss von MipZ am geringsten ist (D). Urheber: Nach [7], Wiedergabe mit Einverständnis des Elsevier Verlags Genomanalysen zeigen, dass MipZ in einer Vielzahl von Bakterien konserviert vorliegt und daher den Prototyp einer neuen Familie von Zellteilungsregulatoren darstellt [8]. Das Lokalisationsmuster von MipZ ist das erste Beispiel für einen stabilen Proteingradienten in Bakterien. Da in bakteriellen Zellen reine Diffusionsgradienten auf Grund ihrer geringen Ausmaße nicht über längere Zeit aufrecht erhalten werden können, müssen in diesem Fall Prinzipien zur Anwendung kommen, die bislang unbekannt sind. Aktuelle Ergebnisse deuten darauf hin, dass trotz der konstanten Dimensionen des Gradienten ein schneller Austausch von MipZ-Molekülen stattfindet, der über die Bindung und Hydrolyse von ATP reguliert wird. Wie diese Dynamik zur Etablierung des beobachteten Gradienten beiträgt, wird gegenwärtig im Detail untersucht. © 2008 Max-Planck-Gesellschaft www.mpg.de Tätigkeitsbericht 2008 Thanbichler, Martin | Regulation der Zellteilung Literaturhinweise [1] M. Thanbichler, L. Shapiro: How bacterial cells move proteins and DNA. Nature Reviews Microbiology 6, 28-40 (2008). [2] J. Lutkenhaus: Assembly dynamics of the bacterial MinCDE system and spatial regulation of the Z ring. Annual Review of Biochemistry 76, 539-562 (2007). [3] J. S. Poindexter: Biological properties and classification of the Caulobacter group. Bacteriological Reviews 28, 231-295 (1964). [4] M. Thanbichler, L. Shapiro: Chromosome organization and segregation in bacteria. Journal of Structural Biology 156, 292-303 (2006). [5] P. H. Viollier, M. Thanbichler, P. T. McGrath, L. West, M. Meewan, H. H. McAdams, L. Shapiro: Rapid and sequential movement of individual chromosomal loci to specific subcellular locations during bacterial DNA replication. Proceedings of the National Academy of Sciences USA 101, 9257-9262 (2004). [6] N. W. Goehring, J. Beckwith: Diverse paths to midcell: Assembly of the bacterial cell division machinery. Current Biology 15, R514-R526 (2005). [7] M. Thanbichler, L. Shapiro: MipZ, a spatial regulator coordinating chromosome segregation with cell division in Caulobacter. Cell 126, 147-162 (2006). [8] W. Margolin: Bacterial division: Another way to box in the ring. Current Biology 16, R881-R884 (2006). Drittmittelfinanzierung DFG, Humboldt-Stiftung, DAAD, Higher Education Commission (Pakistan) www.mpg.de © 2008 Max-Planck-Gesellschaft