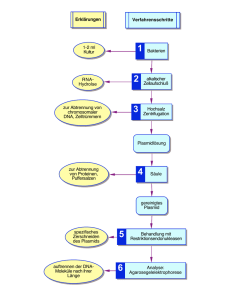
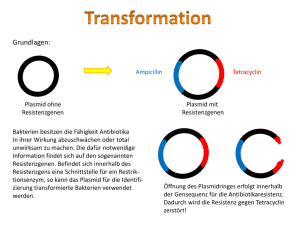
Isolation von Insertionssequenzen mit Hilfe von sacB
Werbung

Isolation von Insertionssequenzen mit Hilfe von sacB Insertionssequenzen (IS) sind transponierbare Elemente, die keine Gene für selektierbare Eigenschaften tragen sondern nur für ihre Transposition kodieren. Eine positive Selektion für solche Elemente ist daher nicht möglich. Da bei Transposition in ein Gen dieses Gen mutiert, kann eine indirekte Identifizierung von IS über die Analyse spontaner Mutanten erfolgen. Zur direkten Selektion spontaner (Insertions-)Mutanten kann das (zumindest in Gram-negativen Bakterien) konditional letale Gen sacB von Bacillus subtilis genutzt werden. Das sacB-Gen kodiert für das Enzym Levansucrase, das durch Saccharose induziert wird und in B. subtilis als Exoenzym vorliegt. Es katalysiert die Reaktion Saccharose + Akzeptor (z.B. Fruktose) → Glukose + Fruktose – Akzeptor. Es vermittelt auf diese Weise die Levansynthese, die mit der Saccharosespaltung gekoppelt ist. In Gram-negativen Bakterien führt die Expression dieses Gens in Medien mit Saccharose zur Lyse der Zellen. Zellen, die das Gen (z.B. auf einem Plasmid) tragen, können folglich auf Medien mit Saccharose keine Kolonien bilden, es sei denn, das sacB-Gen ist durch eine spontane Mutation inaktiviert worden oder das Plasmid und damit das sacB-Gen ist verloren gegangen. Durch plattieren auf Medien mit Saccharose und dem Antibiotikum, mit dem für das Plasmid selektiert wird, wird für das Plasmid mit einem inaktiven sacB-Gen selektiert. Durch die Analyse der Plasmide der Saccharose-resistenten Klone kann zumindest in einem Teil der Mutanten gezeigt werden, dass die Ursache für die Saccharose-Resistenz die Insertion fremder DNA in das sacB-Gen ist. Das sacB-Gen ist außerdem bei einer gezielten Mutagenese hilfreich. Bei dieser Mutagenese wird zunächst das Gen, das man mutagenisieren möchte, in ein Plasmid kloniert, das in dem zu mutagenisierenden Stamm nicht replizieren kann. Danach wird das Gen – meist in vitro – mutiert. Das Plasmid mit dem mutierten Gen wird dann in den zu mutierenden Stamm übertragen. Durch homologe Rekombination kann das Wildtyp-Allel gegen das mutierte Gen ausgetauscht werden. Wenn auf dem Plasmid außerdem sacB liegt, kann auf einem Medium mit Saccharose (aber ohne Antibiotikum zur Selektion für das Plasmid) für die komplette Rekombination (gene replacement) selektiert werden. Praktischer Teil Material: E. coli S17-1 pLU4 (bla, sacB) Medien: LB (Luria Broth, Luria Agar), Komplettmedium für E. coli: 10 g Pepton (tryptisch verdaut) 5 g Hefeextrakt 5 g NaCl (15 g Agar) ad 1000 ml aqua dest. 30 %ige Saccharose-Lösung Antibiotikumkonzentration: - Ampicillin (Amp) 100 µg/ml) Durchführung: E. coli S17-1 PLU4 wird in 3 ml LB in Gegenwart von Ampicillin über Nacht kultiviert die gewachsene Kultur wird morgens 1:10 in LB Amp umgesetzt und nach ca. 4h Inkubation bei 30°C in 0,9 % NaCl verdünnt (10-1, 10-2, 10-3) und je 0,1 ml auf LB Agarmedium mit 15 % Saccharose und Ampicillin (100 µg/ml) ausplattiert zur Bestimmung der Gesamtzellzahl wird eine Verdünnungsreihe angelegt und die 10-4, 10-5, 10-6 Verdünnung ausplattiert (je 0,1 ml) auf LB Amp auf dem Saccharose-Medium gewachsene Kolonien werden noch einmal auf das gleiche Medium gepickt um falsch positive Klone zu identifizieren je 10 Klone werden dann in 4 ml LB Amp über Nacht kultiviert von den gewachsenen Kulturen wird eine Plasmidpräparation durchgeführt zum Vergleich wird vom ursprünglichen Stamm (S17-1 pLU4) ebenso das Plasmid isoliert Auswertung: Bestimmen Sie die Frequenz der sacB Inaktivierung. Vergleichen Sie die isolierten Plasmide. Anhand der beigefügten Restriktionskarte soll die Lokalisation des IS Elements und die Größe bestimmt werden. Dazu sollen entsprechende Restriktionsspaltungen durchgeführt und ausgewertet werden. Anhand von vorhandenen IS Sequenzen soll eine mögliche Zuordnung der erhaltenen IS Elemente in pLU4 ermitt elt werden. KpnI EcoRV (100) EcoRV (6800) KpnI (6400) pnI