Stand der molekularen Diagnostik kardiovaskulärer Erkrankungen
Werbung
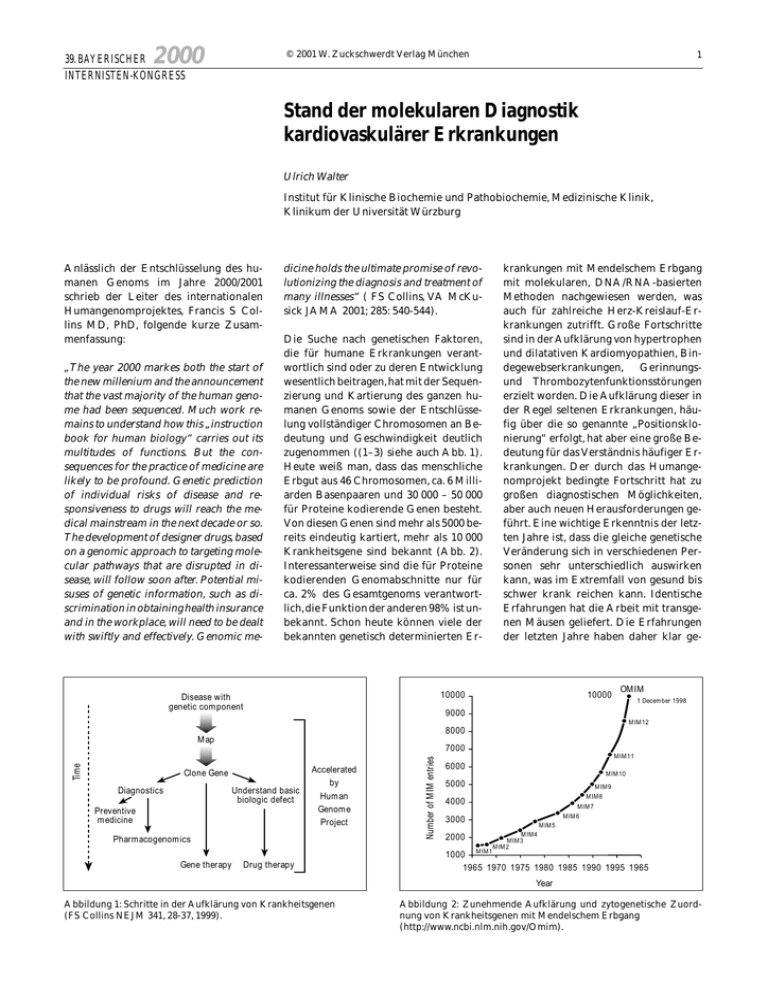
2000 © 2001 W. Zuckschwerdt Verlag München 39. BAYERISCHER INTERNISTEN-KONGRESS 1 Stand der molekularen Diagnostik kardiovaskulärer Erkrankungen Ulrich Walter Institut für Klinische Biochemie und Pathobiochemie, Medizinische Klinik, Klinikum der Universität Würzburg Anlässlich der Entschlüsselung des humanen Genoms im Jahre 2000/2001 schrieb der Leiter des internationalen Humangenomprojektes, Francis S Collins MD, PhD, folgende kurze Zusammenfassung: „The year 2000 markes both the start of the new millenium and the announcement that the vast majority of the human genome had been sequenced. Much work remains to understand how this „instruction book for human biology“ carries out its multitudes of functions. But the consequences for the practice of medicine are likely to be profound. Genetic prediction of individual risks of disease and responsiveness to drugs will reach the medical mainstream in the next decade or so. The development of designer drugs, based on a genomic approach to targeting molecular pathways that are disrupted in disease, will follow soon after. Potential misuses of genetic information, such as discrimination in obtaining health insurance and in the workplace, will need to be dealt with swiftly and effectively. Genomic me- dicine holds the ultimate promise of revolutionizing the diagnosis and treatment of many illnesses“ ( FS Collins, VA McKusick JAMA 2001; 285: 540-544). Die Suche nach genetischen Faktoren, die für humane Erkrankungen verantwortlich sind oder zu deren Entwicklung wesentlich beitragen,hat mit der Sequenzierung und Kartierung des ganzen humanen Genoms sowie der Entschlüsselung vollständiger Chromosomen an Bedeutung und Geschwindigkeit deutlich zugenommen ((1–3) siehe auch Abb. 1). Heute weiß man, dass das menschliche Erbgut aus 46 Chromosomen, ca. 6 Milliarden Basenpaaren und 30 000 – 50 000 für Proteine kodierende Genen besteht. Von diesen Genen sind mehr als 5000 bereits eindeutig kartiert, mehr als 10 000 Krankheitsgene sind bekannt (Abb. 2). Interessanterweise sind die für Proteine kodierenden Genomabschnitte nur für ca. 2% des Gesamtgenoms verantwortlich,die Funktion der anderen 98% ist unbekannt. Schon heute können viele der bekannten genetisch determinierten Er- krankungen mit Mendelschem Erbgang mit molekularen, DNA/RNA-basierten Methoden nachgewiesen werden, was auch für zahlreiche Herz-Kreislauf-Erkrankungen zutrifft. Große Fortschritte sind in der Aufklärung von hypertrophen und dilatativen Kardiomyopathien, Bindegewebserkrankungen, Gerinnungsund Thrombozytenfunktionsstörungen erzielt worden. Die Aufklärung dieser in der Regel seltenen Erkrankungen, häufig über die so genannte „Positionsklonierung“ erfolgt, hat aber eine große Bedeutung für das Verständnis häufiger Erkrankungen. Der durch das Humangenomprojekt bedingte Fortschritt hat zu großen diagnostischen Möglichkeiten, aber auch neuen Herausforderungen geführt. Eine wichtige Erkenntnis der letzten Jahre ist, dass die gleiche genetische Veränderung sich in verschiedenen Personen sehr unterschiedlich auswirken kann, was im Extremfall von gesund bis schwer krank reichen kann. Identische Erfahrungen hat die Arbeit mit transgenen Mäusen geliefert. Die Erfahrungen der letzten Jahre haben daher klar ge- 10000 Disease with genetic component 10000 OMIM 1 December 1998 9000 MIM12 8000 Map Clone Gene Diagnostics Understand basic biologic defect Preventive medicine Accelerated by Human Genome Project Pharmacogenomics Gene therapy Drug therapy Number of MIM entries Time 7000 MIM11 6000 MIM10 5000 MIM9 MIM8 4000 MIM7 MIM6 3000 2000 MIM5 MIM4 MIM3 MIM2 1000 MIM1 1965 1970 1975 1980 1985 1990 1995 1965 Year Abbildung 1: Schritte in der Aufklärung von Krankheitsgenen (FS Collins NEJM 341, 28-37, 1999). Abbildung 2: Zunehmende Aufklärung und zytogenetische Zuordnung von Krankheitsgenen mit Mendelschem Erbgang (http://www.ncbi.nlm.nih.gov/Omim). 2 Tabelle 1: Das genetische Dogma „ein Gen – ein Protein – ein Phänotyp“ gilt nur eingeschränkt. Genetische Heterogenität: Ein klinischer Phänotyp kann durch unterschiedliche Gene bedingt sein (z. B. Kardiomyopathien, Retinitis Pigmentosa etc.) Ein Gen – unterschiedliche Proteine: differentielles Spleißen des Primärtransskripts, RNA-Editing, Posttranslationale Modifikation etc.) Heterogenität der Mutationen: unterschiedlicher Mutationsort (z. B. Intron, Exon) und die Mutationsart (Punktmutation, Non-sense-Mutationen (Stop-Codons), missense-Mutationen (andere AS), Deletionen/Insertionen, Triunkleotid-Repeats Modifier-Gene: Identische Mutationen (Genotyp) können durch die An/Abwesenheit weiterer Modifier-Loci(Gene) entscheidend beeinflußt werden (Mukoviszidose/CFTR; LDL-Rezeptor etc.) Umweltfaktoren: Der Phänotyp von Mutationen wird durch Umweltbedingungen beeinflußt (Thrombose, maligne Hyperthermie, Porphyrie) Tabelle 2: Fließender Übergang von monogenetischen Erkrankungen und Erkrankungen mit komplexen genetischem Hintergrund. – Monogenetische Erkrankungen hoher (100%iger) Penetranz – Monogenetische Erkrankungen wechselnder bis niedriger Penetranz (Modifier-Gene, Umweltfaktoren etc.) – Erkrankungen mit komplexem genetischem Beitrag (Diabetes, Hypertonie, Arteriosklerose, Asthma, Schizophrenie, etc.) Tabelle 3: Auswirkungen der Punktmutation im Faktor V (FV - Leiden). Prävalenz (Deutschland) 7.3% Inzidenz bei Thrombosepatienten Diseased cells or tissue Thromboserisiko Normal cells or tissue cDNA cDNA Upregulated in disease – Heterozygot ca. – Homozytot ca. 80-fach Unchanged in disease Hybridized array Abbildung 3: Molekulare Diagnostik durch Expressionsprofile : c D N A Microarrays (EM Rubin, A Tall. Nature 407, 265, 2000). zeigt, dass die alte Hypothese „Ein Gen – ein Protein – ein Phänotyp“ nur noch eingeschränkt gilt (Tab. 1) und ein fließender Übergang von monogenetischen Krankheiten zu Erkrankungen mit komplexem genetischen Hintergrund besteht (Tab. 2). Die Erfahrungen mit der gut untersuchten Punktmutation im Gerinnungsfaktor V (Faktor V Leiden) und seine Bedeutung für das Thromboserisiko können das bereits gut belegen (Tab. 3). Dies bedeutet, dass eine molekulare Diagnostik ohne ärztliche Untersuchungen und Beratung für monogene Erbkrankheiten und insbesondere für multifaktorielle Krankheiten undenkbar ist. Die größte Herausforderung für die Medizin und die Forschung sind die häufigen,multifaktoriell bedingten (polygenen, komplexen) Erkrankungen, z. B.Arteriosklerose, Asthma, Diabetes, Hypertonie, Thrombose etc. (1–3). Es ist unwahr- scheinlich, dass es hier Gene und Mutationen geben wird, die die Frage nach Erkrankungen einfach mit ja/nein beantworten können.Allerdings werden große Erwartungen an die Chip- und Microarray-Technologie (Abb. 3) und andere Methoden geknüpft, mit denen eine große Zahl von möglichen krankheitsrelevanten Mutationen (besser spricht man von genetischen Polymorphismen) erfaßt werden können. Referenzen 1. 2. 3. Collins FS, McKusick VA. Implications of the Human Genome Project for Medical Science. JAMA 2001; 285: 540-544 Risch NJ. Searching for genetic determinants in the new millenium. Nature 2000; 405: 847-856 Sperling K. Das Humangenomprojekt : Medizin im Licht der Evolution. DMW 2000; 125: A15- A21 7-fach Übersicht: H. Wankmüller et al., Med.Klin. 91 (1996), 308 (1996) Downregulated in disease Printed cDNA array 19–64% Anschrift des Verfassers: Prof. Dr. med. U. Ulrich Walter Institut für Klinische Biochemie und Pathobiochemie, Medizinische Klinik, Klinikum der Universität Josef-Schneider Str. 2 97080 Würzburg











