Proteomic Pathology
Werbung

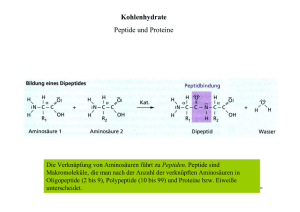
Proteomic Pathology Die pathologische Forschung analysiert die zellulären Vorgänge, die zur Entstehung und Progression von Krankheiten führen. In diesem Zusammenhang werden Zellen und Gewebe zunehmend als ein kompliziertes Netzwerk verstanden, in dem eine Vielzahl biologischer Prozesse gleichzeitig ablaufen und gemeinsam das biologische Verhalten bestimmen. Proteine sind an allen zellulären Prozessen beteiligt und somit sind zum Verständnis der zellulären Funktionen das Verständnis der zeitlich und örtlich differentiellen Protein-Expression, der Protein-Lokalisation, der Protein-Interaktion, der posttranslationalen Protein-Modifikation und der Protein-Aktivität essentiell. Quantitative Massenspektrometrie Die globale Analyse der zellulären Proteinzusammensetzung würde die Sichtweite auf das zelluläre Netzwerk sehr erweitern, ist technisch jedoch anspruchsvoll. Ein großer Durchbruch in diese Richtung war die Einführung von Microarrays zur globalen Quantifizierung der mRNA-Expression. Obwohl dies zu einem starken Anstieg in unserem Wissen über viele zellulären Funktionen geführt hat, wäre die direkte Untersuchung der Proteinexpression häufig vorteilhaft, weil die mRNA- oft nicht gut mit der Proteinexpression korrelliert und posttranslationale Proteinmodifikationen wie Phosphorylierung mit Microarrays nicht erfasst werden können. Die Massenspektrometrie stellt eine Technologie dar, die es erlaubt, Proteine direkt, global und unvoreingenommen zu untersuchen. Das zu untersuchende Proteingemisch wird dabei zunächst proteolytisch verdaut und die Peptide mittels Elektrospray-Ionisierung (ESI) ionisiert. In einem Orbitrap Massenspektrometer oszillieren die Peptide in einer bestimmten, von Masse und Ladung des Peptides abhängigen Frequenz um eine Ionenfalle. Dadurch können die Masse im Verhältnis zur Ladung (m/z) und die Intensität jedes Peptides bestimmt werden. In einem zweiten Messschritt wird die Sequenz des Peptides bestimmt, was als MS/MSAnalyse bezeichnet wird. Dazu werden die Peptide zunächst in eine Kollisionskammer überführt, wo sie fragmentiert werden. Durch die erneute Messung der Masse der Bruchstücke kann die Aminosäuresequenz des Peptides bestimmt werden. Dadurch ergibt sich der besondere Vorteil, dass a priori nicht bekannt sein muss, welche Proteine gemessen werden sollen, sondern das Massenspektrometer potentiell jedes beliebige Protein identifizieren kann. Häufig ist jedoch wichtiger, in welchem Ausmaß sich die Menge eines Proteins in zwei unterschiedlichen Konditionen unterscheidet, als die bloße Anwesenheit eines Proteins nachzuweisen. Eine Möglichkeit, quantitative Messungen durchzuführen, ist das stable isotop labeling of amino acids in cell culture, kurz SILAC. Dabei werden die zu untersuchenden Zellen mit unterschiedlich schweren Aminosäuren kultiviert. Der Massenunterschied wird dabei durch den Einbau von stabilen, schweren Isotopen von Kohlen-, Stick- oder Wasserstoff in die Aminosäuren Arginin und Lysin erreicht. Durch die Kultivierung einer Kontrollzelllinie mit leichten Aminosäuren kann ein relati- ver Vergleich von zwei verschiedenen Versuchsbedingungen erzielt werden, da sich die Peptide durch den resultierenden Massenunterschied im Massenspektrometer unterscheiden lassen. Das SILAC-Prinzip: Durch die Kultivierung von Zellen mit unterschiedlich schweren Aminosäuren, lassen sich die resultierenden markierten Proteine im Massenspektrometer unterscheiden und relativ zueinander quantifizieren. Mit Hilfe des SILAC-Prinzips können zwei Versuchsbedingungen relativ zueinander quantifiziert werden. So kann die Expression zweier Zelllinien global miteinander verglichen werden. Durch die Kombination des SILAC-Prinzips mit spezifischer Proteinpräzipitation können auch Proteininteraktionen global analysiert werden. Dabei wird aus einer Zell-Linie, die mit schweren Aminosäuren kultiviert wurde das Zielprotein präzipitiert, während aus einer Kontrollzelllinie durch eine Pseudo-Präzipitation nur der unspezifische Hintergrund präzipitiert wird. In der massenspektrometrischen Analyse ergeben sich deutliche Unterschiede in der Intensität der Proteine, die nur bei der spezifischen Präzipitation angereichert wurden. Auch die Analyse von posttranslationalen Proteinmodifikationen wie Phosphorylierung ist mittels Massenspektrometrie möglich, da jede Modifikation zu einer definierten Massenänderung führt. Dieser Massenunterschied kann im MS/MS-Modus einer spezifischen Aminosäure zugeordnet werden. Durch die Kombination mit dem SILAC-Prinzip ist es über die reine Identifikation von Phosphorylierungsstellen hinaus auch möglich, Phosphorylierungsunterschiede zwischen verschiedenen Versuchsbedingungen zu bestimmen. Die massenspektrometrischen Analysen erfolgen in enger Kooperation und vielen gemeinsamen Projekten mit Dr. Thomas Oellerich und Prof. Dr. Hubert Serve von der Medizinischen Klinik II, Universitätsklinikum Frankfurt, Prof. Dr. Henning Urlaub von der Bioanalytischen Massenspektrometrie am MPI für biophysikalische Chemie in Göttingen sowie mit Dr. Christof Lenz von der Proteomanalyse-Facility, Universitätsmedizin Göttingen zusammen. Quantitative Tissue-Proteomic Menschliches Gewebe, wie es zur Routine-Diagnostik in die Pathologie kommt, lässt sich nicht mehr mittels des SILAC-Prinzips markieren. Dennoch sind quantitative massenspektrometrische Analysen möglich. Dafür werden zunächst die Proteine aus den Formalin-fixierten, Paraffin-eingebetten Gewebsblöcken isoliert und mit einem Protein-Vergleichsstandard vermischt. Der Proteinvergleichsstandard wird aus Zell-Linien gewonnen, die mittels des SILAC-Prinzips markiert wurden. Es ergibt sich für jedes Protein jeder Gewebeprobe eine Ratio von „Gewebe zu Standard“. Diese Ratios sind untereinander vergleichbar, da jeweils zum gleichen Standard quantifiziert wurde. Mikrodissektion Ein Problem bei der Erforschung der Tumorbiologie stellt die Heterogenität der zu analysierenden Gewebeproben dar. Deshalb arbeiten wir an verschiedenen Möglichkeiten der Mikrodissektion. Zu diesem Zwecke steht uns ein Laser-Capture-Mikrodissektor von Molecular Machines zur Verfügung. Des Weiteren arbeiten wir aber auch an der Etablierung von expressionsbasierten Mikrodissektionsverfahren, bei der Gewebe anhand der Expressionsmusters von immunhistochemischen Markern getrennt wird. Das mikrodissoziierte Gewebe wird anschließend quantitativ massenspektrometrisch analysiert. Expressionsbasierte Mikrodissektion eines NSCLC anhand der Expression von CK5/6. Rechts ist das isolierte Gewebe zu sehen, links das verbleibende, nicht-isolierte Gewebe. Funktionelle Bildgebung – Konfokalmikroskopie Die aktuelle histopathologische Diagnostik beruht neben der morphologischen Begutachtung des zu untersuchenden Gewebes häufig auch auf der immunhistochemischen Expressionsanalyse spezifischer Proteine. Des Weiteren werden in ausgesuchten Fällen gezielt Onkogene auf Mutationen oder Amplifikationen überprüft. In einem gemeinsamen Projekt mit Dr. Gertrude Bunt (UMG, Molecular and Optical Live Cell Imaging Facility) und Prof. Dr. Fred Wouters (UMG, Labor für molekulare und zelluläre Systeme, Institut für Neuro- und Sinnesphysiologie) erschließen wir mit Hilfe hochauflösender konfokaler Mikroskopie und FRET-Verfahren die biologische Funktion relevanter Proteine (Biomarker). Diese liefern neben der alleinigen Analyse der Anwesenheit eines Onkogens oder einer Mutation wertvolle molekular-diagnostische Information. NSCLC gefärbt mit Alexa488 und Cy3. Beispiele für zu etablierende funktionelle Biomarker sind FRET-basierte Phosphorylierungsund Interaktions-Analysen von Onkogenen in ihrem zellulären und histologischen Kontext. Kinaseaktivität – PamGene Die Aktivität eines Proteins wird von einer Vielzahl von Faktoren wie Expression, verschiedenen posttranslationalen Modifikationen oder Interaktion mit anderen Proteinen reguliert. Häufig stellt die abberante Aktivität von onkogen-wirksamen Kinasen eine Schlüsselrolle in der malignen Entartung einer Zelle dar. Ebenso ist es die Aktivität einer Kinase, die entscheidend ist für das Ansprechen auf einen spezifischen Kinase-Inhibitor als gezielte KrebsTherapie. Bei der Kinaseaktivitätsmessung mittels PamChips der Firma PamGene werden Peptide durch die Kinaseaktivität des zu untersuchenden Lysates phosphoryliert und mittels Fluoreszenzfarbstoff-markierter Antikörper sichtbar gemacht. Der Vorteil dabei ist, dass direkt die Aktivität der Kinasen im Lysat bestimmt wird. Ein weiterer Vorteil ist, dass Inhibitoren mit in das Lysat gegeben werden können, so dass direkt das Ansprechen auf einen spezifischen Kinaseinhibitor beobachtet werden kann. Bei der statistischen Auswertung der erhobenen Kinaseaktivitäten arbeiten wir mit Dr. Stefan Lorenzen aus der Abteilung für medizinische Statistik, Universitätsmedizin Göttingen zusammen. Ein PamChip enthält 144 gespottete Peptide, die von der Kinaseaktivität in einem Zell- oder Gewebslysat phosphoryliert werden können. Mittels Fluoreszenzfarbstoff-markierter Antikörper kann die Aktivität der Kinasen im Lysat bestimmt werden. Aktuelle Projekte Aktuell werden in unserer Arbeitsgruppe mit den genannten Methoden verschiedener histologische und molekulare Subtypen des Lungenkrebs analysiert. Dabei arbeiten wir mit Dr. Alexander Emmert, Dr. Mark Hinterthaner, PD Dr. Bernd Danner und Prof. Dr. med. Dipl. Phys. Friedrich A. Schöndube von der Klinik für Thorax-, Herz- und Gefäßchirurgie zusammen. In Zukunft sollen die proteomischen Analysen auf weitere Malignom-Entitäten ausgedehnt werden. Kooperationen Proteomische Massenspektrometrie Dr. T. Oellerich, Medizinischen Klinik II, Universitätsklinikum Frankfurt Prof. Dr. H. Serve, Medizinischen Klinik II, Universitätsklinikum Frankfurt Dr. C. Lenz, Proteomanalyse-Facility, Universitätsmedizin Göttingen Prof. Dr. Henning Urlaub, Bioanalytische Massenspektrometrie, MPI bpc Göttingen Funktionelle Bildgebung Dr. G. Bunt, Molecular and Optical Live Cell Imaging, Universitätsmedizin Göttingen Prof. Dr. F. Wouters, Abteilung Neuro- und Sinnesphysiologie, Universitätsmedizin Göttingen Thorax-Herz-Gefäßchirurgie Dr. A. Emmert, Thorax-, Herz-, Gefäß-Chirurgie, Universitätsmedizin Göttingen Dr. M. Hinterthaner, Thorax-, Herz-, Gefäß-Chirurgie, Universitätsmedizin Göttingen PD Dr. B. Danner, Thorax-, Herz-, Gefäß-Chirurgie, Universitätsmedizin Göttingen Prof. Dr. med. Dipl. Phys. Friedrich A. Schöndube Medizinische Statistik Dr. S. Lorenzen, Medizinische Statistik, Universitätsmedizin Göttingen Ansprechpartner / Kontakt Hanibal Bohnenberger Assistenzarzt Tel: 0551 – 39 6854 Pieper: 919 – 2561 [email protected]