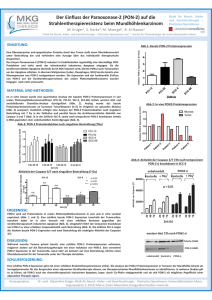
ZIP - Universität Freiburg
Werbung

Aus der Abteilung Allgemeine Pathologie und Pathologische Anatomie des Instituts für Pathologie der Albert-Ludwigs-Universität Freiburg i. Br. Signal Transducer and Activator of Transcription-1 (STAT-1) und STAT-1 assoziierte Proteine (MCM3,MCM5) in epithelialen Ovarialkarzinomen: Expressionsanalysen und Prognostische Relevanz INAUGURAL-DISSERTATION zur Erlangung des Medizinischen Doktorgrades der Medizinischen Fakultät der Albert-Ludwigs-Universität Freiburg i. Br. Vorgelegt 2006 von Yi Shen geboren in Shanghai, China Dekan: Prof. Dr. med. Christoph Peters 1.Gutachter: Prof. Dr. med. Martin Werner 2.Gutachter: PD. Dr. med. Annette Hasenburg Jahr der Promotion: 2006 Inhaltsverzeichnis Inhaltsverzeichnis 1. Zusammenfassung.......................................................................................................................................... 1 2. Einleitung ........................................................................................................................................................ 2 2.1 Ovarialkarzinom......................................................................................................................................... 2 2.1.1. Epidemiologie Onkogenese................................................................................................................ 2 2.1.2 Diagnostik ........................................................................................................................................... 3 2.1.3 Therapie............................................................................................................................................... 5 2.1.4 Prognosefaktoren................................................................................................................................. 6 2.2 STAT1 Signalweg....................................................................................................................................... 8 2.2.1 Übersicht STAT Proteine..................................................................................................................... 8 2.2.2. STAT1: Proteinstruktur....................................................................................................................... 9 2.2.3. STAT1: Aktivierung ......................................................................................................................... 10 2.2.4. STAT1: Funktion ...............................................................................................................................11 2.2.5. Interaktion von STAT1 mit „Minichromosome Maintenance“ Proteinen ........................................ 12 2.3. Fragestellung und Zielsetzung................................................................................................................. 13 3. Materialien und Methoden .......................................................................................................................... 14 3.1 Materialien ............................................................................................................................................... 14 3.1.1 Patientinnen, Gewebeproben und klinische Verlaufdaten ................................................................. 14 3.1.2 Chemikalien, Reagenzien und Geräte ............................................................................................... 14 3.2 Methoden.................................................................................................................................................. 17 3.2.1 Auswahl der Gewebeproben.............................................................................................................. 17 3.2.2 RNA Extraktion................................................................................................................................. 17 3.2.2.1. Gewebevorbereitung und Mikrodissektion ............................................................................... 17 3.2.2.2 RNA Extraktion und Aufreinigung............................................................................................. 18 3.2.2.3 Messungen der RNA-Konzentration .......................................................................................... 19 3.2.3 Quantitative Reverse Transkriptase Polymerase Kettenreaktion (Q-RT-PCR) ................................. 19 3.2.3.1 cDNA Synthese (RT-Schritt) ...................................................................................................... 19 3.2.3.2 Q-(RT)-PCR ............................................................................................................................... 19 Inhaltsverzeichnis 3.2.3.3 Sequenzierung ............................................................................................................................ 22 3.2.4 Immunhistochemie ........................................................................................................................ 23 3.2.4.1 Gewebearray............................................................................................................................... 23 3.2.4.2 Arbeitsschritte Proteinfärbung (Abb. 6) ..................................................................................... 24 3.2.4.3 Auswertung der Färbung ............................................................................................................ 26 3.2.5. Statistische Analyse der mRNA- und Proteinexpressionsdaten..................................................... 26 4. Ergebnisse ..................................................................................................................................................... 27 4.1 STAT1 mRNA Expressionanalyse............................................................................................................ 27 4.1.1 Etablierung der STAT1-spezifischen Q-RT-PCR .............................................................................. 27 4.1.1.1 Optimierung der Primer- und Sondenkonzentration................................................................... 27 4.1.1.2 Bestimmung der Sensitivität und Q-RT-PCR Effizienz.............................................................. 29 4.1.1.3 Bestimmung der Spezifität von Q-RT-PCR - Sequenzierung..................................................... 30 4.1.2 STAT1 mRNA Expression in Ovarialkarzinomen............................................................................. 31 4.2 STAT1, P-STAT1, MCM3 und MCM5 Proteinexpression in Ovarialkarzinomen ................................... 32 4.2.1 Etablierung der Färbungen ................................................................................................................ 32 4.2.2 STAT1 und P-STAT1 Proteinexpression in Ovarialkarzinomen........................................................ 35 4.2.3 MCM3 und MCM5 Proteinexpression in Ovarialkarzinomen .......................................................... 39 4.3 Korrelation der STAT1, PSTAT1, MCM3 und MCM5 Proteinexpression zur Histologie und den klinischen Verlaufsdaten................................................................................................................................. 42 4.3.1 Korrelation der STAT1-, PSTAT1-, MCM3- und MCM5- Expression zu klinischen Verlaufsdaten. 42 4.3.2 Korrelation der STAT1, PSTAT1, MCM3 und MCM5 Expression miteinander............................... 44 5 Diskussion ...................................................................................................................................................... 45 5.1 Die Bedeutung des STAT1 im Signalweges in Ovarialkarzinomen ......................................................... 46 5.2 Die Bedeutung der MCM3 und MCM5 Proteinexpression in Ovarialkarzinomen .................................. 49 5.3 Fazit.......................................................................................................................................................... 51 6 Literaturangaben .......................................................................................................................................... 52 Abkürzungen AA Aminosäure ABC Avidin-Biotin-Complex(immuhistoschemische Färbungsreagenz) ABL Onkogen des Abelson- Leukemia-Virus AEC 3-Amino-9-ethylcarbazol AK Antikörper Aqua dest. destilliertes Wasser Bp Basenpaare BSA Rinderserum Albumin cDNA complementary DNA CT-Wert cycle threshold (Schwellenwert) dNTPs 2’-Desoxyribonukleosidtriphosphate(dATP, dCTP, dGTP, dTTP) EGFR Epidermal Growth Factor Rezeptor ETOH Ethanol F-Primer Vorwärts- Primer H&E Hämatoxylin und Eosin IHC Immunhistochemie JAK Januskinase MCM Minichromosome maintenance M-MLV Moloney Murine Leukemia Virus NLS Nuclear localisation Signal dsNLS dimer-specific NLS OD optische Dichte PBS phosphate buffered saline PCR Polymerase-Kettenreaktion PDGF Platelet-derived Growth Fctor Q-RT-PCR Quantitative-reverse Transkription-PCR rpm rounds per minute R-Primer Rückwärts-Primer SH2 Src-Homology2 siRNA Small inhibitory RNA SRC Onkogen des Rous-Sarkom-Virus STAT signal transducer and activator of transcription TAD Transaktivierungsdomäne VEGF Vascular Epithelial Growth Factor -1- Zusammenfassung 1. Zusammenfassung Das Ovarialkarzinom ist die siebt-häufigste Krebserkrankung bei Frauen und ist, nach dem Mammakarzinom, die häufigste Todesursache gynäkologischer Krebserkrankungen. Die 5-Jahres Überlebensrate ist trotz Fortschritte in der Diagnostik und Behandlung mit ca. 20-25% gering. Die Identifizierung molekularer Prognosemarker könnte verbesserte und individualisierte Therapieprotokolle ermöglichen. Ziel dieser Studie war es, die mRNA und transcription-1“ Proteinexpression von (STAT1) „signal transducer und and STAT1-assoziierter activator of Moleküle („minichromosome-maintenance“: MCM-3, -5) in epithelialen Ovarialkarzinomen des Stadiums IIIc zu untersuchen und auf ihren prognostischen Wert hin zu überprüfen. Dazu wurde auf RNA- und Protein-Ebene die Expression von STAT1 in normalen Ovarien (n=29) und Ovarialkarzinomen (n=114) mittels quantitativer reverser transkriptase Polymerase Kettenreaktion (Q-RT-PCR) und Immunhistochemie (IHC) bestimmt. Zusätzlich wurde die Aktivität von STAT1 mittels eines phospho-spezifischen Antikörpers untersucht und mit der der STAT1-assoziierten MCM3 und MCM5 Proteinexpression korreliert. Der prognostische Wert aller untersuchten Marker wurde, neben den bekannten klinischen Parametern, für das Rezidiv-freie und Gesamtüberleben bestimmt. Die Daten der Studie zeigten einen signifikanten Unterschied der STAT1, PSTAT1, MCM3 und MCM5 Expression zwischen normalen Ovarien und invasiven Karzinomen. In Patientinnen mit adjuvanter Taxol/Carboplatin Therapie war eine hohe STAT1 Proteinexpression mit einem besseren Rezidiv-freien Überleben (p = 0,0221) sowie eine hohe STAT1 (p = 0,0019) und PSTAT1 (p = 0,0325) Proteinexpression mit einem längeren Gesamtüberleben korreliert. Die STAT1 Expression war mit erhöhter MCM3 und MCM5 Proteinexpression verbunden. Der STAT1 Signalweg bei Patientinnen mit fortgeschrittenen epithelialen Ovarialkarzinomen ist aktiviert, mit der Expression von MCM3 und MCM5 korreliert und auch stellt einen neuen prognostischen Marker dar. -2- Einleitung 2. Einleitung 2.1 Ovarialkarzinom 2.1.1. Epidemiologie Onkogenese Das Ovarialkarzinom ist mit einer Inzidenz von ca.15/100,000 pro Jahr in westlichen Ländern die zweit-häufigste gynäkologische Krebserkrankung bei Frauen zwischen dem 40 und 65 Lebensjahr (Wingo et al, 1995). Da das Ovarialkarzinom in frühen Stadien selten symptomatisch ist, wird in ca. 80% aller Frauen ein fortgeschrittenes Stadium diagnostiziert, deren 5-Jahres Überlebensrate daher nur bei ca. 20-25% liegt (Friedlander et al, 1998, Ozols, 2005). Die Ätiologie des Ovarialkarzinoms ist bisher noch unklar, aber sowohl genetische als auch individuelle Faktoren scheinen dabei eine Rolle zu spielen. So ist bekannt, dass das Risiko ein Ovarialkarzinom zu entwickeln mit der Häufigkeit der Ovulation korreliert (Auersperg et al, 2002). Nach der Ovulation proliferiert das oberflächliche Ovarialepithelial stark und das Mutationsrisiko steigt an. Eine Nulliparität, frühe Menarche, späte Menopause gelten daher als Risikofaktoren, während eine frühe Menopause, Schwangerschaft, Stillen und hormonelle Kontrazeption mit einem deutlich geringerem Risiko für die Entstehung eines Ovarialkarzinoms verbunden sind (Baltzer et al, 2004 ). In ca. 10-15% der Ovarialkarzinome liegt ein hereditärer Erkrankungsweg vor (Risch et al, 2001), wobei das Risiko ein Ovarialkarzinom zu entwickeln signifikant ansteigt, wenn in der Familie bereits Ovarialkarzinome aufgetreten sind (Boyd and Rubin, 1997, Auersperg et al, 2002). Hierbei sind Mutationen der Gene BRCA1 (17q21, ca. 80%), BRCA2 (13q12-13, ca. 15%) und DNA Reparaturgene (MMR, ca. 1-5%) (mit)verantwortlich (Frank et al, 1998). Die Häufigkeit von Mutationen von BRCA1 und BRCA2 liegt bei Frauen mit positiver Familienanamnese bei 30-60% und 15%-30% (Ford et al, 1998, Prat et al, 2005), während BRCA1 und BRCA2 Mutationen bei sporadischen Ovarialkarzinomen nur selten (Stratton et al, 1997, Foster et al, 1996) zu finden sind. -3- Einleitung 2.1.2 Diagnostik Klinik Die Diagnose eines Ovarialkarzinoms beruht auf klinischen Symptomen, gynäkologischen Untersuchungen sowie ergänzender Ultraschalluntersuchung und/oder einer Computertomographie (CT) und Magnetresonanztomographie (MRT) des Bauchraumes. Die endgültige Diagnose wird erst nach operativer Entfernung der „Tumor“-Gewebes anhand pathologischer Untersuchungen gestellt, wobei hier auch die histologischen Klassifizierung und Einteilung der Stadien erfolgt. Pathologie / Histologie Über 60% der Ovarialkarzinome entstehen aus Epithelzellen des Oberflächenepithels, die das Ovar umfangen, oder aus Epithelzellen der inneren Seite der Invaginationszyste (Godwin et al, 1992, Auersperg et al, 2002). Die aus Epithelzellen entstandenen Ovarialkarzinome lassen sich histologisch in acht Gruppen unterteilen, wobei serös-papilläre, muzinöse, endometroide und klarzellige Tumoren am häufigsten auftreten(Tabelle 1). Insbesondere die serös-papillären Ovarialkarzinome machen ca. 50% aller diagnostizierten malignen epithelialen Ovarialtumoren aus. Die Stadieneinteilung wird entsprechend den Empfehlungen der „Federation Internationale de Gynecologie et d'Obstetrique“ (FIGO) vorgenommen (Tavassolli und Devilee, 2003), die die Lokalisation und Ausdehnung des Tumors kennzeichnet (Tabelle 2). Die Stadieneinteilung ist für die weitere Behandlung nach operativer Entfernung des Primärtumors sowie für die Prognose wichtig. So bekommen Patientinnen mit Ovarialkarzinomen des Stadiums Ia (Grad1-2) keine zusätzliche adjuvante Therapie und haben 5 Jahres Überlebensrate von 90-95% (Cannistra, 2004). Im Gegensatz dazu liegt bei der meisten diagnostizierten Ovarialkarzinomen ein fortgeschrittenes Stadium vor, bei dem nach operativer Entfernung des Primärtumors meist eine anschließende Chemotherapie. Die Überlebensrate der Patientinnen mit Ovarialkarzinomen der Stadien III-IV haben daher auch eine 5-Jahres Überlebensrate von nur 20-25% (Wingo et al, 1995, Ozols, 2005). -4- Einleitung Histologischer Low Typ Potential Potential (LMP) (HMP) Serös Malignant High Malignant Andere Seröses Zystadenokarzinom Papilläres seröses Zystadenokarzinom Exophytisches papilläres Karzinom Adenofibrom/Zystadenofibrom Muzinös Muzinöses Zystadenokarzinom Adenofibrom/Zystadenofibrom Endometrioid Zystadenokarzinom Adenokarzinom Adenokarzinom mit Plattenepithelmetaplasie (Adenoakanthom) Endometriales Stromasarkom Mesodermaler ("Müller'scher") Mischtumor Klarzellig (Adeno-) Karzinom (mesonephroi de) Maligne Brenner-Tumoren Gemischte epitheliale Tumoren Undifferenzierte Karzinome Unklassifizierte Karzinome Tabelle 1: Histologische Typen der epithelialen Ovarialkarzinome -5- Stadium Ausbreitung I Tumor auf Ovarien begrenzt Ia Einleitung Tumor auf ein Ovar begrenzt; Kapsel intakt, kein Tumor auf der Oberfläche des Ovars, keine malignen Zellen in Aszites oder bei Peritonealspülung. Ib Tumor auf beide Ovarien begrenzt; Kapsel intakt, kein Tumor auf der Oberfläche der beiden Ovarien, keine malignen Zellen in Aszites oder bei Peritonealspülung. . Ic Tumor begrenzt auf ein oder beide Ovarien mit Kapselruptur,Tumor an Ovaroberfläche oder maligne Zellen im Aszites oder positiver Peritoneallavage. II Tumor befällt ein Ovar oder beide Ovarien und breitet sich im Becken aus IIa Ausbreitung auf und/oder Implantate an Uterus und/oder Tuben;keine malignen Zellen in Aszites oder bei Peritonealspülung. . IIb Ausbreitung auf andere Beckengewebe;keine malignen Zellen in Aszites oder bei Peritonealspülung. IIc Ausbreitung im Becken(2a oder 2b)und maligne Zellen im Aszites oder bei Peritonspülung. III Tumor befällt ein oder beider Ovarien mit mikroskopische nachgewiesenen Peritonealmetastasen außerhalb des Beckens und /oder regionären Lymphknotenmetastasen IIIa Mikroskopische Peritonealmetastasen jenseits des Beckens, IIIb Makroskopische Peritonealmetastasen jenseits des Beckens, größte Ausdehung 2 cm oder weniger IIIc Pritonealmetastasen jenseits des Beckens, größte Ausdehnung mehr als 2cm, und /oder regionäre Lymphknotenmetastasen IV Fernmetastasen(ausschließlich Peritonelmetastasen) Tabelle 2: FIGO Stadien des Ovarialkarzinoms 2.1.3 Therapie Die primäre Therapie des Ovarialkarzinoms ist die chirurgische Entfernung der Tumormasse mit maximaler Zytoreduktion („debulking“) und eine anschließende -6- Einleitung adjuvante Chemotherapie bei späten Stadien (IIb-IV) (Greimel et al, 2006). Bei Patientinnen, bei denen entweder keine maximale Zytoreduktion erreicht wurde, oder bei Patientinnen, bei denen keine Primäroperation möglich ist, wird erst nach einer Chemotherapie (2-3 Zyklen) die Operation durchgeführt (Baltzer et al, 2004). Die Chemotherapie soll die nach einer Operation noch im Patienten verbliebenen Tumorzellen vernichten (bei optimal debulkten Patientinnen entspricht dies einer noch verbliebenen Tumormasse < 1 cm), bzw. deren weitere Proliferation und mögliche Metastasierung verhindern. Für die adjuvante Chemotherapie des Ovarialkarzinoms wird als Standard eine Kombination von Substanzen aus der Platinum- (Carboplatin, Cisplatin) und Taxan(Paclitaxel oder Docetaxel) Gruppe angewandt (Du Bois und Pfisterer, 2005). Substanzen der Platinumgruppe greifen in die DNA-, RNA- und Proteininteraktionen ein und sind auf Grund ihrer „DNA cross-linking“ Eigenschaften zytotoxisch (Agarwal und Kaye, 2003). Bei Taxanen beruht der zytotoxische Mechanismus auf der Induktion einer Destabilisierung von Mikrotubuli, einem Zell-Zyklus Arrest und anschließender Apoptose (Agarwal und Kaye, 2003). 2.1.4 Prognosefaktoren Der einzigste akzeptierte prognostische Marker bei Patientinnen mit fortgeschrittenem Stadium ist der „debulking“ Status. Patientinnen mit optimalem „debulking“(<1cm Tumorrest) haben im Vergleich zu Patinetinnen mit nicht optimalem „debulking“ ein signifikant besseres Überleben. Ein wichtiger klinischer Verlaufsparameter für das epitheliale Ovarialkarzinom ist das Protein CA125, welches im Blut nachgewiesen werden kann. Das CA125 Protein wird von epithelialen Zellen des Ovars exprimiert und ist bei über 80% der Frauen mit epithelialen Ovarialkarzinom signifikant erhöht (Meyer and Rustin, 2000, Cannistra, 2004). Die Bestimmung des CA125 Wertes im Serum dient als Verlaufsparameter während der adjuvanten Behandlung und Nachsorge der Patientinnen. Steigende CA125 Werte nach einer Oparation und adjuvanter Behandlung sind dabei indikativ für das Auftreten eines Rezidivs (Meier et al, 1997, Meyer and Rustin, 2000, Santillan et al, 2005). Ein Nachteil des Tumormarkers CA125 ist, dass er weder in -7- Einleitung frühen Stadien des Ovarialkarzinoms noch bei prämenopausalen Frauen sensitiv nachgewiesen werden kann und sich die Werte von denen bei gutartigen Veränderungen (z.B. Endometriose, Myome oder Entzündung) nicht eindeutig unterscheiden lassen (Meyer and Rustin, 2000). Deshalb wird CA125 weder zur Diagnostik noch zur Prognosebestimmung eingesetzt. Daher werden neue, messbare und für maligne Ovarialkarzinome spezifische und signifikant nachweisbare prognostische Tumormarker gesucht werden. Bekannte Onkogene und Tumorsuppressorgene wurden bisher auf ihren prognostischen Wert hin untersucht: Das bei Mammakarzinomen signifikant veränderte Onkogen HER2/neu (ERBB2, 17q24) ist auch in 20-30% der serösen epithelialen Ovarialkarzinomen mit einer schlechten Prognose assoziiert (Hogdall et al, 2003). Andere Studien weisen aber nur in 10% der epithelialen Ovarialkarzinome eine signifikant veränderte HER2/neu Expression auf (Wu et al, 2004). Die Diversität der untersuchten Patientenkollektive sowie kleine Fallzahlen und unterschiedliche Nachweismethoden limitieren den prognostische Wert von HER2/neu bei Ovarialkarzinomen. Ähnlich verhält es sich beim Tumorsuppressorgen p53 (17p13.1), dessen Expression mit fortschreitendem Tumorstadium von serösen Ovarialkarzinomen ansteigt (Kupryjanczyk et al. 1993). Eine negative oder niedrige p53 Expression ist dabei mit einer besseren Prognose bzgl. des 5-Jahres Überlebens korreliert (Geisler et al, 2001). Neben Onkogenen und Tumorsuppressorgenen wurden auch andere Kandidatenmarker auf ihre prognostische Bedeutung beim Ovarialklarzinom hin überprüft. Insbesondere wurden die Proteine analysiert, die bei der Tumor-Stroma Interaktion, bzw. im Tumormilieu eine Rolle spielen, z.B. Matrix-Metalloproteinase-9 (MMP-9; Lengyel et al, 2001, Demeter et al, 2005) und Vascular Epithelial Growth Factor (VEGF; Veikkola et al, 2000). Allerdings bleibt auch deren prognostischer Wert noch unklar. Es ist zu erwarten, dass bei den neuen sgn. Ziel-gerichteten Therapien (z.B. gegen VEGF) die Untersuchung der molekularen Zielstruktur im Primärtumor eine verbesserte prognostische, bzw-. auch prädiktive Aussage treffen lässt. -8- Einleitung 2.2 STAT1 Signalweg 2.2.1 Übersicht STAT Proteine Der „signal transducer and activator of transcription“ (STAT)-Signalweg wurde vor ca. 10 Jahren bei Arbeiten über die Interferon (IFN)-vermittelte Genexpression entdeckt. Dabei fiel auf, dass im Gegensatz zu der Kaskaden-Signalübertragung, intrazelluläre STAT Proteine Signale von Rezeptoren direkt in den Zellkern übermitteln können ohne die Übertragung auf andere intrazelluläre Proteine. (Schindler et al, 1992). Bei Säugetieren besteht die Familie der STAT Proteine aus sieben Mitgliedern, die evolutionär hoch konserviert sind: STAT 1 bis STAT4, STAT5a, STAT5b und STAT6 (Darnell, 1997). Die STAT Proteine sind im Mittel ca. 750-850 Aminosäuren lang (Takeda und Akira, 2000) und bestehen aus fünf funktionellen Proteindomänen (Abb. 1). STATs sind zytoplasmatische Proteine, die mit membranständigen Rezeptoren interagieren und externe Signale in den Zellkern weiterleiten können (Chen et al, 1998, Levy and Darnell, 2002). Durch ihre unmittelbare Rolle bei der Rezeptor-vermittelten Signalübertragung von Zytokinen, Wachstumsfaktoren und Hormonen sind STAT Proteine in verschiedenen essentiellen zellulären Prozessen involviert, wie z.B. der embryonalen Entwicklung und Organogenese, der Zellproliferation, der Zelldifferenzierung, der angeborenen und adaptiven Immunität und der Angiogenese, (Horvath, 2000, Buettner et al, 2002). Der zelluläre Effekt ist dabei abhängig vom aktivierten STAT Familienmitglied: STAT3 und STAT5 stimulieren die Zellproliferation und Angiogenese, STAT1 immunregulatorische Prozesse und Apoptose. Eine aberrante STAT Expression ist in verschiedenen soliden und hämatologischen Tumoren zu beobachten (Bromberg, 2001, Calo et al, 2003). In Tumoren kann eine erhöhte STAT Aktivität durch konstitutive Aktivierung der Tyrosinkinaserezeptoren, z.B. EGF, oder durch eine konstitutive Aktivität intrazellulärer Tyrosinkinasen, z.B. BCR-ABL, erfolgen (Danial and Rothman, 2000, Yu and Jove, 2004, Levy and Darnell, 2002). Die konstitutive STAT Aktivierung stimuliert die Transkription von Zielgenen, die das Zellwachstum, die Zellproliferation und die Angiogense fördern (Yu und Jove, 2004, Calo et al 2003, Buettner et al 2002). Die detaillierte Kenntnis der STAT Proteine und deren -9- Einleitung Funktionen gewinnen daher an prognostischer Bedeutung. 2.2.2. STAT1: Proteinstruktur Die Struktur und Funktion der einzelnen Proteindomänen von STAT1 ist in Abbildung 1 gezeigt: NH2-Terminale Coiled-coil DNABindungsdomäne Linker TAD SH2 PY PS N 134 317 488 576 683 C 701 727 Abbbildung 1: Die STAT1 Proteinstruktur. Gezeigt sind die funktionellen Domänen von STAT1. Die NH2-terminale Region (AA 1-134) stellt eine Protein-Protein-Interaktionsdomäne dar, die für die Bildung von STAT Dimeren zur Aktivierung notwendig sind (Vinkemeier et al, 1996). Die sgn. coiled-coil-Domäne (AA 135- 317) besteht aus vier Helixbündeln und ist an der Interaktion mit regulatorischen Proteinen und Transkriptionsfaktoren beteiligt (Horvath, 2000). Die DNA-Bindungsdomäne (AA318-488) befindet sich im Zentrum des Moleküls und reguliert die spezifische Bindung von STAT an Promotorregionen der DNA(Horvath, 2000). Die Funktion der Linker-Region (AA 489-576) ist bislang noch unklar. Allerdings führen Mutationen in dieser Domäne zu einem Stabilitätsverlust der Binding von STAT an die DNA und zu einer Verminderung der Genaktivierung (Yang et al, 2002). Die Src-Homology2 (SH2) Domäne (AA 577-683) bestimmt die STAT1 Bindungs-Spezifität und vermittelt die Rekrutierung von STAT1 Monomeren an die phosphorylierten Abschnitte von membranständigen Rezeptoren oder intrazelllulärer Tyrosinkinasen (Shuai et al, 1994, Becker et al, 1998), wie in Abbildung 2 gezeigt. Die sgn. “Transaktivierungsdomäne“ (TAD) (AA684-750) liegt am Carboxy-Terminalen Ende und ist zwischen 38-200 Aminosäuren lang. Die Interaktion der TAD mit nukleären Proteinen trägt zur spezifischen Aktivierung von Zielgenen im Zellkern bei. Für STAT1 ist die TAD Domäne eine wichtige Andockstelle für die minichromosome maintenance (MCM) Proteine 3 und 5. Zudem liegen in der TAD Domäne auch die kritischen Aminosäuren Tyrosin-701 und Serin-727, die die STAT1 Aktivität maßgeblich regulieren (Calo et al, 2003, Buettner et al, 2002, Zhang et al, 1998, Bromberg, 2001, Yu and Jove, 2004). - 10 - Einleitung 2.2.3. STAT1: Aktivierung Die Aktivierung der STAT1 Proteine erfolgt durch Phosphorylierung der Aminosäure Tyrosin-701, wobei eine zusätzliche Phosphorylierung der Aminosäure Serin-727(TAD-Domäne, Abb 1) die Transkriptionsaktivität von STAT1 noch steigern kann (Shuai et al 1994, Wen et al 1995). Letzteres ist zwischen den beiden STAT1 Spleißvarianten, STAT1α und STAT1β, unterschiedlich reguliert: STAT1β besitzt eine verkürzte TAD Domäne und hat somit keine Transkriptionsaktivität (Bromberg et al, 1996, Schindler et al, 1992). Die STAT1 Aktivierung kann über drei verschiedene Wege hervorgerufen werden und bestimmt gleichzeitig die zellulären Effekte (Abb. 2, Buettner et al, 2002): Wachstumsfaktorrezeptor Zytokinrezeptor Extrazellulär RTK RTK C SR STAT R JAK STAT JAK R JAK STAT Intrazelluläre Tyrosinkinasen STAT C SR STAT AB L STAT Zytoplasma T STA T STA Pro-apoptose, Anti-Proliferation Zellkern AT ST P21/WAF1,Fas, P53,Caspase I Abbildung 2: Aktivierung des STAT-Signalweges. Das Schema (modifiziert von Buettner et al, 2002) zeigt die drei wichtigsten Aktivierungsmechanismen von STAT1, wie im Text beschrieben. Die durch STAT1-induzierten Gene, bzw. zellulären Funktionen sind angegeben. - 11 - Einleitung Zytokin-Rezeptor vermittelte STAT Aktivierung Zytokinrezeptoren besitzen keine intrinsische Tyrosinkinaseaktivität können aber über die Rekrutierung von Janus Kinasen (JAK) die Konformation ihrer zytoplasmatische Domäne so ändern, dass Andocksstellen für die STAT-spezifischen SH2 Domänen entstehen (Ihle, 2001). Daran können STAT Monomere binden und durch JAK-induzierte Phosphorylierung der Aminosäure Tyrosin-701 aktiviert werden. Danach erfolgt die Bildung von STAT Homo- oder Hetero-Dimeren, die Translokation in den Zellkern und die Gen-spezifische Aktivierung (Yu and Jove, 2004, Buettner et al, 2002, Bromberg, 2001). Membranständige Rezeptortyrosinkinasen-vermittelte STAT Aktivierung Wachstumsfaktor Rezeptoren wie Epidermal Growth Faktor (EGFR) und Platelet-derived growth factor (PDGF) besitzen eine intrinsische Tyrosinkinasenaktivität in ihrer zytoplasmatischen Domäne. Die Aktivierung von STAT Proteinen erfolgt direkt nach Bindung von Wachstumsfaktoren (z.B. EGF oder PDGF) oder zusätzlich durch assoziierte Janus Kinasen (Yu and Jove, 2004). Intrazelluläre Tyrosinkinasen-vermittelte STAT Aktivierung Intrazelluläre Tyrosinkinasen, wie die Onkogene SRC und ABL, können entweder über Wachstumsfaktorrezeptoren oder aber direkt zu einer Aktivierung von STAT Proteinen führen (Parsons, 1997). 2.2.4. STAT1: Funktion STAT1 hat eine primäre Rolle in Zytokin-vermittelten Signalwegen (Darnell et al 1994), z.B. Interferon gamma (IFNγ) oder Interleukin-6 (IL6). Durch Bindung der Zytokine und ihrer Rezeptoren und der dadurch induzierten Rekrutierung von Janus Kinasen, werden STAT1-Proteine phosphoryliert (P-STAT1), die dann Dimere bilden und in den Zellkern wandern. Dort binden sie an DNA-Sequenzen (z.B. die IFN-γ-assoziierte GAS Sequenz, Decker et al 1997) und induzieren die Transkription der STAT1-assoziierten Zielgene P21(bei IFNγ-Stimulation) oder Caspase-1(bei IL-6 Stimulation). Durch die IFNγ-vermittelten Signale, spielt STAT1 eine entscheidende Rolle bei anti-viralen, anti-bakteriellen Immunantworten und bei der immunologischen Tumorüberwachung. So - 12 - Einleitung sind z.B. STAT1-knock out Mäuse durch eine defekte zelluläre Antwort auf IFNγ Stimulation anfällig für virale und bakterielle Infekte (Durbin et al 1996, Meraz et al 1996) und im Vergleich zu Wild-Typ Mäusen auch anfälliger für die chemische Induktion von Tumoren (Kaplan et al 1998). Trotzdem ist in humanen Tumoren und Tumorzelllinien eine erhöhte Expression von STAT1 beobachtet worden (Bromberg et al 1996, Buettner et al 2002). Dies könnte die Immunantwort auf das Tumorgeschehen widerspiegeln und es ist anzunehmen, dass Tumoren mit stark erhöhter STAT1 Expression eine bessere Prognose aufweisen als Tumoren mit schwächerer STAT1 Expression. Beispielhaft konnte dies schon für das Mammakarzinom gezeigt werden (Widschwendter 2002). Auch ist bekannt, dass die chemotherapeutische Substanz Doxorubin die Expression von STAT1 in vitro erhöht und dadurch zu einer vermehrten Apoptose von Tumorzelllinien führen kann (Thomas et al, 2004). STAT1 reguliert die pro-apoptotischen Proteine Caspase-1, Fas und P21 (Battle und Frank, 2002, Kumar et al, 1997, Chin et al, 1996) sowie das Onkogen MYC (Ramana et al, 2000). 2.2.5. Interaktion von STAT1 mit „Minichromosome Maintenance“ Proteinen Die Transkriptionsaktivität von STAT1 wird durch die Transaktivierungsdomäne (TAD) (Darnell et al, 1994) und zusätzlich durch die Interaktion mit nukleären Kofaktoren, z.B. Minichromosome Maintenance Proteinen 3 und 5 (MCM3,-5), bestimmt (Horvai et al 1997, Zhang et al, 1998). Die evolutionäre konservierte MCM Proteinfamilie hat in Säugetieren sechs Mitglieder MCM2 bis MCM7, die hauptsächlich als DNA-Helikasen bei der DNA Replikation fungieren (Ishimi 1997, Levy und Darnell 2002) und auch eine wichtige Rolle bei der DNA-Transkription, DNA Reparatur und Chromatin-Regulation spielen (Forsburg, 2004, Newlon, 1997). MCM3 und MCM5 können einen Komplex bilden (Richter und Knippers 1997, Prokhorova und Blow 2000), der für die transkiptionelle Aktivität von STAT1 wichtig ist (Synder et al, 2005). Dabei vermitteln die Aminosäuren R732 und K734 des MCM5 Proteins die Verbindung zu STAT1 (Zhang et al, 1998). Eine abnorme MCM Expression kann zur DNA-Schädigung und genetischer Instabilität, einem Hauptmerkmal invasiver - 13 - Einleitung solider Tumoren, führen (Tye, 1999, Lei 2005). Die Ko-Expression von STAT1 und MCM Proteinen in Tumoren wurde bislang noch nicht untersucht. 2.3. Fragestellung und Zielsetzung Die Bedeutung des STAT1 Signalweges beim Ovarialkarzinom wurde bislang noch nicht untersucht. Daher stellt sich die Frage, ob die STAT1 Expression, bzw. die STAT1 Aktivität sowie die STAT1-assoziierten Proteine MCM3 und MCM5 mögliche neue molekulare Prognosemarker bei Patientinnen mit fortgeschrittenen Ovarialkarzinomen darstellen können. Ziel dieser Studie war es daher, die mRNA- und Proteinexpression sowie Aktivität von STAT1 in Ovarialkarzinomen des Stadiums IIIc zu bestimmen und auf ihren prognostischen Wert hin zu überprüfen. Zusätzlich sollte die Expression von STAT1 mit den STAT1-assoziierten Markern MCM3 und MCM5 in epithelialen Ovarialkarzinomzellen korreliert und der mögliche prognostische Wert überprüft werden. - 14 - Materialien und Methoden 3. Materialien und Methoden 3.1 Materialien 3.1.1 Patientinnen, Gewebeproben und klinische Verlaufdaten Insgesamt wurden Tumorgewebe von 114 Patientinnen mit epithelialen Ovarialkarzinomen und normale Ovarien von 29 Patientinnen untersucht. Die Patientinnen wurden zwischen 1998 -2005 an der Frauenklinik primär operiert. Die Gewebe waren alle Formalin-fixiert und Paraffin-eingebettet und stammten aus dem Archiv des Pathologischen Institutes, Universitätsklinikum, Freiburg (Ethikvotum #97/05, Ethik-Kommissionn, Albert-Ludwigs-Universität, Freiburg). Die histologische Klassifizierung der Gewebe erfolgte nach den internationalen, diagnostischen Standardkriterien der WHO (Tavassoli und Devilee 2003). Die klinischen Verlaufsdaten wurden von der Frauenklinik, Universitätsklinikum Freiburg bereitgestellt und umfassten folgende Informationen: Debulking Status nach Operation, adjuvante Chemotherapie (Schema, Zyklen, Abbruch) und das Rezidiv-freie und Gesamtüberleben. Alle Studiendaten wurden anonymisiert behandelt. Tabelle 3 fasst das Patientenkollektiv zusammen. 3.1.2 Chemikalien, Reagenzien und Geräte Laborchemikalien, wie z.B. Xylol, Ethanol etc., und Reagenzien wurden von gängigen Laborlieferanten (Sigma-Aldrich) bezogen. Reagenzien für die molekularen Analysen der mRNA Expression umfassten das „Masterpure RNA Kit“ (Epicenter/Biozym), MMLV Reverse Transcriptase (Invitrogen), Hexanucleotide (Roche), Hefe-RNA (Ambion) sowie Reagenzien für die quantitative RT-PCR und Sequenzierungen (Appliedbiosystems). Reagenzien für die immunhistochemischen Untersuchungen schlossen Primärantikörper von verschiedenen Herstellern (Tabelle 4), Sekundärantikörper und -reagenzien (DAKOCytomation) ein. Alle benutzten Geräte sind in Tabelle 5 aufgelistet. Für alle sonstige Analyse wurden standardisierte Puffer und Lösungen des Pathologischen Institutes Freiburg verwendet. - 15 - n Materialien und Methoden % Histologie Stadium 1a 7 6,1 1c 2 1,8 2a 1 0,9 2b 1 0,9 2c 2 1,8 3b 5 4,4 3c 96 84,2 Serös-papillär 85 59,9 Serös 9 6,3 Papillär 16 11,3 Muzinös 3 2,1 Endometrioid 1 0,7 1 4 3,5 2 39 34,2 3 69 60,5 4 2 1,8 Histologie Differenzierungs Grad Klinik Debulking <1 cm 85 75,9 >1 cm 27 24,1 Keine 11 9,8 Taxol/carboplatin 68 60,7 Carboplatin/Topotecan 15 13,4 Carboplatin/Cyclophosphamid 3 2,7 Carboplatin mono 10 8,9 Caelyx 1 0,9 Andere 4 3,6 Rezidiv 61 62,2 Chemotherapie Rezidiv-freies Überleben 15 Monate (0,5 – 47) (Median) Gesamtüberleben (Median) 23 Monate (range 1 – 82) Tabelle 3: Patientenkollektiv – Zusammenstellung der histopathologischen und klinischen Parameter - 16 - Antikörper Materialien und Methoden Klonalität, Tierquelle Hersteller Cell Signaling Technology STAT 1 Inc.(Beverly,MA,USA) monolonal, Maus Zymed Laboratories (South San Phospho-STAT1(Tyr701) Francisco, USA) monolonal, Maus Stressgen Bioreagents (Victoria, BC Anti-MCM3 monolonal, Maus Canada) Anti-MCM5 monolonal, Maus Novus Biologicals, Inc. Polyklonal Ziege Dako Cytomation (Glostrup Dänemark) Goat Anti-Mouse IgG (Biotinyliert) Tabelle 4: Übersicht der immunhistochemischen Antikörper Gerät und Software-Produkte Hersteller Entparaffinierungsgerat Medite Mikroskop,Kamera Zeiss Mikrowelle Siemens Gewebearray: Multi-Tissue-Arrayer Beecher Instruments, Inc. pH-Meter 211 Hanna Instruments Photometer: ND1000 PeqLab, Erlangen PCR: PTC-200,Peltier Thermal Cycler MJ Research Thermomixer comfort, 1,5ml Eppendorf, Hamburg Zentrifuge 5417R Eppendorf (Hamburg) 310 Genetic Analyzer (Kapillar-Sequenzer) + Applied Biosystems 310 Data Collection Software-Version 3.0.0 Q-RT-PCR: 7000 Sequence Detection System + Applied Biosystems ABI Prism 7000 SDS Software Primer Express Software V1.0 Applied Biosystems Sequencing Analysis Version 3.7 Applied Biosystems Tabelle 5: Übersicht der Geräte und Software-Produkte - 17 - Materialien und Methoden 3.2 Methoden 3.2.1 Auswahl der Gewebeproben Basierend auf den pathologischen Befunden, wurden repräsentative Gewebeblöcke (normale Ovarien oder Ovarialkarzinome, alles Formalin-fixierte und Paraffin-eingebettete Gewebe) aus dem Archiv des Pathologischen Institutes selektiert. Um einen direkten Vergleich der Morphologie mit den Studiendaten zu erhalten, wurden pro Gewebeblock erneut je ein 2 μm dicker Paraffinschnitt für eine Hämatoxylin-Eosin Färbung 1 sowie zwei 10 μm Schnitte für die molekulare Ananlyse angefertigt. Die epithelialen Tumorareale, bzw. normalen Ovarialgewebe wurden von Pathologen für die weiteren Analysen markiert. 3.2.2 RNA Extraktion Bei dieser Arbeit lagen bereits isolierte RNA’s von 105 Gewebeproben vor, so dass die RNA Extraktion an 37 Gewebeproben eigenständig nach dem Protokoll des MasterPure RNA Extraktions Kit durchgeführt wurde. 3.2.2.1. Gewebevorbereitung und Mikrodissektion Von allen Formalin-fixierten, Paraffin-eingebetteten Gewebeblöcken wurden jeweils zwei 10μm dicke Gewebeschnitte hergestellt und bei 60°C für mind. eine Stunde getrocknet. Anschließend erfolgte eine Entparaffinierung der Gewebeschnitte mit Inkubation in 2x/20 min in Xylol, 2x/10min 100% Alkohol und je 5min in einer absteigenden Alkoholreihe (95% bis 50%) und zuletzt Aqua dest.. Danach wurden die Gewebeschnitte kurz in Instant-Hämatoxylin (15s) gefärbt, so dass die Zellmorphologie für die Mikrodissektion sichtbar wurde. Mittels feiner, steriler Nadeln wurden die vom Pathologen ausgewählten Tumorareale manuell mikrodissektiert und in ein mit Lyse-Puffer (300 µl Tissue and Cell Lysis Solution mit 1 µl Proteinase K-50µg/µl) vorbereitetes 1,5ml Eppendorfgefäß übertragen. 1 H&E.-Färbung: Hämatoxylin färbt säure Gewebebestandteile blau-violett z.B. Nukleinsäuren, die Zellkern und Ribosomen; Eosin färbt zahlreiche Strukturen rot z.B. Kollagenfasern - 18 - Materialien und Methoden 3.2.2.2 RNA Extraktion und Aufreinigung Die in den Lyse-Puffer mikrodissektierten Gewebeareale wurden in einem Thermomixer bei 65°C mit 350rpm übernacht, bzw. bis zur vollständigen Lyse (bei Bedarf Zugabe von extra 150 µl Lyse-Puffer und 1 µl Proteinase K am 2. Tag) geschüttelt. Zur RNA Isolierung wurden die Prober anschließend für 3-5 min auf Eis gestellt. Danach wurde zu jeder Probe 225 µl MPC Protein Precipitation Reagent gegeben, für 10s kräftig vortext und 10 min bei 4° C (Raumtemperatur) und 8000rpm zentrifugiert. Danach erhielt man ein Pellet, bestehend aus gefällten Proteinen, und eine wässige Phase, die die gelöste Nukleinsäuren enthielt. Die obere Phase wurde vorsichtig in ein neues 1,5ml Eppendorfgefäß überführt und mit 750µl 100% Isopropanol gemischt. Für die RNA Fällung, wurde das RNA/Isopropanol-Gemisch bei 4°C und 13000rpm für 10 min zentrifugiert. Der dabei entstandene Überstand wurde vorsichtig abgenommen und verworfen und das RNA-Pellet mit 450µl 70% Ethanol gewaschen und dann bei Raumtemperatur kurz getrocknet. Um eventuelle kontaminierende Reste von DNA aus dem RNA-Pellet zu entfernen wurde ein DNase-Verdau durchgeführt: Zum getrockneten RNA-Pellet wurden 150µl DNase I Puffer mit 5µl DNase I (1U/µl) zugegeben, das RNA-Pellet komplett gelöst und bei 37°C für 30 min inkubiert. Anschließend erfolgte eine erneute RNA-Aufreinigung durch Zugabe von 200µl Tissue and Cell Lysis Solution und 200µl MPC Protein Precipitation Reagent. Das Gemisch wurde kurz gemischt, für 5 min auf Eis gestellt und dann bei Raumtemperatur und 8000rpm für 10 min zentrifugiert. Der Überstand wurde vorsichtig in ein neues 1,5ml Eppendorfgefäß überführt, mit 500μl Isopropanol gemischt und erneut bei 4°C und 13000rpm für 30 min zentrifugiert. Das dabei entstandene RNA-Pellet wurde noch 2x mit 70% Ethanol gewaschen und bei Raumtemperatur für 10 min getrocknet. Das getrocknete RNA- Pellet wurde abschließend in 20µl TE Puffer gelöst und bei -20° C gelagert, bzw. gleich für die Folgeuntersuchungen eingesetzt. - 19 - Materialien und Methoden 3.2.2.3 Messungen der RNA-Konzentration Zur Bestimmung der RNA Konzentration wurde 1 µl der gelösten RNA auf einem NanoDrop (ND-1000 Spectrophotometer) Photometer gemessen. Das Prinzip der Messung ist die spezifische optische Dichte der RNA bei einer Wellenlänge von 260nm. Die RNA Konzentration wurde RNA-Konzentration(ng/μl)=OD260*40(Faktor für wie RNA). folgt Um die bestimmt: Reinheit der RNA-Präparation zu überprüfen wurde die Ratio OD260 / OD280 gemessen, wobei eine pure RNA-Lösung zwischen 1,8 und 2 liegt. 3.2.3 Quantitative Reverse Transkriptase (Q-RT-PCR) Polymerase Kettenreaktion 3.2.3.1 cDNA Synthese (RT-Schritt) Für jede RNA-Probe wurde cDNA in einem 20µl Ansatz mit der Reverse Transkriptase aus dem Moloney Murine Leukemia Virus (M-MLV) hergestellt. Dafür wurden zunächst 1 µg RNA, 1µl Yeast RNA (10ng/µl), 2µl Hexanucleotide (10x, 50µg/µl) und Wasser add 9 µl gemischt, bei 70°C für 2 min inkubiert (Verminderung der RNA-Sekundärstrukturen) und anschließend bei Raumtemperatur für 10 min abgekühlt. Zusätzlich wurde eine RT-Negativkontrolle nur mit Wasser angesetzt und auf gleiche Weise behandelt. Zu jeder Probe wurde anschließend 11 µl des bereits vorgefertigten Reaktionsmixes (je Probe: 3 µl Wasser, 4µl 5x-Puffer, 1µl dNTP [10nM], 2µl DTT [0,1M] und 1µl M-MLV[200U/µl]) gegeben und für 1 Stunde bei 37°C inkubiert. Die Reaktion wurde danach mit einer Inkubation bei 95°C für 5 min gestoppt. Die cDNA-Proben wurden anschließend kurz auf Eis gestellt und dann bei -20°C bis zur weiteren Analyse aufbewahrt. 3.2.3.2 Q-(RT)-PCR Bei der quantitativen RT-PCR werden Gensequenzen (hier mRNA) mittels spezifischen Primern durch das Enzym Taq-Polymerase vermehrt und so sichtbar gemacht. Dies wird durch den Einsatz einer fluoreszierenden, Gen-spezifischen „Sonde“ möglich, die - 20 - Materialien und Methoden während der Vermehrung kontinuierlich gemessen werden kann (Abb. 3). 1 2 VorwärtsR Primer VorwärtsPrimer Q 5‘ 3‘ 3‘ 5‘ R Q Taq 3‘ 5‘ 3‘ 5‘ RückwärtsPrimer RückwärtsPrimer 3 VorwärtsPrimer 3‘ 4 R VorwärtsPrimer Q Taq 5‘ 3‘ 5‘ R Q Taq 3‘ 5‘ 3‘ 5‘ RückwärtsPrimer RückwärtsPrimer Abbildung 3: Prinzip der Q-RT-PCR. Zum Beginn jedes PCR-Zyklus (1) ist die Reporterfluoreszent (R) der Sonde ist durch den Quencher (Q) unterdrückt. Bei fortschreitender Amplifikation (2-4) wird die Sonde am 5‘-Ende von der Taq-Polymerase (Taq) verdrängt. Die Reporter-Fluoreszenz kann daher am Ende jedes PCR-Zyklus gemessen werden. Die Messwerte der Q-RT-PCR können anschließend für eine absolute (Korrelation zu Standardkurve) oder relative (Korrelation zu einer positiv Kontrolle) Bestimmung der Gensequenz-Menge herangezogen werden. Dies erfolgt mittels des sgn. Schwellenwertes („threshold Cycle“-, CT-Wert), bei dem ein exponentieller Anstieg der Gen-spezifischen Vermehrung auftritt. Für jede Probe wird der CT-Wert des Zielgens sowie der eines „Kontrollgens“ bestimmt und miteinander wie folgt verrechnet: ΔCTProbe =CT ( ZielgenProbe )-CT ( ReferenzgenProbe). Um den „x-fachen“ Anstieg relativ zu einer Kontrolprobe zu bestimmen, wird anschließend die Differenz der ΔCT-Werte zwischen Probe und Kontrolle (ΔCTProbe - ΔCTKontrolle) = ΔΔCT gebildet. Der „x-fache Anstieg berechnet sich dann: x-fach = 2- ΔΔCT. Für diese Studie wurde für die Q-RT-PCR das „ABI7000“ Gerät (Firma Applied Biosystems) mit entsprechender Software (ABI Prism 7000 SDS) benutzt und die STAT1 mRNA mittels spezifischer Primern und Sonde und relativer Quantifizierung bestimmt. - 21 - Materialien und Methoden Die Methodik war im Labor bereits standardisiert und musste nur für die STAT1-spezifische Messung etabliert werden. Das Design der STAT1-spezifischen Primer und Sonde erfolgte anhand der STAT1 Gensequenz (Ensmbl #BC002704) und einer speziellen Software (Primer Express 1.0, Applied Biosystems), wobei die Primer- und Sondenkombination so ausgewählt wurde, dass ein kurzes Fragment (124 bp) der STAT1 mRNA , nicht jedoch DNA gemessen werden konnte (Abb. 4). Die Primer- und Sondensequenzen waren: Vorwärts-Primer: 5′-GAG CAG GTT CAC CAG CTT TAT GAT-3′, Rückwärts-Primer: 5′-AAC GGA TGG TGG CAA ATG-3′ , Sonde: 6-FAM-AAA-GCA AGA CTG GGA GCA CGC TGC-TAMRA. E1 E3 E2 E4 5’...... Prä-mRNA 5’-Cap ...... mRNA E5 ......3’ ...... 3’-PolyA Q-RT-PCR Produkt Protein N NH2-Terminale PY PS Coiled-coil DNABindungsdomäne Linker SH2 C TAD Abbildung 4: Auswahl der STAT1-spezifischen Primer und Sonde für die Q-RT-PCR. Die Vorwärts- und Rückwärts- Primer(schwarzer Pfeil) liegen separat an Exon(E) 3 und Exon 4. Die Sonde liegt an zwei nachbarliche Exon. Das amplifizierte Produkt zieht sich von NH2-Terminale bis Coil-coil Domäne hin. Für diese Studie wurde ein im Labor standardisierter Q-RT-PCR Ansatz und PCR-Programm verwendet (Tabelle 6). Dafür wurden die Reagenzien laut Tabelle 6 gemischt, in eine 96-Lochplatte überführt, mit Folie bedeckt und im TaqMan System mit entsprechendem PCR-Programm (50°C/2min; 95°C/10min und 40 Zyklen mit 95°C/15sec, 60°C/1min) inkubiert. Die Konzentrationen der Primer und Sonden mussten noch etabliert werden (siehe Ergebnisse). - 22 - Reagenz Materialien und Methoden Einsatzmenge (µl) TaqMan Mix 15 Vorwärtsprimer 3 Rückwärtsprimer 3 Sonde 3 H2O 3 cDNA 3 Tabelle 6: Q-(RT)-PCR Ansatz 3.2.3.3 Sequenzierung Um die Spezifität der STAT1 Q-RT-PCR zu überprüfen, wurde bei 4 Proben das nach der Q-RT-PCR erhaltene Produkt sequenziert. Dazu wurden zunächst die PCR Produkte mittels eines kommerziellen Kits nach Angaben des Herstellers (Qiagen PCR Purification Kit, Qiagen) aufgereinigt und anschließend einer Sequenzierungs-PCR, in der fluoreszenz-markierte dNTP’s eingebaut werden, zugeführt. Dies wurde jeweils einmal für den Vorwärts- und Rückwärtsprimer mit dem BigDye Cycle Sequencing Kit (Applied Biosystems) durchgeführt. Der Pipettieransatz sowie das PCR Programm sind in Tabelle 7 aufgelistet. Nach Abschluß der Sequenzierungs-PCR wurden die PCR-Produkte mittels Fällung aufgereinigt: Hierzu wurde das PCR Produkt (20 µl) mit 50μl 100% Ethanol, 2μl 125mM EDTA, 2μl 3M Natriumacetat gemischt, 15 min bei Raumtemperatur inkubiert und dann für 30 min bei 13000rpm zentrifugiert. Das DNA-Pellet wurde anschließend in 70μl 70% Ethanol gewaschen, getrocknet und in 17 µl Sequenzierungs-Puffer („TSR“, Applied Biosystems) aufgenommen. Es folgte die Analyse in einem Kapillarsequenzer (ABI310) nach Standardprotokollen des Labors. Die erhaltene Gensequenz (Abb. 8) wurde mit den entsprechenden Datenbanken korreliert und ergab eine 100% Übereinstimmung. - 23 - Reagenz Materialien und Methoden Einsatzmenge Einsatzmenge (µl, Probe) (µl, pGEM) DNA 1 1 Primer 3,2 1 BigDye Mix 4 4 5x Puffer 4 4 H2O 7,8 10 1. 92°C 25s 2. RAMP 1.0°C/s zu 50°C 3. 50°C 15s 4. 60°C 4min 5. zum Schritt 1 24 Zyklen 6. 10°C immer Tabelle 7: Ansatz und Programm für die Sequenzierungs-PCR 3.2.4 Immunhistochemie Bei der Immunhistochemie können Proteine in situ an Gewebeschnitten dargestellt werden. Für diese Studie wurden für die Proteinfärbungen von STAT1, PSTAT1, MCM3 und MCM5 Schnitte von sgn. Gewebearrays (Abb.5) verwendet und manuell angefärbt. 3.2.4.1 Gewebearray Die Technik der Gewebearrays ermöglicht die gleichzeitige immunhistochemische Analyse von multiplen Gewebeproben und wurde daher für die Analysen der 143 Gewebeproben dieser Studie eingesetzt. Dazu wurden die vom Pathologen markierten, repräsentativen Tumorareale (bzw. normalen Ovarialgewebe) in Gewebearrays übertragen (Abb. 5): Mittels zweier Nadeln wurden aus den Spenderblöcken die entsprechenden Gewebe-Stanzzylinder (Nadel A, Durchmesser = 0,2 µm) in einen zuvor mit Hohl-Stanzen (NADEL B) versehenen „Empfängerblock“ überführt. Die Empfängerblöcke wurden nach Einbringung der Gewebestanzen bei 60°C für 15 min zur Verschmelzung des Gewebes mit dem Paraffin des Empfängerblocks erwärmt. Von den Gewebearrays wurde vor den immunhistochemischen Färbungen noch eine H&E Färbung zur Kontrolle der gestanzten Gewebeareale angefertigt. - 24 - Materialien und Methoden A B Abbildung 5: Herstellung von Gewebemikroarrays. Die Graphik zeigt den Manual-Tissue-Arrayer mit Gewebe-Stanzzylinder (A) und Hohl-Stanzzylinder (B) wie im Text beschrieben und den angefertigten Gewebearray-Block. 3.2.4.2 Arbeitsschritte Proteinfärbung (Abb. 6) Gewebevorbereitung: Für die Färbungen wurden 4μm dicke Gewebeschnitte von den Gewebearray-Blöcken angefertigt, bei 60°C für mind. 1 Stunde getrocknet und dann 2x 10 min in Xylol und 2x 5min in 100% Alkohol entparaffiniert. Blockierung I: Zur Blockierung der endogenen Peroxidase wurden die entparaffinierten Gewebeschnitte in einem Gemisch aus 500μl 30% Wasserstoffperoxid und 50 ml Methanol für 20 min inkubiert und danach in Wasser gespült. Antigendemaskierung: Um die Antigenbindungsstellen für die Antikörper zugänglich zu machen, ist bei fixierten Gewebe eine spezifische Vorbehandlung notwenig, die durch Hitze-induzierte Inkubation und/oder Proteinandau durchgeführt werden kann. Diese „Antigendemaskierung“ (bzw. „Epitope-Retrieval“) sollte für jeden Antikörper optimiert werden. Hier wurden das Erhitzen der Gewebeschnitte in (Mikrowelle) in a) Zitrat Puffer bei pH 6,0 oder b) Tris Puffer bei pH 10 getestet. Danach wurden die Gewebeschnitte in 1x PBS gespült. Blockierung II: Die Gewebeschnitte wurden mit 200μl 2,2%igem Rinderserumalbumin (BSA) zur zusätzlichen Blockierung für 15 min inkubiert. Danach wurde die BSA-Lösung vorsichtig abgeklopft. Primärantikörper: Die Gewebeschnitte wurden anschließend mit 200μl der verdünnten Primärantikörper (Verdünnungen siehe Resultate) bedeckt, in einer - 25 - Materialien und Methoden feuchten Kammer für eine Stunde bei Raumtemperatur inkubiert und danach 3x für je 2 min in 1xPBS gewaschen. Sekundärantikörper: Die Gewebeschnitte wurden mit 200μl einer 1:200 Verdünnung des biotinylierten Sekundärantikörpers bedeckt, 30 min bei Raumtemperatur inkubiert und danach erneut 3x für je 2 min in 1xPBS gewaschen. ABC-Komplex: Der ABC Komplex besteht aus einer Mischung von Avidin und biotinylierter Meerrettichperoxidase in PBS, die 30 min vor Gebrauch laut Herstellerangaben (DAKO) angesetzt wurde. Dieses Gemisch (100µl) wurde auf die Gewebeschnitte gegeben und für 30min bei Raumtemperatur inkubiert. AEC-Färbereaktion: Die Anfärbung der Gewebeschnitte erfolgte mittels AEC (3-Amino -9-ethylcarbazol) Substrat-Chromogenlösung, die auf die Gewebeschnitte getropft wurde, 10 min inkubiert wurde und dann unter fließendem Wasser für 5 min abgespült wurde. Gegenfärbung (Zellkern) und Abdecken: Dies erfolgte mittels Hämalaun (3 min), Waschen in fließendem Wasser und Eindeckeln mit Aquatex und einem Deckglas. 1 2 biotinylierter Sekundärantikörper 3 4 Primärantikörper AEC: Enzymsubstrat biotinylierter Meerrettichperoxidase Avidin Auswertung Abbildung 6: Immunhistochemie (ABC-Methode). 1) Der Primärantiköprer bindet spezifisch an Gewebe, 2) der biotinylierte Sekundärantikörper bindet an den Primärantikörper, 3) Avidin und biotinylierte - 26 - Materialien und Methoden Meerrettichperoxidase (ABC-Komplex) bindet an den Sekundärantikörper, 4) Peroxidase oxidiert Enzymsubstrat (AEC) und färbt Gewebe spezifisch an. 3.2.4.3 Auswertung der Färbung Die Färbungen wurden semi-quantitativ ausgewertet. Für STAT 1 wurde die zytoplasmatische Anfärbung in Stroma- und Tumorzellen bewertet mit 0 = negativ, 1=Stromazellen gefärbt, 2=Tumorzellen gefärbt, 3=Stroma- und Tumorzellen gefärbt. Für P-STAT1 wurde ausschließlich die Anfärbung der Tumorzellen bewertet und zwar mit 0 = negativ, 1= Zytoplasma gefärbt, 2=Zellkern und Zytoplasma gefärbt, 3=Zellkern gefärbt in mehr als 2 „Tumorzellnestern“. Bei MCM3 wurde die Häufigkeit der nukleär positive Tumorzellen prozentual bewertet: 1 =1-25%, 2=26-50%, 3= 51-75%, 4=75-100%. Für MCM5 wurde die Tumorzellexpression mit 0 = negativ, 1= Zytoplasma gefärbt, 2=Zellkern gefärbt, 3= Zellkern und Zytoplasma gefärbt, gewertet. 3.2.5. Statistische Analyse der mRNA- und Proteinexpressionsdaten Die Studiendaten wurden von Frau Dr. Uta Jütting, Institut für Biomathematik und Biometrie, GSF-Forschungszentrum, München ausgewertet. Dabei wurden mittels des Pearsons Korrelations-Koeffizienten und T-Tests die Korrelation der Marker untereinander und mittels Kaplan-Meier Kurven die prognostische Relevanz der Marker geprüft. - 27 - Ergebnisse 4. Ergebnisse 4.1 STAT1 mRNA Expressionanalyse Um die mRNA Expression von STAT1 in normalen Ovarien und in mikrodissektierten invasiven Tumorzellen von Patientinnen mit Ovarialkarzinom zu bestimmen, wurde RNA aus entsprechenden Formalin-fixierten und Paraffin-eingebetteten Gewebeproben isoliert, in cDNA umgeschrieben und mittels quantitativer RT-PCR gemessen (Material und Methoden 3.2.2., 3.2.3.). Während die Reaktionsbedingungen für das mitzuführende Kontrolgen (TBP) bereits im Labor etabliert waren, mussten für diese Arbeit zunächst noch die Reaktionsbedingungen für die STAT1-spezifische Q-RT-PCR bestimmt werden. Danach wurden die bereits im Labor vorhandenen cDNA Proben von 105 Patientinnen sowie die für diese Arbeit speziell angefertigten RNA Extrakte, bzw. cDNA Proben (n=37) in die etablierten STAT1-spezifische Q-RT-PCR eingesetzt. 4.1.1 Etablierung der STAT1-spezifischen Q-RT-PCR Für die STAT1-spezifische Q-RT-PCR wurden anhand der Gensequenz (ensmbl #BC002704) mit Hilfe einer speziellen Primer-Software (Primer Express V1.0, Applied Biosystems) Primer und eine Intron-überspannend Hybridisierungssonde gewählt (Abb. 4, Material und Methode 3.2.3.2). Die Auswahl der Intron-überspannend Sonde gewährleistet die Amplifikation von ausschließlich mRNA. Zur Etablierung der STAT1-spezifischen Q-RT-PCR gehörte die Bestimmung der geeigneten Primer- und Sondenkonzentrationen, der PCR Sensitivität und Effizienz (um die vergleichbare Amplifikation des Kontrollgens und von STAT1 zu validieren) sowie die Sequenzierung des STAT1 Q-RT-PCR Produktes (Spezifität). Für diese Testreihe wurde eine separate Gewebeprobe, bzw. RNA verwendet. 4.1.1.1 Optimierung der Primer- und Sondenkonzentration Für diese Versuche wurden zunächst aus dem RNA-Extrakt der Kontrolgewebeprobe 300µl cDNA hergestellt (Material und Methoden 3.2.3.1), um für alle zu testenden - 28 - Ergebnisse Bedingungen die gleiche Probe vorliegen zu haben. Zur Bestimmung der optimale Primerkonzentration wurde die Konzentration von Sonde (250 nM) und cDNA (5ng/µl) in jedem PCR Ansatz (50µl) konstant gehalten, aber die Konzentration der Vorwärts- und Rückwärts-Primer variiert und kombiniert (50nM, 300nM und 900nM, Tabelle 8). Alle Kombinationen wurden in 4-fach Ansätzen durchgeführt, um gleichzeitig die Reproduzierbarkeit zu testen. Der PCR-Lauf wurde anschließend nach Standardbedingungen durchgeführt (Material und Methode 3.2.3.2.). Nach Abschluß des PCR-Laufes wurden die CT-Werte der verschiedenen Tests als Mittelwert +/Standardabweichung Primerkonzentration der galt 4-fach die, Ansätze die den ermittelt kleinsten (Tabelle 8). CT-Mittelwert Als mit optimale geringster Standardabweichung aufwies. Für die STAT1-spezifsche Q-RT-PCR wurde hier daher eine Vorwärts- und Rückwärts-Primerkonzentration von mit 900/900nM (Endkonzentration/Ansatz) ausgewählt. CT-Werte (n=4) Primerkonzentration (F/R, in nM) Mittelwert Standardabweichung A 50/50 32,3000 0,21494 B 300/50 31,2250 0,55073 C 900/50 31,2630 0,16581 D 50/300 32,4850 0,43493 E 300/300 31,4775 0,3476 F 900/300 31,2167 0,30172 G 50/900 31,9925 0,47105 H 300/900 31,0875 0,2886 I 900/900 30,8550 0,29411 Tabelle 8: Primeroptimierung Um die optimale Sondenkonzentration zu der ausgewählten Primerkonzentration zu wählen, wurde als einziger Faktor die Sondenkonzentration von 50nM bis 250nM (in - 29 - Ergebnisse 50nM Schritten) variiert. Auch hier wurden 4-Fach Ansätze durchgeführt. Die Auswertung erfolgte wie bereits für die Primeroptimierung beschrieben. Tabelle 9 zeigt, dass eine Sondenkonzentration von 250 nM den kleinsten CT-Mittelwert aufwies. Sonden- CT-Werte (n=4) konzentration Mittelwert Standardabweichung (in nM) J 50 33.82 0.90249 K 100 33.07 0.20089 L 150 32.06 0.10985 M 200 31.02 0.32664 N 250 30.65 0.44642 Tabelle 9: Sondenoptimierung. 4.1.1.2 Bestimmung der Sensitivität und Q-RT-PCR Effizienz Die Amplifikation aller zu untersuchenden mRNA Sequenzen (Zielgen und Referenzgen) muß vergleichbar ablaufen, um die relative Quantifizierung auch bei geringen RNA Mengen zu gewährleisten. Daher wurde anhand einer Verdünnungsreihe von RNA die Sensitivität und Effizienz der STAT1-spezifischen Q-RT-PCR überprüft. Hierzu wurden eine RNA Probe in 5 Schritten verdünnt (Tabelle 10) und nach Standardprotokoll in cDNA umgeschrieben (Material und Methoden 3.2.3). Danach wurde eine Q-RT-PCR angesetzt, bei denen die zuvor bestimmten optimalen Primer- und Sondenkonzentrationen verwendet wurden. Die Ergebnisse zeigten eine gute Amplifizierbarkeit der RNA bis hin zu 2,2 ng (Tabelle 10), allerdings war die Reproduzierbarkeit mit einem RNA Einsatz von 11 ng deutlich besser. Die Ermittlung der Q-RT-PCR Effizienz (E) anhand der aufgetragenen CT-Werte (Y-Achse) gegen den Logarithmus der RNA Konzentration (x-Achse) und der Gleichung [e=10(-1/Steigung a)] zeigte eine Effizienz von 2,1 (Abb. 7 ). Dies kommt dem optimalen Wert von „2“ sehr nahe und ist auch mit der bereits vorliegenden Effizienz des TBP-Assays (E=2,1) vereinbar. - 30 - Ergebnisse RNA Einsatz für eine RNA Menge für eine Log [RNA] CT-Wert 20 µl cDNA Synthese Q-RT-PCR Reaktion (Mittelwert Reaktion [ng] [ng] Standardabweichung) 3300 550 2,74 30,2 +/- 0,15 1650 275 2,44 31,8 +/- 0,23 330 55 1,74 33,9 +/- 0,26 66 11 1,04 35,6 +/- 0,46 13,2 2,2 0,34 38,1 +/- 1,01 +/- Tabelle 10: Einsatzmengen und Ergebnisse des Effizienz-Tests 45 y=-3,1096x+39,094 40 R 2 =0,9904 Ct-Werte 35 30 25 -1 0 1 Log RNA 2 3 Abbildung 7: Sensitivität und Effizienz der STAT1 Q-RT-PCR. Gezeigt sind die Ergebnisse der Q-RT-PCR Analyse von STAT1 mRNA in einer Verdünnungsreihe der RNA Einsatzmenge (log RNA). CT-Werte sind auf der y-Achse angegeben. Je weniger RNA Einsatz desto höher der CT-Wert, mit Korrelation von r²=0.9904. 4.1.1.3 Bestimmung der Q-RT-PCR Spezifität - Sequenzierung Nachdem die optimalen Reaktionsbedingungen, die Sensitivität und Effizienz der STAT1-spezifischen Q-RT-PCR geklärt waren, wurde abschließend die Spezifität mittels Sequenzierung (Material und Methode 3.2.2.3) des Q-RT-PCR Produktes überprüft. Wie in Abb. 8 gezeigt, stimmten die Sequenzen mit der Datenbank überein. - 31 - Ergebnisse pGEM S T A T 1 V o rw ä r ts S T A T 1 R ü c kw ärs Abbildung 8: Sequenzierung der STAT1 Q-RT-PCR Produkte. Die Graphik zeigt die Sequenzergebnisse der STAT1-spezifischen Q-RT-PCR Produkte für den Vorwärts- und Rückwärtsprimer, mit pGEM = Positivkontrolle. 4.1.2 STAT1 mRNA Expression in Ovarialkarzinomen Mit dem etablierten STAT1-spezifischen und dem vorhanden TBP-spezifischen Assays wurden die RNA Proben von Ovarialkarzinomen (n=106) und von normalem Ovar (n=29) untersucht. Wie in Abb. 9 gezeigt, war die STAT1 mRNA Expression zwischen normalen Ovarien und mikrodissektierten Tumorzellen von Ovarialkarzinomen ähnlich. Abbildung 9: Vergleich STAT1 mRNA Expression in normalen Ovarien und in Ovarialkarzinomen. Y-Achse zeigt die relative STAT1 mRNA Expression. No Tu - 32 - 4.2 STAT1, P-STAT1, MCM3 Ergebnisse und MCM5 Proteinexpression in Ovarialkarzinomen Nach Bestimmung der STAT1 mRNA Expression wurde mit Hilfe der Immunhistochemie die Proteinexpression von STAT1 (unphosphoryliert/inaktiv), Phospho-STAT1 (P-STAT1, Tyrosin-phophoryliert/aktiv) und den STAT1-assoziierten Proteinen MCM3 und MCM5 in situ an Gewebemikroarrays der Ovarialkarzinome und normalen Ovarien analysiert (Material und Methoden 3.2.4). Für die Immunhistochemie mussten zunächst das Färbeprotokoll für die verwendeten Antikörper etabliert werden. 4.2.1 Etablierung der Färbungen Hierzu wurde ein Test-Gewebearray mit Kontrollgewebe (Lymphknoten, Ovarialkarzinom, Kolonkarzinom und Zervixkarzinom) (Gewebevorbehandlung, benutzt, Antigen-Demaskierung, um die Reaktionsbedingungen Antikörper Verdünnung und Reaktionszeit) zu optimieren. Wie in Abb. 10 A und B gezeigt, waren die optimalen Bedingungen für die STAT1 Immunhistochemie eine Antigen-Demaskierung in Zitratpuffer bei ph6 und eine 1:100 Verdünnung des STAT1 Antikörpers sowie für die P-STAT1 Immunhistochemie eine Antigen-Demaskierung in Tris-Puffer bei ph10 und eine 1:50 Verdünnung des P-STAT1 Antikörpers. Für die MCM3 und MCM5 Färbungen zeigten beide Antikörper eine Vorbehandlung in Zitrat-Puffer ph6,0, eine 1:400 Verdünnung von MCM3 und eine 1:100 Verdünnung von MCM5(Abb.11) die besten Ergebnisse. - 33 - A Ergebnisse B 1:50 A Zitrat (pH 6) 1:100 Zitrat (pH 6) + Pronase 1:200 Tris (pH 10) 1:400 Abbildung 10: Ergebnisse der Färbeoptimierung von STAT1 (A) und PSTAT1 (B). A: Die 1:100 Verdünnung des STAT1 Antikörpers zeigt eine bessere zytoplasmatische Färbung. B: Die Vorbehandlung mit Trispuffer ermöglichte eine Färbung von P-STAT1. - 34 - A Ergebnisse B 1:100 1:50 1:200 1:100 1:400 Abbildung 11: Ergebnisse der Färbeoptimierung von MCM3 (A) und MCM5 (B). A zeigt die Austestung der Konzentrationsreihe von MCM3 auf Kolonkarzinom Schnitten, wobei eine 1:400 Verdünnung eine starke Nukleärfärbung mit geringstem Hintergrund aufwies. B zeigt die Austestung der Konzentrationsreihe von MCM5 auf Schnitten von Zervixkarzinom. Die 1:100 Verdünnung wies eine vergleichbare Zellkernfärbung wie die 1:50 Verdünnung auf, allerdings mit geringerer Hintergrundfärbung. - 35 - Ergebnisse 4.2.2 STAT1 und P-STAT1 Proteinexpression in Ovarialkarzinomen Die Gewebearrays der Kollektivfälle wurden nach den in 4.2.1. etablierten Protokollen für STAT1 und P-STAT1 gefärbt und semiquantitativ ausgewertet (Material und Methoden 3.2.4.3). Die statistische Analyse der unten beschrieben Daten zeigte, dass im Vergleich zu normalen Ovarien sowohl die STAT1 (p < 0,0001) und P-STAT1 (p < 0,0001) Proteinexpression in den Ovarialkarzinomen signifikant erhöht waren. STAT1: Während die normalen Ovarien keine STAT1 Proteinexpression zeigten, waren in den Ovarialkarzinomen verschiedene Muster der STAT1 Proteinexpression zu beobachten (Abb 12): Eine komplett negative Färbung war in 29/107 (27,1%) Fällen zu sehen, der Rest der Fälle zeigte Unterschiede in der Anfärbung von Tumor-assoziierten Stromazellen (Fibroblasten) bzw. Lymphozyten, und von Tumorzellen (Tabelle 11). Bei 28/107 (26,2%) Fällen waren Stromazellen und Lymphozyten, nicht aber die Tumorzellen STAT1 positiv, bei 23/107 (21,5%) der Fälle war das Stroma negativ aber die Tumorzellen positiv und bei 27/107 (25,2%) der Fälle waren sowohl Stroma als auch Tumorzellen STAT1 positiv. Die Lokalisation der STAT1 Färbung war größtenteils zytoplasmatisch, mit der Ausnahme von 4 Fällen in denen eine nukleäre STAT1 Färbung in den Tumorzellen beobachtet wurde. STAT1 negativ Stromazellen Positivität Anzahl der 29/107 Stroma- und Tumorzellen Tumorzellen Zytoplasma Nukleus 28/107 27/107 19/107 4/107 26,2 25,2 17,8 3,7 Fälle (n/N) Prozent (%) 27,1 Tabelle 11: Übersicht der STAT1 Positivität bei 107 Ovarialkarzinomen - 36 - Ergebnisse A B C D E F Abbildung 12: Repräsentative Beispiele der STAT1 Färbung in Ovarialkarzinomen A und B: STAT1 färbt nur Stromazellen zytoplasmatisch, die die negativ gefärbte Tumorzellen begrenzten. C und D: STAT1 färbt Stromazellen und Tumorzellen zytoplasmatisch an. E und F: STAT1 färbt nur Tumorzellen aber zytoplasmatisch und nukleär an. - 37 - Ergebnisse P-STAT1: In normalen Ovarien konnte keine P-STAT1 Proteinexpression beobachtet werden. Im Gegensatz dazu wurde in den untersuchten Ovarialkarzinomen häufig eine P-STAT1 Positivität gefunden (Abb. 13). Diese P-STAT1 Proteinexpression war allerdings zwischen den Ovarialkarzinomen sehr unterschiedlich, insbesondere die zytoplasmatische und nukleäre Lokalisation sowie die Heterogenität innerhalb eines Tumors, wobei die P-STAT1 Positivität auf die Tumorzellen (keine Stromazellen) beschränkt war. Eine negative P-STAT1 Färbung war in 19/105 (18,1%) der Ovarialkarzinome zu sehen. Eine positive Anfärbung der Tumorzellen war in 47/105 (44,8%) der Ovarialkarzinome als ausschließlich zytoplasmatische, ausschließlich nukleäre und in in 21/105 18/105 (20%) der Ovarialkarzinome als (17,1%) der Ovarialkarzinome als zytoplasmatische und nukleäre Färbung zu finden (Tab.12). Interessant war dabei die Anordnung der P-STAT1 nukleär-positiven Tumorzellen (Abb.13e,f,g): Es bildeten sich in unterschiedlichen Arealen des Tumors „Zell-Nester“, bzw. „Zell-Foci“ von nukleär P-STAT1 positiven Tumorzellen, die sich in ihrer Anzahl zwischen den Ovarialkarzinomen mit diesem Merkmal auch unterschieden. In 21/105 Fällen konnten mehr als 2 Foci von P-STAT1 positiven Tumorzellen (pro Areal der Gewebestanze) gezählt werden. In der Nähe dieser Zell-Nester waren vereinzelt auch Lymphozyten nukleär mit P-STAT1 angefärbt, eine Tatsache, die auf einen sehr spezifischen und aktiven Prozess hinweist. P-STAT1 Positivität Tumorzellen negativ Zytoplasma Zytoplasma Nukleus und Nukleus Anzahl der 19 47/105 18/105 21/105 18,1 44,8 17,1 20 Fälle(n/N) Prozent (%) Tabelle 12.: Übersicht der P-STAT1 Proteinexpression in Tumorzellen der Ovarialkarzinome - 38 - Ergebnisse A B C D E F G Abbildung 13: Repräsentative Beispiele der P-STAT1 Färbung in Ovarialkarzinomen A und B: zytoplasmatische Färbung in Tumorzellen C und D: zytoplasmatische und nukleäre Färbung in Tumorzellen E: nukleäre Färbung in Tumorzellen F und G: „Zell-Nester“ von nukleär gefärbten Tumorzellen - 39 - Ergebnisse 4.2.3 MCM3 und MCM5 Proteinexpression in Ovarialkarzinomen MCM3: In normalen Ovarien sowie dem Tumor-assoziierte Stroma, bzw. in den Lymphozyten der Ovarialkarzinome war keine MCM3 Proteinexpression zu beobachten. Im Gegensatz dazu, zeigten die Tumorzellen der Ovarialkarzinome eine starke nukleäre MCM3 Färbung, wobei unterschiedliche Anteile der Tumorzellen betroffen waren. In 27/105 (25,7%) Fällen waren 1-25% positive Tumorzellen, in 26/105 (24,8%) Fällen 26-50% positive Tumorzellen, in 27/105 (25,7%) Fällen 51-75% Tumorzellen und in 25/105 (23,8%) Fällen 75-100% MCM3-positive Tumorzellen zu sehen (Tabelle 13, Abb. 14). MCM3 Tumorzellen Positivität 1-25% 26-50% 51-75% 76-100% Anzahl der Fälle (n/N) 27/105 26/105 27/105 25/105 Prozent (%) 25,7 24,8 25,7 23,8 Tabelle 13 Übersicht der MCM3 Positivität bei 105 Ovarialkarzinomen MCM5: In einigen (4/29) normalen Ovarien wurde eine schwache zytoplasmatische MCM5 Färbung beobachtet. Das Tumor-assoziierte Stroma sowie infiltrierende Lymphozyten in den Ovarialkarzinomen waren dagegen MCM5 negativ. Von den Ovarialkarzinomen, deren Tumorzellen MCM5 positiv waren, wiesen je 49/107 (45,8%) der Fälle entweder eine ausschließlich zytoplasmatische, oder eine zytoplasmatische und nukleäre MCM5 Proteinexpression auf. In 7/107 (6,5%) Fälle war ausschließlich eine starke nukleäre MCM5 Proteinexpression zu sehen (Tab 14, Abb.15). MCM5 Positivität Tumorzellen negativ Zytoplasma Zytoplasma Nukleus und Nukleus Anzahl der Fälle (n/N) 2/107 49/107 49/107 7/107 Prozent (%) 1,9 45,8 45,8 6,5 Tabelle 14: Übersicht der MCM5 Positivität bei 107 Ovarialkarzinomen - 40 - Ergebnisse A B C D E F G H Abb 14: Beispiele der MCM3 Proteinexpression in Ovarialkarzinomen nach dem Anzahl der nukleär gefärbten Tumorzellen A und B: 1-25% C und D: 26-50% E und F: 51-75% G und H: 76-100% - 41 - Ergebnisse Abb 15: Beispiele der MCM5 Färbung in Ovarialkarzinomen A und B: zytoplasmatische Färbung in Tumorzellen C und D: zytoplasmatische und nukleäre Färbung in Tumorzellen E und F: starke nukleäre aber schwache zytoplasmatische Färbung in Tumorzellen G: nur nukleäre Färbung in Tumorzellen - 42 - 4.3 Korrelation der STAT1, Ergebnisse PSTAT1, MCM3 und MCM5 Proteinexpression zur Histologie und den klinischen Verlaufsdaten Die STAT1 mRNA, die STAT1 und P-STAT1 sowie die MCM3 und MCM5 Proteinexpression wurden mit den Patientendaten (Alter), der Histologie (histologischer Sub-Typ, Grading) sowie den klinischen Verlaufsdaten (Rezidiv-freies und Gesamtüberleben) korreliert (Material und Methoden 3.2.5). Es wurde keine signifikanten Korrelationen zu den histopathologischen Daten gefunden. Eine Tatsache, die mit dem einheitlichen Patientenkollektiv zu erklären ist und daher nicht näher beschrieben wird. 4.3.1 Korrelation der STAT1-, PSTAT1-, MCM3- und MCM5- Expression zu klinischen Verlaufsdaten Für die Überlebensanalysen nach Kaplan-Meier (Material und Methoden 3.2.5), wurden die Patientinnen zunächst in zwei Gruppen - optimales (n=85) und nicht-optimales Debulking (n= 27) - unterteilt, da dies per se einen klassischen prognostischen Marker darstellt (vgl. Einleitung 2.1.4). Innerhalb dieser beiden Gruppen zeigte keiner der bestimmten Marker einen signifikanten progostischen Wert, weder für das Rezidiv-freie noch für das Gesamtüberleben. Um bei der prognostischen Aussage auch den Einfluss der unterschiedlichen Chemotherapien zu berücksichtigen, wurde in einem zweiten statistischen Ansatz der prognostische Wert der STAT1, PSTAT1, MCM3 und MCM5 Expression bei Patientinnen mit adjuvanter Taxol/Carboplatin Chemotherapie (n=68) untersucht. Für die anderen Chemotherapie-Gruppen waren die Fallzahlen für eine statistische Analyse zu gering (vgl. Tabelle 3), so dass ein Vergleich des prognostischen Wertes von STAT1 zwischen verschiedene Chemotherapie-Gruppen nicht möglich war. Die Ergebnisse für die Taxol/Carboplatin-Gruppe zeigten, dass eine hohe STAT1 Proteinexpression mit einem längeren Rezidiv-freien Überleben (p=0,0221) korrelierte (Abb.16 A). Des Weiteren waren eine hohe STAT1 (p=0,0019) und PSTAT1 (p=0,0325) Proteinexpression mit einem längeren Gesamtüberleben verbunden (Abb. 16 B,C). - 43 - Ergebnisse A B C Abbildung 16: Kolleration von STAT1 zum Rezidiv-freien (A) und Gesamtüberleben (B,C) bei Patientinnen mit Taxol/Carboplatin (n=68). Die Abb. zeigt Kaplan-Meier Analyse wobei: rote Kurve = niedrige STAT1 Expression, blaue Kurve = Mittelmäßige STAT1 Expression und magenta Kurve = hohe STAT1 Expression. - 44 - Ergebnisse 4.3.2 Korrelation der STAT1, PSTAT1, MCM3 und MCM5 Expression miteinander Um zu klären inwieweit die Expression von STAT1, PSTAT1, MCM3 und MCM5 in den Ovarialkarzinomen miteinander korrelieren, wurden die Marker untereinander verglichen. Dies ergab eine Korrelation zwischen der STAT1 mRNA Expression und der STAT1 (p<0,0001) und PSTAT1 (p= 0,0004) Proteinexpression. Die STAT1 und PSTAT1 Proteinexpression korrelierten untereinander mit p=< 0,0001. Die STAT1 mRNA Expression korrelierte ebenfalls mit der MCM3 (p = 0,0005) und MCM5 (p = 0,0118) Proteinexpression. Auch die STAT1 Proteinexpression korrelierte mit MCM3 (p= 0,0317) und MCM5 (p = 0,0346) Proteinexpression. Im Gegensatz dazu war die PSTAT1 Proteinexpression nicht mit der MCM3 (p =0,8732) oder MCM5 (p = 0,261) Proteinexpression korreliert. - 45 - Diskussion 5 Diskussion Das Ovarialkarzinom ist die siebt-häufigste Krebserkrankung bei Frauen und ist, nach dem Mammakarzinom, die häufigste Todesursache gynäkologischer Krebserkrankungen. Die 5-Jahres-Überlebensrate ist trotz Fortschritte in der Diagnostik und Behandlung mit ca. 20-25% gering. Die Identifizierung molekularer Prognosemarker könnte verbesserte und individualisierte Therapieprotokolle ermöglichen. Die Proteine der „Signal Transducer and Activator of Transcription“ (STAT) Familie übertragen unter streng regulierten physiologischen Bedingungen Signale von Zytokinund Wachstumsfaktor-Rezeptoren sowie von intrazellulären Tyrosinkinasen in den Zellkern und schalten damit die Transkription von spezifischen Genen an. Damit werden definierte zelluläre Funktionen, wie z.B. Zellproliferation, gesteuert. Eine aberrante Expression von STAT Proteinen, bzw. deren dauerhafte Aktivierung, wurde in verschiedenen humanen Tumoren beschrieben (Yu und Jove, 2004). Dabei ist STAT3 bisher am besten untersucht und wird bereits als Onkogen bezeichnet (Calo et al, 2003). Im Gegensatz dazu gibt es zwar einige Daten zu den physiologischen, aber kaum Daten zu den pathologischen Funktionen von STAT1, insbesondere betroffenen maligne Tumoren. So ist bekannt, dass STAT1 sich negativ auf die Proliferation und Metastasierung von Tumorzelllinien auswirkt und zu einer verminderten Angiogenese führen kann (Huang et al, 2002). Des Weiteren war zu beobachten, dass eine erhöhte STAT1 Expression bei gleichzeitigem Vorliegen einer P53 oder BRCA1 Mutation zu einer vermehrten Tumorzellapoptose führte (Welcsh et al, 2001,Townsend et al, 2004). Es konnte für Mammakarzinome gezeigt werden, dass eine hohe STAT1 Aktivität mit einer besseren Prognose einher ging (Widschwendter et al, 2002). Somit könnte STAT1 eher ein Tumorsuppressorgen als ein Onkogen darstellen (Bromberg et al, 1996, Kaplan et al, 1998). Die STAT1 Expression in Ovarialkarzinomen sowie deren mögliche prognostische Bedeutung sind bisher noch nicht bekannt. Ziel dieser Studie war es daher die mRNA und Proteinexpression von STAT1 und den STAT1-assoziierten „MiniChromosome-Maintenance“ Proteinen MCM3 und MCM5 in - 46 - Diskussion epithelialen Ovarialkarzinomen des Stadiums IIIc zu untersuchen und auf ihren prognostischen Wert hin zu überprüfen. Da dies die Auswahl und Untersuchung eines homogenen Patientenkollektives voraussetzte, konzentrierte sich die Diskussion daher auf die mögliche biologische und klinische Bedeutung des STAT1 Signalweges in epithelialen Stadium IIIc Ovarialkarzinomen. Die im Ergebnisteil vorgestellten Daten zeigen, dass die Analyse der ausgewählten Marker sowohl auf mRNA- als auch auf Proteinebene in allen der Formalin-fixierten und Paraffin-eingebetteten Gewebe möglich war. Die Sensitivität, Spezifität und Effizienz der neuen STAT1 Q-RT-PCR wurden bestimmt und zeigten optimale Werte. Für die Proteinanalyse konnten die Färbungen für die ausgewählten Primärantikörper (STAT1,PSTAT1,MCM3,MCM5) an Serienschnitten der Formalin-fixierten und Paraffineingebetten Gewebeproben etabliert werden. 5.1 Die Bedeutung des STAT1 im Signalweges in Ovarialkarzinomen Die Aktivierung des STAT1 Signalweges kann über drei Mechanismen erfolgen (Einleitung, Abb. 2), wobei die Kaskade IFNγ-JAK-STAT1 einen Schwerpunkt einnimmt und hauptsächlich immunologische Kontrollfunktionen, wie z.B. anti-virale Aktivität, sowie anti-proliferative Funktionen stimuliert. Die untersuchten Ovarialkarzinome zeigten in 51,4% der Fälle eine STAT1 positive Proteinexpression in den Tumor-assoziierten Stromazellen mit oder ohne gleichzeitige Expression in den Tumorzellen. Dies betraf vor allem Fibroblasten und Lymphozyten, die die Tumorzellen eingrenzten (vgl. Abb.12 A, B). Es ist anzunehmen, dass der entstehende Tumor eine Stromareaktion mit vermehrter Produktion von IFNγ anregt und so zu einer erhöhten und aktivierten Tumor-assoziierten Lymphozyten bzw. Fibroblasten Population führt. Die genaue Rolle erhöhter STAT1 Proteinexpression in Tumor-assoziierten Lymphozyten oder Fibroblasten ist bis jetzt noch nicht geklärt. Allerdings könnte ein Tumor-Stroma-Milieu vorliegen, das über IFNγ und STAT1 die Lymphozyten-Infiltration weiter unterstützt und so eventuell auch eine Lymphozyten-vermittelte Immunreaktion, die sich gegen den Tumor richtet, ermöglicht. - 47 - Diskussion Tatsächlich hatten in der vorgelegten Studie Ovarialkarzinom-Patientinnen mit adjuvanter Taxol/Carboplatin Therapie und mit STAT1-positiven (Lymphozyten und Tumorzellen) Tumoren ein signifikant längeres Rezidiv-freies und Gesamt-Überleben als Patientinnen mit STAT1-negativen Tumoren. Eine mögliche immunologische Kontrolle von Ovarialkarzinomen mit einhergehender STAT1 Aktivierung und die damit assoziierte stärkere Induktion von Tumorzell-Apoptose ist daher denkbar. Im Gegensatz dazu ist interessant, dass bei einigen Patientinnen (25,2%) ausschließlich Tumorzellen eine zytoplasmatische (21,5%) oder nur nukleäre (3,7%) STAT1 Expression aufwiesen. Dies könnte entweder durch eine Zytokin-vermittelte Reaktion und STAT1 Aktivierung entstanden sein, wie sie z.B. auch durch IFNγ in vitro bei Tumorzelllinien induziert werden kann (Lillemeier et al, 2001), oder auch dadurch, dass die Tumorzellen konstitutiv aktiviertes STAT1 besitzen, wie es z.B. auch für STAT3 bekannt ist (catlette-falcone et al, 1999, Yu und Jove, 2004 ). Ähnlich wie bei der positiven STAT1 Expression, war auch die Phospho-STAT1 Proteinexpression in Ovarialkarzinomen, zumindest bei Patientinnen mit adjuvanter Taxol/Carboplatin Therapie, mit einem längeren Gesamtüberleben korreliert. Interessant waren zwei Beobachtungen: 1. Die P-STAT1 Proteinexpression wurde nur in 37,1% der Fälle im Zellkern der Tumorzellen detektiert. Es ist bekannt, dass nur ca. 1/3 der STAT1 Proteine bei Aktivierung des Signalweges aktiviert werden und somit als P-STAT1 in den Zellkern wandern (Haspel et al, 1996, Vinkemeier, 2004). Auch wurde gezeigt, dass STAT-Proteine zwischen Zytoplasma und Nukleus unabhängig von externer Stimulation pendeln können (Meyer et al, 2002) und dies auch für unphosphoryliertes STAT1 gilt (Chatterjee-kishore et al, 2000). Beide Mechanismen könnten die geringe nukleäre P-STAT1 Positivität erklären. Des Weiteren könnte der nukleäre Import des P-STAT1 in den Tumorzellen blockiert sein. In der STAT1-spezifischen DNA bindungsdomäne befindet sich ein sognannten „nuclear localisation signal“ (NLS) (Melen et al, 2001), das als „dimer-specific“ NLS (dsNLS) für das Einschleusen von (P-)STAT1 in den Zellkern verantwortlich ist (Meyer et al, 2002). Ein Fehlen oder - 48 - Diskussion Blockieren der NLS wäre für eine Akkumulation von P-STAT1 im Zytoplasma verantwortlich und würde die zytoplasmatische P-STAT1 Proteinexpression in 44.8 % der untersuchten Ovarialkarzinome (vgl. Tabelle 12, Abb. 13) erklären. 2. Bei den 20% der Fällen mit nukleärer P-STAT1 Expression war das Färbemuster im Tumorareal nicht homogen, sondern fokal als Tumorzellnester zu sehen (vgl. Abb. 13 E,F,G). Diese fokal auf Tumorzellnester begrenzte P-STAT1 Expression könnte eine spezifische, evtl. auch prognostisch relevante Tumorzell-Subpopulation darstellen, die z.B. durch genetische Veränderungen STAT1 konstitutiv aktiviert. Die Tumorzellnester korrelierten weder mit einer parallelen Expression von MCM3 und MCM5 (vgl. Abb. 14, Abb. 15) noch mit der Infiltration von CD3 oder CD20-positiven T- oder B-Lymphozyten (Daten nicht gezeigt), so dass eine durch das Tumor-Stroma-Milieu verursachte und lokal begrenzte (IFNγ) Stimulation auszuschließen ist und eher an eine konstitutive Tumorzell-spezifische STAT1 Aktivierung zu denken ist. Um zu überprüfen inwieweit die Regulation der STAT1 und P-STAT1 Proteinexpression von der STAT1 mRNA Expression abhängig ist, wurden die STAT1 mRNA Werte mit den STAT1 Proteindaten in den Ovarialkarzinomen korreliert. Die Daten zeigten, dass STAT1 mRNA, Proteinexpression (STAT1) und Aktivität (P-STAT1) miteinander signifikant korrelierten. Allerdings war auf mRNA Ebene weder ein Unterschied zwischen normalen Ovarien und Ovarialkarzinomen noch eine signifikante Korrelation zum Überleben zu sehen. Dies könnte dadurch erklärt werden, dass die STAT1 mRNA Expression nicht so stabil ist (Abril et al,1998) wie die STAT1 Proteinexpression (Haspel et al,1996) und der mögliche schnelle Abbau der STAT1 mRNA in den operierten Ovarialkarzinomen nicht zu erfassen war. Trotzdem unterstützt die enge Korrelation der STAT1 mRNA und Proteinexpression sowie der Aktivität (P-STAT1) in Ovarialkarzinomen die Bedeutung des STAT1-Signalweges für die Entstehung und Progression von Ovarialkarzinomen. Wichtig ist, dass ein deutlicher Unterschied zwischen normalen Ovarien und Ovarialkarzinomen, bzw. insbesondere zwischen Patientinnen mit guter oder schlechter Prognose, nur auf Basis des aktivierten STAT1 Proteins (P-STAT1) und nicht auf STAT1 - 49 - Diskussion mRNA zu sehen ist. Dies würde die mögliche zukünftige Anwendung von STAT1 als zusätzlichen prognostischen Marker erleichtern, da immunhistologische Färbungen einfach in die Routinediagnostik einzubauen wären. Auch wäre zu überlegen, ob man den STAT1 Signalweg therapeutisch nutzen könnte, z.B. durch Steigerung der STAT1 Aktivität mittels IFNγ-Stimulation. Tatsächlich war allein die STAT1 Proteinexpression, bzw. Aktivität prognostisch relevant, nicht jedoch die mRNA Expression. Sowohl die STAT1 als auch P-STAT1 Proteinexpression korrelierten signifikant mit dem Gesamtüberleben. Dabei war eine starke STAT1 und/oder P-STAT1-Proteinexpression in Stroma- und/oder Tumorzellen mit einer besseren Prognose assoziiert. Während die STAT1 Expression in den Stromazellen, wie bereits oben diskutiert, eine mögliche immunregulatorische, anti-Tumor Rolle spielt, wäre die günstige Auswirkung der Tumorzell-spezifischen STAT1 Expression eher mit einer Rolle von STAT1 als Tumorsuppressorgen zu vereinbaren. Somit sollte eher der Verlust, nicht aber die Aktivierung von STAT1 in Tumorzellen zur Progression von Ovarialkarzinomen beitragen. Eine mögliche Rolle von STAT1 als Tumorsuppressor ist nicht auszuschließen, da die STAT1 Aktivität anti-proliferativ wirkt (Ramana et al, 2000). Auch in anderen gynäkologischen Tumoren, wie z.B. dem Mammakarzinom (Widschwender et al, 2002) konnte eine solche prognostische Relevanz der STAT1 Proteinexpression gezeigt werden. 5.2 Die Bedeutung der MCM3 und MCM5 Proteinexpression in Ovarialkarzinomen Neben der STAT1 Expression wurde in der vorgelegten Studie auch die Proteinexpression der STAT1-assoziierten Marker MCM3 und MCM5 untersucht, um hier eine weitere Komponente des STAT1 Signalweges in Ovarialkarzinomen zu bestimmen. Die Funktion der MCM Proteine ist im Wesentlichen die DNA Replikation. MCM Proteine sind Komponenten des DNA Replikations „licensing-systems“, das die einmalige DNA - 50 - Diskussion Replikation jedes Zellzykluses garantiert (Tye, 1999). Somit stellen die MCM Proteine auch einen Marker für den Zellzyklus dar (Ha et al, 2004, Freeman et al, 2003). Insbesondere scheinen die MCM Proteine, vergleichbar mit anderen Chromatin-assoziierten Markern wie cdc6, Zielproteine für die Phosphorylierung durch Zytokin-unabhängige Kinasen zu sein, deren abnorme Regulation zur unkontrollierten DNA Replikation führen kann (Ishimi et al, 2003). In „ruhenden“ Zellen und ausdifferenzierten Zellen nimmt die MCM Proteinexpression stark zu. In normalen Geweben findet sich die MCM Expression fokal auf Proliferationsareale begrenzt. In dysplastischen oder invasiv malignen Zellen ist die Expression von MCM Proteinen sehr prominent (Ishimi et al, 2003, Freeman et al, 2003), wobei z.B. die MCM3 Expression verglichen mit den Proliferationsmarkern Ki-67 in Zervixkarzinomen deutlicher ausgeprägt ist (Ha et al, 2004). Eine Assoziation von MCM Proteinen mit Zellmalignität ist daher anzunehmen. In dieser Studie wurde die MCM3 und MCM5 Proteinexpression in malignen Ovarialkarzinomen bestimmt. Die Daten zeigten eine starke nukleäre MCM3 Proteinexpression in allen Ovarialkarzinomen und auch das MCM5 Protein war in den meisten Ovarialkarzinomen (98,1%) als positive Tumorzellfärbung zu finden. Im Gegensatz dazu, war in allen untersuchten normalen Ovarien sowie in dem Tumor-assozierte Stroma der Ovarialkarzinome keine signifikante MCM3 Proteinexpression zu beobachten. Das MCM5 Protein lag sehr schwach im Zytoplasma der normalen Ovarzellen vor und war im Tumor-assozierte Stroma negativ. Diese Daten deuten daher auf eine Assoziation der MCM3 und MCM5 Proteinexpression mit der Proliferation maligner Ovarialkarzinomzellen hin. Eine weitere wichtige funktionelle Rolle der MCM Proteine ist die Regulation der transkriptionellen Aktivität von STAT1 (Forsburg, 2004). So konnte durch Ausschalten der MCM5 mRNA mittels sgn. „small inhibitory RNA“ (siRNA) eine verminderte Expression der STAT1-induzierbaren Gene, nicht aber von STAT1 Expression oder Aktivierung nachgewiesen werden (Synder et al, 2005). Hier konnte gezeigt werden, dass die MCM3 und MCM5 Proteinexpression in den Tumorzellen der Ovarialkarzinome sehr stark - 51 - Diskussion nukleär ausgeprägt war. Während die STAT1 Expression mit der MCM3 und MCM5 Proteinexpression korrelierte, war dies für PSTAT1 nicht der Fall. Dies könnte daran liegen, dass die Interaktion von STAT1 und MCM Molekülen auch durch die STAT1-Serin Phosphorylierung bestimmt wird, die in dieser Studie nicht untersucht worden ist. Es ist allerdings anzunehmen, dass auch die STAT1 oder STAT1 induzierte Transkkriptionsaktivität die DNA Replikation beeinflusst und die IFNγ−Stimulation des STAT1 Signalweges die Zellproliferation durch Blockierung der G1 zu S Phase verhindern kann. Liegen Mutationen der STAT1 Bindungsstelle (Serin727) für MCM(5) Proteine vor, kann diese Blockierung der Zellproliferation durch den STAT1 Signalweg aufgehoben werden (Zhang et al, 1998, Bromberg et al, 1996). Die Interaktion zwischen STAT1 und MCM Proteinen steuert also zwei verschiede wichtige zelluläre Prozesse, die DNA Replikation und die Zytokin induzierte Transkription (Danvonseca,et al 2001). Die Balance zwischen den Interaktionen ist somit verantwortlich für die Auswirkungen auf die DNA Replikation und Zellproliferation. 5.3 Fazit Diese Studie hat den STAT1-Signalweg als neuen prognostisch-relevanten Marker für Patientinnen mit fortgeschrittenen Ovarialkarzinomen identifiziert. Dabei scheint sich eine hohe STAT1 Aktivität günstig auf das Rezidiv-freie und Gesamtüberleben bei adjuvanter Taxol/Carboplatin Chemotherapie auszuwirken. Des Weiteren besteht eine Assoziation des STAT1 Signalweges mit der Proteinexpression der MCM3 und MCM5 Proteine in Tumorzellen von Patientinnen mit Ovarialkarzinomen. - 52 - Literaturangaben 6 Literaturangaben Abril E, Real LM, Serrano A, Jimenez P, Garcia A, Canton J, Trigo I, Garrido F, Ruiz-Cabello F (1998) Unresponsiveness to interferon associated with STAT1 protein deficiency in a gastric adenocarcinoma cell line. Cancer Immunol Immunother. 47(2):113-20 Agarwal R, Kaye SB (2003) Ovarian cancer: strategies for overcoming resistance to chemotherapy. Nat Rev Cancer 3(7):502-16 Auersperg N, Ota T, Mitchell GW (2002) Early events in ovarian epithelial carcinogenesis: progress and problems in experimental approaches. Int J Gynecol Cancer 12(6):691-703 Baltzer J, Friese K, Graf M, Wolff F (2004) Praxis der Gynäkologie und Geburtshilfe das komplette Praxiswissen in einem Band. 1. Auflage Georg Thieme Verlag Stuttgart. New York pp. 567-577 Battle TE, Frank DA (2002) The role of STATs in apoptosis Curr Mol Med 2:381–92 Becker S, Groner B, Muller CW (1998) Three-dimensional structure of the STAT3b homodimer bound to DNA. Nature (Lond) 394:145–151 Bromberg JF, Horvath CM, Wen Z, Schreiber RD, and Darnell JE Jr (1996) Transcriptionally active Stat1 is required for the antiproliferative effects of both interferon α and interferon γ. Proc Natl Acad Sci. USA, 93: 7673–7678 Bromberg, JF (2001) Activation of STAT proteins and growth control. Bioessays. 2001 23(2): 161-9 Boyd J, Rubin SC. (1997) Hereditary ovarian cancer: molecular genetics and clinical implications. Gynecol Oncol 64(2):196-206 Buettner R, Mora LB, Jove R (2002) Activated STAT signaling in human tumors provides novel molecular targets for therapeutic intervention. Clin Cancer Res. 2002 8(4):945-54 Calo V, Migliavacca M, Bazan V, Macaluso M, Buscemi M, Gebbia N, Russo A (2003) STAT proteins: from normal control of cellular events to tumorigenesis. J Cell Physiol 197(2):157-68 Cannistra SA (2004) Cancer of the ovary. N Engl J Med. 2004 351(24):2519-29 Catlett-Falcone R, Landowski TH, Oshiro MM, Turkson J, Levitzki A, Savino R, Ciliberto G, Moscinski L, Fernandez-Luna JL, Nunez G, Dalton WS, Jove R (1999) Constitutive activation of Stat3 signaling confers resistance to apoptosis in human U266 myeloma cells. Immunity. 10(1):105-15 Chatterjee-Kishore M, Wright KL, Ting JP, Stark GR (2000) How Stat1 mediates constitutive gene - 53 - Literaturangaben expression: a complex of unphosphorylated Stat1 and IRF1 supports transcription of the LMP2 gene. EMBO J. 19(15):4111-22 Chin YE, Kitagawa M, Su WC, You ZH, Iwamoto Y, Fu XY (1996) Cell growth arrest and induction of cyclin-dependent kinase inhibitor p21 WAF1/CIP1 mediated by STAT1. Science 272:719–722 Dafonseca cj, shu f, zhang 2001 DaFonseca CJ, Shu F, Zhang JJ(2001) Identification of two residues in MCM5 critical for the assembly of MCM complexes and Stat1-mediated transcription activation in response to IFN-gamma. Proc Natl Acad Sci U S A. 98(6):3034-9 Danial NN, Rothman P (2000) Jak-Stat signaling activated by Abl oncogenes. Oncogene, 19: 2523–2531 Darnell JE Jr, Kerr IM, Stark GR (1994) Jak-STAT pathways and transcriptional activation in response to IFNs and other extracellular signaling proteins. Science 264(5164):1415-21 Darnell JE Jr (1997) STATs and gene regulation. Science 277:1630±1635 Decker T, Kovarik P, Meinke A (1997) GAS elements: a few nucleotides with a major impact on cytokine-induced gene expression. J Interferon Cytokine Res 17(3):121-34 Demeter A, Sziller I, Csapo Z, Olah J, Keszler G, Jeney A, Papp Z, Staub M (2005) Molecular prognostic markers in recurrent and in non-recurrent epithelial ovarian cancer. Anticancer Res 25(4):2885-9 Du Bois A, Pfisterer J (2005) Future options for first-line therapy of advanced ovarian cancer. Int J Gynecol Cancer 15 Suppl 1:42-50 Durbin JE, Hackenmiller R, Simon, MC, and Levy DE (1996) Targeted disruption of the mouse Stat1 gene results in compromised innate immunity to viral disease. Cell, 84: 443–450 Ford D, Easton DF, Stratton M, Narod S, Goldgar D, Devilee P, Bishop DT, Weber B, Lenoir G, Chang-Claude J, Sobol H, Teare MD, Struewing J, Arason A, Scherneck S, Peto J, Rebbeck TR, Tonin P, Neuhausen S, Barkardottir R, Eyfjord J, Lynch H, Ponder BA, Gayther SA, Zelada-Hedman M (1998) Genetic heterogeneity and penetrance analysis of the BRCA1 and BRCA2 genes in breast cancer families. Am J Hum Genet 62(3):676-89 Forsburg SL (2004) Eukaryotic MCM proteins: beyond replication initiation. Microbiol Mol Biol Rev 68(1):109-31 Foster KA, Harrington P, Kerr J, Russell P, DiCioccio RA, Scott IV (1996) Somatic and germline mutations of the BRCA2 gene in sporadic ovarian cancer. Cancer Res 56:3622– 5 Frank TS, Manley SA, Olopade OI, et al (1998) Sequence analysis of BRCA1 and BRCA2: correlation of - 54 - Literaturangaben mutations with family history and ovarian cancer risk. J Clin Oncol 16:2417– 2425 Freeman A, Morris LS, Mills AD, Stoeber K, Laskey RA, Williams GH, Coleman N(1999) Minichromosome maintenance proteins as biological markers of dysplasia and malignancy. Clin Cancer Res 5(8):2121-32 Friedlander M, Millward MJ, Bell D, Bugat R, Harnett P, Moreno JA, Campbell L, Varette C, Ripoche V, Kayitalire L (1998) A phase II study of gemcitabine in platinum pre-treated patients with advanced epithelial ovarian cancer. Ann Oncol 9(12):1343-5 Geisler HE, Geisler JP, Miller GA, Geisler MJ, Wiemann MC, Zhou Z, Crabtree W (2001) p21 and p53 in ovarian carcinoma: their combined staining is more valuable than either alone. Cancer Aug 15;92(4):781-6 Godwin AK, Testa JR, Handel LM, Liu Z, Vanderveer LA, Tracey PA, Hamilton TC (1992) Spontaneous transformation of rat ovarian surface epithelial cells: association with cytogenetic changes and implications of repeated ovulation in the etiology of ovarian cancer. J Natl Cancer Inst 84(8):592-601 Greimel ER, Bjelic-Radisic V, Pfisterer J, Hilpert F, Daghofer F, du Bois A (2006) Arbeitsgemeinschaft Gynaekologische Onkologie Ovarian Cancer Study Group. Randomized study of the Arbeitsgemeinschaft Gynaekologische Onkologie Ovarian Cancer Study Group comparing quality of life in patients with ovarian cancer treated with cisplatin/paclitaxel versus carboplatin/paclitaxel. J Clin Oncol 24(4):579-86 Ha SA, Shin SM, Namkoong H, Lee H, Cho GW, Hur SY, Kim TE, Kim JW(2004) Cancer-associated expression of minichromosome maintenance 3 gene in several human cancers and its involvement in tumorigenesis. Clin Cancer Res 10(24):8386-95 Haspel RL, Salditt-Georgieff M, Darnell JE Jr (1996) The rapid inactivation of nuclear tyrosine phosphorylated Stat1 depends upon a protein tyrosine phosphatase.EMBO J. 15(22):6262-8 Hogdall EV, Christensen L, Kjaer SK, Blaakaer J, Bock JE, Glud E, Norgaard-Pedersen B, Hogdall CK (2003) Distribution of HER-2 overexpression in ovarian carcinoma tissue and its prognostic value in patients with ovarian carcinoma: from the Danish MALOVA Ovarian Cancer Study. Cancer 98(1):66-73 Horvath CM (2000) STAT proteins and transcriptional responses to extracellular signals. Trends Biochem Sci 25(10):496-502 Huang S, Bucana CD, Van Arsdall M, Fidler IJ (2002) Stat1 negatively regulates angiogenesis, tumorigenicity and metastasis of tumor cells. Oncogene 21(16):2504-12 Ihle JN (2001) The Stat family in cytokine signaling. Curr Opin Cell Biol 2001 13(2):211-7 Ishimi Y, Okayasu I, Kato C, Kwon HJ, Kimura H, Yamada K, Song SY(2003) Enhanced expression of Mcm - 55 - Literaturangaben proteins in cancer cells derived from uterine cervix. Eur J Biochem. 270(6):1089-101 Kaplan DH, Shankaran V, Dighe AS, Stockert E, Aguet M, Old LJ, Schreiber RD (1998) Demonstration of an interferonγ-dependent tumor surveillance system in immunocompetent mice. Proc. Natl. Acad. Sci. USA, 95: 7556–7561 Kumar A, Commane M, Flickinger TW, Horvath CM, Stark GR (1997) Defective TNF-alpha-induced apoptosis in STAT1-null cells due to low constitutive levels of caspases. Science 278:1630–1632 Kupryjanczyk J, Thor AD, Beauchamp R, Merritt V, Edgerton SM, Bell DA, Yandell DW (1993) p53 gene mutations and protein accumulation in human ovarian cancer. Proc Natl Acad Sci U S A. 90(11):4961-5 Lei M (2005) The MCM complex: its role in DNA replication and implications for cancer therapy. Curr Cancer Drug Targets 5(5):365-80 Lengyel E, Schmalfeldt B, Konik E, Spathe K, Harting K, Fenn A, Berger U, Fridman R, Schmitt M, Prechtel D, Kuhn W (2001) Expression of latent matrix metalloproteinase 9 (MMP-9) predicts survival in advanced ovarian cancer. Gynecol Oncol 82(2):291-8 Levy DE, Darnell JE Jr (2002) Stats: transcriptional control and biological impact. Nat Rev Mol Cell Biol 3(9):651-62 Lillemeier BF, Koster M, Kerr IM (2001) STAT1 from the cell membrane to the DNA. EMBO J. 20(10):2508-17 Meraz MA, White JM, Sheehan KC, Bach EA, Rodig SJ, Dighe AS, Kaplan DH, Riley JK, Greenlund AC, Campbell D, Carver-Moore K, DuBois RN, Clark R, Aguet M, Schreiber RD (1996) Targeted disruption of the Stat1 gene in mice reveals unexpected physiologic specificity in the JAK-STAT signaling pathway. Cell, 84: 431–442 Meier W, Baumgartner L, Stieber P, Hashozner U, Fateh-Moghadam A (1997) CA125 based dianosis and therapy in recurrent ovarian cancer. Anticancer Res 17(4B):3019-20 Melen K, Kinnunen L, Julkunen I (2001) Arginine/lysine-rich structural element is involved in interferon-induced nuclear import of STATs. J Biol Chem. 276(19):16447-55 Meyer T, Rustin GJ (2000) Role of tumour markers in monitoring epithelial ovarian cancer. Br J Cancer 82: 1535-1538 Meyer T, Begitt A, Lodige I, van Rossum M, Vinkemeier U (2002) Constitutive and IFN-gamma-induced nuclear import of STAT1 proceed through independent pathways. EMBO J. 21(3):344-54 - 56 - Literaturangaben Newlon CS (1997) Putting it all together: building a prereplicative complex. Cell 91(6):717-20 Ozols RF (2005) Treatment goals in ovarian cancer. Int J Gynecol Cancer 15(Suppl. 1):3–11 Parsons JT, Parsons SJ (1997) Src family protein tyrosine kinases: cooperating with growth factor and adhesion signaling pathways. Current Opinion in Cell Biology 9:187-192 Prat J, Ribe A, Gallardo A (2005) Hereditary ovarian cancer. Hum Pathol 36(8):861-70 Ramana CV, Grammatikakis N, Chernov M, Nguyen H, Goh KC, Williams BR, Stark GR (2000) Regulation of c-myc expression by IFN-gamma through Stat1-dependent and -independent pathways. EMBO J 19: 263–72 Richter A, Knippers R (1997) High-molecular-mass complexes of human minichromosome- maintenance proteins in mitotic cells. Eur J Biochem 247(1):136-41 Risch HA, McLaughlin JR, Cole DE, Rosen B, Bradley L, Kwan E, Jack E, Vesprini DJ, Kuperstein G, Abrahamson JL, Fan I, Wong B, Narod SA (2001) Prevalence and penetrance of germline BRCA1 and BRCA2 mutations in a population series of 649 women with ovarian cancer. Am J Hum Genet. 68:700–710 Santillan A, Garg R, Zahurak ML, Gardner GJ, Giuntoli RL 2nd, Armstrong DK, Bristow RE (2005) Risk of epithelial ovarian cancer recurrence in patients with rising serum CA-125 levels within the normal range. J Clin Oncol 23(36):9338-43 Schindler C, Shuai K, Prezioso VR, Darnell JE Jr (1992) Interferon-dependent tyrosine phosphorylation of a latent cytoplasmic transcription factor. Science 257(5071):809-13 Shuai K, Horvath CM, Huang LH, Quresh SA, Cowburn D, Darnell JE Jr (1994) Interferon activation of the transcription factor Stat91 involves dimerization through SH2-phosphotyrosyl peptide interactions. Cell 76: 821–828 Stratton JF, Gayther SA, Russell P, Dearden J, Gore M, Blake P (1997) Contribution of BRCA1 mutations to ovarian cancer. N Engl J Med 336:1125–30 Snyder M, He W, Zhang JJ (2005) The DNA replication factor MCM5 is essential for Stat1-mediated transcriptional activation. Proc Natl Acad Sci U S A. 102(41):14539-44 Takeda K, Akira S (2000) STAT family of transcription factors in cytokine-mediated biological responses. Cytokine Growth Factor Rev 11:199–207 Tavassoli FA, Devilee P (2003) World Health Organization Classification of Tumours. Pathology and Genetics of Tumours of the Breast and female genital Organs. IARC Press, Lyon, pp. 114-116 - 57 - Literaturangaben Thomas M, Finnegan CE, Rogers KM, Purcell JW, Trimble A, Johnston PG, Boland MP (2004) STAT1: a modulator of chemotherapy-induced apoptosis. Cancer Res 64(22):8357-64 Townsend PA, Scarabelli TM, Davidson SM, Knight RA, Latchman DS, Stephanou A (2004) STAT-1 interacts with p53 to enhance DNA damage-induced apoptosis. J Biol Chem. 279(7):5811-20 Tye BK (1999) MCM proteins in DNA replication. Annu. Rev. Biochem 68, 649–686 Veikkola T, Karkkainen M, Claesson-Welsh L & Atlitalo K (2000) Regulation of angiogenesis via vascular endothelial growth factor receptors. Cancer Res. 60:203-212 Vinkemeier U, Cohen SL, Moarefi I, Chait BT, Kuriyan J, Darnell JE Jr (1996) DNA binding of in vitro activated Stat1α, Stat1β and truncated Stat1: interaction between NH2-terminal domains stabilizes binding of two dimers to tandem DNA sites. EMBO J 15: 5616–5626 Vinkemeier U (2004) Getting the message across, STAT! Design principles of a molecular signaling circuit. J Cell Biol. 167(2):197-201 Welcsh PL, Lee MK, Gonzalez-Hernandez RM, Black DJ, Mahadevappa M, Swisher EM, Warrington JA, King MC (2002) BRCA1 transcriptionally regulates genes involved in breast tumorigenesis. Proc Natl Acad Sci U S A. 99(11):7560-5 Wen Z, Zhong Z, Darnell JE Jr (1995) Maximal activation of transcription by Stat1 and Stat3 requires both tyrosine and serine phosphorylation. Cell 82 (2):241-50 Widschwendter A, Tonko-Geymayer S, Welte T, Daxenbichler G, Marth C, Doppler W (2002) Prognostic significance of signal transducer and activator of transcription 1 activation in breast cancer. Clin Cancer Res 8:3065–74 Wingo PA, Tong T, Bolden S (1995) Cancer statistics 1995. CA Cancer J Clin 45(1):8-30. Wu Y, Soslow RA, Marshall DS, Leitao M, Chen B (2004) Her-2/neu expression and amplification in early stage ovarian surface epithelial neoplasms. Gynecol Oncol. 95(3):570-5 Yang E, Henriksen MA, Schaefer O, Zakharova N, Darnell JE Jr (2002) Dissociation time from DNA determines transcriptional function in a STAT1 linker mutant. J Biol Chem 277(16):13455-62. Yu H, Jove R (2004) The stats of cancer--new molecular targets come of age. Nat Rev Cancer 4(2):97-105 Zhang JJ, Zhao Y, Chait BT, Lathem WW, Ritzi M, Knippers R, Darnell JE Jr (1998) Ser727-dependent recruitment of MCM5 by Stat1alpha in IFN-gamma- induced transcriptional activation. EMBO J 17(23):6963-71 Danksagung Danksagung Ich möchte zuerst Herrn Prof. Martin Werner für das interessante Thema und für die freundliche Übernahme meiner Doktorarbeit bei ihm im Pathologischen Institut und noch für die große Unterstützung beim Antrag an Dr. Heinrich Kircher-Stiftung danken. Weiterhin möchte ich mich bei Frau PD. Dr. Hasenburg dafür bedanken, dass sie mir den ersten Blick auf Operation auf Patientin mit Ovarialkarzinom in Deutschland gezeigt hat und sich als Zweitgutachter meiner Promotion zur Verfügung stellte. Mein ganz besonders Dank gilt Frau Dr. Silke Lassman für die Betreuung meines Aufenthalts in Freiburg und für die große Hilfebereitschaft während der gesamten Doktorarbeit insbesondere bei der Korrektur der Doktorarbeit. Sie ist mir immer ein gutes Vorbild gewesen. Die gemeinsame Arbeit im Labor hat mir viel Freude gemacht und auch viel Kenntnisse gelehrt. Bei Herrn Prof. Axel ZurHausen und Frau Dr. Ulrike Gerlach möchte ich mich für die Begutachtung dessen Archiv und der immuhistochemischen Analyse von Ovarkarzinomen bedanken. Bei Anja Schopflin, Jasmine Roth und Evylen Wätzig möchte ich für die ausgezeichnet Zusammenarbeiten und gute Betreuung während meinem Aufenthalt in Freiburg. Frau Lieselotte Bokle gilt mein Dank für ihre Hilfe bei der Durchführung der histochemischen Färbung. Alle anderen hier namelich nicht genannten Mitarbeiten im pathologischen Institut danke ich für die gute Zusammenarbeit. Ganz besonders bedanken möchte ich mich bei Herrn Rektor Prof. Guobing Wang und Herrn Prof. Zhongbi Wu in China für die große Unterstützung meinen Arbeiten in Freiburg. Sie haben mir die Möglichkeit diesen wissenschaftlichen Austausch gegeben und großen Vertrauen in meinen Arbeiten aufgezeigt. Den größten Dank möchte ich meiner Familie aussprechen. Sie haben mir ihre möglichst Riesenhilfe gegeben und waren immer für mich da gewesen, die Schwierigkeiten zu überwinden.