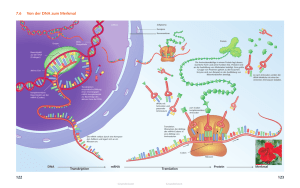
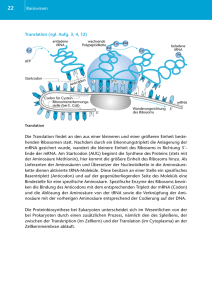
V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert [If you paid for this, you've been ripped off] In eigener Sache: Sollten Fehler egal welcher Art in der Ausarbeitung zu finden sein (davon geh ich mal aus, ich bin ein fauler Noob), bitte eine E-Mail an [email protected] schicken, damit ich's ausbessern kann wenn Zeit ist und die Anfrage sinnvoll klingt. Ähm, in der E-Mail sollt natürlich drin stehen wo was falsch ist … Es wäre vielleicht noch zu sagen, dass man die hier eingefügten Bilder nur zum Privatgebrauch verwenden sollte, weil oft keine Quelle angegeben ist und manche Herausgeber das angeblich übel nehmen. Texte aus den Folien zur Vorlesung sind durch eine andere Schriftfarbe gekennzeichnet, damit man weis welchen Textteilen man eher misstrauen sollte. Die Datei ist aus dem Grund schreibgeschützt, damit niemand den Kopf austauscht, die Mitschrift kopiert und verkauft. Die Mitschrift soll für alle interessierten Personen gratis zu haben sein – oder maximal eine gebrannte CD kosten. Weiters ist noch zu sagen, dass ich in dieser meiner Mitschrift, eine weitere Mitschrift vor allem für den Teil Bläsi verwendet hab (weil ich seine Vorlesung nicht durch'druckt hab…), es finden sich eine ganze Menge Paraphrasen davon hier drin. Allerdings weis ich leider nicht von wem die Mitschrift ist, ich kann daher weder um Erlaubnis fragen noch Credits geben. Sorry, bitte nicht bös sein... Aus den Bläsi Foliensätzen hab ich ein paar Bilder ausgelassen und durch Text ersetzt, weil sie meiner Meinung nach nicht nötig sind. So und jetzt genug mit dem unnötigen Gelaber. Mitschrift zur Vorlesung "Genexpression" von Decker, Bläsi Inhaltsverzeichnis: Inhaltsverzeichnis: .................................................................................................... 1 Vorlesung Genexpression ........................................................................................... 3 Teil Decker: Transkriptionskontrolle ............................................................................. 3 Genexpression ....................................................................................................... 3 Proofreading durch RNA Polymerase ....................................................................... 8 Termination der Transkription................................................................................ 9 Der Promotor .................................................................................................... 10 DNAse I footprinting: ......................................................................................... 13 Systematische Analyse der Transkriptionskontrolle eines durch RNA Pol II transkribierten, eukaryoten Gens .................................................................................................. 26 1. Welche Gene werden durch Stimuli aus der zellulären Umgebung reguliert?............ 26 2. Liegt Transkriptionskontrolle vor? ..................................................................... 29 3. Isolieren des Promoters .................................................................................. 29 4. Wo binden wichtige Transkriptionsfaktoren?....................................................... 34 5. Welche Transkriptionsfaktoren sind an der Regulation beteiligt? ............................ 35 6. Struktur-Funktionsbeziehung im isolierten Transkriptionsfaktor (wo sind funktionelle Domänen?) ....................................................................................................... 37 7. Interaktion des Transkriptionsfaktors mit anderen Proteinen ................................ 38 Transkriptionsfaktoren........................................................................................ 39 Die Initiation der Transkription ............................................................................... 42 RNA Polymerase II Holoenzyme ........................................................................... 45 Bildung einer mRNA CAP Struktur ........................................................................ 47 mRNA Termination und Polyadenylierung .............................................................. 47 1 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Genregulation durch DNA Methylierung.................................................................... 68 Mechanismen zur Regulation der Transkriptionsfaktor Aktivität ................................... 71 Genregulation durch cAMP .................................................................................. 76 Als abschließendes Beispiel Transkriptionskontrolle über Konzentrationsverteilung: ..... 84 Teil Bläsi: Translationskontrolle ................................................................................. 85 transfer RNA is the adapter ................................................................................. 87 tRNA processing ................................................................................................ 88 tRNA charging ................................................................................................... 89 Experiment: Strukturelle Kartierung der 16S tRNA durch Chemical probing. ............... 95 Correct translation initiation requires initiation factors ............................................100 Experiment: Bindungsort von IF1 mittels Chemical probing ermitteln .......................100 Experiment: Reportergen Assay zur Autoregulation von IF3 ....................................102 Toeprinting assay .............................................................................................103 Experiment: Aktivitätsmessung von IF3 ...............................................................103 Experiment: tRNA charging – welches Codon für welche AS ....................................105 Die Shine Dalgarno Sequenz...............................................................................107 Experiment: Biochemischer Nachweis der SD-Anti-SD-Interaktion durch footprinting..108 Experiment: Genetischer Nachweis der SD-Anti-SD-Interaktion ...............................108 Experiment: Biochemischer Nachweis der SD-Anti-SD-Interaktion durch chemical probing ......................................................................................................................109 Parameters affecting translation initiation of a canonical prokaryotic mRNA ...............110 Experiment: Isolierung von mRNA-Fragmenten die S1 binden und deren Sequenzierung ......................................................................................................................111 Experiment: Klärung ob S1 Primär oder Sekundärstrukturen der mRNA bindet ..........111 The downstream box .........................................................................................120 Autoregulation der Translation durch Protein S15 ..................................................122 Experiment: Repressorwirkung des Proteins S15 bei Translation ..............................123 Experiment: Ermittlung der hfq Chaperonaktivität mittels Toeprinting ......................130 Experiment: Spaltung des SodB-RhyB RNA Komplexes durch RNase E......................133 Leaderless mRNA ................................................................................................138 Experiment: Competition Assay zur leaderless mRNA .............................................140 Incorporation of unusual amino acids: the 21st and 22nd amino acids ......................149 Programmed frameshifting .................................................................................152 Experiment: Test auf +1 frame shift:...................................................................152 Experiment: Prinzipeller Test auf Frameshift: ........................................................153 Experiment: Test auf Translationale Introns..........................................................155 Abbau von mRNA ................................................................................................157 Experiment: Nachweis einer bestimmten mRNA in einer Zelle .................................158 Experiment: Nachweis von welchem Ende her mRNA abgebaut wird.........................159 Experiment: Test auf Zusammenhang zwischen effizienter Translation und mRNA Stabilität .........................................................................................................165 Regulation of Gene Expression in Eukaryotes...........................................................168 mRNA processing: Nuclear Splicing .....................................................................168 Group I introns: ...............................................................................................173 3'-end formation...............................................................................................175 Die CAP Struktur ..............................................................................................177 Toeprinting bei Eukaryoten: ...............................................................................181 Experiment: Nachweis interner Initiation durch IRES..............................................183 Eukaryotic translational elongation and termination: ..............................................187 mRNA stability in Eukaryotes ..............................................................................188 Translation in Archaea..........................................................................................198 Experiment: Nachweis, dass Leadersequenz den Transport durch die Membran induziert. ......................................................................................................................205 Experiment: Prinzipielle Funktion des Sec Pathways...............................................208 Import into and export from the nucleus ..............................................................213 Prüfungsfragen ......................................................................................................215 2 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Decker Foliensatz 1 07.03.2006 Vorlesung Genexpression Teil Decker: Transkriptionskontrolle Thomas Decker, Institut f. Mikrobiologie und Genetik Literatur: • Lewin, B.; Genes VIII; Pearson/Prentice Hall • Lodishet al.; Molecular Cell Biology, 4th edition; Freeman • Watson et al.; Molecular Biology of the cell, 4th edition; Garland Science • Watson, J. et al.; Molecular Biology of the gene, fifth edition; Pearson Genexpression All diejenigen Schritte, welche dazu führen, dass der DNA-Code in eine definierte Menge und Aktivität eines Genprodukts (RNA, Protein) übertragen wird. Die Expression von Genen ist über mehrere Ebenen steuerbar. • • • • • • Transkriptionskontrolle 30-35.000 Gene codieren in menschlicher DANN für Proteine. Die Gene werden unterschiedlich stark transkribiert. Housekeeping genes sind wichtige Gene z.B. für Metabolismus oder Zellwachstum, die ständig transkribiert werden. Demgegenüber werden die meisten Gene situationsbedingt transkribiert, z.B. sind Gene der Embryonalentwicklung im adulten Status nicht mehr nötig und werden stillgelegt. Situationsspezifischer Einsatz von Genen Vermeidet Koordinationsschwierigkeiten im Zellinneren und dient der Energieersparnis. Transkriptprozessierung (Splicing, Transport) In Eukaryoten ist Modifizierung (Splicing, PolyA-Schwanz, CAP) und Export der mRNA aus dem Zellkern nötig. Transkriptstabilität Die Stabilität des Transkripts im Cytoplasma ist von mehreren Faktoren abhängig. Translationskontrolle Post-translationale Modifikation (Aktivität des Proteins) Ein Protein kann je nach Verwendungszweck direkt nach der Translation aktiv sein oder durch spätere Veränderung aktiv werden. Proteinstabilität Proteine selbst können stabil oder instabil sein. 3 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Wie 'wachsen' Nukleinsäuren und Polypeptide während ihrer Synthese? Transkription: Der 'template' (=nichtkodierende) Strang der DNA eines Gens wird in 3'-5' Richtung abgelesen um eine 5'-3' mRNA Synthese durchzuführen. Die Polarität und Sequenz der mRNA entspricht der des kodierenden DNA Strangs. Transkription chemisch: sowohl DNA als auch RNA können durch chemische Gegebenheiten nur in 5'-3' Richtung synthetisiert werden. Das 3' OH einer Nucleinsäurekette wird durch einen enzymatisch (Polymerase) dirigierten nucleophilen Angriff unter Abspaltung eines PPi mit dem 5' Ende eines neuen Nucleotids verknüpft. Syntheseraten von Nukleinsäuren und Proteinen • • • Transkription (E. coli): ca. 40 Nucleotide/sec bei 37°C. Translation (E. coli): ca 15 Aminosäuren/sec bei 37°C. DNA Replikation (E. coli): 50.000bp/min; Eukaryoten ca. 2000bp/min. 1AS = 1 Codon = 3 Basen RNA Synthese ist prinzipiell langsamer als die Replikation von DNA. Prokaryoten weisen im Vergleich zu Eukaryoten keinen Nucleus auf. Dadurch kann die mRNA bereits während der fortlaufenden Transkription translatiert werden. Die ersten Schritte der mRNA Synthese: (1) RNA Polymerase muss zunächst am DNA Strang aus der Masse nichtkodierender Basenpaare den Beginn eines Gens finden und an dieser Stelle mit der DNA interagieren. (2) Anschließend ist Trennung der beiden DNA Stränge nötig, um am nichtkodierenden Strang eine mRNA synthetisieren zu können. (3) Das erste synthetisierte Basenpaar ist zumeist eine CG Paarung. (5) Bereits bei der Bindung des dritten Nucleotids an die mRNA muss die RNA Polymerase beginnen, sich am nichtkodierenden DNA Strang entlang zu bewegen und die DNA weiter voneinander zu trennen. 4 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Transkription lässt sich in distincte Phasen gliedern: Initiation … Vorgang von Erkennung und Bindung der RNA Pol an die DNA bis zur Synthese erster Nucleotide ohne Fortbewegung. Elongation … RNA Pol bewegt sich an der DNA entlang und fügt der mRNA Nucleotide hinzu. Termination … spezielle Signale bewirken den Stopp der Transkription und die Lösung der RNA Pol von der DNA. Der beginn der Initiation lässt sich thermodynamisch in einzelne Schritte einteilen: (1) Bildung eines "geschlossenen binären Komplexes" aus ungetrennter DNA und RNA Polymerase. Die Konstante KB beschreibt, wie schnell der Komplex an die DNA bindet. Die Affinität zwischen dem Promotor eines Gens und der RNA Polymerase kann um bis zu einem Faktor 1000 schwanken (106 bis 109 M-1). Die Rückreaktionskonstante ist sehr klein, daher läuft der nächste Schritt sehr schnell ab. (2) Bildung eines "offenen binären Komplexes" in dem die DNA im Bereich des Transkriptionsstarts aufgetrennt wird. Nach dem Aufschmelzen der DNA ist die Ablösung der RNA Polymerase von der DNA von alleine biochemisch praktisch nicht mehr möglich. (3) Bildung eines "ternären Komplexes" (abortive Initiation) indem das erste RNA Nucleotid komplementär zur DNA gebunden wird, dieser Schritt dauert relativ lange. (4) Die Freigabe des Promotors (Promotor clearance) dauert lange, da es sich um einen biochemisch aufwendigen Prozess handelt. Die Anatomie der E. coli RNA Polymerase Das aus den β (rpoB Gen in E.coli), β' (rpoC) und 2α (rpoA) Untereinheiten bestehende 'core' Enzym wird durch Assoziation mit der σUntereinheit (=σFaktor, rpoG) zum Holoenzym. Ein Komplex aus den 2α, β und β' Untereinheiten wird als core enzyme, mit weiteren UE inklusive σ als Holoenzym (holos = ganz) bezeichnet. 5 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Die bakterielle RNA Polymerase RNA Polymerase muss einen Kanal zur Führung der DNA, einen Kanal zu Aufnahme von Nucleotiden, einen weiteren als Ausgang für die synthetisierte mRNA sowie ein aktives Zentrum besitzen, um Nucleotide an der DNA zu mRNA zu synthetisieren. Der Bereich einer DNA, der die Signale und Information zur Synthese eines vollständigen Transkripts enthält, wird als 'transcription unit' bezeichnet Die transcription unit besteht aus einer Startregion über die die RNA Polymerase an die DNA binden kann (Promotor), folgende Sequenzen die für ein (monocistronisch) oder mehrere Gene codieren (polycistronisch), sowie eine Sequenz die der Termination der Transkription dient (Terminator). Als Initiationsstelle wird das Nucleotid bezeichnet, an dem die RNA Polymerase das erste Nucleotid der mRNA bindet. Alles vor der Initiationsstelle, also in Richtung des 5' Endes des nichtkodierenden DNA Stranges, wird als upstream, alles danach, also Richtung des 3' Endes, als downstream bezeichnet. Unterschiede zwischen prokaryotischer und eukaryotischer mRNA: Prokaryotische mRNA kann polycistronisch sein, eukaryotische mRNA ist immer monocistronisch, codiert also immer nur für ein Gen. Die mRNA prokaryotische Gene ist fast immer durchgehend und ermöglicht es, dass Transkription und Translation parallel ablaufen kann. Demgegenüber sind die codierenden Bereiche eukaryotische mRNA (Exons) direkt nach der Transkription noch durch nichtkodierende Bereiche (Introns) getrennt, die mittels Splicing erst entfernt werden müssen um eine kontinuierliche RNA Sequenz für die Translation zu erhalten. 6 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Unterschiedliche Veränderung der DNA Topologie vor und hinter der transkribierenden RNA Polymerase durch die Entwindung der DNA innerhalb der 'transcription bubble': positive und negative supercoils DNA liegt innerhalb der RNA Polymerase in einem Bereich von 25 bp geöffnet vor, die Polymerase überdeckt vor und nach der geöffneten Stelle einen jeweils 10bp langen weiteren Bereich der DNA. In entspanntem Zustand liegt DNA als Doppelhelix mit etwa 10bp pro helikaler Windung vor. Im Zuge der RNA Synthese wird die DNA geöffnet. Dadurch und durch die Bewegung der RNA Polymerase entlang der DNA wird die Windungszahl vor der Polymerase verkleinert (mehr bp pro Windung), während sie hinter der Polymerase größer wird. Im Zuge dieser Spannungen entstehen vor der Öffnungsstelle negative und danach positive Supercoils. Um diese Supercoils aufzulösen ist Energie notwendig, die die RNA Polymerase nicht aufbringen kann. Sie werden daher durch eigene Enzyme, Topoisomerasen bzw. DNA Gyrasen, wieder aufgelöst. 7 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Funktionsweise der Typ I und Typ II Topoisomerasen: Topoisomerase I löst negative Supercoils auf. Sie schneidet einen Strang der DNA (Nick) und läßt entweder den zweiten rotieren, bis die Spannung nachlässt oder zieht den intakten DNA Strang durch den Nick. Anschließend wird der geschnittene Strang wieder ligiert. Topoisomerase II schneidet eine DNA Doppelhelix, führt einen anderen Teil der DNA durch die Öffnung und ligiert die geschnittene DNA Doppelhelix um positive Supercoils aufzulösen. Proofreading durch RNA Polymerase Proofreading dient der Erkennung fehlerhafter Paarung der mRNA Nucleotide mit der DNA. • Pyrophosphorolytic Editing: Das letzte Nukleotid der wachsenden RNA wird durch Pyrophosphat ersetzt. Je länger die Polymerase sich an einer Stelle befindet, desto höher wird die Wahrscheinlichkeit, dass das letzte Nucleotid ausgetauscht wird. Im Vergleich verweilt die Polymerase länger an einem fehlerhaft gepaarten Nucleotid als an einem korrekt gepaarten, die Polymerase unterscheidet dabei aber nicht aktiv zwischen fehlerhafter oder korrekter Basenpaarung. • Hydrolytic Editing: RNA Pol bewegt sich einige Nucleotide rückwärts und entfernt das Stück RNA mit der Fehlpaarung. RNA Viren besitzen eine eigene RNA Polymerase ohne Proofreading. Sie weisen dadurch eine hohe Mutationsrate auf, die es den Viren ermöglichen durch Mutation dem Immunsystem zu entgehen. 8 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Termination der Transkription Termination der Transkription: rho-unabhängige Terminatoren bilden stem-loop Strukturen, gefolgt von einer poly-U Sequenz. In Prokaryoten finden sich zwei Möglichkeiten der Termination. Bei der rho-unabhängigen Termination bildet sich in der mRNA eine so genannte Hairpin Struktur, bei der ein Teil der mRNA mit sich selbst Basenpaarungen ausbildet, die eine Reihe der stabileren CG-Paare enthält. Terminationsvorgang: Direkt im Anschluss an die Hairpin Sequenz wird eine Poly-U Sequenz synthetisiert, die nur schlecht an der DNA bindet. Durch die nun folgende Bildung der stabilen Hairpin Struktur werden die dynamisch instabileren AU Bindungen zwischen DNA und RNA aufgelöst und die mRNA von der DNA abgetrennt. Rho-abhängige Termination der Transkription: Die Rho-abhängige Termination benötigt einen zusätzlichen Faktor. Es handelt sich dabei um einen Proteinkomplex, der eine bestimmte Sequenz der mRNA binden kann. Im Anschluss an die Bindung bewegt es sich an unter ATP Hydrolyse an der mRNA entlang. Am Ende der mRNA wird eine Sequenz transkribiert, die mit sich selbst binden kann und dadurch eine weitere Transkription durch die RNA Polymerase stoppt. Erreicht der Rho-Faktor die RNA Polymerase und findet die Terminationssequenz in der mRNA vor, löst es unter ATP Hydrolyse die mRNA von der DNA ab, zieht sie aus der RNA Polymerase heraus und setzt sie frei. 9 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Der Phage λ benützt Proteine mit antiterminierender Wirkung (N, Q) als Schalter für die frühen und späten Phasen der Genexpression. Genexpression lässt sich auf allen Ebenen regulieren. Für den Lebenszyklus von Phagen ist es essentiell, unterschiedliche Produkte zur passenden Zeit herzustellen. Vollständige Phagen dürfen sich erst dann bilden können, wenn alle Teile des Phagen bereits in ausreichender Menge produziert wurden. Der Phage λ kann mittels spezieller Proteine die Expression seiner Gene auf Ebene der transkriptiven Termination durch so genannte Antitermination steuern. Zunächst wird nur zu so genannten "early genes" des Phagen mRNA erzeugt und anschließend die Transkription abgebrochen. Ein "early gene" hat die Fähigkeit, RNA Polymerase dazu zu bringen Terminationssignal zu überlesen und die Transkription fortzuführen, um die nachfolgend liegenden "late genes" zu transkribieren. Erst wenn dieses Protein in ausreichender Konzentration vorliegt, sind ausreichend viele RNA Polymerasen in der Lage, auch die "late genes" zu transkribieren. Der Promotor • • definiert den Transkriptionsstart eines Gens. enthält DNA Sequenzen (Elemente), welche die Rekrutierung und Bindung der RNA Polymerase an den Transkriptionsstart bewirken. Diese bilden meist Bindungsstellen für Transkriptionsfaktoren. 10 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Wie findet RNA Polymerase einen Promoter? Unterschiedliche Modelle: Würde RNA Polymerase vollständig frei vorliegen und durch freie Diffusion zu den Genen finden, bräuchte man eine zu hohe Konzentration an Polymerasen um ausreichende Transkription sicher zu stellen. - 500-1000 RNA Polymerasen liegen als Core enzyme lose mit DNA assoziiert vor. - 500-1000 RNA Polymerasen liegen als Holoenzym mit der DNA assoziiert vor, das durch den σ-Faktor in der Lage ist, nach Promotoren zu suchen. - Weitere 500-1000 Polymerasen liegen bereits als geschlossene oder offene Komplexe an einem Promotor. - Abschließend finden sich etwa 2500 Core enzyme die Gene transkribieren. - Im Vergleich damit liegt nur eine kleine Anzahl an Polymerasen nicht mit DNA assoziiert vor. Die Bewegung der RNA Polymerase auf der DNA kann durch zwei Modelle beschrieben werden. 1) Die Polymerase diffundiert nach ihrer Synthese nur so lange, bis sie einmal an DNA bindet und wandert solange an ihr entlang, bis sie einen Promotor findet. 2) Die Polymerase diffundiert nach ihrer Synthese nur so lange, bis sie einmal an DNA bindet. Anschließend bleibt sie lose mit der DNA assoziiert und bewegt sich durch binden und lösen von einem DNA Stück zum nächsten bis es einen Promotor findet. Dieses Modell wurde durch experimentelle Daten bestätigt. 11 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Die Assoziation mit σ-Faktor steuert die Fähigkeit von RNA Polymerase, Promoter DNA von anderer DNA zu unterscheiden. Der σ-Faktor erhöht die Affinität einer RNA Polymerase zu spezifischen Promotorsequenzen. Das Core enzym ist in der Lage mit niedriger Affinität d.h. langsam an DNA zu binden. Bindet ein σ-Faktor an das Core enzyme, so wird die Affinität dieses Komplexes für Promotoren erhöht und für zufällige DNA Sequenzen erniedrigt. Der σ-Faktor ist für die Transkription nicht nötig und wird im Zuge der Initiation vom Core enzyme freigesetzt. Der σ-Faktor selbst bewegt sich in freiem Zustand wie RNA Polymerase an DNA entlang, bis es auf ein Core enzym trifft, das nicht transkribiert. Der σ-Faktor erkennt den Promotor eines Gens über bestimmte Sequenzen auf der DNA. Diese Sequenzen werden als Consensussequenzen bezeichnet und finden in Bezug auf die Initiationsstelle bei -10 und -35. Die Anatomie bakterieller Promotoren: Konsensus Sequenzen und Mutationen definieren die Bedeutung der -10 (TAATA) und -35 Bereiche: Durch das TATAAT Motiv bei -10 wird die Consensussequenz auch als TATA-Box bezeichnet. Bei Mutation in diesen Sequenzen findet sich eine veränderte Bindungsaffinität der RNA Polymerase zu den Promotoren. Down Mutation … Verschlechterung der Bindung an den Promotor. Up Mutation … Verbesserung der Bindung an den Promotor. Positionierung des RNA Polymerase Holoenzyms bei der Initiation der Transkription: 12 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Strukturelle Domänen des σ70 Faktors von E.coli Es finden sich mehrere σ-Faktoren, die sich unter anderem durch ihr Molekulargewicht unterscheiden. σ70 weist beispielsweise ein Gewicht von 70kDa auf. Die einzelnen σ-Faktoren ermöglichen es der RNAPolymerase, an Promotoren mit sich unterscheidenden Consensussequenzen zu binden. Je nachdem welche σ-Faktoren vorliegen, können so situationsabhängig unterschiedliche Gene transkribiert werden. σ70 ist der Faktor, der für die Sicherstellung des normalen Stoffwechsels zuständig ist. 14.03.2006 DNAse I footprinting: Ein Verfahren zur Bestimmung von Proteinbindungsstellen auf der DNA: Um zu prüfen, ob ein Protein eine bestimmte DNA Sequenz bindet, wird DNase I Footprintig eingesetzt. DNase I ist ein Enzym, das DNA sequenzunabhängig schneidet. DNA die eine vermutete Proteinbindungsstelle enthält wird vermehrt, mittels eines Enzyms wird am 5' Ende ein radioaktiv markiertes Phosphat angehängt. Anschließend wird die vermehrte DNA mit DNase I in einem berechneten Verhältnis gemischt, so dass jedes DNA Stück genau einmal an einer zufälligen Stelle von DNase I geschnitten wird. Dadurch erhält man unterschiedlich lange Stücke DNA, die sich über ein Gel der Größe nach auftrennen und durch die radioaktive Markierung sichtbar machen lassen. Die DNA Fragmente lassen sich sehr genau auftrennen, jede Bande ist um genau ein nt kürzer als die vorherige. Fügt man das vermutete Bindeprotein hinzu, deckt das Protein seine Bindesequenz auf der DNA ab, DNase kann an dieser Stelle nicht schneiden. Die Gelelektrophorese zeigt im Bereich der Bindesequenz keine Banden und erlaubt so eine Ermittlung derselben. 13 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Kontaktpunkte der RNA Polymerase mit Promoter-DNA werden durch footprinting Verfahren und durch Punktmutationen bestimmt: graue Pfeile … werden diese Stellen verändert, kann RNA-Pol nicht mehr binden. rote Pfeile … Punktmutation hier ändert Bindungsverhalten von Pol an Promotor. Mittels footprinting wurde festgestellt, dass die Polymerase über den σ-Faktor nur an den -35 und -10 Sequenzen des Promotors wirklich fest gebunden ist. Spezifische σ-Faktoren steuern die koordinierte Expression definierter Gengruppen: Wie bereits erwähnt binden unterschiedliche σ-Faktoren jeweils verschiedene Promotorconsensussequenzen, um Gengruppen ereignisgesteuert transkribieren zu können. Unter stationärer Phase versteht man einen Zustand in dem dem Bakterium keine Nahrungsquellen zur Verfügung stehen und der Stoffwechsel bis auf für das Überleben essentielle Gene stillgelegt wird. Phagenkodierte σ-Faktoren steuern das Umschalten von früher auf späte Genexpression beim B. subtilis Phagen SPO1: Für die Transkription der "early genes" dient der σ70-faktor des Wirtsbakteriums. Ein Produkt der "early genes" ist ein phageneigener σ-Faktor. Erst wenn dieser σ-Faktor in ausreichender Konzentration vorliegt, können die "middle genes" transkribiert werden, die einen weitern σFaktor für die "late genes" erzeugen. 14 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Starke Promotoren haben ein zusätzliches UP-Element, welches mit der α-UE der RNA Pol interagiert Man unterscheidet Promotoren je nachdem, wie effektiv sie die Transkription "ihres" Gens ermöglichen. Starke Promotoren weisen zusätzlich zur -35 und -10 Sequenz eine weitere Sequenz auf (UP Element), die mit der C-terminalen Domäne (CTD) der α UE der RNA Polymerase interagiert und dadurch die Bindungswahrscheinlichkeit der Polymerase an den Promotor erhöht. Transkriptionskontrolle in Bakterien: das Operon • • • Ist eine für Prokaryoten typische Anordnung von Genen. Bezeichnet eine Gruppe von Genen, die in direkter Abfolge auf der DNA angeordnet sind. Die im Operon kodierten Gene werden gemeinsam reguliert und in Form einer gemeinsamen, polycistronischen mRNA transkribiert. Operon … physische Abfolge von einander ergänzenden Genen, wodurch eine gemeinsame Regulation ermöglicht wird. Prinzipien der Transkriptionskontrolle: positive und negative Regulation Negative Genregulation … Durch einen starken Promotor werden Gene im Normalzustand transkribiert. Erst die Bindung eines Repressorproteins an den Promotor verhindert eine weitere Transkription, solange das Protein gebunden bleibt. Positive Genregulation … Durch einen schwachen Promotor werden Gene im Normalzustand nicht von RNA Polymerase erkannt. Die Transkription wird erst durch das Binden eines Aktivatorproteins an den Promotor ermöglicht. "gene off by default" "gene turned-on by activator" 15 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Die Lac, Gal und Ara Operonen kodieren für Enzyme, welche Lac, Gal und Ara in die Glykolyse einschleusen Operons zur Erschließung weiterer Zuckerquellen. Das Gal-Operon ermöglicht die einzelnen Schritte zur Erschließung von Glukose aus Galaktose. Das Lac-Operon benötigt den GalOperator. Das Ara-Operon ermöglicht es Pyruvat aus dem 5er Zuckern Arabinose herzustellen. Regulation der Lac mRNA Synthese durch Zugabe oder Entzug des Inducers Lactose: E.coli bevorzugt Glucose als Zuckerquelle, da sie ohne Energieaufwand der Glykolyse zugeführt werden kann. Steht keine Glucose zu Verfügung, können andere Zuckerquellen wie Lactose, Galactose oder Arabinose erschlossen werden, deren Operons aber nur aktiviert werden, wenn Glucose nicht, aber die entsprechenden Zucker vorhanden sind. Lactose ist ein Heterodimer aus Galaktose und Glucose, ß-Galactosidase spaltet Lactose in die beiden Zuckereinheiten. Mittels weiterer Enzyme des Gal-Operons kann Galaktose in Glucose umgewandelt werden. Betrachtet man das Lac Operon, so wird ohne Lactosequelle nur eine sehr geringe Konzentration an Lac mRNA transkribiert (basaler Wert). Als Aktivator der Transkription dient Lactose selbst. Wird dementsprechend eine Lactosequelle erschlossen, steigt die Konzentration der Lac mRNA und der dadurch translatierten Galaktosidase innerhalb von Minuten stark an, steht Lactose nicht mehr zur Verfügung, sinkt die Lac mRNA Konzentration schnell wieder auf das basale Level ab, während die Galaktosidase über einen längeren Zeitraum stabil bleibt. 16 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Die Struktur des Lac Operons Das Lac Operon besteht aus mehreren Promotorsequenzen sowie den drei Genen lacZ, lacY und lacA die für die Genprodukte ß-Galaktosidase, Permease und Transacetylase codieren. ß-Galaktosidase … dient der Spaltung von Lactose in Galaktose und Glucose Permease … dient der Aufnahme von Lactose in die Zelle Transacetylase … modifiziert Lactose, um deren Diffusion aus der Zelle zu verhindern. Der Promotor des Lac Operons weist eine Sequenz auf, die als Operator bezeichnet wird. Operatoren sind Promotorsequenzen in Prokaryoten die vor allem der Bindung von Repressorproteinen dienen. Die Sequenz des Lac Operators liest sich von beiden Seiten her gleich (rotationssymmetrisch palindromisch) und bindet über die beiden identischen, seitenverkehrten Sequenzen ein homotetrameres Protein d.h. ein Protein das aus vier identischen, aneinander gebundenen UE besteht. Lactose wirkt als Inducer, welcher das Repressor-Tetramer in eine nicht an die Operator-DNA bindende Konformation überführt (allosterischer Effekt) Dieses Protein dient an den Operator gebunden als Repressor der Transkription. Es können zwar sowohl RNA Polymerase als auch Repressor gleichzeitig an ihre jeweiligen Promotorsequenzen binden, aber die RNA Polymerase ist dann nicht in der Lage, die DNA aufzutrennen. Die Untereinheiten des Repressortetramers werden durch das Gen lacI erzeugt, das sich nicht im Lac Operon befindet. Die einzelnen UE lagern sich zum Tetramer zusammen und können anschließend den Operator des Lac Operons binden. Lactose ist in der Lage, an die einzelnen UE des Repressors zu binden. Durch die Bindung der Lactose ändert sich die Konformation des Repressorkomplexes (allosterischer Effekt), er ist nicht mehr in der Lage an DNA zu binden. 17 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Der Lac Repressor • • • • Die wesentliche Bindungsstelle für lac Repressor, Operator 1 (O1), überlappt mit der Bindungsstelle für RNA-Polymerase. Die Abbildungen zeigen die Position der Repressorbindungsstelle O1. Es gibt zwei weitere Bindungsstellen, O2 (+410) und O3 (-83). O1, O2 und O3 binden jeweils ein RepressorTetramer. Maximale Repression entsteht dann, wenn ein Repressor Tetramer gleichzeitig an O1 und entweder O2 oder O3 bindet. Der lac-Repressor verhindert nicht die Bindung von RNA-Polymerase, er verstärkt sie sogar. Der lac Repressor verhindert die Trennung der Promoter DNA-Stränge, damit die Initiation der Transkription. Änderung der lac-Repressorstruktur durch Lactose: Das Repressorprotein bindet die Furchen der DNA über eine α-helicale Bindungsdomäne mit Helix-Loop-Helix Struktur. Bei Bindung von Lactose verändert sich die Konformation des Repressorkomplexes, die Helices können räumlich nicht mehr in den Furchen der DNA binden. Positive Kontrolle der Ara, Gal und Lac Operonen durch das catabolite activator protein (CAP oder CRP) Der Promotor der Ara, Gal und Lac Operons weist Bindungssequenzen für die Bindung eines Aktivatorproteins, des CAP (catabolite activator protein), auf. Das CAP dient der positiven Kontrolle des Lac Operons und steigert dessen Transkription, wenn Lactose vorhanden und der Repressor bereits durch Lactose inhibiert ist. CAP liegt immer vor, wird aber nur dann aktiv und bindet an die CAP binding sites, wenn die Zelle hungert und weitere Zuckerquellen als Nahrung erschließen muss. Als Aktivator für CAP dient das Molekül cAMP. 18 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Das Catabolite Activator Protein vermittelt Genregulation durch das 'Hungersignal' cAMP. Es ist vermutlich mit verantwortlich für das Phänomen der 'glucose repression' von Operonen. Es gibt ein Sensorsystem für den aktuellen Nahrungszustand der Zelle. cycloAMP ist indirekt proportional zur ATP Konzentration und dient so als Indikator für Hunger. CAP selbst wird nur dann aktiv, wenn es cAMP gebunden hat. Regulation des Lac Operons durch Glucose und Lactose: Drei Regulationszustände des Lac Operons: Liegen Glucose und Lactose vor, wird das Lac Operon nicht benötigt, kann aber transkribiert werden. Liegt nur Glucose vor, kann das Lac Operon wegen des Repressors nicht transkribiert werden. Liegt nur Glucose vor, kann das Lac Operon aktiviert werden, die Transkription wird durch cAMP weiter begünstigt. Das CAP Protein führt eine Biegung in die Promoter-DNA ein und erleichtert die Interaktion mit der α-UE der RNA Pol: (1) Bindet in Gruben der DNA und bewirkt deren Krümmung. Die Krümmung dient dazu, dass (2) die C-terminale Domäne der α UE der Polymerase in räumliche Nähe des CAP kommt und mit ihm binden kann, wodurch die Gesamtbindung des RNA Polymerasekomplexes an die DNA stabiler und die Transkriptionswahrscheinlichkeit erhöht wird. 19 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Expression des Lac Operons: Glucose + + - Lactose + + Transkription +/+ ++++ Importante: Es findet keine volle Expression statt, wenn Glucose vorhanden ist. Das CAP Protein stabilisiert die DNA-Schlaufe, die sich durch Bindung des Lac Repressors an die beiden Operatoren O1 und O3 bildet. Deshalb ist die Repression durch Lac Repressor in Abwesenheit von Glucose besonders effizient. Der Lac Repressor hat drei verschiedene Operatoren, die sich in ihrer Bindungsstärke unterscheiden. Ist weder Lactose noch Glucose vorhanden, ermöglicht die Bindung des CAP eine effizientere Bindung des Repressors an zwei Operatoren. Durch die DNA-Schlaufe des CAP nähern sich die Repressorprotein an und binden noch effizienter an die Furchen der DNA. Unterschiedliche Wege der positiven und negativen Transkriptionskontrolle durch Inducer- und Korepressor-Moleküle Inducer … Inaktiviert einen Repressor oder aktiviert einen Aktivator. Corepressor … Inaktiviert einen Aktivator oder aktiviert einen Repressor. 20 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Regulation des Trp Operons durch den Ko-Repressor Tryptophan Enzymsynthese des Tryptophanoperons Ist ausreichend Tryptophan vorhanden, dann bindet es an den Repressor, wodurch dieser aktiv wird und weitere Transkription des Trp Operons verhindert. Trp dient damit als Korepressor der eigenen Synthese. Es handelt sich hier um negative Kontrolle. Regulation des Arabinose Operons durch das AraC Protein Repressive loop Das Ara Operon weist zwei voneinander entfernte Aktivatoren auf. - ist Arabinose vorhanden, wirkt es selbst als Inducer des dimeren araC Proteins, das die Bindung der RNA Polymerase unterstützt. - ist Arabinose nicht vorhanden, für das dimere AraC Protein durch Bindung an entfernten Sequenzen der DNA eine Schleifen ein, die eine Bindung von RNA Polymerase verhindert. Das phoR-phoB zwei-Komponentensystem reguliert Gene im Falle einer Erniedrigung der Konzentration an anorganischem Phosphat Der transmembrane Rezeptor phoR dient der Bindung von anorganischem Phosphat im periplasmatischen Raum. Bei Phosphatbindung ist phoR inaktiv, ist kein Phosphat gebunden, ändert sich die Konformation von phoR wodurch es in der Lage ist mit dem cytoplasmatischen Teil Phosphate von ATP abzuspalten. Das freie Phosphat im Cytosol aktiviert phoB, das jetzt als Aktivator weiterer Gene dienen kann. Man spricht in diesem Fall von einem ZweiKomponentensystem. 21 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Regulation der Glutamin-Synthase Gens durch Erniedrigung der Konzentration organisch gebundenen Stickstoffs • • • • Die Proteine NtrB und NtrC bilden ein zwei-Komponenten System zur Regulation von Genen, die zur StickstoffFixierung notwendig sind. Im Falle des glnA Gens ist die mit σ54 assoziierte Polymerase bereits an den Promoter gebunden, kann jedoch den Promoter nicht in eine offene Konformation überführen. Phosphorylierung von NtrC durch NtrB bewirkt Bindung an DNA, Interaktion mit RNA Polymerase und Aktivierung des glnA Promoters. Ähnlich wie bei enhancers eukaryotischer Gene liegt die NtrC Bindungsstelle weit entfernt vom Transkriptionsstart. Bei der Interaktion mit RNA Polymerase bildet die zwischen liegende DNA eine Schlaufe. 2. Bild ... NtrB und σ54 interagieren und bilden DNA Loop. NtrB Protein löst sich nach Transkriptionsbeginn. In Anwesenheit von Quecksilber sorgt das MerR Protein für eine zur RNA Pol Bindung notwendige topologische Positionierung der –35 und –10 Elemente Mer ... Mercury (Quecksilber) Erst durch die Drehung der DNA durch das MerR Protein liegen die -35 und -10 Konsensussequenzen in einer Ausrichtung, die von einer RNA Polymerase gebunden werden kann. 22 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Transkriptionskontrolle in Prokaryoten und Eukaryoten Prokaryoten: Eukaryoten: • • • • • • • • 1 RNA Polymerase Polycistronische mRNA Transkription/Translation gekoppelt relativ einfach strukturierte Promotoren Bindung und korrekte Positionierung durch das RNA Polymerase Holoenzym 3 RNA Polymerasen Monocistronische mRNAs Translation erst nach mRNA Transport und Prozessierung • Komplexe Promoterstruktur, Bindungsstellen für multiple regulatorische Proteine • Ein komplexer Initiationskomplex ist notwendig um RNA Polymerase zu rekrutieren und korrekt zu positionieren. In Prokaryoten ist die Promotorstruktur relativ einfach: -35 und -10 Consensussequenz, dazu Sequenzen für positive oder negative Regulation (Operatoren und Inducer). Eukaryotische Promotorstrukturen sind demgegenüber komplexer. Man unterscheidet bei Eukaryoten zwischen gering regulierten "house keeping genes" (laufend benötigte Gene für Proteine des Cytoskeletts, der Membran, der Organellen, etc.) und Genen die situationsabhängig (Embryonalentwicklung, organspezifische Gene, Reaktion auf Infektionen, etc.) transkribiert werden. Promotorstrukturen und Regulation dieser Gruppen unterscheiden sich grundlegend. Eukaryotische RNA Polymerasen In Eukaryoten finden sich drei unterschiedliche RNA Polymerasen, die vom Aufbau her ähnlich sind, sich aber in einigen Untereinheiten voneinander unterscheiden und dadurch unterschiedliche Tätigkeiten durchführen können. Im Vergleich mit Prokaryoten finden sich Homologe zu ß, ß' und α UE. Nur die eukaryotische RNA Polymerase II weist eine ausgeprägte CTD auf. 23 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Unterscheidungskriterien der 3 eukaryoten RNA Polymerasen Zelluläre Lokalisation Pol I Nucleolus Pol II Nucleoplasma Pol III Nucleoplasma Produkte rRNA Pre-mRNA, snRNAs tRNA, 5SrRNA snRNAs α-Amanitin-Sensitivität (IC50, μg/ml) >1000 0.01-0.05 10-25 Mn2+/Mg2+ Verhältnis der relativen Aktivität 1-2 5-10 2 RNA Polymerase I findet sich im Nukleolus, in dem vor allem die DNA ribosomaler Gene vorliegt und dient, wie erstaunlich, der Transkription von rRNA. RNA Polymerase II: findet sich im Nucleoplasma und transkribiert Gene, einerseits die für Proteine transkribieren und andererseits snRNAs die für das Splicen essentiell sind. Amanitin ist ein Bestandteil des Gifts des Knollenblätterpilzes und zielt auf RNA Polymerasen ab. Pol I ist fast unempfindlich, für Pol II und Pol III ist eine geringe Konzentration ausreichend um die Aktivität zu beeinträchtigen. Über Amantinkonzentration kann so ermittelt werden, welche Polymerase welche Produkte transkribiert. Eine Unterscheidung der Polymerasen ist auch über Mn2+/Mg2+ Konzentration möglich. Die Architektur der RNA Pol II aus Hefe Cramer et al. (2000) Science 288: 640 Eukaryotische RNA Polymerase ist komplizierter als prokaryotische RNA Polymerase, aber schematisch ähnlich. Es finden sich Kanäle für DNA und mRNA und ein aktives Zentrum. Strukturelle Ähnlichkeit zwischen der bakteriellen RNA Polymerase und RNA Pol II aus Hefe 24 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Komplexität der Promotoren aus Eubakterien, Hefe und Säugerorganismen - - Promotoren in Prokaryoten befinden sich möglichst nah am Gen. Hefepromotoren weisen bereits mehr regulatorische Bindungsstellen auf und sind weiter vom Transkriptionsstart entfernt. Promotoren höherer Eukaryoten weisen extrem viele Regulatoren vor und nach dem Transkriptionsstart auf, regulatorische Sequenzen finden sich auch in Introns. Grundstruktur der Promotoren prokaryoter und eukaryoter Gene Eukaryoten weisen keinen einheitlichen σ-Faktor auf, es wird stattdessen eine Vielzahl anderer Proteine erzeugt, die diese Aufgabe übernehmen. Die Promotorstrukturen der einzelnen eukaryotischen Polymerasen lassen sich unterscheiden. Pol I Gene weisen einen so genannten Core Promotor mit einer einzigen URS (upstream regulatory sequence) auf. Es gibt zwei unterschiedliche Pol II Promotoren: - Promotoren für Gene die für Proteine codieren weisen eine der prokaryotischen -10 TATA-Box ähnliche Sequenz bei -20 auf und haben zusätzlich URS. - Promotoren für Gene die für snRNA codieren weisen URS aber keine TATA-Box auf. Pol III Gene werden in drei Gruppen geteilt. 25 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 21.03.2006 gefehlt Systematische Analyse der Transkriptionskontrolle eines durch RNA Pol II transkribierten, eukaryoten Gens 1. Welche Gene werden durch Stimuli aus der zellulären Umgebung reguliert? DNA-microarray, Northern blot, RT-PCR 2. Liegt Transkriptionskontrolle vor? Nuclear run on assay 3. Isolieren des Promoters Genomische Klonierung, Transiente Transfektion, Reportergen assay 4. Wo binden wichtige Transkriptionsfaktoren? Mutations/Deletionskartierung des Promoters, Footprinting, electrophoretic mobility shift assay (EMSA) 5. Welche Transkriptionsfaktoren sind an der Regulation beteiligt? DNA Affinitätschromatographie, Proteinsequenzierung, cDNA Klonierung 6. Struktur-Funktionsbeziehung im isolierten Transkriptionsfaktor (wo sind funktionelle Domänen?). Deletions- und Punktmutagenese der cDNA, Ko-Transfektionsassays 7. Interaktion des Transkriptionsfaktors mit anderen Proteinen Hefe 2-Hybrid-System, Biochemische Verfahren und Massenspektroskopie 1. Welche Gene werden durch Stimuli aus der zellulären Umgebung reguliert? Prinzipiell wird mRNA aus ruhenden, unbehandelten Zellen mit mRNA aus Zellen verglichen, die Stimuli z.B. Wachstumshormone, immunologische Signale etc. ausgesetzt wurden. Durch diesen Vergleich kann überprüft werden, ob die Expression eines bestimmten Gens durch Umweltfaktoren beeinflusst wird. mRNA Bestimmung durch Northern Blot 1. Zelluläre RNA auf Agarosegel auftrennen 2. Transfer der RNA auf nitrozellulose Membran Die Übertragung erfolgt rein durch Saugkräfte. 3. Hybridisieren der Membran mit radioaktiv markierter, genspezifischer Sonde. Genspezifische Sonde ist eine zur mRNA komplementären radioaktive DNA. 4. Ermitteln des Hybridisierungssignals durch Autoradiographie Konnte die Sonde mit mRNA hybridisieren, schwärzt die radioaktive Sonde einen Röntgenfilm und gibt Position und Menge der gesuchten mRNA an. So kann getestet werden, ob ein gesuchtes Gen durch Umweltfaktoren aktiviert, deaktiviert oder die Konzentration des Transkripts verändert wird. DNA Sonden können mit γ-32P-ATP und dem Enzym Polynukleotid Kinase radioaktiv markiert werden Für Nothern Blot essentiell ist die radioaktive Markierung der DNA Sonden. Das Enzym Polynucleotide kinase fügt das γ-Phosphat eines ATPs an das 5' Ende von DNA Einzelsträngen. Man verwendet dabei ATP, das radioaktives 32P enthält. 26 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Reverse Transkription (RT)-PCR RNA-Isolierung Isolierung zwecks Vergleichs wieder aus behandelten und unbehandelten Zellen. È Reverse Transkription d.h. Synthese einer zur RNA komplementären cDNA mittels reverser Transkriptase. È cDNA Amplifikation durch PCR mit genspezifischen Primerpaaren È Analyse des Amplifikationsprodukts durch Agarose-Gelelektrophorese In Bezug auf Quantität einer RNA ist die Analyse mittels RT-PCR eher beschränkt, da sich geringe Mengenunterschiede zwischen behandelter und unbehandelter Zelle nur schwer feststellen lassen, wenn man die PCR zu viele Zyklen durchlaufen lässt. Ist andererseits die Konzentration von mRNA prinzipiell niedrig, wird man bei zu wenigen PCR Zyklen kein sinnvoll verwertbares Signal erhalten. Quantitative, real-time RT-PCR Real time RT PCR wird verwendet, um PCR auch für quantitative Analysen einsetzen zu können. • In die amplifizierte doppelsträngige DNA lagert sich ein Farbstoff ein. Der Farbstoff wird der PCR in ausreichender Konzentration hinzugefügt. • Die durch die Einlagerung in die DNA entstehende Fluoreszenz wird während der PCR in Echtzeit gemessen. Die Fluoreszenz des Farbstoffes entwickelt sich erst, wenn er sich in die dsDNA eingelagert hat. • Damit kann die lineare, quantitative Phase der PCR bestimmt und ausgewertet werden. Die Intensität der Fluoreszenz wird gemessen, sie nimmt mit jedem PCR Zyklus zu, sie ist direkt proportional zur erzeugten DNA Menge. Da die Fluoreszenzintensität während laufender PCR gemessen wird, können die Messwerte behandelter und nichtbehandelter Zellen direkt miteinander verglichen werden und auch geringe quantitative Unterschiede ermittelt werden, während man bei normaler RT-PCR nur das Endprodukt vergleichen kann. Gene Microarrays (=DNA Chips): Die Methode der Wahl zur Genomweiten Analyse von Genexpression. Beispiel: während Wachstums in Glucose bzw. in Ethanol exprimierte Gene in Saccharomyces erscheinen grün bzw. rot. Größe: 18x18mm, enthält alle 6400 Gene aus Saccharomyces an definierten Positionen. Für diese Methode muss das Genom des zu untersuchenden Organismus bekannt sein. Es wird zu jedem Gen im Genom in ausreichender Menge komplementäre DNA erzeugt. Diese komplementäre DNA jedes Gens wird auf einer jeweils genau definierten Position eines Glaschips aufgebracht, sodass jede Stelle dieses Chips einem Gen des Organismus entspricht. Anschließend wird die mRNA einer mit einem Umweltfaktor behandelten Zellkultur isoliert und in fluoreszierend markierte cDNA umgeschrieben. Diese cDNA wird auf den Chip aufgebracht, die cDNA wird nur an den Stellen des Chips binden, die dem korrelierende Gen entsprechen. Auf diese Weise kann einfach ermittelt werden, welche Gene aktiv sind und welche nicht, durch die Intensität der Fluoreszenz ergibt sich die Quantität der Transkripte. Stellt man Testreihen mit unterschiedlichen Umweltfaktoren an und markiert sie mit Fluoreszenzmarkern unterschiedlicher Farbe, kann man auf demselben Chip vergleichen, welche Gene wo wann aktiv sind – im oben genannten Bsp. Grün für Glucose und rot für Ethanol. Mischfarben entsprechen dann einer einheitlichen Expression bei unterschiedlichen Umweltfaktoren, im Bsp. Wäre das Gelb. 27 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Fig. 1. 2-5A induces gene expression changes in DU145 prostate carcinoma cells in a dose-and time-dependent manner Copyright ©2005 by the National Academy of Sciences Malathi, Krishnamurthy et al. (2005) Proc. Natl. Acad. Sci. USA 102, 14533-14538 Im Menschen kann die Chiptechnik dazu verwendet werden, mittels der zelltypspezifischen Transkription von mRNA zu Testen, ob es sich bei einem untersuchten Gewebe um eine bekannte Tumorart handelt. Gen 'Cluster'-Analysen ermöglichen das Auffinden zelltypspezifischer Gengruppen (hier gezeigt anhand von Tumoren unterschiedlichen Ursprungs). In den Zeilen befinden sich RNAs aus unterschiedlichen Tumorarten. Ein Patient kommt daher und hat einen Tumor z.B. in einem Knochen. Dieser Tumor ist zumeist nicht der Primärtumor! Prostatakrebs bildet z.B. oft Metastasen in Knochen. Die Zellen des Knochentumors unterscheiden sich aber nicht von denen des Primärtumors … über Array kann durch die auf die Tumorart spezifischen Marker festgestellt werden, um welche Tumorart es sich handelt und wo der Primärtumor sitzt, der im Moment noch gar keine Beschwerden verursacht. Ist der Primärtumor identifiziert, kann man die aus Erfahrungswerten am besten passende Behandlung bzw. Medikation verschreiben. 28 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 2. Liegt Transkriptionskontrolle vor? Mit den bisher behandelten Methoden kann nur die "steady state" Menge an mRNA gemessen werden d.h. die Menge mRNA die durch das Zusammenspiel an Transkription und mRNA Abbau vorliegt. Man erhält dabei keine Informationen darüber, ob die mRNA eines Gen auf der Ebene der Transkription oder des Abbaus reguliert wird. Der 'nuclear run on assay' Mittels Nuclear run on assay kann geprüft werden, ob ein Gen auf Transkriptionsebene reguliert wird. Dafür werden Zellkerne isoliert und auf Eis gelegt, wodurch die Transkription durch die RNA Polymerasen vollständig gestoppt wird ohne sich von der DNA zu lösen. Durch die Isolation der Zellkerne wird weiters der Abbau durch RNase im Cytosol verhindert. Die Kerne werden anschließend auf 37°C erwärmt und fügt ein Gemisch radioaktiv markierter Nucleotide hinzu, die in die mRNA eingebaut werden, wenn die RNA Polymerasen ihre Tätigkeit wieder aufnehmen. Diese mRNA wird isoliert und auf Makroarrays aufgebracht. Diese Makroarrays sind auf dieselbe Art hergestellt wie Microarrays, mit dem Unterschied, dass sie nur wenige Gene binden können. Auf dem Makroarray wird ein Röntgenfilm aufgebracht, der je nach Konzentration der radioaktiven mRNA schwach bis stark geschwärzt wird. Die Stärke der Schwärzung des Röntgenpapiers ist dementsprechend proportional der Konzentration der mRNA die wiederum proportional der Transkriptionsrate des zu untersuchenden Gens ist. Werden wieder behandelte und unbehandelte Zellen verglichen kann man ermitteln, welche Umweltfaktoren tatsächlich die Transkription und nicht den Abbau beeinflussen. 3. Isolieren des Promoters Genomische Klonierung, Transiente Transfektion, Reportergen assay Für die Isolation eines Promotors wird eine genomische Bibliothek benötigt. Partieller Restriktionsverdau liefert überlappende Fragmente genomischer DNA Zu diesem Zweck muss die genomische DNA zunächst partiell verdaut werden d.h. die Konzentration der Restriktionsnucleasen wird so gewählt, dass man 20kbp lange Stück DNA erhält. 29 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Genomische DNA kann in λ-Phagen DNA kloniert und in λ-Virionen verpackt werden Die partiell verdaute DNA wird in vitro mit den Armstücken des Phagen λ ligiert. Diese Konstrukte lassen sich in vitro mit gereinigten Phagenproteinen verpacken, sodass man infektiöse Phagenpartikel mit der gesamten genomischen DNA erhält und sich in Bakterien vermehren können (so richtig?). Die Phagen werden auf einen Bakterienrasen aufgebracht wo sie die DNA in Bakterien einbringen. Infizierte Bakterien werden durch neu produzierte Phagen lysiert, an den Stellen bilden sich Plaques im Zellrasen. Auf den Zellrasen wird ein Nitrozellulosefilter aufgebracht, der Lysat und Phagen aufnimmt. Mittels alkalischer Lösung werden die Phagen lysiert und die DNA in Einzelstränge aufgetrennt. Der Filter wird mit einer der mRNA entsprechenden radioaktiven Sonde versetzt, sie wird nur bei dem Phagenplaque binden, die das entsprechende Gen enthält. Dabei ist zu sagen, dass die 50nt für eine starke Bindung zwischen cDNA und genomischer DNA ausreichen. In eukaryotischer DNA kann die Sonde stabil an die Exons der genomischen DNA binden. Man erhält so ein 20kbp langes Stück DNA, das mit hoher Wahrscheinlichkeit auch den Promotor des interessanten Gens enthält. Dieser Vorgang ist nur dann notwendig, wenn das Genom des Organismus noch nicht als Datenbank zur Verfügung steht. Genomische Klonierung bei Genomen mit bekannter DNA Sequenz • • • DNA kann aus 'Banken' bezogen werden. DNA wird meist als bacterial artificial chromosome (BAC) vermehrt. Diese enthalten die Replikationssignale aus F Plasmiden und können inserts von bis zu 300kb vermehren. BACs sind oft zu gross für klassische Kloniertechniken. Zur Rekombination wird daher meist die Technik des 'Recombineering' verwendet. 30 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert S1 mapping und 'primer extension' sind zwei Verfahren zur Bestimmung des Transkriptionsstarts 1) Primer extension: das 20kbp Stück wird durchsequenziert, man kann kurze Primer von unterschiedlichen Stellen des nichtkodierenden Strangs synthetisieren. Diese Primer werden mit mRNA versetzt und durch Reverse Transkriptase bis an den Beginn der mRNA verlängert. Werden die unterschiedlich langen Stücke über Gelelektrophorese aufgetrennt, so entspricht das kürzeste Stück dem Primer, der dem Transkriptionsstart am nächsten liegt. 2) S1 nuclease mapping: Es werden längere Stücke des nichtkodierenden Stranges der DNA synthetisiert und mit mRNA vermischt. War das synthetisierte Stück komplementär zur mRNA, wird sie binden können und überstehende Enden erzeugen. Die überstehenden Einzelstränge können durch S1 Endonuclease geschnitten werden, dabei entspricht das Ende der synthetisierten DNA dem Transkriptionsstart. Wird der verbleibende Doppelstrang denaturiert und das synthetisierte Stück über Elektrophorese aufgetrennt, kann festgestellt werden, wie lang das DNA Stück ist und damit wo in der synthetisierten Sequenz sich der Transkriptionsstart befindet. 31 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Deletionskartierung der funktionell wichtigen Promoter-DNA durch Exonuclease Verdau und Religation Promotor bashing: Die ermittelte Promotor wird mehrfach in Plasmide kloniert, wobei die Promotorsequenz bei jedem Plasmid von beiden Enden her sukzessive weiter verkürzt wird. Die Promotorsequenz dient als Promotor für ein Reportergen, das sich ebenfalls auf dem Plasmid befindet. Die einzelnen Plasmide werden in die Zellart eingebracht, in der er vermutlich reguliert wird z.B. Leberzellen. Je nachdem ob das Reportergen sehr gut, weniger gut oder gar nicht mehr exprimiert wird, kann man erkennen, ab wann bei den verkürzten Sequenzen regulatorische Sequenzen dabei waren, die für die Funktion des Promotors von Bedeutung sind. Reportergen-Assays erlauben die Bestimmung der Promoter-Aktivität. Gekoppelt mit gezielten Veränderungen der Promoterstruktur erlauben sie das Auffinden regulatorischer DNA-Sequenzen eines Promoters. Unter einem Reportergen versteht man ein Genprodukt, das sich einfach nachweisen lässt und in der fraglichen Zelle nicht vorkommt. Als Beispiel die Chloramphenicolacetyltransferase (CAT) das normalerweise Bakterien vor dem Antibiotikum Chloramphenicol schützt. Das Reportergen wird mit einem eukaryotischen Promotor in eine eukaryotische Zelle eingebracht und dort exprimiert. Später wird die Zelle lysiert, das Lysat mit radioaktiv markiertem Chloramphenicol versetzt und in weiterer Folge mittels Dünnschichtchromatographie das acetylierte vom unveränderten Chloramphenicol getrennt. Die Menge des acetylierten Chloramphenicol hängt von der Stärke der Transkription des eingebrachten Promotors ab. 32 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Reportergen-assays • • Reportergene kommen in der zur Promoteranalyse verwendeten Zelle nicht vor (meist bakterielle oder Invertebratengene). Häufig verwendete Reportergene: o Chloramphenicol-Acetyl-Transferase (CAT) o Luciferase (aus Glühwürmchen, erzeugt Licht bei der Substratumsetzung) o Green fluorescent protein (GFP; kann zur Fluoreszenz angeregt werden, Expressionsanalyse auch in lebenden Zellen möglich) o β-Galaktosidase (oNPG-Farbreaktion) X-Gal wird durch ß-Galaktosidase in Galaktose und einen blauen Farbstoff gespalten. Gentransfer in Säugerzellen • • • • • CaPO4-Kopräzipitation Es bilden sich Kalziumphosphatkristalle, die DNA mit einschließen. Zellen können die Kristalle aufnehmen und auflösen, wodurch die DNA in der Zelle freigesetzt wird. Elektroporation Zellen werden in einer Küvette unter Spannung gesetzt wodurch die Membran kurzfristig perforiert wird und DNA aus der Lösung in die Zellen eintreten kann. Lipofektion Es werden künstliche Lipidvesikel erzeugt, die DNA enthalten. Diese Vesikel können mit der Zellmembran verschmelzen und die DNA in die Zelle entlassen. Infektion (virale Vektoren) (Mikroinjektion) Sequenzvergleiche homologer DNA Abschnitte in verschiedenen Spezies kann zum Auffinden regulatorischer DNA Bereiche führen 33 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 4. Wo binden wichtige Transkriptionsfaktoren? Mutations/Deletionskartierung des Promoters, Footprinting, electrophoretic mobility shift assay (EMSA). DNase I footprinting und der electrophoretic mobility shift assay (EMSA)zeigen spezifische Bindung eines Transkriptionsfaktors an Promoter DNA. Über DNase I Footprintig kann überprüft werden, ob die RNA Polymerase oder ein Transkriptionsfaktor tatsächlich im Bereich der fraglichen Sequenz bindet. Mit EMSA (elektrophoretic mobility shift assay) kann diese Frage einfacher geklärt werden. Man erzeugt radioaktiv markierte dsDNA des Bereichs, von dem vermutet wird, dass Polymerase oder Transkriptionsfaktor daran binden. Anschließend werden die markierten DNA Stücke mit Kernextrakt versetzt und auf einem Gel getrennt. Handelt es sich tatsächlich um eine Bindungssequenz, werden die entsprechenden Transkriptionsfaktoren aus dem Kern an die DNA binden und auf einem Röntgenfilm zwei Bande hinterlassen – eine für freie DNA und eine für gebundene. Handelt es sich nicht um eine Bindungssequenz, findet sich nur eine Bande für freie DNA. 34 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Saturierende Punktmutagenese enthüllt multiple Bindungsstellen für Transkriptionsfaktoren im Enhancer des SV40 Virus. Durch Punktmutagenese wird überall dort wo eine wichtige Bindungsstelle vermutet wird eine Punktmutation eingeführt und anschließend die Promotoraktivität gemessen. Sinkt die Promotoraktivität ab, war die veränderte Base für die Bindung des Transkriptionsfaktors von Bedeutung. 5. Welche Transkriptionsfaktoren sind an der Regulation beteiligt? DNA Affinitätschromatographie, Proteinsequenzierung, cDNA Klonierung Hat man einen Promotor isoliert und sequenziert, kann man daran gehen beteiligte Transkriptionsfaktoren zu isolieren. 28.03.2006 DNA Affinitätschromatographie Reinigung von Transkriptionsfaktoren durch sequenzspezifische DNAAffinitätschromatographie Zunächst wird ausreichender Zellextrakt erzeugt und die Proteine grob nach Größe getrennt. Die DNA Bindungssequenz des TFs (Transkriptionsfaktors) ist bekannt. Es wird ein kurzes, doppelsträngiges Stück dieser DNA Sequenz synthetisiert. Die synthetisierte DNA wird an eine träge Matrix (meist Sepharose) gekoppelt. Es wird der zuvor aufbereitete Zellextrakt hinzugefügt. Die Lösung wird so gewählt, dass die Bindung anderer Proteine an DNA herabgesetzt wird, damit nur der die synthetisierte DNA Sequenz bindende TF mit der DNA interagiert. Anschließend wird die Säule mit Lösungen ansteigender Ionenstärke (viel Salz) durchgewaschen, um an die DNA gebundene Protein zu lösen. Mit jeder dieser Fraktionen wird ein DNase I Footprint der untersuchten Sequenz durchgeführt. Erhält man einen Footprint (fehlende Banden im Gel durch an DNA gebundenen TF), so enthält die korrelierende Fraktion einen hohen Anteil an gesuchtem TF (im Beispiel entspricht das den Fraktionen 9-12). Diese Fraktionen können mehrmals durch Säulenchromatographie gereinigt werden, um den TF in möglichst reiner Form zu erhalten. 35 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Klonieren einer cDNA durch Expression in E.coli und Detektion mit Hilfe eines spezifischen Antiserums Um das Gen zu finden, das den TF erzeugt wird zunächst das gereinigte Protein in einen Hasen gespritzt, der AK (Antikörper) gegen das körperfremde Protein erzeugen wird. Einige Zeit später wird dem Hasen Blut abgenommen und alle AK isoliert. Es wird eine cDNA Bibliothek des zu untersuchenden Organismus erstellt, wobei jede dieser cDNAs mit einem Promotor versetzt wird, damit die cDNA in Bakterien transkribiert werden kann. Dieses Konstrukt wird mit λ-DNA verknüpft, damit die DNA in Phagenpartikel verpackt werden kann. Mit den Phagen wird wieder ein Zellrasen besprüht, infizierte Bakterien erzeugen nicht nur Phagenproteine, sondern auch die Proteine, die durch die cDNA codiert werden. Es entstehen Phagenplaques, die auch die erzeugten Fremdproteine enthalten. Die Plaques werden abgestempelt und mit den aus dem Hasen gewonnenen AK vermischt. Die AK die im Hasen gegen das eingespritzte Protein erzeugt wurden, werden bei dem Phagenplaque binden, dessen cDNA für dieses Protein codiert. Es werden weitere, radioaktiv markierte AK hinzugefügt, die die konstante Region von AK binden können und daher an zuerst hinzugefügten "Hasen" AKs binden, die wiederum an den gesuchten Proteinen haften. Über einen Röntgenfilm kann der Phagenplaque mit der gesuchten cDNA ermittelt und diese cDNA isoliert, vermehrt und sequenziert werden. Wurde eine cDNA einem TF zugeordnet, muss bewiesen werden, dass es sich dabei tatsächlich um den gesuchten TF handelt. Bestimmung von Transkriptionsfaktoraktivität 1. In vitro Transkription Es wird eine Sequenz konstruiert, die einen Promotor mit mehrfachen Bindungsstellen des ermittelten TFs aufweist. Anschließend wird ein Zellkernextrakt hinzugefügt, der alle notwendigen Faktoren für eine Transkription enthält. Wird der untersuchte TF hinzugefügt, sollte die Transkription erhöht werden bzw. erst überhaupt stattfinden können. 36 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 2. Cotransfektionsassay mit einem Reportergen Um zu prüfen, ob die ermittelte cDNA tatsächlich für ein Protein codiert, das als TF für eine bestimmte Sequenz dient, wird die cDNA dieses Gens mit einem Promotor auf ein Plasmid kloniert. Auf ein zweites Plasmid wird ein Reportergen kloniert, dessen Promotor die fragliche Bindungssequenz des gesuchten TF enthält. Beide Plasmide werden in eine Zelle transfektiert. Das Gen der cDNA wird in jedem Fall erzeugt, handelt es sich dabei tatsächlich um den gesuchten TF, wird er die Transkription des Reportergens auf dem zweiten Plasmid ermöglichen, man erhält das erfreuliche Signal, dass die Arbeit der letzten Monate nicht vergebens war. 6. Struktur-Funktionsbeziehung im isolierten Transkriptionsfaktor (wo sind funktionelle Domänen?) TFs weisen zumindest zwei funktionelle Domänen auf. Die DNA bindende Domäne, die mit der passenden Bindungssequenz interagiert und die so genannte transaktive Domäne, die mit der RNA Polymerase in Kontakt tritt, um die Transkription zu steigern oder zu hemmen. Im Beispiel des Lac Repressors ist eine weitere Domäne für die Funktion notwendig, die Dimerisierungsdomäne, die ein Binden der einzelnen Proteine zur funktionellen Einheit ermöglicht. Struktur-Funktionsanalyse eines Transkriptionsfaktors durch gezielte Deletionsmutagenese und Aktivitätsbestimmung im Reportergenassay Um die einzelnen Domänen zu identifizieren, wird Deletionsmutagenese eingesetzt. Bsp. Der TF Gal4 der in Hefe im Galactosemetabolismus reguliert. Mittels Cotransfektion und EMSA wird zuerst bestätigt, dass der TF an die UAS bindet und die Transkription verstärkt. Anschließend werden Plasmide mit der cDNA des TF erzeugt, die um unterschiedlich lange Stücke DNA am C-Terminus, am N-Terminus oder aus der Mitte verkürzt sind. Wird mit diesen Plasmiden und dem Reportergen erneut Cotransfektion und EMSA durchgeführt, kann ermittelt werden, welche Teile der cDNA für Interaktion mit DNA (kein EMSA Signal bei Deletion und kein Reportergen) oder für Interaktion mit der RNA Polymerase (EMSA Signal, aber kein Reportergen) zuständig sind. Mittels dieser Methode kann man mit ausreichenden Tests Lage und Länge der funktionellen Domänen auf einige AS genau ermitteln. 37 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Viele Transkriptionsfaktoren besitzen einen modulartigen Aufbau aus funktionellen Domänen (DNA-Bindung, Transaktivierung) Es gibt keine Regel, wo im TF sich die DNA bindende und die transaktivierende Domäne befindet. 7. Interaktion des Transkriptionsfaktors mit anderen Proteinen Hefe 2-Hybrid-System, Biochemische Verfahren und Massenspektroskopie Yeast-Two-Hybrid System zur Ermittlung von Proteininteraktionen Es handelt sich um ein Verfahren, bei dem die Domänen eines Proteins getrennt und mit Hilfe von interagierenden Proteinen wieder zusammengesetzt werden. Plasmid 1 codiert für die DNA bindende Domäne, die mit einer so genannten Bait Domäne verbunden ist. Die Bait Domäne selbst darf dabei keine transaktivierende Eigenschaft haben. Plasmid 2 codiert für die transaktivierende Domäne, die mit einer so genannten Fish Domäne verbunden ist. Abschließend benötigt man ein Reportergen, dessen Aktivierungssequenz genau so weit von der Bindungsstelle der RNA Polymerase entfernt ist, dass es nur über das Hybridprotein transkribiert werden kann. Das ganze wird in einen Hefestamm transfektiert, der Histidin nicht synthetisieren kann. Das Reportergen sind in diesem Fall die Histidin synthetisierenden Gene. Wird der Hefestamm auf ein Histidinarmes Medium überführt, werden nur die Hefezellen überleben, die alle Plasmide aufgenommen haben und bei denen das Hybridprotein tatsächlich als TF funktioniert. Sollen Gene mit einer bestimmten DNA bindenden Domäne ermittelt werden, so stellt man eine cDNA Bibliothek her, an jede cDNA wird eine Bait Domäne angefügt und in Plasmide kloniert. Diese Plasmide werden gemeinsam mit den Plasmiden in den Reporterhefestamm eingebracht, die für die transaktivierende + Fischdomäne codieren. Codiert die cDNA auf einem Plasmid für ein Genprodukt, das die fragliche DNA Sequenz binden kann, so wird der Hybrid aus Fish und Bait die Reportergene aktivieren können. Dieser Vorgang kann umgekehrt auch dazu verwendet werden um eine cDNA Bibliothek mit transaktivierenden Domänen zu erstellen. 38 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Identifizieren von Proteininteraktionen: der proteomische Ansatz • • • • • Reinigung des Proteinkomplexes Der zu untersuchende Proteinkomplex wird z.B. mit Hilfe von AK gereinigt. Auftrennen der einzelnen Komponenten (z.B. Gelelektrophorese, 2-D Gelelektrophorese) Im ersten Bild entsprechen die Flecken der über Elektrophorese aufgetrennten einzelnen Proteinkomponenten des Komplexes. In der 2-D Gelelektrophorese werden Proteine einerseits nach ihrer Größe und andererseits in der anderen Richtung nach ihrem isoelektrische Punkt aufgetrennt, man erhält keine Proteinbanden sondern "Spots". Proteaseverdau … Peptide der einzelnen Proteine Die Proteinkomponenten werden aus dem Gel geschnitten und einzeln verdaut. Es wird dabei immer dieselbe Protease z.B. Trypsin verwendet, wodurch die Proteine in eindeutige Stücke gespalten werden. Auftrennen und Ansequenzieren der einzelnen Peptide oder Bestimmung der Peptidmassen durch Massenspektrometrie … charakteristischer 'fingerprint' an Peptidmassen … Datenbank … Zuordnung des Proteins. Massenspektroskopie ermittelt das Verhältnis von Masse zu Ladung der einzelnen Peptidstücke. Die Gesamtheit dieser Verhältnisse ergibt einen 'fingerprint' über den in Datenbanken das zugehörige Protein ermittelt werden kann, wenn es bereits untersucht wurde. Transkriptionsfaktoren Basale oder generelle TFs: • sind Bestandteil des Praeinitiationskomplexes, welcher die RNA Polymerase an den Transkriptionsstart positioniert. Allgemeine TFs: • binden an Bindungsstellen im proximalen oder distalen Promoterbereich (Enhancer). • Beeinflussen die Frequenz, mit der der Praeinitationskomplex gebildet wird, entweder positiv (Aktivatoren) oder negativ (Repressoren). Häufig vorkommende Domänen (eukaryoter) Transkriptionsfaktoren 1. Helix-Turn-Helix Struktur: eine DNA-Bindungsdomäne die man oft in prokaryoten Transkriptionsfaktoren findet (Beispiel: l-Repressor, Lac, Trp Repressoren), die aber auch in eukaryoten Transkriptionsfaktoren vorkommt (Beispiel: HomeodomänenTranskriptionsfaktoren). 2. Zink-Finger Struktur: findet man in einer grossen Zahl eukaryoter Proteine (>1000). Die Zn-Finger Struktur kann, muss aber keine DNA-Bindungsdomäne bilden. Es gibt zwei Typen von Zn-Finger Strukturen. Beiden C2H2 Fingern wird das Zn-Atom durch zwei Cysteine und zwei Histidine komplexiert, beiden C4 Fingern durch vier Cysteine. 3. Leucine-Zipper (LZ) Struktur: Eine amphipathische Helix, welche die Dimerisierung von Transkriptionsfaktoren bewerkstelligt. Der LZ ist mit einer 'basic region' positiv geladener Aminosäuren verbunden. Die basic region vermittelt die DNA Bindung. 4. Helix-Loop-Helix Struktur: Vom Prinzip her ähnlich den LZ Proteinen, jedoch ist die Dimerisierungshelix durch eine Schlaufe (Loop) unterbrochen. 39 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 1. Das helix-turn-helix motif der Homeodomäne des engrailed proteins: Das helix-turn-helix motif als wesentlicher Bestandteil einer DNA bindenden Domäne. Das Motif besteht aus drei Helices; Helix 1 interagiert basenspezifisch über den NTerminus in der kleinen Furche, Helix 3 bindet basenspezifisch in der großen Furche der DNA Diese Motif findet sich häufig in Prokaryoten z.B. im λ Repressor, Lac Repressor, Trp Repressor, sowie vor allem in der entwicklungsbiologischen Genexpression bei Eukaryoten. 2. Die Zn-Finger Struktur: kompakte Anordnung von β-Faltblatt und α Helix durch Komplexierung mit dem Zn2+ Atom Proteine der Zn Finger Struktur bilden die größte Proteinfamilie im eukaryotischen Genom. Das Motif wird neben der DNA Bindung für Protein-Protein Interaktionen verwendet. Die funktionelle Struktur ergibt sich durch die wechselnde Anordnung von Helices und Faltblattstrukturen, die Anordnung wird durch die Bindung des Zn Atoms ermöglicht, das Zn Finger Motif bindet DNA immer in der großen Furche. Es gibt zwei unterschiedliche Zn Finger Strukturen, die DNA binden: Die C2H2 Struktur, bei der ein Zn Atom durch zwei Cysteinen und zwei Histidine gebunden wird und DNA als Monomer bindet. Die C4 Struktur, bei der vier Cysteine ein Zn Atom binden und DNA als Dimer bindet. C2H2 (GL1 Protein) und C4 (Glucocorticoid Rezeptor) Strukturen des Zn-Fingers. • • C2H2 Proteine enthalten 3 oder mehr Finger und binden als Monomere an DNA. C4 Proteine enthalten 2 Finger und binden als Dimere. Die Helix des Zn Fingers kontaktiert die DNA (a) C2H2 (b) C4 … die Bindung als Monomer an DNA ist instabil. 40 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 3. Leucine-Zipper und basic region • • Leucine-Zipper sind der Spezialfall einer durch amphipathische Helices gebildeten coiled-coil Struktur Der L-Zipper dient der Protein Homo- oder Heterodimerisierung In Transkriptionsfaktoren kommen L-Zipper immer im Zusammenhang mit einer basic region vor (basische, d.h. positiv geladene Aminosäuren), welche die Bindung an DNA bewirkt. Die Leucine-Zipper Domäne wird durch eine amphipathische α Helix gebildet, über deren hydrophoben Anteil sich zwei Leucine Zipper in wässriger Umgebung zusammenlagern können. Um DNA binden zu können, benötigt das Protein eine weitere Domäne, die basic region. Zur Interaktion mit der negativ geladenen DNA weist die basic region ausgeprägt basische d.h. positiv geladene AS auf. 4. Die helix-loop-helix Struktur • • Eine durch einen Aminosäureloop unterbrochene Coiled-Coil Struktur Tritt in Transkriptionsfaktoren ebenfalls gekoppelt an eine basic region auf. Die helix-loop-helix Struktur entspricht dem Leucine Zipper, nur dass die Dimerisierungsdomäne durch einen AS Loop unterbrochen ist. Die SH2 Domäne P-SH2 SH2 • verleiht einem Protein die Fähigkeit, an tyrosinphosphorylierte Partner zu binden. • vermittelt die Dimerisierung einer Familie von Trankriptionsfaktoren, den STATS (signal transducers and activators of transcription). Die SH2 Domäne findet sich in Proteinen, die der Signaltransduktion dienen. Die Transkription wird durch TFs mit SH2 Domäne erst dann beeinflusst, wenn das Partnerprotein z.B. als Reaktion auf Umwelteinflüsse phosphoryliert wurde und dadurch ein Dimer gebildet werden kann. Transaktivierungsdomänen (TADs) • • • • Vermitteln Proteininteraktionen, die zur Initiation der Transkription führen Besitzen meist keine klar definierten Sekundär-oder Tertiärstrukturen Können durch das gehäufte Vorkommen bestimmter Aminosäuren klassifiziert werden: - Saure TADs (viel Glu, Asp) - Gln-reiche TADs - Pro-reiche TADs - Ser/Thr-reiche TADs Unterschiedliche Klassen der TADs indizieren vermutlich unterschiedliche Proteininteraktionen bei der Initiation der Transkription. D.h. je nachdem welche AS gehäuft vorkommen, kann eine Aussage über die Partner getroffen werden, mit denen die TADs interagieren werden. 41 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Decker Foliensatz 2 Die Initiation der Transkription • Transkription lässt sich in folgende Phasen unterteilen: - (Prae)Initiation: hier wird ein (Prae)Initiationskomplex gebildet, der RNA Pol an den Promoter rekrutiert. - Clearance: Übergang von einer statischen zu einer sich bewegenden RNA Pol. - Elongation: RNA-Synthese - Termination: Verlassen der DNA Matrize Viele durch RNA Pol II transkribierte Promotoren enthalten eine TATA-Box Basenhäufigkeit im –1/-35 Bereich von insges. 60 Promotoren von Vertebratengenen. DNA Sequenzen, die in Pol II Promotoren an der Bindung des Praeinitiationskomplexes beteiligt sind TFIIB verstärkt basale Transkriptionsfaktoren (TFII bei Pol II). Basale TF … TF die an der Präinitiation beteiligt sind. Viele der Gene die durch RNA Polymerase II transkribiert werden, enthalten eine Sequenz von etwa -26 bis -35, die der TATA-Box in Prokaryoten ähnlich ist. Die oben angeführten Sequenzen dienen der Bildung des Präinitiationskomplexes und können einzeln oder in unterschiedlichen Kombinationen miteinander vorkommen; d.h. jede dieser Sequenzen kann im Promotor eines Gens vorkommen, sie sind aber nicht obligatorisch. Die Sequenzen dienen der direkten oder indirekten Bindung des TFIID Komplexes. Basal (general) transcripton factors of RNA Pol II Name TFIIA TFIID TFIIF TFIIB TFIIE TFIIH Number of subunits 2 1 (TBP)+11 (TAFs) 3 1 2 9 Die meisten basalen TFs sind keine Einzelproteine sondern Komplexe aus unterschiedlich vielen Untereinheiten. 42 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Der (Prae) Initiationskomplex, (P)IC, von RNA PolII Promotoren Der erste Schritt ist die Bindung des basalen Transkriptionsfaktors TFIID an die TAATA-Box. TFIID besteht aus dem TAATA-binding protein (TBP) und den TBP-associated factors (TAFs). -TBP führt eine Biegung in die DNA ein -TFIIH phosphoryliert die carboxyterminale Domäne der Pol II. Neben der phosphorylierung der CTD der RNA Polymerase hat TFIIH DNA Helicaseaktivität, die für das Öffnen des Promotors essentiell ist. Bindung eines TBP Monomers biegt die DNA im Bereich der Initiationsstelle. TBP bindet an die große Furche der DNA, die Bindung führt zu einer Krümmung der DNA, die für die Bindung der RNA Polymerase Voraussetzung ist. Der TBP-TFIIB Komplex mit DNA TBP wird nach der Bindung an die DNA von TFIIB gebunden. TFIIB dient in weiterer Folge als Brücke zwischen TBP, DNA und RNA Polymerase. 43 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert TFIIB verbindet TBP mit der RNA Pol II Nachweis der RNA Pol II CTD Phosphorylierung an polytaenen Chromosomen in Drosophila: phosphorylierte RNA Pol II (rot) findet sich in den puffs, unphosphorylierte RNA Pol II hauptsächlich in den wenig transkribierten Bereichen TFIIH phosphoryliert die CTD von RNA Pol II. In den polytänen Chromosomen der Speicheldrüsen von Drosohiphila finden sich die phosphorylierten RNA Polymerasen vor allem in den stark transkribierten Bereichen, den so genannten Puffs. Demnach ist phosphorylierte RNA Polymerase für die Transkription von Genen verantwortlich. Sichtbar gemacht wurden sie durch Antikörper, die spezifisch nur an phosphorylierte RNA Polymerase binden. Die AK selbst wurden rot fluoreszierend markiert, die rote Farbe findet sich an den Transkribierten Stellen. Wie ist das Holoenzym der eukaryoten RNA Pol II beschaffen? • • • Im Labor experiment ergibt sich ein stufenweiser Aufbau des PIC aus den basalen Transkriptionsfaktoren. Biochemische Analyse der aus Zellen isolierten Pol II legt jedoch den Schluss nahe, dass das Holoenzym ein riesiger Proteinkomplex aus Core-Polymerase, basalen Transkriptionsfaktoren und Kofaktoren ist. Es besteht die Möglichkeit, dass nur TFIID und TFIIA unabhängig an den Promoter binden und dass dieser Komplex nachfolgend das RNA Pol II Holoenzym mit allen weiteren Komponenten rekrutiert. 44 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 04.04.2006 Die sequentielle Bildung des PIC aus basalen Transkriptionsfaktoren kann experimentell mit dem'electrophoretic mobility shift assay' (EMSA) untersucht werden. DNA weist bei einer Gelelektrophorese eine bestimmte Wanderungsgeschwindigkeit (mobility) auf. Wird DNA durch Proteine gebunden, gibt es eine charakteristische Änderung der Wanderungsgeschwindigkeit (mobility shift). Durch diesen Test konnte ermittelt werden, in welcher Reihenfolge die einzelnen Faktoren aneinander bzw. an die DNA binden – wurden die Faktoren nicht in der korrekten Reihenfolge hinzugefügt, kam es zu keiner Bindung an die DNA und zu keinem mobility shift: Reihenfolge: TFIID, dann TFIIA, erst dann RNA Polymerase II Holoenzymkomplex. RNA Polymerase II Holoenzyme Aus: Holstege and Young (1999) PNAS 96:2 Die Bindung des TFIID ist der geschwindigkeitsbestimmende Schritt. SRB Mediatorkomplex ... Proteinkomplex aus vielen Proteinen SWI / snf ... Chromatin remodelling complex TFIIH Komplexe spielen eine Rolle bei der promoter clearance und der DNA Excisions-Reparatur Es gibt drei unterschiedliche TFIIH Komplexe. Core TFIIH: XBP (3'-5'Helicase) Der Core TFIIH Komplex ist das Homolog zur XPD (5'-3'Helicase) Core Polymerase der Prokaryoten. p44, Tfb1, Tfb2 Holo TFIIH 1 (Promoter Clearance): Die CTD Kinase besteht aus zwei UE Core TFIIH+CTD Kinase (CDK7/CycH). CDK7 ist mit (CDK7/CycH) Zellzykluskinasen, CycH mit Zyclinen verwandt. Die Kinase ist für die Phosphorylierung der CTD der RNA Polymerase verantwortlich. Holo TFIIH 2 (DNA-Reparatur): Es gibt einen Zusammenhang zwischen Core TFIIH+Reparaturproteine Transkription und Reparatur, stark (Rad, XPG) transkribierte Bereiche werden schneller repariert als schwach transkribierte. Der Holo TFIIH 2 Komplex dient der Initiation und Durchführung der Reparatur von DNA. 45 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert TFIIH Komplexe bei der promoter clearance und DNA Reparatur Bei der Initation liegt der Komplex als Holo TFIIH 1, bei der Elongation als Holo TFIIH 2 zwecks Reparatur vor. Erreicht die Polymerase im Zuge der Transkription einen beschädigten DNA Bereich, kommt es durch den Komplex aktiv zur Rekrutierung eines größeren Reparaturkomplexes. Phosphorylierung der CTD durch TFIIH und die P-TEFb Kinase reguliert die Bindung von Proteinen, die an der mRNA Prozessierung beteiligt sind Die CTD (carboxyterminale Domäne) wird je nach Phase der Transkription durch die Kinase des Holo TFIIH I Komplex unterschiedlich Phosphoryliert und bindet entsprechend verschiedene Proteinkomplexe, wichtige Phosphorylierungsstellen sind Serine 2 und 5. Die variable Phosphorylierung der CTD erklärt das Assoziieren von Komplexen die an Elongation, RNA Prozessierung und Termination der Transkription beteiligt sind. Ist die RNA Pol II an den Transkriptionsstart gebunden, stimuliert ein Mediatorkomplex die Kinaseaktivität des TFIIH, die CTD wird an Ser5 phosphoryliert. Dadurch können Elongationskomplex und mRNA Cappingkomplex binden. Es kommt zur Promotor clearance und transkription des 5' Endes, gefolgt von einer kurzen Pause in der der mRNA CAP synthetisiert wird. Nach dem Capping wird Ser2 des CTD durch CDK9 phosphoryliert, wodurch die Elongation beginnen kann. Die CTD kommt nur in der RNA Pol II vor, sie kann daher stärker reguliert werden als andere Polymerasen. 46 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bildung einer mRNA CAP Struktur Das γ-Phosphat einer mRNA wird durch eine Triphosphatase gespalten. Mittels Guaninyltransferase erfolgt ein dirigierter nucleophiler Angriff des ß-Phosphats der mRNA auf das α-Phosphat eines Guanins wodurch eine 5'-5' Phosphodiesterbindung entsteht. Abschließend wird das Guanin wird an Position 7 methyliert. Diese CAP Struktur schützt einerseits die mRNA vor Abbau durch Nucleasen vom 5' Ende her und spielt andererseits in der Translationskontrolle eine Rolle. mRNA Termination und Polyadenylierung Die Termination in Eukaryoten unterscheidet sich von der in Prokaryoten. Die transkribierte mRNA weist am 3' Ende nach dem codierenden Teil des Gens spezielle Sequenzen auf (sog. Polyadenylierungssequenzen), an welche die Faktoren CstF und CPSF binden können. Diese Faktoren sind zunächst mit der wiederum spezifisch phosphorylierten CTD assoziiert und wechseln aufdie Poly A Sequenzen. CstF schneidet die mRNA los, es entsteht ein 3' Ende an das mittels CPSF eine spezielle Polymerase binden kann. Diese Poly A Polymerase (PAP) fügt bis zu 200 A Nucleotide an das 3' Ende der mRNA an, bevor PAP und CPSF sich von der mRNA lösen. Am sog. Poly A Schwanz binden Poly A binding Proteins, die die Stabilität der Struktur erhöhen. 47 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Struktureller Aufbau RNA PolIII abhängiger Promotoren RNA Pol III transkribiert 5S rRNA sowie tRNAs und erkennt drei unterschiedliche Promotorsequenzen. Eine dieser Promotorsequenzen ähnelt dem RNA Polymerase II Promotor, die anderen beiden liegen bereits in der codierenden Sequenz. Deletionsanalyse des RNA Pol III abhängigen 5S rRNA Promoters ergibt, dass der kodierende Bereich wichtige regulatorische DNA Sequenzen beinhaltet. Deletionsanalyse: Das Gen für 5S rRNA wurde isoliert und systematisch zerschnitten und mittels Reportergenassay überprüft ob noch Transkription stattfand. Die Analyse ergab, dass das Gen auch noch transkribiert wurde, obwohl bereits ein Teil des Gens deletiert worden war. Der Promotor musste daher innerhalb des Gens liegen. Rolle der Transkriptionsfaktoren TFIIIA, B und C bei der Initiation RNA Pol III abhängiger Transkription von internen Promotoren Es gibt zwei unterschiedliche interne Promotoren. TFIII … Transkriptionsfaktor der RNA Pol III Promotor mit Box A und C: Erst wenn TFIIIA gebunden hat, kann TFIIIC binden. TFIIIC bleibt so lange gebunden, bis oberhalb des Transkriptionsstarts TFIIIB gebunden hat, an das wiederum RNA Pol III binden kann. Promotor mit Box A und B: Es bindet nur TFIIIC an beide Sequenzen, deren Bindung ebenfalls zur Rekrutierung von TFIIIB und weiters zur Bindung der RNA Pol III führt. 48 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Initation der Transkriptionvon RNA Pol I abhängigen Promotoren RNA Pol I Promotoren bestehen aus zwei Sequenzen. Einem upstream control element (UCE) und dem core promotor. Jeweils ein Transkriptions-faktor UBF1 bindet an UCE und core promotor und rekrutiert anschließend an beiden Sequenzen weitere Faktoren, die den SL1 Komplex bilden. Nur wenn beide Komplexe ausgebildet sind, kann die RNA Pol I am core promotor binden. Das TAATA-binding protein (TBP) kann an der Initiation der Transkription durch alle drei eukaryoten RNA Polymerasen beteiligt sein, auch wenn deren Promotoren keine TAATA-Sequenz beinhalten Das TBP findet sich in den Komplexen aller drei eukaryotischen RNA Polymerasen, auch wenn sich in den jeweiligen Promotoren keine TATAA Sequenz findet. Vermutlich wird die Fähigkeit des TBP DNA zu krümmen bei der Bindung aller drei RNA Polymerasen benötigt. 49 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Wie beeinflussen Trankriptionsfaktoren die Transkriptionsrate eines Gens? • • Aktivatoren müssen die Bildung des (P)IC und die Rekrutierung der RNA Polymerase fördern, Repressoren müssen sie unterdrücken. (P)IC ... (pre) initiation complex Es gibt eine Reihe von Möglichkeiten, wie die Bildung des (P)IC durch Transkriptionsfaktoren beeinflusst werden kann: - direkter Kontakt mit Komponenten des (P)IC. - indirekt Kontakt mit dem (P)IC über Kofaktoren oder weitere Transkriptionsfaktoren. - Beeinflussung der Chromatinstruktur. DNA liegt in Eukaryoten meist als DNA-Proteinkomplex vor, es gibt TF die mit den Proteinen der Chromatinstruktur interagieren und sie lockern oder festigen. Direkter und indirekter Kontakt zwischen Transkriptionsfaktoren und dem PIC Kontakt zwischen Transkriptionsfaktoren und RNA Pol II • • • Der (Prae) Initiationskomplex enthält hochmolekulare Proteinkomplexe aus vielen Untereinheiten, die als Kontaktstellen für Transkriptionsfaktoren dienen können. Ein solcher Komplex ist TFIID, der außer TBP die TBP-associated factors (TAFs) enthält. Ein weiterer ist der mit der RNA Pol II CTD assoziierte Mediator Komplex Zwischen Hefe- und humanem Mediatorkomplexen gibt es Nomenklaturunterschiede, weil Proteine durch unterschiedlcihe Versuchsreihen und Experimente entdeckt wurden. Die Menge der UE des Mediatorkomplexes ergibt sich daraus, dass so mit einer hohen Anzahl weiterer unterschiedlicher TFs Interaktionen eingegangen werden können. Dadurch kann eine ausreichend unabhängige Regulation der einzelnen Gene im Genom ermöglicht werden. 50 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Viele Transkriptionsfaktoren, binden weit entfernt vom Transkriptionsstart an 'enhancer' Elemente. Sie können die Bildung des PIC durch direkten Kontakt beeinflussen, wenn die zwischenliegende DNA flexibel ist und die Bildung einer Schlaufe erlaubt. Multiple Kontakte zwischen Transkriptionsfaktoren und Komponenten des PIC am Beispiel des spezifisch in der Lebertranskribierten TTR Gens Im Vergleich zur Anzahl der Gene exisitert eine relativ kleine Anzahl unterschiedlicher TF. Um die Gene unterschiedlich regulieren zu können, werden die TFs in unterschiedlicher Kombination miteinander zur Genregulation eingesetzt. Zu diesem Zweck finden viele gleichzeitige Interaktionen statt. DNA ist flexibel und liegt in Schlaufen vor, daher können auch weiter entfernte Stellen Interaktionen mit dem Komplex eingehen (Enhancer Sequenzen). Wie beeinflussen Transkriptionsfaktoren oder Repressoren die Bildung des Initiationskomplexes? • • • Direkte Interaktion mit basalen Transkriptionsfaktoren Interaktion via Cofaktoren/Coaktivatoren (TAFs, Mediatorproteine, andere) Beeinflussung der Chromatinstruktur: - Binden von Coaktivator/Corepressor-Komplexen mit enzymatischer Aktivität zur Modifikation von Histonen. Modifikationen ... Hinzufügen chemischer Gruppen (Phosphorylierung, Methylierung, Acetylierung) - Umlagern oder Auflösen von Nukleosomen (Chromatin remodelling). 51 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Elektronenmikroskopische Darstellung von mitotischer und Interphasen DNA Rechtes Bild … Chromosom in Metaphase ... maximale Verpackung (1m auf 0,1µm kompaktiert). Übergeordnete DNA Strukturen Das Nukleosom • • • Im Nukleosom sind 146bp DNA knapp 2 mal um ein Histon-Oktamer gewunden. Das Histon ist ein oktamerer Proteinkomplex aus 2x4 UE (2 x H2A, H2B, H3, H4). Zwischen zwei Nukleosomen befindet sich ein DNA 'spacer' von-je nach Species 15 bis 55bp Länge. Histon H1 kann im Bereich der Spacer DNA binden und eine kompaktere Anordnung der Nukleosomen bewirken. Das Protein H1 ist nicht Teil des Nucleosoms, es bindet an DNA und Histon und ist für die Kompaktierung von 10nm auf 30nm Struktur verantwortlich (so richtig?). 52 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Struktur der am Aufbau des Nukleosoms beteiligten Histone H2A, H2B, H3 und H4 Die Strukturen der einzelnen UE sind sich sehr ähnlich. Die N-terminalen Enden der einzelnen UE ragen aus dem Histon heraus und sind so für Modifikationen zugänglich. Geordneter Aufbau eines Nukleosoms Die Bildung eines Nucleosoms läuft geordnet ab, zunächst bindet ein H3/H4 Tetramer an die DNA, anschließend binden zwei H2A/H2B Dimere. Die Anlagerung von H1 führt zu einer kompakteren Struktur nukleosomaler DNA Die Anlagerung von H1 bringt benachbarte Histone so nah zusammen, dass deren N-terminale Enden miteinander interagieren können. Diese beiden Effekte zusammen ergeben die 30nm Struktur. 53 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Was ist Chromatin Remodelling? • Jede Änderung einer vorgegebenen Chromatinstruktur: - Überführung von Eu- in Heterochromatin oder umgekehrt - Auflösen, Bildung oder Umlagerung von Nukleosomen - Einfügen varianter Histone - Chromatin remodelling ist eng an die postranslationale Modifikation von Histonen gekoppelt. Zusätzliche Literatur zum Thema Chromatin: Cell 128: Heft vom 23. Feb. 2007 Remodelingkomplexe müssen in der Lage sein, die Tätigkeit im zweiten Punkt durchzuführen. Histone Variants Involved in Transcription Regulation Histone H3 Variant Forms H3.3 Role(s) in Transcription Transcription activation X chromosome inactivation Localization Structural Features Transcribing region Inactive X chromosome Different from canonical H3 in only four amino acids. H2A macroH2A C-term nonhistone-like region is responsible for most of functions; histone-fold prevents sliding; prefers to form hybrid nucleosome. H2AZ Transcription Promoter, Loop1 differs from H2A, disfavors activation/ heterochromatin formation of hybrid nucleosome; repression boundary C-term α helix is essential for recognition. H2ABbd Transcription Active X Lack of C term; it only organizes activation chromosome 118–130 bp pf DNA and leaves and autosomes each side 10 bp free DNA. H2A.X Repression Canonical in A conserved C-term SQ(E/D) motif yeast, generally that becomes phosphorylated upon distributed DNA damage. Histonvarianten kommen nur in speziellen Bereichen der DNA vor. Diese Varianten werden nicht bei der normalen Bildung von Nucleosomen eingebaut, sondern werden durch spezielle remodelling complexe in bereits bestehende Nucleosome eingefügt. Veränderung der DNA Eigenschaften durch variante Histone Die Histonvarianten H3.3 findet man in transkriptionell aktiven Genen. Die H2AZ Variante in Promotoren oder boundary Elementen. Möglicherweise verhindern sie die Bildung von Heterochromatin. Der Austausch varianter Histone erfolgt durch remodelling complexes (z.B. Tip60 Komplex) 54 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Biochemische Aktivitäten von Chromatin Remodelling Komplexen Chromatin remodelling … DNA liegt in einer bestimmten Form Chromatin vor. Soll ein hier liegendes Gen exprimiert werden, muss die Chromatinstruktur verändert werden. Es kann Eu- in Heterochromatin und umgekehrt überführt werden, sowie lediglich bestimmte Nucleosome umgelagert werden. (a) DNA gleitet am Octamercomplex entlang wodurch Transkriptionsstellen freiwerden. (b) Transfer von Nucleosomen (c) Drehung der DNA vom Nucleosom weg Chromatin Remodelling Komplexe Type SWI / SNF Subunit No Bromodomain /Chromodomain Slide Transfer Restructure 8-11 Bromodomain Yes Yes Yes Kann direkt mit RNA Polymerase assoziiert sein. ISWI 2-4 No Yes No No Mi2 / NuRD 8-10 Chromodomain Yes No No Bromodomain / Chromodomain … Vermittelt die Bindung des Komplexes an Histone. Remodelling Komplexe erkennen Nucleosomen nur dann, wenn die Histone passend modifiziert sind. Rotational und Translational Positioning der DNA um das Nukleosom bestimmen die Zugänglichkeit von Bindungsstellen für Transkriptionsfaktoren Die Positionierung der DNA in Bezug auf die Histone ist in der Hinsicht wichtig, ob die Transkriptionsbindungsstelle nach außen weist und zugänglich ist oder nicht. Rotational positioning beschreibt eine Drehung der DNA im Hinblick auf die Octameroberfläche. Translational positioning … Histone können so verschoben werden, dass unterschiedliche Teil der DNA als Spacer DNA (und damit darin liegende Bindungssequenzen) zugänglich wird. 55 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Enzymatische Modifikation der Histone N-Termini durch Phosphorylierung, Acetylierung und Methylierung. Die N-Termini der Histone ragen aus dem Nukleosom und sind von außen her zugänglich Acetylierung und Deacetylierung von Histonen Histone weisen eine Vielzahl an Lysinen und Argininen auf, die im unmodifizierten Zustand bei neutralem pH positiv geladen vorliegen. Da DNA durch die Phosphatgruppen bei neutralem pH negativ geladen vorliegt, finden sich eine starke elektrostatische Anziehung zwischen DNA und Histonen die eine kompakte Struktur erzeugt. • Histon-Acetyltransferasen (HATs) neutralisieren die positive Ladung von Lysinen. Daher wird die Interaktion mit DNA, geschwächt, die nukleosomale Struktur aufgelockert. Der Promoter wird für Transkriptionsfaktoren zugänglicher. • Histon-Deacetylasen (HDACs) bewirken einen gegenteiligen Effekt und daher transkriptionelle Repression eines Gens. Different Classes of Modifications Identified on Histones Die wichtigsten modifizierbaren AS in Histonen sind Lysin (Acetylierung, Methylierung), Serin (Phosphorylierung) und Arginin (Methylierung). Chromatin Modifications Acetylation Residues Modified K-ac Methylation (lysines) Methylation (arginines) Phosphorylation K-me1 K-me2 K-me3 R-me1 R-me2a R-me2s S-ph T-ph Ubiquitylation Sumoylation ADP ribosylation Deimination Proline Isomerization K-ub K-su E-ar R > Cit P-cis > P-trans 56 Functions Regulated Transcription, Repair, Replication, Condensation Transcription, Repair Transcription Transcription, Repair, Condensation Transcription, Repair Transcription Transcription Transcription Transcription V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Modifikationen der Histone und ihre Korrelation zur Transkription des korrespondierenden Genlocus Histon Code Hypothese: Je nachdem in welcher bestimmten Art die N-Terminis eines Histons modifiziert sind, ergeben sich unterschiedliche zugeordnete Auswirkungen auf die Funktion des Histons. Ist beispielsweise der H3 N-Terminus nicht modifiziert, findet keine Transkription statt, das gebundene Gen ist stillgelegt. Ist er Terminus an Lysin 14 modifiziert, ist das Gen aktiv. Ist es an Lys9 methyliert, wird Heterochromatin gebildet und dadurch das Gen nicht nur abgeschaltet sondern auch die DNA Struktur zusätzlich kompaktiert. Ist es doppelt phosphoryliert, dient das der Chromosomenkondensation während der Mitose. Ist es phosphoryliert und acetyliert, dient das der Expression des Gens usw. Histone modifying enzymes 57 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) HAT Complex Subunit No Catalytic Subunit SAGA PCAF NuA3 NuA4 P300/CBP 15 11 3 6 1 GCN5 PCAF SAS3 ESA1 P300/CBP HDAC Complex Subunit No Catalytic Subunit Sin3 Complex NuRD SIR2 Complex 7 9 3 HDAC1/HDAC2 HDAC1/HDAC2 Sir2 Histone Methylases SUV39 SET1 PRMT Target Histone H3 (Lysine 9) H3 (Lysine 4) H3 (Arginine 3) Bromo/ Chromodomain Bromo Bromo Chromo Bromo Sonnbert Target Histone H3, H2B H3, H4 H3 H4 H2A,H2B,H3,H4 Bromo/ Chromodomain Chromo - Bromo/ Chromodomain Chromo - Bestimmung der Histonacetylierung durch Chromatin-Immunopräzipitation (ChIP) Die Analyse dient dazu, DNA Bereiche zu isolieren, deren Histone in einer Zelle acetyliert vorliegen. Zunächst wird die DNA des Zellkerns chemisch behandelt, um eine Lösung von DNA und Histonen zu verhindern ("cross linking"). Anschließend werden die Chromosomen mittels Ultraschall (Scherkräfte) in 300 bis 1000bp lange Stücke zerteilt. Die Stücke werden mit speziellen Antikörpern versetzt, die spezifisch an acetylierte Histone binden. Das Gemisch wird weiters mit einer Verbindung versetzt, die an den Antikörper binden und dadurch dem Komplex zusätzliches Gewicht verleihen. Unter Immunipräzipitation versteht man das Isolieren dieses schwereren Komplexes durch Zentrifugation. Anschließend wird der isolierte Komplex durch bestimmte Bedingungen so weit aufgelöst, dass die bisher gebundene DNA frei vorliegt. Abschließend wird mit der isolierten DNA PCR durchgeführt, wobei Primer verwendet werden, die auf Promotoren abzielen, um festzustellen, welche Gene acetyliert vorlagen. 58 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Modifikationen der Histone können durch Proteine mit Bromo-und Chromodomänen biologisch umgesetzt werden Eine Bromodomäne bindet spezifisch an acetylierte, eine Chromodomäne spezifisch an methylierte Histone. Über diese beiden Domänen können weitere Proteine spezifisch mit acetylierten oder methylierten Histonen interagieren. Wird ein Histon acetyliert, wird dessen Interaktion mit der DNA geschwächt. Weiters kann dieses Histon von Chromatin Remodelling Komplexen mit Bromodomäne erkannt werden, die die Struktur weiter lockern oder das Nucleosom verschieben. Die Methylierung von Histonen kann Transkription ermöglichen wie verhindern, abhängig davon, welches Lysin bzw. Arginin methyliert wird. Mit modifizierten Histonen interagierende Proteine und ihre Bindungsdomänen Histonmodifikationen regulieren alle biologischen Prozesse an der DNA 59 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Rekrutieren von histonmodifizierenden Enzymkomplexen erfolgt durch transaktivierende und transreprimierende Domänen Zusammenwirken von Transkriptionsfaktoren, Chromatin Remodelling Komplexen und Histonmodifizierenden Komplexen bei der Aktivierung der Transkription 60 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Transkriptionsfaktorbindung kann Voraussetzung oder Konsequenz von Chromatinremodelling und/oder Modifikation sein Ad (a) Ein Aktivator bindet an das kompakte Chromatin und dient dazu, einen HAC zu rekrutieren, der wiederum die folgenden Histone acetyliert und dadurch das kompakte Chromatin auflockert, wodurch TF binden und die Transkription initiieren können. Ad (b) Ein Aktivator bindet an das kompakte Chromatin und dient dazu, einen chromatin remodelling complex zu binden, der in der Lage ist, die Nucleosome so zu verändern, dass TF Zugang zum Promotor erhalten. In diesem Beispiel sind beide Komplexe nötig. DNA binding Protein 1 rekrutiert den chromatin remodelling complex, der das Nucleosom soweit verändert, dass DNA binding protein 2 binden kann, das wiederum HAC binden kann, das die Histone acetyliert und die Struktur auflockert. Chromatin remodelling kann zur kooperativen Bindung von Transkriptionsfaktoren notwendig sein Kooperative Bindung … die Bindung eines Faktors erleichtert bzw. ermöglicht erst die Bindung eines zweiten Faktors. Ad (c) … Die Bindung von Faktor A ermöglicht die Bindung eines Remodelling Komplexes, durch dessen Aktivität die Bindungsstelle für Faktor B frei wird. Ad (d) … Der Faktor A dient selbst dem Remodelling, indem es DNA vom Histon löst und dadurch die DNA Bindungsstelle für den Faktor B freisetzt. 61 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bildung eines Enhancesomes am Beispiel des IFN-beta Promoters Die nucleosomale Struktur muss so verändert werden, dass ein Initiationskomplex überhaupt binden kann. Das Nucleosom wird zugunsten eines sog. Enhanceosoms verschoben, dieses bietet Bindungsstellen für RNA Pol Proteine. Alle Proteine gemeinsam bilden das Enhanceosom, das eine Krümmung der DNA bewirkt, damit RNA Pol binden kann bzw. TFs leichter an die DNA binden können. Interferone (IFN) sind Substanzen, die das Immunsystem produziert, um die Replikation von Viren in Zellen zu unterdrücken. IFN-ß soll daher nur dann gebildet werden, wenn eine Zelle infiziert ist. Rekrutierung von histonmodifizierenden und chromatinremodellierenden Komplexen durch das IFN-beta Enhancosom Rekrutierung von Koaktivatoren, GTFs und RNA Pol II durch den Transkriptionsfaktor PPARγ 62 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Im Promoter des HO Gens (S. cerevisiae cerevisiae) führt die die Bindung von SWI5 zur Auflösung der Nukleosomenstruktur über der SBF Bindungsstelle. Hierdurch kann SBF binden und RNA Pol II über Assoziation mit Mediator dem Promoter zuführen. SWI5 dient sowohl der Rekrutierung eines chromatin remodelling complexes als auch eines HAC, wodurch Bindungsstellen im Promotor des HO Gens frei werden. Im Falle des MMTV Promoters bewirkt die Bindung des Glucocorticoidrezeptors (GR) an nukleosomale DNA eine Änderung des rotational positioning, sodass die Bindungsstellen für NF1 und weitere GR zugänglich werden. MMTV ist ein Retrovirus, das sich in Mäusen in das Genom integrieren und unter entsprechenden hormonellen Bedingungen Krebs hervorrufen kann. In der linear vorliegenden Form der DNA kann der GR nicht binden, ist die DNA des Virus in ein Nucleosom verpackt, kann GR binden. Durch diese bindung findet rotational positioning statt und der TF NF1 kann mit der DNA interagieren und eine Transkription einleiten. Wirkungsweise von transkriptionellen Repressoren Ad (d) … Ein an ein Nucleosom gebundener Repressor kann eine HDAC rekrutieren, die durch Deacetylierung von weiteren Nucleosomen die Chromatinstruktur kompakter und dadurch inaktiv werden läßt. 63 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) - Sonnbert Während der Elongation der Transkription wird die Nukleosomenstruktur nicht vollständig aufgelöst. Das Histon Oktamer löst sich kurzfristig von der DNA um Transkription durch RNA Pol zu ermöglichen. Zusammenspiel der Proteine Fact und PAF während der transkriptionellen Elongation nukleosomaler DNA Epigenetische Genregulation Epigenetic: describing mitotically heritable but reversible modifications to DNA or chromatin that lead to changes in gene expression in the absence of genetic alterations. • Ist Genregulation, die bei identischer DNA in unterschiedlichen Zellen/Organen unterschiedliche und mitotisch stabile Genexpression erzeugt. • Wird bewirkt u.a. durch die Chromatinstruktur und durch DNA –Methylierung. Unter epigenetischer Genregulation versteht man Regulation, die nicht direkt über die DNA Sequenz erfolgt sondern z.B. durch DNA Methylierung bei genetic imprinting. 64 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Eu- und Heterochromatin • • • Euchromatin enthält transkribierte Bereiche der DNA Heterochromatin - ist der transkriptionell inaktivere Bereich des Genoms mit dichter Packung des Chromatins. - kann konstitutiv sein (Telomer- und Centromerbereiche) oder induziert (XChromosom Inaktivierung; Silencing der mating-type loci in Saccharomyces). - die Verpackungsform der DNA ist nicht in jedem heterochromatischen Abschnitt gleich - niedriger Gehalt an acetylierten Histonen. Spezielle DNA Sequenzen (locus control region (LCR), insulator, boundary elements) regulieren die Eingrenzung der Eu- und Heterochromatin Bereiche. Chromosomale Domänen definieren transkribierte und nichttranskribierte Bereiche Insulator: 'isoliert' Heterochromatin von Euchromatin MAR = matrix-attachment region LCR = locus control region Chromosomale Domänen bezeichnen Abschnitte auf dem Chromosom, die entweder als Eu- oder als Heterochromatin vorliegen, die Domänen sind durch "Insulators" voneinander getrennt. LCRs dienen der epigenetischen Kontrolle der Transkription aller Gene innerhalb einer Domäne. Insulators können einem LCR dienen, indem sie Enhancer Sequenzen eines Gens stillegen. Ein Beispiel für LCR sind die unterschiedlichen Hämoglobinklassen, die in den verschiedenen Stadien der Embryogenese exprimiert werden. Hämoglobine eines Stadiums müssen vollständig abgeschalten, die des nächsten Stadiums vollständig aktiviert werden. Führt man in die LCR eines Clusters eine Deletion ein, wird der gesamte Cluster nicht mehr exprimiert. Isolator (Insulator) Elemente 65 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Methylierung des H3K9 durch die Suv39 Methyltransferase bewirkt Bindung des Heterochromatinproteins HP1 • • Die SET Domäne von Suv39 enthält Methyltransferase Aktivität. Die Chromodomäne von HP1 erkennt methyliertes H3. Bildung von Heterochromatin am Beispiel der Rap/Sir Proteine aus Saccharomyces Im Telomerbereich der DNA von Hefe bindet das Protein Rap1, das selbst einen Sir2/3/4 Proteinkomplex bindet. Sir2 ist eine HDAC, die naheliegende Histone deacetyliert. An diesen Histonen können in weiterer Folge weitere Sir2/3/4 Komplexe binden, die in ausreichender Menge miteinander Binden können und so eine kompakte DNA Struktur bilden. Heterochromatinbildung durch Drosophila Polycomb (Pc-G) Proteine Pc-G Proteine in Drosophila dienen der dauerhaften Aufrechterhaltung einer bestehenden Repression. Pc-G Mutanten können den Repressor nicht mehr binden, es kommt zu keiner Akkumulierung von Pc-G und es kann in weiterer Folge Transkription stattfinden, wenn der Repressor verloren geht. 66 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Modelle für die reprimierende Wirkung (gene silencing) durch Polycomb (Pc-G) Proteine Prozesse im Zellkern sind kompartimentiert (factories): dynamische Interaktion mit dem Nucleoplasma und Konzentration der beteiligten Proteine. Einfluss von DNA Schlaufen (loops) auf die Transkription • • • LCRs beeinflussen Transkription von einem oder mehreren Genen durch direkte Interaktion zwischen an LCR gebundenen proteinen und an enhancer gebundenen Proteinen. Interaktion der 3' und 5'Enden erklärt den Einfluss von Terminantionsfaktoren auf die Initiation der Transkription. Interaktionen von boundary (insulator) Elementen können die zwischenliegenden aktiven bereiche von Heterochromatin isolieren. 67 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Interchromosomale Genregulation In einigen Beispielen konnte eine durch Differenzierungs-prozesse gesteuerte Interaktion zwischen Genloci auf unterschiedlichen Chromosomen beobachtet werden, die mit veränderter Genexpression korreliert ist (Erythrocyten, Lymphocytendifferenzierung). 02.05.2006 Genregulation durch DNA Methylierung • • Die meisten Cytosinbasen innerhalb einer CpG Sequenz sind in Säugerzellen am C5-Atom methyliert. Eine Ausnahme bilden die CpG islets, die man oft in den Promotoren von housekeeping Genen findet. Methylierung von DNA korreliert meist (nicht immer) mit transkriptionell inaktiver DNA. DNA Methylierung findet sich sowohl in Pro- als auch in Eukaryoten. In Prokaryoten dient die Methylierung der DNA der Markierung der eigenen DNA, nicht korrekt methylierte DNA wird durch Restriktionsenzyme geschnitten. Demgegenüber dient die Methylierung in Eukaryoten der Genregulation. ad 21.30 … CpG Sequenzen sind rot markiert. Methyliertes Cytosin ist chemisch instabil, durch Deaminierung entsteht aus einem Cytosin ein Uracil. Uracil paart mit Adenin, eine nicht reparierte Deaminierung führt damit eine Mutation ein. CpG Sequenzen finden sich entgegen der Statistik damit nur dort gehäuft im Genom, wo sie einerseits essentiell sind oder andererseits nicht methyliert vorliegen. Nicht methyliert liegen sie in so genannten Housekeeping Genes vor, die laufend transkribiert aber wenig reguliert werden. ad 19.51 … Die "de novo Methylase" Methyliert CpG unabhängig von der DNA Replikation, ist im Zuge der Embryogenese aktiv und inaktiviert im Zuge der Entwicklung nicht mehr benötigte Gene. Die "perpetiation Methylase" fügt im Zuge der DNA Replikation eine Methylierung am neu synthetisierten Strang ein, wenn der ursprüngliche Strang eine Methylierung aufweist und erhält so eine Inaktivierung aufrecht. DNA Demethylasen entfernen Methylierungen z.B. in Gameten, um für Embryogenese wichtige Gene erneut zu aktivieren. Bei der Inaktivierung von Genen durch Methylierung der DNA handelt es sich um epigenetische Genregulation. 68 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert DNA Methylation and transcriptional repression (1) Direct interference von transcription activation factor binding. a) active Transcription [Bild] b) Repression by inhibition of TF binding [Bild] (2) specific transcriptional repressors a) active Transcription Biologische Funktion der DNA Methylierung • • • DNA Methylierung korreliert mit 'gene silencing' aber die Faktenlage (z.B. MeCPkos, DNAMethylaseko) spricht gegen eine direkte kausale Beziehung. Dieselben Mauskos sprechen dafür, dass DNA Methylierung wichtig für das Stilllegen fremder DNA ist (Retrotransposonen, Retroviren, Transgene). Z.B. führt die Abwesenheit von DNA Methylierung zur Aktivierung endogener Retroviren. DNA Methylierung ist positiv mit gene silencing von 'imprinted' loci. In einem Beispiel INHIBIERT Methylierung die Expression der AIR ncRNA, die 'silencing der Gene des 'imprinted' Locus in cis bewirkt. DNA Methylierung ist am Phaenomen des genomic imprinting beteiligt Unterschiedliche imprinted loci unterscheiden sich dadurch, dass entweder das paternale oder das maternale Allel methyliert werden. Bestimmte maternale und paternale Allele liegen in unterschiedlicher Methylierung vor. Es ist immer jeweils ein Allel methyliert und damit inaktiv, um die Gendosis zu kontrollieren, da eine Transkription von beiden Allelen negative Auswirkungen z.B. auf die Embryogenese hätte. Gameten enthalten nur den einfachen Chromosomensatz, damit imprinting korrekt weitergegeben wird, werden alle männlichen Allele durch imprinting deaktiviert, während imprinting in allen weiblichen Allelen entfernt wird. Dadurch ist sichergestellt, dass in einem Embryo erneut ein aktives und ein inaktives Allel vorliegen. 69 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Genomic Imprinting: Methylierung der imprinting control region des paternalen Allels bestimmt die Bindung des CTCF Proteins und damit die Expression der Gene Igf2 und H19. Das maternale Allel liegt nicht methyliert vor, das Isolatorprotein CTCF kann binden. Dadurch wird verhindert, dass die TFs an der Enhancersequenz auf das Gen Igf2 wirken. Gen Igf2 wird nicht transkribiert, der Enhancer wirkt stattdessen auf das Gen H19, das transkribiert werden kann. Das paternale Allel liegt methyliert vor, dadurch steht einerseits der Promotor des H19 Gens nicht für Transkription zur Verfügung, andererseits kann an der Isolatorsequenz das CTCF Protein nicht binden, die TFs an der Enhancersequenz wirken auf Gen Igf2, das transkribiert werden kann. 70 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Mechanismen zur Regulation der Transkriptionsfaktor Aktivität Genexpression kann durch Umweltfaktoren reguliert werden, in diesem Fall spricht man von Signaltransduktion. Die zugehörigen Transkriptionsfaktoren liegen nur dann in aktiver bzw. inaktiver Form vor, wenn die entsprechende Umweltsituation vorherrscht. Folgende molekularen Vorgänge können zur Aktivierung oder Deaktivierung von Transkriptionsfaktoren führen: • Phosphorylierung/Dephosphorylierung (Serin oder Tyrosin) Die am häufigsten vorliegende Modifikation, findet sich auch oft an der AS Threonin. • Proteolyse Durch Spaltung eines Proteins wird seine Aktivität beeinflusst. • Interaktion mit Partnerproteinen Es ist sowohl Aktivitätssteigerung als auch Deaktivierung möglich. • Binden intrazellulärer Liganden (z.B. Steroidhormone) Regulation durch Wechsel des Dimerisierungspartneres am Beispiel des bHLH Proteins Myo D Fehlen der basic region im Id Protein bewirkt mangelnde Affinität zur DNA im MyoD/Id Heterodimer 71 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Aktivierung der Transkriptionsfaktoren TCF (Elk1) und SRF durch Wachstumsfaktoren und den MAPK-Signaltransduktionsweg Serum (aus Rindern hergestellt) war früher beste Möglichkeit Zellkulturen zum Wachsen zu bringen, da es viele Wachstumsfaktoren enthält. Serum wird hergestellt, indem man Blut soweit erhitzt, dass die enthaltenen Zellen gerinnen und dabei Wachstumsfaktoren freisetzen, anschließend werden die Zellen durch Zentrifugation vom Serum getrennt. Das Wachstum (Größe, Zellteilung) von Zellen wird durch Bindung von Wachstumsfaktoren an unterschiedliche Rezeptoren an der Zelloberfläche reguliert. Die unterschiedlichen Rezeptoren vermitteln die Phosphorylierung der MAPK (mitogen activated protein kinase) im Zellinneren. MAPK phosphoryliert eine weitere Kinase (pp90rsk), beide Kinasen wandern in den Nucleus, wenn sie phosphoryliert sind. Im Kern phosphorylieren sie einen an SRE (Serum response element) gebundenen Transkriptionsfaktorkomplex (TCF + SRF), wobei TCF von MAPK, SRF von pp90rsk phosphoryliert wird. Erst wenn dieser Komplex phosphoryliert ist, kann Transkription damit verbundener Gene stattfinden. Die drei Haupt-MAPK Signaltransduktionswege (Erk, Jnk, p38MAPK) werden durch unterschiedliche Stimuli aktiviert (Wachstums bzw. Stresssignale) und verursachen die Phosphorylierung unterschiedlicher Transkriptionsfaktoren. Es sind drei vorherrschende Wege der MAPK Signaltransduktion bekannt. Wege 2 und 3 werden durch Stresssignale (UV, Veränderung osmotische Milieu, etc) ausgelöst. Weiters ist hier zu bemerken, dass es sich bei den Signaltransduktionswegen um Kaskaden handelt. Eine Kinase wird durch eine kleine GTPase (z.B. Ras) aktiviert (MAPKKK z.B. Raf) und aktiviert eine Vielzahl weiterer Kinasen (MAPKK z.B. MEK1), die ihrerseits wiederum viele Kinasen aktiviert (MAPK z.B. ERK … extracellular regulated kinase). Durch die Kaskade erhält man auch bei niedriger Konzentration extrazellulären Liganden bzw. Rezeptoren oder G Proteinen an der Zellmembran eine hohe Konzentration an aktiven TFs im Nucleus. 72 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) • • • Sonnbert Wachstumsfaktorrezeptoren sind Tyrosinkinasen. Bindung des Wachstumsfaktors verursacht Dimerisierung des Rezeptors. Dimerisierte Rezeptoren phosphorylieren sich gegenseitig und erhöhen somit ihre enzymatische Aktivität. Prinzipieller Aufbau eines Wachstumsfaktorrezeptors: Extrazelluläre Domäne zur Bindung des Wachstumsfaktors, transmembrane Domäne zur Verankerung in der Zellmembran, cytosolische Domäne mit katalytischer Sequenz, die bei Bindung des Wachstumsfaktors durch Konformationsänderung weitere Aktionen vermitteln kann z.B. Phosphorylierung weiterer Proteine, im abgebildeten Beispiel kommt es bei Ligandenbindung zunächst zu Dimerisierung und wechselseitiger Phosphorylierung, bevor die eigentliche Aktivität beginnt. Rezeptor-Phosphotyrosine sind Andockstellen für Signaltransduktionsproteine mit SH2 Domänen Proteine mit PTB- und SH2- Domänen erkennen Proteine, wenn diese an bestimmten Tyrosinen phosphoryliert sind und können dadurch Interaktion vermitteln. • • Aktivierte Wachstumsfaktorrezeptoren werden durch Adapterproteine mit dem guanine nucleotide exchange factor SOS verbunden. Das Ras Protein bindet G-Nukleotide und besitzt GTPase Aktivität. SOS sorgt für einen GDP-GTP Austausch. Durch Bindung des EGF (Epidermal growth factor) an Rezeptoren kommt es zur Bildung eines aktiven dimeren Rezeptorkomplexes, das phosphorylierte Tyrosine aufweist. Diese Tyr-P können durch Proteine mit SH2 Domänen erkannt werden, in diesem Fall durch GRB2. GRB2 weist eine weitere Domäne auf (SH3) über die es ein weiteres Protein (SOS) bindet. Über SOS kommt es zur Bindung an die membrangebundene GTPase Ras. Vor der Komplexbildung hat Ras GDP gebunden und ist in diesem Zustand inaktiv. SOS dient als GEF (guanine nucleotide exchange factor) und tauscht bei Bindung an Ras dessen GDP mit GTP aus, wodurch Ras in die aktive Form überführt wird. 73 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Regulation der Ras Aktivität durch guanine nucleotide exchange proteins (GEFs) und GTPase activating proteins (GAPs) GDP gebundenes Ras ist inaktiv, durch GEFs wird GDP mit GTP vertauscht und Ras in die aktive Form überführt. In aktiver Form kann es GAPs binden und aktiviert damit seinerseits GTPase Aktivität in den GAPs. Ras wird durch Spaltung des GTP in GDP + Pi inaktiviert. Diese Spaltung kann erfolgen, wenn Ras GTP über einen längeren Zeitraum gebunden hat und keinen Interaktionspartner findet. Hat aktives Ras hingegen ein GAP gebunden, erhöht sich durch einen allosterischen Effekt die GTPase Aktivität des Ras, wodurch es schneller in die inaktive Form überführt wird. Die MAPK Erk wird durch Phosphorylierung an sowohl Tyrosin als auch Serin durch die dual-specificity kinase Mek (=MAPKK) aktiviert Aktives Ras bindet das GAP Raf. Die GTPase Aktivität von Ras wird erhöht, wenn GTP in GDP gespalten wird, wird Ras in die inaktive und gleichzeitig Raf in die aktive Form überführt. Das aktive Raf aktiviert seinerseits das Protein MEK durch Phosphorylierung. MEK ist selbst eine Kinase und aktiviert nun die MAP Kinase durch Phosphorylierung an einem Tyr und einem Ser. Ein aktiviertes Raf kann viele MEK aktivieren, ein aktiviertes MEK kann eine Vielzahl MAPK aktivieren, man spricht in diesem Zusammenhang von einer Signaltransduktionskaskade. Weiters wird Raf als MAPKKK (MAPK Kinase Kinase) und MEK als MAPKK (MAPK Kinase) bezeichnet, da Raf an der dritten und MEK an der zweiten Stelle über der eigentlichen MAP Kinase stehen. 74 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Phosphorylierung des Transkriptionsfaktors Elk1 durch MAPK verstärkt die Interaktion mit dem Mediator Komplex Kooperation von MAPK Signaltransduktionswegen: - Der JNK-Signaltransduktionsweg verursacht die Phosphorylierung des c-Jun Proteins (bZip). Der Erk-Signaltransduktionsweg stimuliert die Synthese des c-Fos Proteins (bZip). Durch Dimerisierung beider Proteine entsteht der Transkriptionsfaktor AP-1 c-Jun und c-Fos sind Leucin Zipper Proteine. Sind beide durch Phosphorylierung aktiviert, können sie Heterodimere bilden und als TF dienen. 75 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Genregulation durch cAMP Als Beispiel für Signaltransduktion mittels der häufig vorkommenden 7-transmembran Rezeptoren. Diese Art Rezeptoren weisen sieben α-Helices auf, die dadurch mehrmals die Zellmembran durchqueren. Die Loops zwischen den Helices dienen extrazellulär der Bindung der Liganden während sie intrazellulär mit Signaltransduktionsproteinen interagieren. Eine Vielzahl an Botenstoffen des Immunsystems (Cytokine) sowie viele endokrine Hormone (z.B. Glucagon oder Adrenalin) benutzen diese Rezeptoren. Alle dieser Rezeptoren haben gemein, dass sie nach Ligandenbindung über ein ebenfalls membrangebundenes G Protein das membrangebundene Enzym Adenylatcyclase aktivieren, das im aktiven Zustand cAMP erzeugt. Phosphorylierung des bZip Transkriptionsfaktors CREB durch die cAMP-stimulierte Serinkinase PKA cAMP aktiviert unter anderem eine Proteinkinase, die in den Nucleus wandert und dort den DNA gebundenen TF CREB phosphoryliert. CREB liegt mittels Leucine Zipper an die DNA gebunden als Dimer vor und wird an Serine phosphoryliert. CREB kann im phosphorylierten Zustand mit weiteren Faktoren interagieren (CBP dient als Histonacetylase) und dadurch Transkription einleiten. CRE ... cAMP response element CREB. Seven transmembrane Rezeptoren stimulieren cAMP Synthese durch trimere GProteine und Adenylatcyclase Struktur der Adenylatcyclase 76 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Mechanismus der Adenylatcyclase Aktivierung durch trimere G-Proteine Trimere G-Proteine bestehen aus α, β und γ-UE. Es existieren zwei Typen (stimulatorisch und inhibitorische GProteine; Acetylase wirkt stimulatorisch). Das G Protein der Adenylatcyclase hat im inaktiven Zustand GDP, im aktiven GTP gebunden. Das G Protein weist GTPase Aktivität auf, nach einer gewissen Zeit wird gebundenes GTP so in GDP + Pi gespalten und inaktiviert sich dadurch selbst. Mittels des β γ Dimers können seven transmembrane Rezeptoren auch den ERK Signaltransduktionsweg aktivieren. Bestimmte 7 transmembran Rezeptoren aktivieren über G Proteine eine MEK Kinase, die der Aktivierung von MEK dient. Diese Rezeptoren greifen somit auf den MAPK Pathway zurück, wobei sie eine Kaskade der MAPK Phosphorylierung umgehen (Raf). 77 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Viele Cytokinrezeptoren (z.B. für Interferone) verursachen Jak-Stat Signaltransduktion • • Stats sind die einzigen Transkriptionsfaktoren mit SH2 Domäne. Erst nach Dimerisierung können Stats in den Zellkern translozieren. Cytokine regulieren Zellen des Immunsystems. JAK (Januskinase) ... Familie aus vier unterschiedlichen Tyrosinkinasen. JAKs sind an Cytokinrezeptoren gebunden und phosphorylieren bei Cytokinbindung Tyrosine am cytoplasmatischen Teil der Rezeptoren. Der TF Stat (Signal transducer and activator of transcription) weist eine SH2 Domäne auf, über die phosphorylierte Cytokinrezeptoren gebunden und durch JAK selbst phosphoryliert werden können. Zwei phosphorylierte Stats können ein Dimer bilden, wandern in den Nucleus und dienen in dieser Form als Aktivator für ein Response element. Diese Antwort funktioniert sehr schnell, da aktivierte Stats sofort in den Nucleus wandern. Dieser Signaltransduktionsweg dient meist dem Immunsystem als Antwort auf eine Infektion. Stat SH2 Domänen dienen zum Andocken an Rezeptoren und zur Dimerisierung 78 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Der TGF-beta (SMAD) Signaltransduktionsweg • Cytokine der TGF-beta Familie besitzen Rezeptoren mit Serinkinase-Aktivität. Manche SMADs werden direkt durch den Rezeptor phosphoryliert. • Um in den Kern zu gelangen müssen sie mit SMAD4 (=common SMAD) dimerisieren. • Zur Genaktivierung muß das SMAD Dimer mit weiteren Transkriptionsfaktoren interagieren. TGF β ist ein vielfältiges Cytokin mit mehreren Funktionen z.B. Blutgerinnung. Cytokine der TGF ß Familie binden an den Rezeptor Typ II und aktivieren dadurch dessen SerinThreonin-Kinaseaktivität. Die Kinase phosphoryliert den Rezeptor Typ I, wodurch dessen Kinaseaktivität (ebenfalls Ser-Thr) aktiviert wird. Der Rezeptor Typ I kann die TFs SMAD3 bzw SMAD4 phosphorylieren. Sind diese phosphoryliert, können sie jeweils mit SMAD4 ein Dimer binden, das Dimer kann in den Nucleus wandern und dient dort der Aktivierung bestimmter Gene. Der TGF-beta (SMAD) Signaltransduktionsweg II Das Interaktom des TGF-β 79 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Transkriptionsfaktor NFκB wird durch Serin Phosphorylierung und Proteolyse des Inhibitors I-κB aktiviert. NFκB ist an einer Vielzahl immunologischer Prozesse beteiligt. In nicht stimulierten Immunzellen liegt NFκB mittels IκB inaktiviert vor. Durch Stress (UV-Bestrahlung, osmotische Fehlverteilung), Infektion oder Cytokine wird eine Kinase aktiviert, die IκB phosphoryliert. Durch die Phosphorylierung wird IκB für den Abbau markiert, durch den Abbau wird NFκB freigesetzt. Signaltransduktion durch Wnt Liganden Wnt Signaltransduktion ist sowohl entwicklungsbiologisch als auch im adulten Stadium von Bedeutung. Als Signalprotein dient ß Catenin. In der Zelle findet sich ohne aktivierten Pathway der GSK Proteinkomplex (rot, Glykogen Synthase Kinase), der ß Catenin durch Ser-Thr Phosphorylierung für den Abbau markiert. Im Wnt Pathway dient Wnt als extrazellulärer Ligand, der vom "frizzled" Rezeptor gebunden wird. Durch die Bindung von wnt an den Rezeptor kommt es intrazellulär zur Aktivierung des Proteins "Dishevelled", das in der aktiven Form den GSK Komplex inhibiert. Durch diese Inhibierung wird ß Catenin nicht mehr zum Abbau markiert und kann in den Nucleus wandern. Im Nucleus findet sich an Wnt-Pathway abhängigen Genen gebunden ein Proteinkomplex, der eine Histon-Deacetylase ("Groucho") beinhaltet und dadurch die entsprechenden Gene inaktiviert. Wandert ß Catenin in den Nucleus, bindet es an LEF/TCF (T cell factor) des Inhibitorkomplexes, wodurch wiederum die Histon-Deacetylase aus dem Komplex entlassen wird und der verbleibende Komplex als Aktivator dient. 80 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Notch Signaltransduktion Notch ist ein Rezeptor, der in entwicklungsbiologischen Prozessen eine Rolle spielt. Notch ist selbst an die Zellmembran gebunden und interagiert mit Liganden die sich ihrerseits in der Zellmembran befinden. Bei einer Interaktion des Rezeptors mit einem Liganden wird zunächst der extrazelluläre und anschließend der intrazelluläre Teil des Notch Rezeptors abgespalten. Das Peptid im Cytosol wandert in den Nucleus, wo es mit weiteren Proteinen einen Komplex bildet. Dieser Komplex ist in der Lage an bestimmte DNA Sequenzen zu binden und Transkription zu ermöglichen. Wichtig in diesem Zusammenhang ist, dass durch Notch aktivierte Gene durch einen Proteinkomplex gebunden sind, der eine Transkription der Gene inhibiert. Erst wenn Notch an diesen Komplex bindet, wird die Repressorfunktion dieser Proteine in eine Aktivatorfunktion umgewandelt. Aktivierung des SREBP durch Proteolyse • • • SREBP reguliert Gene, die zur Aufnahme und Synthese von Cholesterol benötigt werden. Der Sterolsensor SCAP reguliert die Proteolyse von SREBP. Proteolyse sorgt für die Freisetzung des aktiven Transkriptionsfaktors aus der Membran eines Vesikels des Golgi-Apparats. SRE ... Sterole response element, SREBP … SRE Binding Protein. SREBP wird inaktiv in der Membran des ER erzeugt und bleibt hier gebunden. In der Membran des ER finden sich Rezeptoren für Sterole (SCAP), die eine ebenfalls ER Membran gebundene Protease inaktivieren. Weist die Zelle eine niedrige Sterolkonzentration auf, wird die Protease durch SCAP nicht mehr inaktiviert und schneidet SREBP an einer bestimmten Stelle. Durch diese Proteolyse wird eine weitere Schnittstelle an SREBP zugänglich, durch eine zweite Protease wird ein ein Teil des SREBP im Cytosol freigesetzt. Dieser Teil kann in den Nucleus wandern und dort als TF zur Aktivierung von Genen mit SRE dienen. 81 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) - Sonnbert SREBP ist ein bHLH Protein. SCAP inhibiert seine Proteolyse in Anwesenheit von Sterolen. In Abwesenheit von Cholesterol erfolgt Proteolyse durch zwei aufeinander folgende Proteasen. 09.05.2006 Grundstruktur der Steroidhormone Die Grundsstruktur aller Steroidhormone entspricht dem Cholesterin. Zur Familie der Steroidhormone gehören unter anderem Cortison, Östrogen, Progesteron und Testosteron. Beispiele für Liganden intrazellulärer Rezeptoren aus der Familie der Steroidrezeptoren Steroidrezeptoren können auch Liganden binden, deren Grundstruktur nicht der von Cholesterin entsprechen, so z.B. Retinsäure (aus Vitamin A) und Thyroxin (Schilddrüsenhormon). Die Gemeinsamkeit dieser Liganden liegt in ihrer lipophilen Natur d.h. sie sind in der Lage, ohne Rezeptor durch die Zellmembran zu wandern und intrazelluläre Rezeptoren binden. Cortisol (ein Glucocorticoid) bindet an ligandbindende Domäne (LBD) des Glucocorticoid-Rezeptors und bewirkt die Dissoziation von Hsp90 Hsp90 verhindert, dass der Komplex in den Nucleus wandert. Durch seine lipohilen Eigenschaften kann Cortisol in die Zelle eindringen, die Bindung an den Komplex verdrängt Hsp90 und er kann in Kern wandert. 82 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Steroidhormon-Rezeptoren • • Können im Cytoplasma sein und dort ihren Liganden binden (Beispiel: Glucocorticoidrezeptor, GR). Können im Zellkern und an DNA gebunden vorliegen und dort ihren Liganden binden (Beispiele: RXR-Heterodimere wie TR/RXR, RAR/RXR, VDR/RXR). Grundstruktur der Steroidrezeptor Familie Gelb … strukturell verwandte, DNA bindende Domäne und C-terminal die Liganden-bindende Domäne. Die transaktivierende Domäne am Nterminalen Ende ist in ihrer Länge variabel. Die DNA Bindungsstellen von Transkriptionsfaktoren der SteroidhormonRezeptorfamilie • können entweder inverted repeats (Palindrome) oder direct repeats bilden. Es finden sich zwei verschiedene Arten Steroidhormonrezeptoren: • inverted repeats binden homodimere Rezeptoren (z.B. GR, ER). Die erste Art bindet als Homodimer palindromische Sequenzen auf der DNA d.h. sie binden DNA von beiden Seiten her. Beispiele sind Glucocorticoidrezeptor und Östrogenrezeptor. • direct repeats binden Heterodimere eines Rezeptors (z.B. TR) mit einem weiteren Mitglied der Rezeptorfamilie, RXR. Dabei entscheidet die Anzahl an Basenpaaren zwischen den repeats über die Spezifität. Die zweite Art bindet als Heterodimer zwei hintereinander liegende DNA Sequenzen. (c), (d), und (e) unterscheiden sich nur durch den Nucleotidabstand zwischen den beiden Bindungsequenzen. Beispiele sind der Vitamin D-, Thyroxin- oder der Reinsäure-Rezeptor, die jeweils gemeinsam mit RXR das Heterodimer bilden. Alle Rezeptoren dieser Art dienen selbst als TF. 83 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Die Bindung des Liganden Retinsäure (RA) and den Rezeptor (RAR; gleiches gilt für den T3 Rezeptor) bewirkt die Dissoziation eines Korepressor /HDAc Komplexes und die Assoziation eines Koaktivator/HAT Komplex. Ad (A) .... in Abwesenheit des Hormons bindet das Heterodimer einen Komplex mit Sin3. Sin3 deacetyliert Histone und dient damit ohne Hormon als Repressor der Transkription. Ad (B) ... Liegt Ligand an den Rezeptor gebunden vor, so ist das Heterodimer in der Lage einen HAT Komplex zu rekrutieren, der Histone acetyliert und dadurch DNA für Transkription zugänglich macht. Als abschließendes Beispiel Transkriptionskontrolle über Konzentrationsverteilung: Die Proteine MyoD und E12 weisen ein Helix-Loop-Helix Motiv auf, über das sie in der Lage sind, miteinander ein Heterodimer zu bilden. Sowohl MyoD als auch E12 besitzen weiters eine Region für DNA Bindung. Die Bindung an DNA ist allerdings nur dann möglich, wenn die beiden Proteine als Heterodimer vorliegen, eine einzelne DNA Bindungsdomäne ist für eine Bindung nicht ausreichend. MyoD hat nun die Fähigkeit, mit einem weiteren Protein, Id, ebenfalls ein Heterodimer zu bilden. Nachem Id aber keine Domäne für DNA Bindung aufweist, kann dieses Heterodimer nicht an DNA binden und folglich auch keine Transkription einleiten. Je nachdem in welcher Konzentration also E12 bzw. Id vorliegen, wird das aktive oder das inaktive Heterodimer mit MyoD vorliegen und Transkription stattfinden oder nicht. 84 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bläsi Foliensatz 1 Teil Bläsi: Translationskontrolle Genexpression II: Post-transcriptional Regulation Lit: Lewin / Genes VII - indicated Literature Content: Prokaryotes Eukaryotes Components of the translation machinery 30S/50S ribosomes tRNA Genetic code Translation initiation Elongation Termination mRNA stability protein transport mRNA processing 40S/60S ribosomes tRNAi Translation initiation Elongation Termination mRNA stability protein transport Wir erinnern uns: in Prokaryoten laufen Transkription und Translation gekoppelt ab, während sie bei Eukaryoten getrennt durchgeführt werden. Cistron … Gen. Unter polycistronischer DNA versteht man mehrere hintereinander liegende Gene, die gemeinsam auf eine mRNA transkribiert und erst durch die den einzelnen Genen zugehörigen ORFs (open reading frames) in die eigenständigen Genprodukte translatiert werden. Funktionell zusammengehörige Gene sind bei Prokaryoten in solchen so genannten Operons zusammengefasst. 85 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert In Eukaryoten findet Transkription und Prozessierung von mRNA im Kern statt. Die RNAs, die zunächst transkribiert werden, können nicht direkt in ein Protein translatiert werden. Sie müssen prozessiert werden, bevor sie ins Cytoplasma transportiert werden können, wo die Translation stattfindet. Der Exportschritt der mRNA vom Kern ins Cytoplasma ist ein wichtiger regulatorischer Schritt. Alle Komponenten der Transkriptionsmaschinerie müssen in den Kern transportiert werden. Gelangen bestimmte Transkriptionsfaktoren nicht hinein, können mit ihnen assoziierte Gene nicht transkribiert werden. Translation Systems Prokaryoten weisen im Vergleich mit Eukyaroten wenige Proteine bei der Initiation, aber mehr Proteine bei der Termination auf. Der eigentliche Prozess der Translation ist in Pro- und Eukaryoten relativ ähnlich, der Prozess ist hoch konserviert. Sie unterscheiden sich vor allem durch Regulationsmechanismen, Eukaryoten weisen hier mehr Faktoren auf. Ribosomal recruitment to mRNA in Pro-and Eukaryotes rRNA/mRNA protein/protein AGGAGGU ... Shine Dalgarnosequenz, das auf diese Sequenz folgende AUG dient als Startcodon für die Translation. (Reminder Codon … drei aufeinander folgende Nucleotide, die für eine Aminosäure oder ein Start bzw. Stopsignal codieren). 86 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translation in Prokaryotes Components Initiation Elongation Termination mRNA tRNA ribosomes IF 1-3 Regulatory mechanisms EF: EfTu, EFTs, EFG RF: RF 1-3 RRF Ein kurzer Überblick über die Translation in Prokaryoten: Transkripte in Prokaryoten sind in der Regel polycistronisch, d.h. sie enthalten mehrere Genen (= Cistrons). Die einzelnen Gene werden mittels Ribosomen in Protein translatiert. Ribosomen erkennen Gene an bestimmten, üblicherweise zu Beginn liegenden Sequenzen. Anm.: Im Gegensatz dazu wird bei vielen Viren die virale mRNA in ein Polyprotein umgesetzt, das anschließend so prozessiert wird, dass verschiedene funktionelle Proteineinheiten entstehen. Die Translation dient der Umsetzung des Transkriptes in Protein, man kann drei wichtige Stadien unterscheiden: Initation (geschwindigkeitsbestimmender Schritt), Elongation, Termination. • Bei der Initiation muss das Ribosom die Stelle finden, an der die eigentliche Proteinsequenz beginnt (AUG). Nachdem AUG nicht nur als Startcodon dient, sondern auch für die AS Methionin codiert, kann es mehrfach in einer mRNA vorkommen. Es muss daher in der Umgebung des AUG als Startcodon Signale in der Umgebung geben, durch die das Ribosom das Startcodon erkennen kann. In diesem Zusammenhang sind weiters Phosphorylierungsvorgänge nötig, damit das Ribosom an das Startcodon binden kann. • Während der Elongation werden die Codons der mRNA durch das Ribosom mittels tRNA in die zugehörigen AS umgesetzt und die aufeinander folgenden AS zu einem Polypeptid verknüpft. Auf dieser Ebene finden sich nur wenige Regulationsvorgänge. • Zur Termination kommt es, wenn das Ribosom zu einem Stopcodon gelangt, die nicht in AS übersetzt werden, da für sie keine tRNAs exisiteren. Im Zuge der Termination werden die UE des Ribosoms so zerlegt, dass sie im Anschluss erneut für Translation zur Verfügung stehen. transfer RNA is the adapter Initiator and elongator methionineaccepting tRNAs. Cloverleaf representation of methionineaccepting tRNAs: (A) initiator tRNA and (B) elongator tRNA. The regions important for initiator tRNA identity are highlighted. Jede tRNA bindet über das Anticodon das passend komplementäre Codon auf einer mRNA und stellt damit die Verbindung zwischen Codon und zugehöriger AS her. tRNA wird über das 3' Ende kovalent mit der passenden AS verbunden, eine so beladene tRNA wird als Amino-Acyl-tRNA bezeichnet. 87 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Chromosomal location of tRNA genes in E.coli tRNAs werden in der Regel gemeinsam mit anderen Teilen der Translationsmaschinerie transkribiert, sie liegen meist in der Mitte oder am Ende dieser DNA Sequenzen. Für die meisten AS gibt es mehrere unterschiedliche tRNAs (haben voneinander verschiedene Anticodons), die für sie kodieren. Die tRNA-Codierung in Bakterien ist weiters nicht einheitlich: Sie können einzeln im Chromosom codiert sein oder wie im rrnB Operon (E.coli) zusammen mit der 16S rRNA (Teil der 30S UE) oder der 23S rRNA (Teil der 50S UE) transkribiert und anschließend prozessiert werden. Beim rrnB Operon liegt eine tRNA (2Glu) zwischen der 16S und der 23S RNA, es wird somit eine tRNA gemeinsam mit zwei rRNAs transkribiert. Auf dem E.coli Chromosom finden sich insgesamt sieben vergleichbarer Operons, in jedem dieser Operons liegen einige der tRNAs. tRNA processing [red arrow] RNaseP determines 5'-end. [green arrow] RNaseD determines 3'-end, digests until CCA end is reached. In E.coli dienen die RNasen P und D der tRNA Prozessierung. RNAse P schneidet das 5' Ende, RNase D das 3' der tRNAs aus dem Gesamttranskript frei. tRNAs werden in Pro- und Eukaryoten ähnlich den rRNAs als große Vorläufer-RNAs synthetisiert. Diese Vorläufer-RNA werden anscheinend nach der Ausbildung ihrer Kleeblattstruktur am 5' Ende durch RNase P und am 3' Ende durch RNase D geschnitten. in • • • Prokaryoten: tRNAs werden oft an Nucleotiden modifiziert. tRNAs sind oft am 2' OH methyliert. alle tRNAs weisen am 3' Ende des Akzeptor-Armes eine ACC-Sequenz auf, über die zwei Arten unterschieden werden: o e, die ACC Sequenz wird durch das Gen der tRNA codiert. o l, die ACC Sequenz wird nachträglich durch Nucleotidyl-Transferase angehängt. 88 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert in Eukaryoten: tRNA weisen Introns auf, es ist splicing nötig, das sich vom normalen prä-mRNA splicing unterscheidet. Das Splicing ist ATP abhängig, eine Nuclease schneidet Introns heraus. Die freien Enden der beiden Exons werden durch eine Ligase verknüpft. tRNA charging Das 3' Ende der tRNA wird kovalent an die Carboxylgruppe der zugehörige AS gebunden. Die tRNA wird mit Hilfe von Enzymen, so genannten Aminoacyl-tRNA-Synthetasen, mit der passenden AS beladen; für jede tRNA existiert ein korrelierendes Enzym, das nur die passende AS verarbeitet. Die Synthetase erkennt die zugehörige tRNA über das Anticodon bzw. weiteren eindeutigen Nucleotidsequenzen und verknüpft deren 3' Ende kovalent mit der Carboxylgruppe der passenden AS. Die bereits an die tRNA gebundenen AS können nachträglich verändert werden, nachdem sich das Anticodon der tRNA dabei natürlich nicht ändert wird eine fehlerhafte AS eingebaut. 89 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Glutaminyl-tRNA-synthetase-tRNA-complex Green Arrow … Identity element for tRNAGlu. ad 2. Bild … eingekastelte Stellen sind Identifikationsstellen, die von Enzymen erkannt werden. Der Glutaminyl-tRNA-SynthetaseKomplex ist einer der ersten Komplexe, die röntgenstrukturanalytisch erfasst wurden. tRNA synthetases tRNA Synthetasen beladen tRNAs mit AS. Sie bestehen aus 4 Domänen, die jeweils unterschiedliche Funktionen wahrnehmen. Class I und Class II Synthtasen sind keine Homologe, sie unterscheiden sich beträchtlich. Funktionelle Abschnitte der Synthetase: - tRNA Bindungsdomäne (tRNA Akzeptorhelix) - katalytische Domäne für ATP Bindung - AS Bindungsstelle Class I and Class II Aminoacyl-tRNA synthetases 90 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Synthetasen liegen zwar meist als Monomere oder Homodimere vor, es sind aber auch Heterodimere und Heterotetramere bekannt. Man unterscheidet zwei Synthetase Klassen: Klasse I und Klasse II Synthetasen unterscheiden sich dadurch, auf welcher Seite die tRNA gebunden wird. Klasse I Synthetasen • liegen meist als Monomere vor, kommen aber auch als Homodimere vor. • kontaktieren tRNA über die kleine Furche des Akzeptorarms und am Anticodon, die jeweils in das Protein hineinragen. Klasse II Synthetasen • liegen vorwiegend als Homodimere vor, manche auch als Heterodimere. • kontaktieren tRNA über die große Furche des Akzeptorarms und am Anticodon, die jeweils in das Protein hineinragen. Steps during tRNA charging Die Synthetase enthält Stellen, die eine bestimmte AS sowie die über das Anticodon erkannte tRNA binden. Die Synthetase aktiviert die AS durch Bindung mit ATP, es entsteht ein AS-AMP. Erst jetzt kann ein dirigierter nukleophiler Angriff des 3' OH der tRNA auf die an das AMP gebundene Carboxylgruppe stattfinden und die AS durch Freisetzung von AMP kovalent an die tRNA gebunden werden. Die Synthetase ist in der Lage Fehler zu erkennen und die Bindung zwischen AS und tRNA zu lösen. Beladung einer tRNA: • Bildung von Aminoacyl-AMP • Veresterung der AS mit AMP • Bindung der tRNA an Aminoacyl-AMP • Veresterung der AS mit dem 3' Ende der tRNA 91 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Kinetic Proofreading Kinetic proofreading … Regulation über die Geschwindigkeit, mit der die Reaktion abläuft. "linker Weg" … die Synthetase weist Affinität für eine bestimmte ("ihre") tRNA auf. Die Assoziierung und Komplexbildung (tRNA mit Synthetase) erfolgt schnell, die Dissoziierung langsam. Durch Komplexbildung kommt es zu einer Konformationsänderung der Synthetase, die gebundene tRNA kann ebenfalls schnell mit einer AS beladen werden. "rechter Weg" … Synthetase hat eine schlechtere Affinität zu anderen tRNAs, die Bindung der tRNA ist schwach und führt zu keiner Konformationsänderung. Dadurch kommt es zu einer langsamen Bindung der AS an die tRNA, es besteht die Chance, dass die tRNA vor einer Bindung mit der AS den Komplex verlässt. Das Proofreading der Aminoacyl-tRNA-Synthetasen ist ausgezeichnet: Die Fehlerrate liegt bei nur 1:60000. Dafür gibt es die oben genannten Gründe: • nicht mit dem Enzym assoziierende tRNAs dissoziieren nach anfänglicher Bindung schnell wieder ab, da die Bindung zu unspezifisch ist. Die mit dem Enzym assoziierte tRNA bindet demgegenüber stark und dissoziiert nur schwer wieder ab. • weiters führt die Bindung der assoziierten tRNA an "sein" Enzym zu einer Konformationsänderung desselben, diese Änderung leitet die Aminoacylierung ein. Chemical Proofreading Kommt es zu einer fehlerhaften Bindung einer AS an eine tRNA, kann das durch die Konformation der Synthetase erkannt und die Bindung hydrolysiert werden, zur Fehlererkennung gibt es mehrere Checkpoints. Die tRNA mit passender AS hat im Komplex eine höhere Affinität, es passiert eher selten, dass eine falsche AS an die tRNA angehängt wird. Kommt das trotzdem vor, zerfällt die falsch gekoppelte tRNA schneller als eine korrekte. Warum ist trotzdem nicht schlimm, falls eine inkorrekte AS eingebaut wird? • es gibt viele Proteine eines Transkripts • es gibt nur wenige AS in aktiven Zentren, die Wahrscheinlichkeit das dort eine fehlerhaft beladene tRNA verwendet wird ist gering. 92 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Nonsense suppression Die ersten beiden Nucleotide eines Codons einer AS sind in der Regel konserviert. Gene können nur unter ganz speziellen Bedingungen exprimiert werden. Bei Nonsense Mutation zu einem Stopcodon wird die Translation vorzeitig abgebrochen, das Protein wird nicht vollständig erzeugt. Unter Nonsense Supression versteht man ebenfalls mutierte tRNAs, die per Anticodon ein Stopcodon erkennen, aber mit einer AS beladen sind. Wird diese tRNA eingebaut, kommt es zu keinem Stop der Translation. Allerdings ist es höchst wahrscheinlich, dass dadurch an der mutierten Stelle eine fehlerhafte AS eingebaut wird. Weiters ist es möglich, dass das eigentliche Stopcodon des Gens durch die mutierte tRNA überlesen und die Translation des Gens nicht korrekt abgebrochen wird. Suppression: Findet eine Mutation in einer codierenden Sequenz statt, kann ein normales Triplett in ein Nonsense codon verändert werden (z.B. von einer AS in ein Stopcodon). In einem normalen Bakterienstamm kann diese mRNA nicht mehr korrekt translatiert werden (das Genprodukt wird zu kurz). Einige Bakterienstämme weisen eine Mutation im Anticodon bestimmter tRNAs auf, diese tRNAs codieren durch die Mutation für das Stopcodon, die Nonsense Mutation kann translatiert werden, das Genprodukt wird erzeugt, auch wenn eine AS fehlerhaft ist. 93 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Ribosomes Ribosomen bestehen im Vergleich aus mehr RNA als Protein, es ist allein die RNA für die katalytische Funktion verantwortlich. Die Proteine in Ribosomen haben keine enzymatische Funktion sondern tragen zur Genauigkeit bei. Ribosomen bestehen aus einer kleinen und einer großen Untereinheit und finden sich im Cytoplasma einer Zelle. Die große Untereinheit dient der Verknüpfung der AS mit der Polypeptidkette. Die kleine Untereinheit dient der Führung der mRNA. Prokaryoten: 70S bestehend aus 50S (5S + 23S rRNA) + 30S (16S rRNA) Untereinheiten. Eukaryoten: 80S bestehend aus 50S (5S + 5,8S + 28S rRNA) + 40S (18S rRNA) Untereinheiten. Ribosomen: Das eukaryotische Ribosom ist größer als das prokaryotische und weist weniger RNA Anteil (etwa 60%) auf als das prokaryotische, es wurden mehr Funktionen von Proteinen übernommen. Ribosomen weisen eine A- eine P- und eine E-Site auf. An der A-Site docken neue, beladene tRNAs an, an der P-Site befindet sich die tRNA, welche die Polypeptidkette gebunden hat, von der E-Site aus verlässt die nunmehr unbeladene tRNA das Ribosom. Crystal Structure of the ribosome at 5.5Å resolution Kristallstrukturen sind für die Entwicklung massgeschneiderter Antibiotika von Bedeutung. 94 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Structural mappingof 16S rRNA by chemical probing Die Strukturen der rRNA von Prokaryoten und Eukaryoten sind stark vergleichbar. Viele Teile sind hoch konserviert, nur einzelsträngige Bereiche der rRNA können im Gegensatz zu Loops oder Helices katalytisch sein. Experiment: Strukturelle Kartierung der 16S tRNA durch Chemical probing. Bestimmte Reagenzien modifizieren eine bestimmte Base, wenn sie nicht gepaart vorliegt, z.B. methyliert Kethoxal Guanin, wenn es Einzelsträngig vorliegt: • Kethoxal modifiziert G • DMS modifiziert A • CMTC modifiziert U - Ein Extrakt der Ribosomen enthält, wird mit einer der angeführten Reagenzien versetzt, dadurch werden alle Basen der Ribosomen modifiziert, die einzelsträngig vorliegen. Wird beispielsweise DMS zugesetzt, werden alle freien A modifiziert. - Der Extrakt wird von Proteinen gereinigt und die rRNA denaturiert. - Ist die RNA Sequenz des zu untersuchenden Ribosoms bekannt, werden Primer erzeugt, die an mehreren unterschiedlichen Stellen der rRNA binden. Es werden radioaktiv markierte Oligonucleotide sowie reverse Transkriptase (RT) hinzugefügt, die an den Primern beginnend zur rRNA komplementäre DNA erzeugt. An den modifizierten Basen, im Bsp. A durch DMS, wird die RT die Synthese abgebrochen, es entstehen so unterschiedlich lange DNA Sequenzen. - Die rRNA wird abgebaut, die DNA wird mittels Southern Blot über ein Gel analysiert. Die unterschiedlich langen Stücke der erhaltenen DNA kann ermittelt werden, welche Teile der rRNA doppelsträngig vorliegen, da diese im Gegensatz zu den einzelsträngigen Bereichen vom Primer aus immer vollständig vorliegen. Mittels dieser Informationen kann eine Karte der gepaarten und ungepaarten Regionen der rRNA erstellt werden. 95 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) 23S rRNA Binding of antibiotics to the 30S subunit 50% der Energie in der Zelle wird für die ribosomale Maschinerie aufgewendet. 96 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert … Für Translation benötigte Komponenten. Ein paar lustige Zahlen: Pro E.coli Zelle findet man: • 19.000 Ribosomen • 200.000 tRNAs – jeweils 3000 einer Art • 1500 mRNAs – jeweils 2-3 einer Art Auf jede mRNA kommen somit etwa 12 Ribosomen. Daraus ergibt sich, dass eine mRNA nicht einmal sondern vielfach translatiert wird. Nachdem die Erzeugung dieses Apparats energieaufwenig ist und Bakterien in der Regel auch die AS selbst herstellen (autotroph), muss die Synthese entsprechend reguliert sein, wenn dem Bakterium zu einem Zeitpunkt keine ausreichenden Energiequellen zur Verfügung stehen. Diese Regulation ist sowohl transkriptionell als auch translational realisiert. Adjusting ribosomes and tRNA to the physiological needs The guanosine tetraphosphate cycle relA: ppGpp Synthetase I spoT: ppGpp Synthetase II 3' Pyrophospohydrolase gpp: Phosphohydrolase Die Produktion von Ribosomen und tRNA wird durch den Guanosintetraphosphatzyklus gesteuert. Ist eine AS und damit eine tRNA nicht in ausreichender Konzentration vorhanden, stoppt das Ribosom während der Translation ("iceing reaction"?). Mit dem Ribosom ist ein spezieller Faktor, relA, assoziiert, das pppGpp (Guanosinpentaphosphat) aus GTP erzeugt, wenn das Ribosom stoppt. pppGpp wird in weiterer Folge zu ppGpp (Guanosintetraphosphat) umgewandelt. Ein weiteres Enzym, Guanosintetraphosphatase 2 erzeugt bei niedrigem Kohlenstoffspiegel (vorliegendem Zucker) ebenfalls ppGpp. ppGpp wandert in den Nucleus und inaktiviert Gene die mit der Transkription von rRNA, tRNA sowie der Synthese von AS assoziiert sind; im Beispiel den P1 Promotor des rrnB Operons. 97 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translational autoregulation of ribosomal protein genes S7 ist ein ribosomales Protein, dess Repressoren autoreguliert sind. Ist ausreichend rRNA vorhanden, assoziiert S7 mit einer Sekundärstruktur der 16S rRNA. Ist die S7 Konzentration höher als die der 16S rRNA, bindet das freie S7 an die eigene mRNA an einer der 16S rRNA ähnlichen Sekundärstruktur und inhibiert so die eigene Translation. Dabei ist zu beachten, dass S7 eine höhere Affinität zur 16S rRNA als zur eigenen mRNA aufweist, dadurch wird S7 immer an 16S rRNA gebunden sein, wenn sie in ausreichenden Mengen vorliegt. Translation initiation in Prokaryotes: requires formylated fMet-tRNAfMet Bei Prokaryoten gilt: AUG als Start-Codon, selten auch GUG oder UUG. Für AUG werden zwei unterschiedliche tRNAs erzeugt: eine für das normale AUG-Met sowie eine tRNA mit modifiziertem Met für das ein AUG als Initiationscodon. Diese Initiatior-tRNA (N-formyl-met-tRNA) ist am Akzeptorende im Gegensatz zu anderen tRNAs nicht gepaart. Am Anticodon weist sie drei GC Paare auf, was für tRNAs ebenfalls untypisch ist. 98 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 2. tRNAmMet … erkennt interne AUGs für Elongation Das Anhängen der Formyl-Gruppe ist nicht unbedingt notwendig. Die Formyl-Gruppe erhöht jedoch die Bindungseffizienz von IF2 an die fMet-tRNA. Formylmethionin hat eine wichtige Funktion in der Immunabwehr (Interaktion mit Neutrophilen). Es ist ein Alamon für das Immunsystem, das dadurch eine bakterielle Infektion erkennen kann. In Eukaryoten ist die tRNA an die 40S ribosomale UE gebunden, bevor das Ribosom über Protein Wechselwirkungene an die mRNA bindet. Bei Prokaryoten bindet die 30S UE zunächst mittels RNA-RNA Wechselwirkungen an die mRNA, bevor die fmet tRNA mit Ribosom und 30S UE interagiert. Deformylase ist ein Enzym, das die Formylgruppe nach der Proteinsynthese vom translatierten Protein entfernt. 99 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Correct translation initiation requires initiation factors Functions of IF1-IF3. IF1 occupies the A-site. IF2 tRNAi binding /selection for tRNAi. IF3 kinetic discrimination against elongator tRNAs. conformational changes in 30S subunit prevents association of 30S/50S subunits. Prinzipieller Ablauf der Initiation: • 30S UE komplexieren mit IF1 und IF3. • Bindung von IF2-GTP an den Komplex • IF2 überwachte Bindung von fmet-tRNA an die P-Site. • Bindung der 50S UE, Bildung des 70S Ribosoms unter GTP-Hydrolyse und Konformationsänderung, IF2-3 werden freigesetzt. -) • • • IF1: blockiert mit IF2 die ribosomale A-site IF1 ist essentiell, ein Knockout lethal. Nachweis mittels Chemical probing: Basen in der ASite werden nicht modifiziert, IF1 sitzt folglich hier. -) • • • IF2: erkennt fMet-tRNA Formylgruppe. kontaktiert die tRNAi über die C-terminale Domäne. stimuliert deren Bindung an die P-Site. IF2 ist evolutionär über alle Systeme als Protein konserviert, hat unterschiedliche Funktionen in den einzelnen Systemen. -) • • • IF3: bindet in der Nähe der P-Site Umgebung. diskriminiert gegen alle tRNAs außer tRNAi. diskriminiert gegen alle Startcodons außer AUG IF1 blocks together with IF2 the ribosoA-site during translation initiation IF1 protects sites which are also protected by A-site tRNA. A-Site (Akzeptor) ... Eintrittstelle neuer tRNAs in das Ribosom mit Ausnahme der Initiator-tRNA, wird durch IF1 blockiert. P-Site (Peptidyltransferase) ... beherbergt die tRNA, welche die wachsende Polypeptidkette trägt. Hier bindet die Initiator-tRNA. E-Site (Exit) ... von hier aus wird die tRNA ohne AS aus dem Ribosom entlassen. Experiment: Bindungsort von IF1 mittels Chemical probing ermitteln Durch Chemical Probing kann ermittelt werden, dass IF1 die A-Site besetzt: IF1 zu Ribosomen hinzufügen, DMS dazu, Reverse Transkription, man findet keine Methylierungen an der A-Site der 16S rRNA ... IF1 muss also dort binden. 100 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert IF1 and structural homologues IF2 recognizes fMet tRNA fMet, stimulates binding of it to the P-site and discriminates by virtue of the formyl group IF2 contacts tRNAi via its Cterminal domain. IF2: Mw ~ 80.000 IF2 stimuliert die Bindung der tRNAi an das Ribosom, es bindet die tRNAi über seine Cterminale Domäne. Translation initiation factor 3 MW. 21.700 discriminates against: -non-canonical start codons AUU (start codon of IF3) Dallas and Noller, Molec.Cell (2001) Unterscheidung zwischen Startcodon und NichtStartcodon. Herausgefunden mittels Toeprinting-Assay. 101 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert IF3 translational autocontrol IF3 diskriminiert gegen AUU (bzw. alle Startcodons außer AUG). Eine Bindung von IF3 führt zu Konformationsänderung im Ribosom, AUU wird dadurch nicht mehr als Startcodon erkannt. IF3 diskriminiert gegen alle Startcodons mit Ausnahme von AUG, durch die Konformationsänderung kann nur noch fMet-tRNA an der P-Site binden. Ist die IF3 Konzentration in der Zelle hoch ist, wird AUU als Startcodon nicht akzeptiert. Ist die Konzentration niedrig, wird auch AUU als Startcodon akzeptiert. Nachdem die mRNA die für IF3 codiert selbst ein AUU als Startcodon verwendet, reguliert sich die Synthese von IF3 somit selbst. Experiment: Reportergen Assay zur Autoregulation von IF3 Es wird ein Plasmid mit LacZ-Gen mit AUG als Startcodon in einen WT eingebracht, dadurch wird ß-Galaktosidase erzeugt. Bringt man das Plasmid in eine IF3-Mutante ein, wird ebenfalls ß-Galaktosidase erzeugt. Ändert man das Startcodon des LacZ Gens von AUG auf AUU und bringt es ein: Im WT findet so gut wie keine Translation des LacZ Gens statt. In der IF3 Mutante findet sich etwa ein Drittel so viel Translation wie mit dem ursprünglichen AUG Startcodon. IF3 diskriminiert also gegen alle Startcodons außer AUG - in der IF3 Mutante mit AUU Startcodon findet man aus dem Grund weniger ß-Galaktosidase, weil IF3 im WT eine Konformationsänderung durchführt, durch die AUG viel stabiler gebunden wird. Findet die Konformationsänderung nicht statt, kann AUU zwar binden und die Translation initiieren, allerdings nie so effektiv wie AUG im WT. 102 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Toeprinting assay • • • • • • Eine mRNA mit bekannter Sequenz und rRNA Bindungsstelle verwenden Passend dazu einen Primer mit 32P Markierung synthetisieren. eine hohe Konzentration mRNAs (1012) mit 30S UE und fMet-tRNA mischen und Reverse Transkriptase (RT), markierte Primer und Nucleotide hinzufügen. Die 30S UE bildet mit fMet-tRNA und mRNA einen ternären Komplex. Dieser ist so stabil, dass die Reverse Transkriptase an der Bindungsstelle des Ribosoms abbricht. Ist keine 30S UE an die mRNA gebunden, erhält man mittels der RT eine cDNA, die der Länge der mRNA entspricht. Mit 30S UE und fMet-tRNA erhält eine kürzere cDNA und als Toeprint Signal bezeichnet wird. Das 3' Ende der verkürzten cDNA enspricht der Position, an der die 30S UE an die mRNA gebunden ist. Über Southern Blot kann die verkürzte cDNA mit der normalen cDNA verglichen und die Position bestimmt werden. Je nachdem wie hoch die Ribosomenkonzentration gewählt wird, wird die Masse der mRNAs mehr oder weniger mit einem ternären Komplex besetzt sein. Das wiederum resultiert in unterschiedlichen Toeprinting Signalen. Experiment: Aktivitätsmessung von IF3 Selection of the initiatior tRNA by Esacherichia coli initiation factors Genes and Development (1989) 3:1899 Führt man einen Toeprint-Assay mit 30S UE und einem tRNA-Mix durch, so erhält man viele unterschiedlich lange cDNA-Stücke durch die Reverse Transkriptase, da die 30S UE mit jeweils verschiedenen tRNAs irgendwo auf der mRNA an den den tRNAs passenden Codons einen Komplex bildet. Fügt man zu 30S UE und tRNAs zusätzlich IF3 in ausreichender Konzentration hinzu, erhält man fast ausschließlich gleich lange cDNA Stücke, da IF3 für fMet-tRNA diskriminiert und sich der ternäre Komplex daher nur über dem AUG Startcodon der mRNA bilden wird. Mit Hilfe dieses Tests kann auch auf andere in trans agierende Proteine in Bezug auf translationale Initiation getestet werden. Das abgebildete Experiment zeigt, dass IF3 auch gegen AUGs ohne SD Sequenz diskriminiert und somit tatsächlich nur AUGs als Startcodon zulässt (?). 103 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert mRNA bound to the 30S ribosomal subunit. (A) A 36-nucleotide mRNA is bound to the 30S ribosomal subunit. rRNA is shown in grey, mRNA is shown in yellow, and protein is shown in cyan. The ASD sequence of the 16S rRNA is shown in red to indicate the SD-ASD interaction. The P-site initiation codon is shown in green, and the A-site codon is shown in magenta. Note the kink in the mRNA between the two codons. (B) Close-up of the region indicated in panel A. The upstream and downstream tunnels are marked by arrows. Colors are the same as in panel A. Kinetic discrimination by IF3 104 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Interactions of the initiator tRNA (A) Surface representations of the initiator tRNA. The regions that interact with the indicated component of the translational machinery are highlighted in red, and the nucleotide positions on the tRNA are indicated next to the structure. The structure of the initiator tRNA is derived from PDB entry 2FMT. (B) Detailed view of the interaction between the initiator tRNA and the ribosome. On the left is a surface representation showing the important sites of interaction in red. On the right is the initiator tRNA on the 70S ribosome. The tRNA is shown in red, the mRNA is shown in yellow, and the 16S and 23S rRNA are shown in cyan and grey, respectively. The codon-anticodon interaction is shown. Experiment: tRNA charging – welches Codon für welche AS Deciphering the gentic code Drei Basen = ein Codon = eine AS Durch die 4 Basen erhält man 64 Möglichkeiten der Codierung. Zunächst wird das Enzym Polynucleotidphosphorylase benötigt, das Nucleotide miteinander verknüpft. Nachdem bei einem Gemisch aller vier RNA Basen (A, G, C, U) nicht vorhersagbar ist, welche Sequenz man erhält, wurde zunächst mit homogenen Nucleotiden gearbeitet. Im konkreten Fall mit U, man erhielt Poly U-RNA Sequenzen. Aus E.coli wurden Extrakte isoliert, welche die gesamte Translationsmaschinerie enthielten, mit den Poly U Sequenzen vermischt und anschließend jeweils eine radioaktiv markierte AS hinzugefügt. mRNA!! Daraufhin wurde geprüft, welche AS mit den Poly U-RNAs translatiert wurde indem das Gemisch nach einiger Zeit durch einen Nitrozellulosefilter geschickt wurde. Nicht gebundene tRNA passiert Filter, gebundener ternärer Komplex (Ribosome, tRNA, mRNA) passierten den Filter nicht. Durch radioaktiv markierte AS konnte der Komplex nachgewiesen werden. So wurde ermittelt, dass Poly U für die AS Phe codiert. 105 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The genetic code Codons sind der universelle genetische Code, er ist bis auf wenige Ausnahmen in jedem Organismus gleich. Der genetische Code ist redundant, es gibt meist mehrere tRNAs die für eine AS codieren. Es gibt 64 tRNAs … 61 tRNAs für AS sowie 3 tRNAs (UAA, UAG, UGA) die als Stopcodons dienen. Nicht jedes Codon wird in gleicher Anzahl verwendet bzw. übersetzt. AGA wird im Vergleich mit AGG zu 0,1% zu Arg translatiert. - häufig gebrauchte Produkte … werden häufig vorkommende Codons kodiert. - selten oder in geringer Konzentration benötigte Produkte (z.B. Repressoren) … werden durch selten vorkommende Codons kodiert, es findet sich auch eine entsprechend geringe tRNA Konzentration für dieses Codon (bei Versuchen zu beachten). Wobble Hypothese: An der dritten Stelle des Codons / Anticodons sind neben den normalen GC bzw. AU Bindungen noch andere Paarungen möglich. Aus diesem Grund hat die letzte Stelle eines Codons die geringste Aussage über die zugehörige AS. 106 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Foliensatz 2 Bläsi Die Shine Dalgarno Sequenz Ribosome recruitment signals on prokaryotic mRNA: How to find the right start / reading frame? Die Gene einer polycistronischen mRNA müssen unter Umständen unterschiedlich stark exprimiert werden. Jedes Gen auf der mRNA muss zu diesem Zweck eine Startsequenz und eventuell individuelle Enhancerelemente aufweisen um individuell translatiert werden zu können. AUG dient den meisten Genen als Startcodon. Für das Auffinden des Startcodons eines Gens durch das Ribosom dient die Shine-Dalgarno Sequenz (SD, 5' AGGAGGU 3' als Consensus Sequenz), die bei Prokaryoten immer upstream (5'-seitig) vom eigentlichen Startcodon liegt. Die SD-Sequenz ist komplementär zum 3' Ende der 16S rRNA (Anti-SD-Sequenz) und ermöglicht es dadurch der 30S UE an der passenden Stelle an die mRNA zu binden. Durch diesen Umstand ist es auch möglich, dass sich überlappende Gene translatiert werden, wie es bei Phagen (z.B. Phi) oft der Fall ist. The Shine and Dalgarno / anti-SD Interaction 107 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Biochemischer Nachweis der SD-Anti-SD-Interaktion durch footprinting Footprinting mRNA-ribosome complexes with chemical probes The EMBO Journal vol.13 no.16 pp3892-390? - 1994 Reagiert Sauerstoff mit Fe3+-Ionen, erhält man Wasserstoffperoxid, das weiter zu Wasserstoffhydroxylionen und Hydroxyradikalen reagiert. Hydroxyradikale spalten Ribose an C1 und C4 und greifen damit auch das Rückgrat der RNA an. Mittels Hydroxyradikalen kann dadurch ungeschützt vorliegende RNA an zufälligen Stellen gespalten werden. mRNA wurde an einem Ende markiert und mit Ribosomen und Radikalen vermischt. Aus der erhaltenen Lösung wurden die mRNA Fragmente isoliert und mittels Gel getrennt. Dabei zeigte es sich, dass Banden in einem bestimmten Bereich fehlten – in diesem Bereich also keine Spaltung der mRNA stattgefunden hatte und nur größere oder kleinere mRNA Fragmente vorlagen. Dieser Bereich wurde durch das Ribosom überdeckt und war für die Radikale nicht zugänglich. Der Bereich der vom Ribosom verdeckt war erstreckte sich von -35 bis +20 in Bezug auf das AUG Startcodon der mRNA • • • Experiment: Genetischer Nachweis der SD-Anti-SD-Interaktion Specialized ribosome system: Preferential translation of a single mRNA species by a subpopulation of mutated ribosomes in Escherischia coli Proc.Natl.Acad.Sci.USA Vol.84 pp 4762-4766 July 1987 Zum Nachweis der direkten Interaktion von SD und AntiSD wurde eine komplementäre Mutation in vitro durchgeführt. Es wurde das rru-Operon auf ein Plasmid kloniert. Dabei wurde die Anti-SD Sequenz der 16S rRNA zu einer SD Sequenz verändert. E.coli besitzt sieben Operons für die Transkription von rRNA, es sollen aber nur die durch das Plasmid eingebrachten rRNA Transkripte funktionsfähig sein. Zu diesem Zweck wurde ein weiterer Basenaustausch auf der 16S rRNA des Plasmids vorgenommen (Mutation C → U an Position 1192). Dieser vermittelt eine Resistenz gegen Spectinomycin. Abschließend wurde dem Plasmid ein Reportergen (hGH, menschliches Wachstumshormon) mit einer eine Anti-SD Sequenz hinzugefügt. 108 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Wurde nur das Reportgen mit Anti-SD Startsequenz in wt E.coli eingebracht, fand keine Translation statt, die wt Ribosomen interagierten nicht mit der Anti-SD Sequenz. Wurden Reportergen mit Anti-SD Startsequenz und 16S rRNA mit SD Sequenz und Basenaustausch wt E.coli eingebracht und diese auf Medium mit Spectinomycin gezüchtet, so erhielt man das Hormon. Durch das Spectinomycin waren nur die eingebrachten Ribosomen funktionsfähig, weiters konnte die SD Sequenz auf der 16S rRNA mit der Anti-SD Sequenz des Reportergens interagieren und Translation stattfinden. Ähnlich kann man Protein-Protein-Interaktionen identifizieren. Findet man in einem Protein eine Mutation, die nur mit einer Mutation in einem anderen Protein funktionsfähig ist, kann man Annehmen, dass an dieser Stelle zwischen den beiden Proteinen Interaktion stattfindet. Experiment: Biochemischer Nachweis der SD-Anti-SD-Interaktion durch chemical probing • • • Zunächst werden in ausreichender Menge mRNA (1012-1014) und 30S UE vermischt, es bilden sich Komplexe durch Bindung der SD + Anti-SD Sequenzen auf 30S UE und mRNA. • Anschließend werden Reagenzien hinzugefügt, die nur ungepaarte Nucleotide modifizieren können (sounds familiar). An der Stelle an der mRNA und 30S einen Komplex bilden kann die mRNA nicht modifiziert werden.Dazu ist zu sagen, dass zufällige, einzelne Basen modifiziert werden, es werden nicht alle ungepaarten Basen einer mRNA modifizert. Modifizierung: DMS – A, CMTC – U, Kethocal - G Werden anschließend die Komplexe getrennt und zur mRNA Primer und Reverse Transkriptase hinzugefügt, erhält man je nachdem wo die Transkriptase durch eine zufällige Basenmodifikation abbrechen muss, unterschiedlich lange Stücke cDNA. Wird die cDNA auf einem Gel ausgewertet, erhält man durch die unterschiedliche Länge eine Vielzahl an Banden, allerdings wird sich keine Bande an der Position der SD Sequenz finden, da hier die Reagenzien niemals modifizieren konnten und die Reverse Transkriptase daher hier auch niemals abbrechen musste. Textbook view Translationale Initiation in Prokaryoten: • Bindung der 30S rRNA an die mRNA • Bindung zusätzlicher Faktoren • Bindung der Initiator tRNA • Bindung der 50S rRNA und Bildung des 70S ribosomalen Komplexes 109 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Parameters affecting translation initiation of a canonical prokaryotic mRNA 1. 2. 3. 4. 5. 6. Start codon SD S1 SD < > AUG Secondary stuctures Sequences surrounding the TIR 7. Translational repressors 8. Riboregulation 9. Other control mechanisms Die wichtigsten Parameter, die bei der translationalen Initiation eine Rolle spielen: Das Startcodon, der Abstand zwischen Startcodon und SD Sequenz, Sekundärstrukturen die die Ribosomenbindungsstelle unzugänglich machen können sowie vom Gen abhängig Proteine, die als Aktivatoren oder Repressoren dienen. 1. Flexibility in start codons • • • • > 90% start codons are ATG. Codons other than ATG are found initiating coding sequences. Can infrequently be GTG ("Valine") and TTG ("Leucine"). A methionine is still incorporated as the first amino acid. 2. The strength of the Shine and Dalgarno sequence 3. Ribosomal protein S1 (only r-protein with an assigned function) Ribosome-messenger recognition: mRNA target sites for ribosomal protein S1 Nucl. Acids Res. (1991) 19:155 • essential in E.coli • 61.2 kD protein with an elongated shape • weakly bound to the ribosome by the means of protein-protein interactions • 6 RNA binding domains at the C-terminus • RNA unfolding capacity • required for translation of mRNAs with a canonical RBS S1 ist das größte Protein der 30S UE, ist über Protein S2 mit dem Ribosom verbunden und geht über sechs Domänen Bindungen mit der mRNA eingeht. S1 weist sogenannte RNA unfolding capacity auf, es wird angenommen, dass es einfache mRNA Sekundärstrukturen auflösen kann. Trennt man S1 vom Ribosom, kann dieses nicht mehr an die mRNA binden, es findet keine Translation statt. 110 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Isolierung von mRNA-Fragmenten die S1 binden und deren Sequenzierung • • • mRNA, 30S UE, fMet-tRNA und IF3 werden vermischt, es bilden sich stabile Komplexe. • Anschließend wird Crosslinking mittles UV durchgeführt: Aminogruppen die eine WW mit mRNA gehen werden dadurch permantent mit der mRNA verbunden. • Durch Zugabe von SDS oder EDTA wird der ternäre Komplex aufgetrennt, nur Proteine die im Zuge des Crosslinkings fix mit mRNA verbunden wurden bleiben an der mRNA. • Aus der Mischung wird die mRNA grob über einen Succrosegradienten abzentrifugiert und die mRNA aus dieser Fraktion über eine Sepharose-Protein-A Säule mittels Antikörper gegen Protein S1 getrennt. • Freiliegende mRNA wird durch RNase zernichtet, durch S1 gebundene und damit unzugängliche mRNA bleibt dabei intakt. Die verbleibende mRNA wird am 5' Ende markiert, es wird erneut SDS oder Harnstoff hinzugefügt, wodurch sich S1 mit der mRNA vom Antikörper löst, S1 + mRNA werden isoliert. Abschließend wird die Lösung mit Proteinase K versetzt, die das Protein S1 abbaut, die verbleibende mRNA Sequenz wird sequenziert. Die Sequenzierung der erhaltenen mRNA ergab, dass die Sequenz an der S1 bindet eine Ureiche Sequenz ist, die upstream des translationalen Startpunktes (der SD Sequenz) liegt. Experiment: Klärung ob S1 Primär oder Sekundärstrukturen der mRNA bindet Durch ein spezielles Verfahren (Selex) wird bestimmt, welche Liganden an beispielsweise S1 binden. Es werden verschiedene Liganden erzeugt und deren Bindungseigenschaften geprüft. Das liefert Rückschlüsse auf die bevorzugte Bindungsstelle von S1 durch den Vergleich der Gemeinschaften einzelner Liganden. • Zunächst wird S1 gereinigt. • Es werden Oligonucleotide zufälliger Sequenz erzeugt, Protein S1 wird mit ihnen vermischt. • Komplexe aus S1 und Oligonucleotiden werden durch Filtration gewonnen. • Die Oligonucleotide werden von S1 gelöst und deren analysiert. Das Experiment ergab, das S1 an Pseudoknoten der mRNA und damit Sekundärstrukturen bindet. S1 ist für die translationale Initiation in Bakterien extrem wichtig. Allerdings weisen nicht alle Bakterien S1 auf, diese müssen andere Mechanismen haben, wie die Initiation stabilisiert wird. Es ist auch von der mRNA abhängig, ob sie ohne S1 translatiert werden kann oder nicht. mRNAs die S1 nicht zur Translation benötigen, werden als leaderless mRNA bezeichnet, sie starten direkt mit einem AUG. 111 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Visualization of protein S1 within the 30S ribosomal subunit and its interaction with messenger RNA PNAS (2001) 98:1191 S1 is required for translation initiation of mRNAs with a structured 5'-UTR --------> Steigende Konzentration UTR … untranslated region ompA … Banden werden mit zunehmender S1 Konzentration intensiver. cI … Banden bleiben bei steigender S1 Konzentration in etwa gleich. ompA mRNA … S1 abhängig; Sekundärstruktur an 5' UTR. cI mRNA … S1 unabhängig; keine Sekundärstruktur in 5' UTR. 4. SD < > Start codon Influence of mRNA Determinants on translation inititation in Escherichia coli J. Mol. Biol. (1991) 218, 83-97 Der Abstand zwischen SD und Startcodon beeinflusst die Translation, da das Ribosom mit beiden Sequenzen wechselwirken muss. Der optimale Abstand wäre 1nt In Experimenten wurden Basen zwischen SD Sequenz und Startcodon eingeführt und ermittelt, dass die optimale Distanz 78 Basen beträgt, in der Realität kann Ribosomenbindung in einem Bereich von 4-11 Basen Abstand erfolgen. Toeprint: zwischen Startcodon und SD werden unterschiedlich viele Basen eingesetzt, das stärkste Signal bei 7 Basen. 112 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 5. mRNA 2nd structures can inhibit translation initiation by masking the SD and / or the start-codon Regulation of expression of bacteriophage T4 lysozyme Formation of 2nd structure depends on transcriptional start. Inhibitory stem-loop structures: Different stimuli for opening / closing "Breathing", Bläsi et al., 1989 Sekundärstrukturen sind häufig für die Modulation der translationalen Initiation verantwortlich. Sekundärstrukturen können sich öffnen und wieder schließen, man spricht in diesem Zusammenhang von "Breathing". "Thermosensor", Johansson et al., 2002 "Vitamin perception", Ravnum and Andersson, 2001 113 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The dual-start motif in λS Dual translational initiation sites of the λ S gene The EMBO Journal vol.8 no.11 pp.3501-3510 1989 Das λ S Gen weist zwei Initiationsstellen auf, wodurch zwei unterschiedliche Genprodukte erzeugt werden können. Die Regulation erfolgt über 2 SD Sequenzen: SD-Met1 und SD-Met5 sind unterschiedlich gut für Ribosomen zugänglich; SD-Met1 liegt in einer Sekundärstruktur. Das führt zu unterschiedlicher Expression der zwei Produkte. Im Phagen Lambda liegt die SD Sequenz des Lysegens S in einer solchen Sekundärstruktur, Lyse kann nur stattfinden, wenn die Sekundärstruktur aufgelöst wird. Translational regulation by an RNA thermosensor 114 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translational regulation by temperature mediated changes in mRNA structure of the L. monocytogenes prfA gene Listeria monocytogenes is a human pathogenic bacterium that causes food poisoning. • Listeria regulates virulence genes so they are expressed only within the host (this is true for other pathogens also). • One key feature of being within the host environment is temperature. • PrfA is a transcription factor that activates expression of virulence genes. PrfA is expressed at 37°C but not at 30°C. • Control is at the level of translation, not transcription, by changing the structure of the prfA mRNA. mRNA structure represses prfA translation. The inhibitory structure melts at 37°C 1. 30°C or less, Shine-Dalgarno (SD) sequence paired -no translation. 2. 37°C base pairing melts, ribosome can now bind SD. 3. Translation of prfA can now proceed. 4. Unusual as required no proteins at all for regulation. Neisseria erzeugt als Virulenzgen eine Protease, deren SD Sequenz bei 30° in einer Sekundärstruktur liegt und unter diesen Bedingungen nicht translatiert wird. Wird das Bakterium beispielsweise in einen Wirt aufgenommen, in dem höhere Temperaturen herrschen, öffnet sich die Sekundärstruktur, die Protease kann translatiert werden und seine greuliche Wirkung entfalten. Molecular basis for temperature sensing by an RNA thermometer The ROSE (repressor of heat-shock gene expression) element is present in the 5'-UTR of small heat-shock genes in many Gram- bacteria including Rhizobia, Escherichia coli and Salmonella. The EMBO Journal (2006) 25 Riboswitches transcriptional / translational attenuation ! without protein factors or tRNA molecules ! 115 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Organization of riboswitches Binding domain (Aptamer): • structured binding pocket • high affinity • high specificity Regulatory domain (Expression platform): • reacts upon ligand binding by the aptamer • conformational change alters gene expression Riboswitch Classes e.g.: • vitamin biosynthesis • amino acid metabolism • purine metabolism SAM has different roles in the regulation of methionine biosynthesis gram negative bacteria gram positive bacteria 116 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Confirmation of the transcriptional attenuation model in the metI riboswitch transcriptional attenuation The thi box Thiamine pyrophosphate (TPP), also known as cocarboxylase, is the cofactor of key enzymes of carbon metabolism such as pyruvate dehydrogenase, a-ketoglutarate dehydrogenase, transketolase, pryuvate decarboxylase, and others. Regulation of thiamine (B1) biosynthesis in Escherichia coli (translational control) 117 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Thiamine biosynthetic genes and possible mechanisms of riboswitch control In-Line Probin 6. Secondary structures in TIR Influence of mRNA Determinants on translation initiation in Escherichia coli J. Mol. Biol. (1991) 218, 83-97 118 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sequences of the TIR that affect the translation initiation frequency Bimacombe and McCarty, 1994 119 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The downstream box The initiation of translation in E.coli: apparent base pairing between the 16srRNA and downstream sequences of the mRNA Nucleic Acids Research, Vol.18, No.7 1719 (1990) Mit Hilfe von Mutationen wurde downstream des AUG Startcodons eine Region ausgemacht, die als "downstream-box" bezeichnet wurde. Es wurde postuliert, dass diese mit einem speziellen Bereich (stem-loop) der 16S rRNA interagieren kann; dieser Bereich auf dem Ribsosom wurde als anti-downstreambox bezeichnet. Die downstream-box sollte die Interaktion zwischen SD und Anti-SD Sequenz erleichtern. Es ergeben sich dabei aber zwei logische Probleme: Die Anti-SD Sequenz liegt ungepaart und frei zugänglich vor, während die Anti-downstreambox bereits gepaart in einer Sekundärstruktur vorliegt. Rein aus sterischen Gründen ist damit eine Interaktion zwischen downstream-box und 16S rRNA nicht möglich. Functional importance of RNA interactions in selection of translation initiation codons Molecular Microbiology (1997) 24(1), 19-28 120 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The downstream box does not exist! Evidence angainst an Interaction between the mRNA Downstream Box and 16S rRNA in Translation Inititation Journal of Bacteriology, June 2001, p3499 – 3505 Vol.183 No.11 Eine Röntgenstudie zeigt, dass eine räumliche Interaktion zwischen DS Box und 16S rRNA nicht möglich ist. Durch komplementäre Mutationen in DS Box und 16S rRNA konnte die Existenz dieser Box ausgeschlossen werden. Durch chemical probing kann ebenfalls bewiesen werden, dass es keine Interaktion zwischen DS Box und 16S rRNA gibt. 7. Translational repressors and activators Repressorsysteme: Es lagern sich solange Monomere an die mRNA an bis SD überdeckt ist. Es kommt zu Pseudoknotenbildung (bei Protein Sn15), Ribosome können nicht mehr binden. Aktivatorsysteme: Es kommt zur Bindung von Proteinen upstream eines Hairpins in dem sich SD-Sequenz befindet. SD Sequenz wird frei, Ribosomen können binden. Die SD Sequenz kann auch durch endonucleolytische Schnitte freigesetzt werden. 121 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert T4 gene 32 autoregulation T4 gene 32 encodes a single-stranded nucleic acid-binding protein involved in T4 DNA replication, recombination and repair. Translationale Repressoren dienen häufig der Reglation von akzessorischen Elementen. Bsp: Gen 32 des Phagen T4 codiert für ein Einzelstrangprotein. Dieses Gen ist auf mRNAEbene autoreguliert. Wurden ausreichend Protein 32 Monomere erzeugt, binden diese an sogenannte Pseudoknoten (speziell gefaltete RNA-Strukturen). Nach der Bindung an den Pseudoknoten können weitere Monomere binden, bis die SD Sequenz erreicht wurde, dadurch das Ribosom nicht mehr binden kann und die Translation inhibiert wird. Autoregulation of translation balances ribosomal protein levels with rRNA synthesis Ribosomal protein operons 1. Ribosomal protein genes are organised in operons. 2. Most operons contain genes for both small and large subunit proteins. 3. Some non-ribosomal proteins that have roles in translation process also encoded in these operons. 4. One gene in each operon (dark colour) acts as a regulator. 5. In each case the regulatory protein is an rRNA binding protein. 6. Genes marked + are under translational feedback regulationby the dark coloured gene. Autoregulation der Translation durch Protein S15 Autoregulation of translation balances ribosomal protein levels with rRNA synthesis For each operon there is a regulatory sequence in the ribosomal protein operon that is similar to the sequence on the rRNA. Liegt S15 in höherer Konzentration als die der 16S rRNA vor, inhibiert es seine eigene Translation. 122 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Ribosomal protein S15 from Escherichia coli modulates its own translation by trapping the ribosome on the mRNA initiation loading site Proc. Natl. Acad. Sci. USA, Vol.90, pp.4394-4398, May 1993 Links ternärer Komplex aus Protein 15 mRNA, tRNA und Ribosom. Rechts binärer Komplex, das Ribosom kann zwar binden, aber kein Komplex mit tRNA. S15 bindet bei hoher Konzentration an die eigene mRNA, sodass sich ein Pseudoknoten ausbildet, der AUG und SD Sequenz für das Ribosom unzugänglich macht. Experiment: Repressorwirkung des Proteins S15 bei Translation Verhindert S15 lediglich die Bindung des Ribosoms oder wirkt es anders. Prinzip des Toeprint Assays: • In vitro bindet ein Ribosom an mRNA und bildet gemeinsam mit der fMet-tRNA einen stabilen ternären Komplex. • Fügt man Reverse Transkriptase sowie Primer die am 3' Ende der mRNA binden hinzu, erhält man ein cDNA Signal, das in der Regel 15nt vom A des AUG Startcodons entfernt ist. Je nachdem wie lang das Signal tatsächlich ist, kann ermittelt werden, wie gut das Ribosom gebunden hat. • • • Mischt man lediglich mRNA und 30S UE und fügt keine Initiator tRNA hinzu, bindet die 30S UE nur aufgrund der SD-Anti-SD Interaktion auf der mRNA (binärer Komplex). Mittels Reverser Transkriptase erhält man hier eine cDNA, die bis +10 an das AUG heranreicht (im Gegensatz zu +15 des ternären Komplexes mit Initiator tRNA). Fügt man 30S UE und fMet-tRNA hinzu, erhält man ein Signal bei +17 - es konnte ein stabiler ternärer Komplex gebildet werden. Fügt man 30S UE, fMet-tRNA und S15 hinzu, erhält man ein Signal bei +10, es wird also kein stabiler ternärer Komplex gebildet, aber die 30S UE selbst kann noch an die mRNA binden. binary complex tertiary complex S15 verhindert also die Codon-AnticodonInteraktion der tRNA in der P-Site. Es kann dadurch zu keiner translationalen Initiation kommen. 123 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Prinzipieller Nachweis von Repressorwirkung bzw. Baseninteraktion: 1) Biochemisch: - Hydroxyradikalfootprinting - Toeprinting - RNA Verdau - Reagenzien dazu … wenn Basen nicht modifizierbar … Repressor dran - über Gewicht … Gelelektrophorese 2) Genetisch: - Mutation - Plasmide Threonyl-tRNA-synthetase Jenner et al. Science 1.April 2005, Vol.308 124 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The glgCAP operon The synthesis of ADP-glucose by ADP glucose pyrophosphorylase encoded by the E.coli glgC gene defines both the first committed step of glycogen synthesis and the major rate-controlling step of the pathay: Glucose-1-P + ATP ↔ ADP-glucose + PPi The glg leader Inactivation of CsrA by the non-coding regulatory RNA CsrB Secondary structure prediction for CsrB RNA 125 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) CsrB RNA functions as an antagonist of CsrA action 8. Riboregulation Regulation by antisense RNAs 126 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Two types of riboregulators: cis- and trans-antisense RNAs Cis-encoded small RNAs • • are encoded by the same genetic loci as their target. have the potential to form perfect duplexes. Cis-encoded regulatory RNAs are often used by • plasmids • phages • transposons to • • • • • regulate: plasmid copy number conjugation post-segregational killing temperate phage development transposition Tn10 achieves antisense copy control by overlapping promotors Antisense copy control 127 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Cis-antisense mechanisms: negative regulation Unter Cis-Antisense versteht man, dass zwei RNAs von Promotoren transkribiert werden, die konvergent liegen und dadurch eine sich ergänzende Sequenz aufweisen. Es wird in so einem Fall eine funktionelle mRNA von einem Promotor aus transkribiert. Zusätzlich wird eine weiter RNA von einem weiteren Promotor aus in Gegenrichtung transkribiert, die durch überlappende Sequenzen an die mRNA binden kann und eine Interaktion mit Ribosomen verhindert (negative Regulation). Beispiel: Replikationskontrolle von R1-Plasmiden durch die Antisense RNA copA. Das R1 Plasmid codiert über das Gen repA die eigene Replicase. Upstream des repA Gens liegen die Gene copB sowie tap, es wird von allen drei Genen eine mRNA erzeugt, die als copT bezeichnet wird. In Gegenrichtung wird die Antisense RNA copA transkribiert. copT und copA bilden Sekundärstrukturen aus, über die sie zunächst interagieren, um anschließend eine stabile Doppelstrangbindung einzugehen, wodurch die Translation der Gene tap und repA verhindert wird. Die Translation von repA ist an die Translation von tap gebunden, es findet sich also sowohl Antisense Regulation als auch auch translative Kopplung. Cis-acting antisense RNA mechanisms Antisense RNAs binden oft nicht in linearer Form an die zugehörige mRNA, sondern bilden wie bei der Regulation von repA durch Ausbildung von Sekundärstrukturen sogenannte "kissing loops", über die Antisense und zugehörige mRNA aneinander binden. Ein Beispiel für Antisense RNA findet sich in Bakterien bei der Kontrolle von Toxingenen die sich auf Plasmiden befinden. Es wird dabei sichergestellt, dass nur Bakterien überleben, die das Plasmid behalten. Das als hok bezeichnete Gen kodiert für ein Toxin, das die Zellmembran zerstört. Für die Translation von hok ist ein Upstream liegendes, als mok bezeichnetes Gen notwendig (positive translationale Kopplung). Zu der mok mRNA wird vom Plasmid eine als sok bezeichnete Antisense RNA erzeugt, die an mok bindet und dadurch die Translation von hok verhindert. Im Gegensatz zur mok-hok mRNA ist die sok Antisense RNA sehr instabil und muss vom Plasmid ständig neu transkribiert werden. Geht das Plasmid verloren, wird die sok Antisense RNA schneller abgebaut als die mok-hok mRNA, hok kann translatiert werden and the cell meets the grim reaper. 128 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Trans-encoded regulatory RNAs • are not genetically linked to the loci of their targets • form imperfect duplexes and can affect multiple targets via alternative base pairing. S.aureus erzeugt ein Hämolysin, das Erythrocyten lysiert. Dieses Enzym wird durch das Gen HLA erzeugt. Die HLA mRNA weist im Normalfall eine Sekundärstruktur auf, die die SD Sequenz verbirgt, wodurch das HLA Gen nicht translatiert werden kann und die mRNA schnell wieder abgebaut wird. RNA III kann an die HLA mRNA binden und löst dadurch Bindung die Sekundärstruktur auf, wodurch Translation stattfinden kann. RNA III und HLA mRNA sind nicht überlappend, deswegen wird RNA III als Trans-Antisense RNA bezeichnet. Bacterial stress responses are often mediated by trans-encoded regulatory RNAs Small RNA regulators in bacterial stress responses Small RNAs spielen bei der Genregulation auf translationaler Ebene eine wichtige Rolle. Wird die Zelle Stress ausgesetzt, werden sie transkribiert und dadurch bestimmte Gene im Gegensatz zu RNAi sowohl aus- als auch eingeschalten. Beispiel: RpoS codiert für den Faktor σ38, der in der stationären Phase erzeugt wird und notwendig ist, verschiedene Virulenzfaktoren zu aktivieren. Die rpoS mRNA weist bei der Ribosomenbindungstelle eine Sekundärstruktur auf die Translation verhindert. Die kleine regulatorische RNA DsrA löst die Sekundärstruktur auf und ermöglicht die Synthese von RpoS (positive Translationskontrolle durch DsrA). Beispiel: FlhA mRNA translatiert für einen transkriptionalen Aktivator. Die kleine regulatorische RNA OxyS bindet an die Ribosomenbindungsstelle von FlhA, wodurch dieses nicht mehr translatiert werden kann (negative Translationskontrolle durch OxyS). Kleine regulatorische RNAs spielen in der Kontrolle der Initiation der Translation eine wichtige Rolle, Vorkommen und Wirkungsziele sind dabei nur schwer zu ermitteln. DsrA beispielsweise ermöglicht nicht nur die Translation von RpoS, sondern inhibiert auch die Translation eines transkriptionalen Aktivators (hns), der eine Reihe weiterer Gene in der stationären Phase aktiviert. 129 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert hfq (Host factor Q in E.coli) • • hfq is an Sm-like RNA binding protein It is an abundant, heat-stable, doughnut-shaped hexameric protein hfq was shown to: • interact with several small RNA molecules (i) affect RNA stability (ii) mediate RNA-RNA interactions • interact with RNAP and the ribosomal protein S1 Der Phage Qß benötigt dieses Wirtsenzym, um die eigene RNA zu replizieren. hfq interagiert mit einer Vielzahl kleiner RNAs wie beispielsweise DsrA. In E.coli konnten einige dieser RNAs identifiziert werden, indem hfq in einer Säule gebunden wurde und die RNAs von E.coli durchgeschickt wurden. Experiment: Ermittlung der hfq Chaperonaktivität mittels Toeprinting Das Gen OmpA codiert für das äußere Membranprotein A von E.coli. Mischt man OmpA mRNA mit hfq und führt einen Toeprint Assay durch, so erhält man ein immer geringeres Toeprintsignal, je mehr hfq hinzugefügt wird. hfq behindert demnach das Ribosom bei der Bindung an die mRNA. Dabei sind zwei Ansätze denkbar, wie die ribosomale Bindung verhindert wird. Entweder das Protein besetzt die ribosomale Bindungsstelle oder es verändert die Konformation der mRNA so, dass das Ribosom nicht mehr binden kann. • Zunächst wird ein Toeprint Assay mit OmpA mRNA durchgeführt, indem der mRNA radioaktiv markierte Primer und reverse Transkriptase hinzugefügt wird. Man erhält ein Toeprint Signal, das die Bindung der 30S UE an die mRNA wiederspiegelt. (RT bricht ab, wo 30S UE an mRNA gebunden ist). • Weiters wird OmpA mRNA mit hfq versetzt, hfq wird anschließend mittels Proteinase K wieder abgebaut und in Folge ein Toeprint Assay durchgeführt. Das Signal unterscheidet sich vom ersten Versuch, die 30S UE konnte trotz Abbau von hfq nicht an die mRNA binden. Demnach verändert hfq dauerhaft die Konformation der mRNA. • Um diese Erkenntnis abzusichern wird eine durch hfq in ihrer Konformation veränderte mRNA erhitzt (80°) und langsam abgekühlt, wodurch mögliche Sekundärstrukturen aufgelöst werden. Im Anschluß wird wieder ein Toeprint Assay durchgeführt. Das Signal ergibt, das die 30S UE an mRNA binden kann. Damit steht fest, dass hfq die Struktur von mRNA verändert und nicht durch Bindung an die mRNA Translation verhindert. Hfq protects sRNAs from RNase E cleavage 130 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Hfq acts as an RNA chaperone Hfq stimulates annealing Functional activation and inactivation by trans-encoded regulatory RNAs Inactivation activation 131 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert mRNA silencing: the sodB-RyhB paradigm Wie erwähnt ist Eisen als Cofaktor für aktive Zentren multipler Enzyme essentiell. Entzieht man Bakterien Eisen (z.B. durch Chelierung mittels EDTA), werden sie sich bald den unsichtbaren Engelschören anschließen, denn: Das Gen sodB codiert für das Enzym Superoxid-Dismutase. Dieses wandelt Superoxid in H2O2 um, um die Zelle vor oxidativem Stress zu bewahren. Superoxid-Dismutase benötigt dazu Fe3+. Anm: das produzierte H2O2 wird durch andere Enzyme weiter abgebaut, da H2O2 DNA schädigt. Die Aufnahme von Eisen wird durch das Protein Fur reguliert. hfq reguliert die Translation von Fur und dadurch indirekt die Produktion der Superoxid-Dismutase, die Eisen benötigt. In einem hfq- Stamm ist sowohl die Konzentration von Fur als auch von SuperoxidDismutase erhöht. Bei langsamem Wachstum wird viel hfq produziert, dadurch wird die Konzentration von Fur niedrig gehalten. Eine niedrige Fur Konzentration aktiviert Oberflächenrezeptoren, die Eisen aus der Umgebung aufnehmen. Liegt wenig Eisen und damit inaktives Fur vor, kann die small RNA RhyB transkribiert werden. RhyB inaktiviert Gene, deren Produkte Eisen benötigen z.B. bindet es an die Ribosomenbindungsstelle von sodB und verhindert dessen Translation. Ist RhyB an SodB gebunden, wird sodB leicht durch RNase E gespalten und abgebaut werden. RyhB-SodB base-pairing creates a new RNase E cleavage site in the SodB 5' coding region 132 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Spaltung des SodB-RhyB RNA Komplexes durch RNase E In RNase E Mutanten sind beide RNAs stabiler. • Zunächst wird geprüft, wo sodB ohne RhyB gespalten wird. sodB mRNA wird mit Ribosomen und RNase E sowie speziell erzeugten Sonden vermischt. Diese Sonden sind komplementär zu kurzen Strecken der mRNA. Kann eine Sonde nicht an die mRNA binden, fand an dieser Stelle ein Schnitt durch die mRNA statt, die Sequenz der Sonde ist damit Komplementär zur RNase E Schnittstelle. Es fand sich eine Spaltstelle bei Codon 30. • Dem Gemisch werden hfq und RhyB hinzugefügt und erneut mit Sonden geprüft. Es fand sich dabei in sodB eine andere Spaltstelle bei +12 (nahe am Startcodon). Durch Bindung von hfq und RhyB an die mRNA wird Ribosomenbindung verhindert, eine neue Spaltstelle für RNAse E freigelegt und dadurch die Wahrscheinlichkeit einer Spaltung erhöht. RNaseIII cleavage of RyhB is stimulated by base-pairing with SodB mRNA RhyB kann durch RNase III gespalten werden, wenn es an sodB mRNA gebunden ist. In einem RNase III- Stamm, in dem durch eine Mutation RNase III bei Temperaturerhöhung inaktiviert wird, wird die RhyB RNA bei erhöhter Temperatur wesentlich stabiler im Vergleich zu Temperaturen in denen RNase III funktioniert. Model for sRNA/mRNA turnover Wie werden RNA Komplexe wieder gelöst, nachdem eine Stresssituation erfolgreich überwunden wurde (we shall overcome und so)? Masse et al., 2003 Both sRNA and mRNA are degraded in an RNase E dependent manner 133 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Initial events in sodB and RyhB inactivation RhyB-regulated genes • • • • bfr and ftn encode bacterial ferritins (iron storage proteins) sdhCDAB operon encodes succinate dehydrogenase acnA and fumA encode aconitase and fumarase. sodB encodes an iron-containing superoxide dismutase. Activation by a non-coding RNA 3. mRNA activation: the RpoS-DsrA paradigm 134 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert A network of small non-coding RNAs controls synthesis of outermembrane proteins Small non-coding RNAs and the bacterial outer membrane Current Opinion in Microbiology Vol.9, Iss.6, Dec. 2006, pp.605-611 Basepairing between the homologous regions of RprA and rpoS mRNA opens the hairpin permitting translation 9. Other mechanisms Translationale Kopplung erfolgt häufig bei überlappenden Genen. Negative translationale Kopplung: Liegen die Gene A und B überlappend vor und weist Gen A eine stärkere Ribosomenbindungsstelle als Gen B auf, wird Gen A verhältnismäßig stärker translatiert werden als Gen B. Positive translationale Kopplung: Die Translation eines Gens ist für die Translation eines weiteren Gens notwendig. Beim Phagen Lambda liegen einige Gene nicht überlappend vor, allerdings befinden sich Start und Stopcodon eines Gens in unmittelbarer Nähe des Startcodons eines zweiten Gens, dessen SD Sequenz durch eine Sekundärstruktur der mRNA zunächst nicht zugänglich ist. Wird das erste Gen translatiert, öffnet das Ribosom diese Sekundärstruktur in der sich die SD des zweiten Gens befindet. Dadurch kann das zweite Gen erst translatiert werden, nachdem das erste Gen translatiert wurde (positive translationale Kopplung). 135 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translational control in MS2 phage Translational Attenuation ermC (23S rRNA methylase) → erythromycin resistance cat, cmlA (chloramphenicol acetyltransferase) → chloramphenicol resistance Erythromycin binds to domain V of the 23S rRNA (hydrogen bonds with nucleotides 2058 and 2059) in the 50S subunit of the ribosome and inhibits translocation. Chloramphenicol - Binds to the 50S subunit (23S rRNA) of the ribosome. - Inhibits binding of the amino acid end of the tRNA and formation of the peptide bond. - Toxicity particularly in bone marrow as a result of inhibition of mitochondrial protein synthesis. Translationale Attenuation findet sich häufig bei Resistenzgenen z.B. beim CAT-Gen von B.subtilis. CAT codiert für Chloramphenicolacetyltransferase, das Chloramphenicol inaktiviert, indem es einen Acetylrest anhängt. CAT mRNA liegt immer in ausreichender Konzentration in der Zelle vor, um sofort reagieren zu können, wenn das Bakterium mit Chloramphenicol in Berührung kommt. Würde die mRNA allerdings in Abwesenheit von Chloramphenicol translatiert, würde die Zelle ständig Energie aufwenden müssen. zur Kontrolle der Translation besitzt die mRNA eine Leader Sequenz, die für 6-7 AS codiert. Das Ribsom translatiert diese kurze Sequenz und löst sich anschließend wieder von der mRNA ab. 136 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Chloramphenicol bindet an 23S rRNA und stoppt die Elongation, da es eine Bindung von weiteren tRNAs verhindert. Im Fall der CAT mRNA bewirkt der Stop des Ribosoms, dass eine nachfolgende Sekundärstruktur der mRNA aufgelöst wird, die die SD Sequenz des eigentlichen CAT Gens enthält, dadurch für weitere Ribosomen zugänglich wird und Chloramphenicolacetyltransferase synthetisiert werden kann. Ein ähnlicher Mechanismus findet sich bei der Erythromycin Resistenz. Transcriptional attenuation Transkriptionale Attenuation findet sich häufig bei Genen und Operons, die mit der Biosynthese zu tun haben. Diese Gene bzw. Operons besitzen eine Leadersequenz, die mehrfach für die AS codiert, deren Biosynthese durch die nachfolgenden Gene erfolgt. Bsp: Trp-Operon. Die mRNA des Operons bildet nach der Leader Sequenz eine Sekundärstruktur. Ist ausreichend Trp vorhanden, translatiert das Ribosom die Leadersequenz und löst sich durch die folgende Sekundärstruktur wieder ab. Steht wenig Trp zur Verfügung, ist auch die TrptRNA Konzentration niedrig und das Ribosom muss die Translation der Trp-Leadersequenz stoppen. Der Stop bewirkt eine Konformationsänderung der bereits translatierten mRNA, wodurch sich wiederum die folgende Sekundärstruktur auflöst. Nach dem Auflösen der Sekundärstruktur löst sich das Ribosom nicht von der mRNA sondern translatiert weiter und kann so die Enzyme zur Trp-Biosynthese erzeugen. Es handelt sich hier um eine Mischung zwischen translativer und transkriptiver Attenuation. Summary 137 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bläsi Foliensatz 3 Leaderless mRNA Leaderless mRNA and ribosomal recruitment? • • • • 5'-AUGNNNNNNN-//-3' Bacteria Gram-negative bacteria E. coli (λcI; tetR),C. crescentus, T. thermophilus Gram-positive bacteria Streptococci, Lactococci, Streptomyces, Corynebacteria Mycoplasma pneumonia Archaea Crenarchaeota (S. solfataricus) Euryarchaeota (H. salinarum; M. janaschii) Eukarya (G. lamblia) Organelles (human mitochondria) number leaderless mRNAs 6 -8 ~ 35 > > mehrere 100 5 -10 /> 4 9 -10 Leaderless mRNAs weisen keine SD Sequenz auf, sondern starten direkt mit dem Startcodon AUG. Sie ist in allen Organismen, sowohl in Pro- als auch in Eukaryoten translatiertbar. Sie kommen vor allem in gram+ Bakterien vor, hier vor allem bei Genen, die Resistenz gegen selbst produzierte Antibiotika verleihen. Bringt man leaderless mRNA in gram- Bakterien ein, so kann diese translatiert werden, normale mRNA aus gram- Bakterien kann in gram+ Bakterien allerdings nicht translatiert werden, da sie S1 benötigt, das viele gram+ Bakterien nicht besitzen. Bei Archae z.B. bei Sulfolobus solfataricus sind ca. 70% aller mRNAs monocistronisch und leaderless, es handelt sich dabei um nichtkanonische mRNA. In Mitochondrien sind alle mRNAs leaderless, sie weisen eine bakterienähnliche translationale Maschinerie auf. Translation of a leaderless mRNA in heterologous translation systems cI weist ist eine mit AUG beginnende leaderless mRNA. In Versuchen wurde konnte sie in E.coli, Sulfolobus und Reticulocytenlysat aus Kaninchenaugen translatiert werden. OmpA mRNA aus E.coli ist bei Versuchen im direkten Vergleich damit nur in E.coli, aber nicht in Sulfobolus oder Eukaryoten translatierbar. Es wird daher angenommen, dass es sich bei leaderless mRNA um eine alte Form von mRNA handelt, bevor sich ribosomale Bindungsstellen entwickelten. 138 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The db-adb interaction does not exist Translation Systems IF2 ist ein durch alle Organismen hoch konservierter translationaler Faktor. In Prokaryoten erhöht er die Bindungseffizienz der fMet-tRNA an die P-site des Ribosoms. Da die translationale Maschinerie in Mitochondrien der bakteriellen sehr ähnlich ist, dürfte IF2 hier dieselbe Funktion ausüben. In Archae und Eukaryoten dient IF2 demgegenüber der Bindung der großen ribosomalen UE an die kleine. Im Fall der leaderless mRNA kann ein Ribosom nur dann den Anfang der Translation finden, wenn es über die Initiator-tRNA an an das Startcodon bindet. Zu diesem Zweck muss das Ribosom bereits vor der Bindung an die mRNA einen Komplex mit der tRNA eingegangen sein. Um diese Situation zu begünstigen, kann mit Hilfe von Plasmiden die Konzentration von IF2 in der Zelle erhöht werden. Durch diese Konzentrationserhöhung sollte die Expression von leaderless mRNA gesteigert werden. Are leaderless mRNAs recognized by a 30S -initiator –tRNA complex? Bei geringer IF2 Konzentration findet sich selten fMet-tRNA Bindung an Ribosomen. Bei hoher IF2 Konzentration findet sich häufige fMet-tRNA Bindung an Ribosomen. IF2: accelerates the formation of the codonanticodon interaction by promoting efficient binding of fMet-tRNAfMet to the ribosomal PSite. 139 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Competition Assay zur leaderless mRNA In vitro translation competition assay with increasing concentrations of IF2 OmpA mRNA und cI mRNA werden in vitro translatiert. Abhängig von der hinzugefügten IF2 Konzentration steigt bzw. sinkt die Translation der cI mRNA, während die OmpA mRNA von der IF2 Konzentration unabhängig konstant translatiert wird. Die Konzentration von cI steigt proportional zur IF2 Konzentration, während OmpA von der IF2 Konzentration unabhängig ist. Selection of a 5'-terminal AUG and an internal canonical ribosome binding site in the presence of IF2 and an IF2 mutant protein Roter Punkt … normale mRNA wird in jedem Fall translatiert. Grüner Punkt … leaderless mRNA -) -IF2: keine Bindung von Ribosom und tRNA an leaderless mRNA, keine Translation. -) IF2 wt: Bindung an leaderless mRNA, Translation Es wird eine mRNA erzeugt, die mit einem Startcodon (quasi leaderless) beginnt und zusätzlich downstream eine SD Sequenz mit normalem AUG Startcodon aufweist.Wird damit ein Toeprint Assay durchgeführt (ausreichend mRNA, 30S UE, Initiator-tRNA, anschließend reverse Transkriptase), wird sich folgendes Geschehen auf wundersame Weise zutragen: • ohne IF2 (Spalte –IF2) erhält man ein Toeprint Signal, das der Bildung des ribosomalen Komplexes nur an der downstream liegenden Ribosomenbindungsstelle aufweist. • wird IF2 hinzugefügt (Spalte IF wt), erhält man sowohl ein Toeprint Signal für die Ribosomenbindungsstelle als auch für das einsam liegende Startcodon. • fügt man mutiertes IF2 hinzu (IF R700T), das die Initiator-tRNA nur schlecht an das Ribosom bindet, erhält man so gut wie kein Toeprint Signal für das einsam liegende Startcodon. Damit ist ersichtlich, dass IF2 für die Translation von leaderleass mRNA notwendig ist. 140 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translational control of leaderless mRNA • • • no downstream cis-elements IF2 (stimulates) other components of the translational machinery? Translation initiation factor 3 Fidelity Factor • 70S dissociation factor • Accelerates the rate of codon-anticodon interaction at the Psite. • Discriminates against - non-canonical start codons (AUU) - 30S initiation complexes at 5' AUGs? Sorgt dafür, dass ein Komplex nur dort gebildet wird, wo sich tatsächlich eine Ribosomenbindungsstelle befindet. IF3 diskriminiert gegen alle Codons außer AUG und verändert zu diesem Zweck die Konformation des Ribosoms. Im Versuch mit leaderless mRNAs die als Startcodon AUU benutzen, sinkt die Translation wenn IF3 hinzugefügt wird, da das Ribosom durch die Konformationsänderung leicht von der mRNA abdiffundiert. Experiment: Beweis, dass IF3 gegen leaderless mRNA selektioniert (Toeprint). - Spezielle mRNA mit AUG am 5'-Ende und einem weiteren AUG mit vorgelagerter SD (siehe vorne) 5'-AUG-SD-AUG-3'. + Ribosom + tRNAi. Ein Toeprint assay ergibt zwei Banden für beide AUGs. - Die Zugabe von IF3 mit anschließendem Toeprint Assay ergibt nur eine Bande für das AUG mit SD Sequenz. The translational efficiency of a leaderless mRNA is decreased at elevated levels of IF3 in vivo Relative IF2 and IF3 levels dertermine the translational efficiency of a leaderless reporter gene in vivo Expression von Leaderless mRNA: +IF2, +IF3 … normale Expression -IF2, +IF3 … niedrigste Expression +IF2, -IF3 … höchste Expression 141 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Increased expression of a leaderless tetR-lacZ fusion in infC (IF3) mutant strains Mittels Plasmid wird ein leaderless tetR-LacZ Fusionsgen eingebracht und die Expression der ßGalactosidase überprüft. Im Vergleich zum Wildtyp findet sich in IF3 Mutanten eine höhere Expressionsrate der leaderless mRNA. Kinetic discrimination against 30S initiation complexes at 5' AUGs Links … leaderless mRNAs können im Vergleich nur schwach mit der 30S UE interagieren. Translation initiation pathways in Bacteria Translation initiation of leaderless mRNAs is not intrinsically controlled but by components of the translational machinery. ad A) canonical mRNAs ad B) leaderless mRNAs Are non-dissociated 70S ribosomes capable of initiating translation at 5' terminal start codons? 142 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translation initiation with 70S ribosomes at 5'-terminal start codons but not at internal starts! Translation initiation with 70S ribosomes: an alternative pathway for leaderless mRNAs Nucleic Acids Research, 2004, Vol.32, No.11 pp.3354-3363 Cross-linking of 70S monosomes results in selective translation of leaderless cI mRNA Upon inactivation of ribosome recycling factor the level of 70S monosomes is increased 143 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Under these conditions the leaderless cI mRNA is preferentially translated in vivo: and in vitro: Transfer of growing cells from 37 to 10-15°C triggers the cold shock response • • • Cells growth and protein synthesis stop and resume at lower rates after 1-4h. A subset of cold shock proteins (CspA, CspB, CspG, CspI, ScdA, RbfA) is induced over 10fold. A subset of proteins involved in housekeeping transcriptional / translational control (IF-2, NusA, HN-S, Pnp, GyrA, RecA) is induced 2-10 fold. Bakterien sind als Einzeller stark für Kälte anfällig und müssen sich vor den Folgen tiefer Temperaturen schützen. E.coli besitzt beispielsweise Mechanismen, die es auch noch bei 5°10°C lebensfähig halten, auch wenn dabei der Stoffwechsel verlangsamt wird. Zur Regulation auf Translationsebene werden bei tiefen Temperaturen sogenannte cold shock proteins erzeugt. Es handelt sich dabei um RNA Chaperone bzw. Chaperonine, die verhindern, dass sich RNA bei tiefen Temperaturen fehlerhaft faltet bzw. eine solche Faltung verhindert, um die mRNA translatierbar zu halten. Inefficient translation Major problem: poor translation efficiency • RBS and start codon insufficient to direct ribosome binding • Increased 2° structure • Slow kinetics 144 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert CspA family RNA Chaperon CspA: 7kD; 10% CS protein synthesis • five antiparallel ß strands • RNA-binding motifs in ß2 and ß3 • binds RNA with low affinity, without sequence specificity • aromatic residues on surface important for nucleic acid binding • increases translation rate of mRNA • increases suscepribility of RNA to RNase attack CspB, CspG, CspI: related to CspA; cold shock inducible Cold shock proteins facilitate translation (initiation) by acting as RNA chaperones From initiation to elongation Zu Beginn der Elongation müssen die Initationsfaktoren vom Ribosom gelöst werden. Anschließend bindet eine 50S UE, es wird der 70S Initiationskomplex gebildet, in dem fMet-tRNA an der P-Site gebunden und die A-Site unbesetzt ist, da hier IF2 die Bindung von tRNAs bisher verhindert hat. Für die Elongation sind die Faktoren EF-Tu, EF-Ts und EFG essentiell. Diese Faktoren sind für Bakterien spezifisch, es gibt kaum Homologien zu eukaryotischen Faktoren. Daher wäre hier ein guter Ansatzpunkt für Antibiotika. Viele Imidazolverbindungen stören die Interaktion zwischen EF-Tu und EF-T, Kirromycin bindet EF-Tu an das Ribosom, wodurch die weitere Translation unterbrochen wird. 145 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Each ribosome has a binding site for mRNA and three binding sites for tRNA molecules • • • The P site holds the tRNA carrying the growing polypeptide chain. The A site carries the tRNA with the next amino acid. Discharged tRNAs leave the ribosome at E site. Elongation cycle Elongation factors: • EF-Tu • EF-Ts • EF-G EF-Tu & EF-G are small GTP-binding proteins. Colors: large ribosome subunit, cyan; small subunit, pale yellow; EF-Tu, red; EF-G, blue. tRNAs, gray, magenta, green, yellow, brown. • • Ablauf der Elongation: • Eine aa-tRNA (Aminoacyl-tRNA) kommt daher und wird durch aktives EF-Tu-GTP (rot) gebunden (Ausnahme: fMet-tRNA). EF-Tu-GTP bringt die aa-tRNA zur A-Site eines Ribosoms, dessen P-Site bereits besetzt ist. Bindet eine aa-tRNA in der ASite, wird die tRNA ohne AS aus der E-Site freigesetzt. Das gebundene GTP wird gespalten, EF-Tu-GDP wird freigesetzt, ist in dieser Form inaktiv und bindet deshalb aa-tRNA nicht effizient. Um den Faktor EF-Tu wieder in die aktive Form zu überführen, wird durch den Faktor EFTs das gebundene GDP mit GTP ausgetauscht. Nachdem eine aa-tRNA an der A-Site gebunden hat und EF-Tu sich vom Ribosom gelöst hat, wird die Polypeptidkette von der tRNA in der P-Site mit der neuen AS der tRNA in der A-Site verknüpft. Es gibt keine Peptidyltransferase, die Peptidbindungen knüpft, dieser Vorgang wird von der 50S UE durchgeführt, die rRNA dient als Ribozym. Im Detail wird ein durch das Ribozym dirigierter nucleophiler Angriff durch die AS in der A-Site auf die C-terminale Carboxylgruppe der Peptidkette in der P-Site durchgeführt. Anschließend bindet das Motorprotein EF-G, das das Ribosom unter Hydrolyse von GTP um ein Codon weiterschiebt, EF-G löst sich wieder vom Komplex ab. Die unbeladene tRNA liegt jetzt in der E-Site und kann das Ribosom verlassen, die tRNA mit der Polypeptidkette liegt in der P-Site, die A-Site ist leer. 146 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Elongation of translation in bacteria Das Anticodon der aa-tRNA bindet an die A-Site der 30S UE. Codon-Anticodon-WW bewirken eine Konformationsänderung von EF-Tu und die Hydrolyse von GTP. EF-Tu ist in der Lage, Proofreading zu vermitteln. Bringt EF-Tu eine aa-tRNA zur A-Site, deren Anticodon das aktuelle Codon der mRNA nicht vollständig bindet, liegt EF-Tu nicht korrekt in das Ribosom eingebettet, GTP wird nicht gespalten und der Faktor dissoziiert mit der tRNA wieder ab, bevor die AS in das Peptid eingebaut werden kann. Die Fehlerrate der Translation liegt bei 10-4 (alle 500010000 AS ein Fehler). Fehlerhafte Proteine werden abgebaut, nachdem von jeder mRNA mehrfach Proteine erzeugt werden fallen einige fehlerhafte Proteine üblicherweise nicht ins Gewicht. As the aa-tRNA is deliverd by EF-Tu to the A site on the ribosome, GTP on EF-Tu is hydrolyzed to GDP + Pi, which promotes dissociation of EF-Tu. The hydrolysis depends on codon-anticodon recognition correctly positioning the aa-tRNA in relation to the large ribosomal subunit. EF-Tu colored red. Molecular Mimicry Durch Röntgenstrukturanalyse ist bekannt, dass sich die Faktoren EF-G sowie EF-Tu das aa-tRNA gebunden hat strukturell sehr ähnlich sind. Man spricht in diesem Zusammenhang von molecular mimicry, die ähnliche Form ermöglicht Interaktion mit derselben Site (A-Site). Gemeinsamkeiten von EF-Tu und EF-G: • sind sich strukturell sehr ähnlich. • beide sind aktiv wenn GTP gebunden und inaktiv wenn GDP gebunden ist. • es kann nur entweder EF-Tu oder EF-G an Ribosom binden. 147 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The N on the incoming aminoacid makes a nucleophilic attack on the activated carboxyl group of the growing chain to form the new pepride bond. Das Peptidyltransferasezentrum liegt in der 23S rRNA der 50S Untereinheit. Hier kommen sich Amino- und Carboxylgruppen einander physisch so nah, dass eine Bindung mittels Konformationsänderung der umgebenden RNA vermittelt werden kann. Ribosome Structure and the Mechanism of Translation Cell, Vol.108, 557-572, February 22, 2002, ©2002 by Cell Press Drittes Bild: Anscheinend ist die neu mit der Peptidylkette beladene ehemalige AminoacyltRNA schon irgendwo auf A- UND P-Site. EF-G-GTP sorgt für die endgültige Translokation zur P-Site. 148 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert EF-G-GTP binds in the vicinity of the A site. EF-G-GTP binding my push the tRNA with attached nascent polypeptide from the A site to the P site. Unloaded tRNA that was in the P site shifts to an exit site. Since rRNAs are linked to mRNA by codonanticodon base pairing, the mRNAs would move relative to the ribosome. Wird GTP durch das ähnliche Molekül GMP-PCP ersetzt, ist EF-G-GMP-PCP zwar in der Lage an die A-Site zu binden. Ohne die Hydrolyse von GTP verläuft aber die Translokation sehr langsam und EF-G wird nicht (oder nur schlecht?) freigesetzt. Elongation Cycle of protein biosynthesis Incorporation of unusual amino acids: the 21st and 22nd amino acids Selenocysteineis the major biological form of selenium (Se) Bei Selenocystein handelt es sich um Cystein, das mit Selen modifiziert wurde. Selen wird im aktiven Zentrum einiger Enzyme benötigt; z.B. Deiodinase, Glutathionperoxidase zur Inaktivierung freier Radikale in Säugern, Formatregulon zum Formatabbau in E.coli. Die tRNA für Selenocystein wird folgendermassen erzeugt. Eine spezielle tRNA, sec-tRNA, bindet zunächst über eine spezifische Aminoacyl-tRNA Synthetase Serin. Ein Enzym wandelt die tRNA in Aminoacrylyl-tRNA um. Ein weiteres Enzym hängt Cystein und Selen an, wodurch Selenocysteyl-tRNA entsteht, die jetzt als tRNA für Selenocystein dient. 149 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translational recoding mechanisms Se-Cys Insertion Selenoprotein synthesis A. Böck, K. Forchhammer, J. Heider and C. Baron TIBS (1991) 16:463 Diese Enzyme katalysieren oft Oxidationen / Reduktionen. Enthalten meist ein Selenocystein, das einen Teil des aktiven Bereichs darstellt. The genes coding for these enzymes have stop codons!! Selenocystein wird in der mRNA über ein Stopcodon codiert. Im Normalfall können diese Proteine daher nicht translatiert werden. In Bakterien wurde zusätzlich zu EF-Tu, EFT und EF-G ein weiterer Elongationsfaktor, SELB, gefunden. SELB hat dieselbe Funktion wie EF-Tu, nur dass es einzig Selenocysteyl-tRNA bindet und zur A-Site des Ribosoms transportiert. Das Anticodon der sec-tRNA komplementiert das Stopcodon der mRNA und kann an der A-Site binden. Zur Bindung von SELB an die A-Site des Ribosoms ist noch ein zusätzlicher Faktor nötig, um zwischen normalen Stopcodons und für Selenocystein codierenden Codons unterscheiden zu können. Model of selenocysteine insertion by bacteria Changes in codon meaning can be caused by mutant tRNAs or by tRNAs with special properties. The reading frame can be changed by frameshifting or bypassing, both of which depend on properties of the mRNA. In Bakterien bildet die mRNA direkt nach einem Stopcodon, das für Selenocystein codiert, eine Sekundärstruktur (Hairpin) aus. Erst durch die Bindung an diese Hairpinstruktur ermöglicht es SELB an die A-Site des Ribosoms binden zu können. In Archae und Eukaryoten bildet sich diese Sekundärstruktur am Ende der mRNA aus, SelB bindet daran, führt eine Konformationsänderung der mRNA durch und fügt die Selenocysteyl-tRNA erst bei dem dafür vorgesehenen Stopcodon hinzu. 150 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert tRNAsec and SELB SELB unterscheidet sich von EF-Tu durch eine zusätzliche Domäne am deren C-terminalem Ende, über welche die spezifische Sekundärstruktur der mRNA (SECIS) gebunden wird. Insertion of selenocysteine at UGA codons requires a selenocysteine insertion element (SECIS) Bacteria: One protein, two functions Mammals: Two proteins, Archaea: one function? Methanococcus maripaludis Leibundgut et al. (2005) Embo J. 24:11 151 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Programmed frameshifting +1 Frameshifting In decoding E. coli release factor 2, the frameshifting occurs early in the coding sequence and is required to give a functional product. +1 frameshifting … der Leserahmen verschiebt sich um ein nt downstream. -1 Frameshifting: E. coli dnaX dnaX encodes two subunits (tau and gamma) of DNA polymerase III, the main replicative polymerase. Tau results from standard decoding, and gamma from 50% of the ribosomes shifting to the 21 frame two-thirds of the way through the coding sequence and terminating one codon later at a UGA stop codon. -1 frameshifting … der Leserahmen verschiebt sich um ein nt upstream. Frame shifting ist sowohl in Pro- als auch in Eukaryoten weit verbreitet. Frameshifiting kann der Regulation bestimmter Gene dienen, indem z.B. Stopcodons gelesen oder nicht gelesen werden. Frame shifting wird durch Faktoren z.B. RF2 vermittelt. +1 frameshifting E. coli release factor 2 gene RF2 (Release Factor) bindet in der ASite an UGA Stopcodons und terminiert die Translation, indem die Polypeptidkette auf ein H2O übertragen und dadurch freigesetzt wird. Autoregulation: Im Beispiel terminiert RF2 die Translation der eigenen mRNA an einem nach einigen Codons liegenden UGA relativ rasch, wenn seine Konzentration hoch genug ist. Liegt RF2 in geringer Konzentration vor, stoppt das Ribosom am UGA, ohne dass die Translation sofort durch RF2 abgebrochen wird. Upstream dieses UGA befindet sich eine SD ähnliche Sequenz, das Ribosom bindet an dieser Sequenz neu und startet die Translation neu. Durch diese Wanderung des Ribosoms kommt es zu einem +1 frameshift, wodurch das Ribosom das Stopcodon an dem es zunächst gestoppt war, nicht mehr als UGA sondern als GAC liest und weitertranslatieren kann. Der reading frame stimmt hier also nicht mit dem tatsächlich synthetisierten Produkt überein. Dieser Umstand ist bei Proteinsequenzierungen z.B. mittels Edman-Abbau zu beachten. Experiment: Test auf +1 frame shift: Die SD-ähnliche Sequenz auf der mRNA wurde mit der Anit-SD Sequenz auf einer 16S rRNA, die gegen Spectomyzin resistent ist, vertauscht. Mittels Plasmid werden beide Sequenzen (veränderte mRNA, resistente, veränderte 16S rRNA) in Bakterien eingebracht und diese auf ein Medium mit Spectomycin übertragen. Nachdem man das passende Genprodukt erhält, kann man schließen, das das Ribosom tatsächlich während der Translation erneut bindet und einen frameshift durchführt. (Naja … kann an sich nur funktionieren, wenn man entweder wie im Bild oben angedeutet eine leaderless mRNA hat oder auch die erste SD Sequenz auf der mRNA mit der Anti-SD Sequenz vertauscht. Außerdem muss man noch irgendwie die RF2 Konzentration in den Bakterien beinflussen, damit sie niedrig ist bzw den Test in vitro mit allen nötigen Komponenten + Spectinomycin durchführen. Denk ich mir halt.) 152 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Prinzipeller Test auf Frameshift: Man baut ein Reportergen (z.B. LacZ) in das Gen ein, in dem ein Frameshift vermutet wird. Je nachdem wie man das RG eingbaut - ob die Translation ohne oder nach erfolgtem vermuteten frameshift stattfinden soll – kann über entstandenes Produkt ermittelt werden, ob frameshift stattfand oder nicht. The programmed +1 frameshifting in decoding the Escherichia coli gene for RF2 Frameshifting in the E. coli dnaX gene The τ (normal translation) and γ subunits (–1 frameshifting) are derived from the dnaX gene The programmed –1 frameshifting in decoding the Escherichia coli dnaX gene Normalerweise wird das dnaX Gen vollständig translatiert, als Produkt entsteht die γ UE der DNA Pol III. Es kann sich aber eine Hairpinstruktur ausbilden, an der die Translation verzögert wird. Weiter Upstream findet sich eine SD ähnliche Sequenz. Wenn die 16S rRNA anti-SD Sequenz im Zuge der Verzögerung daran bindet, kommt es zu einem -1 Frameshift. Durch den Frameshift wird ein bisher als GAA gelesenes Codon zu einem UGA, die Translation wird vorzeitig abgebrochen. Das so freigesetze Protein dient als τ UE der DNA Pol III. dnaX Frameshifting cassettes from different bacteria 153 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert +1 frameshifting Mammalian antizyme gene • • • • In Säugern werden Polyamine mittels Ornithindecarboxylase (OCD) aus Ornithin erzeugt. Die Konzentration von Polyaminen muss reguliert werden, da sie an mRNA binden können und dadurch Pseudoknoten bilden, die Ribosomen verzögern und dadurch für frameshifts notwendig sind. OCD wird durch ein Antizym abgebaut. Die mRNA des Antizyms weist ein frühes Stopcodon auf, an dem die Translation üblicherweise vorzeitig abgebrochen wird. Ist die Polyaminkonzentration hoch, erzeugen die Polyamine in der mRNA des Antizyms einen Pseudoknoten, der die Translation vor dem UGA verzögert, das Ribosom führt einen +1 frame shift durch, statt dem Stopcodon wird GAU gelesen, die Translation kann fortgeführt und Antizym erzeugt werden. OCD wird abgebaut, es wird kein neues Polyamin erzeugt. Translational Introns Unter translationalen Introns versteht man mRNA-Bereiche, die vom Ribosom während einer laufenden Translation übersprungen werden. Dabei kann das Ribosom im Leserahmen bleiben d.h. ganze Codons überlesen oder in einen anderen Leserahmen wechseln. Translational introns: an additional regulatory element in gene expression TIBS 18 – August 1993 Rate of translation low > frameshifting high Test: Insert stop codons in non translated region. Synthesis of trans-dominant inhibitor. Beispiel T4 Gen 60: Hier bildet sich im codierenden Bereich der mRNA eine Sekundärstruktur. Das Ribosom ignoriert diesen einige hundert nts langen Bereich und translatiert nur die ungebunden vorliegenden Bereiche der mRNA, ohne die Translation zu unterbrechen. Beispiel Repressor trpR: Der Repressor besitzt am Nterminalen Ende eine Dimerisierungs- am C-terminalen Ende eine DNA-Bindungsdomäne und ist als Dimer aktiv, wenn die mRNA dieses Repressors normal translatiert wird. In 10-15% der Fälle wird ein Teil der mRNA (55nt) nicht translatiert und das Ribosom geht nach em Ende des ignorierten Stückes um -1 versetzt in einen anderen Leserahmen über. Dem auf diese Art erzeugte Repressor fehlt die C-terminale DNA-Bindungsdomäne. Dieser Repressor kann mit normal translatierten Repressoren ein Dimer bilden, das Dimer kann aber nicht mehr an DNA binden und seine Funktion ausüben. Auf diese Art wird die Konzentration des effektiven Repressors in der Zelle reguliert. No other product 154 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Test auf Translationale Introns Vermutet man ein Translationales Intron, führt man im Leserahmen ein Stopcodon in diese Intronsequenz ein. Handelt es sich tatsächlich um ein Intron, wird die Translation nicht unterbrochen, man erhält trotz eingeführtem Stopcodon ein Produkt, da das Intron vom Ribosom übersprungen wird. T4 DNA topoisomerase gene 60 bypassing Requirements: • matching GGA codons flanking an optimally sized 50 nt coding gap • a stop-codon • a nascent peptide signal Bypassing steps Translation termination Bacteria: RF1: UAA / UAG RF2: UAA / UGA RF3: stimulates RF1 + RF2 Bei Prokaryoten finden sich drei Releasefaktoren RF1-3. RF1 bindet die Stopcodons UAA und UAG, RF2 bindet UAA und UGA. RF3 stimuliert die Bindung von RF1 bzw. RF2. Die Releasefaktoren haben strukturelle Ähnlichkeit mit EF-Tu und EF-G, da auch sie an der A-Site binden (molecular mimicry). 155 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert A Posttermination Ribosomal Complex is the Guanine Nucleotide Exchange Factor for Peptide Release Factor RF3 Cell, Vol.107, 115-124, October 5, 2001, Copyright ©2001 by Cell Press ad II) Das Polypeptid wird von der PeptidyltRNA in der P-Site auf H2O übertragen und dadurch freigesetzt. Recycling of ribosomes: The ribosome recycling factor (rrf) rrf ... ribosome recycling factor, ähnelt strukturell den Elongations- und Releasefactoren, im Gegensatz zu diesen wandert er aber nicht in die A-Site, sondern interagiert außerhalb mit dem Ribosom. Ist RF3 vorhanden, bindet rrf an das Ribosom. EFG dringt in die A-Site ein, das Ribosom wird unter GTP Verbrauch vorwärts geschoben, wodurch sich die 50S UE von der 30S UE ablöst und freigesetzt wird. Die 30S UE ist immer noch an die mRNA gebunden. Interagiert sie mit IF3, das ja gegen alle tRNAs außer fMet-tRNA diskriminiert, löst sich auch die 30S UE von der mRNA, da sich an dieser Stelle kein AUG befindet. rrf ist bei einer Temperatur von 28°C aktiv und inaktiviert sich ab einer Temperatur von 42°C. Ab einer Temperatur von 42°C können Ribosomen somit nicht mehr von mRNA gelöst werden, die Zelle wird absterben. rrf kommt in Prokaryoten und Archae vor, nicht aber in Eukaryoten, hier wäre wieder eine gute Ansatzmöglichkeit für Antibiotika. Crystal Structure of Thermotoga maritima Ribosome Recycling Factor: A tRNA mimic Science (1999) 286:2349 (A) RRF zeigt Ähnlichkeit mit tRNA und mit Domäne 3-4 von EF-G. (B) links EF-G, rechts EF-Tu + tRNA 156 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The final steps of 70S disassembly require EF-G + IF3 Schritt 4 … RRF bindet an die A-Site. Schritt 5 … RRF und EF-G bewirken mittels GTP Hydrolyse die Dissoziation der 50S UE vom 70S Ribosom. Schritt 6 … IF3 bindet an die 30S UE und, löst die deacetylierte tRNA ab und verhindert eine Reassoziierung von 30S und 50S UE. Bläsi, Foliensatz 4 Abbau von mRNA Transcription-Translation-Degradation Chemical and functional half-lives of mRNA Chemical half-lives of mRNA transcripts correspond to 50% decrease in the concentration of the corresponding full-length transcripts after transcription has been blocked, e.g., inhibition of RNA polymerase by rifampicin. 157 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Differential mRNA stability in E. coli Differential mRNA stability of: • individual monocistronic mRNAs • segments within polycistronic transcripts In Prokaryoten liegt die Halbwertszeit von mRNA zwischen Sekunden und einigen Minuten, in Eukaryoten kann sie von 30 Minuten bis zu 25 Stunden betragen. Der hohe Turnover in Bakterien ergibt sich durch das schnelle Wachstum. E. coli RNases RNasen sind für Prozessierung von RNA (insbesonders tRNA) sowie für den Abbau von RNA verantwortlich. Exoribonucleasen bauen mRNA vom Ende her ab, Endoribonucleasen spalten mRNA in der Sequenz. Prokaryoten besitzen nur Exoribonucleasen, die mRNA vom 3' Ende her abbauen. Eukaryoten haben sowohl Exoribonucleasen die mRNA vom 3' Ende, als auch solche, die vom 5' Ende her abbauen. Endoribonucleases: RNase E RNase I RNase III RNase HI and HII RNase P Exoribonucleases: PNPase RNase II Oligoribonuclease RNase PH, RNase D RNase R, RNase BN, RNase T Degradosom (E.coli) • bestehend aus RNase E (Endonuclease), PNP (Polynucleotid Phosphorylase), Helicase (RhlB), DnaK. • RNase E ist der Hauptfaktor des Komplexes. Er schneidet Einzelstrang RNA, bevorzugt hinter stem loop Strukturen. • Helicase entwindet Sekundärstrukturen unter ATP Verbrauch. • PNP (Exonuclease) baut mRNA in 3'-5' Richtung ab. Sie ist eine einzelstrangspezifische Nuclease, ihre Funktion wird durch Sekundärstrukturen unterbunden. • Dna K … Protein Chaperon • RNase III ist nicht Teil des Degradosoms, schneidet Doppelstrang RNA und löst damit Sekundärstrukturen. Experiment: Nachweis einer bestimmten mRNA in einer Zelle • • • • • • Voraussetzung ist, dass die Sequenz bekannt ist. Es werden radioaktiv markierte, zur Sequenz komplementäre Oligonucleotide synthetisiert. Zellen werden mit heißem Phenol lysiert, Proteine werden dadurch denaturiert. Das Lysat wird zentrifugiert, im Überstand finden sich Nucleinsäuren. Es werden DNasen hinzugefügt, die vorliegende DNA abbauen. Die verbleibende RNA wird mit Formamid vermischt, wodurch die RNA denaturiert. Die denaturierte RNA wird mit den Oligonucleotiden vermischt und über ein Gel nach Größe getrennt, die amorphe Masse der mRNAs liegt irgendwo zwischen tRNA und rRNA. Befindet sich die gesuchte mRNA unter der Menge der RNAs, wird man mittels Röntgenfilm eine Bande erkennen können. 158 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Experiment: Nachweis von welchem Ende her mRNA abgebaut wird • • • • • • Es werden mRNAs mit einer Mindestlänge von 2knt benötigt, da kürzere mRNAs zu schnell abgebaut werden. Es werden Sonden synthetisiert, die entweder an das 3' oder das 5' Ende einer mRNA binden, bevor diese abgebaut werden. Um eine Aussage über den Abbau von mRNA zu machen, muss die Transkription vollständig gestoppt werden, damit keine neue mRNA mehr produziert wird. Zu diesem Zweck wird Rifampicin verwendet. Zu Zeitpunkt 0 wird Rifampicin hinzugefügt Zu späteren Zeitpunkten (t=0, t=1, t=2, etc) wird die Gesamt-mRNA extrahiert und auf ein gemeinsames Gel in verschiedenen Banden aufgetragen. Mittels Sonden die an den Enden der mRNA binden sieht man, dass die Menge der mRNA mit fortschreitender Zeit abnimmt. Fügt man einer Probe Sonden dazu die entweder nur am 3' oder nur am 5' Ende binden, sollte man nach einiger Zeit das Signal nicht mehr erhalten, von dem Ende her die mRNA abgebaut wird, da diese Enden schlicht und ergreifend (schluchz) bereits abgebaut wurden. mRNA decay in E. coli: RNases Escherichia coli RNase E 159 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Effect of 5' basepairing on the stability of ompA mRNA E. coli polynucleotide phosphorylase (PNPase) Stability determinants of mono-and polycistronic transcripts 160 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) 5' stability determinants 3' stabilizers 3' stem-loop structures protect mRNA from 3' exonucleases mRNA decay in E. coli 161 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Purification of the degradosome components Beim Degradosom handelt es sich eben nicht um eine einzige RNase, sondern um einen Proteinkomplex. E. coli RNA Degradosome The arginine-rich RNA-binding domain of RNase E retains the target RNA until the catalytic domain completes cleavages 162 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert RhlB helicase Binding to RNase E stimulates RhlB ATPase activity Helicase-facilitated degradation of an internal RNA stem-loop structure. Without RNA helicase activity, progression of the exonuclease (yellow) is blocked by the stemloop structure (pausing). The RNA helicase (orange) hydrolyzes ATP to anwind the stem-loop, facilitating RNA degradation by the exonuclease. E. coli Poly (A) polymerase I (PAPI) Poly-A Sequenzen von 10-40nt Länge fungieren in E.coli als Bindungsstelle für Nucleasen. Degradation of stem-loops with different length of poly (A) tails Bei der nicht rhoabhängigen Termination der Transkription bildet sich am 3' Ende der mRNA ein stem-loop, der der mRNA zusätzliche Stabilität verleiht, da Exonucleasen keine doppelsträngige RNA abbauen können. Je länger allerdings ein Poly A Schwanz nach einem Stem Loop ist, desto schneller kann die mRNA abgebaut werden, da die Bindung des Degradosoms an das 3' Ende besser wird und in Folge mittels Helicase den Stem Loop auflösen kann. 163 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 30S ribosomal subunits and endoribonuclease compete for 5' UTRs of E.coli transcripts mRNA stability and translation 1. Transcriptional decoupling (weak ribosome binding sites, T7 RNA polymerase) 2. Trans-translation Unmasking of nucleases-sensitive sites by desynchronization of transcription and translation Sequence and secondary structure of the E. coli ompA 5' UTR 164 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert The ribosome protects from RNase E cleavage at the 5'-end Experiment: Test auf Zusammenhang zwischen effizienter Translation und mRNA Stabilität An efficient Shine-Dalgarno sequence but not translation is necessary for lacZ mRNA stability in Escherichia coli Journal of Bacteriology, Mar. 1994, p.1683-1688, Vol.176, No.6 • • • • • Die SD Sequenz eines lacZ Gens wird durch eine effizientere SD Sequenz ersetzt (gelb markiert). Über die effizientere SD Sequenz bildet sich ein starker ternärer Komplex, Ribosomen sind häufig an mRNA gebunden, die mRNA wird stark translatiert. Das Degradosom kann nicht schneiden, da translatierende Ribosomen Ansatz- und Schnittstellen verdecken. Im Versuch steigt die Halbwertszeit der mRNA mit einer effizienten SD Sequenz auf 2,8 Minuten. Wird die SD Sequenz so verändert, dass Ribosomen nur schlecht daran binden können, verkürzt sich die Halbwertszeit auf 1,3 Minuten. Die Halbwertszeit einer mRNA steigt mit der Translationsrate, je stärker sie translatiert wird, desto stabiler wird sie. Mittels Chloramphenicol, das die Translation verzögert, lässt sich die Stabilität von mRNA ebenfalls erhöhen, da Ribosomen länger an die mRNA gebunden bleiben. 165 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Trans-translation • • • mRNA weist normalerweise ein Stopcodon am Ende der codierenden Sequenz auf. • Exonucleasen die vom 3' Ende her mRNA abbauen entfernen Stopcodons, sie stehen nicht mehr für den Abbruch der Translation zur Verfügung. • Wird eine mRNA ohne Stopcodon von Ribosomen translatiert, bleiben diese am Ende der mRNA gebunden ohne sich zu lösen. • Zur Auflösung dieser Situation werden die speziellen RNAs tmRNA oder SsrA verwendet. Es handelt sich dabei um eine 10S RNA, die wie eine tRNA mit Alanin beladen ist, weiters aber noch eine spezielle RNA Sequenz aufweist ("Tag Sequenz"), die vom Ribosom translatiert werden kann und mit einem Stopcodon endet. tmRNA tritt in die ribosomale A-Site ein (EF-Tu wird benötigt), das bereits translatierte Polypeptid wird auf Alanin übertragen. Anschließend wird die Sequenz der 10S RNA translatiert, über das Stopcodon werden Polypeptid freigesetzt und das Ribosom von der mRNA gelöst. Durch die "Tag" Sequenz ist das Polypeptid für den Abbau durch Proteasen markiert. tmRNA structure Potential mechanisms for SsrA recruitment 166 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Trans-Translation Degradation of a SsrA-tagged substrate by the ClpXP complex Model for binding, spontaneous denaturation and degradation of an SsrA-tagged substrate by Tsp protease Summary 167 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Regulation of Gene Expression in Eukaryotes In Eukaryoten sind Transkription und Translation räumlich getrennt, Transkription findet im Nucleus statt, das Transkript wird modifiziert und ins Cytosol transportiert, wo die Translation stattfindet. Das Transkript von Eukaryoten ist nicht direkt translationsfähig, sondern in codierende Exons und nicht codierende Introns aufgeteilt. Diese Introns müssen zuerst aus der mRNA entfernt werden, bevor eine Translation stattfinden kann. mRNA processing: Nuclear Splicing hn RNA … heterogeneous nuclear RNA Vor der Entfernung der Introns wird diese so genannte prä-mRNA modifiziert. Am 5' Ende wird eine so genannte CAPStruktur angebracht, am 3' Ende werden bis zu 200 A angehängt (PolyA-Schwanz). Diese Modifikationen sind für die Translation der mRNA essentiell. Der Vorgang bei dem die Introns aus der prä-mRNA entfernt und die Exons zusammengefügt werden, wird als Spleißen (splicing) bezeichnet und durch einen Komplex (Spliceosom) durchgeführt, der aus Proteinen und RNAs (hnRNAs, snRNAs) besteht. hallmark of nuclear pre-mRNA introns 5' Splice Site: GT 3' Splice Site: AG short consensus sequences at the exon-intron boundary 168 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Wird eine mRNA aus der gesamten RNA einer eukaryotischen Zelle mittels Northern Blot untersucht und entsprechend markiert, so findet sich nicht eine, sondern mehrere Banden, je nachdem wie viele Introns bereits aus der prä-mRNA gespleißt wurden. Splicing is a two stage process involving many steps • • • Das Spliceosom erkennt die für das Splicen notwendigen 5' (GU) und 3' (AG) Sites auf der mRNA, bringt sie in räumliche Nähe und vermittelt so eine Transesterifizierung mit zwei aufeinander folgenden nucleophilen Angriffen. • Im Intron findet sich eine so genannte Brach Site, die ein A enthält. Das OH dieses As führt einen nucleophilen Angriff auf die Phosphoesterbindung der 5' Site durch. Es entsteht die Lariatstruktur und ein freies OH an der 5' Site. Dieses freie OH der 5' site führt einen dirigierten nucleophilen Angriff auf die 3' Site des Introns durch, wodurch das Lariat freigesetzt und die beiden Exons miteinander verknüpft werden. Das freigesetzte Lariat wird für den Abbau wieder in lineare Form überführt. Lariat ist mittels Gelelektrophorese gut nachzuweisen. 169 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Spleißen ist kein enzymatischer, sondern ein autokatalytischer, das Spliceospm bringt die miteinander interagierenden Teil in räumliche Nähe, damit die Aktion ablaufen kann. Der Vorgang wird durch RNAs vermittelt, die durch gebundene Proteine korrekt positioniert werden. hnRNAs müssen aus dem Zellkern exportiert werden, im Cytosol binden sie an die benötigten Proteine und werden anschließend wieder in den Nucleus importiert. snRNAs des Spliceosoms müssen für Faltung ins Cytoplasma transportiert werden. Für diesen Transport werden zahlreiche Exportproteine benötigt. Im Cytoplasma werden die snRNAs freigesetzt, die Faltung wird durch hfq ähnliche SM Proteine und RNasen durchgeführt. Durch Importproteine werden die gefalteten snRNAs zurück in den Nucleus transportiert. (1) U1 bindet an 5' Splicestelle, U2AF (Auxilary factor) bindet an Pyrimidin (2) U2 bindet mittels ATP Hydrolyse an die Branch site (3) U5/U4/U6 Trimer bindet das Exon an der 5' Site über U5 und U2 über U6. (4) U1 wird freigesetzt, U5 rutscht vom Exon zum Intron, U6 bindet an 5' Site (5) Durch ATP Hydrolyse wird U4 freigesetzt. Das verhindert eine Interaktion von U6 mit U4 (da U6 Bindungsstelle schon von U4 besetzt bzw blockiert) … siehe Fig. 22.12 Paarung von U6 mit U2 - U6/U2 katalysieren Transesterifizierung - U5 bindet Exon an 5' Splicestelle - 5' Site wird geschnitten … es bildet sich Lariat (6) U2/U5/U6 an Lariat gebunden 3' Site wird geschnitten und Exons ligiert. (7) gespleißte RNA wird freigesetzt und Lariast linearisiert (und abgebaut). 170 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Mit Ausnahme von U6, das durch LSM Proteine gebunden wird, interagieren alle snRNAs mit SM Proteinen. SM Proteine binden snRNAs über PuAU3-6Gpu Sequenz. Bei Pflanzen finden sich 30 Faktoren die beim Spleißen eine Rolle spielen. Viele Krankheiten sind auf fehlerhaftes Spleißen zurückzuführen und führen zu teils schwerwiegenden genetischen Erkrankungen. U1-snRNP-Synthese Spliceosomal UsnRNP biogenesis, structure and function Current Opinion in Cell Biology 2011, 13:290-301 Schritt 1) U1 snRNA … Capping Schritt 2) durch Export-Faktoren wird snRNA ins Cytoplasma transportiert Schritt 4) Binden der Sm Proteine an Sm-Sequenz der snRNA Schritt 5) Cap Hypermethylierung, 3' Trimming Schritt 7) Import Schritt 8) Binden der U1 spezifischen Proteine an U1-snRNA + SmProteine 171 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Group I and group II Introns: Organelles and bacteria fold into a typical secondary structure Es gibt drei Splicereaktionen: a) Group I Introns … autokatalytisch, in Organellen und Bakterien b) Group II Introns … autokatalytisch, in Organellen und Bakterien c) nukleares Splicing … benötigt Spliceosom für Splicen (snRNPs) Group I Introns: self splicing Die Exons befinden sich weit auseinander, Introns sind relativ linear und zirkularisieren, sie bilden kein Lariat. • 3' OH Ende eines freien G greift 5' Ende des Introns an • 3' OH Ende von Exon 1 greift 5' Ende von Exon 2 an • 3' Ende von Intron greift 5' von Intron an Group II Introns: self splicing Sie weisen eine spezielle Sekundärstruktur auf, Exons befinden sich dadurch räumlich näher beieinander. Sie spleißen ausschließlich autokatalytisch, es wird ein Lariat gebildet. • OH Gruppe eines freien A attackiert Phosphodiesterbindung zwischen Exon 1 und Intron. • 3' OH greift Bindung zwischen Intron und Exon 2 an. Nuclear Splicing und Group II Splicing ähneln sich bei der Bildung des aktiven Zentrums. 172 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Group I introns: • • • • Nuclei of lower eukayotes Fungal mitochondria Phage T4 Bacteria Self-splicing / autosplicing Das Splicing der Group I Introns benötigt ein monovalentes und ein bivalentes Kation sowie einen Cofaktor. Es wird keine Energie in Form von ATP oder GTP benötigt. • • • 173 OH eines G am 3' Ende greift 3' Ende des Introns an. OH am 3' Ende von Exon A greift 5' Ende von Exon B an OH am 3' Ende des Introns greift 156 bp vor dem eigenen 5' Ende an, es ergeben sich zwei Produkte. V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Group I Introns weisen eine bestimmte Sekundärstruktur auf. Die Ausbildung dieser Struktur ist für den Ablauf des Splicens notwendig. • 9 Helices (P1-P9) … basengepaarte Regionen • P4 aus P und Q … aus Basenpaarungen zwischen konservierten Sequenzen • P7 aus S und R … aus Basenpaarungen zwischen konservierten Sequenzen, der Teil bei P7 entspricht dem Intron core. • Die Region P1 enthält das 3' Ende von Exon 1. • IGS: jener Teil des Introns, der mit Exon 1 paart. • P3, P4, P6, P7 ist die kleinste Region, die autokatalytisches Splicing durchführen kann. Splicing assay in vivo Mit diesem Verfahren wird Mutationsstudie in vivo möglich. Man kann die Auswirkung einer Mutation auf das Splicing anhand von X-Gal-Indikator Platten direkt ablesen. Es wird ein selbstsplicendes Intron in das LacZ Gen eingebracht. Nur wenn das Intron sich tatsächlich selbst aus dem LacZ Gen entfernen kann, wird die Kolonie auf X-Gal-Medium blau. 174 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert 3'-end formation PAP: Poly-A-polymerase CPSF: recognizes AAUAAA CFI, CFII: cleavage factors CFI + II sind Endonucleasen, CstF bindet G-U-reiche Sequenz downstream der Schnittstelle von CFI + II. So genannte Poly A-binding-Proteins (PBP) können an den Poly A Schwanz und weiters an die CAP binden, wodurch die mRNA zirkularisiert wird und sich die Stabilität erhöht, da die Enden nicht für Exonucleasen zugänglich sind. 175 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bläsi, Foliensatz 5 mRNA Translation in Eukaryotes Wie findet das Ribosom den Translationsstart? 40S UE bildet einen Komplex aus Met-tRNAi, eIF2 und GTP. Dieser Komplex bindet an die CAP Struktur der mRNA. Auch in eukaryotischer mRNA finden sich in der Umgebung des Startcodons gewisse Konsensussequenzen (Kozak Sequenz), die dem Ribosom ermöglichen den Start zu erkennen (obwohl eigentlich immer das erste AUG vom 5' Ende her das Startcodon ist). Im Gegensatz zu Prokaryoten werden die Interaktionen zwischen Ribosom und Sequenz nicht zwischen RNA-mRNA sondern ProteinmRNA gebildet. 176 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Die CAP Struktur Die CAP Struktur befindet sich am 5' Ende der mRNA und besteht aus einem methylierten Guanylrest. Diese Struktur ist für die Bindung des Ribosoms an die mRNA nötig und erhöht die Halbwärtszeit der mRNA. Der Guanylrest wird mittels Guanyltransferase angebracht und an Position 7 durch eine Methyltransferase methyliert. In allen höheren Eukaryoten wird weiters die Base, die auf das angebrachte Guanin folgt, an Position 2 mittels O-Methyltransferase methyliert. Eukaryotische mRNA lässt sich über den PolyA schwanz isolieren. Man erzeugt mittels PhenolChlorophorm ein Lysat und bindet die mRNAs über eine Säule mit Poly T-Oligonucleotiden. Durch eine hohe Salzkonzentration kann diese Bindung gelöst und die mRNAs isoliert werden. Der PolyA Schwanz ist sowohl für die Stabilität der mRNA als auch für die Initiation der Translation von Bedeutung. • • • • • • • • Der eukaryotische Intitationsfaktor eIF1 sowie der Faktor aeIF1 in Archae sind homolog dem prokaryotischen Faktor IF3 und diskriminieren somit gegen alle tRNAs außer für Startcodon AUG bzw. unterstützen die Bindung der Initiator-tRNA an die 40S UE. eIF2 ist ein Komplex aus drei UE und transportiert die Initiator-tRNA zum Ribosom. Durch Spaltung von GTP kann es wie IF2 wieder aus dem Ribosom freigesetzt werden. eIF2B recycelt eIF2 indem es bei diesem GDP mit GTP austauscht. Viren können bewirken, dass eIF2B GDP permanent bindet und nicht mehr zur Aktivierung von eIF2 zur Verfügung steht. eIF3 unterstützt die Bindung der 40S UE an die mRNA. eIF4 ist eine Helicase, die Sekundärstrukturen der mRNA aufzulösen, damit das Ribosom die mRNA ungestört translatieren kann. eIF4B fördert die die Aktivität der Helicase. eIF4e bindet die CAP Struktur. eIF4F dient als Adapterprotein mehrerer Faktoren. 177 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Translation inition in Eukaryotes The eukaryotic 40S translation initiation complex+ Regulation 178 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert In Eukaryoten liegen die meisten mRNAs zirkulär vor. Der PolyA Schwanz wird durch PBP (PolyA binding proteins) und das Adapterprotein 4G mit dem CAP verbunden. An 4G binden mehrere Proteine des Initiationsfaktors 3. Regulationen auf Translationsebene laufen in der Regel über Phosphorylierung ab. • Positive Regulation: Das Protein 4E-BP kann 4G binden, das dadurch nicht zur Bindung der CAP zur Verfügung steht. 4E-BP dient so der Regulation der translationalen Initiation. Durch Phosphorylierung von 4E-BP kann die Bindung an 4G wieder gelöst werden, das dadurch zur Bindung an CAP frei wird. • Negative Regulation: elF2α bindet tRNA und bringt sie zum Ribosom. Durch Phosphorylierung der α-UE von eIF2B kann an eIF2 kein GDP-GTP Austausch mehr vorgenommen werden, es wird keine Initiator-tRNA mehr zum Ribosom transportiert. Diese Art der Regulation kann durch Virusinfektion, Hitzschock, Eisenmangel, AS Mangel etc. induziert und weitere Translation verhindert werden. Diese Interaktionen wurden durch Two-Hybrid System, Affinitätschromatographie oder Coimmunpräzipitation nachgewiesen! Ribosomal recruitment to mRNA in Pro-and Eukaryotes • • • Unter Stressbedinungen wird die α UE von eIF2 induziert. GEF kann sich nicht mehr ablösen, durch GEF ist keine Austausch von GTP mit GDP möglich. Es findet keine Translation statt, da eIF2 im inaktiven Zustand keine tRNAi liefern kann. 179 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert RNA Sensors - Regulation of IFN-gamma by negative feedback Human IFN-γ mRNA activates PKR through a pseudoknot sensor to inhibit its own translation. (A) Pseudoknotin the 5'-untranslatedregion of human interferon-γ (IFN-γ) messenger RNA. Base pairing in the pseudoknot stem is shown in red and the start of the open reading frame in green. (B) Autoregulation of IFN-γ synthesis. IFN-γ mRNA translation, which is dependent on eukaryotic initiation factor 2 (eIF2), yields IFN-γ that can induce more RNAdependent protein kinase (PKR) expression in the cell, creating a negative feedback loop. Inactive PKR binds to the pseudoknot, leading to a local PKR activation. Resulting phosphorylation of eIF2α blocks further translation of IFN-γ mRNA. EMBO Rep. 2003 November; 4(11): 1043–1047. Eukaryotic ribosomes require initiation factors 1 and 1A to locate initiation codons Nature Vol 394, 27 August 1998 180 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Mechanisms of translation initiation in eukaryotes Scanning: Die CAP Struktur muss für die 40S UE zugänglich sein. Störende Sekundärstrukturen werden unter ATP Verbrauch mittels Helicase aufgelöst. Wenn die 40S UE an CAP gebunden ist, wandert sie bis zu einem Startcodon oder einer Kozak Sequenz unter ATP-Verbrauch an der mRNA entlang. Sekundärstrukturen werden durch Helicase entwunden. Reinitiation: Kommt ein Ribosom an ein Stopcodon, löst sich die 60S UE ab, die 40S UE bleibt gebunden und wandert unter ATP Verbrauch weiter an der mRNA entlang. Kommt es dabei zu einem Startcodon, kann tRNAi und im Anschluss ein 60S UE binden und Translation erneut aufgenommen werden. "Jumping" ... wurde bisher nicht bewiesen. Das Ribsom bindet am CAP und springt direkt zum AUG. Das wäre vorstellbar, wenn die mRNA nicht linear vorliegt. Internal Initiation: Wurde ursprünglich bei Viren entdeckt (Picornavirus, s.u.). Durch virale Proteasen wird der eukaryotische Translationsfaktor elF4G gespalten, es kann keine CAP abhängige Translation mehr stattfinden. Die viruseigene mRNA ist von CAP unabhängig und initiiert Translation an einer IRES (Internal ribosom entry site). IRES sind Sekundärstrukturen in der 5' NCR (Non coding region, am 5' Ende der mRNA) von Picornaviren, das Ribosom kann an dieser Sekundärstruktur andocken. Toeprinting bei Eukaryoten: Es werden sämtliche Faktoren gereinigt und einzeln zu mRNA, 40S UE und tRNA hinzugefügt. Je nachdem, ob man bei einem zugegebenen Faktor ein Toeprintig Signal erhält oder nicht, hat sich ein stabiler Komplex Ribosom-mRNA gebildet (oder nicht). Bei eIF1 beispielsweise erhält man ein schönes Signal (hurra). 181 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Internal initiation in viral mRNAs *Caspase (apoptosis) viral proteases .. cleave eiF4G: prevents cap-dependent initiation Caspasen spalten im Zuge der Apoptose beinahe an derselben Stelle. Fig.2. Schematic illustration of eucaryotic mRNA translation and major sites of viral regulation. Picornaviral mRNAs have no 5'cap 182 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert IRES: Internal ribosome entry site Interne Initiation benötigt IRES (internal ribosomal entry site). Dabei handelt es sich um komplexe Sekundärstrukturen der mRNA, die gemeinsam mit Proteinfaktoren eine Ribosomenbindung ermöglichen. Denkbar ist, dass Ribosomen über IRES entweder direkt an AUG binden oder von IRES aus auf der mRNA entlang wandern, bis sie an ein AUG gelangen. Die 3D Struktur der IRES wird vermutlich durch RNA-RNA und RNA-Protein Interaktionen bestimmt. RNA-binding Proteins binden an bestimmten Sequenzen der mRNA, dimerisieren und stabilisieren dadurch IRES-Strukturen. Experiment: Nachweis interner Initiation durch IRES • • mRNA mit IRES und Reportergen erzeugen und durch Ligase zirkularisieren. CAP abhängige Initiation ist damit nicht möglich. Da man trotzdem ein Genprodukt erhält, kann man davon ausgehen, dass interne Initiation mittels IRES funktionert. Noch eins: • Es wird ein Plasmid erzeugt, das in Folge ein Thymidin-Kinase-Reportergen, die 5' NCR mit IRES sowie anschließend ein CAT-Reportergen enthält. • Das Plasmid wird mittels Transfektion in Polioinfizierte Kulturzellen eingebracht. • Es kommt zu keiner Thymidin-Kinase-Synthese, da die CAP abhängige Translation durch Polio ausgeschalten ist. • CAT Synthese findet statt, die Translation sollte über IRES initiiert worden sein. 183 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Fig 3. Schematic representation of EMCV, FMDV and TMEV IRES domains H-L, showing binding sites for PTB (thick gray line) and ITAF45. Viral mechanisms of translational reprogramming Translational Control of Viral Gene Expression in Eukaryotes Michael Gale Jr.,1,*Seng-Lai Tan,2and Michael G. Katze2 Microbiology and Molecular Biology Reviews, June 2000, p. 239-280, Vol. 64, No. 2 Leaky scanning and frameshifting 184 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Control by leaky scanning, terminationand re-initiation events Functional recoding at the MuLV gag-pol junction Synthesis of the MuLVGag protein terminates at the gagstop codon (top). However, approximately 5% of the time, the elongating ribosome will read through the gag stop codon to produce the Gag-Pol polyprotein (bottom). During this process, the gagstop codon is redefined to encode glutamine and is shown by the presence of tRNA Gln at the redefined UAG codon. Stop codon redefinition is dependent on specific downstream sequences and a 3'-proximal pseudoknot structure. The elongating ribosome eventually melts out the pseudoknot to complete GagPolysynthesis. Low-frequency gag-pol stop codon redefinition is essential for MuLV replication. Figure adapted with modification from references 12 and 108. Negative regulatory elements in the 5'-UTR of mRNA that may repress translation 185 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Translational pathophysiology: a novel molecular mechanism of human disease Mario CazzolaandRadekC. Skoda Blood, Vol. 95 No. 11 (June 1), 2000: pp. 3280-3288 Regulation of translation initiation in eukaryotes Eisen ist für die katalytische Funktion vieler Enzyme (z.B. Aconitase im Citratzyklus) essentiell. Transferrin dient als Eisentransporter, Ferritin als Eisenspeicher. Bei Eisenmangel werden TransferrinRezeptoren (TR) zur Aufnahme von Eisen benötigt. Hat Aconitase kein Eisen gebunden, weil die Konzentration zu niedrig ist, kann es stabil an das 3' Ende der TR mRNA binden, die mRNA wird stabilisiert, TR können translatiert werden. Weiters bindet Aconitase bei Eisenmangel an das 5' Ende der Ferritin mRNA, wodurch dessen Translation verhindert wird. Bei Eisenüberschuss hat Aconitase Eisen gebunden und kann nicht an die jeweiligen mRNAs binden, Ferritin wird translatiert, TR nicht. 186 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Coordinate regulation of transferrinreceptor and ferritinsynthesis through translational controls operated by the iron-responsive elements (IREs) and by the iron regulatory proteins IRP1 and IRP2. Only one IRE is present in the 5'-UTR of ferritin mRNA. When cellular iron is scarce, IRP molecules are available for binding the 5' IRE; initiation of translation is prevented; and ferritin synthesis is inhibited. By contrast, presence of abundant intracellular iron prevents binding of IRPs to the 5' IRE and allows efficient mRNA translation to proceed (green arrow). Five IREs are present in the 3'-UTR of transferrin receptor (TfR) mRNA. When cellular iron is scarce, binding of one or more IRPs to the IREs in the 3'-UTR stabilizes TfR mRNA and increases TfR translation. Conversely, when iron is abundant, very few IREs are occupied by IRPs, and TfR mRNA is rapidly degraded. Eukaryotic translational elongation and termination: Minor differences between eukaryotes and prokaryotes Eukaryotes: only one release factor Molecular mimikry Ribosome recycling? Die Elongation in Eukaryoten läuft analog zu Prokaryoten ab, es exisitieren Homologe zu EFTu und EF-G. Die Termination in Eukaryoten benötigt nur einen Releasefaktor im gegensatz zu den drei Releasefaktoren in Prokaryoten. Einige bakterielle Toxine können eukaryotische Elongationsfaktoren modifizieren. z.B. das Diphterietoxin überträgt einen Ribosylrest auf die seltene AS Diphtamid, die im Elongationsfaktor eEF2 (Homolog zu EF-G) vorkommt. Dadurch wird eEF2 inaktiviert, die Translation wird gestoppt. EF-G im Bakterium selbst weist kein Diphtamid auf und ist daher nicht vom Toxin betroffen. In der eukaryotischen Translation sind noch viele Punkte ungeklärt, beispielsweise ist noch nicht bekannt, wie Ribosomen von der mRNA dissoziieren, wenn sie das Stopcodon erreicht haben. 187 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert mRNA stability in Eukaryotes Eukaryoten besitzen Endonucleasen sowie Exonucleasen, die mRNA nicht nur vom 3' Ende sondern auch vom 5' Ende her abbauen können. Wie schon erwähnt, ist die Stabilität von eukaryotischer mRNA durch CAP und PolyA im Vergleich mit prokaryotischer mRNA erhöht. mRNA liegt über CAP, Poly A, PABP und eIF4G gebunden zirkulärisiert vor. Je länger der Poly A Schwanz ist bzw.je öfter das 3' UTR motif vorkommt, desto stabiler wird die mRNA. Das 5' Ende ist daher üblicherweise für Exonuclease nicht zugänglich. Für den Abbau muss diese Zirkularisierung gelöst werden. Der PolyA Schwanz wird so weit verkürzt, dass keine Interaktion zwischen PABP und eIF4G mehr stattfinden kann. Anschließend wird der Methyl-7-Guanosyl-Rest des CAP durch ein decapping enyzme entfernt. Erst jetzt kann die mRNA vom 5' Ende oder auch vom 3' Ende her abgebaut werden. Drei Abbauwege von mRNA in Eukaryoten: • Deadenylierung des PolyA, Verkürzung des PolyA durch 3' → 5' Abbau • Deadenylierungsabhängiges Entfernen des CAP mit folgendem 5' → 3' Abbau. • Von Deadenylierung unabhängiges Entfernen des CAPs (PolyA intakt) und folgendem 5' → 3' Abbau. Premature translational termination triggers mRNA decapping Nature (1994) 370 T ½ … Halbwertszeit Frühzeitige Termination führt zum Abbau von mRNA. Wird z.B. in Hefe bei einer mRNA mit bekannter Halbwertszeit ein vorzeitiges Stopcodon eingefügt (2. Gel), sinkt die HWZ der mRNA. Wird diese Mutation in einen Hefestamm eingebracht, der keine funktionelle 5' → 3' Exonuclease besitzt, steigt die HWZ wieder an (3. Gel). Ein Abbau durch vorzeitige Stopcodons wird demnach vom 5' Ende her initiiert. Fehlerhafte Stopcodons lassen sich durch das im Vergleich mit DNA-Polymerase weniger effiziente Proofreading der RNA-Polymerase erklären. Es gibt zwei unterschiedliche Varianten wie der Abbau vorzeitiger Stopcodons eingeleitet werden kann. 188 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert mRNA turnover in yeast Translation initiation is strongly stimulated by interactions between Pab1p and the eIFG4 initiation factor. Transient disruption of these interactions may allow slow deadenylation of the mRNA. Pab1p also interacts with other translation initiation factors, possibly including Mrt1p/Pat1p/Spb10p. After deadenylation to A10 or less, the mRNA is degraded. In the major pathway, Mrt1p probably recruits the Lsm1p/Spb8p protein to the mRNA, presumably as a component of an Lsm complex. This interacts with Dcp1p and Dcp2p, promoting decapping and 5'→3' degradation by Xrn1p. In an alternative pathway, the mRNA is degraded 3'→5' by the exosome complex. This also requires cofactors - the Ski2p, Ski3p and Ski8p proteins - but their order of action and function in recruiting and/or stimulating the exosome complex are unknown. Nonsense-mediated Decay Bereits während dem Spleißen binden Proteinkomplexe (EJC) oberhalb der Splice sites an die mRNA, die anschließend aus dem Nucleus transportiert wird. Im Cytosol binden weitere Proteinfaktoren an die mRNA. Wird dabei ein Stopcodon entdeckt, wandert der Faktor hUpf1 downstream entlang der mRNA. Kommt er dabei an einen EJC, war das Stopcodon vorzeitig, hUpf1 wird phosphoryliert und beschleunigt decapping und Abbau der mRNA. 189 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Eine weitere Variante des Abbaus von mRNA mit vorzeitigen Stopcodons ist, dass der Abbau durch Interaktionen des Ribosoms ausgelöst wird. Kommt das Ribosom während der Translation an ein Stopcodon, prüft es entweder selbst, ob sich downstream cis-aktive Sequenzen befinden (downstream sequence element mit gebundenem Protein), die anzeigen, dass es sich bei dem Stopcodon um einen Fehler handeln muss und initiert in diesem Fall decapping und Abbau von beiden Enden her (bei Hefe). Bei Säugetieren wird der Faktor UPF1 vom Ribosom an einem Stopcodon rekrutiert, der wie oben bereits beschrieben downstream wandert und den Abbau der mRNA initiert, wenn er einen EJC erreicht. 190 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Regulatory RNAs/ siRNAs and miRNAs Nature Biotechnology 21, 1441 -1446 (2003) RNAi - RNA mediated interference In Eukaroyten induziert doppelsträngige dsRNA den sequenz-spezifischen Abbau der mRNA. PTGS: post transcriptionalgene silencing RNAi (RNA mediated interference) Doppelsträngige RNA hat im Cytosol nix zu suchen, es stammt vermutlich von Viren und wird daher durch einen speziellen Proteinkomplex (Dicer) in 21nt lange Stücke zerschnitten. But wait, there's more! Die Teile der dsRNA werden in die Einzelstränge aufgetrennt und von einem weiteren Proteinkomplex (RISC) gebunden. Zumindest eine Hälfte dieser gebundenen RNA ist komplementär zu der von ihr transkribierten mRNA. Der Komplex kann auf diese Art an die sich im Cytosol befindliche mRNA des Virus binden und diese zerschneiden. Man spricht in diesem Zusammenhang auch von PTGS (post transcripitional gene silencing). Unter Gen Knockdown versteht man den Vorgang, die Translation des Zielgens durch RNAi auszuschalten. Dazu bringt man DNA in den Organismus ein, dessen Transkript RNA einerseits einen Teil der mRNA des Zielgens enthält und andererseits so weit mit sich selbst bindet (Antisense RNA), dass dadurch eine scheinbare dsRNA entsteht, die durch RNAi erkannt, geschnitten und verwendet wird, um die mRNA des Zielgens zu zerschneiden. 191 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) siRNA Expression • • • For transient expression: duplex RNA can be delivered to the cell For a stable expression: a vector containing the DNA to produce a hairpin RNA The vector may be plasmid, retrovirus, adenovirus siRNA Delivery • • • • • For cell culture – Lipid-based transfection – Electroporation In vivo – Lipid-based – Conjugations Bacterial phage RNA Cholesterol Atelocollagen – Viral systems (ieretrovirus & adenovirus) siRNA Delivery & Processing Applications for siRNA • • Basic research – Determining protein function – Easier than a knockout and may be used for partial knockdowns Clinical research – You name it – Cancer, hypercholesterolemia, infections, developmental defects 192 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert RNAi= Big Money? Nature Biotechnology 21, 1441 -1446 (2003) What is the Difference between miRNA and siRNA? • • • • • • Function of both species is regulation of gene expression. Difference is in where they originate. siRNA originates with dsRNA. siRNA is most commonly a response to foreign RNA (usually viral) and is often 100% complementary to the target. miRNA originates with ssRNAthat forms a hairpin secondary structure. miRNA regulates post-transcriptional gene expression and is often not 100% complementary to the target. 193 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) miRNA processing Mammalian microRNAs: a small world for fine-tuning gene expression 194 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Another View Processing bodies are sites of storage and/or degradation of mRNA What are the functions of miRNA? • • • • Involved in the post-transcriptional regulation of gene expression. Important in development. Metabolic regulation (miR-375 & insulin secretion). Multiple genomic loci (different expression patterns?). 195 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Differences in miRNAMode of Action Human miRNA function? 196 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert miRNARegistry • • • • • http://www.sanger.ac.uk/Software/Rfam/mirna/index.shtml Latest release contains 1620 2909 predicted and verified miRNAs 227 321 predicted and 131 223 experimentally verified in Homo sapiens Mouse and human are highly conserved Human is not conserved with plants Out of 198 hmiRNAs known, 20% are linked to fragile sites and 50% are in cancer associated regions. 197 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Translation in Archaea Translation initiation factors in the 3 kingdoms Translational initiation pathways in Pro-and Eukaryotes mRNA (SD+) /30S interaction at high and low temperatures 198 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Binary complex formation with S. solfataricus 30S ribosomes is SD-dependent Leaderless mRNA does not bind to ribosomes at 65°C Translation of leaderless mRNAs in S. solfataricus? 199 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Purification and reconstitution of the trimeric aIF2 with purified subunits Stimulation of 35 S-Met-tRNAi binding to ribosomes by aIF2 In contrast to Eukaryotes, binding of tRNAi requires the α-γ subunits of S. solfataricus aIF2 Paola Londei, David Hasenöhrl 200 Sonnbert V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bläsi, Foliensatz 6 Intracellular protein transport Post-translationale Vorgänge: Viele synthetisierte Proteine müssen nach der Translation an ihren Bestimmungsort (Organellen, Nucleus- und Zellmembran, extrazellulärer Raum) transportiert werden. Zu diesem Zweck weisen weisen diese Proteine Peptidequenzen auf, die von Transportproteinen und Proteinen am Bestimmungsort erkannt werden. Es gibt zwei verschiedene Arten: Proteine die zunächst vollständig translatiert und anschließend gefaltet und transportiert werden, und Proteine, deren Translation gestoppt, mitsamt gebundenem Ribosom ans ER transportiert und in das ER hinein fertig translatiert werden. Passage through the membrane Die Phospholipid-Bilayer von Membranen sind im Inneren hydrophob und an der Außenseite hydrophil. Proteine benötigen demnach Poren, um Membranen durchqueren zu können. Die Proteine müssen zu diesem Zweck möglichst linear vorliegen, da sie im gefalteten Zustand nicht durch die Poren passen würden. Diesem Zweck dienen Protein-Chaperone, die sowohl in Pro- als auch Eukaryoten vorkommen. Protein Chaperones Hsp … heat shock protein, Hsp70, Hsp40, GrpE sind Protein Chaperone, Hsp60 und Hsp10 Chaperonine. Hsp70 bzw. Hsp40 kommen in Eukaryoten vor, Dank bzw. DnaJ sind die bakteriellen Homologe. Chaperone sorgen dafür, dass ein Protein eine bestimmte Konformation annimmt oder in einer Konformation bleibt. Zu diesem Zweck binden die Chaperone bereits während der Translation an die entstehende Polypeptidkette. Es ist nicht bekannt, wie Chaperone "ihre" Proteine erkennen. Dna J und anschließend DnaK binden das Polypeptid und Falten es unter ATP Hydrolyse. GrpE (nucleotide exchange factor) setzt DnaK und DnaJ wieder frei. 201 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert GroEL GroEL ist ein Chaperonin, das aus aus mehreren Untereinheiten besteht. Jede dieser UE benötigt für die Funktionalität ATP. Die Untereinheiten schließen sich gemeinsam um ein ungefaltetes Protein und falten es mittels ATP Hydrolyse. Durch Bindung des zusätzlichen Faktors GroES und weiterer ATP Hydrolyse zerfällt der Komplex und setzt das gefaltete Protein frei. GroEL ist für das Überleben einer Zelle essentiell, wird es ausgeknockt, stirbt die Zelle ab. GroEL/GroES Function 202 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Protein export in bacteria Proteins may be targeted to the • inner or outer membrane • periplasmic space • extracellular medium Proteins exported across a membrane have in general a signal sequence Ein zu sezernierendes Protein trägt eine so genannte Leader Sequenz, damit klar ist, wo das Protein hintransportiert werden muss. Nach der Passage durch die Membran wird diese Sequenz üblicherweise von einer Leader Peptidase abgespalten. Diese Sequenz ist in Pround Eukaryoten hoch konserviert, sie lassen sich beispielsweise zwischen Prokaryoten und Hefezellen tauschen. Die Leader Sequenz ist etwa 20-25 AS lang und weist basische AS am N-Terminus sowie eine hydrophobe Sequenz in der Mitte auf. Am C-Terminus befinden sich unterschiedliche Sequenzen für die Leaderpeptidase. Leader Sequenzen von Proteinen, die in Mitochondrien und Chloroplasten importiert werden: • meist hydrophil. • aus ungeladenen AS-Sequenzen, die von basischen AS Sequenzen unterbrochen sind. • keine sauren AS. General mechanism of protein transport • • • Leader Sequenz initiiert die Interaktion des Vorläuferproteins mit Organellenmembran. Proteintransport durch die Membran. Proteolytische Abspaltung der Leader Sequenz ins Innere der Organelle. 203 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bacteria: The sec pathway through the cytoplasmic membrane Sezernierung bei Bakterien: Als Protein Chaperon dient SecB. Als Transportkomplex des zu sezernierenden Proteins dienen SecY und Sec E. SecB erkennt ein Zielprotein an der Leadersequenz und transportiert es vom Ribosom an die Membran, wo es mit dem über SecY an der Innenseite der Membran befestigten SecA interagiert. SecA schiebt das Protein unter ATP Hydrolyse in den aus SecY und SecE gebildeten Kanal. Der weitere Transport wird durch die PMF (Proton motif force) vermittelt. Nachdem das Protein die Membran durchquert hat, wird die Leadersequenz durch eine mit dem Komplex gebundene Leaderpeptidase vom Zielprotein abgespalten. Wieso ist das Leaderpeptid am N-Terminus basisch/ positiv geladen: In Bakterienzellen findet sich zur ATP Erzeugung über die Membran ein elektrochemischer Gradient bzw. ein Membranpotential. Innen ist die Ladung negativ, außen positv. Der basische Bereich des Leaderpeptids erleichtert Interaktionen an der negativ geladenen Innenseite der Membran, um zum Transportapparat zu gelangen (so richtig???). Experiment um Gen für Proteintransport zu finden: 1) ein bestimmtes Protein auswählen … z.B. OmpA (sitzt in äußerer Membran). 2) OmpA auf Plasmid klonieren und OmpA Antikörper dazu. 3) Gel-PAGE, man kriegt zwei Banden, eins für das Protein und eins für das Prä-Protein. Components of the Sec pathway: SecB High selectivity with low specificity: how SecB has solved the paradox of chaperone binding TIBS (1995) 20:65 SecB liegt im Cytosol als Tetramer mit negativer Oberflächenladung vor, das mit Proteinen mit längeren positiv geladenen Bereichen interagieren kann. Als Beispiele weisen das Maltose- und das Galactosebindeprotein (kommen beide im Periplasmaraum vor) lange Sequenzen positiver Ladungen auf und können darüber mit SecB interagieren. 204 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert SecA Promotes Preprotein Translocation by Undergoing ATP-Driven cycles of Membrane Insertion and Deinsertion Cell, Vol 78, September 9, 1994, Copyright © 1994 by Cell Press SecA dient als Motorprotein für die Translokation des Zielproteins aus dem Cytoplasma durch die Membran. SecA interagiert mit SecYE, SecB liefert das Zielprotein an das SecA Protein. Ein Teil von SecA wird durch ATP Hydrolyse sehr flexibel und schiebt das Zielprotein so in den Kanal. Um die Interaktion von SecA + SecB nachzuweisen wird das Zweihybridsystem verwendet. (siehe S38) Experiment: Nachweis, dass Leadersequenz den Transport durch die Membran induziert. Mittels eines speziellen Verfahrens ("French press") werden Bakterien unter hohen Druck gesetzt, die Zellen platzen und es bilden sich so genannte "inside out" Vesikel. Teile der Zellwand können sich in verkehrter Ausrichtung zu Vesikeln zusammenschließen, wodurch auch SecYE mit SecA in verkehrter Ausrichtung in der Vesikelmembran sitzt. Man benötigt die vermutete Leadersequenz an einer nachfolgenden Proteinsequenz, zu der Antikörper existieren, die diese Sequenz binden können. Fügt man SecB, ATP und die modifizierten Proteine hinzu, sollte das Proteinkonstrukt in das Vesikel hineintransportiert werden, wenn die Leadersequenz wie vermutet funktioniert. Anschließend werden mittels Peptidasen alle Peptide außerhalb der Vesikel abgebaut, dann die Vesikel geöffnet und mittels Antikörpern überprüft, ob sich die modifizierten Proteine tatsächlich in den Vesikeln befanden. Escherichia coli translocase: the unravelling of a molecular machine Molecular Microbiology (2000) 37(2), 226-238 Der Sec pathway kommt sowohl bei gram+ als auch bei gram- Bakterien vor. 205 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Two major pathways • • • two major pathways for translocation in bacteria: Sex and SRP pathways both converge at SecYEG translocon and use SecA, a peripherally-bound ATPase that supplies the energy for translocation. SecB binds to nascent chains containing a signal sequence and maintains the preprotein in translocation-competent form, then binds SecA; SRP docks with membrane receptor, FtsY (simpler homologues of eukaryotic SRP and SRP receptor). Membrane targeting and translocation of bacterial hydrogenases Arch Microbiol (2000) 173:319-324 Twin Arg motif at the N-terminus of the signal sequence. TAT pathway: Twin Arginine Translocation. Crossing the cytoplasmic membrane without the sec translocon. Große, bereits gefaltete Proteine wie bakterielle Hydrogenasen können nicht über den SecPathway durch die Membran gebracht werden und müssen auf andere Weise exportiert werden. Für diesen weiteren Pathway ist ein Twin-Arg-Motif (immer zwei aufeinander folgende Arginine) in den zu transportierenden Proteinen typisch. Für den Transport durch die Membran ist ein großer Proteinkomplex aus den Proteinen TatA, E, B, ... nötig, durch den die Zielproteine transportiert werden, der genaue Mechanismus ist nicht geklärt. 206 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Model of Tat targeting and transport In the membrane TatA is depicted in red, TatB in blue, and TatC in yellow. (a) Upon emerging from the ribosome the preprotein must avoid targeting to other pathways such as Sec, which is aided by the characteristics of the signal peptide and mature protein and/or the binding of Tat-specific chaperones (red circles) (70, 129). (b) After folding, any cofactors and/or additional subunits are added prior to targeting to the TatBC receptor complex (c) (d). The proton motive force drives the formation of an active translocaseand the substrate is transported through a pore consisting mainly of TatA (1, 52, 85). (e) Upon removal of the signal peptide the mature protein is released on the periplasmic side of the membrane. Protein export pathways in Bacteria Die meisten Proteine können mittels Sec pathway durch die Membran transportiert werden. Gram- Bakterien besitzen neben der inneren noch eine äußere Membran, durch die Proteine ebenfalls hindurch transportiert werden müssen. Dazu sind vier verschiedene Sekretionssysteme bekannt. 207 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Type I secretion (Sec independent) E. coli: Hemolysine Typ I Sekretionssysteme sind Sec independent und aus drei Proteinkomplexen aufgebaut. • ABC Transporter als Pore in der IM • Membran fusion proteine • Channel forming proteins als Pore in der OM Typ I ist typisch für Hämolysine, Toxine, die Erythrozyten lysieren. Am C-Terminus der Hämolysine befindet sich eine AS Sequenz, die als Exportsignal dient. Durch dieses Exportsignal werden die Hämolysine mittels der Proteine HylB und HylD vom Cytoplasma nach Außen transportiert. Wird die Sequenz vom C-Terminus entfernt, wird das Toxin nicht mehr über die Membranen transportiert. Type II secretion (Sec dependent) Pullulanase in K. oxytoca Proteine des Typ II Sekretionsmechanismus müssen zunächst über den Sec Pathway über die innere Membran in den periplasmatischen Raum transportiert werden. Hier können die Proteine noch modifiziert werden (die endgültige Konformation von Pullanase z.B. wird durch Einführung von Disulphidbrücken mittels DsbA erzeugt). Aus dem Periplasma werden die Proteine über eigene Transportsysteme nach Außen transportiert. Experiment: Prinzipielle Funktion des Sec Pathways Das protein Pullulanase baut außerhalb der Zelle Stärke ab, damit die Monomere der Stärke in die Zelle aufgenommen werden können. Die Zellkultur wird auf reinem Stärkemedium gezogen und kann daher nur überleben, wenn Pullulanase nach außen transportiert wird. Wird die Leadersequenz der Pullulanase oder Proteine des Sec Apparates mutiert, kann ermittelt werden, welche Bestandteile für den Transport essentiell sind, die entsprechenden Kolonien werden auf Stärkemedium absterben. 208 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Type II secretion Das Choleratoxin kann nur freigesetzt werden, wenn das Bakterium vom Phagen CTXΦ befallen ist, da ein Transportkanal des Phagen in der äußeren Membran zur Freisetzung nötig ist. Die Komponenten A und B des Toxins werden über den Sec abhängigen Sekretionsweg in den periplasmatischen Raum transportiert. Hier werden sie mittels DsbA gefaltet und lagern sich zu AB5-Komplexen zusammen. AB5 wird vom Eps Sekretionsapparat erkannt und durch wiederholte Polymerisierung Depolymerisierungsreaktionen von EpsG durch die aus EpsD gebildete Pore in den extrazellulären Raum geschoben. Type III secretion (Sec independent) The Yersinia Yop virulon: a bacterial system for subverting eukaryotic cells Molecular Microbiology (1997) 23(5) 861-867 Typ III erzeugt einen direkten Kanal vom bakteriellen Cytoplasma über IM und OM ins Cytoplasma einer eukaryotischen Zelle. Im Fall von Yersinia Pestis werden so beispielsweise Proteine eingeschleust, die in der eukaryotischen Zelle Apoptose auslösen. An der Bildung des Kanals sind mehr als 20 Proteine beteiligt. Vom prinzipiellen Vorgang her bindet das bakterielle Protein YopN an einen Rezeptor einer eukaryotischen Zelle. Das dient als Auslöser dafür, dass die bakterielle Zelle an die eukaryotische Zelle herangezogen und der Kanal ausgebildet wird. 209 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Type IV secretion Dieses Sekretionssystem dient der Übertagung bakterieller DNA ins Cytoplasma von Eukaryoten und kann sowohl Sec abhängig als auch Sec unabhängig sein. • Chaperon VirE1 bindet VirE2 und verhindert dessen Bindung an T-DNA im eigenen Cytoplasma. • VirE2 sowie mittels VirD2 gebundene T-DNA werden über den P-Pilus zur Zielzelle transportiert. • VirE2 bildet zunächst einen Kanal durch die eukaryotische Zellmembran, weiteres VirE2 und VirD2 mit T-DNA werden in die Zielzelle transportiert. • Im Cytoplasma der Zielzelle liegt VirE2 frei vor und bindet an T-DNA, die auch VirD2 gebunden hat und kann so in den Nucleus wandern. Import of proteins into the ER Es gibt Proteine, die Zwecks Modifizierung oder Verwendungszweck in das ER transportiert werden müssen. Während der Synthese dieser Proteine wird eine Sequenz translatiert (15-30 AS mit hydrophobem Kern), die von einem Protein-RNA Komplex (7S RNA + 6 Proteine), dem SRP (signal recognition particle), erkannt und gebunden wird. Die Bindung verhindert eine weitere Translation des Proteins durch das Ribosom und vermittelt den Transport zum ER. Am ER wird SRP von einem in der ER Membran sitzenden passenden SRP-Rezeptor gebunden. Der Rezeptor wiederum ist mit einer Pore verbunden, in die das bisher translatierte Protein eingeführt wird. Die Translation wird wieder gestartet, die Energie der Translation ist ausreichend, um die Polypeptidkette durch die Pore in das Innere des ER zu schieben. Die Signalsequenz auf dem Peptid wird abgeschnitten, sobald sie im Inneren des ER ankommt. Zur Freisetzung von des SRP vom SRP Rezeptor ist Hydrolyse von GTP notwendig. 210 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Insertion of proteins into the ER Struktur von SRP In Bakterien wurde ein homologer Vorgang entdeckt, der Proteine an und durch die Zellmembran transportiert. Das Homolog zu SRP wird als Ffh Protein, das Homolog zum SRP Rezeptor als FtsY bezeichnet. FtsY ist dabei nicht mit einem eigenen Porenkomplex sondern mit dem Sec-Apparat assoziiert, um das Zielprotein durch die Membran zu schleusen. In Bakterien wird dieser Pathway vor allem von Membranproteinen verwendet. Mitochondrial import of proteins Alle mitochondriale Proteine werden im Nucleus transkribiert, im Cytosol translatiert und müssen anschließend ins Mitochondrium importiert werden. Mitochondrien besitzen eine doppelte Zellmembran zwischen der sich ein Intermembranraum befindet, es findet sich zur ATP Erzeugung ein elektrisches Potential über die innere Membran. Proteine können sowohl für den Intermembranraum als auch für das Lumen des Mitochondrions vorgesehen sein und benötigen dafür unterschiedliche Leadersequenzen. Proteine, die in die Matrix transportiert werden sollen und dort bleiben sollen haben nur ein Matrix targeting Signal. Proteine, deren Zielort der Intermembranraum ist haben zu der Matrixsequenz eine zusätzliche Signalsequenz. Beide Proteinarten werden zunächst in die Matrix transportiert und erst anschließend an ihren tatsächlichen Bestimmungsort weiter transportiert. Sobald ein Protein im Matrixraum angelangt ist, wird die Leadersequenz für den Matrixraum abgeschnitten. Findet sich danach eine weitere Leadersequenz, wird das Protein weitertransportiert. Um Proteine durch die beiden Membranen zu schleusen, dienen so genannte Tom (translocase of the outer mitochondrial membran) und Tim Proteine. 211 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Mitochondrial biogenesis: The Tom and Tim machine Current Biology 1997, 7:R100-R103 • • • Die cytosolischen Chaperone MSF oder cHsp70 binden an eine positiv geladene Leader Sequenz und transportieren das zugehörige Preprotein zu Rezeptoren in der äußeren Mitchondrienmembran. MSF und cHsp70 binden dabei unterschiedliche UE desselben Proteinkomplexes (Tom). Das Preprotein wird mittles der Tom Proteine durch die OM in den Intermembranraum gebracht, wo es an den Tim Proteinkomplex übergeben wird, der es über die IM in den Matrixraum bringt. Bei dem Transport über die Innere Membran hilft der positive Gradient im Intermembranraum dabei, die positive Leadersequenz des Preproteins in die Matrix zu bringen. mtHsp70 nimmt das Protein in der Matrix in Empfang und zieht es mittels ATP Hydrolyse vollständig in die Matrix. In der Matrix wird die Leadersequenz durch MPP abgeschnitten, das Protein durch mtHsp70 gefalten und anschließend freigesetzt. Import through the IM into the mitochondrial lumen requires the pmf and mtHsp70 212 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Common principles of protein translocation across membranes Science (1996) 271:1519 Import into and export from the nucleus Proteine können durch NPCs (nuclear pore complex) importiert und exportiert werden. Proteine wie z.B. Spleißproteine oder Transkriptionsfaktoren müssen importiert werden. An dieser Stelle kann die Transkription eines Gens reguliert werden, je nachdem, ob und wie effizient ein Faktor importiert wird. rRNAs, tRNAs und mRNAs müssen aus dem Kern ins Cytosol exportiert werden. 213 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Mit dem Nucleus assoziierte Proteine weisen entweder eine NES (nuclear export sequence) oder eine NLS (nuclear localisation sequence) auf, je nachdem ob sie exportiert oder importiert werden. Eine NLS ist eine Abfolge positiv geladener AS am NTerminus über die Importproteine (Importine) binden können. Es gibt einfache Diffusion, kleine Moleküle z.B. Ionen können durch die Kernmembran gelangen. Aktiver Transport von Proteinen > 50kDa wird durch NPCs erledigt. Im Cytoplasma findet sich mehr RanGDP, im Kern mehr RanGTP. α + β-Importin dienen als Importproteine. Sie binden an Cargoproteine und Nucleoporine, die wiederum den Import in den Kern selbst vornehmen. Das Trimer α-β-NLS ist in Anwesenheit von RanGDP und damit im Cytosol stabil, während es im Nucleus zerfällt. Exportin-NLS ist hingegen ist in Anwesenheit von RanGTP und damit im Nucleus stabil, während es im Cytosol zerfällt. 214 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Prüfungsfragen Bläsi: 1) wie kann man beweisen, dass die Bindungsstelle des ribosomalen Proteins S1 upstream der Shine Dalgarno Sequenz auf der prokaryotischen mRNA liegt? 2) Wie kann man Interaktion (2 Möglichkeiten) und Funktionsweise der eukaryotischen Translations-Initiationsfaktoren nachweisen? Decker: 1) Wie führt man eine Chromatin-Immunpräzipitation (ChIP) durch und welche Erkenntnisse kann man daraus gewinnen? 2) Skizzieren oder beschreiben sie, wie sich Phosphorylierung der CTD der eukaryotischen RNA Pol II im Laufe des Transkriptionszyklus auswirkt. Bläsi: 1) Wie können sie gentechnisch und biochemisch nachweisen, dass IF3 gegen alle Startcodons außer AUG (GUG) diskriminiert (Skizze) 2) Wie können sie nachweisen (2 Methoden), ob ein translationaler Repressor auf der ShineDalgarno-Sequenz sitzt? Decker: 1) Zwei Komponenten systeme in Bakterien: Response regulator nennen und Zielgene (Skizzen) 2) Wie viele RNA Polymerase gibt es in eukaryotischen Zellen? Welche Gene werden von deisen Polymerasen transkribiert? Wie sehen die typischen Promotoren aus? (skizzen) Bläsi: 1) Welche Parameter beeinflussen die Stabilität prokaryotischer mRNA? wie können sie nachweisen, dass mRNA vom 5' bzw vom 3' Ende her abgebaut wird? 2) Wie können Sie feststellen, ob ein aufgereinigtes Protein als RNA-"molecular chaperone" wirkt? Decker: 1) - Beschreiben sie kurz den Aufbau eines Nukleosoms (Skizze) - Beschreiben sie die Aktivität des chromatin remodelling complexes - Was hat die (De-)Acetylierung der Histone zur Folge 2) Skizzieren sie den kompletten Initiationskomplex bei Transkription durch RNA Polymerase II. Welche dieser Proteine assoziieren mit DNA-bindenden Faktoren. Bläsi: 1) Wie können sie nachweisen, dass der prokaryotische Translationsfaktor IF3 gegen nicht AUG Startcodons diskriminiert? (Toeprint, Plasmid) 2) Wie wird Selenocystein in die wachsende Proteinkette inseriert (Skizzen) Decker: 1) Kurze Beschreibung der Initation der Transkription (Phasen der Initiation) 2) Diffundiert die RNA Polymerase frei zum Promotor? Andere Erklärungsversuche für Promotorauffindung. 3) Wie beeinflusst der Sigma Faktor die Assoziation der RNA Polymerase mit dem Promotor? 215 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Bläsi: 1) Erklärung zweier Experimente die zur Aufklärung des genetischen Codes geführt haben. Wie lässt sich "Recoding" erklären? 2) Erklärung der Funktion von regulatorischen RNAs in prokaryotischer Genexpression. Zwei Nachweise (biochemischer und genetischer) für positive Regulation. Decker: 1) Welche Fragestellung beantworten sie mit ChIP, wie funktioniert das. 2) Aktivierung von Adenylatcyclatase, welche andere Genexpressionswege werden auch durch Adenylatxcyclatase aktiviert? Bläsi: 1) Zwei Ansätze um nachzuweisen, dass IF3 nicht gegen AUG diskriminiert. 2) Skizze wie Translation in Eukaryoten von CAP abhängig ist, wie können Viren dies zerstören? 3) Small regulatory RNAs; Beispiel; Biochemischer und genetischer Nachweis der positiven Kontrolle der Translation. Bläsi: 1) Die RNA III wirkt als translationaler Aktivator des hla-Gens in S.aureus. Wie würden sie das experimentell nachweisen. 2) Die zirkularisation der Hefe-mRNA bewirkt Stabilität (Skizze) 3) Gemeinsamkeiten / Unterschiede in mRNA Abbau in Pro- und Eukaryoten. Decker: 1) Präinitiationskomplex der Eukaryoten bei der Transkription (Skizze). welche Proteine interagieren mit proximalen und distalem Bereich. 2) Mit welchem Experimenten würden sie nachweisen: - TR rate eines Gens im Kontext genomischer DNA - Aktivität eines isolierenden Promotors - Bildung eines TF an DNA - genaue Position der TF Bindungsstelle - für die Bildung wichtiger Basen - TR Start eines Gens Prüfung vom 05.07.2002 Bläsi: 1) Autoregulation von mRNA Bsp: Transkription von rRNA / Translation von ribosomalen Proteinen (in Ecoli im Kontext mit Komponenten des Translationsapparates, Skizze) 2) Wirkungsweise von sRNA als translationalem Aktivator Bsp: Experiment vür Nachweis in vitro / Nachweis in vivo. Decker: 1) Isolieren eines Promotors aus Säugerzelle und Analyse desselben (notwendige Schritte in Stichworten) 2) Regulation des Schilddrüsenhormons TR: Struktur der DNA Bindedomäne, Struktur des TR, Dimerisiernug, Mechanismus der Genaktivierung und -repression. 216 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Prüfung vom 25.10.2002 Bläsi: 1) Welche Parameter beeinflussen die Stabilität von mRNA in Prokaryoten? Wie können sie nachweisen, ob ein Transkript in Bakterien vom 5' bzw. vom 3' Ende her abgebaut wird? 2) Wie würden sie nachweisen, ob ein Protein, das sie gereinigt habe als RNA Chaperon fungiert? Decker: 1) Welche spezifische fragestellung beantworten sie mit folgender Methodik: Nuclear run on assay, real time PCR, Southern Blot, Primer extension, DNase I footprint, Hefe 2 Hybrid System. 2) Welche Mechanismen zum Abschalten eines Gens auf Ebene der Transkription kennen Sie in Pro- und Eukaryoten Organismen? (in Stichworten beantworten und Beispiele nennen). Prüfung vom 03.10.2003 Bläsi: 1) a) Erklären sie die Funktionen von ncRNAs (non coding RNAs) bei Pro- und Eukaryoten und nennen sie Beispiele. b) Erklären sie wie sie nachweisen können, ob eine ncRNA als translationaler Repressor dient? 2) Wie können sie feststellen, ob ein Protein das sie gereinigt haben RNA Chaperon Aktivität hat. Decker: 1) Erklären sie die Begriffe positive und negative Regulation der Transkription und nennen sie je ein Beispiel. 2) Inwiefern unterscheiden sie die drei eukaryotischen RNA Polymerasen (Struktur, biochemische Funktion, Promotorstrukturen,...) Prüfung vom 19.03.2004 Bläsi: 1) Welche Parameter beeinflussen die Stabilität prokaryotischer mRNA? Wie können sie nachweisen, dass mRNA vom 5' bzw. vom 3' ende her abgebaut wird? 2) Wie können sie feststellen, ob ein gereinigtes Protein als RNA Chaperon wirkt. Decker: 1) Beschreiben sie kurz den Aufbau eines Nukleosoms (Skizze) - Beschreiben sie die Aktivität des chromatin remodelling complexes - was hat die (De-)Acetylierung der Histone zur Folge? 2) Skizzieren sie den kompletten Initiationskomplex bei Transkription durch RNA Polymerase II. Welche Proteine assoziieren mit DNA bindenden Faktoren? 217 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Prüfung vom 02.07.2004 Bläsi: 1) Wie kann man Interaktion zwischen SD und anti-SD nachweisen? a) genetischer Ansatz b) biochemischer Ansatz 2) Zwei Experiement skizzieren zum Nachweis, dass ein Protein als translationaler Repressor wirkt. Decker: 1) Y2H a) am Beispiel c-Fos-cJun skizzieren, wie schauen die Fusionsgene aus? b) kann man mit Y2H Interaktion von STATS nachweisen + Begründung. 2) a) Wie bzw. von welchem Co-Faktor wird CREB aktiviert und von welchem Pathway stammt das Signal? b) welcher Co-Aktiviator ist erforderlich und wie wirkt dieser? Prüfung vom 24.09.2004 Bläsi: 1) Wie können sie nachweisen, dass der prokaryotische Translationsfaktor IF3 gegen nicht AUG Startcodons diskriminiert? (Toeprint, Plasmid) 2) Wie wird Selenocystein in die wachsende Proteinkette inseriert (Skizzen) Decker: 1)a) was sind die speziellen Funktionen von TFIIH Komplexen? b) welche Proteinkomplexe vermitteln die Interaktion zwischen RNA Polymerase II und Transkriptionsfaktoren, die spezifische Bindungsstellen eines Promotors erkennen? 2) Welche enzymatischen Aktivitäten von zelloberflächenrezeptoren kennen sie (Bsp). Wie sind die Signaltransduktionswege beschaffen, die diese enzymatische Aktivität letztlich in veränderte Genexpression übertragen (jeweils ein Beispiel in Form einer Skizze). Prüfung vom 19.11.2004 Bläsi: 1) Beschreiben sie zwei experimentelle Ansätze, die zur Aufklärung des genetischen Codes geführt haben. Durch welche Mechanismen kann es während der Translation zu "Recoding" kommen d.h. zu einer nicht ko-linearen Translation des genetischen Codes. 2) Beschreiben sie die Funktion von regulatorischen RNAs in der Genexpression bei Prokaryoten. Wie können sie genetisch und biochemisch nachweisen, dass ein bestimmtes Gen durch eine solche RNA auf translationaler Ebene positiv reguliert wird. Decker: 1) a) Welche Phasen lassen sich bei der Initiation der Transkription unterscheiden (in Stichworten) b) lässt sich das Auffinden eines Promotors durch RNA Polymerase durch freie Diffusion beschreiben? Welche alternativen Erklärungen kommen in Frage? c) Wie beeinflusst der Sigma-Faktor die Assoziation der prokaryotischen RNA Polymerase mit Promotoren? 2) Erklären sie wie entfernt von der Initiationsstelle der Transkription bindende Transkriptionsfaktoren die Bindung der RNA Polymerase beeinflussen können. 218 V1.6.22 Genexpression (Decker, Bläsi) – Sommersemester 2006 (mit Update SS 2008) Sonnbert Prüfung vom 21.01.2005 Bläsi: 1) Zwei Ansätze um nachzuweisen, dass IF3 nicht gegen AUG diskriminiert. 2) Skizze wie Translation in Eukaryoten von CAP abhängig ist, wie können Viren dies zerstören? Decker: 1) Welche Fragestellung beantworten sie mit ChIP, wie funktioniert das. 2) Aktivierung von Adenylatcyclatase, welche andere Genexpressionswege werden auch durch Adenylatxcyclatase aktiviert? Prüfung vom 13.05.2005 Bläsi: 1) Wie können sie biochemisch und genetisch nachweisen, dass der IF3 bei der Translationsinitiation gegen alle Startcodons außer AUG diskriminiert? 2) Wie können sie die Interaktion und die Funktionsweise von Proteinen des Initiationsapparates nachweisen. Decker: 1) DNA Microarray in stichworten, Ablauf, Was sind die Vorteile gegenüber anderen Methoden. 2) Drei verschiedene Mechanismen der positiven Transkriptionskontrolle durch Transkriptionsfaktoren. Prüfung vom 23.09.2005 Bläsi: 1) Small regulatory RNAs? Beispiele; genetischer und biochemischer nachweis der positiven Kontrolle der Translation. 2) Man soll die Abbauwege von mRNAs in Prokaryoten vergleichend gegenüberstellen; Nachweis, von welchem Ende her abgebaut wird. Decker: 1) Warum ist die Phosphorylierung der RNA Pol II wichtig. 2) ? Prüfung vom 20.01.2006 Bläsi: 1) Wie können sie mit Hilfe einer molekularbiologischen Methode nachweisen, wo der prokaryotische Initiationsfaktor IF1 an die kleine ribosomale Untereinheit bindet (Skizze) 2) Skizzieren sie die drei Ereignisse im bereich der Ribosomenbindunsstelle einer prokaryotischen mRNA, welche die Bindung von Ribosomen verhindern können (Skizze). Wie können sie diese "Mechanismen" mit Hilfe von molekularbiologischen Untersuchungen nachweisen? Decker: 1) Skizzieren Sie die Ereignisse die von der Bindung eines Wachstumsfaktors z.B. EGF an seinen Rezeptor zur Induktion von Genexpression führen. Benennen sie Komponenten und relevante Modifikationen die an der Signaltransduktion beteiligt sind. 2) Worin unterscheiden sich Nucleosomen und Enhancosomen? Welche funktionelle Bedeutung hat die Bildung eines Enhanceosoms? 219