Can we predict the direction of cell motion ?
Werbung
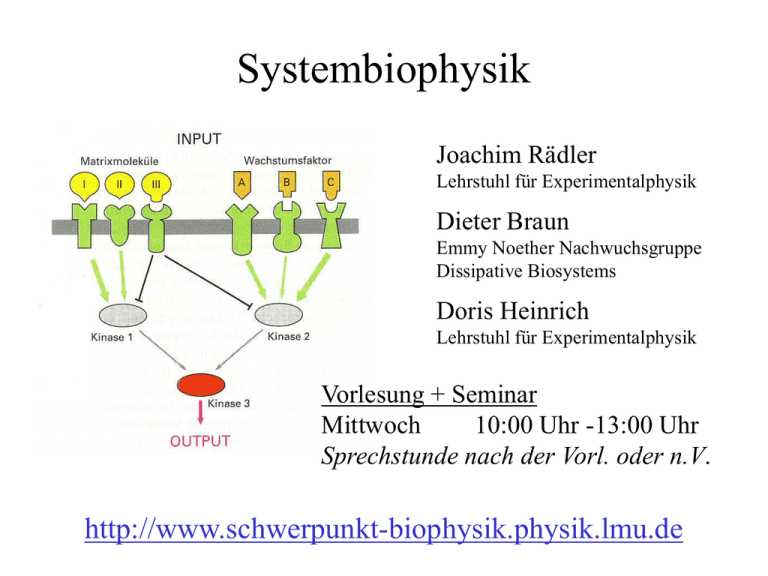
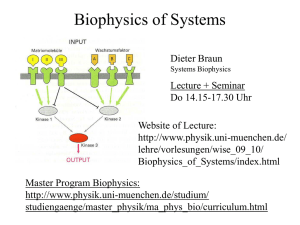
Systembiophysik Joachim Rädler Lehrstuhl für Experimentalphysik Dieter Braun Emmy Noether Nachwuchsgruppe Dissipative Biosystems Doris Heinrich Lehrstuhl für Experimentalphysik Vorlesung + Seminar Mittwoch 10:00 Uhr -13:00 Uhr Sprechstunde nach der Vorl. oder n.V. http://www.schwerpunkt-biophysik.physik.lmu.de Inhalte : Biophysik der Systeme 1. 2. 3. 4. 5. 6. 7. Einleitung Evolution + Spieltheorie Nichtlineare Systemdynamik Raumzeitliche Strukturbildung Biologische Netzwerke Genetische Netzwerke Immunsystem http://www.schwerpunkt-biophysik.physik.lmu.de Makrophage jagt E.coli Bakterium A physical view of the (eukaryotic) cell • • • • www.people.virginia.edu/~rjh9u/cell1.html Macromolecules – 5 Billion Proteins • 5,000 to 10,000 different species – 1 meter of DNA with Several Billion bases – 60 Million tRNAs – 700,000 mRNAs Organelles – 4 Million Ribosomes – 30,000 Proteasomes – Dozens of Mitochondria Chemical Pathways – Vast numbers – Tightly coupled How is a useful approach possible? Biosystems: Rückkopplungen Biosystems: Rückkopplungen Promotoren, Protein-Wechselwirkung Inhibitoren Regulation RNA Interferenz Kompartimente Epigenetics Reaktionsnetzwerke Verstärkung Organellen Cell-Cell Communication Rauschen Diffusion Zum Begriff „Bio-System“ Input Output Eigenschaften * Komponenten (Spezien) * Netzwerkartige Verknüpfungen (kinetische Raten) * Substrukturen (Knoten,Module, Motive) * Funktionelle Input => Output Relation Ziel * Erforschung der „Bauprinzipen“ (reverse engineering) Vorsicht : Bauprinzip nicht „rational“ sondern Ergebnis eines Evolutionprozesses • Erstellung quantitativer Modelle zur Beschreibung des Systems • Überprüfen der Modelle mit experimentellen Daten • Vorhersagen von Systemverhalten Systems Biology Definition • Systems Biology integrates experimental and modeling approaches to study the structure and dynamical properties of biological systems • It aims at quantitative experimental results and building predictive models and simulations of these systems. • Current primary focus is the cell and its subsystems , but the „systems perspective“ will be extended to tissues, organs, organisms, populations, ecosystems,.. Der biochemische Signalpfad in dictyostelium discoideum cAMP + b g Ga b g PIP3 PIP2 PI3K* PTEN RAS Cell polarization pleckstrin homology domain PH Rac/Cdc42 CRAC AcetylcholinAktivierung Actin polymerization Abstrakte Darstellung der Signalübertragung Biochemische Ratengleichung + Definition von Reaktionsräumen + Diffusionsprozesse Reakt.-Diff- Gl. + stochastische Beschreibung Signal-Netzwerke sind „komplex“ Connection Maps: Signal Transduction Knowledge Environment www.stke.org Zum Umgang mit Komplexität Ein klassisches Hilfsmittel für die Analyse Komplexer Systeme System Analyse Quantitative Daten Analyse, Mathematischel Modellierung Simulation Nützliche Analogie zw. Signaltransduktion und elektronischen Schaltkeisen Biologische Signalnetzwerke sind kombinatorisch bzgl. Ein- und Ausgabe Modular view of the chemoattractant-induced signaling pathway in Dictyostelium Peter N. Devreotes et al. Annu. Rev. Cell Dev. Biol. 2004. 20:22 Hierarchische Struktur biologischer Organismen (Z. Oltvai, A.-L. Barabasi, Science 10/25/02) Modular Biology as advocated in the influential paper (Nature 402, Dec 1999) Stochastic Genes From Concentrations to Probabilities Stochastic Genes From Concentrations to Probabilities Inventory of an E-coli: do counting molecules matter? Number of mRNA/cell Number of rRNA/cell Number of tRNA/cell Number of all RNA/cell Number of polysaccharides/cell Number of lipopolysaccharide/cell Number of lipids/cell Number of outer membrane proteins Number of porins (subset of OM) Number of lipoproteins (OM) Number of nuclear proteins Number of ribosomal proteins Number of all proteins in cell Number of external proteins (flag/pili) Number of all proteins 4000 18,000 200,000 222,000 39,000 600,000 25,000,000 300,000 60,000 240,000 100,000 900,000 2,600,000 1,000,000 3,600,000 Note the low number of mRNA ! Wiederholung: Gen-Expression Erbgut ist fixiert, wie kann eine bakterielle Zelle sich an ihre Umgebung anpassen? Antwort: Regulation der Gen-Expression Repressors & Inducers active repressor inactive repressor RNAP inducer no transcription transcription RNAP promoter operator gene promoter operator gene • Inducers that inactivate repressors: – IPTG (Isopropylthio-ß-galactoside) Lac repressor – aTc (Anhydrotetracycline) Tet repressor • Use as a logical Implies gate: Repressor Inducer Output (NOT R) OR I Repressor 0 0 1 1 Inducer 0 1 0 1 Output 1 1 0 1 Der Effekt kleiner Zahlen e.g. Herabsetzen von Transkriptionsrate oder Zellvolumen Proteinlevel konstant Fluktuationen erhöht Stochastic Gen-Expression Extrinsic Noise intrinsisches Rauschen Unterscheide zwischen intrinsischem (biochem. Prozess der Gen-Expression) und extrinsischem (Fluktuationen anderer Zellkomponenten, z.B. Konzentration von RNA Polymerase) Rauschen Idee für Experiment: Gen für CFP (grün fluoreszierendes Protein) und YFP (rot fluoreszierendes Protein) werden durch den gleichen Promotor kontrolliert, d.h. mittlere Konzentration von CFP und YFP in einer Zelle gleich => Wahrscheinlichkeit für Expression sollte sich in einer Zelle nur durch intrinsisches Rauschen unterscheiden Intrinsic Noise A: kein intrinsisches Rauschen => Rauschen korreliert rot+grün=gelb B: intrinsische Rauschen => Rauschen unkorreliert, verschiedene Farben Elowitz, M. et al, Science 2002 Stochastic Gen-Expression Unrepressed LacI Extrinsic Noise Repressed LacI Intrinsic Noise +Induced by IPTG Extrinsic Noise Elowitz, M. et al, Science 2002 Science, 307:1965 (2005)