E-Cell
Werbung

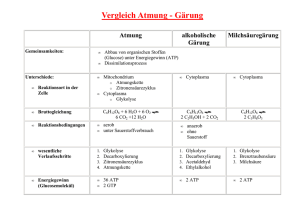
Tomitas E-Cell Software-Umgebung zur Simulation ganzer Zellen Sonja Lorenz Sommerakademie St. Johann 2002 1 Einführung: in vivo - in vitro - in silico 2 Mycoplasma genitalium: Konstruktion einer hypothetischen Minimalzelle 3 E-Cell-Simulationssystem 4 5 3.1 Ontologie 3.2 Mathematische Grundlagen 3.3 Software-Architektur 3.4 Benutzeroberfläche Virtuelle Experimente 4.1 Glykolyse 4.2 Human Erythrocyt Ausblick In vivo 1 1 In vivo In vitro 1 In vivo In vitro In silico 1 Metabolische Pfade (Ausschnitt) 1 Metabolische Pfade (Ausschnitt) 1 Metabolische Pfade (Ausschnitt) 1 Simulation isolierter zellulärer Prozesse •Qualitative Modelle Genregulation und Expression Zellteilungszyklus Mechanismen der Signaltransduktion Circadiane Rhythmik bei Drosophila Bakterielle Chemotaxis •Quantitative Simulation biochemischer Stoffwechselpfade 1 Integrative Modelle ganzer Zellen •Erythrocyten-Modelle (Palsson et al., 1989; Lee et al., 1992; Ni et al., 1996) •DBSolve (Goryanin et al. 1997) •V-Cell (Schaff et al., 1999) •Alliance For Cellular Signaling (AFCS) •Microbial Cell Project (MCP) •Smartcell (Serrano) •E-Cell (Tomita et al.,1997) 1 E.coli Erythrocyt Circadiane Rhythmik E-Reis Zellzyklus E-Cell Projekt E-Neuron Myocard Modell Mycoplasma genitalium Chloroplast Mitochondrium Diabetes mellitus 1 Mycoplasma genitalium: ein genomischer Minimalist •Grampositives, parasitäres Bakterium •Vorkommen im Genital-und Respirationstrakt von Primaten •Totalsequenzierung: Fraser et al., 1995 ! 580 kb Genom ! 2 ca. 470 Gene Das self-surviving Genom Tomita, 2001 2 Überblick: Metabolismus der E-Cell Tomita, 2001 2 Ontologie des E-Cell-Systems Takahashi et al., 1998 3.1 Scomplex: Subkategorie eines Komplexes binding substance binding reaction Scomplex vacant site Takahashi et al., 1998 3.1 Ontologie des E-Cell-Systems Takahashi et al., 1998 3.1 Substanz-Reaktor-Modell Transformation 2-Substrat-2-Produkt-Reaktion Assoziation Dissoziation Takahashi et al., 1998 3.1 Ontologie des E-Cell-Systems Takahashi et al., 1998 3.1 Strukturiertes Substanz-Reaktor-Modell A A B Reaktor in Supersystem Reaktor in Subsystem A A B B A Transmembranärer Transport Reaktor in externem System Takahashi et al., 1998 3.1 Ontologie des E-Cell-Systems Takahashi et al., 1998 3.1 Mathematische Grundlagen ... i Si ... ... j S j ... S1 , S2 ,..., Sn dS1 f1 ( S1 , S 2 ,..., S n ) dt dS 2 f 2 ( S1 , S 2 ,..., S n ) dt dS n f n ( S1 , S 2 ,..., S n ) dt k [ Si ] i i V f [S ] [ A] [ I ] ) (1 K A KI KS [S ] b[ A] a[ I ] (1 ) (1 K A KI (1 [ A] [I ] ) K A KI b[ A] a[ I ] ) K A KI Takahashi et al., 1998 3.2 Objekt-orientiertes MVC-Modell change model control change get data update view Tomita et al., 1997 3.3 Elementare Struktur des E-Cell-Systems experiment controller environment membrane e-cell chromosome cytoplasm interpreter rule file Takahashi et al., 1998 3.3 Informationsfluß in der E-Cell Takahashi et al., 1998 3.3 Simulationsumgebung der E-Cell Takahashi et al., 1998 3.3 Benutzeroberfläche Tomita et al., 1997 3.4 Glykolyse: Übersicht 2 ATP 4.1 Glykolyse: Simulation ATP ? Glc-Entzug Zeit 4.1 4. Glykolyse: im Detail 4.1 Glykolyse: im Detail 4.1 Glykolyse: im Detail 2 Pyruvat 2 C3-Körper 4.1 Glykolyse: im Detail 2 Pyruvat 4 ATP 4 ADP 2 C3-Körper 4.1 Glykolyse: im Detail 2 energieliefernde Schritte Enolbzw. Pyruvat 4.1 Glykolyse: im Detail 2 Pyruvat 4 ATP 4 ADP 2 C3-Körper 4.1 Human Erythrocyt Glykolyse Pentosephosphatweg Membrantransport Nucleotidmetabolismus Tomita, 2001 4.2 Ausblick ! 5 Mangel an quantitativen Daten ! Ausblick ! Mangel an quantitativen Daten ! • Automatisierung der Informationssammlung 5 Ausblick ! Mangel an quantitativen Daten ! • Automatisierung der Informationssammlung • Modellierung der vollständigen Mycoplasma genitalium Zelle 5 Ausblick ! Mangel an quantitativen Daten ! • Automatisierung der Informationssammlung • Modellierung der vollständigen Mycoplasma genitalium Zelle • Identifikation neuer Enzym- und Transportergene 5 Ausblick ! Mangel an quantitativen Daten ! • Automatisierung der Informationssammlung • Modellierung der vollständigen Mycoplasma genitalium Zelle • Identifikation neuer Enzym- und Transportergene • Herstellung lebensfähiger Zellen mit signifikant reduziertem Genom 5 Ausblick ! Mangel an quantitativen Daten ! • Automatisierung der Informationssammlung • Modellierung der vollständigen Mycoplasma genitalium Zelle • Identifikation neuer Enzym- und Transportergene • Herstellung lebensfähiger Zellen mit signifikant reduziertem Genom • Simulation pathologischer Prozesse • Individuelle Behandlungsmethoden (customized medicine) 5 Ausblick ! Mangel an quantitativen Daten ! • Automatisierung der Informationssammlung • Modellierung der vollständigen Mycoplasma genitalium Zelle • Identifikation neuer Enzym- und Transportergene • Herstellung lebensfähiger Zellen mit signifikant reduziertem Genom • Simulation pathologischer Prozesse • Individuelle Behandlungsmethoden (customized medicine) => Ganzheitliches Verständnis der Dynamik des zellulären Metabolismus 5 Numerische Integration Takahashi et al., 2000 3.2 Modellierung von Chromosomen und Genexpression Takahashi et al., 1998 3.1 Human Erythrocyt