Biologie für Mediziner, Zahnmediziner und Lehramtsstudierende
Werbung

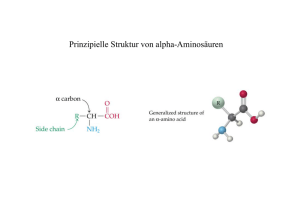
Biologie für Mediziner, Zahnmediziner und Lehramtsstudierende Dr. Kristian M. Müller Institut für Biologie III Termine: Di 8.00-9.00 Mi 8.45-9.45 Do 8.45-9.45 Dozenten: Müller, Baumeister, Baumeister, Fischer-Iglesias, Haller, Laux, Rak, Reth, Schempp, Scherer, Schulze, Wolff Proteine Hydrophobe Aminosäuren Ca Polare und geladene Aminoäsuren Gruppierung der Aminosäuren nach Eigenschaften Saure und basische Eigenschaften einer Aminosäure Nachträgliche Veränderungen der Aminosäuren (Posttranslationale Modifikationen) Proteinogene Aminosäuren Übersicht Bildung und Benennung eines Peptids Amid-Bindung Syntheserichtung des Proteins im Ribosom Übung zu Aminosäuren 1) Nehmen Sie Stift und Papier und schreiben Sie Ihren Namen KRISTIAN 2) Bestimmen Sie den Dreibuchstaben-Code Lys Arg Ile Ser Thr Ile Ala Asn 3) Malen Sie die Struktur Trivial name Symbols Alanine Ala A Arginine Arg R Asparagine Asn N Aspartic acid Asp D Cysteine Cys C Glutamine Gln Q Glutamic acid Glu E Glycine Gly G Histidine His H Isoleucine Ile I Leucine Leu L Lysine Lys K Methionine Met M Phenylalanine Phe F Proline Pro P Serine Ser S Threonine Thr T Tryptophan Trp W Tyrosine Tyr Y Valine Val V Unspecified Xaa X Selenocysteine Sec U Asp /Asn B = Asx Glu /Gln Z = Glx Übung zu Aminosäuren II Finden Sie den Namen in einem Protein (für Fortgeschrittene) LOCUS ZP_00405122 218 aa linear BCT 02-JUN-2005 DEFINITION COG2384: Predicted SAM-dependent methyltransferase [Mycoplasma genitalium G-37]. ACCESSION ZP_00405122 VERSION ZP_00405122.1 GI:66880137 DBSOURCE REFSEQ: accession NZ_AAGX01000005.1 SOURCE Mycoplasma genitalium G37 ORGANISM Mycoplasma genitalium G37 Bacteria; Firmicutes; Mollicutes; Mycoplasmataceae; Mycoplasma. REFERENCE 1 (residues 1 to 218) AUTHORS Rothberg,J.M. and Margulies,M. TITLE Genome Sequencing in Open Microfabricated High Density Picoliter Reactors CDS 1..218 /locus_tag="MgenG_01000472" /coded_by="complement(NZ_AAGX01000005.1:21933..22589)" /db_xref="COG:COG2384" 1 61 121 181 mkkristian knnnnihffv elrsflslns mkhyqcllrv lvqsfnpklv sdgfnnlpel wdivnetlvq yqpkqkpslm ydigcdhsyl ninpkigvia drefiypilv dlkiietlnk tsyliktnqn ltivnsdisk nallsnyqkf glgglkiini isqkenfinr fviqpqsnli ieklkkpfkl tkelvilgpk linfkdkhcl iitsyess Die Peptidbindung Erlaubte Konformationen in der Peptidkette so nicht -> Ramachandran-Diagramm Helices in Proteinen sind rechtsgängig Primär- Sekundär- Tertiär- Quartär-Struktur des Insulin Struktur der b-Lactamase Bändermodell Sekundärstruktur: α-Helices Sekundärstruktur: β-Faltblätter β-Helices werden aus β-Faltblättern gebildet (z.B. Pectat Lyase 2pec) Strukturgebende Elemente Lactamase mit Hydrathülle Strichmodell Lactamase Kalottenmodell Struktur ohne H2O mit K+ und PO43- Die Oberfläche eines Proteins (z.B. Lactamase) Abstand zum Liganden Die Oberfläche eines Proteins II Elektrostatik Hydrophobizität Protein Funktion, Dynamik, Interaktion Epidermal Growth Factor Receptor (EGF-R) EGF Ligand TGF-a Amphiregulin b-cellulin HB-EGF Epiregulin Tyrosinkinase Rezeptor EGF-R EGFR-TK K K PI3-K pY pY GRB2 pY SOS RAS STAT3 PTEN RAF AKT MEK Gene transcription Cell cycle progression Proliferation/ maturation DNA PP Myc JunFos MAPK Cyclin D1 Myc Cyclin D1 Survival (anti-apoptosis) Chemotherapy / Metastasis radiotherapy Angiogenesis resistance Rezeptor mit Ligand 2(EGF-R:EGF) 1IVO, Ogiso et al. Methoden der Proteinbiochemie Protein-Löslichkeit in Abhängigkeit der Ionenstärke Dialyse zum Umpuffern Auftrennung von Proteingemischen: Gelelektrophorese Chromatographie Größenausschlusschromatographie / Gelfiltration Ende