Struktur der DNA - cosbucienii-profesionisti-cluj
Werbung

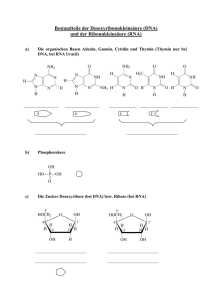
Struktur von DNA Vortrag im Rahmen des biochemischen Seminars für Lehramtskandidaten Referent: Franz Alt Bausteine der Nucleinsäuren Gerüst 5 HO OH O 4 1 3 2 OH OH 5 HO OH O 4 1 3 2 O HO P OH OH OH - β-D-Ribofuranose (RNA) bzw. - β-D-Desoxyribofuranose (DNA) sowie Phosphatgruppen Zucker: Bausteine der Nucleinsäuren Gerüst O O - P O Veresterung jeweils an 3‘ und 5‘ OH O O - OH O O HO HO HO HO O O O O O O P O - O O P O O - O O P O O - O P O O - O Die Phosphodiesterbindung an 3‘ und 5‘ konstituiert das lineare Gerüst der Nucleinsäuren Informationsträger – die Basen O NH2 N HN N N O O N N H H Pyrimidin N N H Cytosin NH2 N N O NH N N H Purin Thymin N N N CH3 Adenin N H Guanin N NH2 Nucleoside NH2 N HO N O N N OH Verknüpfung zum Nucleosid, hier Adenosin (Adenin + Desoxyribose) Jeweils N-9 (Purinderivate) bzw. N-1 (Pyrimidinderivate) mit C-1‘ des Zuckers über N-glykosidische Bindung verknüpft. Die Base liegt oberhalb der Ebene des Zuckermoleküls (auf der Seite der CH2OH-Gruppe), d.h. handelt es sich um eine β-glykosidische Bindung. Wird zusätzlich der C-5‘-Alkohol mit Phosphorsäure verestert, spricht man von Nucleotiden. Verknüpfung von Nucleotideinheiten zur Nucleinsäure O H3C O O 5' P O NH - N O O O Hohe Flexibilität der Nucleinsäure: z.B. freie Drehbarkeit um C-5‘, umklappen der Sesselkonformationen, Ausrichtung der Basen… OH O O N P O N - N O N H O NH2 NH2 3' O O P N O N - N O O O N Sekundärstruktur • Wichtige Hinweise zur Klärung der Struktur der DNA: – 40er Jahre, Erwin Chargaff: Die Zahl der Adeninreste entspricht annähernd der Zahl der Thyminreste, die Zahl der Guaninreste der der Cytosinreste – 50er Jahre, Rosalind Franklin und Maurice Wilkins: Röntgenstrukturanalyse der DNA lässt auf einen helikalen Aufbau mit zwei Wiederholungen entlang der Längsachse schließen (prim. 0,34nm, sek. 3,4nm) • 1953 James Watson / Francis Crick: Einseitige Publikation zur molekularen Struktur der DNA: – Basenpaarung: Zwei gegenläufige Stränge sind durch H-Brücken zwischen Purin- und Pyrimidinbasen verknüpft (A mit T, G mit C). Sie sind komplementär. – Die DNA bildet eine Doppelhelix aus, die Basen befinden sich im Inneren der Helix. – Das Phosphat-Ribose-Gerüst befindet sich außen, was Kationen einen guten Zugang zu den negativen Ladungen der Phosphatbrücken bietet. – Die Basenpaarung weist auf einen möglichen Kopiermechanismus hin. 1962: Nobelpreis für Medizin Basenpaarung • Zusätzlich zu den H-Brücken resultieren zwei Stapelungseffekte aus der Anordnung der Basen: – Die hydrophoben Basengruppen entziehen sich im Inneren der Helix dem Wasser. – Van-Der-Waals-Kräfte zwischen den gestapelten Basenpaaren tragen ebenfalls zur Stabilität bei. DNA kann aufgrund ihrer Flexibilität auch weitere Strukturen bilden, so z.B. die kürzere A-DNA oder die linksgängige Z-DNA. Darüber hinaus können bei entsprechenden Basenabfolgen Haarnadeloder kreuzförmige Strukturen entstehen. Bereiche solcher Strukturen sind wahrscheinlich am DNAStoffwechsel oder der Teilung beteiligt. Haarnadelstruktur und kreuzförmige Struktur von DNA-Abschnitten. aus Palindrom-DNA: umgekehrte Sequenzwiederholung (wie RENTNER, ANNA) mit doppelter Symmetrie. Strukturvarianten der DNA-Doppelhelix (je 36bp). Die B-Form entspricht der WatsonCrick-Strucktur und ist unter physiologischen Bedingungen die stabilste. Tertiärstrukturen • Gesamtlänge der DNA eines menschl. Genoms: ca. 1m, d.h. 2m pro Zelle. • Die DNA muss also einer enormen Packung unterliegen. Superspiralisierung • Die Verdrillung einer verdrillten Struktur nennt man Superspiralisierung. Anschauliches Beispiel ist das Spiralkabel eines Telefons. • Durch Ändern der Verwindungszahl (also der Verdrillung der helikalen Struktur) entsteht eine Spannung im Molekül, welche durch Superspiralisierung oder auch Kreuzstrukturen (an Palindromen) kompensiert wird. Links: Elektronenmikroskopische Aufnahme ringförmiger mitochondrialer DNA Nucleosomen • Nucleosomen sind regelmäßige Strukturen von an die DNA gebundenen Histonkomplexen (Histone: 5 Typen kleiner basischer Proteine) Hintergrund: DNA-Strang an Histonkomplexe (Kugeln) gebunden. Elektronenmikroskopische Aufnahme Histone sind in eukaryotischen Zellen notwendig, um die Superspiralisierung zu erreichen, da keine entsprechenden Enzyme vorhanden sind. Durch die Umwicklung der Histonkerne entstehen negative Spiralen um den Kern und positive Spiralen im freien Teil der DNA. Topoisomerasen können die positiven Spiralen entfernen. Rechts: Modell der Verdichtung durch das Prinzip der Spiralisierung von Spiralen Unten: partiell entfaltetes menschliches Chromosom; DNA-Schleifen sind an einer Gerüststruktur verankert. Literatur • Crick, F. H. C.; Watson, J. D.: Molecular structure of nucleic acids. A structure for desoxyribose nucleic acid. Erschienen in: Nature, 25. April 1953, S.737. • Cox, M. M.; Nelson, D. L.: Lehninger Biochemie. 4., vollständig überarbeitete und erweiterte Auflage. Übersetzung der. 5. amerikanischen Ausflage. Berlin, Heidelberg 2009, Kap. 8 und 24. • Berg, J.M.; Tymoczko, J.L.; Stryer, L.: Stryer Biochemie. 6. Auflage. Heidelberg, 2007, Kap. 4 und 28. • Bildmaterial entnommen aus: Lehninger Biochemie.