Protein-NMR
Werbung

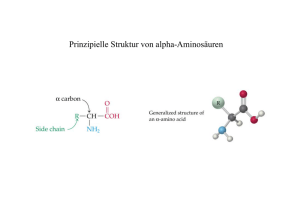
Protein-NMR Vertiefungsfach Analytische Chemie(WS2015/16) Dr.PeterBellstedt – NMRPlattformIAAC&IOMC [email protected] Themenübersicht Termin1am30.11.15:BiochemievonProteinen (Aufbau,Struktur,gentechn.Herstellung) Termin2am07.12.15:NMR-ExperimenteI (Labeling,1D/2D/3D-Exp.fürZuordnungundräuml.Erfassung) Termin3am14.12.15:NMR-ExperimenteII (CSundNOE-basierteStrukturberechnung,prakt.Übung) Termin4am04.01.16:SpezielleNMR-Strategien (>4DExperimente,spez.Labeling-Strategien,RDCs) ProteinesindBiopolymere Esgibt20proteinogeneAminosäuren Säugetiere(inkl.uns) müssen9Aminosäuren mitderNahrung aufnehmen, dasiediesenicht selbstherstellenkönnen. DiemeistenPflanzenund Mikroorganismen sindunshierüberlegen. ProteinebestehenausAminosäuren PeptidbindungenbildendasGrundgerüstvonProteinen Peptide(kurzkettig) undProteine (langkettig) bestehen ausunverzweigten Kettenvon Aminosäuren, diedurch Peptidbindungen miteinander verknüpft sind. [-N-Ca-CO-]bildetdaskonstanteRückgratvonProteinen AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer DieEigenschafteinerAminosäurewirddurchihreSeitenkette(hierunpolar)bestimmt DieEigenschafteinerAminosäurewirddurchihreSeitenkette(hierpolar)bestimmt DieEigenschafteinerAminosäurewirddurchihreSeitenkette(hiergeladen)bestimmt AminosäurensindbeiphysiologischenpHoftZwitterionen Oben: AusMüller-Esterl,Biochemie,2004Elsevier GmbH Unten:AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer EntsprichtderpH-Wertdemisoelektrischen Punkt(pI)istdieNettoladung =0. JedeAminosäure besitzteinen charakteristischenpI – Wert. FürjedeAminosäuregibteseinen1und3-Buchstabencode AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer Co- undposttranslationale ModifizierungvomAminosäuren Enantiomere Formenvonα-Aminosäuren L- und D-Formsindnichtdeckungsgleich. DasCα-AtombildetdasAsymmetriezentrum. ProteinebesteheninderRegelausLAminosäuren. D-Aminosäuren kommen inbakteriellenZellwändenund alsBestandteileinigerAntibiotika und Pilzgiftevor. DieMesomeriederPeptidbindung AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer DurchdenpartiellenDoppelbindungscharakterliegenCO-NHineinerEbene AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer DiePeptidbindung istnichtfreidrehbar undführt (inderRegel)zueinertrans-Stellung derübrigen Substituenten. DadurchwirddieKonformation derPolypeptidkette starkeingeschränkt.DiehiergezeigtenWertebeziehensichaufeine vollständig gestrecktePolypeptidkette. Standardwinkelincis- undtrans-Konformation DefinitionderDihedralwinkel 𝜙 (Phi):N-C𝑎 𝜓 (Psi):C𝑎-CO DasRamachandran-DiagrammzeigtdiemöglichenWinkelkombinationen 𝜙 (Phi):N-C𝑎 𝜓 (Psi):C𝑎-CO AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer Sekundärstrukturen:Parallelesundantiparallelesβ-Faltblatt AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer Wasserstoffbrückenstabilisierendasantiparalleleβ-Faltblatt (a) Aufsicht:CO&NHGruppen sindsoausgerichtet,dasssie aufeinander zeigen (b) Seitenansicht:DieSeitenketten zeigenabwechselnd nach oben undunten (c) „DasFaltblatt“ DasRamachandran-DiagrammzeigtdiemöglichenWinkelkombinationen 𝜙 (Phi):N-C𝑎 𝜓 (Psi):C𝑎-CO AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer Sekundärstrukturen:Rechtgängigeα-Helix DieTertiärstrukturbezeichnetdiedreidimensionaleStruktureinesProteins SchematischeDarstellungvon Sekundärstrukturelementen (Gelb:α-Helix, Rosa:β-Faltblatt(inkl.Richtungvo NzuCTerminus), blau:Übrigeinkl.Kehrenund Schleifen AusLöffler,Petrides,Biochemieund Pathobiochmie,2007Springer TertiärstrukturvonUbiquitin (76AS) Cartoon-Darstellung undOberflächenstruktur vonUbiquitin Quelle:Wikipedia BeispielevonProteinstrukturenausderPDB Quelle:Wikipedia 3D-ModeleinesDNA-Reparaturproteins(NMRbasiert!) ZusammenfassungderStrukturebenen • • • • Primärstruktur:AbfolgederAminosäuren(„Sequenz“) Sekundärstruktur:KleinsteräumlicheStruktureinheit(„lokaleStrukturelemente“) Tertiärstruktur:SummeallerSekundärelemente,3-DimensionaleStruktur Quartärstruktur:KombinationmehrererProteine DNA:DerSpeicherunsererErbinformation „Watson-CrickBasenpaare“: Adenin – Thymin, Guanin – Cytosin DurchBasenpaarungentstehteinerechtsdrehendeDNA-Doppelhelix DieVerpackungsebenendereukaryotischenDNA VonderDNAzumProtein Transkription(DNA->RNA) Durch„Abschreiben“derDNAdurchdasEnzym RNA-PolymeraseentstehtRNA.Anstellevon Thymin wirdinderRNAallerdings Uracil verwendet. EinGenkanngleichzeitigvonmehrerenRNA-Polymerasentranskripiert werden EinGenkanngleichzeitigvonmehrerenRNA-Polymerasentranskripiert werden DerWegvonderDNAzumProteininEukaryontenistetwaskomplexer • RäumlicheTrennungzwischenTranskriptionund Translation(Zellkernvs.Cytoplasma) • CodierendeSequenzderDNAistdurchnichtcodierendeBereiche(„Introns)unterbrochen (Prozessierung notwendig) • FertigeProteinemüssenoftnochinandere KompartimentederZelletransportiertwerden (Sortierung) DerFaltungstrichter MechanistischeModellederProteinfaltung HierarchischesModell (a)vs.Hydrophober Kollaps(b) DieräumlicheStrukturbestimmtdieFunktiondesProteins DasmenschlicheProteom.Auf sonstigeFunktionen entfallenz.B.Motorproteine oder Imunglobuline. DieräumlicheStrukturbestimmtdieFunktiondesProteins DieräumlicheStrukturbestimmtdieFunktiondesProteins • ProteinediechemischeReaktionen katalysierennenntmanEnzyme(„-asen“) BindungenvonLigandenkönnenKonformationsänderungenbewirken DurchKonformationsänderungenkönnenSignaleübertragenwerden DurchwiederholteKonformationsänderungenkönnensichMotorproteinebewegen DurchHydrolysevonAdenosintriphosphat(ATP,der universellenEnergiewährungderZelle)erfährtdas Myosin-ProteineinestrukturelleÄnderung,was wiederumzueinergerichtetenBewegungführt. Ionenkanäle,diePorengrößebestimmtdieSelektivität FehlfaltungenvonProteinenkönnenzuKrankheitenführen • Alzheimer,Parkinson,Prionen-Krankheit(z.B.BSE),Polyglutaminkrankheiten (z.B. ChoreaHuntington),u.v.m. Während das„normale“monomere Prionenprotein vorwiegend ausαHelicesaufgebaut ist,besitztseine pathogene Formeinenhohen Anteil anβ-Faltblättern undbildet schnell neurotoxische Multimere. ZusammenfassungI • Proteinesindunverzweigte BiopolymereausAminosäuren • DieAbfolgevonDNA-Basen(Triplets)bestimmtendiePrimärstruktur(Sequenz) einesProteins • DurchspezifischeWechselwirkungderSeitenkettenuntereinander (Wasserstoffbrücken,IonischeWW,hydrophobeEffekte)bildensichlokale Strukturelementeundschließlicheinemeistkomplexe3-dimensionaleStruktur • DieräumlicheStrukturbestimmtdieFunktioneinesProteins • DurchBindungvonLiganden(oderanderenProteinen)kannsichdieräumliche StruktureinesProteinsändern(Konformationsänderung) • WennmanaufmolekularerEbene(Reaktionsmechanismus)verstehenwill,wie einbestimmtesProteinseineFunktionausübt,mussmanseineräumliche Struktur(en)kennen! • ProteinesindinteressanteForschungsobjekte!(Aberwiebekommtmansie?) WahldesExpressionssystems DieHerstellungeinesrekombinantenExpressionsvektors EinExpressionsvektoristeinPlasmid(circuläre DNA), miteinerKlonierunsgstelle zumEinbringen fremder DNA,einem Promotor zurRegulationderExpression („Herstellung“) undeinem Selektionsmarker (hier Resistenzgegenüber Ampicilin). DurchRestiktion undLigation können unterschiedliche DNA-Fragmente miteinander verknüpft werden.Restiktionsenzyme erkennen spezifischeDNA-Seq. Plasmidkarten liefernallenotwendigenInformationenfürdieKlonierung Plasmidkarten liefernallenotwendigenInformationenfürdieKlonierung Ein„Tag“erleichtertdiespätereIsolation&AufreinigungdesProteins ZellfreieExpressionssysteme Quelle:Promega • • Vorteil:hochreineKomponenten,definiertesSystem Nachteil:OftgeringeAusbeuten,teuer Plasmidkarten liefernallenotwendigenInformationenfürdieKlonierung Daslac-Operon inE.coli:GrundlagefürIPTG-induzierteÜberexpression TranskriptionsaktivierungvonT7-basiertenExpressionssystemenmitIPTG Quellen:ocw.mit.edu/courses/biological-engineering/ http://www.laney.edu/wp/doug_bruce/ • NachZugabevonIPTGindasZellmedium startetdie(Überproduktion) desgewünschtenProteins ProteinreinigunginKurzform(Zellaufschluss,Ni-AffinitätschromatografiebeiHis6-Tag) ProteinreinigunginKurzform(Größenausschlusschromatografie/SEC) Quelle:http://www.gelifesciences.com/ ProblemdernatürlichenHäufigkeit Kern SpinquantenzahlI NatürlicheHäufigkeitin% 1H 1/2 99,99 13C 1/2 1,10 15N 1/2 0,37 17O 5/2 0,04 WielöstmandasProblem der„natural abundance“inderNMR-ProteinStrukturaufklärung? à Auflösung gibt esnächsteStunde. 1H[ppm]