Die Wirksamkeit von Krebsmedikamenten mathematisch vorhersagen
Werbung

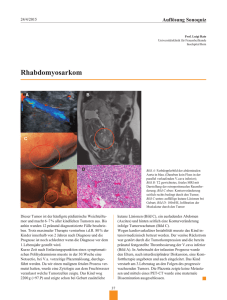
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/die-wirksamkeit-vonkrebsmedikamenten-mathematisch-vorhersagen/ Die Wirksamkeit von Krebsmedikamenten mathematisch vorhersagen Neue Krebsmedikamente werden oft nach dem Prinzip „Versuch und Irrtum“ ausprobiert. Der Grund dafür ist, dass die molekularen Prozesse der Medikamentenwirkung im Tumorgewebe bisher oft nur im Ansatz verstanden werden. Am Stuttgart Research Center Systems Biology (SRCSB) der Universität Stuttgart wird seit dem Jahr 2013 in einem Forschungsverbund daran gearbeitet, die Wirkungsweise solcher Medikamente vorherzusagen. Dabei kombinieren die Wissenschaftler molekularbiologische Experimente mit aufwendigen mathematischen Modellen, um so ein ganzheitliches Verständnis von Erkrankung und Therapie zu erlangen. So soll in Zukunft die Entwicklung von Krebsmedikamenten effizient begleitet werden: Neue, vielversprechende Wirkstoffe aus gentechnisch hergestellten Proteinen könnten dann schneller und kostensparender auf den Markt gebracht werden. Erste Wirkstoffkandidaten wurden bereits entwickelt. PREDICT steht für ein ehrgeiziges Projekt mit dem Namen „Holistic multi-scale modeling of targeted protein therapeutics action: Towards predicting effective treatment of cancer“, an dem seit Anfang 2013 acht Arbeitsgruppen aus vier Instituten der Universitäten Stuttgart und Tübingen, der Robert Bosch Gesellschaft für medizinische Forschung, des Stuttgarter RobertBosch-Krankenhauses sowie der Unternehmen Bayer Technology Services (BTS) und Celonic GmbH arbeiten. Koordiniert wird das Verbundforschungsprojekt, das vom BMBF mit insgesamt 3,5 Millionen Euro gefördert wird, am Stuttgart Research Center Systems Biology (SRCSB). Die Wissenschaftler haben es sich dabei zum Ziel gesetzt, ein ganzheitliches mathematisches Modell zu entwickeln, mit dem sie die Wirkung von Krebsmedikamenten vorhersagen und damit den Entwicklungsprozess in Labor und Klinik beschleunigen können. Parallel dazu arbeiten Molekularbiologen an der Entwicklung solcher Medikamente: Proteintherapeutika als Wirkstoffkandidaten gegen Darmtumoren. Diese mathematischen Modelle werden mit Hilfe einer Multiskalen-Modellierung erstellt „einer Spezialität in Stuttgart“, wie Prof. Dr. Dr. h.c. Matthias Reuss, Teilprojektleiter am SRCSB, erklärt: „Das heißt, einer Modellierung auf mehreren Ebenen. Man beginnt auf der untersten Skala der Moleküle und ihrer Interaktionen, weiter über die Kette von Reaktionen der subzellulären Ebene, dann über die nächsten Ebenen der Zell-Zell-Interaktionen und schließlich von Gewebe und Tumor, der Organ-Ebene, bis hin zum Ganzkörpermodell. Damit lässt sich die Dynamik der Medikamentenverteilung im Körper modellmäßig abbilden.“ 1 Prof. Dr. Klaus Pfizenmaier ist Koordinator des Projekts PREDICT. Er möchte mit seinen Mitarbeitern bis Ende 2015 einen validierten Prototypen für einen Wirkstoff gegen Darmkrebs entwickeln © SRCSB Die Methoden dazu wurden am SRCSB in Kooperation mit Bayer Technology Services selbst entwickelt. Voraussetzung für die mathematischen Modelle sind zunächst unter anderem experimentelle Untersuchungen der Pharmakokinetik - der Verteilung und Aufnahme der Medikamente - und Pharmakodynamik - der Wirkung im Körper. Diese Ergebnisse werden mit biochemischen und histologischen Untersuchungen sowie mit bildgebenden Verfahren am Kleintiermodell gewonnen und dann als Modell ausgearbeitet. „Jeder Tumor ist einzigartig“, so Reuss. „Man kann sie aber gruppieren, und diese Gruppen werden dann in unserem Modell unterschiedlich behandelt.“ Mathematische Modelle unterstützen die Wirkstoffentwicklung 2 Prof. Dr. Dr. h.c. Matthias Reuss ist für die mathematischen Modelle des Projekts PREDICT verantwortlich. © SRCSB Die Entstehung und das Wachstum solcher Tumoren sind äußerst komplexe und individuelle Prozesse: ein dynamisches und vielschichtiges System, das sich nicht so einfach linear beschreiben lässt, sondern eine komplexe mathematische Strukturierung erfordert. Wollte man alle Parameter ganz ohne Vorhersagen rein empirisch untersuchen, wäre dies ein enormer Aufwand, der aus praktischen Gründen nicht zu bewältigen ist und auch wenig Aussicht auf Erfolg hat. Deshalb werden die Lücken zwischen den Experimenten im Labor mit Hilfe von mathematischen Modellen überbrückt. „Wir entwickeln mathematische Modelle der biologischen Prozesse bei der Tumorentwicklung, die uns dann Vorhersagen für die Laborexperimente liefern und damit die Wirkstoffentwicklung wesentlich beschleunigen“, sagt Prof. Dr. Klaus Pfizenmaier, Koordinator von PREDICT und Leiter des Instituts für Zellbiologie und Immunbiologie der Universität Stuttgart. Personalisierte Krebstherapie mit Proteinwirkstoffen Die Modelle, die in Stuttgart entwickelt werden, zielen auf eine neue Klasse von Krebsmedikamenten ab: Proteintherapeutika, die zielspezifisch am Tumor wirken. Solche gentechnisch hergestellten Wirkstoffe sind Fusionsproteine, die die Tumorzellen in den Zelltod schicken. Sie bestehen aus einer von Antikörpern abstammenden Komponente, die den Tumor spezifisch erkennt, und einer Zytokinkomponente, die für den programmierten Zelltod verantwortlich ist. Die tumorselektive Komponente des Fusionsproteins kann auf die speziellen Merkmale einer bestimmten Tumorart zugeschnitten sein. Die zugehörigen mathematischen Modelle sind auf diese neuen Tumortherapeutika ausgerichtet und sollen den Verlauf einer Therapie abschätzen und vorhersagen, bei welchen Patienten das Medikament erfolgreich sein kann. Bei einer solchen personalisierten Krebstherapie wird der Arzt dann aufgrund der patientenspezifischen diagnostischen Parameter mit Hilfe der Modelle entscheiden, welcher Wirkstoff die größte Chance auf Heilung bringt. Im Verlauf der Therapie können zudem aktuelle Daten, zum Beispiel Ergebnisse aus bildgebenden Untersuchungen, dazu benutzt werden, das individuelle Modell noch weiter anzupassen. Vergleichbar sei dies mit der Vorgehensweise bei Wettervorhersagen, so Reuss: „In der Meteorologie werden die Vorhersagen auch ständig mit aktuellen Wetterdaten abgeglichen und entsprechend ans Modell adaptiert. Wir sind momentan dabei, eine solche Verfahrensweise auf die Krebsforschung anzuwenden – aber die Herausforderungen an uns sind groß.“ Grund dafür sind die vielen unterschiedlichen Skalen der Modelle. Schon allein die beiden Skalen „Zeit“ und „Raum“ sorgen für ein enormes Spektrum: „Im biochemischen Bereich liegen diese im Sekundenbereich, bei den klinischen Betrachtungen, ob ein Tumor auf die Behandlung anspricht, dagegen bei Tagen und Wochen“, erklärt Reuss. „Molekulare Wechselwirkungen müssen wir auf einer Mikrometerskala betrachten, den Ganzkörper im Meterbereich. Da muss man sich schon überlegen, wie man so etwas überbrückt.“ Zudem kommt dann noch die Erweiterung auf den dreidimensionalen Raum: „Das ist mathematisch sehr anspruchsvoll“, meint Reuss. Das Spektrum an Skalen hat eine enorme Datenmenge zur Folge, die verarbeitet und gesichtet werden muss. „Glücklicherweise sind wir hier in Stuttgart durch das Hochleistungsrechenzentrum in der Lage, dies zu bewältigen: Seine ca. 30.000 3 Prozessoren ermöglichen es den Wissenschaftlern des SRCSB, die aufwendigen Simulationen parallel durchzuführen." Mit Hilfe der Multiskalen-Modellierung wird die Dynamik der Verteilung von Krebsmedikamenten im Körper simuliert, beginnend auf der Skala der Moleküle und ihrer Wechselwirkungen über die zelluläre Ebene hin zu Geweben und schließlich der Organebene bis hin zum Ganzkörpermodell. © SRCSB Erster Medikamenten-Prototyp zeigt Wirkung Konkrete Ergebnisse gibt es nach nur anderthalb Jahren Forschungsarbeit auch schon: Beispielsweise haben die Biowissenschaftler den Prototypen eines gentechnisch hergestellten Fusionsproteins entwickelt, der in Gewebemodellen und Tierversuchen bereits hervorragende Wirkung zeigte. Außerdem wurde ein Modell auf Einzelzellebene erstellt, das das programmierte Absterben der Tumorzellen zuverlässig beschreibt. Allerdings besteht ein Tumor in der Regel aus Milliarden Zellen, die zu einem bestimmten Zeitpunkt sehr verschieden sind. Die nächste Herausforderung an die Mathematiker wird es deshalb sein, für eine solche Zellpopulation Modelle zu entwickeln. „Plan ist es, bis Ende 2015 mit einem ersten validierten Prototypen für einen Wirkstoff gegen Darmkrebs aufzuwarten“, erklärt Pfizenmaier. „Und wir hoffen, dass wir für dieses Medikament auch einen Partner finden, der bereit ist, den Prototypen weiterzuentwickeln - also die nächsten Schritte von Produktion, präklinischer und klinischer Prüfung zu begleiten. Das ist mit enormen Kosten verbunden. Alles hängt aber ganz davon ab, wie sich die Datenlage im nächsten Jahr gestaltet. Es sieht aber bisher ganz gut aus.“ 4 Fachbeitrag 21.07.2014 pbe BioRegio STERN © BIOPRO Baden-Württemberg GmbH Weitere Informationen Prof. Dr. Klaus Pfizenmaier Stuttgart Research Center Systems Biology (SRCSB) und Institut für Zellbiologie und Immunologie der Universität Stuttgart Allmandring 31 70569 Stuttgart Tel.: 0711 685-66986 E-Mail: klaus.pfizenmaier(at)izi.uni-stuttgart.de Stuttgart Research Center Systems Biology (SRCSB) Der Fachbeitrag ist Teil folgender Dossiers Systembiologie: das Komplexe begreifbar machen 5