Auf der Suche nach den Achillesfersen pathogener
Werbung
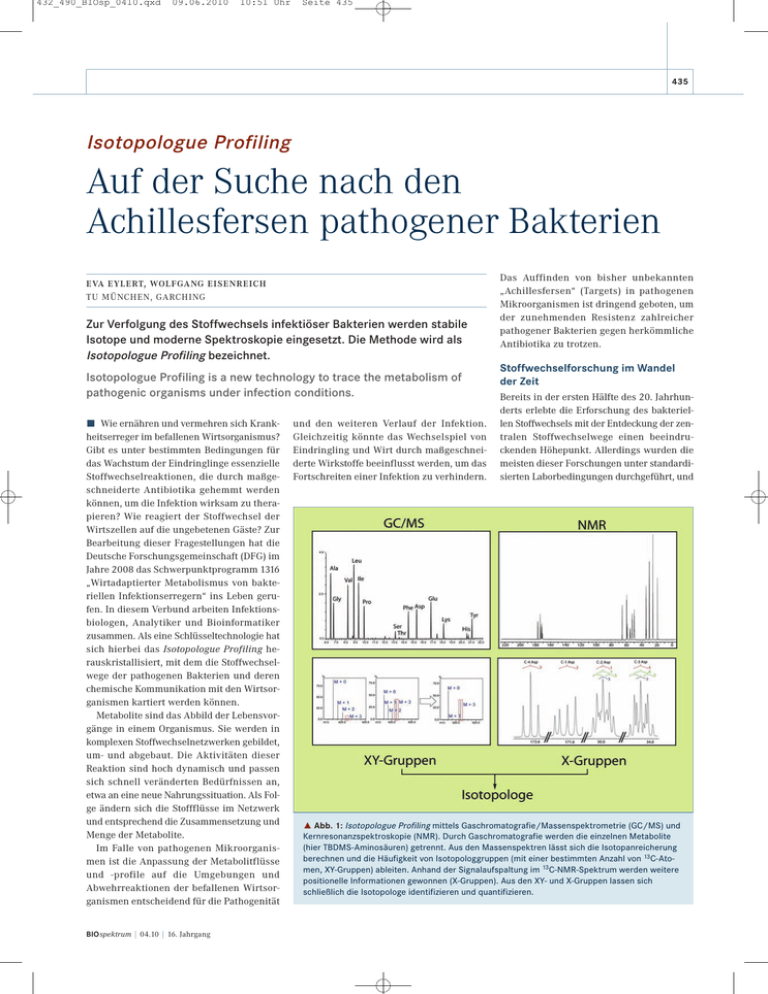
432_490_BIOsp_0410.qxd 09.06.2010 10:51 Uhr Seite 435 435 Isotopologue Profiling Auf der Suche nach den Achillesfersen pathogener Bakterien EVA EYLERT, WOLFGANG EISENREICH TU MÜNCHEN, GARCHING Zur Verfolgung des Stoffwechsels infektiöser Bakterien werden stabile Isotope und moderne Spektroskopie eingesetzt. Die Methode wird als Isotopologue Profiling bezeichnet. Isotopologue Profiling is a new technology to trace the metabolism of pathogenic organisms under infection conditions. ó Wie ernähren und vermehren sich Krankheitserreger im befallenen Wirtsorganismus? Gibt es unter bestimmten Bedingungen für das Wachstum der Eindringlinge essenzielle Stoffwechselreaktionen, die durch maßgeschneiderte Antibiotika gehemmt werden können, um die Infektion wirksam zu therapieren? Wie reagiert der Stoffwechsel der Wirtszellen auf die ungebetenen Gäste? Zur Bearbeitung dieser Fragestellungen hat die Deutsche Forschungsgemeinschaft (DFG) im Jahre 2008 das Schwerpunktprogramm 1316 „Wirtadaptierter Metabolismus von bakteriellen Infektionserregern“ ins Leben gerufen. In diesem Verbund arbeiten Infektionsbiologen, Analytiker und Bioinformatiker zusammen. Als eine Schlüsseltechnologie hat sich hierbei das Isotopologue Profiling herauskristallisiert, mit dem die Stoffwechselwege der pathogenen Bakterien und deren chemische Kommunikation mit den Wirtsorganismen kartiert werden können. Metabolite sind das Abbild der Lebensvorgänge in einem Organismus. Sie werden in komplexen Stoffwechselnetzwerken gebildet, um- und abgebaut. Die Aktivitäten dieser Reaktion sind hoch dynamisch und passen sich schnell veränderten Bedürfnissen an, etwa an eine neue Nahrungssituation. Als Folge ändern sich die Stoffflüsse im Netzwerk und entsprechend die Zusammensetzung und Menge der Metabolite. Im Falle von pathogenen Mikroorganismen ist die Anpassung der Metabolitflüsse und -profile auf die Umgebungen und Abwehrreaktionen der befallenen Wirtsorganismen entscheidend für die Pathogenität BIOspektrum | 04.10 | 16. Jahrgang und den weiteren Verlauf der Infektion. Gleichzeitig könnte das Wechselspiel von Eindringling und Wirt durch maßgeschneiderte Wirkstoffe beeinflusst werden, um das Fortschreiten einer Infektion zu verhindern. Das Auffinden von bisher unbekannten „Achillesfersen“ (Targets) in pathogenen Mikroorganismen ist dringend geboten, um der zunehmenden Resistenz zahlreicher pathogener Bakterien gegen herkömmliche Antibiotika zu trotzen. Stoffwechselforschung im Wandel der Zeit Bereits in der ersten Hälfte des 20. Jahrhunderts erlebte die Erforschung des bakteriellen Stoffwechsels mit der Entdeckung der zentralen Stoffwechselwege einen beeindruckenden Höhepunkt. Allerdings wurden die meisten dieser Forschungen unter standardisierten Laborbedingungen durchgeführt, und ˚ Abb. 1: Isotopologue Profiling mittels Gaschromatografie/Massenspektrometrie (GC/MS) und Kernresonanzspektroskopie (NMR). Durch Gaschromatografie werden die einzelnen Metabolite (hier TBDMS-Aminosäuren) getrennt. Aus den Massenspektren lässt sich die Isotopanreicherung berechnen und die Häufigkeit von Isotopologgruppen (mit einer bestimmten Anzahl von 13C-Atomen, XY-Gruppen) ableiten. Anhand der Signalaufspaltung im 13C-NMR-Spektrum werden weitere positionelle Informationen gewonnen (X-Gruppen). Aus den XY- und X-Gruppen lassen sich schließlich die Isotopologe identifizieren und quantifizieren. 432_490_BIOsp_0410.qxd 436 09.06.2010 10:51 Uhr Seite 436 MET H ODE N & AN WE N DU NGEN ¯ Abb. 2: Isotopologue Profiling am Beispiel des humanpathogenen Bakteriums Listeria monocytogenes. Die linke Seite zeigt die unterschiedlichen Markierungsgrade von Aminosäuren unter Infektions- (intrazellulär) bzw. Kulturbedingungen (in vitro) ausgehend von 13C-Glukose. Anhand dieser Anreicherungen und der positionellen Verteilungen von 13C wurde eine vereinfachte Stoffwechselkarte des Bakteriums (grau hinterlegt) unter Infektionsbedingungen abgeleitet (d. h. wenn sich das Bakterium innerhalb einer Wirtszelle ernährt und vermehrt). Die relevanten Stoffströme sind rot markiert. die Bedeutung der einzelnen Wege unter natürlichen Lebensbedingungen, auch im Wechselspiel mit anderen Organismen, ist keineswegs klar. Ein erneuter Aufschwung der Stoffwechselforschung wird in jüngster Zeit angetrieben durch hochsensitive Analysemethoden zur Messung von Metaboliten in komplexen Mischungen, wie Zellextrakten und biologischen Fluiden (Metabolomics). Ein weiterer Meilenstein auf dem Gebiet der modernen Stoffwechselforschung ist die Messung von metabolen Flüssen am lebenden Objekt (MFA oder Fluxomics). Vor Kurzem wurden nun MFA-Methoden zur Bestimmung der Stoffwechselwege unter komplexen Wachstumsbedingungen entwickelt (Isotopolologue Profiling). Wie funktioniert Isotopologue Profiling? Um die Frage zu klären, welche bakteriellen Stoffwechselwege unter komplexen Umgebungsbedingungen aktiv sind, wird der pathogene Organismus unter Infektionsbedingungen kultiviert. Zusätzlich wird ein Kohlenstoffsubstrat (z. B. Glukose, Fettsäuren oder Aminosäuren) in 13C-markierter Form zugefüttert. 13C ist ein Stabilisotop, das in natürlicher Materie kaum vorkommt und daher durch Zufütterung stark angereichert werden kann. Im Falle von extrazellulär lebenden Pathogenen (z. B. Pseudomonaden) werden diese in einem synthetischen Medium kultiviert, das die chemische Umgebung des Infektionsorts simuliert (wie das Sputum von infi- ziertem Lungengewebe). Im Falle von intrazellulär lebenden Bakterien (z. B. Listerien) werden die markierten Verbindungen den infizierten Wirtszellen angeboten oder den Wirtsorganismen bereits vor der Infektion zugefüttert. Nach einer Wartezeit werden die Bakterien isoliert und stabile Metabolite (z. B. Aminosäuren aus Proteinhydrolysaten) gewonnen. Anschließend wird für jeden Metaboliten die Gesamtanreicherung an 13C sowie die Verteilung des Stabilisotops in unterschiedlichen Isotopologen mittels Kernresonanzspektroskopie (NMR) und Massenspektrometrie (MS) meist in Kopplung mit Gaschromatografie (GC) quantifiziert (Abb. 1). Während sich die NMR besonders durch ihre hohe Positionsauflösung auszeichnet, ist die MS wesentlich empfindlicher, und es können Experimente bereits mit geringen Probenmengen (ab ca. 107 Bakterien) durchgeführt werden. Was lernt man aus den Daten? Die 13C-Anreicherung und das Isotopologmuster der Metabolite zeigen nun spezifisch die jeweilige metabolische Vorgeschichte an. Diese Muster sind somit Fingerabdrücke aller Reaktionen, die zur Biosynthese des untersuchten Metaboliten beigetragen haben. Bei Fütterung von 13C-markierter Glukose kann aus der 13C-Anreicherung beispielsweise in Alanin die Effizienz der Aufnahme von Glukose in die bakterielle Zelle und deren Umbau zu Alanin abgeleitet werden. Da Alanin aus Brenztraubensäure gebildet wird, zeigt das Isotopologmuster von Alanin zusätzlich an, über welchen Stoffwechselweg diese gebildet wurde (z. B. Glykolyse, Pentosephosphatweg oder Entner-Doudoroff-Weg). In ähnlicher Weise können so die Bildungswege für alle Metabolite und deren Bausteine rekonstruiert werden. Die Messdaten werden dann in einen Gesamtfahrplan für den Stoffwechsel des Organismus unter den gewählten Bedingungen zusammengefügt. Beispiel Listeria: Anpassungen an den Wirtsstoffwechsel Die Methode zeichnet sich durch eine breite Anwendbarkeit aus. Derzeit wird die Technik an zahlreichen Mikroorganismen, wie Bakterien und Hefen, aber auch an Pflanzen und Tieren eingesetzt. Im Bereich der Infektionsbiologie wurde die Technik am pathogenen Bakterium Listeria monocytogenes eingeführt. Listeria ist verantwortlich für Listeriose, eine nicht selten tödlich verlaufende Lebensmittelvergiftung, deren Behandlung dringend verbesserte Medikamente benötigt [1, 2]. Im Verlauf der Infektion dringen die Bakterien in die menschlichen Zellen ein. Wie die Mikroben sich unter diesen intrazellulären Bedingungen ernähren, war lange Zeit völlig unklar. Das Isotopologue Profiling konnte schließlich direkt die Anpassung des bakteriellen Stoffwechsels an die komplexen Lebensbedingungen aufdecken. So zeigte das Markierungsmuster der Aminosäuren ausgehend von 13C-Glukose ein völlig verändertes Bild im Vergleich zu einem Referenzexperiment unter Kulturbedingungen (Abb. 2). Die MusBIOspektrum | 04.10 | 16. Jahrgang 432_490_BIOsp_0410.qxd 09.06.2010 10:51 Uhr ter belegten, dass unter intrazellulären Bedingungen mehrere Kohlenstoffsubstrate aus dem Zytosol der Wirtszelle genutzt werden. Insbesondere Aminosäuren und C3-Quellen wie Glycerin werden bevorzugt verwertet. Die Daten zeigten außerdem, dass der Citratzyklus im Stoffwechsel von Listeria nicht geschlossen ist und das Intermediat Oxalessigsäure (Oxa) durch Carboxylierung der Brenztraubensäure (Pyr) entsteht (Abb. 2). Die für diese essenzielle Reaktion involvierte Carboxylase könnte deshalb ein attraktives Target für neue Wirksubstanzen darstellen. Inzwischen werden weitere Krankheitserreger, wie Salmonellen, Staphylokokken, Streptokokken, Yersinien, Pseudomonaden, Legionellen und Chlamydien durch Isotopologue Profiling analysiert. Die ersten Daten zeigen eine enorme Anpassungsfähigkeit der Mikroben an die jeweiligen Lebensumgebungen mit nur wenigen Achillesfersen [3, 4]. Die gezielte Entwicklung von Medikamenten auf diese Targets wird ein vorrangiges Ziel der zukünftigen Infektionsbiologie und medizinischen Chemie sein. ó Literatur [1] Eisenreich W, Slaghuis J, Laupitz R et al. (2006) 13C isotopologue perturbation studies of Listeria monocytogenes carbon metabolism and its modulation by the virulence regulator PrfA. Proc Natl Acad Sci USA 103:2040–2045 [2] Eylert E, Schär J, Mertins S et al. (2008) Carbon metabolism of Listeria monocytogenes growing inside macrophages. Mol Microbiol 69:1008–1017 [3] Schmid A, Neumayer W, Trülzsch K et al. (2009) Crosstalk between type three secretion system and metabolism in Yersinia. J Biol Chem 284:12165–12177 [4] Eisenreich W, Dandekar T, Heesemann J et al. (2010) Carbon metabolism of intracellular bacterial pathogens and possible links to virulence. Nature Microbiol Rev, im Druck Korrespondenzadresse: PD Dr. Wolfgang Eisenreich Dr. Eva Eylert Technische Universität München Department Chemie Lehrstuhl für Biochemie Center of Isotopologue Profiling Lichtenbergstraße 4 D-85747 Garching Tel.: 089-289-13336 Fax: 089-289-13363 [email protected] www.biochemie.ch.tum.de BIOspektrum | 04.10 | 16. Jahrgang Seite 437











