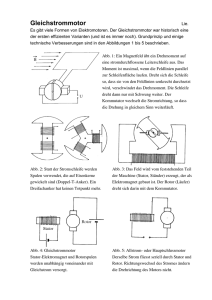
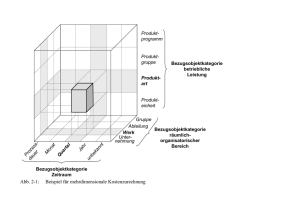
enzymkinetik - AECC Chemie
Werbung

ENZYMKINETIK Das Michaelis-Menten-Gesetz an ausgewählten Beispielen Fachbereichsarbeit im Fach Chemie Verfasst von Bernhard Seyer Betreuer: OStR. Prof. Mag. Dr. Manfred Kerschbaumer Albertus Magnus Gymnasium Vorgelegt im Februar 2009 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Inhaltsverzeichnis 1 DANKSAGUNG ..................................................................................................................................... 4 2 VORWORT ............................................................................................................................................ 5 3 ORGANISCHE GRUNDLAGEN .......................................................................................................... 6 3.1 VON DER AMINOSÄURE ZUM PROTEIN ................................................................................................... 6 3.1.1 Einführung in die Chemie der Aminosäuren ..................................................................................... 6 3.1.2 Bindungen zwischen Aminosäuren .................................................................................................... 8 3.1.3 Peptide und Proteine ....................................................................................................................... 10 3.1.4 Die Struktur der Proteine ................................................................................................................ 11 3.1.4.1 Die Primärstruktur .................................................................................................................................. 11 3.1.4.2 Die Sekundärstruktur .............................................................................................................................. 12 3.1.4.2.1 Die α-Helix ........................................................................................................................................ 13 3.1.4.2.2 Das β-Faltblatt ................................................................................................................................... 15 3.1.4.2.3 Die β-Kehre und die Ω-Schleife ........................................................................................................ 18 3.1.4.3 Die Tertiärstruktur .................................................................................................................................. 19 3.1.4.4 Die Quartärstruktur ................................................................................................................................. 21 4 PHYSIKALISCH-CHEMISCHE GRUNDLAGEN ............................................................................ 23 4.1 KATALYSE ............................................................................................................................................ 23 4.1.1 Einführung in die Katalyse .............................................................................................................. 23 4.1.2 Die Enzymkatalyse .......................................................................................................................... 24 4.1.2.1 4.1.2.2 4.1.2.3 Das aktive Zentrum ................................................................................................................................ 25 Das Schlüssel-Schloss-Prinzip................................................................................................................ 26 Das Induced-fit-Modell .......................................................................................................................... 27 4.2 KINETIK................................................................................................................................................ 27 4.2.1 Einführung in die Kinetik ................................................................................................................ 27 4.2.1.1 4.2.1.2 4.2.1.3 4.2.2 Definition der Reaktionsgeschwindigkeit ............................................................................................... 27 Die Reaktionsordnung ............................................................................................................................ 28 Quasistationarität .................................................................................................................................... 29 Die Michaelis-Menten-Kinetik ........................................................................................................ 30 4.2.2.1 4.2.2.2 4.2.2.3 Michaelis-Menten-Mechanismus............................................................................................................ 30 Michaelis-Menten-Gleichung ................................................................................................................. 31 Lineweaver-Burk-Diagramm .................................................................................................................. 33 4.3 HEMMUNG............................................................................................................................................ 34 4.3.1 Kompetitive Hemmung .................................................................................................................... 35 4.3.2 Nichtkompetitive Hemmung ............................................................................................................ 36 4.3.3 Unkompetitive Hemmung ................................................................................................................ 37 5 ANALYTISCH-CHEMISCHE GRUNDLAGEN ................................................................................ 39 5.1 INSTRUMENTELLE ANALYTIK ............................................................................................................... 39 5.1.1 Einführung in die Photometrie ........................................................................................................ 39 5.1.1.1 5.1.1.2 5.1.1.3 5.1.1.4 6 Apparativer Aufbau eines Photometers .................................................................................................. 39 Prinzip der Messung ............................................................................................................................... 40 Transmission und Extinktion .................................................................................................................. 40 Lambert-Beer„sches Gestetz ................................................................................................................... 41 EIGENE EXPERIMENTE .................................................................................................................. 43 6.1 VORBEREITUNGEN ............................................................................................................................... 43 2 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 6.1.1 Synthese von Cystin aus Cystein ..................................................................................................... 43 6.1.2 Herstellung des Britton-Robinson-Puffers ...................................................................................... 45 6.1.3 Herstellung des Phosphat-Puffers ................................................................................................... 46 6.1.4 Reinigung von Guajakol durch Vakuumdestillation ........................................................................ 47 6.2 CHARAKTERISIERUNG DER MEERRETTICHPEROXIDASE ........................................................................ 49 6.2.1 Thereotische Einführung ................................................................................................................. 49 6.2.1.1 6.2.1.2 6.2.1.3 6.2.2 Organische-chemische Charakteristika des Enzyms ............................................................................... 49 Isolierung des Enzyms aus Kren ............................................................................................................. 51 Lagerfähigkeit und Reinheitszahl ........................................................................................................... 51 Eigene Versuche .............................................................................................................................. 52 6.2.2.1 6.2.2.2 6.2.2.3 Überprüfung der Reinheitszahl ............................................................................................................... 52 Aktivitätsbestimmung mit Guajakol als Elektronendonator ................................................................... 53 Überprüfung der Temperaturempfindlichkeit ......................................................................................... 55 6.3 MESSUNG DER MICHAELIS-MENTEN-KINETIK ..................................................................................... 57 6.3.1 Messung mit ABTS als Elektronendonator ...................................................................................... 57 6.3.2 Bestimmung der Hemmung durch Cystin und Cadmium auf ABTS................................................. 59 6.3.3 Messung mit p-Phenylendiamin als Elektronendonator .................................................................. 61 7 ZUSAMMENFASSUNG ...................................................................................................................... 65 8 SUMMARY .......................................................................................................................................... 65 9 ANHANG.............................................................................................................................................. 66 9.1 9.2 9.3 9.4 9.5 9.6 9.7 VERWENDETE LITERATUTR .................................................................................................................. 66 VERWENDETE ZEITSCHRIFTENBEITRÄGE.............................................................................................. 67 VERWENDETE INTERNETSEITEN ........................................................................................................... 67 E-MAILS ............................................................................................................................................... 68 ABBILDUNGSVERZEICHNIS ................................................................................................................... 68 PROTOKOLL.......................................................................................................................................... 70 ERKLÄRUNG ......................................................................................................................................... 73 3 Albertus Magnus Gymnasium 1 Bernhard Seyer / Enzymkinetik Danksagung Zu Beginn meiner Fachbereichsarbeit möchte ich all jenen danken, die mich beim Verfassen dieser unterstützt und begleitet haben. An erster Stelle ist hier mein Chemieprofessor und Betreuer Prof. Dr. Manfred Kerschbaumer zu nennen. Dieser hat mich nicht nur durch Beratung unterstützt, sondern ist sogar an Wochenenden und in den Ferien in die Schule gekommen, um mir das praktische Arbeiten zu ermöglichen. Doch auch durch die Unterstützung von Dir. Mag. Christian Köhler, welcher den Ankauf von Chemikalien bewilligt hat, ist diese Fachbereichsarbeit erst möglich geworden. Mein besonderer Dank gilt Ao. Univ. Prof. Mag. Dr. Christian Obinger. Dieser hat mir eine detaillierte Arbeitsanleitung für die Extraktion von Meerrettichperoxidase aus Kren und die Charakterisierung derselben übermittelt, welche teilweise theoretisch und teilweise praktisch Eingang in meine Fachbereichsarbeit gefunden hat. Weiters gilt mein Dank Univ. Prof. Dipl.-Ing. Dr. Paul Kosma für die Übermittlung einer Arbeitsanleitung für die Messung der Michaelis-Menten-Kinetik von Meerrettichperoxidase mit o-Phenylendiamin als Elektronendonator. Diese hat mir den Denkanstoß gegeben eine Messung mit p-Phenylendiamin durchzuführen. 4 Albertus Magnus Gymnasium 2 Bernhard Seyer / Enzymkinetik Vorwort Diese Fachbereichsarbeit widmet sich vorwiegend der Enzymkinetik und somit dem Michaelis-Menten-Gesetz. Dabei soll jedoch die Universalität dieses Themas nicht in den Hintergrund geraten. So wird im ersten Kapitel die organische Chemie in den Mittelpunkt gestellt. Im Hinblick auf den Aufbau von Enzymen werden hier Aminosäuren, Peptide, Proteine und deren Eigenschaften näher betrachtet. Das darauf folgende Kapitel beschäftigt sich mit den mathematischen und physikalischchemischen Grundlagen, um so die Basis für die Auswertung der nachfolgenden Messungen zu legen. Nach einer kurzen allgemeinen Einführung in die Kinetik wird die MichaelisMenten-Kinetik hergeleitet und schließlich die Hemmung von Enzymen betrachtet. Das vorletzte Kapitel steht im Zeichen der instrumentellen Analytik und geht besonders auf die Messung von Absorptionsänderungen mittels Photometrie ein, da diese die von mir verwendete Messmethode zur Bestimmung der Enzymkinetik ist. Im abschließenden Kapitel werden die praktischen Messungen dokumentiert, wobei zu Beginn eine theoretische Meerrettichperoxidase Einführung herangezogen, steht. wobei Für diese die zuerst kinetische Messung charakterisiert wird wird und anschließend mit verschiedenen Elektronendonatoren und Inhibitoren photometrische Messungen durchgeführt werden. 5 Albertus Magnus Gymnasium 3 Bernhard Seyer / Enzymkinetik Organische Grundlagen 3.1 Von der Aminosäure zum Protein 3.1.1 Einführung in die Chemie der Aminosäuren Die Grundbausteine der Proteine (und somit auch der Enzyme) sind Aminosäuren 1. „Obwohl es mehr als fünfhundert natürlich vorkommende Aminosäuren gibt, bestehen die Proteine aller Organismen, von den Bakterien bis zum Menschen, zum überwiegenden Teil aus nur zwanzig verschiedenen Aminosäuren.“2 Im Laufe der Evolutionsgeschichte haben sich also nur wenige Arten aus der großen Bandbreite der vielfältigen Aminosäuren herauskristallisiert (siehe Abb. 1). Abb. 1: Die zwanzig biochemisch bedeutendsten Aminosäuren 1 2 Genauer: Aminocarbonsäure; veraltet auch Amidosäure Vollhardt; Schore, 2005, S. 1372 6 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Weshalb eben diese Aminosäuren evolutionär begünstigt wurden, ist nicht vollkommen geklärt, jedoch wird davon ausgegangen, dass dies mehrere Gründe hat. Sowohl die vielfältigen funktionellen Gruppen in den Seitenketten und somit auch mannigfaltigen chemischen Eigenschaften als auch ihre große Reaktivität dürften ausschlaggebend gewesen sein. „Ihre strukturellen und chemischen Eigenschaften decken ein weites Spektrum ab und befähigen Proteine, eine große Bandbreite an Funktionen wahrzunehmen.“3 Des Weiteren ist es auch wahrscheinlich, dass sie im Laufe der präbiotischen Evolution zufällig entstanden sind und somit für die biochemische Evolution verfügbar waren. Den im Folgenden erwähnten Aminosäuren ist gleich, dass sie ein (in der Regel chirales) zentrales Kohlenstoffatom (C, α-Kohlenstoff) mit einer Säuregruppe (–COOH), einer Aminogruppe (–NH2), einem Wasserstoff und einer Seitenkette (R für „Rest“) besitzen. Man spricht von α-Aminosäuren. Da bis auf Glycin (R = H) alle Aminosäuren chiral sind, kann zwischen L- und D-Aminosäuren unterschieden werden (siehe Abb. 2), wobei das L-Isomer zumeist der R-Konfiguration und das D-Isomer zumeist der S-Konfiguration entspricht (siehe Abb. 3). In der Natur kommen aus unbekannten Gründen jedoch nur L-Isomere vor. Abb. 2: Die dreidimensionale Konfiguration der L- und D-Isomere Abb. 3: Dreidimensionale Darstellung der S-Konfiguration „Aminosäuren liegen in Lösung bei neutralem pH-Wert vorwiegend als dipolare Ionen (oder Zwitterionen) vor.“4 Das heißt, dass die Aminogruppe protoniert (–NH3+) und die Carboxylgruppe deprotoniert ist (–COO-). Der pH-Wert, bei dem die Zahl der protonierten Aminogruppen und der deprotonierten Carboxylgruppen gleich ist, wird isoelektrischer Punkt genannt. In elektrischen Feldern bewegen sich Aminosäuren nicht mehr und ihre Löslichkeit hat ein Minimum erreicht. In sauren Lösungen, ist die Aminogruppe protoniert und die Carboxylgruppe noch nicht dissoziiert, in basischen Lösungen ist die Aminogruppe nicht protoniert und die Carboxylgruppe dissoziiert (siehe Abb. 4). 3 4 Berg; Tymoczko; Stryer, 2007, S. 36 Berg; Tymoczko; Stryer, 2007, S. 29 7 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 4: Ionisierungsgrad einer Aminosäure in Abhängigkeit zum pH-Wert Die Beschaffenheit der Seitenketten entscheidet maßgeblich über die Eigenschaften der Aminosäuren. Lange, aliphatische Seitenketten sind hydrophob und daher auch weniger gut wasserlöslich. Polare Seitenketten sind hydrophil und fördern somit die Wasserlöslichkeit der Aminosäure. Aminosäuren werden, um die Sequenz in Proteinen widerzugeben, meist durch drei Buchstaben abgekürzt (z.B. Ala für Alanin, Leu für Leucin, etc.). 3.1.2 Bindungen zwischen Aminosäuren Aminosäuren können unverzweigte, lineare Polymere bilden. Dabei bindet sich eine α- Aminogruppe einer Aminosäure an die α-Carboxylgruppe einer anderen Aminosäure unter Wasserabspaltung. Das Gleichgewicht dieses Prozesses liegt links, daher ist für die Biosynthese dieser Polymere Energie notwendig. Man bezeichnet die resultierenden Verbindungen als Peptide, beziehungsweise Proteine. Diese Art der Bindung wird daher Peptid- oder Amidbindung genannt (siehe Abb. 5). Abb. 5: Ausbildung einer Peptidbindung 8 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Nach ihrer Länge werden Peptide als Di-, Tri-, Tetra,… bis Polypeptide bezeichnet, wobei kurzkettige auch als Oligopeptide oder nur Peptide bezeichnet werden. „Ein Polypeptid besteht aus sich regelmäßig wiederholenden Einheiten, welche die Hauptkette oder das Rückgrat bilden und einem variablen Anteil, den einzelnen Seitenketten.“5 (siehe Abb. 6). Abb. 6: Aufbau einer Polypeptidkette, die Seitenketten sind grün eingezeichnet Natürliche Polypeptideketten besitzen 50 bis 2 000 Aminosäurereste und werden allgemein Proteine genannt. Eines der größten bekannten Proteine, das Muskelprotein Titin (siehe Abb. 7), besteht aus über 27 000 Aminosäuren. Da die durchschnittliche Molekülmasse einer Aminosäure 110 g mol-1 beträgt, liegen die Molmassen der Proteine zwischen 5 500 und 220 000 g mol-1. Statt der Einheit g mol-1 kann auch die Einheit Dalton verwendet werden, welche einem u (= 1,660 538 782 (83) · 10−27 kg) entspricht. B A Abb. 7: Titin. A: Bänderdiagramm; B: Kugel-Stab-Modell. Zur besseren Übersicht wurden einzelne Strukturelemente eingefärbt Zwischen Aminosäuren innerhalb einer Kette können Wasserstoffbrückenbindungen hergestellt werden. Dabei dienen die Cabonylgrupen (C=O) als Wasserstoffakzeptoren und die NH-Gruppen als Wasserstoffdonatoren (einzig Prolin bildet aufgrund ihres Aufbaus keine NH-Gruppen aus, sondern besitzt nur ein an drei Kohlenstoffatome gebundenes N-Atom). Diese Gruppen interagieren nicht nur untereinander, sondern auch mit Gruppen aus den Seitenketten. 5 Berg; Tymoczko; Stryer, 2007, S. 37 9 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Eine weitere Bindungsmöglichkeit ist die Ausbildung von Querbrücken innerhalb der Polypeptidketten. Die häufigste Möglichkeit dafür ist eine Disulfidbrücke (–S–S–). Die Bildung dieser Brücke erfolgt durch Oxidation zweier Moleküle Cystein zu einem Molekül Cystin (siehe Abb. 8). „Extrazelluläre Proteine verfügen oftmals über mehrere Disulfidbrücken, während diese in intrazellulären Proteinen in der Regel fehlen“6 Abb. 8: Bildung von Cystin aus Cystein durch Ausbildung einer Disulfidbrücke 3.1.3 Peptide und Proteine Polypeptidketten sind polar, da sie verschiedene Enden (eine α- Amino- und eine α-Carboxylgruppe) besitzen. Einer vereinbarten Regel folgend wird die Aminosäurensequenz so aufgezählt, dass bei dem aminoständigen Rest begonnen wird. Bei der A-Kette des Rinderinsulins (siehe Abb. 9), welches das erste vollkommen sequenzierte Protein war, ist demnach Glycin der aminoterminale (N-terminale) und Asparagin der carboxyterminale (C-terminale) Rest. Abb. 9: Die Aminosäuresequenz des Rinderinsulins 6 Berg; Tymoczko; Stryer, 2007, S. 38 10 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Die Eigenschaften der Peptide sind durch ihre Aminosäuren bestimmt. Aminosäuren mit hydrophoben Seitenketten helfen dabei, die Struktur der Peptide auch in wässriger Lösung zu erhalten, diverse andere schaffen die Grundlagen für die Reaktivität der Peptide und Proteine. 3.1.4 Die Struktur der Proteine Die Struktur der Proteine kann auf vier Ebenen betrachtet werden: der Ebene der Primär-, der Sekundär-, der Tertiär- und der Quartärstruktur. 3.1.4.1 Die Primärstruktur Die Primärstruktur eines Proteins entspricht dessen Aminosäuresequenz. Diese beruht folglich nur auf der Abfolge der Aminosäuren. Die Sequenz an sich kann somit als quasi eindimensional betrachtet werden, der molekulare Aufbau dieser Kette nimmt jedoch erheblichen Einfluss auf die dreidimensionale Gestalt des Proteins. Die Kenntnis der Primärstruktur (heute sind die Sequenzen von über 100 000 Proteinen bekannt) ist für das Wissen über Eigenschaften und Reaktivität eines Proteins beinahe unerlässlich. Dies zeigt sich besonders im Zusammenhang mit Enzymen und bei der Betrachtung ihrer aktiven Zentren. „Überdies lassen sich Proteine mit neuartigen Eigenschaften erzeugen, indem man Sequenzen bekannter Proteine variiert.“7 Bei der Untersuchung der Struktur des Grundgerüstes lässt sich leicht erkennen, dass die Peptidbindung prinzipiell planar ist, es liegen sowohl die α-Kohlenstoffe beider Aminosäuren als auch die CO- und die NH-Gruppe in einer Ebene. Dies rührt daher, dass die Peptidbindung über einen Doppelbindungscharakter verfügt. Es gibt somit zwei Resonanzstrukturen: eine, bei der sich die Doppelbindung zwischen C und O befindet und eine, bei der sich die Doppelbindung zwischen C und N befindet. Eine Drehung um die erwähnte Bindung wird durch diesen Doppelbindungscharakter verhindert. „Dieser wird auch an der kleinen Bindungslänge deutlich, die mit 132 pm zwischen der einer reinen C–N-Einfachbindung (147 pm) und der einer C–N-Doppelbindung (127 pm) liegt.“8 (siehe Abb. 10) 7 8 Berg; Tymoczko; Stryer, 2007, S. 39 Vollhardt; Schore, 2005, S. 1385 11 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 10: Charakteristische Bindungslängen einer Peptidbindung Durch diese Eigenschaft existieren zwei mögliche Konfigurationen, die cis- und die transKonfigurationen (siehe Abb. 11). Die trans-Konfiguration ist gegenüber der cis-Konfiguration jedoch deutlich bevorzugt, sodass fast alle Peptidbindungen in trans-Konfiguration geknüpft sind. „Diese Bevorzugung von trans gegenüber cis lässt sich dadurch erklären, dass eine cisVerknüpfung durch sterische Kollisionen zwischen den Gruppen am α-Kohlenstoff gestört würde, was bei der trans-Konfiguration nicht der Fall ist.“9 Einzig zwischen Prolin und einer beliebigen Aminosäure ist die Bindung cis-geknüpft. Dies hat den Grund, dass durch den cyclischen Aufbau des Prolin bei beiden Konfigurationen mit sterischer Hinderung zu rechnen ist, jedoch ist diese bei cis-Konfiguration schwächer. Abb. 11:Trans- (links) und cis-Isomere Die Bindung zwischen dem α-Kohlenstoff und der Amino-, beziehungsweise Carbonylgruppe besitzt reinen Einfachbindungscharakter. Somit kann um diese Bindung frei gedreht werden und es kann zu einer Faltung des Proteins kommen. 3.1.4.2 Die Sekundärstruktur „Die Sekundärstruktur kommt teilweise durch Disulfidbrücken, hauptsächlich jedoch aufgrund der Starrheit der Amidbindung und der Maximierung der Wasserstoffbrücken und 9 Berg; Tymoczko; Stryer, 2007, S. 40 12 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik anderen nichtkovalenten Bindungen entlang der Kette(n) zustande.“10 Auf Basis dieses Wissens postulierten Linus Pauling und Robert Corey 1951 zwei periodische Polipeptidstrukturen, die α-Helix und das β-Faltblatt. Später wurden diese beiden Strukturen mittels Röntgenstrukturanalyse nachgewiesen und noch weitere Elemente wie Kehren und Schleifen, welche jedoch keinen periodischen Aufbau besitzen, entdeckt. Diese Strukturen werden meist durch Wasserstoffbrücken zwischen den C=O- und den N–H-Gruppen der Aminosäuren gebildet. Allgemein werden solche Abschnitte Sekundärstruktur genannt. 3.1.4.2.1 Die α-Helix Die erste von Pauling und Corey entdeckte Struktur war die annähernd stabförmige α-Helix11. Das Rückgrat der Aminosäuresequenz ist dabei schraubenförmig aufgerollt, die Reste weisen stachelartig nach außen (siehe Abb. 12). Abb. 12: Aufsicht einer α -Helix, die stachelartig abstehenden Reste (grün) sind besonders gut zu erkennen α-Helices werden durch Wasserstoffbrückenbindungen zwischen der CO- und der NHGruppe gebildet, wobei jede CO-Gruppe an die vier Reste entfernte NH-Gruppe bindet. „Folglich sind außer bei den Aminosäuren am Ende der α-Helices alle CO- und NH-Gruppen der Hauptkette an den Wasserstoffbrücken beteiligt. Jeder Rest ist gegen den nächsten um 0,15 nm entlang der Helixachse verschoben, auch Translation genannt, und um 100° verdreht, sodass eine volle Umdrehung der Helix 3,6 Aminosäurenresten entspricht.“12 Die Ganghöhe 10 Vollhardt; Schore, 2005, S. 1389 α, da es sich um die als erstes entdeckte Struktur handelt 12 Berg; Tymoczko; Stryer, 2007, S. 44 11 13 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik der Helix (das Produkt aus Schiebung und Windung) beträgt also 0,54. Grundsätzlich kann der Drehsinn einer Helix nach rechts oder links weisen, beide Konformationen sind erlaubt, jedoch ist aus sterischen Gründen die rechtsgängige Konformation energetisch günstiger und daher in fast allen Fällen gegenüber der linksgängigen bevorzugt. Diese zunächst nur theoretisch vorhergesagte Struktur wurde sechs Jahre später mittels Röntgenstrukturanalyse an der Myoglobinstruktur (siehe Abb. 13) tatsächlich bestätigt. Abb. 13: Dreidimensionale Struktur des Myoglobin. A) Bänderdiagramm B) Kalottenmodell. Eine Helix wurde zur besseren Vergleichbarkeit der beiden Darstellungen blau gefärbt Diese und die Struktur des Hämoglobins wurden 1960 von dem Österreicher Max Ferdinand Perutz und dem Briten John Cowdery Kendrew aufgeklärt. Für eben diese Leistung erhielten die beiden Chemiker 1962 den Nobelpreis für Chemie. Der Anteil der α-Helices an der Gesamtstruktur eines Proteins kann von beinahe null bis annähernd 100 Prozent gehen, wobei eine einzelne α-Helix selten länger als 4,5 nm ist. Des Weiteren bestehen 25 Prozent aller in Wasser löslichen Proteine aus α-Helices, welche durch Kehren und Schleifen verbunden sind (siehe Abb. 14). Abb. 14: Ein größtenteils α-helicales Protein 14 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Bei der schematischen Darstellung werden α-Helices als gedrehte Bänder dargestellt, wobei auch eine Darstellung als Stab möglich ist (siehe Abb. 15). In manchen, seltenen Fällen werden die Seitenketten der α-Helix zusätzlich zu dem Bänderdiagramm herausgehoben und eingezeichnet, um gewisse stereochemische oder für Reaktionen relevante Eigenschaften zu verdeutlichen. Abb. 15: Darstellung einer α-Helix. A) Kugel-Stab-Modell B) Bänderdiagramm C) Zylindrische Darstellung 3.1.4.2.2 Das β-Faltblatt Bei β-Faltblättern13 (β-sheet, β-pleated sheet) liegt die Polypeptidkette, in diesem Fall als βStrang bezeichnet, nicht aufgerollt, sondern beinahe vollkommen ausgestreckt vor, wobei die Seitenketten benachbarter Aminosäuren in entgegengesetzte Richtungen weisen (siehe Abb. 16). Der Abstand zwischen zwei benachbarten Aminosäuren beträgt 0,35 nm, also um 0,2 nm mehr als bei der α-Helix. 13 β, da es sich um die als zweites entdeckte Struktur handelt 15 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 16: Struktur eines β-Stranges Durch diese Gegebenheiten wird ein breites Spektrum an flächigen Strukturen möglich. Sie sind in ihrer Struktur vielfältiger als α-Helices und trotz ihres flächigen Aufbaus meist leicht in sich gedreht (siehe Abb. 17). Abb. 17: In sich gedrehte β-Stränge. A) Kugel-Stab-Modell, B) Darstellung als Pfeile C) um 90° gedrehte Darstellung von B) Innerhalb eines β-Faltblattes können sich zwei β-Stränge durch Wasserstoffbrücken miteinander verbinden. Dies geschieht entweder parallel, das heißt, dass beide Stränge dieselbe Richtung aufweisen (siehe Abb. 18), oder antiparallel, das heißt, dass die Stränge entgegengesetzte Richtungen haben (siehe Abb. 19). Verbinden sich drei β-Stränge zu einem 16 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik β-Faltblatt, wobei sich zwei Stränge zueinander parallel und zwei zu einander antiparallel verhalten, spricht man von einem gemischten β-Faltblatt (siehe Abb. 20). β-Faltblätter besitzen vor allem bei Fettsäure bindenden Proteinen große Bedeutung. Abb. 18: Ein paralleles β-Faltblatt Abb. 19: Ein antiparalleles β-Faltblatt Abb. 20: Ein gemischtes β-Faltblatt Bei der schematischen Darstellung werden β-Stränge zumeist als breite Pfeile dargestellt, wobei ihre Spitzen in Richtung des carboxyterminalen Endes weisen. Dadurch kann der Typ des Faltblattes (parallel/antiparallel/gemischt) angezeigt werden (siehe Abb. 21). Abb. 21: Ein größtenteils aus β-Faltblättern bestehendes Protein 17 Albertus Magnus Gymnasium 3.1.4.2.3 Bernhard Seyer / Enzymkinetik Die β-Kehre und die Ω-Schleife Die β-Kehre, oft auch Haarnadelkehre genannt (reverse, β-turn, hairpin bend), ermöglicht eine abrupte Richtungsänderung innerhalb eines Polypeptidstranges. Dabei ist eine CO-Gruppe einer Aminosäure mit der NH-Gruppe der in der Kette dritten nachfolgenden Aminosäure verknüpft (siehe Abb. 22). Abb. 22: Struktur einer Kehre, die Wasserstoffbrückenbindung ist grün strichliert eingezeichnet Kompliziertere Strukturen, welche einen Richtungswechsel der Polypeptidkette ermöglichen, sind Schleifen, oft auch wegen ihrer Struktur als Ω-Schleifen (Ω-loops) bezeichnet. Kehren und Schleifen sind ihm Gegensatz zu α-Helices und β-Faltblättern keine periodischen, aber definierte Strukturen. Sie liegen immer an der Proteinoberfläche und sind oft an Interaktionen des Proteins beteiligt (siehe Abb. 23). Abb. 23: Schleifen an der Oberfläche eines Proteins (rot eingezeichnet) 18 Albertus Magnus Gymnasium 3.1.4.3 Bernhard Seyer / Enzymkinetik Die Tertiärstruktur „Die Gesamtanordnung der Polypeptidkette eines Proteins wird als Tertiärstruktur bezeichnet.“14 Der Aufbau der Tertiärstruktur, also die Abfolge und Lage der soeben vorgestellten Sekundärstrukturen, ist zumeist komplex und nicht symmetrisch. Die Verteilung von polaren und unpolaren Gruppen sagt viel über die Eigenschaften eines Proteins aus. Myoglobin, der Sauerstoffträger im Muskel, zum Beispiel besitzt in seinem Inneren außer zweier Histine, welche für die Bindung von Eisen und Sauerstoff verantwortlich sind, nur unpolare Reste. Das Äußere hingegen besitzt sowohl polare als auch unpolare Areale (siehe Abb. 24). Abb. 24: Verteilung der Aminosäuren im Myoglobin. A) Kalottenmodell des Myoglobins: hydrophobe Aminosäuren sind gelb, hydrophile blau und andere weiß eingezeichnet B) Durch einen Querschnitt wird ersichtlich, dass der Großteil der hydrophoben Aminosäuren im Inneren des Proteins liegen In wässrigem Milieu werden sich hydrophobe Reste allgemein nach innen kehren, um dem Wasser auszuweichen, hydrophile werden außen bleiben. Es kommt in wässriger Lösung also zu einer spontanen Faltung der Polypeptidkette. α-Helices und β-Stränge sind meist amphipatisch, dass heißt sie besitzen sowohl einen hydrophoben Anteil, welcher in der Regel im Inneren des Proteins verbleiben wird, als auch einen hydrophilen Anteil, welcher der Außenseite des Proteins zugewandt sein wird. Auch nehmen ungepaarte CO- und NHGruppen Einfluss auf die Struktur. „Eine ungepaarte NH- oder CO-Gruppe der Peptidbindung zieht Wasser einem unpolaren Medium vor.“15 Besitzen sie jedoch hydrophobe Seitenketten 14 15 Berg; Tymoczko; Stryer, 2007, S. 50 Berg; Tymoczko; Stryer, 2007, S. 51 19 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik und werden über Wasserbrücken (z.B. zu α-Helices oder β-Faltblättern) gepaart, so sind auch sie in hydrophober Umgebung stabil. Ein weiterer Effekt, der die Teritiärstruktur eines Proteins beeinflusst sind die Van-der-Waals-Kräfte, welche zwischen den eng aneinander liegenden Polypeptidketten herrschen. Wenn es die Funktion des Proteins erfordert, kann jedoch auch gegen die oben genannten Regeln verstoßen werden. Es liegt also der hydrophile Bereich im Proteininneren und der hydrophobe außen. Dies ist zum Beispiel bei Porinen, Molekülen in der Membran von Bakterien, welche Kanäle in der durch vor allem Alkane geprägten Permiabilitätsbarrieren bilden, der Fall. Der hydrophobe, aliphatische Teil der Porine interagiert mit dieser, das polare Innere ist mit Wasser gefüllt und stellt somit einen Kanal dar (siehe Abb. 25). Abb. 25: Struktur des Porin Gewisse Kombinationen von Sekundärstrukturen, wie zum Beispiel das Helix-Kehre-Helix Motiv, sind sehr häufig zu finden. Solche Einheiten werden Strukturmotive oder Supersekundärstrukturen genannt. Auch falten sich einige Polypeptidketten zu Strukturen, welche wie Perlen auf einer Kette wirken. Diese kompakten Einheiten, die Domänen, welche die Perlen darstellen und durch flexible Polypetidsequenzen miteinander verbunden sind, bestehen aus 30 bis 400 Aminosäuren. 20 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Durch das große Repertoire an Eigenschaften, welche die 20 Aminosäuren bilden, ist es so möglich das Innere eines Proteins unter bester Ausnützung der Van-der-Waals-Kräfte möglichst gut auszufüllen, die definierte, fixe Form eines Proteins sicherzustellen und diesem dabei gewünschte Funktionen und Eigenschaften zu verleihen. Eine Zerstörung der Tertiärstruktur, nicht aber der Primär- und Sekundärstruktur, wird Denaturierung genannt. Sie kann als Umkehrung der spontanen Faltung von Proteinen betrachtet werden (siehe Abb. 26). Dabei kann das Protein seine Eigenschaften verändern und somit seine Wirkung verlieren. Abb. 26: Spontane Faltung eines Proteins 3.1.4.4 Die Quartärstruktur Viele Proteine verfügen über mehr als nur eine Polypeptidkette, welche auch als Untereinheit bezeichnet wird. Die räumliche Anordnung all dieser Untereinheiten und die Wechselwirkungen innerhalb dieser wird als Quartärstruktur bezeichnet. Besteht die Quartärstruktur aus zwei identischen Ketten, so wird diese als Dimer bezeichnet. Doch dies ist nur selten der Fall. Meist ist die Quartärstruktur weitaus komplexer und enthält viele verschiedene Untereinheiten. Das menschliche Hämoglobin zum Beispiel besteht aus je zwei identen Untereinheiten, den α- und den β-Ketten (siehe Abb. 27). 21 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 27: Das α2β2-Tetramer des menschlichen Hämoglobins. A) Bänderdiagramm B) Kalottenmodell In diesem Fall spricht man von einem α2β2-Tetramer. Oft bilden auch viele Kopien derselben Art eines Proteins Untereinheiten, welche mit Untereinheiten anderer Proteinspezies komplexere Strukturen wie zum Beispiel Hüllen von Viren bilden (siehe Abb. 28). Abb. 28: Hülle des menschlichen Rhinovirus, die Untereinheiten sind in verschiedenen Farben dargestellt 22 Albertus Magnus Gymnasium 4 Bernhard Seyer / Enzymkinetik Physikalisch-chemische Grundlagen 4.1 Katalyse 4.1.1 Einführung in die Katalyse „Als Katalysator16 bezeichnet man eine Substanz, deren Anwesenheit eine Reaktion beschleunigt, ohne dass sie selbst eine (Netto)reaktion eingeht.“17 Hierbei senkt der Katalysator die Aktivierungsenergie, indem er einen energetisch günstigeren Reaktionsweg ermöglicht. Der Energieunterschied ΔG‡ des Übergangszustandes X‡, wobei G die GibbsEnergie (auch freie Enthalpie)18, ein thermodynamisches Potential ist, X für einen beliebigen Zustand steht und das Symbol ‡ kurzlebige Zustände oder Produkte markiert (siehe Abb. 29). Allgemein wird zwischen homogener, heterogener und Enzymkatalyse unterschieden. Bei der homogenen Katalyse befinden sich Katalysator und Reaktionsmischung in der gleichen Phase, bei der heterogenen in verschiedenen. Die dritte Art der Katalyse, auf welche im Folgenden näher eingegangen wird, besitzt sowohl Charakteristika der homogenen Katalyse als auch der heterogenen Katalyse: die Enzymkatalyse. Abb. 29: Vergleich des Reaktionsverlaufs zwischen einer katalysierten und einer unkatalysierten Reaktion 16 Griech. Καταλύειν, sich auflösen, losbinden, aufheben Atkins; Paula, 2006, S. 930 18 Nach Eyring ist die freie Enthalpie, also nicht lediglich nur die Enthalpie, sondern auch die Entropie bei Reaktionen allgemein und so auch bei katalysierten Reaktionen bedeutend 17 23 Albertus Magnus Gymnasium 4.1.2 Bernhard Seyer / Enzymkinetik Die Enzymkatalyse Ein Großteil aller biochemischen Prozesse wird durch Enzyme beschleunigt, gesteuert, oder sogar erst ermöglicht. Enzyme sind spezialisierte Proteine, welche in großer Varietät in fast allen natürlichen Matrizes vorhanden sind. Ist eine katalytische Wirkung durch RNAMoleküle gegeben, so spricht man von Ribozymen. RNA ist eines der ersten biochemisch bedeutenden Moleküle. Sie dürfte neben der Speicherung von Informationen auch katalytische Aufgaben übernommen haben und kann somit als „Urenzym“ angesehen werden. „In einer eleganten Versuchsreihe wies die Arbeitsgruppe um Thomas Cech nach, dass sich die RNA in dieser [Anm.: Bezug auf die Entfernung eines Introns] Reaktion selbst spleißt und das Intron herrausschneidet.“19 Da jedes Enzymen nur in der Lage ist eine bestimmte Menge an Substratmolekülen zu binden, stehen bei enzymkatalysierten Reaktion nur eine begrenzte Anzahl an Bindungsstellen zur Verfügung, was dazu führt, dass sich bei gleichbleibender Enzymkonzentration und wachsender Substratkonzentration die Reaktionsgeschwindigkeit einer maximalen Geschwindigkeit annähert, welche dann erreicht ist, wenn alle Bindungsstellen der Enzyme besetzt sind (siehe Abb. 30). Dieses Charakteristikum hat die Ezymkatalyse mit der heterogenen Katalyse gemein. Abb. 30: Reaktionsgeschwindigkeit in Abhängigkeit von der Substratkonzentration. 19 Berg; Tymoczko; Stryer, 2007, S.948 24 Albertus Magnus Gymnasium 4.1.2.1 Bernhard Seyer / Enzymkinetik Das aktive Zentrum Enzyme besitzen ein aktives Zentrum, welches Bindungen mit Substraten eingehen kann (Enzym-Substrat-Komplex) und diese dort zu den Produkten umsetzt. Dieses muss nicht nur aus Aminosäuren bestehen, sondern kann auch Cofaktoren besitzen. Diese können kleine organische Moleküle (Coenzyme) oder Metalle bzw. Metallionen sein. Ist ein Coenzym an das zugehörige Enzym fest gebunden, so nennt man dies prosthetische Gruppe. „Ein Enzym ohne Cofaktor wird als Apoenzym bezeichnet; das vollständige katalytisch aktive Enzym heißt Holoenzym.“20 In Tabelle 1 sind einige Enzyme mit ihren Coenzymen bzw. ihren Metallen aufgelistet. Enzym Cofaktor Coenzym Acetyl-CoA-Carboxylase Coenzym A (CoA) Glycogen-Phosphorylase Pyridoxalphosphat Lactat-Dehydrogenase Nicotinamidadenindinucleotid (NAD+) Monoamin-Oxidase Flavinadeninnucleotid Pyruvat-Carboxylase Biotin Pyruvat-Dehydrogenase Thiaminpyrophosphat Metall Carboanhydrase Zn2+ Gluthation-Peroxidase Se Hexokinase Ni2+ Nitrat-Reduktase Mo Propionyl-CoA-Carboxylase K+ Superoxid-Dismutase Mn2+ Urease Mg2+ Tab. 1: Enzyme mit ihren Cofaktoren 20 Berg; Tymoczko; Stryer, 2007, S.230 25 Albertus Magnus Gymnasium 4.1.2.2 Bernhard Seyer / Enzymkinetik Das Schlüssel-Schloss-Prinzip Enzyme sind hochspezifisch, ihre aktive Position ist auf das umzusetzende Substrat und die Art der Reaktion abgestimmt. Das Substrat bildet mit dem aktiven Zentrum des Enzyms Wechselwirkungen wie Van-der-Waals-Kräfte oder Wasserstoffbrückenbindungen aus. Der so resultierende Komplex wird Enzym-Substrat-Komplex (kurz ES-Komplex) genannt (siehe Abb. 31). Das Schlüssel-Schloss-Prinzip versucht diesen Vorgang zu erklären, in dem es die dreidimensionalen Strukturen des Enzyms und des Substrats mit einem Schlüssel bzw. Schloss vergleicht, welche ideal ineinander passen, ohne dass dazu strukturelle Anpassungen vorgenommen werden müssen (siehe Abb. 32). Experimentelle Untersuchungen haben jedoch gezeigt, dass dies nicht vollkommen der Wahrheit entspricht und das Induced-fit-Modell die Realität eher beschreibt. Abb. 31: Ein Enzym-Substrat-Komplex (bei Cytochrom-P450-Oxidase) Abb. 32: Schema des Schlüssel-Schloss-Prinzips 26 Albertus Magnus Gymnasium 4.1.2.3 Bernhard Seyer / Enzymkinetik Das Induced-fit-Modell Das Induced-fit-Modell oder auch Anpassungsmodell erklärt den Katalyseprozess, indem es davon ausgeht, dass durch die Bindung des Substrats die Konformation, also die tatsächliche und absolute räumliche Anordnung der Atome, im aktiven Zentrum geändert wird, um eine optimale Bindung ausbilden zu können (siehe Abb. 33). Abb. 33: Schema des Induced-fit-Modells 4.2 Kinetik 4.2.1 Einführung in die Kinetik Das Gebiet der Kinetik beschäftigt sich mit den Geschwindigkeiten chemischer Reaktionen und deren Gesetzmäßigkeiten. Dabei ist es grundlegend notwendig die Stöchiometrie der Reaktion und mögliche Nebenreaktionen zu identifizieren. Dies geschieht im ersten Schritt empirisch, wobei sich aus experimentellen Ergebnissen das Verhältnis der Konzentrationen der Produkte und Edukte ermitteln lässt, welche schließlich zu allgemein gültigen Differenzialgleichungen führen, aus denen sich bestimmte Parameter ausdrücken und ableiten lassen. 4.2.1.1 Definition der Reaktionsgeschwindigkeit Allgemein ist die Geschwindigkeit einer Reaktion als Anzahl der umgesetzten Teilchen pro Zeiteinheit definiert. Für eine Reaktion A → B + C gilt also für die Hinreaktion: 27 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 𝜈=− 𝑑𝐴 𝑑𝑡 (1) Das negative Vorzeichen rührt daher, dass die Abnahme der Konzentration von A, also –[A] betrachtet wird. Dies wäre aber äquivalent mit den Ausdrücken: 𝜈= 4.2.1.2 𝑑𝐵 𝑑𝑡 und 𝜈 = 𝑑𝐶 𝑑𝑡 (2), (3) Die Reaktionsordnung In den meisten Fällen stellt sich jedoch heraus, dass die Reaktionsgeschwindigkeiten proportional zu einer experimentell bestimmbaren Potenz der Stoffkonzentrationen sind. Für eine beliebige Reaktion gilt also: 𝜈 = 𝑘[𝐴]𝑎 [𝐵]𝑏 … (4) „Die Potenz, in der die Konzentration eines bestimmten Teilchens im Geschwindigkeitsgesetz einer Reaktion erscheint, ist die Ordnung der Reaktion im Bezug auf dieses Teilchen.“21 Für a = 1 würde also gelten, dass die Reaktion im Bezug auf den Stoff A erster Ordnung ist. Die Addition aller Exponenten gibt die Gesamtordnung einer Reaktion an. Sind zwei Stoffe beteiligt, deren Exponenten beide 1 sind, so ist das Geschwindigkeitsgesetz also gesamt zweiter Ordnung. Bei manchen Reaktionen kann auch ein Geschwindigkeitsgesetz nullter Ordnung auftreten. Diese Reaktionsgeschwindigkeiten sind also unabhängig von jeglicher Teilchenkonzentration. Für sie gilt: 𝜈=𝑘 (5) Das Geschwindigkeitsgesetz kann für jede Reaktion experimentell ermittelt werden, wobei jeder Reaktionsordnung ein allgemein gültiges differentielles Geschwindigkeitsgesetz zugeordnet werden kann. 21 Atkins; Paula, 2006, S. 880 28 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Oft wird für die Ermittlung dieser auf Vereinfachungen zurückgegriffen, wie die des Geschwindigkeitsgetzes pseudo-erster Ordnung. Ist ein Geschwindigkeitsgesetz von den Reaktanten A und B abhängig, so kann beispielsweise B in so großem Überschuss zugesetzt werden, sodass annäherungsweise [B] = [B] 0 während des gesamten Reaktionsverlaufes gilt, dass sich die Konzentration von B nicht merklich ändert. Werden bei einer Reaktion, an der mehrere Stoffe beteiligt sind, alle Stoffe bis auf einen im Überschuss zugesetzt, so kann die Reaktionsordnung in Bezug auf diesen festgestellt werden. Man spricht von der Isoliermethode. 4.2.1.3 Quasistationarität Bei mehrstufigen Reaktionen, das heißt Reaktionen, die über mehrere Zwischenstufen laufen, steigt der mathematische Rechenaufwand von Zwischenstufe zu Zwischenstufe stark an. „Ein aus zahlreichen Schritten bestehender Mechanismus ist analytisch nahezu nicht mehr nicht [sic!] beschreibbar, sodass wir nach anderen Lösungsmethoden suchen müssen.“22 Um eine Möglichkeit zu finden, dennoch zu auswertbaren Gesetzmäßigkeiten zu kommen, bedient man sich der Näherung des quasistationären Zustandes. Bei diesem wird angenommen, dass die Konzentration der Intermediate, also der kurzlebigen Zwischenprodukte (X), die sich im Laufe einer Reaktion bilden, erst in einer Induktionsperiode von null ausgehend ansteigt, sich im weiteren Verlauf jedoch nicht relevant ändert. Damit gilt: 𝑑[𝑋] ≈0 𝑑𝑡 (6) Dies führt in weiterer Folge zu einer grundlegenden Vereinfachung vieler mathematischer Schritte, die für die kinetische Betrachtung komplexerer Reaktionen relevant sind. Hier sind als typische Intermediate Radikale oder Enzym-Substrat-Komplexe zu nennen. 22 Atkins; Paula, 2006, S. 899 29 Albertus Magnus Gymnasium 4.2.2 Bernhard Seyer / Enzymkinetik Die Michaelis-Menten-Kinetik Für enzymkatalysierte Reaktionen gelten bestimmte Bedingungen und Gesetzmäßigkeiten, welche von der kanadischen Medizinerin Maud Menten und dem deutsch-US-amerikanischen Biochemiker und Mediziner Leonor Michaelis 1913 in der nach ihnen benannten MichealisMenten-Theorie formuliert wurden. 4.2.2.1 Um Michaelis-Menten-Mechanismus die Kinetik einer enzymatischen Reaktion zu untersuchen, wird die Anfangsgeschwindigkeit der durch geringe Mengen eines Enzyms beschleunigten Produktbildung in einer Lösung gemessen. Die hohe Effizienz des Enzyms zeigt sich unter anderem dadurch, dass selbst, wenn die Konzentration des Enzyms drei Größenordnungen unter der des Substrats liegt noch eine merkliche Beschleunigung feststellbar ist. „Die grundlegenden Merkmale enzymatischer Reaktionen sind: 1. Bei gegebener Anfangskonzentration [S]0 des Substrats ist die Anfangsgeschwindigkeit der Produktbildung proportional zur Gesamtkonzentration des Enzyms, [E] 0. 2. Bei gegebenem [E] 0 und niedrigen Werten von [S] 0 ist die Geschwindigkeit der Produktbildung proportional zu [S] 0. 3. Bei gegebenen Werten von [E] 0 und hohen Werten von [S] 0 wird die Geschwindigkeit der Produktbildung unabhängig von [S] 0 und erreicht einen Höchstwert, die Maximalgeschwindigkeit νmax.“23 Diese Grundlagen werden im Michaelis-Menten-Mechanismus wiedergegeben. Dabei wird im ersten Schritt reversibel ein Enzym-Substrat-Komplex gebildet. Im zweiten Schritt setzt das Enzym das Produkt frei und ist damit entweder unverändert oder modifiziert (Induced-fitModell) für die Bildung eines Enzym-Substratkomplexes wieder frei. Diesen Schritten werden die Geschwindigkeitskonstanten ka der Bildung des Enzym-Substrat-Komplexes, bzw. ka‘ für die Rückreaktion und kb der Produktbildung zugeordnet: 23 Atkins; Paula, 2006, S. 932 30 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 𝐸+𝑆 ka 𝐸𝑆 𝑘𝑎 , 𝑘𝑎′ (7) 𝑘𝑎′ 𝑘𝑏 𝐸𝑆 → 𝐸 + 𝑃 (8) 𝑘𝑏 Zwischen den beiden oben genannten Schritten liegt noch ein Zwischenschritt, in dem das Produkt an das Enzym gebunden ist, der Enzym-Produkt-Komplex. Dieser ist jedoch für weitere Berechnungen vernachlässigbar. 4.2.2.2 Michaelis-Menten-Gleichung Wenn die in 4.2.1.2 definierte Beziehung nun angewandt wird und dabei die in 4.2.2.1 festgelegten Eigenschaften der enzymatischen Reaktionen berücksichtigt werden, erhält man für die Geschwindigkeit der Produktbildung: 𝜈 = 𝑘𝑏 [𝐸𝑆] (9) Die Bildungsgeschwindigkeit des Enzym-Substrat-Komplexes ergibt sich analog dazu als: 𝜈 = 𝑘𝑎 𝐸 [𝑆] Die Zerfallsgeschwindigkeit des Enzym-Substrat-Komplexes (10) ergibt sich aus der Geschwindigkeit der Rückreaktion und der Produktbildungsgeschwindigkeit und ist demnach: 𝜈 = 𝑘𝑎′ 𝐸𝑆 + 𝑘𝑏 [𝐸𝑆] (11) Werden nun die Bildungs- und die Zerfallsgeschwindigkeit kombiniert und das Prinzip der Quasistationarität herangezogen und der Enzym-Substrat-Komplex als Intermediat betrachtet, so ergibt sich für die Konzentration des Enzymsubstratkomplexes: 𝑑[𝐸𝑆] = 𝑘𝑎 𝐸 𝑆 − 𝑘𝑎′ 𝐸𝑆 − 𝑘𝑏 𝐸𝑆 = 0 𝑑𝑡 (12) 31 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Durch Umformung folgt: 𝐸𝑆 = 𝑘𝑎′ 𝑘𝑎 + 𝑘𝑏 (13) 𝐸 [𝑆] Dabei sind [E] und [S] die Konzentrationen des freien Enzyms und des freien Substrats. Aus der obigen Beziehung lässt sich nun die so genannte Michaelis-Konstante KM definieren: 𝑘𝑎′ + 𝑘𝑏 𝐸 [𝑆] 𝐾𝑀 = = 𝑘𝑎 [𝐸𝑆] (14) Daraus ist ersichtlich, dass die Einheit der Michaelis-Konstante eine molare Konzentration ist. Um nun ein von den Ausgangskonzentrationen des Enzyms [E] 0 und des Substrats [S] 0 abhängiges Geschwindigkeitsgesetz zu finden, müssen zwei Überlegungen angestellt werden: Erstens gilt, dass sich die Ausgangskonzentration des Enzyms aus der Summe der Konzentration des freien Enzyms und der Konzentration des Enzym-Substrat-Komplexes ergibt: [𝐸]0 = 𝐸 + [𝐸𝑆] (15) Zweitens ist, da das Substrat relativ zum Enzym im Überschuss vorliegt, die Konzentration des freien Substrats näherungsweise die Anfangskonzentration des Substrats: 𝑆 ≈ [𝑆]0 (16) Daraus ergibt sich durch Umformen und Einsetzen in die Gleichung der Michaelis-Konstante: 𝐸𝑆 = [𝐸]0 − [𝐸𝑆] [𝑆]0 𝐾𝑚 (17) Löst man nach [ES] auf, erhält man: 𝐸𝑆 = [𝐸]0 [𝑆]0 [𝑆]0 + 𝐾𝑀 (18) 32 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Das Einsetzen in die Gleichung für die Geschwindigkeit der Produktbildung führt zu: 𝜈 = 𝑘𝑏 [𝐸]0 [𝑆]0 [𝑆]0 + 𝐾𝑀 (19) Die Maximalgeschwindigkeit ist dann erreicht, wenn alle Enzyme durch Substrate gesättigt sind, also [ES] = [E] 0 erfüllt ist. Somit gilt: 𝜈𝑚𝑎𝑥 = 𝑘𝑏 [𝐸]0 (20) Einsetzen der Gleichung 20 in die Gleichung 19 liefert die Michaelis-Menten-Gleichung: 𝝂= 4.2.2.3 𝒗𝒎𝒂𝒙 [𝑺]𝟎 𝑲𝒎 + 𝑺𝟎 (21) Lineweaver-Burk-Diagramm Es gibt die Möglichkeit, die Michaelis-Menten-Gleichung in eine Form zu bringen, welche für die Datenauswertung durch lineare Regression geeignet ist: 𝟏 𝟏 𝑲𝑴 = + 𝝂 𝝂𝒎𝒂𝒙 𝝂𝒎𝒂𝒙 𝟏 𝑺𝟎 (22) Trägt man nun 1 / ν gegen 1 / [S] 0 in einem Diagramm auf, erhält man eine Gerade mit der Steigung KM / νmax, dem y-Achsenabschnitt 1 / νmax und dem x-Achsenabschnitt -1 / KM. Des Weiteren lässt sich kb aus den so erhaltenen Daten berechnen. Dieses Diagramm wird als Lineweaver-Burk-Diagramm bezeichnet (siehe Abb. 34). 33 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 34: Linweaver-Burk-Diagramm 4.3 Hemmung Es gibt Stoffe, welche die Fähigkeit aufweisen, ein Enzym zu hemmen (inhibieren) und so seine Wirkung zu schwächen. Diese Hemmung kann reversibel oder irreversibel sein. Handelt es sich um eine irreversible Hemmung, führt dies zu einer permanenten Inhibierung, wobei das Enzym seine Wirkung verliert. Nach der Enzymform (freies Enzym, Enzym-Substrat-Komplex) werden drei Arten von Hemmungen unterschieden: die kompetitive, die nichtkompetitive und die unkompetitive Hemmung (siehe Abb. 35). Auch bei zu hohen Substratkonzentrationen kann es zu einer Hemmung kommen. „Je nachdem, an welche Enzymform (E, ES, EP usw. im katalytischen Kreisprozess) sich der Inhibitor anlagert, resultiert in der Darstellung nach Lineweaver und Burk eine Änderung der Steigung der Geraden oder des Ordinatenabschnittes oder von beiden…“24 24 Wollenberger; Renneberg; Bier; Scheller, 2003, S. 28 34 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 35: Schematische Darstellung der drei Hemmtypen im Vergleich zu einem ungehemmten Enzym-SubstratKomplex 4.3.1 Kompetitive Hemmung Bei der kompetitiven Hemmung konkurriert der Inhibitor mit dem Substrat um die Bindungsstelle im aktiven Zentrum des Moleküls. Es findet also nicht nur eine Reaktion zwischen Enzym und Substrat statt, sondern auch eine Reaktion zwischen Inhibitor und Substrat, welche zur Bildung eines Enzym-Inhibitor-Komplexes führt, der ebenfalls mit dem Substrat reagieren kann (siehe Abb. 36). Abb. 36: Bei einer kompetitiven Hemmung stattfindende Reaktionen In einem Lineweaver-Burk-Diagramm kann man diesen Hemmungstyp daran erkennen, dass vmax unverändert ist, also der Schnittpunkt mit der Ordinate gleich ist, jedoch Km und somit die Steigung deutlich erhöht ist (siehe Abb. 37). 35 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 37: Lineweaver-Burk-Diagramm einer kompetitiven Hemmung Eine kompetitive Hemmung kann durch eine höhere Substratkonzentration ausgeglichen werden, da die Stärke der Hemmung nur vom Verhältnis der Substratkonzentration zur Inhibitorkonzentration abhängig ist. 4.3.2 Nichtkompetitive Hemmung Nichtkompetitiv hemmende Inhibitoren können sowohl an das freie Enzym als auch an den Enzym-Substrat-Komplex binden. Der Inhibitor bindet dabei nicht an das aktive Zentrum, sondern an eine andere Stelle am Enzym und hindert das Substrat dabei sich an das Enzym anzulagern. Bindet der Inhibitor an einen Enzym-Substrat-Komplex, kann es nicht zu einer Umwandlung des Substrates und somit nicht zu einer Freisetzung des Produktes kommen (siehe Abb. 38). Abb. 38: Bei einer nichtkompetitiven Hemmung stattfindende Reaktionen 36 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik In einem Lineweaver-Burk-Diagramm kann eine nichtkompetitive Hemmung daran erkannt werden, dass KM und somit der Schnittpunkt mit der Abszisse unverändert bleibt, jedoch vmax erniedrigt und somit die Steigung erhöht wird (siehe Abb. 39). Abb. 39: Lineweaver-Burk Diagramm einer nichtkompetitiven Hemmung In der Regel kommt es zu der Beeinträchtigung der Wirkung durch eine Deformierung der räumlichen Struktur des aktiven Zentrums. Die relative Hemmwirkung hängt dabei nicht von der Konzentration des Substrates ab. 4.3.3 Unkompetitive Hemmung Im Fall einer unkompetitiven Hemmung bindet der Inhibitor nur an den Enzym-SubstratKomplex. Auch hier erfolgt die Bindung nicht an die aktive Position, kann jedoch nur bei Vorhandensein eines an das Enzym gebundenen Substrates erfolgen, wodurch es zur Ausbildung eines Enzym-Substrat-Inhibitor-Komplexes kommt. Dieser ist nicht in der Lage ein Produkt freizusetzen (siehe Abb. 40). Abb. 40: Bei einer unkompetitiven Hemmung stattfindende Reaktionen 37 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik In einem Lineweaver-Burk-Diagramm wirkt sich die unkompetitive Hemmung in der Form aus, dass KM und vmax um den gleichen Betrag vermindert werden, die Steigung also gleich bleibt, sich jedoch die Schnittpunkte mit Abszisse und Ordinate verändern (siehe Abb.41). Abb. 41: Lineweaver-Burk Diagramm einer unkompetitiven Hemmung 38 Albertus Magnus Gymnasium 5 Bernhard Seyer / Enzymkinetik Analytisch-chemische Grundlagen 5.1 Instrumentelle Analytik Um eine Messung der Konzentrationsänderung eines Substrates abhängig von der Zeit durchzuführen, bedient man sich analytischer Verfahren, welche auf verschiedenen Grundlagen basieren können. Dies kann zum Beispiel durch optische Dedektion erreicht werden, jedoch stehen auch nasschemische Methoden wie Titration, Biosensoren oder elektrochemische Methoden wie Voltammetrie oder Chronoamperometrie zur Verfügung. Dabei hat jedes dieser Verfahren Nachteile und ist nur eingeschränkt nutzbar. Elektrochemische Methoden sind nur bei elektrochemisch aktiven Substanzen wie Reduktions- oder Oxidationsmitteln durchführbar, Methoden, die auf optischen Messungen beruhen nur bei Substanzen, welche durch die Umsetzung durch das Enzym ihre Farbe ändern. Zweitgenannte Methode kommt aufgrund mehrerer Vorteile, wie die Möglichkeit, Echtzeitmessungen durzuführen, dort wo es möglich ist, oft zur Anwendung. Im Hinblick auf den praktischen Teil dieser Arbeit wird im Folgenden eine dieser optischen Methoden, die Photometrie, näher erläutert. 5.1.1 Einführung in die Photometrie In der Photometrie, welche ein Teilbereich der Spektroskopie ist, wird die Absorption (in seltenen Fällen auch die Emission) des sichtbaren Lichts meistens in Lösung gemessen. Daraus kann dann auf die Konzentration des untersuchten Stoffes geschlossen werden. Das Gerät mit dem solche Messungen durchgeführt werden, wird Photometer genannt. 5.1.1.1 Apparativer Aufbau eines Photometers Im Prinzip sind Photometer aus einer Strahlungsquelle, welche meist eine Wolframbandlampe ist, aber in letzter Zeit auch durch Halogenlampen ersetzt wird, einem Probenraum, einem Monochromator, welcher aus dem Licht der Strahlungsquelle Licht mit nur einer Wellenlänge erzeugt, einem Empfänger und einer Register- und Auswerteeinheit aufgebaut. Das System wird zumeist auch durch Blendensysteme erweitert. Die flüssige Probe befindet sich dabei in einer Glas- oder Kunststoffküvette im Probenraum. (siehe Abb. 42) 39 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 42: Schematischer Aufbau eines Photometers 5.1.1.2 Prinzip der Messung Beim Durchgang des monochromatischen Lichtes durch die Probe erfährt dieses aus mehreren Gründen eine Intensitätsschwächung. Zum einen durch Absorption und Streuung der Strahlung durch die Probe, zum anderen aber auch durch Reflexion und Absorption durch das Küvettenmaterial. Streulicht, Reflexion und Küvettenabsorption werden jedoch durch eine Küvette, welche nur das Lösungsmittel, jedoch nicht die Probe enthält, korrigiert. Man spricht von einem Blindwert beziehungsweise dem „Blank“. 5.1.1.3 Transmission und Extinktion Die Absorptionsintensität kann durch die Transmission und die Extinktion (auch Absorption) charakterisiert werden. Dabei ist die Transmission (T) ein Maß für das Verhältnis der durchgelassen Strahlungsintensität (I) zur einfallenden (I0) und kann daher folgendermaßen berechnet werden: 𝑇= 𝐼 𝐼0 (23) Auch eine prozentuelle Auswertung ist möglich: 𝑇[%] = 𝐼 100 𝐼0 (24) 40 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Die Extinktion (E) berechnet sich über den dekadischen Logarithmus des Kehrwertes der Transmission, also folgt: 𝐸 = 𝑙𝑜𝑔 𝐼0 𝐼 (25) Statt der Variablen E ist auch die Variable A für Adsorption gebräuchlich. Wird die Absorption bzw. die Emission in Abhängigkeit von der Wellenlänge gemessen, so ergibt sich ein Absorptions- bzw. Emissionsspektrum, aus dem sich das für Messungen und Berechnungen oft notwendige Absorptions- bzw. Emissionsmaximum ablesen lässt. 5.1.1.4 Lambert-Beer‘sches Gestetz „Untersuchungen von BOUGUER (1698 bis 1758) und LAMBERT (1728 bis 1777) zeigten, daß [sic!] die Extinktion von der Schichtdicke der Küvette abhängt. Die Abhängigkeit der Extinktion von der molaren Konzentration wurde von BEER (1825-1863) gefunden. Die Kombination beider Gesetzmäßigkeiten führt zum Gesetz von BOUGUER, LAMBERT und BEER oder einfach nur LAMBERT-BEERsches Gesetz …“25 Das Lambert-Beer‟sche Gesetz erlaubt es, die Strahlungsschwächung in Abhängigkeit vom zurückgelegten Weg zu berechnen: 𝐼 = 𝐼0 10−𝜀𝑐𝑑 (26) Dabei sind ε der Extinktionskoeffizient, eine empirisch ermittelte Stoffkonstante, c die Konzentration des absorbierenden Stoffes und d die Schichtdicke der Probe. „Die Abnahme dI der Intensität I, die eintritt, wenn die Strahlung die Strecke dl26 zurücklegt, die die Konzentration [J]27 der absorbierenden Teilchen J enthält, muss proportional zur Länge des Weges, der Konzentration der Teilchen und der Intensität des Lichtstrahls sein.“28 25 Otto, 2006, S. 285 Anm.: in meiner Fachbereichsarbeit als d bezeichnet 27 Entspricht c 28 Atkins, Paula; 2006, S. 485 f. 26 41 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Wenn man diesen Proportionalitätsfaktor als κ definiert, ergibt sich also: 𝑑𝐼 = − 𝜅 𝑐 𝐼 𝑑 (27) 𝑑𝐼 =−𝜅𝑐𝑑 𝐼 (28) Durch Umformung erhält man: „Dieser Ausdruck gilt für jede unendlich dünne Schicht, durch die der Strahl hindurchtritt.“29 Für eine endlich dicke Probe mit der Länge d muss also die Summe aller unendlich dünnen Schichten gebildet werden, um die Abnahme der Intensität I0 zu berechnen. Dies führt zu einem bestimmten Integral: 𝐼 𝐼0 𝑑𝐼 =−𝜅 𝐼 𝐼 (29) 𝑐𝑑 𝐼0 Bleibt die Konzentration c überall in der Probe gleich groß, so ergibt sich als Lösung des Integrals: 𝑙𝑛 𝐼 =−𝜅𝑐𝑑 𝐼𝑂 (30) Durch Umwandlung des natürlichen in den dekadischen Logarithmus nach ln(x) = (ln10) log(x) und Ersetzen von κ durch ε ln10 erhält man das Lambert-Beer‟sche-Gesetz: 𝐸= 𝜀𝑐 𝑑 29 (31) Atkins; Paula, 2006, S. 486 42 Albertus Magnus Gymnasium 6 Bernhard Seyer / Enzymkinetik Eigene Experimente Um die Michaelis-Menten-Kinetik nachzuvollziehen und zu messen wurden anhand eines Enzyms einige Experimente und Messungen durchgeführt. Dabei diente Meerrettichperoxidase30 als Enzym, als Substrat Wasserstoffperoxid (H2O2) und als Elektronendonatoren drei verschiedene Substanzen. Diese waren: ABTS31 (siehe Abb. 43), Guajakol32 (siehe Abb. 44) und Para-Phenylendiamin33 (siehe Abb. 45). Abb. 43: ABTS Abb. 45: pPhenylendiamin Abb. 44: Guajakol 6.1 Vorbereitungen 6.1.1 Synthese von Cystin aus Cystein Verwendete Chemikalien L-Cystein (kristallin) Verwendete Geräte Magnetrührer „IKA-WERKE RCTB“ (siehe Abb. 46) KI-Lösung (10%) I2 (kristallin) 30 Auch Krenperoxidase, horseradish peroxidase, HRP (im Weiteren auch meist mit dieser Abkürzung bezeichnet) 31 Nach IUPAC 2,2‘-Azino-bis-(3-ethylbenzothiazolin-6-sulfonsäure)-Diammoniumsalz 32 Auch Guajacol, nach IUPAC 2-Methoxy-phenol 33 Auch p-Phenylendiamin, PPD, nach IUPAC 1,4-Diaminobenzen 43 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 46: Magnetrührer IKA-WERKE RCTB Das bereits im theoretischen Teil erwähnte Cystin ist einer der möglichen Inhibitoren für HRP. Es lässt sich leicht aus Cystein durch Oxidation mit einem milden Oxidationsmittel gewinnen. Ein mögliches Oxidationsmittel hierfür ist Iod. Ein Mol Iod (I2) wird zu zwei Mol Iodid (I-) reduziert und oxidiert dabei zwei Mol Cystein zu einem Mol Cystin (siehe 3.1.2, Abb. 8). Im Gegensatz zu Cystein ist Cystin schlecht in Wasser löslich und lässt sich daher leicht abfiltrieren. L-Cystein wurde gelöst und mit KI-Lösung versetzt. Unter ständigem Rühren mit einem Magnetrührer wurden I2-Kristalle zugegeben. Dabei konnte eine Entfärbung des gelösten Iods beobachtet werden, was auf dessen Reduktion zu Iodid schließen ließ. Es fielen weiße CystinKristalle aus, welche durch eine Glasfritte abfiltriert, mehrmals gewaschen, getrocknet und zur weiteren Verwendung aufbewahrt wurden (siehe Abb. 47). Abb. 47: Das synthetisierte Cystin 44 Albertus Magnus Gymnasium 6.1.2 Bernhard Seyer / Enzymkinetik Herstellung des Britton-Robinson-Puffers Verwendete Chemikalien Verwendete Geräte Phosphorsäure (85%) Magnetrührer „IKA-WERKE RCTB“ Essigsäure (98%) pH-Meter „WTW pH 315i“ (siehe Abb.48) Borsäure (kristallin) NaOH, 2 mol/L (8 g L-1) Abb. 48: pH-Meter „WTW pH 315i“ 2,73 mL Phosphorsäure, 2,40 mL Essigsäure und 2,472 g Borsäure wurden mit Deionat auf einen Liter aufgefüllt und danach mit NaOH auf pH 5,0 eingestellt, wobei der pH-Wert kontinuierlich mit einem pH-Meter kontrolliert wurde (Abb. 49). 45 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 49: Einstellen des Britton-Robinson-Puffers 6.1.3 Herstellung des Phosphat-Puffers Verwendete Chemikalien Verwendete Geräte KH2PO4 (kristallin) Magnetrührer „IKA-WERKE RCTB“ Na2HPO4 (kristallin) pH-Meter „WTW pH 315i“ NaOH, 2 mol/L (8 g L-1) Nach der Henderson-Hasselbalch-Gleichung lässt sich das Konzentrationsverhältnis zwischen Säure und Base in einem konjungierten Puffer bei gewünschtem pH-Wert berechnen: 𝑝𝐻 = 𝑝𝐾𝑆 − 𝑙𝑔 𝑆ä𝑢𝑟𝑒 𝐵𝑎𝑠𝑒 (31) 46 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik In diesem speziellen Fall ist der gewünschte pH-Wert 7, die Säure H2PO4-, die konjugierte Base Na2HPO4 und somit der pKs (von H2PO4-) 7,21. Setzt man nun die Konzentration der Base willkürlich 1 mol/L, so erhält man: 7 = 7,21 − 𝑙𝑔 𝑆ä𝑢𝑟𝑒 (32) Durch Umformung erhält man für die Säurekonzentration 1,6 mol/L. Das Verhältnis von Säure zu Base, also von KH2PO4 zu Na2HPO4, müsste also 1,6 zu 1 (8:5) betragen. Es wurde eine der Stoffmenge äquivalente Masse der beiden Substanzen im oben genannten Verhältnis eingewogen und in Deionat gelöst. Der geringfügig von 7,0 abweichende pH-Wert wurde wie in 6.1.2 beschrieben mit NaOH korrigiert. 6.1.4 Reinigung von Guajakol durch Vakuumdestillation Guajakol ist ein für die Aktivitätsbestimmung von HRP gut geeigneter Elektronendonator. Einer der Nachteile ist jedoch, dass es nach einiger Zeit an der Luft oxidiert und sich dabei von farblos nach braunviolett färbt. Da nun eine durch Oxidation verunreinigte Substanz vorlag, ergab sich die Notwendigkeit der Reinigung eben dieser. Die Methode der Umkristallisation war aufgrund des Schmelzpunktes, welcher bei Normaldruck (760 Torr34) zwischen 28°C und 32°C liegt, jedoch auszuschließen. So war eine Destillation der nächste Gedanke. Aufgrund des Siedepunktes, welcher bei Normaldruck 205°C beträgt, ergab sich die Notwendigkeit einer Vakuumdestillation. Aus Baukastenteilen wurde daher eine Vakuumdestillationsapparatur aufgebaut (siehe Abb. 50). 34 760 Torr = 760 mmHg = 1 atm (Atmosphäre) = 101 325 Pa (Pascal) = 1,01325 bar 47 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik D H C I F E B A J G Abb. 50: Vakuumdestillationsapparatur Diese bestand aus einem Zweihalskolben (siehe Abb. 50, A), in dem sich das verunreinigte Guajakol befand, einer Kapillare35 (siehe Abb. 50, B), einem einfachen Destillationsaufsatz (siehe Abb. 50, C), einem Thermometer (siehe Abb. 50, D), einem Liebig-Kühler (siehe Abb. 50, E), einer Spinne mit drei Kolben (siehe Abb. 50, F) und einer Woulfeschen Flasche36 (siehe Abb. 50, G) mit Manometer (siehe Abb. 50, H). Das Vakuum wurde mittels Wasserstrahlpumpe (siehe Abb. 50, I) erzeugt, geheizt wurde mit einer Heizhaube (siehe Abb. 50, J). Da durch den niedrigen Schmelzpunkt des Destillats jedoch ein Festkörper im Kühler entstehen würde, wurde dieser mit Warmwasser beschickt. Die Destillation wurde bei 88°C und einem Druck von 12 Torr durchgeführt. Nach der Clausius-Claperyon‟schen Gleichung für den Phasenübergang flüssig/gasförmig kann der Dampfdruck in Abhängigkeit von der Temperatur oder der Siedepunkt in Abhängigkeit von dem Druck berechnet werden. Sie lautet: 35 Kapillaren dienen bei Vakuumdestillationen zur Vermeidung von Siedeverzügen Woulfesche Flaschen dienen bei Erzeugen eines Vakuums mittels Wasserstrahlpumpe als Sicherheitsflaschen, um ein Zurückschlagen des Wassers in die Apparatur zu verhindern 36 48 Albertus Magnus Gymnasium 𝑙𝑛 Bernhard Seyer / Enzymkinetik 𝑝1 ∆𝑉 𝐻 1 1 = − 𝑝2 𝑅 𝑇2 𝑇1 (33) Hierbei sind p1 und p2 die Dampfdrücke des Stoffes bei den Temperaturen T1 und T2 (bzw. sind T1 und T2 die Siedepunkte bei den Drücken p1 und p2), ∆𝑉 𝐻 die Verdampfungsenthalpie des Stoffes und R die Gaskonstante (= 8,31447 J K-1 mol-1 = 62,3637 dm3 Torr K-1 mol-1). Im Fall von Guajakol beträgt die Siedetemperatur bei 760 Torr 205°C, wobei in die Gleichung jedoch K37 statt °C eingesetzt werden muss. Die Verdampfungsenthalpie von Guajakol beträgt 45,92 kJ mol-1 (= 45 920 J mol-1). Möchte man nun bei einer Temperatur von 90°C destillieren und setzt die oben genannten Werte in die Gleichung ein, so erhält man den Druck, auf den man herabsenken muss, um dies zu ermöglichen. Dieser beträgt 19,6 Torr. Für 88°C würde er 18,0 Torr betragen. Es wäre theoretisch also ein weniger stark reduzierter Druck notwendig gewesen als in der Praxis, der Siedepunkt liegt also höher (theoretisch müsste der Siedepunkt bei 12 Torr bei rund 79°C liegen). Diese Siedepunkterhöhung ist vermutlich auf die Verunreinigung des Guajakols durch die Oxidationsprodukte, aber auch durch andere Substanzen wie Wasser zurückzuführen. Das Destillat, welches vermutlich durch eine leichte Verunreinigung mit Wasser nicht auskristallisierte, wurde in eine braune Flasche abgefüllt und für die weitere Verwendung aufbewahrt. 6.2 Charakterisierung der Meerrettichperoxidase 6.2.1 6.2.1.1 Thereotische Einführung Organische-chemische Charakteristika des Enzyms Meerrettichperoxidase38, eine Oxireduktase, katalysiert die Oxidation organischer Substanzen unter Verwendung von H2O2 als Substrat. Dabei findet allgemein folgende Reaktion statt: 𝐻2 𝑂2 + 𝐸𝑙𝑒𝑘𝑡𝑟𝑜𝑛𝑒𝑛𝑑𝑜𝑛𝑎𝑡𝑜𝑟 2 𝑒 − + 2 𝐻 + 37 38 HRP 2 𝐻2 𝑂 + 𝐸𝑙𝑒𝑘𝑡𝑟𝑜𝑛𝑒𝑛𝑑𝑜𝑛𝑎𝑡𝑜𝑟 2+ (34) Kelvin, K = °C + 273,15 Auch Krenperoxidase, horseradish peroxidase, HRP 49 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Es sind jedoch auch Donatoren denkbar, welche nur ein oder mehr als zwei Elektronen abgeben können. Dementsprechend müssten die stöchiometrischen Koeffizienten angepasst werden. Als Donatoren kommen organische Moleküle wie Guajakol, ABTS, Luminol, Pyrogallol oder o-Phenylendiamin in Frage. Meerrettichperoxidase ist ein aus 308 Aminosäuren bestehendes Polymer mit einer Masse von ca. 44 000 Dalton. Als Cofaktor besitzt das Protein ein Häm b-Molekül. Neben der Polypeptidkette sind des Weiteren noch zwei Ca2+ und mehrere Kohlenhydrat-Moleküle enthalten (siehe Abb. 51). Abb. 51: Struktur der Meerrettichperoxidase, Ca2+-Atome grün, Fe2+ orange eingezeichnet Eine Hemmung des Moleküls kann sowohl durch einige organische als auch durch mehrere anorganische Verbindungen auftreten. Beispiele für organische Inhibitoren sind Cystin, p-Aminobenzoesäure oder Harnstoff. Beispiele für anorganische Inhibitoren sind Hydroxylamin, Vanadate, aber auch Schwermetallionen wie Pb2+, Cd2+, Cu2+ oder Fe3+ (das Zentralatom des Cofaktors des Enzyms, das Häm b, ist Fe2+). 50 Albertus Magnus Gymnasium 6.2.1.2 Bernhard Seyer / Enzymkinetik Isolierung des Enzyms aus Kren Eine labortechnisch oft angewandte Methode, um Meerrettichperoxidase zu gewinnen ist deren Extraktion aus Kren. Dabei müssen mehrere Wurzeln ca. 2 Tage lang durch ständiges Berieseln mit Wasser gequollen werden. Danach wird die gequollene Wurzel gerieben und über einige Stunden mit Wasser extrahiert. Nach Sättigung mit Ammonsulfat wird nun das Rohextrakt zentrifugiert. Das so erhaltene Produkt wird nun zweimal einer Dialyse unterworfen und danach abermals zentrifugiert. Durch Kationentausch wird schließlich das isolierte Enzym erhalten. Diese Prozedur wurde jedoch nicht praktisch durchgeführt, da die Vorrichtungen für das Zentrifugieren und die Dialyse nicht vorhanden waren und das Verfahren auf Grund seines großen Aufwandes den Rahmen einer Fachbereichsarbeit sprengen würde. 6.2.1.3 Lagerfähigkeit und Reinheitszahl Meerrettichperoxidase ist gegenüber höheren Temperaturen und besonders hohen und niedrigen pH-Werten sensibel. So verliert eine Enzymlösung bereits binnen weniger Tage einen größeren Teil (bis zu 20 %) seiner Aktivität, wenn es bei einem zu hohen pH-Wert gelagert wird. Erhitzt man es über einen kurzen Zeitraum auf höhere Temperatur, so verliert es schon in wenigen Minuten einen Großteil seiner Aktivität, bei hohen Temperaturen (zwischen 90°C und 100°C) ist das Enzym binnen kürzester Zeit vollkommen denaturiert. Am stabilsten ist eine HRP-Lösung im pH-Bereich von 5 bis 9, wobei das pH-Optimum bei 6 bis 6,5 liegt. Die Reinheitszahl gibt Auskunft über den Häm b – Gehalt der Peroxidase, jedoch nicht über seine Aktivität. Die Reinheitszahl ist das Verhältnis der Absorption bei den Wellenlängen 275 nm und 403 nm. Dies rührt daher, dass Häme bei 403 nm und das Trägerprotein bei 275 nm absorbieren. 51 Albertus Magnus Gymnasium 6.2.2 6.2.2.1 Bernhard Seyer / Enzymkinetik Eigene Versuche Überprüfung der Reinheitszahl Verwendete Chemikalien HRP-Lösung, 0,25 g/L Verwendete Geräte Photometer „Thermo Genesys 10-S“ (siehe Abb. 52) Notebook „HP EliteBook 8530w“ Abb. 52: Photometer „Thermo Genesys 10-S“ Um die Reinheitszahl des erworbenen Enzyms (siehe Abb. 53) zu überprüfen, wurde eine photometrische Messung einer Lösung desselben (siehe Abb. 54) bei den Wellenlängen 275 nm und 403 nm durchgeführt. Dabei wurde ein Notebook an das Photometer angeschlossen, über das dieses mittels der Software VISIONlite 2.1 bedient werden konnte. Als Blindwert diente eine mit Deionat gefüllte Küvette. Laut Herstellerangabe sollte diese ca. 3 betragen, der Mittelwert mehrmaliger Messungen ergab eine Reinheitszahl von 3,048. Nach einigen Wochen Lagerung der HRP-Lösung bei 4°C wurde abermals eine Messung der Reinheitszahl durchgeführte, welche zu dem Wert 2,117 führte. Die Abnahme ist vermutlich auf die lange Lagerungszeit zurückzuführen. 52 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 54: Meerrettichperoxidase in Deionat gelöst Abb. 53: Meerrettichperoxidase im ungelöstem Zustand 6.2.2.2 Aktivitätsbestimmung mit Guajakol als Elektronendonator Verwendete Chemikalien Verwendete Geräte HRP-Lösung, 0,25 g/L Magnetrührer „IKA-WERKE RCTB“ Phosphat-Puffer, pH 7,0 pH-Meter „WTW pH 315i“ Guajakol Photometer „Thermo Genesys 10-S“ H2O2, 30% Notebook „HP EliteBook 8530w“ Die Aktivität eines Enzyms ist ein beliebig definierter Wert U (unit). Ein U steht hierbei für die Zunahme der Absorption um 0,01 pro Minute bei einer bestimmen Enzymkonzentration und unter bestimmten Bedingungen. Häufig wird die Aktivität auch als U / ml Enzym angegeben. Für die Bestimmung derselben kommen oft Pyrogallol oder Guajakol als Elektronendonatoren zum Einsatz. 53 Albertus Magnus Gymnasium Zum Zwecke einer Aktivitätsbestimmung wurde eine Bernhard Seyer / Enzymkinetik Lösung von Guajakol mit einer Konzentration von 20 mmol/L in einer Phosphatpufferlösung hergestellt. Die 30%ige H2O2-Lösung wurde mit Deionat auf 12 mmol/L verdünnt. Um solch geringe Mengen qualitativ überführen zu können, mussten Mikropipetten verwendet werden (siehe Abb. 55). Abb. 55: Mikropipetten mit selbstkonstruierter Pipettierhilfe Es wurden 3 Teile Puffer-Lösung mit je einem Teil Guajakol- und WasserstoffperoxidLösung gemischt und bei 470 nm der Blindwert mit dieser Lösung gemessen. Danach wurde diese mit der HRP-Lösung versetzt und photometrisch über einen Zeitraum von 1 Minute die Absorptionsänderung bestimmt. Guajakol färbt sich dabei von farblos zu rostbraun (siehe Abb. 56). Mit dem resultierenden Wert, welcher 0,0057 dA/min betrug, ergab sich demnach eine Aktivität von 0,57 U. Abb. 56: Guajakol vor (links) und nach (rechts) der Umsetzung mit HRP 54 Albertus Magnus Gymnasium 6.2.2.3 Bernhard Seyer / Enzymkinetik Überprüfung der Temperaturempfindlichkeit Verwendete Chemikalien Verwendete Geräte HRP-Lösung, 0,25 g/L Photometer „Thermo Genesys 10-S“ Britton-Robinson-Puffer, 0,04 mol/L, pH 7,0 Notebook „HP EliteBook 8530w“ ABTS H2O2, 10 mmol/L Um die Tempertaturempfindlichkeit der Peroxidase zeigen zu können, wurde ein Teil einer Enzymlösung über einen Zeitraum von 15 min konstant auf 50°C gehalten. Mit einem anderen Teil derselben Lösung wurde eine Aktivitätsbestimmung im Bezug auf ABTS durchgeführt. Dabei wurden 27,4 mg ABTS (siehe Abb. 57) in 10 mL Britton-RobinsonPuffer gelöst, sodass sich eine Lösung mit der Konzentration 5 mmol/L ergab (siehe Abb. 58). Abb. 57: ABTS, ungelöst Abb. 58: ABTS, gelöst in Britton-Robinson-Puffer 2,9 mL von dieser wurden mit 50 μL HRP-Lösung, welche nicht hohen Temperaturen ausgesetzt war, und 100 μL H2O2-Lösung versetzt und nach einiger Inkubationszeit ein Spektrum im Bereich von 400 nm bis 800 nm aufgenommen (siehe Abb. 59), woraus sich ein Absorptionsmaximum bei 420 nm ablesen ließ. Als Blindwert diente eine Küvette mit reinem Britton-Robinson-Puffer. 55 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 59: Spektrum des ABTS Weitere 2,9 mL wurden ebenfalls mit 50 μL HRP-Lösung versetzt. Von dieser wurde der Blindwert gemessen. Danach wurden 100 μL H2O2-Lösung zugesetzt und eine photometrische Bestimmung bei 420 nm durchgeführt. Es ergab sich eine Aktivität von 11,91 U. Mit der den höheren Temperaturen ausgesetzten Lösung wurde ebenfalls eine Bestimmung der Aktivität durchgeführt. Hier ergab sich eine Aktivität von 1,89 U. Damit wurde ein Aktivitätsverlust von 84,13 % bestimmt. Auch eine Erhitzung auf knapp 100°C wurde durchgeführt. Eine Messung ergab dabei einen 100 %igen Aktivitätsverlust. Es wurde also nachgewiesen, dass Meerrettichperoxidase gegen hohe Tempertaturen sehr empfindlich ist und bereits nach wenigen Minuten bei 50°C zum Großteil und bei 100°C vollkommen denaturiert ist. 56 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 6.3 Messung der Michaelis-Menten-Kinetik 6.3.1 Messung mit ABTS als Elektronendonator Verwendete Reagenzien: Verwendete Chemikalien Verwendete Geräte HRP-Lösung, 0,25 g/L Photometer „Thermo Genesys 10-S“ Britton-Robinson-Puffer, 0,04 mol/L, pH 7,0 Notebook „HP EliteBook 8530w“ ABTS H2O2, 10 mmol/L ABTS reagiert unter Einwirkung von HRP und H2O2 unter Abgabe eines Elektrons zu einem grünen Farbstoff, wobei pro Mol H2O2 2 Mol ABTS umgesetzt werden. Es findet also folgende Reaktion statt: Abb. 60: Reaktion von ABTS mit HRP und H2O2 Die ABTS-Lösung wurde mit Britton-Robinson-Puffer hergestellt. Aus dieser Stammlösung mit der Konzentration 10 mmol/Lwurden durch Verdünnung mit Britton-Robinson-Puffer Lösungen verschieden hoher Konzentrationen (5 mmol/L, 1 mmol/L, 0,5 mmol/L, 0,1 mmol/L, 0,05 mmol/L) gewonnen (siehe Abb. 61). Abb. 61: ABTS in verschieden hoher Konzentration. V.l.n.r.: 10 mmol/L, 5 mmol/L, 1 mmol/L, 0,5 mmol/L, 0,1 mmol/L, 0,05 mmol/L, reiner Britton-Robinson-Puffer. 57 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Danach wurden Stammlösungen und die verdünnten Lösungen mit 100 μL H2O2- und 50 μL HRP-Lösung versetzt und die Absorption eine Minute lang jede Sekunde gemessen (siehe Tab. 2). Dabei diente immer die mit HRP-Lösung versetzte Probe als Blindwert. [ABTS] (mmol/L) 10 5 1 0,5 0,1 0,05 dA/min 0,6060 0,3485 0,0812 0,0415 0,0083 0,0042 Tab. 2: Messwerte der Umsetzung von ABTS Daraus konnte mittels Lambert-Beer‟schem Gesetz dank bekanntem Extinktionskoeffizienten (ε420 = 43,2∙103 [L mol-1 cm-1]) die Geschwindigkeit in d[ABTS]/min und das LineweaverBurk-Diagramm (siehe Abb. 62) errechnet und durch die doppeltreziproke Darstellung die Michaelis-Menten-Konstante und die Maximalgeschwindigkeit berechnet werden. Für KM ergab sich 24,47 mmol dm-3 und für vmax 0,0478 mmol dm-3 min-1. 12 10 1 / v (1 / mmol dm-3 min-1) 8 -1 6 ABTS 4 Regressionsgerade 2 0 -0,5 0 0,5 1 1,5 2 2,5 -2 -4 1 / [ABTS] (1 / mmol1 dm-3) Abb. 62: Lineweaver-Burk-Diagramm der Umsetzung von ABTS 58 Albertus Magnus Gymnasium 6.3.2 Bernhard Seyer / Enzymkinetik Bestimmung der Hemmung durch Cystin und Cadmium auf ABTS Verwendete Chemikalien Verwendete Geräte HRP-Lösung, 0,25 g/L Photometer „Thermo Genesys 10-S“ Britton-Robinson-Puffer, 0,04 mol/L, pH 7,0 Notebook „HP EliteBook 8530w“ ABTS Cystin Cd(NO3)2 H2O2, 10 mmol/L Zur Bestimmung der Hemmung von HRP wurden zwei Lösungen der Inhibitoren Cystin und Cadmium (Cd2+) hergestellt. Als Problem ergab sich dabei die schlechte Wasserlöslichkeit des Cystins. Es wurde daher eine übersättigte Lösung hergestellt, von dieser ein festkörperfreier Teil entnommen und mit den in 6.3.1 genannten Substratlösungen gemischt. Nach der in 6.3.1 erwähnten Methode wurde die Reaktionsgeschwindigkeit mit dem Inhibitor bestimmt. Die Prozedur wurde mit einer Cd2+-haltigen Lösung wiederholt. Es ergaben sich dadurch verringerte Geschwindigkeiten (siehe Tab. 3). ABTS ungehemmt Hemmung durch Cadmium Hemmung durch Cystin [ABTS] (mmol/L) dA/min [ABTS] (mmol/L) dA/min [ABTS] (mmol/L) dA/min 10 0,6060 10 0,1857 10 0,1801 5 0,3485 5 0,1153 5 0,1513 1 0,0812 1 0,0265 1 0,0619 0,5 0,0415 0,5 0,0134 0,5 0,0357 0,1 0,0083 0,1 0,0028 0,1 0,0081 0,05 0,0042 0,05 0,0014 0,05 0,0042 Tab. 3: Messwerte der Umsetzung von ABTS ohne und mit Inhibitoren 59 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Die Daten wurden umgerechnet und in ein Lineweaver-Burk-Diagramm übertragen, wodurch festgestellt wurde, dass es sich bei der Hemmung der HRP bei Cystin um eine unkompetitive und bei Cd2+ um eine nichtkompetitive Hemmung handelt (siehe Abb. 63). 35 30 1 / v (1 / mmol dm-3 min-1) 25 -1 y = 15,592x + 0,7018 ABTS ungehemmt 20 15 Hemmung durch Cadmium Hemmung durch Cystin y = 5,1134x + 1,8623 10 y = 5,1122x + 0,2089 5 Regressionsgerade 0 -0,5 -5 0 0,5 -10 1 1 / [ABTS] 1,5 2 2,5 (1 / mmol1 dm-3) Abb. 63: Lineweaver-Burk-Diagramm der Umsetzung von ABTS, zur besseren Erkennbarkeit wurde der Bereich um die Nullstelle vergrößert Sowohl die Messung ohne Inhibitoren als auch die Messungen mit den beiden Inhibitoren lieferten sehr eindeutige und gut verwertbare Ergebnisse. Bei beiden Inhibitoren ließ sich die Art der Hemmung problemlos feststellen, wobei die sehr starke Aktivitätsverringerung durch Cadmium auffällt. Es scheint einen Teil der Proteinstruktur zu zerstören. Dabei dürfte es mit Seitenketten von Aminosäuren so interagieren, dass sich eine neue Faltung ergibt, wodurch das Enzym einen Teil seiner Wirkung einbüßt. Cystin hingegen scheint nur in der Lage zu sein, mit dem Enzym-Substrat-Komplex in Wechselwirkung zu treten, wobei nur ein geringer Aktivitätsverlust festzustellen ist. 60 Albertus Magnus Gymnasium 6.3.3 Bernhard Seyer / Enzymkinetik Messung mit p-Phenylendiamin als Elektronendonator Verwendete Chemikalien Verwendete Geräte HRP-Lösung, 0,25 g/L Photometer „Thermo Genesys 10-S“ p-Phenylendiamin, kristallin Notebook „HP EliteBook 8530w“ H2O2, 10 mmol/L p-Phenylendiamin, nach IUPAC 1,4-Diaminobenzen, ist ein farbloser, giftiger Festkörper. Es ist an der Luft leicht oxidabel und nimmt dabei eine bräunliche Farbe an (siehe Abb 64). Abb. 64: Oxidiertes, kristallines p-Phenylendiamin Auch hier wurde nach dem gleichen Verfahren wie in 4.2.1 vorgegangen. Um die Adsorptionsveränderung und somit die Geschwindigkeit besser messen zu können, mussten die Substratkonzentrationen jedoch erhöht werden (Tab. 4). [PPD] (mmol/L) 200 80 40 20 10 5 2 1 dA / min 0,0142 0,0110 0,0153 0,0549 0,0459 0,0365 0,0227 0,0137 Tab. 4: Messwerte der Umsetzung von p-Phenylendiamin 61 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Es wurde ein Lineweaver-Burk-Diagramm gezeichnet (siehe Abb. 65), wobei auffiel dass bei zu hohen Konzentrationen eine Hemmung durch Substratüberschuss auftrat. Aus den verwertbaren Ergebnissen, also die, bei denen die Substratkonzentration unter 20 mmol/L betrug, wurde abermals ein Lineweaver-Burk-Diagramm (Abb. 66) aufgestellt. Die Michaelis-Menten-Konstante wurde mit 3,65 mmol dm-3 berechnet. Die errechnete Maximalgeschwindigkeit beträgt 0,0636 dA min-1. 100 90 80 1 / v (1 / dA min-1) 70 60 50 40 30 20 10 0 0 0,02 0,04 0,06 0,08 1 / [PPD] (1 / mmol1 dm-3) 0,1 0,12 Abb. 65: Lineweaver-Burk-Diagramm der Umsetzung von p-Phenylendiamin, die Hemmung durch Substratüberschuss ist gut erkennbar 80 70 60 1 / v (1 / dA min-1) 50 40 30 20 10 0 -0,6 -0,4 -0,2 -10 0 0,2 0,4 0,6 0,8 1 1,2 -20 1 / [PPD] (1 / mmol1 dm-3) Abb. 66: Lineweaver-Burk-Diagramm der Umsetzung von p-Phenylendiamin 62 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Die Messwerte waren trotz der sehr leichten Oxidierbarkeit von p-Phenylendiamin überraschend genau und sehr zufriedenstellend. Es ist im Gegensatz zu ABTS kein Extinktionskoeffizient bekannt gewesen, wodurch nur die Geschwindigkeit in Bezug auf die Absorptionsänderung und nicht auf die Stoffmengenänderung gemessen und berechnet werden konnte. Ein Problem, das bei der Untersuchung mit p-Phenylendiamin als Elektronendonator jedoch auftrat, war, dass keine Information aus der Literatur vorhanden und somit der Mechanismus und die Stöchiometrie der Reaktion gänzlich unbekannt waren. Es konnte einzig auf eine Arbeitsanleitung, welche das o-Isomere des Phenylendiamins verwendet. Im Falle von diesen reagieren jedoch zwei Moleküle zu einem (siehe Abb. 67), was bei para-Stellung nicht möglich ist, wodurch sich ein vollkommen anderer Reaktionsmechanismus ergibt. Abb. 67: Reaktion des o-Phenylendiamins mit HRP und H2O2 Durch den Einsatz von Pb2+ als Inhibitor konnte ein Intermediat wahrscheinlich gemacht werden und somit auf einen zweistufigen Reaktionsmechanismus geschlossen werden (siehe Abb. 68). Abb. 68: Reaktion von p-Phenylendiamin. Es ist eine grünliche Zwischenstufe zu erkennen, welche allmählich zum bräunlichen Endprodukt übergeht (das Endprodukt ist dunkelbraun) Daher gehe ich davon aus, dass im ersten Schritt dem Elektronenpaar eines Stickstoffatoms ein Elektron entzogen wird und dieses eine H+-Ion abgibt. Dieses Radikal ist bedingt stabil und reagiert sehr schnell weiter. Im zweiten Schritt wird das andere Stickstoffatom auf 63 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik gleiche Weise oxidiert und gibt ebenfalls ein H+-Ion ab. Durch die neue Elektronenverteilung wird nun die Ausbildung des stabilen p-Chinondiimins ermöglicht (siehe Abb. 69). Abb. 69: Vermutete Reaktion von p-Phenylendiamin mit HRP und H2O2 64 Albertus Magnus Gymnasium 7 Bernhard Seyer / Enzymkinetik Zusammenfassung In meiner Fachbereichsarbeit, in welcher die Kinetik enzymkatalysierter Reaktionen, deren Messung und mathematische Auswertung im Mittelpunkt steht, beschäftigte ich mich zuerst mit der Betrachtung der Aminosäuren aus dem Blickwinkel der organischen Chemie. Von den Eigenschaften der Aminosäuren beginnend wurde ein roter Faden bis hin zu komplexen Proteinen verfolgt. In den weiteren Kapiteln fand eine Auseinandersetzung mit den mathematischen, physikalischen und schließlich den analytischen Grundlagen statt. Dies beinhaltete die Herleitung der Michaelis-Menten-Kinetik und eine kurze Einführung in die Photometrie. Nachdem die theoretischen Grundlagen gelegt worden waren, widmete sich das abschließende Kapitel der praktischen Messung der Enzymkinetik und einer mathematischen Auswertung der Messergebnisse. Dabei kam es zu sehr zufriedenstellenden Ergebnissen, wobei die Messung mit ABTS als Elektronendonator den Hauptanteil ausmachte, jedoch auch p-Phenylendiamin als Substrat im Mittelpunkt des Interesses stand. Ich entdeckte zufällig, dass sich diese Substanz auch mit HRP umsetzen lässt, konnte mich jedoch nicht auf Fakten aus der Literatur stützen. Daher musste ich den Reaktionsmechanismus und alle mit diesem Substrat in Verbindung stehenden Ergebnisse mehr durch Spekulation als durch belegbare Fakten erklären. 8 Summary At the beginning of my “Fachbereichsarbeit” that centres on the kinetics of enzyme-catalyzed reactions, their measurement and mathematical analysis are taking center stage, I dealt with the examination of amino acids from the perspective of organic chemistry. Starting with the properties of amino acids a route up to complex proteins has been followed. In the following chapters there was a study of mathematical, physical and ultimately analytical basic principles. This included the derivation of the Michaelis-Menten kinetics and a brief introduction to photometry. Having laid the theoretical foundations I devoted the final chapter to practical measurements of enzyme kinetics and its mathematical analysis. This led to very satisfactory results, whereby measurement with ABTS as electron donor had the major share. However, p-Phenylenediamine was also in the center of interest. Only by chance I found out that this substance can react with HRP too, but I could not verify that in the available literature. Therefore, I had to explain the reaction mechanism and all results related to this substrate more on the basis of speculation than of verifiable facts. 65 Albertus Magnus Gymnasium 9 Bernhard Seyer / Enzymkinetik Anhang 9.1 Verwendete Literatutr Atkins, Peter W.; Paula, Julio de: Physikalische Chemie. Vierte, vollständig überarbeitete Auflage. – Weinheim: WILEY-VCH, 2006. Becker, Heinz G. O. et al.: Organikum. Organisch-chemisches Grundpraktikum. 22., vollständig überarbeitete und aktualisierte Auflage. – Weinheim: WILEY-VCH, 2005. Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert: Biochemie. 6. Auflage. – Heidelberg: Spektrum Akademischer Verlag, 2007. Beyer, Hans; Walter, Wolfgang; Francke, Wittko: Lehrbuch der Organischen Chemie. 24. Auflage. – Stuttgart: S. Hirzel Verlag, 2004. D‟Ans, Jean; Lax, Ellen: Taschenbuch für Chemiker und Physiker. 3. Auflage. Bd. 2.– Berlin, Göttingen, Heidelberg: Springer – Verlag, 1964 Jeromin, Günter E.; Bertau, Martin: Bioorganikum. Praktikum der Bioanalyse. – Weinheim: WILEY-VCH, 2005. Otto, Mathias: Analytische Chemie. Dritte, vollständig überarbeitete und erweiterte Auflage. – Weinheim: WILEY-VCH, 2006. Vollhardt, K. Peter C.; Schore, Neil E.; Butenschön, Holger (Übersetzungs-Herausgeber): Organische Chemie. Vierte Auflage. – Weinheim: WILEY-VCH, 2005. Wollenberger, Ulla; Renneberg, Reinhard; Bier, Frank F.; Scheller, Frieder W.: Analytische Biochemie. Eine praktische Einführung in das Messen mit Biomolekülen. – Weinheim: WILEY-VCH, 2003. 66 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 9.2 Verwendete Zeitschriftenbeiträge Hamilton, T. M.; Dobie-Galuska, A. A.; Wietstock, S. M.: The o-PhenylenediamineHorseradish Peroxidase System: Enzyme Kinetics in the General Chemistry Laboratory. In: Journal of Chemical Education, Vol. 76, No. 5, 1999, S. 642 ff. - Bellmawr, NJ: Division of Chemical Education of the American Chemical Society. 9.3 Verwendete Internetseiten Enzyme Explorer: Peroxidase Enzymes. http://www.sigmaaldrich.com/lifescience/metabolomics/enzyme-explorer/analytical-enzymes/peroxidase-enzymes.html, 20. 11. 2008. Giffhorn: Biologie Grundpraktikum für Bioinformatiker. http://www.unisaarland.de/fak8/heinzle/de/teaching/GrundPraktikum_Bioinf/V5_Giffhorn_Kinetik_2007.pdf, 16. 10. 2008. Information on EC 1.11.1.7 – peroxidase: http://www.brendaenzymes.org/php/result_flat.php4?ecno=1.11.1.7, 20. 1. 2009. Inherent properties, identifiers and references (für Guajakol): http://www.chemspider.com/Chemical-Structure.447.html, 1. 2. 2009. Max Ferdinand Perutz: http://de.wikipedia.org/wiki/Max_Ferdinand_Perutz, 28. 11. 2008. Meerrettichperoxidase: http://de.wikipedia.org/wiki/Meerrettichperoxidase, 10. 11. 2008. Morawski, Birgit; Lin, Zahnglin; Cirino, Pat; Joo, Hyun; Bandara, Geethani; Arnold, Frances H.: Functional expression of horseradish peroxidase in Saccharomyces cerevisiae and Pichia pastoris, http://peds.oxfordjournals.org/cgi/content/full/13/5/377, 1. 2. 2009- Peroxidase Substrates. http://www.sigmaaldrich.com/life-science/biochemicals/biochemicalproducts.html?TablePage=15845077, 20. 11. 2008. 67 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 9.4 E-Mails Kosma, Paul: Kren-Peroxidase. [email protected], 11. 12. 2008. 9.5 Abbildungsverzeichnis Titelbild: Struktur der Meerrettichperoxidase, http://www.tifr.res.in/~shyamal/research.html; Adsorptionsspektrum, Screenshot aus VISIONlite; Geschwindigkeitsmessung; Screenshot aus VISIONlite; Lineweaver-Burk-Diagramm, erstellt mit Microsoft Excel 2007; MichaelisMenten-Gleichung Abb. 1: http://www.neb.com/nebecomm/tech_reference/general_data/amino_acid_structures.asp Abb. 2: Berg; Tymoczko; Stryer, 2007, S. 29 Abb. 3: Berg; Tymoczko; Stryer, 2007, S.29 Abb. 4: Berg; Tymoczko; Stryer, 2007, S.30 Abb. 5: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 6: Berg; Tymoczko; Stryer, 2007, S. 38 Abb. 7: Bernhard Seyer, Zeichnung nach 1TIT. ppd (http://www.rcsb.org), erstellt mit jmol, 2009 Abb. 8: Berg; Tymoczko; Stryer, 2007, S. 38 Abb. 9: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 10: Stryer, Biochemie S. 41 Abb. 11: Bernhard Seyer, erstellt mit Symyx Draw 3.1 und jmol, 2009 Abb. 12: Berg; Tymoczko; Stryer, 2007, S. 43 Abb. 13: Berg; Tymoczko; Stryer, 2007, S. 50 Abb. 14: Berg; Tymoczko; Stryer, 2007, S. 45 Abb. 15: Berg; Tymoczko; Stryer, 2007, S. 44 Abb. 16: Berg; Tymoczko; Stryer, 2007, S. 45 Abb. 17: Berg; Tymoczko; Stryer, 2007, S.47 Abb. 18: Berg; Tymoczko; Stryer, 2007, S. 46 Abb. 19: Berg; Tymoczko; Stryer, 2007, S. 46 Abb. 20: Berg; Tymoczko; Stryer, 2007, S. 46 68 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 21: Berg; Tymoczko; Stryer, 2007, S. 47 Abb. 22: Berg; Tymoczko; Stryer, 2007, S. 47 Abb. 23: Berg; Tymoczko; Stryer, 2007, S. 48 Abb. 24: Berg; Tymoczko; Stryer, 2007, S. 51 Abb. 25: Berg; Tymoczko; Stryer, 2007, S. 52 Abb. 26: Berg; Tymoczko; Stryer, 2007, S. 61 Abb. 27: Berg; Tymoczko; Stryer, 2007, S. 53 Abb. 28: Berg; Tymoczko; Stryer, 2007, S. 53 Abb. 29: Berg; Tymoczko; Stryer, 2007, S. 236 Abb. 30: Berg; Tymoczko; Stryer, 2007, S. 237 Abb. 31: Berg; Tymoczko; Stryer, 2007, S. 237 Abb. 32: Berg; Tymoczko; Stryer, 2007, S. 239 Abb. 33: Berg; Tymoczko; Stryer, 2007, S. 240 Abb. 34: Berg; Tymoczko; Stryer, 2007, S. 245 Abb. 35: Berg; Tymoczko; Stryer, 2007, S. 251 Abb. 36: Berg; Tymoczko; Stryer, 2007, S. 253 Abb. 37: Berg; Tymoczko; Stryer, 2007, S. 254 Abb. 38: Berg; Tymoczko; Stryer, 2007, S. 254 Abb. 39: Berg; Tymoczko; Stryer, 2007, S. 255 Abb. 40: Berg; Tymoczko; Stryer, 2007, S. 253 Abb. 41: Berg; Tymoczko; Stryer, 2007, S. 255 Abb. 42: http://de.wikipedia.org/wiki/Fotometrie Abb. 43: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 44: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 45: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 46: Bernhard Seyer, Wien, Jänner 2008 Abb. 47: Bernhard Seyer, Wien, Jänner 2008 Abb. 48: Bernhard Seyer, Wien, Jänner 2008 Abb. 49: Bernhard Seyer, Wien, Jänner 2008 69 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik Abb. 50: Bernhard Seyer, Wien, Jänner 2008 Abb. 51: Bernhard Seyer, Zeichnung nach 6ATJ. ppd (http://www.rcsb.org), erstellt mit jmol, 2009 Abb. 52: Bernhard Seyer, Wien, Jänner 2008 Abb. 53: Bernhard Seyer, Wien, Jänner 2008 Abb. 54: Bernhard Seyer, Wien, Jänner 2008 Abb. 55: Bernhard Seyer, Wien, Jänner 2008 Abb. 56: Bernhard Seyer, Wien, Jänner 2008 Abb. 57: Bernhard Seyer, Wien, Jänner 2008 Abb. 58: Bernhard Seyer, Wien, Jänner 2008 Abb. 59: Screenshot aus VISIONlite, 2008 Abb. 60: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 61: Bernhard Seyer, Wien, Jänner 2008 Abb. 62: Bernhard Seyer, erstellt mit Microsoft Excel 2007, 2008 Abb. 64: Bernhard Seyer, Wien, Jänner 2008 Abb. 65: Bernhard Seyer, erstellt mit Microsoft Excel 2007, 2008 Abb. 66: Bernhard Seyer, erstellt mit Microsoft Excel 2007, 2008 Abb. 67: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 Abb. 68: Bernhard Seyer, Wien, Jänner 2008 Abb. 69: Bernhard Seyer, erstellt mit Symyx Draw 3.1, 2008 9.6 Protokoll 3. Juli 2008 1. Besprechung: Möglichkeiten für Themen, Einigung auf Enzymkinetik 10. Juli 2008 2. Besprechung: Literaturinformationen 12. Juli 2008 Besorgung von fehlender Literatur 26. August 2008 3. Besprechung: Grobdisposition der Arbeit (Titel, Enzyme als Stoffe, Enzyme als Katalysatoren, physikalische Chemie) 70 Albertus Magnus Gymnasium 29. August 2008 11. September 2008 12. September 2008 Bernhard Seyer / Enzymkinetik 4. Besprechung: Arbeit und Überlegungen in Richtung Enzymkinetik und Photometrie 5. Besprechung: Festlegung und Einreichung des Titels und der Grobdisposition Erstellung eines Inhaltsverzeichnisses 6. Besprechung: Konkrete Besprechung des Katalyse-Experimentes, 25. September 2008 Rechercheauftrag für eine Arbeitsvorschrift für die Extraktion von Peroxidase aus Kren, Rohfassung für das 1. Kapitel (Aminosäuren, Proteine), Vorlage des Inhaltsverzeichnisses 6. Oktober 2008 18. Oktober 2008 23. Oktober 2008 6. November 2008 Kontaktaufnahme zu Universität Wien und Universität für Bodenkultur bezüglich oben genannter Arbeitsvorschrift 7. Besprechung: Besprechung des 1. schriftlichen Teils, Hinweis auf Copyright-Problematik, Weiterarbeit in Richtung Proteinstruktur 8. Besprechung: Erste Versuche mit Photometer, Kombination Photometer-Laptop 9. Besprechung: Installation der Software für Photometer, Aufnahme der Spektren von Farbstoffen 8. November 2008 10. Besprechung: Herstellung von Lösungen 17. November 2008 11. Besprechung: Erste Versuche zur Michaelis-Menten Kinetik 12. Besprechung: Messung der Kinetik von HRP mit ABTS als 20. November 2008 Elektronendonor, Besprechung der Arbeitsanleitung von o-Phenylendiamin, Auftrag zur Recherche von Reaktion mit p-Phenylendiamin 20. - 25. November 2008 27. November 2008 29. November 2008 30. November 2008 4. Dezember 2008 Eingehende Recherchen zu PPD, keine Literatur bezüglich Reaktion mit HRP gefunden 13. Besprechung: Weitere Messungen, Mitteilung über Recherchenergebnisse, Recherche bezüglich Inhibitoren 14. Besprechung: Abschluss der Messungen mit ABTS als Elektronendonor ohne Inhibitoren Herstellung von Cystin aus Cystein 15. Bersprechung: Erste Versuche mit PPD, Hinweis auf zweistufigen Prozess 71 Albertus Magnus Gymnasium 6. Dezember 2008 11. Dezember 2008 12. Dezember 2008 Bernhard Seyer / Enzymkinetik 16. Besprechung: Messungen der Michaelis-Menten Kinetik mit PPD, Vorlage weiterer Teile der theoretischen Kapitel Erhalt der Arbeitsvorschrift zu Extraktion von Peroxidase 17. Besprechung: Besprechung der Arbeitsvorschrift, Feststellung der Undurchführbarkeit, theoretische Behandlung 16. Dezember 2008 Kontaktaufnahme mit Spektrum-Verlag bezüglich Copyright 30. Dezember 2008 Messungen mit PPD und ABTs (Hemmung durch Cystin) 10. Jänner 2009 14. + 15. Jänner 2009 Charakterisierung des Enzyms, Vorlage des physikalisch-chemischen und analytisch-chemischen Kapitels Vakuumdestillation von Guajakol Erneute Kontaktaufnahme mit Springer-Verlag bezüglich Rechte, 15. Jänner 2009 Weiterleitung zu W.H. Freeman & Co. Verlag, Kontaktaufnahme mit W.H. Freeman & Co, Erlaubnis zur Verwendung 17. Jänner 2009 23. Jänner 2009 29. Jänner 2009 Messung der Aktivität von HRP mit Guajakol, vorlage des praktischen Teils Messungen mit ABTS und Cadmium als Inhibitor 18. Besprechung: Besprechung des theoretischen und des praktischen Teils 9. Februar 2009 19. Besprechung: Besprechung letzter organisatorischer Punkte 13. Februar 2009 Abgabe der Fachbereichsarbeit 72 Albertus Magnus Gymnasium Bernhard Seyer / Enzymkinetik 9.7 Erklärung Ich erkläre, dass ich diese Fachbereichsarbeit selbst verfasst und ausschließlich die angegebene Literatur verwendet habe Ort und Datum Unterschrift des Schülers 73