petrinetze und anwendungsmöglichkeiten für
Werbung

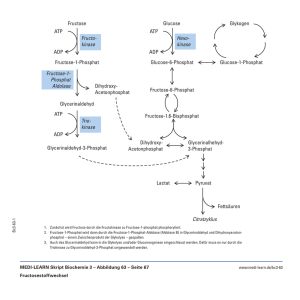
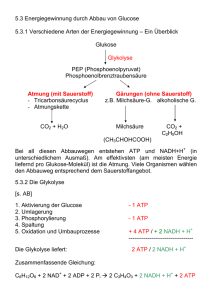
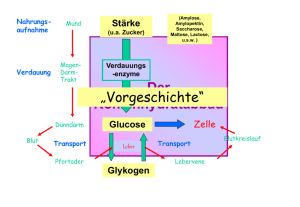
Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke BTU COTTBUS INSTITUT FÜR SOFTWARE ENGINEERING & PETRI NETS INFORMATIK PETRINETZE UND ANWENDUNGSMÖGLICHKEITEN FÜR METABOLISCHE NETZWERKE MONIKA HEINER August ‘98 Problem software Petrinetz Petri net properties properties [email protected] http://www.informatik.tu-cottbus.de metabolische Netzwerke [email protected] 1 / 25 [email protected] 2 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke August ‘98 TOOL OVERVIEW press informal specification hierarchical Petri Net Editor with output filters PED functional testing protocols PEDVisor (distributed) animation tool elevating rotary table (rapid prototyping) functional requirements safety requirements qualitative analysis protocols INA PEP PROD SMV qualitative Petri net analyzers performance requirements feed belt (belt 1) travelling crane robot deposit belt (belt 2) Production cell: arm 1 arm 2 hierarchy browser quantitative analysis protocols INA TimeNet (non-stochastic) (stochastic) quantitative Petri net analyzers execution protocols FUNLite motion lib execution tool [email protected] 3 / 25 [email protected] 4 / 25 August ‘98 NADH 1,3-BPG Glucose6-phosphat ADP Fructose6-phosphat 16 14 13 Ribose5-phosphat 3PG Fructose1,6-bisphosphat 2 NADP+ 17 DHAP 12 ATP 8 F6P E4P Glycerinaldehyd3-phosphat CO2 Ribose5-phosphat 2. Möglichkeit 1. Möglichkeit 18 ADP Ribulose5-phosphat PEP Ribulose5-phosphat CO2 Reddy, V. N.; Liebman, M. N.; Mavrovouniotis, M. L.: Qualitative Analysis of Biochemical Reaction Systems; Computers in Biology and Medicine 26(96), 9-24. Lac NAD + ADP ATP 20 NADH Pyr 4 GSH 2 NADPH Glucose6-phosphat Fructose6-phosphat Ribose5-phosphat Fructose1,6-bisphosphat 2 NADP+ 2 NADPH Glucose6-phosphat Dihydroxyacetonphosphat Glycerinaldehyd3-phosphat Ribulose5-phosphat CO2 Ribose5-phosphat Fructose6-phosphat 3. Möglichkeit Fructose1,6-bisphosphat Dihydroxyacetonphosphat Glycerinaldehyd3-phosphat 2 ATP [Reddy 96] 19 ATP 10 G6P Gluc 2 NADP+ 2 1 9 3 2 NADPH 2 GSSG 2 NADP+ ADP R5P ATP F6P 11 GAP 2 NADPH Glucose6-phosphat 2PG FBP 7 S7P 5 BEISPIEL [REDDY 96] [email protected] August ‘98 PHOSPHATZYKLUS Dihydroxyacetonphosphat 6 Xu5P Ru5P 4 Petrinetze & metabolische Netzwerke BEISPIEL PENTOSE- 15 GAP NAD+ + Pi Petrinetze & metabolische Netzwerke Stryer, L.: Biochemie; Spektrum der Wissenschaft 1988, S. 450. 5 / 25 [email protected] Pyruvat 4. Möglichkeit 6 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke PETRINETZE, BASICS 1 August ‘98 PETRINETZE, BASICS 2 (3) MARKEN (1) KNOTEN Plätze (bewegliche Objekte, Fahrzeuge, Werkstücke, Daten, Steuerflußzeiger, ..., Stoffeinheiten (z. B. Mol), ...) Transitionen Bedingung ist nicht erfüllt “passive Elemente” Bedingungen Zustände “chem. Verbindungen” “aktive Elemente” Ereignisse Aktionen “chem. Reaktionen” Bedingung ist (einmal) erfüllt n Bedingung ist n-mal erfüllt (2) KANTEN Vorbedingungen Nachbedingungen (4) MARKIERUNG (Systemzustand, Stoffverteilung) 5 Wieviele Marken befinden sich jeweils auf einem Platz? 3 Ereignis -> Anfangsmarkierung [email protected] 7 / 25 [email protected] 8 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke August ‘98 PETRINETZE, BASICS 3 BEISPIELE, REAKTIONSGLEICHUNG (5) MARKENFLUß ❑ FÜR LICHTINDUZIERTE PHOSPHORYLIERUNG 2 NAD+ + 2 H2O -> 2 NADH + 2 H+ + O2 ❑ ein Ereignis kann stattfinden, wenn -> alle Vorbedingungen (entsprechend den Kantengewichten) erfüllt sind; 2 2 2 ❑ wenn ein Ereignis stattfindet, dann werden H2O -> von allen Vorbedingungen (entsprechend den Kantengewichten) Marken entfernt, und 2 H+ r1 O2 ❑ AUS DER PHOTOSYNTHESE -> zu allen Nachbedingungen (entsprechend den Kantengewichten) Marken hinzugefügt; 2 CO2 + H2S + 2 H2O -> 2 (CH2O) + H2SO4 CO2 ❑ Stattfinden eines Ereignisses (Schalten/Feuern einer Transition) 2 2 CH2O H2S -> atomar 2 -> zeitlos [email protected] NADH NAD+ H2O 9 / 25 [email protected] r2 H2SO4 10 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke August ‘98 METABOLISCHE PETRINETZE TYPISCHE GRUNDSTRUKTUREN (1) PLÄTZE ❑ REAKTIONSKETTEN -> beteiligte Stoffe / chem. Verbindungen r1 MB1 MB2 r2 MB3 ❑ Substrate (Randplätze), ❑ (FREE-CHOICE) VERZWEIGUNG r1 InSub Input-Substrat MB2 MB1 r2 r1 OutSub Output-Substrat z. B. Glukose, Laktat; r2 MB3 ❑ Metabolite, z. B. Glukose-6-Phosphat ❑ VERZWEIGUNG MIT NEBENBEDINGUNG r1 ❑ Nebenbedingungen für Reaktionen, z. B. Elektronen-Carrier, Phosphoryl-Carrier; MB2 ❑ ggf. Enzyme MB1 r2 [email protected] MB3 11 / 25 [email protected] 12 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke METABOLISCHE PETRINETZE 2 METABOLISCHE PETRINETZE 3 (2) TRANSITIONEN ❑ ❑ August ‘98 (3) KANTENANSCHRIFTEN spontane Reaktionen -> Anzahl der durch die Reaktion betroffenen Stoffeinheiten enzym-katalysierte Reaktionen, zwei Modellierungsvarianten: (4) MARKENANZAHL ohne Berücksichtigung der Enzymkonzentration -> Anzahl verfügbarer Stoffeinheiten MB1 Enzym MB2 (5) ANFANGSMARKIERUNG mit Berücksichtigung der Enzymkonzentration x Enzym x MB1 -> Anfangsstoffverteilung x enzym-katal. Reaktion MB2 Σ METABOLISCHES PETRINETZ (MPN): x - Anzahl der notwendigen Stoffeinheiten, damit die Reaktion stattfinden kann; ❑ Menge aller Wege von den Input- zu den Output-Substraten unter Berücksichtigung der stöchiometrischen Verhältnisse; ggf. Transportschritte, -> inhomogene Stoffverteilung; [email protected] 13 / 25 [email protected] 14 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke August ‘98 BEISPIEL [REDDY 96] ALS PETRINETZ, VERSION 1 BEISPIEL [REDDY 96] ALS PETRINETZ, VERSION 2 Gluc ATP ADP Ru5P G6P Xu5P NADP+ S7P E4P 2 2 NADPH Ru5P NADPH GSSG GSSG F6P GAP 2 ATP F6P ADP R5P 2 2 Xu5P GSH 2 GAP S7P F6P E4P FBP 2 GSH R5P 2 NADP+ GAP DHAP NAD+ DHAP ATP ADP ATP Pi ADP NADH 1,3-BPG ADP NAD+ NADH ATP ATP GAP G6P Gluc F6P FBP ADP ATP NAD+ 3PG Pi 2PG ADP NADH PEP ADP Lac Pyr PEP 2PG 3PG ATP 1,3-BPG Pyr NADH NAD+ glukose1.ped Lac [email protected] 15 / 25 [email protected] glukose2.ped 16 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke BEISPIEL [REDDY 96] ALS PETRINETZ, VERSION 3 August ‘98 TYPISCHE PETRINETZFRAGEN Gluc ATP ADP (1) Wieviele Marken können sich maximal auf einem Platz befinden? Ru5P G6P NADP+ 2 2 NADPH 2 ATP Xu5P R5P GSSG F6P ❑ (0, 1, k, oo) -> BESCHRÄNKTHEIT GSH 2 ADP GAP S7P FBP F6P E4P (2) Wie oft kann eine Transition schalten? GAP NAD+ DHAP Pi 1,3-BPG ❑ (0-mal, n-mal, oo-mal) -> LEBENDIGKEIT ADP NADH 1,3-BPG ATP ADP 3PG ATP NADH 2PG (3) Ist ein bestimmter Systemzustand NAD+ Lac ❑ immer wieder erreichbar? -> FORTSCHRITTSEIGENSCHAFT PEP ADP ATP Pyr ❑ niemals erreichbar? -> SICHERHEITSEIGENSCHAFT NADH NAD+ glukose3.ped [email protected] Lac 17 / 25 [email protected] 18 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke BEISPIEL [REDDY 96] ALS PETRINETZ, VERSION 4 August ‘98 ANNAHMEN IN VERSION4 Gluc b2 b1 ❑ die zwei Auftreten von GAP können separiert werden (kein logischer Knoten) ATP ADP Ru5P G6P a2 NADP+ 3 3 2 2 2 b1 NADPH a2 2 b2 a1 Xu5P R5P GSSG F6P a1 ❑ die Verzweigungswahrscheinlichkeiten an den Konflikten von G6P und Ru5P sind bekannt und können durch die Verhältnisse G6P - 3:1 Ru5P - 2 : 1 charakterisiert werden 2 GSH ATP 2 ADP S7P GAP FBP F6P E4P GAP NAD+ DHAP Pi NADH 1,3-BPG ADP ATP 4 7 Lac Gluc start NADH 7 7 7 NAD+ ATP ADP Pi Lac glukose3_zyk.ped [email protected] 19 / 25 [email protected] 20 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke TYPISCHE ANALYSETECHNIKEN 1 August ‘98 TYPISCHE ANALYSETECHNIKEN 2 (5) NETZINVARIANTEN (1) MARKENSPIEL (?) ❑ P-INVARIANTEN -> Menge von Plätzen mit (gewichteter) konstanter Markensumme; (2) ERREICHBARKEITSGRAPH Knoten: Systemzustände Kanten: schaltende Transition mPn: Meta-Stofferhaltungsregeln, alle Elektronencarrier z1 t4 t1 z2 Bsp: P-Invariante (Pi ,..., Lac); d.h. es kann nur so viel Lactose entstehen, wie Pi eingeht; t2 z3 t3 ❑ T-INVARIANTEN z4 -> Menge von Transitionen, die eine Markierung reproduzieren; (3) REDUZIERTE ERREICHBARKEITSGRAPHEN mPn: Reaktionsketten, die eine Stoffverteilung reproduzieren; BND, LIVE mPn: Elementarmodi [Schuster 93], [Schuster 96] (4) STRUKTURANALYSEN z. Bsp: konservativ -> beschränkt [email protected] Bsp: Forward/Backward-Reaktion der Triosephosphat-Isomerase 21 / 25 [email protected] 22 / 25 Petrinetze & metabolische Netzwerke August ‘98 Petrinetze & metabolische Netzwerke QUALITATIVE ANALYSIS August ‘98 INTEGRATION VON QUALITATIVER AND QUANTITATIVER ANALYSE TECHNIQUES NET REDUCTION STRUCTURAL PROPERTIES LINEAR PROGRAMMING static analysis ❑ ZEITVERBRAUCH place / transition invariants state / trap equation REACHABILITY ANALYSIS Zeit (complete) reachability graph compressed state spaces OBDDs, ONDDS Kronecker products reduced state spaces coverability graph dynamic analysis ❑ VERZWEIGUNGSWAHRSCHEINLICHKEITEN (model checking) p1 symmetry p2 stubborn / sleep sets p1 + p2 = 1 branching processes concurrent automaton [email protected] 23 / 25 [email protected] 24 / 25 Petrinetze & metabolische Netzwerke August ‘98 MODEL CLASSES PETRI NETS PLACE/TRANSITION PETRI NET context checking by Petri net theory (COLOURED PN) verification by temporal logics TIME-DEPENDENT PN NON-STOCHASTIC PETRI NET STOCHASTIC PETRI NET worst-case evaluation performance prediction reliability prediction [email protected] 25 / 25