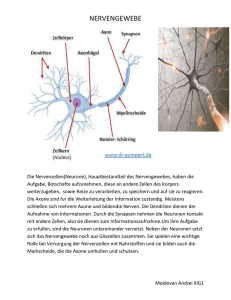
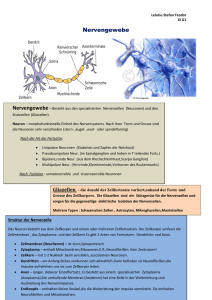
Press release - the Bernstein Center for Computational
Werbung

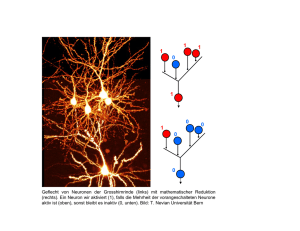
Max-Planck-Institut für Dynamik und Selbstorganisation MPI für Dynamik und Selbstorganisation ● Postfach 2853 ● D 37018 Göttingen -PressestelleAm Faßberg 17, 37077 Göttingen Telefon: (0551) 5176 - 668 oder 0173 3958625 Telefax: (0551) 5176 - 463 Datum: 8. Oktober 2012 Ein Blick auf den Schaltplan des Gehirns Mit einem neuen Verfahren lassen sich Verbindungen zwischen Nervenzellen kartografieren. Unser Gehirn vollbringt seine bemerkenswerten Leistungen durch das Zusammenspiel einer unvorstellbaren Vielzahl von Nervenzellen, die in komplexen Netzwerken miteinander verschaltet sind. Ein Team von Wissenschaftlern des Max-Planck-Instituts für Dynamik und Selbstorganisation (MPIDS), der Universität Göttingen und des Bernstein Center for Computational Neuroscience Göttingen hat nun eine Methode entwickelt, mit der sich neuronale Schaltpläne entschlüsseln lassen. Aus Messungen der gesamten neuronalen Aktivität können sie bestimmen, mit welcher Wahrscheinlichkeit jeweils zwei Nervenzellen miteinander verbunden sind. Die Studie, die helfen soll, die Funktionsweise des Gehirns besser zu verstehen, ist in der Fachzeitschrift PLoS Computational Biology erschienen. Aus rund 80 Milliarden Nervenzellen besteht das menschliche Gehirn und keine von ihnen lebt für sich allein. Die Nervenzellen (auch Neuronen genannt) bilden ein eng verknüpftes Netzwerk, durch welches sie untereinander Signale austauschen. Die Verbindungen sind dabei alles andere als zufällig arrangiert und ein Verständnis davon, welche Neuronen miteinander Verbindungen eingehen und welche nicht, verspricht wertvolle Informationen über die Funktionsweise des Hirns. Das Verbindungsnetzwerk direkt aus der Gewebestruktur zu ermitteln, ist eine bis auf weiteres praktisch unmögliche Aufgabe - selbst in Zellkulturen mit nur einigen tausend Neuronen. Hingegen gibt es heutzutage weit entwickelte Methoden, um dynamische Aktivitätsmuster von Neuronen aufzuzeichnen. Ein solches Muster gibt an, welches Neuron zu welchem Zeitpunkt ein Signal weitergeleitet hat. Es ist somit eine Art neuronales Gesprächsprotokoll. Das Göttinger Team rund um Theo Geisel, Direktor am MPIDS, hat sich nun eines dieser Aktivitätsmuster zunutze gemacht. Die Wissenschaftler verwenden Daten aus sogenannten Kalzium-Fluoreszenz-Messungen, die in Zusammenarbeit mit der Universität Barcelona aufgenommen wurden. Diese Bildgebungsmethode erlaubt es, die Aktivität von Tausenden von Neuronen in einer Zellkultur (oder im lebenden Hirn) gleichzeitig aufzuzeichnen. Denn für das Weiterleiten eines elektrischen Signals sind am Neuron Ionen wie etwa Kalzium-Ionen verantwortlich. Mit Licht einer bestimmten Wellenlänge bestrahlt, beginnen diese zu leuchten und verraten so, welche Neuronen gerade aktiv sind. Jedoch sind die zeitlichen Abläufe in Neuronen zu schnell, um direkt mitverfolgen zu können, wie ein Impuls „abgefeuert“ wird. Es lässt sich also nicht unmittelbar nachzuvollziehen, wie eine einzelne Zelle eine andere beeinflusst oder ob eine Verbindung direkt ist oder über mehrere Stationen verläuft. Mit dem im Team von Theo Geisel nun entwickelten Algorithmus lassen sich aber aus den gemessenen Daten dennoch bemerkenswert genaue Informationen über die Linienführung im Verbund der Nervenzellen gewinnen. „Unsere Methode basiert auf der so genannten Transferentropie“, erklärt Olav Stetter vom MPIDS, Erstautor der nun erschienenen Publikation. Die Transferentropie ist eine Größe aus der Informationstheorie, mit der gerichtete Informationsflüsse quantitativ bestimmt werden können. Mit anderen Worten: Es kann berechnet werden, wie wahrscheinlich es ist, dass das Signal von einem bestimmten Neuron eine Aktivität in einem anderen zur Folge hatte. „Wir haben mithilfe der Transferentropie ein robustes Verfahren entwickelt, das zuverlässig zwischen wahren kausalen Zusammenhängen unterscheiden kann und solchen, die nur scheinbar bestehen und durch indirekte Verbindungen oder durch Messfehler entstanden sind“, sagt Stetter. Die Wissenschaftler haben ihre neue Methode zunächst auf künstliche Daten angewandt. Sie sind dabei von einem gegebenen Netzwerk ausgegangen und haben die Signale, welche in einem Kalzium-Fluoreszenz-Experiment zu erwarten wären, in realistischer Qualität berechnet. Wie die Wissenschaftler zeigen, sind die kausalen Zusammenhänge im Netzwerk zeitlich variabel und abhängig vom Zustand der Aktivität des Netzwerkes. Nur in ruhigeren Phasen mit relativ geringer Aktivität stimmten die kausalen Zusammenhänge mit dem tatsächlichen Aufbau des Netzwerks überein. Diese Tatsache konnten sich die Göttinger Forscher für ihre Vorhersagen nutzbar machen. In Phasen hoher Aktivität hingegen sind so viele Nervenzellen gleichzeitig am neuronalen Gesprächsfluss beteiligt, dass sich der Weg einer Information nicht mehr gut nachvollziehen lässt. In der nun publizierten Arbeit wurden auch echte Neuronen untersucht. Die Analyse mit der neuen Methode lieferte somit bereits konkrete Resultate und deckte eine ungewöhnliche Konzentration von Verbindungen rund um einzelne Zellen auf. Die Forscher sind zuversichtlich, dass sich ihr Verfahren auf eine breite Klasse von Systemen anwenden lässt: „Unsere Methode ermöglicht eine Rekonstruktion von neuronalen Netzwerken ohne spezifische Annahmen über deren Beschaffenheit. Vielmehr lassen wir uns von den Daten leiten“, erklärt MPIDS-Mitarbeiter Demian Battaglia. Dies lässt hoffen, dass der Algorithmus der Göttinger Wissenschaftler es erlaubt, in breitem Umfang neuronale Schaltpläne zu berechnen, in gezüchteten Netzwerken ebenso wie in natürlichen. Die gesammelten Informationen aus einer Vielzahl von verschiedenen neuronalen Netzwerken können dann helfen zu verstehen, wann und wo die Neuronen Verbindungen eingehen und nach welchen Kriterien sie ihre Partner auswählen. Abbildung 1: Aus der Aktivität der Neuronen, die sich in Fluoreszenz-Messungen zeigt (im Bild links), können die Wissenschaftler darauf schließen, wie die Nervenzellen miteinander verschaltet sind (im Bild rechts angedeutet). Grafik: MPIDS Originalveröffentlichung: Olav Stetter, Demian Battaglia, Jordi Soriano, Theo Geisel: Model-free Reconstruction of Excitatory Neuronal Connectivity from Calcium Imaging Signals, PLoS Computational Biology, 8(8): e1002653 (23. August 2012), doi:10.1371/journal.pcbi.1002653 Kontakt: Dr. Birgit Krummheuer Presse- und Öffentlichkeitsarbeit Max-Planck-Institut für Dynamik und Selbstorganisation Tel.: +49 (0)551 5176-668 mobil: +49 (0)173 3958625 E-Mail: [email protected] Olav Stetter Max-Planck-Institut für Dynamik und Selbstorganisation Tel.: +49 (0)551 5176-444 E-Mail: [email protected] Prof. Dr. Theo Geisel Max-Planck-Institut für Dynamik und Selbstorganisation Tel.: +49 (0)551 5176-401 E-Mail: [email protected]