bacTrace
Werbung

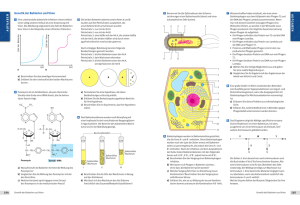
bacTrace-Vision Der bei der BacTrace GmbH in Entwicklung befindliche Schnelltest bacTrace verwendet natürliche Bakteriophagen, um Bakterien nachzuweisen. Der „normale“ Bakteriophage weist ein sehr enges Spektrum von Bakterien auf, die er als host (Wirtsbakterium) verwendet - im Prinzip sollte jede Bakteriophagenart eine einzige Bakterienart als host akzeptieren. Häufig ist diese Bakteriophagen-Bakterien-Reaktion sogar noch spezifischer: Bakteriophagen reagieren dann nur mit einem mehr oder weniger kleinen Teil einer Bakterienart und nicht mit allen Bakterien einer Art. Es existieren aber auch sog. broad host Phagen, die ein breites Spektrum von hosts für ihre Fortpflanzung benutzen. Dabei handelt es sich in der Regel um eng verwandte Bakterienarten. Im Folgenden soll aufgezeichnet werden, dass für bacTrace Überlegungen im Gange sind, die natürlichen Grenzen der BakteriophagenBakterien-Reaktion mit Mitteln der modernen Biologie und Gentechnik aufzuweichen. 2014 In der SINAMIRA AG entsteht die Idee für einen Schnelltest zum Nachweis von Bakterien unter Verwendung von Bakteriophagen: Erste Vorversuche, Beginn der Entwicklung neuer Spektrometer mit bisher unbekannter Empfindlichkeit gemeinsam mit der rgb GmbH. 1 2015 Gemeinsam mit der DSMZ1 und dem Lehrstuhl für medizinische Biotechnologie2 werden von der SINAMIRA AG richtungsweisende Versuche mit dem E. coli-Phagen TB54 eingeleitet, die beweisen, dass die Idee, dass fluoreszierende Bakteriophagen auf ihren hosts nachgewiesen werden können. Das europäische Patent wird im November beantragt. 2016 Entwicklung erster einwandfrei funktionierender Vorläufer der bacTrace-Messgeräte. Gründung der BacTrace GmbH. Einreichung des weltweiten Patents im Dezember. Grundlagen Für die weiteren Ausführungen werden die wissenschaftlichen Grundlagen von bacTrace als bekannt vorausgesetzt. In bacTrace derzeit zum Einsatz kommende Bakteriophagen werden aus ihren natürlichen Umgebungen entnommen und dann unverändert - allerdings mit Fluoreszenz versehen – in hoher Konzentration als Detektoren für ihre natürlichen Wirtsbakterien verwendet. Bakteriophagen kommen in Meerwasser natürlicherweise in einer Konzentration von 107 Phagen/ml3 vor, in der Reaktionskammer von bacTrace werden dagegen Konzentrationen von 109 bis 1011 Phagen pro ml eingesetzt. Dazu bedarf es ausgeklügelter Zucht- und Reinigungsprozesse im Herstellungsprozess. Die Verwendung fluoreszierender ansonsten aber unveränderter natürlicher ganzer Phagen zum Nachweis von ausgewählten Bakterien ist sozusagen Schritt eins in der Entwicklung von bacTrace. Wenn bacTrace geeignet für einen breiten Einsatz in Medizin und Technik sein soll, müssen zumindest die in diesen Bereichen wichtigen (häufigen oder Probleme verursachenden) Bakterien nachgewiesen werden können – allein im humanmedizinischen Bereich sind dies 1 Leibniz Institut DSMZ - Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH in Braunschweig, eines der größten Bioressourcenzentren weltweit 2 Lehrstuhl für Medizinische Biotechnologie (Prof. Dr. Dr. Oliver Friedrich) im Department Chemie- und Bioingenieurwesen an der Technischen Fakultät der Friedrich-Alexander Universität 3 Bakteriophagen. Vielfalt, Anwendung und ihre Bedeutung für die Wissenschaft vom Leben. C. Rhde Und J. Sikorski. Naturwissenschaftliche Rundschau 64. Jahrg., Heft 1, 2011 (5-14) 2 mindestens 35 bakteriellen Erreger4, unter Einschluss selten als Infektionserreger auftretender Bakterien kann diese Zahl auf 150 Bakterienarten oder mehr ansteigen. Die im Internet frei zugängliche Virus-Host Datenbank5 verzeichnet im Januar 2016 300 Bakterienarten von klinischer Relevanz, für die Bakteriophagen bekannt sind. Bisher wurden weltweit ca. 6.000 Bakteriophagenarten genau beschrieben. Diese Zahl mag sich groß anhören, in Wirklichkeit sind bisher aber nur für einen Teil klinisch oder technisch wichtiger Bakterien Bakteriophagen entdeckt worden. Dabei ist es fraglich, ob z.B. asiatische Phagen aus der Datenbank in Kyoto „deutsche“ hosts akzeptieren. Die gute Nachricht dabei ist, dass man bei uns auch nicht gezielt nach Phagen gesucht hat - schlicht, weil kein Anlass dafür bestand. Allerdings ist es aufgrund der Gültigkeit des NAGOJA-Protokolls in der BRD seit 2016 quasi unmöglich, Bakteriophagen aus anderen Ländern (als Ausnahmen sind Holland, Belgien und ………… zu nennen) zu importieren6 und evtl. für bacTrace zu verwenden. Aber die grundsätzliche Verfügbarkeit ist mindestens Zeichen, dass für diese Bakterien Bakteriophagen zu finden sein werden – wir müssen sie eben suchen! Als Konsequenz aus dem NAGOJA-Protokoll könnten wir zwar Bakteriophagen aus der Sammlung der DSMZ verwenden, deren Zahl mit 55 Bakteriophagen (davon 20 mit mehr oder weniger großer Bedeutung für die Humanmedizin) allerdings begrenzt ist. Deshalb hinaus wird die BacTrace GmbH (Stand Jan. 2016) ein über die nächsten Jahre laufendes Suchprogramm für die wichtigsten Bakteriophagen-Arten gemeinsam mit der DSMZ initiieren, um Bakteriophagen der wichtigsten klinischen bakteriellen Erreger zu identifizieren. Das sollte für die in Tabelle 1 aufgeführten Bakterienarten gelingen. 4 Antibiotika. Stille. 2012 Mihara, T., Nishimura, Y., Shimizu, Y., Nishiyama, H., Yoshikawa, G., Uehara, H., Hingamp, P., Goto, S., and Ogata, H.; Linking virus genomes with host taxonomy. Viruses 8, 66. (Laboratory of Chemical Life Science, Kyoto University Bioinfomatics Center, Institute for Chemical Research) 5 6 „Bei Mikroorganismen macht das Nagoya-Protokoll viele zu Verlierern“. Stellungnahme des DSMZ auf der Website des Instituts 3 Häufig auftretende Infektionserreger Weniger häufig auftretende wichtige Infektionserreger Staphylococcus aureus Staphylocossus epidermidis Streptococcus pneumoniae Haemophilus influenzae Haemophilus parainfluenzae Klebsiella pneumoniae Escherichia coli Enterobacter cloacae Enterococcus faecalis Pseudomonas aeruginosa Proteus mirabilis Proteus vulgaris Staphylococcus saprophyticus Bacteroides fragilis Entercoccus faecium Enterobacter aerogenes Serratia marcescens B-Streptokokken (agalactiae) Chlamydia trachomatis Ureaplasma urealyticum Mycoplasma hominis Neisseria gonorrhoeae Citrobacter freundii Moraxella catarrhalis Stenotrophomonas maltophilia Pasteurella multocida Acinetobacter baumannii Providencia rettgeri Bordetella pertussis Legionella pneumophilia Tabelle 1. Klinisch wichtige Bakterienarten Außerdem ist vorgesehen, im Internet eine BacTrace-Phagen-Datenbank zu entwickeln, in der weltweit Forscher die von ihnen entdeckten Bakteriophagen eintragen und beschreiben können, wo und wie sie sie gefunden und isoliert haben – auch dies hilft uns weiter, zu definieren, ob und für welche Phagen Chancen bestehen, sie mit welcher Methode auch in Deutschland zu finden. Es ist in diesem Projekt nicht vorgesehen, diese Phagen physisch zu sammeln. Entwicklungslinie 1 Es gibt viele Bakteriophagenarten, die zudem sehr unterschiedlich aufgebaut sein können. Aber das Bauprinzip der Bakteriophagen ist im Endeffekt einfach und einander ziemlich ähnlich. Fast alle Phagen bestehen aus einem Kopf mit DNA oder RNA, einer Kragen- und HalsRegion und einer Endplatte, an der die Beine ansetzen. 4 Abb. 1. Darstellung eines typischen Bakteriophagen und eine stark vereinfachte Darstellung Bakteriophagen können ihre Wirtsbakterien weder auf - auch nur geringe - Entfernung wahrnehmen, noch sich Ihnen gezielt nähern. Nur wenn eines der Bakteriophagen-Beine zufällig auf das richtige Bakterium trifft, setzt eine - dann allerdings sehr schnelle und sehr feste (irreversible) - Bindung ein. Als nächstes finden auch die anderen Füße des Bakteriophagen auf dem Wirtsbakterium ihren irreversiblen Halt. Dann wird die Bakteriophagen-DNA resp. -RNA in das Bakterium gespritzt, und die Verwandlung des Bakteriums in einen Bakteriophagen-produzierenden „Zombie“ setzt ein. Alles, was dann kommt, ist eine spannende „Geschichte“, die uns für bacTrace aber nicht weiter interessieren muss. Für bacTrace ist einzig und allein wichtig, dass die BakteriophagenHülle auch nach der Injektionsphase unverrückbar fest auf der Bakterienwand hängen bleibt. Die in bacTrace zur Verwendung kommenden Bakteriophagen sind bekanntlich mit Fluoreszenzfarbstoffen gekennzeichnet worden. Die durch ein Anregungslicht an den Fluoreszenzfarbstoffen induzierten emittierten Frequenzen werden verwendet, um die Bakterienarten zu identifizieren. Abb. 2. bacTrace-Bakteriophagen werden an Kopf- und Halsregion mit Fluoreszenzfarbstoffen gekennzeichnet - jede Bakteriophagenart mit einem anderen Fluoreszenzfarbstoff. Die Füße werden dagegen nicht mit Fluoreszenz versehen, um deren biologische Reaktionsfähigkeit (die Bindungsreaktion an das host-Bakterium) nicht zu beeinträchtigen. 5 Heute verwenden wir in der bacTrace-Reaktionskammer die gesamten Bakteriophagen, also Kopf, Hals und Beine. Den Bakteriophagen Kopf benötigt bacTrace im Moment nur, um als Träger von Fluoreszenz-Molekülen zu dienen. In einem nächsten Schritt ist vorgesehen, unter Verwendung neuer Fluoreszenzfarben den Bakteriophagen den Kopf „abzuschneiden“. Er wird in der von bacTrace verwendeten Bindungsreaktion nicht benötigt. Und er beinhaltet genetisch aktives Material, dass wir auch nicht brauchen und das potenziell eventuell … nein, nicht gefährlich ist, aber wir brauchen es einfach nicht! Genetisch aktives Material wie DNA und RNA könnte von Regulationsbehörden irgendwann einmal als „gefährlich“ angesehen werden – und wenn es nur darum geht, ob die bacTrace-Küvetten irgendwann einmal als Sondermüll eingestuft werden sollten oder nicht. Abb. 3. Fluoreszierender Phagenrest ohne Kopf Es ist also vorgesehen, zu prüfen, ob kopflose Bakteriophagen„Reste“ die biologischen Reaktionen auszuführen in der Lage sind, die wir in bacTrace brauchen. Falls ja, und nichts spricht aus heutiger Sicht dagegen, werden wir kopflose Bakteriophagen einsetzen, die nur aus Hals und Füßen bestehen. Die Fluoreszenz wird dann nur noch am Hals angebracht. Wir planen dafür ein ca. 1jähriges Forschungsprojekt. Im dritten (logischen) Schritt, der sich heutzutage andeutet, verzichten wir dann bei geeigneten Bakteriophagen-Arten auch noch auf die Bakteriophagen-Halsregion und verwenden nur noch die an der Basisplatte zusammenhängenden Beine der Bakteriophagen, die immer noch in der Lage sein sollten, ihre biologische Reaktion auszuführen. 6 Abb. 4. Mit Fluoreszenz versehene Basisplatte und Beine eines Bakteriophagen Die Fluoreszenz wird an der Basisplatte angebracht. Die von der BacTrace GmbH selbst entwickelten und in bacTrace verwendeten Spektrometer sind empfindlich genug, um mit der Restfluoreszenz ein für die Detektion der Bakterien ausreichendes Signal zu registrieren. Dieses Projekt ist Teil des o.g. Forschungsprojektes. Abb. 5. Letztes Entwicklungsstadium: Verzicht auf die Bakteriophagen – es werden nur noch die Bindungsmoleküle (evtl. mit einem anderen Fluoreszenzfarbstoff) verwendet Als letztes Entwicklungsstadium werden wir versuchen, die technisch eleganteste Lösung für bacTrace zu entwickeln, und auch noch auf die Bakteriophagen-Beine zu verzichten – um nur noch fluoreszierende Bindungsmoleküle der Bakteriophagen-Füße zu verwenden. Damit hätte diese Entwicklungslinie von bacTrace ihr Endstadium erreicht. Die zweite Entwicklungslinie setzt auf die Gentechnik Die Natur hat uns die fantastische Bakteriophagen-Host Reaktion beschert, die wir in bacTrace einsetzen. Für die Natur mag diese Reaktion perfekt sein – sie tut schließlich alles, was Bakteriophagen und Bakterien benötigen, um Milliarden Jahre nebeneinander zu existieren. 7 Man darf auch nicht vergessen, dass nicht nur die Bakteriophagen in dem Zusammenspiel Bakteriophagen-Bakterien Gewinner sind, weil sie sich in den Bakterien fortpflanzen, nein, auch die Bakterien als hosts gewinnen in der der Reaktion – das zu erläutern, würde an dieser Stelle in die falsche Richtung führen. Aber der Mensch will ja meistens mehr, als die Natur ihm kostenlos anbietet. Auch für bacTrace wäre es sehr interessant, die Grenzen der Natur hinsichtlich der zur Verfügung stehenden Phagen zu sprengen, und über Phagen zu verfügen, die mehr können als die natürlich vorkommenden, z.B. Phagen, die alle Bakterien einer Art als host akzeptieren Phagen, die ausgewählte Gram-positive oder Gram-negative Bakterien als host akzeptieren Phagen, die z.B. Antibiotika-Resistenzen erkennen – z.B. MRSA Broad host Phagen, die artübergreifend eine Reihe von Bakterien als host akzeptieren, die typischerweise bestimmte Infektionen verursachen Phagen, die besonders lange in bestimmten Flüssigkeiten existieren, ohne ihre biologische Reaktionsfähigkeit zu verlieren Es ist heute ohne weiteres möglich Bakteriophagen gentechnisch so zu verändern, dass sie bestimmte neue Aufgaben erfüllen, für die sie die Natur eigentlich nicht vorgesehen hatte – z.B. als „Fähren“ für Gene funktionieren oder chemische Substanzen in Zellen hinein transportieren. Bakteriophagen sind selber (sehr kleine) Gentechniker, die die DNA von Bakterien z.B. im Zuge der sog. lysogenen Vermehrung7 verändern können. Bei diesem Prozess kann es zu „Unfällen“ kommen, die entweder von internen Reparaturmechanismen „geheilt“ werden oder die DNA der Nachfolgegenerationen dauerhaft verändern – und wenn der nicht reparierte „Unfall“ merkbare Veränderungen bei Bakteriophagen oder Bakterien ergibt, nennt man das in der Natur… Evolution! Wenn man bedenkt, dass sich ein E. coli unter guten Bedingungen alle 20 Minuten reproduziert (und das seit fer Entstehung des Lebens vor 3,x Milliarden Jahren), kann man sich vorstellen das sich über die Zeit bei theoretisch ca. 8*1013 Generationen vom 1. Bakterium bis zum heutigen E. coli schon viele „Fehler“ eingeschlichen haben können. Noch einmal: das ist dann Evolution, der Prozess, der über viele Abzweiger auch zur Entwicklung des modernen Menschen geführt hat. Und wenn der Mensch in den Prozess inzwischen irgendwie steuernd eingreift, nennt man das Gentechnik. 7 Vermehrungszyklus von Bakteriophagen, bei der das Wirtsbakterium nicht zerstört wird. Die Bakteriophagen-DNA wird in die bakterielle DNA integriert und bei der Zellteilung des Bakterium mit vermehrt. 8 Es sollte also grundsätzlich möglich sein, Bakteriophagen gentechnisch so zu verändern, dass sie die o.g. Ansprüche erfüllen. Abb. 6. Es ist heute möglich, mit drei richtig ausgewählten „natürlichen“ Staphylococcus aureus-Bakteriophagen fast 100% aller S. aureus-Stämme zu detektieren. Unser Ziel sollte es sein, 100% aller S. aureus-Stämme mit einem gentechnisch veränderten Phagen zu erreichen. Dasselbe gilt für alle anderen Bakterienarten Derzeit sind wir dabei, Labors zu identifizieren, die diese Aufgaben im Auftrag von BacTrace GmbH ausführen können. Wenn es gelungen ist, Phagen gentechnisch so zu verändern, dass sie neue Aufgaben zu erfüllen, können wir darangehen, wie oben beschrieben, nicht mehr den ganzen Phagen in bacTrace einzusetzen, sondern nur noch die notwendigen biologisch aktiven Teile. Insbesondere der Verzicht auf die Kopfregion mit DNA oder RNA könnte zulassungstechnisch Vorteile resp. Vereinfachungen bringen. 9