Bio 5
Werbung

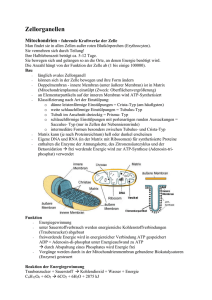
Endoplamatisches Retikulum • Glattes Endoplasmatisches Retikulum (sER) • Rauhes Endoplasmatisches Retikulum (rER) Aufgaben des sER • Synthese der Membranlipide • Entgiftungsfunktion (Cytochrom P450) • Synthese von Steroidhormonen • Proteinreifung • Vesikeltransport zum Golgi-Appart • Sonderform: sarkoplasmatisches Retikulum in Muskulatur (Ca2+-Speicher) Aufgaben des rER • Proteinsynthese: – Für Sekretion Plamaproteine zB Albumine, Immunglobuline strukturelle Proteine (Kollagen) enzymatische und proteolytische Enzyme (Trypsin) – Für intrazelluläre Funktion – Für Zellmembranen Proteinsynthese bei Eukaryonten • erfolgt im Cytoplasma an freien Ribosomen • oder an Ribosomen am rauhen ER • bei Vorliegen einer kurzen spezifischen Aminosäuresequenz am Amino- (NH2) terminalen Ende wird der jeweilige weitere Transport veranlaßt Typische Signalpeptide zB für • Import in das ER: • Verbleiben im ER: • Import in Mitochondrien: • Import in den Kern: ((-Pro-Pro-Lys- Lys-Lys-Arg-Lys-Val …nicht Details der Aminosäuresequenz lernen!) • Import in Peroxisomen usw. Freie und membrangebundene Ribosomen • ribosomale Untereinheiten lagern sich an mRNA • abhängig von der gebildeten ASSequenz erfolgt die weitere Synthese im Cytoplasma oder am ER „signal recognition particle“ SRP • das SRP (Signal-Erkennungspartikel) erkennt das kurze Signalpeptid eines für das ER bestimmten Proteins • bindet dieses • verhindert zunächst die weitere Synthese • bindet (nach Diffusion spezifisch) an den SRP Rezeptor des ER • dann erfolgt durch ein Translokatorprotein (wird durch Ribosomenbindung geöffnet) die weitere Synthese und Übertragung des Proteins in das Lumen des ER Qualitätskontrolle im ER • im ER werden falsch gefaltete Proteine durch Chaperon-Proteine zurückgehalten • gegebenfalls nachgefaltet oder sofern dies nicht gelingt, ins Cytoplasma abgegeben und abgebaut Glykosylierung in ER • Proteine werden im ER Lumen meist sofort glykosyliert → es werden Zuckergruppen, Glukose, Mannose usw. an NH2 Gruppen von Asparagin angehängt (NGlykosylierung) Synthese eines Transmembranproteins • Proteine, welche als Transmembranproteine in die Phospholipidmembran eingebaut werden, weisen eine (oder mehrere) Start-Transfer- und Stop-Transfer-Peptidsequenz(en) auf Aufgaben des Golgi-Apparats I • Modifikation der N-Glykosylierung • O-Glykosilierung (an OH-Gruppen von Serinund Threonin-Seitengruppen durch Glycosyltransferasen) • Modifikation und Verteilung von Proteinen, Lipiden, Proteoglykanen für sekretorische Vesikel, Plasmamembran und Lysosomen Aufgaben des Golgi-Apparats II • Bildung der extrazellulären Matrix • unterschiedlichen Sekreten aus Glykoproteinen, Proteoglykane und Peptidoglykanen • Synthese von Bestandteilen der Glykokalix • Bindung von Sulfatgruppen an Proteoglykane Oligosaccharidtypen • Mannose-reiche Oligosaccharide • komplexe Oligosaccharide (aus verschiedenen Zuckerresten) Glykosilierung in ER- und Golgi-Lumen • In einem mehrstufígen Prozess werden zuerst Zuckergruppen von der N-Glykosylierung durch Glucosidasen und Mannosidasen noch im ER abgespalten • im Golgi-Apparat werden zunächst weitere Mannosereste abgespaltet und dann nach Phosphorilierung weitere Zuckergruppen angefügt, dabei auch O-Glykosylierung Sekretion • konstitutiv (kontinuierlich) oder • reguliert (zB. über Rezeptoren vermittelt) Ribosomen • aus großer und kleiner Untereinheit aufgebaut • mit A-(Aminoacyl-tRNA)-, -P-(Peptidyl-tRNA)-, und E-(Exit)-Bindungsstellen und Peptidyltransferase an großer Untereinheit und mRNA-Bindungsstelle an kleiner Untereinheit eukaryontische Ribosomen • aus 28 S, 5.8 S und 5 S rRNA wird große Untereinheit mit (etwa 50) Proteinen aufgebaut • die kleine Untereinheit besteht aus der 18 S rRNA und (etwa 30) Proteinen • die 45 S rRNA (für 28,18,5.8 S rRNAs wird an der NOR an kurzen Armen der akrozentrischen Chromosomen gebildet • die 5 S rRNA an Gencluster an Chromosom 1 (Angaben in Klammer auf dieser Folie nicht lernen) Translation • Initiation • Elongation • Termination Translation • an mRNA in 5‘ - 3‘ Richtung • bei eukaryontischen Ribosomen etwa 2 Aminosäuren/Sekunde in Peptidkette eingebaut (in Prokaryonten etwa 10 x so schnell) • nach Initiation, Elongation bis zur Termination durch Binden eines Release-Faktors (Freisetzungsfaktors) an ein Stop-codon (Angaben in Klammer auf dieser Folie nicht lernen) Initiation • die Initiator-tRNA (mit gebundenem Methionin und Initiationsfaktoren) bindet an die kleine Untereinheit (UE) • die mRNA bindet an die kleine UE (über 5‘ Cap) und letztere sucht von 5‘- 3‘ nach dem ersten AUG • Intitiationsfaktoren lösen sich und die große UE bindet mit P-Stelle an Initator-tRNA Elongation • anschließend bindet die erste Aminoacyl-tRNA an die A-Stelle und in der PeptidyltransferaseStelle • der großen UE wird eine Peptidbindung zwischen den ersten zwei Aminosäuren geknüpft • dann bewegt sich die kleine UE um ein Codon nach 5‘ wodurch die A-Stelle für die nächste Aminoacyl-tRNA frei wird und der Vorgang wird wiederholt Proteinstabilität • sehr variabel, für bestimmte Proteine sogar extrem unterschiedlich • bestimmte Strukturproteine, wie in Knorpel, Knochen usw. sind für Monate bis Jahre stabil → Stoffwechselenzyme und Wachstumsfaktoren sind oft nur Sekunden bis Tage stabil • Proteinfaltung erfolgt spontan oder durch Chaperone • Proteinabbau durch Proteasen bzw. in Proteasomen Proteasomen • Neben den Lysosomen kommen im Cytoplasma eukaryontischer Zellen die Proteasomen für den Proteinabbau vor • Sie stellen kleine (etwa 10 nm Durchmesser) in wesentlichen rohrförmige Gebilde dar, die zum Abbau von überschüssigen, fehlerhaften oder „alten“ Proteinen dienen • der Import erfolgt über einen Ubiquitinabhängigen Transportmechanismus in das Innere der Proteasomen • In das Lumen der Proteasomen sind wandständige Proteasen und Peptidasen eingebaut, welche eingebrachte Proteine in die Einzelbausteine, also Aminosäuren abbauen und wieder in das Cytoplasma abgeben • Bei einer schweren Krankheit, dem Angelman Syndrom, sind bei einem Teil der Patienten Mutationen in Ubiquitin-Proteinligase E3A-Gen gefunden wurden. Das genannte Enzym ist für das „Anhängen“ von Ubiquitin an Proteine, und damit für deren Import und Abbau wesentlich Kernporen • bestehen aus vielen Proteinen → bilden achteckigen Kernporenkomplex • der Durchmesser der Öffnung liegt bei etwa 9 nm (kann aktiv bis auf nahezu 30 (26) nm) erweitert werden • Fibrillen an der cytoplasmatischen Seite und ein „nukleärer Käfig“ an der Innenseite sind für die Funktion wesentlich • liegen in großer Zahl an der Kernmembran vor • sind für den Stoffaustausch zwischen Cytoplasma und Karyoplasma sehr wichtig • ua. werden durch Kernporen ribosomale und viele weitere Proteine (zB. Enzyme, wie DNA-und RNA-Polymerasen) in den Kern transportiert und mRNA, Ribosomenuntereinheiten aus dem Kern transportiert Passage durch Kernporen • Kleine Komponenten können passiv durch Diffusion durch die Kernporen • Größere Komponenten müssen aktiv, mit ATP Verbrauch unter Erweiterung des Kernporenkomplexes transportiert werden • Signalsequenzen der Proteine für den Kernimport und Rezeptormoleküle sind beteiligt Kernlamina • Aufbau aus Intermediärfilamenten (Lamine) • Stabilisierung der Kernhülle Änderung des Kerns während (mitotischem) Zellzyklus • Im mitotischen Zellzyklus wird der Zellkern in der S (Synthese)-Phase auf etwa den doppelten Inhalt vergrößert • Am Anfang der Mitose wird die Kernhülle aufgelöst (zerfällt unter Phosphorilierung der Kernlamina in kleine Fragmente) • am Ende der Mitose wird sie durch Dephosphorilierung der Kernlaminafragmente wieder aufgebaut