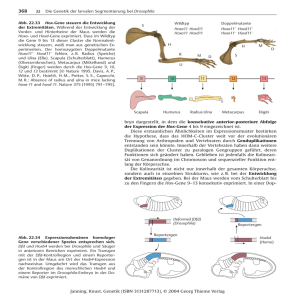
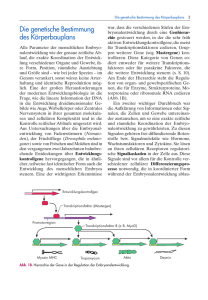
Dokument_46
Werbung

Evolution früher Faktoren der Segmentierungskaskade: Funktionelle Untersuchungen in Bruchidius, Tribolium und Oncopeltus Der Naturwissenschaftlichen Fakultät der Friedrich-Alexander-Universität Erlangen-Nürnberg zur Erlangung des Doktorgrades Dr. rer. nat. vorgelegt von Irene Schnellhammer aus Fürth Als Dissertation genehmigt von der Naturwissenschaftlichen Fakultät der Friedrich-Alexander Universität Erlangen-Nürnberg Tag der mündlichen Prüfung: 5.7.2012 Vorsitzender der Promotionskommission: Prof. Dr. Fink Erstberichterstatter: PD Dr. Michael Schoppmeier Zweitberichterstatter: Prof. Dr. Frasch Inhaltsverzeichnis Zusammenfassung VII Summary IX Abbildungsverzeichnis X Tabellenverzeichnis XII Abkürzungen XIII Allgemeine Einleitung . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1 Segmentierung in Drosophila . . . . . . . . . . . . . . . . . . . . . . . . . 1 Kurzkeim- und Langkeimentwicklung . . . . . . . . . . . . . . . . . . . . 4 Segmentierung in anderen Insektenspezies . . . . . . . . . . . . . . . . . . 4 Übersicht der Projekte und Modellorganismen . . . . . . . . . . . . . . . . 7 I. Kapitel: hunchback in Bruchidius obtectus 8 Einleitung . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 10 Die Funktion von Hunchback in Langkeim-Insekten . . . . . . . . . . . . . 10 Die Funktion von Hunchback in Kurzkeim-Insekten . . . . . . . . . . . . . 12 Ziele dieses Projekts . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 14 Material & Methoden . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 16 Bruchidius obtectus: Haltung und Zucht . . . . . . . . . . . . . . . . . . . 16 Die Separierung verschiedener Entwicklungsstadien . . . . . . . . . . . . . 16 Unterscheidung zwischen Männchen & Weibchen . . . . . . . . . . . . . . 17 Fixierung der Bruchidius-Embryonen . . . . . . . . . . . . . . . . . . . . 18 Klonierung von Bruchidius hunchback und paired . . . . . . . . . . . . . . 18 III Inhaltsverzeichnis In-situ-Hybridisierung von Bruchidius-Embryonen . . . . . . . . . . . . . 19 Parentale RNAi in Bruchidius . . . . . . . . . . . . . . . . . . . . . . . . 21 Ovarpräparation von Bruchidius . . . . . . . . . . . . . . . . . . . . . . . 22 Ergebnisse . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 23 Das Bruchidius Hunchback-Ortholog . . . . . . . . . . . . . . . . . . . . . 23 Die Expression von maternalem hunchback im Bruchidius-Ovar . . . . . . 24 Die Expression von Bruchidius hunchback im Laufe der Embryogenese . . 25 hb-RNAi in Bruchidius resultiert in einem starken Gapgen-Phänotyp . . . . 29 Die Expression des Paarregelgens paired im B.o Wildtyp . . . . . . . . . . 31 Die Expression von B.o. paired in B.o. hb-RNAi-Embryonen . . . . . . . . 34 Diskussion . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 36 Kanonische Gapgen Funktion von B.o. hb in der anterioren Musterbildung . 36 B.o. hunchback ist wichtig für die posteriore Musterbildung . . . . . . . . . 37 Bruchidius und Tribolium Hunchback, ein Vergleich . . . . . . . . . . . . . 38 Die Hunchback-Funktion ist in Langkeimern konserviert . . . . . . . . . . 39 II. Kapitel: Das Mex-3/Cad System 41 Einleitung . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 42 Funktion und Ursprung von Bicoid . . . . . . . . . . . . . . . . . . . . . . 42 Regulationsmöglichkeiten von Caudal außerhalb Drosophila . . . . . . . . 44 Ziele dieser Arbeit . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 47 Material & Methoden . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 48 Oncopeltus fasciatus: Haltung und Zucht . . . . . . . . . . . . . . . . . . . 48 Die Separierung verschiedener Entwicklungsstadien . . . . . . . . . . . . . 48 Die Unterscheidung Männchen/Weibchen in Oncopeltus . . . . . . . . . . 49 Fixierung der Oncopeltus-Embryonen . . . . . . . . . . . . . . . . . . . . 50 Klonierung von mex-3, caudal und zen . . . . . . . . . . . . . . . . . . . . 50 In-situ-Hybridisierung von Oncopeltus . . . . . . . . . . . . . . . . . . . . 51 Parentale RNAi in Oncopeltus . . . . . . . . . . . . . . . . . . . . . . . . 52 Ergebnisse . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 53 Die Caudal- und Mex-3-Orthologe in Bruchidius und Oncopeltus . . . . . . 53 Posteriore Expression von B.o. caudal während der Embryogenese . . . . . 54 IV Inhaltsverzeichnis B.o. cad spielt eine wichtige Rolle in der Embryonalentwicklung . . . . . . 55 Bruchidius zen wird dorsal im Ei exprimiert . . . . . . . . . . . . . . . . . 57 Expression von Bruchidius mex-3 während der Embryogenese . . . . . . . 58 Bruchidius mex-3-RNAi beeinträchtigt die Segmentierung . . . . . . . . . 60 Posteriore Expression von O.f. caudal während der Embryogenese . . . . . 62 O.f. cad-RNAi führt zu einem frühzeitigen Abbruch der Segmentierung . . 64 Die Analyse des Markergens engrailed nach Oncopeltus cad-RNAi . . . . 65 Die Expression von mex-3 in der Oncopeltus-Embryogenese . . . . . . . . 65 O.f. mex-3 ist wichtig für die anteriore u. posteriore Musterbildung . . . . . 67 Die Expression von engrailed nach mex-3-RNAi in Oncopeltus . . . . . . . 71 Die Expression von even-skipped nach mex-3-RNAi in Oncopeltus . . . . . 71 caudal-Expression nach mex-3-RNAi in Bruchidius u. Oncopeltus . . . . . 75 Diskussion . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 76 Wichtige Rolle für caudal in der Bruchidius- u. Oncopeltus-Embryogenese 76 Die Funktion von mex-3 im Rahmen der Oncopeltus-Embryogenese . . . . 79 B.o. mex-3 wird für die Anlage thorakaler u. abd. Segmente benötigt . . . . 81 Ist das Mex-3/Caudal System innerhalb der Insekten konserviert? . . . . . . 85 Ausblick . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 85 III. Kapitel: mlpt und svb in Tribolium 85 Einleitung . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 86 Rolle der Gapgene in der frühen Embryonalentwicklung von Drosophila . . 86 Gapgen-Homologe in der frühen Embryonalentwicklung von Tribolium . . 87 Die Expression und Funktion von mlpt in Tribolium . . . . . . . . . . . . . 89 Die mlpt-Peptide . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 90 Die Interaktion von Polished-rice/Tarsaless und Shavenbaby in Drosophila . 93 Ziele dieser Arbeit . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 96 Material & Methoden . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 97 Tribolium castaneum: Haltung und Zucht . . . . . . . . . . . . . . . . . . 97 Etablierung des mille-pattes-Überexpressionssystems . . . . . . . . . . . . 97 Durchführung des Hitzeschocks zur Aktivierung der mlpt-Überexpression . 103 Nachweis der mlpt-Überexpression nach dem Hitzeschock via ISH . . . . . 104 V Inhaltsverzeichnis mlpt-RNAi in einer mlpt-Überexpressionslinie . . . . . . . . . . . . . . . . 104 Die Klonierung des Transkriptionsfaktors shavenbaby . . . . . . . . . . . . 105 Pupale Injektion von dsRNA in Tribolium (Posnien et al., 2009) . . . . . . 105 Ergebnisse . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 107 Etablierung des mlpt-Überexpressionssystems in Tribolium . . . . . . . . . 107 Ektopische Expression von mlpt in transgenen mlpt-Linien nach Hitzeschock 107 Die Überexpression von mlpt hat keine offensichtlichen phänotypischen Auswirkungen auf larvale Strukturen . . . . . . . . . . . . . . . . . . . 108 Der mlpt-RNAi Effekt kann durch mlpt-Überexpression in den hsp68-mlptLinien nicht gemildert werden . . . . . . . . . . . . . . . . . . . . 111 Die Expression von shavenbaby in Tribolium . . . . . . . . . . . . . . . . 112 Vergleich zwischen svb- und mlpt-Expression in Tribolium . . . . . . . . . 115 Die Funktion von T.c. svb während der Embryogenese . . . . . . . . . . . 117 Die Analyse von Markergenen nach parentaler svb-RNAi . . . . . . . . . . 119 Diskussion . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 123 Ektopische Expression von T.c. mlpt hat keine phänotypischen Auswirkungen 123 mlpt-Überexpression mildert nicht den RNAi-Phänotyp . . . . . . . . . . . 124 Die Expressionsmuster von T.c. mlpt und T.c. svb unterscheiden sich . . . . 125 svb-RNAi resultiert in ähnlichem Gapgen-Phänotyp wie mlpt-RNAi . . . . 127 Der Einfluss von Svb auf die Regulation anderer Segmentierungsgene . . . 128 Der Einfluss von Svb auf das Segmentpolaritätsgen gooseberry . . . . . . . 130 Shavenbaby - ein neues Gapgen in Tribolium? . . . . . . . . . . . . . . . . 130 Ausblick: Die Funktion von mlpt in Bruchidius obtectus . . . . . . . . . . 132 Anhang 133 Literaturverzeichnis 142 Erklärung 158 Danksagung 159 VI Zusammenfassung Die Segmentierung der Taufliege Drosophila melanogaster wird von einer hierarchischen Genkaskade gesteuert. Ein Großteil der dabei beteiligten Gene spielt auch eine wichtige Rolle in der Embryogenese phylogentisch weit entfernter Insekten. Jedoch lassen sich nicht alle Erkenntnisse, die aus der Untersuchung der Drosophila-Embryogenese gewonnen wurden, auf die Embryonalentwicklung anderer Insektenspezies übertragen, da Drosophila in vielfacher Hinsicht einen abgeleiteten Entwicklungstyp repräsentiert. Die Untersuchung verschiedener Insekten-Modellorganismen dient dazu, alternative Strategien der Embryonalentwicklung zu verstehen und allgemein gültige Prinzipien der Insektenentwicklung abzuleiten. Im Rahmen dieser Arbeit habe ich das Gapgen hunchback im Langkeimkäfer Bruchidius obtectus untersucht (Projekt 1). Durch in-situ-Hybridisierung, Generierung von Knockdown Phänotypen sowie der Analyse des Paarregelgens paired in B.o. hb-RNAi-Embryonen bestätigte sich, dass die Gapgen-Funktion von hunchback an den Entwicklungsmodus der Langkeimentwicklung gekoppelt ist, in Bruchidius also eher der hb-Funktion in Drosophila entspricht als der in Kurzkeiminsekten wie Tribolium. Während hunchback-Homologe in allen bisher untersuchten Insekten gefunden werden konnten, ist Bicoid, das auch für die Repression von Caudal im anterioren Bereich der Fliegen-Embryonen notwendig ist, auf die cyclorrhaphen Fliegen beschränkt. In Tribolium wurde gezeigt, dass das KH-Domänen-Protein Mex-3 die Translation von caudal reprimiert. Meine Analyse der Funktion von Mex-3 im Käfer Bruchidius obtectus und in der Wanze Oncopeltus fasciatus (Projekt 2) zeigte, dass mex-3 auch für die Segmentierung dieser Spezies wichtig ist, dies also wohl den ursprünglichen Mechanismus der Caudal-Repression darstellen dürfte. Schließlich habe ich noch das atypische Gapgen mille-pattes im Kurzkeimer Tribolium untersucht, das für mehrere kleine Peptide codiert (Projekt 3). Das mille-pattes-Homolog polished-rice spielt in der Drosophila-Segmentierung keine Rolle und repräsentiert daher möglicherweise eine Kurzkeim-spezifische Musterbildungsfunktion. Da die Funktionsweise der Mille-pattes-Peptide in Tribolium unbekannt war, habe ich zunächst transgene mlptLinien hergestellt, bei denen durch Hitzeaktivierung mlpt zu bestimmten Zeitpunkten der Embryonalentwicklung überexprimiert werden konnte. Leider zeigte jedoch die Überexpres- VII Zusammenfassung sion von mille-pattes alleine keine phänotypischen Auswirkungen, weshalb die Vermutung nahe lag, dass die Mlpt-Funktion an einen lokalisiert exprimierten Effektor gebunden ist. In Drosophila ist das der Transkriptionsfaktor shavenbaby und ich konnte für T.c. shavenbaby zeigen, dass dieser mit mlpt koexprimiert wird und sein Knockdown zu mlpt-ähnlichen Phänotypen führt. VIII Summary Segmentation in Drosophila melanogaster is regulated by a hierarchical gene cascade. Most of the genes involved also play important roles during embryogenesis of other insect species. However, many aspects of Drosophila embryogenesis are highly derived, i.e. not all findings in this species also apply to other insect taxa. Thus, the investigation of other insect model organisms is necessary in order to identify different strategies of embryonic development and to understand general principles behind diverse modes of insect development. I investigated the Drosophila homolog hunchback in the long-germ beetle Bruchidius obtectus (project 1). In-situ Hybridisation, larval RNAi phenotypes and expression analysis of the pair-rule gene paired in B.o. hb-RNAi knockdown embryos, showed that the gap gene function of B.o. hunchback is more similiar to that of distantly related long-germ Drosophila rather than with the more closely related short-germ Tribolium. Hunchback homologs can be found in all insects studied so far. By contrast, Bicoid which is also responsible for the repression of the gene caudal in the anterior part of the embryo - is restricted to the cyclorrhaphan flies. In Tribolium the KH-domain protein Mex-3 represses caudal translation. My analysis of mex-3 in the beetle Bruchidius obtectus and in the bug Oncopeltus fasciatus (project 2) showed that mex-3 is necessary also for the segmentation process in these species, which supports the idea that Mex-3 represents the ancestral mode of caudal repression in insects. Finally, I investigated the atypical gap gene mille-pattes in the short-germ beetle Tribolium (project 3). mille-pattes codes several small peptides and is in Drosophila not necessary for the segmentation process. Thus, Mille-pattes peptides might represent a feature specific to short-germ segmentation, but their function in Tribolium was unclear. I generated transgenic lines where mlpt can be overexpressed via activation from a heat-shock promoter. However, after heat-shock no phenotypes could be observed at the cuticle level. This could indicate that the function of Mlpt is connected to a spatially regulated effector protein, which would restrict the effect of overexpressing mlpt alone. Based on work in Drosophila, a candidate interacting gene was the transcription factor shavenbaby. I showed that T.c. shavenbaby is coexpressed with mlpt, and that its knockdown results in RNAi effects very similar to those of mlpt RNAi. This indicates that Shavenbaby also in Tribolium segmentation is the likely target gene of Mlpt peptides. IX Abbildungsverzeichnis 1 Übersicht der Embryonalentwicklung in Drosophila . . . . . . . . . . 2 2 Individualentwicklung von Oncopeltus, Bruchidius u. Tribolium . . 9 3 Morphologische Unterschiede männlicher u. weiblicher Bruchidien 17 4 Alignment von HB-Orthologen verschiedener Arthropoden . . . . . 23 5 Bruchidius hb wird maternal im Ovar exprimiert . . . . . . . . . . . 25 6 Die Embryonalentwicklung von Bruchidius . . . . . . . . . . . . . . . 26 7 Die Expression von Bruchidius hb während der Embryogenese . . . 28 8 Segmentverlust nach Bruchidius hunchback RNAi . . . . . . . . . . 30 9 Alignment der konservierten Domänen von Paired-Homologen . . . 32 10 Expression von Bruchidius paired im Wildtyp . . . . . . . . . . . . . 33 11 Expression des Paarregelgens paired in Bruchidius nach hb-RNAi . 35 12 Allgemeine phylogenetische Übersicht der Bilateria . . . . . . . . . 45 13 Unterscheidung von Männchen und Weibchen in Oncopeltus . . . 49 14 Alignment der funktionellen Domänen von Caudal bzw. Mex-3 . . 54 15 Expression von caudal in Bruchidius . . . . . . . . . . . . . . . . . . 55 16 Vergleich von Hoechst gefärbten WT und B.o. cad -RNAi-Embryonen 56 17 Die Expression von Bruchidius zen im Blastoderm . . . . . . . . . . 58 18 Die Expression von mex-3 in der Bruchidius-Embryogenese . . . . 59 19 Beeinträchtigung der Segmentierung nach B.o. mex-3 -RNAi . . . 61 20 Übersicht der Oncopeltus-Embryogenese . . . . . . . . . . . . . . . . 63 21 Posteriore caudal -Expression in Oncopeltus . . . . . . . . . . . . . . 63 22 Posteriore Segmentierungsstörung in Oncopeltus nach cad -RNAi . 66 23 O.f. cad -RNAi führt zur Störung der posterioren Invagination . . . 67 24 Die Expression von mex-3 in der Oncopeltus-Embryogenese . . . . 68 25 Phänotypische Reihe nach mex-3 -RNAi in Oncopeltus . . . . . . . 70 X Abbildungsverzeichnis 26 Beeinträchtigung der posterioren Entwicklung nach O.f. mex-3 -RNAi 72 27 even-skipped -Expression im O.f. WT und nach mex-3 -RNAi . . . . 74 28 Vergleich der Funktion von mex-3 in Bruchidius und Tribolium . . 84 29 Vergleich der Gapgen-Expression zwischen Drosophila u. Tribolium 89 30 Struktur der polycistronischen Tribolium mlpt-RNA . . . . . . . . . 91 31 Zusammenfassung der Dentikelbildung in Drosophila . . . . . . . . 95 32 Übersicht der Klonierungsstrategie von mlpt in den piggyBac Vektor 98 33 mlpt-Überexpression nach Aktivierung des hsp86-mlpt Konstrukts 34 mlpt-Überexpression hat keine Auswirkungen auf Kutikula-Phänotyp111 35 Die Expression von svb in der Embryogenese von Tribolium . . . . 114 36 Doppel-ISH von T.c. gooseberry und T.c. svb . . . . . . . . . . . . 115 37 Vergleich der T.c. mlpt- und T.c. svb-Expressionsdomänen . . . . . 116 38 svb-RNAi-Phänotypen nach parentaler RNAi . . . . . . . . . . . . . 119 39 Tribolium giant-Expression im Wildtyp und nach svb-RNAi . . . . . 121 40 Kernfärbung und gsb-Färbung in svb-RNAi Embryonen . . . . . . . 122 41 Gapgenexpression in Tribolium . . . . . . . . . . . . . . . . . . . . . . 131 42 pSLfa[Tc’hsp5’3’UTR]fa u. pBac[3xP3-DsRedaf] . . . . . . . . . . . 140 43 pBac[3xP3-DsRedTc’hsp5’-mlpt-hsp3’] . . . . . . . . . . . . . . . . . 141 XI 109 Tabellenverzeichnis 1 Degenerierte Primer für das Bruchidius hb-Projekt . . . . . . . . . 19 2 Verteilung der Phänotypen nach Bruchidius hb-RNAi (c=3 µg/µl) 30 3 Primer für das Mex-3/Cad-Projekt . . . . . . . . . . . . . . . . . . . 51 4 Verteilung der Phänotypen nach B.o. mex-3 -RNAi (c=300 ng/µl) 62 5 Verteilung der Phänotypen nach O.f. caudal -RNAi (c=3 µg/µl) . . 65 6 Verteilung der O.f. mex-3 -RNAi Phänotypen (c=2 µg/µl) . . . . . 69 7 Entwicklungsdauer von Tribolium bei unterschiedlichen Temperaturen 97 8 Primer für die Klonierung von T.c. shavenbaby . . . . . . . . . . . . 105 9 Embryonalen Injektion zur Generierung der hsp68-mlpt-Linien . . . 107 10 Verteilung des Stummelbeinphänotyps in der hsp68-mlpt-Linie 8 . 110 11 Auswirkungen der mlpt-Überexpression auf den mlpt-RNAi-Effekt 112 12 Verteilung der svb-RNAi Phänotypen (c=2 µg/µl). . . . . . . . . . 118 XII Abkürzungen A abdominales Segment Abd A Abdominal A Ag Auge Ant Antenne Antp Antennapedia B.o. Bruchidius obtectus bw backward cad caudal Dfd Deformed en engrailed eve even-skipped fw forward gsb gooseberry hb hunchback Ic interkalares Segment ISH in-situ-Hybridisierung Lb Labium Lbr Labrum mex-3 muscle-excess 3 Md Mandibel mlpt mille-pattes Mx Maxille ORF Open Reading Frame O.f. Oncopeltus fasciatus pal-1 posterior alae in males XIII Abkürzungen prd paired PCR Polymerase Chain Reaction pri polished-rice RT Raumtemperatur SB San Bernadino Scr Sex combs reduced svb shavenbaby T thorakales Segment T.c. Tribolium castaneum Ubx Ultrabithorax WT Wildtyp XIV Allgemeine Einleitung Allgemeine Einleitung Die vorliegende Arbeit beschäftigt sich mit drei verschiedenen Themenschwerpunkten, die in unterschiedlichen Kapiteln vorgestellt werden. Alle bearbeiteten Projekte verbindet die Fragestellung, wie Segmentierung auf molekularer Ebene im Zuge der Embryonalentwicklung in verschiedenen Insekten funktioniert und welche allgemeingültigen Prinzipien sich für die Embryogenese der Insekten daraus ableiten lassen. Segmentierung in Drosophila Unter Segmentierung versteht man die Unterteilung eines Organismus entlang der anterioren-posterioren Achse in sich wiederholende strukturelle Einheiten. Sie ist vermutlich eine Anpassung an die Notwendigkeit, physiologische Prozesse in größeren Vielzellern besser zu koordinieren (Couso, 2009). Die Vorgänge welche nötig sind, dass aus einem befruchteten Insektenei eine segmentierte Larve entstehen kann, sind am besten in der Fruchtfliege Drosophila melanogaster (Ordnung: Diptera) verstanden. Sie sollen im Folgenden sowohl unter dem morphologischen als auch molekularen Gesichtspunkt beschrieben werden. Auf morphologischer Ebene finden zunächst im Zentrum des befruchteten DrosophilaEies zahlreiche Kernteilungen statt (Abb.1: A). Der Großteil der Zellkerne wandert an die Peripherie des Eies und bildet das Blastoderm aus. Zu diesem frühen Zeitpunkt sind die Zellkerne von keiner Zellmembran umgeben (Hartenstein, 1993). Die noch im Eiinneren verbleibenden Kerne werden polyploid und unterstützen als sogenannte Vitellophagen die Ernährung des zukünftigen Embryos (Callaini et al., 1990). Wenn die Kerne des Blastoderms schließlich mit Zellmembranen umgeben werden, und somit das Stadium des zellularisierten Blastoderms erreicht ist, sind bei einer Temperatur von 25 °C , seit der Befruchtung des Eies gerade mal drei Stunden vergangen. Daran schließt sich die Ausbildung des Keimrudiments auf der ventralen Eiseite entlang der anterior-posterioren Achse an, das im Zuge der Keimstreifstreckung nach dorsal-anterior auswächst. Der gestreckte Keimstreif verkürzt sich erneut, die lateralen Seiten des Embryos beginnen zu wachsen und abschließend wird der restliche Dotter internalisiert, während sich die lateralen Seiten auf der dorsalen Seite des Embryos schließen. Somit ist nach Ablauf von 13 Stunden die larvale Grundgestalt ausgebildet und nach einer Entwicklung von insgesamt 24 Stunden ist die Drosophila-Larve schlüpfbereit (Foe et al., 1993; Hartenstein, 1993). 1 Allgemeine Einleitung Was passiert nun im Zuge dieser morphologischen Entwicklung auf molekularer Ebene? Eine sogenannte hierarchische Genkaskade (Abb.1: B) führt zur Aktivierung verschiedener Transkriptionsfaktoren und ist für die Segmentierung in Drosophila verantwortlich (RiveraPomar und Jäckle, 1996). Die ersten Schritte werden bereits im Rahmen der Oogenese eingeleitet, wenn die sich entwickelnde Oocyte ihre initiale Polarität erhält, die beispielsweise festlegt, wo im Ei sich der Kopf bilden wird (Roth und Lynch, 2009). Diese ersten Schritte beinhalten die Lokalisierung von maternaler bicoid-mRNA am Cytoskelett des anterioren Eipols (Berleth et al., 1988) und am posterioren Ende die Positionierung von nanos-mRNA via Oskar (Ephrussi et al, 1991). Neben diesen lokalisierten, maternalen mRNAs gibt es auch homogen verteilte mRNA im Ei, wie das z.B. für maternales hunchback und caudal gezeigt werden konnte (Tautz et al., 1987; Mlodzik und Gehring, 1987). Abb. 1: Übersicht der Embryonalentwicklung in Drosophila A) Die morphologische Zusammenfassung skizziert, wie sich aufgrund mehrerer Kernteilungen aus der Zygote, über das Kernscharstadium, das einschichtige Blastoderm bildet. Nach der Ausbildung der Zellmembranen um die Zellkerne, formt sich das Keimrudiment, das im Zuge der Keimstreifstreckung zunächst nach dorsal anterior auswächst (roter Pfeil) und schließlich im Keimstreifrückzug wieder verkürzt wird (roter Pfeil). Parallel dazu strecken sich die lateralen Seitenwände nach dorsal (grüne Pfeile) und wachsen dort zusammen (nach Foe et al., 1993; Hartenstein; 1993). B) An der Spitze der hierarchischen Segmentierungskaskade von Drosophila stehen die maternalen Effektgene, welche die Gapgene von anterior nach posterior aktivieren. Die Paarregelgene werden noch vor der Zellularisierung des Blastoderms exprimiert, die Segmentpolaritätsgene danach, während der Keimstreifstreckung (nach Gilbert, 2006). 2 Allgemeine Einleitung Nach der Befruchtung und Eiablage löst sich die lokalisierte bicoid-mRNA vom anterioren Pol und bildet an der Peripherie des Eies einen von anterior nach posterior verlaufenden Gradienten, der sich nach der Translation auf der Proteinebene widerspiegelt (Lipshitz, 2009). Am posterioren Pol wird die lokalisierte nanos-mRNA ebenfalls translatiert und ein zum Bicoid-Gradienten gegenläufiger Nanos-Protein-Gradient bildet sich aus (Gavis und Lehmann, 1992). Die Translation von maternalem hunchback wird von Nanos am posterioren Eipol reprimiert (Wreden et al., 1997), weshalb sich die Hunchback-Expression zunächst auf den anterioren Bereich des Eies beschränkt. Am anterioren Eipol ist Bicoid für die translationale Repression von caudal zuständig, sodass von posterior nach anterior ein CaudalProtein-Gradient entsteht (Mlodzik und Gehring, 1987). Zusammengefasst basiert die initiale Musterbildung in Drosophila aus verschiedenen, gegenläufigen Proteinexpressionsdomänen, welche nun die nächste Stufe der Segmentierungskaskade regulieren, die zygotischen Gapgene (Driever und Nüsslein-Volhard, 1988). Gapgene wie beispielsweise hunchback, Krüppel, knirps und giant werden entlang der anterioren-posterioren Achse in breite, sich teilweise überlappende Bereiche exprimiert (Jäckle et al., 1986, Rivera-Pomar et al., 1995). Die Klassifizierung eines Gens als Gapgen bezieht sich zum einen auf die Deletion mehrerer aneinander grenzender Segmente nach dem Verlust dieses Gens und zum anderen, auf seine Eigenschaft die Expression der nachgeschalteten Paarregelgene zu steuern (Hülskamp und Tautz, 1991). Paarregelgene wie even-skipped (Frasch et al., 1987), fushi-tarazu (Hafen et al., 1984) und paired (Gutjahr et al., 1993) werden ebenfalls im Drosophila-Blastoderm aktiviert, und spiegeln durch ihre Expression zum ersten Mal im Laufe der Entwicklung die Unterteilung des Embryos in sich wiederholende Einheiten wider, da sie in sieben aufeinander folgenden Streifen exprimiert sind. Diese primären Paarregelgene werden durch eine verschiedene Kombination der Transkriptionsfaktoren von maternalen Genen und Gapgenen gesteuert. Ein gut untersuchtes System stellt dabei die Regulierung des zweiten even-skipped-Streifens dar. Für die Aktivierung der Expression ist die Bindung von Bicoid und Hunchback im Promotor Bereich notwendig; für die Ausbildung der anterioren und posterioren Grenzen des Streifens, eine Repression durch die zusätzliche Bindung der Transkriptionsfaktoren Krüppel und Giant (Small et al., 1992). Während der Keimstreifstreckung aktivieren die Paarregelgene die Transkription der Segmentpolaritätsgene u.a. engrailed (Kornberg, 1981) und wingless (Heuvel et al., 1989), die für die Ausbildung und Aufrechterhaltung der vierzehn 3 Allgemeine Einleitung Segmentgrenzen benötigt werden. Als letzter Schritt ist die Festlegung der Segmentidentität wichtig, welche durch die Hoxgene realisiert wird (Lewis, 1978). Die Drosophila-Segmentierungskaskade zeigt anschaulich, dass eine hierarchische Genaktivierung für die Gliederung des Embryos notwendig ist. Sie ist unidirektional, das heißt, dass die Gene die oben in der Segmentierungskaskade stehen meistens eine aktivierende oder reprimierende Wirkung auf alle nachfolgenden Gene haben, umgekehrt ist jedoch keine Regulierung möglich (Rivera-Pomar und Jäckle, 1996). Hingegen ist eine starke Interaktion der Gene auf den jeweiligen Segmentierungsebenen untereinander vorhanden (Vincent und Lawrence, 1994; Sanchez und Thieffry, 2003; Jäger et al., 2004). Kurzkeim- und Langkeimentwicklung Die soeben beschriebene Segmentierungskaskade von Drosophila im Rahmen der Embryonalentwicklung ist nicht repräsentativ für die Entwicklung aller Insekten. Drosophila gehört zu den sogenannten Langkeimern, was bedeutet, dass bereits im Blastoderm Kopf, Thorax und abdominale Segmente mehr oder weniger gleichzeitig angelegt werden (Akam, 1987; Davis und Patel, 2002). Im Gegensatz dazu steht die Kurzkeimentwicklung welche beinhaltet, dass bis zum Ende des syncytialen Blastoderms nur die anteriorsten Segmente, also Kopf und meistens thorakale Segmente angelegt worden sind, während alle bis dahin noch fehlenden Segmente nach der Gastrulation sequentiell aus einer posterioren Wachstumszone in zellulärer Umgebung gebildet werden (Davis und Patel, 2002). Folglich muss in Kurzkeimern die Segmentierung, vor allem für die der posterioren Segmente, einem anderen Mechanismus unterliegen (Liu und Kaufman, 2005; Choe et al., 2006), als dem aus Drosophila bekannten, der als Grundlage die freie Diffusion von Transkriptionsfaktoren im syncytialen Blastoderm besitzt (Hülskamp und Tautz, 1991; Liu und Kaufman, 2005). Die Kurzkeimentwicklung wird als der ursprüngliche Segmentierungsmechanismus angesehen, da alle hemimetabolen Insekten die Kurzkeimentwicklung durchlaufen und die Segmentierung mittels der Langkeimentwicklung auf die später entstandenen holometabolen Insekten beschränkt ist (Patel et al., 1994). Da in den holometabolen Insektenordnungen wie Hemiptera, Coleoptera und Lepidoptera sowohl Kurzals auch Langkeimer beschrieben worden sind, wird angenommen, dass die Langkeimentwicklung mehrmals unabhängig voneinander entstanden ist (Davis und Patel, 2002; Savard et al., 2006). 4 Allgemeine Einleitung Segmentierung in anderen Insektenspezies Nach der Entschlüsselung der grundlegenden Segmentierungsmechanismen in Drosophila, ging man verstärkt dazu über, Drosophila-Orthologe der Segmentierung auch in anderen Insektenarten zu untersuchen (Peel et al., 2005; Rosenberg et al., 2009; Lynch et al., 2012). Für alle großen Gruppen der holometabolen Insekten gibt es mittlerweile mindestens einen sehr gut untersuchten Vertreter, wie z.B. Nasonia vitripennis (Hymenoptera), Tribolium castaneum (Coleoptera), Bombyx mori (Lepidoptera) und alternativ zu Drosophila für die Dipteren, Megaselia abdita oder Episyrphus balteatus (Werren und Loehlin, 2009; Klingler, 2004; Terenius et al., 2010; Stauber et al., 2000; Lemke et al., 2010). Auch unter den hemimetabolen Insekten sind Modellorganismen wie Oncopeltus fasciatus (Hemiptera) oder Gryllus bimaculatus (Orthoptera) bereits etabliert (Liu und Kaufman, 2004; Mito und Noji, 2006). Anhand einiger Beispiele soll veranschaulicht werden, dass die Orthologe der Drosophila-Segmentierungskaskade in diesen Spezies teilweise in ihrer Expression und Funktion zu Drosophila identisch sind – trotz einer größeren phylogenetischen Distanz – dass aber auch zum Teil gravierende Unterschiede bestehen können (Peel et al., 2005). Die für die Segmentidentiät wichtigen Hoxgene, sind innerhalb der Insekten stark konserviert (Hughes und Kaufman, 2002). Auch Segmentierungsgene wie engrailed und wingless fungieren außerhalb Drosophila als Segmentpolaritätsgene was zahlreiche Studien in Gryllus (Niwa et al., 1997; Miyawaki et al., 2004), Oncopeltus (Liu und Kaufman, 2004; Angelini und Kaufman, 2005), Nasonia (Pultz et al., 1999), Tribolium (Brown et al., 1994; Nagy und Carroll, 1994) und Manduca (Kraft und Jäckle, 1994; Saenko et al., 2010) belegen. Auf der Ebene der Paarregelgene machen sich jedoch bereits einige Unterschiede in den verschiedenen Insektenspezies bemerkbar. In den holometabolen Organismen kommen hierbei die unterschiedlichen Segmentierungsmodi – sprich Kurz- und Langkeimentwicklung – zum Tragen. In Drosophila und im Käfer Bruchidius werden sieben bzw. sechs even-skippedStreifen gleichzeitig im syncytialen Blastoderm gebildet. Später, im Zuge der Ausbildung des Keimrudiments wird eve in beiden Organismen segmental exprimiert (Frasch et al., 1987; Schnellhammer, Diplomarbeit). Im Kurzkeimkäfer Tribolium sind bis zum Ende des blastodermalen Stadiums nur die ersten drei even-skipped-Streifen vorhanden, während die noch fehlenden nach der Gastrulation, aus der Wachstumszone heraus gebildet werden (Patel et al., 1994). Zwar ist die Funktion von Tribolium eve als Paarregelgen konserviert, jedoch ist auf- 5 Allgemeine Einleitung grund des unterschiedlichen Segmentierungsmodus, ein zeitlicher Unterschied hinsichtlich des Expressionsmusters zu beobachten. In der hemimetabolen Wanze Oncopeltus fasciatus, ebenfalls ein Kurzkeimer, wird im Blastoderm zunächst eine breite eve-Expressionsdomäne angelegt, aus der anschließend sechs eve-Streifen hervorgehen (Liu und Kaufman, 2005a). Auf den ersten Blick erscheint dies widersprüchlich, da im Oncopeltus-Blastoderm die Segmentierung nur bis einschließlich T3 stattfindet, aber es stellte sich heraus, dass Oncopeltus eve nicht als Paarregelgen fungiert, sondern eine frühe Funktion als Gapgen und eine spätere als Segmentpolaritätsgen inne hat (Liu und Kaufman, 2005a). Bei der Untersuchung der Gapgen-Homologen erkannte man, dass auch in dieser Genklasse gewisse Unterschiede bezüglich der Funktion in den verschiedenen Insektenspezies bestehen können. In Drosophila wird Krüppel im Bereich der zukünftigen thorakalen und der ersten fünf abdominalen Segmente im Blastoderm exprimiert (Knipple et al., 1985). Bei Krüppel-Mutanten kann eine Deletion der Segmente von T1 bis einschließlich der anterioren Grenze von A5 beobachtet werden (Wieschaus et al., 1984). In den Kurzkeimern Tribolium und Oncopeltus ist die Krüppel-Expressionsdomäne kleiner und erstreckt sich ausschließlich über die drei thorakalen Segmente (Liu und Kaufman, 2004; Cerny et al., 2005). Was die Funktion betrifft, resultiert Krüppel-RNAi in Oncopeltus in dem aus Drosophila bekannten Gapphänotyp, welcher die Deletion des zweiten thorakalen Segments bis einschließlich des vierten abdominalen Segments beinhaltet (Liu und Kaufman, 2004). In Tribolium hingegen ist der Krüppel-RNAi Effekt im anterioren Bereich des Embryos eine homeotische Transformation thorakaler Segmente hin zu gnathaler Identität, welche mit einer starken posterioren Verkürzung der Embryonen einhergeht (Cerny et al., 2005). Die beobachtete homeotische Transformation im thorakalen Bereich von Tribolium-Embryonen nach Krüppel-RNAi erklärt sich dadurch, dass Krüppel einen regulatorischen Einfluss auf die homeotischen Gene Dfd, Scr und Ubx ausübt (Cerny et al., 2005). Auch in Drosophila konnte eine Regulation der Hoxgene durch Krüppel gezeigt werden (Harding und Levine, 1988), allerdings werden mögliche homeotische Transformationen in Drosophila Krüppel-Mutanten, von den bereits oben beschriebenen Deletionen überdeckt (Wieschaus et al., 1984; Preiss et al., 1985). Ähnlich liegt der Fall in dem aus Drosophila bekannten Gapgen hunchback, das in Drosophila in einer breiten von anterior nach posterior verlaufenden Domäne exprimiert wird und dessen Verlust bei den stärksten Phänotypen in der Deletion gnathaler, thorakaler und abdominaler Segmente resultiert (Lehmann und Nüsslein-Volhard, 1987, Tautz und Pfeifle, 6 Allgemeine Einleitung 1989; Wu et al., 2001). Hier scheint die Funktion in Abhängigkeit des Segmentierungsmodus konserviert zu sein. Im Langkeimer Nasonia wird hunchback ebenfalls in einer breiten, anterioren Domäne exprimiert (Pultz et al., 2005). Ein Verlust von Nasonia hunchback führt zu einem kanonischen Gapphänotyp, so dass alle Kopfsegmente - bis auf das Labrum - sowie ein Großteil der abdominalen Segmente nicht angelegt werden (Pultz et al., 1999). In den Kurzkeimern Tribolium und Oncopeltus ist hunchback jeweils anterior in einer Domäne exprimiert, welche die Kopfanlagen umspannt, sowie in der posterioren Wachstumszone (Wolff et al., 1995; Liu und Kaufman, 2003). hunchback-RNAi führt in beiden Organismen nicht zu dem aus Langkeimern bekannten kanonischen Gapphänotyp, sondern zu einer Transformation thorakaler in abdominale Strukturen, ebenfalls aufgrund einer Fehlregulation der Hoxgene, sowie einer drastischen Verkürzung der Embryonen (Liu und Kaufman, 2003; Marques-Souza et al., 2008). Das bekannteste Beispiel, dass die Segmentierung in Drosophila nicht repräsentativ für die frühe Musterbildung in Insekten ist, stellt Bicoid dar. Bicoid ist einerseits bedeutend für die Aktivierung der Transkription von Segmentierungsgenen und zum anderen für die translationale Repression von caudal-mRNA im anterioren Bereich der Fliegenembryonen (Ephrussi und St Johnston, 2004). Jedoch ist Bicoid erst unmittelbar vor der Radiation der cyclorrhaphen Fliegen aus einer Duplikation des Hox-3-Gens hervorgegangen (Stauber et al., 1999) und deswegen neben Drosophila, auf die höheren Dipteren wie beispielsweise Episyrphus und Megaselia beschränkt (Lemke et al., 2010; Stauber et al., 2000). Was die Regulation von caudal außerhalb der cyclorrhaphen Fliegen angeht, konnte gezeigt werden, dass im Käfer Tribolium das KH-Domänen-Protein Mex-3 die caudal-Translation inhibiert. In der frühen Musterbildung von Drosophila spielt mex-3 jedoch keine Rolle (Schoppmeier et al., 2009). Die soeben aufgeführten einzelnen Beispiele sollen zeigen, dass das Drosophila-System nicht auf die Entwicklung aller Insekten anwendbar ist. Ein langfristiges Ziel ist es herauszufinden, welche allgemeingültigen Prinzipien hinter der Segmentierung von Insekten stehen. Ein besonderes Augenmerk liegt dabei auf den bereits erwähnten, unterschiedlichen Segmentierungsmechanismen von Kurzkeimern und Langkeimern. Mit der Aufklärung der grundlegenden Mechanismen der Kurz- und der Langkeimentwicklung, wäre es dann ebenfalls möglich zu untersuchen, welche Änderungen des genetischen Netzwerks im Laufe der Evolution stattgefunden haben, um aus einem Kurzkeimentwicklungsmodus einen Langkeimentwicklungsmodus zu entwickeln (Liu und Kaufman, 2005b). 7 Allgemeine Einleitung Übersicht der Projekte und Modellorganismen Im Rahmen dieser Doktorarbeit wurden drei unterschiedliche Fragestellungen bearbeitet: Bisherige Daten deuten darauf hin, dass die Funktion von hunchback an den Entwicklungsmodus gekoppelt ist, da wie bereits erläutert, in Langkeimern wie Nasonia und Drosophila, hunchback als klassisches Gapgen fungiert (Pultz et al., 2005; Lehmann und NüssleinVolhard, 1987; Tautz und Pfeifle, 1989), während in den Kurzkeimern Oncopeltus und Tribolium, hunchback bei der Festlegung der Segmentidentität eine dominante Rolle spielt und die Generierung der abdominalen Segmente dirigiert (Liu und Kaufman, 2003; Marques-Souza et al., 2008). Der erste Teil der Arbeit beschäftigt sich mit der Untersuchung der Expression und Funktion des Gapgens hunchback im Langkeimkäfer Bruchidius (Abb.2: B). Aufgrund der nahen phylogenetischen Verwandtschaft zum Kurzkeimkäfer Tribolium, soll damit die Frage beantwortet werden, ob die hunchback-Funktion tatsächlich an den Entwicklungsmodus gekoppelt ist oder ob die aus Tribolium bekannte hunchback-Funktion typisch für die Ordnung der Käfer ist. Der zweite Teil der Arbeit widmet sich der Fragestellung, ob das in Tribolium gefundene Mex-3/Cad System innerhalb der Insekten konserviert ist und somit, im Gegensatz zum Bicoid/Cad System, den ursprünglichen Mechanismus der caudal-Repression der Insekten darstellt (Schoppmeier et al., 2009). Als Modellsysteme dienen hierfür der Käfer Bruchidius obtectus und die Wanze Oncopeltus fasciatus (Abb.2: A). Schließlich soll auch die Wirkungsweise des neu entdeckten Gapgens mille-pattes in Tribolium (Abb.2: C) untersucht werden (Savard et al., 2006), das in der frühen DrosophilaSegmentierung keine Rolle spielt und somit einen Beitrag zum weiteren Verständnis der Kurzkeimsegmentierung in Tribolium liefern könnte. 8 Allgemeine Einleitung Abb. 2: Individualentwicklung von Oncopeltus, Bruchidius u. Tribolium Die Eier von A) Oncopeltus fasciatus, B) Bruchidius obtectus und C) Tribolium castaneum. Während die hemimetabole Entwicklung in Oncopeltus auf einer Aneinanderreihung nymphaler Stadien beruht (D, G), durchläuft Bruchidius im Rahmen der holometabolen Entwicklung verschiedene larvale Stadien E) die in einer Puppenform resultieren H). Das gleiche gilt für Tribolium: Die larvale Phase F) grenzt an ein Puppenstadium an I). J), K) und L) zeigen die adulte Morphologie der jeweiligen Spezies. 9 I. Kapitel: hunchback in Bruchidius obtectus Einleitung Das folgende Kapitel hat als Themenschwerpunkt die Analyse der Funktion des aus Drosophila bekannten Gapgens hunchback in Bruchidius obtectus. Hunchback ist innerhalb der Metazoen konserviert und wurde in zahlreichen Organismen sehr genau untersucht: Von Anneliden und Nematoden, über diverse Insektenarten bis hin zu den Wirbeltieren. Seine Funktion bei der Ausbildung extraembryonaler Membranen sowie der Spezifizierung des zentralen Nervensystems scheint zwischen den Lophotrochozoa und den Ecdysozoa konserviert zu sein (Pinnell et al., 2006). Das Säuger Homolog von Hunchback heißt Ikaros und ist in der Hämatopoese (Georgopoulos et al., 1994) sowie der Unterdrückung von Tumoren involviert (Mullighan et al., 2008). Speziell innerhalb der Gruppe der Insekten spielt hunchback eine zentrale Rolle in der Segmentierung, welche im Folgenden an einigen Beispielen dargestellt werden soll. Die Funktion von Hunchback in Langkeim-Insekten Hunchback ist ein Zinkfinger-Transkriptionsfaktor (Tautz et al., 1987) und wurde ursprünglich während des Drosophila-Screens Anfang der 80 iger Jahren entdeckt (Nüsslein-Volhard und Wieschaus, 1980). Die Expression von hunchback-RNA ist zunächst maternalen Ursprungs (Schröder et al., 1988) und im frühen Embryo ubiquitär nachweisbar (Tautz und Pfeifle, 1989). Auf Proteinebene bildet sich eine von anterior nach posterior verlaufende Expressionsdomäne aus, da die posteriore Translation von hunchback durch den Nanos/Pumilio-Komplex reprimiert wird (Irish et al., 1989; Wreden et al., 1997). Die zygotische Transkription von Hunchback findet zum einen durch die Aktivierung von Bicoid statt (Struhl et al., 1989, Driever et al., 1989), zum anderen ist Hunchback in der Lage seine eigene Transkription autoregulatorisch zu aktivieren (Simpson-Brose et al., 10 I. Kapitel: Bruchidius hunchback - Einleitung 1994). Auch konnte der Einfluss von Hunchback auf weitere Gapgene wie Krüppel und knirps (Hülskamp et al., 1990), Paarregelgene (Small et al., 1992; Rivera-Pomar und Jäckle, 1996), Segmentpolaritätsgene (Zuo et al., 1990) und Hoxgene (Harding und Levine, 1988) gezeigt werden, so dass Hunchback auf alle Ebenen der Drosophila-Segmentierungskaskade wirkt. Im Zuge der fortschreitenden Embryogenese wird die breite anteriore Expressionsdomäne durch einen schmaleren Streifen ersetzt, welcher die Region des Parasegments 4 abdeckt und auch am posterioren Eipol findet nun eine hb-Expression statt (Tautz und Pfeifle, 1989; Wu et al., 2001). In Abhängigkeit davon, welche hb-Allele betroffen sind, können unterschiedliche Effekte nach dem Verlust beobachtet werden (Hülskamp et al., 1994). In schwachen Drosophila hbMutanten wurde eine Transformation von Segmenten beschrieben (Lehmann und NüssleinVolhard, 1987), was durch die Missexpression der Hoxgene Antp und Ubx erklärt wird (Harding und Levine, 1988). Dieser homeotische Effekt wird in stärkeren Phänotypen überdeckt, da sich bei einem Verlust von maternalem und zygotischem hunchback Larven entwickeln, denen das Labium und thorakale Segmente fehlen. Zusätzlich können am posterioren Ende Fusionen zwischen dem siebten und achten abdominalen Segment beobachtet werden (Lehmann und Nüsslein-Volhard, 1987). Diese Segmentdeletionen sind auf eine frühe Störung der Expression der Paarregelgene zurückzuführen, da beispielsweise paired anterior statt in Streifen, in einer breiten Domäne exprimiert wird und posterior der siebte paired-Streifen vollständig fehlt (Gutjahr et al., 1993). Ein ähnliches Bild für die Expression und Funktion von hunchback zeichnet sich in Megaselia abdita ab, ebenfalls eine Vertreterin der cyclorrhaphen Fliegen wie Drosophila. Eine zunächst ubiquitäre hb-Expression im Ei wird von einer breiten anterioren und einer schmaleren posterioren Expressionsdomäne abgelöst. Nach hb-RNAi weisen die Larven Deletionen in den gnathalen, thorakalen und den Segmenten A1-A3 sowie dem achten abdominalen Segment auf (Stauber et al., 2000). Welche Funktion erfüllt hunchback nun außerhalb der Dipteren? In der Schlupfwespe Nasonia vitripennis (Hymenoptera), ein Langkeimer, wird hunchback ebenfalls maternal exprimiert und eine gleichmäßige hb-Expression ist in frühen Embryonalstadien zu finden. Im Blastodermstadium erscheinen ebenfalls eine breite anteriore Expressionsdomäne sowie eine schmale posteriore Domäne. Zu diesem Zeitpunkt kann HB-Protein nur im anterioren Bereich des Embryos nachgewiesen werden, im posterioren Bereich findet - ebenfalls wie 11 I. Kapitel: Bruchidius hunchback - Einleitung in Drosophila - eine hb-Repression auf translationaler Ebene durch Nanos statt (Pultz et al., 2005; Lynch und Desplan, 2010). Der Verlust von maternalem und zygotischem hunchback führt zu drastischeren Phänotypen als das in Drosophila der Fall ist. Für Nasonia wurde beschrieben, dass alle anterioren Strukturen, bis auf das Labrum, sowie Pro-, Meso- und Metathorax, die abdominalen Segmente A7/A8 und die posterioren Strukturen fehlen (Pultz et al., 2005). Unterstützt werden diese Befunde auf molekularer Ebene durch die fehlende Expression des Segmentpolaritätsgens engrailed im anterioren Bereich von hb-mutanten NasoniaEmbryonen (Pultz et al., 1999). Eine Erklärung für den schwächeren Effekt von hunchback auf die Drosophila-Embryogenese ist, dass Bicoid - welches ausschließlich in der Gruppe der cyclorrhaphen Fliegen vorkommt (Stauber et al., 1999) - einen Teil der anterioren Musterbildungsfunktion von hunchback übernommen hat, da ein Knockout von Drosophila bicoid und hunchback in einem identischen Deletionsmuster wie in Nasonia hb-Mutanten resultiert (Pultz et al., 2005). Zusammengefasst handelt es sich bei dem hunchback-Phänotyp in den eben beschriebenen Langkeiminsekten um einen typischen kanonischen Gapphänotyp, da die Expressionsdomänen auf RNA- und Proteinebene mit der Deletion aneinandergrenzender Segmente auf Kutikulaebene zusammen fallen. Die Funktion von Hunchback in Kurzkeim-Insekten Zwar spielt hunchback auch in der Segmentierung der Kurzkeim-Insekten eine wichtige Rolle, jedoch unterscheidet diese sich von der soeben skizzierten Funktion in den LangkeimInsekten: Maternales hb-Transkript kann auch in Tribolium nachgewiesen werden, genau wie eine frühe anteriore und eine posteriore Domäne (Wolff et al., 1995; Wolff et al., 1998). Allerdings ist die anteriore Expressionsdomäne in Tribolium nicht homolog zu derjenigen von Langkeim-Insekten, da im anterioren Bereich von Tribolium kein embryonales Gewebe angelegt wird, sondern die extraembryonale Serosa (Falciani et al., 1996). Die posteriore Expressionsdomäne splittet sich im Zuge der Embryogenese auf und wird in zwei breiten Domänen im Keimrudiment exprimiert, welche die Region des Gnathums sowie des ersten thorakalen Segments abdecken. Während der Keimstreifstreckung wird hunchback in der posterioren Wachstumszone von Tribolium, sowie in einem neuronalen Muster exprimiert (Wolff et al., 1995; Wolff et al., 1998). In starken hb-RNAi-Phänotypen wird eine Transfor12 I. Kapitel: Bruchidius hunchback - Einleitung mation von gnathalen und thorakalen Segmenten hin zu abdominaler Identität beschrieben. Dieser Transformationsphänotyp ist zusätzlich von der sukzessiven Deletion posteriorer Segmente begleitet. Dementsprechend findet man in solchen Embryonen keinen Verlust der anterioren Paarregelexpressionsmuster wie paired oder even-skipped, dafür ist die Expression der Hoxgene Antp, Ubx und Abd A nach anterior verschoben (Marques-Souza et al., 2008). In diesem Falle liegt also eine Kombination aus einem anterioren Transformationsphänotyp mit einer posterioren Deletion vor. Dass dieser anterior vor der Expressionsdomäne beobachtete Transformationsphänotyp charakteristisch für die Funktion von hunchback in Kurzkeimern ist, spiegelt sich auch in den Studien zur hb-Expression und -Funktion in den Insekten Oncopeltus und Gryllus wider (Liu und Kaufman, 2004, Mito et al., 2005). Die zunächst homogene Expression von Oncopeltus hb wird mit der Ausbildung des Blastoderms auf eine zentrale Expressionsdomäne beschränkt, welche mit der Region der zukünftigen Mandibeln, Maxillen sowie des Labiums zusammen fällt. Noch im Blastodermstadium verschwindet diese Domäne wieder und im jungen Keimstreif ist keine anteriore hunchbackExpression mehr vorhanden, jedoch tritt im Bereich der posterioren Wachstumszone eine neue Expressionsdomäne auf. Die nach parentaler hb-RNAi erhaltenen Phänotypen sind denen von Tribolium sehr ähnlich. Es wurde eine Transformation der gnathalen und thorakalen Segmente hin zu abdominaler Identität beobachtet, die von einer Expansion des Hoxgens Abd A hervorgerufen wird. Zusätzlich tritt in stärkeren Phänotypen eine Verkürzung des Abdomens auf (Liu und Kaufman, 2004). Von Gryllus ist bekannt, dass hunchback im Blastoderm in einem Streifen exprimiert wird, welcher sich vom mandibulären bis zum labialen Segment erstreckt und während der Keimstreifstreckung tritt eine posteriore Expression in der Wachstumszone auf (Mito et al., 2005). Die funktionale Analyse mittels RNAi resultiert ebenfalls in einer homeotischen Transformation der gnathalen und der thorakalen Segmente hin zu abdominaler Identität und auch in Gryllus hb-RNAi-Embryonen sind posteriore Verkürzungen zu finden. Das Markergen even-skipped wird in den transformierten anterioren Segmenten wie im Wildtyp exprimiert, allerdings ist eine Verschiebung des Hoxgens Abd A vom ersten abdominalen Segment nach anterior in hb-RNAi-Embryonen zu beobachten (Mito et al., 2005). Auch die Expressionsdaten für die Orthopteren Schistocera und Locusta legen eine ähnliche Funktion von hunch13 I. Kapitel: Bruchidius hunchback - Einleitung back nahe - eine schwache Expression in den Kopflappen und gnathalen Segmenten bis einschließlich dem ersten thorakalen Segment - wobei für diese beiden Organismen noch keine funktionellen Analysen vorhanden sind (Patel et al., 2001). Zusammenfassend für die Rolle von hunchback in der Kurzkeimentwicklung ergibt sich somit das Bild eines nicht kanonischen Gapgens. Statt Segmentdeletionen im Bereich der anterioren hb-Expression, wie das in den Langkeim-Insekten beschrieben worden ist, kommt es in starken hb-Phänotypen von Kurzkeiminsekten, lediglich zu Segmenttransformationen, die sich zudem über die ursprüngliche Expressionsdomänen hinaus erstrecken sowie zur Deletion eines Großteils der abdominalen Segmente (Marques-Souza et al., 2008). Ziele dieses Projekts Für die Beschreibung der hunchback-Funktion in Kurz- und Langkeimorganismen wurde in den oberen Abschnitten eine kleine Auswahl an untersuchten Modellorganismen vorgestellt welche zeigt, dass die Funktion dieses Gapgens bereits recht gut verstanden ist. Nun stellt sich die Frage, welche zusätzlichen Erkenntnisse die Untersuchung von hunchback im Bohnenkäfer Bruchidius obtectus noch bringen kann. Die in den oberen Abschnitten beschriebene Hunchback-Funktion in verschiedenen Kurzund Langkeimern verdeutlicht, dass Hunchback wahrscheinlich in Abhängigkeit des Segmentierungsmodus eine unterschiedliche Funktion in der frühen Embryonalentwicklung erfüllt (Marques-Souza et al., 2008). Obwohl Nasonia (Hymenoptera) und Drosophila (Diptera) phylogenetisch weit auseinander liegen (Wiegmann et al., 2009) und den Segmentierungsmechanismus der Langkeimentwicklung vermutlich konvergent ausgebildet haben, ist die Hunchback-Funktion konserviert (Pultz et al., 2005; Lehmann und Nüsslein-Volhard, 1987). Innerhalb der Coleopteren, die phylogenetisch zwischen den Hymenopteren und Dipteren stehen (Savard et al., 2006), ist der Mechanismus der Langkeimentwicklung wahrscheinlich ebenfalls unabhängig entwickelt worden. Eine Analyse der Hunchback-Funktion im Langkeimkäfer Bruchidius obtectus kann dazu beitragen zu verstehen, in wieweit es eine gemeinsame Segmentierungsstrategie von Langkeiminsekten gibt. Zudem eröffnet sich zum ersten Mal die Möglichkeit, die hb-Funktion innerhalb der Coleopteren, zwischen Kurz- und Langkeimentwicklung zu vergleichen, da diese im KurzkeimKäfer Tribolium bereits gut untersucht ist (Wolff et al., 1995; Wolff et al., 1998; MarquesSouza et al., 2008). Durch die Klonierung, sowie Analyse der Expression und Funktion von 14 I. Kapitel: Bruchidius hunchback - Einleitung Bruchidius hb soll geklärt werden, ob hunchback im Rahmen der Langkeimentwicklung von Bruchidius ebenfalls als kanonisches Gapgen wirkt, wie in den bis dato untersuchten anderen Langkeiminsekten aus verschiedenen Insektenordnungen, oder doch als nicht kanonisches Gapgen wie das im Kurzkeim-Käfer Tribolium gezeigt werden konnte (Marques-Souza et al., 2008). 15 I. Kapitel: Bruchidius hunchback - Material & Methoden Material & Methoden Die Liste an verwendeten Puffern und Lösungen ist im Anhang A zu finden. Die Bruchidius obtectus Population wurde seit den 80-iger Jahren von dem mittlerweile emeritierten Prof. Dr. Jürgen Büning in Erlangen aufrechterhalten und mir für meine Experimente zur Verfügung gestellt. Bruchidius obtectus: Haltung und Zucht Die Haltung und Zucht basiert auf den Beschreibungen von Emil Jung (Jung, 1965) und Jürgen Büning (Büning, 1980). Eine ausführlichere Zusammenfassung ist in meiner Diplomarbeit nachzulesen (Schnellhammer, 2007). Nahrung: Die Bohnenkäfer werden in Plastikkästen (10 cm x 10 cm x 6 cm) auf angefeuchteten Wachtelbohnen (Palatina, Mühle der Pfalz) gezogen. Die L1-Larven fressen sich durch die Schale in die Bohne und bleiben über sämtliche larvale und pupale Stadien hinweg in derselben. Nach Beendigung der Metamorphose schlüpfen die adulten Käfer aus der Bohne und leben ohne weitere Nahrungsaufnahme noch ca. 3 Wochen. Temperatur: Die Aufzucht der Bohnenkäfer vom Ei bis zum adulten Tier findet im Inkubator bei 32 °C statt und ist nach 4 Wochen abgeschlossen. Für die Eiablage werden die Tiere in einen Inkubator auf 25 °C gestellt. Die Embryogenese ist bei dieser Temperatur nach 5 Tagen beendet. Luftfeuchtigkeit: Während die adulten Käfer relativ unempfindlich auf die Luftfeuchtigkeit reagieren, ist es bei dem initialen Zuchtansatz sehr wichtig, die Wachtelbohnen anzufeuchten (ein paar Tropfen Wasser genügen) bevor die Eier darauf gegeben werden, damit die Larven sich leichter in die Bohnen hineinfressen können. Die Separierung verschiedener Entwicklungsstadien Eier: Zur Gewinnung von Eiern, werden einige Wachtelbohnen und etwa 200 Käfer in eine verschließbare Plastikbox gegeben, die unten mit einem Netz der Maschenweite 1 x 1 mm abgeschlossen wird. Die Bohnenkäfer werden durch den Geruch der Wachtelbohnen zur 16 I. Kapitel: Bruchidius hunchback - Material & Methoden Eiablage stimuliert und legen die Eier neben diesen ab. Die Eier fallen durch das Netz auf ein ausgelegtes farbiges Papier und können mit einem Pinsel aufgesammelt werden. Puppen: Die Puppen werden aus Bohnen herausgeschnitten, die aus einer 3 Wochen alten Aufzuchtbox entnommen sind. Ob eine Bohne von Puppen besetzt ist oder nicht lässt sich gut an der Schale erkennen, da diese transparente Stellen aufweist, unter denen die Puppen unmittelbar zu finden sind. Meistens genügt es die Schale an diesen Stellen abzuziehen und die Puppen fallen aus der Bohne. Andernfalls muss mit einem Pinsel nachgeholfen werden. Adulte Käfer: Für die Zusammenstellung einer Ablagebox zur Eigewinnung müssen die Käfer von den Bohnen aus den Anzuchtboxen getrennt werden. Dazu eignet sich ein herkömmliches Küchensieb mit einer Maschenweite von 2 x 2 mm, groß genug, dass die Käfer in eine bereit gestellte Schüssel fallen und die Bohnen zurückgehalten werden können. Unterscheidung zwischen Männchen u. Weibchen Für pupale und adulte RNAi sowie die Präparation von Ovaren aus adulten Käfern ist die Unterscheidung von männlichen und weiblichen Bruchidien nötig. Die Geschlechter können im Puppenstadium anhand der posterior gelegenen Genitalpapillen leicht unterschieden werden: Die Genitalpapillen der Weibchen sind rundlich und zeigen drei kleine Erhebungen, die Genitalpapillen der Männchen sind eher langgestreckt (Abb. 3: A). Abb. 3: Morphologische Unterschiede männlicher u. weiblicher Bruchidien A) Weibliche Puppe. Der Pfeil markiert die Lage der Genitalpapillen. Die unterschiedliche Form bei Weibchen und Männchen ist schematisch dargestellt. B) Bruchidius Männchen. Der Pfeil weist auf die schmale Stelle am letzten Hinterleibssegment. Die Skizze verdeutlicht die unterschiedliche Breite des letzten Segments anhand derer die Geschlechter unterschieden werden können. 17 I. Kapitel: Bruchidius hunchback - Material & Methoden Die adulten Käfer kann man anhand der Größe, Weibchen sind meistens etwas größer als Männchen, sowie der Form des letzten Hinterleibssegments unterscheiden. Die Hinterleibssegmente der Weibchen sind alle ungefähr gleich breit, während bei Männchen das letzte Hinterleibssegment deutlich schmaler ist (Abb. 3: B). Fixierung der Bruchidius-Embryonen Dechorionisieren: Bruchidius-Eier werden von der Ablagebox gesammelt, in ein Schraubsiebchen der Maschenweite 200 µm gegeben und für 60 min in 100% Klorix (DanKlorix) gestellt. Nach Ablauf dieser Zeit hat sich von einem Großteil der Eier das Chorion aufgelöst und die Eier werden mit Leitungswasser gründlich gewaschen um Klorix-Rückstände zu entfernen. Fixierung: Die Bruchidius-Embryonen werden mit einem feinen Pinsel in ein Szintillationsgläschen gegeben, das 6 ml Heptan, 2 ml PEMS sowie 300 µl Formaldehyd (37%) enthält und mindestens für eine Stunde auf einen Schüttler gespannt. Devitellinisierung: Da die Devitellinisierung der Eier bei Bruchidius nicht mit einem osmotischen Methanolschock erreicht werden kann, ist es nötig die Eier einzeln von Hand mit feinen Minutiennadeln zu schälen. Dazu werden die Eier aus dem Fixierungsgläschen in ein Blockschälchen mit PBT transferiert. Die Vitellinhülle lässt sich gut erkennen, da sie unter dem Binokular glänzend wirkt, während devitellinisierte Eier matt schimmern. Am leichtesten lässt sich diese Hülle entfernen wenn man das Ei zwischen den Minutiennadeln leicht quetscht, so dass ein Riss entsteht der dann mit den Nadeln vergrößert werden kann. Ist die Vitellinhülle entfernt, kann das Ei in ein Eppendorf-Röhrchen mit 50% Methanol überführt werden. Bevor die Embryonen für unbestimmte Zeit auf - 20 °C gelagert werden, werden sie in 100% Methanol überführt. Klonierung von Bruchidius hunchback und paired Zunächst wurde ein Bruchidius hb-Fragment kloniert um die Expression und Funktion des Gens zu untersuchen. Zur exakteren Auswertung der Phänotypen war es notwenig ein weiteres Segmentierungsgen, das Paarregelgen paired, zu klonieren. Die Isolierung von Bruchidius-RNA aus 0-24 bzw. 24-48 h alten Embryonen erfolgte mit dem Trizol Reagenz von 18 I. Kapitel: Bruchidius hunchback - Material & Methoden Invitrogen und die Herstellung von Bruchidius-cDNA mit dem Superscript II RT Kit (Invitrogen). Anschließend wurden die gewünschten DNA-Fragmente durch PCR mit den in Tabelle 1 aufgeführten Primern isoliert. Bei den verwendeten Primern handelt es sich um degenerierte Primer, da das Bruchidius-Genom noch nicht sequenziert ist. Die Primer sind von Metabion. Die 5’ und 3’ Enden der erhaltenen DNA-Fragmente werden mit Hilfe der T4-DNAPolymerase (NEB) aufgefüllt und durch den Einsatz der Polynukleotidkinase (NEB) phosphoryliert. Nach einer Aufreinigung durch Gelextraktion (Min Elute Extraction Kit, Qiagen) findet die Ligation in den pBluescriptvektor durch die Verwendung der T4-DNA-Ligase (NEB) statt. Nach der Transformation des Vektors in den E.coli-Stamm TOP 10 One Shot wird durch Colony-PCR ein geeigneter Klon ausgewählt. Die Sequenzierung wird von der Firma Seqlab (Göttingen) übernommen. Insgesamt wurde bei allen Schritten gemäß den Herstellerangaben verfahren. Die Sequenzen finden sich unter Anhang B. Tab. 1: Degenerierte Primer für das Bruchidius hb-Projekt Primername Sequenz 5’-3’ Tm Länge B.o. fw hb 1 B.o. bw hb 1 AAC TAC AAT CTG AGC ATC TG CCC ATR TGD ATN GTR TAC AT (54 °C) 840 bp (54 °C) B.o. fw prd 1 B.o. bw prd 1 CAR TAY AAR MGN GAR AAY CC TCN CKN GTR TAD ATR TCN GG (55 °C) 311 bp (57 °C) In-situ-Hybridisierung von Bruchidius-Embryonen Das Protokoll ist eine Kombination aus einem Parhyale- und dem Glomeris-in-situ-Protokoll (Rehm et al., 2009, Prpic und Tautz, 2003). Die benötigten Lösungen sind in Anhang A festgehalten, die Synthese der Dig-gelabelten Sonden erfolgt nach Standardprotokoll. Tag 1: 1. Die in Methanol aufgewahrten devitellinisierten Embryonen werden schrittweise in PBT überführt. 2. PBT wird durch eine Detergenzlösung ersetzt und die Embryonen darin für 20 Minuten gewaschen. 19 I. Kapitel: Bruchidius hunchback - Material & Methoden 3. Im Anschluss erfolgt 4 x Waschen mit PBT. 4. PBT entfernen und 500 µl Bruchidius-Hyb-B zugeben und für 5 min bei 55 °C inkubieren. 5. Die Bruchidius-Hyb-B Lösung durch 500 µl Bruchidius-Hyb-A Lösung ersetzen und für 3 Stunden bei 55 °C inkubieren. 6. Soviel Bruchidius-Hyb-A Lösung entfernen, dass die Embryonen gerade noch bedeckt sind und 3 - 5 µl Sonde zu pipettieren und über Nacht bei 55 °C inkubieren. Tag 2: 7. Bruchidius-Hyb-A Lösung entfernen, mit 500 µl Bruchidius-Hyb-B Lösung ersetzen und für 10 min bei 55 °C inkubieren. 8. Erneut mit 500 µl Bruchidius-Hyb-B Lösung waschen und für 30 min bei 55 °C inkubieren. 9. 75% Bruchidius-Hyb-B und 25% 2x SSC, für 15 min bei 55 °C waschen. 10. 50% Bruchidius-Hyb-B und 50% 2x SSC, für 15 min bei 55 °C waschen. 11. 25% Bruchidius-Hyb-B und 75% 2x SSC, für 15 min bei 55 °C waschen. 12. 2 x mit 2x SSC, für 15 min bei 55 °C waschen. 13. 2 x mit 0.2x SSC, für 30 min bei 55 °C waschen. 14. 50% 0.2x SSC und 50% PBT, für 15 min bei RT waschen. 15. 25% 0.2x SSC und 75% PBT, für 15 min bei RT waschen. 16. 2 x mit PBT für 15 min bei RT waschen. 17. Blocking Schritt: 200 µl BBR (2.5%) und 800 µl PBT für 2 Stunden bei RT. 18. Zugabe von Anti-DIG-AP Antikörper (Roche): 1:1000 und über Nacht bei 4 °C inkubieren. 20 I. Kapitel: Bruchidius hunchback - Material & Methoden Tag 3: 19. 6 x mit PBT für jeweils 10 min waschen. 20. 4 x mit PBT für 30 min waschen. 21. 3 x mit Färbepuffer 1 für jeweils 5 min waschen. 22. Zum Färben auf 1 ml Färbepuffer 15 µl NBT/BCIP (Roche) zugeben. 23. Die Färbung durch mehrmaliges Waschen mit PBT abstoppen. Die Embryonen können mit Hoechst 33342 (Endkonzentration: c=2.5 µg/ml, Sigma Deisenhofen) gegen gefärbt und bei 4 °C in PBT aufgehoben werden. Zur weiteren Auswertung mit dem Mikroskop werden die Embryonen auf Objektträger in einen Tropfen 50% Glycerol gebracht. Parentale RNAi in Bruchidius Die Synthese der Doppelstrang-RNA wird mit dem T7-Megaskript Kit (Ambion) durchgeführt, wobei nach Herstellerangaben verfahren wird. Im Wesentlichen läuft die Injektion von weiblichen Bruchidius-Puppen und adulten Tieren so ab, wie in Kapitel 3 für Tribolium beschrieben. Ein Unterschied der bei der Injektion von Bruchidius-Puppen berücksichtigt werden muss ist, dass Bruchidius-Puppen voluminöser und weicher sind als Tribolium-Puppen. Deswegen werden die Bruchidius-Puppen nicht auf einen Objektträger aufgeklebt, sondern einzeln auf einen Knetgummisockel gelegt, mit dem Zeigefinger der linken Hand festgehalten und ein wenig gedrückt. Sobald sich die Kutikula spannt wird auf der ventralen Seite lateral in das Abdomen injiziert. Durch die injizierte Flüssigkeit streckt sich die Puppe. Es darf aber nur so viel injiziert werden, dass sich die am Bauch gefalteten Elytren nicht anheben, andernfalls ist die Überlebensrate extrem schlecht. Nachdem die gewünschte Anzahl an Puppen injiziert worden ist, werden sie zusammen mit Männchen und einigen Wachtelbohnen in ein verschließbares Fliegenröhrchen gegeben, das mit einem Netz nach unten abschließt. Nach Ablauf von 4 Tagen ist die Metamorphose der injizierten Puppen abgeschlossen und dann dauert es meistens nochmal 3-4 Tage bis die erste Eiablage stattfindet. 21 I. Kapitel: Bruchidius hunchback - Material & Methoden Wie für die Ablagebox beschrieben, fallen die Eier durch das Netz durch - in diesem Fall in eine Petrischale - und können dann mit einem Pinsel aufgesammelt werden. Die Embryonen reifen bis zum Erreichen des gewünschten Stadiums bei 25 °C nach und werden dann wie oben beschrieben fixiert. Alternativ fertigt man nach Abschluss der Embryogenese Kutikulapräparate an. Dafür hat es sich bewährt die Eier ebenfalls 1 h in 100% Klorix zu geben, dann mit Minutiennadeln die Eihüllenreste in PBT zu entfernen und schließlich die Larven mit einem Pinsel in einen Tropfen Hoyers:Milchsäure (1:1) zu geben. Die Klärung der Präparate findet im Inkubator bei 60 °C über Nacht statt. Ovarpräparation von Bruchidius Bruchidius-Weibchen werden in ein Blockschälchen mit PBT gegeben, dekapiert und mit Hilfe zweier Uhrmacherpinzetten auf der ventralen Seite des Abdomens geöffnet. Die Ovarien werden auf Eis für 30 Minuten in 4% Formol und PBT fixiert. Anschließend werden die Aktinhüllen der Ovarien mit Minutiennadeln entfernt und ein weiterer Fixierungsschritt für 15 min erfolgt, ebenfalls in PBT das 4% Formol enthält. Die in-situ-Hybridisierung erfolgt nach dem Standard-Laborprotokoll (Bäumer et al., 2011; Tautz & Pfeifle, 1989), das als einzige Abwandlung zur embryonalen ISH eine Ovar-Hybridisierungslösung sowie nach erfolgter Hybridisierung Waschschritte mit einem MABT-Puffer vorsieht (Zusammensetzungen s.h. Anhang A). 22 I. Kapitel: Bruchidius hunchback - Ergebnisse Ergebnisse In dem Kurzkeim-Käfer Tribolium wurde hunchback als nicht kanonisches Gapgen beschrieben (Marques-Souza et al., 2008), während in den Langkeim-Insekten Nasonia und Drosophila, hunchback als kanonisches Gapgen agiert (Pultz et al., 2005; Lehmann und NüssleinVolhard, 1987). Mit Bruchidius soll untersucht werden, welche Rolle hunchback in einem Langkeim-Käfer spielt und somit die Frage beantwortet werden, ob die Funktion von hunchback an den Entwicklungsmodus gekoppelt ist. Das Bruchidius Hunchback-Ortholog Hunchback ist ein Zinkfinger Transkriptionsfaktor (Typ: C2H2), welcher in Drosophila aus sechs Zinkfingerdomänen besteht, die in zwei N-terminale, vier mittelständige sowie zwei C-terminale Zinkfinger eingeteilt werden (Tautz et al., 1987). Das klonierte Bruchidius hbFragment ist 840 bp lang und umfasst die mittleren Zinkfingerdomänen zwei, drei und vier. Abb. 4: Alignment von HB-Orthologen verschiedener Arthropoden Der Ausschnitt zeigt ein Alignment der mittleren Zinkfinger 2-4 von Bruchidius obtectus (B.o.), Tribolium castaneum (T.c.: NP_001038093), Nasonia vitripennis (N.v.: NP_001128392), Drosophila melanogaster (D.m.: AAN13395) und dem Krebs Artemia franciscana (A.f.: CAJ20042). 23 I. Kapitel: Bruchidius hunchback - Ergebnisse Ein Sequenzvergleich (Abb. 4) der klonierten mittleren Bruchidius Hunchback-Zinkfingerdomänen mit den entsprechenden Sequenzen verschiedener Arthropoden zeigt, dass Bruchidius Hunchback sehr gut konserviert ist, da die Übereinstimmung der Sequenzen zwischen Bruchidius und Drosophila ca. 78,7% und zwischen Bruchidius und Tribolium ebenfalls 78,7% beträgt. Die Expression von maternalem hunchback im Bruchidius-Ovar In allen bisher untersuchten Spezies wird hunchback zunächst maternal exprimiert: In Gryllus- und Oncopeltus-Ovaren wurde hunchback in den sich entwickelnden, noch unbefruchteten Eiern nachgewiesen (Mito et al., 2005; Liu und Kaufmann, 2003). Auch in Drosophila und Tribolium kann kurz nach der Befruchtung des Eies, noch bevor die zygotische Genexpression aktiviert ist, hunchback bereits detektiert werden (Tautz und Pfeifle, 1989; Wolff et al., 1995). Es ist deswegen sehr wahrscheinlich, dass Bruchidius hunchback ebenfalls maternal exprimiert wird. Während Drosophila ein polytrophes Ovar besitzt, was bedeutet, dass 15 Nährzellen eine Eizelle umgeben und im Zuge ihrer Reifung durch das Ovar mit wandern (Bastock und St Johnston, 2008), bildet Bruchidius (Büning, 1978) - genau wie Tribolium (Trauner und Büning, 2007) - ein telotrophes Ovar aus. Die sich entwickelnden Eizellen grenzen nicht direkt an die Nährzellen an, sondern stehen mit diesen über sogenannte Nährstränge in Verbindung, da die Nährzellen während der gesamten Eizellreifung im Tropharium verbleiben. B.o. hb-mRNA kann bereits in den Ovariolen nachgewiesen werden (Abb. 5). Im Tropharium ist eine starke hb-Expression im Zentrum zu finden, während die Spitze und der posteriore Bereich nur ganz leicht gefärbt sind. Zieht man den Analogieschluss zu Tribolium (Trauner und Büning, 2007) müssten sich im posterioren Teil des Trophariums somatisches Gewebe sowie ganz junge Oocyten befinden. In den wachsenden Oocyten akkumuliert die hb-RNA und ist ubiquitär in den Eiern verteilt. Die an den Oocytenseiten anliegenden somatischen Follikelzellen bleiben sowohl in jüngeren als auch älteren Stadien ungefärbt (Abb. 5: A vergrößerter Ausschnitt, Pfeilköpfe), jedoch scheint hunchback in den Follikelzellen welche sich zwischen den einzelnen Eikammern befinden, exprimiert zu sein (Abb. 5: A, Sterne). Dort ist die Expression vor allem zu Beginn der Oogenese sehr kräftig. B.o. hunchback wird also ebenfalls maternal exprimiert und anschließend im heranreifende Ei deponiert. 24 I. Kapitel: Bruchidius hunchback - Ergebnisse Abb. 5: Bruchidius hb wird maternal im Ovar exprimiert B.o. hb-Expression in einer telotrophen Bruchidius Ovariole. A) B.o. hb wird im Zentrum des Trophariums und in den wachsenden Oocyten ubiquitär exprimiert. Die Vergrößerung verdeutlicht, dass in den lateralen Follikelzellen keine Expression statt findet (Pfeilköpfe); hingegen ist in den Follikelzellen zwischen den Eikammern eine starke Expression erkennbar (Sterne). A’) Kernfärbung der gleichen Ovariole wie in A, zur Darstellung der Morphologie. Die Expression von Bruchidius hunchback im Laufe der Embryogenese Um die B.o. hb-Expressionsdaten besser einordnen zu können, soll zunächst die BruchidiusEmbryogenese kurz zusammengefasst werden. Die frühe Embryonalentwicklung von Bruchidius ist durch zahlreiche mitotische Kernteilungen im Zentrum des Eies charakterisiert (Jung, 1965; Schnellhammer Diplomarbeit, 2007). Zu diesem Zeitpunkt sind die Kerne, die sogenannten Energiden, nur von Zytoplasma umgeben nicht jedoch von einer Zellmembran (Mazumdar und Mazumbar, 2002; Schowalter, 2006) (Abb. 6: A, B). Ein Großteil dieser Energiden wandert an den Rand des Eies (Abb. 6: C), die im Zentrum des Eies verbleibenden Kerne polyploidisieren und sind als Vitellophagen an der Ernährung des zukünftigen Embryos beteiligt. Im jungen Blastoderm sind die an der Peripherie des Eies befindlichen Zellkerne noch relativ weit voneinander getrennt (Abb. 6: D), durch weitere Mitosen werden sie jedoch immer dichter gepackt und am posterioren Ende heben sich die Polzellen ab, die später für die Entwicklung der Keimbahn zuständig sind (Abb. 6: E). Nach der Ausbildung der Zellmembranen wandern im differenzieren Blastoderm die meisten Zellen zur ventralen Eiseite und bilden das embryonale Gewebe, das die gesamte Länge der anterior-posterioren Achse einnimmt. Auf der dorsalen Seite des Ei- 25 I. Kapitel: Bruchidius hunchback - Ergebnisse es bleiben weniger Zellen, dort entwickelt sich die Serosa, eine extraembryonale Membran (Abb. 6: F). Die weitere Embryogenese von Bruchidius beinhaltet die Keimstreifstreckung, während der sich das posteriore Ende des Embryos nach dorsal-anterior ausdehnt (Abb. 6: G). Schließlich beginnt der Keimstreifrückzug (Abb. 6: H); der Embryo wird kompakter, lateral wachsen die Seitenwände nach dorsal und der Rückenschluss findet statt (Abb. 6: I) (Jung, 1965). Abb. 6: Die Embryonalentwicklung von Bruchidius Die Embryonen sind mit der anterioren Seite nach links orientiert. Der Messbalken spiegelt die Länge 200 µm wider. A) Zygote. B) Kernscharstadium. C) Frühes Blastoderm. Ein Großteil der Energiden wandert an die Peripherie des Eies. D) Die mitotischen Teilungen setzen sich fort, so dass die Kerne im Blastoderm dichter gepackt sind. E) Am posterioren Pol sind die Vorläufer der Keimbahn, die Polzellen zu erkennen (Pfeil). F) Die lateralen Seitenplatten fusionieren ventral und bilden das Keimrudiment. G) Gestreckter Keimstreif, die Kopflappen, sowie Mandibeln und Maxillen sind morphologisch gut zu erkennen. H) Keimstreifrückzug. Die Pfeilspitzen deuten auf die Tracheenöffnungen. I) Embryo während des Rückenschlusses. An der dorsalen Seite ist das Dorsalorgan (DO) zu erkennen, welches sich aus Resten der extraembryonalen Membranen zusammen setzt (Aufnahmen aus Diplomarbeit Schnellhammer, 2007). 26 I. Kapitel: Bruchidius hunchback - Ergebnisse Bruchidius hunchback ist während der gesamten Embryogenese nachweisbar (Abb. 7). Bis zum Stadium des frühen Blastoderms ist B.o. hb homogen im Ei verteilt und vermutlich noch maternalen Ursprungs (Abb. 7: A, A’). Die erste distinkte Expression lässt sich in älteren, blastodermalen Stadien ausmachen, da im posterioren Drittel des Eies keine hb-mRNA mehr nachweisbar ist und B.o. hb eine breite anteriore Expressionsdomäne bildet (Abb. 7: B, B’). Kurz vor Ende der Blastodermphase verschwindet die Expression von B.o. hb am anterioren Eipol, so dass zu diesem Zeitpunkt B.o. hb ausschließlich in einem breiten Streifen in der Mitte des Eies exprimiert wird (Abb. 7: C, C’). Mit der Ausbildung des Keimrudiments entsteht neben der weiterhin vorhanden zentralen Expression eine zusätzliche hunchback-Expressionsdomäne an der posterioren Eispitze (Abb. 7: D). Betrachtet man den Embryo von der dorsalen Seite sieht man, dass in der dort befindlichen extraembryonalen Membran kein hunchback exprimiert wird (Abb. 7: D’; Pfeil). Im Keimrudiment schließlich, wird hunchback in drei Bereichen exprimiert; zum einen im Kopfbereich sowie in einer breiten zentralen Domäne die wahrscheinlich thorakale Segmente überspannt und schließlich weiterhin am posterioren Ende des Keimstreifs (Abb. 7: E). Während die beiden anterioren Expressionsbereiche im Zuge der Keimstreifstreckung verschwinden, bleibt die posteriore Expressionsdomäne die gesamte Keimstreifstreckung über bestehen (Abb. 7: F). Im vollständig elongierten Keimstreif beschränkt sich die B.o. hunchback-Expression auf vermutlich neuronale Vorläuferzellen, welche sich im Kopfbereich und entlang der ventralen Mittellinie befinden (Abb. 7: G). Diese neuronale Expression bleibt auch während des Rückenschlusses weiter bestehen (Abb. 7: H). 27 I. Kapitel: Bruchidius hunchback - Ergebnisse Abb. 7: Die Expression von Bruchidius hb während der Embryogenese 28 I. Kapitel: Bruchidius hunchback - Ergebnisse Die anteriore Seite der Embryonen zeigt nach links. A) Frühe, ubiquitäre Expression von hunchback. A’) Kernfärbung zur näheren Charakterisierung des Stadiums: frühes Blastoderm. B) Breite anteriore hb-Expressionsdomäne. B’) Kernfärbung: spätes Blastoderm. C) Die initial breite anteriore hb-Domäne wird durch eine breite, zentrale Expressionsdomäne ersetzt. C’) Entwicklungsstadium: Spätes Blastoderm. D) Zusätzlich zur zentralen Expressionsdomäne tritt noch eine zweite Domäne am posterioren Pol des Eies auf. Die dorsale Seite des Embryos ist ohne hb-Expression. (Pfeil). D’) Gleiches Ei wie D, dieses Mal von dorsal. Der Bereich der dorsalen Serosa bleibt von hunchback-Expression frei (Pfeil). E) Wachsender Keimstreif mit hb-Expression im Kopf, im thorakalen Bereich sowie in der posterioren Streckungszone. F) Keimstreifstreckung mit beginnender neuronaler und posteriorer hb-Expression. G) Vollständig gestreckter Keimstreif. Die Expression von hunchback beschränkt sich auf die Kopflappen und auf ein segmentales, neuronales Muster (Pfeile weisen auf neuronale Expression). H) Fast vollständig entwickelter Embryo, mit beginnendem Rückenschluss, von ventral aufgenommen. Deutlich zu sehen ist die kräftige neuronale Expression entlang der ventralen Mittellinie (Pfeil). hb-RNAi in Bruchidius resultiert in einem starken Gapgen-Phänotyp Durch Injektion von hb-dsRNA in weibliche Bruchidius-Puppen und die Analyse der daraus resultierenden Nachkommen ist es möglich, B.o. hunchback auf funktionaler Ebene zu untersuchen. Zur Kontrolle wurden auch einige weibliche Bruchdius-Puppen lediglich mit Wasser injiziert, in einem separaten Ansatz verpaart und ausgewertet. Die Bruchidius-Wildtyp Larve (Abb. 8: A) ist ca. 600 µm lang. An den Kopf grenzen drei thorakale Segmente sowie acht abdominale Segmente an. Thorakale und abdominale Segmente sind ventral von Zähnchenbändern besetzt. Das erste thorakale Segment hat ein breites Zähnchenband, das in kurzem Abstand von einem schmaleren gefolgt wird (Abb. 8: A). An den Segmenten T2-A8 befindet sich jeweils nur ein ventrales Zähnchenband. Sowohl das zweite thorakale Segment, als auch alle abdominalen Segmente bilden lateral Tracheenöffnungen aus. Alle Segmente des Rumpfes sind mit langen Haaren bewachsen (Abb. 8: A). In der unteren Hälfte des Bruchidius-Kopfes (Abb. 8: A’) sind die auffallendsten Strukturen die stark autofluoreszierenden Mandibeln. Darunter liegen die paarigen Maxillen und das unpaare Labium. Oberhalb der Mandibeln befindet sich das Labrum, sowie die kleinen Antennen, zwischen denen zwei lange, prominente Kopfborsten auffallen (Abb. 8: A’, Sternchen). hb-RNAi in Bruchidius resultiert in einem drastischen Phänotyp, dessen Häufigkeit bei 58,7% liegt (Tabelle 2). Dieser Phänotyp (Abb. 8: B) beinhaltet die Deletion der Kopf und thorakalen Segmente, sowie einen Großteil der abdominalen Segmente. Die Segmentdeletionen bewirken, dass die hb-RNAi-B.o. Larven mit ca. 300 µm Länge kürzer als 29 I. Kapitel: Bruchidius hunchback - Ergebnisse Tab. 2: Verteilung der Phänotypen nach Bruchidius hb-RNAi (c=3 µg/µl) Phänotyp WT hb-RNAi Phänotyp unentwickelt hb-dsRNA 11 (17,5%) 37 (58,7%) 15 (23,8%) Kontrolle 0 13 (28,9%) 32 (71,1%) Abb. 8: Segmentverlust nach Bruchidius hunchback RNAi A) WT Kutikulapräparat von Bruchidius. Die hell leuchtenden Strukturen am Kopf sind die Mandibeln. An drei thorakale Segmente mit jeweils einem Beinpaar, schließen sich acht abdominale Segmente an. Thorakale und abdominale Segmente sind jeweils mit einem ventral liegenden Zähnchenband besetzt. Tracheenöffnungen liegen lateral an T2, sowie allen abdominalen Segmenten. A’) Frontale Ansicht der Bruchidius Kopfstrukturen (Diplomarbeit, Schnellhammer 2007). Die Sternchen markieren zwei prominente Kopfborsten, daneben sitzen die kurzen Antennen. Das Labrum befindet sich mittig zwischen den paarigen Mandibeln. Unter den Mandibeln liegen die Maxillen und das Labium. B) hb-RNAi-Bruchidius Phänotyp: Die Kopfstrukturen fehlen bis auf das Labrum völlig. Die thorakalen Segmente sind nicht angelegt, an der anteriorsten Seite befinden sich noch ein paar übrige Zähnchenbandreste, die evtl. zu den thorakalen Segmenten gehören könnten. Entlang der anterioren-posterioren Achse sind noch vier stark unstrukturierte Zähnchenbänder erkennbar (weiße Pfeile). B’) Vergrößerte Ansicht der anterioren Seite von A. Bis auf die zwei Kopfborsten (Sternchen) und das Labrum fehlen alle restlichen Kopfstrukturen. 30 I. Kapitel: Bruchidius hunchback - Ergebnisse die Wildtyp-Larven sind. An der anterioren Seite des Embryos erkennt man noch Reste von Zähnchenbänder, an welche im Schnitt meistens noch vier abdominalen Zähnchenbänder angrenzen (Abb. 8: B, Pfeile), deren Morphologie stark beeinträchtigt ist und auch ihre Identität kann nicht mehr eindeutig zugeordnet werden. Die genauere Analyse der in den hb-RNAiKutikulas erkennbaren anterioren Struktur ergab, dass es sich dabei um die Insektenoberlippe - das Labrum - handelt (Abb. 8: B’). Die über dem Labrum befindlichen langen Borsten sind noch entwickelt (Abb. 8: B’, Sternchen), ansonsten fehlen sowohl Antennen als auch alle übrigen Mundwerkzeuge. Der Bruchidius hunchback-RNAi Phänotyp sieht genau so aus wie der hb-mutante-Phänotyp in Nasonia vitripennis bei dem ebenfalls nur das Labrum sowie einige wenige abdominale Segmente entwickelt sind (Pultz et al., 2005). Die Expression des Paarregelgens paired im B.o Wildtyp Um zu untersuchen, ob die nach hb-RNAi resultierenden Phänotypen in Bruchidius auf einen frühen Defekt im Rahmen der Embryogenese zurückzuführen sind, wurde ein 311 bp langes Fragment des Drosophila-Orthologs paired kloniert. Drosophila paired codiert einen Transkriptionsfaktor, welcher aus einer Paired-Box besteht und einer weiter C-terminal gelegenen Homeodomäne (Bopp et al., 1986; Jun und Desplan, 1996). Diese funktionalen Domänen sind gut konserviert, da die Sequenzidentität (Abb. 9) der Paired-Box zwischen Tribolium und Bruchidius bei 97% und zwischen Bruchidius und Drosophila bei 71% liegt; die der Homeodomäne zwischen Tribolium und Bruchidius sogar bei 100% und zwischen Bruchidius und Drosophila mit 86% ebenfalls sehr stark konserviert ist. D.m. paired ist ein Paarregelgen und wird initial in Form einer anterioren Kappe im Blastoderm exprimiert (Gutjahr et al., 1993). Anschließend erfolgt die Expression eines breiten anterioren Streifens, aus welchem die Streifen eins und zwei hervorgehen. Dahinter bilden sich mit einer kleinen zeitlichen Verzögerung aus zwei initial ebenfalls breit angelegte Expressionsdomänen, die Streifen drei und vier sowie fünf bis sieben aus. Mit Erreichen des zellulären Blastoderms erscheint am posterioren Ende ein achter Expressionsstreifen und es splitten sich die Streifen zwei bis sieben auf, indem die Expression in der Mitte des jeweiligen Streifens herunter reguliert wird, so dass paired nun in einem segmentalen Muster exprimiert ist (Kilchherr et al., 1986; Gutjahr et al., 1993). 31 I. Kapitel: Bruchidius hunchback - Ergebnisse Abb. 9: Alignment der konservierten Domänen von Paired-Homologen Der erste konservierte Abschnitt stellt die Paired Box dar (schwarzer Balken); der zweite konservierte Bereich die Homeodomäne (schwarzer Balken) von Nasonia vitripennis (N.v.: ACT79975), Drosophila melanogaster (D.m.: NP_523556), Tribolium castaneum (T.c.: XP_966652) und Bruchidius obtectus. Die früheste paired-Expression in Bruchidius findet im Präblastoderm statt, wenn sich an der posterioren Eiseite die Polzellen deutlich abheben (Abb. 10: A’, Pfeil). An der anterioren Seite des Eies bildet sich ein schmaler Streifen der sich von den beiden lateralen Seiten über die ventrale Seite erstreckt (Abb. 10: A). Kurze Zeit später erscheinen zeitgleich nach dem ersten Expressionsstreifen, drei zusätzlich breite Streifen, die sich über das Zentrum des Eies erstrecken (Abb. 10: B, B’). Unmittelbar nach dem Auftreten der primären paired-Streifen splitten diese sich in sekundäre Streifen auf, scheinbar durch die Herunterregulierung der paired-Expression im Zentrum des jeweiligen initialen breiten Streifens (Abb. 10: B, Pfeil). Mit dem Erreichen des Keimrudiments ist paired segmental in allen gnathalen, thorakalen und abdominalen Segmenten exprimiert (Abb. 10: C, C’). 32 I. Kapitel: Bruchidius hunchback - Ergebnisse Abb. 10: Expression von Bruchidius paired im Wildtyp Die anteriore Seite der Embryonen zeigt nach links, die dorsale Seite, sofern nicht anders angegeben, nach oben. A) Die erste detektierbare prd-Expression findet im späten Blastoderm statt (Pfeil zeigt auf die posterioren Polzellen) und umfasst einen anterioren Streifen der wahrscheinlich mit der Region der zukünftigen Mandibeln zusammen fällt. A’) Hoechstfärbung des gleichen Stadiums mit deutlich sichtbaren Polzellen (Pfeil). B) Die Streifen zwei bis vier treten zeitgleich auf und fangen an sich in sekundäre Streifen aufzusplitten, kurz nachdem die Polzellen wieder in das Innere des Embryos verlagert worden sind. B’) Hoechstfärbung. C) Im Keimrudiment findet eine segmentale Expression in den Mandibeln sowie Maxillen statt und setzt sich über die thorakalen bis zu den bereits vorhandenen abdominalen Segmenten fort. C’) In der Kernfärbung des gleichen Stadiums kann man gut zwischen der dorsalen Serosa (S) und dem embryonalen Gewebe unterscheiden. D) Im gestreckten Keimstreif bleibt die Kopfexpression in den Mandibeln bestehen, die segmentalen Streifen sind verschwunden und nur noch im letzten, dem achten abdominalen Segment, ist eine prd-Expression zu finden. D’) Selber Embryo wie D, von ventral fotographiert. 33 I. Kapitel: Bruchidius hunchback - Ergebnisse In allen bisher beschriebenen Stadien ist die paired-Expression auf die embryonalen Anlagen beschränkt, die dorsal gelegene Serosa bleibt stets frei von Expression (Abb. 10: C, S). Im gestreckten Keimstreif bleibt nur die Expression in den Mandibeln und im achten abdominalen Segment bestehen. Alle anderen Streifen verschwinden gleichzeitig (Abb. 10: D, D’). Insgesamt deutet die Expression von Bruchidius paired darauf hin, dass wie für Drosophila beschrieben (Gutjahr et al., 1993), Bruchidius paired im Blastodermstadium die Funktion eines Paarregelgens inne hat und später im Keimrudiment als Segmentpolaritätsgen wirkt. Die Expression von B.o. paired in B.o. hb-RNAi-Embryonen Bruchidius hunchback-RNAi wirkt sich auf die Expression von Bruchidius paired sowohl in frühen als auch späteren Entwicklungsstadien aus: Im Blastoderm findet die Ausbildung des ersten paired-Streifens nicht statt. Lediglich eine breite diffuse Expression kann beobachtet werden, welche sich vom posterioren Eipol bis in das vordere Drittel des Eies erstreckt (Abb. 11: A, A’, Pfeil). In einem etwas älteren Blastodermstadium sind drei breite paired-Expressionsdomänen ausgebildet, allerdings sind sie nur rudimentär angelegt (Abb. 11: B, B’). Zwar scheint sich das Keimrudiment ventral noch entwickeln zu können, jedoch findet eine deutliche Keimstreifstreckung in hb-RNAi-Embryonen nicht statt. Dies wird ersichtlich durch die stark verkürzten Embryonen bei denen trotzdem noch morphologisch zwischen einem Kopfbereich und abdominalen Strukturen unterschieden werden (Abb. 11: C’, Pfeil). Allerdings ist in diesem Stadium weder in den gnathalen und thorakalen Segmenten, noch posterior eine paired-Expression zu finden (Abb. 11: C). Diese Befunde bestätigen, dass der auf Kutikulaebene gefundene Phänotyp nach Bruchidius hunchback-RNAi, welcher in dem kompletten Verlust von Kopf und dem Großteil der abdominalen Segmente basiert, tatsächlich auf Segmentdeletionen und der Störungen der frühen Musterbildung zurückzuführen ist. 34 I. Kapitel: Bruchidius hunchback - Ergebnisse Abb. 11: Expression des Paarregelgens paired in Bruchidius nach hb-RNAi Die anteriore Seite der Embryonen ist nach links orientiert. A) Nach hb-RNAi ist im Blastoderm eine schwache leicht verwaschene paired-Expression vorhanden. A’) Kernfärbung mit Hoechst. B) In einem etwas älteren Stadium sind vier rudimentäre paired-Streifen vorhanden, die allerdings nach anterior verschoben sind. B’) Kernfärbung mit Hoechst. C) Nach dem Erreichen des Keimrudimentstadiums ist keine paired-Expression mehr zu detektieren. C’) Die Keimstreifstreckung fehlt vollständig, jedoch können mittels Kernfärbung Rudimente von Kopfstrukturen und der abdominale Bereich voneinander unterschieden werden (Pfeile). 35 I. Kapitel: Bruchidius hunchback - Diskussion Diskussion Durch die Adaption von Standardmethoden, welche in anderen Modellorganismen zur Untersuchung der Embryonalentwicklung Verwendung finden, an den Langkeimer Bruchidius obtectus, steht nun dem Kurzkeimer Tribolium bezüglich der frühen Embryonalentwicklung ein interessanter Vergleichsorganismus zur Seite. Die Analyse des Gapgens hunchback in Bruchdius zeigt bereits anschaulich wie groß die Unterschiede der Segmentierung zwischen zwei Spezies innerhalb einer Ordnung aufgrund ihrer verschiedenen Entwicklungsmodi sein können. Kanonische Gapgen Funktion von B.o. hb in der anterioren Musterbildung Initial wird Bruchidius hunchback maternal im Ei deponiert (Abb. 5: A). Im frühen Blastoderm ist B.o. hb homogen im Ei verteilt und wird kurze Zeit später in einer breiten anterioren Domäne exprimiert (Abb. 7: A, B). Auch im Keimrudiment besteht die anteriore Expression fort, welche sich von den Kopflappen bis ins anteriore Abdomen hinein zieht (Abb. 7: E). Parentale hb-RNAi in Bruchidius resultiert in einem sehr starken anterioren und posterioren Segmentierungsphänotyp (Abb. 8), welcher mit dem beschriebenen Phänotyp aus Nasonia mutanten hunchback-Embryonen nahezu identisch ist (Pultz et al., 1999, Pultz et al., 2005). Anterior fehlen die Antennen, Mundwerkzeuge sowie die thorakalen Segmente (Abb. 8: B’). Als einzige anteriore Struktur, sowohl in Nasonia als auch in Bruchidius, ist das Labrum noch entwickelt. Das Labrum stellt die anteriorste Kopfstruktur dar, wobei es sich vermutlich um nicht segmentales anteriores Gewebe handelt (Posnien et al., 2009). Aus Drosophila ist bekannt, dass der sowohl anterior als auch posterior im Ei aktivierte Torso-Signalweg im anterioren Bereich u.a. für die Entwicklung des Labrums zuständig ist (Klingler et al., 1988). Es konnte auch gezeigt werden, dass der Torso-Signalweg eine Repression von hunchback am vorderen Eipol bewirkt, was für die Entwicklung des Labrums nötig ist (Janody et al., 2000). Das könnte eine Erklärung dafür sein, dass sich diese anteriore Struktur nach Bruchidius hunchback-RNAi normal entwickelt, da im Wildtyp die Ausbildung des Labrums nur in Abwesenheit von hunchback stattfinden kann. Eine Funktion von Torso im anterioren und posterioren Bereich des Tribolium-Eies konnte bereits gezeigt werden (Schoppmeier und Schröder, 2005), so dass die Möglichkeit besteht, dass der Torso- 36 I. Kapitel: Bruchidius hunchback - Diskussion Signalweg auch in Bruchidius konserviert und an der Repression von hunchback im Wildtyp beteiligt ist. Während es sich in dem beobachteten anterioren Phänotyp in Tribolium um eine Transformation von gnathalen und thorakalen Segmenten hin zu abdominaler Identität handelt (Marques-Souza et al., 2008), sind die anterioren Entwicklungsdefekte in Bruchidius tatsächlich auf den Verlust von Segmenten zurückzuführen, was sich auf der Ebene des untersuchten Paarregelgens B.o. paired widerspiegelt. Der erste anteriore paired-Streifen wird in Bruchidius hb-RNAi-Embryonen nicht ausgebildet; weiter posterior gelegene Streifen treten nur rudimentär und mit einer anterioren Verschiebung auf (Abb. 11: B). Im Keimrudiment ist schließlich überhaupt keine paired-Expression mehr nachweisbar (Abb. 11: C). Insgesamt erfüllt damit hunchback in Bruchidius die Funktion eines kanonischen Gapgens: breite Expressionsdomänen, welche deckungsgleich mit aneinander grenzenden Segmentdeletionen nach einem Knockdown von hunchback sind. B.o. hunchback ist wichtig für die posteriore Musterbildung Im stärksten Kutikulaphänotyp nach hunchback-RNAi sind neben dem Labrum nur noch vier ventrale Zähnchenbänder als distinkte Strukturen erkennbar (Abb. 8: B). Die Identität kann man mangels morphologischer Merkmale nicht eindeutig zuordnen, doch ist es anzunehmen, dass es sich dabei um die abdominalen Segmente A3 bis einschließlich A6 handelt, da im Wildtyp die letzten Segmente der Bruchidius-Larve posterior deutlich schmaler werden, was in den B.o. hb-RNAi Kutikulas nicht beobachtet werden kann. Somit würden die posteriorsten Segmente A7 und A8 fehlen. Dies wäre konsistent mit der Beobachtung, dass hunchback im Keimrudiment an der posterioren Spitze und im gestreckten Keimstreif am posterioren Ende in einem schmalen Streifen exprimiert wird. Diese zusätzliche abdominale Funktion von hunchback ist ebenfalls aus Drosophila bekannt. Ist zunächst noch die Reprimierung von maternalem hunchback via dem Nos/Pum Komplex nötig (Irish et al., 1989; Wreden et al., 1997), wird die spätere posteriore Regulierung von hunchback durch den Torso-Signalweg durchgeführt, welcher in diesem Fall im Gegensatz zur anterioren Eiseite - durch die Aktivierung der Gapgene des terminalen Systems tailless und huckebein (Brönner und Jäckle, 1991), die Expression von posteriorem hunchback steuert. Hunchback wiederum erfüllt an dieser Stelle eine Funktion als sogenanntes sekundäres Gapgen (Margolis et al., 1995). Fehlende posteriore hunchback-Aktivität bewirkt in schwachen Phänotypen die Fusion des siebten und achten abdominalen Segments; 37 I. Kapitel: Bruchidius hunchback - Diskussion in stärkeren Phänotypen die Deletion der selbigen sowie den partiellen Verlust der posterioren Filzkörper (Lehmann und Nüsslein-Volhard, 1987). In Nasonia trifft man auf die gleiche Situation. Eine später im Blastoderm auftretende posteriore Expressionsdomäne, die für die Spezifizierung der posteriorsten Segmente nötig ist (Pultz et al., 2005). Aus den bisher verfügbaren Bruchidius-Daten geht also hervor, dass auch die posteriore hunchback-Funktion zwischen den verschiedenen Langkeiminsekten konserviert ist. Bruchidius und Tribolium Hunchback, ein Vergleich Obwohl Bruchidius und Tribolium beide der Ordnung Coleoptera zugehören, spiegelt sich diese Verwandtschaft weder in den Expressionsmustern noch in den funktionalen Daten von hunchback wider. Während in Bruchidius eine anteriore Kopfexpression und eine Expression in den thorakalen Segmenten auftreten, beschränkt sich die anteriore Expressionsdomäne von Tribolium hunchback auf die Serosa (Wolff et al., 1995). Die Expression von Tribolium hunchback im Gnathum und T1 lässt sich zwar teilweise mit der Bruchidius-Expression vergleichen, welche sich vom Kopf bis wahrscheinlich A2 hinzieht, allerdings ist die Funktion von Hunchback in diesem Bereich unterschiedlich, was sich auf phänotypischer Ebene widerspiegelt. Nach Bruchidius hunchback-RNAi fehlen sämtliche Kopfstrukturen (Abb. 8: B), bis auf das Labrum, während nach Tribolium hunchback-RNAi die Entwicklung von Labrum, Antennen und Mandibeln unbeeinflusst bleibt (Marques-Souza et al., 2008). In Bruchidius ist hunchback nötig, dass das Paarregelgen paired exprimiert wird, da in hb-RNAi-Embryonen die Expression dieses Gens stark gestört ist, was mit dem Fehlen der Kopf, Thorax und einem Großteil der abdominalen Segmente korreliert (Abb. 11). In Tribolium sind die fehlenden gnathalen und thorakalen Segmente jedoch auf einen anderen Effekt zurückzuführen, da nach hb-RNAi in Tribolium sowohl die anterioren Streifen der Paarregelgene even-skipped als auch runt wie im Wildtyp exprimiert werden, was ein Hinweis darauf ist, dass die gnathalen und thorakalen Segmente in Tribolium initial angelegt werden (Marques-Souza et al., 2008). Jedoch zeigte die Analyse der Hoxgenexpression nach T.c. hb-RNAi, dass eine anterioren Verschiebung der Expression von Antp, Abd A und Ubx, welche im WT nur in den abdominalen Segmenten koexprimiert werden, stattfindet so dass die gnathalen und thorakalen Segmente eine falsche Identität entwickeln (Marques-Souza et al., 2008). 38 I. Kapitel: Bruchidius hunchback - Diskussion Auch die posteriore Funktion von hunchback in beiden Käfern ist unterschiedlich. Zwar resultiert der RNAi-Effekt in beiden Organismen in abdominalen Störungen der Musterbildung und auf den ersten Blick sind diese posterioren Phänotypen ähnlich, allerdings ist der Effekt in Tribolium deutlich stärker als in Bruchidius und auf eine Störung der Wachstumszone sowie einen damit verbundenen vorzeitigen Segmentierungsabbruch zurückzuführen (Marques-Souza et al., 2008). Ein gegenteiliger Effekt kann durch die Überexpression von hunchback in Tribolium erzielt werden, da dieser in einer einer zusätzlichen Anzahl an abdominalen Segmenten resultiert (Distler und Klingler, in Vorbereitung). Aufgrund der Langkeimentwicklung ist in Bruchidius eine posteriore Wachstumszone nicht existent und aus Drosophila weiß man, dass eine posteriore ektopische Expression von hunchback zu der Deletion von abdominalen Segmenten führt (Irish et al, 1989), so dass die Ursache für das Fehlen der posterioren Segmente in Bruchidius wahrscheinlich eine andere ist als in Tribolium. Die Hunchback-Funktion ist in Langkeimern konserviert Der Vergleich der frühen Embryonalentwicklung verschiedener Langkeiminsekten wie Drosophila und Episyrphus aus der Ordnung der Dipteren, oder den Hymenopteren Nasonia und Apis zeigt, dass die frühen genetischen Interaktionen für die Segmentierung der anteriorenposterioren Achse teilweise sehr unterschiedlich sind, auch bei der Zugehörigkeit zur gleichen Insektenordnung (Lemke und Schmidt-Ott, 2009; Wilson und Dearden, 2011). Ein großer Unterschied beim Vergleich der Langkeimentwicklung zwischen Dipteren und Hymenopteren ist die Bicoid, welches in der anterioren Musterbildung bei den Dipteren eine prominente Rolle spielt, während in den Hymenopteren Nasonia und Apis aufgrund des Fehlens von Bicoid (Stauber et al., 1999), vor allem otd-1 und hb maßgeblich an der anterioren Segmentierung beteiligt sind (Lynch et al., 2006; Wilson und Dearden, 2011). Doch auch der Vergleich der Segmentierung zwischen den näher verwandten Arten Nasonia und Apis bringt Unterschiede zu Tage. Beispielsweise wirkt Nasonia otd-1 vermutlich reprimierend auf caudal, während in Apis gezeigt werden konnte, dass otd-1 eindeutig aktivierend auf die caudal-Expression wirkt (Lynch et al., 2006; Wilson und Dearden, 2011). Ein anderes Beispiel ist das Gapgen giant: Nasonia giant-mRNA ist zunächst maternalen Ursprungs und bildet einen von anterior nach posterior verlaufenden Gradienten aus, welcher die Expression weitere Gapgene reguliert. Später wird Nasonia giant auch zygotisch exprimiert durch 39 I. Kapitel: Bruchidius hunchback - Diskussion die Aktivierung von Hunchback (Brent et al., 2007). In Apis hingegen wird das Gapgen giant ausschließlich durch Hunchback zygotisch aktiviert (Wilson und Dearden, 2011). Eine Begründung für diese Unterschiede ist, dass es sich vor allem bei den frühen Vorgängen der Segmentierung um sehr schnell evolvierende Prozesse handelt (Kalinka et al., 2010). Neben den soeben exemplarisch aufgeführten Unterschieden gibt es auch Gemeinsamkeiten bezüglich der frühen Segmentierung. Schon die früheren Untersuchungen des Gapgens hunchback in den Langkeimern Nasonia, Drosophila, Megaselia etc. zeigten, dass die Funktion von hunchback in der Segmentierung dieser Langkeimer konserviert ist (Lehmann und Nüsslein-Volhard, 1987; Pultz et al., 1999; Pultz et al., 2005; Stauber et al., 2000). Mit Bruchidius ein Vertreter der Langkeimer aus der Ordnung der Coleoptera, konnte nun in einer weiteren holometabolen Insektenordnung gezeigt werden, dass hunchback sowohl in der Expression als auch seiner Funktion stark konserviert ist. Damit ist man bei der Entschlüsselung gemeinsamer Prinzipien der Langkeimentwicklung einen kleinen Schritt weiter und es wäre interessant zu wissen, wie groß die Übereinstimmung der Expression und Funktion weiterer Gene der frühen Segmentierungskaskade in Bruchidius im Vergleich zu den anderen Langkeiminsekten ist. Durch das Verständnis ob und inwieweit es eine gemeinsame Strategie der Langkeimentwicklung gibt, könnte auch die Frage neu diskutiert werden welcher Entwicklungsmodus an der Basis der holometabolen Insekten steht: In Anbetracht der Tatsache, dass alle hemimetabolen Insektenordnungen den Modus der Kurzkeimentwicklung für die Segmentierung nutzen und in den später entstandenen holometabolen Insekten sowohl Kurz- als auch Langkeimentwicklung existiert, ist die Vermutung dass der ursprüngliche Segmentierungsmodus der holometabolen Insekten die Kurzkeimentwicklung ist, während die Langkeimentwicklung konvergent in den Hymenopteren, Coleopteren, Lepidopteren und Dipteren entstanden ist (Davis und Patel, 2002; Peel, 2008). Obwohl es sich bei den Ergebnissen bezüglich der Expression und Funktion von Bruchidius hunchback um einen ersten Datenpunkt handelt, ist doch die Ähnlichkeit zur Funktion und Expression von Nasonia hunchback, Apis hunchback und Episyrphus hunchback erstaunlich (Pultz et al., 2005; Stauber et al., 2000; Wilson und Dearden, 2011). In Abhängigkeit davon in welchem Umfang weitere Gemeinsamkeiten der frühen Segmentierung innerhalb der Langkeiminsekten gefunden werden können, zieht dies die Frage nach sich ob nicht auch an der Basis der holometabolen Insekten die Langkeimentwicklung 40 I. Kapitel: Bruchidius hunchback - Diskussion stehen könnte und die Kurzkeimentwicklung innerhalb der holometabolen Insekten sekundär erneut entstanden ist. Um jedoch eine zufriedenstellende Antwort zu finden, müsste die frühe Segmentierung von Bruchidius weiter untersucht werden um zusätzliche Parallelen oder auch Unterschiede zum Langkeimmodus anderer Insekten finden zu können. Zudem ist die Untersuchung weiterer Segmentierungsstrategien in verschiedenen holometaboler Kurzkeiminsekten - neben Tribolium - nötig um die Möglichkeit der sekundären Kurzkeimentwicklung zu bestätigen oder auszuschließen. 41 II. Kapitel: Das Mex-3/Cad System Einleitung Das Mex-3/Cad System soll neben dem im vorigen Kapitel bereits vorgestellten Bohnenkäfer Bruchidius, auch in der Wanze Oncopeltus fasciatus untersucht werden. Zunächst wird erläutert was es mit dem Mex-3/Cad System auf sich hat, wobei den Ausgangspunkt für die Fragestellung erneut Drosophila bildet. Funktion und Ursprung von Bicoid Bicoid nimmt in der Drosophila-Segmentierung eine zentrale Stellung ein, da es maßgeblich in die anteriorer Musterbildung involviert ist (Ephrussi und St Johnston, 2004). Die Nachkommen von bicoid-mutanten Fliegenweibchen bilden keinen Kopf und keine thorakalen Strukturen aus, stattdessen entstehen Embryonen mit zusätzlichen posterioren Strukturen am eigentlich anterioren Pol (Frohnhöfer und Nüsslein-Volhard, 1986). bicoid-mRNA wird noch während der Oogenese im heranreifenden Ei deponiert, indem ein Komplex aus den Proteinen Dynein und Swallow, bicoid-mRNA an das anteriore Mikrotubuliskelett der Zelle verankert (Schnorrer et al., 2000). Nach der Befruchtung und der Eiablage löst sich ein Teil der lokalisierten bicoid-mRNA und bildet an der Peripherie des Eies einen von anterior nach posterior verlaufenden Gradienten aus (Spirov et al., 2009), der schließlich in einen Proteingradienten übersetzt wird (Driever und Nüsslein-Volhard, 1988a). Bicoid ist ein Homeodomänenprotein (Gehring, 1987) und erfüllt zwei Aufgaben: Zum einen wirkt es als Morphogen und Transkriptionsregulator durch die konzentrationsabhängige Regulierung nachgeschalteter Genen, so dass deren Aktivierung - entsprechend der erhaltenen Positionsinformation - von anterior nach posterior möglich ist (Driever und NüssleinVolhard, 1989). Beispielsweise benötigen anterior exprimierte Gapgene wie orthodenticle für die Transkriptionsaktivierung eine hohe Konzentration des Bicoid-Proteins (Finkel- 42 II. Kapitel: Mex-3/Cad System - Einleitung stein und Perrimon, 1990), während weiter posterior exprimierte Gapgene wie Krüppel oder knirps nur durch geringe Bicoid-Konzentrationen aktiviert werden müssen (Hülskamp und Tautz, 1991; Rivera-Pomar et al., 1995). Die zweite Aufgabe von Bicoid ist die eines Translationsinhibitors. Dies beinhaltet die Bindung von Bicoid in der 3’ UTR der caudal-mRNA (Rivera-Pomar et al., 1996a), welche homogen im frühen Embryo verteilt ist (Mlodzik et al., 1985). In Folge dessen wird das Protein dEHP rekrutiert, das eine Interaktion mit der am 3’ Ende der mRNA befindlichen Kappe vermittelt (Cho et al., 2005). Durch diese Bindung ist es nicht mehr möglich caudal-mRNA im anterioren Bereich des Embryos zu translatieren, weswegen sich die caudal-Translation ausschließlich im posterioren Teil des Embryos abspielt und dort die Gapgene der posterioren Gruppe aktivieren kann (Mcdonald and Struhl, 1986; Rivera-Pomar et al., 1995). Diese strikte translationale Repression von Drosophila caudal im anterioren Bereich des Embryos ist für die frühe Musterbildung sehr wichtig, da eine Überexpression von Drosophila caudal in Embryonen resultiert, die eine gestörte Kopfinvolution sowie zahlreiche Segmentfusionen entlang der a-p Achse aufweisen. In den stärksten Phänotypen tritt sogar der vollständige Verlust der Kopf- und thorakalen Segmente auf (Frohnhöfer und Nüsslein-Volhard, 1986; Mlodzik et al., 1990). Neben Drosophila wurde Bicoid noch in den cyclorrhaphen Fliegen Megaselia, Platypeza, Lonchoptera sowie Episyrphus gefunden und ist dort ebenfalls an der Aktivierung der Gapgene hunchback, otd und Krüppel beteiligt (Stauber et al., 1999; Lemke et al., 2008; Lemke et al., 2010). In Episyrphus wurde gezeigt, dass sich nach bicoid-RNAi die caudalExpression nicht mehr auf den posterioren Bereich des Embryos beschränkt sondern caudal ubiquitär im Ei exprimiert wird, so dass auch hier Bicoid als translationaler Repressor von caudal fungiert (Lemke und Schmidt-Ott, 2009; Lemke et al., 2010). Obwohl Bicoid diese wichtige Funktion in der anterioren Musterbildung innerhalb der cyclorrhaphen Fliegen inne hat (McGregor, 2005), blieb die Suche nach Bicoid-Homologen in anderen Insektenordnungen wie Coleoptera (Brown et al., 2001) oder Hymenoptera (Lynch et al., 2006) erfolglos und selbst innerhalb der Dipteren konnte außerhalb der cyclorrhaphen Fliegen kein BicoidHomolog gefunden werden (Stauber et al., 2001). Der Grund dafür ist der Zeitpunkt der Entstehung von Bicoid, welcher unmittelbar vor der Entstehung der cyclorrhaphen Fliegen liegt: In ursprünglichen Insekten wie dem Ofenfischchen Thermobia domestica liegt innerhalb des Hoxclusters das Hox-3-Gen. Es ist wie ein typisches Hoxgen für die Festlegung der 43 II. Kapitel: Mex-3/Cad System - Einleitung Segmentidentität zuständig, wird aber zusätzlich auch in den extraembryonalen Membranen exprimiert (Hughes et al., 2004). Die Vermutung ist, dass dieses homeotische Hox-3-Gen im Laufe der Evolution seine homeotische Funktion gänzlich verloren hat, direkt in das Gen zerknüllt übergangen ist und nur noch für die Spezifizierung der extraembryonalen Membran zuständig ist (Hughes et al., 2004). Durch eine spätere Duplikation des Gens zerknüllt unmittelbar vor der Radiation der cyclorrhaphen Fliegen entstand schließlich das Gen bicoid das somit auf diese Gruppe beschränkt ist (Stauber et al., 2002). Die Frage ist nun, wie die Caudal-Regulation - ohne Bicoid - in anderen Organismen durchgeführt wird. Regulationsmöglichkeiten von Caudal außerhalb Drosophila Im Gegensatz zu Bicoid, ist Caudal innerhalb der Bilateria sehr gut konserviert. In allen bisher untersuchten Gruppen von Mollusken (Le Gouar et al., 2003) über Nematoden (Draper et al., 1996) und Arthropoden (Schulz et al., 1998; Copf et al., 2004; Shinmyo et al., 2005; Olesnicky et al., 2006; Stauber et al., 2008 ) bis hin zu Vertebraten (Marom et al., 1997; Pillemer et al., 1998), ist Caudal an der posteriore Musterbildung beteiligt (Abb. 12). Im Folgenden soll anhand des Nematoden C.elegans und einigen Beispielen aus dem Reich der Insekten Bicoid unabhängige Möglichkeiten der Caudal-Regulation aufgezeigt werden. Das C.elegans-Homolog von D.m. caudal heißt pal-1 und seine Regulation findet - wie das Bicoid/Caudal System in Drosophila - auch auf translationaler Ebene statt (Draper et al., 1996). Die frühe Entwicklung von C. elegans basiert auf holoblastischen Furchungen, wobei auf die befruchtete Eizelle ein zwei Zell-Stadium, dann ein vier Zell-Stadium etc. folgen. Die einzelnen Zellen werden Blastomere genannt (Rose and Kemphues, 1998). Während C.elegans pal-1-mRNA in allen vier Blastomeren verteilt ist, ist Pal-1-Protein nur in den zwei posterioren Blastomeren nachweisbar. Ein KH-Domänen Protein (Siomi et al., 1993) namens Mex-3 ist für die Repression des C.elegans Homologs von Caudal in den anterioren Blastomeren des frühen Embryos zuständig. In mex-3-mutanten C.elegans-Embryonen findet eine Expansion von Pal-1 in die anterioren Blastomeren statt und es kommt statt zur Ausbildung einer pharyngalen Zelllinie zur Ausbildung von Muskeln (mex-3 = muscle excess), welche sich im Wildtyp nur auf die posterioren embryonalen Bereiche beschränken (Draper et al., 1996). 44 II. Kapitel: Mex-3/Cad System - Einleitung Abb. 12: Allgemeine phylogenetische Übersicht der Bilateria Darstellung der phylogenetischen Beziehungen zwischen den Bilateria (nach Gilbert, 2006). Hervorgehoben sind die Tierstämme, in welchen eine konservierte Funktion von caudal in der posterioren Musterbildung bereits nachgewiesen wurde. Die Expression von C.elegans Pal-1 ist also wichtig für die Festlegung der Identiät der posterioren Blastomeren (Hunter und Kenyon, 1996). Auch innerhalb der Insekten ist die Funktion von caudal in der posteriore Musterbildung konserviert. Im Langkeiminsekt Nasonia vitripennis konnte gezeigt werden, dass caudal bereits im Rahmen der Oogenese posterior im Ei deponiert wird. Von dort ausgehend bildet sich nach der Eiablage ein von posterior nach anterior verlaufender cad-mRNA-Gradient aus, welcher wahrscheinlich allein auf die posteriore Lokalisierung der cad-mRNA zurückzuführen ist (Olesnicky et al., 2006). Diese These wird von dem Befund gestützt, dass die frühe Entwicklung in Nasonia sehr stark von der Lokalisierung maternaler mRNAs abhängt wie beispielsweise otd- oder giant-mRNA, welche ebenfalls durch die Fixierung am Cytoskelett der Eipole Gradienten bilden können (Lynch et al., 2006; Brent et al., 2007). Trotzdem ist nicht auszuschließen, dass ein weiterer Faktor im anterioren Bereich für die Repression der Nasonia caudal-mRNA zuständig ist. Interessanterweise zeigt die funktionale Analyse von cad in Nasonia, dass caudal in der posterioren Segmentierung eine etwas größere Rolle spielt als das in Drosophila der Fall ist. Der Knockdown sowohl von maternalem als auch zygotischem caudal durch parentale RNAi resultiert in Embryonen die früh sterben und keine Kutikula ausbildeten. Nur die Injektion niedrigste Konzentrationen an cad-dsRNA ermöglicht die Erstellung einer phänotypischen Serie die zeigt, dass nach cad-RNAi Nasonia-Embryonen einen Segmentierungsabbruch ab 45 II. Kapitel: Mex-3/Cad System - Einleitung T3 aufweisen. In Drosophila hingegen setzt die Störung der posterioren Segmentierung erst ab dem zweiten abdominalen Segment ein (Olesnicky et al., 2006; Mcdonald und Struhl, 1986). Die Expression und Funktion von caudal wurde auch in den Kurzkeiminsekten Gryllus bimaculatus und Tribolium castaneum untersucht (Shinmyo et al., 2005; Copf et al., 2004). Aufgrund der posterioren Ausbildung des Keimrudiments in diesen Insekten wäre ein von anterior verlaufender Morphogengradient bei der Musterung des embryonalen Gewebes nicht sinnvoll, da dieser am posterioren Eipol nicht mehr ausreichend Information vermitteln würde, weshalb vermutet wird, dass die Musterbildung in Kurzkeiminsekten mit nur einem Gradienten auskommen könnte. Dieser würde dementsprechend von posterior nach anterior verlaufen (Rosenberg et al., 2009). Caudal wäre dafür ein geeigneter Kandidat, ob es jedoch - analog zu Bicoid (Ephrussi und St Johnston, 2004) - eine Morphogenfunktion inne haben kann ist umstritten, da aus Drosophila bekannt ist, dass Caudal zwar einen Gradienten ausbildet, aber die Aktivierung der Zielgene nicht von der Caudal-Konzentration abhängt, sondern nur von der An- oder Abwesenheit von Caudal (Mcdonald und Struhl, 1986). In Gryllus bimaculatus wird Caudal eine Eigenschaft als Morphogen zugeschrieben, weil beispielsweise nach cad-RNAi das Zielgen hunchback in einer weiter posterior gelegenen Domäne exprimiert wird. Auch ist die Funktion von caudal in Gryllus ausgeprägter als in Drosophila, da starke cad-RNAi-Phänotypen keine Kutikula ausbilden und schwächere Phänotypen eine Deletion von gnathalen, thorakalen und abdominalen Segmenten aufweisen (Shinmyo et al., 2005). Tribolium caudal ist zunächst ubiquitär im Ei exprimiert. Im Blastoderm beschränkt sich dann die Expression auf den posterioren Bereich des Eies, wo sich das embryonale Gewebe entwickelt (Schulz et al., 1998; Copf et al., 2004). Im Gegensatz zu Gryllus werden jedoch die Zielgene von Caudal in Tribolium nicht konzentrationsabhängig aktiviert (Schoppmeier et al., 2009). cad-RNAi in Tribolium resultiert ebenfalls in Embryonen, deren gesamte posteriore Entwicklung - bis auf den anterioren Kopfbereich gestört ist. Vor kurzem konnte nachgewiesen werden, dass das aus C.elegans bereits bekannte KH-Domänen-Protein Mex3 (Draper et al., 1996) auch in Tribolium für die Repression der caudal-Translation zuständig ist, in dem es an der 3’ UTR der cad-mRNA bindet (Schoppmeier et al., 2009). Wird mex-3 durch RNAi herunter reguliert, fehlen in schwächeren Phänotypen das Labrum und die Antennen während in den stärksten Phänotypen der gesamte Kopf der Tribolium-Larven nicht entwickelt ist. Die Analyse weiterer Segmentierungsgene wie wingless (Nagy und Car46 II. Kapitel: Mex-3/Cad System - Einleitung roll, 1994) und even-skipped (Brown et al., 1997) in Tribolium mex-3-RNAi-Embryonen ergab, dass im Blastoderm der im Wildtyp im anterioren Kopfbereich exprimierte winglessStreifen fehlt und von den normalerweise drei even-skipped-Streifen im Blastoderm nur der am posterioren Pol gelegene, vollständig ausgebildet wird. Diese Ergebnisse belegen, dass der Kopfdefekt nach mex-3-RNAi auf eine frühe Beeinträchtigung der anterioren Segmentierung zurückzuführen ist (Schoppmeier et al., 2009). Der Grund dafür ist, dass nach mex-3-RNAi die posteriore Caudal-Expression im Vergleich zum Wildtyp nach anterior verschoben ist, so dass nun Caudal auch in dem Bereich der Kopfanlagen zu detektieren ist (Schoppmeier et al., 2009). Der anteriore Bereich, dort wo sich die Serosa bilden wird (van der Zee et al., 2005), bleibt jedoch nach mex-3-RNAi weiterhin von Caudal-Expression frei. Es zeigte sich, dass Zen-2 dort für die Repression von Caudal verantwortlich ist, denn nach zen-2-RNAi kann Caudal auch im anterioren Bereich des Eies nachgewiesen werden (Schoppmeier et al., 2009). Da wie bereits oben beschrieben, zen-2 aus dem Hox-3-Gen hervorgegangen ist (Hughes et al., 2004) und aufgrund der Duplikation von zen-2, bicoid entstanden ist (Stauber et al., 1999), welches in Drosophila die caudal-Translation im anterioren Bereich des Eies unterdrückt, geht die Vermutung dahin, dass die ursprüngliche Funktion von Hox-3 in der Repression der caudal-Translation lag (Schoppmeier et al., 2009). Ziele dieser Arbeit In Anbetracht der Tatsache, dass das Mex-3/Cad System sowohl im Nematoden C.elegans als auch innerhalb der Insekten in Tribolium castaneum während der frühen Embryonalentwicklung eine wichtige Rolle spielt, stellt sich die Frage in wie weit dieser Mechanismus bei den Ecdysozoa und evtl. sogar innerhalb der Bilateria konserviert ist (Draper et al., 1996; Schoppmeier et al., 2009). Zunächst soll im Rahmen dieses Projekts die Frage geklärt werden, ob das Mex-3/Cad System innerhalb der Insekten konserviert ist. Dazu werden die Orthologe von mex-3 und caudal zum einen in Bruchidius obtectus untersucht, bei dem aufgrund der nahen phylogenetischen Verwandtschaft zu Tribolium - beide der Ordnung Coleoptera zugehörig - das Mex-3/Cad System konserviert sein könnte. Gesetzt den Fall, dass das Mex-3/Caudal System eine spezifische Anpassung von Kurzkeimern darstellt, werden ebenfalls die Homologe von mex-3 und caudal in der hemimetabolen Wanze Oncopeltus fasciatus analysiert, welche den gleichen Segmentierungsmodus wie Tribolium aufweist. 47 II. Kapitel: Mex-3/Cad System - Material & Methoden Material & Methoden Im Rahmen dieses Projekts wurde ebenfalls mit dem im vorangegangenen Kapitel vorgestellten Bohnenkäfer Bruchidius obtectus gearbeitet. Die verwendeten Methoden wurden darin bereits näher beschrieben. Neu hingegen ist der Modellorganismus Oncopeltus fasciatus, eine in der Natur an Pflanzen saugende Wanze, deren Handhabung im Folgenden zusammengefasst wird. Der Zuchtansatz wurde von Kristen Panfilio aus Köln bereit gestellt. Oncopeltus fasciatus: Haltung und Zucht Die Haltung von Oncopeltus fasciatus gestaltet sich als unkompliziert und die Population vermehrt sich sehr schnell. Nahrung: Oncopeltus fasciatus wird in verschließbaren Plastikboxen (15 cm x 15 cm x 12 cm) gehalten. Für eine ausreichende Luftzufuhr haben die Plastikboxen am Deckel eine Öffnung, die mit einem engmaschigen Netz verklebt ist. Der Boden der Box ist mit einem Papiertuch ausgelegt, so dass sich die Wanzen verstecken können. Der Austausch dieses Papiertuchs erleichtert auch eine regelmäßige Säuberung, da damit ein großer Teil des Wanzenkots entfernt wird. Ein kleines Petrischälchen mit Sonnenblumenkernen (Seeberger) dient als Nahrungsquelle. Zum Trinken reicht ein kleiner, mit Wasser gefüllter Erlenmeyerkolben, wobei ein zusammengefaltetes Papiertuch als Docht dient. Andernfalls fallen die Wanzen in den Wasserbehälter und ertrinken. Für die Eiablage wird ein Wattebausch aus Baumwolle zugegeben. Dieser wird sofern keine Eier benötigt werden, alle 3-4 Tage entfernt und eingefroren wodurch die Zuchtgröße halbwegs konstant bleibt. Temperatur: Die Wanzenzuchtboxen werden in einen Inkubator auf 25 °C gestellt. Vom Ei bis zur Nymphe dauert es 6-7 Tage, bis zum adulten geschlechtsreifen Tier 4-5 Wochen. Pflege: Die Zuchtkäfige werden im regelmäßigen Abstand von 2-3 Wochen komplett gesäubert, da sie von den Tieren stark verunreinigt werden. Dabei entfernt man auch Exuvien und tote Tiere. 48 II. Kapitel: Mex-3/Cad System - Material & Methoden Die Separierung verschiedener Entwicklungsstadien Eier: Oncopeltus fasciatus legt seine Eier in die ausgelegte Watte. Die Eier sind knapp 2 mm lang. Junge Eier sind eher gelblich; mit zunehmendem Altern werden sie orange und schließlich dunkelrot. Dadurch heben sie sich immer gut von der weißen Watte ab und können mit einer Minutiennadel vorsichtig von dieser abgelöst werden. Nymphen und adulte Wanzen: Für die Durchführung von RNAi-Experimenten werden die Nymphen aus den Anzuchtboxen entnommen. Es werden die ältesten nymphalen Stadien genommen, in Männchen und Weibchen sortiert (Unterscheidung, siehe weiter unten) und separat in Anzuchtboxen gehalten, bis sich die letzte Häutung zum adulten Tier vollzogen hat. Die Unterscheidung Männchen/Weibchen in Oncopeltus Männchen und Weibchen können anhand ihrer ventralen Musterung an den abdominalen Segmenten voneinander unterschieden werden. Oncopeltus-Weibchen bilden sowohl am vorletzten als auch am letzten abdominalen Segment als Nymphen einen schwarzen Fleck aus, während sie als adulte Tiere ventral einen breiten Strich, gefolgt von zwei großen schwarzen Flecken ausbilden. Oncopeltus-Männchen haben als Nymphen einen schwarzen Fleck am letzten Hinterleibssegment, als adulte Tiere ventral zwei durchgehende Streifen (Abb. 13: A, B). Tendenziell sind die Weibchen sowohl im letzten Nymphenstadium als auch als adulte Tiere etwas größer als die Männchen. Abb. 13: Unterscheidung von Männchen und Weibchen in Oncopeltus 49 II. Kapitel: Mex-3/Cad System - Material & Methoden A) Ventrale Ansicht einer männlichen Nymphe. Posterior ist der schwarze Fleck erkennbar (Pfeil und nebenstehende Skizze). Weibchen haben posterior zwei schwarze Flecken (s.h. Skizze). B) Ventrale Ansicht eines adulten Weibchens mit charakteristischer ventraler Zeichnung. Rechts daneben, Skizzen der Musterung im Vergleich Männchen/Weibchen. Fixierung der Oncopeltus-Embryonen Initiale Hitzefixierung: Nachdem die Oncopeltus-Eier aus der Watte abgesammelt worden sind, werden sie in ein Szintillationsgläschen mit Wasser gegeben. Dieses Gläschen stellt man mit samt den Eiern für exakt 2 min in einen kleinen Topf mit kochendem Wasser. Anschließend werden mit einem Pinsel die auf der Wasseroberfläche schwimmenden Eier aufgenommen und in ein Fixierungsgläschen mit dem Standard Fixierungsansatz (2 ml PEMS, 6 ml Heptan, 300 µl Formol (37%)) überführt. Dieser Ansatz kommt dann zum Fixieren für 20 Minuten auf einen Schüttler. Diese Fixierungszeit sollte nach Möglichkeit nicht überschritten werden, weil sonst das Chorion und die Vitellinhülle sehr fest werden und die weitere Bearbeitung zeitaufwendiger wird. Dechorionisierung und Devitellinisierung: Die mittlerweile an fixierten Eier werden in ein Salznäpfchen mit 100% Methanol transferiert. Nach ungefähr 5 Minuten ist ein Großteil der Eihüllen durch das eindringende Methanol geplatzt. Die Entfernung der Eihüllenreste erfolgt mit zwei Minutiennadeln unter dem Binokular. Finale Fixierung: Auf diese Art gehäutete Eier kommen erneut in ein Fixierungsgläschen und werden noch einmal nachfixiert, dieses Mal für 45 Minuten. Danach können die Eier in 100% Methanol bei -20 °C aufbewahrt werden. Klonierung von mex-3, caudal und zen Da beide Organismen zum Bearbeitungszeitpunkt noch nicht sequenziert worden waren, wurden erneut degenerierte Primer erstellt. Einzige Ausnahme ist Oncopeltus caudal: Vor einigen Monaten wurde für diesen Organismus ein maternales Transkriptom erstellt und mir wurde die exakte O.f. caudal-Sequenz von dem Bioinformatiker Ho-Ryun Chung zur Verfügung gestellt. Die Primer sind von Metabion. Die Herstellung von Oncopeltus-cDNA und Bruchidius-cDNA, sowie die einzelnen Klonierungsschritte richten sich nach den Standardmethoden, die bereits in Material & Methoden des ersten Kapitels skizziert wurden. Onco- 50 II. Kapitel: Mex-3/Cad System - Material & Methoden peltus zen wurde nicht kloniert, da bereits bekannt ist, dass es in der frühen Segmentierung keine Rolle spielt (Panfilio et al., 2006). Die Sequenzen finden sich unter Anhang B. Tab. 3: Primer für das Mex-3/Cad-Projekt Primer Sequenz 5’-3’ Tm Länge O.f. fw mex-3 O.f. bw mex-3 GAR CAY GTN GCN GAR ATH GT ATR TAN GTR TGN GTY TGR TGY TG (58 °C) (60 °C) 930 bp O.f. fw cad O.f. bw cad ACC ATC TGG AAC AGC CCG TGC GAG GCG AGC TCC GAT TTT CGC C (65 °C) 283 bp (68 °C) B.o. fw mex-3 CAR AAY ATG ACN GAR TGY GTN CC (63 °C) 1035 bp B.o. bw mex-3 CAT NGC DAT RTG NGC YTC DAT YTC (64 °C) B.o. fw cad B.o. bw cad CAR WSN CAR CCN AAY CC YTT NCK NCK RTT YTG RAA CC (54 °C) 189 bp (56 °C) B.o. fw zen B.o. bw zen TNA RYT NGA RAA RGA RTT YC YTT NCK NCK RTT YTG RAA CC (52 °C) (56 °C) 115 bp In-situ-Hybridisierung von Oncopeltus Das Protokoll stammt von Ariel Chipman. Die dafür benötigten Lösungen sind im Anhang A zusammengefasst. Die Herstellung der DIG-markierten Sonden richtet sich nach Standardmethoden. Die engrailed- und even-skipped-Klone von Oncopeltus wurden von Paul Liu zur Verfügung gestellt. Tag 1: 1. Die in 100% igem Methanol befindlichen Embryonen schrittweise in PBT überführen: 25%, 50%, 75%, 100%. 2. 3 x in 100% PBT waschen. 3. In 1 ml O.f Hyb-Puffer für 4 h (!!) bei 60 °C im Wasserbad prähybridisieren. 4. Hyb-Puffer auf 500 µl reduzieren und 3-5 µl Sonde zugeben. 5. Über Nacht bei 60 °C im Wasserbad hybridisieren. 51 II. Kapitel: Mex-3/Cad System - Material & Methoden Tag 2: 6. 30 min in 1 ml frischen, vorgewärmten Hyb-Puffer waschen (60 °C). 7. 2 x 15 min in 1 ml Hyb-Puffer waschen (60 °C). 8. 1 x 15 min in 2x SSC Lösung inkubieren (60 °C). 9. 3 x 12 min in 0.2x SSC Lösung waschen (60 °C). 10. Bei RT 3 x 5 min in PBT waschen; für 2 h blocken (200 µl BBR (2.5%)/800 µl PBT). 11. Zugabe von 1 µl Anti-Dig AP Fab (Roche). Über Nacht bei 4 °C im Kühlschrank stehen lassen. Tag 3: 12. 5 x 20 min in PBT bei RT waschen. 13. 2 x 5 min in Färbepuffer 1 waschen und mit 15 µg/µl NBT/BCIP Färbung entwickeln. Nach dem Abschluss der Färbereaktion, 3-4 Mal in PBT waschen und mit Hoechst (1:20) gegen färben. Parentale RNAi in Oncopeltus Die verwendete Doppel-Strang-RNA wurde mit dem T7-Megaskript Kit (Ambion) synthetisiert. Nachdem die wie oben beschrieben vorsortierten weiblichen Nymphen ihre finale Häutung zum adulten Tier abgeschlossen haben, werden sie nach Standardprotokoll injiziert. Es hat sich bewährt die Tiere in durchsichtige Küchenfolie einzuwickeln, da sie dadurch völlig regungslos sind. Man dreht die Tiere auf die dorsale Seite und reißt mit einer Pinzette ein kleines Loch in die Folie, so dass man mit der Injektionsnadel lateral zwischen die hinteren abdominalen Segmente einstechen kann. Das injizierte Volumen an dsRNA beträgt soviel, dass sich das Abdomen der Wanze leicht streckt. Das Anfärben der dsRNA mit grüner Lebensmittelfarbe hilft zu erkennen, ob die Injektion erfolgreich war, da diese sich sehr gut vom orangen Abdomen abhebt. Nachdem das entsprechende Tier injiziert ist, wird es zusammen mit einem Männchen in ein Fliegenröhrchen mit ausreichend Wasser, Sonnenblumenkerne und Baumwollwatte gesetzt. Bis zur ersten Eiablage dauert es rund fünf Tage. 52 II. Kapitel: Mex-3/Cad System - Ergebnisse Ergebnisse Das in der Einleitung vorgestellte Mex-3/Cad System soll nun in zwei anderen Insekten, Bruchidius und Oncopeltus, untersucht werden. Dazu wurden zunächst aus diesen Organismen jeweils mex-3 und caudal kloniert. Anschließend fand die Analyse der Expression und Funktion der Gene statt, welche im Folgenden für die beiden Modellorganismen separat dargestellt wird. Die Caudal- und Mex-3-Orthologe in Bruchidius und Oncopeltus Drosophila caudal codiert ein Homeodomänenprotein, welches als Transkriptionsfaktor im Rahmen der frühen Embryogenese fungiert (Mlodzik et al., 1985). Der klonierte Bereich des caudal-Homologs für Oncopeltus beträgt 283 bp, der für Bruchidius 189 bp. Vergleicht man die Aminosäurensequenzen der Caudal-Homeodomäne zwischen den beiden Organismen (Abb. 14: A), zeigt sich, dass Bruchidius und Tribolium Caudal eine Sequenzähnlichkeit von 84% aufweisen und die Übereinstimmung von Bruchidius und Oncopeltus Caudal sogar 86% beträgt. Sowohl für Oncopeltus als auch für Bruchidius wurde mit Hilfe von degenerierten Primern jeweils ein mex-3-Fragment kloniert (B.o. mex-3: 1035 bp; O.f. mex-3: 930 bp), das die beiden beschriebenen KH-Domänen umspannt (Draper et al., 1996). KH-Domänen bestehen aus ca. 50 Aminosäuren und sind dafür bekannt RNA binden zu können (Siomi et al., 1994; Pagano et al., 2009). Ein Ausschnitt des Alignments auf Proteinebene zeigt die erste KH-Domäne von verschiedenen Spezies (Abb. 14: B). Zwischen Bruchidius und Tribolium beträgt die Übereinstimmung der Aminosäuresequenz 100%, bei der zweiten KH-Domäne unterscheiden sie sich nur in einem einzigen Aminosäureaustausch (nicht gezeigt). Auch ist Sequenzähnlichkeit zwischen Bruchidius und Oncopeltus sehr hoch. Ist in der ersten KHDomäne ein Aminosäureaustausch zu verzeichnen, sind es in der zweiten KH-Domäne vier (nicht gezeigt). Die erste und zweite KH-Domäne sind durch eine kurze Aminosäuresequenz verbunden (Abb. 14: B, blau unterstrichen), welche zwischen den verschiedenen Organismen ebenfalls stark konserviert ist, so dass davon auszugehen ist, dass es sich bei den klonierten Bruchidius und Oncopeltus Sequenzen tatsächlich um mex-3-Homologe von Tribolium bzw. C.elegans handelt. 53 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 14: Alignment der funktionellen Domänen von Caudal bzw. Mex-3 A) Alignment der Caudal-Homeodomäne von O.f. (Oncopeltus fasciatus), B.o. (Bruchidius obtectus), T.c. (Tribolium castaneum; NP_001034498.1) und D.m. (Drosophila melanogaster ; AAF53923.1). B) Alignment der ersten Mex-3 KH-Domäne von Bruchidius u. Oncopeltus. Schwarz unterlegt ist die erste KH-Domäne; der Bereich zwischen erster und zweiter KH Domäne, blau. Der Vergleich der Aminosäuresequenz von T.c. (Tribolium castaneum; NP_001137201.1), B.o. (Bruchidius obtectus), O.f. (Oncopeltus fasciatus) sowie C.e. (Caenorhabditis elegans; AAC47313.1) stellt die konservierte Sequenzidentität der KH-Domäne dar. Posteriore Expression von B.o. caudal während der Embryogenese B.o caudal wird im Blastoderm (Abb. 15: A’) in einer breiten Domäne exprimiert, die sich von der posterioren Eispitze bis in den anterioren Bereich des Eies erstreckt, so dass die caudal-Expressionsdomäne ca. 75% des Eies umfasst. Es scheint dabei so, dass am posterioren Pol die Expression deutlich stärker ausgeprägt ist und nach anterior hin graduell abfällt 54 II. Kapitel: Mex-3/Cad System - Ergebnisse (Abb. 15: A). Im Zuge der Keimstreifbildung wird die caudal-Expression auf einen schmalen Streifen am posterioren Ende des Embryos begrenzt (Abb. 15: B), welcher dort während der gesamten Keimstreifstreckung in gleicher Intensität und Ausdehnung bestehen bleibt (Abb. 15: C) und wahrscheinlich an der Entwicklung der Hinterdarmanlagen beteiligt ist, wie das ebenfalls in Drosophila gezeigt werden konnte (Wu und Lengyel, 1998). Abb. 15: Expression von caudal in Bruchidius Die anteriore Seite der Embryonen zeigt nach links. A) Im Blastoderm Stadium wird B.o. cad in einer breiten, posterioren Domäne exprimiert. A’) Hoechstfärbung zur Charakterisierung des Stadiums. B) Im jungen Keimstreif beschränkt sich die Expression auf eine schmale Bande im hinteren Bereich des Embryos. C) Dieser distinkte Expressionsbereich bleibt während der gesamten Keimstreifstreckung bestehen. B.o. caudal spielt eine wichtige Rolle in der frühen Embryonalentwicklung Die Analyse der Expression lässt bereits vermuten, dass Bruchidius caudal eine zentrale Funktion in der Segmentierung inne hat, was die B.o. caudal-RNAi-Experimente widerspiegeln. Es wurden unterschiedliche Konzentrationen an caudal Doppelstrang-RNA in weibliche Puppen injiziert, doch selbst bei sehr niedrigen Konzentrationen wie 100 ng/µl entwickeln B.o. cad-RNAi-Embryonen keine Kutikula, so dass keine phänotypische Reihe erstellt werden kann. 55 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 16: Vergleich von Hoechst gefärbten WT und B.o. cad -RNAi-Embryonen Die anteriore Seite der Embryonen ist nach links orientiert. Es wird die Entwicklung des Wildtyps, mit Embryonen nach parentaler cad-RNAi verglichen. A), B) zeigen ein früheres und ein späteres Blastoderm-Stadium im Wildtyp. Im Vergleich dazu unterscheiden sich cad-RNAi-Embryonen zu diesen Entwicklungszeitpunkten nicht (A’, B’). Mit dem Beginn der Zellkondensation an der ventralen Seite zur Ausbildung des Keimrudiments (C, Pfeile) werden die ersten Unterschiede zu cad-RNAi-Embryonen sichtbar, da hier eine Zellbewegung nach posterior stattfindet (C’, Pfeile). Das gestreckte Keimstreifstadium D) (weißer Balken markiert die Kopflappen) wird nach cadRNAi nicht erreicht. Am posterioren Pol kommt es zu einer Ansammlung von Gewebe wobei es sich um Ansätze von Kopfstrukturen handeln könnte (D’, weißer Balken). Die Bruchidius-WT-Bilder sind aus meiner Diplomarbeit entnommen (Diplomarbeit, Schnellhammer 2007). 56 II. Kapitel: Mex-3/Cad System - Ergebnisse Jedoch konnte durch Kernfärbung der embryonalen Entwicklungsstadien nach B.o. cadRNAi die Beeinträchtigung der frühen Musterbildung dokumentiert werden. Laufen die anfänglichen Kernteilungen bis zum Erreichen des Blastodermstadiums noch regelmäßig und wie im Wildtyp ab (Abb. 16: A bis B’), werden die ersten Defekte bei der Ausbildung des Keimrudiments sichtbar. Normalerweise wandert der Großteil der Zellen in Richtung ventral und bildet dort die embryonalen Strukturen aus. Auf der dorsalen Seite sind weniger Zellkerne, welche die Anlage der extraembryonalen Membran darstellen (Abb. 16: C, S). Nach caudal-RNAi kann anstatt einer koordinierten Zellbewegung auf die ventrale Seite (Abb. 16: C, Pfeile) eine Verdichtung der Zellen am posterioren Pol beobachtet werden (Abb. 16: C’, Pfeile). Aufgrund dessen wird das Stadium des gestreckten Keimstreifs (Abb. 16: D) nicht erreicht, sondern es bildet sich schließlich ein Geweberest aus. Dieser befindet sich in der posterioren Eispitze, durchläuft keine Keimstreifstreckung und stellt vermutlich Reste von Kopfstrukturen dar (Abb. 16: D’). Zusammengefasst zeigen sowohl die Expressionsdaten als auch die funktionalen Daten von Bruchidius caudal, dass caudal in der Embryonalentwicklung dieses Langkeimkäfers eine wichtige Rolle spielt. Nun stellt sich die Frage nach der caudal-Regulation und welchen Einfluss Bruchidius zen und mex-3 darauf haben. Bruchidius zen wird dorsal im Ei exprimiert In Tribolium ist zen-2 im anterioren Drittel des Eies exprimiert. In diesem Bereich entwickelt sich die Serosa, eine extraembryonale Membran (Van der Zee et al., 2005). Zen-2 ist in die anteriore Caudal-Repression involviert, da ein Knockdown von zen-2 via RNAi in einer Expansion von Caudal in diese Region resultiert (Schoppmeier et al., 2009). In Bruchidius wurde auch ein zen-Ortholog gefunden und es stellte sich heraus, dass Bruchidius zen ebenfalls sehr früh exprimiert ist. Im Gegensatz zu Tribolium wird B.o zen in einer engen distinkten Domäne die sich über die dorsale Seite des ganzen Embryo erstreckt exprimiert (Abb. 17: A, A’) und ist vermutlich an der Initiation der dorsalen Serosaanalge beteiligt, wie das bereits für andere Insekten gezeigt werden konnte (Rushlow et al., 1987; Van der Zee et al., 2005). Da sich im Langkeiminsekt Bruchidius die Serosa vor allem auf der dorsalen Seite entwickelt, ist nicht anzunehmen dass eine Repression von Caudal durch Zen entlang der anterioren-posterioren Achse eine Rolle spielt, wie das in Tribolium der Fall ist (Schoppmeier et al., 2009). zen-RNAi in Bruchidius resultiert in Larven, welche eine Trans57 II. Kapitel: Mex-3/Cad System - Ergebnisse formation des ersten abdominalen Segments in ein thorakales Segment aufweisen (nicht gezeigt). Dies scheint aber ein Off-Target Effekt zu sein, da nur ein sehr kleiner Ausschnitt der zen-Homeodomäne kloniert worden ist und durch RNAi wahrscheinlich ein anderes Hoxgen herunterreguliert wurde. Da die vermutliche Funktion von zen in der Entwicklung der Serosa im Zusammenhang mit dem gestellten Thema nicht von Interesse war, wurde davon abgesehen ein längeres zen-Fragment zu klonieren um somit einen spezifischen RNAi-Effekt zu erzielen. Abb. 17: Die Expression von Bruchidius zen im Blastoderm Die anteriore Seite des Embryos zeigt nach links; die dorsale Seite des Eies zeigt zum Betrachter. A) Bruchidius zen kann im Blastoderm auf der dorsalen Eiseite in einem schmalen Streifen detektiert werden, der mit der zukünftigen Serosaanlage zusammen fällt. A’) Hoechstfärbung zur Charakterisierung des Stadiums: Blastoderm. Expression von Bruchidius mex-3 während der Embryogenese Bruchidius mex-3 wird während der gesamten Embryonalentwicklung hindurch in einem dynamischen Muster exprimiert: Im Kernschar-Stadium, wenn die Kernteilungen sich noch auf das Zentrum des Eies beschränken, ist eine leichte anteriore Expression von B.o. mex-3 zu finden, die sich bis zur Hälfte des Eies erstreckt (Abb. 18: A, A’). Da zu diesem Zeitpunkt noch keine zygotische Transkription stattfindet, könnte es sich um eine maternale Expression von B.o. mex-3 handeln. Mit der Ausbildung des frühen Blastoderm Stadiums breitet sich mex-3 über das ganze Ei hinweg aus (Abb. 18: B, B’) und wird erst nach der Wanderung der zukünftigen embryonalen Zellen auf die ventrale Seite des Eies in einem schmalen anterioren 58 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 18: Die Expression von mex-3 in der Bruchidius-Embryogenese 59 II. Kapitel: Mex-3/Cad System - Ergebnisse Dargestellt ist die Expression von mex-3 in verschiedenen Bruchidius-Entwicklungsstadien. A’-D’ zeigen Hoechst-Färbungen zur näheren Charakterisierung des Stadiums. Noch während der ersten Kernteilungen kann eine schwache anteriore Expressionsdomäne von mex-3 in Bruchidius beobachtet werden (A, A’), die sich im frühen Blastoderm über das gesamte Ei ausbreitet (B, B’). Mit der Ausbildung der ventralen Keimanlage ist eine definierte anteriore Expressionsdomäne zu sehen (C, C’). Diese wird in einem jungen Keimstreif von einer in Ansätzen segmentalen Musterung begleitet (D, D’), die auch in älteren Stadien bestehen bleibt und von einer posterioren Expression begleitet wird (E). Im gestreckten Keimband ist eine kräftige Expression in den Kopflappen und entlang der ventralen Mittellinie zu finden (F). Während des Rückenschlusses bleibt die Expression im Kopf bestehen (G) und auch eine schwache Expression in den ventralen Zellklustern ist vorhanden (H). Bereich des Embryos exprimiert, der mit den Kopfanlagen zusammenfällt (Abb. 18: C, C’). Diese anteriore Kopfdomäne wird in einem älteren Stadium von einer fast segmental erscheinenden posterioren Expressionsdomäne begleitet (Abb. 18: D, D’, E). Die extraembryonale Membran, die dorsal gelegene Serosa (S), welche anhand der weiter voneinander liegenden Zellkernen leicht ausgemacht werden kann und der kleine Teil der Serosa, die an der vordersten Eispitze gelegen ist, bleiben in allen Stadien frei von der mex-3-Expression (Abb. 18: C, D). Im gestreckten Keimstreif ist die Kopfexpression weiterhin präsent und entlang der ventralen Mittellinie entsteht eine durchgehende Expression von mex-3, die mit der Ausbildung neuronalen Zellen zusammen fallen könnte (Abb. 18: F). Mit dem Einsetzen des Rückenschlusses bleibt die ventrale neuronale Expression erhalten und auch die Expression im Kopfbereich ist weiterhin detektierbar (Abb. 18: G, H). Bruchidius mex-3-RNAi beeinträchtigt die Segmentierung Die Funktion von mex-3 in der Bruchidius-Embryogenese wurde mittels parentaler RNAi untersucht. Es konnte eine phänotypische Reihe erstellt werden, die in Abb. 19 dargestellt ist. Die ventrale Ansicht der Wildtyplarve zeigt die stark autofluoreszierenden Mandibeln im Kopfbereich, drei thorakale und acht abdominale Segmente (Abb. 19: A). In schwachen mex-3-RNAi-Phänotypen der Kategorie I (Abb. 19: B) findet man Deletionen im Bereich der ersten abdominalen Segmenten, sowie eine Störung der Musterung im posterioren Bereich (Pfeil). In stärkeren Phänotypen der Kategorie II (Abb. 19: C) sind die thorakalen Segmente T2 und T3 nur noch partiell vorhanden und die gesamte abdominale Segmentierung ist beeinträchtigt, da nur noch Reste von Zähnchenbändern zu erkennen sind. Schließlich ist im stärksten Phänotyp der Kategorie III (Abb. 19: D) lediglich der Kopf vollständig entwickelt 60 II. Kapitel: Mex-3/Cad System - Ergebnisse und einzelne Beine von T1 und T2 sind vorhanden, ansonsten ist die gesamte Segmentierung gestört. Bei diesem Versuch war die Penetranz der erhaltenen Phänotypen relativ gering (Tabelle 4), da der Anteil an mex-3-RNAi-Phänotypen bei 15,5% lag. Um auszuschließen, dass mex-3-RNAi in einer verminderten Eiablage oder einer erhöhten frühen Letalität der Embryonen resultiert, weswegen ein großer Teil der Phänotypen auf Kutikulaebene nicht analysiert werden kann, wurden alle gelegten Eier nach mex-3-RNAi gezählt und ausgewertet, wie viele Embryonen sich noch entwickeln konnten. Von insgesamt 437 Eiern blieben 102 Eier unentwickelt, was bedeutet dass diese Eier nur aus der Eihülle und Dotterresten bestanden. Insgesamt entspricht das einem Hintergrund an defekten Eiern von ca. 23,3%. Der Hintergrund an defekten Eiern in Bruchidius und Tribolium liegt auch in Wildtypablagen meist zwischen 20 und 30%. Dies zeigt, dass nach mex-3-RNAi in Bruchidius weder die Oogenese beeinträchtigt ist, noch eine erhöhte frühe Letalität der Embryonen festgestellt werden kann. Abb. 19: Beeinträchtigung der Segmentierung nach B.o. mex-3 -RNAi Alle Kutikulapräparate zeigen die ventrale Ansicht der Larven. Der Messbalken stellt die Länge 100 µm dar. A) Bruchidius Wildtyp Larve, bestehend aus Kopf, den thorakalen Segmenten und den acht abdominalen Segmenten, mit den charakteristischen Zähnchenbändern. B) Schwacher Phänotyp der Kategorie I, Deletionen im zentralen Bereich der Larve sowie eine Störung in der posterioren Musterbildung bei A8 (Pfeil). C) Stärkere Phänotypen zeigen eine zunehmende Deletion posteriorer Segmente, während der stärkste Phänotyp der Kategorie III nur noch aus einem intakten Kopf sowie Rudimenten der ersten zwei thorakalen Segmente besteht (D). 61 II. Kapitel: Mex-3/Cad System - Ergebnisse Tab. 4: Verteilung der Phänotypen nach B.o. mex-3 -RNAi (c=300 ng/µl) Phänotyp WT I II III unentwickelt mex-3 -dsRNA 58 (55.7%) 9 (8.7%) 6 (5.8%) 1 (1%) 30 (28.4%) Kontrolle 0 0 0 10 (29.4%) 24 (70.6%) Eine Möglichkeit die Penetranz zu erhöhen wäre, ein weiteres Fragment zu klonieren, welches andere Bereiche der mex-3-Sequenz umspannt, was sich aber im Rahmen dieser Arbeit nicht durchführen ließ, da keine funktionierenden degenerierten Primer anhand von Konsensus-Sequenzen außerhalb der stark konservierten KH-Domänen designed werden konnten. Was die Gründe für die geringe Anzahl an Phänotypen im Rahmen der Bruchidius mex-3-RNAi-Experimente sein könnte wird in der Diskussion näher erörtert. Posteriore Expression von O. f. caudal während der Embryogenese Die Oncopeltus-Embryogenese beginnt ebenfalls mit einem Kernscharstadium bei dem sich die Kernteilungen auf das Zentrum des Eies beschränken (Butt, 1949; Liu und Kaufman, 2004). Daran schließt sich ein Blastodermstadium an. Die Bildung des Keimrudiments verläuft etwas anders als bei Bruchidius, da in Oncopeltus die sogenannten lateralen Seitenplatten, welche durch eine Verdichtung der Zellkerne entlang der anterioren-posterioren Achse entstehen (Abb. 20: A, gestrichelte Linie), nicht auf der ventralen Seite fusionieren, sondern nach posterior wandern, dort fusionieren und in den Dotter eintauchen (Abb. 20: B). Durch diesen Vorgang, Anatrepsis genannt, liegt der Kopf des Embryos nun auf der posterioren Seite des Eies und der abdominale Bereich ist zur anterioren Eispitze hin orientiert (Abb. 20: C). Nach der Keimstreifstreckung erfolgt eine Rotation des Embryos (Abb. 20: D), welche als Katatrepsis bezeichnet wird (Panfilio et al., 2006), so dass gegen Ende der Embryogenese der Embryo wieder mit dem Kopf zur anterioren Eiseite zeigt (Butt, 1949; Liu und Kaufman, 2004). Oncopeltus caudal wird im Blastoderm (Abb. 21: A’) relativ schwach und in einer breiten Domäne exprimiert (Abb. 21: A). Nur in der anterioren Eispitze ist caudal nicht aktiv. Während des Keimstreifwachstums wird O.f. cad durchgehend an der posterioren Spitze des Keimstreifs in einer schmalen Bande exprimiert (Abb. 21: B, C). 62 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 20: Übersicht der Oncopeltus-Embryogenese Alle Bilder sind so ausgerichtet, dass die anteriore Eiseite nach links zeigt. Bei A und D handelt es sich um eine Kernfärbung mit Hoechst, bei B und C um eine mex-3 in-situ-Färbung, wobei der Fokus auf den embryonalen Anlagen liegt. A) Laterale Ansicht eines Blastoderms mit beginnender Seitenplattenbildung, welche durch die gestrichelte Linie hervorgehoben ist. B) Dorsale Ansicht der posterior in den Dotter einwandernden lateralen Seitenplatten (Pfeile deuten die Bewegungsrichtung der Einwanderung an). Die Kopflappen (KL) wandern zum Schluss ein, so dass in C) der Embryo mit dem Kopf zur posterioren Eiseite hin orientiert ist. D) Gegen Ende der Embryogenese dreht sich der Embryo erneut, relativ zur Eiachse, so dass der Kopf wieder nach anterior zeigt. Abb. 21: Posteriore caudal -Expression in Oncopeltus 63 II. Kapitel: Mex-3/Cad System - Ergebnisse A) Im Blastoderm ist caudal schwach von posterior nach anterior bis zum ersten Viertel der Eispitze exprimiert. A’) Die Hoechst Färbung des gleichen Embryos wie A, zeigt das noch undifferenzierte Blastoderm. Während der Keimstreifstreckung ist caudal durchgehend in der posterioren Wachstumszone am äußersten Ende des Embryos detektierbar. B) zeigt einen jüngeren Keimstreif, C) einen etwas älteren mit beginnender Extremitätenbildung. Während der Keimstreifstreckung wird caudal in der posterioren Wachstumszone exprimiert. Dieser Bereich des Embryos stellt die Wachstumszone dar, aus welcher der Kurzkeimer Oncopeltus alle abdominalen Segmente heraus bildet (Liu und Kaufman, 2005a), so dass Oncopeltus caudal während der gesamten Embryogenese eine wichtige Rolle in der Wachstumszone zu spielen scheint. O.f. cad-RNAi führt zu einem frühzeitigen Abbruch der Segmentierung Zeigen bereits die in-situ-Hybridisierungen von Oncopeltus caudal, dass die Funktion von caudal in der posterioren Musterbildung vermutlich auch im Kurzkeimer Oncopeltus konserviert ist, untermauern cad-RNAi-Experimente dies zusätzlich. Als Kontrolle wurden parallel Oncopeltus-Weibchen statt mit Doppelstrang-RNA mit Wasser injiziert. Alle daraus resultierenden Nachkommen entwickelten sich wie im Wildtyp und schlüpften nach Ablauf von einer Woche (25 °C) aus den Eiern (Tabelle 5). Die Präparation einer Oncopeltus-Wildtyp-Kutikula (Abb. 22: A) zeigt deutlich die stechend saugenden Mundwerkzeuge der Wanze. Im Inneren des Kopfes befinden sich, wie zu zwei Fadenrollen aufgewickelt, die Mandibeln und Maxillen welche nach Abschluss der Entwicklung nach außen verlagert und vom langen rüsselförmigen Labium eingeschlossen werden. An die thorakalen Segmente mit den langgliedrigen Beinen schließen sich acht abdominale Segmente an. Nach parentaler cad-RNAi stellt sich mit zunehmender Stärke der Phänotypen eine posteriore Verkürzung ein. Die Phänotypen können in zwei Kategorien eingeteilt werden, welche ungefähr gleich häufig auftreten (Tabelle 5). Phänotypen der Kategorie I (Abb. 22: B) bilden zwar einen intakten Kopf und Thorax aus, haben aber eine verringerte Anzahl an abdominalen Segmenten. Im gezeigten Beispiel wären dies nur noch A1 und A2. In den stärksten Phänotypen der Kategorie II (Abb. 22: C) bricht die Segmentierung nach der Anlage der anteriorsten Kopfstrukturen, welche Antennen und Augen beinhalten, komplett ab. Der Anteil an unentwickelten Eier, die nur aus der Eihülle und Dotterresten bestehen, liegt nach cad-RNAi in Oncopeltus bei ca. 1,6% (Tabelle 5). 64 II. Kapitel: Mex-3/Cad System - Ergebnisse Tab. 5: Verteilung der Phänotypen nach O.f. caudal -RNAi (c=3 µg/µl) Phänotyp WT I II unentwickelt cad -dsRNA 24 (4,73%) 206 (40,63%) 269 (53,1%) 8 (1,6%) Kontrolle 207 (100%) 0 0 0) Die Analyse des Markergens engrailed nach Oncopeltus cad-RNAi Um die auf Kutikulaebene erhaltenen Phänotypen weiter zu analysieren wurde die Expression von engrailed nach caudal-RNAi in Oncopeltus untersucht. Im Wildtyp wird engrailed in Oncopeltus als Segmentpolaritätsgen exprimiert (Liu und Kaufman, 2004), was bedeutet dass in jedem Segment, beginnend von den Antennen über die gnathalen bis hin zu den abdominalen Segmenten, engrailed exprimiert ist (Abb. 22: D). In schwächeren cad-RNAi-Phänotypen ist die Expression in den Segmenten, von den Antennen beginnend bis hin zu A1 vorhanden, wirkt aber bereits etwas ungeordnet und die Streifen in den jeweiligen Segmenthälften scheinen nicht exakt aneinander anzugrenzen (Abb. 22: E). Noch drastischer ist die Situation in den stärksten Phänotypen (Abb. 22: F): Zwar ist das Expressionsmuster von engrailed in den Antennen bis hin zum interkalaren Segment intakt, allerdings wird in weiter posterior gelegenen Segmenten die Expression nur noch rudimentär angelegt und keine durchgehenden Streifen sind mehr zu beobachten. Ein weiterer früher Effekt der nach cad-RNAi beobachtet werden kann ist, dass der Embryo nicht mehr an der posterioren Seite des Eies in den Dotter eintaucht (Abb. 23: A, Pfeil), sondern an einer beliebigen Stelle im hinteren Bereich des Eies. Im gezeigten Beispiel ist es die dorsale Eiseite, aber auch das Eintauchen an den lateralen Seiten wurde beobachtet (nicht gezeigt). Die Folge davon ist, dass der Embryo nicht mehr richtig auswachsen kann und eine stark verkrümmte Form annimmt (Abb. 23: B, Pfeil). Die funktionale Analyse von Oncopeltus caudal zeigt, dass caudal sowohl für die Ausbildung gnathaler und thorakaler Strukturen, die Positionierung des Keimbandes während der Invagination und in der posterioren Wachstumszone für die geordnete Bildung neuer Segmente verantwortlich ist. 65 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 22: Posteriore Segmentierungsstörung in Oncopeltus nach cad -RNAi Gezeigt werden Kutikulapräparationen und die engrailed-Expression nach cad-RNAi. A) Wildtyp Kutikula von Oncopeltus. Am Kopf fallen die stechend-saugenden Mundwerkzeuge auf (Md, Mx und Lb), am Thorax drei lange Beinpaare an die acht abdominale Segmente angrenzen. B) Schwacher cad-RNAi-Phänotyp der Kategorie I. Die Kopfstrukturen sind normal entwickelt, das Abdomen umfasst hingegen nur noch zwei abdominale Segmente. C) Sehr starker cad-RNAi Effekt der Kategorie II. Die gesamte posteriore Entwicklung ist gestört, lediglich die anteriorsten Strukturen wie Antennen und Augen sind erkennbar. Den Abbruch der Segmentierung spiegelt auch eine Färbung mit dem Segmentpolaritätsgen engrailed wider, welches im Wildtyp in regelmäßigen Streifen von den Antennen beginnend, über das interkalare Segment, den Anlagen der gnathalen und thorakalen sowie den abdominalen Segmenten exprimiert wird (D). In einem schwächeren Phänotyp der ebenfalls in die Kategorie I der phänotypischen Serie eingeordnet werden kann, ist die initiale engrailed-Färbung von den antennalen Segmenten bis zum ersten abdominalen Segment zwar vorhanden, wirkt aber unregelmäßig und bricht nach dem A1-Streifen ab (E). In stärkeren Phänotypen ist nur noch die engrailed-Musterung in den anterioren Strukturen korrekt (Ant, Ag und Ic) ansonsten ist sie nur noch rudimentär vorhanden (F). 66 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 23: O.f. cad -RNAi führt zur Störung der posterioren Invagination Dargestellt sind engrailed in-situ-Hybridisierungen nach O.f. cad-RNAi. Die Streifen sind bereits alle mit dem Keimrudiment in das Ei invaginiert und deswegen nicht zu sehen. Im Wildtyp (A) findet die Invagination des Keimrudiments an der posterioren Eispitze statt (Pfeil) und der Keimstreif streckt sich nach anterior. Nach cad-RNAi hingegen (B) kommt es häufig vor, dass der Embryo an einer beliebigen Stelle in das Ei hinein taucht (Pfeil) und daraufhin ungeordnet und faltig in den Dotter hineinwächst. Die Expression von mex-3 in der Oncopeltus-Embryogenese Nachdem im vorherigen Abschnitt die Bedeutung von Oncopeltus caudal in der Embryogenese untersucht worden ist, soll nun der Einfluss von mex-3 auf die Oncopeltus-Entwicklung näher analysiert werden. Im Kernscharstadium sowie früh im Blastoderm wird Oncopeltus mex-3 nicht exprimiert (nicht gezeigt). Mit der Ausbildung des späten Blastoderms (Abb. 24: A’), welches aus einer anterioren und dorsalen Serosaanlage sowie zwei lateralen Seitenplatten besteht, wird mex-3 in einem breiten von posterior nach anterior verlaufenden Bereich exprimiert (Abb. 24: A); nur die anteriore und dorsale Serosaanlage bleibt frei von Expression (Abb. 24: B). Sobald der Embryo anfängt in den Dotter einzutauchen wird mex-3 entlang des Keimrudiments exprimiert, vor allem anterior, im zukünftigen Kopfbereich (Abb. 24: C, D). Im Zuge der Keimstreifstreckung ist mex-3 in den Kopflappen sowie im posterioren Bereich des Embryos zu finden. Auch eine beginnende vermutlich neuronale Expression entlang der ventralen Mittellinie tritt auf (Abb. 24: E). Im vollständig elongierten Keimstreif (Abb. 24: F) besteht die Kopfexpression und die neuronale Expression fort. Zusätzlich treten an den Extremitäten distinkte Banden auf und auch an der posteriorsten Spitze des Keimbandes sind zwei kleine Expressionsbereiche erkennbar. Während der abschließenden Keimstreifverkürzung (Abb. 24: G) wird die Expression von mex-3 in den Antennen besonders deutlich und auch die Expression in den Beinen besteht fort. 67 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 24: Die Expression von mex-3 in der Oncopeltus-Embryogenese A) Im späten Blastoderm ist eine breite Expression von mex-3 erkennbar. A’) Hoechstfärbung von A. B) Dorsale Ansicht: Nur die anteriore und dorsale Serosaanlagen (S) bleiben frei von Expression. C) Mit dem Fortschreiten der Keimbandinvagination beschränkt sich die mex-3 -Expression auf die lateralen Seitenplatten und den zukünftigen Kopfbereich (Pfeil). D) Ist der Embryo fast vollständig in den Dotter eingetaucht, ist mex-3 weiterhin im anterioren Kopfgewebe aktiv. E) Im sich streckenden Keimstreif ist eine etwas stärkere Kopfexpression und abdominal eine mex-3 Domäne erkennbar. Auch entlang der ventralen Mittellinie wird mex-3 exprimiert (Pfeil). F) Im vollständig gestreckten Keimstreif bleibt die Kopfexpression erhalten, in den Appendices tritt eine Expression auf (kleine Pfeile), die neuronale mex-3 -Expression wird kräftiger und an der posteriorsten Spitze des Embryos sind zwei kleine Expressionsdomänen erkennbar (unterstrichen). G) Während der Keimstreifverkürzung bleibt die Expression in den Beinen bestehen und mex-3 wird in den distalen Antennenbereichen exprimiert (Pfeil). 68 II. Kapitel: Mex-3/Cad System - Ergebnisse O.f. mex-3 ist wichtig für die anteriore u. posteriore Musterbildung Die funktionale Analyse von O.f. mex-3 via parentale RNAi resultiert in einer Reihe von Phänotypen, die anhand ihrer Ausprägung in vier verschiedene Kategorien eingeteilt worden sind (Tabelle 6). Im Vergleich zum Wildtyp (Abb. 25: A) fallen in schwachen mex-3-RNAiPhänotypen der Kategorie I (Abb. 25: B) deutlich die fehlenden Mandibeln und Maxillen auf. Ansonsten scheint die Entwicklung normal verlaufen zu sein. Stärkere Kopfdefekte der Kategorie II beinhalten eine Verkürzung der Antennen; Augen, Mandibeln und Maxillen fehlen ebenfalls (Abb. 25: C). Neben den deutlich sichtbaren Kopfdefekten treten aber auch Embryonen auf, bei denen die anteriore Musterbildung vollständig funktioniert hat. Jedoch führt ein frühzeitiger Segmentierungsabbruch nach der Ausbildung von T3 zu einer drastischen Verkürzung der Embryonen (Abb. 25: D). Schließlich gibt es noch Phänotypen der Kategorie IV, die beide Merkmale sprich eine Störung der anterioren Musterbildung sowie einen posterioren Segmentierungsdefekt in Kombination aufweisen (Abb. 25: E). Insgesamt lässt sich der mex-3-RNAi-Phänotyp in Oncopeltus durch zwei Merkmale beschreiben; zum einen liegt eine Störung der anterioren Musterung und zum anderen eine Fehlentwicklung der posterioren Segmentierung vor. Beide Effekte sollen nun anhand von Markergenexpressionen näher analysiert werden. Tab. 6: Verteilung der O.f. mex-3 -RNAi Phänotypen (c=2 µg/µl) Phänotyp WT I II III IV n.e. mex-3-dsRNA 61(43,7%) 18(12,9%) 19(13,5%) 25(17,8%) 17(12,4%) 0 Kontrolle 139(100%) 0 0 0 0 0 69 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 25: Phänotypische Reihe nach mex-3 -RNAi in Oncopeltus A) WT Kutikula. B) Schwacher mex-3 -RNAi Phänotyp der Kategorie I. Mandibeln und Maxillen fehlen. C) In stärkeren Phänotypen der Kategorie II ist die Kopfmusterbildung stärker beeinträchtigt. Von den Antennen sind nur noch ein paar Borsten erkennbar. Augen, Labrum, Mandibeln und Maxillen fehlen. Ab dem Labium scheint die Entwicklung normal zu verlaufen. D) mex-3 RNAi Phänotyp bei dem die Kopfentwicklung wie im Wildtyp verlaufen ist. Ein vorzeitiger Segmentierungsabbruch führte lediglich zur Ausbildung der Segmente bis einschließlich T3. E) Eine Kombination aus fehlenden anterioren Strukturen wie Antennen, Augen, Mandibeln und Maxillen sowie Kompaktierung des Abdomens (Doppelpfeil). 70 II. Kapitel: Mex-3/Cad System - Ergebnisse Die Expression von engrailed nach mex-3-RNAi in Oncopeltus Im Oncopeltus Wildtyp-Blastoderm werden sechs engrailed-Streifen angelegt (Liu u. Kaufman, 2004). Diese wachsen durch die posteriore Invagination koordiniert in das Eiinnere hinein, so dass im invaginierten Keimrudiment sechs engrailed-Streifen präsent sind, welche die Segmente von den Mandibeln bis einschließlich des dritten thorakalen Segments repräsentieren (Abb. 26: A). Im Anschluss daran beginnt die für Kurzkeimer typische schrittweise ablaufende Segmentierung aus der posterioren Wachstumszone heraus. Diese führt schließlich zur Ausbildung von zehn abdominalen engrailed-Streifen im gestreckten Keimstreif (Abb. 26: E). Nach mex-3-RNAi werden wie im Wildtyp sechs engrailed-Streifen im Keimrudiment exprimiert, allerdings ist das Keimrudiment im Vergleich zum Wildtyp verkürzt und Fusionen einzelner engrailed-Streifen können beobachtet werden wie beispielsweise zwischen dem labialen und ersten thorakalen Segment (Abb. 26: B, Pfeil) oder dem ersten thorakalen und zweiten thorakalen Segment (Abb. 26: C, Pfeil). In stärkeren mex-3-RNAi-Phänotypen sind die engrailed-Streifen immer noch angelegt, jedoch nur rudimentär und die lateralen Seitenplatten scheinen ungleichmäßig in den Dotter eingewachsen zu sein, da die linke und rechte Hälfte des Embryos nicht exakt zusammenpasst, was in einer relativen Verschiebung der engrailed-Streifen zueinander resultiert (Abb. 26: D). In mex-3-RNAi-Embryonen die bereits das Stadium des gestreckten Keimstreifs erreicht haben, kann die Beobachtung gemacht werden, dass das dritte thorakale Segment fehlt, die weiter posterior liegenden Segmente zwar alle angelegt werden, allerdings ist das Abdomen viel schmaler und gekrümmter als in der Wildtypsituation (Abb. 26: F). Vergleicht man die Gestalt des Kopfes jüngerer und älterer Embryonen erkennt man, dass zwar im mex-3-RNAi-Embryo das Kopfgewebe noch vorhanden ist, aber reduzierter und ungeordneter wirkt als im Wildtyp (Abb. 26: D, F; schwarzer Balken). Dies ist nicht auf einen Präparationsartefakt zurückzuführen sondern ebenfalls eine Ausprägung des mex-3-RNAi-Phänotyps. Um den anterioren Effekt näher zu analysieren wurde eine even-skipped in-situ-Hybridisierung durchgeführt und frühe Stadien noch vor der Keimstreifinvagination näher analysiert. 71 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 26: Beeinträchtigung der posterioren Entwicklung nach O.f. mex-3 -RNAi Die en-Expression im Wildtyp wird mit der en-Expression in mex-3 -RNAi-Embryonen verglichen. A) Frei präpariertes Keimrudiment kurz nach der Invagination in den Dotter. Alle sechs im Blastoderm angelegten en-Streifen sind mit dem embryonalen Gewebe in das Innere des Eies verlagert worden und repräsentieren die Segmente von den Mandibeln bis hin zum dritten thorakalen Segment. B) Vergleichbares Stadium wie unter A, allerdings nach mex-3 -RNAi. Es ist zu erkennen, dass das Keimrudiment kürzer ist als das vergleichbare Wildtypstadium, das labiale und erste thorakale Segmente fusioniert zu sein scheinen (Pfeil) und das dritte thorakale Segment nur partiell angelegt worden ist. C) Keimrudiment nach mex-3 -RNAi mit einer Fusion von T1 und T2 (Pfeil). D) Starker mex-3 -RNAi Phänotyp. Zwar werden alle sechs engrailed-Streifen ausgebildet, allerdings nur rudimentär und die Fusion der lateralen Seitenplatten scheint asymmetrisch verlaufen zu sein. Das anteriore Kopfgewebe ist stark reduziert (schwarzer Balken). E) Wildtyp engrailedFärbung in einem vollständig elongierten Keimstreif. F) Vergleichbares Stadium wie E, nur nach mex-3 -RNAi. Es fällt auf, dass sowohl das Kopfgewebe unstrukturiert wirkt, ein Segmentierungsdefekt nach T2 vorhanden ist und die restlichen abdominalen Segmente angelegt werden. Jedoch ist die morphologische Gestalt des Keimstreifes stark gestört. 72 II. Kapitel: Mex-3/Cad System - Ergebnisse Die Expression von even-skipped nach mex-3-RNAi in Oncopeltus even-skipped erfüllt in Oncopeltus nicht die Funktion eines Paarregelgens wie das aus anderen Organismen bekannt ist, sondern wirkt als Segmentpolaritätsgen (Liu und Kaufman, 2005). Die initiale eve-Expression besteht aus einer breiten von posterior nach anterior verlaufenden Domäne, die sich bis ins vordere Drittel des Eies erstreckt. An der anterioren Grenze beginnt sich dann der erste anteriore Streifen abzuspalten (Liu und Kaufman, 2005) (Abb. 27: A, A’). Anschließend formen sich nacheinander die verbleibenden fünf Streifen, so dass am Ende des Blastodermstadiums sechs even-skipped-Streifen vorhanden sind (Abb. 27: B, B’). Diese repräsentieren die Segmente welche die Mandibeln, Maxillen und Labium sowie die ersten drei thorakalen Segmente umspannen. Nach mex-3-RNAi ist das regelmäßige Streifenmuster beeinträchtigt. Die ersten zwei eveStreifen scheinen enger aneinander zu grenzen, was auch damit zusammen hängen mag, dass die Streifen selbst breiter und verwaschener sind als in der Wildtyp-Expression (Abb. 27: C, C’). Es konnten auch mex-3-RNAi-Embryonen gefunden werden, bei denen eve vom mandibulären Segment bis hin zum dritten thorakalen Segment exprimiert wird, allerdings sind die Streifen in ihrer Ausprägung deutlich reduziert (Abb. 27: D, D’). Die stärkste Veränderung die gefunden werden konnte war eine Reduktion der Streifenanzahl von sechs auf vier eve-Streifen. Dass bereits Streifen in das Ei eingewandert sind kann ausgeschlossen werden, da keine posteriore Invagination sichtbar ist. Aufgrund der posterioren Lage des ersten eve-Streifens kann man vermuten, dass die zwei vorhergehenden eve-Streifen, welche die Mandibeln und Maxillen kennzeichnen, fehlen (Abb. 27: E, E’). Auch die dazugehörige Kernfärbung zeigt, dass im anterioren Bereich des Eies die Anlage des embryonalen Gewebes reduziert ist und aufgrund des großen Zellkernabstands in dieser Region vermutlich die Serosa expandiert ist (Abb. 27: E’). 73 II. Kapitel: Mex-3/Cad System - Ergebnisse Abb. 27: even-skipped -Expression im O.f. WT und nach mex-3 -RNAi 74 II. Kapitel: Mex-3/Cad System - Ergebnisse A-E zeigt die even-skipped-Expression nach mex-3 -RNAi, A’-E’ die entsprechenden Stadien mit Hoechstfärbung. A) Früheste Expression von even-skipped im WT Blastoderm. Die Pfeilköpfe markieren den Beginn der ersten bereits sichtbaren Streifenbildung. A’) Hoechstfärbung, Blastoderm. B) Ausbildung aller sechs even-skipped-Streifen, posterior ist der Beginn der Invagination zu sehen. B’) Blastoderm mit Kernfärbung. C) mex-3 -RNAi-Embryo, die Platzierung der Streifen wirkt ungeordneter als im Wildtyp und auch der Streifenabstand (Doppelpfeil) sowie die Streifenbreite ist verwaschener (Pfeilkopf). C’) Gleicher Embryo mit Kernfärbung. D) Stärkerer mex-3 -RNAi Phänotyp: Die Ausbildung der even-skipped-Streifen ist noch stärker beeinträchtigt, da einzelne Streifen fast nicht mehr voneinander zu unterscheiden sind. D’) Hoechstfärbung des Blastoderms mit beginnender Invagination. E) mex-3 -RNAi-Embryo mit nur vier angelegten eve-Streifen. Eine bereits beginnende Einwanderung der Streifen kann ausgeschlossen werden, da keine posteriore Invagination zu sehen ist. E’) Die Kernfärbung des gleichen Embryos zeigt eine expandierte Serosa, gekennzeichnet durch einen größeren Zellkernabstand an der anterioren Seite des Eies (beschriebener Bereich ist mit weißem Balken unterlegt). caudal-Expression nach mex-3-RNAi in Bruchidius u. Oncopeltus Da in Tribolium gezeigt werden konnte, dass sich die Caudal-Proteinexpressionsdomäne nach mex-3-RNAi nach anterior verschiebt und somit von einem regulatorischen Einfluss von Mex-3 auf caudal ausgegangen werden kann (Schoppmeier et al., 2009), sollte sofern das Mex-3/Cad System in Bruchidius und Oncopeltus konserviert ist, nach mex-3-RNAi ebenfalls ein Effekt auf die Caudal-Expression zu sehen sein. In Bruchidius konnte der direkte Zusammenhang zwischen den beobachteten Phänotypen und einer möglichen Expansion von Caudal nicht untersucht werden. Zum einen kreuzreagierte der Tribolium-Antikörper gegen Caudal in Bruchidius nicht und zum anderen stellte sich das Problem, dass die Phänotypenfrequenz nach mex-3-RNAi sehr gering war (s.h. auch Diskussion). Auch in Oncopeltus kreuzreagierte der Tribolium-Caudal-Antikörper nicht, was auch damit zusammenhängen könnte, dass die Oncopeltus-Embryonen Hitze fixiert werden. Zahlreiche Versuche die Fixierung der Embryonen ohne Hitzeschritt durchzuführen, brachten nicht den gewünschten Erfolg, da die Morphologie durch das Devitellinisieren der Eier mit der Hand immer sehr stark beeinträchtigt wurde. Deswegen wurde alternativ eine caudal in-situHybridisierung in Oncopeltus mex-3-RNAi-Embryonen durchgeführt. Jedoch konnte weder in frühen noch in späteren embryonalen Stadien eine Veränderung der caudal-Expression im Vergleich zum Wildtyp beobachtet werden (nicht gezeigt). 75 II. Kapitel: Mex-3/Cad System - Diskussion Diskussion Die Regulation von Caudal auf translationaler Ebene durch bicoid in Drosophila ist sehr gut untersucht und spielt in der frühen Embryonalentwicklung eine entscheidende Rolle (RiveraPomar et al., 1996a; Ephrussi und St Johnston; 2004; Cho et al., 2005). bicoid gibt es allerdings nur in der Gruppe der cyclorrhaphen Fliegen (Stauber et al., 2002; Lemke et al., 2008), während caudal-Homologe in allen bisher untersuchten Bilateria gefunden wurden und eine Funktion in der posterioren Musterbildung inne haben (Lall und Patel, 2001; Copf et al., 2004). Aus dem Stamm der Nematoden ist von C.elegans bekannt, dass das KH-DomänenProtein Mex-3 die Translation von caudal-mRNA inhibiert (Draper et al., 1996). Vor einigen Jahren konnte auch innerhalb der Insekten, im Mehlkäfer Tribolium castaneum, gezeigt werden, dass mex-3 in der frühen anterioren Musterbildung eine Rolle spielt, indem es die Translation von caudal-mRNA im Bereich der zukünftigen Kopfanlagen reprimiert (Schoppmeier et al., 2009). Mit der Untersuchung der Gene mex-3 und caudal im holometabolen Käfer Bruchidius obtectus und der hemimetabolen Wanze Oncopeltus fasciatus soll die Frage beantwortet werden, inwieweit das im Käfer Tribolium verwendete Mex-3/Cad-System auch in anderen Insektenspezies konserviert ist. Wichtige Rolle für caudal in der Bruchidius- u. Oncopeltus-Embryogenese Sowohl im Langkeimer Bruchidius als auch im Kurzkeimer Oncopeltus wird caudal in einer breiten Domäne im Blastoderm exprimiert. Während in Bruchidius die caudal-Expression von posterior nach anterior hin schwächer wird (Abb. 15: A), ist im Oncopeltus-Blastoderm die Expression homogen und nur die anteriore Eispitze, wo sich die Serosaanlage entwickelt (Butt, 1947), bleibt frei von caudal-Expression (Abb. 21: A). Nach cad-RNAi in Bruchidius verläuft die frühe Embryogenese noch bis zum Stadium des frühen Blastoderms, dann aber scheint die Ausbildung des Keimrudiments beeinträchtigt zu sein, da die Zellen anstatt auf die ventrale Seite zu wandern, eher eine Ansammlung auf der posterioren Seite zeigen, was nach einiger Zeit zum Abbruch der Embryonalentwicklung führte (Abb. 16). Insgesamt ist die Funktion von caudal in Bruchidius deutlich stärker als die beschriebene Funktion von caudal in Drosophila, da sich in Drosophila auch in Abwesenheit von caudal 76 II. Kapitel: Mex-3/Cad System - Diskussion Kopf, Thorax und ansatzweise abdominale Strukturen ausbilden können (Macdonale und Struhl, 1986). Hingegen sind in Episyrphus, Nasonia, Tribolium und Gryllus ähnlich starke Phänotypen nach cad-RNAi beobachtet worden, wie sie in Bruchidius auftreten: die meisten Embryonen entwickeln keine Kutikula und nur vereinzelte Embryonen zeigen Reste der anterioren Kopfstrukturen (Lemke und Schmidt-Ott, 2009; Olesnicky et al., 2006; Copf et al., 2004; Shinmyo et al., 2005). Eine mögliche Erklärung für die schwächere Funktion von caudal bei der anterioren-posterioren Musterbildung in Drosophila könnte darin liegen, dass bicoid im anterioren Bereich des Embryos die Aktivierung von Gapgenen übernommen hat, die in Nasonia und Gryllus noch von Caudal reguliert werden. Beispielsweise wird das Gapgen Krüppel in Drosophila von Bicoid aktiviert, während die Krüppel-Expression sowohl in Nasonia (Olesnicky et al., 2006), als auch in Gryllus (Shinmyo et al., 2005) von Caudal aktiviert wird. Somit besitzt vermutlich auch Bruchidius caudal einen stärken Einfluss auf die Regulation der Gapgene als das in Drosophila der Fall ist. Nach cad-RNAi in Oncopeltus entwickeln selbst die stärksten Phänotypen auf Kutikulaebene noch Antennen, Augen und anteriores Kopfgewebe (Abb. 22: C). caudal wurde bereits in einem anderen hemimetabolen Kurzkeimer untersucht, nämlich Gryllus bimaculatus (Ordnung: Ensifera). Trotz der nahen phylogenetischen Verwandtschaft besteht ein scheinbarer Unterschied zwischen diesen beiden Organismen hinsichtlich der caudal-Funktion, da für Gryllus beschrieben worden ist, dass der Großteil der Embryonen nach cad-RNAi keine Kutikula mehr ausbildet (Shinmyo et al., 2005). Dies war in Oncopeltus nicht der Fall, da nur bei ca. 2% der Embryonen keine Kutikulapräparate angefertigt werden konnten (Tabelle 5). Eine Möglichkeit ist, dass die Sekretion von Kutikula in Oncopeltus weniger störanfällig ist als in Gryllus, so dass die verbleibenden anterioren Reste in Oncopeltus meistens präpariert werden können. Die Analyse des Gryllus cad-RNAi-Effekts mittels wg-ISH unterstützt diese Theorie, da dort 45% der untersuchten Embryonen nur noch die anteriorsten Kopfstrukturen aufwiesen (Shinmyo et al., 2005), was nahezu deckungsgleich mit den von mir erzielten Resultaten auf Kutikulaebene in Oncopeltus ist (Tabelle 5). Eine weitere Gemeinsamkeit hinsichtlich der frühen Caudal-Funktion zwischen Bruchidius und Oncopeltus besteht auch bei der Ausbildung des Keimrudimens. Wie bereits erwähnt bildet sich bei Bruchidius nach cad-RNAi das Keimrudiment nicht mehr an der ventralen Seite aus, sondern eher an der posterioren Eispitze (Abb. 16: C’, D’). Ein ähnliches Phä77 II. Kapitel: Mex-3/Cad System - Diskussion nomen kann bei Oncopeltus beobachtet werden. Anstatt dass die lateralen Seitenplatten an der posterioren Eispitze fusionieren und dann in den Dotter eintauchen, findet die Fusion an einer beliebigen Stelle im Ei statt und das Keimrudiment taucht an einer falschen Stelle in den Dotter ein (Abb. 23). Somit scheint bei beiden Organsimen, Caudal die Richtung für die korrekte Zellwanderung zur Ausbildung des embryonalen Gewebes vorzugeben. Neben der Funktion von Caudal im Blastoderm tritt während der Keimstreifstreckung in beiden Organismen eine posteriore Expressionsdomäne auf (Abb. 15: B, C; Abb. 21: B, C), die jedoch aufgrund der unterschiedlichen Entwicklungsmodi keine analoge Funktion besitzt. Die im Keimstreif von Bruchidius später auftretende Domäne der caudal-Expression ist vermutlich für die Ausbildung der Hinterdarmanlage zuständig, wie das bereits für Drosophila herausgefunden worden ist (Wu und Lengyel, 1998). Caudal aktiviert dort zum einen die Zielgene forkhead sowie wingless und ist zugleich für die Invagination der Darmanlage zuständig, was die Expression von folded gastrulation widerspiegelt (Wu und Lengyel, 1998). Die posteriore Expressionsdomäne (Abb. 21: B, C) während der Keimstreifstreckung in Oncopeltus deutet darauf hin, dass caudal in der dort befindlichen Kurzkeim-spezifischen Wachstumszone benötigt wird, was sich auch auf funktionaler Ebene zeigt. caudal-RNAi in Oncopeltus resultiert nämlich auch in cad-RNAi-Embryonen, welche die BlastodermPhase ohne größere Entwicklungsstörung durchlaufen und erst im Bereich des Abdomens Segmentierungsdefekte aufweisen (Abb. 22: B). Eine mögliche Erklärung für dieses Phänomen wäre, dass eine geringe Menge an caudal-mRNA noch vorhanden und ausreichend für die blastodermale Musterbildung ist, aber nicht mehr für die Funktion von caudal in der posterioren Wachstumszone aus der die abdominalen Segmente generiert werden. Die posteriore Funktion von caudal in der Wachstumszone wurde beispielsweise auch für den Kurzkeimer Tribolium beschrieben (Copf et al., 2004) und selbst in Vertebraten spielen die caudal-Homologe cdx1, cdx2 und cdx4 in der posterioren Musterbildung eine Schlüsselrolle (Lohnes, 2003). Dass Oncopeltus caudal ebenfalls für die Spezifizierung der Hinterdarmanlage benötigt wird ist wahrscheinlich, jedoch konnte diesbezüglich keine gesonderte Expression während der Keimstreifstreckung beobachtet werden. Zusammenfassend kann man sagen, dass die frühe, blastodermale Funktion von caudal unabhängig vom Entwicklungsmodus konserviert ist. Alle Segmente die im Blastoderm definiert werden (in Bruchidius wären das Kopf, thora78 II. Kapitel: Mex-3/Cad System - Diskussion kale und abdominale Segmente; in Oncopeltus nur Kopf und thorakale Segmente) benötigen während ihrer Entwicklung im Blastoderm ein gewisses Level an cad. Anders verhält es sich mit der späteren Funktion von caudal während der Keimstreifstreckung. Im Langkeimer Bruchidius ist diese posteriore caudal-Domäne nicht für die weitere Bildung abdominaler Segmente zuständig und daher vermutlich an der der Spezifizierung des hinteren Darmsystems beteiligt, während in Oncopeltus, caudal eine zusätzliche Kurzkeimspezifische Funktion in der Wachstumszone inne hat, so dass weitere abdominale Segmente gebildet werden können. Die Funktion von mex-3 im Rahmen der Oncopeltus-Embryogenese Oncopeltus mex-3 wird früh im Blastoderm in einer breiten Domäne exprimiert (Abb. 24: A). Später kann mex-3 im Bereich der zukünftigen Kopflappen und entlang der lateralen Seitenplatten nachgewiesen werden (Abb. 24: C). Die Expression in den Kopflappen besteht während der Keimstreifstreckung fort, entlang der ventralen Mittellinie wird mex-3 in einem neuronalen Muster exprimiert und am posterioren Ende des Keimstreifs ist eine breite mex3-Expressionsdomäne zu beobachten (Abb. 24: E). Der Knockdown von mex-3 durch RNAi in Oncopeltus führt einerseits zu Deletionen im anterioren Bereich, die in stärkeren Phänotypen fast im vollständigen Verlust des Kopfes auf Kutikulaebene resultieren (Abb. 25: B, C) und andererseits zur Beeinträchtigung der abdominalen Musterbildung, was zu einer drastischen Verkürzung der Nymphen führt (Abb. 25: D). Die Expressionsdaten und funktionalen Daten von Oncopeltus mex-3 zeigen, dass O.f. mex-3 sowohl in der anterioren als auch in der posterioren Segmentierung eine wichtige Rolle spielt. Oncopeltus mex-3 in der anterioren Segmentierung: Ursächlich für die anterioren Deletionen ist eine frühe Störung der Segmentierung: Die mandibulären bis thorakalen engrailed-Streifen, welche bereits im Oncopeltus-Blastoderm spezifiziert werden (Liu und Kaufman, 2004), sind nach mex-3-RNAi entweder schwächer exprimiert oder partiell reduziert (Abb. 26). Auch die even-skipped-Expression im Blastoderm von Oncopeltus mex-3-RNAiEmbryonen ist beeinträchtigt, da teilweise keine distinkten even-skipped-Streifen mehr gebildet werden und in stärkeren Phänotypen fehlt die even-skipped-Expression in den mandibulären und maxillären Bereichen des Blastoderms vollständig (Abb. 27: C, D, E). Zu- 79 II. Kapitel: Mex-3/Cad System - Diskussion dem ist das anteriore Kopfgewebe reduziert, was sich im blastodermalen Stadium durch eine deutliche Expansion der Serosa darstellt (Abb. 27: E’) und in den Keimstreifpräparaten als verkleinerte Kopfstrukturen (Abb. 26). Die beobachten Kopfdefekte in Oncopeltus gleichen den beschriebenen mex-3-RNAiPhänotypen in Tribolium. In Tribolium konnte ebenfalls gezeigt werden, dass die Anzahl der eve-Streifen in mex-3-RNAi-Embryonen im Blastoderm reduziert ist und dass ein Grund dafür die anteriore Expansion der Caudal-Expression ist (Schoppmeier et al., 2009). Die Analyse der caudal-mRNA-Expression nach mex-3-RNAi in Oncopeltus ergab, dass caudal sowohl im Blastoderm als auch während der Keimstreifstreckung wie im Wildtyp exprimiert wird. Da jedoch Tribolium Mex-3 auf der Ebene der Translation wirkt, indem es an caudal-mRNA bindet und die Translation verhindert (Schoppmeier et al., 2009), könnte es durchaus sein, dass eine Expansion von Caudal nur auf Proteinebene nachgewiesen werden kann, was aber aufgrund des Fehlens eines funktionierenden Antikörpers im Rahmen dieser Arbeit nicht möglich war. Aus Xenopus ist bekannt, dass ein Knockdown von Mex-3B unter anderem zur Reduktion der Expression von otx2 führt (Takada et al., 2009), das ein Säugerhomolog von otd ist (Simeone et al, 1993). Diese reduzierte Expression von otx2 in Xenopus ist vermutlich auf eine Derepression posteriorer Signale zurückzuführen und wirkt sich auf phänotypischer Ebene dahingehend aus, dass Xenopus-Embryonen mit verkleinerten Augen entstehen (Takada et al., 2009). In zahlreichen Insekten wie Nasonia, Drosophila oder Episyrphus spielt otd1 eine wichtige Rolle in der anterioren Segmentierung und führt bei einem Verlust zu fehlenden Kopfsegmenten (Lynch et al., 2006; Finkelstein und Perrimon, 1990; Lemke und SchmidtOtt, 2009). Kürzlich wurde die Expression und Funktion von Genen welche bei der anterioren Segmentierung in Drosophila eine zentrale Rolle spielen auch in Oncopeltus untersucht (Birkan et al., 2011). Beispielsweise resultieren collier, crocodile oder cap ’n’ collar nach RNAi in relativ schwachen Kopfphänotypen. Einzig Oncopeltus otd führte zu einem starken Phänotyp, der in dem Verlust von Antennen, Augen und gnathalen Strukturen besteht (Birkan et al., 2011). Also ein vergleichbarer Phänotyp, der auch nach O.f mex-3-RNAi auftritt, so dass wie in Xenopus, eine vermutlich indirekten Regulation von O.f. otd durch O.f. mex-3 bestehen könnte. Durch den Knockdown von O.f. mex-3 könnte es zu einer Expansion der posterioren Signale - möglicherweise Caudal - kommen, so dass otd nicht mehr exprimiert wird und die Aktivierung wichtiger Gene der anteriore Musterbildung unterbleibt. 80 II. Kapitel: Mex-3/Cad System - Diskussion Oncopeltus mex-3 in der posterioren Segmentierung: Die Analyse des Segmentpolaritätsgens engrailed nach mex-3-RNAi ergab, dass bei einem Teil der mex-3-RNAi-Embryonen die sechs Segmente, also mandibuläres bis einschließlich drittes thorakales Segment im Blastoderm angelegt worden sind, auch die Invagination in den Dotter findet statt, allerdings ist in den daraus entstehenden Keimstreifen im Bereich der thorakalen Segmentanlagen und in der Wachstumszone häufig eine Gewebsdeformation zu sehen (Abb. 26: B, D). Letzteres scheint die Generierung der Segmente aus der Wachstumszone heraus zu beeinträchtigen, da es entweder zu einem frühen Segmentierungsabbruch kommt oder aber sich abdominale Segmente bilden, jedoch deutlich schmaler als im Wildtyp sind und eher an einen fadenförmigen Fortsatz erinnern (Abb. 26: F). Dies führt dann schließlich zu drastischen Verkürzungen auf Kutikulaebene. Auch hier könnte ein mögliches Mex-3/Caudal System zum Tragen kommen. In Tribolium konnte gezeigt werden (Schoppmeier, persönliche Mitteilung), dass nach mex-3-RNAi die im Bereich der posterioren Wachstumszone befindliche Caudal-Expressionsdomäne expandiert und folglich die koordinierte posteriore Segmentierung gestört ist. Zudem besteht die Möglichkeit dass O.f. mex-3 bei der Bildung abdominaler Segmente unabhängig von Caudal agiert. Einen Hinweis dafür wie mex-3 posterior wirken könnte, liefert erneut die Untersuchung zur Funktion von Mex-3B im Frosch Xenopus (Takada et al., 2009). Dort konnte gezeigt werden, dass die Überexpression von mex-3 in verkürzten Embryonen resultiert, was mit einer Interaktion von Mex-3B und dem FGF-Signalweg zusammenhängt. Mex-3B bindet an die mRNA einzelner Komponenten des FGF-Signalwegs, wie sdc2 sowie ets1b und führt zu deren Destabilisierung, weswegen die Zelle weniger sensitiv auf das ihr zugedachte Signal reagiert (Takada et al., 2009). Im Kurzkeimer Tribolium wird in der posterioren Wachstumszone ebenfalls Fgf8 exprimiert (Beermann und Schröder, 2008), so dass hier eine mögliche Interaktion von Mex-3 mit dem Fgf8-Signalweg konserviert sein könnte. Über Fgf-Signalwege in Oncopeltus ist bis jetzt noch nichts bekannt, aber möglicherweise handelt es sich bei den posterioren Verkürzungen in Oncopeltus ebenfalls um eine Fehlregulation des FGF-Signalwegs nach mex3-RNAi. Insgesamt deuten die Oncopeltus mex-3-Daten sowohl bezüglich der anterioren, als auch der posterioren Musterbildung darauf hin, dass die Funktion von mex-3 zwischen den beiden Kurzkeimern Oncopeltus und Tribolium konserviert sein könnte, wobei eine direkte Interaktion von Oncopeltus Mex-3 mit caudal spekulativ bleibt. 81 II. Kapitel: Mex-3/Cad System - Diskussion B.o. mex-3 wird für die Anlage thorakaler u. abdominaler Segmente benötigt B.o. mex-3 wird während der gesamten Embryogenese exprimiert (Abb. 18). Die früheste Expression von mex-3 in einer breiten anterioren Domäne ist wahrscheinlich maternalen Ursprungs, während dem sich differenzierenden Blastoderm ist mex-3 in den zukünftigen Kopflappen detektierbar, an die sich posterior eine segmentale Expression anschließt (Abb. 18: A, B, C). Im Zuge der Keimstreifstreckung und dem abschließend erfolgenden Rückenschluss ist mex-3 in einem neuronalen Muster in den Kopflappen und entlang der ventralen Mittellinie zu finden (Abb. 18: F). Nach parentaler mex-3-RNAi weisen die Embryonen Segmentierungsstörungen in Form von Deletionen im Bereich der thorakalen und abdominalen Segmente auf. Daraus resultieren stark verkürzten Larven, bei denen nur noch der Kopf wie im Wildtyp entwickelt ist (Abb. 19). Als problematisch bei der Analyse der Funktion von Bruchidius mex-3 stellte sich die geringe Penetranz der Phänotypen nach mex-3-RNAi heraus (Tabelle 4). Dafür gibt es mehrere mögliche Erklärungen. Für die Ordnung Lepidoptera ist bekannt, dass die RNAi-Effizienz, ohne offensichtliche Gründe, zwischen den verschiedenen Spezies stark variieren kann (Terenius et al., 2011). Die Versuche an Bruchidius ergaben, dass RNAi im Bohnenkäfer zwar funktioniert, jedoch die Effizienz schlechter ist als in Tribolium. Eine weitere Möglichkeit für die Schwierigkeit mex-3-RNAi-Phänotypen zu generieren könnte sein, dass mex-3 in Bruchidius ebenfalls einen effizienten, autoregulatorischen Feedback Loop bildet, wie das für Xenopus mex-3b gezeigt worden ist: In der 3’ UTR Region von Xenopus mex-3b kann Mex3b Protein binden und somit die Translation seiner eigenen mRNA inhibieren (Takada et al., 2009). Die Folge ist, dass die Injektion von mex-3-Morpholinos in Xenopus-Embryonen in einem erhöhten Level an endogener mex-3-mRNA resultiert. Die Generierung von Phänotypen durch sehr geringe Konzentrationen an dsRNA (300600 ng/µl) schließen einen Off-Target Effekt vermutlich aus (Terenius et al., 2011), weswegen die gefunden Phänotypen durchaus die Funktion von mex-3-RNAi in Bruchidius repräsentieren könnten. Die B.o. mex-3-RNAi-Phänotypen sind auf den ersten Blick überraschend, da sich lediglich die Kopfsegmente wie im Wildtyp entwickeln, wohingen in T.c. mex-3-RNAi Phänotypen die Kopfsegmente die stärksten Entwicklungsdefekte aufweisen (Schoppmeier et al., 2009). Diese scheinbare widersprüchliche Funktion von mex-3 in den beiden Käferspezies lässt sich jedoch mit den Anlageplänen der beiden Käfer erklären: Im 82 II. Kapitel: Mex-3/Cad System - Diskussion Langkeimer Bruchidius ist der Großteil der Segmentanlagen proportional im Blastoderm repräsentiert, welcher den gesamten Raum entlang der a-p Achse einnimmt (Abb. 17: A). Im Kurzkeimer Tribolium hingegen, wird im anterioren Bereich des Eies extraembryonales Gewebe gebildet, die Kopfanlagen befinden sich in der Mitte des Eies und posterior davon die ersten thorakalen Segmente, sowie die Wachstumszone (Abb. 17: B). In Bruchidius wird mex-3 sowohl im Kernscharstadium als auch mit der Ausbildung des differenzierten Blastoderms entlang der a-p Achse exprimiert (Abb. 17: C), im differenzierten Blastoderm von Tribolium lediglich im zentralen Bereich des Eies, welcher mit den Kopfanlagen zusammen fällt (Abb. 17: D). Gesetzt den Fall, dass die Funktion der zentralen mex-3-Expression in Tribolium und Bruchidius konserviert ist, betreffen die mex-3-RNAi Phänotypen unterschiedliche embryonale Anlagen: die Kopfstrukturen in Tribolium, in Bruchidius eher thorakale und abdominale Segmente (Abb. 17: E, F). Somit sind ist die Funktion von mex-3 in beiden Käferspezies vermutlich konserviert, auch wenn es auf den ersten Blick auf phänotypischer Ebene nicht so erscheint. Ursächlich für die Segmentdeletionen in Tribolium ist u.a. die anteriore Expansion der CaudalExpressionsdomäne im Blastoderm (Schoppmeier et al., 2009). Aufgrund der niedrigeren Penetranz der Bruchidius mex-3-RNAi Phänotypen konnte ein ähnlicher Effekt auf die Bruchidius Caudal-Regulation nicht gezeigt werden. Jedoch liegt die Vermutung nahe, dass Bruchidius Mex-3 ebenfalls Caudal regulieren kann, da nach mex-3-RNAi Segmentdeletionen im zentralen Bereich des Eies beobachtet werden wie in Tribolium. Somit könnte in beiden Käfer-Spezies, das Mex-3/Cad System konserviert sein, obwohl auf phänotypischer Ebene ein unterschiedlicher Effekt zu sehen ist, der jedoch auf die verschiedenen Anlagepläne zurückgeführt werden kann. Neben der Regulation von Caudal könnte mex-3 sowohl in Tribolium als auch Bruchidius noch zusätzliche ebenfalls konservierte Funktionen im Blastoderm haben und für die direkte oder indirekte Regulation weiterer früher Musterbildungsgene verantwortlich sein, wie das auch für Oncopeltus mex-3 diskutiert wurde, was aber mangels weiterer Daten noch offen ist. 83 II. Kapitel: Mex-3/Cad System - Diskussion Abb. 28: Vergleich der Funktion von mex-3 in Bruchidius und Tribolium A, C, E: mex-3 in Bruchidius; B, D, F: mex-3 in Tribolium (nach Schoppmeier et al., 2009) A) Der frühe Anlageplan von Bruchidius im Blastoderm; ein Großteil der Segmente ist bereits im Blastoderm spezifiziert. C) mex-3 wird über den gesamten Embryo hinweg in einem segmentalen Muster exprimiert. Die zentrale Region, welche später auch einen RNAi-Effekt wiederspiegelt, ist unterstrichen. E) Die gestrichelte Linie schließlich markiert die deletierte Region auf Kutikulaebene im stärksten Bruchidius mex-3 -RNAi-Phänotyp. B) Als Kurzkeimer sind im Tribolium-Blastoderm nur die Kopf und ein Teil der Thoraxsegmente angelegt, die fehlenden Segmente bilden sich nach der Gastrulation aus der Wachstumszone (WZ). D) Im Vergleich zu Bruchidius zeigt mex-3 in Tribolium eine breite Domäne, welche sich über den Bereich der zukünftigen Kopfanlagen spannt. F) Die gestrichelte Linie markiert die fehlenden Kopfstrukturen nach mex-3 -RNAi in Tribolium. 84 II. Kapitel: Mex-3/Cad System - Diskussion Ist das Mex-3/Caudal System innerhalb der Insekten konserviert? Die momentane Datenlage deutet darauf hin, dass die Regulation von Caudal durch Mex3 im Blastoderm zwischen Tribolium, Bruchidius und Oncopeltus konserviert sein könnte. Der Vergleich der Phänotypen nach mex-3-RNAi von Tribolium und Oncopeltus zeigt, dass in Kurzkeiminsekten die anterioren Strukturen in ihrer Entwicklung beeinträchtigt sind, während im Langkeimkäfer Bruchidius eher thorakale und abdominale Strukturen betroffen sind. Ist die Repression von Caudal durch Mex-3 konserviert, besteht auf phänotypischer Ebene ein Unterschied, der mit dem unterschiedlichen segmentalen Anageplan im Blastoderm von Kurz- und Langkeimern zu erklären sein könnte. Insgesamt scheint mex-3 für die Segmentierung eine wichtige Rolle zu spielen, welche aber im Laufe der Evolution in Drosophila verloren gegangen ist, da in Drosophila keine frühe Funktion von mex-3 vorhanden ist. Lediglich in späteren Entwicklungsstadien ist eine neuronale Expression von Drosophila mex-3 zu sehen (Schoppmeier et al., 2009), wie das auch in Tribolium (Schoppmeier et al., 2009), Bruchidius und Oncopeltus gezeigt werden konnte, so dass eine späte Funktion von mex-3 in der Neurogenese konserviert zu sein scheint. Ausblick Die Analyse von mex-3 in Bruchidius und Oncopeltus zeigte, dass mex-3 ein wichtiges Gen der anteriore Musterbildung ist. Da in Drosophila mex-3 keine Rolle in der Segmentierung spielt (Schoppmeier et al., 2009), ist es für die Fragestellung von Interesse, welche Mechanismen außerhalb des Drosophila-Paradigmas in der Embryonalentwicklung konserviert sind. Bei der weiteren Bearbeitung des Projekts wäre zunächst wichtig, Spezies-spezifische Caudal-Antikörper zu generieren um einen möglichen Effekt auf die Caudal-Translation nachweisen zu können und zu beweisen dass die beobachteten Phänotypen auf eine Derepression von Caudal zurückzuführen sind. Ein weiterer interessanter Gesichtspunkt ist, dass abhängig vom Anlageplan bzw. vom Modus der Segmentierung unterschiedliche, embryonale Strukturen betroffen sind. Während im Langkeimer Bruchidius Deletionen im thorakalen und abdominalen Bereich zu beobachtet werden können, fehlen in Tribolium und Oncopeltus vor allem die Kopfsegmente und die aus der posterioren Wachstumszone entstehenen abdominalen Segmente nach mex-3-RNAi. Die Analyse von mex-3 in anderen Langkeim- sowie Kurzkeiminsekten würde zeigen, in wie weit sich diese Theorie verallgemeinern lässt. 85 III. Kapitel: mlpt und svb in Tribolium Einleitung Das letzte Kapitel ist einem mittlerweile sehr gut etablierten Modellorganismus gewidmet, nämlich dem Reismehlkäfer Tribolium castaneum (Klingler, 2004). Nach Drosophila melanogaster ist wohl seine Embryogenese innerhalb der Insekten, die am besten untersuchte. Trotzdem gibt es zahlreiche offene Fragen, etwa auf dem Gebiet der Segmentierung (Rosenberg et al., 2008) die daher rühren, dass es sich bei Tribolium um einen Kurzkeimer handelt (Tautz et al., 1994; Davis und Patel, 2002). Während in Drosophila alle Segmentanlagen bereits im Blastoderm vorhanden und in einer syncytialen Umgebung entstehen können, bilden sich in Tribolium die meisten posterioren Segmente nach der Gastrulation, in zellulärer Umgebung, aus einer so genannten Wachstumszone heraus (Liu und Kaufmann, 2005). Die meisten Gene der hierarchischen Segmentierungskaskade von Drosophila (Akam, 1987; Ingham, 1988; Rivera-Pomar und Jäckle, 1996), wurden auch in Tribolium untersucht. Dabei stellte sich heraus, dass für die in Drosophila maßgeblich an der initialen Regionalisierung des Embryos beteiligten Gapgene (Niessing et al., 1997) zwar Homologe in Tribolium gefunden werden konnten, die Funktion der Gapgene in Tribolium aber keinesfalls denen von Drosophila entspricht (s.u.). Rolle der Gapgene in der frühen Embryonalentwicklung von Drosophila Die Gapgene wurden initial in dem berühmten Drosophila-Screen von Nüsslein-Volhardt und ihren Mitarbeitern entdeckt (Nüsslein-Volhard und Wieschaus, 1980). Sie bekamen ihren Namen daher, dass Drosophila-Larven, die eine Mutation in einem Gapgen aufweisen, in ihrer Segmentierung stark beeinträchtigt sind, da mehrere aneinander unmittelbar angrenzende Segmente nicht ausgebildet werden. Gapgene sind die ersten zygotisch exprimierten Gene während der Embryonalentwicklung und codieren Transkriptionsfaktoren (Jäger, 2011). 86 III. Kapitel: mlpt und svb in Tribolium - Einleitung Für die Kopfmusterung sind vor allem hunchback (Lehmann und Nüsslein-Volhard, 1987), buttonhead (Wimmer et al., 1995), empty spiracles (Walldorf und Gehring, 1992) sowie orthodenticle (Finkelstein und Perrimon, 1990) zuständig, während die thorakale und abdominale Musterbildung stark von den Gapgenen hunchback (Lehmann und Nüsslein-Volhard, 1987; Wu et al., 2001), Krüppel (Gaul und Jäckle, 1987; Jäckle et al., 1988), knirps (Nauber et al., 1988) und giant (Petschek et al., 1986; Mohler et al., 1989) abhängt. Terminale Strukturen unterliegen der Musterung von tailless und huckebein (Weigel et al., 1990). Die Expression der Gapgene wird auf verschiedenen Ebenen beeinflusst: Zum einen werden sie durch die maternalen Gene reguliert (Struhl et al., 1989), zum anderen ist eine wichtige Eigenschaft der Gapgene, dass sie ihre Expression gegenseitig regulieren können (Surkova et al., 2009; Papatsenko und Levine, 2011). Die über das ganze Blastoderm hinweg überlappend exprimierten Gapgene (Abb. 29: A) können dann wiederum direkt Paarregelgene (Carroll and Vavra, 1989), Segmentpolaritätsgene (Gutjahr et al., 1993) und Hoxgene (Harding and Levine, 1988) regulieren und spielen somit eine große Rolle für Metamerisierung und Segmentidentität. Gapgen-Homologe in der frühen Embryonalentwicklung von Tribolium Lässt sich die gerade vorgestellte Segmentierungskaskade von Drosophila auf den Kurzkeimkäfer Tribolium übertragen? Es konnte gezeigt werden, dass auch in Tribolium maternale Gene an der Aktivierung der ersten zygotischen Gene involviert sind (Wolff et al., 1998; Schoppmeier und Schröder 2005). Überraschenderweise hat sich aber durch die Untersuchung der Gapgen-Homologe in Tribolium herausgestellt, dass sie bei der Segmentierung keinen so großen Einfluss auf die Entstehung der Segmente, sondern vielmehr eine verstärke Rolle in der Festlegung der Segmentidentität spielen. Die Aktivierung von Paarregelgenen durch die Gapgene - sowohl im anterioren Kopfbereich, als auch im Rumpf - spielt in Tribolium möglicherweise eine untergeordnete Rolle (Bucher und Klingler, 2004; Cerny et al., 2008; Marques-Souza et al., 2008). Vor allem bei der Segmentierung des Abdomens ist die Meinung verbreitet, dass sich die Paarregelgene in einem ”pair-rule circuit” selbst aktivieren und die Gapgene bezüglich der Paarregelexpression eher eine permissive Rolle übernehmen, denn eine instruktive (Choe et al., 2005). Andererseits konnte gezeigt werden, dass die Gapgene einen sehr großen Einfluss auf die Expression der homeotischen Gene nehmen und somit für die Segmentidentität eine wichtige 87 III. Kapitel: mlpt und svb in Tribolium - Einleitung Rolle spielen (Bucher und Klingler, 2004; Cerny et al., 2008; Marques-Souza et al., 2008). Dass die Expression und die Funktion von Gapgen Homologen zwischen den beiden Arten oft nicht konserviert ist, sollen folgende Beispiele kurz beleuchten. Empty spiracles, buttonhead und orthodenticle, die in Drosophila als wichtige Gapgene für die Kopfmusterung identifiziert werden konnten (Walldorf und Gehring, 1992; Wimmer et al., 1995; Finkelstein und Perrimon, 1990), werden in Tribolium zwar ebenfalls anterior aktiviert, allerdings in viel schmaleren Domänen und auch nicht überlappend wie in Drosophila. Auch die funktionalen Analysen zeigen, dass T.c. empty spiracles lediglich für die Spezifizierung des posterioren Ocellensegments sowie des antennalen Segments zuständig ist (Schinko et al., 2008). Für Tribolium buttonhead konnte keine Funktion gefunden werden, hingegen scheint die späte Funktion von otd-1, bezüglich der Ausbildung der okkularen und antennalen Segmente in Tribolium und Drosophila konserviert zu sein. (Schinko et al., 2008). Die anterioren Expressionsdomänen von T.c. hunchback, T.c. Krüppel, und T.c. giant sind hinsichtlich ihrer Funktion nicht konserviert. Der Verlust dieser Gapgene führt zu Transformationen anteriorer Segmente. Die initialen Segmentanlagen werden aber normal gebildet, was sich in der weitgehend korrekten Ausbildung der Paarregel- bzw. Segmentpolaritätsstreifen widerspiegelt. Insgesamt führt der Knockdown dieser Gene in Tribolium meist nicht zu Lücken im Segmentmuster wie in Drosophila (Bucher und Klingler, 2004; Cerny et al., 2008; Marques-Souza et al., 2008), vielmehr kommt es zum Abbruch der Segmentierung im Thorax oder Abdomen. Einzig für T.c. knirps konnte eine Deletion benachbarter Segmente - Antennen und Mandibeln - gefunden werden (Cerny et al., 2008), ein Phänotyp der sogar stärker ist als in Drosophila (Gonzalez-Gaitan et al., 1994). Das für die Aktivierung des terminalen Systems wichtige Gapgen tailless in Drosophila, wird in Tribolium zwar in einem kurzen Zeitfenster posterior exprimiert, hat aber offenbar keinen Einfluss auf die Expression weiterer Segmentierungsgene. Der Unterschied in der Gapgen-Expression und -Funktion zwischen Drosophila und Tribolium setzt sich auch in der thorakalen und abdominalen Musterbildung fort. Vergleicht man die Expressionsdomänen der untersuchten abdominalen Gapgene von Drosophila mit den bekannten Expressionsdomänen in Tribolium fällt auf (Abb. 29: A, B), dass ab dem zweiten abdominalen Segment in Tribolium keine Expression von Gapgenen mehr nachgewiesen wurde. Lediglich hunchback wird später in der Wachstumszone aktiv (Marques-Souza et 88 III. Kapitel: mlpt und svb in Tribolium - Einleitung al., 2008, Wolff et al., 1995). Daraus ergeben sich nun zwei mögliche Hypothesen. Zum einen könnte es sein, dass die abdominale Musterbildung in Tribolium, ebenso wie die anteriore Musterbildung, ohne wesentlichen Einfluss der Gapgene abläuft. Zum anderen ist es denkbar, dass für die posteriore Musterung in Tribolium Kurzkeim-spezifische Gapgene eine Rolle spielen, die nicht von der Drosophila-Situation her abgeleitet werden können (Cerny et al., 2008). Ein solches Kurzkeim-spezifische Gapgen könnte das von Savard et al., (Savard et al., 2006) identifizierte Gen mille-pattes sein. Abb. 29: Vergleich der Gapgen-Expression zwischen Drosophila u. Tribolium A) Die Graphik zeigt die Expression von fünf Gapgenen (tailless, giant, knirps, Krüppel und hunchback) während der Drosophila-Embryogenese entlang der a-p Achse. Es soll veranschaulicht werden, dass alle Segmente im Embryo von verschiedenen, teilweise auch stark überlappenden Gapgen-Expressionsdomänen überspannt werden (nach Cerny et al., 2008). B) Der Segmentanlageplan von Tribolium. Am posterioren Ende befindet sich die Wachstumszone (WZ), aus der die noch fehlenden abdominalen Segmente gebildet werden. Während im anterioren Bereich bis zum zweiten abdominalen Segment Gapgen-Expressionen nachgewiesen werden konnten, ist ab dem dritten abdominalen Segment (grau unterlegter Bereich) keine Expression von Gapgenen bekannt. Einen Teil der Musterbildung in diesem Bereich wird möglicherweise von dem Gen mille-pattes (schwarzer Balken) übernommen, welches aus der frühen Drosophila-Embryogenese nicht bekannt ist (nach Cerny et al., 2008; Savard et al., 2006). 89 III. Kapitel: mlpt und svb in Tribolium - Einleitung Die Expression und Funktion von mlpt in Tribolium mille-pattes (mlpt) wurde im Rahmen eines EST Screens identifiziert (Savard, 2004). Die weitere Charakterisierung und Analyse des Gens fand wenig später statt (Savard et al., 2006) und soll im Folgenden kurz zusammengefasst werden. mille-pattes wird bereits im Blastoderm exprimiert. Die früheste Expressionsdomäne überspannt die zukünftigen Kopflappen; später im differenzierten Blastoderm ist mlpt in einer weiteren Domäne am posterioren Pol des Embryos zu finden. Im Laufe der Keimstreifstreckung bleibt mlpt in einer verkleinerten anterioren Domäne exprimiert, die mit der posterioren Grenze des mandibulären Segments zusammen fällt, sowie einer zentralen Expressionsdomäne die sich vom zweiten thorakalen bis zum dritten abdominalen Segment hin zieht. Eine dritte Expressionsdomäne tritt später wieder im Bereich der Wachstumszone auf. Schließlich wird mlpt in einer vierten Domäne im siebten abdominalen Segment exprimiert. Zudem konnte gezeigt werden, dass mlpt in neuronalen Zellclustern sowohl in den Kopflappen als auch im gesamten thorakalen und abdominalen Bereich aktiviert wird. Schließlich ist auch eine deutliche Expression in den Gelenken der zukünftigen Extremitäten auszumachen (Savard et al., 2006). Parentale RNAi-Experimente zeigen, dass nach einem Knockdown von mlpt zum einen die Segmentidentität von Tribolium stark gestört ist, da ein Großteil der abdominalen Segmente eine thorakale Identität annimmt und Larven mit bis zu zehn Beinpaaren entstehen. Auch konnte eine posteriore Verkürzung der Larven beobachtet werden, was nahe legt, dass mlpt in irgendeiner Weise Einfluss auf die Wachstumszone in Tribolium nimmt. Diese beiden Befunde, also die Transformation von Segmenten, sowie die Deletion abdominaler Segmente, sind Indizien dafür, dass mlpt sich wie ein Tribolium-typisches Gapgen verhält. Untermauert wurde diese Erkenntnis noch von der Entdeckung, dass mlpt in der Lage ist, andere Gapgene zu regulieren; beispielsweise kann in mlpt-RNAi Embryonen die posteriore giantDomäne nicht mehr ausgebildet werden und die Krüppel-Expression expandiert in posteriore Bereiche des Embryos. Umgekehrt haben auch andere Gapgene Einfluss auf die mlptExpression. In giant-RNAi Embryonen ist die zentrale mlpt-Expressionsdomäne viel stärker ausgeprägt und nach anterior verschoben (Savard et al., 2006). Schon die Tatsache, dass mlpt aus der frühen Drosophila-Entwicklung nicht bekannt ist und offenbar eine kurzkeimspezifische Musterbildungsfunktion inne hat, macht es hoch interessant. Hinzu kommt noch, dass mlpt keinen Transkriptionsfaktor codiert, sondern mehrere Polypeptide. 90 III. Kapitel: mlpt und svb in Tribolium - Einleitung Die mlpt-Peptide mlpt codiert für eine polycistronische mRNA, welche wiederum für drei kleine Peptide codiert, die als ORF1A (10 Aminosäuren), ORF2A (12 Aminosäuren) und ORF3A (15 Aminosäuren) bezeichnet werden. Allen drei ORFs ist gemein, dass sie das gleiche Konsensusmotiv - LDPTGLY - enthalten (Abb. 30). Ein von Savard et al., (2006) beschriebener ORF1B, der ebenfalls auf der mRNA liegen soll, wurde in späteren Studien als wahrscheinlich nicht funktionell diskutiert (Galindo et al., 2007). Abb. 30: Struktur der polycistronischen T.c. mlpt-RNA Die codierende Region umfasst 598 bp und enthält 3 ORFs die translatiert werden (mod. Abb. nach Savard et al., 2006 und Galindo et al., 2007). Die ORFA Mlpt-Peptide sind erstaunlich gut konserviert und konnten neben Tribolium in anderen holometabolen Insekten wie Drosophila und Bombyx, sowie in hemimetabolen Insekten wie Aphis oder Locusta, aber auch in Crustaceen wie Daphnia nachgewiesen werden. Die Anzahl der ORFA Paralogen zwischen den verschiedenen Spezies variiert allerdings. Es wird angenommen, dass ursprünglich zwei ORFA Paraloge vorlagen - wie das bei Daphnia der Fall ist - und dass daraus im Laufe der Evolution bis zu 11 Paraloge entstanden sind, wie im Seidenspinner Bombyx mori (Lepidoptera) (Galindo et al., 2007). Mille-pattes ist nicht nur als potentielles neues Gapgen in Tribolium interessant, sondern auch wegen seiner ungewöhnlichen Genstruktur, da es polycistronisch organisiert ist und sehr kleine Peptide codiert (Savard et al., 2006). Polycistronische mRNA, das heißt die Anordnung mehrerer Open Reading Frames auf einer mRNA, wie zum Beispiel das Lac-Operon in E. coli (Jacob und Monod, 1961), ist vor allem in Prokaryoten eine häufig genutzte Art der Genregulation. In Eukaryoten ist dies eher die Ausnahme, da die Translation bei Eukaryoten vorsieht, dass die 40S Ribosomenuntereinheit am 5’ Ende der mRNA bindet und bis zum ersten, auf der mRNA vorhanden Startcodon vorwandert. Unterstützt wird die RibsomenmRNA Bindung durch einen Initiationsfaktor, der sowohl mit dem Ribosom, als auch mit 91 III. Kapitel: mlpt und svb in Tribolium - Einleitung der 5’ mRNA Cap in Wechselwirkung tritt (Kozak, 1999; Pestova et al., 2001). Interne Ribosomen Bindestellen wie sie für Prokaryoten beschrieben wurden und es Ribosomen ermöglichen, weiter 3’ gelegene ORFs zu translatieren, werden in Eukaryoten noch diskutiert (Gilbert, 2010). Es gibt zwar Beispiele von bicistronischen mRNAs in denen ein kurzer ORF einem langen ORF vorangeht (z.B. Insulin im Hühnchen; Mansilla et al., 2005), allerdings dient dieser kurze ORF eher einer Translationsinhibition für den längeren ORF. Dieser kann dann z.B. durch alternatives Splicen aktiviert werden, welches den kleinen 5’ ORF entfernt und dann eine effizientere Translation des längeren ORF ermöglicht (Kozak, 2005, Mansilla et al., 2005). Alternatives Splicen ist auch das Mittel der Wahl um aus polycistronischen mRNAs - die immerhin 25% des C.elegans Genoms codieren - monocistronische mRNA zu prozessieren. Diese wird dann auf konventionellem Weg translatiert (Lawrence, 2002; Evans und Blumenthal, 2000). Dass alle drei ORFAs in Tribolium translatiert werden ist bis jetzt zwar nur eine Vermutung, allerdings konnte für die ORFAs in Drosophila anhand von GFP Markierungen der einzelnen ORFs gezeigt werden, dass in der Tat alle ORFs translatiert werden (Galindo et al., 2007; Kondo et al., 2007). Ein Analogieschluss zur Tribolium Situation ist deshalb durchaus zulässig. Ein Unterschied zwischen den einzelnen ORFs in Drosophila betrifft die Translationseffizienz: ORFs, die näher am 5’ Ende der mRNA lokalisiert sind, werden offenbar mit einer höheren Effizienz translatiert als diejenigen, welche sich näher am 3’ Ende befinden (Galindo et al., 2007). Aus dieser Erkenntnis resultiert auch die Vermutung, dass die Mlpt-Peptide mit Hilfe eines Reinitiationsmechanismus translatiert werden. Dieser bewirkt, dass nach der Translation des ersten Peptides die 40S Untereinheit des Ribosoms nicht von der mRNA abfällt, sondern bis zum nächsten Startcodon weiterwandert und sich dort der Translationskomplex von neuem aufbaut (Kozak 2005). Die Voraussetzung dass es sich bei den vorangegangenen Transkripten um kleine ORFs handelt, ist bei mlpt erfüllt und auch die Tatsache, dass die Effizienz der Translation zum 3’ Ende hin abnimmt, passt zu diesem Modell (Galindo et al., 2007). Eine weitere interessante Eigenschaft der Mlpt-Peptide liegt in ihrer Größe begründet und der damit verbundenen Frage, auf welcher Ebene sie in die Embryonalentwicklung von Tribolium eingreifen. Kleine Peptide sind aus anderen biologischen Kontexten bekannt und gut untersucht. Ein Beispiel wären Neuropeptide, die 50 AS lang sind und durch Modifikation großer Vorläuferproteine im Golgiapparat prozessiert und schließlich aus den Nervenzellen 92 III. Kapitel: mlpt und svb in Tribolium - Einleitung sezerniert werden. Neuropeptide sind sowohl in Vertebraten als auch Invertebraten gefunden worden und dienen vor allem der Zellkommunikation (Penzlin, 7. Auflage, 2005; Burbach, 2011). Ein Beispiel dafür, dass kleine Peptide auch zur Zelldifferenzierung beitragen, sind die Kopf- und Fuss-Aktivatoren in Hydra. Die Bildung der Kopf-Fuß-Achse hängt von der Präsenz dieser kleinen Peptide ab, die wahrscheinlich eine ursprüngliche Form von ZellSignalwegen darstellen. Das Peptid Hym-323 wird in allen epithelialen Zellen außer Kopf und Fuss exprimiert, und ist nach seiner Freisetzung aus diesen Zellen in der Lage, die Bildung eines Hydra-Fußes zu steuern (Bosch und Fujisawa, 2001). Abschließend sei noch erwähnt, dass kleine Peptide auch in der Arabidopsis-Forschung immer mehr in den Fokus geraten, da auch hier gezeigt werden konnte, dass sie einen wichtigen Einfluss auf Zellkommunikation und Differenzierung nehmen (Matsubayashi, 2011). Nun stellt sich die Frage, wie mlpt in Tribolium seine Funktion als Gapgen erfüllt. Eine mögliche Schlüsselrolle könnte dabei ein Gen namens shavenbaby spielen, das in Drosophila durch das ein mlpt-Homolog - polished rice alias tarsaless - reguliert wird. Die Interaktion von Polished-rice/Tarsaless und Shavenbaby in Drosophila Ein Drosophila-Embryo hat auf der ventralen Seite Trichome, zähnchenartige Fortsätze der Epidermiszellen, die durch Aktinbündel am apikalen Pol ausgestülpt werden (Kondo et al., 2007; Delon et al., 2003). Diese Dentikel sind in der anterioren Hälfte der Segmente lokalisiert (Abb. 31: A, B) und bestehen aus sechs bis sieben aneinander grenzenden Reihen; die posteriore Hälfte der Segmente trägt nackte Kutikula (Herth und Sander, 1972; Vogel, 1977). Dieses Muster zieht sich vom thorakalen über den gesamten abdominalen Bereich des Drosophila-Embryos hinweg. Die ventralen Zähnchenbänder dienen vor allem der Fortbewegung der Drosophila-Larve. Auf der dorsalen Seite befinden sich ebenfalls in regelmäßigen Abständen Trichome, welche nicht als Dentikel, sondern als Haare bezeichnet werden. Die Funktion von shavenbaby: Shavenbaby, welches für einen Zinkfinger Transkriptionsfaktor codiert (Mével-Ninio et al., 1991), wurde im Zusammenhang mit der TrichomBildung in Drosophila intensiv untersucht und es stellte sich heraus, dass es eine Schlüsselrolle in der Trichomdifferenzierung einnimmt (Delon und Payre, 2004; Chanut-Delalande et al., 2006). Shavenbaby ist segmental exprimiert (Garfinkel et al., 1994) und wird vom DER-Signalweg aktiviert (DER =Drosophila-EGF-Receptor) (Payre, 2004; O´Kefee et al., 93 III. Kapitel: mlpt und svb in Tribolium - Einleitung 1997). Dies führt zur Bildung der Trichome im anterioren Abschnitt des Segments, während der Wg-Signalweg die Expression von Shavenbaby reprimiert (Noordermeer et al., 1992), was in den nackten Kutikulaabschnitten im posterioren Bereich eines Segments resultiert (Abb. 31: D). Shavenbaby ist an der Transkriptionsaktivierung zahlreicher anderen Gene wie shavenoid oder minature beteiligt, die für die Morphologie der Trichome entscheidend sind (Chanut-Delalande et al., 2006). Das Ausschalten von Shavenbaby führt zum vollständigen Verlust der Dentikel, während ein Ausfall der Zielgene von Shavenbaby lediglich zu verschiedenen morphologischen Veränderungen derselben führt (Chanut-Delalande et al., 2006). Die Funktion von polished-rice/tarsaless: Eine andere Mutation die in einer Störung der Trichombildung resultiert, ist auf das Gen polished-rice (pri) (Kondo et al. 2007) zurückzuführen, das mlpt Homolog in Drosophila. Polished-rice wurde im Zusammenhang mit der Beinentwicklung identifiziert (Galindo et al., 2007). Es wird früh in den Beinimaginalscheiben exprimiert und spielt eine Rolle bei der Ausbildung der tarsalen Segmente (deswegen ist in der Literatur auch der Name tarsal-less zu finden). Nicht nur die Bildung der tarsalen Glieder, sondern auch die Entwicklung der Gelenke zwischen den einzelnen, tarsalen Beinabschnitten ist auf die pri-Aktivität zurückzuführen (Pueyo und Couso, 2011). Zudem ist es wichtig für die Ausbildung des Tracheennetzwerks (Kondo et al., 2007). Im folgenden Abschnitt soll jedoch näher auf den Epidermisphänotyp von pri-Mutanten eingegangen werden, der dem von svb-Mutanten gleicht. Das Zusammenspiel von Shavenbaby und Polished-rice: Es wurde untersucht, ob pri und svb möglicherweise in der Trichommusterbildung interagieren (Kondo et al., 2010). Die segmentalen Expressionsdomänen von polished-rice stimmen mit denen von shavenbaby genau überein. Allerdings konnte in shavenbaby-Mutanten keine Änderung der Expression von polished-rice festgestellt werden, und auch in pri-Mutanten ist shavenbaby weiterhin wildtypisch exprimiert; lediglich die Zielgene von Shavenbaby wurden nicht mehr exprimiert (Kondo et al., 2007). Daraus wurde die Schlussfolgerung gezogen, dass Polished-Rice und Shavenbaby irgendwie parallel wirken müssen. Von Shavenbaby war bekannt, dass das Gen mehrere Isoformen codiert (Garfinkel et al., 1992, Garfinkel et al., 1994). Nämlich Shavenbaby welches die längste Isoform ist, gefolgt von OvoA das einen verkürzten N-Terminus 94 III. Kapitel: mlpt und svb in Tribolium - Einleitung hat und schließlich OvoB das die kürzeste Isoform darstellt (Abb. 31: C). Abb. 31: Zusammenfassung der Dentikelbildung in Drosophila A) Ventro-laterale Ansicht einer Drosophila-Larve. Der Kopf zeigt nach links. Die ventralen Zähnchenbänder sind unterstrichen. B) Schemazeichnung der ventralen Epidermis (modifiziert nach Chanut-Delalande et al., 2006). Sechs bis sieben Reihen von Trichom-bildenden Zellen werden von epidermalen Zellen ohne Trichome gefolgt. C) Schematische Darstellung der Shavenbaby/Ovo Isoformen (modifiziert nach Kondo et al., 2010). Die Isoformen unterscheiden sich in der unterschiedlichen Länge der N-terminalen Domäne. Eine dort befindliche Repressordomäne (rot) ist für die Transkriptionsinhibition zuständig. D) Zusammenfassung der im fortlaufenden Text beschriebenen genetischen Interaktionen zur Aktivierung der Trichombildung (modifiziert nach Chanut-Delalande et al., 2006 und Kondo et al., 2010). OvoA und OvoB werden normalerweise nur in der Keimbahn des Embryos exprimiert und sind dort für die Differenzierung der Polzellen hin zu den weiblichen Keimbahn von großer Bedeutung (Garfinkel et al., 1992; Garfinkel et al., 1994). Die ektopische Expression von OvoA führt zu einer Repression der Dentikelmusterung, wohingegen die ektopische Expression der kürzesten Isoform - OvoB - zur zusätzlichen Bildung von Trichomen beiträgt (Kondo et al., 2010). Die Schlussfolgerung ist, dass die Länge des Ovo/Shavenbaby-Proteins dafür verantwortlich ist, ob es eine aktivierende oder eine reprimierende Wirkung auf die Zielgenexpression hat (Kondo et al., 2010; Andrews et al., 2000). Durch eine Reihe von Ver95 III. Kapitel: mlpt und svb in Tribolium - Einleitung suchen konnte gezeigt werden, dass Polished-Rice-Peptide die inaktive, reprimierende Form von Shavenbaby - wahrscheinlich durch die Aktivierung einer Protease - in die aktivierende Form umwandeln können, indem das initial lange Shavenbaby-Protein posttranslational am N-Terminus gekürzt wird und so durch den Wegfall einer Repressordomäne zu einem Transkriptionsaktivator wird. Es ist also die direkte oder indirekte Interaktion von Pri-Peptiden und Shavenbaby-Protein nötig, um die Aktivierung der Dentikelbildung zu erreichen (Kondo et al., 2010). Die Aktivierung von Shavenbaby durch Pri-Peptide konnte kurze Zeit später auch im Zusammenhang mit der bereits erwähnten Entwicklung der tarsalen Gelenke nachgewiesen werden (Pueye und Couso, 2011), allerdings scheint die Pri-abhängige Tracheenentwicklung ohne die Anwesenheit von Shavenbaby abzulaufen (Kondo et al., 2010), so dass es noch weitere, bis her unbekannte Interaktionspartner von Pri geben muss. Ziele dieser Arbeit Obwohl mlpt in Tribolium initial entdeckt worden ist (Savard et al., 2006) und sein beschriebener Gapgenphänotyp für das Verständnis der Segmentierung eines Kurzkeimers von großer Bedeutung sein könnte, gibt es bis jetzt noch kein Modell, wie mlpt als Nichttranskriptionsfaktor in Tribolium Einfluss auf die Segmentierung nimmt. Im Rahmen dieses Teilprojekts sollte zum einen herausgefunden werden, wie eine Überexpression von mlpt die TriboliumEntwicklung beeinflusst. Dazu wurde das vor kurzem in Tribolium etablierte HitzeschockÜberexpressionssystem verwendet (Berghammer et al. 1999; Lorenzen et al., 2003). mlpt sollte mit dem Hitzeschockpromotor hsp68 gekoppelt in den Transformationsvektor piggyBac kloniert und in die Keimbahn von Tribolium verbracht werden. Durch die Aktivierung des Hitzeschockpromotors kann dann eine mlpt-Überexpression in den Embryonen dieser transgenen Linien hervorgerufen werden. Nach Abschluss der Embryogenese sollten dann die phänotypische Effekte der mlpt-Überexpression untersucht werden. Ein weiteres Ziel dieser Arbeit ist es den Mechanismus zu entschlüsseln, wie mlpt als Nicht-Transkriptionsfaktor die Expression von anderen Gapgenen beeinflussen kann. Ein mögliches Ziel der mlpt-Funktion ist der aus Drosophila bekannte Transkriptionsfaktor Shavenbaby, auf den die Drosophila-Homologen der Mlpt-Peptide einen regulatorischen Einfluss haben (Kondo et al., 2010). Dieser Transkriptionsfaktor sollte kloniert und sowohl seine Expression durch in-situ-Hybridisierung (Tautz und Pfeifle, 1989), und die seine Funktion mittels parentaler RNAi (Bucher et al., 2002) näher untersucht werden. 96 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden Material & Methoden Eine ausführliche Liste der verwendeten Puffer und Lösungen befindet sich in Anhang A. Die Experimente werden an zwei Käferstämmen durchgeführt: Tribolium castaneum San Bernadino und Tribolium castaneum vermillionwhite (Lorenzen et al., 2002). Da sie sich in der Haltung und Zucht nicht unterscheiden, wird im Folgenden die Mehlkäferhaltung im Allgemeinen kurz zusammen gefasst. Tribolium castaneum: Haltung und Zucht Nahrung: Die Mehlkäfer befinden sich in verschließbaren Plastikboxen (15 cm x 15 cm x 12 cm) in einem Mehl (doppelgriffiges Rosenmehl, Typ 405, Wiener Griessler)/Hefegemisch (Bierhefepulver, Cenovis). Pro kg Mehl werden 50 g Hefe zugesetzt sowie 0.3 g Fumidil, um Pilzentwicklung zu vermeiden. Temperatur: Die Generationszeit kann durch die Haltung der Käfer bei 25 °C bzw. 32 °C beeinflusst werden. Tab. 7: Entwicklungsdauer von Tribolium bei unterschiedlichen Temperaturen Temperatur 25 °C 32 °C Embryonalentwicklung Larvale Stadien Puppenstadium Gesamte Entwicklungsdauer (nach Sokoloff, Vol.2, Seite 70) 6.8 d 31.2 d 10.2 d 48 d 2.9 d 14.6 d 4.6 d 22 d Luftfeuchtigkeit: 45-50%. Separierung verschiedener Entwicklungsstadien: Um Käfer, Puppen, Eier und Mehl voneinander zu trennen, kommen verschiedene Siebsätze zum Einsatz. Siebe (Analysesiebe, Retsch) mit einer Maschenweite von 700 µm dienen zum Absammeln von Käfer oder Puppen; Siebe mit einer Maschenweite von 300 µm zur Gewinnung der Eier. 97 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden Etablierung des mille-pattes-Überexpressionssystems Die Erstellung eines mille-pattes-Überexpressionssystems untergliedert sich in drei verschiedene Arbeitsschritte (Herstellung des Vektors, embryonale Injektion sowie Etablierung der transgenen Linien), die im Folgenden dargestellt werden. Herstellung des Mille-pattes Überexpressionsvektors: Ausgangsmaterial ist ein pBluescript-Vektor, der die gesamte, kodierende Region des T.c. mlpt Gens (616 bp) enthält (Accession: AM269505.1) und freundlicherweise von J. Savard zur Verfügung gestellt wurde (Savard et al., 2006). Um mille-pattes während verschiedener, embryonaler Entwicklungsstadien überexprimieren zu können, wird das mlpt Gen zunächst mit dem Hitzeschockpromotor hsp68 versehen und schließlich in einen piggyBac Transformationsvektor kloniert (Horn und Wimmer, 2000). Die Vektorkarten der verwendeten Plasmide befinden sich im Anhang C. Abb. 32: Übersicht der Klonierungsstrategie von mlpt in den piggyBac Vektor 98 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden Die Klonierung des mlpt-Gens erfolgt in 3 Schritten. Zunächst wurden mittels PCR an das 5’und das 3’-Ende Restriktionsschnittstellen angefügt, sodass eine direktionale Klonierung in den Hitzeschockvektor pSLfa stattfinden kann, aus dem das gesamte Konstrukt (Promotor, 5’UTR, mlpt-cDNA, 3’UTR) dann in den Transformationsvektor piggyBac subkloniert wurde. I. Einfügen von Restriktionsschnittstellen an die 5’- und 3’-Enden des mlpt-Gens im pBluescriptvektor: Da die mlpt-cDNA gerichtet in verschiedene Vektoren kloniert werden sollte, war es nötig sie am 5’- und 3’-Ende mit Restriktionsschnittstellen zu versehen. Alle verwendeten Primer sind von der Firma Metabion. PCR mit mlpt-Fusionsprimern fw Primer (fw1-mlpt-Not1-Xba1): 5’-GCT CTA GAG CGG CCG CTA AAT TCC CAG CGC GAG TCG TGC CAA GTT ATG TCA GGT CTC G-3’ (Tm=62 °C) bw Primer (bw1-mlpt-AsiS1-Xho1): 5’-CCC CTC GAG GCG ATC GCA TTT TAT TCA TTT CCA AG-3’ (Tm=42 °C) Reaktionsansatz: 3 µl Phusion HF Reaktionspuffer (5x, Finnzymes), 3 µl dNTPs (2mM, Peqlab), 1.5 µl fw Primer (20 µM, Metabion), 1.5 µl bw Primer (20 µM, Metabion), 0.5 µl Taq-Phusion DNA Polymerase (2 U/µl, Finnzymes), 2 µl Template (mlpt-pBluescript; 50 ng/ µl), 18.5 µl Wasser. PCR-Programm: 98 °C/2 min; (98 °C/30s, 40 °C/45s, 72 °C/60s) x 30; 72 °C/7 min. Zur Entfernung von Primern, Oligonukleotiden etc. aus dem PCR Ansatz wird anschließend eine Gelextraktion mit dem Min Elute Gel Extraction Kit 50 (Qiagen) durchgeführt (nach den Herstellerangaben). Da das Schneiden von endständigen Restriktionsschnittstellen sich als ineffizient erwiesen hatte, wird das modifizierte mlpt-Gen (mlpt-mod) erneut in pBluescript kloniert. Die Klonierung erfolgt nach Standardprotokoll (vgl. auch Kapitel I, Material & Methoden). 99 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden II. Restriktionsverdau des aufgereinigten mlpt-mod pBluescript Vektors sowie des pSLfa[Tc’hsp5’3’UTR]fa Vektors: Doppelverdau des mlpt-mod pBluescript Vektors: 1 µl Xba1 (20.000 U/ml, NEB), 1 µl Xho1 (20.000 U/ml, NEB), 3 µg mlpt-mod pBluescript Vektor, 5 µl NEB Puffer 4, 0.5 µl BSA (10 mg/ml), mit Wasser auf 50 µl auffüllen. Trippelverdau des pSLfa[Tc’hsp5’3’UTR]fa Vektors: 1 µl Xba1 (20.000 U/ml, NEB), 1 µl Xho1 (20.000 U/ml NEB), 1 µl Not1 HF (20.000 U/ml, NEB), 3 µg pSLfa[Tc’hsp5’3’UTR]fa Vektor, 5 µl NEB Puffer 4, mit Wasser auf 50 µl auffüllen. Beide Ansätze werden über Nacht bei 37 °C inkubiert und schließlich bei 65 °C inaktiviert. Durch eine abschließende Gelextraktion mit dem Min Elute Gel Extraction Kit (Qiagen) isoliert man das ca. 632 bp lange mlpt-mod Gen, sowie das 4236 bp lange Backbone des Hitzeschockvektors. III. Ligation des mlpt-mod-Gens mit pSLfa[Tc’hsp5’3’UTR]fa: Für die spätere Aktivierung des mlpt-Gens durch Hitzeschockpromotoren, ligiert man den geschnittenen pSLfa[Tc’hsp5’3’UTR]fa Vektor mit dem ebenfalls geschnittenen mlpt-mod Gen. Zur Optimierung der Ligationseffizienz, hat es sich bewährt, die Mengenverhältnisse von Fragment und Vektor nach folgender Formel zu berechnen: MasseFragment (ng) = 5 x Masse Vektor x Länge Fragment (bp)/Länge Vektor (bp) Daraus resultiert folgender Ligationsansatz: 300 ng pSLfa[Tc’hsp5’3’UTR]fa Vektor, 48 ng mlpt-mod Fragment, 4 µl Ligationspuffer (NEB), 1 µl T4 DNA-Ligase (400 U/µl, NEB), mit Wasser auf 20 µl auffüllen. Die Ligation findet über Nacht bei 16 °C statt. Nach Transformation elektrokompetenter Bakterien (TOP 10) nach Standardprotokoll (vgl. auch Kapitel I, Material & Methoden) und der Auswahl eines pSLfa[Tc’hsp5’-mlpt-mod-3’UTR]fa positiven Klons findet erneut eine Anreicherung des Vektors und eine abschließende Aufreinigung mit dem Qiafilter Plasmid Midi Kit (Qiagen) statt. 100 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden IV. Verdau pSLfa[Tc’hsp5’-mlpt-mod-3’UTR]fa und pBac[3xP3-DsRedaf]: Doppelverdau des pSLfa[Tc’hsp5’-mlpt-mod-3’UTR]fa Vektors: 1 µl AscS1 (10.000 U/ml, NEB), 1 µl Fse (8.000 U/ml NEB), 3 µg Vektor, 5 µl NEB-Puffer 4, 0.5 µl BSA (100 mg/ml), mit Wasser auf 50 µl auffüllen. Doppelverdau des pBac[3xP3-DsRedaf] Transformationsvektors: Der pBac[3xP3-DsRedaf] Transformationsvektor wurde von Tobias Merkel zur Verfügung gestellt (Tobias Merkel, Masterarbeit 2010). 1 µl AscS1 (10.000 U/ml, NEB), 1 µl Fse (8.000 U/ml, NEB), 3 µg Vektor, 5 µl NEB-Puffer 4, 0.5 µl BSA (100 mg/ml), mit Wasser auf 50 µl auffüllen. Beide Ansätze werden eine Stunde bei 37 °C verdaut. Den Abschluss bildet eine 20 minütige Hitzeinaktivierung der Enzyme bei 65 °C. Das gewünschte hsp5’-mlpt-mod-3’UTR Fragment ist 1709 bp lang, das pBac[3xP3-DsRedaf] 6423 bp. Mittels einer Gelextraktion mit dem Qiagenkit werden die beiden Fragmente isoliert. V. Ligation des hsp5’-mlpt-mod-3’UTR Fragments mit dem pBac[3xP3-DsRedaf] backbone: Die Ligation wird genauso angesetzt, wie bereits ausführlicher unter III. beschrieben: 100 ng geschnittenes pBac[3xP3-DsRedaf] backbone, 150 ng hsp5’-mlpt-mod-3’UTR Fragment, 4 µl Ligationspuffer (NEB), 1 µl T4 DNA-Ligase (400 U/µl, NEB), mit Wasser auf 20 µl auffüllen. Für 16 Stunden bei 16 Grad ligieren. Schließlich erfolgt die Transformation von E.colis, TOP 10 One Shot. Mit einer ausgewählten Kolonie wird eine Anreicherung des pBac[3xP3DsRedTc’hsp5’-mlpt-mod-hsp3’] Plasmids und eine Aufreinigung mit dem Qiafilter Plasmid Midi Kit (Qiagen) durchgeführt. 101 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden VI. Sequenzierung und Verifizierung des neuen pBac[3xP3-DsRedTc’hsp5’-mlpt-modhsp3’] Vektors: 2 Sequenzieransätze wurden zur Firma Seqlab nach Göttingen geschickt. Einmal wurde mit dem bereits unter Abschnitt I vorgestellten bw Primer (bw1-mlpt-AsiS1-Xho1) sequenziert um zu kontrollieren, ob sich tatsächlich das mlpt-Gen im Vektor befindet. Die zweite Sequenzierung fand mit folgendem Sequenzierprimer (Metabion) statt, der von der piggyBac L-Site aus startet und von Jutta Distler zur Verfügung gestellt wurde. Seqprim: 5’ - GCT TGT TGG TGA GGA TTC TG - 3’. Beide Sequenzierergebnisse zeigten 100% Übereinstimmung mit dem mlpt-Gen und der anschließenden Hitzeschockpromotor 5’ UTR bzw. mit der piggyBac L Site, so dass von einer erfolgreichen Fertigstellung des Konstrukts ausgegangen werden kann. Embryonale Injektion des Mille-pattes Überexpressionsvektors: Das Protokoll der embryonalen Injektion richtet sich im Wesentlichen nach Berghammer et al., (2009). Der Vollständigkeit halber wird ein kurzer Überblick über den Versuchsablauf gegeben. Für diesen Versuch wird der Tribolium vermillionwhite Stamm verwendet. vermillion codiert eine Tryptophan Oxygenase, die bei der Pigmentierung der Augen eine Rolle spielt. Käfer des vermillionwhite Stammes haben deshalb - im Gegensatz zum San Bernadino Stamm weiße, unpigmentierte Augen (Lorenzen et al., 2002). Die fehlende Pigmentierung erleichtert im weiteren Versuchsablauf, die Detektion des rot fluoreszierenden Transformationsmarkers dsRed, welcher an einen Augen-spezifischen 3xP3 Promotor gekoppelt ist. Somit ist eine schnelle Unterscheidung von transformierten und nicht-transformierten Käfern möglich. Der vermillionwhite Stamm wird für einige Tage von 25 °C auf 32 °C gestellt, so dass sich die Tiere an die Temperatur adaptieren können. Am Tag der Injektion werden jeweils 1h Gelege gemacht, die dann für eine weitere Stunde bei 25 °C nachreifen. Diese Eier transferiert man in ein Schraubsiebchen mit einer Maschenweite von 200 µm und wäscht sie gründlich mit ca. 1l Leitungswasser, das Zimmertemperatur hat um Mehlreste zu entfernen. Schließlich werden die Eier unter dem Binokular mit Hilfe eines feinen Pinsels (Leonardy, 0) auf einem Objektträger (Roth) aufgereiht. Dabei ist zu beachten, dass die posteriore Eiseite (schmale, spitze Seite) stets zur Längskante des Objektträgers zeigt. 102 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden Die Injektion der aufgereihten Käfereier findet unter dem Mikroskop auf der posterioren Seite statt. Die Injektionslösung setzt sich wie folgt zusammen: 500 ng/µl des Transformationsvektors (pBac[3xP3-DsRedTc’hsp5’-mlpt-mod-hsp3’]), 350 ng/µl Helperplasmid (codiert die Transposase), 10% Phenolrot (Sigma), mit Wasser auf ein Gesamtvolumen von 40 µl auffüllen. Um zu verhindern dass später kleinen Partikel die Injektionsnadel (Borosilikat Glasnadel, World Precision Instruments) verstopfen, wird dieser Ansatz kurz zentrifugiert und beim Befüllen der Nadel darauf geachtet, die Lösung von der Mitte des Eppendorfröhrchens aufzunehmen. Die Nadel mit der Injektionslösung spannt man in einen Mikromanipulator ein. Da die Glasnadel an der Spitze durch den Schmelzprozess zugeklebt ist, ist es wichtig, sie an der Kante des Objektträgers vorsichtig zu brechen. Anschließend werden die unter dem Mikroskop befindlichen Embryonen auf dem Objektträger an der Nadelspitze vorbeigeführt. Die Nadel wird bei jedem Embryo in die posteriore Seite eingestochen und so lange Injektionslösung reingedrückt, bis sich der Embryo leicht rot färbt. Sind alle Embryonen auf diese Art und Weise injiziert, werden sie mit samt dem Objektträger auf eine flache Schale gelegt und in einer feuchten Kammer (21 cm x 15 cm x 15 cm, 100 ml Wasser mit 15% NaCL Lösung) für 3-4 Tage bei 32 °C gelagert. Danach wird die Schlupfrate bestimmt, die geschlüpften Larven auf Mehl gegeben und bis zum Erreichen des pupalen Stadiums erneut bei 32 °C gehalten. Ist das pupale Stadium erreicht, werden Einzelverpaarungen angesetzt. Die aus dieser G0-Generation resultierenden Nachkommen (G1 Generation) sollten - bei erfolgreicher Transformation der Keimbahn der Elterntiere - das mlpt-Überexpressionsplasmid in ihr Genom integriert haben. Selektion und Etablierung transgener Mille-pattes Linien: Die G1 Generation der angesetzten Einzelkreuzungen untersucht man unter dem Fluoreszenzbinokular (Nikon SMZ 1500) mit dem dsRed Filter. Rotleuchtende Augen zeigen ein positives Transformationsereignis an, während das Fehlen der Fluoreszenz dahingehend zu interpretieren ist, dass keine Insertion des mlpt-Überexpressionskonstrukts in die Keimbahn der Elterntiere stattgefunden hat. Die dsRed positiven Nachkommen der verschiedenen Einzelkreuzungen werden in separate, verschließbare Röhrchen mit Mehl gegeben und im 4-6 Wochen Rhythmus nach selektiert, so dass man langfristig gesehen stabile mlpt-Überexpressionslinien erhält. 103 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden Durchführung des Hitzeschocks zur Aktivierung der mlpt-Überexpression Zur Analyse der phänotypischen Auswirkungen einer erhöhten Konzentration der Mlpt Peptide, muss der endogene Hitzeschockpromotor am 5’ bzw. 3’ Ende des mlpt-Gens der transgenen Linien aktiviert werden. Dazu überführt man die Eier der zu analysierenden Linien in verschließbare Szintillationsgläschen. Diese werden in einem Styroporschwimmer befestigt und für exakt 10 Minuten in ein Wasserbad (Haake S) mit einer Temperatur von 46 °C gestellt. Nach Ablauf dieser Zeit kommen die Eier zum Nachreifen in den 32 °C Inkubator (Schinko et al., 2010). 4 Tage später werden Kutikulapräparate angefertigt. Nach dem Entfernen von Mehl und Chorion durch 50% Danklorix (2 x 3 min), nimmt man die Embryonen mit einem Pinsel auf und überführt sie in einen Tropfen Hoyers/Milchsäure (50% Hoyers Medium/50% Milchsäure). Die Präparate werden über Nacht bei 60 °C geklärt und können aufgrund ihrere Autofluoreszenz am nächsten Tag mit Hilfe des Fluoreszenzmikroskops (Zeiss, Axiophot) ausgewertet werden. Nachweis der mlpt-Überexpression nach dem Hitzeschock via ISH Die Aktivierung des mlpt-Überexpressionskonstrukt durch den Hitzeschock soll anhand einer in-situ-Hybridisierung überprüft werden. Für eine ausreichende Menge an Embryonen hitzeschockt man über mehrere Tage hinweg 0-24h alte Embryonen. Nach jedem Hitzeschock reifen die Embryonen für 45 Minuten bei 32 °C nach. Dieser Zeitraum ist nötig, damit das Hitzeschockkonstrukt exprimiert werden kann (Merkel, Masterarbeit 2010). Die Fixierung der Embryonen, die Sondensynthese der DIG-markierten Sonden und in-situHybridisierung erfolgen gemäß Standardprotokollen. mlpt-RNAi in einer mlpt-Überexpressionslinie Es soll zudem ermittelt werden, ob der von Savard et al. (2006) beschriebene mlpt-Phänotyp in Tribolium sich aufgrund der Überexpression der mlpt Linien nach einem Hitzeschock kompensieren lässt. Dazu werden ca. 50 Weibchen aus einer Hitzeschocklinie aussortiert und diese mit mlpt-dsRNA injiziert (ds-RNA Synthese: T7-Megaskript Kit (Ambion); Injektion: Bucher et al., 2002; Posnien et al., 2009). Alle injizierten Weibchen werden dann auf Mehl bei 32 °C gehalten. Die Zeiträume der Eiablage betragen 0-8h bzw. 8-16h. Von den Eiern einer jeweiligen Ablage werden 50% gehitzeschockt und dann zum Nachreifen für 4 104 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden Tage auf 32 °C gestellt, während die anderen 50% nicht gehitzeschockt werden und ebenfalls auf 32 °C gebracht werden. Nach Ablauf von 4 Tagen werden von beiden Ansätzen Kutikulapräparte angefertigt und die Phänotypen unter dem Fluoreszenzmikroskop ausgezählt. Die Klonierung des Transkriptionsfaktors shavenbaby Die Klonierung in den pBluescript-Vektor erfolgte nach Standardprotokoll (s.h. auch Material & Methoden, Kapitel I). Mit Hilfe von NCBI wird die Tribolium svb Sequenz (Accession: XM_969788.1) ermittelt und mit dem Programm Primer3 (Steve Rozen and Helen J. Skaletsky, 2000) potentielle Primerpaare herausgesucht. Die einzelnen Zwischenschritte der Klonierung sind in Material & Methoden des ersten Kapitels zusammengefasst und richten sich nach Standardprotokollen. Die Sequenzen finden sich unter Anhang B. Tab. 8: Primer für die Klonierung von T.c. shavenbaby Primername Sequenz 5’-3’ Tm Länge fw-svb-Primer AAT ACG CCC CTA TCG TAC CC (60 °C) 434 bp bw-svb-Primer ACC CGA TTG AGA ATT TGT CG (56 °C) Pupale Injektion von dsRNA in Tribolium (Posnien et al., 2009) Um die Phänotypen von svb und mlpt miteinander zu vergleichen, werden pupale Injektionen durchgeführt. Die Synthese von dsRNA erfolgt mit dem T7-Megaskript Kit von Ambion. Beide Gene wurden mit einer Konzentration von 2 µg/µl bzw. 4 µg/µl injiziert. Für die pupale Injektion werden weibliche Tribolium-Puppen aus dem San Bernadino Stamm aussortiert. Man reiht sie an der Längskante eines Objektträgers so auf, dass die posterioren Enden der Puppen zur Kante hin zeigen. Die Puppen werden für die Injektion zunächst fixiert: Man zieht mit einem Kleber (Fixogum, Marabu) eine dünne Linie und schiebt die Puppen zügig mit einer Federstahlpinzette in den Kleber. Dabei ist zu beachten, dass nur das letzte abdominale Segment in den Kleber eintaucht. Wird die Puppe zu weit in den Kleber geschoben, besteht die Gefahr, dass beim adulten Käfer der Hinterleib so stark verklebt ist, dass er nicht aus der letzten Puppenhülle heraus kommt und verstirbt. Sind alle Puppen aufgereiht, wird mit Hilfe des Mikromanipulators die ds-RNA (Synthese erfolgte mit dem T7-Megaskript Kit (Ambion)) lateral ins Ab- 105 III. Kapitel: mlpt und svb in Tribolium - Material & Methoden domen injiziert. Es wird so viel Volumen injiziert, dass sich die Puppe leicht streckt und sich die am Bauch gefalteten Elytren etwas anheben. Schließlich werden die Objektträger mit den injizierten Puppen auf Mehl gelegt, die vorsortieren Puppen Männchen dazu gegeben und bei 32 °C zu adulten Käfern hochgezogen. Mehrtägige Eiablagen (über das Wochenende) werden für Kutikulapräparate verwendet, ansonsten finden Fixierungen von 0-24h alten und 24-28h alten Embryonen statt. Auf die fixierten mlpt- bzw. svb-RNAi-Embryonen werden zur weiteren Analyse Einzel- und Doppel-ISHs gemacht, wobei die Fixierung der Tribolium-Embryonen sowie die ISH nach Standardprotoll (Tautz & Pfeifle, 1989) durchgeführt wurde. 106 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Ergebnisse Etablierung des mlpt-Überexpressionssystems in Tribolium Der im Rahmen des Projekts generierte Vektor pBac[3xP3-DsRedTc’hsp5’-mlpt-mod-hsp3’] wird zusammen mit dem Helperplasmid, welches die piggyBac-Transposase codiert, durch Injektion in den posterioren Pol von frisch abgelegten Eiern in die Keimbahn von Tribolium eingebracht. Etwas 10% der überlebenden Embryonen/Larven wiesen Insertionen im Genom der Urkeimzellen auf, die auf Nachkommen übertragen wurden, so dass stabile hsp68-mlptÜberexpressionslinien resultierten. Die Ergebnisse der embryonalen Injektion sind in Tabelle 9 zusammengefasst. Tab. 9: Embryonalen Injektion zur Generierung der hsp68-mlpt-Linien Injizierte Eier Schlupfrate in % Transgene Linien 340 8.7% (86 Larven) 8 Durch einen Hitzeschock sollte nun die Überexpression von mlpt in diesen acht transgenen Linien (Benennung: hsp68-mlpt-Linie 1-8) ausgelöst werden und die Wirkung von ektopischem mlpt in Kutikulapräparaten bzw. mittels in-situ-Hybridisierung näher analysiert werden. Ektopische Expression von mlpt in transgenen mlpt-Linien nach Hitzeschock Der erste Schritt bei der Analyse der hsp68-mlpt-Linien war zu untersuchen, ob durch den Hitzeschock tatsächlich Überexpression von mlpt induziert wurde, das Konstrukt also wie geplant funktionierte. Dazu wurden 0-24h alte vermillionwhite - Embryonen und 0-24h alte Embryonen der Hitzeschocklinien in parallelen Ansätzen gleichzeitig einem zehn minütigem Hitzeschock unterzogen, für 45 min zum Nachreifen gestellt, fixiert und für die mlpt in-situ-Hybridisierung eingesetzt. Da der hsp68-Promotor nach seiner Aktivierung offenbar in allen Geweben aktiv ist (Schinko et al., 2010), war die Erwartung, dass nach dem Hitzeschock der hsp68-mlpt-Embryonen mlpt-Transkript in allen Zellen nachweisbar wäre. Eine Kontrolle mit vermillionwhite -Embryonen sollte ausschließen, dass evtl. auftretende Hintergrundfärbung mit einer mlpt-Überexpression verwechselt wird. 107 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 33 zeigt die mlpt-Expression in hitzebehandelten vermillionwhite -Embryonen ab dem Keimrudimentstadium, welche dem Muster entspricht, das von Savard et al., (2006) detailliert beschrieben wurde. Im Keimrudiment ist mlpt anterior in einem schmalen Streifen exprimiert, der sich (laut Savard et al.) mit der posterioren Grenze des mandibulären Segments deckt. Eine zweite Domäne überlappt mit dem zweiten thorakalen Segment und erstreckt sich etwa bis zum dritten abdominalen Segment (Abb. 33: A, B). Während die anteriore Expressionsdomäne verschwindet, streckt sich die posteriore Domäne zeitgleich mit dem wachsenden Keimstreif, und eine weitere mlpt-Expressionsdomäne tritt im Bereich der Wachstumszone auf (Abb. 33: C). Während der beginnenden Keimstreif-Verkürzung verschwinden diese mlpt-Domänen und das Gen wird nunmehr in einem neuronalen Muster ausgeprägt (Abb. 33: D). Hitzebehandelte Embryonen aller acht hsp68-mlpt-Linien zeigen hingegen eine deutliche Expression von mlpt im gesamten Embryo (Abb. 33: A’-E’). Bis zum Stadium des differenzierten Blastoderms ist allerdings keine ektopische Expression von mlpt feststellbar (nicht gezeigt). Die früheste Detektion der Überexpression ist im Keimrudiment möglich (Abb. 33: A’) und bleibt dann in allen Stadien der Keimstreifstreckung hindurch nachweisbar (Abb. 33: B’-D’). Die Hitzeschock-Expression ist jedoch nicht übermäßig stark, da die endogenen Expressionsdomänen vor diesem Hintergrund weiterhin deutlich zu erkennen sind. Insgesamt zeigt dieses Experiment, dass das mlpt-Überexpressionskonstrukt funktional ist und eine zeitlich definierte Überexpression der Mlpt-Peptide nach dem Blastodermstadium möglich ist. Der nächste Abschnitt widmet sich der Fragestellung wie sich die ektopische Expression von mlpt auf das phänotypische Erscheinungsbild der Tribolium-Larven auswirkt. Die Überexpression von mlpt hat keine offensichtlichen phänotypischen Auswirkungen auf larvale Strukturen Parentale mlpt-RNAi in Tribolium resultiert in der homeotischen Transformation abdominaler Segmente zu thorakaler Identität und der Deletion abdominaler Segmente. Auch eine Verkürzung der Beine ist beschrieben. Dieser Phänotyp wurde jedoch nicht näher analysiert (Savard et al., 2006). Um die Funktion der Mlpt-Peptide während des Segmentierungsprozesses besser verstehen zu können, sollten die Effekte der mlpt-Überexpression in allen acht hsp68mlpt-Linien auf Kutikulaebene untersucht werden. Nach der Induktion der Überexpression mittels Hitzeschock, wurden die Embryonen bis zum Erreichen des larvalen Stadiums auf 32 108 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 33: mlpt-Überexpression nach Aktivierung des hsp86-mlpt Konstrukts mlpt in-situ-Hybridisierung während der Keimstreifstreckung in hitzebehandelten Embryonen des vermillionwhite -Stammes (A-D) sowie vergleichbare Entwicklungstadien ebenfalls hitzebehandelter Embryonen der hsp68-mlpt-Überexpressionslinien (A’-D’). Im Keimrudiment wird mlpt in vermillionwhite -Embryonen in einer schmalen anterioren Domäne exprimiert, sowie in einer breiteren posterioren Domäne (A, B). Im weiteren Verlauf der Entwicklung verschwindet die anteriore Expressionsdomäne und wird durch ein neuronales Kopfmuster ersetzt (Pfeil) (C, D). Während mlpt zunächst noch eine dritte Domäne in der posterioren Wachstumszone des Embryos bildet (C), ist nach Beendigung der Keimstreifstreckung mlpt in einem ausschließlich neuronalen Muster (Pfeil) aktiv. Im Vergleich dazu, ist mlpt in hsp68-mlpt-Linien nach dem Hitzeschock ubiquitär verteilt (A’-D’). 109 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse °C gehalten, eingebettet und auf Kutikula-Phänotypen hin untersucht. Als Kontrolle dienten erneut Embryonen des vermillionwhite -Stammes. Von den acht getesteten hsp68-mlpt-Linien zeigten sieben Linien trotz Aktivierung des Hitzeschocks keinen spezifischen Phänotyp. Seltene phänotypische Defekte wie die Deletion thorakaler Segmente (Abb. 34: C, D), oder fehlende Kopfanhänge sind auf Entwicklungsdefekte zurückzuführen, die durch den Hitzeschock während der frühen Embryogenese verursacht werden (Merkel, Masterarbeit 2010). Lediglich eine Linie (Nr.8) zeigt einen spezifischen Phänotyp (Abb. 34: D). Diese Larven zeigen eine deutliche Reduktion aller drei Beinpaare auf: Nur Coxa und Trochanter sind entwickelt, die restlichen Beinsegmente (Femur, Tibia und Tarsus) fehlen. Am posterioren Ende fehlen außerdem die terminalen Strukturen, die so genannten Pygopodien. Eine genauere Untersuchung dieser Linie zeigt jedoch, dass dieser Phänotyp auch ohne Hitzeschock auftritt (Tabelle 10) und zwar mit einer Häufigkeit von ca. 25%, die unabhängig von der induzierten mlpt-Überexpression war. Da der Stummelbeinphänotyp stark an den für Distalless beschriebenen Phänotyp in Tribolium erinnert (Beermann et al., 2001), wurde ein Komplementationstest mit einer Distal-less/godzilla-Linie durchgeführt (Sa-8/god), die Martin Klingler zur Verfügung stellte. Die Analyse der F1 Generation ergab, dass hier ebenfalls der Stummelbeinphänotyp auftritt. Das bedeutet, dass keine Komplementation stattgefunden hat und dass in der hsp68-mlptLinie 8 offenbar die Insertion des Konstrukts das Distal-less Gen inaktiviert hat. Zusammengefasst kann konstatiert werden, dass in allen untersuchten hsp68-mlpt-Linien die Überexpression von mlpt zu keiner phänotpyischen Ausprägung führt. Tab. 10: Verteilung des Stummelbeinphänotyps in der hsp68-mlpt-Linie 8 Embryonen gesamt WT-Embryonen Stummelbeinphänotyp Phänotypenhäufigkeit mit Hitzeschock ohne Hitzeschock 14 11 3 21.4% 37 29 8 21.6% 110 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 34: mlpt-Überexpression hat keine Auswirkungen auf Kutikula-Phänotyp A) Tribolium WT: Kopf, drei thorakale Segmente (T1-T3) sowie acht abdominale Segmente (A1-A8) an welche eine terminalen Struktur - die sogenannten Pygopoden (Py) angrenzen. Im Vergleich dazu zeigen C) und D) Embryonen nach dem Hitzeschock mit fehlenden thorakalen Strukturen. D) Ein starker Beinphänotyp kann in einer hsp68-mlpt-Linie gezeigt werden. Die Beinentwicklung endet mit dem Trochanterring (Pfeil). Alle thorakalen Segmente und abdominalen Segmente sind ausgebildet, lediglich die terminale Struktur fehlt. Der mlpt-RNAi Effekt kann durch mlpt-Überexpression in den hsp68-mlpt-Linien nicht gemildert werden In früheren Arbeiten (Bucher und Klingler, 2004) konnte gezeigt werden, dass die Stärke von RNAi-Phänotypen von der initialen Konzentration der injizierten Doppelstrang-RNA abhängen kann: niedriger konzentrierte dsRNA führt zu schwächeren Phänotypen als höher konzentrierte dsRNA. Um herauszufinden ob die Hitze-induzierte Expression des mlpt-Transkripts überhaupt funktionelle Peptide generiert, sollte getestet werden ob eine Überexpression von mlpt zu einer Abschwächung des mlpt-RNAi-Effekts führen kann, im Vergleich zu RNAi-Embryonen bei denen keine Überexpression ausgelöst worden war. Es wurden ca. 30 adulte Käferweib- 111 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse chen der hsp68-mlpt-Linie Nr.2 mit 2 µg/µl mlpt-dsRNA injiziert. Von den Eiablagen wurden jeweils 50% hitzebehandelt und mit den nicht hitzebehandelten Ablagen zur weiteren Entwicklung auf 32 °C gebracht. Nach Beendigung der Embryogenese wurden Kutikulapräparate angefertigt und auf zwei Kritierien hin untersucht: Zahl der Beinpaare und der Segmente pro Larve (Tabelle 11). Tab. 11: Auswirkungen der mlpt-Überexpression auf den mlpt-RNAi-Effekt mlpt-RNAi-Embryonen der hsp68-mlpt-Linie Beinpaare* thorakale und abdominale Segmente* Ohne HS (n=40) Mit HS (n=47) 5.03 +/- 0.24 4.62 +/- 0.21 7.38 +/- 0.24 6.96 +/- 0.36 *angegeben sind jeweils Mittelwert sowie Standardabweichung Die Tabelle zeigt, dass im Schnitt die Zahl der Beinpaare in mlpt-RNAi-Embryonen nach der Aktivierung der mlpt-Überexpression durch den Hitzeschock geringer ist (4.62 Beinpaare), als in der Kontrolle (5.03 Beinpaare), die nicht hitzebehandelt war. Allerdings ist der Unterschied wohl nicht signifikant. Ein marginaler Unterschied konnte auch in der Segmentanzahl gefunden werden. mlpt-RNAi-Embryonen die nicht hitzebehandelt wurden, bildeten im Schnitt 7.38 Segmente aus; dem gegenüber stehen 6.96 Segmente bei Embryonen, die einem Hitzeschock unterzogen wurden. Aber auch hier waren die Unterschiede nicht signifikant, weswegen dieser Versuchsansatz nicht weiter verfolgt worden ist. Die nun folgenden Ergebnisse beziehen sich auf einen potentiellen Mlpt-Interaktionspartner, den Transkriptionsfaktor Shavenbaby (Kondo et al., 2010). Die Expression von shavenbaby in Tribolium In Drosophila sind viele Gene an der Ausbildung der Trichome beteiligt. Unter anderem das T.c. mlpt Homolog polished-rice und der Transkriptionsfaktor shavenbaby. Für diesen Transkriptionsfaktor konnte kürzlich gezeigt werden, dass er normalerweise als Repressor, nach proteolytischer Modifikation aber als Aktivator seiner Zielgene wirken kann. Der ”switch” zwischen Repressor und Aktivator scheint von Polished-rice-Peptiden vermittelt zu werden (Kondo et al., 2010). Da die Funktion von Mlpt-Peptiden in Tribolium bisher noch völlig 112 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse unverstanden ist, untersuchte ich, ob das in Drosophila gezeigte Zusammenspiel zwischen Polished-rice und Shavenbaby auch in Tribolium zu finden ist. Dazu wurde ein Fragment des shavenbaby-Homologs in Tribolium kloniert und zunächst die Expression mittels in-situHybridisierung näher charakterisiert. Svb ist bereits früh in der Embryogenese, kurz nach der Ausbildung der Serosagrenze in Tribolium exprimiert (Abb. 35: A’). Es bildet eine kappenförmige Expressionsdomäne am posterioren Eipol (Abb. 35: A). Diese Expressionsdomäne bleibt während der posterioren Invagination des Embryos bestehen und ist auch im frühen Keimrudiment zu beobachten (Abb. 35: B), wobei die Expression in einem transversalen Streifen erhöht wird. Während der Keimstreifstreckung dehnt sich die posteriore Domäne zunächst aus (Abb. 35: C) und zieht sich schließlich auf eine definierte Region in der posterioren Wachstumszone zurück (Abb. 35: D, E), wo sie zum Abschluss der Segmentierung einen schmalen Streifen im Telsonbereich ausbildet (Abb. 35: F). Zu diesem dynamischen posterioren Expressionsmuster bildet sich anterior, im Bereich der Kopflappen, ein neuronales Muster aus (Abb. 35: E, F, G). Dieses setzt sich im Zuge der Keimstreifverkürzung (Abb. 35: E) in den thorakalen und abdominalen Segmenten in einer Pünktchen förmigen Expression fort. Zusätzlich ist auch noch eine deutliche Expression von svb in den distalen Enden der zukünftigen Mundwerkzeuge und der Beine zu sehen (Abb. 35: F, Sternchen). Um die posteriore Expressionsdomäne genauer charakterisieren zu können, wurde eine Doppelfärbung mit svb und dem Segmentpolaritätsgen gooseberry (gsb) durchgeführt. Die Ergebnisse dieser Doppelfärbung sind in Abb. 36 zusammengestellt. Während der posterioren Invagination ist svb hinter dem letzten gsb-Streifen exprimiert (Abb. 36: A, A’): anschließend kommt es zu einer minimalen Überschneidung zwischen dem dritten gsb-Streifen und der posterioren svb-Domäne (Abb. 36: B, C). Die svb-Expression ist zu diesem Zeitpunkt bereits unmittelbar vor dem jüngsten gsb-Streifen zu detektieren, zieht sich über selbigen hinweg und erstreckt sich bis zum Hinterende des Keimrudiments. Im weiteren Verlauf der Segmentierung gibt es dann aber keinen Überlapp mehr zwischen den gsb-Streifen und der svb-Expression, da diese sich auf den hinteren Teil der Wachstumszone beschränkt (Abb. 36: D, E). 113 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 35: Die Expression von svb in der Embryogenese von Tribolium Die anteriore Seite der Embryonen zeigt stets nach links. A) Die früheste Detektion von svb ist im differenzierten Blastoderm möglich (A’, Pfeil markiert Serosa - Embryogrenze). Svb wird dann am posterioren Ende des Eies exprimiert, im Bereich der zukünftigen Wachstumszone, B) wo das Gen auch im Keimrudiment aktiv bleibt. C) Im Zuge der fortschreitenden Keimstreifstreckung (D, E) zieht sich die svb-Expression schließlich auf einen schmalen posterioren Streifen zurück (F). Diese posteriore Expression in der Wachstumszone wird in älteren Stadien von einer neuronalen Expression in den Kopflappen (E, F, G, Pfeilmarkierung) begleitet; während des Auswachsens der Mundwerkzeuge und der Beine ist svb zudem an den distalen Enden dieser Appendizes nachweisbar (F, Sternchen). 114 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 36: Doppel-ISH von T.c. gooseberry und T.c. svb Dargestellt ist eine Doppelfärbung der Expressionen von gooseberry (lila) und shavenbaby (orange). Während der Keimstreifinvagination ist svb hinter dem zuletzt gebildeten gsb-Streifen exprimiert (A, A’). B) Im Keimrudiment und im jungen Keimstreif (C) ist die svb-Expression unmittelbar vor dem letzten gsb-Streifen zu beobachten und zieht sich vom letzten gsb-Streifen bis über den gesamten posterioren Bereich des Embryos hin. D) Mit fortschreitender Entwicklung erscheint zwischen dem jüngsten gsb-Streifen und der svb-Expression eine kleine Lücke. Nach dem Abschluss der Segmentierung und der Ausprägung aller gsb-Streifen verschwindet die posteriore svb-Domäne (E). Vergleich zwischen svb- und mlpt-Expression in Tribolium Da die Frage beantwortet werden soll, ob mlpt und shavenbaby während der Embryonalentwicklung in Tribolium interagieren können, sollen die bereits diskutierten Färbungen von mlpt bzw. svb nochmals im direkten Vergleich gezeigt werden (Abb. 37). Es ist zu erkennen, dass mlpt und svb vom differenzierten Blastoderm beginnend bis hin zum gestreckten Keimstreif in der posterioren Wachstumszone koexprimiert sind (Abb. 37: A-D’). 115 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 37: Vergleich der T.c. mlpt- und T.c. svb-Expressionsdomänen 116 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Die anteriore Seite der Embryonen zeigt nach links. A und A’ sind laterale Ansichten, ansonsten wird die ventrale Seite der herauspräparierten Embryonen gezeigt. Die schwarzen Balken deuten an, dass beide Gene in der gleichen Region exprimiert werden, die roten Balken signalisieren, dass es für eine mlpt-Expressionsdomäne keine entsprechende svb-Expression gibt. Im Blastoderm (A, A’) ist am posterioren Pol eine Überschneidung zwischen mlpt und svb zu beobachten. Im Keimrudiment (B, B’) ist die posteriore Expression beider Gene fast deckungsgleich. Das gleiche gilt während der Keimstreifstreckung (C, C’); dagegen hat die anteriore mlpt-Expression im mandibulären Segment keine svb-Entsprechung. Auch in späteren Stadien bleiben beide Gene in der Wachstumszone exprimiert (D, D’). Die breite zentrale mlpt-Expressionsdomäne (D) hat jedoch keine svb-Entsprechung. Auch die punktförmigen Expressionen in den Kopflappen ähneln sich (Abb. 37: D-E), ebenso wie die Muster, die später in den Extremitäten und in bestimmten (neuronalen?) Zellen die in allen Segmenten auftreten (Abb. 37: E, E’). Es fällt aber auch auf, dass mlpt in einigen Bereichen während der Embryogenese von Tribolium exprimiert wird, die keine svb Entsprechung haben. Schon früh in der Entwicklung (Abb. 37: A) hat die breite anteriore mlpt-Expressionsdomäne die sich von der Serosagrenze bis zur Mitte des Keimrudiments zieht, kein svb-Pendant (Abb. 37: A’). Während der Streckung des Keimrudiments bildet lediglich mlpt eine mandibuläre Expressionsdomäne aus, die in svb gefärbten Embryonen ebenfalls nicht zu finden ist (Abb. 37: B-C’). Zeitgleich mit dem Erscheinen der neuralen Kopfexpression, ist in mlpt gefärbten Embryonen eine breite zentrale Expression zu finden, die in keinem Stadium der svb gefärbten Embryonen vorhanden ist. Zusammengefasst kann man sagen, dass nur im posterioren Bereich, sowie evtl. in bestimmten neuronalen Vorläuferzellen eine Überschneidung der Genexpressionen von mlpt und svb evident ist. Die Funktion von T.c. svb während der Embryogenese Der nun folgende Abschnitt widmet sich der Frage, welche Funktion shavenbaby in der Tribolium-Embryonalentwicklung hat. Die initialen pupalen Injektionen führten zu einer hohen Letalität der Puppen. Fast 80% starben nach der Injektion. Dass es sich dabei um einen genspezifischen Effekt handelt, konnte mit der Kontrolle (Injektion von Wasser) gezeigt werden, bei der alle injizierten Tiere überlebten. Ein technisches Problem mit der Injektion ist somit wohl auszuschließen. Interessanterweise liegt die Überlebensrate von mit mlpt-dsRNA injizierten Puppen ebenfalls bei knapp 20%. Um einen möglichen Einfluss von svb-RNAi auf die Metamorphose zu umgehen, war für die weitere Analyse der Genfunktion adulte RNAi 117 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse das Mittel der Wahl, also Injektion von dsRNA in adulte Weibchen. Injiziert wurde svbdsRNA in unterschiedlichen Konzentrationen (1 µg/µl - 4 µg/µl). Allerdings war dabei kein Unterschied in der Stärke der resultierenden, larvalen Phänotypen zu finden, so dass für die weiteren Experimente stets eine Konzentration von 2 µg/µl verwendet wurde. Die phänotypischen Auswirkungen adulter svb-RNAi sind auf Abbildung 38 zusammengestellt. Dabei variierten die erhaltenen Phänotypen nur darin, wie viele abdominale Segmente in thorakale Segmente transformiert bzw. wie viele abdominale Segmente noch ausgebildet wurden. Im Vergleich zum WT (Abb. 38: A) bilden schwächere svb-RNAi Phänotypen (Kategorie I) vier ”thorakale” Segmente mit Beinpaaren aus (Abb. 38: B). Alle Beine wirken gedrungener als das Wildtypbein und die Klaue ist verbreitert. An die Segmente mit thorakalem Charakter schließen sich drei beinlose abdominale Segmente an. Die terminalen Strukturen fehlen ganz. Bemerkenswert ist ebenfalls, dass kein Rückenschluss stattfindet und die Larven dorsal offen sind. Ein etwas stärkerer Phänotyp (Kategorie II) (Abb. 38: C) weist fünf Beinpaare auf, wobei nicht alle zusätzlichen Beine voll ausgebildet sind. Oft sind sie stark verkürzt. Diese Segmente haben auch Tracheenöffnungen, was bei thorakalen Segmenten des Wildtyps nur in T2 der Fall ist. An die transformierten Segmente schließen sich noch zwei beinlose abdominale Segmente an. Auch hier findet kein Rückenschluss statt. Die Beine sind im Vergleich zum Wildtyp deutlich verkürzt und erscheinen dicker, mit z.T. undeutlich ausgebildeten Gelenken (Abb. 38: D-F). In solchen svb-RNAi-Embryonen sind zwar alle Beinregionen Coxa, Trochanter, Femur, Tibiotarsus und Prätarsus ausgebildet, jedoch von der Gesamtheit her verkürzt und mit Falten, die den distalen Podomergrenzen/Gelenken ähneln. Die folgende Tabelle spiegelt die Verteilung der Phänotypen in knapp 200 ausgezählten svb-RNAi-Larven wider. Am häufigsten (zu fast 63%) sind die stärkeren Phänotypen der Kategorie II, während schwächer Phänotypen nur zu knapp 22% ausgebildet werden. Tab. 12: Verteilung der svb-RNAi Phänotypen (c=2 µg/µl). Phänotyp WT I II Kutikularest Larven (%) 27 (14%) 43 (22%) 121 (63%) 1 (0.5%) 118 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 38: svb-RNAi-Phänotypen nach parentaler RNAi Die Wildtyp Larve von Tribolium (A), mit drei thorakalen Segmenten, acht abdominalen Segmenten und den terminalen Urogomphi. svb-RNAi-Phänotypen der Kategorie I (B) sind deutlich kürzer als die Wildtyp-Larve. Die drei thorakalen Segmente werden von einem transformierten Segment gefolgt (T4), das ebenfalls ein Beinpaar trägt. Schließlich werden noch drei abdominale Segmente ausgebildet (Kennzeichnung der Tracheenöffnungen mit Sternchen). Bei diesen Larven findet kein Rückenschluss statt. Alle ausgebildeten Beine wirken im Vergleich zum Wildtyp verdickt. In stärkeren Phänotypen der Kategorie II sind zwei abdominale Segmente partiell zu thorakaler Identität transformiert (C). Den drei thorakalen Segmenten folgen zwei Segmente, die zwar Tracheenöffnungen tragen (Kennzeichnung: Sternchen) - und damit potentiell abdominalen Charakter aufweisen, jedoch ist zumindest entweder ein Beinstummel präsent, oder aber auch ein ganzes Bein ausgebildet. Die Beine wirken dicker und krummer als im Wildtyp. Auch in diesen Larven findet kein Rückenschluss statt. Schematische Zeichnung (Quelle: Johannes Scholten, Diplomarbeit) eines WT-Käferbeins (D), bestehend aus Coxa, Trochanter, Femur, Tibiotarsus und Prätarsus. Ein WT-Käferbein ist in (E) und ein svb-RNAi Käferbein in (F) vergrößert dargestellt. Alle Beinregionen können identifiziert werden, jedoch ist das Bein insgesamt kürzer und stämmiger als im Wildtyp. Die Analyse von Markergenen nach parentaler svb-RNAi Savard et al., 2006 konnten zeigen, dass sich mlpt-RNAi auf die Expression von Gapgenen wie hunchback, Krüppel oder giant auswirkt. So wird beispielsweise die posteriore hunchback-Domäne verzögert ausgebildet und die posteriore Krüppel-Expressionsgrenze wird nicht definiert, so dass die Krüppel-Domäne in mlpt-RNAi Embryonen mehr Segmente umfasst als in der Wildtyp-Situation. Für giant stellte sich heraus, dass zwar die anteriore 119 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Kopfdomäne normal exprimiert wird, die posteriore Domäne im Keimstreif jedoch in allen untersuchten Stadien fehlt (Savard et al., 2006). Mlpt-Peptide können also sowohl aktivierenden als auch reprimierenden Einfluss auf die Expression von Gapgenen haben. Gesetzt den Fall, dass Mlpt-Peptide den Transkriptionsfaktor Shavenbaby in Tribolium aktivieren wäre zu erwarten, dass nach svb-RNAi ähnliche Verschiebungen der Gapgenmuster auftreten, wie das für mlpt gezeigt worden ist. Deswegen wurden Markergene wie hunchback, Krüppel und giant nach svb-RNAi näher analysiert, ebenso wie das Paarregelgen even-skipped und das schon oben erwähnte Segmentpolaritätsgen gooseberry. Trotz sorgfältiger Analysen konnte allerdings keine Veränderung der frühen Expressionsdomänen von hunchback, Krüppel und even-skipped gefunden werden. Interessanterweise wurde jedoch ein Effekt auf die giant-Expression in svb-RNAi Embryonen gefunden. Im Wildtyp wird giant sowohl in einem anterioren Kopfmuster, als auch in einer posterioren Domäne exprimiert, die sich im Keimrudiment in zwei Streifen aufspaltet (Abb. 39: A-C), von denen der posteriore etwas länger persistiert (Abb. 39: E-G). Die anteriore Kopfdomäne hingegen ist bis in den gestreckten Keimstreif hindurch präsent und wird in einem neuronalen Muster exprimiert (Bucher und Klingler, 2004). In svb-RNAi Embryonen konnte früh in der Entwicklung, im Blastoderm und im Keimrudiment, keine veränderte giant-Expression gefunden werden. Sowohl die Kopfdomäne als auch die abdominale Expressionsdomäne wurde in allen untersuchten Embryonen korrekt angelegt. Erst während der Keimstreifstreckung fiel auf, dass die posteriore Domäne vorzeitig verschwindet (Abb. 39: D) bzw. nur noch eine leichte, rudimentäre Expression am posterioren Ende vorzufinden ist (Abb. 39: F, Pfeil). Die anteriore Kopfdomäne war in allen untersuchten svb-RNAi Stadien hindurch wie im Wildtyp exprimiert. Kernfärbungen im gestreckten Keimstreif zeigen deutlich die zusätzlich angelegten Beinpaare (Abb. 40: A-B’). Mit Hilfe des Segmentpolaritätsgens gooseberry kann der Segmentierungsdefekt in älteren Keimstreifstadien sichtbar gemacht werden. Im Vergleich zum Wildtyp, der zehn abdominale gsb-Streifen zeigt (Abb. 40: C), sind in svb-RNAi Embryonen ein Segmentierungsdefekt je nach Phänotypstärke ab dem dritten abdominalen Segment zu beobachten. Zwar wird noch eine größere Zahl von gsb-Streifen angelegt, allerdings in einem ungeordneten Muster. Auch die Streckung des Keimstreifes ist beeinträchtigt, bzw. weist Gewebsverdickungen und Verkrümmungen auf (Abb. 40: D, E). Diese Ergebnisse sind konsistent mit den oben gezeigten Kutikula-Phänotypen, bei denen die posterioren Segmente stark beeinträchtigt sind. 120 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 39: Tribolium giant-Expression im Wildtyp und nach svb-RNAi Die Embryonen sind in allen Bildern mit anterior nach links orientiert. A) Im differenzierten Blastoderm wird giant im gesamten Keimrudiment und in der posterioren Einfaltung exprimiert. Im Keimrudiment B) hebt sich deutlich die anteriore Expressionsdomäne in den Kopflappen ab und am posterioren Ende ist ein breite Expressionsdomäne sichtbar, der ein etwas kräftigerer Expressionsstreifen voran geht. Während der Keimstreifstreckung C) bleibt die Expression in den Kopflappen bestehen und die posteriore Expression bildet zwei schmale, deutlich voneinander abgegrenzte Streifen. Während die anteriore Kopfdomäne wie im Wildtyp gebildet wird, fehlt in svb-RNAi Embryonen die posteriore Expressionsdomäne (D). Im Zuge der Keimstreifstreckung (E) wird in Wildtyp Embryonen die posteriore giant-Domäne immer schmaler und ist unmittelbar vor der Wachstumszone lokalisiert. F) zeigt einen svb-RNAi Embryo, der etwas älter ist als der Wildtyp in E), und bei dem die posteriore giant-Domäne nur noch rudimentär ausgeprägt ist (Pfeil). 121 III. Kapitel: mlpt und svb in Tribolium - Ergebnisse Abb. 40: Kernfärbung und gsb-Färbung in svb-RNAi Embryonen Die Kernfärbung eines gestreckten Wildtyp-Embryos zeigt die Morphologie eines normalen Keimstreifes (A, A’); im Vergleich dazu erscheint ein svb-RNAi Embryo im vergleichbaren Stadium deutlich kürzer und gedrungener (B, B’). Auffallend ist die gestörte abdominale Segmentierung am posterioren Ende, die auch in einer Verdickung und Verkürzung des Keimstreifs im Abdomen resultiert (B). Der vergrößerte Ausschnitt des thorakalen Bereiches (B’) zeigt die zusätzlichen Beinanlagenpaare (T4 + T5), die in diesem Stadium wie die Beinknospen des Wildtyps aussehen. Einen vollständig gestreckten Keimstreif, bei dem alle Segmentanlagen mit dem Segmentpolaritätsgen gsb gefärbt sind, zeigt Bild C. Es sind zehn abdominale Segmentanlagen sichtbar (später fusionieren A9 und A10 mit den terminalen Strukturen, so dass die Tribolium-Larve nur über acht abdominale Segmente verfügt). In svb-RNAi Embryonen ist die gsb-Expression bis in den anterioren Bereich des Abdomens regelmäßig. Ab dem zweiten (D, Pfeilkopf) oder dritten abdominalen Segment (E) wird gsb zwar weiterhin exprimiert, aber die Streifen wirken versetzt und ungleichmäßig. Jedoch scheint sich gegen Ende der Keimstreifstreckung wieder ein regelmäßiges Muster zu entwickeln (E, Pfeil). 122 III. Kapitel: mlpt und svb in Tribolium - Diskussion Diskussion Die ektopische Expression von T.c. mlpt hat keine phänotypischen Auswirkungen In einer früheren Arbeit (Savard et al., 2006) konnte gezeigt werden, dass mlpt eine Gapgenähnliche Funktion in Tribolium ausübt. Ein Knockdown von mlpt via parentaler RNAi resultiert in der Transformation abdominaler Segmente hin zu thorakalen Segmenten, in einer Deletion abdominaler Segmente, sowie in Störungen beim Rückenschluss und einer nicht näher beschriebenen morphologischen Verkürzung der Beine. Im Rahmen dieses Projekts sollte herausgefunden werden, wie sich eine Überexpression von mlpt auf die Embryogenese in Tribolium auswirkt. Durch Keimbahntransformation wurde ein zusätzliches mlpt-Gen ins Tribolium-Genom eingebracht, das von einem Hitzeschockpromotor reguliert wird (Tabelle 9). Dadurch ist es möglich, die Transkription dieses Gens durch Temperaturerhöhung in spezifischen embryonalen Stadien zu aktivieren. Von Überexpressionsexperimenten mit dem Gapgen hunchback ist bekannt, dass Hitzeschock-induzierte ektopische Expression einen Phänotyp auslöst, der dem Phänotyp von Knockdown Experimenten genau entgegengesetzt ist: während beim hunchback-Knockdown thorakale zu abdominalen Segmenten transformiert - und zusätzlich abdominale Segmente deletiert werden (Marques-Souza et al., 2008; Cerny, 2007), erhalten nach einer hb-Überexpression abdominale Segmente thorakale Identität und es werden zusätzlich abdominale Segmente gebildet (Distler und Klingler, in Vorbereitung). Entsprechende Effekte waren auch für die Überexpression von mlpt in Tribolium erwartet worden: da ein Verlust von mlpt-Aktivität die Transformation von abdominalen Segmenten zu thorakaler Identität und auch den Verlust von abdominalen Segmenten bewirkt, hätte die ektopische Expression von mlpt eine Transformation von thorakalen Segmenten zu abdominalem Schicksal bewirken sollen und zudem wäre vielleicht auch hier mit der Entstehung zusätzlicher abdominaler Segmente zu rechnen gewesen. Auch eine Veränderung der Beinmorphologie hätte man erwarten können. Allerdings konnte in allen allen sieben hsp68-mlpt Hitzeschocklinien kein mlpt-spezifischer Effekt auf die larvalen Kutikulastrukturen ausgemacht werden. Bis auf leichte Defekte, die vom Hitzeschock selbst resultierten (und auch in den Kontrollen auftreten), hatten die Larven Wildtyp-Morphologie (Abb.: 34). Dabei erscheint unwahrscheinlich, dass nicht das richtige Zeitfenster erwischt worden ist, da meist 123 III. Kapitel: mlpt und svb in Tribolium - Diskussion 0-16h Ablagen bzw. 0-24h Ablagen hitzebehandelt wurden (weil die Stämme noch nicht genügend expandiert waren, wären bei kürzeren Zeiträumen nicht genügend Eier vorhanden gewesen). Da in-situ-Hybridisierung nach dem Hitzeschock zeigte, dass in der Tat mlpt überexprimiert worden ist, ist klar, dass das Konstrukt im Prinzip funktioniert (Abb.: 33 A’-D’). Trotzdem können technische Probleme an diesem Punkt natürlich nicht hundertprozentig ausgeschlossen werden. Vielleicht war die Stärke der Expression doch zu gering, oder vielleicht konnten die Mlpt-Peptide aus irgendwelchen Gründen nicht effizient translatiert werden. Wahrscheinlicher ist allerdings, dass die mlpt-Überexpression in gar keinem Phänotyp resultiert. Im Laufe dieses Projekts konnte eine andere Arbeitsgruppe (Kondo et al., 2010) zeigen, dass mlpt in Drosophila seine Funktion indirekt über den Transkriptionsfaktor shavenbaby ausübt. Die Präsenz von Mlpt bewirkt, dass Shavenbaby von einem Transkriptionsinhibitor in einen Transkriptionsaktivator umgewandelt wird und so seine Zielgene aktivieren kann. Gesetzt den Fall, dass die Funktion von Mlpt ebenfalls an ein Effektorprotein gekoppelt ist, wäre in der Tat zu erwarten, dass die alleinige Überexpression von Mlpt keine phänotypische Veränderung bewirkt. Nur die gleichzeitige Überexpression von Effektor und Mlpt zusammen würde dann einen zum RNAi-Knockdown komplementären Phänotyp produzieren. mlpt-Überexpression mildert nicht den RNAi-Phänotyp Eine weitere Idee die Funktionalität des hsp86-mlpt-Konstrukts zu testen, bestand darin, parentale RNAi in einer mlpt- Hitzeschocklinie durchzuführen und die Eier einer Ablage (0-8h oder 8-16h) einem Hitzeschock zu unterziehen. Anschließend wurde die Phänotypenstärke nach der Beendigung der Embryonalentwicklung mit der nicht Hitze-behandelten Kontrolle verglichen (Tabelle 11). Die Erwartung war, dass aufgrund eines höheren Levels an mlptTranskript nach dem Hitzeschock in diesen Larven, eine Abschwächung des Phänotyps im Vergleich zu den Kontrollembryonen einher geht. Die Stärke des RNAi-Effekts ist nämlich unter anderem davon abhängig, wie hoch die injizierten Konzentrationen an dsRNA sind (Bucher und Klingler, 2004), d.h. der Knockdown ist normalerweise nicht vollständig. Es stellte sich in diesen Experimenten allerdings heraus, dass nur marginale Unterschiede in der Beinpaaranzahl oder in der Segmentanzahl zwischen den beiden Versuchsansätzen bestand, die wohl nicht signifikant waren. Dieses negative Ergebnis muss allerdings aus verschiede124 III. Kapitel: mlpt und svb in Tribolium - Diskussion nen Gründen noch als vorläufig betrachtet werden: zum einen waren die Eiablagen relativ lange Intervalle - falls die Phänotyp-Rettung nur in einem sehr engen Zeitfenster möglich sein sollte, würde ein evtl. signifikanter Unterschied in der Phänotypstärke unter Umständen in der Masse nicht optimal behandelter Embryonen untergehen. Zweitens geht aus der in-situ-Hybridisierung von gehitzeschockten mlpt-Embryonen hervor, dass die endogene mlpt-Expression deutlich stärker ist, als die mlpt-Expression, die von dem Hitzeschock herrührt (Abb.: 33 A’-D’). Daher ist gut möglich, dass das Level des nach RNAi verbliebenen Transkripts in den endogenen mlpt Expressionsdomänen nach Hitzeschock nicht signifikant ansteigt, so dass auch keine deutliche Abschwächung des mlptRNAi-Phänotyps erzielt werden kann. Ein weiterer Ansatz der diese technischen Probleme umgehen würde, könnte darin bestehen, dass RNAi gegen die Sequenzen des endogenen mlpt-Gens gerichtet würde, die im hsp68-mlpt-Konstrukt nicht vorhanden sind. So könnte man endogene 5’-UTR oder 3’-UTR Sequenzen von mlpt in einem hsp68-mlpt-Konstrukt mit anderen 5’-UTRs bzw. 3’-UTRs ersetzen. RNAi gegen endogene UTR-Sequenzen würde dann das Transgen nicht beeinträchtigen, da es in Tribolium offenbar kein ”spreading” des RNAi-Effektes auf benachbarte Transkriptbereiche gibt (Arakane et al., 2005). Da sich der RNAi-Effekt damit nur auf endogene mlpt-mRNA auswirken würde, sollte die mit Hilfe eines Hitzeschocks überexprimierte mlpt-mRNA mit veränderter UTR den Phänotyp aufheben bzw. deutlich abmildern. Da hierfür aber weitere transgene Konstrukte notwendig gewesen wären, für die im Rahmen dieser Arbeit keine Zeit verblieb, konnte dieser Ansatz noch nicht verfolgt werden. Der nächste Abschnitt wird sich nun mit der Frage beschäftigen, ob Shavenbaby auch in Tribolium der Effektor der mlpt-Funktion ist. Die Expressionsmuster von T.c. mlpt und T.c. svb unterscheiden sich In Drosophila spielt die posttranslationale Modifikation von Shavenbaby sowohl für die Bildung der ventralen Dentikel (Kondo et al., 2010), als auch für die Ausbildung der tarsalen Gelenke eine zentrale Rolle (Pueyo und Couso, 2011). Diese proteolytische Verkürzung des N-Terminus, die durch Pri/Tal-Peptide vermittelt wird, schaltet die Funktion von Svb von Transkriptionsinhibior zu Transkriptionsaktivator. Allerdings scheint es auch noch andere Interaktionspartner von Pri/Tal Peptiden zugeben, die bisher noch nicht bekannt sind. Beispielsweise sind Pri/Tal-Peptide in Drosophila für die 125 III. Kapitel: mlpt und svb in Tribolium - Diskussion Entstehung des Tracheennetzwerks erforderlich, obwohl Shavenbaby hier nicht exprimiert ist (Kondo et al., 2010). Dennoch war shavenbaby ein interessanter Kandidat für ein Zielgen der Mlpt-Peptide und wurde deshalb kloniert. svb wird in Tribolium bereits ab dem differenzierten Blastoderm in einer posterioren Domäne exprimiert (Abb. 35: A, A’). Eine Doppelfärbung mit dem Segmentpolaritätsgen gooseberry zeigte, dass die Expressionsdomäne zunächst mit dem jeweils jüngsten segmentalen Streifen überlappt und sich von dort bis zur posterioren Spitze der Wachstumszone erstreckt (Abb. 36). Ab der Entstehung des zweiten abdominalen Segments ist die Expressionsdomäne dann ausschließlich auf die Wachstumszone beschränkt (Abb. 35: B-E). Im gestreckten Keimstreif kommt es zu einer schmalen Expressionsdomäne unmittelbar vor der Wachstumszone (Abb. 35: F). Aufgrund dieses dynamischen Expressionsmusters kann man bereits vermuten, dass auch svb in der Embryogenese von Tribolium eine wichtige Rolle spielt. Aber ist die Funktion von Shavenbaby tatsächlich an Mlpt gekoppelt? Eine Voraussetzung dafür wäre, dass Svb und Mlpt koexprimiert sind, oder zumindest in unmittelbarer Nachbarschaft (falls die Mlpt-Peptide eine gewisse Distanz diffundieren können). Der Vergleich der Expressionsmuster der beiden Gene zeigt, dass sie in der Tat weitgehend überlappen, was eine Interaktion wie in Drosophila beschrieben, nahe legt (Abb. 37). Das gilt für die posterioren, blastodermalen Domänen (Abb. 37: A, A’) und auch die Domänen in der Wachstumszone überschneiden sich (Abb. 37: B-D’). Allerdings haben weder die anteriore, blastodermale noch die breite zentrale mlpt-Domäne im Keimstreif, eine Entsprechung in der svb-Expression (Abb. 37: B, C, D). Zwar ist von klonalen Analysen aus Drosophila bekannt, dass Mlpt nicht-zellautonom agieren kann, d.h. die Mlpt-Peptide können über mehrere Zellen hinweg diffundieren (Kondo et al., 2007), allerdings ist der Abstand der anterioren mlpt-Expressionsdomäne hin zur posterioren svb-Expression vermutlich zu groß, als dass hier von einer Interaktion zwischen diesen beiden Domänen ausgegangen werden kann. Allerdings hat die anteriore mlpt-Domäne laut Savard et al., 2006 auch gar keine Funktion, da mlpt-RNAi Larven anterior normal differenzieren. Eine interessante Frage ist allerdings, inwieweit die zentrale Domäne in sich streckenden Keimstreifen funktionell relevant ist. Hier muss man aber berücksichtigen, dass in dieser Abbildung mRNAs verglichen werden - es könnte durchaus sein, dass in den Zellen, die hier noch mlpt aber nicht mehr svb exprimieren, durchaus noch Svb-Protein vorhanden ist. Alternativ wäre auch denkbar, dass diese 126 III. Kapitel: mlpt und svb in Tribolium - Diskussion späte mlpt-Expression gar keine Funktion innehat. Anhand dieser Expressionsdaten kann man also schließen, dass svb durchaus ein Interaktionspartner von Mlpt-Peptiden sein könnte, während Hinweise auf zusätzliche Interaktionspartner wie für Drosophila vermutet nicht erkennbar sind. svb-RNAi resultiert in einem ähnlichen Gapgen-Phänotyp wie mlpt-RNAi Injektion von svb-dsRNA in Tribolium-Puppen führt zu einer sehr hohen Letalität, sodass für die Ermittlung der svb-Funktion adulte RNAi verwendet wurde. Eine ähnlich hohe Letalität injizierter Puppen konnte bei mlpt beobachtet werden. Dagegen war die Überlebensrate von injizierten adulten Käfern bei beiden Genen nahezu 100%. Der RNAi-Effekt von svb in Tribolium resultiert in der Transformation abdominaler Segmente hin zu Segmenten mit thorakaler Identität, sowie in einer starken Verkürzung der Larven aufgrund der Deletion von abdominalen Segmenten (Abb. 38: B, C). Weitere phänotypische Veränderungen konnten in der Beinstruktur beobachtet werden. Zwar sind alle Beinsegmente vorhanden, allerdings ist die Form der Klauen verändert, da sie im Vergleich zum Wildtyp verkürzt sind (Abb. 38: E, F). Die übrigen, mehr proximal gelegenen Beinsegmente wirken gestaucht und die Kutikula erscheint zerknittert. Schließlich ist noch auf den fehlerhaften Rückenschluss hinzuweisen, der in allen svb-RNAi-Larven zu finden ist. Diese Defekte nach T.c. svb-RNAi ähneln damit weitgehend den von Savard et al., 2006 beobachten Phänotypen nach mlpt-RNAi. Allerdings gibt es deutlich quantitative Unterschiede zwischen svb-RNAi und mlpt-RNAi Phänotypen, da die Anzahl der transformierten Segmente in mlpt-RNAi-Embryonen deutlich höher lag als in den svb-RNAi Tieren. Diese zeigten maximal die Transformation der ersten beiden abdominalen Segmenten hin zu thorakaler Identität, während bei mlpt-RNAi Larven nahezu alle abdominalen Segmente zu thorakaler Morphologie umgewandelt werden können. Als stärkster Phänotyp wurden mlptLarven beschrieben, die zehn Beinpaare ausbildeten (Savard et al., 2006). Dies impliziert, dass diese Larven mindestens sieben der normalerweise zehn abdominalen Segmenten ausbilden; in svb-RNAi-Larven konnten maximal noch vier abdominale Segmente ausgemacht werden. Eine mögliche Interpretation dieser phänotypischen Unterschiede ist, dass der svb-RNAiEffekt quantitativ stärker ist, und dass daher aufgrund der drastisch verkürzten Larven weniger transformierbare abdominale Segmente verbleiben als dies in mlpt-Larven der Fall ist 127 III. Kapitel: mlpt und svb in Tribolium - Diskussion - auch wenn der Transformationsphänotyp in mlpt-RNAi Larven auf den ersten Blick spektakulärer wirkt als der bloße Verlust von Segmenten. Die These, dass der svb-RNAi Effekt einfach quantitativ stärker ausfällt als der mlpt-RNAi Effekt wird noch untermauert durch die Beobachtung, dass alle svb-RNAi Larven keinen Rückenschluss machen, während dies bei mlpt-RNAi Larven zwar erwähnt wurde (Savard et al., 2006), in meinen eigenen Kontrollversuchen aber nur hin und wieder beobachtet werden konnte. Auch die Beinmorphologie scheint in svb-RNAi-Larven stärker betroffen zu sein als in mlpt-RNAi Larven, bzw. eine veränderte Beinstruktur nicht bei allen mlpt-RNAi-Larven beobachtet werden konnte. Falls diese Interpretation stimmt, könnte der stärkste Phänotyp bei svb ein RNAi-Phänomen sein, d.h.vielleicht ist der Knockdown von svb mittels RNAi einfach effizienter. Alternativ ist aber auch denkbar, dass der Knockdown des Effektors Svb aus biochemischen Gründen einen drastischeren Effekt hat, als ein Knockdown seines Aktivators Mlpt. So wäre etwa denkbar, dass Mlpt-Peptide eine Protease aktivieren, die dann wiederum Svb aktiviert. Wenn nun diese Protease eine gewisse Basis-Aktivität hätte, könnte dies erklären, weshalb selbst eine vollständige mlpt-Inaktivierung nicht zu einer kompletten Svb-Inaktivierung führen würde, der mlpt-Phänotyp also zwangsläufig schwächer wäre. Eine andere Möglichkeit wäre auch, dass die Phänotypen von mlpt und svb sich nicht nur quantitativ sondern auch qualitativ unterscheiden; dies wäre etwa zu erwarten, falls mlpt noch andere Zielpeptide außer Svb regulieren würde. In diesem Fall würde man allerdings eher erwarten, dass der svb-RNAi-Phänotyp schwächer wäre, was unwahrscheinlich erscheint. Eine weitere Möglichkeit ist ernster zu nehmen: es könnte sein, dass Svb nicht nur aktivierende Funktionen bei der Segmentierung ausübt (Funktion des aktivierten Peptids), sondern zusätzlich auch als Inhibitor fungieren kann. Diese Funktion wäre dann mlpt-unabhängig, was den stärkeren Phänotyp von svb-RNAi-Larven gut erklären könnte. Diese Option muss daher bei der zukünftigen Analyse dieser beiden Gene im Auge behalten werden. Der Einfluss von Svb auf die Regulation anderer Segmentierungsgene Falls Svb tatsächlich ein Zielprotein der Mlpt-Aktivität ist, müssten die von Savard beobachteten Effekte auf die Gapgene Krüppel, hunchback und giant nach mlpt-RNAi (Savard et al., 2006) in ähnlicher Form auch in svb-RNAi-Embryonen gefunden werden. Um einen direkten Vergleich zu haben, wurden mlpt-RNAi- und svb-RNAi-Embryonen parallel gefärbt und untersucht. Frühe Effekte auf die Gapgenexpression im Blastoderm wurden für 128 III. Kapitel: mlpt und svb in Tribolium - Diskussion mlpt-RNAi-Embryonen nicht beschrieben und auch in svb-RNAi-Embryonen erschienen die blastodermalen Gapgen-Expressionsmuster wie im Wildtyp. Diese Befunde sind deckungsgleich mit den Kutikula-Phänotypen, welche zeigen, dass Kopf- und thorakalen Strukturen keinen RNAi-Effekt zeigen. Die blastodermale Gapgenexpression wird deswegen im Folgenden nicht weiter diskutiert, vielmehr liegt das Augenmerk auf der Expression der Gapgene im Zuge der Keimstreifstreckung. Krüppel wird im Wildtyp-Keimstreif in einer zentralen Domäne exprimiert, die sich vom labialen Segment bis hin zum dritten thorakalen Segment erstreckt (Cerny et al., 2005). Diese breite Domäne expandiert in mlpt-RNAi-Embryonen nach posterior bis in die Wachstumszone hinein (Savard et al., 2006). Eine entsprechende Expansion von Krüppel in svbRNAi-Embryonen habe ich jedoch nicht beobachtet. Shavenbaby hat also offenbar keinen regulatorischen Einfluss Krüppel. Im Fall von hunchback erstreckt sich im Wildtyp-Keimrudiment eine Expressionsdomäne von den Kopflappen bis hin zum dritten thorakalen Segment. Während der Keimstreifstreckung entsteht eine kräftige Expressionsdomäne in der posterioren Wachstumszone (Wolff et al., 1995). Für mlpt-RNAi Embryonen wurde beschrieben, dass die Expressionsdomäne in der posterioren Wachstumszone zwar vorhanden ist, aber zeitlich verzögert exprimiert wird (Savard et al., 2006). Ein ähnlicher Effekt konnte in svb-RNAi-Embryonen von mir nicht beobachtet werden. Die hunchback-Expression in der posterioren Wachstumszone scheint zeitlich richtig exprimiert zu sein. T.c. giant wird in Wildtyp-Keimstreifembryonen sowohl in einer anterioren, als auch in zwei posterioren Domänen exprimiert. Die anteriore Domäne erstreckt sich von den Kopflappen bis zum maxillären Segment; der erste Streifen der posterioren Domäne umfasst das dritte thorakale Segment und der zweite, das zweiten abdominalen Segment (Bucher und Klingler, 2004). Für mlpt-RNAi Embryonen war bekannt, dass sich die anteriore Kopfdomäne normal ausbildet, die posterioren Domänen hingegen fehlten. Dasselbe konnte ich für svbRNAi-Embryonen beobachten (Abb.: 39). Die anteriore Domäne in den Kopflappen wurde korrekt exprimiert, während die posterioren Streifen entweder nur rudimentär angelegt waren oder vollständig fehlten (Abb. 39: D, G). Bei der Weiterführung dieses Projekts sollten die soeben diskutierten Versuche wiederholt werden, damit ausgeschlossen werden kann, dass evtl. die für die in-situ-Färbungen verwendeten svb-RNAi-Phänotypen schwächer waren, als die auf Kutikula-Ebene untersuchten. Zusammenfassend sind die Ergebnisse bisher, 129 III. Kapitel: mlpt und svb in Tribolium - Diskussion dass Svb auf die Regulation von hunchback und Krüppel keinen Einfluss zu nehmen scheint, hingegen für die posteriore Aktivierung der giant-Domäne benötigt wird. Der Einfluss von Svb auf das Segmentpolaritätsgen gooseberry Die Segmentierungsdefekte in der Kutikula von svb-RNAi-Larven werden schon in Keimstreif-Embryonen im Expressionsmuster von gooseberry abgebildet. Ab dem zweiten abdominalen Segment bilden die entstehenden Streifen ein ungeordnetes Muster (Abb. 40: D, E). Das Fehlen der posterioren giant-Domäne könnte diese Muster-Störungen evtl. erklären. Dies könnte ein weiterer Hinweis darauf sein, dass Gapgene in Tribolium eher eine permissive Funktion in der Wachstumszone ausüben, als eine instruktive (Marquez-Souza et al., 2008; Choe et al., 2005). Ein ähnliches Resultat wurde auch nach giant-RNAi in Tribolium beschrieben (Bucher und Klingler, 2004). Als segmentaler Marker diente hier engrailed (Brown et al. 1994) und Segmentierungsstörungen wurden vom ersten thorakalen bis zum letzten abdominalen Segment beobachtet - wobei allerdings die thorakalen Entwicklungsstörungen im Zuge der Keimstreifstreckung kompensiert zu werden scheinen, so dass ältere Embryonen nur noch im posterioren Bereich ein unregelmäßiges engrailed-Muster ausbilden (Bucher und Klingler, 2004) - ähnlich wie das von mir auch für gooseberry in svb-RNAi Embryonen gefunden wurde. Shavenbaby - ein neues Gapgen in Tribolium? Die Analyse der Gapgene nach svb-RNAi ergab, dass svb zumindest einen Einfluss auf ein Gapgen hat, giant. Gemäß der Definition für Gapgene in Tribolium, kann Shavenbaby damit als neues Gapgen bezeichnet werden: der RNAi-Effekt beinhaltet anteriore Transformationen und posteriore Deletionen und zudem ist eine Interaktion mit einem anderen Gapgen gezeigt worden. Nimmt man als Kritierium für Gapgene noch die molekulare Funktion als Transkriptionsfaktor dazu, läuft Shavenbaby Mlpt gewissermaßen den Rang ab, so dass vielleicht eher svb das eigentliche neue Gapgen ist und nicht mlpt wie von Savard et al., postuliert. Deswegen kann das in der Einleitung bereits vorgestellte Gapgenmodell in Tribolium weiter modifiziert werden, indem svb hinzugefügt wird (Abb. 41). Allerdings konnte im Rahmen dieser Dissertation noch nicht geklärt werden, ob Svb auch Paarregelgene reguliert (das ist allerdings sehr wahrscheinlich). Vor allem aber bleibt die Fra- 130 III. Kapitel: mlpt und svb in Tribolium - Diskussion ge noch unbeantwortet, ob die Aktivierung des Transkriptionsfaktors Svb durch kleine und vermutlich durch den Zellverband diffundierende Peptide ein für Kurzkeim-Embryonen essentieller molekularer Mechanismus ist, oder eher eine Tribolium-spezifische Besonderheit. Um diese Fragen zu klären, müssen nicht nur einige der hier begonnenen Experimente wiederholt und verfeinert werden, es werden wahrscheinlich auch neue methodische Ansätze, etwa räumlich regulierte Missexpression, zur Anwendung gebracht werden müssen. Damit könnte z.B. die Diffusionsreichweite der Mlpt-Peptide im Embryo gemessen werden, und es könnte die Wirkung von Svb bzw. Mlpt auf jeden einzelnen abdominalen Paarregelstreifen untersucht werden, ohne die Entwicklung insgesamt, v.a. die Keimstreifstreckung selbst, zu beeinträchtigen. Abb. 41: Gapgenexpression in Tribolium Gezeigt ist der Segmentanlagenplan in Tribolium nach der Gastrulation. Für die Funktion von mlpt ist svb nötig. Die eingezeichneten Expressionsdomänen spiegeln nur die örtliche Expression, nicht aber die volle zeitliche Dynamik wider. So treten die posterioren Expressionsdomänen von svb in Wirklichkeit erst auf, wenn die anteriore Domäne bereits wieder verschwunden ist (nach Cerny et al., 2008; Savard et al., 2006). 131 III. Kapitel: mlpt und svb in Tribolium - Diskussion Ausblick: Die Funktion von mlpt in Bruchidius obtectus Für Drosophila konnte gezeigt werden, dass in Nullmutanten von tarsal-less ein Transgen, das Bombyx mori tarsal-less exprimiert, die Mutation kompensieren kann, sodass die mutanten Drosophila-Larven wie Wildtyp aussehen (Galindo et al., 2007). Dies ist darauf zurückzuführen, dass das Kernmotiv der mlpt ORFs in allen untersuchten Arten sehr gut konserviert ist. Interessant wäre es nun zu wissen, welche Funktion mlpt in Bruchidius hat. Es wird angenommen, dass die Funktion von mlpt in der Aktivierung von Shavenbaby bei Bildung der Trichome in Drosophila eine Dipteren-spezifische Rekrutierung darstellt, und dass die ursprüngliche Funktion von mlpt in der Steuerung der Beinentwicklung lag (Pueyo und Couso, 2011). Bruchidius obtectus (Coleoptera) wäre ein geeignetes Modell, denn diese Larven haben im Gegensatz zu Tribolium ventrale Dentikel, welche möglicherweise ebenfalls durch svb/mlpt-System gebildet werden könnten. Vor allem aber wäre es interessant zu sehen ob dieses System wie in Tribolium auch in die Segmentierung von Bruchidius involviert ist, also eine Konservierung zwischen den beiden Käferarten Bruchidius und Tribolium darstellt oder ob es womöglich doch eine spezielle Adaption in der Wachstumszone des Kurzkeimkäfers Tribolium ist. In einem Quick- and Dirty Versuch wurde Tribolium mlpt-dsRNA in Bruchidius injiziert und geschaut, ob ein Effekt auftritt. Es konnte beobachtet werden, dass tatsächlich ein sehr milder Effekt auftritt: Die Dentikel sind weiterhin vorhanden, allerdings konnte eine Fusion der Dentikelfelder auf der abdominalen, ventralen Seite beobachtet werden. Es scheint also, dass mlpt auch in die Segmentierung des Langkeimkäfers Bruchidius involviert ist. Inwieweit dabei das svb/mlpt-System konserviert ist und ob es ebenfalls einen Einfluss auf die Gapgene von Bruchidius hat, wäre ein weiterer Forschungsaspekt für die Zukunft. 132 Anhang Anhang A: Puffer und Lösungen Alle Chemikalien stammen sofern nicht anders gekennzeichnet von der Firma Carl Roth GmbH aus Karlsruhe. B.o. Detergenzlösung für 20 ml: 1% SDS, 0.5% Tween, 50mM Tris-HCl (pH=7.5), 1mM EDTA (pH=8), 150mM NaCl, mit H2 O auf Endvolumen auffüllen. Färbepuffer 1 (ISH mit NBT/BCIP) für 1 ml: 500 µl TrisHCl (1M, pH= 9.5), 250 µl MgCl2 (1M), 20 µl NaCl (5M), 5 µl Tween (20%), mit H2 O auf Endvolumen auffüllen. Färbepuffer 2 (ISH mit INT/BCIP) für 1 ml: 100 µl TrisHCL (1M, pH=9.5), 50 µl MgCl2 (1M), 20 µl NaCl, 5 µl Tween (20%), mit H2 O auf Endvolumen auffüllen. Hyb-A (Bruchidius) für 20 ml: 10 ml Formamid, 5 ml 20xSSC (pH=5.5 mit HCl), 400 µl Lachshoden DNA (10 mg/ml), 100 µl tRNA (20mg/ml), 20 µl Heparin (50 mg/ml), mit H2 O auf Endvolumen auffüllen. Hyb-B (Bruchidius) für 20 ml: 10 ml Formamid (Calbiochem), 5 ml 20xSSC (pH=5.5), mit H2 O auf Endvolumen auffüllen. B.o. Hyb-Ovar für 50 ml: 25 ml Formamid, 12.5 ml SSC (20x), 1 g SDS, 1 g BBR, 100 µl tRNA (20 mg/ml), 20 µl Heparin (50 mg/ml), 250 mg Chaps, 500 µl Tween (20%), 500 µl EDTA (0.5 M, pH=7.5), mit H2 O auf Endvolumen auffüllen. 133 Anhang Ovar-Hyb(Wash) 50% Formamid, 2 x SSC, 1% SDS 5x MABT für 1l: 58 g Maleinsäure, 32 g NaOH Pellets, 43.8 g NaCl pH= 7.5, auf 1l mit H2 O auffüllen. PBS (10x) für 1l: 80 g NaCl, 2g KCl, 2 g KH2 PO4 , 11.5 g Na2 HPO4 , mit H2 O auf 1 l auffüllen. PEMS 0.1 M Pipes, 1 mM MgSO4 , 1 mM EDTA, pH=6.9. Anhang B: Sequenzen B.o. paired Proteinsequenz: 1 QYKRENPSIF SWEIRDRLVM EGICDRSTAP SVSAISRLLR GKGGDCDGDD 51 KSSENEGGSD CESEPGIPLK RKQRRSRTTF TAHQLDELEK AFERTQYPDI 101 YTR DNA-Sequenz: 1 CAGTATAAGA GGGAGAACCC GTCAATTTTC AGTTGGGAGA TTAGGGACCG 51 ATTGGTGATG GAAGGCATCT GTGACAGGAG TACAGCCCCT AGCGTATCCG 101 CTATCTCTAG GTTGCTTAGA GGCAAAGGTG GCGACTGCGA CGGAGACGAT 151 AAATCCAGTG AAAACGAAGG AGGGTCTGAC TGCGAAAGTG AGCCGGGTAT 201 CCCGTTGAAG AGAAAGCAAC GACGATCCCG AACTACCTTC ACCGCTCACC 251 AGTTAGACGA GCTTGAAAAA GCATTCGAAC GTACTCAGTA TCCTGACATA 301 TACACCCGCG A 134 Anhang B.o. hunchback Proteinsequenz: 1 KHHLEYHLRN HTGSKPFSCT QCSYSCVNKS MLNSHLKSHS NIYQYRCSDC 51 SYATKYCHSL KLHLRKYGHK PAMVLNPDGT PNPLPIIDVY GTRRGPKLKS 101 QEQRPVEEQR SPQPEQVIPF PLNQFMLNAQ SQLPFAAFPF FAGFPGPLAN 151 TLLMQNLEKI ARERQSSVLN REEPQPVEKQ PQDEVLDLSK PEEPSQNRRV 201 PQQDDSSGDE EEDDTHTTMF SNVEMVENKE AKAKEEEMTN NNKEDFNCQF 251 CKISFGDLVL YTMHMGYHGY KDPFTCNMC DNA-Sequenz: 1 CCGCACATGT TGCAAGTGAA TGGATCTTTG TAGCCGTGGT ACCCCATGTG 51 CATGGTGTAC AGCACGAGAT CACCAAAGCT AATTTTGCAG AACTGACAGT 101 TGAAATCTTC TTTATTGTTA TTCGTCATCT CCTCTTCTTT GGCTTTGGCT 151 TCTTTGTTTT CAACCATTTC CACATTGCTG AACATGGTTG TATGGGTATC 201 ATCTTCTTCT TCATCTCCTG AAGAGTCATC TTGTTGGGGC ACCCTTCTGT 251 TCTGGGACGG CTCTTCAGGT TTGCTGAGGT CTAGTACCTC GTCTTGGGGT 301 TGTTTCTCGA CTGGCTGTGG TTCTTCTCTA TTTAAAACTG AACTCTGCCT 351 TTCCCTTGCA ATTTTCTCCA GATTTTGCAT GAGCAAAGTG TTTGCTAGTG 401 GGCCAGGGAA TCCAGCAAAG AAGGGGAACG CGGCGAATGG TAGCTGGGAT 451 TGAGCATTCA ACATAAACTG ATTCAATGGG AACGGTATTA CTTGCTCGGG 501 TTGCGGACTT CTTTGCTCCT CTACTGGCCT TTGCTCTTGG CTCTTCAGTT 551 TTGGACCTCG CCTGGTACCA TAGACGTCTA TTATAGGTAA CGGATTTGGT 601 GTACCGTCTG GATTGAGCAC CATCGCTGGC TTGTGGCCGT ACTTGCGCAG 651 ATGAAGCTTC AACGAATGGC AGTACTTAGT GGCGTAACTA CAATCTGAGC 701 ATCTGTATTG GTATATGTTG GAGTGCGATT TCAGGTGGGA GTTCAACATA 751 GATTTGTTCA CACAAGAGTA ACTGCATTGG GTGCAACTGA ACGGTTTCGA 801 ACCGGTGTGG TTCCTCAGGT GATACTCCAG GTGATGCTT 135 Anhang B.o. zen Proteinsequenz: 1 EKEFHTNKYL CRPRRIQMAQ DLNLTERQIK IWFQNRRK DNA-Sequenz: 1 CGAGAAGGAG TTCCACACTA ACAAGTACCT GTGCCGACCA AGACGTATCC 51 AAATGGCTCA AGACTTGAAT CTGACGGAAC GTCAGATCAA AATCTGGTTC 101 CAAAACCGTC GCAAG B.o. caudal Proteinsequenz: 1 QRQPNPGKTR TKDKYRVVYT DHQRIELEKE FTFNNKYITI RRKSELATNL 51 GLSERQIKIW FQNSRK DNA-Sequenz: 1 CAAAGGCAGC CGAACCCTGG CAAGACCCGC ACCAAAGACA AGTACCGCGT 51 GGTATACACT GATCACCAAC GCATCGAACT GGAGAAAGAG TTCACCTTTA 101 ACAACAAATA CATCACGATT CGTCGCAAGT CGGAGCTGGC GACTAACCTA 151 GGACTCTCCG AACGCCAAAT CAAAATTTGG TTCCAGAACA GCCGCAAG B.o. mex-3 Proteinsequenz: 1 QNMTECVPVP SSEHVAEIVG RQGCKIKALR AKTNTYIKTP VRGEEPVFVV 51 TGRKEDVAKA KREILSAAEH FSQIRASRKN NLAGLGSGAS TPPGPPANIP 101 GHVTIQVRVP YRVVGLVVGP KGATIKRIQH QTHTYIVTPS RDKEPVFEVT 151 GLPESVEAAR REIEAHIAMR TGNAAGGLNG LPPLDETDLI ASLYKSGFSS 201 ILNYMEPTGG QASVDTFPAM TSSTGSSGAF SSSGSCSSSS SSNGRDLGAI 251 WSGSDRDEGL GDSPTLFDTS AALSSIWTYP ASAGGANPAN AASVSAPTRP 301 SPSSSTSPAD SLLSSATKCL VCSDAKVTHA LVPCGHNFFC MECAN 136 Anhang DNA-Sequenz: 1 CAGAACATGA CTGAGTGTGT GCCAGTGCCC AGTTCCGAGC ATGTGGCCGA 51 AATCGTCGGC AGACAAGGTT GCAAAATCAA GGCCCTGCGA GCGAAGACAA 101 ACACCTACAT CAAAACGCCA GTACGTGGAG AAGAACCAGT GTTCGTAGTG 151 ACAGGCCGCA AAGAGGATGT AGCCAAAGCG AAGAGGGAAA TCCTTTCCGC 201 GGCAGAACAC TTCTCACAGA TCAGAGCATC GCGCAAAAAT AATCTCGCAG 251 GTCTCGGTTC AGGCGCCAGC ACACCTCCAG GACCGCCCGC AAACATCCCC 301 GGTCACGTAA CCATTCAGGT TCGAGTCCCG TATCGCGTAG TAGGCCTGGT 351 AGTGGGACCC AAGGGTGCCA CGATCAAGAG GATCCAGCAC CAGACGCACA 401 CGTACATCGT CACACCTAGC AGGGACAAGG AACCAGTTTT CGAGGTGACC 451 GGACTCCCGG AGAGCGTTGA AGCAGCTCGC AGAGAGATCG AAGCCCACAT 501 CGCGATGCGT ACCGGCAACG CTGCCGGAGG ACTGAACGGC CTGCCCCCTT 551 TGGACGAAAC CGACTTGATC GCTTCTCTGT ACAAGTCGGG TTTTAGCTCA 601 ATCCTGAACT ACATGGAGCC GACGGGCGGT CAGGCCTCAG TGGACACCTT 651 TCCCGCCATG ACCTCGTCGA CGGGAAGCAG CGGCGCTTTT TCTAGCTCAG 701 GATCGTGCTC CAGCAGCTCC AGCAGTAACG GAAGAGATCT GGGCGCCATC 751 TGGAGCGGAA GCGACAGGGA TGAGGGACTT GGAGACTCGC CAACGCTATT 801 CGACACATCT GCCGCTTTGA GCAGCATCTG GACGTACCCC GCGTCCGCGG 851 GCGGTGCCAA CCCTGCCAAC GCGGCCTCAG TCTCCGCCCC AACGCGCCCC 901 TCCCCTTCCA GCAGCACCTC CCCTGCTGAT TCGTTGCTCA GCTCGGCCAC 951 CAAATGTCTG GTGTGCTCCG ATGCCAAGGT CACCCACGCA CTGGTGCCCT 1001 GCGGCCATAA CTTTTTTTGC ATGGAATGCG CCAACAG 137 Anhang O.f. mex-3 Proteinsequenz: 1 EHVAEIVGRQ GCKIKSLRAK TNTYIKTPVR GEEPVFVVTG RKEDVAKAKK 51 EILSAADHFS QIRASRKNNL GGCGGLSPPG PPTNVPGNVT IQVRVPYRVV 101 GLVVGPKGAT IKRIQHQTHT YIVTPSRDKE PVFDVTGLPE HVEAARRDIE 151 AHIALRTGAG ALPGLIGSSG TTGIMDESHE IIASLCKSGL GSLFSSNSWL 201 DSLPSQTDTN MGFSSAFSAG SSGCSSASSS SSNSGRTGLG DLGSIWSMSL 251 DRDEGLGDSP SFETANASSS IWSFPTVTRP SPSSSASPTE SLSGAKRECV 301 VCCEREVTAA DNA-Sequenz: 1 GAGCACGTTG CTGAGATCGT TGGACGCCAG GGATGCAAGA TTAAATCTCT 51 GAGAGCAAAA ACCAACACCT ACATCAAGAC ACCAGTCAGA GGAGAAGAGC 101 CTGTATTTGT GGTCACTGGC AGAAAGGAGG ATGTGGCCAA GGCCAAGAAA 151 GAGATTTTGT CAGCTGCTGA TCATTTTTCT CAGATTAGAG CTTCAAGGAA 201 AAATAATCTT GGCGGATGCG GTGGATTGTC ACCACCTGGG CCTCCCACAA 251 ATGTCCCAGG CAATGTCACT ATTCAAGTCC GAGTACCTTA TCGTGTGGTT 301 GGCCTTGTTG TTGGACCTAA AGGTGCTACT ATCAAAAGAA TTCAACACCA 351 GACTCATACA TACATTGTCA CACCCTCAAG AGATAAGGAA CCTGTGTTTG 401 ATGTTACCGG TCTTCCGGAA CATGTTGAAG CTGCTCGTCG TGATATTGAG 451 GCTCATATCG CATTAAGAAC AGGTGCTGGT GCTCTTCCAG GACTTATTGG 501 TTCAAGTGGT ACAACTGGCA TCATGGATGA AAGTCATGAG ATAATTGCTT 551 CACTCTGCAA GAGTGGACTT GGAAGTCTTT TTTCTTCTAA CTCTTGGCTG 601 GATTCTCTTC CATCGCAGAC TGACACTAAT ATGGGATTCT CTTCTGCATT 651 TAGTGCTGGG AGTTCCGGTT GCAGCTCAGC ATCATCGAGC TCCTCCAATT 701 CAGGCCGAAC CGGGCTTGGA GACTTGGGAT CCATCTGGAG TATGTCTTTG 751 GACAGAGATG AAGGCCTAGG TGACTCTCCT TCTTTCGAAA CAGCTAACGC 801 CTCTTCAAGC ATCTGGTCAT TTCCTACTGT GACCAGGCCA TCTCCATCCA 851 GTTCTGCTTC TCCTACAGAA TCCCTTAGTG GTGCCAAGAG AGAATGCGTT 901 GTTTGCTGTG AAAGAGAAGT CACGGCTGCT C 138 Anhang O.f. caudal Proteinsequenz: 1 HLEQPVHRVP QSRSPYEWMK KPSYQSHPTP GKTRTKDKYR VVYTDHQRLE 51 LEKEFHYSKY ITIRRKSELA DNA-Sequenz: 1 ACCATCTGGA ACAGCCCGTG CATAGAGTGC CGCAGAGTAG GAGTCCTTAC 51 GAGTGGATGA AGAAACCCAG TTACCAGTCA CACCCGACTC CAGGAAAAAC 101 TAGAACTAAA GACAAATATC GTGTTGTGTA TACAGATCAT CAGAGACTGG 151 AGTTAGAGAA AGAGTTCCAC TACAGTAAAT ACATAACAAT AAGGCGAAAA 201 TCGGAGCTCG CCTC T.c. shavenbaby Proteinsequenz: 1 YAPIVPPDKV TLPVSKKPEN TPKPQNLAPV RQFSVIQRTP KTTKRDEDKD 51 ARLPTAPLVI QEPEQEQPID YHIPKRRGET EDENEEKRIR EQRRSHCSKV 101 SKPLISVRAI VGKLPVTMSA AAGHGRGSST GQNSGSQSTN SQSG DNA-Sequenz: 1 AATACGCCCC TATCGTACCC CCGGACAAAG TCACTCTTCC TGTGTCAAAG 51 AAACCGGAAA ATACCCCCAA ACCACAAAAC CTAGCCCCCG TACGACAATT 101 CTCCGTTATT CAACGAACCC CAAAAACGAC AAAACGCGAC GAAGACAAAG 151 ATGCGCGGCT TCCGACAGCC CCTTTGGTCA TACAAGAGCC GGAACAGGAA 201 CAGCCGATAG ATTATCACAT TCCGAAACGG CGGGGAGAGA CGGAAGATGA 251 AAATGAAGAG AAGAGGATTC GGGAGCAGAG ACGGAGTCAC TGTTCCAAAG 301 TGAGCAAACC GTTAATTTCA GTCCGGGCGA TTGTTGGGAA GCTTCCGGTG 351 ACTATGTCGG CGGCGGCGGG GCACGGGAGG GGCTCCTCCA CGGGGCAAAA 401 CTCCGGGAGC CAGTCGACAA ATTCTCAATC GGGT 139 Anhang Anhang C: Vektorkarten Abb. 42: pSLfa[Tc’hsp5’3’UTR]fa u. pBac[3xP3-DsRedaf] A) Der Vektor pSLfa [Tc’hsp5’3’UTR]fa trägt die 5’ und 3’ Hitzeschockpromotoren (grün markiert) sowie die Restriktionsschnittstellen XbaI und XhoI. B) Der Transformationsvektor pBac[3xP3-DsRedaf] mit dem Transformationsmarker dsRed sowie dazugehörigem, augenspezifischen 3xP3-Promotor und den Erkennungssequenzen für die Transposase: pBacL und pBacR. 140 Anhang Abb. 43: pBac[3xP3-DsRedTc’hsp5’-mlpt-hsp3’] Vektorkarte des mlpt-Überexpressionsvektors. Die schwarzen Boxen markieren die für das Transformationsereignis relevanten piggyBac L(eft) bzw. piggyBac R(right)-Bereiche. Die Lage des fluoreszierenden Transformationsmarkers dsRed wird durch die rote Box symbolisiert, der dazu gehörige 3xP3-Promotor durch das hellrote Dreieck. Neben dem SV-40 Promotor beinhaltet der Vektor noch die Hitzeschockpromotoren hsp5’ und hsp3’ welche durch die gelben Boxen dargestellt werden, dazwischen findet sich schließlich das T.c. mlpt-Gen, das überexprimiert werden soll. 141 Literaturverzeichnis Akam, M., 1987. The molecular basis for metameric pattern in the Drosophila embryo. Development 101: 1-22. Andrews, J., Garcia-Estefania, D., Delon, I., Lü, J., Mével-Ninio, M., Spierer, A., Payre, F., Pauli, D. and Oliver, B., 2000. Ovo transcription factors function antagonistically in the Drosophila female germline. Development 127: 881-892. Angelini, D. R. and Kaufman, T.C., 2005. Functional analyses in the milkweed bug Oncopeltus fasciatus (Hemiptera) support a role for Wnt signaling in body segmentation but not appendage development. Developmental Biology, Vol. 283, Issue 2: 409-423. Arkane, Y., Muthukrishnan, S. Beeman R. W., Kanost, M. R. and Kramer, K. J., 2005. Laccase 2 is the phenoloxidase gene required for beetle cuticle tanning. PNAs, 102 (32): 1133711342. Bastock, R. and St Johnston, D., 2008. Drosophila oogenesis. Current Biology, Vol. 18, No 23: R1082-R1087. Bäumer, D., Trauner, J., Hollfelder, D., Cerny, A. and Schoppmeier, M., 2011. JAK-STAT signalling is required throughout telotrophic oogenesis and short-germ embryogenesis of the beetle Tribolium. Developmental Biology 350: 169-182. Beermann, A., Jay, D. G., Beeman, R. W., Hülskamp, M., Tautz, D. and Jürgens, G., 2001. The short antennae gene of Tribolium is required for limb development and encodes the orthologue of the Drosophila Distal-less protein. Development 128: 287-297. Beermann, A. and Schröder, R., 2008. Sites of Fgf signalling and perception during embryogenesis oft the beetle Tribolium castaneum. Dev Genes Evol 218: 153-167. Bender, M., Horikami, S., Cribbs, D. and Kaufman, T.C., 1988. Identification and Expression of the Gap segmentation gene hunchback in Drosophila melanogaster. Developmental Genetics 9: 715-732. Berghammer, A. J., Weber, M., Trauner, J. and Klingler, M., 2009. Red Flour Beetle (Tribolium): Germline Transformation and Insertional Mutagenesis. CSH Protocols; doi: 10.1101/pdb.prot5259. Berleth, T., Burri, M., Thoma, G., Bopp, D., Richstein, S., Frigerio, G., Noll, M. and NüssleinVolhard, C., 1988. The role of localization of bicoid-mRNA in organizing the anterior pattern of the Drosophila embryo. The EMBO Journal, Vol.7, No.6: 1749-1756. 142 Literaturverzeichnis Birkan, M., Schaeper, N. D. and Chipman, A. D., 2011. Early patterning and blastodermal fate map of the head in the milkweed bug Oncopeltus fasciatus. Evolution & Development, 13; 5: 436-447. Bopp, D., Burri, M., Baumgartner, S., Frigerio, G. and Noll, M., 1986. Conservation of a large protein domain in the segmentation gene paired and in functionally related genes of Drosophila. Cell, Vol. 47: 1033-1040. Bosch, T. C. G. and Fujisawa, T., 2001. Polyps, peptides and patterning. BioEssays 23: 420427. Brent, A. E., Yucel, G., Small, S. and Desplan, C., 2007. Permissive and instructive anterior patterning rely on mRNA localization in the wasp embryo. Science, Vol. 315: 1841-1843. Brönner, G. and Jäckle, H., 1991. Control and function of terminal gap gene activity in the posterior pole region of the Drosophila embryo. Mechanisms of Development, 35: 205211. Brown, S. J., Patel, N. H. and Denell, R. E., 1994. Embryonic Expression of the single Tribolium engrailed homolog. Developmental Genetics 15: 7-18. Brown, S. J., Parrish, J. K., Beeman R. W. and Denell, R. E., 1997. Molecular characterization and embryonic expression of the even-skipped ortholog of Tribolium castaneum. Mechanisms of Development: 165-173. Brown, S., Fellers, J., Shippy, T., Denell, R., Stauber, M. and Schmidt-Ott, U., 2001. A strategy for mapping bicoid on the phylogenetic tree. Current Biology, 11: 34-44. Bucher, G., Scholten, J. and Klingler, M., 2002. Parental RNAi in Tribolium (Coleoptera). Curr Biol 12: 85-86. Bucher, G. and Klingler, M., 2004. Divergent segmentation mechanism in the short germ insect Tribolium revealed by giant expression and function. Development 131: 1729-1740. Büning, J., 1978. Development of telotrophic-meroistic Ovarioles of polyphage beetles with special reference to the formation of nutritive cords. Journal of Morphology 156: 237-256. Büning, J., 1980. RNA-Synthesen in der Frühembryogenese von Bruchidius obtectus SAY. Habilitationsschrift. Burbach, J. P., 2011. What are neuropeptides? Methods Mol Biol. 789: 1-36. Butt, F. H., 1947. Embryology of the milkweed bug, Oncopeltus fasciatus (Hemiptera). Agriculture Experiment Station Memoir 283, Cornell University. Callaini, G., Dallai, R. and Riparbelli, M. G., 1990. Behaviour of yolk nuclei during early embryogenesis in Drosophila melanogaster. Bolletino di Zoologia, 57, 3: 215-220. Carroll, S. B. and H. Vavra, S. H., 1989. The zygotic control of Drosophila pair-rule gene expression. Development 107: 673-683. Cerny, A. C., Bucher, G., Schröder, R. and Klingler, M., 2005. Breakdown of abdominal patterning in the Tribolium krüppel mutant jaws. Development 132: 5353-5363. 143 Literaturverzeichnis Cerny, A. C., 2007. Regulation von Segmentierungs- und Hoxgenen durch Gap-Gene im Kurzkeim-Embryo von Tribolium castaneum. Dissertation, FAU. Cerny, A. C., Grossmann, D., Bucher, G. and Klingler, M., 2008. The Tribolium ortholog of knirps and knirps-related is crucial for head segmentation but plays a minor role during abdominal patterning. Developmental Biology 321: 284-294. Chanut-Delalande H., Fernandes, I., Roch, F., Payre, F. and Plaza, S., 2006. Shavenbaby couples patterning to Epidermal cell shape control. PLoS Biol 4(9): e290.DOI:10.1371/journal.pbio.0040290. Cho, P. F., Poulin, F., Cho-Park, Y. A., Cho-Park, I. B., Chicoine, J. D., Lasko, P. and Sonenberg N., 2005. A New Paradigm for Translational Control: Inhibition via 5´-3´ mRNA Tethering by Bicoid and the eIF4E Cognate 4EHP. Cell, Vol. 121: 411-423. Choe, C. P., Miller, S. C. and Brown, S. J., 2005. A pair-rule gene circuit defines segments sequentially in the short-germ insect Tribolium castaneum. PNAS, Vol. 103; No.17: 65606564. Copf, T., Schröder, R. and Averof, M., 2004. Ancestral role of caudal genes in axis elongation and segmentation. PNAS, Vol. 101; No. 51: 17711-17715. Couso, J. P., 2009. Segmentation, metamerism and the Cambrian explosion. Int. J. Dev. Biol. 53: 1305-1316. Davis, G. K. and Patel, N. H., 2002. Short, long, and beyond: molecular and embryological approaches to insect segmentation. Annu. Rev. Entomol. 47: 669-699. Delon, I., Chanut-Delalande, H. and Payre, F., 2003. The Ovo/Shavenbaby transcription factor specifies actin remodelling during epidermal differentiation in Drosophila. Mechanisms of Development, Volume 120, Issue 7: 747-758. Delon, I. and Payre, F., 2004. Evolution of larval morphology in flies: get in shape with shavenbaby. Trends in Genetics, Volume 20, Issue 7: 305-313. Draper, C. W, Mello, C. C, Bowerman, B., Hardin, J. and Priess, J. R., 1996. Mex-3 is a KH Domain Protein that regulates Blastomere identity in early C. elegans Embryos. Cell, Vol. 87, Issue 2: 205-216. Driever, W. and Nüsslein-Volhard, C., 1988a. A Gradient of Bicoid Protein in Drosophila Embryos. Cell, Vol. 54: 83-93. Driever, W. and Christiane Nüsslein-Volhard, C., 1988. The Bicoid protein determines position in the Drosophila embryo in a concentration-dependent manner. Cell 54(1): 95-104. Driever, W. and Nüsslein-Volhard C., 1989. The Bicoid protein is a positive regulator of hunchback transcription in the Drosophila embryo. Nature Vol.337: 138-143. Driever, W., Thoma, G. and Nüsslein-Volhard, C., 1989. Determination of spatial domains of zygotic gene expression in the Drosophila embryo by the affinity of binding sites for the Bicoid morphogen. Nature Vol. 340: 363-367. 144 Literaturverzeichnis Ephrussi, A., Dickinson, L. K. and Lehmann, R., 1991. Oskar organizes the germ plasm and directs localization of the posterior determinant nanos. Cell, Vol.66: 37-50. Ephrussi, A. and St Johnston, D., 2004. Seeing is believing: The Bicoid Morphogen Gradient matures. Cell, Vol. 116, 143-152. Evans, D. and Blumenthal, T., 2000. Trans-splicing of polycistronic Caenorhabditis elegans pre-mRNAs: Analysis of the SL2 RNA. Mol Cell Biol. 20 (18): 6659-6667. Falciani, F., Hausdorf, B., Schröder, R., Akam, M., Tautz, D., Denell, R. and Brown, S., 1996. Class 3 Hox genes in insects and the origin of zen. PNAS, Vol. 93: 8479-8484. Finkelstein, R. and Perrimon, N., 1990. The orthodenticle gene is regulated by bicoid and torso and specifies Drosophila head development. Nature Vol 346: 485-488. Frasch, M., Hoey, T., Rushlow, C., Doyle, H. and Levine, M., 1987. Characterization and localization of the Even-skipped protein of Drosophila. The EMBO Journal vol. 6, no. 3: 749-759. Frohnhöfer, H. G. and Nüsslein-Volhard, C., 1986. Organization of anterior pattern in the Drosophila embryo by the maternal gene bicoid. Nature Vol. 324: 120-125. Galindo, M. I., Pueyo, J. I., Fouix, S., Bishop, S. A., Couso, J. P., 2007. Peptides encoded by short ORFs control development and define a new eukaryotic gene family. PLoS Biol 5(5): e106. doi: 10.1371/journal.pbio.0050106 Garfinkel, M. D., Lohe, A. R. and Mahowald, A. P., 1992. Molecular genetics of the Drosophila melanogaster ovo locus, a gene required for sex determination of germline cells. Genetics, Vol. 130, 791-803. Garfinkel, M. D., Wang, J., Liang, Y. and Mahowald, A. P., 1994. Multiple products from the shavenbaby-ovo Gene Region of Drosophila melanogaster: Relationship to genetic complexity. Molecular and Cellular Biology, Vol. 14, No.10: 6809-6818. Gavis, E. R. and Lehmann, R., 1992. Localisation of nanos RNA controls embryonic polarity. Cell, Vol. 71: 301-313. Gonzales-Gaitan, M., Rothe, M., Wimmer, E. A., Taubert, H. and Jäckle, H., 1994. Redundant functions of the genes knirps and knirps-related for the establishment of anterior Drosophila head structures. Developmental Biology Vol. 91: 8567-8571. Georgopoulos, K., Bigby, M., Wang, J. H., Molnar, A., Wu, P., Winandy, S. and Sharpe, A., 1994. The ikaros gene is required for the development of all lymphoid lineages. Cell 79: 143-156. Gaul, U. and Herbert Jäckle, H., 1987. Pole region-dependent repression of the Drosophila gap gene krüppel by maternal gene products. Cell, Vol.51: 549-555. Gehring, W. J., 1987. Homeo Boxes in the study of Development. Science Vol., 236: 12451252. Gilbert, S., 2006. Developmental Biology. Palgrave Macmillan; Auflage: 8th rev. ed. (10. Mai 2006). 145 Literaturverzeichnis Gilbert, W. V,. 2010. Alternative Ways to think about cellular internal ribosome entry. The Journal of biological chemistry Vol. 285, No. 38: 29033-29038. Le Gouar, M., Lartillot, N., Adoutte, A. and Vervoort, M., 2003. The expression of a caudal homologue in a mollusc, Patella vulgata. Gene Expression Patterns 3 : 35-37. Gutjahr, T., Frei, E. and Noll, M., 1993. Complex regulation of early paired expression: initial activation by gap genes and pattern modulation by pair-rule genes. Development 117:609623. Hafen, E., Kuroiwa, A. and Gehring, W. J., 1984. Spatial distribution of transcripts from the segmentation gene fushi tarazu during Drosophila embryonic development. Cell, Vol. 37: 833-841. Harding, K. and Levine, M., 1988. The Gap genes define the limits of Antennapedia and Bithorax gene expression during early development in Drosophila. The EMBO Journal vol. 7 no.1 pp.205-214. Hartenstein, V., 1993. The interactive fly: Atlas of Drosophila Development. Cold Spring Harbor Laboratory Press. Herth, W. and Sander, K., 1973. Mode and Timing of Body Pattern Formation (Regionalization) in the early embryonic development of Cyclorrhaphic Dipterans (Protophormia, Drosophila). Wilhelm Roux’ Archiv 172, 1-27. van den Heuvel, M., Nusse, R., Johnston, P. and Lawrence, P. A., 1989. Distribution of the Wingless Gene Product in Drosophila embryos: A protein involved in Cell-Cell Communication. Cell, Vol. 59: 739-749. Horn, C. and Wimmer, E. A., 2000. A versatile vector set for animal transgenesis. Dev Genes Evol 210: 630-637 Hughes, C. L. and Kaufman, T. C., 2002. Hox genes and the evolution of the arthropod body plan. Evolution & Development, 4, No. 6: 459-499. Hughes, C. L., Liu, P. Z. and Kaufman, T. C., 2004. Expression patterns of the rogue Hox genes Hox3/zen and fushi tarazu in the apterygote insect Thermobia domestica. Evolution & Development, 6, No. 6: 393-401. Hülskamp, M., Pfeifle, C. and Tautz, D., 1990. A morphogenetic gradient of Hunchback protein organizes the expression oft the gap genes krüppel and knirps in the early Drosophila embryo. Nature 346: 577-580. Hülskamp, M. and Tautz, D., 1991. Gap genes and gradients - the logic behind the gaps. BioEssays Vol. 13, No.6: 261-268. Hülskamp, M., Lukowitz, W., Beermann, A., Glaser, G. and Tautz, D., 1994. Differential Regulation of Target Genes by Different Alleles of the Segmentation Gene hunchback in Drosophila. Genetics 138: 125-134. Ingham, P. W., 1988. The molecular genetics of embryonic pattern formation in Drosophila. Nature Vol. 335: 25-33. 146 Literaturverzeichnis Irish, V., Lehmann, R. and Akam, M., 1989. The Drosophila posterior-group gene nanos functions by repressing hunchback activity. Nature 338: 646-648. Jäckle, H., Tautz, D., Schuh, R., Seifert, E. and Lehmann, R., 1986. Cross regulatory interactions among the gap genes of Drosophila. Nature Vol. 324: 668-670. Jäckle, H., Gaul, U. and Redemann, N., 1988. Regulation and putative function of the Drosophila gap gene krüppel. Development 104, Supplement: 29-34. Jacob, F. and Monod, J., 1961. Genetic regulatory mechanisms in the synthesis of proteins. J.Mol.Biol 3: 318-356. Jäger, J., 2011. The gap gene network. Cell, Mol, Life Sci 68: 243-274. Janody, F., Reischl, J. and Dostatni, N., 2000. Persistence of Hunchback in the terminal region of the Drosophila blastoderm embryo impairs anterior development. Development 127: 1573-1582. Jun, S. and Desplan, C., 1996. Cooperative interactions between Paired domain and homeodomain. Development 122: 2639-2650. Jung, E., 1965. Entwicklungsgeschichtliche Ergebnisse zur Kennzeichnung des Eitypus. Z. Morph. Ökol. Tiere 56: 444-480. Kalinka, A. T., Varga, K. M., Gerrard, D. T., Preibisch, S., Corcoran, D. L., Jarrells, J., Ohler, U., Bergman, C. M. and Tomancak, P., 2010. Gene expression divergence recapitulates the developmental hourglass model. Nature 468: 811-814. O’Keefe, L., Dougan, S. T., Gabay, L., Raz, E., Shilo, B. Z. and DiNardo, S., 1997. spitz and wingless, emanating from distinct borders, cooperate to establish cell fate across the engrailed domain in the Drosophila epidermis. Development 124: 4837-4845 Kilchherr, F., Baumgartner, S., Bopp, D., Frei, E. and Noll, M., 1986. Isolation of the paired gene of Drosophila and its spatial expression during early embryogenesis.Nature Vol. 321: 493-499. Klingler, M., Erdélyi, M., Szabad, J. and Nüsslein-Volhard, C., 1988. Function of torso in determining the terminal anlagen of the Drosophila embryo. Nature Vol. 335: 275-277. Klingler, M., 2004. Tribolium. Curr. Biol. Vol 14, No. 17: R639-640. Knipple, D. C., Seifert, E., Rosenberg, U. B., Preiss, A. and Jäckle, H., 1985. Spatial and temporal patterns of krüppel gene expression in early Drosophila embryos. Nature Vol. 317: 40-44. Kondo, T., Hashimoto, Y., Kato, K., Inagaki, S., Hayashi, S. and Kageyama, Y., 2007. Small peptide regulators of actin-based cell morphogenesis encoded by a polycistronic mRNA. Nature Cell Biology, Vol. 9, No. 6: 660-665. Kondo, T., Plaza, S., Zanet, J., Benrabah, E., Valenti, P., Hashimoto, Y., Kobayashi, S., Payre, F. and Kageyama, Y., 2010. Small peptides switch the transcriptional activity of Shavenbaby during Drosophila embryogenesis. Science 329:336-339. 147 Literaturverzeichnis Kornberg, T., 1981. engrailed: A gene controlling compartment and segment formation in Drosophila. Proc. Natl. Acad. Sci USA 78: 1095-1099. Kotkamp, K., Klingler, M. and Schoppmeier, M., 2010. Apparent role of Tribolium orthodenticle in anteroposterior blastoderm patterning largely reflects novel functions in dorsoventral axis formation and cell survival. Development 137: 1853-1862. Kozak, M., 1999. Review: Initiation of translation in prokaryotes and eukaryotes. Gene 234: 187-208. Kozak, M., 2005. Regulation of translation via mRNA structure in prokaryotes and eukaryotes. Gene 361: 13-37. Kraft, R. and Jäckle, H., 1994. Drosophila mode of metamerization in the embryogenesis oft the lepidopteran insect Manduca sexta. PNAS, Vol. 91: 6634-6638. Lall, S. and Patel, N. H., 2001. Conservation and divergence in molecular mechanisms of axis formation. Annu. Rev. Genet. 35: 407-37. Lawrence, J. G., 2002. Shared strategies in gene organization among prokaryotes and eukaryotes. Cell, Vol. 110: 407-413. Lehmann, R. and Nüsslein-Volhard, C., 1987. hunchback, a gene required for segmentation of an anterior and posterior region of the Drosophila embryo. Developmental Biology 119: 402-417. Lemke, S., Stauber, M., Shaw, P. J, Rafiqi, A. M., Prell, A. and Schmidt-Ott, U. 2008. Bicoid occurrence and Bicoid-dependent hunchback regulation in lower Cyclorrhaphan flies. Evolution & Development Vol. 10, No. 4: 413-420. Lemke, S. and Schmidt-Ott, U., 2009. Evidence for a composite anterior determinant in the hover fly Episyrphus balteatus (Syrphidae), a cyclorrhaphan fly with an anterodorsal serosa anlage. Development 136, 507 doi: 10.1242/dev.033530. Lemke, S., Busch, S. E., Antonopoulos, D. A., Meyer, F., Domanus, M. H. and Schmidt-Ott, U., 2010. Maternal activation of gap genes in the hover fly Episyrphus. Development 137: 1709-1719. Lewis, E. B., 1978. A gene complex controlling segmentation in Drosophila. Nature Vol. 276: 565-570. Lipshitz, H. D., 2009. Follow the mRNA: a new model for Bicoid gradient formation. Nature Reviews, Molecular Cell Biology, Vol 10: 509-512. Liu, P. Z and Kaufman, T. C., 2003. hunchback is required for suppression of abdominal identity, and for proper germband growth and segmentation in the intermediate germband insect Oncopeltus fasciatus. Development 131: 1515-1527. Liu, P. Z and Kaufman, T. C., 2004. krüppel is a gap gene in the intermediate germband insect Oncopeltus fasciatus and is required for development of both blastoderm and germbandderived segments. Development 131: 4567-4579. 148 Literaturverzeichnis Liu, P. Z and Kaufman, T. C., 2005a. even-skipped is not a pair-rule gene but has segmental and gap-like functions in Oncopeltus fasciatus, an intermediate germband insect. Development 132: 2081-2092. Liu, P. Z and Kaufman, T. C., 2005b. Short and long germ segmentation: unanswered questions in the evolution of a developmental mode. Evolution & Development 7:6: 629-646. Lohnes, D., 2003. The Cdx1 homeodomain protein: an integrator of posterior signaling in the mouse. BioEssays 25: 971-980. Lorenzen, M. D., Brown, S. J., Denell, R. E. and Beeman, R. W., 2002. Cloning and Characterization of the Tribolium castaneum Eye-Color Genes encoding Tryptophan Oxygenase and Kynurenine 3-Monooxygenase. Genetics 160: 225-234 Lorenzen, M. D., Berghammer, A. J., Brown, S. J., Denell, R. E., Klingler, M. and Beeman, R. W., 2003. piggyBack - mediated germline transformation in the beetle Tribolium castaneum. Insect Molecular Biology 12 (5): 433-440. Lynch, J. and Desplan, D., 2003. Evolution of Development: Beyond Bicoid. Current Biology, Vol. 13: R557-R559. Lynch, J. A. and Desplan, D., 2010. Novel modes of localization and function of nanos in the wasp Nasonia. Development 137: 3813-3821. Lynch, J. A., Brent, A. E., Leaf, D. S., Pultz, M. A. and Desplan, C., 2006. Localized maternal orthodenticle patterns anterior and posterior in the long germ wasp Nasonia. Nature 439: 728-732. Lynch, J. A., El-Sherif, E. and Brown, S. J., 2012. Comparisons of the embryonic development of Drosophila, Nasonia and Tribolium. WIREs Dev. Biol., 1: 16-39. Mansilla, A., Lopez-Sanchez, C., de la Rosa, E. J., Garcia-Martinez, V., Martinez-Salas, E., de Pablo, F. and Hernandez-Sanchez, C., 2005. Developmental regulation of a proinsulin messenger RNA generated by intron retention. EMBO reports Vol.6, no 12: 1182-1187. Margolis, J. S, Borowsky, M. L., Steingrimsson, E., Shim, C. W., Lengyel, J. A. and Posakony, J. W., 1995. Posterior stripe expression of hunchback is driven from two promoters by a common enhancer element. Development 121: 3067-3077. Marom, K., Shapira, E. and Fainsod, A., 1997. The chicken caudal genes establish an anteriorposterior gradient by partially overlapping temporal and spatial patterns of expression. Mechanisms of Development 64: 41-52. Marques-Souza, H., Aranda, M. and Tautz, D., 2008. Delimiting the conserved features of hunchback function for the trunk organization of insects. Development 135: 881-888 doi: 10.1242/dev.018317. Matsubayashi, Y., 2011. Small Post-Translationally Modified Peptide Signals in Arabidopsis. The Arabidopsis Book: e0150. doi: 10.1199/tab.0150. Mazumdar, A. and Manjari Mazumdar, M., 2002. How one becomes many: blastoderm cellularization in Drosophila melanogaster. BioEssays, Vol.24, Issue 11: 1012-1022. 149 Literaturverzeichnis Mcdonald, P. M. and Struhl, G. 1986. A molecular gradient in early Drosophila embryos and its role in specifying the body pattern. Nature Vol. 324: 537-545. Merkel, T., 2010. Missexpressionssysteme in Tribolium castaneum. Masterarbeit, FAU, unveröffentlicht. Mével-Ninio, M., Terracol, R. and Kafatos, F. C., 1991. The ovo gene of Drosophila encodes a zinc finger protein required for female germ line development. EMBO J. 10: 2259-2266. Mito, T., Sarashina, I., Zhang, H., Iwahashi, A., Okamoto, H., Miyawaki, K., Shinmyo, Y., Ohuchi, H. and Noji, S., 2005. Non-canoncial functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development 132: 2069-2079. Mito, T. and Noji, S. 2006. Evolution of developmental systems underlying segmented body plans of bilaterian animals: insights from studies of segmentation in a cricket. Paleontological Research, Vol. 10, No.4: 337-344. Miyawaki, K., Mito, T., Sarashina, S., Zhang, H., Shinmyo, Y., Ohuchi, H. and Noji, S., 2004. Involvement of Wingless/Armadillo signaling in the posterior sequential segmentation in the cricket, Gryllus bimaculatus (Orthoptera), as revealed by RNAi analysis. Mechanisms of Development 121:119-130. Mlodzik, M., Fjose, A. and Gehring, W. J., 1985. Isolation of caudal, a Drosophila homeo boxcontaining gene with maternal expression, whose transcripts form a concentration gradient at the pre-blastoderm stage. The EMBO Journal Vol. 4 No.11: 2961-2969. Mlodzik, M. and J. Gehring, W. J., 1987. Expression of the caudal Gene in the germ line of Drosophila: Formation of an RNA and Protein Gradient during early embryogenesis. Cell, Vol. 46: 465-476. Mlodzik, M., Gibson, G. and Gehring, W. J., 1990. Effects of ectopic expression of caudal during Drosophila development. Development 109: 271-277. Mohler, J., Eldon, E. D. and Pirrotta, V., 1989. A novel spatial transcription pattern associated with the segmentation gene, giant, of Drosophila. The EMBO Journal vol. 8 no.5: 15391548. Mullighan, C. G., Miller, C. B., Radtke, I., Phillips, L. A., Dalton, J., Ma, J., White, D., Hughes, T. P., Le Beau, M. M., Pui, C., Relling, M. V., Shurtleff, S. A. and Downing, J. R., 2008. BCR-ABL1 lymphoblastic leukaemia is characterized by the deletion of Ikaros. Nature Vol. 453: 110-115. Nagy, L. M. and Carrol, S., 1994. Conservation of wingless patterning functions in the shortgerm embryos of Tribolium castaneum. Nature 367: 460-463. Nauber, U., Pankratz, M. J., Kienlin, A., Seifert, E., Klemm, U. and Jäckle, H., 1988. Abdominal segmentation of the Drosophila embryo requires a hormone receptor-like protein encoded by the gap gene knirps. Nature Vol. 336: 489-492. Niessing, D., Rivera-Pomar, R., La Rosée, A., Häder, T., Schöck, F., Purnell, B. A. and Jäckle, H., 1997. A cascade of transcriptional control leading to axis determination in Drosophila. Journal of cellular physiology 173: 162-167. 150 Literaturverzeichnis Niwa, N., Saitoh, M., Ohuchi, H., Yoshioka, H. and Noji, S., 1997. Correlation between distalless expression patterns and structures of appendages in development of the two-spottet cricket, Gryllus bimaculatus. Zoological Science 14: 115-125. Noordermeer, J., Johnston, P., Risewijk, F., Nusse, R. and Lawrence, P. A., 1992. The consequences of ubiquitous expression of the wingless gene in the Drosophila embryo. Development 116: 711-9. Nüsslein-Volhard, C. and Wieschaus, E., 1980. Mutations affecting segment number and polarity in Drosophila. Nature Vol. 287: 795 - 801. Olesnicky, E. C., Brent, A. E., Tonnes, L., Walker, M., Pultz, M. A., Leaf, D. and Desplan, C., 2006. A caudal mRNA gradient controls posterior development in the wasp Nasonia. Development 133: 3973-3982. Pagano, J. M., Farley, B. M., Essien, K. I., and Ryder, S. P., 2009. RNA recognition by the embryonic cell fate determinant and germline totipotency factor Mex-3. PNAS, Vol. 106, No.48: 20252-20257. Panfilio, K. A., Liu, P. Z., Akam, M. and Kaufman, T. C., 2006. Oncopeltus fasciatus zen is essential for serosal tissue function in katatrepsis. Developmental Biology Vol. 292, Issue 1: 226-243. Papatsenko, D. and Michael Levine, M., 2011. The Drosophila Gap Gene Network is composed of two parallel toggle switches. PLoS ONE 6(7): e21145.doi:101371/journal.pone.0021145 Patel, N. H., Condron, B. G. and Zinn, K., 1994. Pair-rule expression patterns of even-skipped are found in both short- and long-germ beetles. Nature Vol. 367: 429-434. Patel, N. H., Hayward, D. C., Lall, S., Pirkl, N. R., DiPietro, D. and Ball, E. E., 2001. Grasshopper hunchback expression reveals conserved and novel aspects of axis formation and segmentation. Development 128: 3459-3472. Pearson, J. C., Lemons, D. and McGinnis, W., 2005. Modulating Hox Gene Functions during animal body patterning. Nature Vol. 6, 893-904. Peel, A. D., 2008. The evolution of developmental gene networks: lessons from comparative studies on holometabolous insects. Phil. Trans. R. Soc. B. 363: 1539-1547. Penzlin, H., 2005. Lehrbuch der Tierphysiologie. Spektrum Akademischer Verlag; 7. Auflage: 193-196. Pinnell, J., Lindeman, P. S., Colavito, S., Lowe, C. and Savage, R. M., 2006. The divergent roles of the segmentation gene hunchback. Integrative and Comparative Biology, Vol. 46, No. 4: 519-532. Posnien, N., Bashasab, F. and Bucher, G., 2009. The insect upper lip (labrum) is a nonsegmental appendage-like structure. Evolution & Development Vol.11, No.5: 480-488. Preiss, A., Rosenberg, U. B., Kienlin, A., Seifert, E. und Jäckle, H., 1985. Molecular genetics of Krüppel, a gene required for segmentation of the Drosophila embryo. Nature, Vol. 313: 27-32. 151 Literaturverzeichnis Pultz, M. A., Pitt, J. N. and Alto, N. M., 1999. Extensive zygotic control of the anteroposterior axis in the wasp Nasonia vitripennis. Development 126: 701-710. Pultz, M. A., Westendorf, L., Gale, S. D., Hawkins, K., Lynch, J., Pitt, J. N., Reeves, N. L., Yao, J. C. Y., Small, S., Desplan, C. und Leaf, D. S., 2005. A major role for zygotic hunchback in patterning the Nasonia embryo. Development 132: 3705-3715. Panfilio, K. A., Liu, P. Z., Akam, M. and Kaufman, T. C., 2006. Oncopeltus fasciatus zen is essential for serosal tissue function in katatrepsis. Dev. Biol. 292: 226-243. Patel, N. H., Hayward, D. C., Lall, S., Pirkl, N. R., Di Pietro, D. and Ball, E. E., 2001. Grasshopper hunchback expression reveals conserved and novel aspects of axis formation and segmentation. Development 128: 3459-3472. Pestova, T. V., Kolupaeva, V. G., Lomakin, I. V., Pillpenko, E. V., Shatsky, I. N., Agol, V. I. and Hellen, C. U. T., 2001. Molecular mechanisms of translation initiation in eukaryotes. PNAS Vol. 98; no. 13: 7029-7036. Petschek, J. P., Perrimon, N. and Mahowald, A. P., 1987. Region-specific defects in l(1)giant Embryos of Drosophila melanogaster. Developmental Biology 119:175-189. Rivera-Pomar, R., Lu, X., Perrimon, N., Taubert, H. and Jäckle, H., 1995. Activation of posterior gap gene expression in the Drosophila blastoderm. Nature Vol. 376: 253-256. Rivera-Pomar, R., Niessing, D., Schmidt-Ott, U., Gehring, W. J. and Jäckle, H., 1996a. RNA binding and translational suppression by Bicoid. Nature Vol. 379: 746-749. Rivera-Pomar, R. and Jäckle, H., 1996. From gradients to stripes in Drosophila embryogenesis: filling in the gaps. Trends Genet. Vol. 12: 478-483. Payre, F., 2004. Genetic control of epidermis differentiation in Drosophila. Int. J. Dev. Biol. 48: 207 - 215. Peel, A. D., Chipman, A. D. and Akam, M. (2005). Arthropod segmentation: Beyond the Drosophila paradigm. Nature Reviews Genetics Vol. 6: 905-916. Pillemer, G., Epstein, M., Blumberg, B., Yisraeli, J. K., De Robertis, E. M., Steinbeisser, H. and Fainsod, A., 1998. Nested expression and sequential downregulation of the Xenopus caudal genes along the anterior-posterior axis. Mechanisms of Development 71: 193-196. Posnien, N., Schinko, J., Grossmann, D., Shippy, T. D., Konopova, B. and Bucher, G., 2009. RNAi in the Red Flour Beetle (Tribolium). CSH Protocols ; doi : 10.1101/pdb.prot 5256 Prpic, N. M. and Tautz, D., 2003. The expression of the proximodistal axis patterning genes distal-less and dachshund in the appendages of Glomeris marginata (Myriapoda: Diplopoda) suggests a special role of these genes in patterning the head appendages. Dev. Biol. Vol: 260 (1): 97-112. Pueyo, J. I. and Couso, J. P., 2011. Tarsal-less peptides control Notch signaling through the Shavenbaby transcription factor. Dev. Biol., doi: 10.1016/j.ydbio.2011.03.033 152 Literaturverzeichnis Rehm, E. J., Hannibal, R. L., Chaw, C. R., Vargas-Vila, M. A. and Patel, N. H., 2009. In Situ Hybridization of labeld RNA Probes to fixed Parhyale hawaiensis Embryos. Cold Spring Harb Protoc: doi10.1101/pdb.prot5130. Rose, L. S. and Kemphues, K. J., 1998. Early patterning of the C.elegans embryo. Annu. Rev. Genet. 32: 521-45. Rosenberg, M., Lynch, J. and Desplan, C., 2009. Heads and tails: Evolution of antero-posterior patterning in insects. Biochim. Biophys. Acta; doi: 10.1016/j.bbagrm. Roth, S. and Lynch, J. A., 2009. Symmetry Breaking during Drosophila Oogenesis. Cold Spring Harb Perspect Bio; 1: a001891. Rozen, S. and Skaletsky, H. J., 2002. Primer3 on the WWW for general users and for biologist programmers. Humana Press: pp 365-386 Rushlow, C., Frasch, M., Doyle, H. and Levine, M., 1987. Maternal regulation of zerknüllt: a homoeobox gene controlling differentiation of dorsal tissues in Drosophila. Nature Vol. 330: 583-586. Saenko, S. V., Brakefield, P. M. and Beldade, P., 2010. Single locus affects embryonic segment polarity and multiple aspects of an adult evolutionary novelty. BMC Biology, 8: 111 Sánchez, L. and Thieffry, D., 2003. Segmenting the fly embryo: a logical analysis of the pairrule cross-regulatory module. Journal of theoretical biology, 224; 4: 517-537. Sander, K., 1997. Pattern formation in insect embryogenesis: the evolution of concepts and mechanisms. Inf. J. Insect Morpho. & Embryol., Vol. 25, No.4: 349-367. Savard, J. 2004. A genomic approach to the study of ´Tribolium castaneum genetics, development and evolution. PhD thesis, University of Cologne, Germany. Savard, J., Tautz, D., Richards, S., Weinstock, G. M., Gibbs, R. A., Werren, J. H., Tetteling, H. and Lercher, M. J., 2006. Phylogenomic analysis reveals bees and wasps (Hymenoptera) at the base of the radiation of holometabolous insects. Genome Research 16: 1334-1338. Savard, J., Marques-Souza, H., Aranda, M. and Tautz, D., 2006. A Segmentation Gene in Tribolium produces a polycistronic mRNA that codes for multiple conserved Peptides. Cell 126: 559-569 Schäper, N. D., Pechmann, M., Damen, W. G. M., Pripic, N. M. and Wimmer, E. A., 2010. Evolutionary plasticity of collier function in head development of diverse arthropods. Developmental Biology 344: 363-376. Schinko, J. B., Kreuzer, N., Offen, N., Posnien, N., Wimmer, E. A. and Bucher, G., 2008. Divergent functions of orthodenticle, empty spiracles and buttonhead in early head patterning of the beetle Tribolium castaneum (Coleoptera). Developmental Biology 317: 600-613. Schinko, J. B., Weber, M., Viktorinova, I., Kiupakis, A., Averof, M., Klingler, M., Wimmer, E. and Bucher G., (2010). Functionality of the GAL4/UAS system in Tribolium requires the use of endogenous core promoters. BMC Dev Biol 10: 53 153 Literaturverzeichnis Schnellhammer, I. 2007. Einführung von Bruchidius obtectus als Modellorganismus in die Entwicklungsbiologie. Diplomarbeit, FAU, unveröffentlicht. Schnorrer, F., Bohmann, K. und Nüsslein-Volhard, C., 2000. The molecular motor Dynein is involved in targeting Swallow and bicoid RNA to the anterior pole of Drosophila oocytes. Nature Cell Biology, Vol.2: 185-190. Schoppmeier, M. and Schröder, R., 2005. Maternal Torso Signaling controls Body axis Elongation in a short germ insect. Current Biology, Vol. 15: 2131-2136. Schoppmeier, M., Fischer, S., Schmitt-Engel, C., Löhr, U. and Klingler, M., 2009. An Ancient anterior patterning system promotes caudal repression and head formation in Ecdysozoa. Current Biology, Vol. 19, Issue 21: 1811-1815. Schowalter, T. D., 2006. Insect ecology: An ecosystem approach. Academic Press: ISBN 012088772X, 9780120887729. Schröder, C., Tautz, D., Seifert, E. and Jäckle, H., 1988. Differential regulation of the two transcripts from the Drosophila segmentation gene hunchback. The EMBO Journal Vol. 7, No.9: 2881-2887. Schröder, R., 2003. The genes orthodenticle and hunchback substitute for bicoid in the beetle Tribolium. Nature, Vol. 422: 621-625. Schulz, C., Schröder, R., Hausdorf, B., Wolff, C. and Tautz, D., 1998. A caudal homologue in the short germ band beetle Tribolium shows similarities to both, the Drosophila and the vertebrate caudal expression patterns. Dev Genes Evol 208: 283-289. Shinmyo, Y., Mito, T., Matsushita, T., Sarashina, I., Miyawaki, K., Ohuchi, H. and Noji, S., 2005. caudal is required for gnathal and thoracic patterning and for posterior elongation in the intermediate-germband cricket Gryllus bimaculatus. Mechanisms of Development 122: 231-239. Simeone, A., Acampora, D., Mallamaci, A., Stornaiuolo, A., D´Apice, M. R., Nigro, V. and Boncinelli, E., 1993. A vertebrate gene related to orthodenticle contains a homeodomain of the bicoid class and demarcates anterior neuroectoderm in the gastrulating mouse embryo. The EMBO Journal, Vol.12, No.7: 2735-2747. Simpson-Brose, M., Treisman, J. and Desplan, C., 1994. Synergy between the Hunchback and Bicoid Morphogens is required for anterior patterning in Drosophila. Cell, Vol. 78: 855865. Siomi, H., Matunis, M. J., Michael, W. M. and Dreyfuss, G., 1993. The pre-mRNA binding K protein contains a novel evolutionarily conserved motif. Nucleic Acids Research, Vol. 21, No. 5: 1193-1198. Siomi, H., Choi, M., Siomi, M. C., Nussbaum, R. L. and Dreyfuss, G., 1994. Essential role for KH Domains in RNA Binding: Impaired RNA Binding by a mutation in the KH Domain of FMRI that causes fragile X Syndrome. Cell, Vol. 77: 33-39. Small, S., Blair, A. and Levine, M., 1992. Regulation of even-skipped stripe 2 in the Drosophila embryo. EMBO J., 11 (11): 4047-4057. 154 Literaturverzeichnis Sokoloff, A., 1974. The Biology of Tribolium - with special emphasis on genetic aspects. Oxford at the clarendon press, Volume 2 Spirov, A., Fahmy, K., Schneider, M., Frei, E., Noll, M. and Baumgartner, S., 2009. Formation oft the Bicoid morphogen gradient: a mRNA gradient dictates the protein gradient. Development 136: 605-614. Stauber, M., Jäckle, H. and Schmidt-Ott, U., 1999. The anterior determinant bicoid of Drosophila is a derived Hox class 3 gene. PNAS, Vol. 96: 3786-3789. Stauber, M., Taubert, H. and Schmidt-Ott, U., 2000. Function of bicoid and hunchback homologs in the basal cyclorrhaphan fly Megaselia (Phoridae). PNAS Vol. 97, No.20: 1084410849. Stauber, M., Lemke, S. and Schmidt-Ott, U., 2008. Expression and regulation of caudal in the lower cyclorrhaphan fly Megaselia. Dev Genes Evol 218: 81-87. Struhl, G., Struhl, K. and Macdonald, P. M., 1989. The gradient morphogen bicoid is a concentration-dependent transcriptional activator. Cell 57: 1259-1273. Struhl, G., Johnston, P. and Lawrence, P. A., 1992. Control of Drosophila body pattern by the Hunchback morphogen gradient. Cell 69 (2): 237-249. Surkova, M. S., Spirov, A. V., Gursky, V. V., Janssens, H., Kim, A. R., Radulescu, O., VanarioAlonso, C. E., Sharp, D. H., Samsonova, M. and Reinitz, J., 2009. Canalization of Gene Expression in the Drosophila Blastoderm by Gap Gene Cross Regulation. PLoS Biol 7 (3): e1000049. Doi:101371/journal-pbio.1000049 Takada, H., Kawana, T., Ito, Y., Kikuno, R. F., Mamada, H., Araki, T., Koga, H., Asashima, M. and Taira, M., 2009. The RNA-binding protein Mex3b has a fine-tuning system for mRNA regulation in early Xenopus development. Development 136: 2413-2422. Tautz, D., Lehmann, R., Schnürch, H., Schuh, R., Seifert, E., Kienlin, A., Jones, K. and Jäckle, H., 1987. Finger protein of novel structure encoded by hunchback, a second member of the gap class of Drosophila segmentation genes. Nature Vol. 327: 383-389. Tautz, D. and Pfeifle, C., 1989. A non-radioactive in situ hybridization method for the localization of specific RNAs in Drosophila embryos reveals translational control of the segmentation gene hunchback. Chromosoma 98: 81-5. Tautz, D., Friedrich, M. and Schröder, R., 1994. Insect embryogenesis - what is ancestral and what is derived? Development Supplement: 193-199. Terenius, O., Papanicolaou, A., Garbutt, J. S., Eleftherianos, I., Huvenne, H., Kanginakudru, S., Albrechtsen, M., An, C., Aymeric, J. L., Barthel, A., Bebas, P., Bitra, K., Bravo, A., Chevalier, F., Colligne, D. P., Crava, C. M., de Maagd, R. A., Duvic, B., Erlandson, M., Faye, I., Felföldi, G., Fujiwara, H., Futahashi, R., Gandhe, A. S., Gatehouse, H. S., Gatehouse, L. N., Giebultowicz, J. M., Gómez, I., Grimmelikhuijzen, C. J. P., Groot, A. T., Hauser, F., Heckel, D. G., Hegedus, D. D., Hrycaj, S., Huang, L., Hull, J. J., Iatrou, K., Iga, M., Kanost, M. R., Kotwica, J., Li, C., Li, J., Liu, J., Lundmark, M., Matsumoto, S., MeyeringVos, M., Millichap, P. J., Monteiro, A., Mrinal, N., Niimi, T., Nowara, D., Ohnishi, A., 155 Literaturverzeichnis Ozaki, C. O. K., Papakonstantinou, M., Popadic, A., Rajam, M. V., Saenko, S., Simpson, R. M., Soberón, M., Strand, M. R., Tomita, S., Toprak, U., Wang, P., Wee, C. W., Shyard, S., Zhang, W., Nagaraju, J., French-Constant, R. H., Herrero, S., Gordon, K., Swevers, L. and Smagghe, G., 2011. RNA interference in Lepidoptera: An overview of successful and unsuccessful studies and implications for experimental design. Journal of Insect Physiology, 57: 231-245. Trauner, J. und Büning, J., 2007. Germ-cell cluster formation in the telotrophic meroistic ovary of Tribolium castaneum (Coloeptera, Polyphaga, Tenebrionidae) and its implication on insect phylogeny. Dev Genes Evol 217: 13-27. Vincent, J. P. and Lawrence, P. A., 1994. Drosophila wingless sustains engrailed expression only in adjoining cells: Evidence from mosaic embryos. Cell, Vol.77; 6: 909-915. Vogel, O., 1977. Regionalisation of Segment-forming capacities during early embryogenesis in Drosophila melanogaster. Roux´s Archives 182, 9-32. Walldorf, U. and Gehring, W. J., 1992. empty spiracles, a gap gene containing a homeobox involved in Drosophila head development. EMBO Journal Vol., 11 no.6: 2247-2259. Weigel, D., Jürgens, G., Klingler, M. and Jäckle, H., 1990. Two Gap genes mediate maternal terminal pattern information in Drosophila. Science Vol. 248: 495-498. Werren, J. H. and Loehlin, D. W., 2009. The Parasitoid Wasp Nasonia: An Emerging Model System with Haploid Male Genetics. Cold Spring Harb. Protoc: doi: 10.1101/pdb.emo 134. Wieschaus, E., Nüsslein-Volhard, C. and Kluding, H., 1984. krüppel, a gene whose activity is required early in the zygotic genome for normal embryonic segmentation. Developmental Biology 104: 172-186. Wiegmann, B. M., Trautwein, M. D., Kim, J. W., Cassel, B. K., Bertone, M. A., Winterton, S. L. and Yeates, D. K., 2009. Single-copy nuclear genes resolve the phylogeny of the holometabolous insects. BMC Biology, 7: 34. Wilson, M. J. and Dearden, P. K., 2011. Diversity in insect axis formation: two orthodenticle genes and hunchback act in anterior patterning and influence dorsoventral organization in the honeybee (Apis mellifera). Development 138: 3497-3507. Wimmer, E. A., Simpson-Brose, M., Cohen, S. M., Desplan, C. and Jäckle, H., 1995. Transand cis-acting requirements for blastodermal expression of the head gap gene buttonhead. Mechanisms of Development 53: 235-245. Wolff, C., Schröder, R., Schulz, C., Tautz, D. and Klingler, M., 1998. Regulation of the Tribolium homologues of caudal and hunchback in Drosophila: evidence for maternal gradient systems in a short germ embryo. Development 125: 3645-3654 Wolff, C., Sommer, R., Schröder, R., Glaser, G. and Tautz, D., 1995. Conserved and divergent expression aspects of the Drosophila segmentation gene hunchback in the short germ band embryo of the flour beetle Tribolium. Development 121: 4227-4236. 156 Literaturverzeichnis Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E. and Strickland, S., 1997. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development 124: 3015-3023. Wu, L. H. and Lengyel, J. A., 1998. Role of caudal in hindgut specification and gastrulation suggests homology between Drosophila amnioproctodeal invagination and vertebrate blastopore. Development 125: 2433-2442. Wu, X., Vasisht, V., Kosman, D., Reinitz, J. and Small, S., 2001. Thoracic patterning by the Drosophila gap gene hunchback. Developmental Biology 237: 79-92. van der Zee, M., Berns, N. and Roth, S., 2005. Distinct Functions of the Tribolium zerknüllt genes in Serosa specification and dorsal closure. Current Biology Vol.15, Issue 7: 624-636. Ping Zuo, P., Dusan Stanjevic, D., John Colgan, J., Kyuhyung Han, K., Michael Levine, M. and James L. Manley, J. L., 1991. Activation and repression of transcription by the gap proteins Hunchback and Krüppel in cultured Drosophila cells. Genes Dev. 5: 254-264. 157 Erklärung Ich erkläre hiermit ehrenwörtlich, dass ich die vorliegende Dissertation ”Evolution früher Faktoren der Segmentierungskaskade: Funktionelle Untersuchungen in Bruchidius, Tribolium und Oncopeltus” selbst verfasst und dabei keine anderen als die angegebenen Hilfsmittel und Quellen verwendet habe. Ich erkläre weiterhin, dass ich nie versucht habe die vorliegende Dissertation in gleicher oder ähnlicher Form in einem Prüfungsverfahren vorzulegen. Ich habe früher außer den mit dem Zulassungsantrag urkundlich vorgelegten Graden keine weiteren akademischen Grade erworben oder zu erwerben versucht. Erlangen, im April 2012 Irene Schnellhammer 158 Danksagung Prof. Dr. Martin Klingler und PD Dr. Michael Schoppmeier danke ich, dass ich die Möglichkeit hatte, an mehreren Modellorganismen und verschiedenen Themen zu arbeiten, so dass die tägliche Laborarbeit sehr abwechslungsreich war. Speziell bei Martin möchte ich mich für seine Geduld bedanken und die angenehmen fachlichen Gespräche; bei Michael für den Ansporn zur Teilnahme an zahlreichen Tagungen sowie die Krapfen-, Schokoladen- und Gummibärchenberge. Danke an Herrn Prof. Dr. Frasch für die Übernahme des Zweitgutachtens. Bei Herrn Prof. Dr. Brandstätter und Frau Prof. Dr. Dietrich möchte ich mich für die Abnahme der mündlichen Prüfung bedanken. Großer Dank gebührt meinen beiden Laborgenossen Christian und Fabi: Christian, der mich als junge Doktorandin unter seine Fittiche genommen hat, mit mir zahllose biologische Themen diskutiert und meine lange Miene bezüglich seiner Musikauswahl geduldig ertragen hat. Fabi, der immer guter Laune ist, schwere Kartons und Kanister für mich gehoben hat und tonnenweise Tribolium-Embryonen fixiert hat. Jutta, meiner Paralleldoktorandin und Reisegefährtin auf sämtlichen Tagungen, danke ich für die gute Zusammenarbeit und den täglichen Schwatz zwischen Tür und Angel. Ralph Rübsam möchte ich danken, für viele interessante Vogelexkursionen, Gespräche über Gott und die Welt, sowie seine Vorbildfunktion als Forscher und Biologe. Angela und Holli möchte ich für die lustigen Mensagänge danken und den gesicherten Nachschub an yellow/white-Drosophila-Stämmen. Tina danke ich für meine Urlaubsvertretung in der Bruchidius-Zucht und ihre Hilfe, wenn man mal wieder irgendein Spezialsiebchen, Töpfchen oder was auch immer gesucht hat. Vielen Dank Claudia, für die Nachbestellung sämtlicher Verbrauchsgegenstände und deine Besuche in meinem Labor um mal kurz vorbei zu schauen. Schade, dass das mit dem Zwischenfenster zwischen unseren beiden Räumen nie geklappt hat. Danke an Upal und Nicole für frischen Wind im Labor und die Erweiterung meiner englischen Sprachkünste. Bedanken möchte ich mich auch bei allen Studenten, die im Laufe der Jahre in meinem Labor gewerkelt haben (Nadi, Tobi, Franzi, Lissy, Romina, Anna, Jessi, Klaus, Doro, Denise, Fany und Ahmad), für die immer gute Atmosphäre und neuen Denkanstöße. Maria danke ich für ihr heiteres Temperament und den Freitagssatz: Jetzt haben wir es dann bald geschafft, Irene! 159