Biofocus - BIOspektrum
Werbung

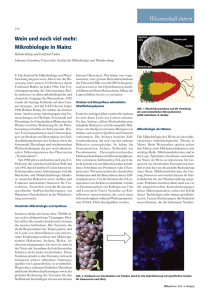
Biofocus 39 B I O S P E K T R U M • 1. 0 1 • 7. J A H R G A N G Habilitierte stellen sich vor Redox-getriebene Protonentranslokation in methanogenen Archaea Uwe Deppenmeier, Institut für Mikrobiologie und Genetik, Universität Göttingen 쑺 Extrem sauerstoffempfindliche, Methan-produzierende Organismen werden in der Gruppe der methanogenen Archaea zusammengefasst. Sie spielen eine wichtige Rolle in der anaeroben Nahrungskette an sulfatarmen Standorten, da sie Endproduk- te aus Gärungsvorgängen zur Methanogenese verwenden und die gasförmigen Produkte (CH4 und CO2) dem Kohlenstoffkreislauf zuführen. Die methylotrophen Vertreter dieser Gruppe wie etwa Methanosarcina mazei können Methanol, Methylamine, Acetat Abb. 1: Darstellung der anaeroben Atmungskette aus Methanosarcina mazei. FeS, Eisen-Schwefel-Zentren; MPhen, Methanophenazin, HS-CoM, Coenzym M, HS-CoB, Coenzym B Biofocus 40 B I O S P E K T R U M • 1. 0 1 • 7. J A H R G A N G oder H2/CO2 umsetzen. Nicht nur wegen der ökologischen Relevanz und der Bedeutung des Methans als Treibhaus- und Biogas haben die methanogenen Archaea ein großes mikrobiologisches Interesse gefunden, sondern auch auf Grund ihrer ungewöhnlichen Cofaktoren, Enzyme und Katalysemechanismen [1]. An dieser Stelle seien nur die Verbindungen Coenzym M und Coenzym B erwähnt, die als Überträger von C1-Verbindungen beziehungsweise als Elektronendonatoren im Zuge der Methanogenese dienen. Der zentrale Elektronenüberträger im Cytoplasma ist das Coenzym F420, das eine ähnliche Funktion wie NAD+ in Bakterien und Eukaryoten einnimmt. Die Prozesse der Methanbildung [1, 2] sind sehr komplex und können an dieser Stelle nicht im Detail beschrieben werden. Hervorzuheben ist jedoch, dass alle Stoffwechselwege zur Synthese eines gemischten Disulfids aus Coenzym M und Coenzym B führen, das als Heterodisulfid (CoM-S-SCoB) bezeichnet wird. Es fungiert als Elektronenakzeptor von verschiedenen membrangebundenen Elektronentransport-Systemen. Als Elektronendonatoren können H2, reduziertes F420 (F420H2) oder reduziertes Ferredoxin genutzt werden. Die anaeroben Atmungsketten [2], die an der Umsetzung der Substrate beteiligt sind, werden im Folgenden näher beschrieben: Reduziertes F420 wird über ein neuartiges membrangebundenes Enzym, das als F420H2-Dehydrogenase bezeichnet wurde, reoxidiert. Die Reduktionsäquivalente werden von der Heterodisulfid-Reduktase zur reduktiven Spaltung des Heterodisulfids verwendet (Abb. 1). Dieses System ist von essentieller Bedeutung beim Wachstum auf Methanol und Methylaminen. F420H2 + CoM-S-S-CoB 씮 HS-CoM + HS-CoB + F420 (∆G0'= -30,9 kJ/mol) In Anwesenheit von H2 (Wachstum auf H2 + CO2) dient eine membrangebundene Hydrogenase zur Bereitstellung von Reduktionsäquivalenten, die ebenfalls auf die Heterodisulfid-Reduktase übertragen werden (Abb. 1). H2 + CoM-S-S-CoB 씮 HS-CoM + HS-CoB (∆G0' = -40 kJ/mol) Der Transfer von Elektronen zwischen der Hydrogenase beziehungsweise F420H2Dehydrogenase und der Heterodisulfid-Reduktase wird von dem in meiner Arbeitsgruppe entdeckten Cofaktor Methanophenazin katalysiert [3]. Hierbei handelt es sich um ein 2-Hydroxyphenazin-Derivat, das über eine Etherbindung mit einer Pentaisoprenoid-Einheit verknüpft ist. Kürzlich gelang uns die chemische Synthese dieses ungewöhnlichen Elektronenüberträgers [4]. Es zeigte sich, dass der Cofaktor die Funktion von Chinonen übernimmt, die in methanogenen Archaea nicht vorkommen. Die Redox-Reaktionen in der Membran von Methanosarcina mazei sind mit Protonentranslokationen über die Cytoplasmamembran verbunden [5, 6]. Dabei ist die Reduktion des Methanophanzins durch die membrangebundene Hydrogenase beziehungsweise durch die Dehydrogenase als auch die Oxidation des reduzierten Cofaktors durch die Heterodisulfid-Reduktase mit einem Transfer von zwei Protonen gekoppelt (Abb.1). Der entstehende elektrochemische Protonengradient wird von einer A1Ao-ATP Synthase zur Bildung von ATP genutzt [2]. Interessanterweise zeigen die initialen Enzyme der Elektronentransportketten von Methanosarcina mazei große Ähnlichkeiten zu Atmungskomponenten von Bakterien. So ist die membrangebundene Hydrogenase homolog zu Hydrogenasen zahlreicher Gramnegativer Bakterien [2, 6]. Die Organismen enthalten ein b-Typ-Cytochrom, das mit dem Kern-Enzym, bestehend aus der großen und kleinen Untereinheit, verbunden ist. Das aktive NiFe-Zentrum zur H2-Oxidation befindet sich mit hoher Wahrscheinlichkeit auf der Außenseite der Membran, so dass es im Zuge der H2-abhängigen Methanophenazin-Reduktion zur Freisetzung von zwei skalaren Protonen kommt. Überraschend war auch die Erkenntnis, dass die F420H2-Dehydrogenase ähnlich aufgebaut ist wie die protonentranslozierende NADHDehydrogenase (Komplex 1 der Atmungskette) aus E. coli [7]. Das methanogene Enzym setzt sich wie das bakterielle Gegenstück aus drei Subkomplexen zusammen und enthält FAD und verschiedene EisenSchwefel-Zentren [5]. FpoF stellt das InputModul dar und ist für die Oxidation von F420H2 verantwortlich (Abb. 1). Das membranassoziierte Modul (FpoB, C, D und I) und der membranintegrale Subkomplex (FpoA, H, J, K, L, M, und N) sind völlig homolog zu dem entsprechenden Modulen von bakteriellen NADH-Dehydrogenasen. Die Elektronen aus der F420H2-Oxidation werden vermutlich auf Methanophenazin übertragen (Abb. 1). Der Mechanismus der Protonentranslokation ist bisher unbekannt. Die detaillierte Untersuchung der F420H2- Dehydrogenase soll fortgesetzt werden und zur Aufklärung des Reaktionsmechanismus von Komplex I aus Bakterien und Mitochondrien beitragen. Literatur [1] Thauer, R. K. (1998) Biochemistry of methanogenesis: a tribute to Marjory Stephenson. Microbiology 144, 2377-2406 [2] Deppenmeier, U., Lienard, T. & Gottschalk, G. (1999) Novel reactions involved in energy conservation by methanogenic archaea. FEBS Lett. 457, 291-297 [3] Abken, H. J., Tietze, M., Brodersen, J., Bäumer, S., Beifuss, U. & Deppenmeier, U. (1998) Isolation and characterization of methanophenazine and function of phenazines in membrane-bound electron transport of Methanosarcina mazei Gö1, J. Bacteriol. 180, 2027-2032 [4] Beifuss, U., Tietze, M., Bäumer, S. & Deppenmeier, U. (2000) Methanophenazine: structure, total synthesis and function of a new cofactor from methanogenic archaea, Angew. Chem. Int. Ed. 39, 2470-2472 [5] Bäumer, S., Ide, T., Jacobi, C., Johann, A., Gottschalk, G. & Deppenmeier, U. (2000). The F420H2 dehydrogenase from Methanosarcina mazei Gö1 is a redox-driven proton pump closely related to NADH dehydrogenases, J. Biol. Chem. 275, 1796817973 [6] Ide, T., Bäumer, S. & Deppenmeier, U. (1999). Energy conservation by the H2:heterodisulfide oxidoreductase from Methanosarcina mazei Gö1. J. Bacteriol. 181, 4076-4080 [7] Friedrich, T. (1998) The NADH:ubiquinone oxidoreductase (complex I) from Escherichia coli, Biochim. Biophys. Acta 1364, 134-146 Korrespondenzadresse PD Dr. Uwe Deppenmeier Institut für Mikrobiologie & Genetik Georg-August-Universität Göttingen Grisebachstr. 8 D- 37077 Göttingen Tel.: 0551-393812 Fax: 0551-393793 eMail: [email protected] Uwe Deppenmeier Jahrgang 1962, Biologiestudium (1982-1988) an der Universität Göttingen. 1988-1991 Promotion bei Prof. Dr. G. Gottschalk am Institut für Mikrobiologie in Göttingen. 1991-1992 Postdoktorand im Labor von Prof. R. P. Gunsalus, UCLA, Los Angeles, USA. Seit 1993 Wissenschaftlicher Assistent am Institut für Mikrobiologie und Genetik, Universität Göttingen. 1999 Habilitation für das Fachgebiet Mikrobiologie an der Georg-August Universität Göttingen.