08 - Enzymkinetik - Ruhr
Werbung

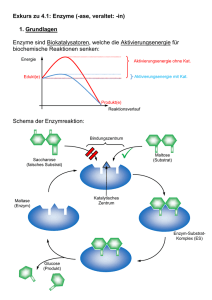
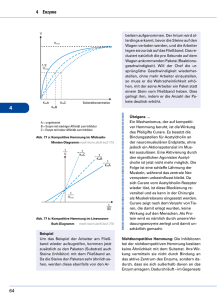
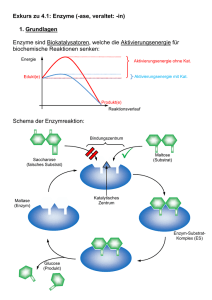
53 GRUNDLAGEN DER ENZYMKINETIK: MICHAELIS-MENTEN-GLEICHUNG, HEMMTYPEN BIOCHEMISCHE GRUNDLAGEN Die katalytischen Eigenschaften eines Enzyms können durch verschiedene Faktoren beeinflusst werden. Dazu gehören vor allem Temperatur, pH-Wert und Ionenstärke des Reaktionsmilieus. Die Geschwindigkeit einer enzymatischen Reaktion ist in bestimmten Grenzen auch von der Konzentration des Substrates [S] abhängig. In der Regel nimmt die Reaktionsgeschwindigkeit mit steigender Substratkonzentration zu, bis bei hohen Konzentrationen ein Sättigungswert (Maximalgeschwindigkeit Vmax) erreicht wird. Das Erreichen dieser Maximalgeschwindigkeit ist ein wesentliches Charakteristikum enzymatisch katalysierter Reaktionen. Im einfachsten Fall, in dem nur ein Substrat umgesetzt wird, lässt sich die Abhängigkeit der Reaktionsgeschwindigkeit von der Substratkonzentration mit Hilfe der von Michaelis und Menten angegebenen Gleichung beschreiben: v 0 d[S] dt V K S S max m Dabei bedeutet: [S]: Substratkonzentration bei Beginn der Reaktion V0: Anfangsgeschwindigkeit der Reaktion bei der entsprechenden Substratkonzentration [S] t: Zeit Vmax: Maximalgeschwindigkeit der Reaktion, die bei Substratsättigung erreicht wird Beachte: Vmax ist proportional zur gesamten Enzymkonzentration [E]0: Vmax ~ [E]0 Km: Michaelis-Konstante; bei dieser Substratkonzentration läuft die Reaktion mit halbmaximaler Geschwindigkeit ab. Oft ist die Michaelis-Konstante ein Maß für die Affinität des Enzyms zum Substrat. Die Ermittlung der Michaelis-Konstante hat eine große praktische Bedeutung für die Beurteilung eines Enzyms und der Wirkungsweise von Enzyminhibitoren. 54 Die Bestimmung der Maximalgeschwindigkeit spielt im klinisch-chemischen Labor eine große Rolle. Da die Maximalgeschwindigkeit Vmax proportional zur Enzymkonzentration ist, lässt sich aus der Maximalgeschwindigkeit die Enzymkonzentration (z.B. im Blut) ermitteln. Die Michaelis-Konstante lässt sich graphisch ermitteln, wenn man die Anfangsgeschwindigkeit der Reaktion bei verschiedenen Substratkonzentrationen unter sonst identischen Bedingungen misst und die Werte V0 gegen [S] aufträgt (Auftragung nach Michaelis und Menten). Dabei ergibt sich eine Hyperbel, die bei hohen Substratkonzentrationen der Maximalgeschwindigkeit Vmax zustrebt. Im Bereich hoher Substratkonzentrationen ist also die Reaktionsgeschwindigkeit praktisch unabhängig von der Substratkonzentration, im Bereich niedriger Substratkonzentrationen ist V0 dagegen stark von der Substratkonzentration abhängig. Aus der Kurve lässt sich die halbmaximale Geschwindigkeit und die zugehörige Substratkonzentration, die der Michaelis-Konstante des Enzyms entspricht, ablesen. S K m V wenn v 0 max 2 Diese Methode zur Bestimmung der Michaelis-Konstante ist jedoch verhältnismäßig ungenau, da sich Vmax in den wenigsten Fällen eindeutig festlegen lässt. Die graphische Bestimmung wird wesentlich vereinfacht durch eine Auftragung nach Lineweaver und Burk, der eine einfache Umformung der Michaelis-Menten-Gleichung zugrunde liegt: K 1 1 m v V S V 0 max max Bei einer Auftragung von 1/V0 gegen 1/[S] erhält man eine Gerade, die die Y-Achse im Punkt 1/Vmax und die X-Achse im Punkt -1/Km schneidet. Aus diesen Schnittpunkten lassen sich sowohl die Maximalgeschwindigkeit als auch die Michaelis-Konstante bestimmen. Im normalen Stoffwechsel spielt die Hemmung enzymatischer Reaktionen - ebenso wie die Aktivierung - eine bedeutende Rolle bei der Regulation von metabolischen Prozessen. Daneben lassen sich in vielen Fällen Vergiftungserscheinungen oder auch die Wirkung bestimmter Pharmaka auf eine Hemmung spezifischer enzymatischer Reaktionen zurückführen. Grundsätzlich unterscheidet man bei der Hemmung von Enzymen zwischen irreversibler und reversibler Hemmung. Im Gegensatz zur irreversiblen Hemmung kann bei der reversiblen Hemmung der Inhibitor vom Enzym dissoziieren (z.B. durch Verdünnen). 55 REVERSIBLE HEMMTYPEN Man unterscheidet drei Hemmtypen: kompetitive Hemmung, nichtkompetitive Hemmung und unkompetitive Hemmung. Bei der kompetitiven Hemmung wird der Inhibitor reversibel am aktiven Zentrum des Enzyms gebunden und verhindert so die Bindung des Substrats. Durch erhöhte Substratkonzentrationen kann der Inhibitor jedoch verdrängt werden, so dass bei sehr hohen Substratkonzentrationen die Maximalgeschwindigkeit der enzymatischen Reaktion erreicht werden kann. Die scheinbare Michaelis-Konstante des Enzyms nimmt dagegen in Anwesenheit des kompetitiven Inhibitors zu. Die Auftragung der Kehrwerte der Geschwindigkeit (1/V0) gegen die Kehrwerte der Substratkonzentration (1/[S]) nach Lineweaver und Burk ergibt eine Gerade, die die Gerade der ungehemmten Reaktion auf der Y-Achse im Punkt 1/Vmax schneidet. Aus dem Schnittpunkt mit der X-Achse lässt sich die scheinbare (oder auch apparente) Michaelis-Konstante (Km app) der gehemmten Reaktion errechnen, die mit der Inhibitor-Konstante Ki über folgende Formel verknüpft ist: K m app 1 I K m Ki Dabei ist [I] die molare Konzentration des Inhibitors. Die Inhibitor-Konstante Ki ist ein Maß für die Affinität zwischen Enzym und Inhibitor. Sie gibt diejenige Konzentration des Inhibitors an, bei der die apparente Michaelis-Konstante der gehemmten Reaktion gerade doppelt so groß ist wie die Michaelis-Konstante der ungehemmten Reaktion. Bei der nichtkompetitiven Hemmung wird der Inhibitor nicht am aktiven Zentrum, sondern an einer anderen Stelle des Enzymproteins gebunden. Dadurch wird die Aktivität des Enzyms herabgesetzt oder vollständig unterdrückt. Da in diesem Fall der Inhibitor nicht durch das Substrat vom Enzym verdrängt werden kann, wird selbst bei sehr hohen Substratkonzentrationen die Maximalgeschwindigkeit des Enzyms nicht erreicht. Für den speziellen Fall, dass die Michaelis-Konstante für das Substrat unverändert bleibt, ergibt sich bei der Kehrwertauftragung nach Lineweaver und Burk bei der nichtkompetitiven Hemmung eine Gerade, welche die X-Achse im gleichen Punkt schneidet wie die Gerade der ungehemmten Reaktion. Die Maximalgeschwindigkeit der gehemmten Reaktion lässt sich aus dem Schnittpunkt mit der Y-Achse bestimmen. Die Dissoziationskonstante des EnzymInhibitor-Komplexes (Ki) berechnet man mit Hilfe der folgenden Formel: V V app max 1 I Ki 56 Hierbei ist Vapp die in Anwesenheit des Inhibitors erhaltene Maximalgeschwindigkeit, [I] die molare Konzentration des Inhibitors und Vmax die Maximalgeschwindigkeit der ungehemmten Reaktion. Ki entspricht der Inhibitorkonzentration, bei der die Maximalgeschwindigkeit der gehemmten Reaktion gerade halb so groß ist wie die Maximalgeschwindigkeit der nicht gehemmten Reaktion. Wird der Inhibitor nur von dem Enzym-Substrat-Komplex und nicht vom freien Enzym gebunden, handelt es sich um eine unkompetitive Hemmung. Bei diesem Hemmtyp wird der Enzym-Substrat-Inhibitor-Komplex nicht oder nur verlangsamt zum Enzym-Produkt-Komplex umgesetzt. In der Auftragung nach Lineweaver und Burk ergibt eine enzymatische Reaktion in Gegenwart eines unkompetitiven Inhibitors eine Gerade, die parallel zur Geraden für die ungehemmte Reaktion verläuft. Die Inhibitorkonstante lässt sich nach folgenden Formeln ermitteln: V V app max 1 I K oder K m app i K 1 m I K i wobei Km app bzw. Vapp die in Anwesenheit des Inhibitors erhaltene Michaelis-Konstante und Maximalgeschwindigkeit, [I] die molare Konzentration des Inhibitors und Km und Vmax die entsprechenden Konstanten der ungehemmten Reaktion sind. Ein unkompetitiver Inhibitor reduziert in gleichem Maße sowohl Vmax als auch Km einer enzymatischen Reaktion. Häufig wird auch das Auftreten von gemischten Hemmtypen beobachtet, die sich nicht eindeutig einer dieser drei Grundarten zuordnen lassen. ALLGEMEINE FRAGESTELLUNGEN Enzyme als Biokatalysatoren; Substratspezifität, Klassifizierung der Enzyme; Grundlagen der Messung einer Reaktionsgeschwindigkeit im optischen Test; Michaelis-Menten-Gleichung; Mechanismen der Hemmung von Enzymen; Interpretation von Km, Vmax, Ki; Medizinische Bedeutung der Enzyme. 57 58 ZIELSETZUNG DER EXPERIMENTE 1. Bestimmung der Michaelis-Konstante der Lactatdehydrogenase aus Kaninchen-Skelettmuskeln Die Lactatdehydrogenase (L-Lactat: NAD Oxidoreductase, EC 1.1.1.27) (LDH) katalysiert den letzten Schritt der anaeroben Glykolyse LDH CH3-CHOH-COO + NAD+ L (+) Lactat CH3-CO-COO + NADH + H+ Pyruvat Das Gleichgewicht dieser Reaktion liegt auf der Seite der Lactatproduktion. Die Gleichgewichtskonstante K(25°C) = Pyruvat NADH H 2 ,7 10 12 Lactat NAD Unter den Reaktionsbedingungen in diesem Versuch liegt das gesamte Enzym in Form eines LDH-NADH Komplexes vor. Die Michaelis-Konstante Km für Pyruvat beschreibt in diesem Falle die Wechselwirkung zwischen Pyruvat und dem LDH-NADH Komplex. Die Reaktionsgeschwindigkeit wird mit dem photometrischen Test nach Otto Warburg gemessen, wobei der Unterschied im Absorptionsmaximum von NAD+ und NADH ausgenutzt wird. Nikotinamid-Adenin-Dinukleotide (NAD) sind als Coenzyme von Dehydrogenasen in der Übertragung von Reduktionsequivalenten beteiligt. Dabei geht die oxidierte Form, NAD+, in die reduzierte Form, NADH, über: RH2 + NAD+ R + NADH + H+ 59 Bei der Dehydrogenierung wird ein H-Hydridion (1 Proton + 2 Elektronen) auf die C4-Position des Pyridinringes übertragen. Der zweite Substratwasserstoff wird als Proton H+ freigesetzt. Dieser Prozess ist schematisch am Beispiel der LDH-Reaktion dargestellt: NADH besitzt neben dem Absorptionsmaximum bei 260 nm ein zusätzliches Maximum bei 340 nm, das der oxidierten Form, dem NAD+, fehlt. In der Praxis wird die Messung statt bei 340 nm oft bei 366 nm durchgeführt, da Wolframdraht-Glühlampen in diesem Bereich benutzt werden können. 60 Der Extinktionskoeffizient für NADH beträgt in Abhängigkeit von der Wellenlänge: 340 = 6,2 · 103 (ℓ · mol-1 · cm-1) 366 = 3,3 · 103 (ℓ · mol-1 · cm-1) (Bei Verwendung von zur Berechnung ist neben der verwendeten Wellenlänge die Dimension von unbedingt zu berücksichtigen.) HEMMUNG DER LACTATDEHYDROGENASE DURCH OXAMAT In diesem Versuch wird die Hemmung des Lactatdehydrogenase-NADH – Komplexes durch Oxamat untersucht. Substrat ist dabei Pyruvat. Man kann aber auch NADH als Substrat der Lactatdehydrogenase betrachten. Entsprechend könnte auch die Hemmung des Umsatzes von NADH durch Oxamat untersucht werden. Aus rein praktischen Gründen ist dieses Experiment mit den im Praktikum zur Verfügung stehenden Mitteln nicht möglich: Wegen der hohen Affinität von NADH, also dem niedrigen Km-Wert für die Bindung von NADH an Lactatdehydrogenase, müssten die Messungen bei NADH-Konzentrationen durchgeführt werden, die so niedrig sind, dass ein photometrische Bestimmung nicht möglich ist. Bei geeigneter Versuchsanordnung (Messung der NADHKonzentrationen durch Fluoreszenz) würde sich herausstellen, dass Oxamat nur an den Lactatdehydrogenase–NADH–Komplex, aber nicht an die Lactatdehydrogenase bindet. Oxamat (Halbamid der Oxalsäure) Fragen: Vergleichen Sie die Struktur des Inhibitors Oxamat mit der Struktur der Substrate Pyruvat und NADH Welchen Hemmtyp erwarten Sie für die Hemmung der Lactatdehydrogenase durch Oxamat a) bezüglich Pyruvat? b) bezüglich NADH? 61 VERSUCHSDURCHFÜHRUNG UND VERSUCHSPROTOKOLL Mitbringliste: 30cm langes Lineal, Taschenrechner, Kittel Messung der Reaktionsgeschwindigkeit der LDH-Reaktion in Abhängigkeit von der Pyruvat-Konzentration 1. In Abwesenheit von Oxamat: Pipettierplan A Küvette Nr. ml Na-Phosphat-Puffer (Glaskolben) (0,1 mol/ℓ, pH 7.4) ml NADH (gelbes Eppendorf-Gefäß) (5 mmol/ℓ) ml Na-Pyruvat (blaues Eppendorf-Gefäß) (10 mmol/ℓ) ml H2O 1 2 3 4 5 2,00 2,00 2,00 2,00 2,00 0,15 0,15 0,15 0,15 0,15 0,02 0,05 0,10 0,20 0,40 0,81 0,78 0,73 0,63 0,43 0,02 0,02 0,02 0,02 0,02 START der Reaktion ml LDH* (rotes Eppendorf-Gefäß) Die Messung erfolgt gegen eine mit 3ml Wasser gefüllte Küvette. Der Küvetteninhalt wird durch dreimaliges Umdrehen der Küvette (bitte vorher mit Parafilm verschließen) gründlich gemischt und die Anfangsextinktion (E0) gemessen. Nach 30 sec wird überprüft, ob E0 unverändert bleibt. Die Küvette wird aus dem Photometer herausgenommen. Das Enzym (jeweils 0,02 ml) wird in die Küvette pipettiert. Der Küvetteninhalt wird sofort mit Parafilm verschlossen und durch dreimaliges Wenden der Küvette gemischt. Nach Entfernen des Parafilms wird die Küvette in das Photometer eingesetzt. Für einen Zeitraum von 2 min wird in Abständen von 15 sec die Extinktion abgelesen. 62 2. In Anwesenheit von Oxamat: Pipettierplan B Küvette Nr. ml Na-Phosphat-Puffer (Glaskolben) (0,1 mol/ℓ, pH 7.4) ml NADH (gelbes Eppendorf-Gefäß) (5 mmol/ℓ) ml Na-Pyruvat (blaues Eppendorf-Gefäß) (10 mmol/ℓ) ml Oxamat (grünes Eppendorf-Gefäß) (1 mmol/ℓ) ml H2O 6 7 8 9 10 2,00 2,00 2,00 2,00 2,00 0,15 0,15 0,15 0,15 0,15 0,02 0,05 0,10 0,20 0,40 0,24 0,24 0,24 0,24 0,24 0,57 0,54 0,49 0,39 0,19 0,02 0,02 0,02 0,02 0,02 START der Reaktion ml LDH* (rotes Eppendorf-Gefäß) Der Küvetteninhalt wird durch dreimaliges Umdrehen der Küvette (bitte vorher mit Parafilm verschließen) gründlich gemischt und die Anfangsextinktion (E0) gemessen. Nach 30 sec wird überprüft, ob E0 unverändert bleibt. Die Küvette wird aus dem Photometer herausgenommen. Das Enzym (jeweils 0,02 ml) wird in die Küvette pipettiert. Der Küvetteninhalt wird sofort mit Parafilm verschlossen und durch dreimaliges Wenden der Küvette gemischt. Nach Entfernen des Parafilms wird die Küvette in das Photometer eingesetzt. Für einen Zeitraum von 2 min wird in Abständen von 15 sec die Extinktion abgelesen. 63 Messergebnisse zu Pipettierplan A Zeit Küvette 1 2 3 4 5 Vor Zugabe von LDH 0 30“ Start durch Zugabe von LDH (0,02 ml/Küvette; mischen) 15“ 30“ 45“ 1’ 1’15“ 1’30“ 1’45“ 2' Messergebnisse zu Pipettierplan B Zeit Küvette 6 7 8 9 Vor Zugabe von LDH 0 30“ Start durch Zugabe von LDH (0,02 ml/Küvette; mischen) 15“ 30“ 45“ 1’ 1’15“ 1’30“ 1’45“ 2' 10 64 AUSWERTUNG UND FEHLERDISKUSSION Die Anfangsgeschwindigkeiten (NADH Verbrauch pro Zeiteinheit) werden mit Hilfe des Lambert-Beer Gesetzes errechnet: E = · c · d E = ermittelte Extinktionsänderung = molarer Extinktionskoeffizient von NADH bei 366 nm = 3,3·103 l · mol-1· cm-1 d = Schichtdicke, 1 cm c = Änderung der NADH-Konzentration Für die mit Hilfe des Steigungsdreiecks für ein Zeitintervall von t min ermittelte Anfangsgeschwindigkeit der Reaktion, v0, ergibt sich dann: v 0 c t E d t E mol 1 min - 1 3,3 103 t Für t = 1 min folgt daraus: v 0 E 0 , 30 µmol ml 1 min 1 65 Aufgaben: 1. Stellen Sie die Zeitverläufe der Extinktion der Versuche 3 und 8 jeweils in einem Graphen dar. 2. Bestimmen Sie Extinktionsabnahme pro min ab dem Messpunkt 15sec. 3. Stellen Sie die Abhängigkeit der Anfangsgeschwindigkeiten von den Substratkonzentrationen in An- und Abwesenheit des Inhibitors jeweils in einem Graphen nach Michaelis-Menten (Anfangsgeschwindigkeit gegen Substratkonzentration) und Lineweaver-Burk (1/Anfangsgeschwindigkeit gegen 1/Substratkonzentration) dar. Bestimmung der Substratkonzentrationen [S] (Pyruvatkonzentrationen): Die Konzentration der Na-Pyruvat-Lösung beträgt 10 mmol/ℓ (siehe Pipettierplan A und B). Diese Na-Pyruvat-Lösung wird durch das Pipettieren um folgenden Faktor verdünnt: 𝑃𝑦𝑟𝑢𝑣𝑎𝑡 − 𝐿ö𝑠𝑢𝑛𝑔 𝑉𝑜𝑙𝑢𝑚𝑒𝑛 𝑑𝑒𝑠 𝑔𝑒𝑠𝑎𝑚𝑡𝑒𝑛 𝐾ü𝑣𝑒𝑡𝑡𝑒𝑛𝑖𝑛ℎ𝑎𝑙𝑡𝑒𝑠 (= 3 𝑚𝑙) Folglich beträgt die Konzentration [S] der Na-Pyruvat-Lösung in den einzelnen Küvetten: [𝑆] = 𝐾𝑜𝑛𝑧𝑒𝑛𝑡𝑟𝑎𝑡𝑖𝑜𝑛 𝑑𝑒𝑟 𝑁𝑎 − 𝑃𝑦𝑟𝑢𝑣𝑎𝑡 − 𝐿ö𝑠𝑢𝑛𝑔 = 10 𝑚𝑚𝑜𝑙/ℓ ∙ 𝑃𝑦𝑟𝑢𝑣𝑎𝑡 − 𝐿ö𝑠𝑢𝑛𝑔 𝑉𝑜𝑙𝑢𝑚𝑒𝑛 𝑑𝑒𝑠 𝑔𝑒𝑠𝑎𝑚𝑡𝑒𝑛 𝐾ü𝑣𝑒𝑡𝑡𝑒𝑛𝑖𝑛ℎ𝑎𝑙𝑡𝑒𝑠 𝑃𝑦𝑟𝑢𝑣𝑎𝑡 − 𝐿ö𝑠𝑢𝑛𝑔 (𝑚𝑙) 3 𝑚𝑙 4. Ermitteln Sie für die ungehemmte und die gehemmte Reaktion jeweils Vmax, Km und Km app 5. Welcher Typ der Enzymhemmung durch Oxamat liegt vor? 6. Berechnen Sie mit Hilfe der im Text für den jeweiligen Hemmtyp angegebenen Formel die Inhibitor-Konstante, Ki. 66 Tabelle Küvette Nr. Extinktionsabnahme pro Minute Anfangsgeschwindigkeit 30“ – 1‘30“ v 0 E min 1 v0 0 , 30 * Substratkon -zentration [S] (in der Küvette) 1 [S] E min µmol ml min ml min µmol 1 2 3 4 5 6 7 8 9 10 * siehe Berechnung mmol/ mmol 67 BERECHNUNGEN 68 HÄUFIGSTE FEHLER BEIM EXPERIMENT UND SEINER AUSWERTUNG 1. Experiment - Photometer nicht gemäß der Anleitung am Arbeitsplatz eingestellt - Mangelhafte Pipettierung (Merke: Pipettenspitze soll bei Entnahme aus dem Vorrat und beim Pipettieren in die Küvette schräg aufstehen. Pipetten senkrecht halten, Küvetten schräg! - Durchmischung des Küvetteninhalts vergessen - Ungenaue Ablesung vom Photometer (zu früh oder zu spät) 2. Auswertung - Messpunkte nicht vollständig, unklar oder falsch eingetragen - Dimension nicht angegeben, Koordinatenbezeichnung in Abbildungen ungenügend. 69 SCHLUSSFOLGERUNGEN