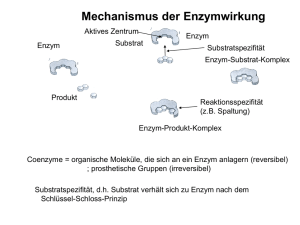
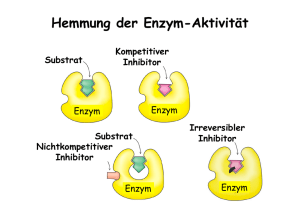
Enzym
Werbung

Enzyme Enzyme im Studyguide steht diese Folie -hier- ● sind Proteine ● sind Biokatalysatoren ● sind bereits in kleiner Menge ausreichend wirksam ● werden bei der Reaktion nicht verbraucht ● sind sehr spezifisch ● erniedrigen die Aktivierungsenergie ● erhöhen die Umsetzungsgeschwindigkeit ● verschieben NICHT das chemische Gleichgewicht ??? auch RNA kann Enzymfunktion haben Aktivierungsenergie Die Wellen entsprechen der thermischen (Bewegungs) Energie der Moleküle. Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.12 + 3.14a Verminderung der Aktivierungsenergie erhöht die Wahrscheinlichkeit einer Reaktion Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.13 Schematische Darstellung der Funktion von Enzymen Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb 3.15 Reaktionsketten youtube.com/watch?v=_Y3V5hg5EkE Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.14b Enzyme verschieben das Gleichgewicht nicht Enzyme beeinflussen die Lage des Gleichgewichts nicht. Sie erhöhen jedoch die Reaktionsgeschwindigkeit (in beide Richtungen!) Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.25 Halbwertszeit einer Reaktion Einheitenzeichen = s ! 1 ms Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH 1 µs Mechanismen der Enzymkatalyse ● Das aktive – katalytische – Zentrum des Enzymes bindet das Substrat (Emil Fischer, SchlüsselSchloss-Prinzip) ● Sowohl das Substrat als auch das Enzym ändern dabei ihre Konformation (Daniel Koshland, induzierte Passform – ‚induced fit‘) Mechanismen der Enzymkatalyse Die Wechselwirkung zwischen Enzym und Substrat erfolgt ● durch Ausbildung von Wasserstoff-Brücken ● über Ionenbindungen zwischen geladenen Gruppen von z.B. Lysin, Arginin, Aspartat, Glutamat, Imidazolgruppe von Histidin ● durch elektrostatische Wechselwirkungen ● über hydrophobe Interaktionen ● manchmal auch durch Ausbildung einer kovalenten Bindung Einige allgemeine Strategien der enzymatischen Katalyse induced fit Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb.4.32 einige häufige Enzymarten Hydrolasen relevant hydrolytische Spaltungsreaktionen, allgemein Nucleasen Abbau von DNA und RNA Proteasen Abbau von Proteinen – Spaltung der Peptidbindung ATPasen Synthasen Hydrolyse von ATP z.B. im anabolen Stoffwechsel – Kondensation zweier Moleküle Polymerasen Synthese von z.B. DNA oder RNA Isomerasen Neuanordnung von Bindungen Kinasen Phosphorylierung – Addition von PO43- Phosphatasen Dephosphorylierung – Abspaltung von PO43- Oxidoreduktasen Redoxreaktionen – Oxidasen, Reduktasen, Dehydrogenasen Offizielle Nomenklatur der Enzymarten Ergänzung The International Union of Biochemistry and Molecular Biology has developed a nomenclature for enzymes, called EC numbers. It describes each enzyme using a sequence of four numbers, preceded by "EC." The first number broadly classifies the enzyme based on its function into six major categories: EC 1 Oxidoreductases catalyze oxidation/reduction reactions, which involve electron transfer. EC 2 Transferases transfer a chemical group called a functional group (e.g., methyl or phosphate) from one substance to another. EC 3 Hydrolases catalyze the cleavage of chemical bonds through the addition of a water molecule. EC 4 Lyases cleave various bonds by means other than hydrolysis and oxidation. EC 5 Isomerases transfer a group within a single molecule to form an isomer. EC 6 Ligases join two molecules with covalent bonds. Offizielle Nomenklatur der Enzymarten Ergänzung Die EC-Nummern (Enzyme Commission numbers) bilden ein numerisches Klassifikationssystem für Enzyme aus vier durch Punkte voneinander getrennten Zahlen. Genaugenommen wird nicht das Enzym selbst, sondern die Reaktion, die es katalysiert, kategorisiert. So können nicht miteinander verwandte Enzyme, welche die gleiche Reaktion katalysieren, dieselbe EC-Nummer haben. Wipipedia 2013-01 Offizielle Nomenklatur der Enzymarten Ergänzung Synthasen Synthasen heißen in der Biochemie seit 1984 Enzyme, die die Herstellung eines bestimmten Stoffes katalysieren; dieser Stoff wird im Namen explizit genannt. Synthasen haben keine eigene EC-Nummern-Zuordnung. Vor 1984 war Synthase ein Synonym für Ligase. Beispiele sind die Citrat-Synthase (eine Transferase) und die Argininosuccinat-Synthase (eine Ligase). Der ähnliche Name Synthetase ist eine veraltete Bezeichnung für Enzyme, die bei ihrer Reaktion ein Nucleosidtriphosphat verbrauchen. Diese Enzyme werden inzwischen als Ligasen bezeichnet. Synthetase Als Synthetase werden Enzyme aus der Klasse der Ligasen bezeichnet, welche bei der Ligation Adenosintriphosphat (ATP) spalten. Wie bei der Bezeichnung Synthase für ATP-unabhängige Ligasen wird durch diese Benennung die „synthetisierende“ (erzeugende) Rolle, also eine Beteiligung im Anabolismus betont. Aufgrund der Verwechslungsgefahr zwischen Synthetase und Synthase wird diese Benennung vom Nomenclature Committee of the International Union of Biochemistry (NC-IUB) nicht mehr empfohlen und gilt als veraltet. Synthetasen werden inzwischen als Ligasen bezeichnet. Isoenzyme … ● katalysieren die gleiche Reaktion ● stammen oft aus unterschiedlichen Zellen oder Geweben ● sind meistens Produkte verschiedener Gene ● unterscheiden sich in ihren physikalischen und katalytischen Eigenschaften ● lassen sich daher z.B. elektrophoretisch trennen Substrat / Cosubstrat ● Substrat – Molekül an dem ein Enzym ‚arbeitet‘ Zusätzlich oft benötigt: ● Coenzym / Cosubstrat niedermolekulare organische Moleküle reversibel an Enzym gebunden Beispiele: NAD+, NADP+, CoA ● Prosthetische Gruppe ‚Coenzym‘, das kovalent an Enzym gebunden ist Beispiel: Pyridoxalphosphat NAD+ / NADH Nicotinamid-Adenin-Dinukleotid NAD+ dient dem Organismus meist als Oxidationsmittel (und wird dabei reduziert). Daher ist das Verhältnis NAD+/NADH groß (>>1) Dinukleotid Nicotinamid Adenin Redoxreaktion: NAD+ kann durch Aufnahme von zwei Elektronen (e−) und einem Proton (H+) zu NADH reduziert werden. NADH ist die 'energiereiche' Form Grafik: Wikipedia Ribose NADP+ / NADPH Nicotinamid-Adenin-Dinukleotid-Phosphat NADPH dient dem Organismus meist als Reduktionsmittel (und wird dabei oxidiert). Daher ist das Verhältnis NADP+/NADPH klein (<<1). NADP+ oxidierte Form NADPH reduzierte Form H- ? NADP+ NADPH NAD+ / NADH ~ katabole Reaktionen Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.34 modifiziert EM NADP+ / NADPH ~ anabole Reaktionen Coenzym A (CoA) Das Coenzym des Carbonsäure-Stoffwechsels CoA ist ein Derivat der Panthotensäure (= Vitamin B5) Adenin Cysteamin β-Alanin Pantoinsäure Diphosphat Pantothensäure Ribose-3‘-Phosphat Molecular Biology of the Cell (© Garland Science 2008) Acetyl-Coenzym A (CoA) Acetyl-CoA ist die 'energiereiche' Form Acetyl-Gruppe Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.36 Coenzyme basierend auf Vitaminen Mechanismen der Enzymkatalyse ● Säure-Basen Katalyse Aminosäurereste von beispielsweise Histidin oder Glutaminsäure reagieren als Säure oder Base, indem sie während einer Reaktion H+-Ionen aufnehmen oder abgeben. ● Kovalente Katalyse Aminosäurereste (häufig Lysin) oder Koenzyme (Pyridoxalphosphat) gehen kovalente Bindungen mit einem Substrat ein und bilden ein kurzlebiges Zwischenprodukt. ● Metallionenkatalyse Metallionen können z.B. als strukturstabilisierende Koordinationszentren (Zn), Redox-Partner (Fe, Cu) oder als Lewis-Säuren (Zn) die Katalyse unterstützen. Säure-Basen Katalyse Lysozym ist ein Teil des angeborenen Immunsystems. Es katalysiert die hydrolytische Spaltung von Polysaccharidketten in der Zellwand von Bakterien. youtube.com/watch?v=TK7onDEElYc Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.30 Ereignisse im aktiven Zentrum von Lysozym Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.31 Säure-Basen Katalyse Auch die Imidazolgruppe des Histidins kann als H+-Donor bzw. Akzeptor dienen. Grafik: Wikipedia Kovalente Katalyse am Beispiel des Reaktionsmechanismus der Fructose-1,6-bisphosphat-Aldolase (=Aldolase) Aldolase katalysiert die Spaltung von Fructose-1,6-bisphosphat in Dihydroxyacetonphosphat (DHAP) und Glycerinaldehyd-3-Phosphat (GADP). Diese Reaktion ist ein Teilschritt der Glycolyse und daher unentbehrlich für die Verwertung von Kohlenhydraten in allen Lebewesen. Grafik: Wikipedia, modifiziert EM Kovalente Katalyse Lysin bindet kovalent an die Reaktionsintermediate, die dadurch vorübergehend stabilisiert werden während das Cystein und das Histidin in einer SäureBasen-Katalyse Reaktion als Wasserstoff-Donor bzw. Akzeptor fungieren. Seitenketten der an der Reaktion beteiligten Aminosäuren im aktiven Zentrum des Enzyms in rot, grün und blau. Grafik: Wikipedia, modifiziert EM Reaktionsmechanismus der Aldolase Metallionen-Katalyse am Beispiel der Carboanhydrase Das Enzym katalysiert die Hydratisierung von CO2 zu Kohlensäure (= Hydrogencarbonat) und umgekehrt. CO2 lässt sich im Körper leichter in Form von Hydrogencarbonat transportieren, daher ist eine reversible Umwandlung sinnvoll. Außerdem wird über die Reaktion der pHWert des Blutes geregelt. Zink komplexiert ein Hydroxylion, an das CO2 addiert wird. Grafik: Wikipedia Enzymkinetik Diffusion: ... ‘eine Zufallswanderung‘ = random walk youtube.com/watch ?v=mB1yIAFsLAA& list=PL86FB286677 14C01D&index=11 &feature=plpp_vide o s ∼ t Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.22 s= youtube.com/watch ?v=PtYP8uoN0lk Zusammenhang zwischen Substratkonzentration und Geschwindigkeit enzymkatalysierter Reaktionen Löffler/Petrides, Biochemie und Pathobiochemie, 6. Auflage 1998 Michaelis-Menten-Enzymkinetik (1) mathematisch-quantitative Behandlung Enzym-katalysierter Reaktionen Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.15 E = Enzym, S = Substrat, [ES] = Enzym-Substrat-Komplex, [EP] = EnzymProdukt-Komplex, P = Produkt, k = Geschwindigkeitskonstanten Grafik: Wikipedia Michaelis-Menten-Enzymkinetik (2) Grafik: Wikipedia ● ES EP kann man nicht messen, das Entstehen von P aber sehr wohl ● in der Zelle im Fließgleichgewicht ~ keine Rückreaktion P + E EP ● im Reagenzglas am Anfang nur E + S, kein P, daher ist zu diesem Zeitpunkt die Reaktion P + E EP unbedeutend ● Michaelis-Menten Kinetik = numerische Lösung der Differentialgleichungen mit den ‘vereinfachten’ kinetischen Parameter Michaelis-Menten-Enzymkinetik (3) numerische Lösung der Differentialgleichungen Leonor Michaelis Maud Menten Grafik: Wikipedia Michaelis Menten Gleichung v Vmax [S] Km Umsatzgeschwindigkeit maximale Umsatzgeschwindigkeit Substratkonzentration Michaelis Menten-Konstante Maß für die Affinität des Enzyms zum Substrat Michaelis-Menten-Enzymkinetik (4) Grafik: Wikipedia Km ist jene Substratkonzentration (mol / l) bei der das Enzym mit der Hälfte seiner maximal möglichen Geschwindigkeit arbeitet Für [S] = Km gilt [E] = [ES] und daher vmax ist nicht direkt messbar aber eben berechenbar. Michaelis-Menten-Enzymkinetik (5) Messung von Substratkonzentrationen modiziert nach dem Skriptum „Ergänzung …“, Hans Goldenberg Messung von Enzymkonzentrationen Michaelis-Menten-Enzymkinetik (3) ES ⇌ E + S m Zur Bedeutung des KM-Wertes: 1) Substratkonzentration bei halbmaximaler Geschwindigkeit Km = [S] wenn v = vmax/2 2) Km hat die Dimension einer Substratkonzentration (mol/l) 3) Km ist ist ein Maß für die Affinität des Enzyms für das Substrat: Gleichgewichtskonstante für den Zerfall des Enzym-Substrat-Komplexes ES Affinitätskonstante für die Bildung von ES Je kleiner Km, desto größer die Affinität des Enzyms zum Substrat 4) Wenn [S] = Km [E] = [ES] 50%ige Substratsättigung (“Halbsättigung”) des Enzyms Lineweaver-Burk Diagramm -eine- Umformung der Michaelis Menten Gleichung entspricht der Geradengleichung y = kx + d Grafik: Wikipedia Einfluss von pH und Temperatur auf die Enzymaktivität pH-Wert Optimum am Beispiel der Verdauungs-Enzyme Pepsin (im Magen) und Trypsin (im Darm). Temperatur Optimum der Enzymaktivität, typischerweise bei 37°C irreversible Denaturierung bei zu hoher Temperatur Fieber Einzelne Enzyme können durchaus davon abweichen, etwa die Taq-Polymerase in der PCR Maßeinheiten der Enzymaktivität SI-Einheit: 1 katal (1 kat) Umsatz von 1 mol Substrat / sec üblicher Bereich: µkat, nkat 1 unit (U) Umsatz von 1 µmol Substrat / min Wechselzahl (µmol Substratumsatz) / (min x µmol Enzym) entspricht vmax / Menge Enzym Hemmung der Enzymaktivität Bedeutung in der Medizin: Wirkung vieler Pharmaka (= Medikamente ^^) Irreversible Hemmung – Reversible Hemmung Produkthemmung – Rückkopplung Zwei charakteristische Hemmtypen - kompetitive Hemmung - nicht-kompetitive Hemmung Mischformen, z.B. unkompetitive Hemmung Kompetitive Hemmung Inhibitor ist meist Substrat-Analoges, das mit dem Substrat um die gleiche Bindungsstelle am Enzym konkurriert. Michaelis-Menten-Kinetik: KM wird größer ‚Gegenmaßnahme‘: Erhöhung der Substratkonzentration vmax .[S ] v= [I ] + [ ] + ) S K m (1 Ki Kapp = K m (1 + [I] ) Ki [I] ... Konzentration des Inhibitors Ki ... Affinitätskonstante des Inhibitors, das ist analog zur Michaelis-Konstante die Dissoziationskonstante des Enzym-InhibitorKomplexes. Diese ist bei einem wirksamen Inhibitor wesentlich kleiner als Km. Kapp ... Apparenter (scheinbarer) KM-Wert Kompetitive Hemmung Als kompetitiv wird eine Hemmung der Enzymaktivität bezeichnet, wenn ein Agonist und ein Antagonist um die Besetzung des aktiven Zentrums konkurrieren, wobei der Antagonist keine biochemische Wirkung hat. Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 3.28 Nicht-kompetitive Hemmung Der Inhibitor bindet an einer Stelle des Enzyms, die nicht die Substrat-Bindungsstelle ist ( Konformationsänderung) Inhibitor bindet an freies Enzym ( EI) und an den ESKomplex ( EIS). Ausnahme: Bindung von Substrat und Inhibitor schließen sich wechselweise aus. Michaelis-Menten-Kinetik: vmax wird kleiner Nicht-kompetitive Hemmung vmax wird in Gegenwart des Inhibitors kleiner, Km bleibt aber unverändert. http://www.mymcat.com/ wiki/Enzyme_Inhibition Grafik: Wipipedia E = Enzym, S = Substrat, P = Produkt, I = Inhibitor, ES = Enzym-Substrat-Komplex, EI = Enzym-Inhibitor-Komplex, ESI = EnzymSubstrat-Inhibitor-Komplex Wikipedia http://www.med4you.at/laborbefunde/techniken/enzyme/hemmung_nichtkomp.gif Wikipedia http://www.med4you.at/laborbefunde/techniken/enzyme/hemmung_komp.gif Vergleich der beiden Typen von Enzymhemmung Kompetitiv I Kapp = K m (1 + ) Ki v max, ungeh Nicht-kompetitiv v max, geh = [I] (1 + ) Ki Unkompetitive Hemmung ● Inhibitor wird nur vom Enzym-Substrat-Komplex gebunden, nicht jedoch vom freien Enzym. ● Der Enzym-ProduktKomplex wird verlangsamt gebildet. ● Michaelis-Menten-Kinetik: sowohl KM als auch vmax werden verändert. Grafik: Wikipedia E = Enzym, S = Substrat, P = Produkt, I = Inhibitor, ES = Enzym-Substrat-Komplex, EI = Enzym-Inhibitor-Komplex, ESI = EnzymSubstrat-Inhibitor-Komplex Wikipedia med4you.at/laborbefunde/techniken/enzyme/hemmung_nichtkomp.gif Wikipedia Ergänzung: Vergleich kompetitive / nicht-kompetitive Hemmung med4you.at/laborbefunde/techniken/enzyme/hemmung_komp.gif Hemmung der Enzymaktivität – Ergänzung Regulationsmechanismen von Enzymen Mechanismus Signal Selbstregulation Allosterische Hemmung Allosterische Hemmung Substratkonzentration Reaktionsprodukt Endprodukt (feed back) oder anderer Metabolit (crosstalk zwischen Stoffwechselwegen) Allosterische Aktivierung Ausgangsstoff (feed forward) Allosterische Modifikation (±) Regulator als Stoffwechselsignal Allosterische Modifikation (±) Interaktion zwischen Enzymen Interkonversion, Proteolyse Proteolytische Kaskade Interkonversion, Phosphorylierung/ Dephosphorylierung Kontrolle der Genexpression Totalabbau von Enzym Phosphorylierungskaskade, allosterische Modifikation Aktivierung/Deaktivierung von Transkription oder Translation Aktivierung der Ubiquitinierung Ansprechzeit Millisekunden Millisekunden Millisekunden Millisekunden Millisekunden Millisekunden Sekunden bis Minuten Sekunden bis Minuten Stunden Stunden Feedback-Hemmung in nur einem Biosyntheseweg Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.34 Vielfältige FeedbackHemmung Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.35 Allosterische Regulation Die Bindung eines Effektor-Moleküls ‘X’ an die allosterische Region eines Proteins (nicht im aktiven Zentrum) verändert die Konformation des Proteins wodurch die Bindung von ‘Glucose’ im aktiven Zentrum erleichtert wird. Es gibt allosterische Aktivatoren aber auch allosterische Inhibitoren (vgl. nicht-kompetitive Enzym-Hemmung). Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.37 Feedback-Hemmung durch Konformationsänderung Aspartat-Transcarbamoylase (ATC) Das Endprodukt Cytidintriphosphat (CTP) deaktiviert ATC “T-Form“ (T = tensed) Die Substrate Carbamoylphosphat und Aspartat aktivieren ATC “R-Form“ (R = relaxed) 2. Teil ist hardcore, 1. sehr gut youtube.com/watch?v=5aW0C3-IHVo Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH Kinetik allosterischer Enzyme in Anwesenheit positiver oder negativer Effektoren v / vmax = K-TYP 1 1 + ( K / [ S ] )n n … Hill-Koeffizient (Kooperativitätskoeffizient) V-TYP Löffler/Petrides, Biochemie und Pathobiochemie, 6. Auflage 1998 Sauerstoff als allosterischer Aktivator von Hämoglobin – Ergänzung http://cgi.chemie.tu-darmstadt.de/akplenio/moproc/eisen/Haemoglobin/Haemo%20zusammenfassung.htm Sauerstoff als allosterischer Aktivator von Hämoglobin – Ergänzung Aktivitätsänderung von Enzymen durch limitierte Proteolyse Aktivierung der Proteasen Trypsin und Chymotrypsin durch limitierte Proteolyse a) b) Durch Abspaltung eines N-terminalen Hexapeptides entsteht aus Trypsinogen Trypsin Durch Abspaltung zweier Dipeptide entsteht aus Chymotrypsinogen Chymotrypsin. Hierfür werden Trypsin und Chymotrypsin benötigt. Aktivitätsänderung von Proteinen durch Phosphorylierung In einer typischen Eukaryontenzelle sind viele tausend Proteine durch kovalente Bindung einer Phosphatgruppe modifiziert. Dabei werden in den meisten Fällen auch ihre Eigenschaften (Aktivität, Bindung, Lokalisation etc.) verändert. Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.38 GTP-bindende Proteine als molekulare Schalter Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.39 Konformationsänderung nach Hydrolyse eines Nukleotids EF-Tu spielt eine wesentliche Rolle bei der Translation von mRNA in Protein; GTP-Hydrolyse setzt die mit Aminosäure beladene tRNA frei, die anschließend die wachsende Polypeptidkette verlängert. hardcore: Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH www.youtube.com/watch?v=rukzo81MGfk&list=PLCi0K MllW4KcYecYkr-ZRJULb2PFuBNrq&index=28 Ein allosterisches Motorprotein Durch ATP-Hydrolyse wird die Bewegung eines Motorproteins entlang einer ‚Faser‘ (auch Filament) zielgerichtet, hier nach rechts. Solche Proteine dienen zum Stofftransport in Zellen. Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.42 Eine 'Protein-Maschine' ATPase Animation der Folie war ohnehin schlecht youtube.com/watch?v=iD3RUlK9rXU&feature=BFa&list= ULuR4Eysyk5N0&lf=mfu_in_order allgemein zum Thema Proteinmaschinen youtube.com/watch?NR=1&v=FJ4N0iSeR8U&feature=e ndscreen Kinesin http://www.youtube.com/watch?v=YAva4g3Pk6k up eventually Dynein, Myosin Alberts, Lehrbuch der Molekularen Zellbiologie © 2012 Wiley-VCH, Abb. 4.43 look