Aminosäuren, Proteine, Enzyme, Enzymkinetik & Energetische
Werbung

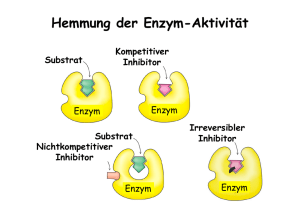
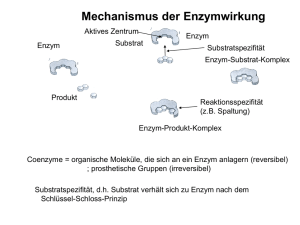
Aminosäuren, Proteine, Enzyme, Enzymkinetik & Energetische Kopplung • Gegenstandskatalog: 4.1 4.1.1 4.1.2 4.1.3 Enzyme Einteilung, Wirkweise und Bedeutung von Enzymen für den Stoffwechsel; Coenzyme, Beziehung zu den Vitaminen Beeinflussung der Enzymaktivität Ribozyme 4.3 4.3.1 Grundzüge des Stickstoffwechsels Aminosäuren und Proteine 4.5.1 Energetische Kopplung, Prinzip und Bedeutung Aminosäuren • Alle Aminosäuren besitzen ein a-C-Atom, das als funktionelle Gruppe eine Carboxyl- und eine Aminogruppe, sowie eine variable Seitenkette trägt. • Bis auf Glycin (R = H) sind alle proteinogenen AS chiral und gehören zur L-Reihe nach Fischer bzw. 2S-Reihe nach C.I.P. (Ausnahme: Cystein L/2R) • AS sind Ampholyte, da sie sowohl ein Proton auf-, als auch abgeben können • Beim physiologischen pH-Wert liegt die – Aminogruppe protoniert als –NH3+ – Caroboxylgruppe deprotoniert als –COOvor. • Der isoelektrische Punkt ist der pH-Wert, an dem die AS neutral vorliegt. • AS können nach folgenden Kriterien unterteilt werden: – Chemische Eigenschaften (Seitenkette) – Verwendung in der Proteinbiosynthese (proteinogen (20)/nichtproteinogen(>100)) – Fähigkeit des Organismus zur Biosynthese • Essentiell: Valin, Leucin, Isoleucin, Phenylalanin, Tryptophan, Lysin, Methionin, Threonin • Semi-essentiell: Histidin (nur für Säuglinge) Peptide und Proteine • Peptide und Proteine sind kettenförimge Makromoleküle, in denen die 20 (bzw. 21) proteinogenen Aminosäuren sequenzspezifisch kovalent miteinander verknüft sind. • Die AS-Sequenz ist dabei genetisch vorgeschrieben • 2-10 AS: Oligopeptide (Dipeptid/Tripeptid/…) • 10-100 AS: Polypeptid • >100 AS: Protein • Ort der Proteinbiosynthese: Ribosomen – Freie Ribosomen: lösliche und für die Zelle selbst bestimmte Proteine – Raues ER: Exportproteine und Membranproteine • Die einzelnen AS sind durch die sog. Peptidbindung verknüpft. • Die Peptidbindung enteht formal durch Wasserabspaltung zwischen der Carboxylgruppe der einen und der Aminogruppe der nächstfolgenden AS. Peptidbindung entspricht einer Säureamidbindung. • Aufgrund ihres partiellen Doppelbindungscharacters ist die Peptidbindung planar. • An einem Ende der AS-Kette befindet sich die freie a-Aminogruppe N-Terminus • Am anderen Ende befindet sich die freie Carbxylgruppe C-Terminus • Nach Konvention wird die AS-Sequenz eines Proteins immer vom N-Terminus aus begonnen. • Bei Polypeptiden und Proteinen unterscheidet man folgende Strukturen: – Primärstruktur – Sekundärstruktur – Tertiärstruktur – Quartärstruktur Primärstruktur • Die AS-Sequenz wird als Primärstruktur bezeichnet, sie gibt keinerlei Auskunft über die Raumstruktur des Proteins. Sekundärstruktur • Räumliche Anordnung der AS-Kette aufgrund von H-Brückenbildung zwischen C=O- und NHGruppen. • Wichtige Sekundärstrukturen sind: – a-Helix – b-Faltblatt – b-Schleifen a-Helix • Polypeptidkette liegt in Form einer rechtsgewundenen Schraube vor • Pro Windung 3,6 AS • Ganghöhe beträgt 0,54nm • Prolin kann aufgrund seiner Struktur zu einer Unterbrechung der Helix führen Knick • Beispiele: a-Keratine b-Faltblatt • Aufbau aus mehreren benachbarten b-Strängen aufgebaut, bei denen die Seitenketten der AS abwechselnd ober- und unterhalb der Faltblattebene liegen • Beispiel: b-Keratin der Haut/Nägel Tertiärstruktur • Die TS beschreibt die Ausbildung der stabilen Raumstruktur monomerer Proteine. • Sie beruht auf kovalenten (Disulfidbrücken) und nichtkovalenten Wechselwirkungen (HBrücken, hydrophope WW, Ionenbindungen) der Aminosäureseitenketten. Quartärstruktur • Bei der Assoziation mehrerer identischer oder nichtidentischer Polypeptidketten (Untereinheiten) mit jeweils eigener Primär-, Sekundär- und Tertiärstruktur zu einer funktionellen Gruppe spricht man von der Quartärstruktur eines Proteins. • Erst durch Raumstruktur wird die biologische Aktivität eines Proteins ermöglicht. • Bsp.: Keratinkinase (dimer), Hämoglobin (tetramer) • Unterteilung von Proteinen: – Sphäroproteine: Wasserlösliche, reaktionsfähige Proteine (Bsp.: Enzyme) – Skleroproteine: Wasserunlösliche, reaktionsarme Proteine (Bsp.: Strukturproteine wie Kollagen) – Proteine können prinzipiell auch andere Gruppen als AS tragen: • • • • Lipide Lipoproteine Kohlenhydrate Glykoproteine Phosphoratome Phosphoprotein Metalle Metallprotein Abbau von Proteinen • Proteinasen: neben Amylasen wichtigste hydrolytische Enzyme; C-NHydrolasen • → Spaltung der Peptidbindung • Proteinasen: – Endopeptidasen: spalten Peptidbindungen innerhalb des Proteinmoleküls → Zerlegung in kleinere Peptide – Exopeptidasen: spalten fortschreitend vom Ende eines Proteinmoleküls her Aminosäuren ab → Carboxypeptidasen, Aminopeptidasen • Proteinasen wirken nicht spezifisch auf bestimmte Substrate (Proteine) – Hemmung durch Inhibitorproteine – Proteinasen, die sich extrazellulär im Blut und in den extrazellulären Flüssigkeiten befinden, üben dort spezifische regulatorische Funktionen aus (→ Blutgerinnung, Fibrinolyse, Aktivierung von Komplementfaktoren) – innerhalb der Zellen finden sich Proteinasen v. a. in Lysosomen Abbau von Aminosäuren • Kohlenstoffskelett wird i. A. über den Citronensäurezyklus weiterverarbeitet • Stickstoffanteil gesonderter Weg: Abspaltung der Aminogruppe vom Kohlenstoffgerüst. • Aminogruppen werden auf α-Ketosäure übertragen (Transaminierung), meist auf α-Ketoglutarsäure. Dabei entsteht Glutaminsäure und die jeweilige αKetosäure, die als Substrat in den Citratzyklus eingeht. Auf diesem Weg können die verschiedenen Aminosäuren je nach Bedarf auch ineinander umgewandelt werden. Abbau von Aminosäuren • Der endgültige Abbau der AS erfolgt in der Leber. Über das Blut gelangen überschüssige AS in der Transportform Alanin zur Leber. • Dort wird die Aminogruppe in Form von Glutamat fixiert. • Anschließend wird Glutamat oxidativ desanimiert und liefert NH3 in den Harnstoffzyklus. • Über die Nieren wird der Stickstoff letztlich in Form von Harnstoff ausgeschieden. Enzyme - Biokatalysatoren • Als reaktionsspezifische Katalysatoren sind in lebenden Systemen zwei unterschiedliche Makromoleküle vorhanden: – Katalytisch aktive Proteine (Enzyme) – Katalytisch aktive Ribonukleinsäuren (Ribozyme) • • • • Die weithäufigsten Biokatalysatoren sind Enzyme. Ribozyme kommen in Spleißosomen und Ribosomen vor. Sie trennen Introns aus Primärtranskripten heraus und verknüpfen die Exons oder katalysieren die Peptidverknüpfung in den Ribosomen Wie alle Katalysatoren beschleunigen sie die Einstellung der Gleichgewichtslage einer Reaktion, ohne das Gleichgewicht zu ändern. Sie katalysieren sowohl Hin- als auch Rückreaktion E+S → ES → EP → E+P • Die Reaktion verläuft i.A. nach folgendem Schema: 1. Substrat bindet kovalent/nichtkovalent an das Enzym Enzym-Substrat-Komplex 2. Katalysierte Reaktion führt zum Enzym-ProduktKomplex 3. Enzym-Produkt-Komplex zerfällt in Produkt und Enzym (unverändert) • Die Bindung des Substrats an das Enzym findet dabei am katalytischen Zentrum des Enzyms statt. Enzyme katalysieren immer nur eine einzige bzw. nur sehr wenige Reaktionen. Sie sind reaktionsspezifisch bzw. wirkungsspezifisch. Dabei setzen sie immer nur bestimmte Substrate um. Die Substratspezifizität beruht auf folgenden Modellen Schlüssel-Schloss-Prinzip: Modell des „induced fit“: Aktives Zentrum des ungebundenen Enzym verändert seine Form Enzyms hat eine zum Substrat (Konformation) bei der komplementäre Gestalt Substratbindung, aktives Zentrum hat die zum Substrat komplementäre Gestalt erst nach dessen Bindung Enzymklassifikation • Enzyme werden nach ihrer Wirkspezifität in sechs Gruppen unterteilt: Hauptklasse Katalysierte Reaktion Oxidoreduktasen Redoxreaktionen Beispiele Lactatdehydrogenase Glutamatdehydrogenase Succinatdehydrogenase Pyruvatdehydrogenase Transferasen Gruppenübertragungen Hexokinase Glycogenphosphorylase Hydrolasen Hydrolytische Abspaltung von Gruppen Proteasen, Petidasen Esterasen Glycosidasen Lyasen Nichthydrolytische Abspaltung von Gruppen Aldolase Transketolase Fumarase Isomerasen Umwandlungen isomerer Verbindungen Retinalisomerase Triosephopsphat-Isomerase UDP-Galaktose-4-Epimerase Ligasen Energieabhängige Verknüpfung von Bindungen Pyruvatcarboxylase Acyl-CoA-Synthetase Glutaminsynthetase • • Enzyme, die verschiedene Primär- und Raumstrukturen besitzen, aber die gleiche Reaktion mit dem gleichen Substrat und dem gleichen Produkt katalysieren, werden Isoenzyme genannt. Enzyme, die mehrere katalytische Zentren besitzen, werden Multienzymkomplexe gennant (Bsp.: Pyruvatdehydrogenase, DANN-Replikase) • Viele Enzyme benötigen Cofaktoren für ihre Aktivität. Diese gehen nicht unverändert aus der Reaktion hervor und müssen regeneriert werden. • Das vollständig funktionelle Enzym mit der niedermolekularen Gruppe wird als Holoenzym bezeichnet • Das Enzym ohne die niedermolekulare Gruppe wird Apoenzym genannt • Die niedermolekulare Gruppe wird als Coenzym (= Cosubstrat), und wenn sie fest mit dem Enzym verbunden ist prosthetische Gruppe genannt. • Da Cosubstrate regeneriert werden, werden sie nur in geringen Mengen benötigt. Menschen können Cosubstrate häufig nur teilweise synthetisieren, sie müssen bestimmte Vorstufen der Cosubstrate mit der Nahrung aufnehmen. Diese Vorstufen werden Vitamine genannt. • Es werden fettlösliche Vitamine (E, D, K, A) und wasserlösliche Vitamine (C, H, Vitamine des B-Komplexes) unterschieden. Herkunft und Funktion der Coenzyme Coenzym Funktion Vitamin Ascorbat Hydroxylierungen Ascorbat Redoxsystem Vitamin C Decarboxylierungen Thiamin Aldehydgruppentransfer Vitamin B1 Wasserstoffübertragung Riboflavin Thiaminpyrophosphat Flavinmononukleotid (FMN); Flavinadenindinucleotid (FAD) Vitamin B2 + Nicotinamidadenindinucleotid(-phosphat) NAD ; NADP Pyridoxalphosphat + Wasserstoffübertragung Nicotinsäure Transaminierungen Pyridoxin Decarboxylierungen Vitamin B6 a,b-Elimination Coenzym A Acylübertragung Pantothensäure Biotinyl-Lysyl-Enzym Carboxylierung Biotin Lipoyl-Lysyl-Enzym Wasserstoffübertragung Lionsäure Acylgrupenübertragung Tetrahydrofolat C1-Gruppenübertragung Folsäure 5'-Adenosylcobalamin 1,2 Verschiebungen von Alklgruppen Cobalamin Difarnesylnaphthochinon Carboxylierungen von Glutamylresten Vitamin B12 Ubichinon Wasserstoffübertragung Naphthochinon Cytochrome Elektronenübertragung Vitamin K Adenosintriphosphat Phosphatübertragungen - Adenylübertragungen Cytidindiphosphat Phospholipidübertragungen - Uridindiphosphat Saccharidübertragungen - S-Adenosylmethionin Methylgruppenübertragungen - Phoshoadenosyl-Phosphosulfat (PAPS) Sulfatübertragungen - Beeinflussung der Enzymaktivität • Die Aktivität der Enzyme wird beeinflusst von verschiedenen Faktoren: – pH-Wert (meist Optimum beim pH = 7,4) – Temperatur (meist Körpertemperatur) – Affinität zum Substrat – Effektoren: • Aktivatoren • Inhibitoren Enzymkinetik • Die Geschwindigkeit der Enzymkatalyse hängt von der Substratkonzentration ab. • Die Michaelis-Menten-Gleichung beschreibt die Abhängigkeit der Reaktionsgeschwindigkeit von der Substratkonzentration. Die Michaeliskonstante KM gibt die Substratkonzentration an, bei der ein Enzym mit halbmaximaler Geschwindigkeit arbeitet. Sie ist ein Maß für die Affinität eines Enzyms zu einem Substrat. • Je steiler die Kurve, desto höher ist die Affinität zum Substrat • KM ist unabhängig von der Enzymkonzentration • Um den KM-Wert exerimentell zu bestimmen bedient man sich einer doppelt reziproken Auftragungsart der Michaelis-Menten-Gleichung im Lineweaver-Burk-Diagramm. • Aus diesem kann KM direkt ablesen Enzymeffektoren Effektoren Aktivatoren Inhibitoren Irreversible Inhibitoren Suizidinhibitoren Reversible Inhibitoren Kompetitive Inhibitoren Nichtkompetitive Inhibitoren Unkompetitve Inhibitoren Irreversible Inhibition • Inhibitor bindet kovalent an das Enzym und beeinträchtigt dadurch dessen Funktionsfähigkeit. • Ein besonderer Fall sind die Suizidinhibitoren. Dabei wandelt das Enzym den potentiellen Inhibitor enzymatisch in den eigentlichen Inhibitor um, der die kovalente Bindung mit dem Enzym eingeht. • Bsp.: Cyanid Kompetetive Inhibition • Substrat und Inhibitor strukturell ähnlich, beide haben Affinität zum aktiven Zentrum desselben Enzyms, beide konkurrieren um reversible Bindung an aktives Zentrum des Enzyms • Verdrängung des Inhibitors durch hohe Substratkonzentrationen möglich • Vmax unverändert • KM-Wert höher → es muss eine höhere Substratkonzentration vorhanden sein, um die halbmaximale Geschwindigkeit zu erreichen Nichtkompetetive Inhibition • • • • • i.d.R. keine strukturelle Ähnlichkeit von Substrat und Inhibitor Inhibitor bindet an Enzym oder ES-Komplex → Komplex aus Enzym, Substrat und Inhibitor; Inhibitor bindet nicht am aktiven Zentrum, sondern an eine eigene Stelle keine Verdrängung des Inhibitors möglich (daher sind z. B. Schwermetallionen giftig) Vmax erniedrigt → mit Inhibitor besetzte EnzymMoleküle sind weniger oder gar nicht aktiv KM-Wert unverändert Unkompetetive Inhibition • Inhibitoren binden ausschließlich an ESKomplex • KM und Vmax erniedrigt • Bsp.: Hemmung der Cytochromoxidase durch Azid Regulation der Enzymaktivität • Eine weitere Unterscheidung von Inhibitionstypen bieten die Bindungsstellen der Inhibitoren: – Isosterische Inhibition • Inhibitor und Substrat binden an das katalytische Zentrum. • Meist kompetetive Hemmung – Allosterische Inhibition • Substrat und Inhibitor binden an unterschiedlichen Stellen • Bestehen häufig aus einer katalytisch wirksamen Untereinheit (Substratbindungsstelle) und einer weiteren, assoziierten Regulationsuntereinheit (Inhibitorbindungsstelle) Regulation der Enzymaktivität • Neben der Enzymsynthese und dem Enzymabbau wird die Stoffwechselleistung einer Zelle durch die Enzymaktivität der vorhandenen Enzymmoleküle reguliert. • Produkthemmung – Enzym wird durch das Produkt der Reaktion, die es katalysiert, gehemmt – überwiegend Typ der kompetitiven Hemmung – Bei komplexen Stoffwechselvorgängen werden Schlüsselenzyme reguliert (Bsp.: Glykolyse/Gluconeogenese) • Substrathemmung – Substrat hemmt bei erhöhten Konzentrationen seinen eigenen Umsatz – typisch bei Enzymen, bei denen das Substrat zunächst an periphere Stellen gebunden und dann an die Bindungsstelle im aktiven Zentrum „geleitet“ wird Energetische Kopplung • Energie kommt in verschiedenen Formen vor: – – – – – Mechanische (kinetische, potentielle) Energie Elektrische Energie Wärme Strahlungsenergie Energie einer chemischen Bindung • Die beiden letztgenannten sind für den lebenden Organismus von essentieller Bedeutung • Strahlungsenergie ist die Aufnahmeform der Energie während der Photosynthese • Chemische Energie ist die Speicherform der Organismen • Um energiearme Substrate im Rahmen eines Stoffwechselprozesses umzuwandeln, bedarf es eines zweiten Energie liefernden Prozesses, derart, dass die Energie verbrauchende (endergonische) Reaktion an eine Energie liefernde (exergonische) Reaktion gekoppelt wird. • Die Energie liefernde Verbindung fällt dabei auf einen energieärmeren Zustand und muss anschließend regeneriert werden. ( Photosynthese) Energiereiche Verbindungen • Nukleotide – Adenosintriphosphat (ATP) – Guanosintriphosphat (GTP) – Uridintriphosphat (UTP) • Acylphosphate • Enolphosphate • Thiocarbonsäuren/-ester ATP • • • • • • 2 Phosphorsäureanhydrid-Bindungen des ATP energiereich Anhydrid-Bindungen leicht hydrolysierbar, da partielle Ladungen der benachbarten Phosphor- bzw. Sauerstoffatome sich gegenseitig abstoßen Aktivierung energiearmer Substrate: endständiger Phosphat-Rest oder ADP-Rest bzw. Diphosphat-Rest oder AMP-Rest wird übertragen auch GTP oder UTP, allerdings seltener „Gruppenübertragungspotential des ATP“ • Acylphosphate energiereich: Carboxy-Gruppe und Phosphorsäure-Rest über Anhydrid-Bindung verbunden • Enolphosphate: höherer Energiegehalt als Anhydrid-Bindung, z. B. des Phosphoenolpyruvats (PEP); Oxo-Enol-Tautomerie und Phosphorsäureester-Bindung • Thiocarbonsäureester / Thioester: keine Resonanzstabilisierung wie in Sauerstoffestern, daher energiereich