Prädiktive Marker - des Klinikums
Werbung

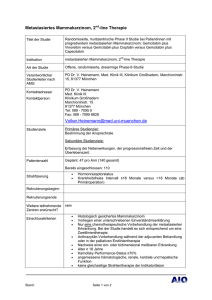
Pankreaszentrum München: Optimierung der Patientenversorgung Prädiktoren zum Therapieansprechen beim Pankreaskarzinom Dr. med. Stefan Böck Medizinische Klinik und Poliklinik III Klinikum der Universität München – Campus Großhadern www.pankreaszentrum-muenchen.de 14.05.2011 Definitionen Prognostischer Marker: „Prognostic factors are patient and tumor factors that predict patient outcome (usually survival) and are independent of treatment administered.” Prädiktiver Marker: „Predictive factors are clinical, cellular, and molecular markers that predict response of the tumor to treatment (either in terms of tumor shrinkage or a survival benefit from treatment).” - Prädiktive Marker für Therapie-assoziierte Toxizitäten - Marker sind i. d. R. determinierbar vor Therapie-Start ( z. B. Tumormarker-Kinetik) Shepherd FA & Tsao MS, J Clin Oncol 2006; 24: 1219 Prädiktive Marker: Gemcitabin dFdC: 2,2-Difluoro-Deoxycytidin hENT-1: Human Equilibrative Nucleoside Transporter 1 Ko AH et al, Gastroenterology 2009; 136: 43 Prädiktive Marker: Gemcitabin & hENT-1 Farrell JJ et al, Gastroenterology 2009; 136: 187 Prädiktive Marker: Gemcitabin Prädiktive Marker für Effektivität und Toxizität Marker Prädiktor Referenz hENT-1 (Human Equilibrative Nucleoside Transporter 1) Effektivität Farrell et al. Gastroenterology 2009 hCNT-3 (Human Concentrative Nucleoside Transporter 1) Effektivität Marechal et al. Clin Cancer Res 2009 Haptoglobin hämatolog. Toxizität Matsubara et al. JCO 2009 CDA*3 SNP (CDA 208 G>A [Ala70Thr]) Toxizität Ueno et al. BJC 2009 CDA SNP (79A>C [Lys27Gln]) hämatolog. Toxizität Farrell et al. ASCO-GI 2009 Prädiktive Marker: Gemcitabin Gemcitabin vs. CO-1.01: rand. Phase II Gemcitabin (dFdC) h-ENT1 unabhängige intrazelluläre Aufnahme Passive Diffusion? CO-1.01 Prädiktive Marker: Gemcitabin Limitationen: Meist retrospektive Studien, n < 50 Keine homogene Therapie, kein Kontrollarm/Randomisation Uneinheitliche Methodik (Protein/Gen-Level: IHC, mRNA,…) Kim R et al, Cancer 2011; Epub ahead of print; Marechal R et al, Cancer 2010; 116: 5200 Prädiktive Marker: Gemcitabin Prädiktive Marker: EGFR Pathway EGF – Rezeptor - Protein-/Genexpression - Polymorphismen RAS PI3-K pY K K SOS pY GRB2 MEK pY PTEN RAF STAT AKT MAPK mTOR Proliferation und Differenzierung Gen-Transkription Zellzyklus-Progression Apoptose G2 M S G1 Angiogenese Metastasierung Prädiktive Marker: EGFR Pathway beim PC 1 Lee - k-ras Mutation (Exon 2, Codon 12 + 13)1: 50 % - 90 % - EGFR Gen Amplifikation1,2: 40 % - PTEN Expression („loss of expression“)3: (CRC und MBC ca. 40%) 40 % - (aktivier.) EGFR Mutationen (Exon 18-21)1,4,5,6: 0 % - 3 % - b-raf Mutation6: 0%-5% - erbB-3 Überexpression7: 50 % J et al, Cancer 2007; 109: 1561; 2 Tzeng CWD et al, J Surg Res 2007; 143: 20; 3 Tao J et al, JHUSTMS 2006; 26: 444; 4 Kwak EL et al, Clin 7 Friess H et al, Cancer Res 2006; 12: 4283; 5 Tzeng CDW et al, Surgery 2007; 141: 464; 6 Immervoll H et al, Virchows Arch 2006; 448: 788; Clin Cancer Res 1995; 1: 1413 Prädiktive Marker: Cetuximab SWOG Phase III - Studie, 1st Line, 745 Patienten Stratifikation: ECOG, Stadium, vorherige Operation Gemcitabin + Cetuximab Gemcitabin wöchentlich x 7 für 8 Wochen dann wöchentlich x 3 alle 4 Wochen R Cetuximab400 mg/m2 loading dose dann 250 mg/m2 wöchentlich Gemcitabin Gemcitabin wöchentlich x 7 für 8 Wochen dann wöchentlich x 3 alle 4 Wochen primärer Endpunkt = medianes Überleben 33% ↑ Philip PA et al, J Clin Oncol 2010; 28: 3605 Prädiktive Marker: Cetuximab EGFR Expression (n=595): - 547 Pat. (92%) EGFR-IHC pos. - Überleben für EGFR pos. Pat. in beiden Therapiearmen identisch (6 Monate) Philip PA et al, J Clin Oncol 2010; 28: 3605 Prädiktive Marker: Erlotinib PA.3 Studie: 1st Line, 569 Patienten, Stratifikation: ECOG, Stadium, Zentrum Gemcitabin + Erlotinib Gemcitabin wöchentlich x 7 für 8 Wochen dann wöchentlich x 3 alle 4 Wochen R Erlotinib 100 mg täglich p.o. (n=48: Eskalation auf 150 mg/d) Gemcitabin + Placebo Gemcitabin Placebo wöchentlich x 7 für 8 Wochen dann wöchentlich x 3 alle 4 Wochen täglich p.o. primärer Endpunkt = medianes Überleben 33% ↑ Moore MJ et al, J Clin Oncol 2007; 25: 1960 Prädiktive Marker: Erlotinib N = 162 / 569 Moore MJ et al, ASCO 2005: Abstract # 1 Prädiktive Marker: Erlotinib Überleben im Gem/Erlotinib Arm nach KRAS Status: N = 569 KRAS: n = 117 (wt 21%) KRAS mut 6.0 Monate (n=49) KRAS wt 6.1 Monate (n=15) EGFR FISH: n = 107 da Cunha Santos G et al, Cancer 2010; 116: 5599 Prädiktive Marker: Erlotinib Gemcitabin + Erlotinib → Capecitabin Gemcitabin (Gem) 1000 mg/m2 (30 Min.) weekly x 7 for 8 Wochen, dann weekly x 3 alle 4 Wochen R Erlotinib (E) 150 mg täglich (po) n = 281 Capecitabin + Erlotinib → Gemcitabin Capecitabin (Cap) 2 x 1000 mg/m2 po d 1-14 alle 3 Wochen Erlotinib (E) 150 mg täglich (po) Stratifikation – Zentrum – Stadium (lokal fortgeschritten vs. metastasiert) Rekrutierung: 46 Deutsche Zentren, von Mai 2006 bis Dezember 2008 ClinicalTrials.gov Identifier: NCT00440167 Prädiktive Marker: Erlotinib Translationale Analysen (AIO-PK0104) • Formalin-fixierte, Paraffin-eingebettete (FFPE) Tumorblöcke • n = 208 FFPE Tumorblöcke verfügbar (74%) • Zentrale Analyse im Labor für Molekularpathologie der LMU München (PD Dr. A. Jung, Prof. Dr. T. Kirchner) • KRAS Mutation (Exon 2, Codon 12+13, PCR): 176 / 204 Patienten erfolgreich analysiert 53 Wildtyp (wt, 30%), 123 Mutation (mut, 70%) Boeck S et al, ASCO 2010; Abstract # LBA4011 Prädiktive Marker: Erlotinib All patients, by KRAS status 1.0 KRAS wt: n = 53, 40 events, median = 8 mo KRAS mut: n = 123, 105 events, median = 6.6 mo Rate without Event 0.8 Hazard Ratio: 1.62 (1.11 – 2.35) p = 0.011 0.6 0.4 0.2 0.0 0 4 8 12 16 20 24 28 32 Time [Month] => prognostischer (und/oder prädiktiver) Marker? Boeck S et al, ASCO 2010; Abstract # LBA4011 Prädiktive Marker: VEGF Pathway Gem + Erlotinib vs. Gem + Erlotinib + Bev (AViTA) Van Cutsem E et al, J Clin Oncol 2009; 27: 2231 Prädiktive Marker: Bevacizumab VEGFR-1 Polymorphismus (AViTA) n = 154 / 607; post-hoc SNP-Analyse Lambrechts D et al, ECCO/ESMO 2009: Abstract # 16LBA Prädiktive Marker: Bevacizumab VEGFR-1 Polymorphismus (AViTA) n = 154 / 607; post-hoc SNP-Analyse Lambrechts D et al, ECCO/ESMO 2009: Abstract # 16LBA Genetische Subtypen des Pankreaskarzinoms? Collisson EA et al, Nat Med 2011; 17: 500 Zusammenfassung & Ausblick • Im klinischen Alltag spielen molekulare Prädiktoren für das Therapieansprechen beim Pankreaskarzinom bisher keine klar definierte Rolle • Gemcitabin: hENT-1 als bisher am besten validierter Marker für Effektivität • Anti-EGFR Therapie: Bedeutung von KRAS noch nicht eindeutig definiert • Anti-VEGF Therapie: VEGFR-SNPs als mögliche Prädiktoren? Dringend nötig: Validierung im Rahmen (randomisierter) klinischer Studien, einheitliche Methodik, innovative Biostatistik